蛋白水解酶(肽酶)可依据催化类型和活性中心氨基酸种类分为六大类,分别由丝氨酸、苏氨酸、半胱氨酸、天冬氨酸、谷氨酸或金属基团在催化过程中起主导作用。其中,丝氨酸蛋白水解酶的催化活性中心包含有丝氨酸残基,该残基上的羟基可作为亲核试剂攻击肽键而使其水解[1-2]。在MEROPS分类系统中,依据氨基酸序列相似性已划分出超过50类的丝氨酸蛋白水解酶,再依据三级结构和催化活性位点顺序,这50类中的大部分可归入14个家族[3]。
多种来源的丝氨酸蛋白水解酶已从不同角度得到研究和应用。哺乳动物丝氨酸蛋白水解酶在消化、凝血、凋亡和免疫系统中的作用被深入研究,对其催化机制的研究和相应抑制剂类药物的研发对人类健康具有重要意义[4-6]。植物丝氨酸蛋白水解酶中最受关注的是枯草杆菌素,其参与蛋白质折叠、植物发育、与环境的相互作用等,在植物防御反应、对抗各种病原体过程中的作用为关注重点[7]。昆虫丝氨酸蛋白水解酶在其依赖先天免疫抵御病原生物的过程中发挥巨大作用,其结构特征、催化机制等被大量研究,以帮助了解昆虫媒介传播疾病和控制有害生物[8]。微生物丝氨酸蛋白水解酶在医药、农业、工业等领域均具有显著的研究和应用价值。在医药领域,针对病原体致病机制所涉丝氨酸蛋白水解酶进行抑制剂的筛选,是开发抗真菌、抗细菌和抗病毒类药物的有效途径[9]。农业领域,食线虫真菌来源的致病相关丝氨酸蛋白水解酶是开发生物杀虫剂的理想选择[10]。工业领域,酶制剂市场份额最大的产品为微生物来源的枯草杆菌蛋白酶(碱性蛋白酶)以及动物来源的胰凝乳蛋白酶等,均属于丝氨酸蛋白水解酶家族[11]。鉴于微生物的多样性及其所蕴藏酶资源的丰富性,微生物来源丝氨酸蛋白水解酶的认知、挖掘和应用具有显著科学意义和应用价值。
已知,黑曲霉(Aspergillus niger)具有丰富的蛋白水解酶系,现有研究多集中在其代表性的酸性蛋白酶方面,且其工业应用价值也已被深度开发研究[12-13]。相反,对碱性蛋白酶的研究与挖掘多集中于细菌来源[14-16]。在前期的研究中发现,黑曲霉同样具有合成碱性蛋白酶的可能性,其中的蛋白酶CpyC可能是一种功能和应用性能皆未解析的碱性丝氨酸蛋白酶。为此,本文就此酶的生化特征进行较全面的研究,并探索其在蛋白质生物加工领域的可能应用。
1 材料与方法
1.1 菌种与质粒
黑曲霉CICIM F0215为前期分离鉴定的野生菌株,保藏于中国高校工业微生物资源与信息中心。大肠杆菌JM109、毕赤酵母GS115和质粒pPIC9k,用于本研究中目的基因的克隆与表达。黑曲霉和大肠杆菌分别采用PDA和LB培养基进行培养[17];毕赤酵母及其重组菌培养参照Pichia遗传操作手册进行。
1.2 主要试剂
限制性内切酶和LA Taq DNA聚合酶,大连宝生物工程有限公司;T4 DNA连接酶、质粒小量提取试剂盒、TRIzol总RNA提取试剂、cDNA合成试剂盒、小量DNA产物纯化回收试剂盒,ThermoFisher公司;大豆分离66蛋白,上海源叶生物科技有限公司;BODIPY FL Ester,ThermoFisher;苯甲基磺酰氟(phenylmethylsulfonyl fluoride,PMSF)、抑肽素、E-64、EDTA,Sigma公司;肽类分子质量标准品细胞色素(12 500 Da)、抑肽酶(6 500 Da)、杆菌肽(1 450 Da),Gly-Gly-Tyr-Arg(451 Da)、Gly-Gly-Gly(189 Da),中国计量科学研究院;其余生化试剂为分析纯,国药化学试剂有限公司。
1.3 黑曲霉蛋白水解酶基因克隆和表达
按照试剂盒使用说明提取黑曲霉总RNA并制备cDNA。基因扩增、连接、转化与鉴定等按照常规实验方法进行[18]。基因扩增引物由生工生物工程(上海)股份有限公司合成,上游引物(5′→3′)为GTACTGGTAGAGATAGGTCTTGGGTTTCCG,下游引物(5′→3′)为TGCTCTAGATTAAAGCCCCGATAACTTGCCG(下划线部分为限制性酶切位点)。
按照毕赤酵母操作手册所述方法进行重组酶的发酵制备,终止发酵后离心(4 ℃,6 000 r/min,15 min)收集发酵上清液,制得粗酶液,再经盐析、脱盐获得初步纯化的重组酶蛋白。酶蛋白纯度分析采用SDS-PAGE法[17]。
1.4 酶活力测定与酶学特征分析
1.4.1 酶活力检测方法
依照前期建立的均相检测法测定重组酶活力,底物为经BODIPY NHS Ester标记的大豆分离蛋白[19]。基本步骤为:荧光底物以缓冲液溶解至10 μg/mL,与适度稀释的酶液分别预热至50 ℃,等体积加入96孔板,于50 ℃下反应10 min,用Tecan infinite 200酶标仪(瑞士Tecan)在激发波长500 nm、发射波长530 nm条件下检测荧光。以底物和酶液作为对照。用相对荧光强度(relative fluorescence units,RFU)代表酶活力,计算公式为:
RFU=FC-(FA+FB)
式中:FC,样品荧光值;FA,底物荧光值;FB,酶液荧光值。
1.4.2 温度和pH对酶活力的影响
以不同温度值替代1.4.1节方法中的反应温度进行酶活力测定,以酶活力最高值为100%,计算相对酶活力,确定重组酶的最适作用温度。用乙酸-乙酸钠缓冲液(0.2 mol/L)、磷酸氢二钠-磷酸二氢钠缓冲液(0.2 mol/L)、Tris-盐酸缓冲液(0.05 mol/L)和碳酸钠-氢氧化钠缓冲液(0.025 mol/L)分别配制pH 5.0~11.0的缓冲液,将酶液分别用这些缓冲液稀释后进行反应,然后按照1.4.1节的方法进行酶活力测定,以酶活力最高者为100%,计算相对酶活力,确定重组酶的最适作用pH。在最适反应pH下,将酶液在不同温度保持1 h后检测酶活力,以原始酶样活力值为100%,计算相对酶活力,确定重组酶的热稳定性;在最适反应温度下,将酶液在不同pH保持1 h后检测酶活力,以原始酶样活力值为100%,计算相对酶活力,确定重组酶的pH稳定性。
1.4.3 金属离子对酶活力的影响
在反应体系中分别加入终浓度为1 mmol/L的Ca2+、Zn2+、Mg2+、Ba2+、Fe3+、Na+、K+、Cu2+、Co2+和Mn2+,测定其酶活力,以未加金属离子的酶液活力为100%,计算相对酶活力。
1.4.4 抑制剂对酶活力的影响
选择PMSF、抑肽素、E-64和EDTA等4种抑制剂,相应活力抑制检测及抑制率计算方法参见文献[20]。
1.5 酶水解大豆蛋白的水解特征分析
向10 g/L大豆分离蛋白溶液中以10 U/mL加酶量加入CpyC,在pH 8.0和50 ℃下孵育,在不同时间间隔取样,进行SDS-PAGE。蛋白水解样本同步采用HPLC分析其肽谱变化,HPLC在Agilent 1200系统上进行,采用TSKgel G2000SWXL色谱柱(300 mm×7.8 mm,Tosoh,Japan),柱温为30 ℃,流动相为乙腈、水和三氟乙酸(体积比为45∶55∶0.1),流速为0.5 mL/min,使用波长280 nm的紫外光学检测器。
1.6 生物信息学分析
黑曲霉基因组开放阅读框分析,使用在线分析软件ORF Finder进行(https://www.ncbi.nlm.nih.gov/orffinder/);信号肽分析使用软件SignalP 6.0进行(https://services.healthtech.dtu.dk/service.php?SignalP);序列比对分析使用在线分析程序BLAST进行(https://blast.ncbi.nlm.nih.gov/Blast.cgi);遗传发育树构建使用MEGA 11软件辅助(Neighbour-joining法);氨基酸序列比对分析使用在线分析软件ESPript进行(https://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)。
2 结果与讨论
2.1 黑曲霉基因组中存在1个功能未知的丝氨酸蛋白酶CpyC
使用ORF Finder并配合使用BLAST等工具分析A.niger CBS 513.88基因组序列,获得了一个疑似编码丝氨酸蛋白水解酶的开放阅读框XM_001400743,重新命名其编码基因为cpyC,编码区大小为1 392 bp,编码由463个氨基酸残基组成的多肽CpyC,且不含典型的信号肽序列。将CpyC与来自14个不同丝氨酸蛋白水解酶家族的代表性氨基酸序列进行比对分析并构建遗传发育关系,结果显示,来自不同家族的丝氨酸蛋白水解酶的自展值大多小于50%,与其较低的氨基酸序列相似度相吻合(图1)。CpyC与分类归属于S08类的酶蛋白遗传距离相对接近,初步预测CpyC属于丝氨酸蛋白水解酶SB家族S08类(图1)。

图1 CpyC在丝氨酸蛋白水解酶家族中的遗传距离
Fig.1 Genetic distances of CpyC in the serine peptidase clan
注:线段的长度表示用MEGA 11计算的遗传距离;分支节点上的数字代表的是bootstrap百分比,小于50%的bootstrap值未显示;除CpyC外,其余各丝氨酸蛋白水解酶以其在UniProt数据库中的登录号表示,括号中标注为各丝氨酸蛋白水解酶在MEROPS数据库中的分类号。各丝氨酸蛋白水解酶来源为:P00780,地衣芽胞杆菌(Bacillus licheniformis);P27487,人(Homo sapiens);Q05523,嗜热脂肪芽胞杆菌(Bacillus stearothermophilus);Q1RF98,大肠杆菌(Escherichia coli);P20350,黑腹果蝇(Drosophila melanogaster);P0A7C2,大肠杆菌(E.coli);P0A9M0,大肠杆菌(E.coli);P35049,尖孢镰刀菌(Fusarium oxysporum);P16753,人巨细胞病毒(Human cytomegalovirus);Q9HTZ1,铜绿假单胞菌(Pseudomonas aeruginosa);P42790,假单胞菌(Pseudomonas sp.);P02788,人(H.sapiens);P06875,大肠杆菌 (E.coli);P0A7C6,大肠杆菌(E.coli)。
鉴于丝氨酸蛋白水解酶S08类成员的氨基酸序列仍然具有较大的差异性,学者进一步根据催化活性位点附近基序的保守性,将其分为S08A、S08B和S08C三个亚类[1]。为进一步确定本研究CpyC分类归属,将其与6种已知结构的丝氨酸蛋白水解酶S08类成员进行了氨基酸序列详细比对分析,结果如图2所示。S08类丝氨酸蛋白水解酶的催化活性中心包括天冬氨酸、组氨酸和丝氨酸[1],在CpyC分子中依次为Asp188、His226和Ser381。

图2 CpyC与丝氨酸蛋白水解酶S08类成员的氨基酸序列比对
Fig.2 Multiple sequence alignment of CpyC and members from serine peptidase S08 family
注:除CpyC外,其余各丝氨酸蛋白水解酶以其在UniProt数据库中的登录号表示;α和β分别代表α-螺旋和β-折叠;TT代表β-转角;红色背景代表该处残基相同;红色字母蓝色边框代表其中残基相对保守;白色、红色和黑色字母分别代表相同、相似和一致性低的残基。
S08类丝氨酸蛋白水解酶属于SB家族,该家族的一个共同特征是催化活性中心丝氨酸位于Gly-Thr-Ser-Xaa-Xaa-Xbb-Pro基序中,其中Xaa代表脂肪族氨基酸,Xbb代表丝氨酸、苏氨酸、甘氨酸或丙氨酸[1]。在CpyC分子中,其催化活性位点Ser381所在的相应基序为Gly379-Thr-Ser-Ile-Ala-Thr-Ala385,除基序最后一位的疏水性氨基酸由脯氨酸替换为丙氨酸外,其余位点均符合家族特征。丝氨酸蛋白水解酶S08类的催化活性中心天冬氨酸通常位于Asp-Thr/Ser-Gly基序[1],在CpyC中相应基序为Asp188-Thr-Gly190,符合此分类特征。S08类丝氨酸蛋白水解酶不同于其他分类的另一个特征是,其催化活性中心丝氨酸总是由TCX(X为任意碱基)密码子编码,cpyC中相应密码子为TCC,符合此分类特征。
在现有分类体系中,S08B亚类丝氨酸蛋白水解酶在其催化活性中心附近具有包含Asp139、Arg172和Ala324的特征基序,S08A和S08C亚类的相应特征基序包含Thr/Ser139、His172和Met324,而S08C亚类又以Asn140、Ser175和Cys330为单独特征[1]。比对结果显示,CpyC仅以Thr189和His229符合S08A和S08C亚类共同特征基序的前两个位点类型,不符合S08B和S08C亚类各自的特征基序位点类型。另外,比对结果中二级结构特征显示CpyC的催化结构域包含交替出现的α-螺旋和β-折叠结构,这也符合S08类丝氨酸蛋白水解酶的典型特征。
综合以上结果,来源于黑曲霉的CpyC,其分类特征相对接近丝氨酸蛋白水解酶SB家族S08A亚类,但存在细微差异,CpyC应为SB家族的一个新类。
2.2 cpyC基因的克隆表达及重组酶的生化特征
以cDNA途径克隆并表达黑曲霉F0215 cpyC。经核苷酸序列测定与分析后发现,所克隆的核苷酸序列与A.niger CBS 513.88中相应序列略有差异,对应编码的氨基酸序列差异为K13N、R251G和Y244F,此差异未涉及催化活性中心位点和其他已知特征基序。按照毕赤酵母操作手册进行电击转化、高拷贝转化子筛选和摇瓶发酵,进一步对粗酶液进行纯化,获得经SDS-PAGE分析验证为单一条带的纯化酶蛋白,用于后续酶学特征分析。
对CpyC的最适反应温度、pH及相关稳定性进行研究,结果如图3所示。CpyC的最适反应温度为50 ℃,在40~60 ℃保持50%以上的酶活力。CpyC在常温下具有较好的稳定性,在50 ℃以下1 h内可保持70%以上的酶活力。CpyC的最适反应pH值为8.0,在pH 6.5~9.5保持50%以上的酶活力。在pH 6.0~9.5维持1 h,CpyC可保持50%以上的酶活力。现有研究中的微生物碱性蛋白酶主要来自细菌,尤其是芽胞杆菌属[14-16],本研究新鉴定的碱性真菌蛋白酶CpyC是对微生物碱性蛋白酶研究的有益补充。
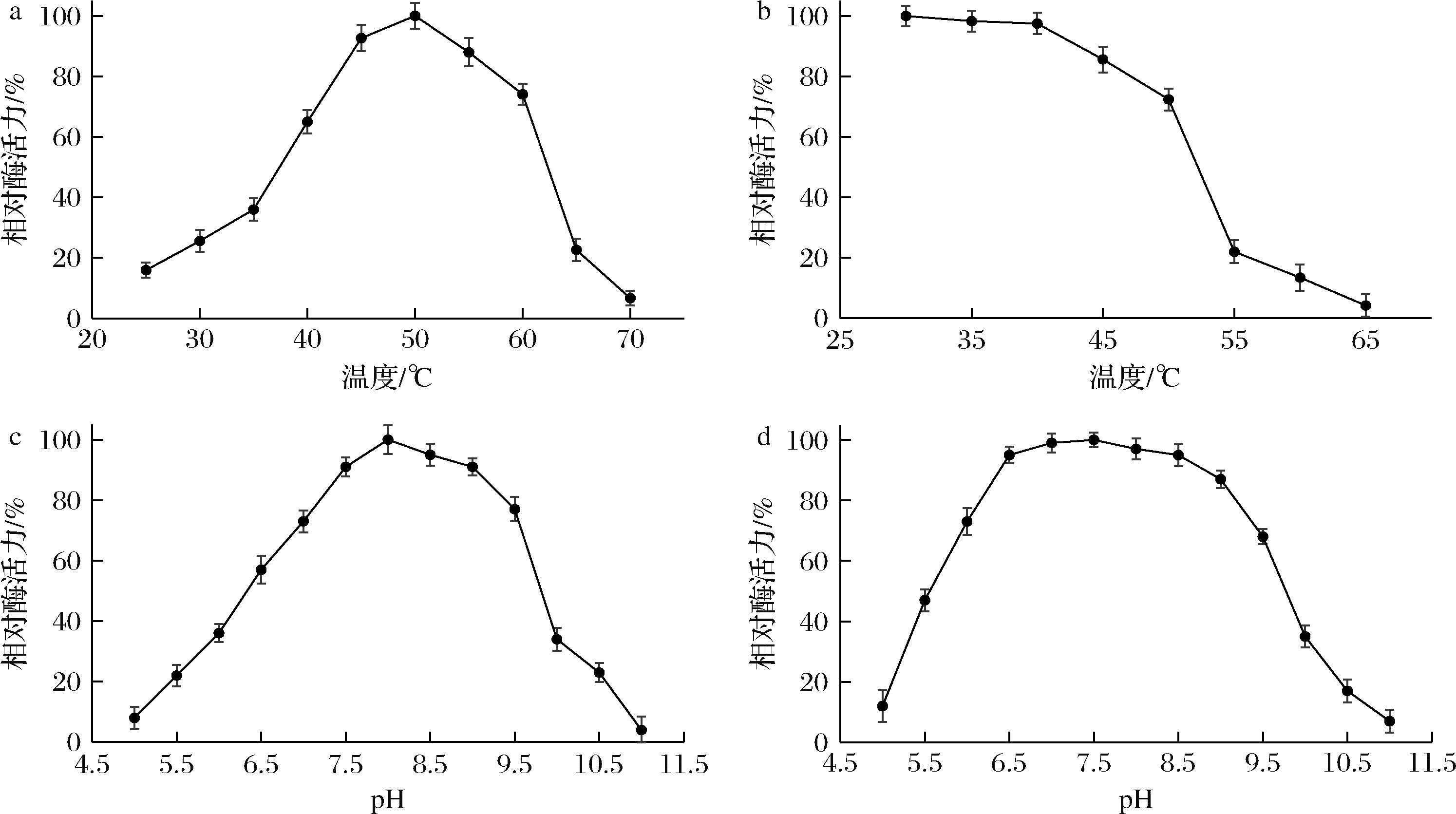
a-最适温度;b-温度稳定性;c-最适pH;d-pH稳定性
图3 温度和pH对CpyC活力的影响
Fig.3 Effects of temperature and pH on the activity of CpyC
金属离子对CpyC活力的影响如表1所示,不同金属离子对CpyC分别表现出激活或抑制作用。其中,Ca2+的激活作用最为显著,Zn2+和Mg2+也具有相对较弱的激活作用,Co2+具有最为显著的抑制作用,Cu2+的抑制作用相对较弱。其余金属离子对CpyC无明显激活或抑制作用。
表1 金属离子对CpyC活力的影响
Table 1 Effects of metal ions on the activity of CpyC
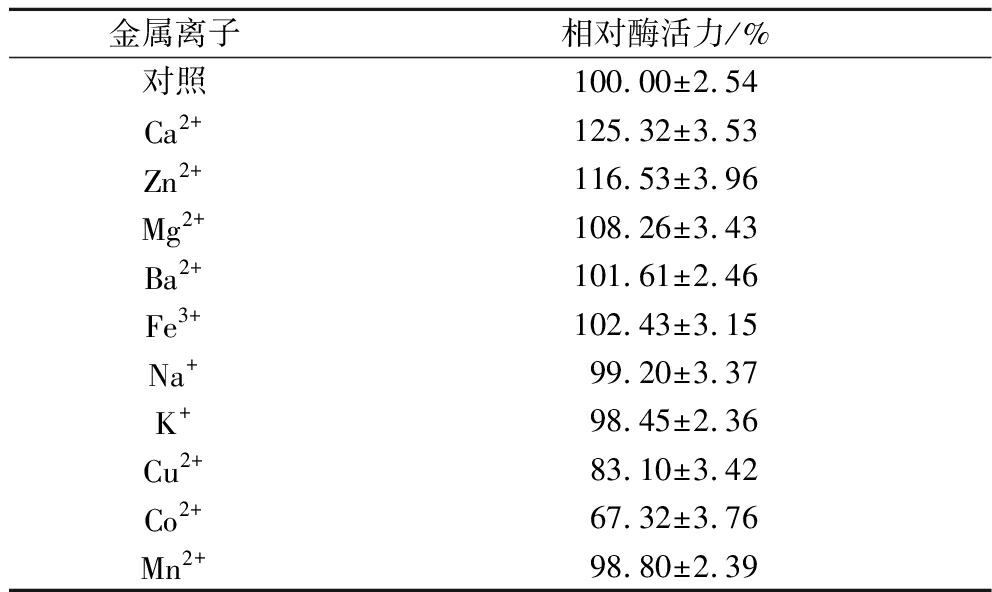
金属离子相对酶活力/%对照100.00±2.54Ca2+125.32±3.53Zn2+116.53±3.96Mg2+108.26±3.43Ba2+101.61±2.46Fe3+102.43±3.15Na+99.20±3.37K+98.45±2.36Cu2+83.10±3.42Co2+67.32±3.76Mn2+98.80±2.39
使用不同种类和浓度的抑制剂对CpyC进行酶活力抑制实验,结果见图4。丝氨酸蛋白水解酶特异性抑制剂PMSF在0.1 mol/L浓度水平即可抑制90%以上的CpyC活力,PMSF浓度升至1 mol/L浓度可彻底抑制CpyC活力;0.1 mol/L和1 mol/L的EDTA分别可抑制超过20%和40%的CpyC活力,再次说明金属离子对CpyC存在激活作用;不同浓度的抑肽素(天冬氨酸蛋白水解酶家族的特异性抑制剂)和E-64均未抑制CpyC活力。上述结果显示,黑曲霉来源的蛋白酶CpyC是一种丝氨酸蛋白酶。
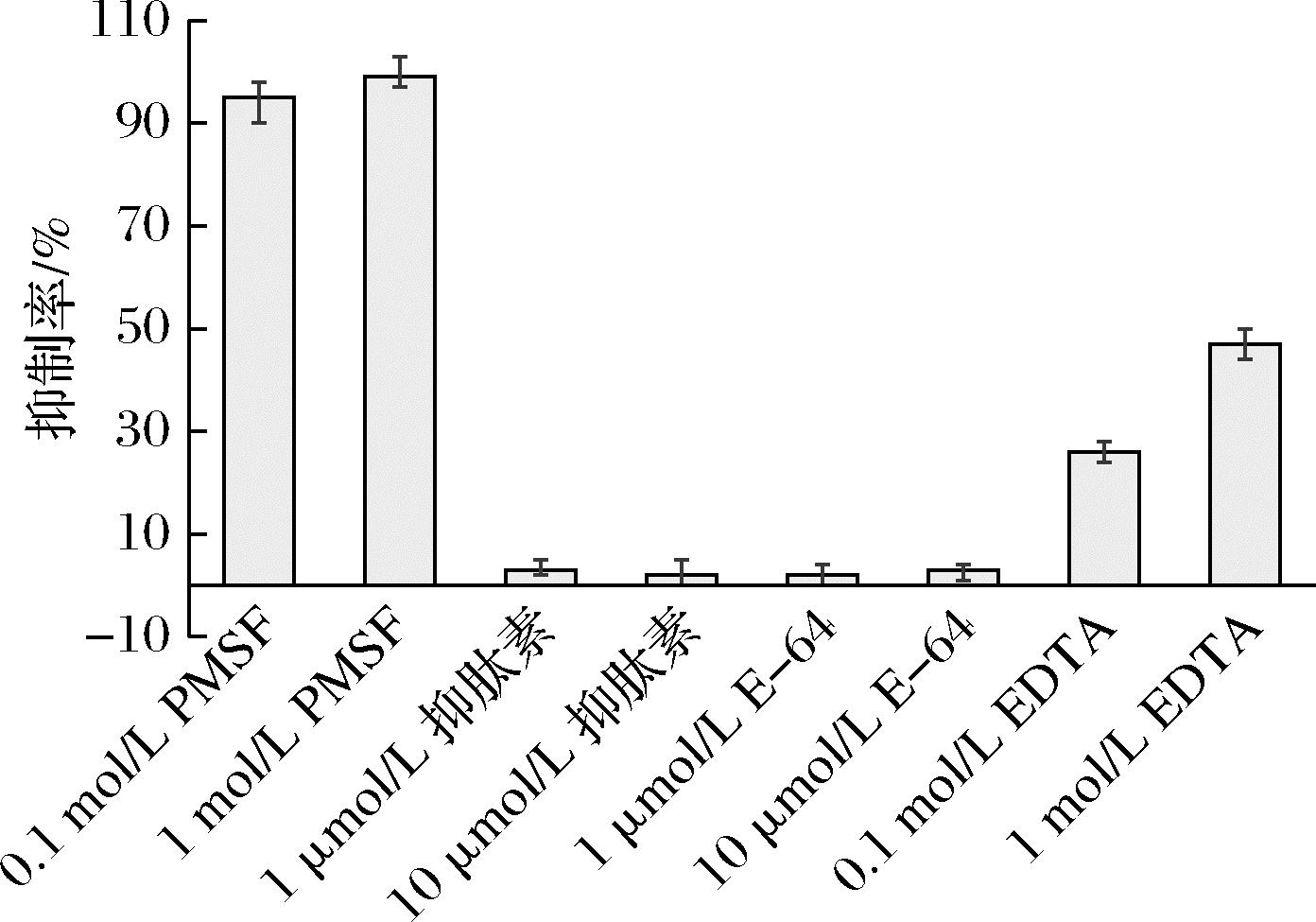
图4 抑制剂对CpyC活力的影响
Fig.4 Effects of inhibitors on the activity of CpyC
2.3 重组CpyC水解大豆分离蛋白特征分析
为了进一步解析CpyC的水解作用特征,将其用于大豆分离蛋白水解并对产物进行分析,结果如图5所示。
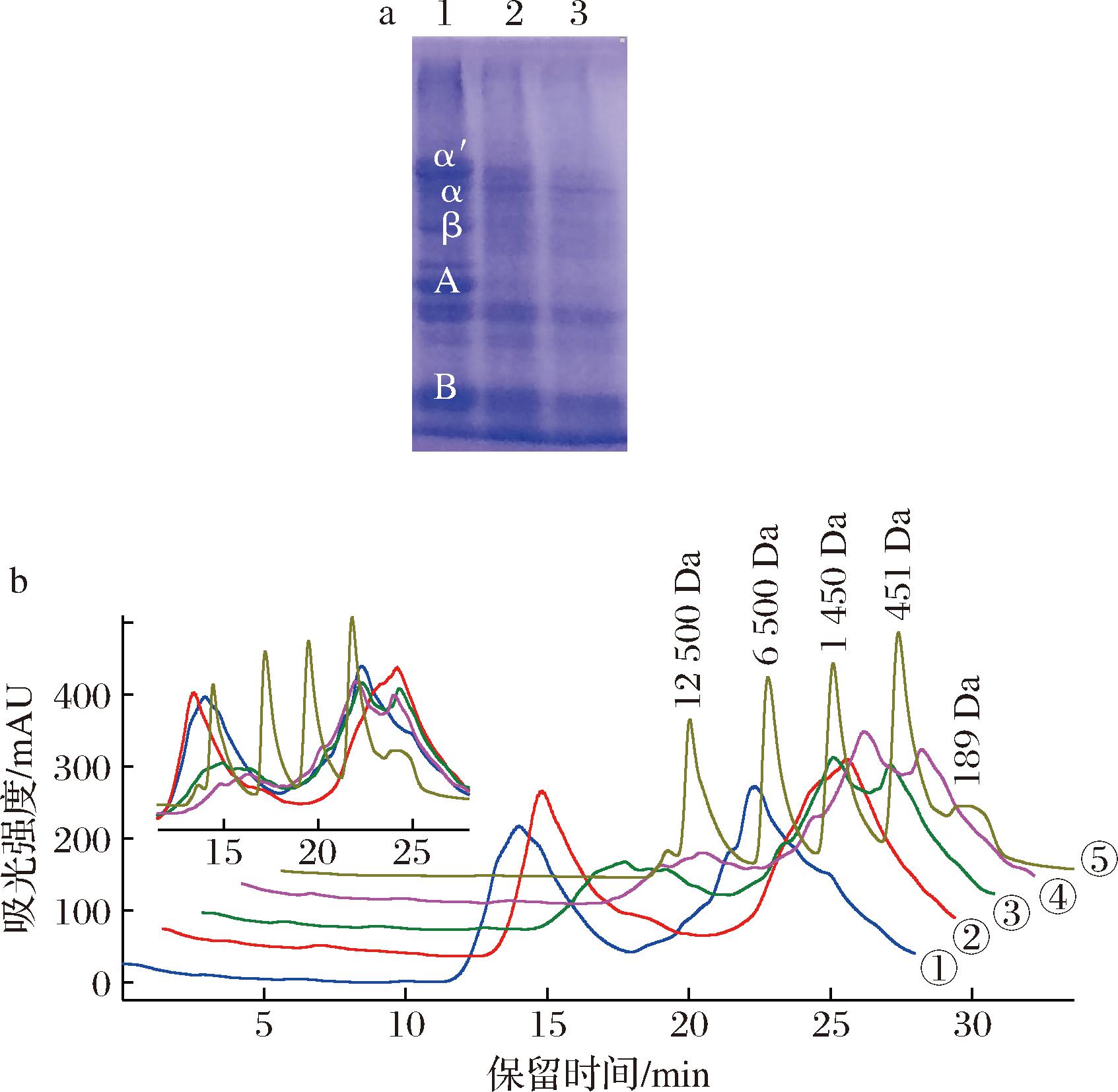
a-SDS-PAGE分析酶解产物(1、2、3分别为反应0.25、6、12 h后样品);b-HPLC分析酶解产物(①为大豆分离蛋白样品,②~④依次为反应0.25、6、12 h后样品,⑤为肽类分子质量标准品)
图5 CpyC水解大豆分离蛋白的产物分析
Fig.5 Product analysis of soybean protein isolate hydrolyzed by CpyC
由图5-a可知,CpyC对大豆分离蛋白的5种主要组成亚基均具有显著的水解作用,其中β-伴大豆球蛋白的3种组成亚基(α′、α和β)水解相对较快,大豆球蛋白的两种组成亚基(A和B)水解相对较慢。
图5-b展示了酶解产物的变化历程,分析显示大豆分离蛋白主要成分集中于两个分子质量范围,分别为12 500 Da和451 Da附近。CpyC在0.25 h内对分子质量12 500~451 Da的底物成分作用明显,此分子质量区间内组分明显减少,而分子质量低于451 Da的组分明显增加,说明此段时间CpyC主要作用于长度为4~100个氨基酸残基的多肽底物。反应进行到6 h以后,分子质量大于12 500 Da的底物组分显著减少,而分子质量在6 500~451 Da的组分明显增加,说明此段时间CpyC主要作用于长度在100个氨基酸残基以上的多肽,生成了分子质量在6 500~451 Da的产物。反应进行到12 h,分子质量在12 500 Da附近的多肽组分含量继续降低,而增加的产物主要位于1 450~451 Da。特别的是,分子质量在189 Da附近的组分含量在6 h相比0.25 h明显减少,再反应至12 h,含量仅有小幅下降,说明CpyC对二肽具有转化作用,具体作用方式有待进一步研究。
3 结论
黑曲霉蛋白水解酶CpyC接近丝氨酸蛋白水解酶SB家族S08类,为一种真菌来源的新型碱性蛋白酶,具有水解蛋白质为多肽/寡肽的水解特征和应用潜力。
[1] RAWLINGS N D, SALVESEN G S.Handbook of Proteolytic Enzymes[M].3rd Edition.London:Academic Press, 2013.
[2] RAWLINGS N D.Twenty-five years of nomenclature and classification of proteolytic enzymes[J].Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics, 2020, 1868(2):140345.
[3] RAWLINGS N D, BARRETT A J, THOMAS P D, et al.The MEROPS database of proteolytic enzymes, their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database[J].Nucleic Acids Research, 2018, 46(D1):D624-D632.
[4] WEISS S A I, REHM S R T, PERERA N C, et al.Origin and expansion of the serine protease repertoire in the myelomonocyte lineage[J].International Journal of Molecular Sciences, 2021, 22(4):1658.
[5] AKULA S, FU Z R, WERNERSSON S, et al.The evolutionary history of the Chymase locus-a locus encoding several of the major hematopoietic serine proteases[J].International Journal of Molecular Sciences, 2021, 22(20):10975.
[6] PATEL S.A critical review on serine protease:Key immune manipulator and pathology mediator[J].Allergologia et Immunopathologia, 2017, 45(6):579-591.
[7] FIGUEIREDO J, SOUSA SILVA M, FIGUEIREDO A.Subtilisin-like proteases in plant defence:The past, the present and beyond[J].Molecular Plant Pathology, 2018, 19(4):1017-1028.
[8] VEILLARD F, TROXLER L, REICHHART J M.Drosophila melanogaster clip-domain serine proteases:Structure, function and regulation[J].Biochimie, 2016, 122:255-269.
[9] BURCHACKA E, PITA P,  UPICKA-S
UPICKA-S OWIK A.Recent advances in fungal serine protease inhibitors[J].Biomedicine &Pharmacotherapy, 2022, 146:112523.
OWIK A.Recent advances in fungal serine protease inhibitors[J].Biomedicine &Pharmacotherapy, 2022, 146:112523.
[10] 汤丽燕, 杨乐, 邢新婧, 等.食线虫真菌致病相关丝氨酸蛋白酶的研究进展[J].微生物学通报, 2017, 44(4):911-919.
TANG L Y, YANG L, XING X J, et al.Advances in pathogenicity-related serine proteases in nematophagous fungi[J].Microbiology China, 2017, 44(4):911-919.
[11] SALWAN R, SHARMA V.Trends in extracellular serine proteases of bacteria as detergent bioadditive:Alternate and environmental friendly tool for detergent industry[J].Archives of Microbiology, 2019, 201(7):863-877.
[12] PEL H J, DE WINDE J H, ARCHER D B, et al.Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513.88[J].Nature Biotechnology, 2007, 25(2):221-231.
[13] BUDAK S O, ZHOU M M, BROUWER C, et al.A genomic survey of proteases in Aspergilli[J].BMC Genomics, 2014, 15(1):523.
[14] BARZKAR N.Marine microbial alkaline protease:An efficient and essential tool for various industrial applications[J].International Journal of Biological Macromolecules, 2020, 161:1216-1229.
[15] SHARMA K M, KUMAR R, PANWAR S, et al.Microbial alkaline proteases:Optimization of production parameters and their properties[J].Journal of Genetic Engineering and Biotechnology, 2017, 15(1):115-126.
[16] BHATT H B, SINGH S P.Cloning, expression, and structural elucidation of a biotechnologically potential alkaline serine protease from a newly isolated haloalkaliphilic Bacillus lehensis JO-26[J].Frontiers in Microbiology, 2020, 11:941.
[17] 诸葛健,王正祥. 工业微生物实验技术手册[M].北京:中国轻工业出版社, 1994.
ZHU G J, WANG Z X.Industrial Microbiology:A Laboratory Manual[M].Beijing:China Light Industry Press, 1994.
[18] SAMBROOK J, RUSSELL D W.Molecular Cloning:A Laboratory Manual[M].3rd ed.Cold Spring Harbor, N.Y.:Cold Spring Harbor Laboratory Press, 2001.
[19] 宋鹏, 程磊, 田康明, 等.工业蛋白水解酶快速检测方法的建立与初步应用[J].食品与发酵工业, 2019, 45(17):216-220.
SONG P, CHENG L, TIAN K M, et al.A novel method for rapid determination of industrial protease and its preliminary application[J].Food and Fermentation Industries, 2019, 45(17):216-220.
[20] 程磊, 王正祥.黑曲霉寡肽酶YpsA的生化特征解析[J].食品与发酵工业, 2023, 49(1):19-24.
CHENG L, WANG Z X.Biochemical characteristics of oligopeptidase YpsA from Aspergillus niger[J].Food and Fermentation Industries, 2023, 49(1):19-24.