随着不可再生能源的消耗,以小麦麸皮、玉米秸秆等木质纤维素为原料的生物炼制技术得到了快速发展[1-2]。农作物小麦麸皮、玉米秸秆等是可再生的生物质资源,生物质是一种良好的替代品,有诸多优势,地球上最丰富的可再生绿色能源是从植物中提取的木质纤维素生物质,其结构成分主要是纤维素、半纤维素、木质素等[3-4]。其中,纤维素和半纤维素是由糖单体聚合而成[5-6],而木质素由3种不同的苯丙烷基单元组成的一种结构复杂的聚合物[7]。由于木质素结构稳定、难以分解,在木质纤维素生物质的生物转化过程中,木质素会阻碍纤维素和半纤维素的有效水解[8]。在过去的几十年里,已报道了许多常用的木质素降解预处理方法,例如:物理法[9-11]、化学法[12-15]和生物法[16-17],将木质素降解生成有价值的化合物[18-19],其中生物法主要是采用微生物或其产生的生物酶对木质素进行降解,是一种经济、高效、安全的预处理方法[20]。对木质素降解环境中的微生物进行筛选,发现了细菌和真菌菌株对木质素降解能力不同。真菌表现出比细菌更强大的木质素降解活力,学者们已对真菌的木质素降解酶系进行了广泛研究[21]。然而,使用细菌降解木质素比真菌更有优势,因为细菌的基因组较小,易于遗传操作,重要降解酶的重组表达可以大规模进行[22]。
染料脱色过氧化物酶(dye-decolorizing peroxidases EC.1.11.1.19,DyPs)是一种新发现的含有血红素的过氧化物酶[23],虽然其一级结构氨基酸序列与其他过氧化物酶(如木质素过氧化物酶、锰过氧化物酶)存在很大差异,但DyPs中含有的血红素基团也具有催化反应能力,它们的物理化学性质与传统的血红素酶蛋白相似,如紫外-可见光特征吸收、分子质量或等电点等[24-25]。因此,学者们为这类酶建立了一个新的血红素过氧化物酶家族——染料脱色过氧化物酶[23]。
研究表明,DyPs是由α-螺旋包裹着反平行β-折叠构成的铁氧化还原蛋白类似的折叠,从而呈现一种类似于三明治的结构[26]。DyPs酶的特异性较低,它们对典型的过氧化物酶底物均有氧化作用,如ABTS、刚果红、酸性红151、活性黑5等[27]。DyPs是具有氧化活性和水解活性的双功能酶,其分子质量为40~60 kDa[28]。DyPs含有远端天冬氨酸残基,而不是组氨酸残基,在酸性条件下,使用H2O2作为电子受体时,DyPs对多种底物均具有活性,包括单酚类化合物、合成染料、木质素和木质素模型化合物等[29-31]。
近年来,越来越多的学者对微生物DyPs酶进行了深入研究,发现其不仅具有较好的染料脱色效果,在木质素的降解方面也具有突出优势。本文综述了木质素的结构特征、DyPs酶的分类和结构特征、DyPs在木质素降解中的应用及其降解机制以及基因改造技术对DyPs性状改善的研究等。
1 木质素的结构特征
木质纤维素生物质的结构成分主要是纤维素、半纤维素、木质素等(如图1所示)。其中,纤维素和半纤维素是由糖单体聚合而成,而木质素是一种结构复杂的聚合物,含有丰富的芳香族化合物,存在于植物维管组织的细胞壁中,主要作用是为植物结构提供机械强度[32-33]。木质素结构由3种不同的苯丙烷基单元组成,如图2所示,它们分别是来自前体对香豆醇的对羟基苯基单元、来自前体松柏醇的愈创木基单元以及来自前体芥子醇的丁香基单元[34-35]。由于木质素结构稳定、难以分解,在木质纤维素的生物转化过程中,木质素会阻碍纤维素和半纤维素的有效水解[8]。木质素中主要的键合方式有β-O-4、β-5、β-β等,其中最常见的键合方式是β-O-4键[36]。
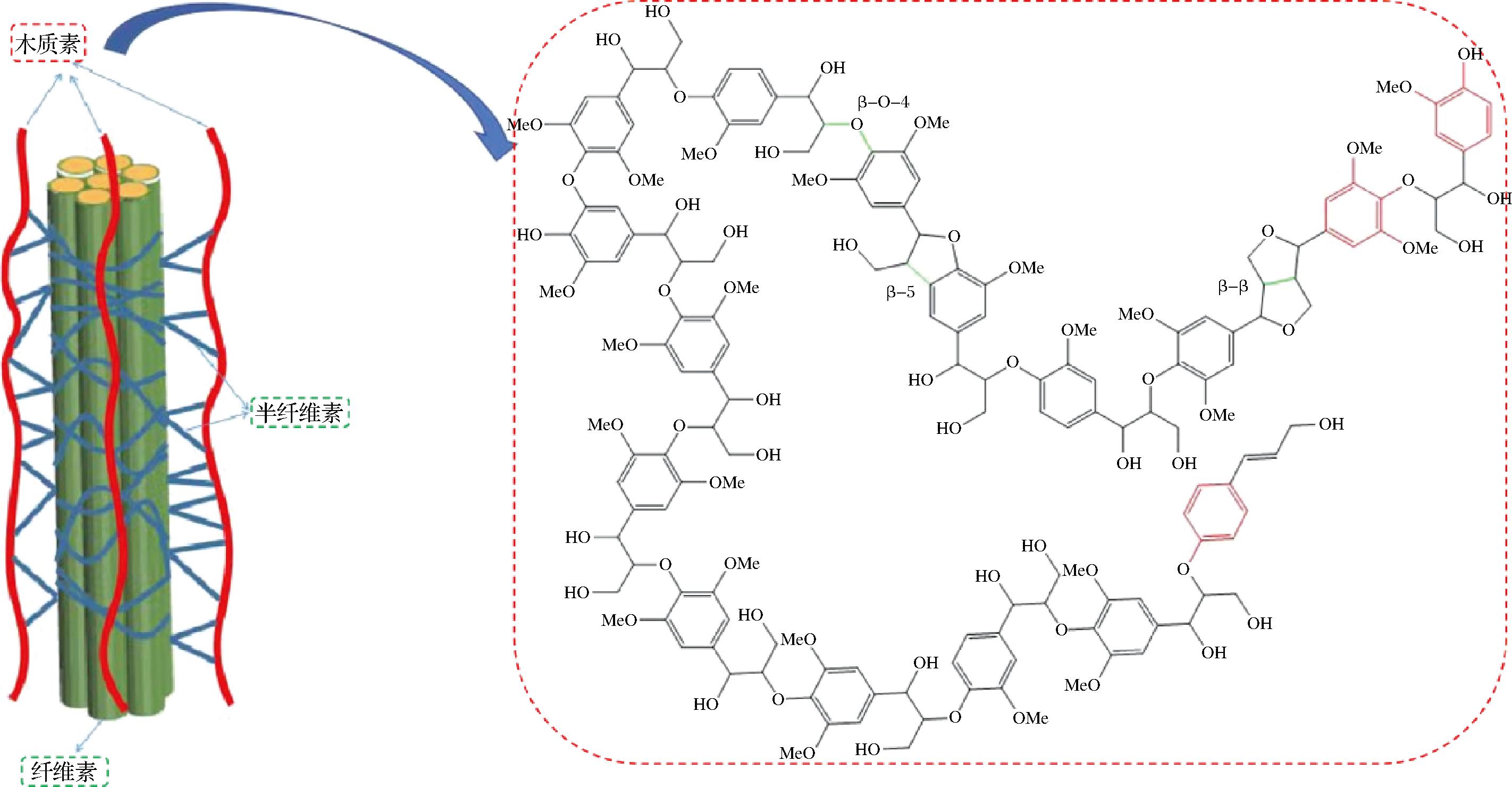
图1 植物生物质资源结构示意图
Fig.1 Structure diagram of plant biomass resources
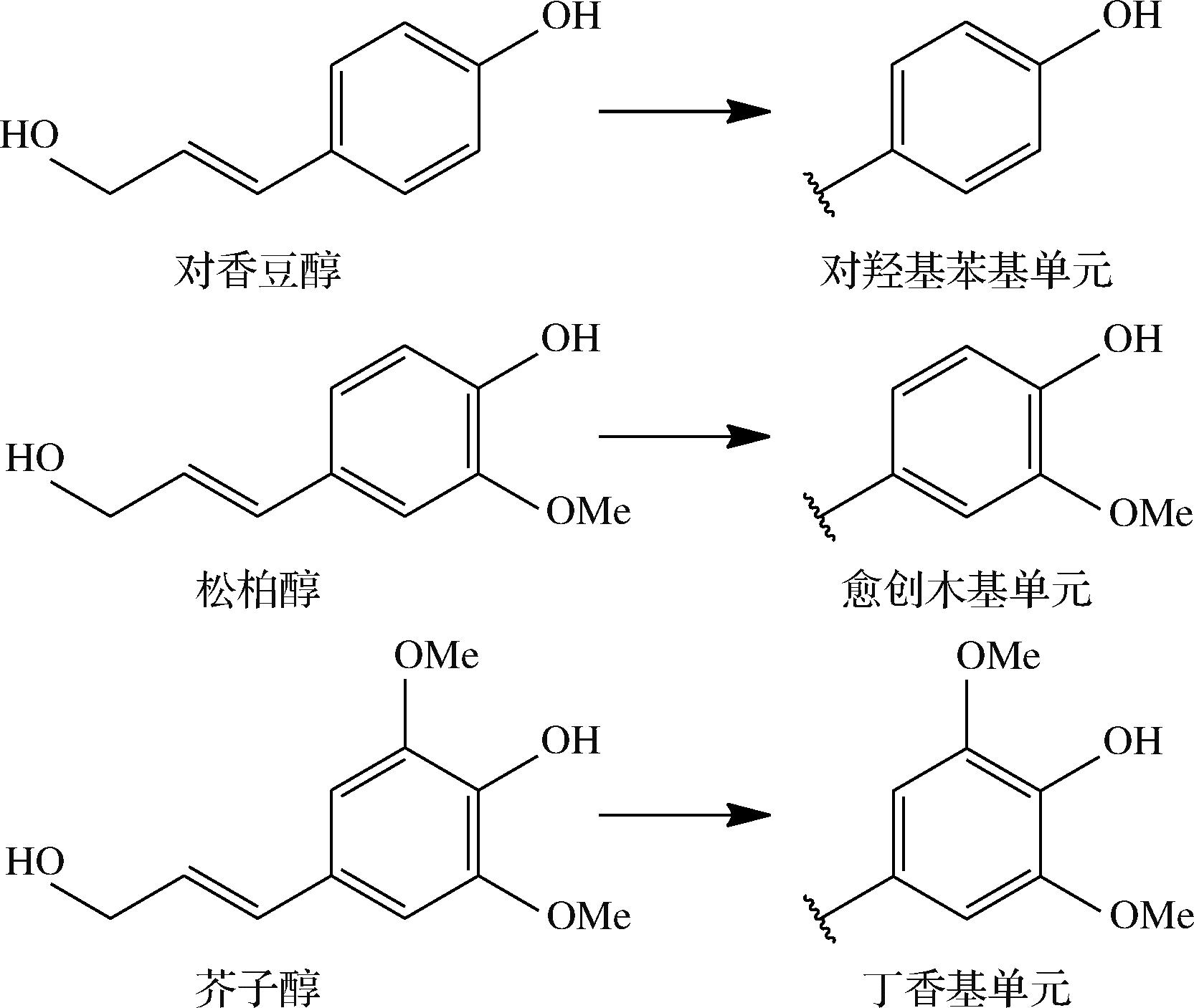
图2 木质素的结构单元
Fig.2 The structural unit of lignin
2 DyPs的分类和结构特征
2.1 DyPs的来源和分类
染料脱色过氧化物酶最早是在真菌中发现的,因其对各种染料具有脱色和降解的能力而得名。1999年,KIM等[37]从真菌Geotrichum candidum Dec 1中分离纯化出具有染料脱色作用的蛋白,随后,在其它真菌中也发现了一些DyPs,并发现它们具有木质素降解性能,如Termitomyces albuminosus[38]、Auricularia auricula-judae[27]、Irpex lacteus[39]、Thermobifida fusca[40]等。对分离纯化得到的DyPs进行表征,发现其均含有血红素辅助因子[41-42],但其一级结构氨基酸序列与已发现的血红素过氧化物酶存在很大差异,因此,学者们为这类酶建立了一个新的血红素过氧化物酶家族——染料脱色过氧化物酶[23]。
将GenBank数据库中搜索的真菌、细菌和古细菌中发现的典型DyPs进行汇总列表(如表1所示),并对其氨基酸序列进行多序列比对,建立系统发育树(如图3所示),根据聚类结果以及前期报道可知,DyPs可分为A、B、C和D亚族。A、B、C型DyPs主要存在于细菌中,如厚壁菌门、放线菌门以及α-、β-、γ-、δ-变形菌门等,而D型DyPs大多存在于真菌中[32, 43]。A型DyPs含有Tat依赖的信号序列,表明其参与胞质外的生理活动,还未报道B型和C型DyPs的蛋白质序列有分泌信号肽,因此,推定B型和C型DyPs为胞质酶,参与细胞内代谢[44],D型DyPs主要包含真菌变体。
表1 不同类型的染料脱色过氧化物酶
Table 1 Different types of dye-decolorizing peroxidases

编号DyPs名称GenBank VERSION来源1TfuDyPAAZ57111.1Thermobifida fusca2MvaABM12972.1Mycobacterium vanbaalenii PYR-13Bsu168CAB15852.1Bacillus subtilis 1684PdeABL69832.1Paracoccus denitrificans PD12225RpaABD87513.1Rhodopseudomonas palustris BisB186DyPPaADF43017.1Pseudomonas aeruginosa7CteKF-1EED66859.1Comamonas testosteroni KF-18PpDyPpdb|7QZA|HPseudomonas putida9YfeXBAE76711.1E.coli K-1210DdiEAL70759.1Dictyostelium discoideum AX411AcspCAG67144.1Acinetobacter sp.ADP112RjoDyPBpdb|3VEE|CRhodococcus jostii13BtDyPpdb|2GVK|ABacteroides thetaiotaomicron VPI-548214CcoEAT98288.1Campylobacter concisus 1382615ChuABG59511.1Cytophaga hutchinsonii ATCC 4918516CyspACK71272.1Cyanothece sp.PCC 742417CviAAQ59612.1Chromobacterium violaceum ATCC 1247218OanABS17389.1Ochrobactrum anthropi ATCC 4918519PsspABQ94167.1Psychrobacter sp.PRwf-120MxDyPWP_201424458.1Myxococcus xanthus21AnaBAB77951.1Anabaena sp.PCC 712022AmspDyP2pdb|4G2C|BAmycolatopsis sp.Atcc 3911623Lbi2EDR12662.1Laccaria bicolor S238 N-H8224PplEED79944.1Postia placenta Mad-698-R25PchCAP99029.1Penicillium rubens Wisconsin 54-125526BjaDyPBAA77283.1Bjerkandera adusta27AauDyP1pdb|5IKG|AAuricularia auricula-judae28PosCAK55151.1Pleurotus ostreatus29Msp1CAP53934.1Marasmius scorodonius30PpaALL41008.1Phakopsora pachyrhizi31VvDyPAKU04643.1Volvariella volvacea32SvDyPAGT48269.1Saccharomonospora viridis33StDyPRYJ30411.1Streptomyces sp.L-9-1034CMfCAA9891472.1Candidatus Methylobacter favarea35PbaKIH83555.1Pseudomonas batumici36DbDyPTHU88578.1Dendrothele bispora CBS 962.96
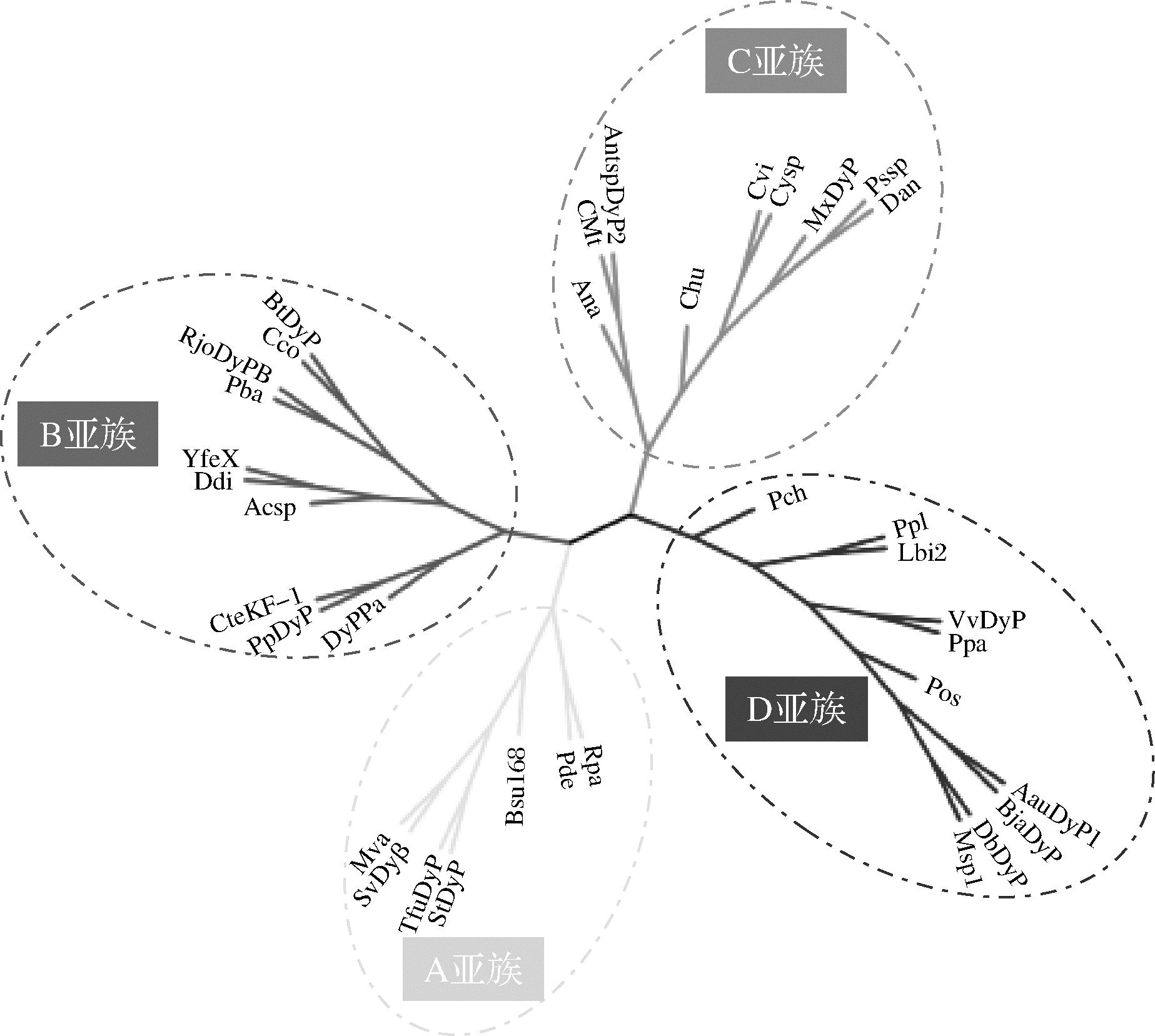
图3 A~D亚族染料脱色过氧化物酶系统发育树
Fig.3 Phylogenetic tree of dye-decolorizing peroxidases of the A-D subgroup
注:黄色表示A亚族;红色表示B亚族;绿色表示C亚族;蓝色表示D亚族。
2.2 DyPs的结构特征
根据PeroxiBase数据库,到目前为止,构成染料脱色过氧化物酶超家族的序列只有237个,包括26个A型DyPs蛋白,49个B型DyPs蛋白,24个C型DyPs蛋白,而D型DyPs蛋白占一半以上,达到138个[43]。据报道,DyPs的空间结构具有一致性,包含两个结构域,一个是α-螺旋,另一个是反平行的β-折叠,血红素辅因子作为辅基处于两个结构域之间的空腔中[45]。DyPs是由脱辅基蛋白和血红素通过非共价键形式进行结合[46]。DyPs只有在脱辅基蛋白与血红素结合之后构成全蛋白,并以H2O2作为电子受体,才能发挥DyPs的生物学功能[47]。因此,在DyPs得到高效表达的同时,尽可能提高酶与血红素的结合尤为重要。
研究表明,DyPs是由α-螺旋包裹着反平行β-折叠构成的铁氧化还原蛋白类似的折叠,从而呈现一种类似于三明治的结构(如图4所示),DyPs含有高度保守的GXXDG基序,以近端的组氨酸、精氨酸和远端保守的天冬氨酸作为血红素的配体[48]。D型DyPs远端天冬氨酸在蛋白内长距离电子转移中起着关键作用,远端精氨酸在稳定血红素/蛋白质基质相互作用中起着结构作用[49]。据报道,C型DyPs具有较高的Mn2+氧化活性,可与真菌锰过氧化物酶(MnP)相媲美[50]。
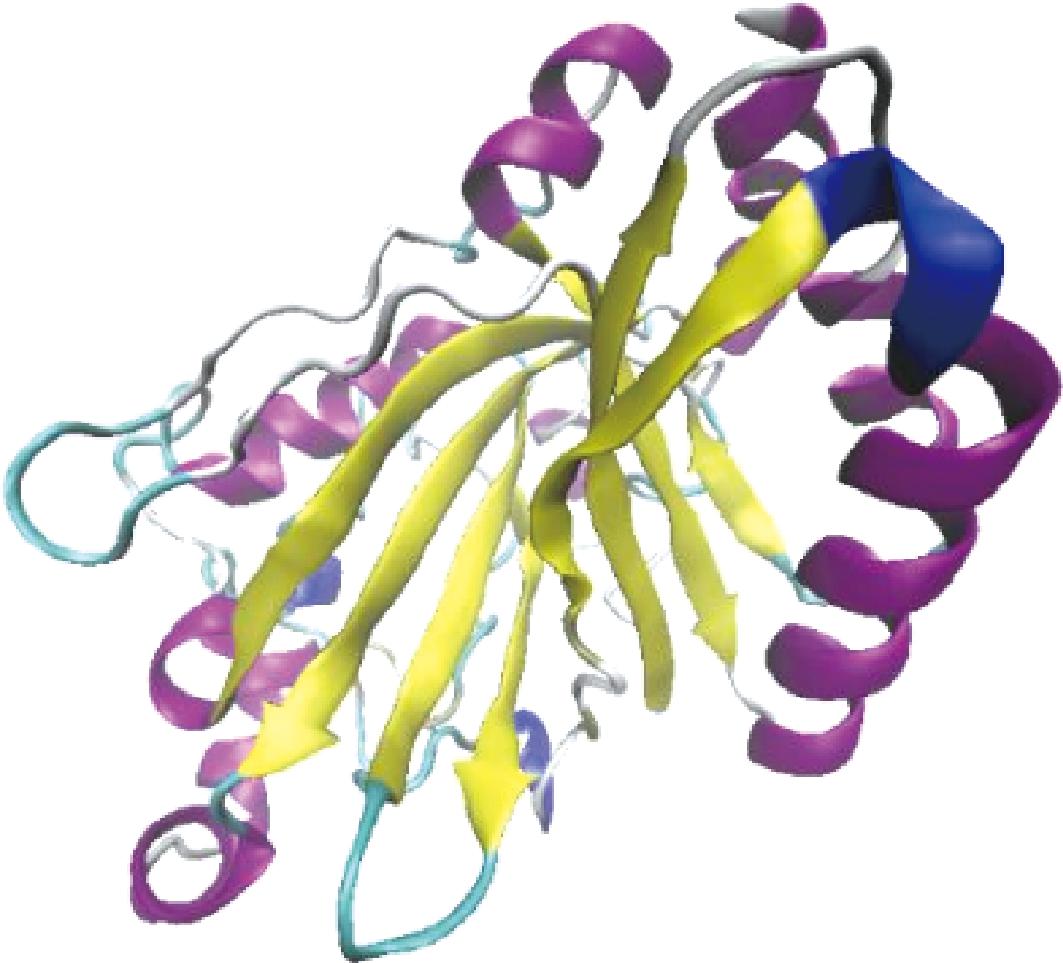
图4 PmDyP蛋白的三级结构[51]
Fig.4 Tertiary structure of PmDyP protein
2.3 DyPs的物理化学性质
在所有鉴定出的DyPs中,除少数细菌能产生分子质量小于40 kDa的DyPs外,其他酶的分子质量均集中在50~60 kDa,含有血红素蛋白。DyPs主要以单体、二聚体、四聚体和六聚体的形式存在。大量研究表明,以ABTS为底物,DyPs在酸性条件下仍能保持较高的活力。在酶活力测试中,DyPs的最佳pH一般在4.0~5.0。例如,当BsDyP、PfDyP和PpDyP以ABTS、2,6-二甲氧基苯酚为底物时,DyPs的最佳pH值变化主要分布在2.0~4.0、4.0~7.2和3.0~5.5[52]。在DyP的实际应用中,最佳pH必须保证DyPs的高活性,并考虑DyPs的稳定性。
此外,温度对酶活力的影响较大。DyPs的最佳温度主要集中在40~60 ℃。据报道,以二氯苯酚为底物,在pH值为5.5,温度为45~60 ℃时,TfuDyP反应速率最快,且反应速率随温度升高呈线性下降[29]。
之前研究了各种离子对B型DyPs的影响,发现Ca2+、Zn2+、Mn2+都能使胞外漆酶活性提高,Zn2+的作用增强最为显著[53]。一些金属离子如Fe2+、Hg2+对DyPs也有较强的抑制作用。Fe2+的抑制作用可能是破坏DyPs的过氧化氢转移机制,使DyPs本身活化,而其他离子可能也参与此反应过程。这些金属离子占据DyPs的活性中心,改变DyPs的结构,阻断底物与DyPs的结合,从而抑制酶的活性。
3 DyPs氧化催化的应用研究进展
DyPs独特的结构和催化特性表明其在实际应用中具有广泛的用途[54-55]。DyPs在H2O2的催化下,酶作为电子受体,可直接将具有高氧化还原电位的底物降解,是一种极具潜力的木质素降解酶。
3.1 DyPs对木质素的降解研究
木质素是一种高度交联的芳香杂环化合物,由苯丙烷基通过各种醚键和C—C键聚合而成,极难生物降解[56]。DyPs的一些性质表明,它们可能参与了木质素的降解。例如,当AauDyp氧化大体积底物时,表面会形成暴露的活性自由基,类似于木质素过氧化物酶和多功能过氧化物酶催化色氨酸时产生的活性自由基[57]。
已有研究表明,DyPs对木质素的处理具有积极作用,转化产物可用于医药、食品、能源等方面,可将木质素转化为高价值产品[55, 58-59]。在研究DyPs对木质素降解过程中,常用两种木质素二聚体模型化合物愈创木酚基甘油-β-愈创木基醚(guaiacylglycerol-β-guaiacyl ether,GGE)和藜芦基甘油-β-愈创木基醚(veratrylglycerol-β-guaiacyl ether,VGE)。CHEN等[60]在嗜热放线菌Thermomonospora curvata中发现了A型DyP酶TcDyP,研究表明,TcDyP在降解模型化合物GGE时,生成了愈创木酚、4-甲基愈创木酚和羟基化愈创木酚五聚体,降解机理如图5-a所示,推测降解的关键步骤是TcDyP使GGE中Cα和Cβ形成的C—C发生裂解。与此同时,TcDyP具有广泛的底物作用范围,可以使非酚木质素底物对甲氧基扁桃酸MMA脱羧,产生茴香醛作为最终产物。脱羧通常在木质素底物降解中起着至关重要的作用。MIN等[61]发现了Bacillus subtilis KCTC2023中的染料脱色过氧化物酶BsDyP,在其对木质素降解机制的研究中,如图5-b所示,发现BsDyP在50 ℃下可以将VGE降解转化为藜芦醛和愈创木酚,2 h转化率达到53.5%,藜芦醛目前应用于医药、食用香料等行业[62-63]。除此之外,如图5-c所示,用B型DyPs处理GGE还可以得到愈创木酚、愈创木酚三聚体和香兰素。其中,愈创木酚低聚物的产生是由于在血红素过氧化物酶的存在下,愈创木酚可以发生自由基重组,进而转化为愈创木酚低聚物。同时,当Mn2+加入时,DyPB酶的活力提高5~23倍[26]。由此可得,Mn2+的加入可以提高DyPs对木质素底物的催化效率。对比发现,上述降解过程中的关键步骤均是Cα和Cβ形成的C—C发生裂解。从细菌Rhodococcus jostii RHA1中发现的DyPB酶,其突变体N246A对硬木硫酸盐木质素及其溶剂萃取组分进行降解,如图5-d所示,在Mn2+的辅助下,其中两种主要降解产物分别为2,6-二甲氧基-1,4-苯醌和4-羟基-3,5-二甲氧基苯甲醛(丁香醛)[64]。丁香醛目前广泛应用于医药、香料、农药化工等行业[65-66]。
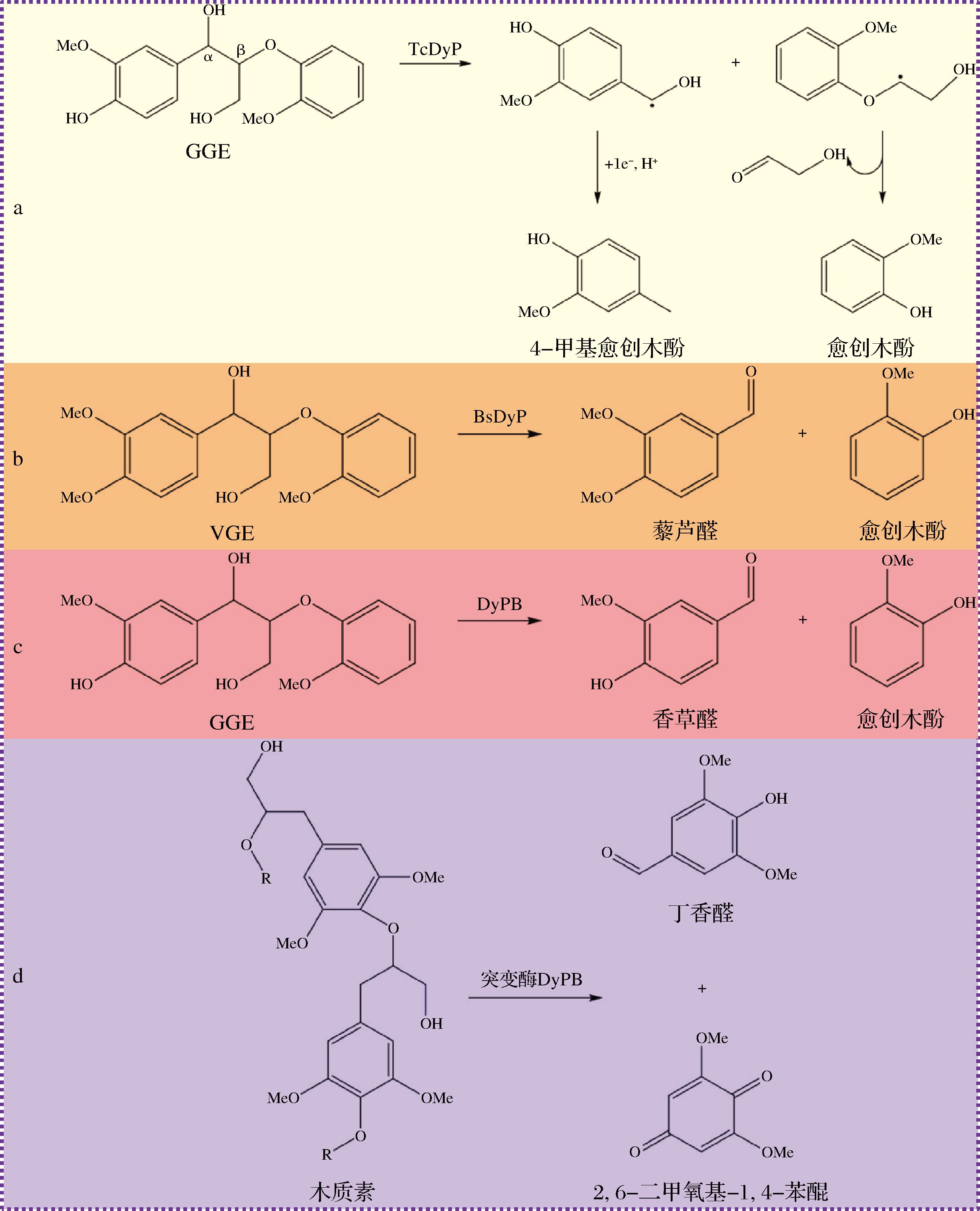
图5 染料脱色过氧化物酶降解木质素机制
Fig.5 Mechanism of lignin degradation by dye-decolorizing peroxidases
上述研究揭示了DyPs降解木质素的生物技术潜力,证明了木质素可以作为DyPs的底物。虽然已经证实DyPs能降解木质素,但关于降解木质素的机理和分子机制的报道较少。随着对DyPs的进一步研究,DyPs将适用于更多的木质素及其衍生物底物。同时,遗传代谢工程技术的进步也加速了DyPs在木质素上的普遍适用性,例如荧光假单胞菌DyPs工程增强了硫酸盐木质素降解的酶催化作用[67],使它克服了物理和化学因素在应用中的局限性。
3.2 DyPs的其他潜在应用
纺织印染废水因其水量大、颜色深、有机物浓度高,处理起来一直是最具挑战性的工业废水。处理困难的一个重要因素是它含有各种类型的染料,其中大多数含有芳香环结构,具有生物毒性[68]。染料废水的处理方法主要有物理法、化学法和生物法。生物法无二次污染,成本低,一直是染料废水处理研究的重点。已有研究表明,DyPs对不同类型染料的处理具有积极作用[69],如偶氮染料、蒽醌染料、三芳基甲烷染料等。这种差异通常是由于染料芳香环上电子供体取代基吸收电子的能力不同造成的。不同菌株的DyPs脱色效率也有较大差异。
野生型DyPs在酸性条件下通常表现出较高的活性[52],而酶本身在发挥作用后仍然是蛋白质,蛋白质水解物可用于微生物生长培养基或生物肥料,也可用于动物饲料,有利于循环经济的发展[70]。DyPs在循环经济系统中是一个有意义的研究方向,它将产生巨大的经济效益,减缓环境问题。
DyPs的底物范围广,耐酸性强,在卤化、环氧化、羟基化等反应中发挥稳定作用,成为造纸、涂料、环保、医疗、生物能源等行业的研究热点[71-74]。除了对木质素的降解作用和对不同类型染料的高效脱色外,DyPs还可应用于食品领域。SCHEIBNER等[75]发现,真菌中的一种DyPs能有效降解β-胡萝卜素。β-胡萝卜素是一种黄色脂溶性化合物,稳定存在于自然界中,通过有效降解β-胡萝卜素,使含有乳清的食品和饮料变白。Krahe在2020年发现了一种由皂荚扁豆产生的具有烯烃降解能力的新型DyP PsaPOX,可以裂解芳烯(E)-甲基异丁香酚、α-甲基苯乙烯和反式茴香醇。茴香醛等裂解产物作为香料添加剂,广泛应用于食品工业[76]。
4 基因改造技术对DyPs性状改善的研究进展
DyPs在实际应用过程中常常受到环境因素的影响。例如酶活力不高,最适pH范围较窄,限制了DyPs的实际应用价值。定向进化技术和基因重组技术的发展为解决DyPs的实际应用提供了无限可能。基因数据库的建立和系统发育分析揭示了DyPs之间的组学关系,并阐明了基因功能。这表明利用分子生物学对DyPs的研究可以达到一个新的阶段[77-78]。
采用基因工程技术,可对DyPs进行高效异源表达。YU等[61]从Saccharomonospora viridis基因文库中克隆了一个DyPs基因SviDyP,其序列与微生物染料脱色过氧化物酶相似。该蛋白在中性至碱性条件下能对三芳基甲烷染料、蒽醌染料和偶氮染料进行有效脱色。重构的SviDyP的最佳pH值为7.0,最适温度为70 ℃。此外,与其他DyPs相比,它具有广泛的耐碱性(pH 4.0~9.0)和耐热性(37~80 ℃)。
同时,定向进化和修饰技术在DyPs酶学性质改善中也得到了广泛应用。RAHMANPOUR等[26]对嗜热单孢菌Thermobifida fusca中TfuDyP酶的D203A和R315Q位点进行诱变,测定酶活得出,突变体D203A对酚类底物的催化活性丧失,且对ABTS的催化活性降低,突变体R315Q对酚类和ABTS均没有活性,表明D203和R315位点对酶催化活性有重要影响。UCHIDA等[79]在确定霍乱弧菌中VcDyP酶的活定位点时,将两个保守的氨基酸Asp144和Arg230分别突变为Val和Leu,突变体D144V和R230L均不能将染料脱色,表明D203和R315是维持酶活的关键碱基。研究结果说明远端保守残基Asp-Arg很可能是DyPs的催化活性中心,且对酶催化活力有重要影响。BRISSOS等[80]对Pseudomouas.putida MET94的E188K、A142V和H125Y位点进行诱变,得到P.putida 6E10。PpDyP的活性位点以及突变位点的详细视图(如图6所示),保守氨基酸残基为H197、D132、N136、R214,其中E188K突变位于血红素辅因子的侧链附近,处在表面环I179-A196上,位于血红色通道入口5 Å处,A142V突变位于α-螺旋E140-A146上且与含有保守氨基酸残基N136、D132的环D117-D139和突变位点H125Y相连接,此突变可能会对α-螺旋的性质有影响,而H125Y突变处在离保守氨基酸残基D132很近的位置,此突变可能对活性中心的空间结构造成影响。突变体对木质素模型二聚体GGE、硫酸盐木质素的氧化性能显著提高,尤其是对2,6-二甲氧基苯酚的催化效率提高了100倍。同时,与野生型菌株产生的DyPs不同,重组PpDyP更倾向于在pH 8.5条件下参与反应,大大提高了耐碱能力[61]。
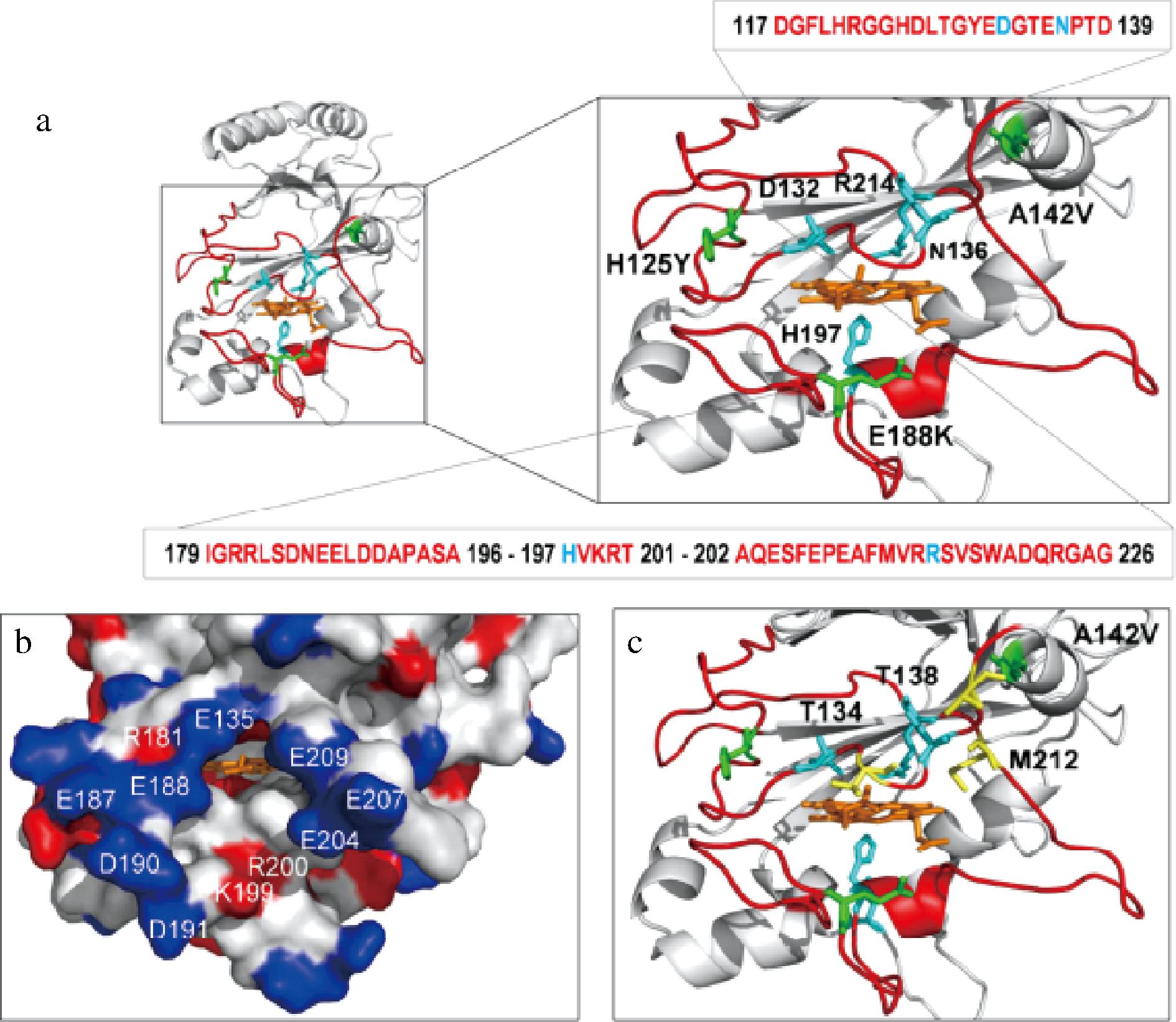
图6 PpDyP的活性位点及突变位点的三级结构图[80]
Fig.6 Tertiary structure diagram of the active and mutated sites of PpDyP
酶的定向进化技术也在不断创新。ALESSA等[81]率先利用细菌胞外蛋白分泌系统,简单高效地实现了DyPs的进化,加速了平菇菌株PC15产生DyP4蛋白。对可降解性较高的DyPs进行改造和挖掘,DyPs可作为木质素生物降解的预处理工艺,在DyPs对木质素降解过程中存在大量芳香族单体。基因编辑技术对DyPs影响的另一个方面是,在阐明DyPs降解机制的基础上,主动修改菌株产生DyPs的代谢途径,以完成最终产物的靶向生产。
5 结语
虽然国内对染料脱色过氧化物酶的研究相对较晚,但它因氧化性强、耐酸性强等优点在农作物副产物加工应用方面表现出极大潜力,不仅可以提高副产物的综合利用价值,还可以提高粮食利用率,实现节粮减损,创造经济效益。但目前DyPs在木质素降解中发挥的作用机理以及活性作用位点等尚不明确,有待进一步探索和确认。此外,通过化学工程和蛋白质工程对DyPs进行人工修饰或构建,可获得更高效的酶。随着DyPs催化效率的提高,产量的增加,环境耐受性的增强,DyPs在工业中的应用前景将更加广阔。
[1] 吴凡, 蔡红燕, 沈汪洋, 等.小麦麸皮的营养特性与应用进展[J].食品工业, 2020, 41(5):284-287.
WU F, CAI H Y, SHEN W Y, et al.Nutritional characteristics and application progress of wheat bran[J].The Food Industry, 2020, 41(5):284-287.
[2] 杨传文, 邢帆, 朱建春, 等.中国秸秆资源的时空分布、利用现状与碳减排潜力[J].环境科学, 2023, 44(2):1149-1162.
YANG C W, XING F, ZHU J C, et al.Temporal and spatial distribution, utilization status, and carbon emission reduction potential of straw resources in China[J].Environmental Science, 2023, 44(2):1149-1162.
[3] SINGHANIA R R, PATEL A K, RAJ T, et al.Lignin valorisation via enzymes:A sustainable approach[J].Fuel, 2022, 311:122608.
[4] UPTON B M, KASKO A M.Strategies for the conversion of lignin to high-value polymeric materials:Review and perspective[J].Chemical Reviews, 2016, 116(4):2275-2306.
[5] ZHOU C H, XIA X, LIN C X, et al.Catalytic conversion of lignocellulosic biomass to fine chemicals and fuels[J].Chemical Society Reviews, 2011, 40(11):5588-5617.
[6] 王超, 李宗全.水解液中半纤维素分离提纯的研究进展[J].造纸科学与技术, 2015, 34(4):37-41;36.
WANG C, LI Z Q.A review of the separation and purification of hemicelluloses from hydrolysate[J].Paper Science &Technology, 2015, 34(4):37-41;36.
[7] BUGG T D, RAHMANPOUR R.Enzymatic conversion of lignin into renewable chemicals[J].Current Opinion in Chemical Biology, 2015, 29:10-17.
[8] SADAQAT B, KHATOON N, MALIK A Y, et al.Enzymatic decolorization of melanin by lignin peroxidase from Phanerochaete chrysosporium[J].Scientific Reports, 2020, 10(1):20240.
[9] ZAKARIA M R, FUJIMOTO S, HIRATA S, et al.Ball milling pretreatment of oil palm biomass for enhancing enzymatic hydrolysis[J].Applied Biochemistry and Biotechnology, 2014, 173(7):1778-1789.
[10] JACQUET N, MANIET G, VANDERGHEM C, et al.Application of steam explosion as pretreatment on lignocellulosic material:A review[J].Industrial &Engineering Chemistry Research, 2015, 54(10):2593-2598.
[11] WANG K Q, XIONG X Y, CHEN J P, et al.Comparison of gamma irradiation and steam explosion pretreatment for ethanol production from agricultural residues[J].Biomass and Bioenergy, 2012, 46:301-308.
[12] 杜琨, 林明喜, 黄立杰, 等.碱性双氧水法提取甘蔗渣中的纤维素[J].现代盐化工, 2019, 46(6):17-20.
DU K, LIN M X, HUANG L J, et al.Extraction of cellulose from bagasse by alkaline hydrogen peroxide method[J].Modern Salt and Chemical Industry, 2019, 46(6):17-20.
[13] GLI SKA K, GITALT J, TORRENS E, et al.Extraction of cellulose from corn stover using designed ionic liquids with improved reusing capabilities[J].Process Safety and Environmental Protection, 2021, 147:181-191.
SKA K, GITALT J, TORRENS E, et al.Extraction of cellulose from corn stover using designed ionic liquids with improved reusing capabilities[J].Process Safety and Environmental Protection, 2021, 147:181-191.
[14] LI J, ZHANG M, WANG D H.Enhancing delignification and subsequent enzymatic hydrolysis of corn stover by magnesium oxide-ethanol pretreatment[J].Bioresource Technology, 2019, 279:124-131.
[15] CAPUTO D, FUSCO C, NACCI A, et al.A selective cellulose/hemicellulose green solvents extraction from buckwheat chaff[J].Carbohydrate Polymer Technologies and Applications, 2021, 2:100094.
[16] KUMAR R, WYMAN C E.Effects of cellulase and xylanase enzymes on the deconstruction of solids from pretreatment of poplar by leading technologies[J].Biotechnology Progress, 2009, 25(2):302-314.
[17] 张循海, 宋贺明, 贾宏葛, 等.漆酶系统提取玉米秸秆中的纤维素[J].齐齐哈尔大学学报(自然科学版), 2020, 36(5):43-44;49.
ZHANG X H, SONG H M, JIA H G, et al.The laccase extracting cellulose systems from corn straw[J].Journal of Qiqihar University (Natural Science Edition), 2020, 36(5):43-44;49.
[18] CHEN S S, WANG W C, LI X, et al.Regulating the nanoscale intimacy of metal and acidic sites in Ru/γ-Al2O3 for the selective conversions of lignin-derived phenols to jet fuels[J].Journal of Energy Chemistry, 2022, 66:576-586.
[19] WANG J, FAN Y X, WANG H L, et al.Promoting efficacy and environmental safety of photosensitive agrochemical stabilizer via lignin/surfactant coacervates[J].Chemical Engineering Journal, 2022, 430:132920.
[20] NGUYEN L T, PHAN D P, SARWAR A, et al.Valorization of industrial lignin to value-added chemicals by chemical depolymerization and biological conversion[J].Industrial Crops and Products, 2021, 161:113219.
[21] SIJINAMANOJ V, MUTHUKUMAR T, MUTHURAJA R, et al.Ligninolytic valorization of agricultural residues by Aspergillus nomius and Trichoderma harzianum isolated from gut and comb of Odontotermes obesus (Termitidae)[J].Chemosphere, 2021, 284:131384.
[22] SETHUPATHY S, MURILLO MORALES G, GAO L, et al.Lignin valorization:Status, challenges and opportunities[J].Bioresource Technology, 2022, 347:126696.
[23] SUGANO Y.DyP-type peroxidases comprise a novel heme peroxidase family[J].Cellular and Molecular Life Sciences, 2009, 66(8):1387-1403.
[24] HOFRICHTER M, ULLRICH R, PECYNA M J, et al.New and classic families of secreted fungal heme peroxidases[J].Applied Microbiology and Biotechnology, 2010, 87(3):871-897.
[25] LIERS C, ARANDA E, STRITTMATTER E, et al.Phenol oxidation by DyP-type peroxidases in comparison to fungal and plant peroxidases[J].Journal of Molecular Catalysis B:Enzymatic, 2014, 103:41-46.
[26] RAHMANPOUR R, REA D, JAMSHIDI S, et al.Structure of Thermobifida fusca DyP-type peroxidase and activity towards Kraft lignin and lignin model compounds[J].Archives of Biochemistry and Biophysics, 2016, 594:54-60.
[27] SUGANO Y, MURAMATSU R, ICHIYANAGI A, et al.DyP, a Unique Dye-decolorizing Peroxidase, Represents a Novel Heme Peroxidase Family asp171 replaces the distal histidine of classical peroxidases[J].Journal of Biological Chemistry, 2007, 282(50):36652-36658.
[28] LIERS C, BOBETH C, PECYNA M, et al.DyP-like peroxidases of the jelly fungus Auricularia auricula-judae oxidize nonphenolic lignin model compounds and high-redox potential dyes[J].Applied Microbiology and Biotechnology, 2010, 85(6):1869-1879.
[29] AHMAD M, ROBERTS J N, HARDIMAN E M, et al.Identification of DypB from Rhodococcus jostii RHA1 as a lignin peroxidase[J].Biochemistry, 2011, 50(23):5096-5107.
[30] LON AR N, COLPA D I, FRAAIJE M W.Exploring the biocatalytic potential of a DyP-type peroxidase by profiling the substrate acceptance of Thermobifida fusca DyP peroxidase[J].Tetrahedron, 2016, 72(46):7276-7281.
AR N, COLPA D I, FRAAIJE M W.Exploring the biocatalytic potential of a DyP-type peroxidase by profiling the substrate acceptance of Thermobifida fusca DyP peroxidase[J].Tetrahedron, 2016, 72(46):7276-7281.
[31] 朱竹兵, 孙亚武, 唐蕾.褐色嗜热裂孢菌脱色过氧化物酶的表达及发酵条件优化[J].食品与发酵工业, 2019, 45(13):23-30.
ZHU Z B, SUN Y W, TANG L.Expression and fermentation optimization of dye-decolorizing peroxidase from Thermobifida fusca[J].Food and Fermentation Industries, 2019, 45(13):23-30.
[32] DE GONZALO G, COLPA D I, HABIB M H M, et al.Bacterial enzymes involved in lignin degradation[J].Journal of Biotechnology, 2016, 236:110-119.
[33] SAINSBURY P D, HARDIMAN E M, AHMAD M, et al.Breaking down lignin to high-value chemicals:The conversion of lignocellulose to vanillin in a gene deletion mutant of Rhodococcus jostii RHA1[J].ACS Chemical Biology, 2013, 8(10):2151-2156.
[34] DE VRIES L, GUEVARA-ROZO S, CHO M, et al.Tailoring renewable materials via plant biotechnology[J].Biotechnology for Biofuels, 2021, 14(1):167.
[35] VANHOLME R, DE MEESTER B, RALPH J, et al.Lignin biosynthesis and its integration into metabolism[J].Current Opinion in Biotechnology, 2019, 56:230-239.
[36] CAO Y, CHEN S S, ZHANG S C, et al.Advances in lignin valorization towards bio-based chemicals and fuels:Lignin biorefinery[J].Bioresource Technology, 2019, 291:121878.
[37] KIM S J, SHODA M.Purification and characterization of a novel peroxidase from Geotrichum candidum dec 1 involved in decolorization of dyes[J].Applied and Environmental Microbiology, 1999, 65(3):1029-1035.
[38] JOHJIMA T, OHKUMA M, KUDO T.Isolation and cDNA cloning of novel hydrogen peroxide-dependent phenol oxidase from the basidiomycete Termitomyces albuminosus[J].Applied Microbiology and Biotechnology, 2003, 61(3):220-225.
[39] SALVACH A D, PRIETO A, MART
A D, PRIETO A, MART NEZ
NEZ  T, et al.Characterization of a novel dye-decolorizing peroxidase (DyP)-type enzyme from Irpex lacteus and its application in enzymatic hydrolysis of wheat straw[J].Applied and Environmental Microbiology, 2013, 79(14):4316-4324.
T, et al.Characterization of a novel dye-decolorizing peroxidase (DyP)-type enzyme from Irpex lacteus and its application in enzymatic hydrolysis of wheat straw[J].Applied and Environmental Microbiology, 2013, 79(14):4316-4324.
[40] VAN BLOOIS E, TORRES PAZMI O D E, WINTER R T, et al.A robust and extracellular heme-containing peroxidase from Thermobifida fusca as prototype of a bacterial peroxidase superfamily[J].Applied Microbiology and Biotechnology, 2010, 86(5):1419-1430.
O D E, WINTER R T, et al.A robust and extracellular heme-containing peroxidase from Thermobifida fusca as prototype of a bacterial peroxidase superfamily[J].Applied Microbiology and Biotechnology, 2010, 86(5):1419-1430.
[41] LI J, LIU C, LI B Z, et al.Identification and molecular characterization of a novel DyP-type peroxidase from Pseudomonas aeruginosa PKE117[J].Applied Biochemistry and Biotechnology, 2012, 166(3):774-785.
[42] ZUBIETA C, KRISHNA S S, KAPOOR M, et al.Crystal structures of two novel dye-decolorizing peroxidases reveal a beta-barrel fold with a conserved heme-binding motif[J].Proteins, 2007, 69(2):223-233.
[43] GRANJA-TRAVEZ R S, PERSINOTI G F, SQUINA F M, et al.Functional genomic analysis of bacterial lignin degraders:Diversity in mechanisms of lignin oxidation and metabolism[J].Applied Microbiology and Biotechnology, 2020, 104(8):3305-3320.
[44] 皮倩, 夏荣, 唐蕾.红球菌染料脱色过氧化物酶的异源表达及活性分析[J].食品与发酵工业, 2021, 47(18):86-91.
PI Q, XIA R, TANG L.Heterologous expression and activity analysis of dye-decolorizing peroxidase from Rhodococcus jostii[J].Food and Fermentation Industries, 2021, 47(18):86-91.
[45] COLPA D I, FRAAIJE M W, VAN BLOOIS E.DyP-type peroxidases:A promising and versatile class of enzymes[J].Journal of Industrial Microbiology &Biotechnology, 2014, 41(1):1-7.
[46] COLPA D I, FRAAIJE M W.High overexpression of dye decolorizing peroxidase TfuDyP leads to the incorporation of heme precursor protoporphyrin IX[J].Journal of Molecular Catalysis B:Enzymatic, 2016, 134:372-377.
[47] LAYER G, REICHELT J, JAHN D, et al.Structure and function of enzymes in heme biosynthesis[J].Protein Science, 2010, 19(6):1137-1161.
[48] LAUBER C, SCHWARZ T, NGUYEN Q K, et al.Identification, heterologous expression and characterization of a dye-decolorizing peroxidase of Pleurotus sapidus[J].AMB Express, 2017, 7(1):164.
[49] ZITARE U A, HABIB M H, ROZEBOOM H, et al.Mutational and structural analysis of an ancestral fungal dye-decolorizing peroxidase[J].The FEBS Journal, 2021, 288(11):3602-3618.
[50] BROWN M E, BARROS T, CHANG M C Y.Identification and characterization of a multifunctional dye peroxidase from a lignin-reactive bacterium[J].ACS Chemical Biology, 2012, 7(12):2074-2081.
[51] 杨趁仙. 木质素降解细菌的筛选及其酶对木质素降解机制的研究[D].杨凌:西北农林科技大学, 2018:85-87.
Yang C X.The isolation of lignin-degrading bacteria and the degradation mechanism of enzymes[D].Yangling:Northwest A&F University, 2018:85-87.
[52] SANTOS A, MENDES S, BRISSOS V, et al.New dye-decolorizing peroxidases from Bacillus subtilis and Pseudomonas putida MET94:Towards biotechnological applications[J].Applied Microbiology and Biotechnology, 2014, 98(5):2053-2065.
[53] AZEVEDO A M, MARTINS V C, PRAZERES D M, et al.Horseradish peroxidase:A valuable tool in biotechnology[J].Biotechnology Annual Review, 2003, 9:199-247.
[54] BUGG T D H, WILLIAMSON J J, RASHID G M M.Bacterial enzymes for lignin depolymerisation:New biocatalysts for generation of renewable chemicals from biomass[J].Current Opinion in Chemical Biology, 2020, 55:26-33.
[55] ALRUWAILI A, RASHID G M M, BUGG T D H.Application of Rhodococcus jostii RHA1 glycolate oxidase as an efficient accessory enzyme for lignin conversion by bacterial Dyp peroxidase enzymes[J].Green Chemistry, 2023, 25(9):3549-3560.
[56] RALPH J, LAPIERRE C, BOERJAN W.Lignin structure and its engineering[J].Current Opinion in Biotechnology, 2019, 56:240-249.
[57] LIERS C, PECYNA M J, KELLNER H, et al.Substrate oxidation by dye-decolorizing peroxidases (DyPs) from wood- and litter-degrading Agaricomycetes compared to other fungal and plant heme-peroxidases[J].Applied Microbiology and Biotechnology, 2013, 97(13):5839-5849.
[58] KAMIMURA N, SAKAMOTO S, MITSUDA N, et al.Advances in microbial lignin degradation and its applications[J].Current Opinion in Biotechnology, 2019, 56:179-186.
[59] 李家琦, 邓佳钦, 朱艺, 等.酶促水解木质纤维素高值转化研究现状[J].湖南林业科技, 2023, 50(1):115-122.
LI J Q, DENG J Q, ZHU Y, et al.Current status of high-value conversion of lignocellulose by enzymatic hydrolysis[J].Hunan Forestry Science &Technology, 2023, 50(1):115-122.
[60] CHEN C, SHRESTHA R, JIA K M, et al.Characterization of dye-decolorizing peroxidase (DyP) from Thermomonospora curvata reveals unique catalytic properties of A-type DyPs[J].Journal of Biological Chemistry, 2015, 290(38):23447-23463.
[61] YU W, LIU W, HUANG H, et al. Application of a novel alkali-tolerant thermostable DyP-type peroxidase from Saccharomonospora viridis DSM 43017 in biobleaching of eucalyptus kraft pulp[J]. Plos One, 2014, 9(10): e110319.
[62] 杨兆国. 异香兰素及藜芦醛的合成研究[D].长春:吉林大学, 2008:4-5.
YANG Z G.Study on synthesis of isovanillin and veratraldehyde[D].Changchun:Jilin University, 2008:4-5.
[63] 符成华. 藜芦醛及其衍生物的合成研究[D].安庆:安庆师范大学, 2020:1-13.
FU C H.Study on synthesis of veratraldehyde and its derivatives[D].Anqing:Anqing Normal University, 2020:1-13.
[64] SINGH R, GRIGG J C, QIN W, et al.Improved manganese-oxidizing activity of DypB, a peroxidase from a lignolytic bacterium[J].ACS Chemical Biology, 2013, 8(4):700-706.
[65] 赵雪梅, 伍时华, 龙秀锋, 等.丁香醛对酿酒酵母乙醇发酵过程和理化特性的影响[J].食品与发酵工业, 2022, 48(16):188-195.
ZHAO X M, WU S H, LONG X F, et al.Effects of syringaldehyde on ethanol fermentation and physicochemical properties of Saccharomyces cerevisiae[J].Food and Fermentation Industries, 2022, 48(16):188-195.
[66] 方雪祥, 袁慧慧, 单艳超, 等.丁香酚与丁香醛缩二甲醇联合抗氧化作用研究[J].食品工业科技, 2020, 41(2):21-26.
FANG X X, YUAN H H, SHAN Y C, et al.Research on synergistic antioxidant activity of eugenol and syringaldehyde dimethylacetal[J].Science and Technology of Food Industry, 2020, 41(2):21-26.
[67] RAHMAN POUR R, EHIBHATIOMHAN A, HUANG Y L, et al.Protein engineering of Pseudomonas fluorescens peroxidase Dyp1B for oxidation of phenolic and polymeric lignin substrates[J].Enzyme and Microbial Technology, 2019, 123:21-29.
[68] ROUTOULA E, PATWARDHAN S V.Degradation of anthraquinone dyes from effluents:A review focusing on enzymatic dye degradation with industrial potential[J].Environmental Science &Technology, 2020, 54(2):647-664.
[69] KAUSHIK P, MALIK A.Fungal dye decolourization:Recent advances and future potential[J].Environment International, 2009, 35(1):127-141.
[70] NAVONE L, MOFFITT K, HANSEN K A, et al.Closing the textile loop:Enzymatic fibre separation and recycling of wool/polyester fabric blends[J].Waste Management, 2020, 102:149-160.
[71] NAWAZ M Z, SHANG H R, SUN J Z, et al.Genomic insights into the metabolic potential of a novel lignin-degrading and polyhydroxyalkanoates producing bacterium Pseudomonas sp.Hu109A[J].Chemosphere, 2023, 310:136754.
[72] SHIN S K, HYEON J E, JOO Y C, et al.Effective melanin degradation by a synergistic laccase-peroxidase enzyme complex for skin whitening and other practical applications[J].International Journal of Biological Macromolecules, 2019, 129:181-186.
[73] BARBOSA C, SILVEIRA C M, SILVA D, et al.Immobilized dye-decolorizing peroxidase (DyP) and directed evolution variants for hydrogen peroxide biosensing[J].Biosensors and Bioelectronics, 2020, 153:112055.
[74] KONG L Y, GUO D S, ZHOU S Y, et al.Cloning and expression of a toxin gene from Pseudomonas fluorescens GcM5-1A[J].Archives of Microbiology, 2010, 192(7):585-593.
[75] SCHEIBNER M, HÜLSDAU B, ZELENA K, et al.Novel peroxidases of Marasmius scorodonius degrade β-carotene[J].Applied Microbiology and Biotechnology, 2008, 77(6):1241-1250.
[76] KRAHE N K, BERGER R G, ERSOY F.A DyP-type peroxidase of Pleurotus sapidus with alkene cleaving activity[J].Molecules, 2020, 25(7):1536.
[77] SUGANO Y, YOSHIDA T.DyP-type peroxidases:Recent advances and perspectives[J].International Journal of Molecular Sciences, 2021, 22(11):5556.
[78] TIAN J H, POURCHER A M, KLINGELSCHMITT F, et al.Class P dye-decolorizing peroxidase gene:Degenerated primers design and phylogenetic analysis[J].Journal of Microbiological Methods, 2016, 130:148-153.
[79] UCHIDA T, SASAKI M, TANAKA Y, et al.A dye-decolorizing peroxidase from Vibrio cholerae[J].Biochemistry, 2015, 54(43):6610-6621.
[80] BRISSOS V, TAVARES D, SOUSA A C, et al.Engineering a bacterial DyP-type peroxidase for enhanced oxidation of lignin-related phenolics at alkaline pH[J].ACS Catalysis, 2017, 7(5):3454-3465.
[81] ALESSA A H A, TEE K L, GONZALEZ-PEREZ D, et al.Accelerated directed evolution of dye-decolorizing peroxidase using a bacterial extracellular protein secretion system (BENNY)[J].Bioresources and Bioprocessing, 2019, 6(1):20.