基因组编辑是指在基因组尺度对生物体进行精确设计与高效改造。基因组编辑技术自诞生以来就成为生命科学领域的有力工具,经历了从锌指核酸内切酶(zinc-finger nucleases, ZFNs)、转录激活效应子核酸酶(transcription activator like effector nucleases, TALENs)到成簇规律间隔短回文重复序列CRISPR/Cas (Clustered regularly interspaced short palindromic repeats/CRISPR-associated protein)的3次技术革新[1]。这些技术都是利用限制性内切酶在同源结合蛋白或RNA的指导下在目标位置引入DNA双链断裂(double-stranded DNA break, DSB)。
根据Cas蛋白的不同,CRISPR/Cas系统可分为3个不同的类型(I型、II型与III型),其中II型CRISPR/Cas系统的结构最为简单,只需要核酸酶Cas9、成熟的crRNA(CRISPR-derived RNA)与tracrRNA(trans-activating RNA)就可以实现对特定外源DNA序列的切割[2-3]。由CRISPR/Cas产生的DSB会引起细胞产生两种主要的DNA修复途径:一种是通过与DNA模板进行同源重组(homology-directed repair, HR)来修复,从而引入特定的突变;另一种是在没有DNA模板的情况下,真核生物可以通过非同源末端连接(non-homologous End Joining, NHEJ)修复DSB,由NHEJ的易错性而引入插入或缺失(insertions/deletions, InDels)[4]。因此,精确的基因组编辑在很大程度上依赖于DNA转化和HR整合外源DNA模板,导致细胞存活率低、多重编辑困难,甚至在某些情况下基因组编辑失败,就点突变或单碱基转换而言,它的精度仍然值得怀疑[5]。
为了规避这些限制,DAVID和AKIHIKO等研究团队相继发布了基于CRISPR/Cas9衍生的碱基编辑系统。将灭活的Cas蛋白(dead Cas9, dCas9;保留Cas9结合DNA能力,但不切割DNA)或有切割单链DNA(single-stranded DNA, ssDNA)活性的Cas蛋白(Cas9 nickase, nCas9;保留Cas9结合DNA的能力,但仅切割ssDNA)与碱基脱氨酶或者逆转录酶融合,整合蛋白可以在目标基因组位点直接将一个碱基对转换为另一个碱基对,在不引入DSB、不添加外源DNA或依赖HR的情况下在靶位点进行点突变[6]。目前,已开发的碱基编辑器主要有:(a)胞嘧啶碱基编辑器(cytidine base editors, CBEs),能够实现胞嘧啶(cytosine, C)到胸腺嘧啶(thymine, T)或T-to-C的转换;(b)腺嘌呤碱基编辑器(adenine base editor,ABEs),实现腺嘌呤(adenine, A)到鸟嘌呤(guanin, G)或G-to-A的转换[7];(c)糖基化酶碱基编辑器(glycosylase base editors, GBEs)能产生C-to-G或C-to-A之间的颠换[8];(d)Prime碱基编辑器(prime editors, PEs)可以实现任意碱基之间的转换[9];(e)双功能碱基编辑器(dual base editors, DBEs)是在CBEs和ABEs的基础上把2种脱氨酶融合到nCas9/dCas9,它可以同时诱导C-to-T和A-to-G转换[10]。目前,碱基编辑技术在动物、植物和微生物中应用广泛,可以在纠正致病突变、加速基因组修饰和生物育种等研究中发挥重要作用。本文对碱基编辑器最新研究进展进行综述,针对该技术目前所存在的问题讨论未来发展方向。
1 基于CRISPR/Cas系统碱基编辑器的开发
1.1 CBE编辑器
CBE技术原理是将dCas9或nCas9与胞嘧啶脱氨酶融合形成融合蛋白,单一向导RNA(single guide RNA,sgRNA)引导融合蛋白靶向目标序列,胞嘧啶脱氨酶可催化胞嘧啶(C)脱氨基转化为尿嘧啶(uracil, U),尿嘧啶(U)与腺嘌呤(A)相配对,在DNA复制过程中腺嘌呤(A)又与胸腺嘧啶(T)配对,从而实现C-to-T的取代(图1-a)。2016年,KOMOR等[11]率先研发第一代单碱基编辑系统(BE1),评估4种不同来源的胞嘧啶脱氨酶(hAID、hAPOBEC3G、rAPOBEC1和pmCDA1)的单链DNA(single-stranded DNA, ssDNA)脱氨效果,其中的来自鼠源的rAPOBEC1活性最高。将rAPOBEC1融合到dCas9的N端,当dCas9和sgRNA形成的复合物与目标DNA结合时,打开DNA双链结构,由rAPOBEC1连接的胞苷脱氨基可将单链目标C∶U转化为G∶U异源双链,在DNA复制或DNA修复后可永久转化为A∶T。BE1有效脱氨窗口约为5个碱基(nt),通常在原型间隔序列毗邻基序(protospacer adjacent motif, PAM)上游-13~-17位,在体内碱基编辑的效率为总DNA序列的0.8%~7.7%。与体外碱基编辑相比,效率降低了5~36倍,推测细胞内碱基编辑效率大幅下降的原因可能是尿嘧啶DNA糖基化酶(uracil DNA glycosylase, UDG)催化细胞DNA中U的去除并启动碱基切除修复(base-excision repair, BER),最常见的结果是U∶G逆转为C∶G。之后发现尿嘧啶DNA糖基化酶抑制剂(uracil DNA glycosylase inhibitor, UGI)是枯草杆菌噬菌体PBS1的残基蛋白,能有效地阻断UDG活性,将UGI融合到BE1的C末端,创建了二代碱基编辑器BE2(rAPOBEC1-XTEN-dCas9-UGI),发现BE2在人类细胞中的编辑效率平均比BE1高3倍。为进一步提高碱基编辑效率,他们恢复了BE2中Cas9HNH结构域的第840位催化的组氨酸残基,从而产生切割活性,同时还保留UGI以抑制BER,最终形成第三代碱基编辑器[BE3, rAPOBEC1-XTEN-dCas9(A840H)-UGI],BE3保留Cas9中的D10A突变,防止dsDNA切割,编辑效率相比于BE2提高了2~6倍。

a-CBE系统;b-ABE系统;c-GBE系统;d-PE系统;e-DBE系统
图1 碱基编辑器原理示意图
Fig.1 Schematic diagram of the principle of the base editor
和BE1系统类似,同年,NISHIDA等[12]将七鳃鳗来源的脱氨酶PmCDA1与来自化脓性链球菌的dCas9或nCas9进行融合,开发出新的CBE系统——Target-AID。由于脱氨酶的来源不同,两种碱基编辑器的编辑窗口和编辑效率也不同,Target-AID的编辑窗口为PAM上游-15~-19位,在Target-AID复合体中,发现nCas9-AID比dCas9-AID复合体编辑效率略高,同时加入UGI,阻断尿嘧啶碱基切除修复途径的初始步骤,在哺乳动物细胞中的编辑效率可达到15%~55%。基于BE1~BE3和Target-AID这2种碱基编辑器在生物体的成功应用,研究者通过设计多种Cas9突变体或者利用Cas9直系同源物,对脱氨酶的结构域进行改造,增加UGI或核定位信号(nuclear localization signal, NLS)的数量等一系列优化,开发出不同的CBE系统[13](表1)。
表1 CBE主要结构、编辑窗口和编辑效率
Table 1 The main structure, editing window and editing efficiency of CBE
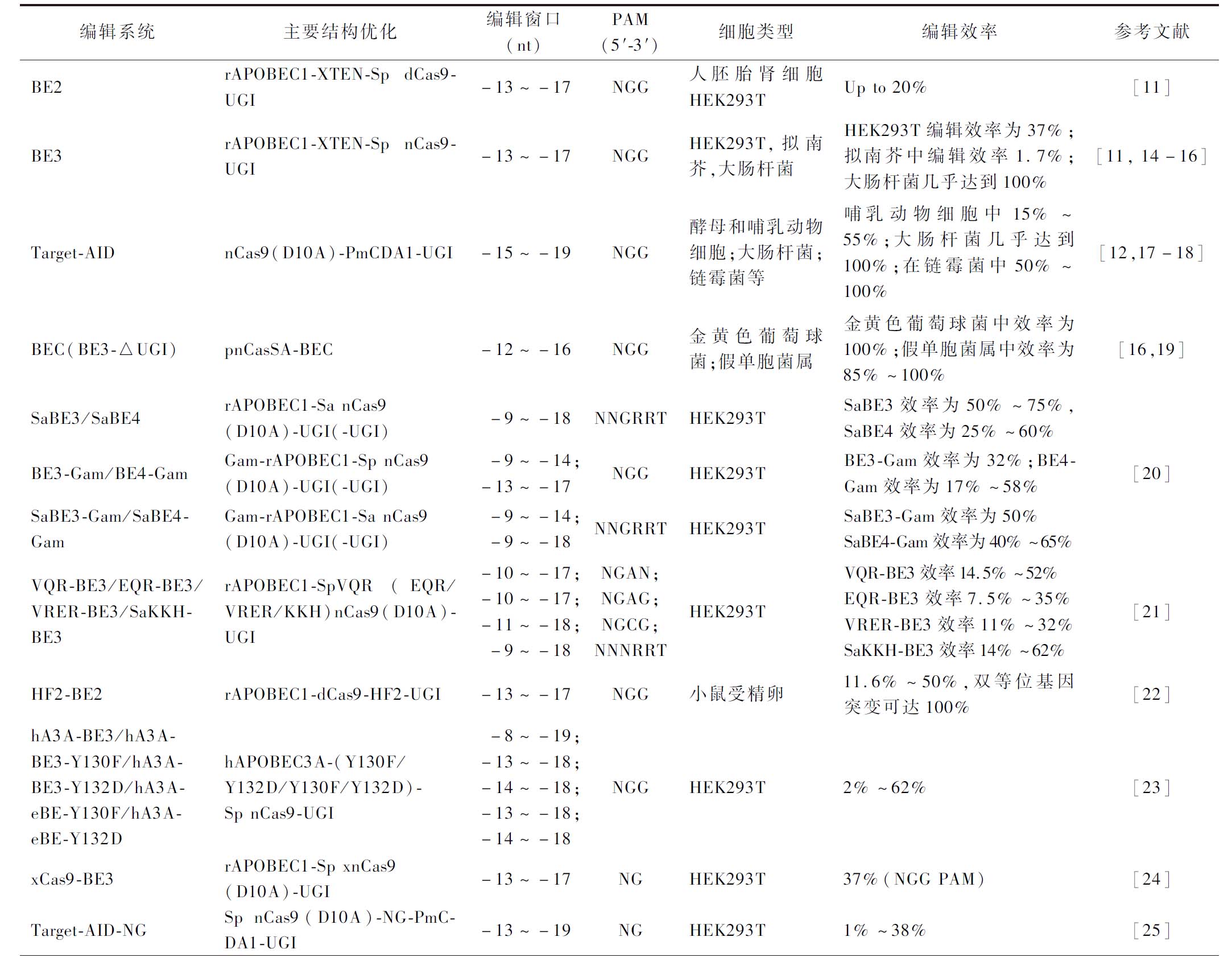
续表1
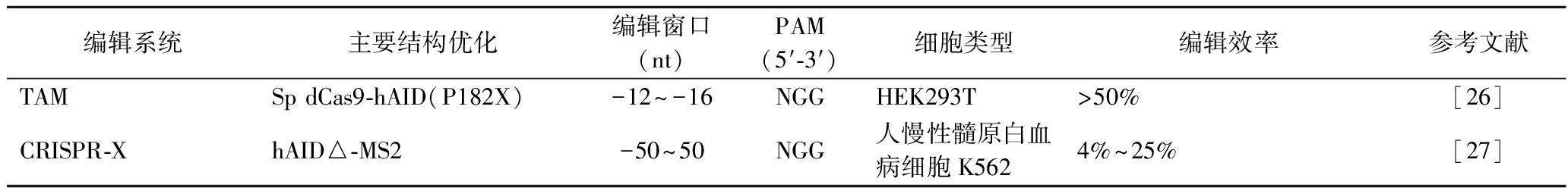
编辑系统主要结构优化编辑窗口(nt)PAM(5′-3′)细胞类型编辑效率参考文献TAMSp dCas9-hAID(P182X)-12~-16NGGHEK293T>50%[26]CRISPR-XhAID△-MS2-50~50NGG人慢性髓原白血病细胞K5624%~25%[27]
1.2 ABE编辑器
ABE技术原理与CBE相似,腺嘌呤脱氨酶与nCas9或dCas9组成融合蛋白,通过sgRNA引导融合蛋白到特定靶点,使非靶链的腺苷脱氨,暴露为ssDNA,不同的是腺嘌呤脱氨酶将目标序列的A脱氨之后生成一个次黄嘌呤(inosine, I),在接下来的DNA复制和修复过程中I被识别为G,进而与对应的C配对,最终实现A-to-G的转换(图1-b)。与CBE相比,ABE不需要UGI,可能是因为DNA中的I不能被任何已知的哺乳动物DNA糖基化酶有效去除。
2017年,GAUDELLI等[28]开发出在基因组DNA中介导A∶T转化为G∶C的腺嘌呤碱基编辑器。首先用天然腺嘌呤脱氨酶(大肠杆菌TadA、人源ADAR2、小鼠ADA和人源ADAT2)替换BE3的rAPOBEC1成分,发现nCas9与脱氨酶融合质粒与相应的sgRNA共转入HEK293T细胞,没有发生A∶T到G∶C编辑,说明天然腺嘌呤脱氨酶无法直接使用。作者发现TadA是一种tRNA腺嘌呤脱氨酶,可将tRNAArg的单链反密码子环中的腺嘌呤转化为I。大肠杆菌TadA与CBE编辑器中使用的rAPOBEC1具有同源性,并且TadA催化的脱氨基反应不需要小分子激活剂,因此选择大肠杆菌TadA作为DNA腺嘌呤脱氨酶的改造对象。在氯霉素选择下,发现TadA D108处或附近的突变使TadA能够在DNA底物上进行A脱氨,将突变后的TadA*(TadA A106V和D108 N)与nCas9进行融合,并在C端添加一个NLS,构建出了第一代腺嘌呤碱基编辑器ABE1.2(TadA*-XTEN-nCas9-NLS)。之后进行7轮的筛选和进化,得到目前具有广泛序列兼容性的高活性碱基编辑器ABE7.10(TadA-TadA*-nCas9),对HEK293T细胞中6个基因组位点测试,平均编辑效率为(58±4.0)%,是ABE1.2的29倍,脱靶效率<0.1%,编辑活性窗口可覆盖sgRNA的第4~9位。
在Gaudelli的基础上,越来越多的ABE版本被开发出来[29-30](表2),然而腺嘌呤脱氨酶与SpCas9以外的Cas同源物的亲和性有限,限制了ABE的应用。因此,RICHTER等[31]利用噬菌体辅助的非连续和连续进化(PANCE和PACE)对ABE7.10的脱氨酶组分进行修改,产生了ABE8e(TadA-TadA*-Sp nCas9)。通过在TadA-8e结构域中引入8个额外的突变,使得Cas9依赖性和非依赖性DNA脱靶编辑以及转录组范围内RNA脱靶都得到改善,活性是ABE7.10的590倍,编辑效率为18%~86%,增强了ABE的有效性和适用性。HUA等[32]用VQR-Cas9、VRER-Cas9和SaKKH-Cas9来替换碱基编辑器中SpCas9,开发了新的腺嘌呤碱基编辑器ABE-P3、ABE-P4和ABE-P5,VQR-Cas9可以识别NGA PAM序列,VRER-Cas9可以接受NGCG PAM序列,SaKKHCas9可以诱导含有NNGRRT和NNNRRT PAM序列区域的编辑,增大了在水稻基因组中的靶向位点。
表2 ABE主要结构、编辑窗口和编辑效率
Table 2 The main structure, editing window and editing efficiency of ABE
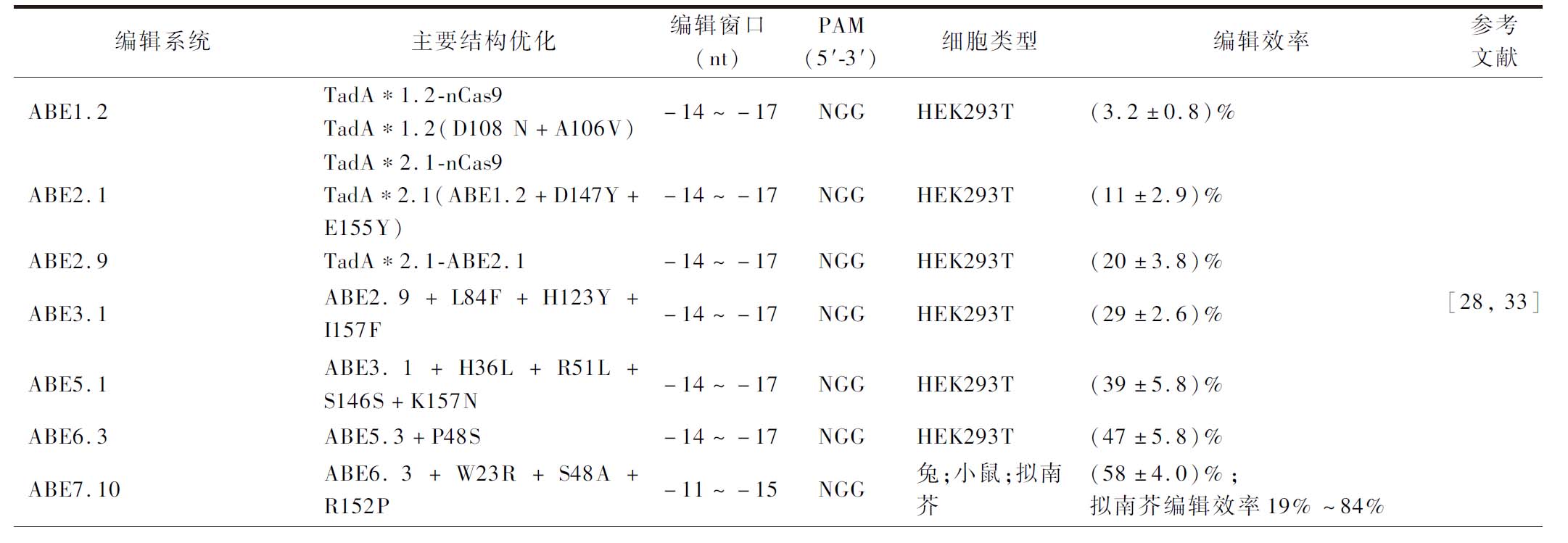
续表2

编辑系统主要结构优化编辑窗口(nt)PAM(5′-3′)细胞类型编辑效率参考文献ABE8eTadA-TadA∗-Sp nCas9-13~-17NGGHEK293T18%~86%[31]pABEABE7.10-spCas9D10A-13~-17NGG大肠杆菌,金黄色葡萄球菌大肠杆菌效率71%;金黄色葡萄球菌编辑效率50%~100%[34]CRISPR-aBESTecTadA-Cas9n(D10A)-12~-17NGG链霉菌属0%~40%[35]xABETadA-TadA∗-Sp xCas9(D10A)-14~-17NGHEK293T69%(NGG PAM)[24]PABE-7TadA-TadA∗-nCas9-14~-17NGG水稻;小麦59.1%[36]ABE-P2(ABEsa)/ABE-P3(VQR-ABE)/ABE-P4(VRER-ABE)/ABE-P5(SaKKH-ABE)TadA-TadA∗-Sa nCas9(VQR nCas9/VRER nCas9/SaKKH nCas9)-9~-15;-11~-18;-13~-17;-13~-17NNGRRT;NGA;NGCG;NNNNGT水稻ABE-P2(ABEsa)编辑效率25%~45%ABE-P3(VQR-ABE)编辑效率30%~74.3%ABE-P4(VRER-ABE)编辑效率2.6%ABE-P5(SaKKH-ABE)编辑效率6.5%[32]zABE7.10zTadA-zTadA∗-znCas9-14~-16NGG斑马鱼15%~25%[37]ABEmaxTadA-TadA ∗-Sp nCas9-11~-15NGGHEK293T27%~52%[38]VQR-ABE/SaKKH-Cas9TadA-TadA∗-nVQRCas9(Sa KKH nCas9)-10~-16;-9~-15NGA;NNGRRT小鼠VQR-ABE编辑效率20%SaKKH-Cas9编辑效率16%[39]
1.3 GBE编辑器
2020年之前,BE只能催化嘧啶和嘧啶、嘌呤和嘌呤(C-to-T和A-to-G)之间的转换,不能产生嘧啶和嘌呤之间的颠换。此外,传统的CBE在脱氨酶AID的作用下,将DNA单链上的C转变为U,经过多次DNA复制才转化为T。针对上述问题,ZHAO等[8]研发出新型碱基编辑器GBE1.0,能够在大肠杆菌中引起C-to-T转换和在哺乳动物细胞中引起C-to-G转换。GBE1.0技术原理是由nCas9、胞嘧啶脱氨酶AID和大肠杆菌中尿嘧啶糖基转移酶(Escherichia coli-derived uracil DNA N-glycosylase, eUNG)组成,eUNG-nCas9-AID特异性地与靶DNA结合,胞嘧啶脱氨酶脱氨产生U,eUNG切除脱氨酶产生的U碱基并形成启动DNA修复过程的无嘌呤/脱嘧啶(apurinic/apyrimidinic site, AP)位点,在DNA修复下形成C-to-A转换(图1-c)。为在哺乳动物细胞中使用,用大鼠APOBEC1替换AID,构建了APOBEC1-nCas9-eUNG,此GBE1.0可以在间隔区的第6位进行C-to-G颠换。
KURT等[40]开发出C-to-G碱基转换的CGBE1和miniCGBE1碱基编辑器,用大鼠胞嘧啶脱氨酶变体APOBEC1(R33A)进一步优化GBE系统。CGBE1由nCas9、eUNG和APOBEC1(R33A)组成,miniCGBE1在CGBE1基础上去掉了eUNG结构域。CGBE1和miniCGBE1不但可有效诱导目标C-to-G碱基转换,还降低不需要的C-to-W(W=A或T)和Indel突变的水平。为提高GBE1.0编辑效率,SUN等[41]构建GBE2.0[APOBEC(R33A)-nCas9-Rad51-UNG1],用酿酒酵母UNG1代替GBE1.0中eUNG,保留GBE1.0对脱氨酶突变;Rad51是参与DNA修复的关键蛋白,可增加核苷酸脱氨酶与ssDNA底物之间的亲和力,GBE2.0通过融合Rad51提高了GBE编辑效率。哺乳动物细胞中GBE2.0编辑效率为8.70%~72.1%。DONG等[42]将VP64(反式激活因子transactivation modules,TM)融合到GBE中,在HEK293T细胞中Vp64-GBE比GBE1.0表现出更高的效率,VP64作为反式激活因子,可促进凝聚染色质的重塑和展开,增加CGBE对目标位点的靶向性,将编辑效率提高到31.03%;作者还将SunTag系统(由多个GCN4肽和单链可变片段scFv抗体组成的标记系统SunTag,被用来扩大CBE的编辑窗口)与GBE融合构建了SunTag-GBEs,GBE1.0主要诱导哺乳动物细胞目标区域C6位点的C-to-G颠换,而SunTag-GBEs将GBE1.0靶向范围扩展到C6-C8,平均编辑效率为30.88%;进一步将SpRY-Cas9变体与和Vp64-GBE进行融合,构建的Vp64-SpRY-GBE在非NGG PAM的基因组位点上平均编辑效率为11%,成功地扩展了GBE靶向范围(表3)。
表3 GBE主要结构、编辑窗口和编辑效率
Table 3 The main structure, editing window and editing efficiency of GBE
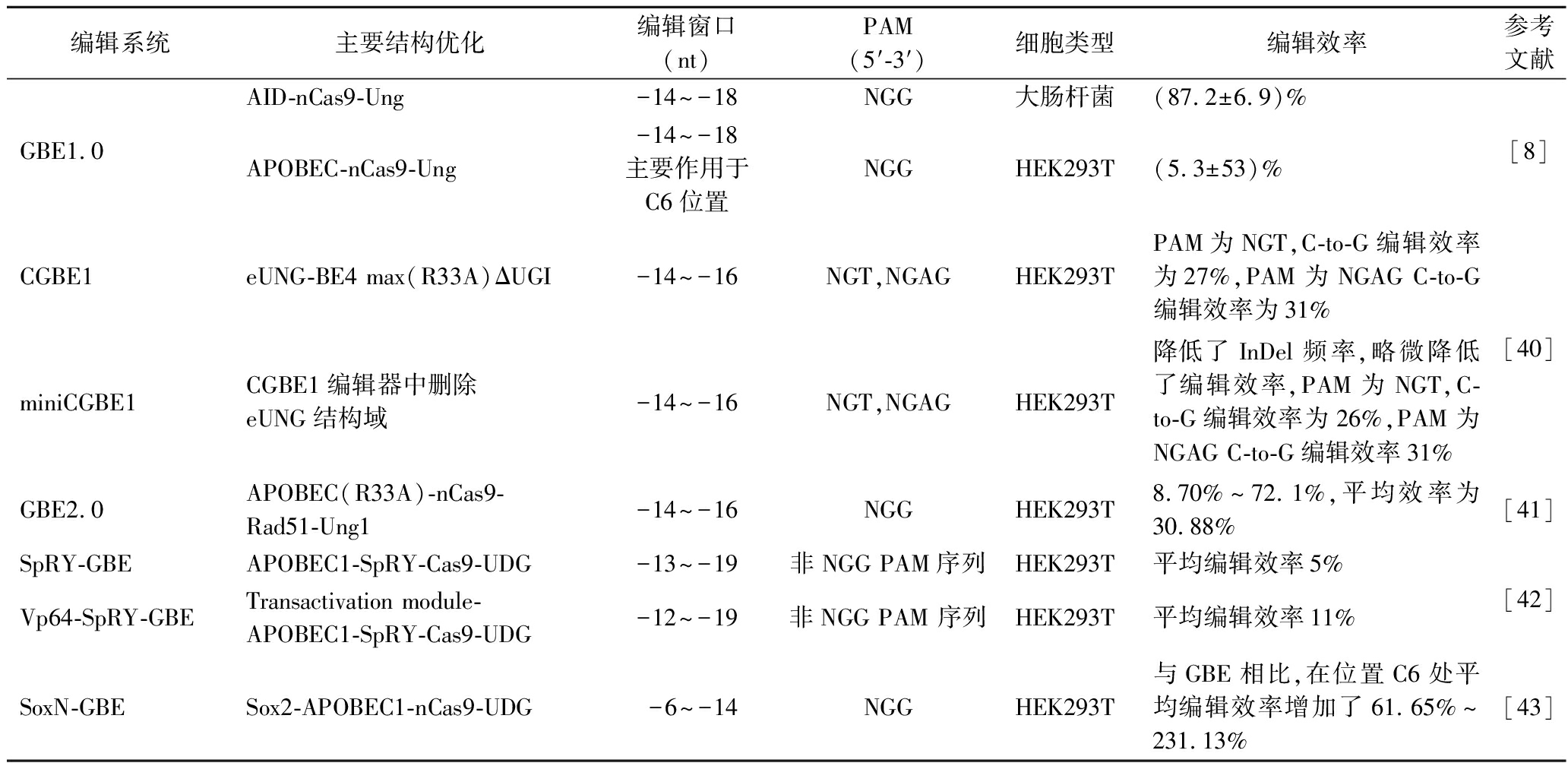
编辑系统主要结构优化编辑窗口(nt)PAM(5′-3′)细胞类型编辑效率参考文献GBE1.0AID-nCas9-Ung-14~-18NGG大肠杆菌(87.2±6.9)%APOBEC-nCas9-Ung-14~-18主要作用于C6位置NGGHEK293T(5.3±53)%[8]CGBE1eUNG-BE4 max(R33A)ΔUGI-14~-16NGT,NGAGHEK293TPAM为NGT,C-to-G编辑效率为27%,PAM为NGAG C-to-G编辑效率为31%miniCGBE1CGBE1编辑器中删除eUNG结构域-14~-16NGT,NGAGHEK293T降低了InDel频率,略微降低了编辑效率,PAM为NGT,C-to-G编辑效率为26%,PAM为NGAG C-to-G编辑效率31%[40]GBE2.0APOBEC(R33A)-nCas9-Rad51-Ung1-14~-16NGGHEK293T8.70%~72.1%,平均效率为30.88%[41]SpRY-GBEAPOBEC1-SpRY-Cas9-UDG-13~-19非NGG PAM序列HEK293T平均编辑效率5%Vp64-SpRY-GBETransactivation module-APOBEC1-SpRY-Cas9-UDG-12~-19非NGG PAM 序列HEK293T平均编辑效率11%[42]SoxN-GBESox2-APOBEC1-nCas9-UDG-6~-14NGGHEK293T与GBE相比,在位置C6处平均编辑效率增加了61.65%~231.13%[43]
1.4 PE编辑器
CBE和ABE可以实现4种突变(C-to-T、G-to-A、A-to-G和T-to-C),GBE可以实现C-to-G和C-to-A的转换,但这些基础编辑器仅限于单个碱基替换,只能实现某些核苷酸的改变,不能进行小片段的精准插入删除,也缺乏高效编辑工具。ANZALONE等[9]开发出第一代Prime编辑器PE1.0,与其他报道的碱基编辑器相比,PE更加通用和更大的灵活性,能够提供其余6种类型的碱基转换和小片段碱基的插入和删除。
PE在人类细胞中介导靶向插入、缺失,实现所有12种可能的碱基到碱基转换及其组合,无需DSB和供体DNA模板。PE1.0编辑系统主要由nCas9(H840A)与工程化改造的逆转录酶(reverse transcriptase, RT)组成的融合蛋白、PBS(primer binding site, PBS)序列和RT模板(RT template, RTT)序列的pegRNA(prime editing guide RNA)组成。pegRNA引导nCas9蛋白结合到目标位点对DNA进行切割,暴露出ssDNA,PBS序列与暴露的ssDNA互补结合,在RT酶作用下进行逆转录,该过程以RTT为模板,RTT序列信息被逆转录形成新的ssDNA,在DNA断裂处出现3′-flap和5′-flapDNA单链结构的动态转换,但细胞修复机制会倾向切除5′-flap,而3′-flap在DNA连接酶作用下被整合到基因组,最终实现目标基因组碱基的变化(图1-d)。
研究者通过增加RT热稳定性、对Cas蛋白元件、pegRNA结构和靶向范围等方面对PE进行优化,使其效率得到了极大提升[44-45](表4)。ANZALONE团队[9]对PE1.0中的RT进行蛋白质工程化改造,在PE1.0基础上对5个氨基酸残基突变,得到PE2.0 RT[(nCas9(H840A)-M-MLV RT(D200 N/L603 W/T330P/T306K/W313F)],PE2.0点突变效率是PE1.0的1.6~5.1倍,并且能够与更短的PBS序列兼容。然而,PE2.0在DNA错配修复和连接过程中,含有PAM的ssDNA链上整合编辑有50%机率被切断,使编辑效率受到影响。因此,构建了第三代PE3.0,包含一个额外的sgRNA来切割未编辑的ssDNA链,以最小化DSB,PE3.0系统在HEK293T细胞中的编辑效率比PE2.0又提高1.5~4.2倍。为最大限度地减少Indel,设计带有间隔区的sgRNAs,使其与编辑过的ssDNA链匹配,但与原始等位基因不匹配,构建得到PE3b系统,在HEK293T细胞中编辑效率可达20%~50%,目标位点InDel的突变率仅为0.74%。CHEN等[46]研究参与DNA修复过程的476个基因对PE2.0编辑结果的影响,发现DNA错配修复(mismatch repair, MMR)基因显著影响PE编辑效率,并促进Indel的形成。经过筛选,发现MSH2、MSH6、MLH1和PMS2是影响MMR复合物的编码基因,敲除这些基因使PE2.0的编辑效率提高了2.3倍。作者还将功能缺失的MLH1蛋白变体(MLH1 dn)与PE2.0融合形成PE4.0(PE2+MLH1 dn),与PE3.0融合形成PE5.0(PE3+MLH1 dn),降低了MLH1对目标位点的修复识别,PE4.0比PE2.0、PE5.0比PE3.0的编辑效率分别提高了7.7倍和2.0倍。PE碱基编辑器在有丝分裂后小鼠皮层神经元、人诱导多能干细胞和小鼠胚胎细胞等多种哺乳动物细胞、水稻和小麦等单子叶植物及少数微生物中都已成功应用[47-48]。LU等[49]将哺乳动物PE系统整合到植物双元载体中,生成pCXPE01编辑器,对番茄进行精确的基因组修饰。除在动植物中的测试外,2021年TONG等[50]将PE编辑器用于大肠杆菌,发现PE可在大肠杆菌进行单碱基对替换,在最佳条件下1 bp缺失的效率高达40%。此外,使用该工具在大肠杆菌基因组中成功删除97 bp和插入33 bp,为开发其他微生物PE系统提供借鉴。
表4 PE主要结构、编辑窗口和编辑效率
Table 4 The main structure, editing window and editing efficiency of PE
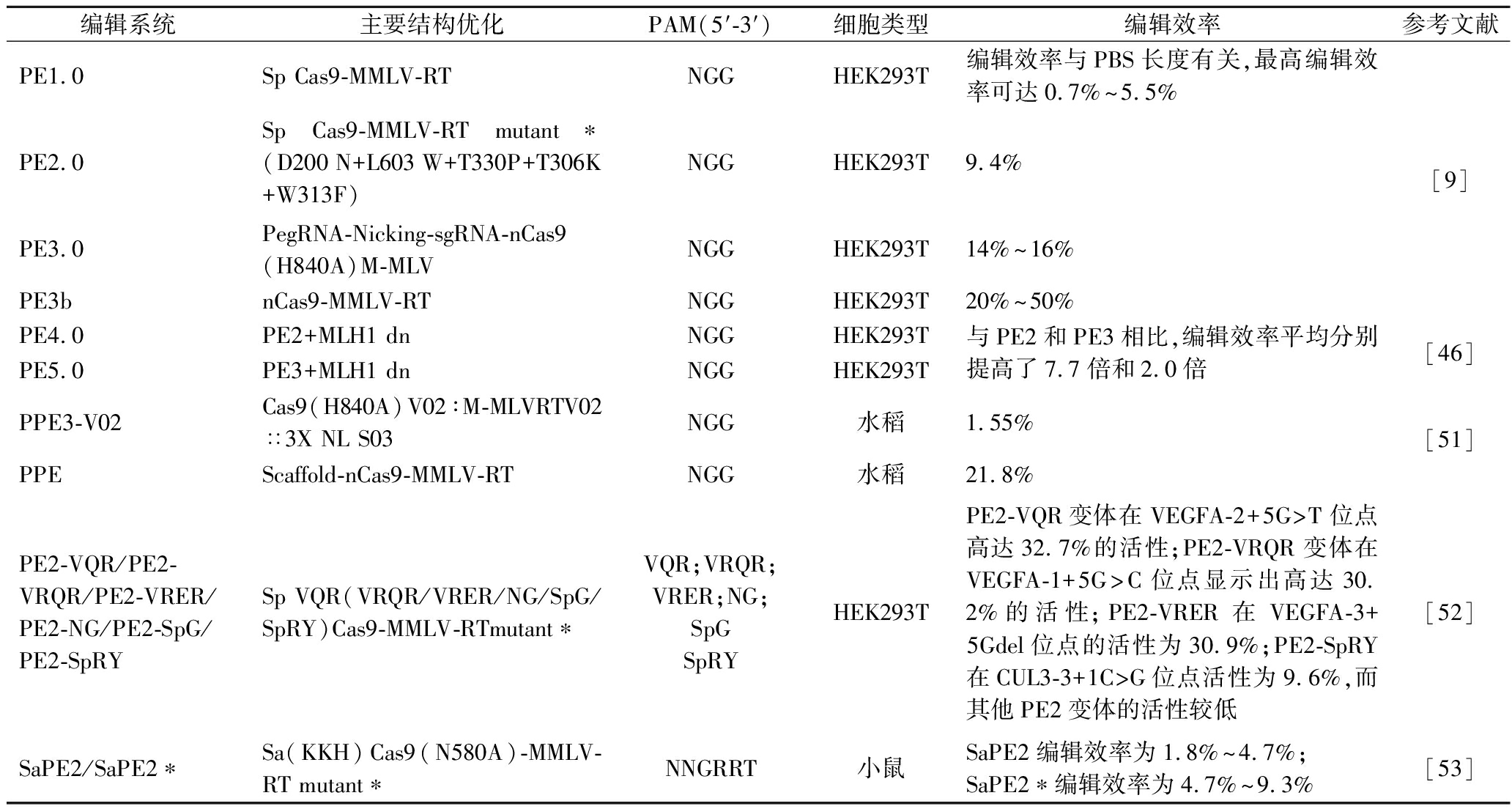
编辑系统主要结构优化PAM(5′-3′)细胞类型编辑效率参考文献PE1.0Sp Cas9-MMLV-RTNGGHEK293T编辑效率与PBS长度有关,最高编辑效率可达0.7%~5.5%PE2.0Sp Cas9-MMLV-RT mutant∗(D200 N+L603 W+T330P+T306K+W313F)NGGHEK293T9.4%PE3.0PegRNA-Nicking-sgRNA-nCas9(H840A)M-MLVNGGHEK293T14%~16%PE3bnCas9-MMLV-RTNGGHEK293T20%~50%[9]PE4.0PE2+MLH1 dnNGGHEK293TPE5.0PE3+MLH1 dnNGGHEK293T与PE2和PE3相比,编辑效率平均分别提高了7.7倍和2.0倍[46]PPE3-V02Cas9(H840A)V02∶M-MLVRTV02∷3X NL S03NGG水稻1.55%PPEScaffold-nCas9-MMLV-RTNGG水稻21.8%[51]PE2-VQR/PE2-VRQR/PE2-VRER/PE2-NG/PE2-SpG/PE2-SpRYSp VQR(VRQR/VRER/NG/SpG/SpRY)Cas9-MMLV-RTmutant∗VQR;VRQR;VRER;NG;SpGSpRYHEK293TPE2-VQR变体在VEGFA-2+5G>T位点高达32.7%的活性;PE2-VRQR变体在VEGFA-1+5G>C位点显示出高达30.2%的活性;PE2-VRER在VEGFA-3+5Gdel位点的活性为30.9%;PE2-SpRY在CUL3-3+1C>G位点活性为9.6%,而其他PE2变体的活性较低[52]SaPE2/SaPE2∗Sa(KKH)Cas9(N580A)-MMLV-RT mutant∗NNGRRT小鼠SaPE2编辑效率为1.8%~4.7%;SaPE2∗编辑效率为4.7%~9.3%[53]
1.5 DBE编辑器
CBE和ABE可以实现C-to-T和A-to-G的转换,GBE可以实现C-to-G和C-to-A的颠换,但都只催化单一类型的核苷酸转化,这限制了产物的多样性。PE可用于转换任何核苷酸或同时修饰多个碱基,可在更广泛的区域诱导碱基替换,但PE编辑效率远低于CBE和ABE,导致PE难以广泛应用。2020年,ZHANG等[10]开发出双碱基编辑器(A&C-BEmax),将rAPOBEC1、TadA与nCas9融合,可在同一位点诱导同时发生C-to-T和A-to-G的转换。A&C-BEmax在同一等位基因A/C同时突变率为2%~30%,C-to-T的编辑效率5.3%~82.6%,在腺嘌呤上的活性略有降低,A-to-G的编辑效率为0.2%~10%。此外,与ABEmax相比,A&C-BEmax C-to-T编辑窗口扩展到-4~-19nt,A-to-G编辑窗口扩展到-12~-18nt,还降低RNA脱靶活性(表5)。
表5 DBE主要结构、编辑窗口和编辑效率
Table 5 The main structure, editing window and editing efficiency of DBE
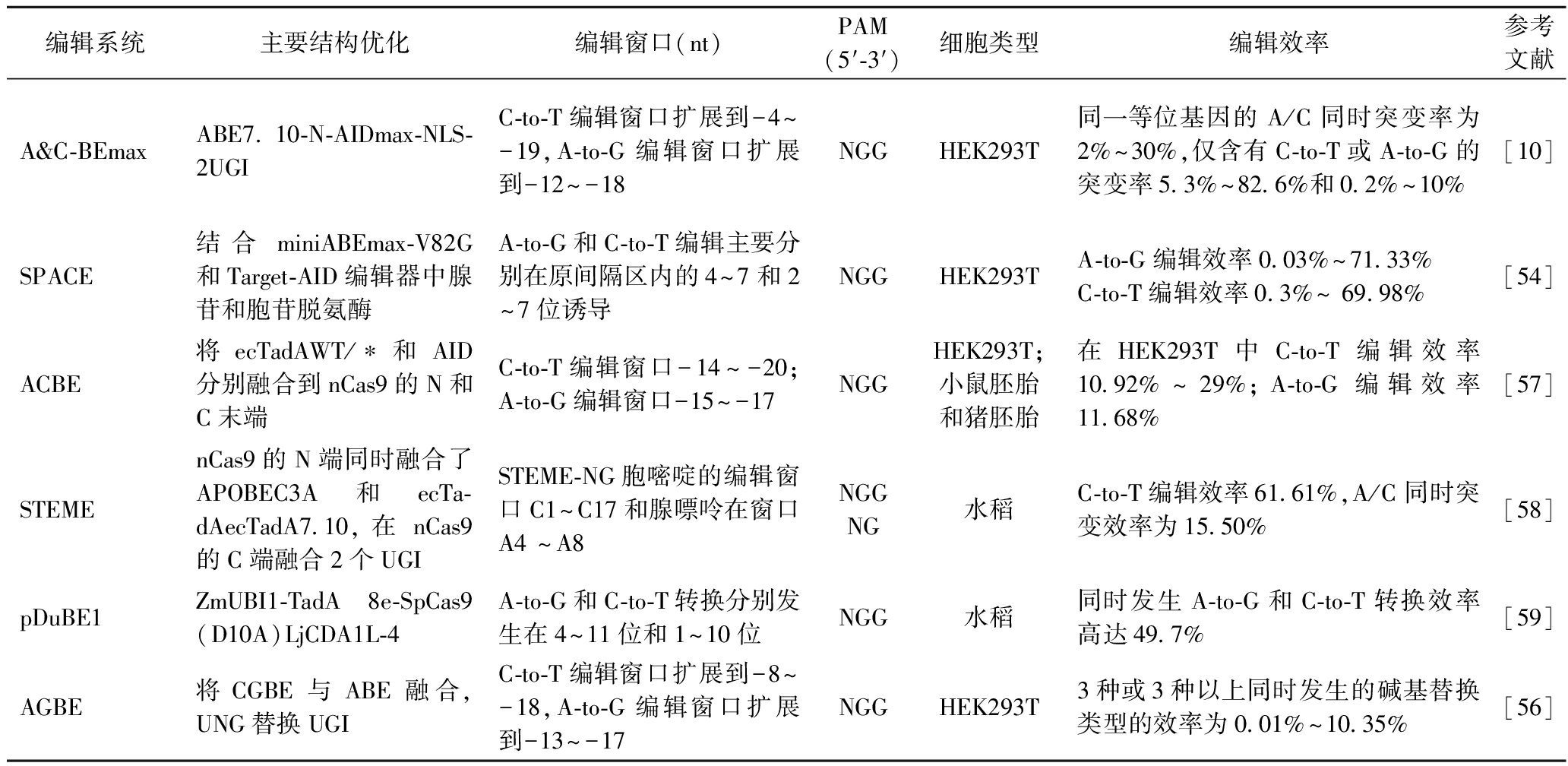
编辑系统主要结构优化编辑窗口(nt)PAM(5′-3′)细胞类型编辑效率参考文献A&C-BEmaxABE7.10-N-AIDmax-NLS-2UGIC-to-T编辑窗口扩展到-4~-19,A-to-G编辑窗口扩展到-12~-18NGGHEK293T同一等位基因的A/C同时突变率为2%~30%,仅含有C-to-T或A-to-G的突变率5.3%~82.6%和0.2%~10%[10]SPACE结合miniABEmax-V82G和Target-AID编辑器中腺苷和胞苷脱氨酶A-to-G和C-to-T编辑主要分别在原间隔区内的4~7和2~7位诱导NGGHEK293TA-to-G编辑效率0.03%~71.33%C-to-T编辑效率0.3%~ 69.98%[54]ACBE将ecTadAWT/∗和AID分别融合到nCas9的N和C末端C-to-T编辑窗口-14~-20;A-to-G编辑窗口-15~-17NGGHEK293T;小鼠胚胎和猪胚胎在HEK293T中C-to-T编辑效率10.92%~29%;A-to-G编辑效率 11.68%[57]STEMEnCas9的N端同时融合了APOBEC3A和ecTa-dAecTadA7.10,在nCas9的C端融合2个UGISTEME-NG胞嘧啶的编辑窗口C1~C17和腺嘌呤在窗口A4 ~A8NGGNG水稻C-to-T编辑效率61.61%,A/C同时突变效率为15.50%[58]pDuBE1ZmUBI1-TadA 8e-SpCas9 (D10A)LjCDA1L-4A-to-G和C-to-T转换分别发生在4~11位和1~10位NGG水稻同时发生A-to-G和C-to-T转换效率高达49.7%[59]AGBE将CGBE与ABE融合,UNG替换UGIC-to-T编辑窗口扩展到-8~-18,A-to-G编辑窗口扩展到-13~-17NGGHEK293T3种或3种以上同时发生的碱基替换类型的效率为0.01%~10.35%[56]
DBE系统都是由nCas9与胞嘧啶脱氨酶和腺嘌呤脱氨酶融合而成,以A&C-BEmax为例[10],将2种脱氨酶和nCas9的组合生成了5种结构,经测试发现TadA仅在与nCas9相邻的N端融合时起作用,与ABE7.10的N端融合的胞嘧啶脱氨酶在同一等位基因中诱导了同步A/C显著水平突变。胞嘧啶脱氨酶与nCas9融合时活性显著增加,其中hAID比rAPOBEC1表现出更高的活性(图1-e)。通过优化密码子、增加NLS和添加2个UGI等一系列优化,显著提高A&C-BEmax的碱基转换效率和A/C同时转换活性。
GRÜNEWALD等[54]开发出能够有效地同时引入A-to-G和C-to-T碱基编辑器SPACE,增加了60个现有单碱基编辑工具CBE和ABE无法创建的额外密码子更改。SPACE可用于创建或恢复多核苷酸变体(MNVs),与单一脱氨酶碱基编辑器相比,SPACE产生的突变组合更加多样,使其在饱和诱变筛选、定向进化和蛋白质工程具有应用前景。SAKATA等[55]将PmCDA1、TadA和nCas9结合开发出Target-ACEmax碱基编辑器。在47个基因组靶标上显示出较高的同时编辑C-to-T和A-to-G活性,在哺乳动物中C-to-T编辑效率高达50%,A-to-G编辑效率为40%。LIANG等[56]报道一种新型双脱氨酶介导的碱基编辑器AGBE,将糖基化酶碱基编辑器CGBE与腺嘌呤碱基编辑器ABE融合,UGI被UNG取代,不仅实现同一目标位点同时产生C-to-T和A-to-G的转化,而且有效诱导靶向C-to-G碱基颠换以及C-to-T和C-to-A转换,从而增加了目标位点DNA链中变化的多样性(表5)。
2 总结与展望
碱基编辑器通过更换脱氨酶元件、改变sgRNA长度和设计多种Cas9突变体,使编辑效率、目标范围和编辑产品的纯度都取得显著效果,但碱基编辑器仍存在以下问题:(a)脱靶问题,脱靶现象是所有基因组编辑工具的普遍缺点,碱基编辑器可能会导致大量无法预测的脱靶突变[60];(b)普适性问题,特别是PE在多倍体物种和普通小麦等农业粮食作物中没有广泛应用,甚至不能应用[61];(c)递送效率和编辑稳定性问题,目前碱基编辑器主要通过质粒形式来实现编辑,但递送效率较低。虽然碱基编辑器质粒能在细胞中高效表达和实现有效编辑,但在细胞中长期表达存在脱靶和编辑效率低等问题。如何克服上述问题成为碱基编辑器今后研究的重点。
碱基编辑器在动植物和微生物中具有广阔的应用前景,未来(a)可进一步扩大编辑窗口的宽度、扩大同时编辑的基本类型和提高编辑效率;(b)基于碱基编辑器开发出更强大的人工定向编辑工具,在指定的目标DNA区域内随机生成大量点突变,筛选出所需的有益突变;(c)通过结合密码子扩展等新技术,将碱基编辑工具应用于基因组工程、蛋白质进化和非规范氨基酸生产等需求。碱基编辑器的出现仅8年时间,但其基础和应用研究发展迅猛。随着碱基编辑器不断的优化和工程化设计,我们有理由相信碱基编辑器将成为食品科学和生命科学等领域的高效精准基因组编辑工具。
[1] LIANG Y H, CHEN F B, WANG K P, et al.Base editors:Development and applications in biomedicine[J].Frontiers of Medicine, 2023, 17(3):359-387.
[2] ZHOU L, PENG R X, ZHANG R, et al.The applications of CRISPR/Cas system in molecular detection[J].Journal of Cellular and Molecular Medicine, 2018, 22(12):5807-5815.
[3] CHO S, SHIN J, CHO B K.Applications of CRISPR/cas system to bacterial metabolic engineering[J].International Journal of Molecular Sciences, 2018, 19(4):1089.
[4] WANG Y, LIU Y, ZHENG P, et al.Microbial base editing:A powerful emerging technology for microbial genome engineering[J].Trends in Biotechnology, 2021, 39(2):165-180.
[5] 余传照, 莫健新, 赵鑫, 等.基于CRISPR/Cas系统的DNA碱基编辑技术及其在生物医学和农业中的应用[J].生物工程学报, 2021, 37(9):3071-3087.
YU C Z, MO J X, ZHAO X, et al.CRISPR/Cas-mediated DNA base editing technology and its application in biomedicine and agriculture[J].Chinese Journal of Biotechnology, 2021, 37(9):3071-3087.
[6] WU W Y, YANG Y H, LEI H T.Progress in the application of CRISPR From gene to base editing[J].Medicinal Research Reviews, 2019, 39(2):665-683.
[7] KIM J S.Precision genome engineering through adenine and cytosine base editing[J].Nature Plants, 2018, 4(3):148-151.
[8] ZHAO D D, LI J, LI S W, et al.Glycosylase base editors enable C-to-A and C-to-G base changes[J].Nature Biotechnology, 2021, 39(1):35-40.
[9] ANZALONE A V, RANDOLPH P B, DAVIS J R, et al.Search-and-replace genome editing without double-strand breaks or donor DNA[J].Nature, 2019, 576(7785):149-157.
[10] ZHANG X H, ZHU B Y, CHEN L, et al.Dual base editor catalyzes both cytosine and adenine base conversions in human cells[J].Nature Biotechnology, 2020, 38(7):856-860.
[11] KOMOR A C, KIM Y B, PACKER M S, et al.Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage[J].Nature, 2016, 533(7603):420-424.
[12] NISHIDA K, ARAZOE T, YACHIE N, et al.Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems[J].Science, 2016, 353(6305):aaf8729.
[13] ZENG H Z, YUAN Q C, PENG F, et al.A split and inducible adenine base editor for precise in vivo base editing[J].Nature Communications, 2023, 14(1):5573.[PubMed]
[14] CHEN Y Y, WANG Z P, NI H W, et al.CRISPR/Cas9-mediated base-editing system efficiently generates gain-of-function mutations in Arabidopsis[J].Science China.Life Sciences, 2017, 60(5):520-523.
[15] ZHENG K, WANG Y, LI N, et al.Highly efficient base editing in bacteria using a Cas9-cytidine deaminase fusion[J].Communications Biology, 2018, 1:32.
[16] GU T N, ZHAO S Q, PI Y S, et al.Highly efficient base editing in Staphylococcus aureus using an engineered CRISPR RNA-guided cytidine deaminase[J].Chemical Science, 2018, 9(12):3248-3253.
[17] BANNO S, NISHIDA K, ARAZOE T, et al.Deaminase-mediated multiplex genome editing in Escherichia coli[J].Nature Microbiology, 2018, 3(4):423-429.
[18] ZHAO Y W, TIAN J Z, ZHENG G S, et al.Multiplex genome editing using a dCas9-cytidine deaminase fusion in Streptomyces[J].Science China Life Sciences, 2020, 63(7):1053-1062.
[19] CHEN W Z, ZHANG Y, ZHANG Y F, et al.CRISPR/Cas9-based Genome Editing in Pseudomonas aeruginosa and Cytidine Deaminase-Mediated Base Editing in Pseudomonas Species[J].iScience, 2018, 6:222-231.
[20] KOMOR A C, ZHAO K T, PACKER M S, et al.Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C:G-to-T:A base editors with higher efficiency and product purity[J].Science Advances, 2017, 3(8):eaao4774.
[21] KIM Y B, KOMOR A C, LEVY J M, et al.Increasing the genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions[J].Nature Biotechnology, 2017, 35(4):371-376.
[22] LIANG P P, SUN H W, SUN Y, et al.Effective gene editing by high-fidelity base editor 2 in mouse zygotes[J].Protein &Cell, 2017, 8(8):601-611.
[23] WANG X, LI J N, WANG Y, et al.Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion[J].Nature Biotechnology, 2018, 36(10):946-949.
[24] HU J H, MILLER S M, GEURTS M H, et al.Evolved Cas9 variants with broad PAM compatibility and high DNA specificity[J].Nature, 2018, 556(7699):57-63.
[25] NISHIMASU H, SHI X, ISHIGURO S, et al.Engineered CRISPR-Cas9 nuclease with expanded targeting space[J].Science, 2018, 361(6408):1259-1262.
[26] MA Y Q, ZHANG J Y, YIN W J, et al.Targeted AID-mediated mutagenesis (TAM) enables efficient genomic diversification in mammalian cells[J].Nature Methods, 2016, 13(12):1029-1035.
[27] HESS G T, FRÉSARD L, HAN K, et al.Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells[J].Nature Methods, 2016, 13(12):1036-1042.
[28] GAUDELLI N M, KOMOR A C, REES H A, et al.Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage[J].Nature, 2017, 551(7681):464-471.
[29] SRETENOVIC S, GREEN Y, WU Y C, et al.Genome- and transcriptome-wide off-target analyses of a high-efficiency adenine base editor in tomato[J].Plant Physiology, 2023, 193(1):291-303.
[30] YANG S P, ZHU X X, QU Z X, et al.Production of MSTN knockout porcine cells using adenine base-editing-mediated exon skipping[J].In Vitro Cellular &Developmental Biology-Animal, 2023, 59(4):241-255.
[31] RICHTER M F, ZHAO K T, ETON E, et al.Phage-assisted evolution of an adenine base editor with improved Cas domain compatibility and activity[J].Nature Biotechnology, 2020, 38(7):883-891.
[32] HUA K, TAO X P, ZHU J K.Expanding the base editing scope in rice by using Cas9 variants[J].Plant Biotechnology Journal, 2019, 17(2):499-504.
[33] KANG B C, YUN J Y, KIM S T, et al.Precision genome engineering through adenine base editing in plants[J].Nature Plants, 2018, 4(7):427-431.
[34] ZHANG Y, ZHANG H Y, WANG Z P, et al.Programmable adenine deamination in bacteria using a Cas9-adenine-deaminase fusion[J].Chemical Science, 2020, 11(6):1657-1664.
[35] TONG Y J, WHITFORD C M, ROBERTSEN H L, et al.Highly efficient DSB-free base editing for streptomycetes with CRISPR-BEST[J].Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(41):20366-20375.
[36] LI C, ZONG Y, WANG Y P, et al.Expanded base editing in rice and wheat using a Cas9-adenosine deaminase fusion[J].Genome Biology, 2018, 19(1):59.
[37] QIN W, LU X C, LIU Y X, et al.Precise A·T to G·C base editing in the zebrafish genome[J].BMC Biology, 2018, 16(1):139.
[38] KOBLAN L W, DOMAN J L, WILSON C, et al.Improving cytidine and adenine base editors by expression optimization and ancestral reconstruction[J].Nature Biotechnology, 2018, 36(9):843-846.
[39] YANG L, ZHANG X H, WANG L R, et al.Increasing targeting scope of adenosine base editors in mouse and rat embryos through fusion of TadA deaminase with Cas9 variants[J].Protein &Cell, 2018, 9(9):814-819.
[40] KURT I C, ZHOU R H, IYER S, et al.CRISPR C-to-G base editors for inducing targeted DNA transversions in human cells[J].Nature Biotechnology, 2021, 39(1):41-46.
[41] SUN N X, ZHAO D D, LI S W, et al.Reconstructed glycosylase base editors GBE2.0 with enhanced C-to-G base editing efficiency and purity[J].Molecular Therapy, 2022, 30(7):2452-2463.
[42] DONG X X, YANG C, MA Z Z, et al.Enhancing glycosylase base-editor activity by fusion to transactivation modules[J].Cell Reports, 2022, 40(3):111090.
[43] YANG C, DONG X X, MA Z Z, et al.Pioneer factor improves CRISPR-based C-to-G and C-to-T base editing[J].Advanced Science, 2022, 9(26):e2202957.
[44] TESTA L C, MUSUNURU K.Base editing and prime editing:Potential therapeutic options for rare and common diseases[J].BioDrugs, 2023, 37(4):453-462.
[45] FU Y D, HE X Y, GAO X D, et al.Prime editing:Current advances and therapeutic opportunities in human diseases[J].Science Bulletin, 2023, 68(24):3278-3291.
[46] CHEN P J, HUSSMANN J A, YAN J, et al.Enhanced prime editing systems by manipulating cellular determinants of editing outcomes[J].Cell, 2021, 184(22):5635-5652.
[47] GAO P, LYU Q, GHANAM A R, et al.Prime editing in mice reveals the essentiality of a single base in driving tissue-specific gene expression[J].Genome Biology, 2021, 22(1):83.
[48] ZHU G N, ZHU H L.Modified gene editing systems:Diverse bioengineering tools and crop improvement[J].Frontiers in Plant Science, 2022, 13:847169.
[49] LU Y M, TIAN Y F, SHEN R D, et al.Precise genome modification in tomato using an improved prime editing system[J].Plant Biotechnology Journal, 2021, 19(3):415-417.
[50] TONG Y J, JØRGENSEN T S, WHITFORD C M, et al.A versatile genetic engineering toolkit for E.coli based on CRISPR-prime editing[J].Nature Communications, 2021, 12(1):5206.
[51] JIN S, LIN Q P, LUO Y F, et al.Genome-wide specificity of prime editors in plants[J].Nature Biotechnology, 2021, 39(10):1292-1299.
[52] KWEON J, YOON J K, JANG A H, et al.Engineered prime editors with PAM flexibility[J].Molecular Therapy, 2021, 29(6):2001-2007.
[53] LIU P P, LIANG S Q, ZHENG C W, et al.Improved prime editors enable pathogenic allele correction and cancer modelling in adult mice[J].Nature Communications, 2021, 12(1):2121.
[54] GRÜNEWALD J, ZHOU R H, LAREAU C A, et al.A dual-deaminase CRISPR base editor enables concurrent adenine and cytosine editing[J].Nature Biotechnology, 2020, 38(7):861-864.
[55] SAKATA R C, ISHIGURO S, MORI H, et al.Base editors for simultaneous introduction of C-to-T and A-to-G mutations[J].Nature Biotechnology, 2020, 38(7):865-869.
[56] LIANG Y H, XIE J K, ZHANG Q J, et al.AGBE:A dual deaminase-mediated base editor by fusing CGBE with ABE for creating a saturated mutant population with multiple editing patterns[J].Nucleic Acids Research, 2022, 50(9):5384-5399.
[57] XIE J K, HUANG X Y, WANG X, et al.ACBE, a new base editor for simultaneous C-to-T and A-to-G substitutions in mammalian systems[J].BMC Biology, 2020, 18(1):131.
[58] LI C, ZHANG R, MENG X B, et al.Targeted, random mutagenesis of plant genes with dual cytosine and adenine base editors[J].Nature Biotechnology, 2020, 38(7):875-882.
[59] XU R F, KONG F N, QIN R Y, et al.Development of an efficient plant dual cytosine and adenine editor[J].Journal of Integrative Plant Biology, 2021, 63(9):1600-1605.
[60] 张雅玲, 王锌和, 李构思, 等.新型DNA碱基编辑器的研究进展[J].华南农业大学学报, 2022, 43(6):1-16;193.
ZHANG Y L, WANG X H, LI G S, et al.Research advances in novel DNA base editors[J].Journal of South China Agricultural University, 2022, 43(6):1-16;193.
[61] LI J Y, ZHANG C, HE Y B, et al.Plant base editing and prime editing:The current status and future perspectives[J].Journal of Integrative Plant Biology, 2023, 65(2):444-467.