细菌发酵过程中存在着代谢副产物多、产物抑制、环境胁迫等问题,导致发酵成本高、产量低,限制了工业菌株的发展。解析并优化细菌的代谢途径、提高它们的环境适应能力可以从根本上改善这些问题,促进发酵工业的发展。
细菌中普遍存在一类长度在50~400个核苷酸的较短转录本,它们以RNA的形式在转录后水平调节基因表达,这些具有调控功能的短的RNA通常被称为小RNA(small RNA, sRNA)[1-2]。sRNA主要通过碱基配对影响靶mRNA的翻译和稳定性,在细胞生长代谢、适应环境变化、应激反应、群体感应、毒力调节等各种细胞过程中发挥核心作用[2-3]。利用sRNA调控机制,可以对工程菌进行改造使其适应工业生产条件或减弱细菌毒素的表达。例如,在乳酸乳球菌中sRNA s042和s015都可以提高菌株的耐酸能力,可缓解其发酵过程中受到的pH限制,间接提高发酵产物乳酸链球菌素的产量[4-5]。在产气肠杆菌中过表达sRNA RyhB并调节NADH水平可以优化代谢途径,显著提高工业产品2,3-丁二醇的产量[6]。
细菌应对复杂的环境变化时,对基因的调控往往是多层次的,与已经广泛研究的转录因子、双组分系统等面向DNA的调控元件不同,sRNA的作用特性使其提供了另一个丰富的RNA调节层,形成对DNA调节层的拓展和补充,共同构成细菌中庞大复杂的调控网络。本文将从sRNA来源的多样性、作用机制的多样性及sRNA与其他调控组分的复杂关联来阐述sRNA在细菌复杂调控网络中的重要角色和作用,为解决发酵工业存在的问题提供理论参考。
1 sRNA来源的多样性
随着对sRNA研究的深入,sRNA的来源也不仅仅局限于传统意义上的顺式编码和反式编码(基因间区编码),越来越多的反式作用sRNA被发现由mRNA的5′非翻译区(5′ untranslated region, 5′UTR)和3′非翻译区(3′ untranslated region, 3′UTR)衍生出来,它们往往和原位基因使用共同的转录起始位点或终止位点[7]。丰富的来源使sRNA在细菌生命过程中发挥的作用更加广泛和精细,不同来源的sRNA及其特点汇总在表1中。
1.1 顺式编码的sRNA
顺式编码的sRNA从基因反义链上转录,因此又被称作反义sRNA(antisense RNA, asRNA),它们与靶mRNA的一部分完全互补,起到迅速调节的作用[22]。sRNA GadY,编码在相邻基因酸调节转录因子gadX的相反链,与gadX mRNA 3′UTR碱基互补并增加其稳定性,导致gadX mRNA的积累,进而促进下游谷氨酸脱羧酶系统中相关基因的表达,有利于大肠杆菌的耐酸生长[8-9]。lexA基因在金黄色葡萄球菌中是 SOS反应的主要调节因子,临床分离的一种突变株在lexA相反链的下游能够产生sRNA lexA-asRNA,是由于上游基因sbrB内部的转录终止子(transcriptional terminator, TT)自然突变而导致的转录通读产生的,能在碱胁迫的环境下抑制lexA表达并激活SOS反应[23]。虽然该突变在金黄色葡萄球菌中并不保守,但是类似的TT自然突变导致的转录通读现象并不罕见,它们代表了一种新的转录调控和遗传进化的方式[23]。
1.2 反式编码的sRNA
最初发现的几个sRNA是研究相邻基因时偶然发现的(如MicF,DsrA),因此sRNA被认为由基因间区产生[24]。除了基因间区编码,越来越多的sRNA被发现从基因mRNA两端的非翻译区衍生而来,5′UTR和3′UTR正逐渐成为新的sRNA候选库。
1.2.1 基因间区编码的sRNA
基因间区编码的sRNA与其他基因间没有重叠,独立转录出来发挥作用。大肠杆菌中的RyhB是一个经典的反式作用sRNA,受到铁调控因子Fur的抑制,在缺铁环境中,RyhB借助分子伴侣Hfq控制50多个参与铁摄取和铁利用的基因来维持细胞内铁稳态,发挥关键作用[12]。类似地,最近在金黄色葡萄球菌中也鉴定出了与维持铁稳态相关的sRNA IsrR,为应对宿主体内的缺铁环境,它能抑制非必需途径中含铁酶的表达,并将铁进行重新分配[13]。IsrR与RyhB发挥的功能非常相似,但IsrR并不是RyhB的同源物,也不需要Hfq协助发挥作用[13]。这说明在细菌生命进化的过程中,无论革兰氏阴性菌还是阳性菌,sRNA都在快速应对环境变化中发挥相当重要的作用。
1.2.2 由5′UTR衍生的sRNA
5′UTR是基因mRNA 5′末端的非翻译区,不参与蛋白质的合成,但最近的研究证明5′UTR可以通过核糖核酸酶(RNase)等的剪切被加工成独立的sRNA发挥作用。铜绿假单胞菌的群体感应系统相关基因rhlI的5′UTR区域能衍生出一种具有活性的sRNA RhlS,它能够激活rhlI的翻译并诱导正丁酰基高丝氨酸内酯合酶产生,还能调节编码铁载体受体的基因fpvA的mRNA[10]。沙门氏菌的编码内膜转运蛋白的oppABCDF基因簇的5′UTR中发现了一种RNA海绵OppX,它能通过隔离sRNA MicF的种子区域来减弱MicF的抑制作用,促进膜孔蛋白OmpF的合成,从而调节细菌包膜的渗透性和物质运输等[14]。OppX仅仅作为RNA海绵将MicF隔离在一个不具有活性的复合体中,而不影响它的水平和稳定性[14]。这意味着细菌sRNA在复杂调控中往往不是单独发挥作用,而是与其他调节元件共同发挥作用。
1.2.3 由3′UTR衍生的sRNA
与5′UTR类似,3′UTR是mRNA 3′末端的非翻译区,通过核糖核酸内切酶或外切酶的加工,能产生具有丰富调节功能的sRNA[25-26]。乳酸乳球菌基因argR的3′UTR中被鉴定出一种sRNA ArgX,它有独立的启动子并受到精氨酸浓度和转录因子CcpA的诱导,可以调节精氨酸脱亚胺酶途径中的arc mRNA水平[18]。此外实验还证明了ArgX与转录因子ArgR拥有相同的靶标,从转录和转录后水平协同发挥作用[18]。在肠杆菌中,CpxP是一种在内膜蛋白错误折叠引起的应激反应中起重要作用的蛋白质,sRNA CpxQ从cpxP mRNA的3′UTR切割下来并依赖Hfq发挥作用[19]。CpxQ几乎涵盖了整个cpxP 3′UTR,该区域在肠杆菌中极为保守,可以抑制包括Na+/H+逆转运蛋白NhaB在内的几种蛋白质的合成,从而减少应激下膜电位的损失[19]。
表1 不同来源sRNA的特点及举例
Table 1 Characteristics and examples of sRNA from different sources

sRNA来源特点举例物种靶标生理功能参考文献顺式编码编码在靶标基因的对链,通常只有一个靶标,与靶标完全碱基互补。GadYEscherichia coligadX耐酸[8-9]asRhlSPseudomonas aeruginosarhlI群体感应[10]SR4Bacillus subtilisbsrG抗毒素[11]反式编码基因间区编码5′UTR衍生3′UTR衍生编码在基因间区或由mRNA两端的非翻译区加工而来,与靶标有短而不完美的碱基互补,通常有多个靶标。RyhBEscherichia colisodB、isc、shiA等维持铁稳态、毒力[12]IsrRStaphylococcus aureusfdhA、gltB2等维持铁稳态[13]MicFEscherichia coliompF细菌包膜应激[14]DsrAEscherichia colirpoS、hns、mreB等参与多种应激反应[15]RhlSPseudomonas aeruginosarhlI群体感应[10]OppXSalmonella typhimuriumsRNA MicF调节细胞膜渗透性[14]Vsr217Vibrio tasmaniensis LGP32fbp促进糖酵解[16]UpsMRhodobacter sphaeroidesdcw调节细胞分裂[17]ArgXLactococcus lactisarc精氨酸代谢[18]CpxQEscherichia colinhaB、skp等细菌包膜应激[19]DapZSalmonella typhimuriumdpp、opp合成赖氨酸[20]RsaGStaphylococcus aureustcaR、sarA、ccpA、sRNA RsaI氧化还原应激[21]
2 sRNA调控机制的多样性
随着对sRNA研究的深入,其多种多样的作用机制也逐渐被解析出来,以便更好理解这些数量庞大、简单而又复杂的调控组分如何发挥作用。迄今为止发现的sRNA作用机制主要包括与靶标RNA碱基配对、与蛋白质直接结合以及编码小蛋白质发挥双重功能(图1)。
2.1 sRNA与靶mRNA碱基配对
碱基配对是sRNA最普遍的作用方式,它们通过碱基配对来激活或抑制靶mRNA的翻译,或者提高或降低靶mRNA的稳定性,来影响基因表达。
2.1.1 影响靶mRNA与核糖体结合
sRNA可以通过与Shine-Dalgarno(SD)序列或起始密码子碱基配对来隔离靶mRNA的核糖体结合位点(ribosome-binding site, RBS),还可以与mRNA的5′UTR或编码序列(coding sequence, CDS)相互作用来释放RBS,进而影响核糖体的结合和mRNA翻译[1]。鼠伤寒沙门氏菌中sRNA RaiZ从编码核糖体失活蛋白的基因raiA的3′末端衍生而来,可以与编码HU-α蛋白的hupA mRNA的RBS结合形成双链,以阻止30S核糖体负载,抑制其翻译[27]。而在肠出血性大肠杆菌中,转录激活蛋白PchA的CDS序列与自身mRNA 5′UTR配对,隔离了RBS,在缺氧条件下,sRNA DicF通过与pchA CDS配对从而释放RBS,促进pchA表达并增强了有利于定殖宿主的关键毒力因子EHEC Ⅲ型分泌系统的表达[28]。
2.1.2 影响靶mRNA的稳定性
sRNA另一种常见的作用机制是提高或降低靶mRNA的稳定性,具体形式包括促进或抑制mRNA的降解、使转录提前终止从而抑制mRNA合成等等。枯草芽孢杆菌中的sRNA RoxS,能在苹果酸的存在下通过富C区结合在yflS mRNA的5′末端,使其避免被核酸外切酶RNase J1切割,延长半衰期,同时增强该mRNA与核糖体的结合促进苹果酸转运蛋白YflS的表达[29]。大肠杆菌的多顺反子galETKM操纵子中,sRNA Spot42通过碱基配对结合在galT的终止密码子附近,引起转录提前终止和核酸内切酶RNase Ⅲ或RNase E介导的转录物切割,产生2种长度不同的galET mRNA[30]。与此同时,Spot 42还会下调galK的转录和翻译,并促进galKM mRNA的降解,这些调控共同导致了galET的表达增加和galKM的表达减少,引起半乳糖代谢的变化从而优化大肠杆菌的生长[30]。类似的sRNA调节多顺反子差异表达的现象似乎在细菌中普遍存在,在多种生命过程中起到重要过渡作用。
2.1.3 其他配对机制
革兰氏阳性菌中还有一些不同于上述配对机制的调节实例。最近在李斯特菌中发现prsA2 mRNA的5′UTR能与hly mRNA(编码李斯特菌溶血素O)的3′UTR直接配对,从而防止RNase J1介导的prsA2 mRNA的降解,该mRNA-mRNA作用有利于李斯特菌毒力因子的表达[31]。此外,sRNA与其他sRNA也可以通过直接配对相互作用。(详见下文3.4节内容)
2.2 sRNA与蛋白质直接结合
除了在转录后水平通过碱基配对调控靶标,一些sRNA可以直接结合蛋白质发挥作用,其中最具有代表性的是6S RNA和CsrB/CsrC。6S RNA最先在大肠杆菌中被鉴定出来,在细菌中广泛存在,它通过与RNA聚合酶的全酶形式特异性结合,下调许多依赖σ因子的基因转录,有助于帮助细菌应对营养不良的环境,缺乏6S RNA还会对细胞的存活表型造成影响[32-33]。大肠杆菌中CsrA蛋白可以调节碳通量、生物膜形成和运动相关基因,通常结合mRNA结构环内的GGA基序影响翻译或改变靶标mRNA的稳定性[34-35]。CsrA的调节活性主要被sRNA CsrB和CsrC调节,它们分别含有22个和13个重复GGA基序,可以作为RNA海绵隔离CsrA使其远离靶标[34-35]。此外,sRNA McaS也能通过隔离CsrA调节其活性[36]。在许多细菌中都发现了Csr系统或其同源物,如假单胞菌属中含有Rsm系统,以类似的机制发挥作用[35]。
2.3 双功能sRNA编码小蛋白质
sRNA能够以非编码RNA形式结合mRNA发挥作用,有些sRNA本身还包含小的开放阅读框(open reading frame,ORF),可以翻译成小的编码蛋白发挥作用,这样的sRNA称为双功能sRNA[37]。双功能sRNA的转录后调控和翻译蛋白2种功能之间可能会存在竞争关系,进一步丰富了sRNA介导的复杂调控网络。
大肠杆菌sRNA AzuR(以前被称作IsrB)能够通过碱基配对抑制cadA和galE的mRNA的表达,同时它还编码含有28个氨基酸的蛋白质AzuC,AzuC能与需氧甘油-3-磷酸脱氢酶GlpD相互作用增加其活性[38]。研究发现,AzuR的碱基配对区域和AzuC的编码区域相重合,这意味着它们之间存在互相干扰和竞争[38]。此外,另一个sRNA FnrS也被证明可以抑制AzuC的翻译,增加了AzuCR RNA作用的灵活性[38]。在其他研究中,大肠杆菌sRNA SgrS、Spot42以及位于金黄色葡萄球菌毒力调控中心的sRNA RNAⅢ也被证明具有双功能属性[37,39-40]。
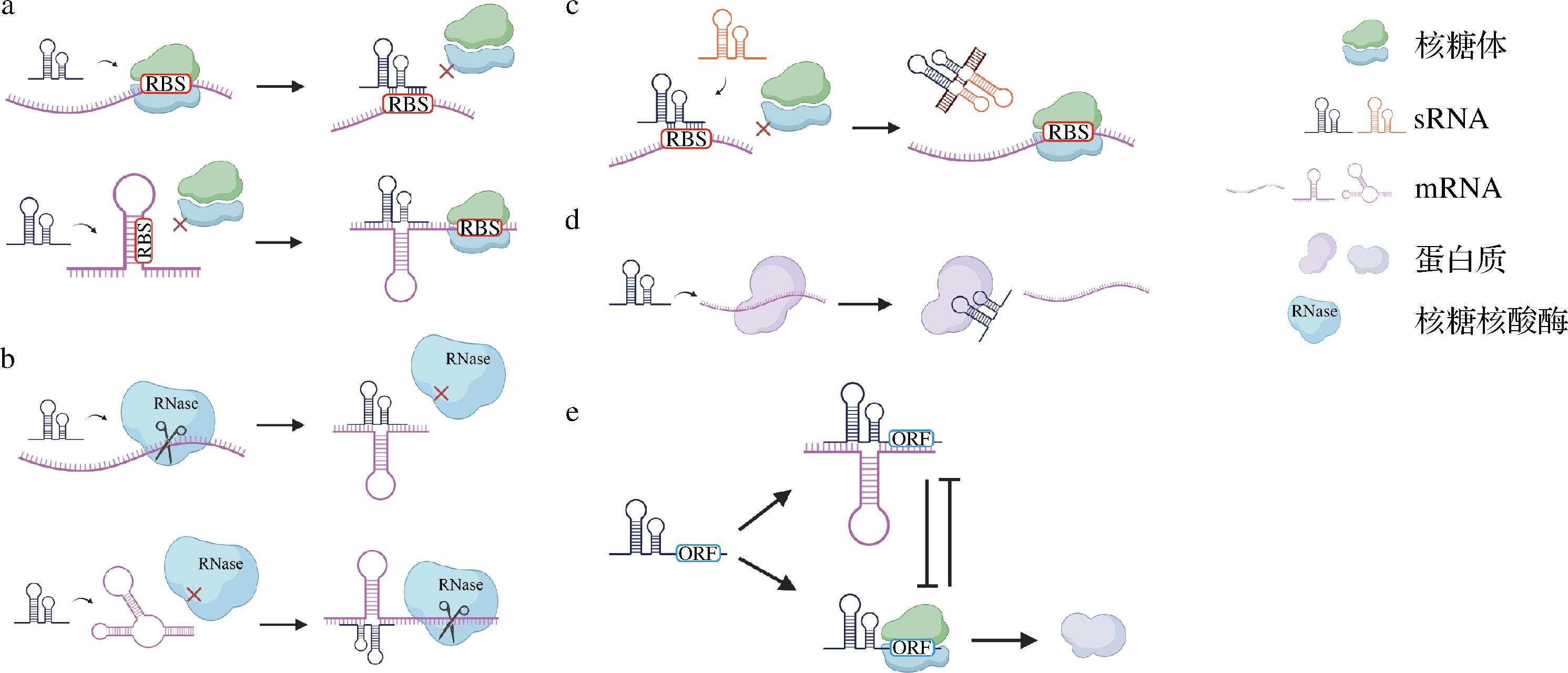
a-sRNA通过阻止或促进核糖体结合RBS来抑制或激活翻译;b-sRNA提高或降低mRNA稳定性;c-sRNA通过隔离其他sRNA干扰其活性; d-sRNA通过隔离蛋白降低其活性;e-双功能sRNA结合mRNA或编码小蛋白质
图1 sRNA调控作用机制示意图
Fig.1 Mechanism of sRNA regulation
3 sRNA与其他组分共同构成复杂调控网络
细菌在面对环境压力和环境变化时,往往需要同时对多种途径进行调节,涉及一系列靶标基因,可以同时调节多个靶标的sRNA在其中发挥了巨大的作用。在以往的研究中,基因调控可以通过核糖开关、转录因子、双组分系统等从转录或其他层面发挥作用,这些调控与sRNA介导的转录后调控必然是错综复杂交织在一起的,共同形成庞大的调控网络,使细菌以最优效率完成对环境的适应。sRNA通过与各个元件之间的相互影响,在调控网络中起到重要的纽带作用。
3.1 sRNA与核糖开关
核糖开关是另一类存在于5′UTR的调控性非编码RNA,可以通过响应代谢物或离子的变化从而改变自身结构来调节下游基因的表达[41]。一些sRNA内部含有核糖开关,可以从代谢物和转录后水平同时调控靶标基因。
粪肠球菌的乙醇胺(ethanolamine, EA)代谢eut基因簇中含有双组分系统EutVW,其中组氨酸激酶EutW负责感知环境中的EA,并磷酸化转录因子EutV使其成为具有活性的二聚体形式,随后EutV通过结合eutG前终止子附近的双发夹结构来阻止eut转录终止[42]。sRNA EutX编码在基因eutT和eutG之间,它内部含有一个响应腺苷钴胺素(adocbl)的核糖开关,还含有可供EutV结合的另一个双发夹结构[42]。Adocbl是EA代谢的关键辅因子,当没有Adocbl时,EutX可以反式发挥作用,通过双发夹结构隔离EutV,使eut转录提前终止,关闭下游基因通路;当Adocbl存在时,EutX内部核糖开关构象的改变使双发夹结构消失,不能隔离EutV,导致eut基因簇多个基因转录通读,开启EA代谢(图2)[42]。这种包含核糖开关的结构的优势可能是sRNA的数量不需要过多的变化,核糖开关的存在使sRNA能通过代谢物来感知环境的变化,代谢物介导的构象改变也更为迅速和直接,使sRNA能更快速地参与到下游基因的调控中。
3.2 sRNA与双组分系统
双组分系统是原核生物适应环境的重要信号传导系统,通过协调细菌毒力、运动、抗生素耐药性、代谢、形态分化、应激反应等各种生物过程,来维持体内平衡和各种细胞功能[43-46]。双组分系统通常包含2种组分,一个是称为组氨酸激酶的传感器元件,负责感知环境信号;另一个是称为响应调节器的效应元件,负责根据信号产生细胞响应[47]。
双组分系统中的效应器可以通过结合DNA来调节基因的表达,因此也可以促进sRNA的产生。例如,大肠杆菌RcsB是FixJ/NarL家族的响应调节器,磷酸化的RcsB可以同源二聚化并调节sRNA基因rprA、osmC和ftsZ的启动子,促进其转录,产生的sRNA RprA还可以进一步调节调控因子RpoS,激活一系列靶标基因表达[48]。霍乱弧菌中,双组分系统EnvZ/OmpR是肠道定殖所必需的[49]。研究发现,在高渗透压和酸胁迫的条件下,OmpR直接与sRNA CoaR的启动子结合促进其表达,CoaR能通过解除TcpI对菌毛蛋白亚基TcpA的抑制从而促进TcpA的表达,增强霍乱弧菌的肠道定殖能力[49]。
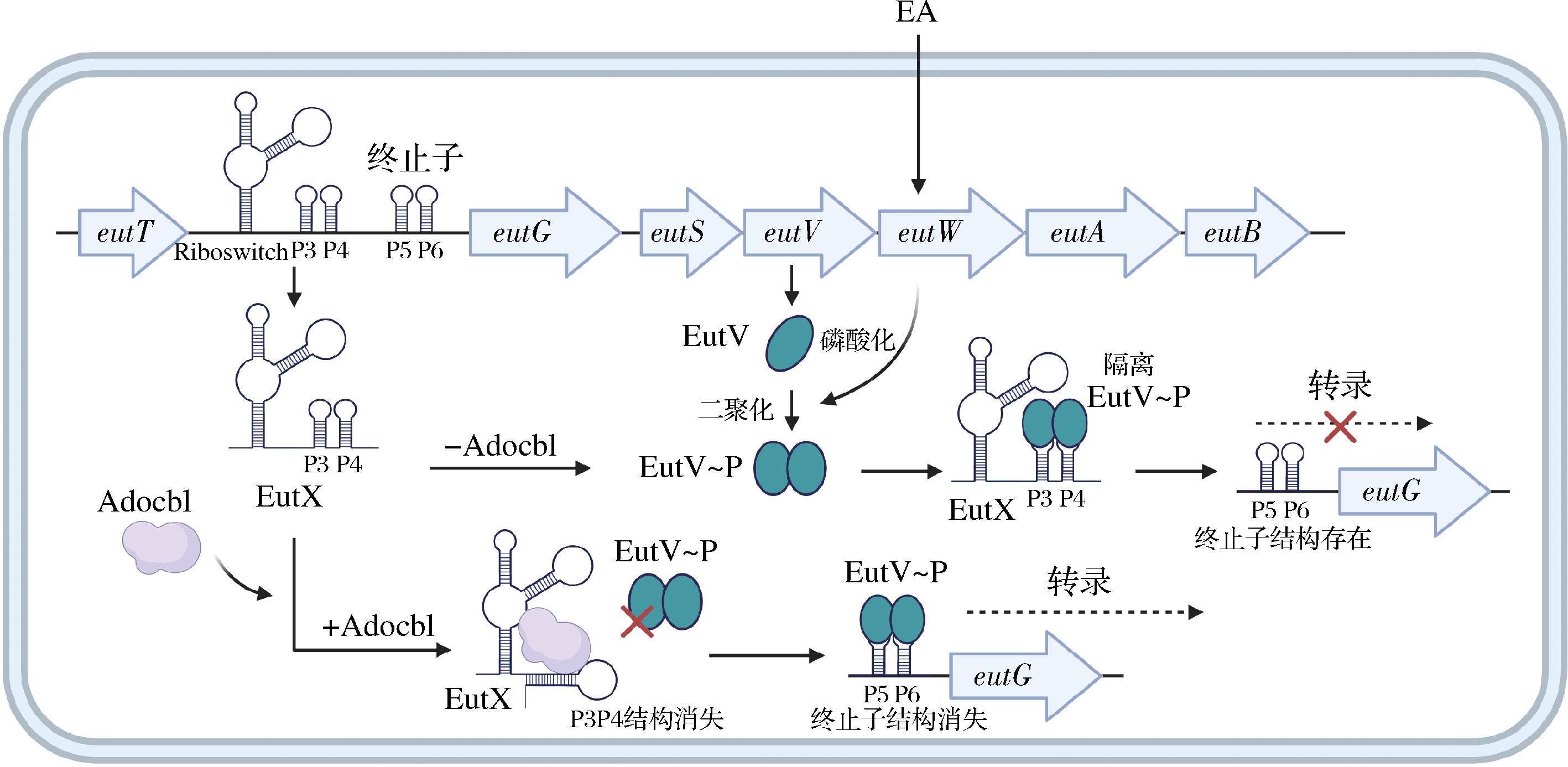
图2 粪肠球菌sRNA EutX调节乙醇胺代谢示意图[42]
Fig.2 sRNA EutX regulates ethanolamine metabolism in Enterococcus faecalis[42]
双组分系统的合成也可以受到sRNA的调控。大肠杆菌NarP/NarQ双组分系统参与调节无氧呼吸基因的表达,研究显示narP mRNA受到sRNA SdsN137和RprA的抑制[50]。此外,大肠杆菌中的另一个双组分系统EnvZ/OmpR可以直接激活sRNA OmrA/B的启动子,但同时envZ-ompR mRNA本身也是OmrA/B的下调靶标,它们共同形成了一种负反馈的调节机制[51]。sRNA OmrA/B对envZ-ompR的抑制似乎仅影响OmpR的总量但不影响具有活性的磷酸化OmpR的量,这意味着OmpR调节子可能存在着多层次的调控[51]。
3.3 sRNA与转录因子
转录因子是广为人知的一大类调控因子,通过识别目标启动子处特定的DNA序列,招募或阻断RNA聚合酶,从而上调或下调该启动子的转录起始[52]。
sRNA的转录通常受到转录因子的调节。例如,正常情况下枯草芽孢杆菌中的sRNA RoxS受到转录因子Rex的抑制,而在苹果酸存在时,RoxS会暂时摆脱这种抑制,并反式作用于编码苹果酸转运蛋白的yflS mRNA,增强其表达,促进苹果酸代谢(图3)[29]。另一个sRNA RosA也受到转录因子CcpA的抑制,CcpA是枯草芽孢杆菌中碳代谢的关键调节因子,通过抑制RosA间接调控sRNA RoxS,使RoxS可以响应不同碳源代谢调控,发挥关键作用(图3)[53-54]。可以看出,sRNA受转录因子调节,可以在转录后水平扩展转录因子的影响范围,使相关基因的调节更加全面。
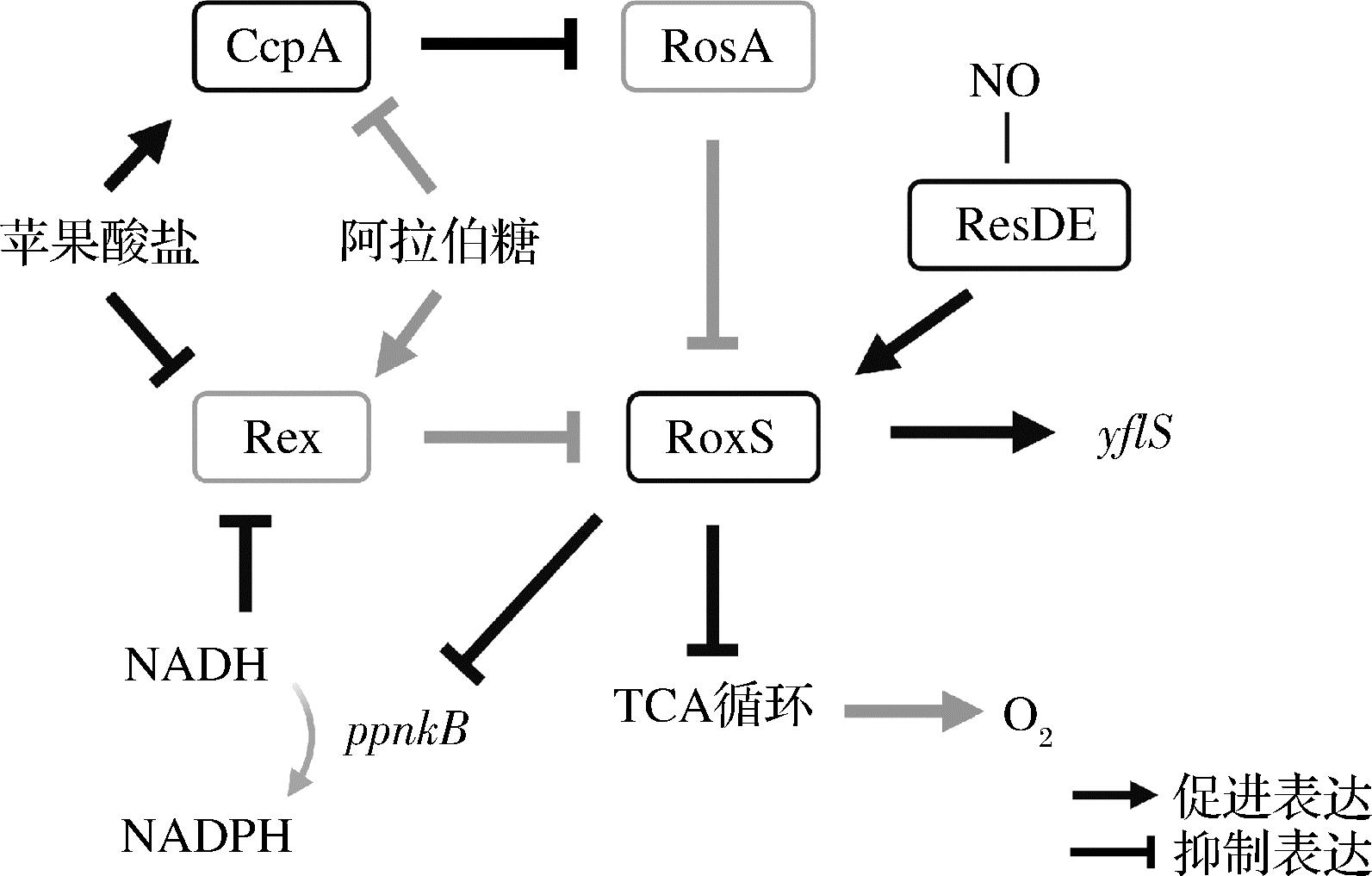
CcpA, Rex-转录因子;RosA, RoxS-sRNA;ResDE-双组分系统; yflS-编码苹果酸转运蛋白;ppnkB-编码NAD激酶; 黑色表示苹果酸盐为碳源时的代谢调控途径, 灰色表示阿拉伯糖为碳源时的代谢调控途径。
图3 枯草芽孢杆菌sRNA RoxS调节不同碳源 代谢途径示意图[29,53-54]
Fig.3 sRNA RoxS regulates metabolic pathways of different carbon sources in Bacillus subtilis[29,53-54]
反过来转录因子也可以作为靶标受到sRNA的转录后调节。flhDC操纵子编码转录因子FlhD4C2,它是大肠杆菌一系列运动相关基因的主要激活因子,受到编码在其对链上的反义sRNA AsflhD的精确调控,过多或过少的AsflhD都会抑制其中flhD的翻译,进而影响一系列运动相关基因使细菌运动减缓[55]。大肠杆菌RpoS是应激反应的关键调节因子,可调节约500个基因,它的表达受到3个sRNA,ArcZ、DsrA、RprA的转录后激活,还受到sRNA CyaR的转录后抑制,涉及多种机制的联合作用[56]。
3.4 sRNA与sRNA
除了研究广泛的mRNA靶标,sRNA还可以作为RNA海绵直接作用于其他sRNA,干扰它们的活性和稳定性。sRNA-sRNA作用与sRNA-mRNA作用之间存在着竞争关系,有助于细菌对基因表达微调以应对环境压力[56]。
大肠杆菌应激调控因子RpoS既受到sRNA ArcZ、DsrA、RprA的激活,又受到sRNA CyaR的抑制,这其中包含了sRNA ArcZ与CyaR之间的相互作用,ArcZ以一种非全长的形态直接与CyaR配对,诱导CyaR被RNase E降解并减轻它对RpoS的抑制[56]。运动发酵单胞菌在乙醇胁迫下,sRNA Zms4和Zms6通过调控多个乙醇抗性相关的靶标基因,对细菌生长、乙醇耐受性和产量都有显著影响[57]。Zms4和Zms6可以不需要Hfq直接结合,通过竞争或结构变化影响与各自靶标的结合,从而精准调控代谢途径以适应环境变化[57]。
sRNA RosA是第一个在革兰氏阳性菌中经证实的RNA海绵,它通过碱基配对与至少2个sRNA,RoxS和FsrA相互作用,其中RosA-RoxS的相互作用不仅影响RoxS的水平,还影响其稳定性和调节活性(图3)[53]。RosA作为RNA海绵在其中主要有2个作用:一方面是以富G区域与RoxS中发挥调节作用的富C区结合,降低其活性;另一方面RosA还能刺激RoxS 5′茎环的打开来促进其降解,这种作用使RoxS的半衰期大大缩短,使调控更为迅速和精准[53]。后来在金黄色葡萄球菌中也发现了类似的RNA海绵。一种前噬菌体编码的sRNA SprY,能够与金黄色葡萄球菌中的核心毒力调节sRNA RNAⅢ进行碱基配对,阻止RNAⅢ调节其靶标,从而降低金黄色葡萄球菌的溶血活性和毒力[58]。
3.5 sRNA多靶标的调控顺序
sRNA介导的调控通常都会有多个靶标,一些靶标属于同一代谢途径可能会被同时调控,但另一些不同作用的靶标就涉及到调控顺序的问题,优先调控哪些靶标可能取决于细菌所处环境的变化。
在中华根瘤菌中,sRNA rnTrpL来源于色氨酸合成基因trpE(G)上游的色氨酸转录衰减子中,含有一个小的ORF trpL,编码含有14个氨基酸的前导肽peTrpL[59]。当色氨酸充足时,trpL的翻译会导致转录提前终止,释放rnTrpL,使其下调trpDC操纵子,减少色氨酸的生物合成;然而,当抗生素四环素存在时,rnTrpL会产生与色氨酸无关的积累,此时rnTrpL会和其他2种分子peTrpL及rplUrpmA mRNA形成抗生素依赖性的核糖核蛋白复合物,降低编码核糖体蛋白L21和L27的rplUrpmA mRNA稳定性[59]。研究表明,这2种靶标出现竞争关系时,四环素和peTrpL的存在会导致sRNA rnTrpL特异性向rplU转录物转移,说明在抗生素存在的环境,sRNA rnTrpL需要优先处理与抗生素耐药性相关的靶标[59]。
4 总结与展望
sRNA在细菌的多种生命活动,尤其是涉及毒力、应激反应和适应环境变化的过程中发挥着重要的作用。然而,sRNA不是单独发挥作用的,它与各个调节模块间有着不可或缺的紧密联系。核糖开关的存在拓展了sRNA响应代谢物和离子的能力;转录因子和双组分系统通过激活sRNA的转录,进一步拓展了它们在转录后水平的调控范围,使相关基因的控制更为精准和全面;反过来sRNA也可以作为上游去调控转录因子和双组分系统基因的表达,进而引发更大的调控链;sRNA还可以作为海绵调节其他sRNA的活性和稳定性。此外,面对环境的变化,sRNA的靶标和发挥的作用也会及时的调整,以达到最优效果。
不同调节模块相互联结共同形成了细菌中庞大的调控网络,增强了细菌在各种复杂环境中的生命力,sRNA在其中发挥着异常重要的连接和补充作用。通过了解sRNA及其调控网络的调控机制,有利于辅助我们进行代谢路径的设计,构建更安全、更高效的工程菌株,解决发酵过程中副产物多、产物抑制等问题,助力发酵工业的生产和发展。目前对包括细菌、酵母等在内的工业微生物的sRNA研究仍然不够充分,许多发酵过程中具有显著表达差异的sRNA还未被解析,相信在未来,sRNA的研究会在工程菌的运用、致病菌感染治疗等方面发挥更多、更大的作用。
[1] STORZ G, VOGEL J, WASSARMAN K M.Regulation by small RNAs in bacteria:Expanding frontiers[J].Molecular Cell, 2011, 43(6):880-891.
[2] NITZAN M, REHANI R, MARGALIT H.Integration of bacterial small RNAs in regulatory networks[J].Annual Review of Biophysics, 2017, 46:131-148.
[3] DUTTA T, SRIVASTAVA S.Small RNA-mediated regulation in bacteria:A growing palette of diverse mechanisms[J].Gene, 2018, 656:60-72.
[4] WU H, SONG S Y, TIAN K R, et al.A novel small RNA s042 increases acid tolerance in Lactococcus lactis F44[J].Biochemical and Biophysical Research Communications, 2018, 500(3):544-549.
[5] QI J K, CAIYIN Q, WU H, et al.The novel sRNA s015 improves nisin yield by increasing acid tolerance of Lactococcus lactis F44[J].Applied Microbiology and Biotechnology, 2017, 101(16):6483-6493.
[6] WU Y, CHU W Y, YANG J Y, et al.Metabolic engineering of Enterobacter aerogenes for improved 2, 3-butanediol production by manipulating NADH levels and overexpressing the small RNA RyhB[J].Frontiers in Microbiology, 2021, 12:754306.
[7] MENARD G, SILARD C, SURIRAY M, et al.Thirty years of sRNA-mediated regulation in Staphylococcus aureus: From initial discoveries to in vivo biological implications[J].International Journal of Molecular Sciences, 2022, 23(13):7346.
[8] OPDYKE J A, KANG J G, STORZ G.GadY, a small-RNA regulator of acid response genes in Escherichia coli[J].Journal of Bacteriology, 2004, 186(20):6698-6705. [9] NEGRETE A, SHILOACH J.Constitutive expression of the sRNA GadY decreases acetate production and improves E.coli growth[J].Microbial Cell Factories, 2015, 14:148.
[10] THOMASON M K, VOICHEK M, DAR D, et al.A rhlI 5′ UTR-derived sRNA regulates RhlR-dependent quorum sensing in Pseudomonas aeruginosa[J].mBio, 2019, 10(5):e02253-e02219.
[11] JAHN N, BRANTL S.One antitoxin:Two functions:SR4 controls toxin mRNA decay and translation[J].Nucleic Acids Research, 2013, 41(21):9870-9880.
[12] CHAREYRE S, MANDIN P.Bacterial iron homeostasis regulation by sRNAs[J].Microbiology Spectrum, 2018, 6(2).DOI:10.1128/microbiolspec.RWR-0010-2017.
[13] CORONEL-TELLEZ R H, POSPIECH M, BARRAULT M, et al.sRNA-controlled iron sparing response in Staphylococci[J].Nucleic Acids Research, 2022, 50(15):8529-8546.
[14] MATERA G, ALTUVIA Y, GEROVAC M, et al.Global RNA interactome of Salmonella discovers a 5′ UTR sponge for the MicF small RNA that connects membrane permeability to transport capacity[J].Molecular Cell, 2022, 82(3):629-644.e4.
[15] WU P Z, LIU X D, YANG L N, et al.The important conformational plasticity of DsrA sRNA for adapting multiple target regulation[J].Nucleic Acids Research, 2017, 45(16):9625-9639.
[16] LUO X, ESBERARD M, BOULOC P, et al.A small regulatory RNA generated from the malK 5′ untranslated region targets gluconeogenesis in Vibrio species[J].mSphere, 2021, 6(3):e0013421.
[17] GRÜTZNER J, REMES B, EISENHARDT K M H, et al.sRNA-mediated RNA processing regulates bacterial cell division[J].Nucleic Acids Research, 2021, 49(12):7035-7052.
[18] VAN DER MEULEN S B, HESSELING-MEINDERS A, DE JONG A, et al.The protein regulator ArgR and the sRNA derived from the 3′-UTR region of its gene, ArgX, both regulate the arginine deiminase pathway in Lactococcus lactis[J].PLoS One, 2019, 14(6):e0218508.
[19] CHAO Y J, VOGEL J.A 3’ UTR-derived small RNA provides the regulatory noncoding arm of the inner membrane stress response[J].Molecular Cell, 2016, 61(3):352-363.
[20] CHAO Y J, PAPENFORT K, REINHARDT R, et al.An atlas of Hfq-bound transcripts reveals 3′ UTRs as a genomic reservoir of regulatory small RNAs[J].The EMBO Journal, 2012, 31(20):4005-4019.
[21] DESGRANGES E, BARRIENTOS L, HERRGOTT L, et al.The 3′UTR-derived sRNA RsaG coordinates redox homeostasis and metabolism adaptation in response to glucose-6-phosphate uptake in Staphylococcus aureus[J].Molecular Microbiology, 2022, 117(1):193-214.
[22] EVGUENIEVA-HACKENBERG E.Riboregulation in bacteria:From general principles to novel mechanisms of the trp attenuator and its sRNA and peptide products[J].Wiley Interdisciplinary Reviews.RNA, 2022, 13(3):e1696.
[23] BASTET L, BUSTOS-SANMAMED P, CATALAN-MORENO A, et al.Regulation of heterogenous LexA expression in Staphylococcus aureus by an antisense RNA originating from transcriptional read-through upon natural mispairings in the sbrB intrinsic terminator[J].International Journal of Molecular Sciences, 2022, 23(1):576.
[24] ARGAMAN L, HERSHBERG R, VOGEL J, et al.Novel small RNA-encoding genes in the intergenic regions of Escherichia coli[J].Current Biology, 2001, 11(12):941-950.
[25] PONATH F, HÖR J, VOGEL J.An overview of gene regulation in bacteria by small RNAs derived from mRNA 3′ ends[J].FEMS Microbiology Reviews, 2022, 46(5):fuac017.
[26] DESGRANGES E, MARZI S, MOREAU K, et al.Noncoding RNA[J].Microbiology Spectrum, 2019, 7(2).
[27] SMIRNOV A, WANG C A, DREWRY L L, et al.Molecular mechanism of mRNA repression in trans by a ProQ-dependent small RNA[J].The EMBO Journal, 2017, 36(8):1029-1045.
[28] MELSON E M, KENDALL M M.The sRNA DicF integrates oxygen sensing to enhance enterohemorrhagic Escherichia coli virulence via distinctive RNA control mechanisms[J].Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(28):14210-14215.
[29] DURAND S, BRAUN F, HELFER A C, et al.sRNA-mediated activation of gene expression by inhibition of 5′-3′ exonucleolytic mRNA degradation[J].eLife, 2017, 6:23602.
[30] JEON H J, LEE Y, MONFORD PAUL ABISHEK N, et al.sRNA-mediated regulation of gal mRNA in E.coli:Involvement of transcript cleavage by RNase E together with Rho-dependent transcription termination[J].PLoS Genetics, 2021, 17(10):e1009878.
[31] IGNATOV D, VAITKEVICIUS K, DURAND S, et al.An mRNA-mRNA interaction couples expression of a virulence factor and its chaperone in Listeria monocytogenes[J].Cell Reports, 2020, 30(12):4027-4040.e7.
[32] WASSARMAN K M.6S RNA, a global regulator of transcription[J].Microbiology Spectrum, 2018, 6(3).DOI:10.1128/microbiolspec.RWR-0019-2018.
[33] CAVANAGH A T, WASSARMAN K M.6S RNA, a global regulator of transcription in Escherichia coli, Bacillus subtilis, and beyond[J].Annual Review of Microbiology, 2014, 68:45-60.
[34] QUENDERA A P, SEIXAS A F, DOS SANTOS R F, et al.RNA-binding proteins driving the regulatory activity of small non-coding RNAs in bacteria[J].Frontiers in Molecular Biosciences, 2020, 7:78.
[35] DJAPGNE L, OGLESBY A G.Impacts of small RNAs and their chaperones on bacterial pathogenicity[J].Frontiers in Cellular and Infection Microbiology, 2021, 11:604511.
[36] JØRGENSEN M G, THOMASON M K, HAVELUND J, et al.Dual function of the McaS small RNA in controlling biofilm formation[J].Genes &Development, 2013, 27(10):1132-1145.
[37] VANDERPOOL C K, BALASUBRAMANIAN D, LLOYD C R.Dual-function RNA regulators in bacteria[J].Biochimie, 2011, 93(11):1943-1949.
[38] RAINA M, AOYAMA J J, BHATT S, et al.Dual-function AzuCR RNA modulates carbon metabolism[J].Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(10):e2117930119.
[39] WADLER C S, VANDERPOOL C K.A dual function for a bacterial small RNA:SgrS performs base pairing-dependent regulation and encodes a functional polypeptide[J].Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(51):20454-20459.
[40] AOYAMA J J, RAINA M, ZHONG A S, et al.Dual-function Spot 42 RNA encodes a 15-amino acid protein that regulates the CRP transcription factor[J].Proceedings of the National Academy of Sciences of the United States of America, 2022, 119(10):e2119866119.
[41] BÉDARD A S V, HIEN E D M, LAFONTAINE D A.Riboswitch regulation mechanisms:RNA, metabolites and regulatory proteins[J].Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms, 2020, 1863(3):194501.
[42] DEBROY S, GEBBIE M, RAMESH A, et al.Riboswitches.A riboswitch-containing sRNA controls gene expression by sequestration of a response regulator[J].Science, 2014, 345(6199):937-940.
[43] S NCHEZ DE LA NIETA R, SANTAMAR
NCHEZ DE LA NIETA R, SANTAMAR A R I, D
A R I, D AZ M.Two-component systems of Streptomyces coelicolor:An intricate network to be unraveled[J].International Journal of Molecular Sciences, 2022, 23(23):15085.
AZ M.Two-component systems of Streptomyces coelicolor:An intricate network to be unraveled[J].International Journal of Molecular Sciences, 2022, 23(23):15085.
[44] SULTAN M, ARYA R, KIM K K.Roles of two-component systems in Pseudomonas aeruginosa virulence[J].International Journal of Molecular Sciences, 2021, 22(22):12152.
[45] QIN S G, XIAO W, ZHOU C M, et al.Pseudomonas aeruginosa:Pathogenesis, virulence factors, antibiotic resistance, interaction with host, technology advances and emerging therapeutics[J].Signal Transduction and Targeted Therapy, 2022, 7:199.
[46] BROWN A N, ANDERSON M T, BACHMAN M A, et al.The ArcAB two-component system:Function in metabolism, redox control, and infection[J].Microbiology and Molecular Biology Reviews:MMBR, 2022, 86(2):e0011021.
[47] JACOB-DUBUISSON F, MECHALY A, BETTON J M, et al.Structural insights into the signalling mechanisms of two-component systems[J].Nature Reviews Microbiology, 2018, 16(10):585-593.
[48] WALL E, MAJDALANI N, GOTTESMAN S.The complex rcs regulatory cascade[J].Annual Review of Microbiology, 2018, 72:111-139.
[49] XI D Y, LI Y J, YAN J X, et al.Small RNA coaR contributes to intestinal colonization in Vibrio cholerae via the two-component system EnvZ/OmpR[J].Environmental Microbiology, 2020, 22(10):4231-4243.
[50] BROSSE A, BOUDRY P, WALBURGER A, et al.Synthesis of the NarP response regulator of nitrate respiration in Escherichia coli is regulated at multiple levels by Hfq and small RNAs[J].Nucleic Acids Research, 2022, 50(12):6753-6768.
[51] BROSSE A, KOROBEINIKOVA A, GOTTESMAN S, et al.Unexpected properties of sRNA promoters allow feedback control via regulation of a two-component system[J].Nucleic Acids Research, 2016, 44(20):9650-9666.
[52] BROWNING D F, BUTALA M, BUSBY S J W.Bacterial transcription factors:Regulation by pick “N” mix[J].Journal of Molecular Biology, 2019, 431(20):4067-4077.
[53] DURAND S, CALLAN-SIDAT A, MCKEOWN J, et al.Identification of an RNA sponge that controls the RoxS riboregulator of central metabolism in Bacillus subtilis[J].Nucleic Acids Research, 2021, 49(11):6399-6419.
[54] DURAND S, BRAUN F, LIOLIOU E, et al.A nitric oxide regulated small RNA controls expression of genes involved in redox homeostasis in Bacillus subtilis[J].PLoS Genetics, 2015, 11(2):e1004957.
[55] LEJARS M, CAILLET J, SOLCHAGA-FLORES E, et al.Regulatory interplay between RNase III and antisense RNAs in E.coli:The case of AsflhD and FlhD, component of the master regulator of motility[J].mBio, 2022, 13(5):e0098122.
[56] KIM W, LEE Y.Mechanism for coordinate regulation of rpoS by sRNA-sRNA interaction in Escherichia coli[J].RNA Biology, 2020, 17(2):176-187.
[57] HAN R H, HANING K T, GONZALEZ-RIVERA J C, et al.Multiple small RNAs interact to co-regulate ethanol tolerance in zymomonas mobilis[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:155.
[58] LE HUYEN K B, GONZALEZ C D, PASCREAU G, et al.A small regulatory RNA alters Staphylococcus aureus virulence by titrating RNAIII activity[J].Nucleic Acids Research, 2021, 49(18):10644-10656.
[59] MELIOR H, LI S Q, STÖTZEL M, et al.Reprograming of sRNA target specificity by the leader peptide peTrpL in response to antibiotic exposure[J].Nucleic Acids Research, 2021, 49(5):2894-2915.