β-半乳糖苷酶是一种重要的酶,具有水解乳糖和合成低聚半乳糖的功能,通常来源于细菌、酵母菌、霉菌等微生物[1]。β-半乳糖苷酶在食品工业中广泛应用于制造乳糖水解物(供乳糖不耐受患者使用)以及形成糖基化产物,具有重要意义。牛乳中乳糖质量分数为4.5%~5.0%,乳糖经过人体小肠黏膜细胞中分泌的β-半乳糖苷酶水解后才能被机体吸收,缺乏该酶会导致乳糖不耐受。在非洲国家乳糖不耐症人群高达90%~100%,在亚洲占70%[2]。目前,解决乳糖不耐症的最佳方法是用β-半乳糖苷酶水解乳糖生产低乳糖或无乳糖乳制品[3]。常用的商业化β-半乳糖苷酶的最适反应温度大多较高,对pH的要求比较严格,从而存在生产成本较高、消耗能量高等问题。但是,在乳制品生产工业中,很多操作工艺都是在低温下进行,因此,理想化的β-半乳糖苷酶是能够在低温下具有较高酶活性的适冷酶。本文从β-半乳糖苷酶的微生物来源、特性、催化特性的研究现状进行综述。进一步对适冷β-半乳糖苷酶的来源、特性、耐冷机制及工业化应用进行详细的阐述。并对适冷β-半乳糖苷酶前景进行了展望,为该酶改造的深入探索与应用开发提供参考。
1 β-半乳糖苷酶来源、特性及催化机制
1.1 β-半乳糖苷酶来源及特性
β-半乳糖苷酶(β-galactosidase,EC:3.2.1.23)全称为β-D-半乳糖苷半乳糖水解酶(β-D-galactoside galactohydrolase),俗称乳糖酶,具有催化半乳糖和葡萄糖之间β-糖苷键功能,也参与合成半乳糖化产物或低聚半乳糖。根据基因序列、结构、底物特异性和反应机理不同,β-半乳糖苷酶分为糖苷水解酶(glycoside hydrolases,GH)GH1、GH2、GH35和GH42家族。大部分已报道的微生物来源的β-半乳糖苷酶属于GH2和GH42家族。
β-半乳糖苷酶来源广泛,主要来源于微生物,如细菌、酵母和丝状真菌。其中,酵母菌、霉菌是商业化β-半乳糖苷酶的主要来源,有曲霉菌属(Aspergillus)和克鲁维酵母菌属(Kluyveromyces)[4]。不同来源的β-半乳糖苷酶在酶学特征上存在较大差异。一般霉菌来源β-半乳糖苷酶的最适pH值为2.5~5.4,最适温度最高达到50 ℃。霉菌来源的β-半乳糖苷酶是胞外酶,便于应用于水解乳糖。相反,酵母菌来源的β-半乳糖苷酶是胞内酶,乳糖先通过透性酶转运至细胞内再被水解代谢[5]。来自乳酸克鲁维酵母的β-半乳糖苷酶最适pH值为4.8,最适温度是50 ℃;来自脆壁克鲁维酵母和黑曲霉的β-半乳糖苷酶的最适pH值为4.0~4.5,pH值为3.0~7.0时较为稳定,最适温度为55 ℃[6]。乳酸菌作为一般认为安全的微生物,也是β-半乳糖苷酶的较好来源。从植物乳杆菌BFP32中所分离得到的β-半乳糖苷酶的最适温度为60~65 ℃;从德氏乳杆菌和母乳中筛选出的发酵乳杆菌中的β-半乳糖苷酶的最适温度为40 ℃[7-8]。从保加利亚乳杆菌L2和嗜热链球菌S2 中分离出来的β-半乳糖苷酶,在42 ℃条件下发酵脱脂乳6 h时,其乳糖分解率均高于65%,乳糖残存量均低于2%[9]。MAISCHBERGER等[10]在戊糖乳杆菌中筛选的β-半乳糖苷酶的最适pH值为7.5,最适温度为55 ℃;以乳糖为底物,最适pH值为8.0,最适温度为60~65 ℃。从乳酸菌筛选出的β-半乳糖苷酶的活性相对高,但是,在中温或高温条件下其酶活性和水解乳糖能力相对高。
1.2 β-半乳糖苷酶的催化机制
1.2.1 分解乳糖的作用机制
乳糖不耐症在世界多数地区都存在,亚洲地区成年人中有乳糖不耐症占70%以上[2]。目前,乳制品用β-半乳糖苷酶预先处理后,乳制品中的乳糖含量可降低70%~80%。利用β-半乳糖苷酶生产低乳糖牛奶等乳制品成为解决乳糖不耐受问题最有效的途径[3]。除此之外,水解乳糖可减少由于乳糖的低溶解度而引起的结晶问题,并增加了乳制品的甜度。β-半乳糖苷酶对乳糖的水解分为糖基化和去糖基化两个步骤(图1-a)。首先是糖基化,乳糖通过催化反应将其分解成葡萄糖和酶-半乳糖共价中间复合物。其次是去糖基化,葡萄糖分离后使半乳糖的异头碳原子与水分子中的羟基基团生成半乳糖分子,从而达到把乳糖水解成为葡萄糖和半乳糖的目的[11]。
1.2.2 合成低聚半乳糖的作用机制
由半乳糖连接成聚合度为2~9的,通常在还原端含有一个葡萄糖单位的低聚糖为低聚半乳糖。β-半乳糖苷酶还可在人体内通过转糖苷作用生成低聚半乳糖,作为一种膳食纤维,它在肠道内作为增殖因子仅被双歧杆菌利用,却不会被腐败细菌利用,可大大减少肠道有害毒素的产生,对预防便秘和腹泻有很重要的作用[12]。与其他熟知的功能性低聚糖(如低聚果糖和低聚异麦芽糖)相比,低聚半乳糖是唯一能够被人体全部肠道益生菌利用的益生元。并且,低聚半乳糖具有轻微甜味,是蔗糖甜味的30%~60%。转半乳糖化是β-半乳糖苷酶的固有性质,在乳糖水解过程中会形成低聚糖。将乳糖的半乳糖部分转移到含有羟基的受体分子中,可产生具有不同聚合度和不同糖苷键的低聚糖。合成低聚半乳糖有以下3个步骤:1)释放葡萄糖残基,剩下的酶与半乳糖基残基复合体以进一步反应;2)将该复合体转移到含有羟基的受体中,溶液中低浓度的乳糖会刺激水分子充当受体并产生半乳糖;3)含有高浓度乳糖溶液中的乳糖作为受体,并与该复合物结合,最终产生低聚半乳糖[13](图1-b)。生产低聚半乳糖的商业化β-半乳糖苷酶来自于乳酸克鲁维酵母、环状芽孢杆菌、两岐双歧杆菌、米曲霉、嗜热链球菌等[14]。RABIU等[15]在各种双歧杆菌(如角双歧杆菌、两双歧杆菌BB-12、青年双歧杆菌ANB-7、婴儿双歧杆菌DSM-20088、假长双歧杆菌DSM-20099)的纯培养物中提取β-半乳糖苷酶合成低聚半乳糖。PANESAR等[16]研究了对底物配比、乳糖初始浓度、温度等因素对β-半乳糖苷酶催化乳糖转半乳糖化生成低聚半乳糖的影响。但β-半乳糖苷酶的转糖苷活性低,并且由于同时存在底物水解反应,导致产物往往包括多样化糖苷聚合度及糖苷键构成类型,并最终限制了低聚半乳糖合成量和纯度。
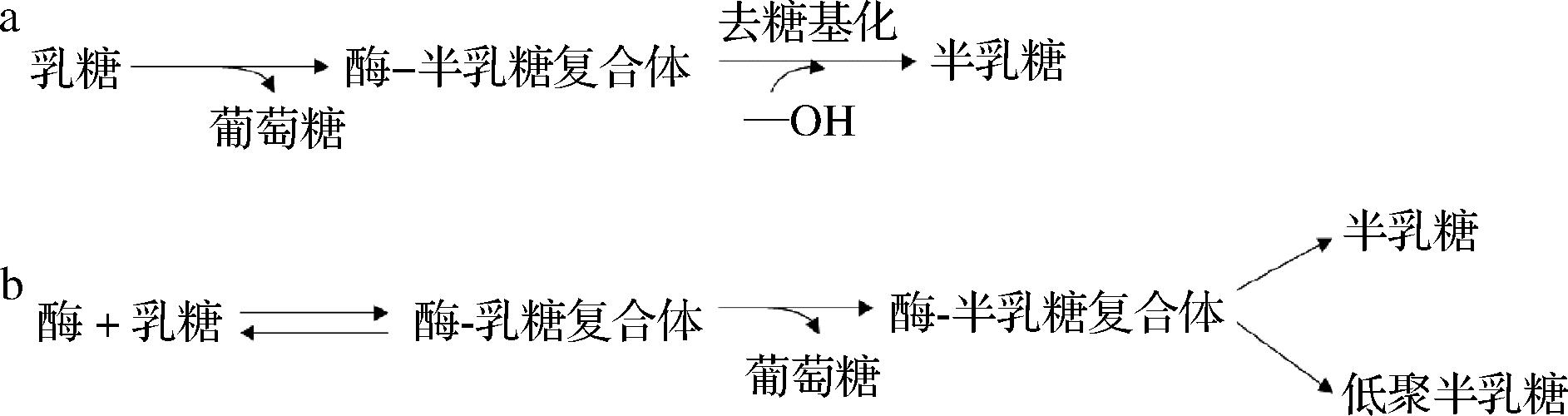
a-水解乳糖;b-合成低聚半乳糖
图1 β-半乳糖苷酶催化水解乳糖和合成低聚半乳糖的机理
Fig.1 Mechanisms of β-galactosidase catalyzed hydrolysis of lactose and synthesis of galactooligosaccharide
2 适冷β-半乳糖苷酶
2.1 适冷β-半乳糖苷酶的来源及特性
酶的催化活性及稳定性受温度的明显影响。根据温度对酶的活性及稳定性的影响,将酶分成适冷酶、中温酶和高温酶。适冷酶是一类在低温(一般在0~30 ℃)条件下具有高催化活性的酶,并且可降低结合过程中酶的变构作用和催化底物变化所需要的活化能,因此,适冷酶在低温时需要较少的能量就能完成催化过程。适冷酶的最适反应温度与嗜温酶相比要低20~30 ℃。目前,适冷β-半乳糖苷酶主要从适冷微生物中分离得到,这些微生物从极地、深海、高山地区等地方筛选,如节杆菌属(Arthrobacter)、动物球菌属(Planococcus)、假交替单胞菌属(Pseudoalteromonas)、盐单胞菌属(Halomonas)、拉恩氏菌属(Rahnella)、副球菌属(Paracoccus)、噬盐古细菌(Halorubrum lacusprofundi)[17-20],其酶学性质如表1所示。除了来自Arthrobacter sp.C2-2的β-半乳糖苷酶具有六聚体结构、来自副球菌属32 d的具有二聚体以外,适冷β-半乳糖苷酶一般都属于GH2家族,具有四聚体结构[17,19,30]。有些适冷微生物含有2种或多种编码β-半乳糖苷酶的基因,属于不同的家族。比如,节杆菌属含有2种β-半乳糖苷酶,分别属于GH2和GH42家族;适冷杆菌(Cryobacterium sp.LW097)含有4种β-半乳糖苷酶,其中1种属于GH35、3种属于GH42[29]。这些适冷性β-半乳糖苷酶的筛选,主要通过含有X-gal的培养基来进行筛选,β-半乳糖苷酶可以分解X-gal形成蓝色化合物。根据不同微生物选择不同培养基,如YPD培养基中或者乳清培养基加入X-gal[34-35]。
表1 不同微生物来源适冷β-半乳糖苷酶的酶学性质
Table 1 Enzymatic characteristics of β-galactosidase from different microorganisms
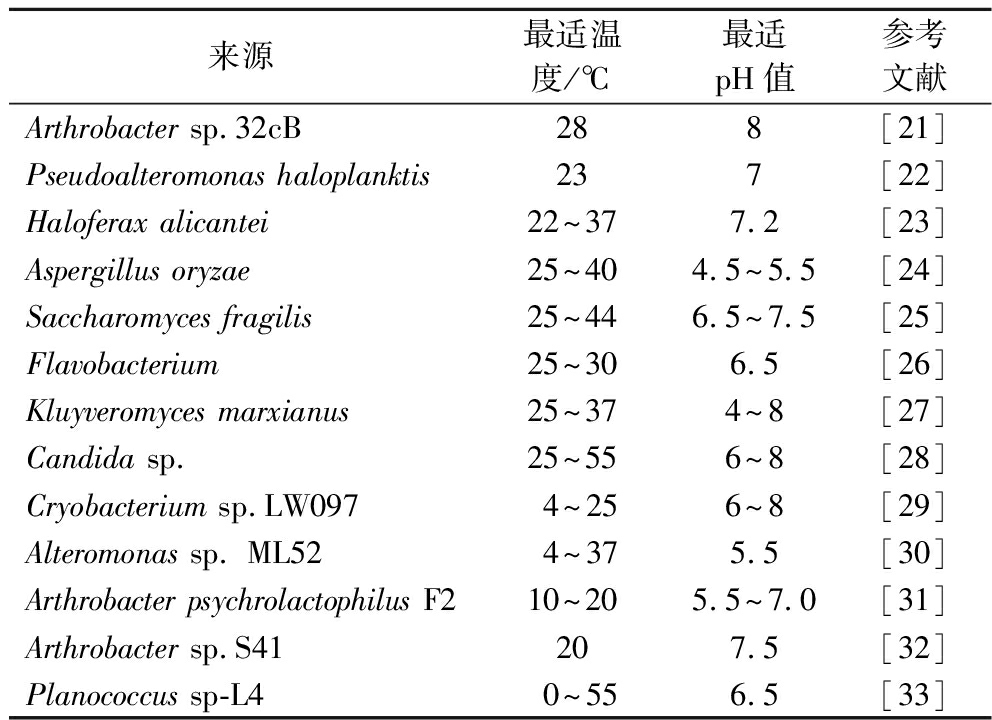
来源最适温度/℃最适pH值参考文献Arthrobacter sp.32cB288[21]Pseudoalteromonas haloplanktis237[22]Haloferax alicantei22~377.2[23]Aspergillus oryzae25~404.5~5.5[24]Saccharomyces fragilis25~446.5~7.5[25]Flavobacterium25~306.5[26]Kluyveromyces marxianus25~374~8[27]Candida sp.25~556~8[28]Cryobacterium sp.LW0974~256~8[29]Alteromonas sp. ML524~375.5[30]Arthrobacter psychrolactophilus F210~205.5~7.0[31]Arthrobacter sp.S41207.5[32]Planococcus sp-L40~556.5[33]
除了从天然适冷菌中分离出适冷性β-半乳糖苷酶,可以通过DNA重组技术和基因突变的技术获得具有适冷性能的β-半乳糖苷酶。CIE LI
LI SKI等[36]在大肠杆菌中表达了来自拟交替单胞菌(Pseudoalteromonas) 22b的β-半乳糖苷酶,观察到90%的牛奶乳糖在30 ℃下约6 h和15 ℃下28 h内被水解[36]。来自伊氏碱性乳杆菌(Alkalilactibacillus ikkense)的低温β-半乳糖苷酶,该重组酶在0 ℃时活性能够保持60%,在10 ℃或更低温度下稳定100 h以上[37]。此外,在冷休克蛋白(cspD2)基因启动子的控制下,从盐蓝菌(Halorubrum lacusprofundi)中扩增并克隆了嗜极 β-半乳糖苷酶基因,并在盐红菌(Halorubrum sp.NRC-1)中表达,表达量提高20倍,经纯化后在4 ℃条件下能够保持10%的酶活性[20]。NIE等[34]在毕赤酵母(Pichia pastoris)中表达了皱纹乳杆菌(Lactobacillus crispatus B470)的异二聚体β-半乳糖苷酶,并对重组β-半乳糖苷酶进行了纯化,也在20~30 ℃下保持20%的活性。从节杆菌(Alteromonas sp.ML117)分离的β-半乳糖苷酶在大肠杆菌中的表达,在10 ℃条件下24 h内能够降解牛奶中86%的乳糖[38]。来自海洋交替单胞菌(Alteromonas sp.QD01)的β-半乳糖苷酶基因在大肠杆菌中表达时,金属离子促进了β-半乳糖苷酶的活性。该酶的最适 pH 值和温度分别为 8.0 和 40 ℃,30 ℃时维持60%的酶活性[39]。WANG等[40]构建了一个土壤宏基因组文库,并成功分离到一个新的 β-半乳糖苷酶基因,并在毕赤酵母中表达和纯化,纯化后的酶在 38 ℃和pH=7.0 时表现出最高的酶活性,并在 0 ℃ 条件下可维持11%的活性,被认为是适冷β-半乳糖苷酶。
SKI等[36]在大肠杆菌中表达了来自拟交替单胞菌(Pseudoalteromonas) 22b的β-半乳糖苷酶,观察到90%的牛奶乳糖在30 ℃下约6 h和15 ℃下28 h内被水解[36]。来自伊氏碱性乳杆菌(Alkalilactibacillus ikkense)的低温β-半乳糖苷酶,该重组酶在0 ℃时活性能够保持60%,在10 ℃或更低温度下稳定100 h以上[37]。此外,在冷休克蛋白(cspD2)基因启动子的控制下,从盐蓝菌(Halorubrum lacusprofundi)中扩增并克隆了嗜极 β-半乳糖苷酶基因,并在盐红菌(Halorubrum sp.NRC-1)中表达,表达量提高20倍,经纯化后在4 ℃条件下能够保持10%的酶活性[20]。NIE等[34]在毕赤酵母(Pichia pastoris)中表达了皱纹乳杆菌(Lactobacillus crispatus B470)的异二聚体β-半乳糖苷酶,并对重组β-半乳糖苷酶进行了纯化,也在20~30 ℃下保持20%的活性。从节杆菌(Alteromonas sp.ML117)分离的β-半乳糖苷酶在大肠杆菌中的表达,在10 ℃条件下24 h内能够降解牛奶中86%的乳糖[38]。来自海洋交替单胞菌(Alteromonas sp.QD01)的β-半乳糖苷酶基因在大肠杆菌中表达时,金属离子促进了β-半乳糖苷酶的活性。该酶的最适 pH 值和温度分别为 8.0 和 40 ℃,30 ℃时维持60%的酶活性[39]。WANG等[40]构建了一个土壤宏基因组文库,并成功分离到一个新的 β-半乳糖苷酶基因,并在毕赤酵母中表达和纯化,纯化后的酶在 38 ℃和pH=7.0 时表现出最高的酶活性,并在 0 ℃ 条件下可维持11%的活性,被认为是适冷β-半乳糖苷酶。
乳酸菌是多种发酵食品的发酵剂。目前,关于乳酸菌中适冷β-半乳糖苷酶的研究极少,只有罗伊氏乳杆菌(Lactobacillus reuteri)L461和L103[41]。因此,通过基因突变或蛋白质结构变化提高β-半乳糖苷酶的耐冷性也是一个新的研究方向。
2.2 适冷β-半乳糖苷酶的适冷机制
目前,研究已报道关于适冷酶结构的一些变化:如酶分子具有更大的柔韧性和较小的功能域;减少了亚基表面之间的相互作用并增加了暴露于溶剂中的疏水残基的数量;酶的活性中心更容易接近底物,且酶与底物的亲和力更大等[42]。与中温酶、高温酶相比,适冷酶的催化腔中所有反应侧链的序列都具有高度保守型,这表明酶的催化机制并没有发生改变。但是,与同种酶的中温酶和高温酶相比,适冷酶的一级序列和高级结构存在差异,如甘氨酸含量增加,脯氨酸和精氨酸含量减少,盐桥、芳香环相互作用和疏水作用减弱;分子间、亚基间以及结构域间的相互作用减弱;与溶液间的相互作用增强;环状结构增加,使蛋白质分子具有较高的柔韧性和较低的热稳定性[43]。酶活性位点的亲和性改变也是适冷酶的一种特点,具有大的、开放式的催化裂口[44]。虽然适冷酶的亲和机理尚未清楚,但一些研究表明,适冷酶可在低能量消耗状态下与底物亲和,酶的构象改变。因此,适冷酶分子的整体结构变化对适冷酶在低温下的催化效果起到重要作用。
一般情况下,温度降低10 ℃,酶的反应速度会降低2~3倍。研究表明,与中温和高温酶相比,适冷酶可以降低反应活化能,在低温下的催化速率(kcat)增加,其Km有所下降。然而,一般适冷酶在低温下的活性提高是通过优化Km值来实现,其通过改变蛋白质序列及结构增加蛋白质的柔韧性。MANGIAGALLI等[45]通过减弱盐桥、芳香环相互作用和疏水作用及降低金属结合亲和力的方法提高适冷酶的活性。适冷酶与中温和高温酶相比,在低温下具有较高的比活力和较低的最适催化温度(Topt,20~45 ℃)。虽然有些适冷酶的Topt与嗜热酶同系物没有差别[46]。尤其是,适冷酶的失活温度低于蛋白质的变性温度(Tm),这表明酶的活性中心比整体结构更不耐热[47]。然而,中温和高温酶的失活温度与蛋白质变性温度相同(Topt=Tm)。此外,与中温酶和高温酶相比,适冷酶具有较低的热稳定性。FELLER[48]测定来自适冷、中温和高温微生物的α-淀粉酶分别在50 ℃下完全失活的时间,其中适冷酶60 min后完全失去活性,而中温和高温酶100 min后还保持60%和90%的活性。
适冷酶与中温酶同系物之间的特性比较如图2所示。

a-最适反应温度;b-热稳定性;c-蛋白质在折叠/伸展转变的 差示扫描量热法图谱;d-圆二色光谱或荧光光谱法
图2 适冷酶与中温酶同系物的特性分析
Fig.2 Characteristic analysis of a homologue of cold-adaptive enzyme and mesophilic enzyme
图2-a比较了适冷酶和中温酶的最适反应温度,表示适冷酶在低温下更活跃,适冷酶的最适温度在20~45 ℃,而中温酶在50~60 ℃。图2-b比较了特定温度下的酶的热稳定性,适冷酶的热稳定性低,在Topt温度下,酶活性能维持90 min左右。相比之下,中温酶和高温酶在相同温度下可维持更长时间。图2-c和2-d用差示扫描量热法和圆二色光谱或荧光光谱法测定了蛋白质在折叠/伸展转变时的热量。适冷酶具有较低的Tm值、量热焓(ΔHcal)[49]。
3 适冷β-半乳糖苷酶的优点及工业化应用
在食品生产中利用适冷酶的最大优点是能够保护耐冷性生物活性成分。适冷β-半乳糖苷酶主要用于乳糖的水解和低聚糖的生产。β-半乳糖苷酶主要用来治疗乳糖不耐受症,处理加工牛乳、乳清,生产低乳糖牛奶和乳制品。
3.1 适冷β-半乳糖苷酶在低乳糖和无乳糖乳制品工业中的应用
β-半乳糖苷酶被广泛用于生产无乳糖乳制品,是专门为消化系统中缺乏β-半乳糖苷酶的乳糖不耐受患者设计的。此外,β-半乳糖苷酶用于冰淇淋和炼乳生产,以避免乳糖结晶,并提高这些产品的甜度和奶油味[3]。无乳糖牛奶是在牛奶储存期间(分批过程)或在超高温处理(无菌过程)之后添加可溶性β-半乳糖苷酶生产的(图3)。在无乳糖乳制品生产工业中,理想化的β-半乳糖苷酶在4~8 ℃具有较高的酶活性,且能够耐受Na+、Ca2+、葡萄糖的抑制[50]。目前,用于低温水解乳糖的适冷β-半乳糖苷酶来自于交替单胞菌属、低温杆菌、路氏肠杆菌(Enterobacter ludwigii)、欧文氏菌属微生物(Erwinia amylovora)等[51]。含有适冷β-半乳糖苷酶的菌株可以在低于30 ℃的温度下以乳糖作为唯一碳源生长,如假交替单胞菌DFR1和DFR6可以在添加1%乳糖的营养肉汤中生长,因此特性该菌株也被应用于大规模水解乳糖[6]。WIERZBICKA-WOS等[31]从副球菌属32 d中分离出来的适冷β-半乳糖苷酶,也适用于在冷藏温度下水解牛奶中的乳糖。FAN等[18]将来自耐冷菌拉恩氏菌属R3的编码适冷β-半乳糖苷酶的基因在大肠杆菌中表达,发现该酶在4 ℃下也具有高活性,可分解冷藏牛奶中的乳糖,并在巴氏杀菌时失活,最大限度保存牛奶的营养成分。适冷β-半乳糖苷酶在低温下可水解牛奶中的乳糖保持原有味道,也提高对乳糖不耐症人群的营养价值。此外,适冷β-半乳糖苷酶乳糖水解能力高,可缩短水解时间,也可以减少细菌污染的风险。因此,在较低温度和较大pH范围下具有高活性的耐冷β-半乳糖苷酶,对于降低成本、节约能源,具有重要的应用价值。
3.2 适冷β-半乳糖苷酶在奶酪乳清乳糖分解中的应用
奶酪乳清是奶酪制作过程的主要副产品之一。乳清是酪蛋白凝聚和凝乳分离后得到的液体相,含有乳糖、蛋白质、脂类和矿物盐。乳清在生产中常常被当做废液直接排放,不仅浪费资源,而且污染环境。乳清可作为微生物发酵或化学修饰生产高价值化合物的乳糖来源。在此情况下,先用β-半乳糖苷酶进行预处理,以获得葡萄糖和半乳糖,更适合作为微生物生长的底物(图3)。但目前利用适冷β-半乳糖苷酶水解乳清的研究极少,来自拟交替单胞菌的适冷β-半乳糖苷酶水解乳清中的乳糖生产低热量天然甜味剂——D-塔格糖。在此过程中,乳糖在23 ℃下进行水解,不需要额外的进行冷却或加热工艺[22]。
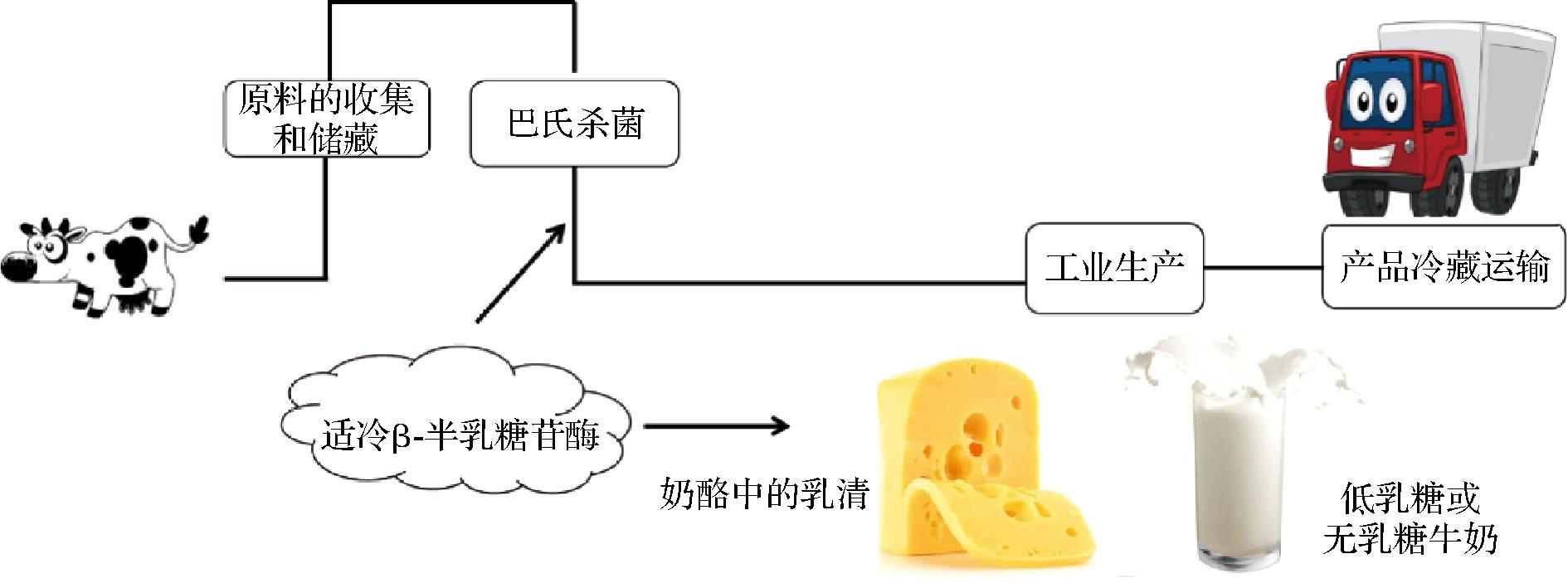
图3 适冷β-半乳糖苷酶在低乳糖或无乳糖牛奶和 奶酪乳清中分解乳糖的应用
Fig.3 Application of cold-adapted β-galactosidase in lactose hydrolysis of low-lactose or non-lactose milk and cheese whey
3.3 适冷β-半乳糖苷酶在低聚半乳糖合成中的应用
低聚半乳糖是一种益生元,可以促进有益的肠道细菌(如乳杆菌或双歧杆菌)的生长,抑制病原菌在胃肠道中的定殖。并且,在化妆品和食品工业中分别用作添加剂和甜味剂。来自南极细菌Alteromonas sp., 南极节杆菌属的适冷β-半乳糖苷酶可应用于低聚半乳糖的合成[52-53]。此外,可利用来自节杆菌属的适冷β-半乳糖苷酶合成各种寡糖,如乳果糖、低聚木糖、低聚阿拉伯糖[53]。
4 展望
目前,对β-半乳糖苷酶的特性分析研究较多,也广泛应用于乳糖的水解和低聚半乳糖的合成。具有较高β-半乳糖苷酶活性的菌株有利于提高食品中乳糖的水解率,减少乳糖不耐受的影响,合成低聚半乳糖刺激人体小肠中有益细菌的增殖。由于实际乳制品生产操作在低温下进行,在低温下具有较高酶活性的适冷β-半乳糖苷酶的研究和应用也越来越多。然而,适冷β-半乳糖苷酶研究不够成熟。目前,适冷β-半乳糖苷酶主要从极地、深海、高山地区等地方筛选出的适冷微生物中分离得到。与同种酶的中温酶和高温酶相比,分析它们的结构特征推理耐冷机制,可将其小规模的应用到乳糖、乳清的水解和低聚半乳糖合成中。对适冷β-半乳糖苷酶,从以下几点进行深入研究:1)挖掘更多的适冷β-半乳糖苷酶的微生物菌株。乳酸菌是各种乳制品中的主要微生物,可作为适冷β-半乳糖苷酶的主要微生物。可通过将耐冷微生物的β-半乳糖苷酶基因导入到乳酸菌中,获得带有适冷β-半乳糖苷酶的乳酸菌,应用到发酵食品中。也可通过对乳酸菌中的β-半乳糖苷酶基因或氨基酸序列进行突变或蛋白质结构变化提高其耐冷性。2)通过分子技术提高适冷β-半乳糖苷酶在低温下的活性及热稳定性研究。体外蛋白质定向改造是获得适冷酶的一种新型方法。LAYE等[54]最新研究对南极耐盐性β-半乳糖苷酶的几个氨基酸进行突变,改变蛋白质的水合层提高耐冷性。研究显示,很多与耐冷性有关的蛋白质结构特性尚未发现,需要不断研究耐冷蛋白质及其机制进行研究。3)扩大适冷β-半乳糖苷酶的应用范围。适冷β-半乳糖苷酶可应用于炼乳、酸奶等乳制品加工中。在炼乳加工过程中,水解乳糖使得浓缩时避免结晶,产品更细腻、减少蔗糖用量。制造低乳糖酸奶,可加速和提高发酵效率,使酸奶具有独特的风味。
[1] DAMIN B I S, KOVALSKI F C, FISCHER J, et al.Challenges and perspectives of the β-galactosidase enzyme[J].Applied Microbiology and Biotechnology, 2021, 105(13):5281-5298.
[2] CATANZARO R, SCIUTO M, MAROTTA F.Lactose intolerance:An update on its pathogenesis, diagnosis, and treatment[J].Nutrition Research, 2021, 89:23-34.
[3] ABDEL WAHAB W A, AHMED S A, KHOLIF A M M, et al.Rice straw and orange peel wastes as cheap and eco-friendly substrates:A new approach in β-galactosidase (lactase) enzyme production by the new isolate L.paracasei MK852178 to produce low-lactose yogurt for lactose-intolerant people[J].Waste Management, 2021, 131:403-411.
[4] DE P DUA ALVES É, MORIOKA L R I, SUGUIMOTO H H.Comparison of bioethanol and beta-galactosidase production by Kluyveromyces and Saccharomyces strains grown in cheese whey[J].International Journal of Dairy Technology, 2019, 72(3):409-415.
DUA ALVES É, MORIOKA L R I, SUGUIMOTO H H.Comparison of bioethanol and beta-galactosidase production by Kluyveromyces and Saccharomyces strains grown in cheese whey[J].International Journal of Dairy Technology, 2019, 72(3):409-415.
[5] DE CARVALHO C T, DE OLIVEIRA S D Jr, DE BRITO LIMA W B, et al.Potential of “coalho” cheese whey as lactose source for β-galactosidase and ethanol co-production by Kluyveromyces spp.yeasts[J].Preparative Biochemistry &Biotechnology, 2020, 50(9):925-934.
[6] XAVIER J R, RAMANA K V, SHARMA R K.β-galactosidase:Biotechnological applications in food processing[J].Journal of Food Biochemistry, 2018, 42(5):e12564.
[7] HEMMARATCHIRAKUL J, JATURAPIREE P, PRUEKSASRI S, et al.Production of galactooligosaccharide by β-galactosidase from Lactobacillus pentosus var.plantarum BFP32[J].International Food Research Journal, 2015, 22(6):2550.
[8] GOMAA E.β-galactosidase from Lactobacillus delbrueckii and Lactobacillus reuteri:Optimization, characterization and formation of galactooligosaccharides[J].Indian Journal of Biotechnology, 2018, 17:407-415.
[9] RHIMI M, AGHAJARI N, JAOUADI B, et al.Exploring the acidotolerance of β-galactosidase from Lactobacillus delbrueckii subsp.bulgaricus:An attractive enzyme for lactose bioconversion[J].Research in Microbiology, 2009, 160(10):775-784. [10] MAISCHBERGER T, LEITNER E, NITISINPRASERT S, et al.Beta-galactosidase from Lactobacillus pentosus:Purification, characterization and formation of galacto-oligosaccharides[J].Biotechnology Journal, 2010, 5(8):838-847.
[11] SHAFI A, AHMED F, HUSAIN Q.β-Galactosidase mediated synthesized nanosupport for the immobilization of same enzyme:Its stability and application in the hydrolysis of lactose[J].International Journal of Biological Macromolecules, 2021, 184:57-67.
[12] SOUZA A F C E, GABARDO S, COELHO R J S.Galactooligosaccharides:Physiological benefits, production strategies, and industrial application[J].Journal of Biotechnology, 2022, 359:116-129.
[13] SAQIB S, AKRAM A, HALIM S A, et al.Sources of β-galactosidase and its applications in food industry[J].3 Biotech, 2017, 7(1):79.
[14] BOTVYNKO A, BEDN
 OV
OV A, HENKE S, et al.Production of galactooligosaccharides using various combinations of the commercial β-galactosidases[J].Biochemical and Biophysical Research Communications, 2019, 517(4):762-766.
A, HENKE S, et al.Production of galactooligosaccharides using various combinations of the commercial β-galactosidases[J].Biochemical and Biophysical Research Communications, 2019, 517(4):762-766.
[15] RABIU B A, JAY A J, GIBSON G R, et al.Synthesis and fermentation properties of novel galacto-oligosaccharides by beta-galactosidases from Bifidobacterium species[J].Applied and Environmental Microbiology, 2001, 67(6):2526-2530.
[16] PANESAR P S, KAUR R, SINGH R S, et al.Biocatalytic strategies in the production of galacto-oligosaccharides and its global status[J].International Journal of Biological Macromolecules, 2018, 111:667-679.
[17] PAWLAK-SZUKALSKA A, WANARSKA M, POPINIGIS A T, et al.A novel cold-active β-d-galactosidase with transglycosylation activity from the Antarctic Arthrobacter sp.32cB-Gene cloning, purification and characterization[J].Process Biochemistry, 2014, 49(12):2122-2133.
[18] FAN Y T, HUA X, ZHANG Y Z, et al.Cloning, expression and structural stability of a cold-adapted β-galactosidase from Rahnella sp.R3[J].Protein Expression and Purification, 2015, 115:158-164.
[19] RUTKIEWICZ-KROTEWICZ M, PIETRZYK-BRZEZINSKA A J, SEKULA B, et al.Structural studies of a cold-adapted dimeric β-D-galactosidase from Paracoccus sp.32 d[J].Acta Crystallographica Section D Structural Biology, 2016, 72(9):1049-1061.
[20] RAM K, CAPES MELINDA D, PRIYA D, et al.Cloning, overexpression, purification, and characterization of a polyextremophilic β-galactosidase from the Antarctic haloarchaeon Halorubrum lacusprofundi[J].BMC Biotechnology, 2013, 13(1):3.
[21] RUTKIEWICZ M, WANARSKA M, BUJACZ A.Mapping the transglycosylation relevant sites of cold-adapted β-d-galactosidase from Arthrobacter sp.32cB[J].International Journal of Molecular Sciences, 2020, 21(15):5354.
[22] VAN DE VOORDE I, GOIRIS K, SYRYN E, et al.Evaluation of the cold-active Pseudoalteromonas haloplanktis β-galactosidase enzyme for lactose hydrolysis in whey permeate as primary step of D[J].Process Biochemistry, 2014, 49(12):2134-2140.
[23] KIXMÜLLER D, STRAHL H, WENDE A, et al.Archaeal transcriptional regulation of the prokaryotic KdpFABC complex mediating K+ uptake in H.salinarum[J].Extremophiles, 2011, 15(6):643.
[24] GUERRERO C, VERA C, ILLANES A.Fed-batch operation for the synthesis of lactulose with β-galactosidase of Aspergillus oryzae[J].Bioresource Technology, 2017, 237:126-134.
[25] MORIOKA L R I, DOS SANTOS VIANA C, DE P DUA ALVES É, et al.Concentrated beta-galactosidase and cell permeabilization from Saccharomyces fragilis IZ 275 for beta-galactosidase activity in the hydrolysis of lactose[J].Food Science and Technology, 2019, 39(3):524-530.
DUA ALVES É, et al.Concentrated beta-galactosidase and cell permeabilization from Saccharomyces fragilis IZ 275 for beta-galactosidase activity in the hydrolysis of lactose[J].Food Science and Technology, 2019, 39(3):524-530.
[26] KUSCHEL B, RIEMER F, PFOST D, et al.Large-scale purification of epilactose using a semi-preparative HPLC system[J].European Food Research and Technology, 2017, 243(3):391-402.
[27] OZARSLAN S, KÖK TA T.Treatment of lactose by fermentation:Production process on β-galactosidase using Kluyveromyces marxianus isolated from kefir grains[J].International Food Research Journal, 2022, 29(4):864-871.
T.Treatment of lactose by fermentation:Production process on β-galactosidase using Kluyveromyces marxianus isolated from kefir grains[J].International Food Research Journal, 2022, 29(4):864-871.
[28] MONIER M, YOUSSEF I, ABDEL-LATIF D A.Synthesis of photo-responsive chitosan-cinnamate for efficient entrapment of β-galactosidase enzyme[J].Reactive and Functional Polymers, 2018, 124:129-138.
[29] WANG L J, MOU Y, GUAN B, et al.Genome sequence of the psychrophilic Cryobacterium sp.LW097 and characterization of its four novel cold-adapted β-galactosidases[J].International Journal of Biological Macromolecules, 2020, 163:2068-2083.
[30] SUN J J, YAO C Y, WANG W, et al.Cloning, expression and characterization of a novel cold-adapted β-galactosidase from the deep-sea bacterium Alteromonas sp.ML52[J].Marine Drugs, 2018, 16(12):469.
[31] WIERZBICKA-WOS A, CIE LI
LI SKI H, WANARSKA M, et al.A novel cold-active β-D-galactosidase from the Paracoccus sp.32 d-gene cloning, purification and characterization[J].Microbial Cell Factories, 2011, 10:12.
SKI H, WANARSKA M, et al.A novel cold-active β-D-galactosidase from the Paracoccus sp.32 d-gene cloning, purification and characterization[J].Microbial Cell Factories, 2011, 10:12.
[32] HENNESSY R C, NIELSEN S D, GREVE-POULSEN M, et al.Discovery of a bacterial gene cluster for deglycosylation of toxic potato steroidal glycoalkaloids α-chaconine and α-solanine[J].Journal of Agricultural and Food Chemistry, 2020, 68(5):1390-1396.
[33] MAHDIAN S M A, KARIMI E, TANIPOUR M H, et al.Expression of a functional cold active β-galactosidase from Planococcus sp-L4 in Pichia pastoris[J].Protein Expression and Purification, 2016, 125:19-25.
[34] NIE C M, LIU B, ZHANG Y H, et al.Production and secretion of Lactobacillus crispatus β-galactosidase in Pichia pastoris[J].Protein Expression and Purification, 2013, 92(1):88-93.
[35] DE F TIMA MATOS DE FREITAS M, HORT
TIMA MATOS DE FREITAS M, HORT NCIO L C, ALBUQUERQUE T L, et al.Simultaneous hydrolysis of cheese whey and lactulose production catalyzed by β-galactosidase from Kluyveromyces lactis NRRL Y1564[J].Bioprocess and Biosystems Engineering, 2020, 43(4):711-722.
NCIO L C, ALBUQUERQUE T L, et al.Simultaneous hydrolysis of cheese whey and lactulose production catalyzed by β-galactosidase from Kluyveromyces lactis NRRL Y1564[J].Bioprocess and Biosystems Engineering, 2020, 43(4):711-722.
[36] CIE LI
LI SKI H, KUR J, BIA
SKI H, KUR J, BIA KOWSKA A, et al.Cloning, expression, and purification of a recombinant cold-adapted β-galactosidase from Antarctic bacterium Pseudoalteromonas sp.22b[J].Protein Expression and Purification, 2005, 39(1):27-34.
KOWSKA A, et al.Cloning, expression, and purification of a recombinant cold-adapted β-galactosidase from Antarctic bacterium Pseudoalteromonas sp.22b[J].Protein Expression and Purification, 2005, 39(1):27-34.
[37] SCHMIDT M, STOUGAARD P.Identification, cloning and expression of a cold-active beta-galactosidase from a novel Arctic bacterium, Alkalilactibacillus ikkense[J].Environmental Technology, 2010, 31(10):1107-1114.
[38] YAO C Y, SUN J J, WANG W, et al.A novel cold-adapted β-galactosidase from Alteromonas sp.ML117 cleaves milk lactose effectively at low temperature[J].Process Biochemistry, 2019, 82:94-101.
[39] LI D D, LI S Y, WU Y H, et al.Cloning and characterization of a new β-galactosidase from Alteromonas sp.QD01 and its potential in synthesis of galacto-oligosaccharides[J].Marine Drugs, 2020, 18(6):312.
[40] WANG K, LI G, YU S Q, et al.A novel metagenome-derived β-galactosidase:Gene cloning, overexpression, purification and characterization[J].Applied Microbiology and Biotechnology, 2010, 88(1):155-165.
[41] NGUYEN T H, SPLECHTNA B, STEINBÖCK M, et al.Purification and characterization of two novel beta-galactosidases from Lactobacillus reuteri[J].Journal of Agricultural and Food Chemistry, 2006, 54(14):4989-4998.
[42] DONG Y W, LIAO M L, MENG X L, et al.Structural flexibility and protein adaptation to temperature:Molecular dynamics analysis of malate dehydrogenases of marine molluscs[J].Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(6):1274-1279.
[43] ÖTEN A M, ATAK E, TAKTAK KARACA B, et al.Discussing the roles of proline and glycine from the perspective of cold adaptation in lipases and cellulases[J].Biocatalysis and Biotransformation, 2023, 41(4):243-260.
[44] PARVIZPOUR S, HUSSIN N, SHAMSIR M S, et al.Psychrophilic enzymes:Structural adaptation, pharmaceutical and industrial applications[J].Applied Microbiology and Biotechnology, 2021, 105(3):899-907.
[45] MANGIAGALLI M, BROCCA S, ORLANDO M, et al.The “cold revolution”.Present and future applications of cold-active enzymes and ice-binding proteins[J].New Biotechnology, 2020, 55:5-11.
[46] PISCHEDDA A, RAMASAMY K P, MANGIAGALLI M, et al.Antarctic marine ciliates under stress:Superoxide dismutases from the psychrophilic Euplotes focardii are cold-active yet heat tolerant enzymes[J].Scientific Reports, 2018, 8:14721.
[47] SO AN J, PURG M, ÅQVIST J.Computer simulations explain the anomalous temperature optimum in a cold-adapted enzyme[J].Nature Communications, 2020, 11:2644.
AN J, PURG M, ÅQVIST J.Computer simulations explain the anomalous temperature optimum in a cold-adapted enzyme[J].Nature Communications, 2020, 11:2644.
[48] FELLER G.Protein stability and enzyme activity at extreme biological temperatures[J].Journal of Physics:Condensed Matter, 2010, 22(32):323101.
[49] MANGIAGALLI M, LOTTI M.Cold-active β-galactosidases:Insight into cold adaption mechanisms and biotechnological exploitation[J].Marine Drugs, 2021, 19(1):43.
[50] CHANALIA P, GANDHI D, ATTRI P, et al.Purification and characterization of β-galactosidase from probiotic Pediococcus acidilactici and its use in milk lactose hydrolysis and galactooligosaccharide synthesis[J].Bioorganic Chemistry, 2018, 77:176-189.
[51] CHEN Q M, WU Y C, HUANG Z L, et al.Cold-active enzymes in the dairy industry:Insight into cold adaption mechanisms and their applications[J].Trends in Food Science &Technology, 2022, 125:126-135.
[52] LI S, ZHU X, XING M.A new β-galactosidase from the Antarctic bacterium Alteromonas sp.ANT48 and its potential in formation of prebiotic galacto-oligosaccharides[J].Marine Drugs, 2019, 17(11):599.
[53] PAWLAK-SZUKALSKA A, WANARSKA M, POPINIGIS A T, et al.A novel cold-active β-D-galactosidase with transglycosylation activity from the Antarctic Arthrobacter sp.32cB-Gene cloning, purification and characterization[J].Process Biochemistry, 2014, 49(12):2122-2133.
[54] LAYE V J, DASSARMA S.Double mutations far from the active site affect cold activity in an Antarctic halophilic β-galactosidase[J].Protein Science, 2022, 31(3):677-687.