近些年来,随着碳中和概念的提出和环境保护意识的逐渐增强,通过全生物法合成工业化学品原料得到了广泛的关注。伴随着近年来代谢工程和合成生物学的不断发展,利用如大肠杆菌(Escherichia coli)、恶臭假单胞菌(Pseudomonas putida)等模式微生物合成二元羧酸的研究取得了一定的进展[1-3]。己二酸是一种中长链二元羧酸,可应用于尼龙材料、食品添加剂以及生物可降解塑料等多种工业制品中[4-5]。
生物法生产己二酸主要有3种路径:逆/顺β-氧化途径结合ω-氧化途径、逆己二酸降解途径以及α-酮庚二酸途径。在逆/顺β-氧化途径结合ω-氧化途径中,CLOMBURG等[6]通过在大肠杆菌中过表达硫解酶、3-羟基乙酰辅酶A脱氢酶/脱水酶、反式烯酰辅酶A还原酶、ω-羧化酶、醇脱氢酶以及醛脱氢酶编码基因构建该途径,通过敲除与己二酸合成途径竞争碳流的部分基因,合成了170 mg/L己二酸。DENG等[7]发现一株嗜热放线菌(Thermobifida fusca) B6可以通过逆己二酸降解途径天然产己二酸,该途径包括β-酮硫解酶、3-羟酰基辅酶A脱氢酶、3-羟基己二酰辅酶A脱氢酶、5-羧基-2-戊烯酰辅酶A还原酶以及己二酰辅酶A合成酶[2],在大肠杆菌中异源表达上述6个酶后,更换启动子提高限速酶5-羰基-2-戊烯酰辅酶A还原酶的表达量,敲除ldhA、atoB和sucD基因减少乳酸以及乙酸积累,增加丁二酰辅酶A积累,使己二酸产量达到1.8 g/L。α-酮庚二酸途径包括高柠檬酸合成酶、高柠檬酸脱水酶、顺高乌头酸脱水酶、苏式异高柠檬酸脱氢酶、α-酮庚二酸脱羧酶以及转氨酶,TURK等[8]以此构建了一条6-氨基己酸的合成路线,然而副产物己二酸的产量(0.32 g/L)远远高于目标产物6-氨基己酸(0.02 g/L),推测是大肠杆菌中内源的半醛脱氢酶催化己二酸半醛生成了己二酸。总的来看,逆己二酸降解途径是一种有效的己二酸合成代谢途径[9],但存在反应途径较长、外源基因表达不平衡等问题,导致难以对其进行改造调控。
在微生物中过表达外源基因往往会对菌体生长产生代谢负担[10],在模式微生物中代谢途径基因大多利用T7启动子进行表达,而该启动子的转录以及翻译强度较强,对菌体造成的代谢负担较大从而导致目标产物产量无法提高[11],LIU等[12]在3-羟基丙酸代谢途径中利用T7启动子分别表达一种丙二酰辅酶A还原酶的两个表达模块,3-羟基丙酸产量仅达到0.15 g/L。后续通过平衡两个模块的表达水平,3-羟基丙酸的产量达到3.72 g/L,较之前有了显著提升。XU等[13]通过更换启动子调节紫色杆菌素代谢途径基因的表达水平,使该产物的产量较对照提升了3.2倍。同样,LIU等[14]通过更换启动子强度确定了玉米黄质合成途径中SgcE10与SgcE表达水平的最优比例,高于或低于该比例都会导致玉米黄质产量显著下降。此外,还可以通过更换、优化复制子、质粒结构等[15-17]方法对代谢途径基因的表达水平进行优化。
为协调逆己二酸降解途径基因表达,本研究将实验室前期构建的逆己二酸降解途径分为4个模块,选取不同强度的启动子构建相应质粒并进行组合,利用构建的己二酸生物传感器开展高通量筛选验证,最终最优组合产量达到120.60 mg/L。在此基础上,开展发酵条件优化,己二酸产量达到240.49 mg/L,较对照提升了16.65倍。
1 材料与方法
1.1 实验材料
1.1.1 菌株
大肠杆菌(Escherichia coli)JM109、DH5α为构建质粒的宿主菌株;大肠杆菌K12 MG1655为己二酸传感器宿主菌株以及扩增paaJ、paaH、paaF基因的菌株;大肠杆菌K12 MG1655 ΔatoB ΔsucD ΔpflB ΔadhE ΔarcA ΔldhA ΔpoxB Δpta 为表达逆己二酸降解途径的宿主菌株。
1.1.2 培养基
LB培养基(g/L):NaCl 10,胰蛋白胨10,酵母粉5;
TB培养基(g/L):K2HPO4·3H2O 16.34,KH2PO4 2.31,胰蛋白胨 12,酵母粉 24;
Modified TB培养基(g/L):K2HPO4·3H2O 8.17,KH2PO4 2.31,胰蛋白胨12,酵母粉 24,NH4Cl 10.70,CuSO4·5H2O 2.5×10-6。
1.1.3 试剂
酵母提取物、胰蛋白胨,英国Oxoid公司;抗生素(卡那霉素以及链霉素),生工生物工程(上海)股份有限公司;其余所有试剂购自中国医药集团有限公司或北京伊诺凯科技有限公司。
1.1.4 仪器与设备
ETC811 PCR仪,德国Eppendorf公司;SYNERGY H1多功能酶标仪,美国Bio-Tek有限公司;ST 16R冷冻离心机,赛默飞世尔科技(中国)有限公司;1260 Infinity II高效液相色谱,美国安捷伦有限公司;Waters Acquity UPLC超高效液相色谱、BEH C18色谱柱(2.1 mm×150 mm, 1.7 μm)、MALDI SYNAPT Q-TOF MS质谱,美国沃特世公司;HPX-87H色谱柱,美国伯乐公司;UV-1800紫外可见分光光度计,翱艺仪器(上海)有限公司。
1.2 实验方法
1.2.1 质粒构建
诺唯赞2×Rapid taq Master Mix以及2×Phanta Flash Mix (Dye Plus)被分别用于菌落PCR和基因/启动子扩增。密码子优化后的acot8、cat1、ter、benM基因由苏州金唯智科技有限公司合成。paaJ、paaH以及paaF基因由大肠杆菌K12 MG1655(实验室保藏)扩增而来。组成型启动子PrpsU以及Pmdh源自ZHOU等[18]构建的组成型启动子库。组成型启动子PPL818、PPL3230、PPL2536、PPL106、PPL729、PPL1937、PPL3206以及PPL2787源自ZHAO等[19]等构建的组成型启动子库。空载质粒pCDFDuet-1以及pRSFDuet-1被作为载体表达逆己二酸降解途径基因。
质粒pCDF-PPL729-cat1-paaF-PPL3206-ter的构建过程如下:首先,以pCDF- Pmdh-cat1-paaF-PrpsU-ter为模板,在需要更换启动子的位点(Pmdh)设计响应引物进行PCR扩增并回收片段。其次,以组成型启动子PPL729片段为载体,回收片段为插入片段进行同源重组构建pCDF- PPL729-cat1-paaF-PrpsU-ter质粒。随后,以pCDF- PPL729-cat1-paaF-PrpsU-ter,在需要更换启动子的位点(PrpsU)设计响应引物进行PCR扩增并回收片段,再以组成型启动子PPL3206片段为载体,回收片段为插入片段进行同源重组构建pCDF-PPL729-cat1-paaF-PPL3206-ter质粒。其余质粒均按以上方法进行构建。
1.2.2 摇瓶及孔板发酵方法
摇瓶发酵:将保存在-80 ℃的菌液或平板上的单菌落接种于含有10 mL LB培养基的50 mL 三角瓶中,37 ℃, 250 r/min培养12~16 h后,以2%接种量转接至含有50 mL新鲜培养基的250 mL三角瓶中,开始发酵,培养条件为37 ℃, 200 r/min。
孔板发酵:将平板上的单菌落接种至48孔板中,37 ℃, 250 r/min培养12~16 h后,以2%接种量转接至24孔板中进行发酵,培养条件为37 ℃, 250 r/min。
1.2.3 高通量筛选
初筛:从平板上挑选的单菌落在24孔板发酵48 h后,将发酵液于12 000 r/min离心10 min,取上清液500 μL以1∶1的配比加入培养至对数生长期(OD600=0.6~0.8)的己二酸传感器发酵液中,30 ℃, 250 r/min培养4 h后测定荧光。将荧光值高的菌种保藏于-80 ℃冰箱中。
复筛:将荧光值高的菌种接种于含有10 mL LB培养基的50 mL 三角瓶中,37 ℃, 250 r/min培养12~16 h后,以2%接种量转接至含有50 mL新鲜培养基的250 mL三角瓶中,37 ℃, 200 r/min培养72 h,将发酵液处理后利用高效液相色谱测定己二酸产量以进一步筛选己二酸高产菌株。
1.3 分析方法
菌体OD600值由紫外可见分光光度计测定。
发酵样品首先于12 000 r/min离心10 min,取500 μL上清液用10 mmol/L稀硫酸稀释后,再于12 000 r/min离心10 min。随后,用0.2 μm水系滤膜过滤。己二酸产量由高效液相色谱测定,检测器为紫外检测器(210 nm),流动相为5 mmol/L稀硫酸,流速为0.6 mL/min,测定温度为35 ℃,进样量为20 μL。
为对己二酸进行定性定量分析,本研究采用LC-MS进行检测。液相部分,检测器为光二级体检测器(210 nm),温度为45 ℃,流速为0.3 mL/min,进样量为5 μL。流动相为0.1%(体积分数)甲酸以及纯乙腈。梯度洗脱程序:在0~7.3 min,流动相配比为100% 0.1%甲酸;在7.3~8.2 min,流动相配比为70% 0.1%甲酸以及30%纯乙腈;在8.2~9.1 min,流动相配比为20% 0.1%甲酸以及80%纯乙腈;在9.1~10 min,流动相配比为100% 0.1%甲酸。质谱部分由Waters MALDI SYNAPT Q-TOF MS检测器检测[20]。
2 结果与分析
2.1 逆己二酸降解途径的构建、验证与优化
以实验室保藏的八敲大肠杆菌K12 MG1655 (ΔatoB ΔsucD ΔpflB ΔadhE ΔarcA ΔldhA ΔpoxB Δpta)为底盘宿主,选择源自大肠杆菌K12 MG1655的β-酮硫解酶、3-羟酰基辅酶A脱氢酶以及3-羟基己二酰辅酶A脱氢酶基因(paaJ、paaH、paaF)[21]、源自齿垢密螺旋体(Treponema denticola)的5-羧基-2-戊烯酰辅酶A还原酶基因(ter)[22]以及源自小鼠(Mus musculus)的己二酰辅酶A合成酶基因(acot8)[23]构建逆己二酸降解途径。另外,选择源自克氏梭菌(Clostridium kluyveri)的辅酶A转移酶基因(cat1)[24]合成丁二酰辅酶A以提高途径前体供应量。通过吉布森组装,首先构建质粒pRSF-Pmdh-paaJ-paaH以及pCDF-Pmdh-cat1-paaF,最后构建质粒pRSF-Pmdh-paaJ-paaH-PrpsU-acot8(pAD2)以及pCDF-Pmdh-cat1-paaF-PrpsU-ter(pAD4)。将质粒转入八敲大肠杆菌中得到Δ8-pAD24并进行摇瓶发酵(图1)。如图1-b所示,72 h发酵样品的LC-MS结果与己二酸标样一致,产量达到14.44 mg/L。
为调控途径基因在底盘细胞中的表达水平,本研究首先将途径基因模块化,进而更换各模块启动子以优化逆己二酸降解途径,从而平衡途径基因在宿主中的表达水平。首先,将逆己二酸降解途径分为4个模块(图2-a);其次,从ZHAO等[19]构建的组成型启动子库选取8个组成型启动子,其中每个模块分别用高低两种表达水平的启动子启动,以此构建相应质粒(图2-b)。构建完成后采用全因子法[25]对质粒进行组合(图2-a),因该启动子库中启动子序列同源性较强,在构建过程中易发生同源重组导致构建失败[26],最终成功构建除pRSF-PPL818-paaJ-paaH-PPL106-acot8以外的7种质粒,得到12种组合以进行下一步的研究。
2.2 结合己二酸生物传感器筛选最优组合
为避免因基因异质性导致己二酸产量降低从而干扰实验结果,使用实验室构建的高通量方法对每种组合开展验证。该高通量筛选方法分为3步:转化、初筛和复筛。首先,从平板上挑出转化子,菌落PCR验证正确后进行24孔板发酵,以己二酸生物传感器筛选荧光值较高的菌株,再转入摇瓶中发酵以进行复筛,以高效液相色谱验证己二酸产量。初筛过程中使用生物传感器为转录因子型传感器(抑制型),以BenM (TISNO-120)[27]为转录因子,由PPL1986启动子控制。其工作原理与乳糖操纵子类似,当BenM未与己二酸结合时,其与BenO的结合方式会组织对RNA聚合酶的转录,而当BenM与己二酸结合时,该转录因子的构象会发生改变,RNA聚合酶从而能够结合在BenO上,以激活下游绿色荧光蛋白表达。
将每种组合的转化子各挑24个在孔板中进行发酵后测定荧光,结果如图3-a所示,荧光值最高的为高表达模块1(paaJ, paaH)和模块2 (acot8)与低表达模块3 (cat1, paaF)和模块4(ter)的组合。摇瓶复筛验证各组合的结果如图3-b所示,各组合复筛的产量与初筛得到的结果能够相互印证,高表达模块1和2,低表达模块3和模块4的组合己二酸产量最高,达到120.60 mg/L,产量相较于Δ8-pAD24提高8.35倍。其次,可以发现过表达模块1的组合己二酸产量较高,由此可以确定逆己二酸降解途径的关键酶为β-酮硫解酶以及3-羟基酰辅酶A脱氢酶。最后,高表达4种基因的组合己二酸产量最低,仅达到52.39 mg/L,从该结果可以看出,在构建代谢途径过程中,全部高表达途径基因不是最优选择,平衡各途径基因的表达水平能显著提高目标产物产量。以最优组合为基础,本研究继续开展发酵条件优化实验。

a-逆己二酸降解途径;b-Δ8-pAD24 LC-MS分析;c-基因敲除验证;d-pAD2和pAD4验证
图1 逆己二酸降解途径的构建与验证
Fig.1 Construction and verification of reverse adipate degradation pathway 注:M为marker。
都
2.3 发酵优化提高己二酸产量
2.3.1 接种量
接种量不同会在不同程度影响菌体的生长条件和产物合成水平[28],同时己二酸生物合成为生长偶联型,需要菌体达到较好生长状况才能更有效的合成己二酸,因此本研究以2%接种量为对照,选择4种不同接种量作为实验组,优化接种量以提高己二酸产量。结果如图4所示,从图4-b可以看出,在接种量为4%时,己二酸产量最高,达到而在该接种量下,菌体的生长情况在各组中也为最优。同时,当接种量大于4%后,己二酸产量是随着接种量增加而减少的,由此可见过高的接种量对于产物合成有负面影响。综上,选择4%接种量进行下一步的研究。
2.3.2 铵盐
无机氮源是大肠杆菌偏好吸收的氮源[29],为优化培养基成分改善菌体生长状况,进一步提高己二酸产量,在TB培养基有机氮源(酵母粉以及胰蛋白胨)的基础上,选择不同浓度的NH4Cl作为无机氮源,对产己二酸培养基成分进行优化。从图5-b可以看出,随着NH4+浓度的增加,己二酸产量明显减少,但添加了20 mmol/L NH4Cl后己二酸产量(157.74 mg/L)较对照组(120.59 mg/L)有明显提升,因此选择20 mmol/L NH4Cl作为无机氮源进行后续的实验。
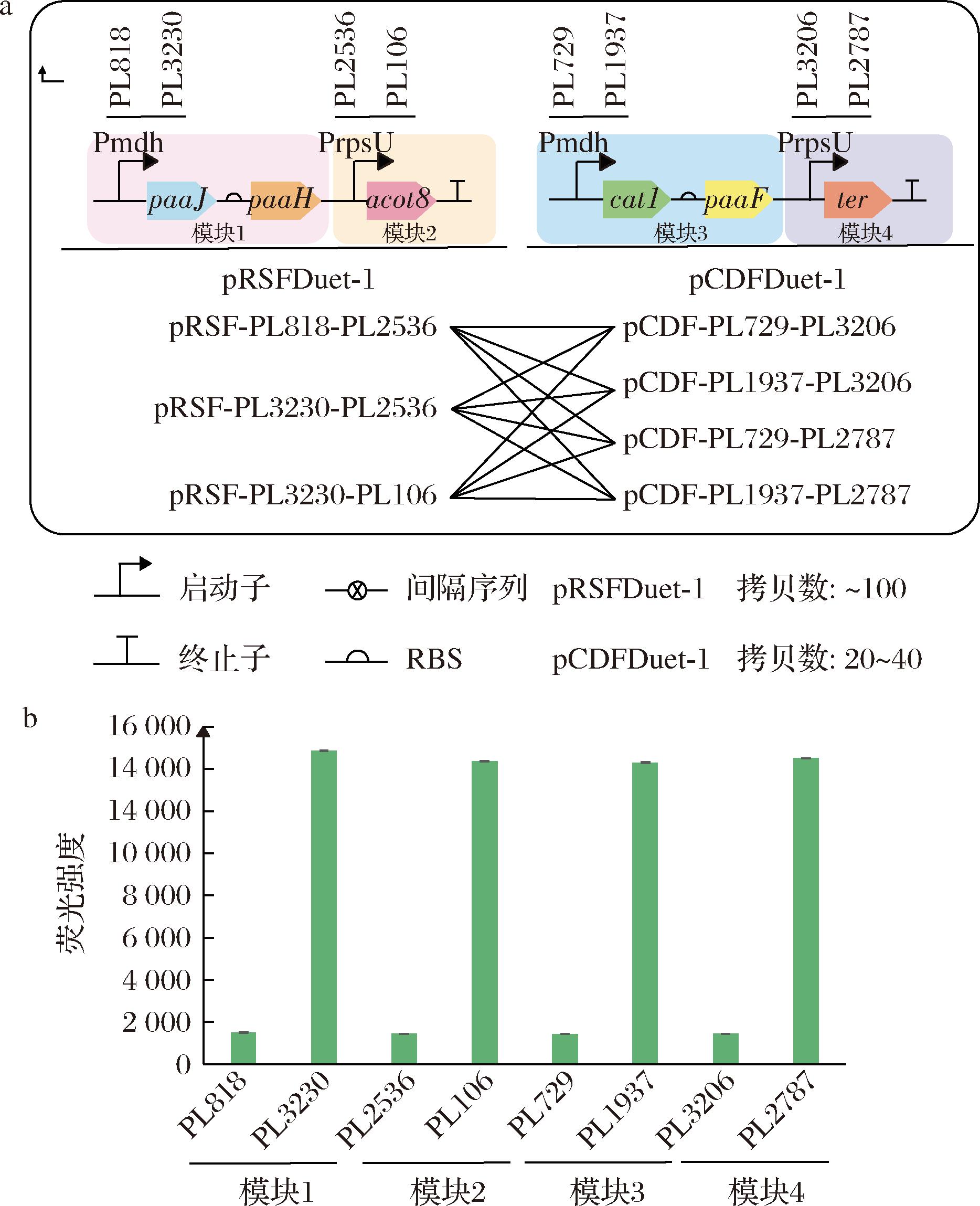
a-逆己二酸降解途径的模块化与不同质粒的组合方法; b-各模块对应的组成型启动子强度
图2 逆己二酸降解途径模块化优化
Fig.2 The modularization optimization of the reverse adipate degradation pathway 注:荧光强度由sfGFP/OD600值表征(下同)。
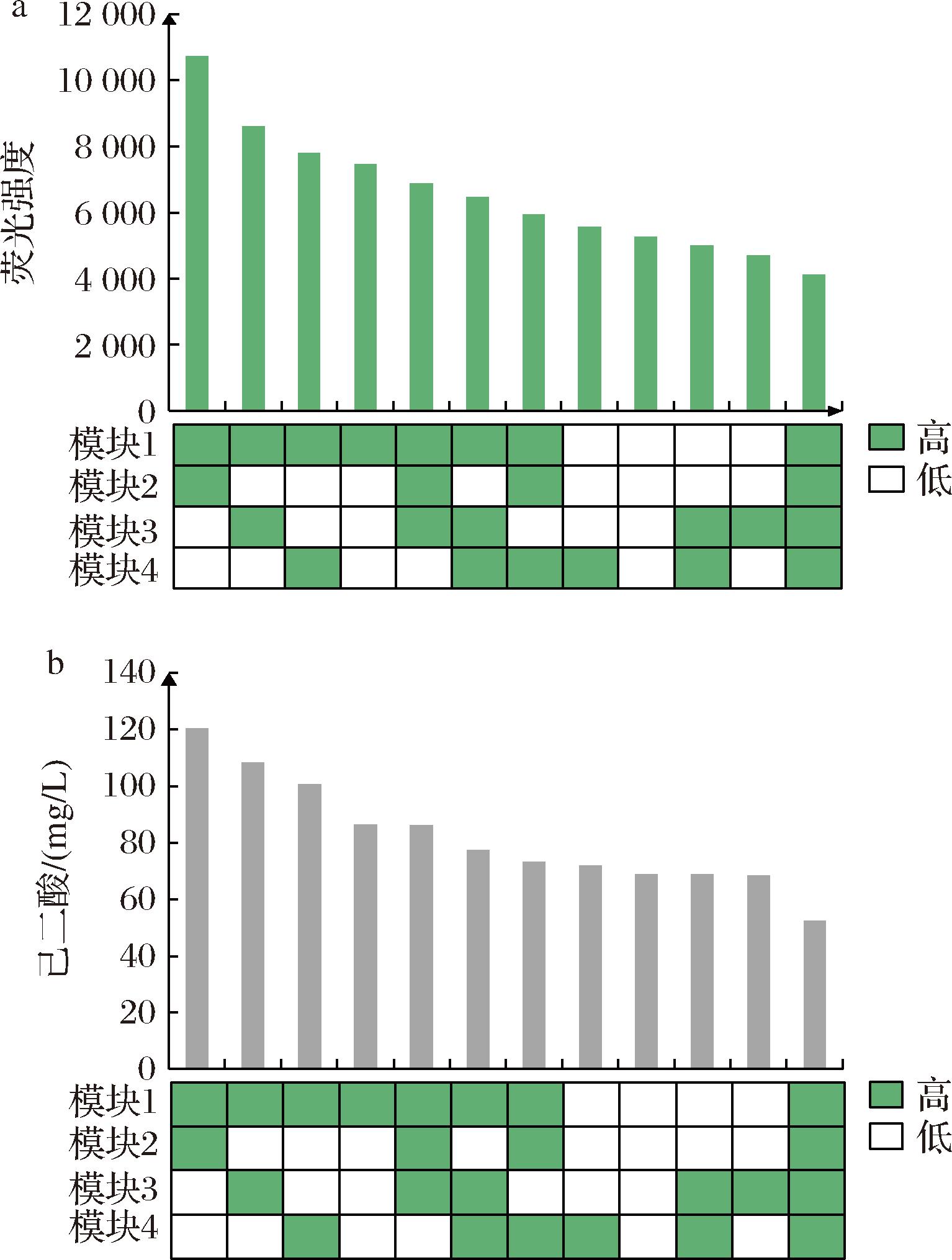
a-初筛结果;b-复筛结果
图3 不同组合的高通量筛选结果
Fig.3 The screening results of the high-throughput screening method of different combinations
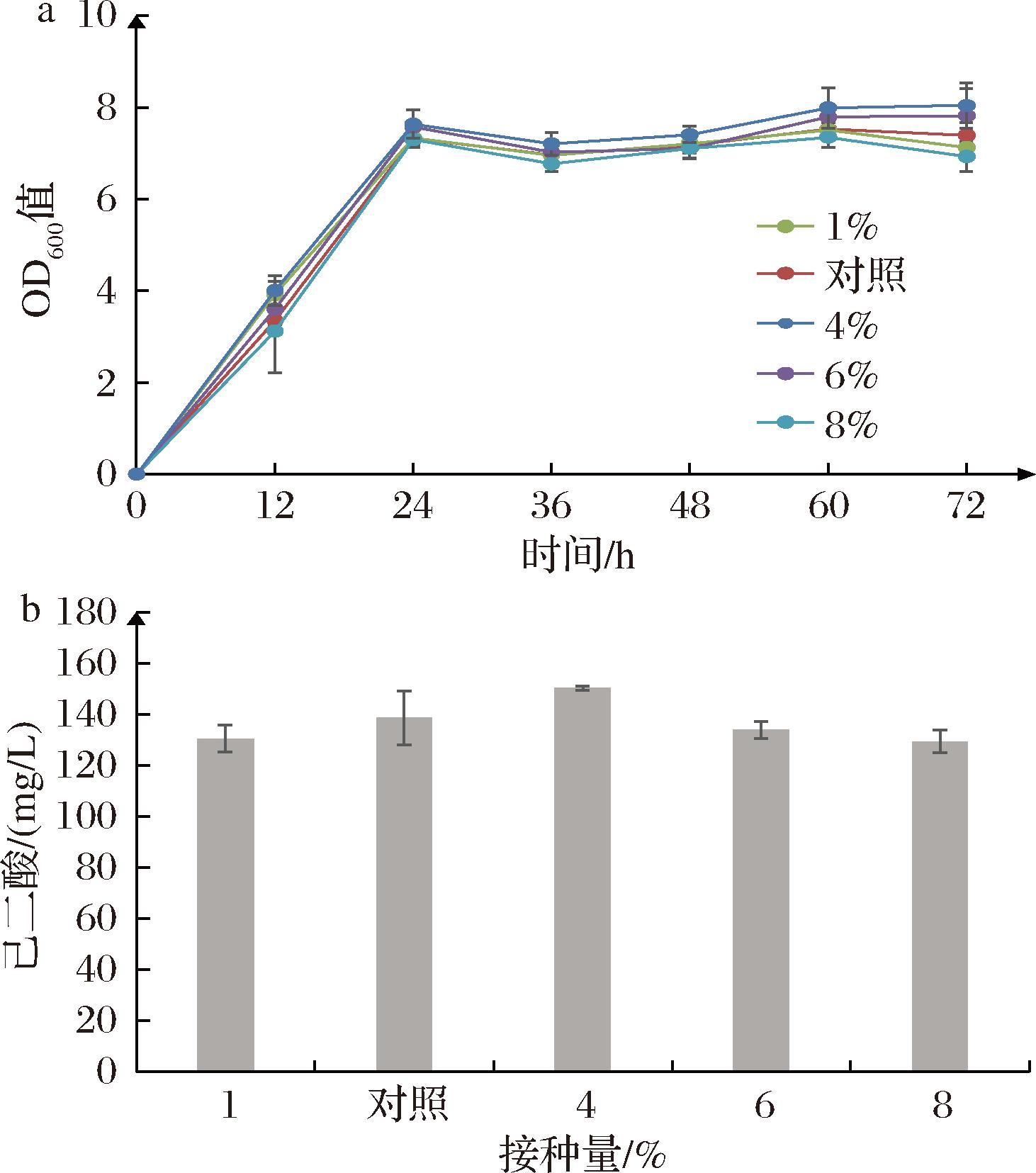
a-菌体生长情况;b-己二酸产量
图4 接种量对己二酸合成的影响
Fig.4 Effects of inoculation on the biosynthesis of adipic acid
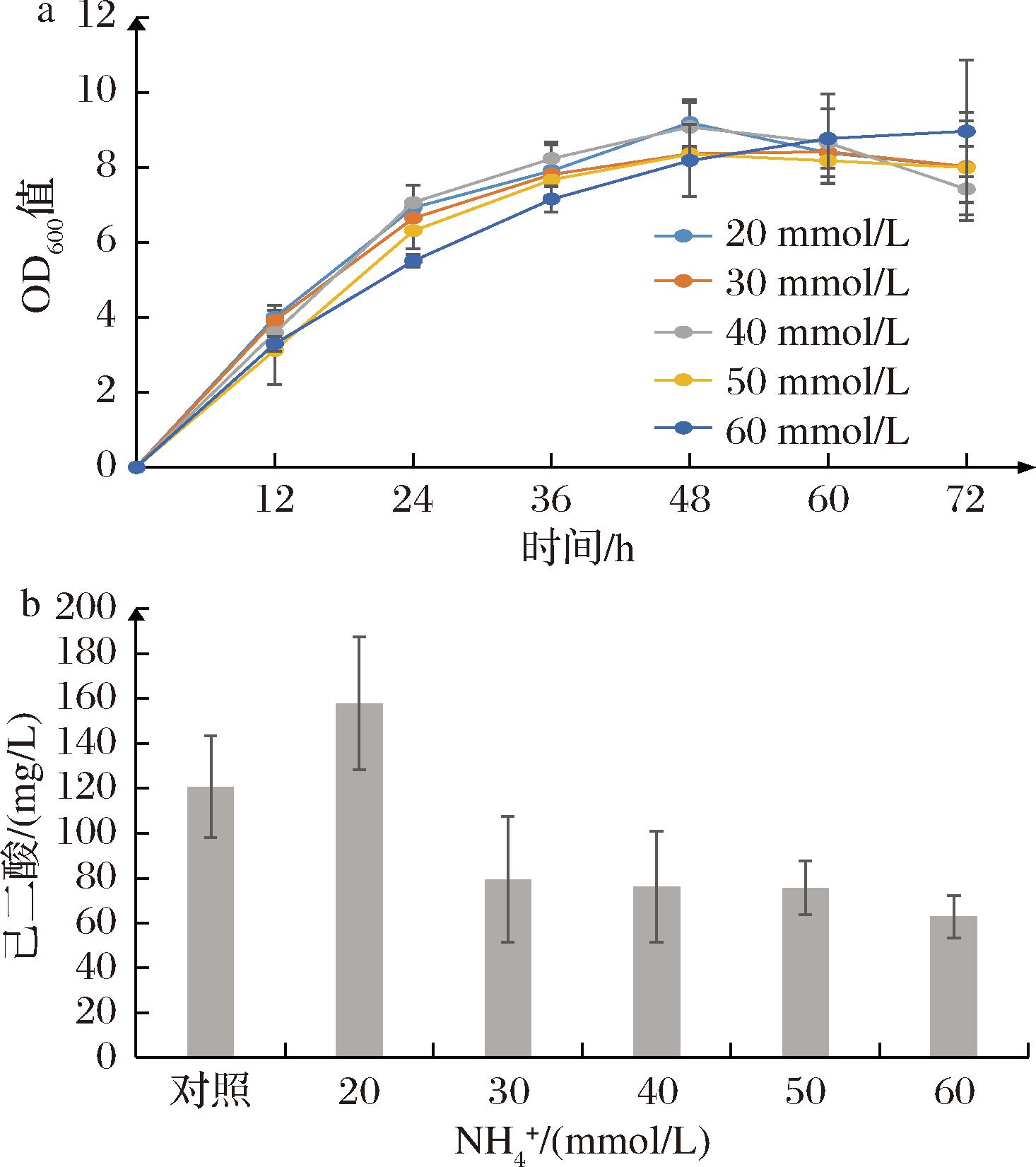
a-菌体生长情况;b-己二酸产量
图5 铵盐对己二酸合成的影响
Fig.5 Effects of ammonium on the biosynthesis of adipic acid
2.3.3 磷酸氢盐
磷酸氢盐是一类缓冲物质,有助于在发酵过程中维持发酵环境的pH值,促进菌体的基础代谢。因此,优化了TB培养基中磷酸氢盐的用量,以7 mmol/L磷酸氢盐为对照组,选择不同浓度的磷酸氢盐添加进培养基。结果如图6所示,随着磷酸氢盐浓度的逐渐添加,己二酸的产量逐渐下降,当磷酸氢盐浓度为3.5 mmol/L时,己二酸产量达到178.09 mg/L,因此选择该浓度继续进行实验。
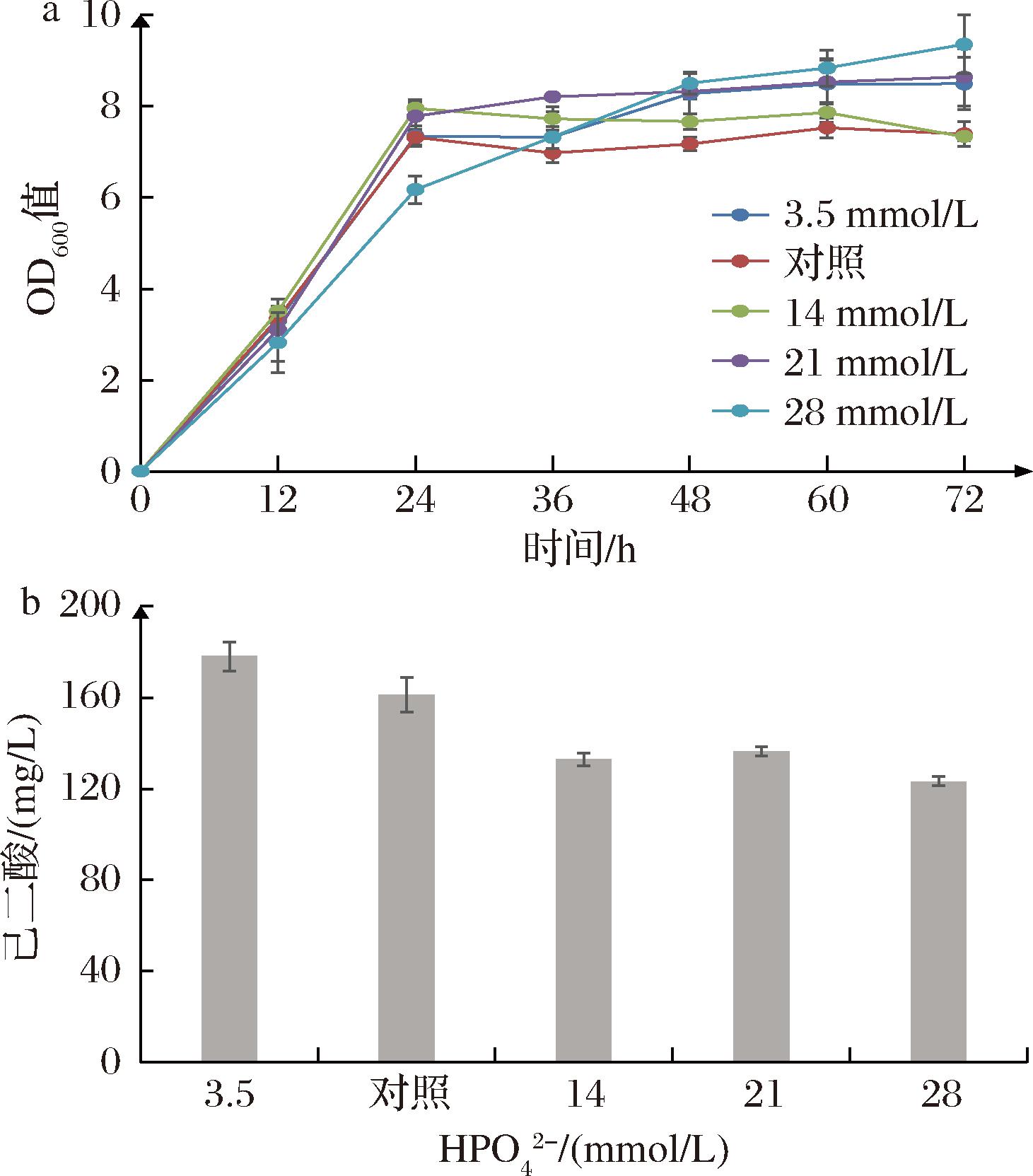
a-菌体生长情况;b-己二酸产量
图6 磷酸氢盐对己二酸合成的影响
Fig.6 Effects of hydrogen phosphate on the biosynthesis of adipic acid
2.3.4 金属离子
金属离子如Mn2+、Zn2+、Ca2+等是酶常见的辅因子,在培养基中添加金属离子有助于增加代谢途径酶的酶活性,提高目标产物产量[30]。同时,添加金属离子也可以在一定程度上维持发酵环境的pH稳定。因此,选取Mn2+、Ca2+、Zn2+、Fe2+、Cu2+分别添加进优化后的培养基中进行发酵。实验结果如图7所示,金属离子的添加有效提高了己二酸的产量,其中添加了Cu2+的培养基己二酸产量最高,达到240.49 mg/L。
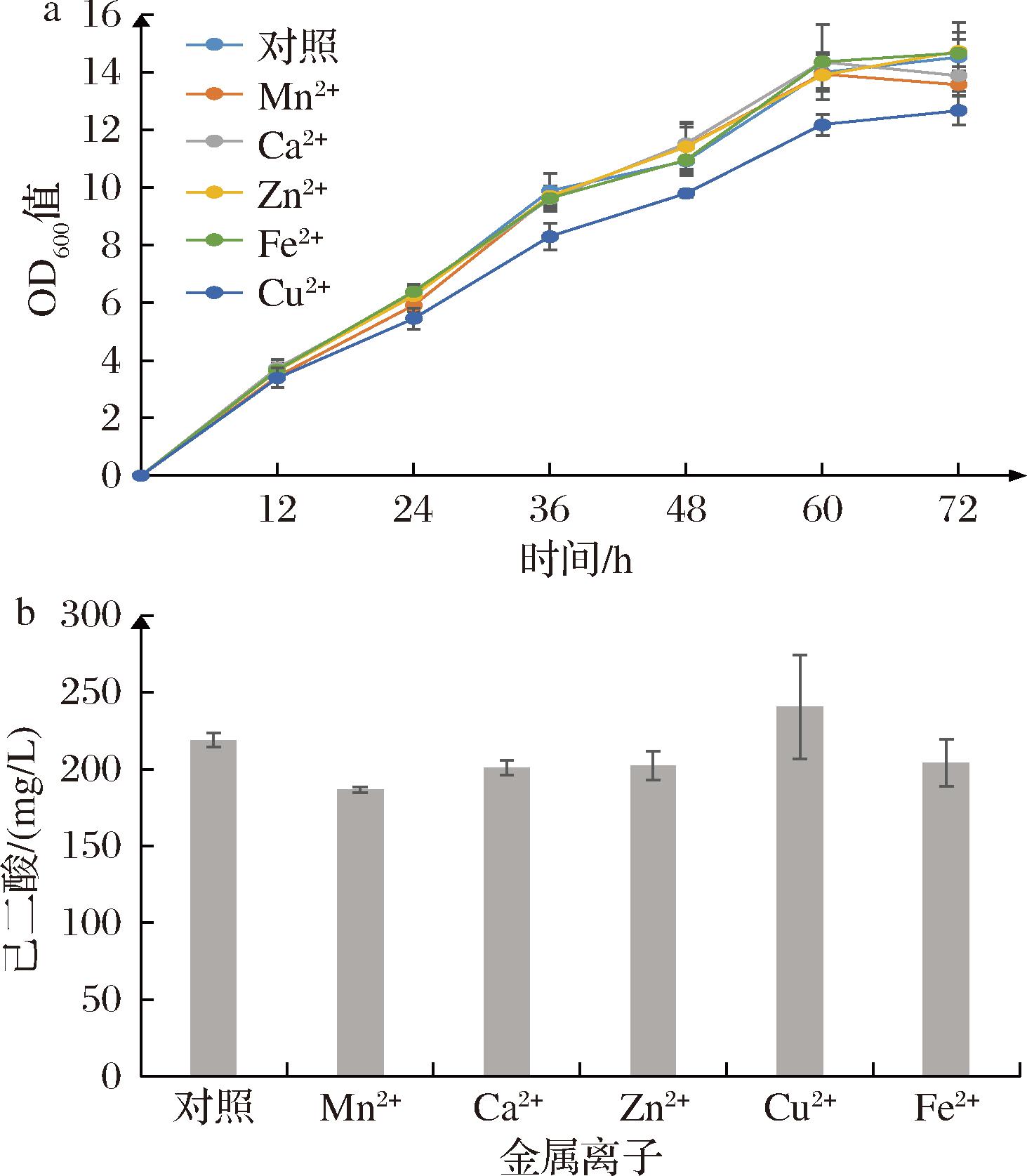
a-菌体生长情况;b-己二酸产量
图7 金属离子优化结果
Fig.7 Effects of metal ions on the biosynthesis of adipic acid
3 结论与讨论
本研究首先将逆己二酸降解途径模块化,通过更换不同启动子协调逆己二酸降解途径各基因表达强度,最后通过高通量筛选方法对组合进行验证,确定了途径关键酶PaaJ以及PaaH,对逆己二酸降解途径基因在底盘细胞中的表达水平实现了成功调控,使己二酸产量达到120.66 mg/L,产量有了明显提高,为后续优化代谢途径的研究提供了借鉴。同时,在最优结果基础上进行了发酵工艺优化,己二酸产量提高了16.65倍,达到240.49 mg/L。在后续研究中,可以从挖掘途径同工酶、基因元件选择以及发酵过程参数等方面通过合成生物学以及代谢工程策略进行进一步探究,提高己二酸生物合成的水平。
[1] LI W N, SHEN X L, WANG J, et al.Engineering microorganisms for the biosynthesis of dicarboxylic acids[J].Biotechnology Advances, 2021, 48:107710.
[2] ZHAO M, HUANG D X, ZHANG X J, et al.Metabolic engineering of Escherichia coli for producing adipic acid through the reverse adipate-degradation pathway[J].Metabolic Engineering, 2018, 47:254-262.
[3] NIU W, WILLETT H, MUELLER J, et al.Direct biosynthesis of adipic acid from lignin-derived aromatics using engineered Pseudomonas putida KT2440[J].Metabolic Engineering, 2020, 59:151-161.
[4] SKOOG E C, SHIN J, S EZ-JIMÉNEZ V, et al.Biobased adipic acid - The challenge of developing the production host[J].Biotechnology Advances, 2018, 36(8):2248-2263.
EZ-JIMÉNEZ V, et al.Biobased adipic acid - The challenge of developing the production host[J].Biotechnology Advances, 2018, 36(8):2248-2263.
[5] BART J C J, CAVALLARO S.Transiting from adipic acid to bioadipic acid.part II.Biosynthetic pathways[J].Industrial &Engineering Chemistry Research, 2015, 54(2):567-576.
[6] CLOMBURG J M, BLANKSCHIEN M D, VICK J E, et al.Integrated engineering of β-oxidation reversal and ω-oxidation pathways for the synthesis of medium chain ω-functionalized carboxylic acids[J].Metabolic Engineering, 2015, 28:202-212.
[7] DENG Y, MAO Y.Production of adipic acid by the native-occurring pathway in Thermobifida fusca B6[J].Journal of Applied Microbiology, 2015, 119(4):1057-1063.
[8] TURK S C H J, KLOOSTERMAN W P, NINABER D K, et al.Metabolic engineering toward sustainable production of nylon-6[J].ACS Synthetic Biology, 2016, 5(1):65-73.
[9] HAO T T, LI G H, ZHOU S H, et al.Engineering the reductive TCA pathway to dynamically regulate the biosynthesis of adipic acid in Escherichia coli[J].ACS Synthetic Biology, 2021, 10(3):632-639. [10] RUGBJERG P, SOMMER M O A.Overcoming genetic heterogeneity in industrial fermentations[J].Nature Biotechnology, 2019, 37(8):869-876.
[11] WEHRS M, TANJORE D, ENG T, et al.Engineering robust production microbes for large-scale cultivation[J].Trends in Microbiology, 2019, 27(6):524-537.
[12] LIU C S, DING Y M, ZHANG R B, et al.Functional balance between enzymes in malonyl-CoA pathway for 3-hydroxypropionate biosynthesis[J].Metabolic Engineering, 2016, 34:104-111.
[13] XU P, RIZZONI E A, SUL S Y, et al.Improving metabolic pathway efficiency by statistical model-based multivariate regulatory metabolic engineering[J].ACS Synthetic Biology, 2017, 6(1):148-158.
[14] LIU Q, WU K Y, CHENG Y B, et al.Engineering an iterative polyketide pathway in Escherichia coli results in single-form alkene and alkane overproduction[J].Metabolic Engineering, 2015, 28:82-90.
[15] ROUCHES M V, XU Y S, CORTES L B G, et al.A plasmid system with tunable copy number[J].Nature Communications, 2022, 13(1):3908.
[16] YOUNG E M, ZHAO Z, GIELESEN B E M, et al.Iterative algorithm-guided design of massive strain libraries, applied to itaconic acid production in yeast[J].Metabolic Engineering, 2018, 48:33-43.
[17] XU P, VANSIRI A, BHAN N, et al.ePathBrick:A synthetic biology platform for engineering metabolic pathways in E.coli[J].ACS Synthetic Biology, 2012, 1(7):256-266.
[18] ZHOU S H, DING R P, CHEN J, et al.Obtaining a panel of cascade promoter-5′-UTR complexes in Escherichia coli[J].ACS Synthetic Biology, 2017, 6(6):1065-1075.
[19] ZHAO M, YUAN Z Q, WU L T, et al.Precise prediction of promoter strength based on a de novo synthetic promoter library coupled with machine learning[J].ACS Synthetic Biology, 2022, 11(1):92-102.
[20] LI L L, CHANG M, TAO G J, et al.Analysis of phospholipids in Schizochytrium sp.S31 by using UPLC-Q-TOF-MS[J].Analytical Methods, 2016, 8(4):763-770.
[21] TEUFEL R, MASCARAQUE V, ISMAIL W, et al.Bacterial phenylalanine and phenylacetate catabolic pathway revealed[J].Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(32):14390-14395.
[22] TUCCI S, MARTIN W.A novel prokaryotic trans-2-enoyl-CoA reductase from the spirochete Treponema denticola[J].FEBS Letters, 2007, 581(8):1561-1566.
[23] WESTIN M A K, HUNT M C, ALEXSON S E H.The identification of a succinyl-CoA thioesterase suggests a novel pathway for succinate production in peroxisomes[J].Journal of Biological Chemistry, 2005, 280(46):38125-38132.
[24] SÖHLING B, GOTTSCHALK G.Molecular analysis of the anaerobic succinate degradation pathway in Clostridium kluyveri[J].Journal of Bacteriology, 1996, 178(3):871-880.
[25] GILMAN J, WALLS L, BANDIERA L, et al.Statistical design of experiments for synthetic biology[J].ACS Synthetic Biology, 2021, 10(1):1-18.
[26] HOSSAIN A, LOPEZ E, HALPER S M, et al.Automated design of thousands of nonrepetitive parts for engineering stable genetic systems[J].Nature Biotechnology, 2020, 38(12):1466-1475.
[27] SNOEK T, CHABERSKI E K, AMBRI F, et al.Evolution-guided engineering of small-molecule biosensors[J].Nucleic Acids Research, 2020, 48(1):e3.
[28] STREHAIANO P, MOTA M, GOMA G.Effects of inoculum level on kinetics of alcoholic fermentation[J].Biotechnology Letters, 1983, 5(2):135-140.
[29] LOZANO TEROL G, GALLEGO-JARA J, SOLA MART NEZ R A, et al.Engineering protein production by rationally choosing a carbon and nitrogen source using E.coli BL21 acetate metabolism knockout strains[J].Microbial Cell Factories, 2019, 18(1):1-19.
NEZ R A, et al.Engineering protein production by rationally choosing a carbon and nitrogen source using E.coli BL21 acetate metabolism knockout strains[J].Microbial Cell Factories, 2019, 18(1):1-19.
[30] DENG X, ZHENG Y C, LI Q B.Effect of ambient conditions on simultaneous growth and bioaccumulation of mercuric ion by genetically engineered E.coli JM109[J].Journal of Hazardous Materials, 2006, 136(2):233-238.