随着化石燃料的日益减少和CO2的大量排放,世界各国都在寻求并开发低污染、可再生的原料。甲醇是煤炭以及天然气的副产品[1],价格便宜,到2017年为止,年产量超过1亿t[2]。同时,随着科技的进步,甲醇可以通过电催化[3]或者光催化[4]等绿色的方式获得。因此,利用甲醇作为底物完成生物转化成为专家学者关注的方向。研究人员在改造现有的模式生物为甲基营养微生物中取得进展[5],但合成甲基微生物距离工业化应用尚存在技术难题,所以天然甲基微生物成为最佳研究对象。其中,嗜甲醇巴斯德毕赤酵母(Pichia pastoris,即Komagataella phaffii)凭借其自身优势在一众天然甲基微生物中脱颖而出。2009年,人们完成了毕赤酵母的全基因组测序[6],为该酵母的代谢工程研究奠定了基础。因其完善的翻译后修饰系统[7-8]以及生物安全性[9],毕赤酵母已被广泛应用于一系列重组蛋白的生产[10]。此外,毕赤酵母是Crabtree阴性酵母,适用于高密度发酵[11]。
尽管毕赤酵母已经成功应用于多种蛋白质及化合物的工业化生产,但以甲醇为唯一碳源进行工业化生产还面临着甲醇生物转化率低下的问题,仅有30%~50%的甲醇被同化为生物量[12-13],这意味着需要通过两步发酵法(先利用甘油等碳源迅速积累生物量,再用甲醇诱导目的基因表达)的方式来完成工业生产,这种发酵方式极大地增加了生产的时间成本以及物力成本[14]。因此,深入了解甲醇代谢机理,确定关键酶基因对后续甲醇利用途径的改造与优化有重大意义。
在最近的研究中,有团队发现毕赤酵母的同化途径发生在过氧化物酶体中(图1),并从过氧化物酶体中分离出与细胞质中D-核酮糖-5-磷酸-3-表异构酶1(D-ribulose-5-phosphate 3-epimerase,Rpe1-1),核糖-5-磷酸异构酶1(ribose-5-phosphate isomerase,Rki1-1),转醛醇酶1(transaldolase,Tal1-1),果糖-1,6-二磷酸醛缩酶1(fructose-1,6-bisphosphate aldolase,Fba1-1)等酶对应的同工酶:Rpe1-2,Rki1-2,Tal1-2,Fba1-2[15]。这些酶主要参与木酮糖-5-磷酸(xylulose 5-phosphate,XU5P)的再生,而XU5P作为甲醛受体,对于甲醇同化至关重要。因此本研究对这些过氧化物酶体同工酶进行详细分析,期望能从中找到甲醇同化的关键酶基因,以期为后续甲醇利用途径的改造优化提供靶标。
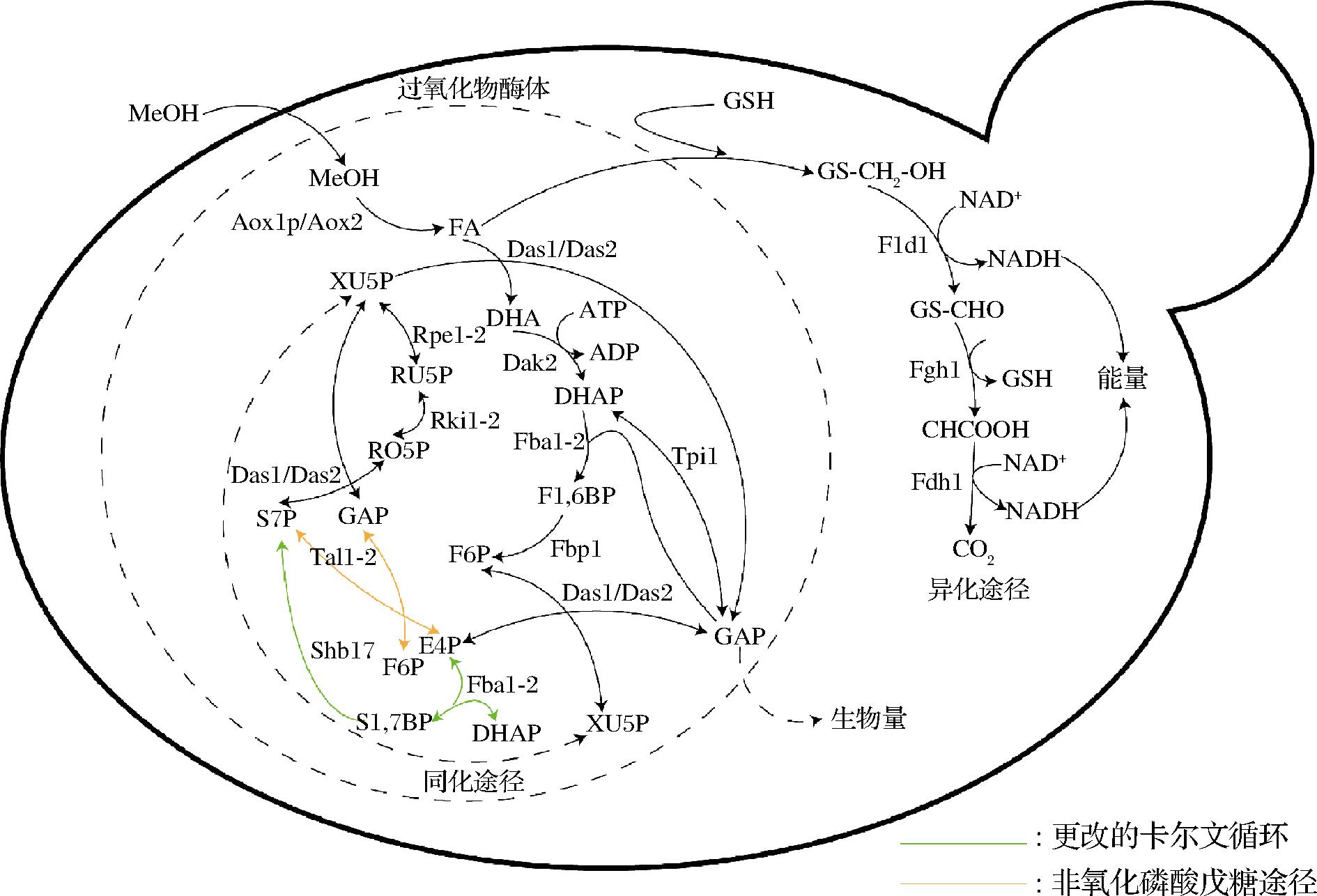
图1 甲醇利用途径示意图
Fig.1 The diagram of methanol utilization pathway
1 材料与方法
1.1 实验材料
实验中所用的菌株及质粒见表1。文中所需的引物由苏州金唯智生物科技有限公司合成,并在电子版增强出版附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.035425)中展示。
表1 菌株和质粒
Table 1 Strains and plasmids

菌株/质粒特性来源E.coli XL10-Gold实验室保存P.pastoris GS115his-实验室保存P.pastoris KO-RPE1-2GS115,RPE1-2Δ本研究构建P.pastoris KO-RKI1-2GS115,RKI1-2Δ本研究构建P.pastoris KO-TAL1-2GS115,TAL1-2Δ本研究构建P.pastoris KO-FBA1-2GS115,FBA1-2Δ本研究构建P.pastoris KO-4GS115,RPE1-2Δ,RKI1-2Δ,TAL1-2ΔFBA1-2Δ本研究构建P.pastoris KO-SHB17GS115,SHB17Δ本研究构建P.pastoris HB-RPE1-2KO-4,HIS4::PRPE1-2-RPE1-2-TRPE1-2本研究构建P.pastoris HB-RKI1-2KO-4,HIS4::PRKI1-2-RKI1-2-TRKI1-2本研究构建P.pastoris HB-TAL1-2KO-4,HIS4::PTAL1-2-TAL1-2-TTAL1-2本研究构建P.pastoris HB-FBA1-2KO-4,HIS4::PFBA1-2-FBA1-2-TFBA1-2本研究构建P.pastoris HB-C4KO-4,HIS4::PRPE1-1-RPE1-1-TRPE1-1,PRKI1-1-RKI1-1-TRKI1-1,PTAL1-1-TAL1-1-TTAL1-1,PFBA1-1-FBA1-1-TFBA1-1本研究构建P.pastoris HB-C4PKO-4,HIS4::PRPE1-1-RPE1-1-PTS1-TRPE1-1,PRKI1-1-RKI1-1-PTS1-TRKI1-1,PTAL1-1-TAL1-1-PTS1-TTAL1-1,PFBA1-1-FBA1-1-PTS1-TFBA1-1本研究构建P.pastoris GFPGS115,游离质粒表达PAOX1-GFP-TAOX1本研究构建P.pastoris GFP-PTS1GS115,游离质粒表达PAOX1-GFP-PTS1-TAOX1本研究构建P.pastoris GFP-RPE1-2GS115,游离质粒表达PAOX1-GFP-RPE1-2-TAOX1本研究构建P.pastoris GFP-RKI1-2GS115,游离质粒表达PAOX1-GFP-RKI1-2-TAOX1本研究构建P.pastoris GFP-TAL1-2GS115,游离质粒表达PAOX1-GFP-TAL1-2-TAOX1本研究构建P.pastoris GFP-FBA1-2GS115,游离质粒表达PAOX1-GFP-FBA1-2-TAOX1本研究构建P.pastoris GFP-RPE1-1GS115,游离质粒表达PAOX1-GFP-RPE1-1-TAOX1本研究构建P.pastoris GFP-RKI1-1GS115,游离质粒表达PAOX1-GFP-RKI1-1-TAOX1本研究构建P.pastoris GFP-TAL1-1GS115,游离质粒表达PAOX1-GFP-TAL1-1-TAOX1本研究构建P.pastoris GFP-FBA1-1GS115,游离质粒表达PAOX1-GFP-FBA1-1-TAOX1本研究构建P.pastoris GFP-C4GS115,HIS4::PRPE1-1-GFP-RPE1-1-TRPE1-1,PRKI1-1-GFP-RKI1-1-TRKI1-1,PTAL1-1-GFP-TAL1-1-TTAL1-1,PFBA1-1-GFP-FBA1-1-TFBA1-1本研究构建P.pastoris GFP-C4PGS115,HIS4::PRPE1-1-GFP-RPE1-1-PTS1-TRPE1-1,PRKI1-1-GFP-RKI1-1-PTS1-TRKI1-1,PTAL1-1-GFP-TAL1-1-PTS1-TTAL1-1,PFBA1-1-GFP-FBA1-1-PTS1-TFBA1-1本研究构建sgRNA-Cas9Zeocin+实验室保存sgRNA-RPE1-2Zeocin+,RPE1-2基因sg质粒本研究构建sgRNA-RKI1-2Zeocin+,RKI1-2基因sg质粒本研究构建sgRNA-TAL1-2Zeocin+,TAL1-2基因sg质粒本研究构建sgRNA-FBA1-2Zeocin+,FBA1-2基因sg质粒本研究构建sgRNA-SHB17Zeocin+,SHB17基因sg质粒本研究构建hb-RPE1-2Zeocin+,HIS4,PRPE1-2-RPE1-2-TRPE1-2本研究构建hb-RKI1-2Zeocin+,HIS4,PRKI1-2-RKI1-2-TRKI1-2本研究构建
续表1

菌株/质粒特性来源hb-TAL1-2Zeocin+,HIS4,PTAL1-2-TAL1-2-TTAL1-2本研究构建hb-FBA1-2Zeocin+,HIS4,PFBA1-2-FBA1-2-TFBA1-2本研究构建hb-C4Zeocin+,HIS4,PRPE1-1-RPE1-1-TRPE1-1,PRKI1-1-RKI1-1-TRKI1-1,PTAL1-1-TAL1-1-TTAL1-1,PFBA1-1-FBA1-1-TFBA1-1本研究构建hb-C4PZeocin+,HIS4,PRPE1-1-RPE1-1-PTS1-TRPE1-1,PRKI1-1-RKI1-1-PTS1-TRKI1-1,PTAL1-1-TAL1-1-PTS1-TTAL1-1,PFBA1-1-FBA1-1-PTS1-TFBA1-1本研究构建
1.2 主要培养基及培养条件
大肠杆菌使用LLB培养基(g/L)(蛋白胨10,酵母提取物5,NaCl 5)培养,抗生素使用博来霉素,工作浓度为25 μg/mL,在37 ℃,220 r/min的摇床中培养。毕赤酵母在30 ℃,200 r/min摇床中培养,使用YPD培养基(g/L)(酵母提取物10,蛋白胨20,葡萄糖20)扩大培养,使用YPM培养基(g/L)[酵母提取物10,蛋白胨20,甲醇1.5%(体积分数)]进行荧光定位实验。抗生素使用博来霉素,工作浓度为75 μg/mL;当测定生长曲线时,使用MMH培养基(g/L)(无氨基酵母氮源13.4,甲醇1.5%,组氨酸0.004),并且每24 h 补加0.5%(体积分数)甲醇。
1.3 质粒的构建
1.3.1 sgRNA质粒的构建
共构建sgRNA-RPE1-2、sgRNA-RKI1-2、sgRNA-TAL1-2、sgRNA-FBA1-2、sgRNA-SHB17 5个sgRNA质粒。以sgRNA-RPE1-2质粒的构建为例构建其他4个质粒sgRNA-RPE1-2的构建:通过CHOPCHOP网站(http://chop-chop.cbu.uib.no/)在线设计sgRNA序列,将RPE1-2基因的DNA序列输入网站并选择菌种为Pichia pastoris,挑选打分较高的sgRNA序列用于构建质粒。根据对应的sgRNA序列设计部分互补的引物对,退火粘合,通过Golden Gate assembly连接至sgRNA-Cas9骨架质粒的Bsa I位点,随后转化至E.coli XL10-Gold感受态,通过sg-verify-F/R引物进行菌落PCR验证并挑选阳性克隆扩大培养,提取质粒进行下步操作。
1.3.2 回补质粒的构建
1.3.2.1 过氧化物酶体同工酶回补质粒的构建
通过PCR从GS115菌株的基因组扩增得到RPE1-2、RKI1-2、TAL1-2、FBA1-2等基因的完整表达框,然后通过Gibson assembly分别将各个基因的表达框组装至ZH01质粒(图2)的位点1,得到回补质粒hb-RPE1-2、hb-RKI1-2、hb-TAL1-2、hb-FBA1-2。
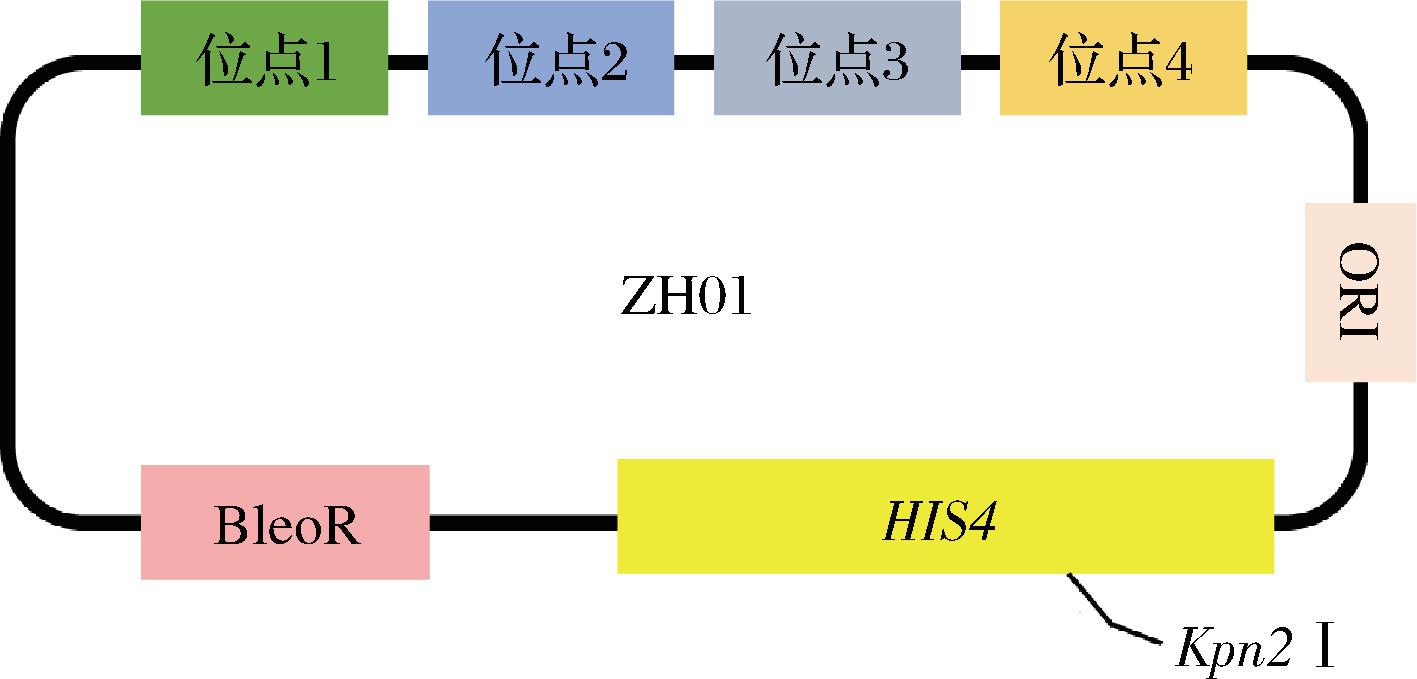
图2 ZH01质粒示意图
Fig.2 The diagram of ZH01 plasmid
1.3.2.2 细胞质同工酶回补质粒的构建
通过PCR从GS115菌株的基因组扩增得到RPE1-1、RKI1-1、TAL1-1、FBA1-1等基因的完整表达框,然后通过Gibson assembly将RPE1-1表达框组装至ZH01质粒的位点1,RKI1-1表达框组装至位点2,TAL1-1表达框组装至位点3,FBA1-1表达框组装至位点4,得到hb-C4回补质粒。
在hb-C4质粒的基础上,通过Gibson assembly在各个细胞质同工酶基因的3’端添加过氧化物酶体定位信号1(peroxisome targeting signal type 1,PTS1)序列,即得到定位至过氧化物酶体的细胞质同工酶回补质粒hb-C4P。
1.4 菌株的构建
1.4.1 基因编辑菌株的构建
使用实验室现有的CRISPR-Cas9基因编辑系统(实验结果未发表)完成KO-RPE1-2,KO-RKI1-2,KO-TAL1-2,KO-FBA1-2,KO-4、KO-SHB17等菌株的构建。KO-RKI1-2、KO-TAL1-2、KO-FBA1-2、KO-4、KO-SHB17等菌株的构建方法请参考KO-RPE1-2的构建。
KO-RPE1-2菌株的构建:首先利用RPE1-2-HAUP-F/ RPE1-2-HAUP-R引物对和RPE1-2-HADN-F/RPE1-2-HADN-R引物对分别扩增RPE1-2基因上下游各1 000 bp的同源臂片段,然后利用RPE1-2-HAUP-F/RPE1-2-HADN-R引物对将上下游同源臂片段进行融合PCR,获得2 000 bp的上下游同源臂融合片段。最后将1 μg上下游同源臂片段与1 μg sgRNA-RPE1-2质粒混合转化至GS115感受态细胞,待转化板形成单克隆菌株后随机挑取菌株进行基因组PCR验证,选择验证正确的菌株供后续实验使用。
1.4.2 回补菌株的构建
1.4.2.1 过氧化物酶体同工酶回补菌株的构建
利用BspE I限制性内切酶线性化1.3.2.1节构建的hb-RPE1-2、hb-RKI1-2、hb-TAL1-2、hb-FBA1-2等质粒上的HIS4基因,分别电转化至KO-4菌株的感受态细胞,得到HB-RPE1-2、HB-RKI1-2、HB-TAL1-2、HB-FBA1-2等回补菌株。
1.4.2.2 细胞质同工酶回补菌株的构建
利用BspE I限制性内切酶线性化1.3.2.2节构建的hb-C4及hb-C4P质粒上的HIS4基因,分别电转化至KO-4菌株的感受态细胞,得到在KO-4菌株细胞质中过表达4个细胞质同工酶的HB-C4菌株以及在过氧化物酶体中回补4个细胞质同工酶的HB-C4P菌株。
1.5 同工酶亚细胞定位验证
为了区分过氧化物酶体定位与细胞质定位的不同,将表达GFP蛋白和表达GFP-PTS1(在GFP的C-端融合表达PTS1)蛋白的两株毕赤酵母细胞分别在YPM培养基中培养18 h,并用磷酸盐缓冲液(pH 7.4)洗涤两次并重悬。取10 μL制片,用共聚焦激光扫描显微镜(TCS SP8,德国徕卡)在488 nm的波长下观察GFP蛋白细胞质定位及过氧化物酶体定位的表型变化。随后,将GFP蛋白分别融合到Rpe1-2、Rki1-2、Tal1-2、Fba1-2、Rpe1-1、Rki1-1、Tal1-1、Fba1-1等蛋白的N-端并在毕赤酵母中表达,观察荧光表型以确定同工酶的亚细胞定位。
1.6 毕赤酵母总RNA的提取和cDNA的合成
使用购自北京康为世纪科技有限公司的超纯RNA提取试剂盒提取毕赤酵母总RNA,随后定量至1 μg并使用南京诺唯赞生物科技股份有限公司的HiScript Ⅲ RT SuperMix for qPCR (+gDNA wiper)试剂盒完成逆转录,获得cDNA片段。RNA提取步骤及cDNA合成步骤请参照相应试剂公司的官方说明书。
1.7 实时荧光定量PCR (real-time quantitative PCR,RT-qPCR)引物设计
使用NCBI网站(https://www.ncbi.nlm.nih.gov/tools/primer-blast/index.cgi?LINK_LOC=BlastHome)的Prime-BLAST功能设计各基因的RT-qPCR引物。将待测基因的开放阅读框输入网页中的“PCR Template”框,Tm值设为58~62 ℃,PCR产物长度设为80~120 bp,将“Organism”选项切换为“Komagataella pastoris GS115”,“Database”选项设置为“Refseq mRNA”,点击“Get Primers”即可获得合适的RT-qPCR引物片段。
1.8 甲醇含量的测定
将样品在12 000 r/min下离心10 min,吸取上清液进行HPLC测定。使用Aminex HPX-87H色谱柱(9 μm,1 300 mm×7.8 mm,美国伯乐)进行分析。以5 mmol/L H2SO4溶液为流动相,流速设置为0.6 mL/min,柱温设置为50 ℃运行30 min,运行期间HPLC信号采集使用示差检测器(RID-10A, 日本岛津)。
2 结果与分析
2.1 同工酶定位验证
通过观察细胞内无定位信号的GFP蛋白与定位在过氧化物酶体的GFP-PTS1蛋白的荧光分布,可以发现,无定位信号的GFP蛋白荧光弥散在整个胞质中,而加有PTS1定位信号的GFP蛋白呈点状聚集在过氧化物酶体中(图3)。随后将GFP-Rpe1-1、GFP-Rki1-1、GFP-Tal1-1、GFP-Fba1-1、GFP-Rpe1-2、GFP-Rki1-2、GFP-Tal1-2、GFP-Fba1-2等融合蛋白在毕赤酵母中表达。如图3所示,GFP-Rpe1-1、GFP-Rki1-1、GFP-Tal1-1、GFP-Fba1-1等蛋白的荧光分布在整个细胞质中,而GFP-Rpe1-2、GFP-Rki1-2、GFP-Tal1-2、GFP-Fba1-2等蛋白的荧光呈点状聚集在过氧化物酶体中。因此,Rpe1-1、Rki1-1、Tal1-1、Fba1-1属于细胞质同工酶,而Rpe1-2、Rki1-2、Tal1-2、Fba1-2属于过氧化物酶体同工酶。
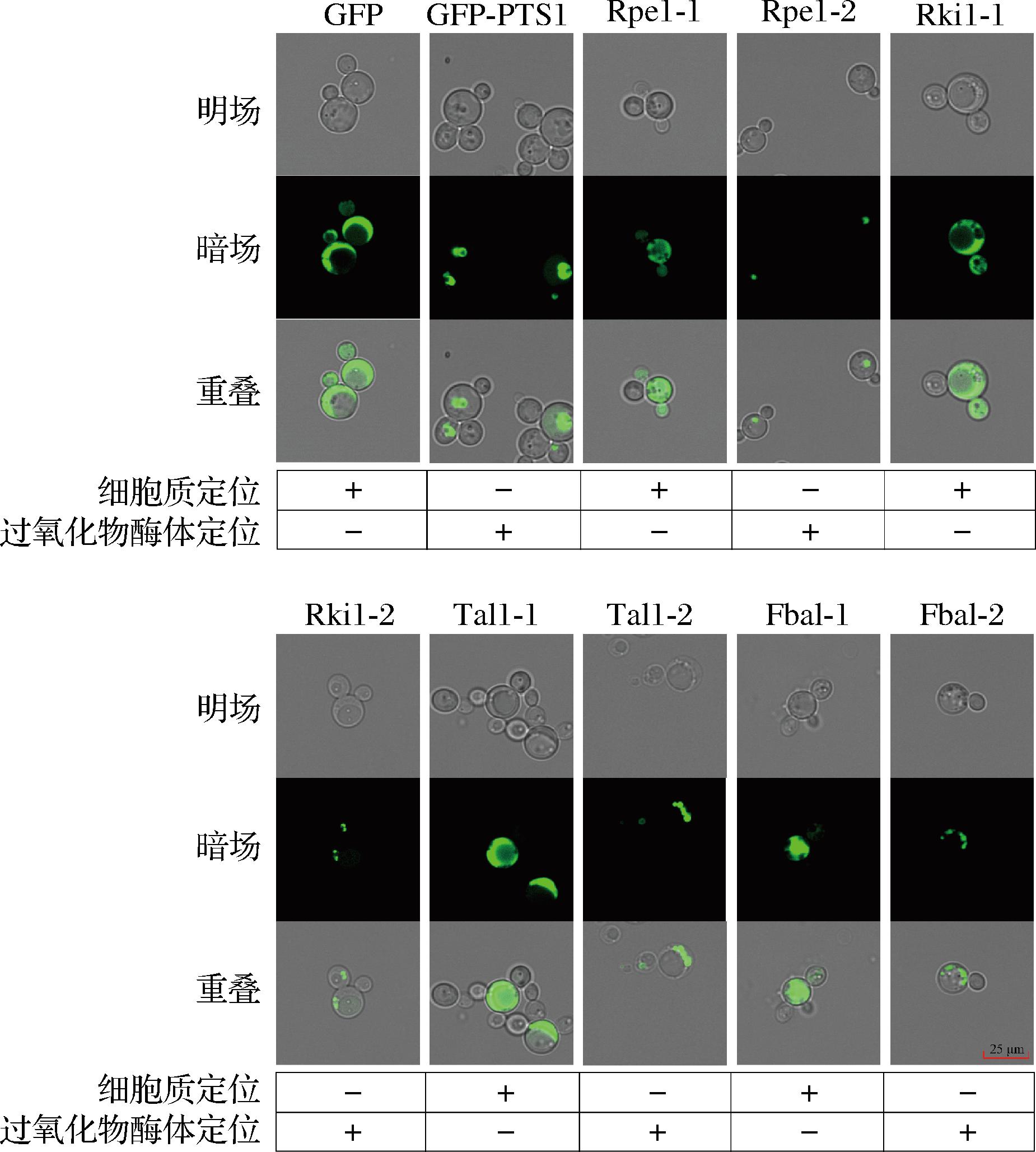
图3 同工酶的定位观察
Fig.3 Observation of isozymes location 注:“+”表示相应蛋白所定位的细胞器,“-”表示相应 蛋白未定位的细胞器。
2.2 过氧化物酶体同工酶参与甲醇代谢的验证
为了验证过氧化物酶体同工酶参与甲醇代谢,分别测定RPE1-1、RKI1-1、TAL1-1、FBA1-1、RPE1-2、RKI1-2、TAL1-2、FBA1-2在YPD及YPM培养基下的转录水平变化,如图4所示,4个细胞质同工酶基因在YPD培养基和YPM培养基中转录水平均无显著性变化,而过氧化物酶体同工酶基因在YPM培养基中的转录水平显著高于其在YPD培养基中的转录水平,该结果说明过氧化物酶体同工酶响应甲醇并参与甲醇代谢。值得注意的是,FBA1-2基因在甲醇培养基中的转录水平提升最为显著,这意味着Fba1-2可能在甲醇代谢过程中发挥重要作用。
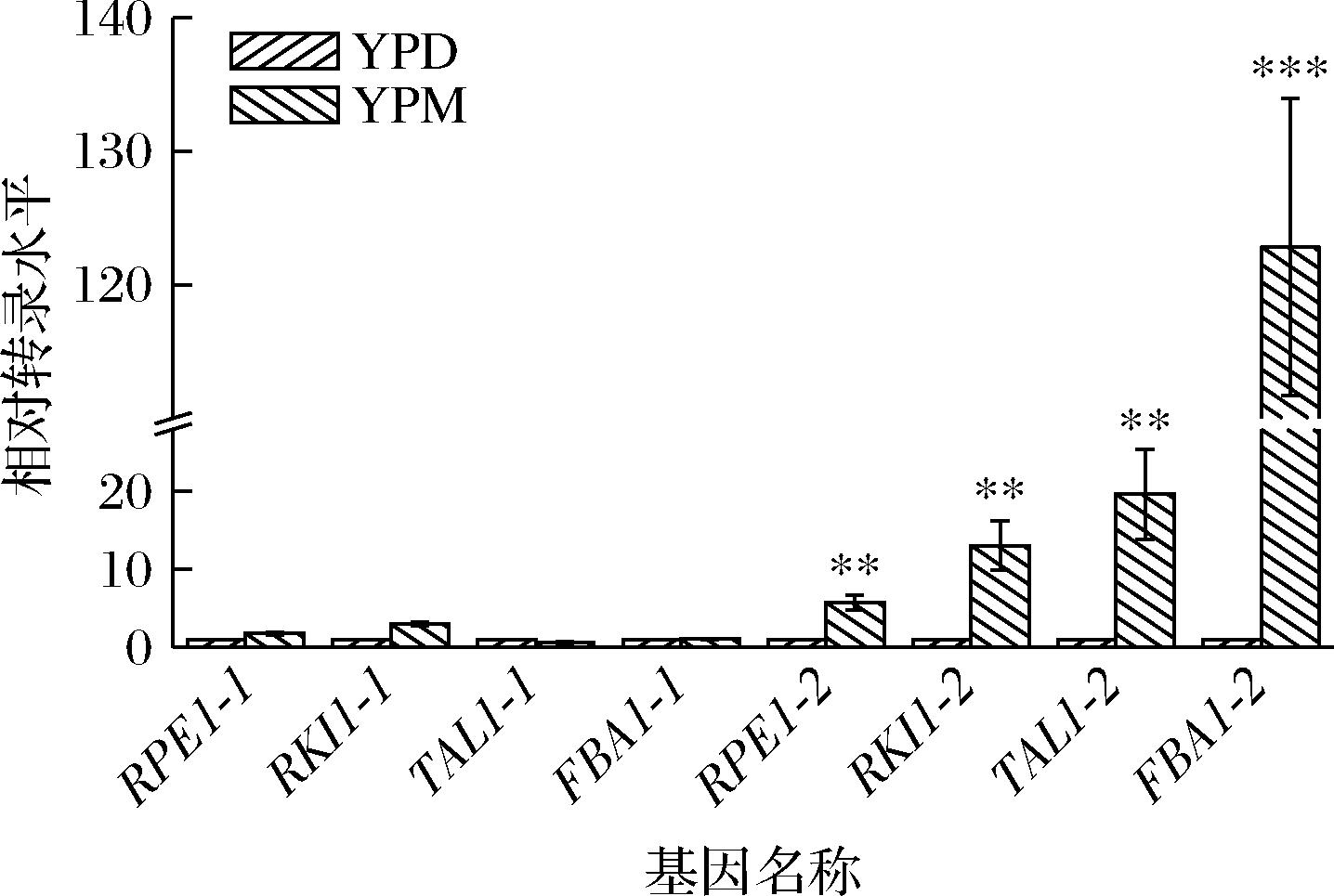
图4 同工酶基因在YPD及YPM培养基中的转录水平变化
Fig.4 Transcriptional changes of isozyme genes in YPD and YPM medium 注:**表示差异显著(0.000 1≤P<0.001),***表示差异 极显著(P<0.000 1)
2.3 过氧化物酶体同工酶的敲除与回补对甲醇代谢的影响
将1.4.1节构建的5株过氧化物酶体同工酶敲除菌在MMH培养基中培养。如图5-a,图5-b所示,RPE1-2、RKI1-2、TAL1-2等基因的敲除对毕赤酵母在甲醇培养基中的生长几乎没有影响,而FBA1-2的敲除会造成轻微的生长抑制,并且其甲醇利用速率也略有减缓,这可能是由于Fba1-2参与了同化途径的两步反应(图1),因此它在整个甲醇代谢过程中发挥重要作用。值得注意的是,虽然4个过氧化物酶体同工酶的单独敲除对毕赤酵母的甲醇生长影响不大,但将4个同工酶同时敲除会造成细胞在甲醇培养基中的生物量急剧下降(图5-a),该结果进一步反映了同化途径的过氧化物酶体区室化对于甲醇代谢的重要性。
为了明确4个过氧化物酶体同工酶中的关键酶,将Rpe1-2、Rki1-2、Tal1-2、Fba1-2单独回补到KO-4菌株中,如图5-c,图5-d所示,Fba1-2的回补能够最大程度恢复四敲菌株在甲醇培养基中的生物量以及甲醇利用速率,该结果说明Fba1-2属于甲醇代谢过程中的关键酶,它可以成为后续酶改造等优化的靶标。
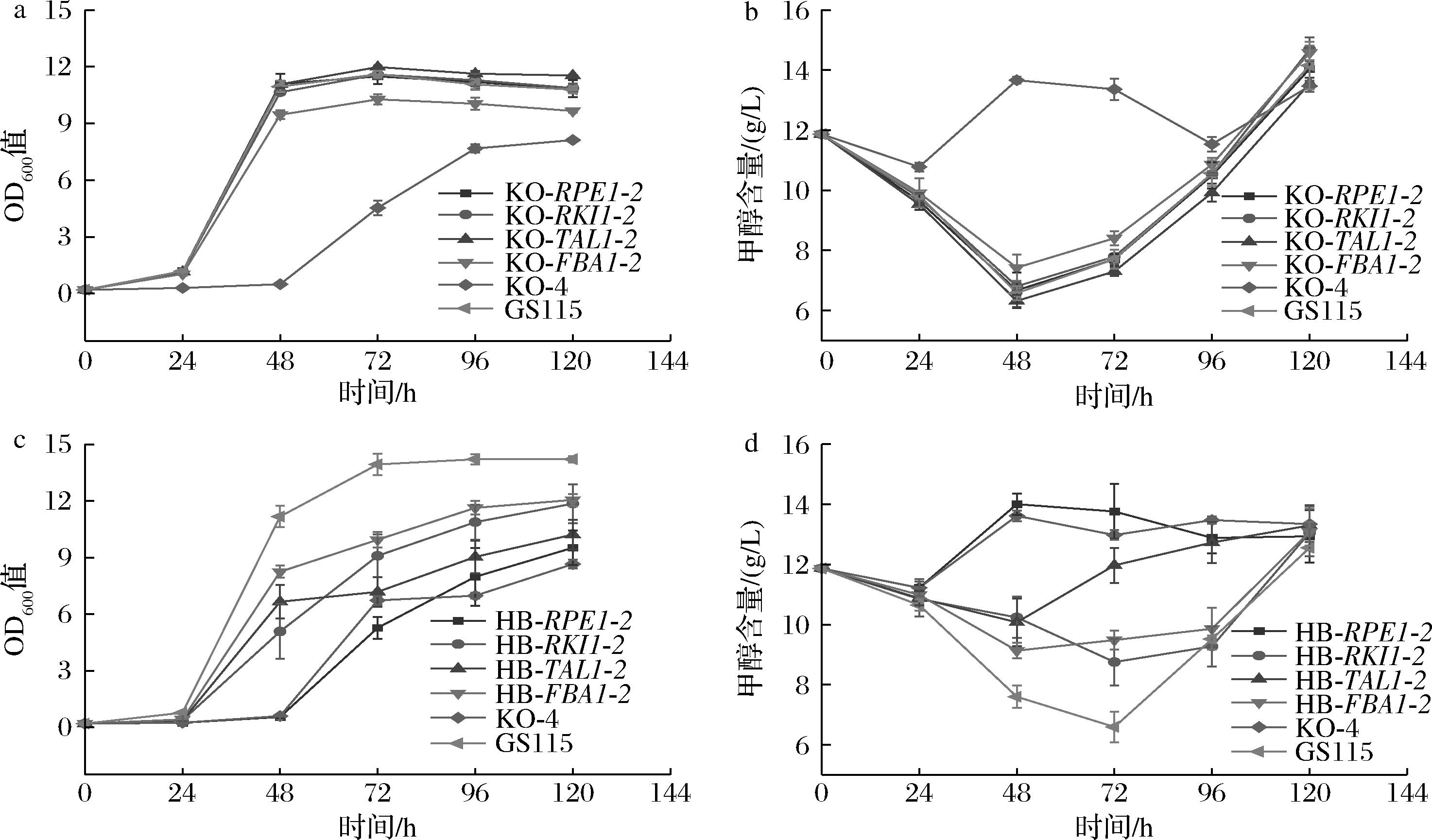
a-过氧化物酶体同工酶敲除对生长的影响;b-过氧化物酶体同工酶敲除对甲醇利用的影响;c-四敲除菌中回补过氧化物酶体 同工酶敲除对生长的影响;d-四敲除菌中回补过氧化物酶体同工酶敲除对甲醇利用的影响
图5 过氧化物酶体同工酶的敲除及回补对表型的影响
Fig.5 Effects of peroxisomal isoenzyme knockout and complement on phenotype
2.4 XU5P主要再生途径的确认
Tal1-2是XU5P再生途径中非氧化磷酸戊糖途径支路的关键酶,其缺失应对甲醇利用造成较大影响。但在结果2.3节中,Tal1-2的缺失和回补对于毕赤酵母的甲醇生长几乎没有影响。该结果表明非氧化磷酸戊糖途径可能不是XU5P再生的主要途径。为了验证这一猜测,本研究又敲除了另一条推测的XU5P再生途径中的关键酶基因,即更改的卡尔文循环中的SHB17基因。如图6所示,SHB17的敲除会造成较严重的生物量下降以及甲醇利用率减缓。这意味着在毕赤酵母中,非氧化磷酸戊糖途径和更改的卡尔文循环是两条互补的XU5P再生途径,其中更改的卡尔文循环为主要途径。
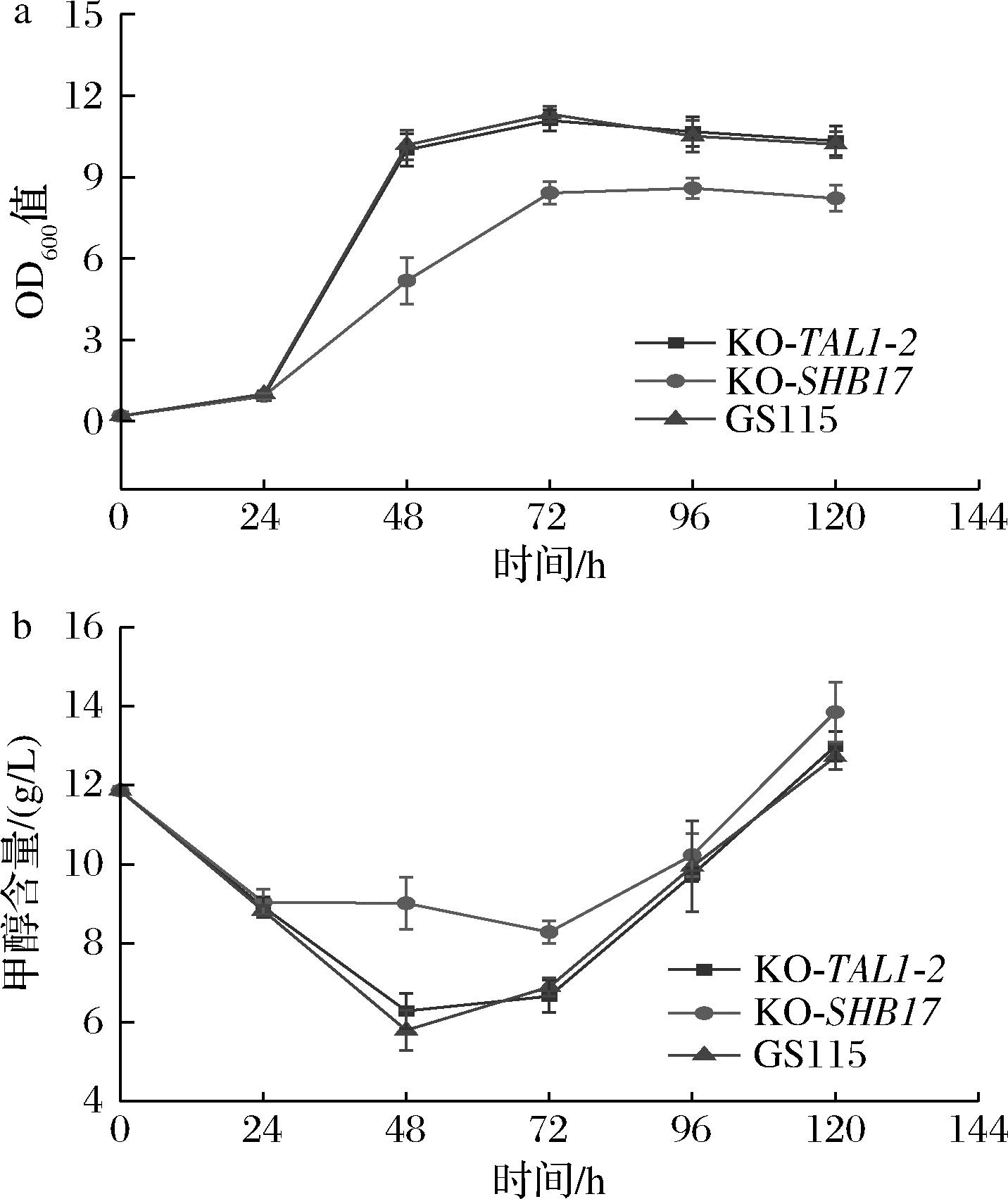
a-TAL1-2或SHB17缺失对生长的影响; b-TAL1-2或SHB17缺失对甲醇利用的影响
图6 TAL1-2或SHB17缺失对表型的影响
Fig.6 Effects of TAL1-2 or SHB17 deletion on phenotype
2.5 四敲除菌中细胞质同工酶的回补
由于同时敲除4个过氧化物酶体同工酶后,毕赤酵母仍然能在甲醇培养基中生长,根据该结果可推测细胞质同工酶可能在过氧化物酶体同工酶缺失后替代其作用。于是,分别在4敲除菌的细胞质及过氧化物酶体中过表达4个细胞质同工酶,得HB-C4和HB-C4P菌株,如图7-a所示,HB-C4菌株的4个细胞质同工酶均定位在细胞质,而HB-C4P菌株中4个加有PTS1定位信号的细胞质同工酶呈点状聚集在过氧化物酶体。随后测定这些菌株在MMH培养基中的生长情况,发现细胞质同工酶无论定位在细胞质还是定位在过氧化物酶体,均无法改善四敲除菌的生长缺陷(图7-b)。该结果说明细胞质同工酶在甲醇代谢过程中起到的作用有限,不能完全替代过氧化物酶体同工酶的功能。上述结果也进一步反映了过氧化物酶体同工酶在进化过程中的独特性以及同化途径的过氧化物酶体区室化对甲醇代谢的重要性。
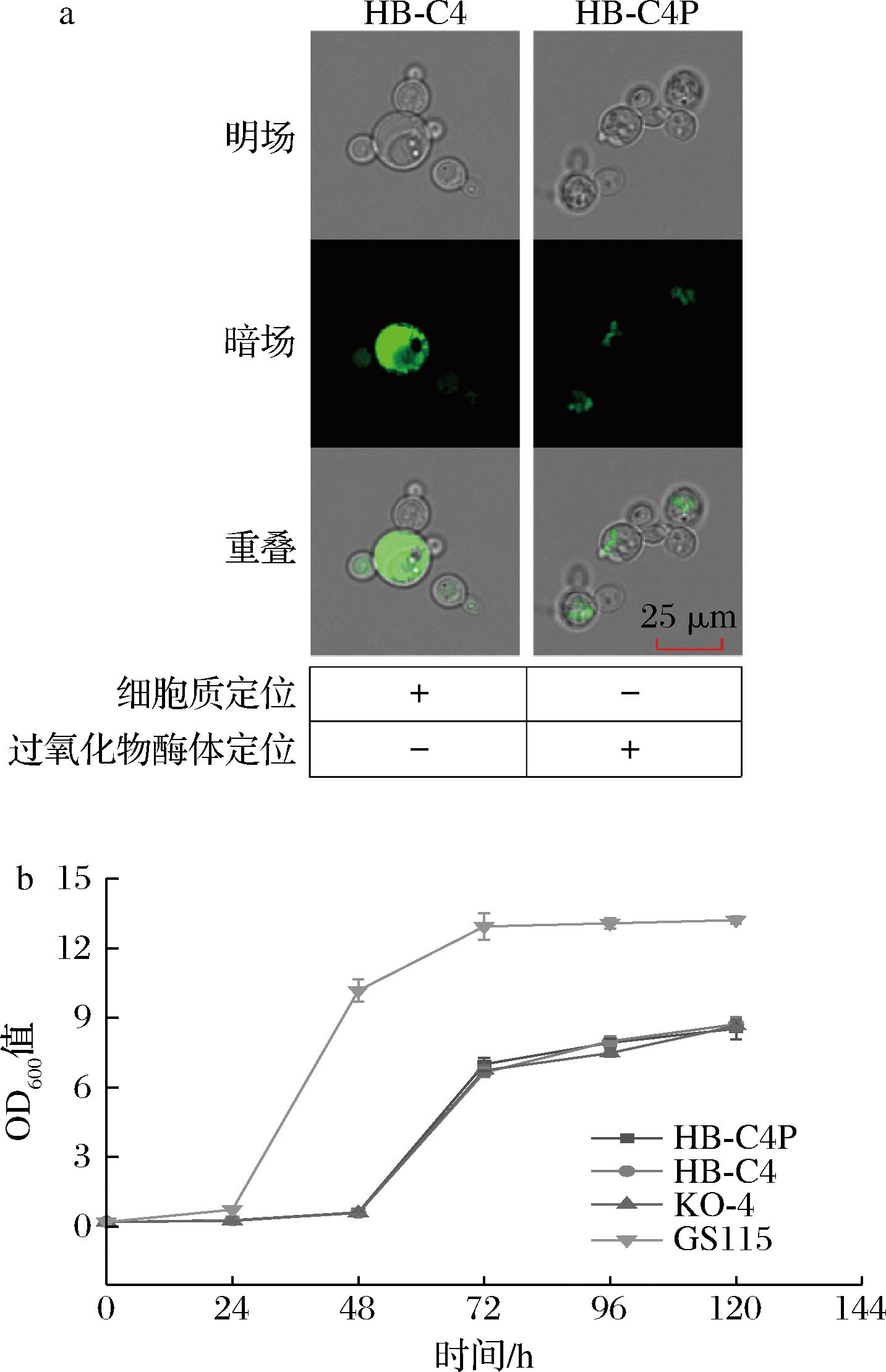
a-同工酶定位验证;b-细胞质同工酶的过表达 对四敲除菌生长的影响
图7 细胞质同工酶的过表达
Fig.7 Overexpression of cytoplasmic isoenzymes
3 结论与讨论
与RUßMAYER等[15]研究一致,本研究发现Rpe1-2、Rki1-2、Tal1-2、Fba1-2等同工酶存在于过氧化物酶体,且参与甲醇代谢。为了进一步明确甲醇代谢机理以及甲醇同化途径的关键酶基因,本研究对4个过氧化物酶体同工酶进行了系统性的研究。
通过对4个过氧化物酶体同工酶进行单敲除、多敲除以及回补实验发现:RPE1-2、RKI1-2、TAL1-2等基因的缺失对毕赤酵母的甲醇代谢几乎没有影响。而与之前的研究结果一致[16],FBA1-2基因的敲除会对菌株的生长与甲醇利用率产生轻微的影响,这意味着FBA1-2可能是重要的同化基因。在之后的过氧化物酶体同工酶回补实验中,这一猜测得到验证。本研究还发现,虽然各个过氧化物酶体同工酶的单敲除对毕赤酵母在甲醇中的生长影响都不大,但如果这4个同工酶同时缺失将会对其生长产生巨大的抑制作用,这一结果反映了同化途径的过氧化物酶体区室化对毕赤酵母甲醇利用的重要性。先前的研究推测,Tal1-2酶所在的非氧化磷酸戊糖途径对XU5P的再生只起到了补充作用[17]。本研究通过比较KO-TAL1-2菌株与KO-SHB17菌株的生长水平发现,Tal1-2的缺失对菌株的生长不会产生可见的影响,而Shb17的缺失却会造成严重的生长缺陷,该结果表明Shb17酶所在的更改的卡尔文循环是同化过程中甲醛受体(XU5P)的主要再生途径。最后,本研究根据在4敲除菌株中过表达细胞质同工酶的结果得出结论:毕赤酵母在长久的进化过程中获得的过氧化物酶体同工酶具有其独特性,对于甲醇代谢扮演着举足轻重的作用。
综上所述,本研究通过对过氧化物酶体同工酶的系统性研究,进一步明确了同化途径过氧化物酶体区室化的重要性,确定了甲醇同化过程中的关键酶基因FBA1-2以及XU5P的主要再生途径为更改的卡尔文循环。菌株的优化是发酵工业的重要一环,本研究可为后续优化天然/非天然甲基微生物甲醇利用效率提供理论依据,有助于合成生物学与发酵工业的进一步发展。
[1] OLAH G A.Beyond oil and gas:The methanol economy[J].Angewandte Chemie International Edition, 2005, 44(18):2636-2639.
[2] BASILE A, DALENA F.Methanol:Science and Engineering[M].Amsterdam:Elsevier, 2018.
[3] AL-ROWAILI F N, JAMAL A, BA SHAMMAKH M S, et al.A review on recent advances for electrochemical reduction of carbon dioxide to methanol using metal-organic framework (MOF) and non-MOF catalysts:Challenges and future prospects[J].ACS Sustainable Chemistry &Engineering, 2018, 6(12):15895-15914.
[4] LAIS A, GONDAL M A, DASTAGEER M A, et al.Experimental parameters affecting the photocatalytic reduction performance of CO2 to methanol:A review[J].International Journal of Energy Research, 2018, 42(6):2031-2049.
[5] ZHU T C, ZHAO T X, BANKEFA O E, et al.Engineering unnatural methylotrophic cell factories for methanol-based biomanufacturing:Challenges and opportunities[J].Biotechnology Advances, 2020, 39:107467.
[6] DE SCHUTTER K, LIN Y C, TIELS P, et al.Genome sequence of the recombinant protein production host Pichia pastoris[J].Nature Biotechnology, 2009, 27(6):561-566.
[7] CEREGHINO J L, CREGG J M.Heterologous protein expression in the methylotrophic yeast Pichia pastoris[J].FEMS Microbiology Reviews, 2000, 24(1):45-66.
[8] LI P Z, ANUMANTHAN A, GAO X G, et al.Expression of recombinant proteins in Pichia pastoris[J].Applied Biochemistry and Biotechnology, 2007, 142(2):105-124.
[9] SCHWARZHANS J P, LUTTERMANN T, GEIER M, et al.Towards systems metabolic engineering in Pichia pastoris[J].Biotechnology Advances, 2017, 35(6):681-710. [10] FICKERS P.Pichia pastoris:A workhorse for recombinant protein production[J].Current Research in Microbiology and Biotechnology, 2014, 4(2):354-363.
[11] ATA Ö, REBNEGGER C, TATTO N E, et al.A single Gal4-like transcription factor activates the Crabtree effect in Komagataella phaffii[J].Nature Communications, 2018, 9(1):4911.
[12] JORD J, DE JESUS S S, PELTIER S, et al.Metabolic flux analysis of recombinant Pichia pastoris growing on different glycerol/methanol mixtures by iterative fitting of NMR-derived 13C-labelling data from proteinogenic amino acids[J].New Biotechnology, 2014, 31(1):120-132.
J, DE JESUS S S, PELTIER S, et al.Metabolic flux analysis of recombinant Pichia pastoris growing on different glycerol/methanol mixtures by iterative fitting of NMR-derived 13C-labelling data from proteinogenic amino acids[J].New Biotechnology, 2014, 31(1):120-132.
[13] JORD J, ROJAS H C, CARNICER M, et al.Quantitative metabolomics and instationary 13C-metabolic flux analysis reveals impact of recombinant protein production on trehalose and energy metabolism in Pichia pastoris[J].Metabolites, 2014, 4(2):281-299.
J, ROJAS H C, CARNICER M, et al.Quantitative metabolomics and instationary 13C-metabolic flux analysis reveals impact of recombinant protein production on trehalose and energy metabolism in Pichia pastoris[J].Metabolites, 2014, 4(2):281-299.
[14] 战春君. 甘油抑制巴斯德毕赤酵母PAOX1机制研究[D].无锡:江南大学, 2018. ZHAN C J.Mechanism of glycerol repression on PAOX1 in Pichia pastoris[D].Wuxi:Jiangnan University, 2018.
[15] RUßMAYER H, BUCHETICS M, GRUBER C, et al.Systems-level organization of yeast methylotrophic lifestyle[J].BMC Biology, 2015, 13:80.
[16] FUKUOKA H, KAWASE T, OKU M, et al.Peroxisomal Fba2p and Tal2p complementally function in the rearrangement pathway for xylulose 5-phosphate in the methylotrophic yeast Pichia pastoris[J].Journal of Bioscience and Bioengineering, 2019, 128(1):33-38.
[17] HOU R, GAO L H, LIU J H, et al.Comparative proteomics analysis of Pichia pastoris cultivating in glucose and methanol[J].Synthetic and Systems Biotechnology, 2022, 7(3):862-868.