β-葡聚糖主要存在于广泛应用于酿造、饲料等工业中的大麦、高粱等谷物的胚乳细胞壁中[1]。由于家禽缺乏相应的肠酶来有效分解胚乳细胞壁的主要多糖:β-葡聚糖[2],谷物应用于家禽饲料中往往营养价值较低。β-葡聚糖遇水会膨胀,黏度增加,不仅导致家禽肠道中的食糜黏稠度增高、养分吸收受阻,而且含有β-葡聚糖的黏性粪便极大增加了养鸡场清洁工作的劳动量,并带来一定的环境污染问题[3]。在饲料中加入β-葡聚糖酶有利于改善以上问题。由于家禽特殊的摄食习惯,其饲料需要保持较高的颗粒稳定性(particle stability,PDI)。在造粒生产环节阶段中采用高温处理使饲料中的淀粉充分糊化是提高饲料PDI的主要方法。张亮等[4]研究了造粒温度对颗粒饲料质量的影响,发现温度的升高(80~90 ℃)可以改善颗粒饲料的质量,其中造粒温度达到90 ℃时获得的饲料的PDI等指标显著优于采用85 ℃或80 ℃获得的饲料。目前市场上尚没有能耐受80 ℃以上高温的β-葡聚糖酶。本课题组在前期工作中,发现了一种来源于黑曲霉的高耐热葡聚糖酶AnEglA6[5-6],这种酶在乳酸克鲁维酵母中的表达产物AnEglA6K能短时耐受85 ℃高温[6]。序列比对发现,AnEglA6分子由两部分组成,前端311个氨基酸残基形成催化功能结构域,而羧基端的36个氨基酸残基则构成纤维素结合结构域(cellulose binding domain,CBD)[6],两者之间是由44个氨基酸残基组成的连接桥。在葡聚糖酶系中,CBD往往与结晶型纤维素的水解有关[6-7],但AnEglA6K对微晶纤维素只具有很微弱的水解能力。碳水化合物结合模块(carbohydrate binding module,CBM)是一类多模块酶蛋白,其功能是与纤维素、木聚糖等碳水化合物底物结合[7],CBM通常与催化多糖水解反应的催化结构域(catalytic domains,CD)连接[8]。CBD是最早被发现的碳水化合物结合模块[9]。部分CBM家族具有与结晶型纤维素结合的能力,广泛应用于碳水化合物生物材料的功能化,促进异源蛋白质的表达、纯化、稳定和固定化等[10]。纤维素酶应用于畜禽饲料中能提高仔鸡、肉牛等动物营养物质的消化率[11]。替换或增加CBD是提高纤维素酶降解结晶型纤维素活性的方法之一[12]。本课题组在前期工作中,将不同微生物来源的碳水化合物结合模块CBM更换AnEglA6自身的CBD,结果发现所获得的融合体酶的催化活性基本丧失或大幅度下降,但AnEglA6与来自嗜热菌(Caldicellulosiruptor kristjanssonii)的木聚糖酶的碳水化合物结合模块CBM9[13]以及来自荧光假单胞菌(Pseudomonas fluorescens)的木聚糖酶的模块CBM10[14]重组获得的融合酶保持了较高的葡聚糖酶活力,两者均获得了明显的降解微晶纤维素的能力,后者的热稳定性显著提高。本文主要研究上述融合酶的获得方法及应用性能。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
含有eglA6基因的质粒pPIC9K-eglA6为实验室前期构建[5]。表达载体pPIC9K、宿主菌巴斯德毕赤酵母GS115为Invitrogen公司产品,为本课题组保藏。含有碳水化合物结合模块CBM9[13]和CBM10[14]的编码基因按文献报道的序列由苏州金唯智生物科技有限公司合成,片段合成后插入载体pUC57后分别获得重组质粒pUC-CBM9和pUC-CBM10。
1.1.2 菌株和质粒
限制性内切酶、DNA聚合酶PrimerStar、核酸标准分子质量,宝日医(北京)有限公司;无缝克隆连接试剂盒,宝日医公司;质粒提取和DNA片段回收试剂盒,苏州康宁生物科技有限公司;蛋白标准分子质量,翌圣生物科技有限公司;茯苓多糖、酵母葡聚糖、大麦葡聚糖、桦树木聚糖、CMC-Na,MegaZyme公司;热凝胶、甘露聚糖、微晶纤维素,上海源叶生物技术公司;蛋白纯化镍亲、层析填料,无锡天演技术有限公司。
1.1.3 主要培养基与试剂的配制
LB培养基(luria-bertani medium)、酵母浸出粉胨葡萄糖培养基(yeast extract peptone dextrose medium, YPD)、MM基本培养基(mineral medium)、甘油缓冲复合培养基(buffered glycerol-complex medium, BMGY) 和诱导表达培养基(buffered methanol-complex medium, BMMY)均参考文献[5]。
柠檬酸-磷酸二氢钠缓冲液参考文献[5]。
1.1.4 引物及基因的合成
根据eglA6的核苷酸序列(NCBI登录号:MG913988)以及CBM9和CBM10的核苷酸序列[11-12]设计引物,序列如下:
Peg01:5′-CTGAAGCTTACGTAGAATTCAGTCCG-AGGGCTAAGCGGTC-3′
Peg02:5′-GCGAATTAATTCGCGGCCGCCTATTA-atggtgatggtgatggtgCAAACACTGCGAATACCAC-3′
Peg9:5′-CTTACATATATCCTCTTTGCCGCAGCT-ACAGCCGTCGATG-3′
Peg10:5′-CAATTACATTGCTGGTTACCCGCAGCTACAGCCGTCGATG-3′
其中Peg01和Peg02用于扩增eglA6完整的成熟肽编码区。Peg01和Peg9、Peg10用于扩增eglA6中不含纤维素结合结构域编码区的部分。引物中单下划线部分为内切酶EcoR Ⅰ和Not I识别位点,双下划线部分与CBM9、CBM10编码区5′-端互补,用于片段间的无缝连接。单下划线小写字母部分为His-tag编码区。
根据核苷酸序列[13-14]设计扩增CBM9、CBM10的引物如下:
P901:5′-GCAAAGAGGATATATGTAAG-3′
P902:5′-GCGAATTAATTCGCGGCCGCCTATTA-atggtgatggtgatggtgGGTAACCAGCAATGTAATTG-3′
P1001:5′-GGTAACCAGCAATGTAATTG-3′
P1002:5′-GCGAATTAATTCGCGGCCGCCTATTA-atggtgatggtgatggtgGCCGCTCCCCACAATACCAA-3′
双下划线部分与CBM基因5′-端一致,用于CBM基因的扩增,同时也用于与eglA6扩增片段的无缝连接。
1.2 实验方法
1.2.1 融合酶表达载体构建
以质粒pPIC9K-eglA6作为模板,用引物Peg01、Peg9扩增eglA6基因中不含纤维素结合结构域编码区的片段。PCR条件为:98 ℃预变性 3 min;94 ℃变性 30 s,54 ℃退火30 s,72 ℃延伸 1 min,30个循环。扩增片段大约为1.1 kb。再以pUC-CBM9为模板,以引物P901、902扩增片段CBM9。PCR条件为:98 ℃预变性 3 min,94 ℃变性 30 s,54 ℃退火30 s,72 ℃延伸 40 s,30个循环。上述PCR扩增产物均以胶回收方法进行纯化。载体pPIC9K以EcoR Ⅰ和Not Ⅰ酶切线性化,以PCR产物纯化试剂盒纯化后与两次PCR获得的片段混合,用无缝连接试剂盒进行连接。连接产物转化大肠杆菌JM109,转化子提取质粒后经酶切电泳鉴定,获得重组质粒pPIC9K-eglA6-CBM9。用引物Peg01、Peg10扩增eglA6基因,以pUC-CBM10为模板,以P1001、P1002为引物扩增片段CBM10,扩增产物约为0.15 kb。上述扩增片段与线性化的载体pPIC9K连接获得重组质粒pPIC9K-eglA6-CBM10。操作方法与质粒pPIC9K-eglA6-CBM9构建方法一致。用引物Peg01、Peg02扩增eglA6基因中包含纤维素结合结构域的完整编码区的片段,由于该片段末端带有His-tag标签,为便于和已有文献报道[5]区别,将该片段命名为eglA6T。上述扩增产物纯化后直接与经EcoR I和Not I双酶切的pPIC9K连接。
1.2.2 酵母转化及筛选
将1.2.1节中获得的重组质粒用Bgl Ⅱ线性化后分别电转化毕赤酵母GS115,毕赤酵母的电转化及转化子筛选方法参考文献[15]。获得酵母转化子后取单菌落划线分离后再取单菌落参考文献[5]的方法摇瓶发酵检测葡聚糖酶酶活力。
1.2.3 融合酶的纯化
离心并收集重组菌发酵上清液。由于融合酶C端带有His-tag标签,用镍柱进行蛋白纯化,方法参考文献[16]。SDS-PAGE及蛋白浓度测定方法均参考文献[5]。
1.2.4 融合酶酶学性质的测定
酶活力检测、酶学性质分析等方法均与文献[6]相同。其中酶活力定义:以大麦葡聚糖为底物,每分钟分解底物产生的还原糖,其还原力相当于1 μmol葡萄糖所需的酶量,用1 U表示。滤纸纤维素酶检测方法[17]:将滤纸裁剪成0.5 cm×0.5 cm大小的正方形,在2 mL的离心管中放入2片裁剪好的新华滤纸,加入400 μL的柠檬酸-磷酸二氢钠缓冲液和100 μL酶液,在最适温度、pH的反应条件下反应1 h立即加入750 μL的DNS溶液,沸水浴5 min。每分钟降解滤纸释放1 μmol 葡萄糖所需的酶量,定义为一个滤纸纤维素酶活力单位U。
1.2.5 融合酶水解产物的分析
取葡萄糖、麦芽糖、麦芽三糖和麦芽六糖混合后用体积分数75%的乙醇配制成10 mg/mL的溶液,作为薄层层析的标准糖溶液。展层溶剂体积比为乙酸乙酯∶冰醋酸∶水=2∶2∶1,显色剂为1 g二苯胺、1 mL苯胺和5 mL磷酸溶于50 mL丙酮。将融合酶与大麦葡聚糖于75 ℃下进行充分反应,待酶将底物水解彻底后进行薄层层析,操作方法与文献[6]相同。
2 结果与分析
2.1 重组质粒的构建
以pPIC9K-eglA6为模板,Peg01、Peg9为引物,扩增获得1.1 kb左右的片段,与eglA6基因中不含CBD编码区的片段大小一致。以pUC-CBM为模板,以P901、P902为引物,PCR扩增获得约0.6 kb的片段,与CBM9编码区大小一致。上述2个扩增产物与经双酶切的表达载体pPIC9K混合后进行无缝连接并转化大肠杆菌后获得转化子,提取质粒以EcoR I、Not I双酶,电泳结果如图1中泳道2所示。由电泳图可见,该重组质粒酶切后得到大约9.3 kb和1.7 kb的片段,分别与pPIC9K及片段eglA6-CBM9一致,该质粒命名为pPIC9K-eglA6-CBM9。采用同样的方法构建重组质粒pPIC9K-eglA6-CBM10,其酶切电泳图如图1泳道3所示。以Peg01、Peg02为引物扩增获得的含完整编码区的eglA6T片段直接与经双酶切的载体pPIC9K连接,获得重组质粒pPIC9K-eglA6T,其酶切电泳图如图2泳道1所示。
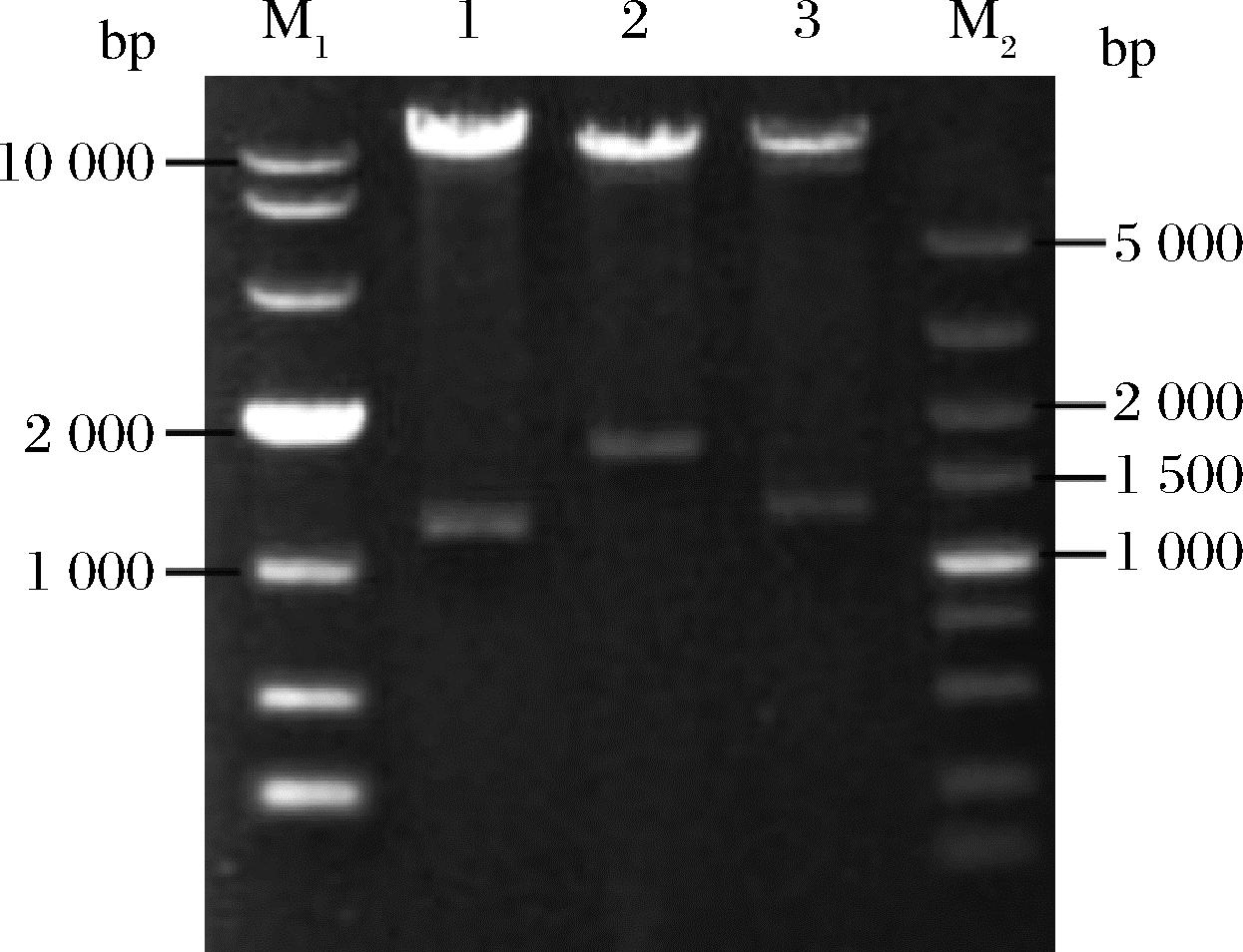
M1-10 000 bp的核酸Marker;M2-5 000 bp的核酸Marker; 1-重组质粒pPIC9K-eglA6T以EcoR I、Not I双酶切产物; 2-重组质粒pPIC9K-eglA6-CBM9以EcoR I、Not I双酶切产物; 3-重组质粒pPIC9K-eglA6-CBM10以EcoR I、Not I双酶切产物
图1 重组质粒酶切验证
Fig.1 Recombinant plasmid digestion verification
2.2 重组酵母的构建、融合酶的表达与纯化
上述3种重组质粒线性化后电转化毕赤酵母GS115,在MM平板上筛选转化子。3种重组质粒的转化子各任取20个,划线分离后取单菌落经摇瓶发酵检测酶活力。结果显示,重组质粒pPIC9K-eglA6T转化子的酶活力大约在500~900 U/mL,与韩冰等[5]报道的采用类似方法构建的表达同一条基因的酵母转化子酶活力基本一致。pPIC9K-eglA6-CBM9转化子的酶活力大约为150~250 U/mL,pPIC9K-eglA6-CBM10转化子的酶活力大约在600~750 U/mL。其中pPIC9K-eglA6-CBM9转化子中编号为11的转化子发酵酶活力最高,命名为Pichia pastoris GS115/pPIC9K-eglA6-CBM9 YL11,简称AnEg-CB9 YL11,所表达的融合酶命名为AnEg-CBM9用于后续实验。质粒pPIC9K-eglA6-CBM10转化子中酶活力最高的为Pichia pastoris GS115/pPIC9K-eglA6-CBM10 YL05,简称AnEg-CB10 YL05,所表达的融合酶命名为AnEg-CBM10。质粒pPIC9K-eglA6T转化子中酶活力最高的为Pichia pastoris GS115/pPIC9K-eglA6T YL19,简称AnEglA6T YL19,所表达的融合酶命名为AnEglA6T。
上述菌种的发酵上清液按1.2.3节进行纯化。纯化产物蛋白电泳如图2所示。用图像分析软件Image Lab 4.0对电泳条带进行推算,结果显示,AnEg-CBM9和AnEg-CBM10的表观分子质量分别为81 kDa和59 kDa。含野生酶完整结构的AnEglA6T的表观分子质量为52 kDa,与本课题组前期研究获得的AnEglA6的分子质量基本一致[5]。根据核苷酸序列推算,AnEg-CBM9、AnEg-CBM10的理论分子质量分别为62 kDa和43 kDa,AnEglA6T理论分子质量为41 kDa。显然3种酶的表观分子质量均明显高于理论分子质量,这是真核生物分泌表达的常见现象。真核生物的胞外蛋白在分泌表达过程中往往在内质网中被糖基化修饰,因此胞外蛋白的表观分子质量往往大于理论分子质量。将上述3种纯化得到的融合酶分别用于酶学性质的研究。
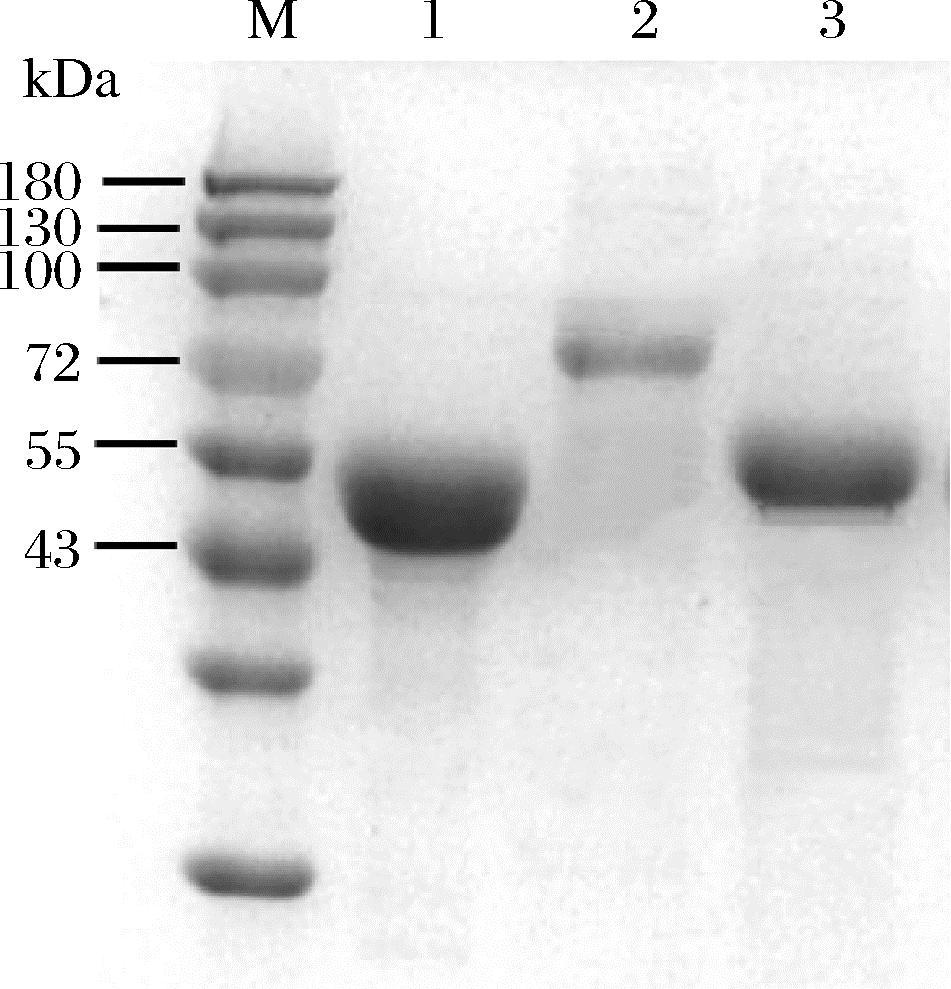
M-Protein Ladder;1-AnEglA6T;2-AnEg-CBM9; 3-AnEg-CBM10
图2 融合酶SDS-PAGE分析
Fig.2 SDS-PAGE analysis of fusion enzymes
2.3 酶学性质
2.3.1 融合酶的葡聚糖酶性质
以大麦葡聚糖为底物,在pH值为4.5的条件下,测定60~90 ℃下2种融合酶的相对酶活力,结果如图3-a所示。这3种酶的催化活性与温度的关系非常相似,最适温度均为75 ℃,在90 ℃时只有AnEg-CBM10有酶活力,AnEg-CBM9和AnEglA6T的酶活力几乎为0。AnEglA6T的性质与韩冰等[5]报道的eglA6基因在巴斯德毕赤酵母中的表达产物AnEglA6的性质基本一致。
在最适温度75 ℃下,测定pH 3~7 时2种融合酶的相对酶活力。结果如图3-b所示。2种融合酶活力与pH的关系有一定的差异。AnEglA6T的最适pH值为4,这与韩冰等[5]的报道一致,而融合体酶AnEg-CBM9和AnEg-CBM10的最适pH值分别是4.5和5.0,略有改变。
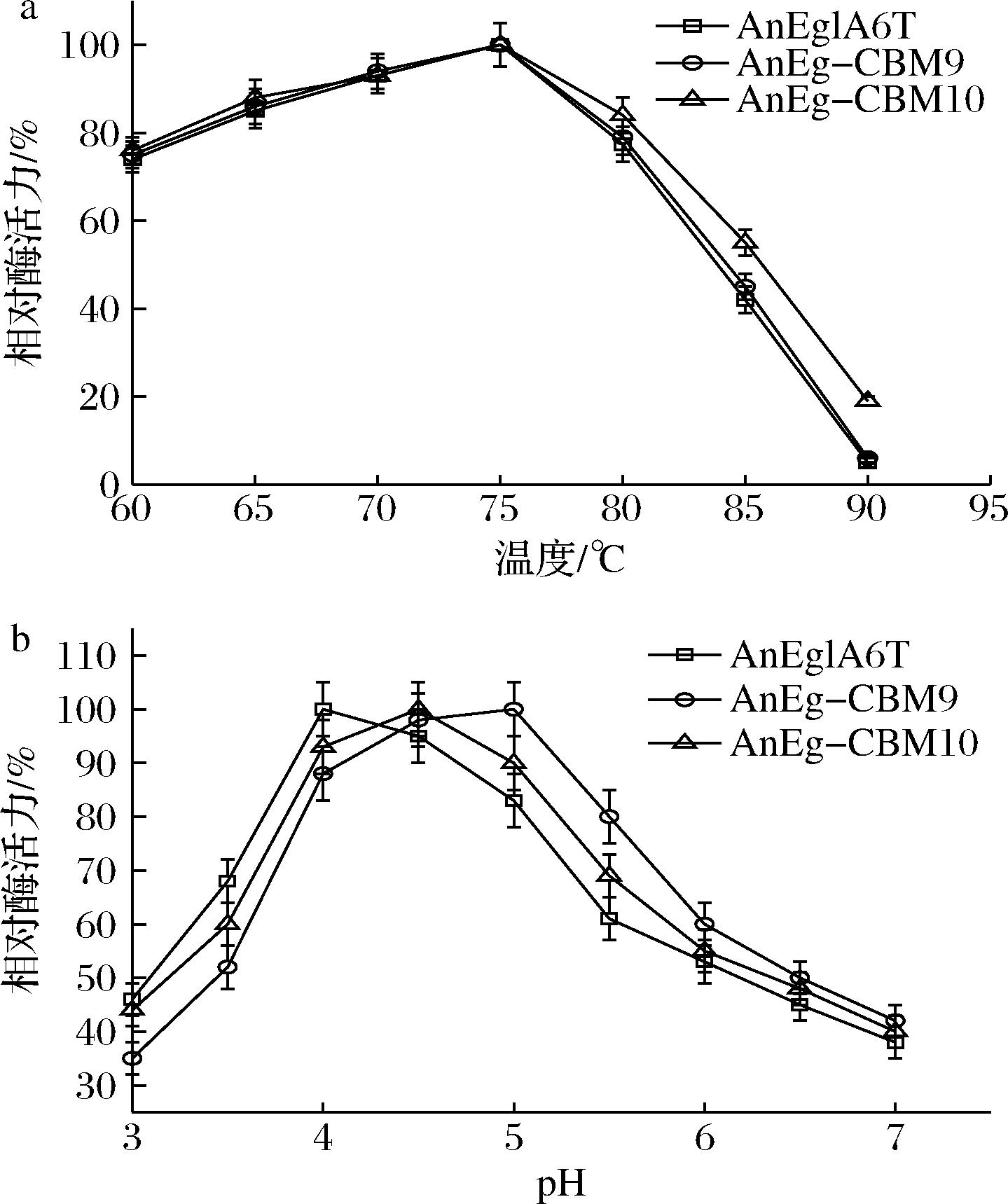
a-温度;b-pH
图3 温度和pH对融合酶葡聚糖活性的影响
Fig.3 Effect of temperature and ph on the glucan activity of fusion enzymes
2.3.2 融合酶的稳定性
在各自的最适pH条件下,分别测定了融合酶在75、80、85 ℃下的相对酶活力变化。由图4可见,3种酶的热稳定性有相当大的差异。其中,AnEglA6T和AnEg-CBM9的耐热性十分接近,AnEg-CBM10的热稳定性显著高于AnEglA6T和AnEg-CBM9。在85 ℃高温下,AnEglA6和AnEg-CBM9在保温1 h后残余酶活力均只有10%左右,而同样条件下AnEg-CBM10的残余酶活力约为30%。进一步检测显示,85 ℃下AnEg-CBM10的酶活力半衰期大约为40 min,另两种酶均不足5 min。在90 ℃下,AnEglA6T和AnEg-CBM9快速失活,大约在5 min后即完全失活。AnEg-CBM10可以短时耐受90 ℃高温,其90 ℃下的酶活力半衰期大约为7 min。
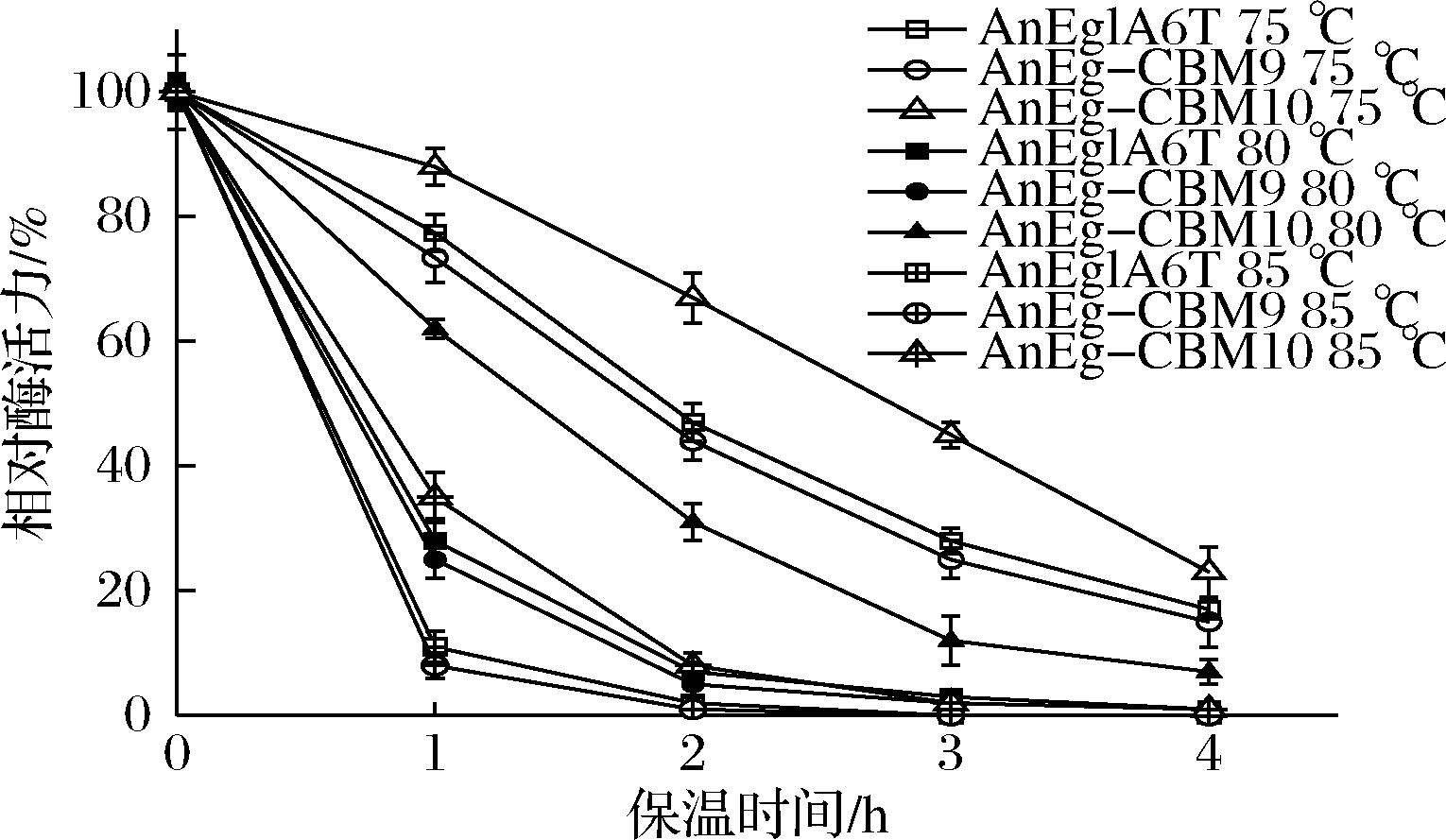
图4 融合酶的热稳定性
Fig.4 Thermal stability of fusion enzymes
2.3.3 融合酶水解产物分析
将融合酶与大麦葡聚糖混合反应后检测反应产物,结果如图5所示。3种葡聚糖酶与底物反应后的产物为不同分子质量的寡糖,主体产物均比较接近麦芽糖和麦芽三糖,也形成少量分子质量稍大的寡糖。
内切型葡聚糖酶的特点就是产生大量不同分子质量的寡糖,AnEglA6因这一特点被判定为内切型葡聚糖酶[5]。由图5可知,融合酶AnEg-CBM9和AnEg-CBM10的水解产物与AnEglA6基本相同,可见AnEg-CBM9和AnEg-CBM10也是内切型葡聚糖酶。
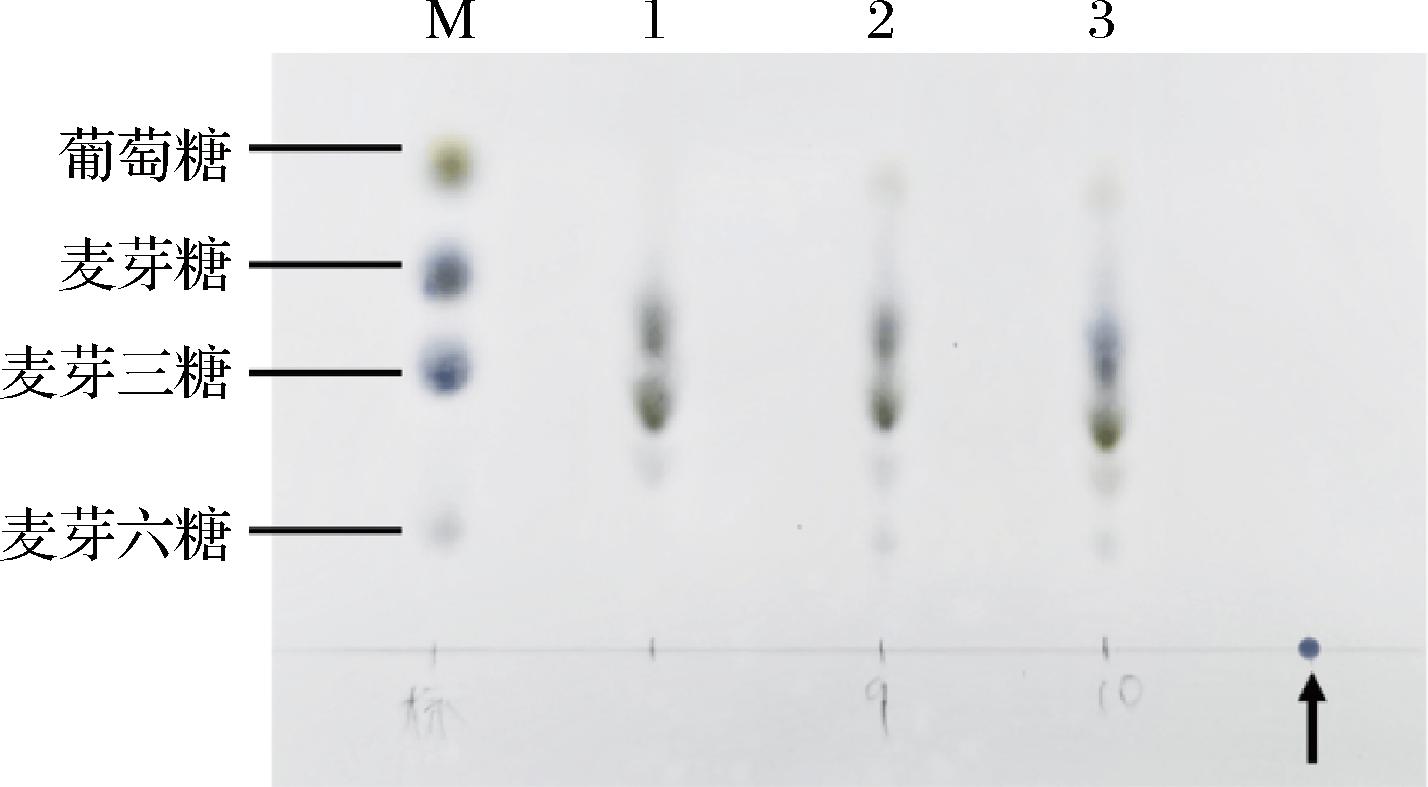
M-标准糖溶液(葡萄糖、麦芽二糖、麦芽三糖和麦芽六糖的混合 溶液);1-AnEglA6T的水解产物;2-AnEg-CBM9的水解产物; 3-AnEg-CBM10的水解产物;箭头所指处为5 g/L大麦葡聚糖
图5 重组葡聚糖酶水解产物的薄层层析
Fig.5 Thin layer chromatography of recombinant glucanase hydrolysate
2.3.4 融合酶的底物特异性
在最适条件下,将2种融合酶分别与10 g/L的不同底物反应并测定比酶活力,结果如表1所示。与野生型酶AnEglA6T相比,两种融合酶的底物特异性有明显的变化。大麦葡聚糖和燕麦葡聚糖为底物时,3种酶的比酶活力基本一致。以地衣多糖为底物时,两种融合酶的比酶活力均比野生型酶AnEglA6有较大幅度的下降。以CMC-Na为底物时,两种融合酶的比酶活力明显高于AnEglA6T,其中AnEg-CBM10的比酶活力几乎达到AnEglA6T的2倍。内切型β-1,3-1,4-葡聚糖酶的特点是水解葡聚糖分子内部β-1,4-糖苷键,但优先水解与1,3-键相邻的β-1,4键[1],因此这一类酶对β-1,3-键丰富的地衣多糖具有更高的水解效率,而对只有β-1,4-键的CMC-Na水解效率较低。由表1可知,3种酶以地衣多糖为底物时比酶活力最高,而以CMC-Na为底物时的活性较低,结合前文对水解产物的研究结果可知,3种酶均具有内切型β-1,3-1,4-葡聚糖酶的特点,但是它们对不同底物的水解效率又表现出明显的差异。两种融合酶的一个特点是对地衣多糖的水解活性明显低于AnEglA6T,而对CMC-Na的活性又明显高于AnEglA6T,可见β-1,3-键对两种融合酶的影响相对较小。两种融合酶的另一个特点是其底物范围明显扩大,均对微晶纤维素表现出明显的水解活性,具有纤维素酶的特点,这显然是纤维素结合结构域赋予融合酶的新功能。同时,这两种融合酶还获得了水解酵母葡聚糖和茯苓多糖的能力,这是一个意外的结果,因为提供催化结构域的AnEglA6和提供纤维素结合结构域的两种木聚糖酶均不具有水解β-1,3型葡聚糖的能力。两种融合体酶对木聚糖、甘露聚糖和半乳聚糖等非葡萄糖单元构成的多糖没有表现出水解活性,由此推测融合体酶可能只限于水解葡萄糖单元构成的底物。以滤纸为底物对两种融合体酶的反应条件进行检测,结果显示,AnEg-CBM9和AnEg-CBM10的最适温度均为60 ℃,最适pH值分别为5.5和6.0。进一步以纤维素酶为依据检测融合酶的热稳定性,结果与采用葡聚糖酶为依据获得的结果基本一致。实验结果看,融合体酶在高温下对结晶型纤维素的催化活性显著下降,但这一变化与酶的高温失活并不存在必然关系。
表1 底物特异性分析
Table 1 Substrate specificity analysis
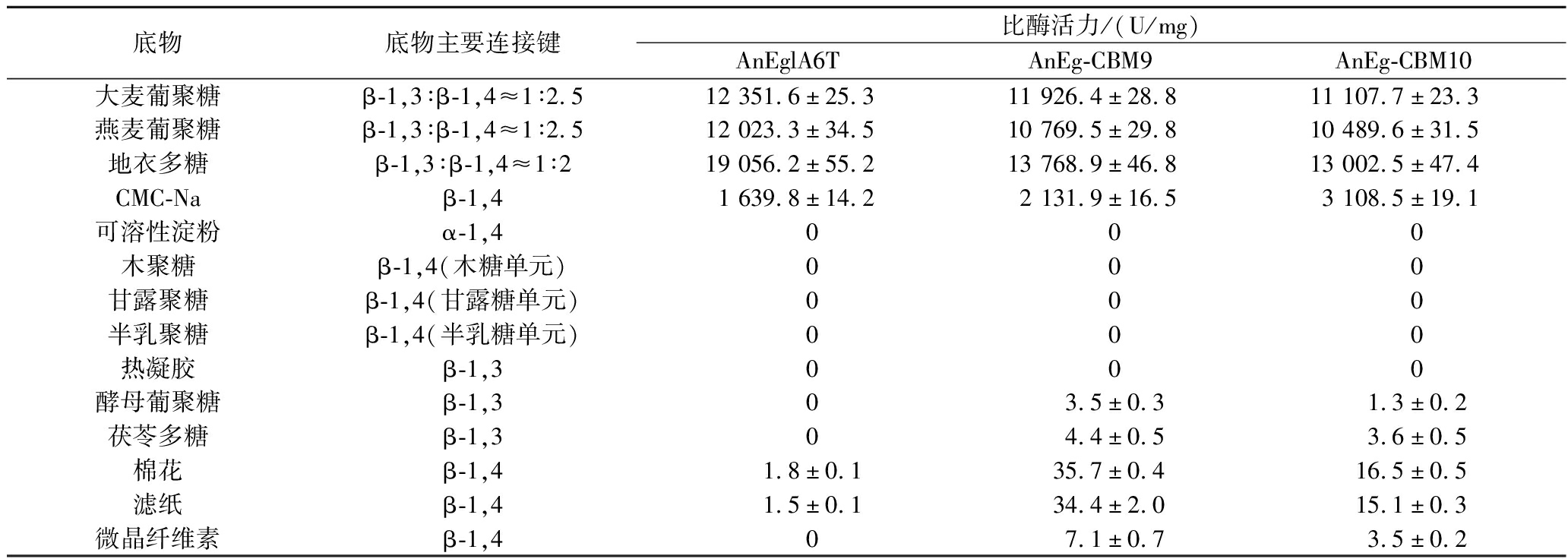
底物底物主要连接键比酶活力/(U/mg)AnEglA6TAnEg-CBM9AnEg-CBM10大麦葡聚糖β-1,3∶β-1,4≈1∶2.512 351.6±25.311 926.4±28.811 107.7±23.3燕麦葡聚糖β-1,3∶β-1,4≈1∶2.512 023.3±34.510 769.5±29.810 489.6±31.5地衣多糖β-1,3∶β-1,4≈1∶219 056.2±55.213 768.9±46.813 002.5±47.4CMC-Naβ-1,41 639.8±14.22 131.9±16.53 108.5±19.1可溶性淀粉α-1,4000木聚糖β-1,4(木糖单元)000甘露聚糖β-1,4(甘露糖单元)000半乳聚糖β-1,4(半乳糖单元)000热凝胶β-1,3000酵母葡聚糖β-1,303.5±0.31.3±0.2茯苓多糖β-1,304.4±0.53.6±0.5棉花β-1,41.8±0.135.7±0.416.5±0.5滤纸β-1,41.5±0.134.4±2.015.1±0.3微晶纤维素β-1,407.1±0.73.5±0.2
注:以棉花、滤纸和微晶纤维素为底物时,AnEg-CBM9和AnEg-CBM10的酶活力检测均为在纤维素酶最适条件下的检测结果。
2.3.5 融合酶结构变化影响催化活性改变的初步分析
利用I-Tasser对融合酶进行同源建模,底物口袋使用网站POCASA 1.1进行预测,结果如图6所示。浅灰色区域为蛋白结构部分,深灰色区域为预测的蛋白质底物口袋。初步测算显示两种融合体酶的底物结合口袋显著大于AnEglA6T,这可能间接说明了这3种酶对底物利用能力的改变。融合酶AnEg-CBM9的结合结构域CBM9也显示了两个可能的底物结合口袋,如图6箭头所指,这可能证明了CBM的不同改变了重组蛋白的结构,赋予了融合酶一些新的功能[18]。在上述同源建模的基础上可以进一步预测可能的催化反应的配体以及与配体结合的氨基酸残基位点,结果如图7及表2所示。
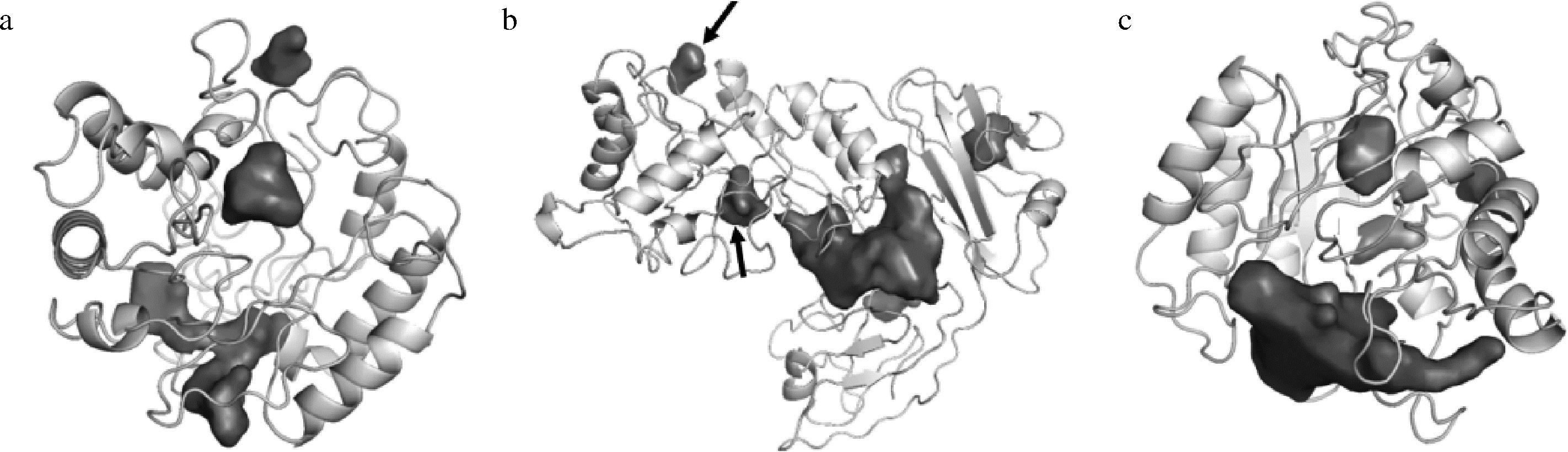
a-AnEglA6T;b-AnEg-CBM9;c-AnEg-CBM10
图6 融合酶同源建模模型及底物口袋预测
Fig.6 Fusion enzyme homologous modeling model and substrate pocket prediction
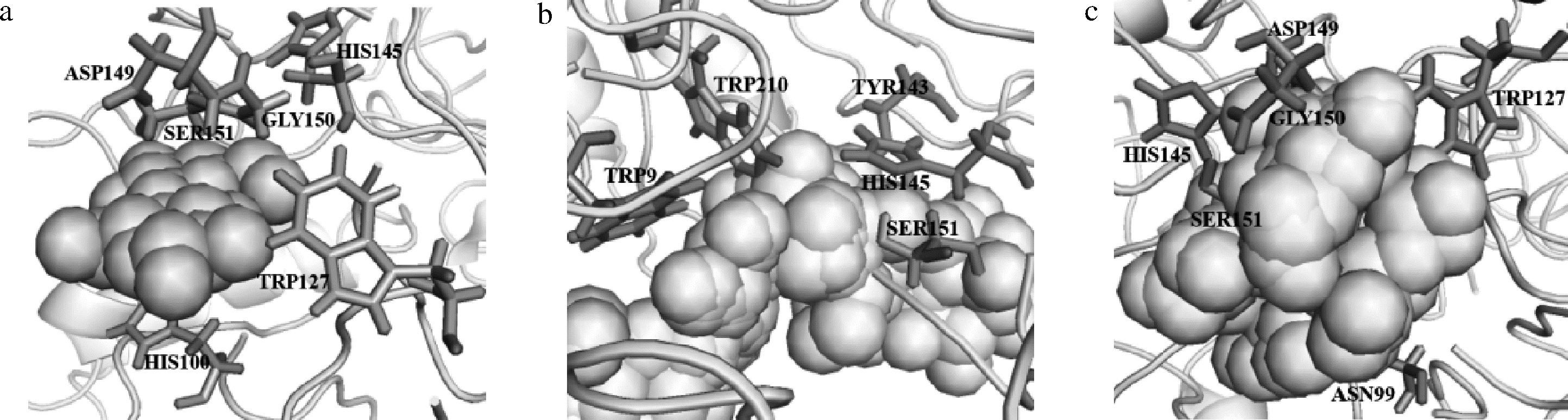
a-AnEglA6T;b-AnEg-CBM9;c-AnEg-CBM10
图7 融合酶配体结合位点残基
Fig.7 Fusion enzyme ligand binding site residues
表2 融合酶配体名称及配体结合位点残基
Table 2 Ligand names and ligand binding site residues of fusion enzymes

名称配体配体结合位点残基AnEglA6TCBI(β-1,4-D-葡聚糖)HIS100、TRP127、HIS145、ASP149、GLY150、SER151AnEg-CBM91HF6100(纤维三糖)TRP9、TYR143、HIS145、SER151、TRP210AnEg-CBM10CBI(β-1,4-D-葡聚糖)ASN99、TRP127、HIS145、ASP149、GLY150、SER151
所预测结果中,AnEglA6T与AnEg-CBM10的预测的最适配体均为β-1,4-D-葡聚糖,与配体结合的氨基酸残基均为6个,其中仅有一个残基不同,由His100变为了ASN99。AnEg-CBM9的预测的最适配体为纤维三糖。从结构分析结果看,两种融合体酶的催化活性中心均与AnEglA6T有差异,这在一定程度上解释了融合体酶底物范围发生变化的原因。AnEg-CBM9预测的活性中心变化更大,显示其底物特异性发生变化的可能性也较大,这与表1所列底物特异性分析结果基本相符,AnEg-CBM9对微晶纤维素、茯苓多糖等新底物的活性显著高于AnEg-CBM10。上述预测结果与实验结果也有一定的差异,例如两种融合体酶预测的所有配体中均没有β-1,3-葡聚糖。结构预测与实验结果之间出现差异的一个重要原因可能是酵母表达的酶有糖基化修饰但规律尚不完全清楚,因此分析时还难以考虑这方面的影响。
3 讨论
本文将不同来源的纤维素、木聚糖等多糖水解酶类的碳水化合物结合结构域更换黑曲霉耐热型葡聚糖酶AnEglA6T本身的纤维素结合结构域,筛选到两种保留了较高葡聚糖水解活性的融合体酶,AnEg-CBM9和AnEg-CBM10。检测结果表明这两种酶都具有明显的水解微晶纤维素的能力,并且对滤纸的水解能力也有大幅度提高,可见CBM结构域对葡聚糖酶能否水解结晶型纤维素具有关键性的作用。本文最有价值的研究结果是发现AnEglA6的CBD被来源于荧光假单胞菌的木聚糖酶的CBM10取代后得到的融合体酶AnEg-CBM10的热稳定性相较于AnEglA6T有显著的提高。AnEg-CBM10在85 ℃下半衰期达到40 min左右,而AnEglA6T在同样条件下半衰期不足5 min,同一条基因在乳酸克鲁维酵母中的表达产物AnEglA6K在85 ℃下半衰期大约为15 min[5]。本文构建AnEg-CBM9的CBM9来源于极端嗜热菌C.kristjanssonii,但获得的融合体酶的耐热性并没有提高,这可能是由于CBM9的分子过大。有研究发现,较大的CBM在改善融合体的热稳定性方面并不具有优势[19]。较大的 CBM 内部通常包含多个较小的串行子模块,这些子模块具有不同的功能[20],后续研究可以考虑分别选取CBM9的部分模块来构建融合体,进一步探索获得高稳定性葡聚糖酶的方法。与之相反,CBM10来源于假单胞菌,本身并不具有优越的耐热性,但获得的融合体AnEg-CBM10的热稳定性却有显著的提高,这也在一定程度上说明较小的CBM可能更有利于耐热酶的构建。通过添加或更换CBM提高酶的耐热性的研究已有报道,THONGEKKAEW等[21]将来自于里氏木霉的纤维素酶的CBD融合到隐球酵母的脂肪酶的羧基端,融合酶在80 ℃保温2 h还具有80%的活性,而野生酶在80 ℃下保温30 min就几乎完全失活。王春娟等[22]将来自海栖热袍菌的CBM27与宇佐美曲霉来源的β-甘露聚糖酶(Au Man5A)进行融合,使后者的热稳定性提高了8 ℃。从文献报道的研究结果看,融合体酶的耐热性有时比两种供体来源的酶更加耐热,本文获得的AnEg-CBM10也具有这一特点,因此利用CBM探索构建耐热型融合体酶时不一定要局限于使用耐热酶来源的CBM。
4 结论
本文将真菌高耐热葡聚糖酶AnEglA6催化结构域与荧光假单胞菌P.fluorescens的纤维素结合结构域CBM10融合获得的融合体酶AnEg-CBM10在85 ℃下酶活力半衰期达到40 min,能短时耐受90 ℃高温,并且对结晶型纤维素也有一定的水解能力,与AnEglA6相比,融合体酶在禽类饲料加工中具有更大的应用潜力。
[1] 夏许寒, 朱成林, 李诚.β-1,3-1,4-葡聚糖酶研究进展[J].食品科学, 2016, 37(19):289-295. XIA X H, ZHU C L, LI C.Advances in research on β-1,3-1,4-glucanase [J].Food Science, 2016, 37(19):289-295.
[2] BOYCE A, WALSH G.Production, purification and application-relevant characterisation of an endo-1, 3(4)-β-glucanase from Rhizomucor miehei[J].Applied Microbiology and Biotechnology, 2007, 76(4):835-841.
[3] 谭会泽, 冯定远.饲料中的β-葡聚糖和β-葡聚糖酶的应用[J].畜禽业, 2005(3):18-21. TAN H Z, FENG D Y.Application of β-glucan and β-glucanase in feed[J].Livestock and Poultry Industry, 2005(3):18-21.
[4] 张亮, 杨在宾, 杨维仁, 等.不同制粒温度对颗粒饲料制粒加工工段水分变化的影响[J].饲料研究, 2013(9):85-87. ZHANG L, YANG Z B, YANG W R, et al.Effect of different granulating temperature on moisture change in granulating processing section of pellet feed[J].Feed Research, 2013(9):85-87.
[5] 韩冰, 王施岚, 德青美朵, 等.一种黑曲霉高耐热β-葡聚糖酶基因的克隆、表达及重组酶性质分析[J].食品与发酵工业, 2018, 44(11):55-62. HAN B, WANG S L, DE Q M D, et al.Gene cloning, expression and characterization analysis of a highly thermal stable β-glucanase gene from Aspergillus niger[J].Food and Fermentation Industries, 2018, 44(11):55-62.
[6] 王施岚, 徐楚涵, 姚雁, 等.黑曲霉β-葡聚糖酶基因eglA6在乳酸克鲁维酵母中的表达及重组酶性质[J].食品与生物技术学报, 2021, 40(12):65-74. WANG S L, XU C H, YAO Y, et al.Expression of β-glucanase gene eglA6 of Aspergillus niger in Kluyveromyces lactis and characterization of recombinant enzyme[J].Journal of Food Science and Biotechnology, 2021, 40(12):65-74.
[7] AÏSSA K, NOVY V, NIELSEN F, et al.Use of carbohydrate binding modules to elucidate the relationship between fibrillation, hydrolyzability, and accessibility of cellulosic substrates[J].ACS Sustainable Chemistry &Engineering, 2019, 7(1):1113-1119.
[8] JAGADEESWARAN G, GAINEY L, MORT A J.An AA9-LPMO containing a CBM1 domain in Aspergillus nidulans is active on cellulose and cleaves cello-oligosaccharides[J].AMB Express, 2018, 8(1):1-13.
[9] TOMME P, WARREN R A J, MILLER R C Jr, et al.Cellulose-Binding Domains:Classification and Properties[M].ACS Symposium Series.Washington, DC:American Chemical Society, 1996:142-163. [10] OLIVEIRA C, CARVALHO V, DOMINGUES L, et al.Recombinant CBM-fusion technology:Applications overview[J].Biotechnology Advances, 2015, 33(3-4):358-369.
[11] 刘欢, 朱安南, 孟勇, 等.纤维素酶在畜禽生产中的研究进展[J].饲料研究, 2022, 45(16):148-153. LIU H, ZHU A N, MENG Y, et al.Research progress of cellulase on livestock and poultry production[J].Feed Research, 2022, 45(16):148-153.
[12] 谌恩华, 吴华伟, 李相前.结晶纤维素的降解[J].草业科学, 2016, 33(11):2367-2374. SHEN E H, WU H W, LI X Q.Advance in research on degradation of crystalline cellulose[J] Pratacultural Science, 2016, 33(11):2367-2374.
[13] KRSKA D, MAZURKEWICH S, BROWN H A, et al.Structural and functional analysis of a multimodular hyperthermostable xylanase-glucuronoyl esterase from Caldicellulosiruptor kristjansonii[J].Biochemistry, 2021, 60(27):2206-2220.
[14] RAGHOTHAMA S, SIMPSON P J, SZAB L, et al.Solution structure of the CBM10 cellulose binding module from Pseudomonas Xylanase A[J].Biochemistry, 2000, 39(5):978-984.
L, et al.Solution structure of the CBM10 cellulose binding module from Pseudomonas Xylanase A[J].Biochemistry, 2000, 39(5):978-984.
[15] 王兵波, 沈微, 钱灵紫, 等.一种密码子优化的酸性普鲁兰酶基因在巴斯德毕赤酵母中的高效表达[J].食品与发酵工业, 2016, 42(7):9-15. WANG B B, SHEN W, QIAN L Z, et al.High expression of a codon-optimized acid-resistant pullulanase-enconding gene in Pichia pastoris[J].Food and Fermentation Industries, 2016, 42(7):9-15.
[16] 祝冬君, 张锦雯, 姚雁, 等.枯草芽孢杆菌普鲁兰酶基因的分泌表达及其在粉丝制作中的应用[J].食品与生物技术学报, 2022, 41(2):82-93. ZHU D J, ZHANG J W, YAO Y, et al.Secretory expression of pullulanase gene from Bacillus subtilis and its application to vermicelli production[J].Journal of Food Science and Biotechnology, 2022, 41(2):82-93.
[17] 赵玉萍, 杨娟.四种纤维素酶酶活测定方法的比较[J].食品研究与开发, 2006, 27(3):116-118. ZHAO Y P, YANG J.Comparison of four cellulase activity measurement methods [J].Food Research and Development, 2006, 27(3):116-118.
[18] 李恒, 唐双焱.碳水化合物结合结构域研究进展[J].微生物学报, 2017, 57(8):1160-1167. LI H, TANG S Y.Carbohydrate-binding modules:Assisted polysaccharide recognition[J].Acta Microbiologica Sinica, 2017, 57(8):1160-1167.
[19] LIU L W, ZENG L Y, WANG S Y, et al.Activity and thermostability increase of xylanase following transplantation with modules sub-divided from hyper-thermophilic CBM9_1-2[J].Process Biochemistry, 2012, 47(5):853-857.
[20] LIU L W, CHENG J, CHEN H G, et al.Directed evolution of a mesophilic fungal xylanase by fusion of a thermophilic bacterial carbohydrate-binding module[J].Process Biochemistry, 2011, 46(1):395-398.
[21] THONGEKKAEW J, IKEDA H, IEFUJI H.Increases thermal stability and cellulose-binding capacity of Cryptococcus sp.S-2 lipase by fusion of cellulose binding domain derived from Trichoderma reesei[J].Biochemical and Biophysical Research Communications, 2012, 420(1):183-187.
[22] 王春娟, 李剑芳, 唐诗涵, 等.融合β-甘露聚糖酶基因的构建、表达及酶学性质的分析[J].食品与生物技术学报, 2016, 35(11):1135-1141. WANG C J, LI J F, TANG S H, et al.Expression and enzymatic properties analysis of a fusion β-mannanase[J].Journal of Food Science and Biotechnology, 2016, 35(11):1135-1141.