酯酶属于α/β水解酶超家族,通常催化酯键的裂解和形成,包括酯化和酯交换等反应,一般情况下优先水解短链酯,在结构上具有α/β水解酶典型的催化三联体结构和相对保守的催化域[1-3]。1999年,ARPIGNY等将细菌源酯酶分类成8个家族[4]。其中,酯酶的第Ⅳ家族(HSL家族)有典型的帽结构域[5],通常由4~5个α螺旋组成,覆盖活性位点[6],并且其N末端部分(帽结构域的一部分)可能与底物特异性和催化效率的调节有关[7]。
脂解酶作为一种工业生物催化剂应用广泛[8-9],工业上对高酶活酯酶的需求也日益增大,因此对酯酶的研究有巨大的价值。有的研究者通过直接分离产酯酶的菌株,在菌种中分离所需的酯酶[10],再去扩展被表征的高活性酯酶[11],但大多数产酯酶的菌株及其酶会由于培养条件受限而难以被发现[12]。于是,有的研究者直接基于活性去筛选酯酶[13]。现在,利用宏基因组序列筛选或功能筛选新型酯酶成为了一种趋势[14],KANG等[15]便在宏基因组库中分离得到1种新型热稳定酯酶。
本试验在以三丁酸甘油酯为指示底物的琼脂平板上基于活性功能筛选到了1个高活性的阳性克隆,构建亚克隆测序验证后,对其进行了异源表达和酶学性质的表征,展现了其在工业应用方面的价值。
1 材料与方法
1.1 材料
1.1.1 材料采集与处理
本实验室已构建的珠穆朗玛峰土壤宏基因组文库[16]:2014年2月取样于珠穆朗玛峰(北纬28.21°,东经86.56°,海拔高度4 276 m)。文库宿主:Escherichia coli EPI100;文库载体:Cosmid pWEB-TNC。
1.1.2 菌株、质粒及培养基材料等
EPI100-T1R、Cosmid pWEB-TNC、E.coli BL21(DE3)均由本实验室保藏。LB培养基(g/L):酵母提取物5,胰蛋白胨10,NaCl 10。三丁酸甘油酯筛选培养基:12.5 μg/mL氯霉素、100 μg/mL氨苄青霉素、体积分数为1%甘油三丁酸酯,体积分数为1%阿拉伯树胶的LB固体培养基。
1.2 酯酶的阳性克隆的筛选
在三丁酸甘油酯筛选培养基平板上铺约包含400~500个克隆的文库菌液,37 ℃培养1周,挑出具有透明圈的阳性克隆后,水平转移铺板验证。
1.3 亚克隆的构建
提取阳性克隆质粒后,用限制性内切酶Sau 3AI部分酶切,胶回收,去磷酸化后,将酶切基因片段连接到经完全酶切、去磷酸化后的pWEB-TNC载体中,连接产物化转至E.coli EPI100-T1R,涂布在三丁酸甘油酯筛选培养基上,37 ℃培养1周,挑出阳性亚克隆测序验证。
1.4 酯酶基因的生物信息学分析
提取阳性亚克隆质粒并进行酶切检验后送公司进行双末端测序,拼接后的结果使用ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)进行开放阅读框分析。序列相似性使用BLASTp(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行分析,通过Clustal W(https://www.ebi.ac.uk/Tools/msa/clustalo/)和在线工具ESPript 3.0(http://espript.ibcp.fr/ESPript/ESPript/) 进行多序列比对及同源序列分析。使用MEGA6.0 基于邻接法构建系统发育树,自展值设为1 000,系统发育树的美化采用在线工具 ITOL(https://itol.embl.de/)。使用ExPASy(https://web.expasy.org/protparam/)进行蛋白理化性质分析预测蛋白的理论分子质量和等电点,使用SignalP 4.0 (http://www.cbs.dtu.dk/services/SignalP-4.0/)预测蛋白是否含有信号肽序列,使用TMHMMOL/L(https://dtu.biolib.com/DeepTMHMMOL/L)预测是否存在跨膜区域,使用PDB (http://www.ebi.ac.uk/thornton-srv/databases/pdbsum/Generate.html)寻找相似三维结构,使用SWISS MODEL(https://swissmodel.expasy.org/)进行同源蛋白三维结构预测和模型构建,使用SAVES v5.0(https://saves.mbi.ucla.edu/)对模型进行可信度评价。图像处理使用软件AdobeIllustrator。数据处理使用软件EXCEL。采用AutoDock Vina 1.1.2软件进行分子对接,使用PyMol 2.5.2 对对接结果进行可视化分析。
1.5 异源表达和纯化
设计目的基因上带有酶切位点的上下游引物(AesZF899-R:5′-ATATCTCGAGCGCCTTGGACTTCA-3′, AesZF899-F:5′-ATATGAATTCATGCCGATCGTAC-TCGA-3′),通过PCR(条件:95 ℃ 5 min;95 ℃ 45 s;62 ℃ 45 s;72 ℃ 3 min;72 ℃ 10 min)扩增得到目的片段序列,然后将其与带有相同酶切位点 Nco I和 Xho I 的表达载体相连接,建立pET-28a(+)-AesZF899重组质粒。通过序列分析和三丁酸甘油酯底物验证,将阳性重组质粒分别转化到表达宿主E.coli BL21(DE3)中进行表达。挑取已验证正确的重组克隆子E.coli BL21(DE3)-AesZF899于LB液体培养基中,37 ℃,220 r/min振荡培养至OD600值为0.6~0.8时,添加异丙基硫代半乳糖苷(isopropyl β-D-thiogalactoside ,IPTG)使其终浓度为0.5 mmol/L,18 ℃,200 r/min,诱导表达20 h后离心弃上清液,用裂解液重悬菌体后超声波破碎30 min(6 mmol/L超声探头,1 s超声,间隔2 s),高速离心(12 000 r/min,20 min)取上清液过偶联镍离子的亲和层析纯化。将纯化后的蛋白样品与5×蛋白上样缓冲液按相应比例混合,98 ℃反应10 min使蛋白变性,选择体积分数为12%的聚丙烯酰胺分离凝胶和体积分数为5%浓缩胶来检测纯化效果,采用考马斯亮蓝法染色脱色。
1.6 酯酶活力的测定
采用对硝基苯酚(p-nitrophenol,p-NP)比色法测定酯酶活力,以1 mmol/L的p-NP为标准液,稀释不同浓度,用酶标仪读取405 nm处的OD值。以吸光度值为横坐标,p-NP 浓度为纵坐标,绘制标准曲线。在40 ℃、pH 9.0条件下,每分钟降解底物对硝基苯酯生成1 μmol对硝基苯酚所需酶量为一个酶活力单位(U)。反应混合物(500 μL)包含最终浓度为50 mmol/L的pH值为8.0的Tris-HCl和0.2 mmol/L对硝基苯酯底物,除非另有说明,还包括纯化的AesZF899,所有反应都平行测定3次,酶反应时间5 min。
1.7 底物特异性的测定
以4种对硝基苯酚酯:对硝基苯乙酸酯(pNPC2)、对硝基苯丁酸酯(pNPC4)、对硝基苯己酸酯(pNPC6)、对硝基苯辛酸酯(pNPC8)为底物,评估纯化的AesZF899的底物特异性。
1.8 酯酶的酶学性质的测定
温度对AesZF899活性的影响:在pH 8.0条件下,设置温度梯度20~100 ℃,按照酯酶酶活力测定方法,依次计算各个温度酶活力大小,从而得到该酶作用的最适温度。将最高酶活力的实验组的酶活力记为100%。
pH对AesZF899活性的影响:在温度40 ℃条件下,设置pH梯度(甘氨酸缓冲液pH 9.0、10.0,Tris缓冲液pH 8.0、9.0,磷酸盐缓冲液pH 6.0、7.0、8.0,柠檬酸缓冲液pH 3.0、4.0、5.0、6.0),按照酯酶酶活力测定方法,依次计算各个pH下酶活力大小,从而得到该酶作用的最适pH。将最高酶活力的实验组的酶活力记为100%。
温度对AesZF899稳定性的影响:在pH 9.0条件下,将酶液分别置于30、40、50 ℃保温,期间每隔一段时间测定其在最适条件下(40 ℃、pH 9.0)残余酶活力大小。将未经保温操作的对照组在最适条件下(40 ℃、pH 9.0)测得的酶活力记为100%。
pH对AesZF899稳定性的影响:在pH 9.0条件下,将酶液分别置于不同缓冲液中(柠檬酸缓冲液pH 3.0、4.0、5.0、6.0,磷酸盐缓冲液pH 6.0、7.0、8.0,Tris缓冲液pH 8.0、9.0,甘氨酸缓冲液pH 9.0、10.0)4 ℃ 保温2 h,期间每隔一段时间测定其在最适条件下(40 ℃、pH 9.0)残余酶活力大小。将未经保温操作的对照组在最适条件下(40 ℃、pH 9.0)测得的酶活力记为100%。
金属离子和有机溶剂对 AesZF899 活性的影响:在最适条件下(40 ℃、pH 9.0),在缓冲液中加入10 mmol/L的不同的金属离子(Cu2+、K+、Na+、Ni+、Mg2+、Fe3+、Mn2+、Li+)及体积分数为1%和10%的化学溶剂[SDS、二甲基亚砜(dimethyl sulfoxide, DMSO) 、甲醇、乙醇、丙醇、正丁醇、异戊醇],测定酯酶活力。将添加有机溶剂但未添加酶液的对照组作为空白,将添加酶液但未添加有机溶剂的对照组在最适条件下(40 ℃、pH 9.0)测得的酶活力记为100%。
2 结果与分析
2.1 酯酶的克隆、表达、序列分析和系统发育
本研究基于宏基因组功能筛选了约200万个文库菌克隆后获得了多个周围有透明圈的阳性克隆,选择一个透明圈半径最大的酯酶阳性克隆进行后续实验。对该阳性克隆进行复筛,确认该克隆具有酯酶活性后进行亚克隆的构建,挑选具有酯酶活性的亚克隆,酶切验证酯酶阳性亚克隆构建成功,并对该阳性亚克隆质粒测序。
亚克隆测序结果经ORF Finder在线工具分析后,定位一个长度为945 bp的开放阅读框,命名为AesZF899,可编码314个氨基酸,预测蛋白大小34 kDa,等电点是5.49,显示其不含信号肽,不存在跨膜区域,提示AesZF899为胞内酶。NCBI在线工具BLASTp中的UniProtKB/Swiss-prot (swissprot)数据库序列比对结果表明,与来自Afipia sp. P52-10中编码313个氨基酸的α/β水解酶(GenBank登录号为WP_034470467.1)一致性达76.43%,但该酶的酶学性质尚未表征,Graphic Summol/Lary中提示AesZF899可能编码一种乙酰基酯酶。将AesZF899提交到GenBank数据库中,登录号为PRJNA932869。
利用MEGA X构建了包含Arpigny和Jaeger的分类中8个家族的代表序列邻接系统发生树(图1-a),以对AesZF899进行相关的分类,显示AesZF899与酯酶第四家族密切相关。
对酯酶AesZF899及同家族的triacylglycerol lipase [Moraxella sp.](CAA37862)、esterase [Renibacterium salmoninarum ATCC 33209](ABY25125)和Arylesterase(B5BLW5.1)进行多序列同源比对,结果如图1-b所示,在AesZF899的氨基酸序列中存在酯酶第四家族(HSL家族)酯酶的保守基序HGGG和GDSAG,比对分析推测活性位点丝氨酸(Ser162)、保守天冬氨酸(Asp242)和组氨酸(His88)可能组成了催化三联体。
2.2 AesZF899的结构建模与分子对接
二级结构分析结果表明AesZF899 有10个α螺旋、8个β折叠、3个无规则卷曲。在SWISS-MODEL上进行同源建模[17-21],根据Est8(PDB:4YPV,序列覆盖度43.87%)的结构建模得到AesZF899的结构,结果如图2 所示。GMQE的可信度范围为0~1,值越大表明质量越好;QMEAN4评估待测蛋白与模板蛋白的匹配度,值越大表示匹配越好,该结构的GMQE值是0.83,QMEAN4值是0.79。ERRAT分析不同原子类型之间非键合相互作用的统计数据,结果表明,AesZF899有91.77%的得分。VERIFY 3D用于确定原子模型(3-dimension,3D)的准确性,其中的结果是通过比较已经被晶体学或核磁共振(nuclear magnetic resonance,NMR)解析的结构。结果表明,AesZF899有96.15%的氨基酸残基得分。以上结果显示,同源建模得到的结构合理,适合于下一步的分子对接。此外,还可以看到在AesZF899的三维结构中存在α/β水解酶折叠子,即多个α螺旋和β折叠组成,并且可以看到上方存在一个CAP结构域。
分子对接的对硝基苯乙酸酯(Cas号:830-03-5)的3D结构从PUBCHEM数据库下载获得,并在MMOL/LFF94力场下进行能量最小化。在对接开始之前,使用PyMol 2.5.22对受体蛋白进行处理,包括去除水分子、盐离子以及小分子。结果如图3所示,从右边的相互作用细节图中看到对硝基苯乙酸酯与AesZF899上的Leu-210、Asp-208发生氢键作用,和Lys-23发生阳离子pi共轭作用;此外,还可以观察到对硝基苯乙酸酯和蛋白的Phe-16、Leu-210 发生疏水作用。氢键作用为最强的非共价作用之一,在此表明发生氢键作用的Leu-210、Asp-208为对结合起重要贡献的氨基酸。结合亲和力为负数表示存在结合的可能,通常数值小于-6 kcal/mol被认为结合可能性较大。在该复合物中,对接软件给出对硝基苯乙酸酯与AesZF899结合亲和力评分为-6.0 kcal/mol,意味着对硝基苯乙酸酯与AesZF899蛋白具备结合可能性。
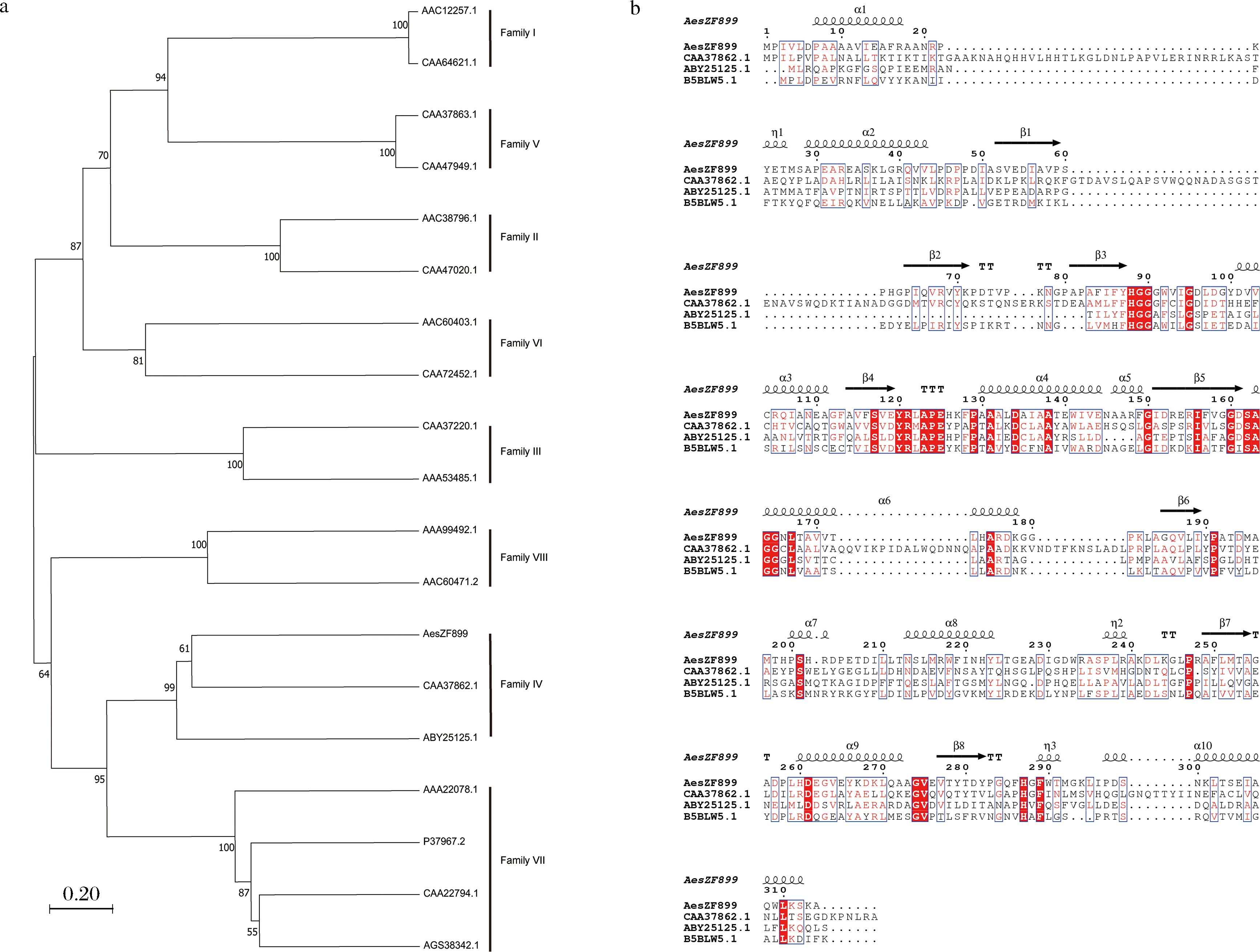
a-AesZF899的系统发育树;b-AesZF899的多重序列比对
图1 AesZF899的生物信息学分析
Fig.1 Bioinformatics analysis of AesZF899
注:基于最大似然法建立的系统发育树,以GenBank中获得全基因组序列登录号建立;NCBI登录号CAA37862、ABY25125和B5BLW5.1为 AesZF899同家族酯酶,相同或相似的残基用蓝框划出,α代表α螺旋、β代表β折叠、η代表不规则卷曲。
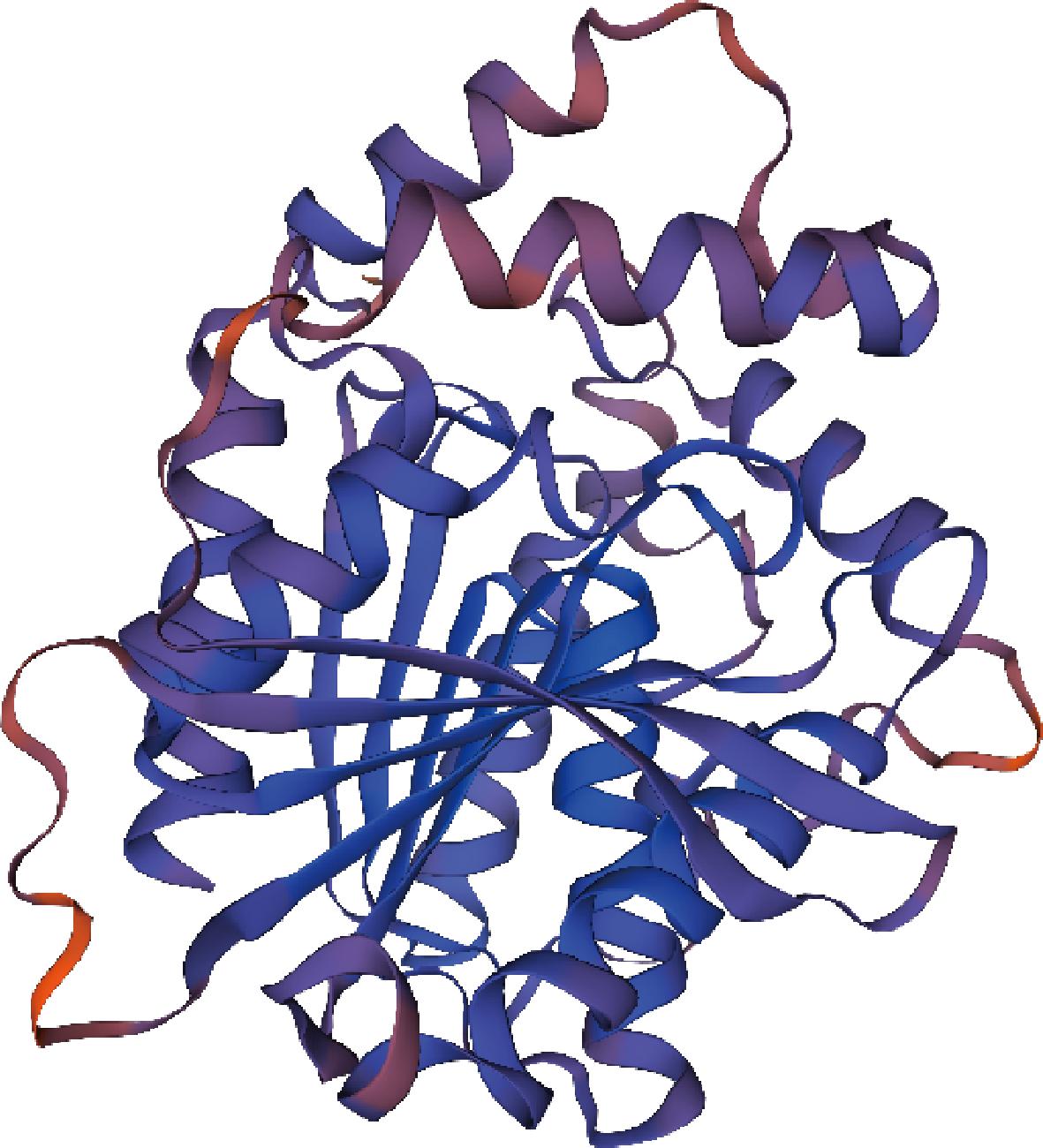
图2 AesZF899的三维结构
Fig.2 3D structure of AesZF899
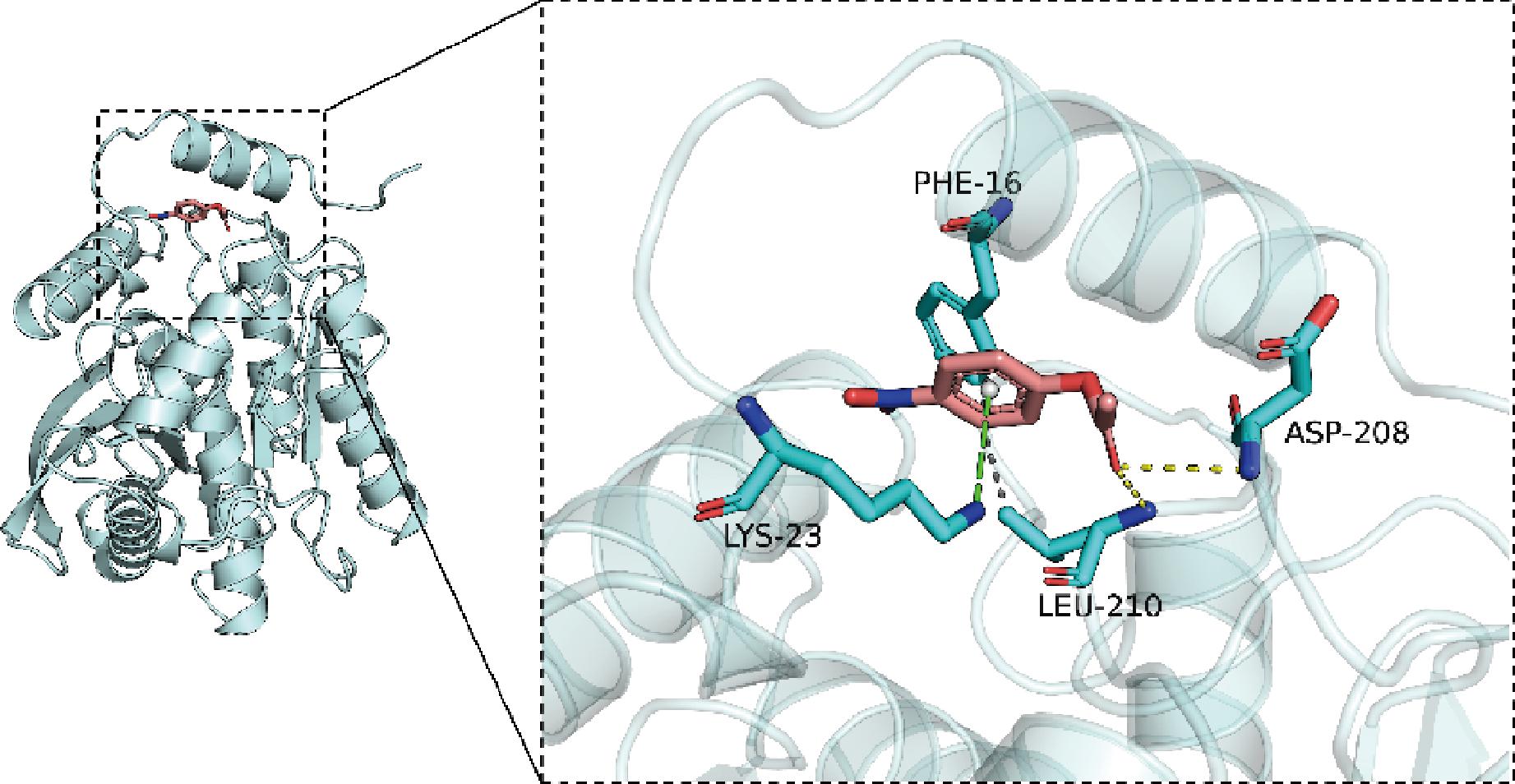
图3 AesZF899与对硝基苯乙酸酯的分子对接图
Fig.3 Molecular docking diagram of AesZF899 with p-nitrophenylacetate
注:右图是左图整体视图中的局部放大图,小分子是琥珀色, 发生相互作用的氨基酸是青色,疏水作用用灰色虚线, 氢键用黄色虚线表示,阳离子Π共轭用绿色虚线表示。
2.3 重组酯酶的表达和纯化
N端包含His-Tag的重组AesZF899在大肠杆菌BL21 (DE3)中成功表达,并经Ni+亲和层析纯化,SDS-PAGE结果如图4所示,AesZF899的大小与理论大小34 kDa相符。
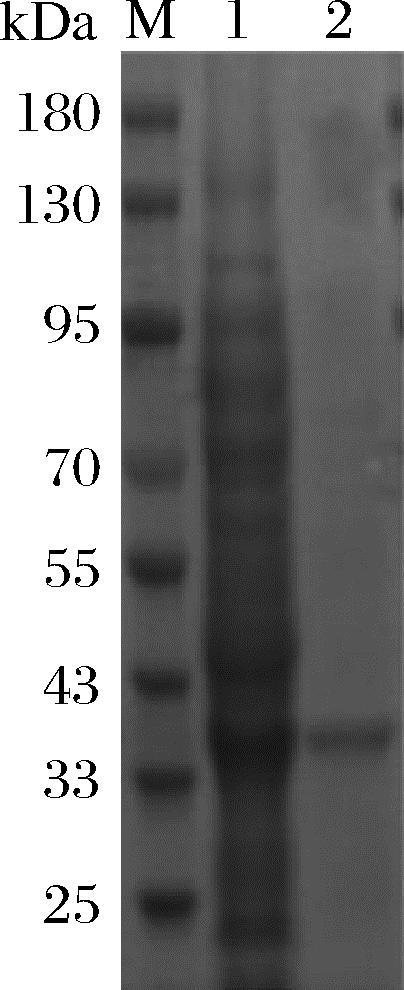
M-蛋白Marker;1-粗酶液;2-纯酶
图4 AesZF899的SDS-PAGE分析
Fig.4 SDS-PAGE analysis of AesZF899
2.4 酯酶的酶学特性分析
酶的最适底物如图5所示,在对对硝基苯乙酸酯(pNPC2)、对硝基苯丁酸酯(pNPC4)、对硝基苯己酸酯(pNPC6)和对硝基苯辛酸酯(pNPC8) 4种底物的底物特异性实验中,AesZF899对对硝基苯乙酸酯(pNPC2)的酶活性最佳,底物活性谱是酯酶活性的特征,即酯酶通常仅在短链酯上具有活性,因此,AesZF899是一个酯酶,而不是脂肪酶。
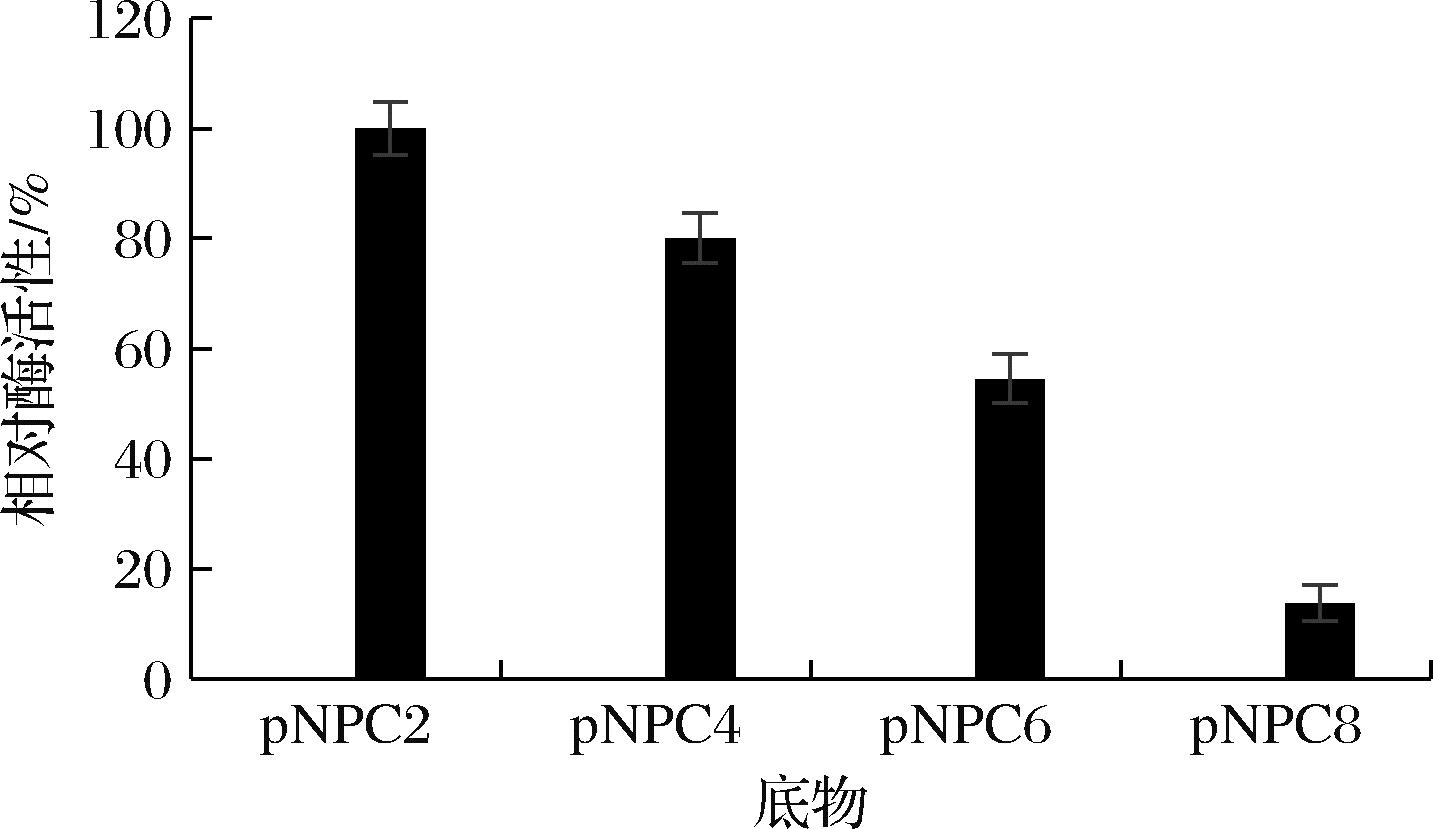
图5 底物特异性
Fig.5 Specificity of substrate
酶的最适温度如图6所示,酶活性在40 ℃时达到最大值,酶促反应理想温度在40~50 ℃,反应温度大于60 ℃后,相对酶活性不足20%,酶的作用温度区间较窄,反映了AesZF899是一个中温酶。
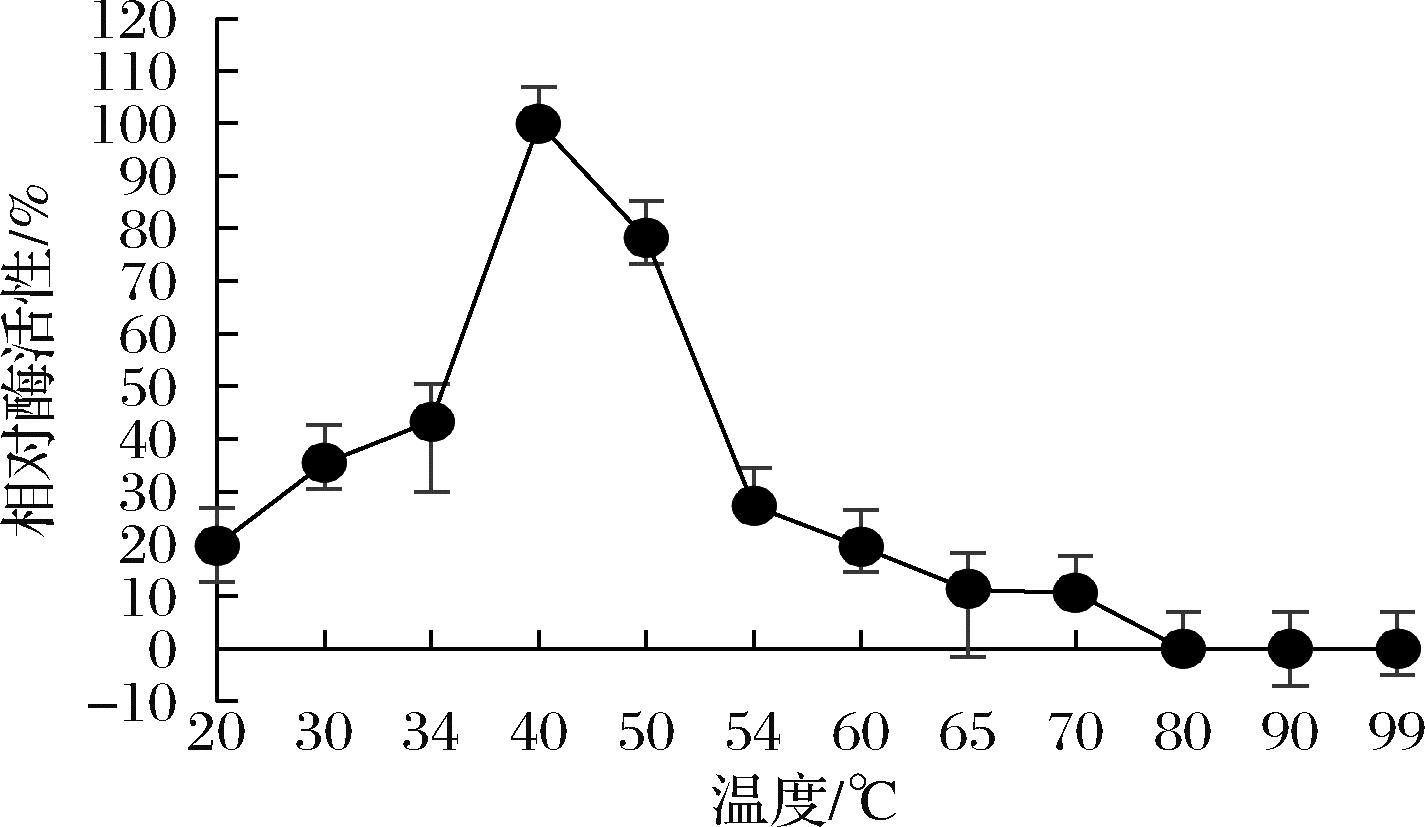
图6 温度对酶活性的影响
Fig.6 Effect of temperature on enzyme activity
酶的温度稳定性如图7所示,在30 ℃温育8 h后仍可保持初始活性,酯酶在温度35、40 ℃的半衰期分别是7、4 h,当温育温度大于50 ℃后,温育半小时后相对酶活性仅50%,温育1 h后酶活性几乎完全丧失,反映了AesZF899并不耐高温。
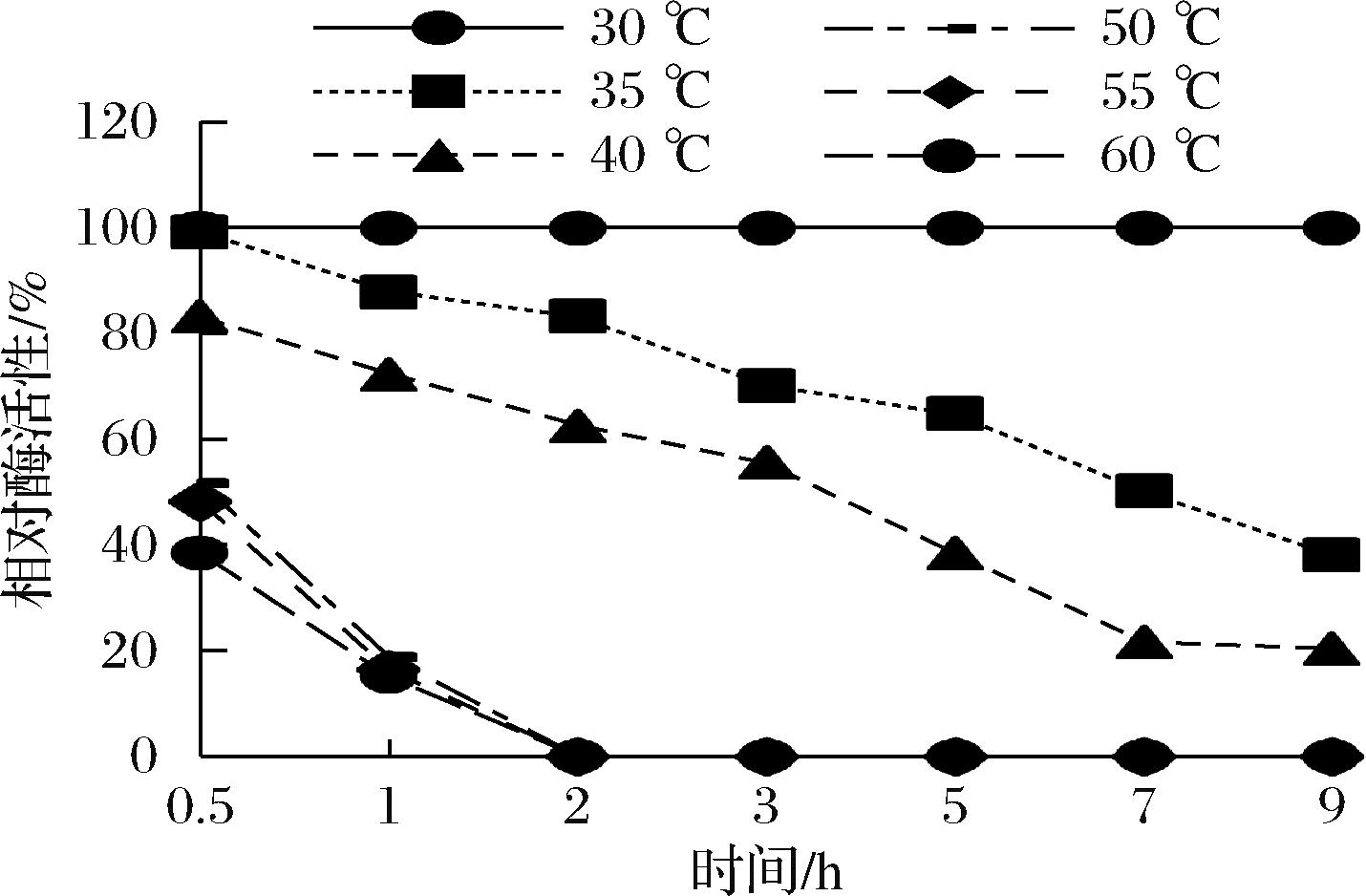
图7 时间对酶稳定性的影响
Fig.7 Effect of temperature on enzyme stability
酶的最适pH如图8所示,酶活性随着酶促反应的pH值的上升先升后降,pH值小于5.0时酯酶几乎无活性,pH值在9.0时酯酶的活性最大,反映了AesZF899是一个碱性酶。
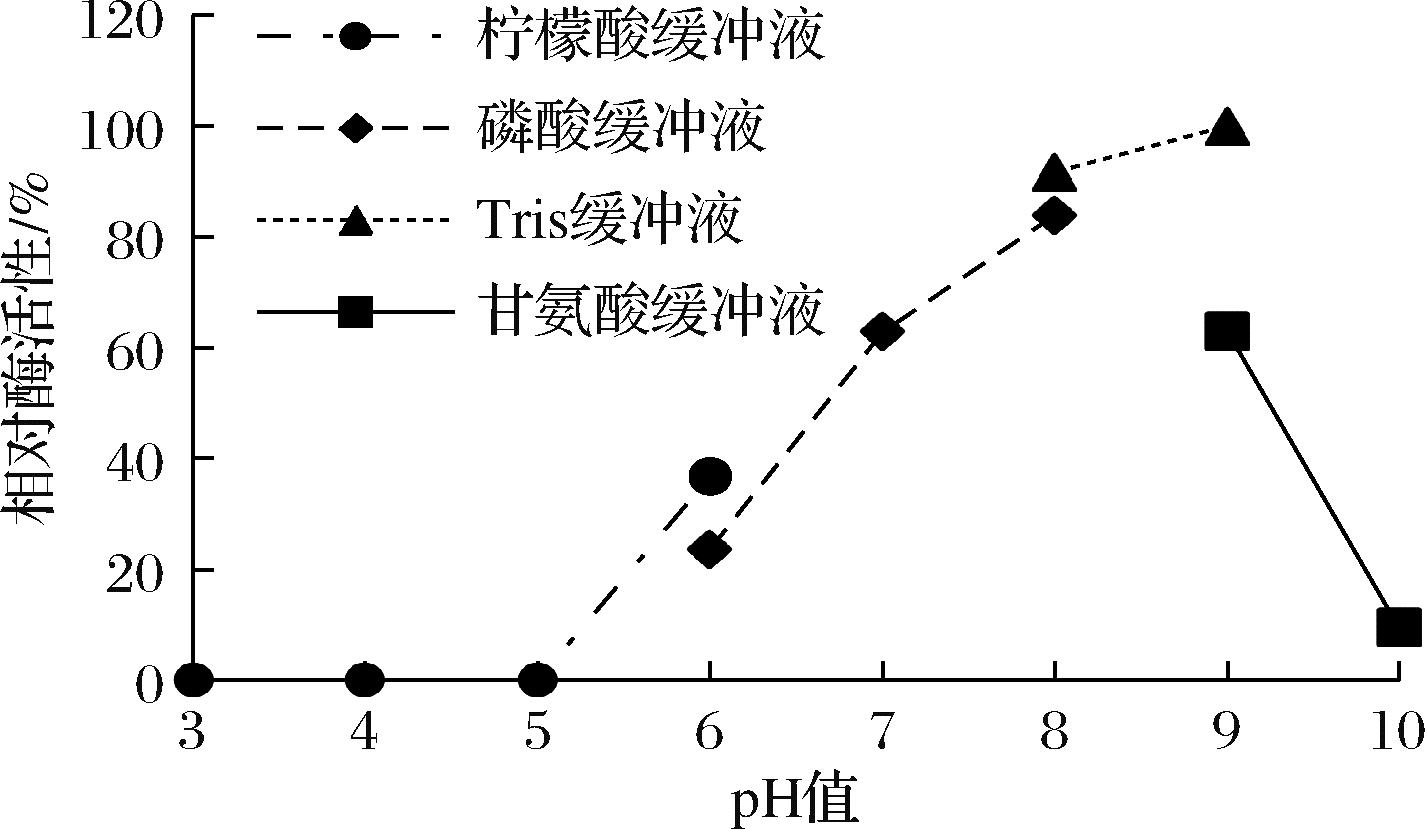
图8 pH 对酶活性的影响
Fig.8 Effect of pH on enzyme activity
pH值对酶稳定性的影响如图9所示,过酸或过碱的环境都会降低酶的稳定性,中度偏碱的环境更适合酶的稳定,酶在pH 9的情况下可维持65%的活性。
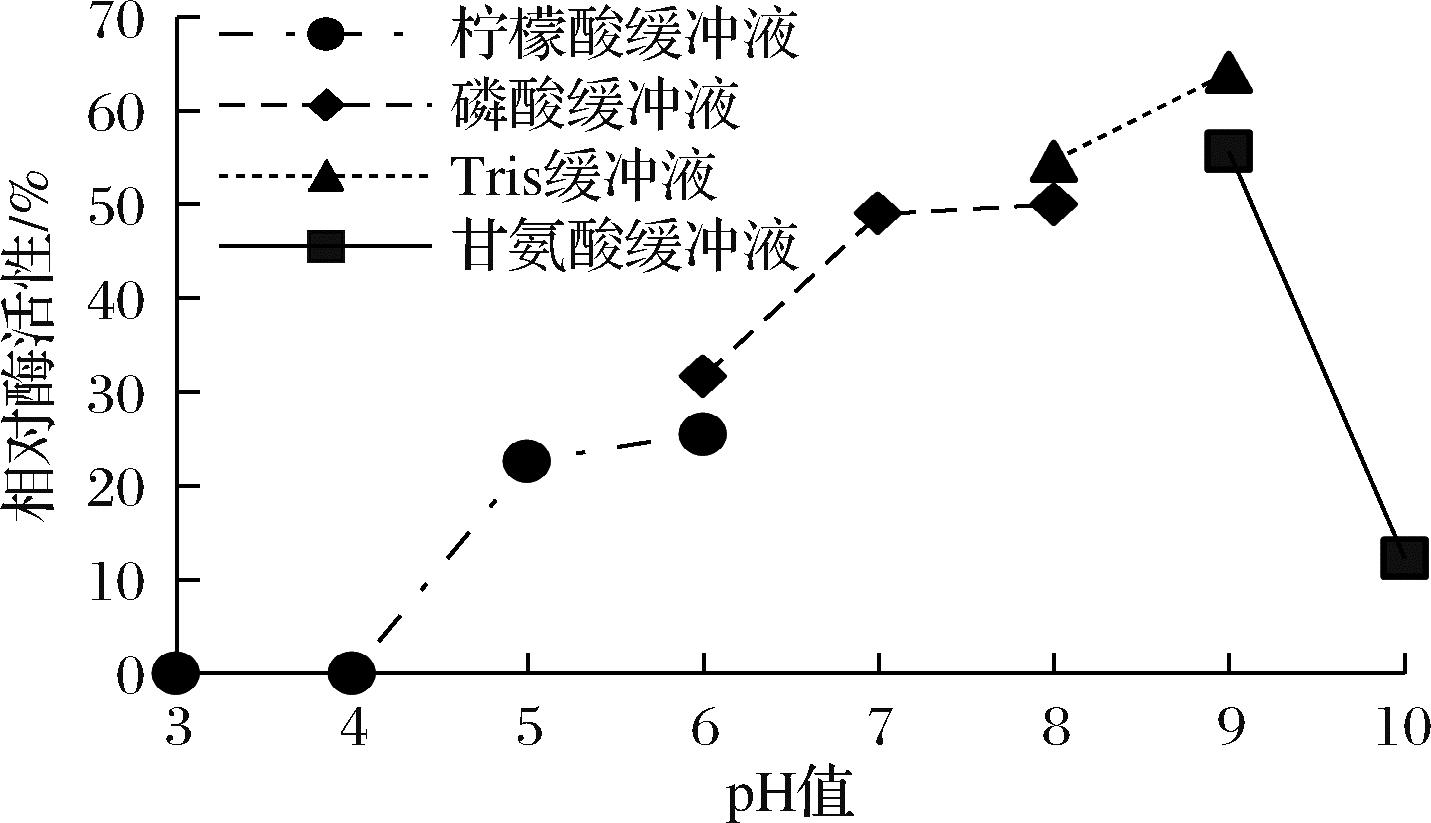
图9 pH 对酶稳定性的影响
Fig.9 Effect of pH on enzyme stability
金属离子对酶活性的影响如图10所示,Fe3+可以增强酶活性,Mn2+和Cu2+对AesZF899的酶活性抑制作用较弱,K+和Ca2+对AesZF899的酶活性抑制作用较强,Na+、Li+和Mg2+对AesZF899的酶活性抑制作用最强,反应体系中加入Na+、Li+和Mg2+可使AesZF899的相对酶活性降低到50%以下。
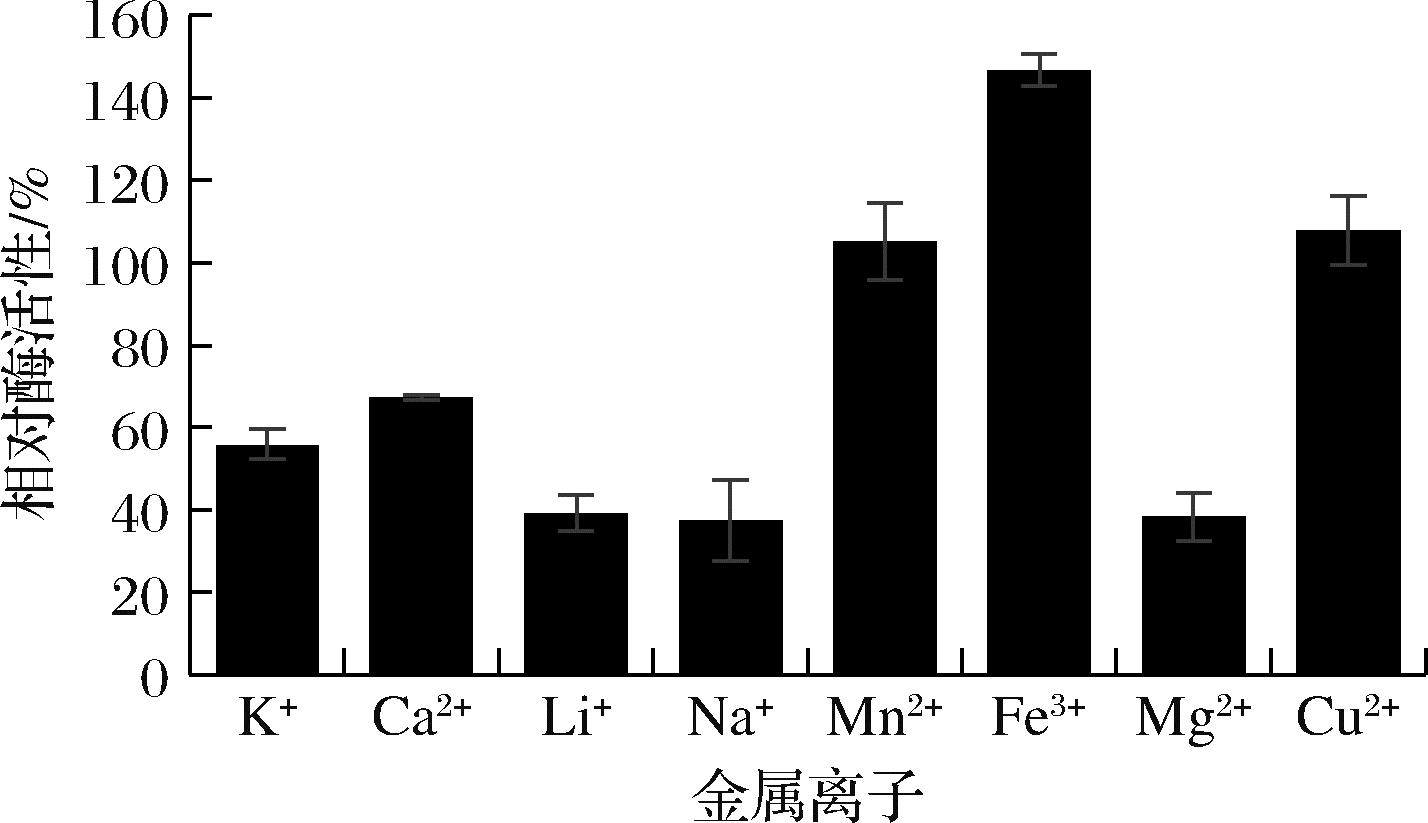
图10 金属离子对酶活性的影响
Fig.10 Effect of metal ions on enzyme activity
有机溶剂对酶活性的影响如图11所示,酶在10%甲醇和10%丙醇溶剂中活性明显降低,可能是由于有机溶剂分子阻碍底物和酶活性位点的接触[22];酶在1% DMSO中活性明显增强,可能是由于其使酶的柔性部位增加[23];酶在1%和10%的异戊醇中活性稍微增加,可能是由于二级结构的改变[24]。
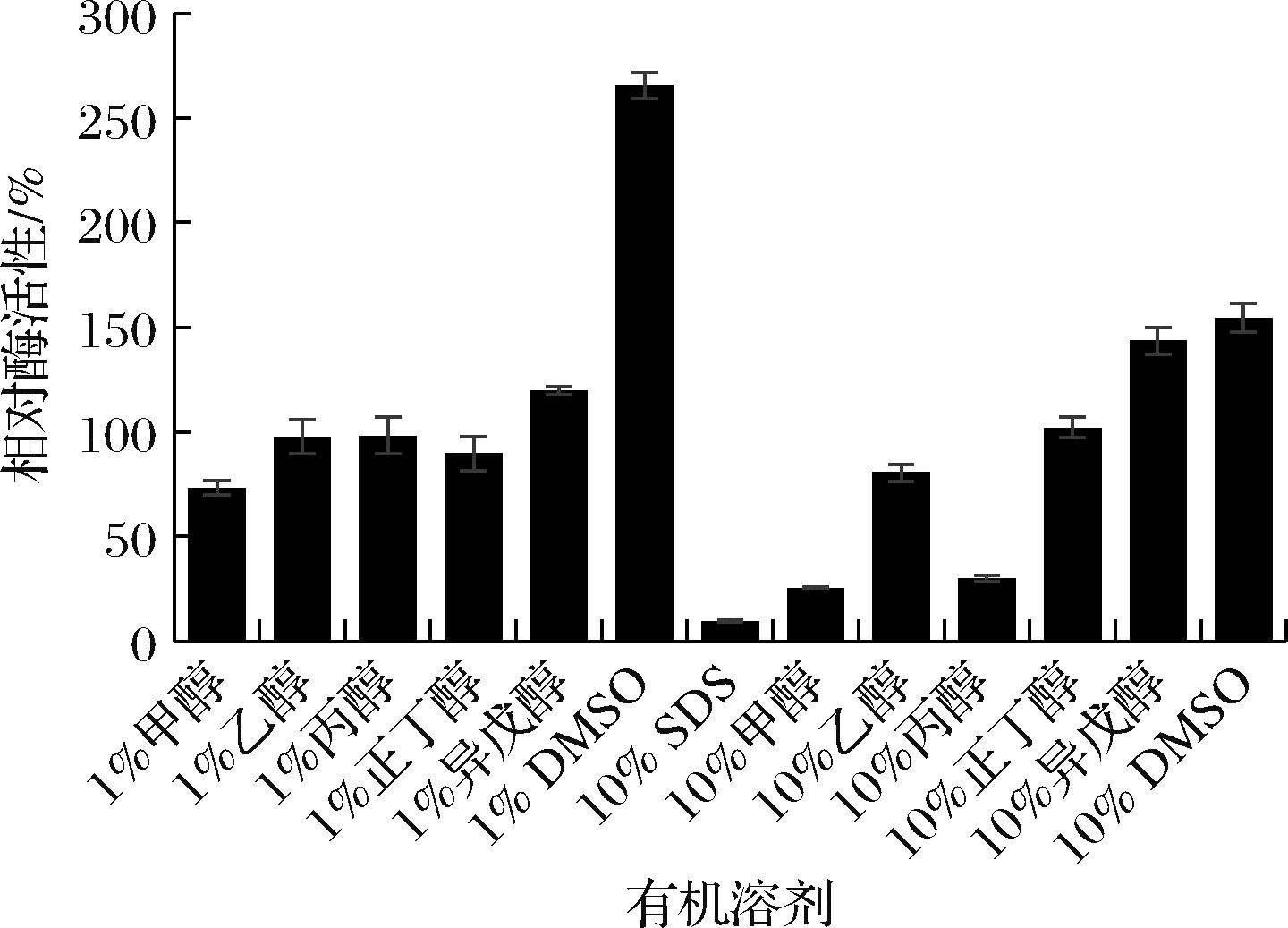
图11 有机溶剂对酶活性的影响
Fig.11 Effect of organic solvent on enzyme activity
在最佳条件下,即酶促反应温度40 ℃,pH值为9.0,底物是对硝基苯乙酸酯,反应体系不额外加金属离子,不加有机溶剂的情况下,酯酶AesZF899的比活力可高达(3 957±3) U/mg。
3 结果与讨论
本研究通过宏基因组手段从前期建立的珠穆朗玛峰土壤文库中,筛选了约200万个文库菌克隆后获得了多个周围有透明圈的阳性克隆,选择一个透明圈半径最大的酯酶阳性克隆,即酯酶活性最大的一个阳性克隆,构建正确的阳性亚克隆后在NCBI上比对序列信息,并利用基因工程技术实现了该基因在大肠杆菌中的异源表达,通过镍柱亲和层析纯化后得到纯度较高的纯酶,在脱盐柱上脱盐后进行酶学性质的测定。通过对进化树的分析,推测该酯酶是酯酶第四家族。选取进化树上与该酯酶相近的分支的序列进行多重序列比对,发现该酯酶的序列具有保守基序HGGG和GDSAG,符合酯酶第四家族GDSAG亚族的特点。采用了测定酯酶活性的通用底物对硝基苯酯测定该酶的底物特异性,该酶在底物特异性实验中偏好短链酯,最佳底物是对硝基苯乙酸酯,最佳pH值为9.0,偏碱性,最佳温度40 ℃。在最佳条件下的该酯酶的比活力可达3 957 U/mg。同时,Fe3+可以增强酶活性,适用于工业中铁容器材质。
通常采用在同等条件下释放的对硝基苯酚的含量去衡量酯酶的活性大小,这是因为酯酶水解酯键,产生游离酚重新激活显色基团或荧光基团,进一步通过分光光度计定量测定,从而反应水解能力的大小[25]。AesZF899的比活力可达3 957 U/mg,Sigma-Aldrich提供的商业酯酶的活性也仅为104~105 U/g,虽然商业酯酶的活力通常小于天然酯酶的活力,大于人工设计的酯酶活力[26]。在一些实验中为了获得具有较高催化活性的酶,会对其底物结合口袋的形成、相互作用和疏水性进行合理的定向突变[27]。在未来的研究中,可以对AesZF899进行突变获得更高活性的酯酶,或者可以通过突变底物分子周围重要的氨基酸从而改造酶的分子结构以提高酶的稳定性以及增强酶的选择性、改变酶的表面特性等。酯酶主要催化短链的脂肪酸酯及醇的反应,且生产成本低于脂肪酶,成为了化学合成中重要的催化剂[28]。有研究者从豌豆中分离得到酯酶,并根据底物特异性将其归类为羧酸酯酶,用于酶抑制法检测农药残留[29]。所以根据底物特异性可以将AesZF899也归类为酯酶,用于生物催化剂等工业领域。同时,AesZF899在30 ℃的稳定性很好,利于工业酶制剂的常温储存,保持高效的酶活性,减少工业酶制剂的贮存成本。
[1] JOCHENS H, HESSELER M, STIBA K, et al.Protein engineering of α/β-hydrolase fold enzymes[J].ChemBioChem, 2011, 12(10):1508-1517.
[2] OLLIS D L, CHEAH E, CYGLER M, et al.The α/β hydrolase fold[J].Protein Engineering, Design and Selection, 1992, 5(3):197-211.
[3] BOURNE Y, MARCHOT P.Hot spots for protein partnerships at the surface of cholinesterases and related α/β hydrolase fold proteins or domains:A structural perspective[J].Molecules, 2017, 23(1):35.
[4] ARPIGNY J L, JAEGER K E.Bacterial lipolytic enzymes:Classification and properties[J].The Biochemical Journal, 1999, 343(1):177-183.
[5] RAUWERDINK A, KAZLAUSKAS R J.How the same core catalytic machinery catalyzes 17 different reactions:The serine-histidine-aspartate catalytic triad of α/β-hydrolase fold enzymes[J].ACS Catalysis, 2015, 5(10):6153-6176.
[6] MANDRICH L, PEZZULLO M, ROSSI M, et al.SSoNΔ andSsoNΔlong:Two thermostable esterases from the same ORF in the archae on Sulfolobus solfataricus?[J].Archaea, 2006, 2(2):109-115.
[7] BEN CHEIKH R, BERTICAT C, BERTHOMIEU A, et al.Characterization of a novel high-activity esterase in Tunisian populations of the mosquito Culex pipiens[J].Journal of Economic Entomology, 2008, 101(2):484-491.
[8] JEGANNATHAN K R, NIELSEN P H.Environmental assessment of enzyme use in industrial production:A literature review[J].Journal of Cleaner Production, 2013, 42:228-240.
[9] KIM T D.Bacterial hormone-sensitive lipases (bHSLs):Emerging enzymes for biotechnological applications[J].Journal of Microbiology and Biotechnology, 2017, 27(11):1907-1915. [10] BERLEMONT R, SPEE O, DELSAUTE M, et al.Novel organic solvent-tolerant esterase isolated by metagenomics:Insights into the lipase/esterase classification[J].Revista Argentina De Microbiologia, 2013, 45(1):3-12.
[11] KONO Y, TOMITA T.A role of highly active carboxylesterase in an organophosphorus insecticide resistant strain of Culex pipiens molestus[J].Medical Entomology and Zoology, 1993, 44(1):15-21.
[12] 黄循柳, 黄仕杰, 郭丽琼, 等.宏基因组学研究进展[J].微生物学通报, 2009, 36(7):1058-1066. HUANG X L, HUANG S J, GUO L Q, et al.Advances of metagenomics[J].Microbiology, 2009, 36(7):1058-1066.
[13] SANTILLAN J Y, DETTORRE L A, LEWKOWICZ E S, et al.New and highly active microbial phosphotriesterase sources[J].FEMS Microbiology Letters, 2016, 363(24):276.
[14] 时玉萍. 基于功能筛选的酯酶基因的克隆及其酶学性质的研究[D].上海:上海海洋大学, 2013. SHI Y P.Gene cloning, expression, and characterization of two novel esterase:Screening based on functional method[D].Shanghai:Shanghai Ocean University, 2013.
[15] KANG C H, OH K H, LEE M H, et al.A novel family VII esterase with industrial potential from compost metagenomic library[J].Microbial Cell Factories, 2011, 10:41.
[16] WANG S C, GAO X, GAO Y J, et al.Tetracycline resistance genes identified from distinct soil environments in China by functional metagenomics[J].Frontiers in Microbiology, 2017, 8:1406.
[17] WATERHOUSE A, BERTONI M, BIENERT S, et al.SWISS-MODEL:Homology modelling of protein structures and complexes[J].Nucleic Acids Research, 2018, 46(W1):W296-W303.
[18] BIENERT S, WATERHOUSE A, DE BEER T A P, et al.The SWISS-MODEL Repository:New features and functionality[J].Nucleic Acids Research, 2017, 45(D1):D313-D319.
[19] GUEX N, PEITSCH M C, SCHWEDE T.Automated comparative protein structure modeling with SWISS-MODEL and Swiss-PdbViewer:A historical perspective[J].Electrophoresis, 2009, 30(Suppl 1):S162-S173.
[20] STUDER G, REMPFER C, WATERHOUSE A M, et al.QMEANDisCo:Distance constraints applied on model quality estimation[J].Bioinformatics, 2020, 36(6):1765-1771.
[21] BERTONI M, KIEFER F, BIASINI M, et al.Modeling protein quaternary structure of homo- and hetero-oligomers beyond binary interactions by homology[J].Scientific Reports, 2017, 7:10480.
[22] SECUNDO F, CARREA G, TARABIONO C, et al.Activity and enantioselectivity of wildtype and lid mutated Candida rugosa lipase isoform 1 in organic solvents[J].Biotechnology and Bioengineering, 2004, 86(2):236-240.
[23] 何平, 黄卓烈, 巫光宏, 等.二甲基亚砜对木瓜蛋白酶活性与构象的影响[J].华南农业大学学报, 2008, 29(3):37-41. HE P, HUANG Z L, WU G H, et al.Effect of dimethyl sulfoxide on the activity and conformation of papain[J].Journal of South China Agricultural University, 2008, 29(3):37-41.
[24] 刘隽, 郑忠亮, 左振宇, 等.微水相中氨基酰化酶催化拆分反应的研究[J].食品工业科技, 2009, 30(8):143-145. LIU J, ZHENG Z L, ZUO Z Y, et al.Study on the resolution catalysed by aminoacylase in micro-aqueous phase[J].Science and Technology of Food Industry, 2009, 30(8):143-145.
[25] 刘喆, 李家霖, 白利平.微生物酯酶研究进展[J].微生物学报, 2023, 63(2):451-464. LIU Z, LI J L, BAI L P.Research advances in microbial esterases[J].Acta Microbiologica Sinica, 2023, 63(2):451-464.
[26] LI G L, XU L, ZHANG H J, et al.A de novo designed esterase with p-nitrophenyl acetate hydrolysis activity[J].Molecules, 2020, 25(20):4658.
[27] MA J, MA Y P, LI Y Q, et al.Characterization of feruloyl esterases from Pecoramyces sp.F1 and the synergistic effect in biomass degradation[J].World Journal of Microbiology and Biotechnology, 2022, 39(1):1-12.
[28] 马春辉. 酯酶在化学合成中的应用[J].化工设计通讯, 2018, 44(6):68. MA C H.Application of esterase in chemical synthesis[J].Chemical Engineering Design Communications, 2018, 44(6):68.
[29] 黄嫒, 马志强, 王玮, 等.豌豆酯酶的纯化和酯酶活性表征及其对农药的敏感性[J].食品与发酵工业, 2019, 45(11):78-84. HUANG A, MA Z Q, WANG W, et al.Purification, characterization and pesticides-sensitivity of plant-esterase from Pisum sativum Linn[J].Food and Fermentation Industries, 2019, 45(11):78-84.