自1780年瑞典化学家舍勒从酸奶中分离得到乳酸以来,乳酸作为一种酸味剂、增味剂和防腐剂,在食品、制药、化妆品和其他化学工业中占据了重要地位。近年来,乳酸作为生产聚乳酸的原料而备受关注。聚乳酸作为一种可生物降解的聚合物,可替代石油基塑料用于包装和医疗领域。此外,光学纯L-乳酸或/和D-乳酸聚合成的聚乳酸具有更好的热稳定性和机械性,有望在未来应用于汽车和电子领域[1]。乳酸的市场需求每年以10%的速度增长,且2022年全球乳酸产能已达到99.5万t。因此,具有高光学纯度和低发酵成本等优势的微生物发酵法生产乳酸受到广泛关注。
为了构建具有高产量、高产率和高生产率的高效乳酸合成微生物细胞工厂,研究人员开发了多种代谢工程策略,包括乳酸脱氢酶(lactate dehydrogenase,LDH)的表达调控、糖酵解途径优化、副产物途径的阻断、氧化还原平衡调节等。与此同时,通过拓宽微生物细胞工厂中可利用的碳源种类降低乳酸发酵成本,提高菌株工业鲁棒性,为放大发酵奠定了坚实的基础。此外,在基因编辑、多基因同时调控、基因动态调控、高通量筛选等新技术的辅助下,乳酸合成微生物细胞工厂的构建得到了显著加速。在这个过程中,不同菌种在乳酸生产方面具有不同的优势和生产能力。表1为不同菌种高产乳酸的研究现状总结。
表1 不同菌种的乳酸生产能力研究现状
Table 1 Research status of lactic acid production capacity of different strains
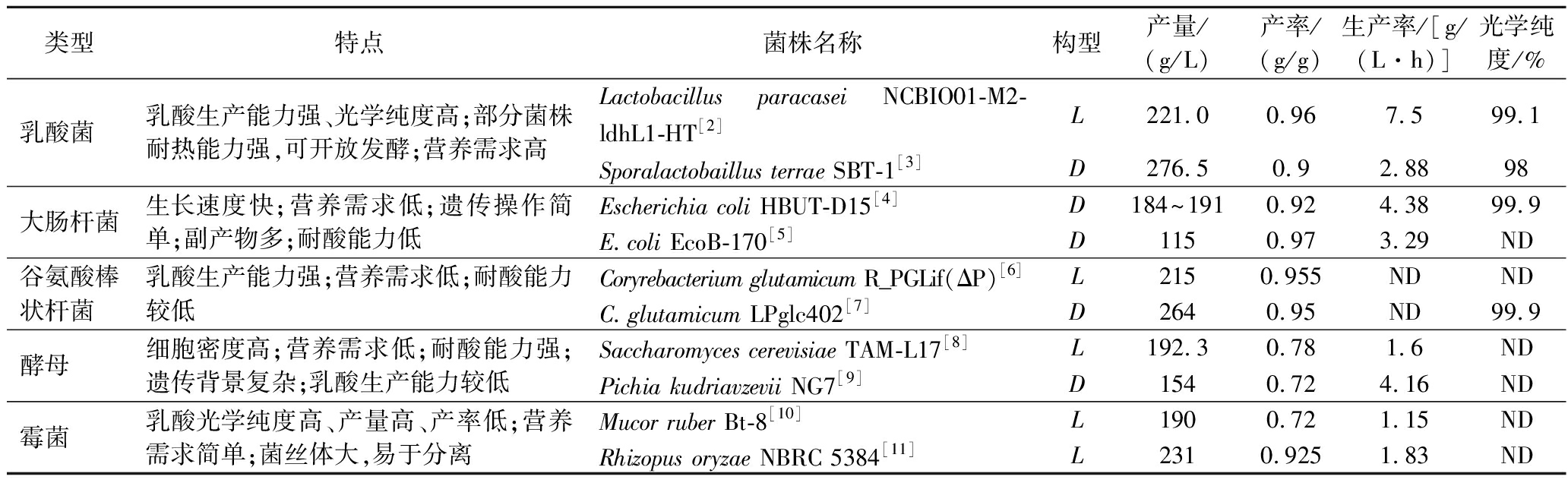
类型特点菌株名称构型产量/(g/L)产率/(g/g)生产率/[g/(L·h)]光学纯度/%乳酸菌乳酸生产能力强、光学纯度高;部分菌株耐热能力强,可开放发酵;营养需求高Lactobacillus paracasei NCBIO01-M2-ldhL1-HT[2]L221.0 0.967.599.1Sporalactobaillus terrae SBT-1[3]D276.50.92.8898大肠杆菌生长速度快;营养需求低;遗传操作简单;副产物多;耐酸能力低Escherichia coli HBUT-D15[4]D184~1910.924.3899.9E.coli EcoB-170[5]D1150.973.29ND谷氨酸棒状杆菌乳酸生产能力强;营养需求低;耐酸能力较低Coryrebacterium glutamicum R_PGLif(ΔP)[6]L2150.955NDNDC.glutamicum LPglc402[7]D2640.95ND99.9酵母细胞密度高;营养需求低;耐酸能力强;遗传背景复杂;乳酸生产能力较低Saccharomyces cerevisiae TAM-L17[8]L192.30.781.6NDPichia kudriavzevii NG7[9]D1540.724.16ND霉菌乳酸光学纯度高、产量高、产率低;营养需求简单;菌丝体大,易于分离Mucor ruber Bt-8[10]L1900.721.15NDRhizopus oryzae NBRC 5384[11]L2310.9251.83ND
注:ND为未查到。
本文总结了近年来利用合成生物学和代谢工程技术构建微生物细胞工厂合成乳酸的研究进展,并对当前面临的挑战及未来的研究方向进行了探讨。
1 代谢通路的构建与优化
要构建高产量、高产率和高生产率的乳酸合成微生物细胞工厂,乳酸代谢通路的构建与优化是不可或缺的。LDH的表达调控、糖酵解途径的强化以及副产物代谢途径的敲除,可为乳酸的形成提供更多代谢通量;阻断L-乳酸或D-乳酸对映体的生成,实现了高光学纯度乳酸的生产。此外,新兴的基因组规模代谢网络模型(genome-scale metabolic model, GSMM)从全局水平指导乳酸生产菌株的改造,为乳酸产量的提高提供了新的思路(图1)。
1.1 LDH的表达调控
LDH催化丙酮酸生成乳酸是微生物合成乳酸的关键步骤。在天然乳酸生产菌中,可使用强启动子取代LDH的启动子和增加基因拷贝数来提高乳酸产量[13]。而对于非天然乳酸生产菌株或合成量较少的菌株,需筛选高酶活力的LDH来构建异源代谢通路。目前未见研究来系统评估来源不同的LDH催化活性及同一个LDH在不同的宿主存在活性差异,研究人员仍需检测多个不同来源的LDH来挑选具有较高活性的酶,不同微生物细胞工厂使用的LDH来源如表2所示。随着合成生物学工具和策略的发展,一些基因表达元件或方法可以用于提高LDH的表达水平和催化效率。ZHOU等[14]通过启动子工程策略,调整ldhA上游的潜在转录区域来微调基因表达,实现了菌株生长速率和乳酸产量的同时提高。田康明等[15]使用温度诱导型启动子PR-PL作为遗传开关来调控E.coli中乳酸的合成。前期低温发酵以保证高细胞密度,后期高温发酵则诱导LDH表达。与之类似,HWANG等[16]利用Pnar启动子将E.coli发酵过程分为高氧诱导生长阶段和低氧诱导乳酸合成,这两种LDH的动态调控方法均能有效提高乳酸产量(图1-a)。此外,REIDER APEL等[17]利用CRISPR/Cas9技术构建了一个无克隆的工具包,可以用于S.cerevisiae中LDH的整合位点和启动子的选择。目前单纯通过LDH的表达调控尚不能达到乳酸工业化生产的要求,还需进行其他代谢工程改造。

a-LDH的表达调控;b-糖酵解途径的强化;c-副产物途径的敲除;d-乳酸光学纯度的提升;e-GSMM指导菌株改造[12]
图1 代谢通路的构建与优化策略
Fig.1 Strategies for the construction and optimization of metabolic pathways
表2 不同宿主中异源表达LDH的来源
Table 2 Source of heterologously expressed LDH in different hosts

宿主L-LDH的来源D-LDH的来源S. cerevisiaePelodiscus sinensis; Lactobacillus lactis; Bacillus coagulans; 牛源Leuconostoc mesenteroides; E. coli; Leuconostoc pseudomesenteroidesKluyveromyces marxianusLactobacillus plantarum; Plasmodium falciparum; Bacillus megateriumL. plantarumE. coliBacillus coagulans; Streptococcus bovis; B. subtilisLactobacillus bulgaricus; Pediococcus acidilacticiC. glutamicum内源Lactobacillus delbrueckii; L. bulgaricus; E. coliBacillus内源L. delbrueckii; 嗜热菌
1.2 糖酵解途径的强化
乳酸的合成前体是丙酮酸,而丙酮酸是糖酵解的终产物。因此,为了实现高效的乳酸生产,需要加强糖酵解途径(图1-b)。磷酸果糖激酶(phosphofructokinase,PFK)和丙酮酸激酶(pyruvatekinase,PYK)是糖酵解的限速酶[18]。在E.coli KO20中,通过pfk和异源pyk基因的过表达可使乳酸产量增加9倍[19]。在C.glutamicum CRZ2中,过表达pfk基因乳酸产量仅能提高15%,过表达pyk则会导致产量下降,而过表达负责葡萄糖转运的葡萄糖激酶基因glk,乳酸产量提高了98%[20]。酵母中pfk和pyk基因的过表达虽能增加酶的表达,但葡萄糖的消耗率仍保持不变。因此,YAMADA等[18]通过全局代谢工程技术将不同启动子的13种糖酵解相关基因随机整合到酵母基因组中的δ位点,显著提高葡萄糖消耗率,并在后续研究中将这种方法应用于乳酸高产菌株的构建[21]。总之,强化糖酵解途径来提升乳酸的产量的瓶颈可能并非由单一酶引起,且不同菌株中糖酵解相关基因的过表达对乳酸合成的影响也不同。因此,需要从糖酵解途径的全局出发,系统地提高葡萄糖消耗率,进而提升乳酸的产量。
1.3 副产物途径的敲除
乳酸发酵包括同型乳酸发酵、异型乳酸发酵以及混合酸发酵,其中同型乳酸发酵只产生乳酸,无副产物途径,理论产率可达100%。而在异型乳酸发酵或混合酸发酵过程中,阻断或减少副产物合成路径能使更多的碳通量流向乳酸合成。异型乳酸发酵会产生乙醇、乙酸和CO2等副产物(图1-c),阻断磷酸戊糖酮解酶途径,将碳通量重新定向到磷酸戊糖途径,可以减少副产物的形成,提高乳酸产量。在混合酸发酵时,阻断丙酮酸代谢旁路可提高乳酸产量(图1-c)。在大肠杆菌中,丙酮酸甲酸裂解酶PFLB、乙酸激酶ACKA和磷酸转乙酰酶PTA是丙酮酸代谢中影响乳酸产量的关键酶,对E.coli MG1655分别单基因缺失后乳酸产量可增加39.6%、25.2%和39.4%[22]。对于酵母而言,乙醇是丙酮酸代谢的主要产物。失活丙酮酸脱羧酶基因pdc和乙醇脱氢酶基因adh可减少乙醇积累,增加乳酸积累量[23],阻止二羟丙酮磷酸合成甘油可将碳通量引向乳酸合成途径,进一步提高乳酸产量。除大肠杆菌和酵母外,不同生产菌株还需阻断其他副产物代谢途径,如Kluyveromyces pneumoniae需在失活丙酮酸代谢关键基因的基础上再失活2,3-丁二醇合成途径[24]。
1.4 乳酸光学纯度的提升
光学纯L-乳酸或/和D-乳酸能够合成高结晶度和高熔点的聚乳酸,从而用于生产纤维、薄膜和液晶,具有广阔的应用前景[1]。因此,提高乳酸的光学纯度对聚乳酸的应用至关重要。乳酸的光学纯度受宿主选择和底物复杂性的影响(图1-d)。野生型乳酸生产菌株通常同时产生L-乳酸和D-乳酸。为了提高D-乳酸的光学纯度,失活L-LDH是最常用的方法[25]。由于菌株通常含有多个L-LDH,给实际操作带来困难,近期发表的CRISPR胞脱氨酶辅助碱基编辑器可以同时失活L.lactis NZ9000中的4个L-LDH,为乳酸菌的乳酸光学纯度提升提供了新技术[26]。当存在乳酸外消旋酶基因larA时,还需敲除larA[25]。此外,甲基乙二醛旁路会同时产生L-乳酸和D-乳酸,通过失活甲基乙二醛合成酶基因mgsA,也可以提高D-乳酸光学纯度[27]。最后,当过表达L-乳酸氧化酶基因loxL时,可以消耗L-乳酸,从而在被L-乳酸污染的复杂培养基中也能实现高光学纯D-乳酸的发酵[28]。提高L-乳酸纯度时,以上策略也适用。总之,由于菌株的特异性和培养基的不同,通常采用多种策略联合使用,以满足聚合级乳酸的要求。
1.5 基因组规模代谢网络模型指导菌株改造
基于基因组测序分析构建的GSMM,通过对微生物基因、蛋白以及代谢反应之间的关系进行表征,来预测模拟不同系统水平的代谢反应通量,为解析复杂代谢网络和调控机制、高效定向改造微生物奠定了基础(图1-e)。GSMM已广泛用于预测细胞表型、指导代谢工程、预测药物靶点、检测生物标志物等方面[29]。
目前,基于GSMM的代谢工程改造已经在酵母和大肠杆菌中得到了应用。2000年,报道了首个大肠杆菌GSMM—iJE660[30]。2003年,基于iJE660首次使用OptKnock算法对大肠杆菌高产乳酸的基因敲除靶点进行预测,ack、pta、pfk、fba、adhE以及glk基因的联合缺失可得到18.13 mmol/h的最佳乳酸生产率[31]。随着GSMM的改进和算法的更新,研究人员陆续探索了ABCMOMA、BATMOMA以及CSMMOMA等混合算法,用于预测乳酸产率的最优解。然而,以上的设计基因敲除策略需要其他基因的上调或下调以匹配所需的通量分布,因此又提出了一种基于约束的算法GeneReg[32]。经该算法预测,酵母需要至少36个基因的上调或下调,以实现酵母中乳酸的高效生产,最大乳酸合成通量为56.66 mmol/(g DW·h)。GSMM全面和系统化的代谢网络分析,为乳酸提供了更加经济和可持续的生产方式。随着各种微生物GSMM的构建和完善,GSMM将用于多种乳酸菌、谷氨酸棒杆菌等微生物的代谢工程,以实现乳酸的高效生产。但目前预测精度仍不高,需要进行“湿实验”来验证预测,并很少获得成功的应用。
2 氧化还原平衡的调节
细胞内的氧化还原状态是由多个氧化还原反应组成的复杂网络维持的。当涉及氧化还原反应的代谢途径发生改变或引入新代谢途径时,往往会导致细胞内的氧化还原失衡,从而降低工业微生物的生长性能和产物合成能力[33]。因此,调节氧化还原状态是提高乳酸产量的有效途径之一。
通过在培养基中添加甘油、山梨醇、L-半胱氨酸盐酸盐以及Na2S等化合物,降低底物的氧化还原电位,并提供更多的还原当量,从而显著提高乳酸的生产率[34]。此外,NADH/NAD+和NADPH/NADP+等氧化还原辅因子在分解代谢、合成代谢以及能量产生的耦合中发挥着关键作用。氧化还原辅因子的扰动对产物分布以及细胞内代谢物含量均有显著的影响。主要还原副产物(如乙醇和甘油)代谢途径的敲低、甘油醛-3-磷酸脱氢酶基因gapdh的过表达[35]以及NADH脱氢酶基因nde的失活[36],均可提高胞内NADH水平,进而提高乳酸产量。此外,通过对大肠杆菌氧化还原相关反应的全基因组分析,发现D-乳酸的产量还与核苷酸和氨基酸代谢相关的脱氢酶基因(如guaB、pyrD和serA)有关。这些基因的单基因缺失可以增加乳酸产量[37]。
除氧化还原相关酶以外,全局转录调控因子Rex可以通过响应细胞内NADH/NAD+比率的变化来调节细胞内的氧化还原平衡[38]。失活Caldicellmlosiruptor bescii中的rex基因会使细胞内氧化还原电位降低,从而使乳酸等代谢产物积累增加[39]。但是rex基因的缺失在不同菌株中可能会产生不同的代谢效应,例如,在Thermoanaerobacterium saccharolyticum中,rex基因的缺失解除了对adhE和adhA的表达调控,导致乙醇产量增加两倍,而乳酸产量减少[38]。因此,rex基因的缺失的不同代谢效应可能取决于其调控的靶点不同而导致的,因此仍需进一步研究。总之,全面了解不同菌株氧化还原代谢将有助于设计氧化还原系统以促进乳酸的高效生产。
3 底物谱的拓宽
乳酸发酵底物成本约占总成本的40%~70%,是影响乳酸生产经济性的重要因素。为了降低乳酸的发酵成本,需要拓宽乳酸生产菌株的底物谱,促进廉价的农业工业废弃物在乳酸发酵中的应用。
碎米、废木薯渣以及废姜黄等农业废弃物中富含淀粉。然而大多数乳酸菌无法直接利用淀粉类物质,通常需要对淀粉原料进行预处理或者对菌株进行改造。研究者通过表达α-淀粉酶基因amyA,实现了利用玉米淀粉和糙米同步糖化发酵乳酸[25]。此外,木质纤维素也广泛存在于农业废弃物中,如玉米秸秆、大豆残渣和甘蔗渣等。该类原料的使用可有效避免食品资源的浪费,是理想的廉价乳酸发酵底物。木质纤维素水解产物中木糖占总糖的30%,但大部分乳酸菌缺乏木糖同化的能力,使木质纤维素的应用受到限制。将来源于Lactobacillus pentosus和Lactobacillus lactis IO-1的木糖同化基因引入乳酸菌后,从而使其能够利用木糖作为碳源进行乳酸发酵[40]。对于纤维素类生物质,通常需要使用昂贵的纤维素酶进行预处理。为了解决这一问题,GANDINI等[41]在乳杆菌中整合β-葡萄糖苷酶基因bglA和内切葡聚糖酶基因engD,构建了纤维素酶系统。该菌株可利用纤维素低聚糖作为底物一步发酵生产乳酸,向光学纯乳酸的低成本可持续生物发酵迈出了重要一步。
另外,廉价农业工业废弃物的水解产物中的碳源大多是混合糖,乳酸生产菌株在混合糖存在的情况下,首先消耗葡萄糖,抑制碳分解代谢,导致糖未能充分利用,从而降低发酵效率并增加生产成本。为了解决这一问题,LU等[42]对大肠杆菌编码葡萄糖转运磷酸转移酶系统的主要酶IIBCglc中的ptsG基因进行失活。ptsG基因缺失导致磷酸转移酶(phosphotransferase system,PTS)系统被破坏,葡萄糖只能依靠半乳糖转运蛋白GalP和Mgl等低效率的替代系统进行转运,进而消除了分解代谢抑制。LI等[43]在阻断PTS系统和激活Gal系统的同时,将木糖分解的限速酶木糖异构酶基因xylA替换为B.coagulans的xylABC。该双通道工艺使木糖利用率增加46.3%。WANG等[44]通过研究基于阻遏物的木糖传感/调控基因线路,开发了一种高通量筛选方法,成功获得了木糖转运蛋白HXT14的突变体,其木糖转运能力提高了6.5倍,为提高菌株中木糖的利用率提供了新的解决方案。
以上方法虽然有助于提高底物利用率和乳酸发酵效率,但当底物复杂度增加时,这些方法的适用性会受到限制,仍需要进一步的优化和改进,以提高其在实际工业应用中的可行性和效率。
4 乳酸生产菌株的工业鲁棒性
乳酸工业生产过程中面临各种恶劣的工业条件,包括低pH值、高温、高糖以及来自廉价生物质水解物中的有毒抑制剂等。微生物鲁棒性是指面对各种扰动时保持稳定和高效表型的能力。通过理性设计加定向进化可提高菌株对恶劣工业条件和有毒抑制剂的胁迫耐受性,从而提高乳酸的产量和产率。
4.1 菌株酸胁迫耐受性的提高
微生物发酵过程中乳酸的积累导致pH值逐渐降低,当pH值低于乳酸的pKa时,游离乳酸会扩散到细胞内,由于胞内pH值高于乳酸的pKa,游离乳酸会解离成酸性阴离子和质子,对细胞造成损伤[9]。目前解决这一问题的主要方法是添加中和剂[如NaOH、Ca(OH)2、CaCO3和氨水等],但这会增加发酵成本和下游的分离纯化成本。因此,工业菌株的酸胁迫是目前乳酸工业生产面临的主要问题之一。
微生物对酸胁迫的反应是一个复杂的过程。目前,生物化学、蛋白组学和遗传学的研究已证实了酸胁迫耐受性与各种细胞代谢调节过程密切相关。菌株对乳酸胁迫的转录反应很大程度上受转录因子的影响(图2-a)。BAEK等[23]在S.cerevisiae中表达全局转录因子Haa1可促使其亚细胞定位由胞浆转移至胞核,并诱导其靶基因ygp1、gpg1和spi1的表达,从而介导酵母对乳酸的胁迫适应[23]。sRNA可以通过与mRNA相互作用快速调节目标基因表达,在环境压力响应过程中发挥关键的调控作用[45](图2-b)。GAIDA等[46]在E.coli中过表达3种sRNAs:DsrA、RprA和ArcZ,将乳酸耐受性提高1 270倍。为了减少乳酸在胞内的积累,表达乳酸阴离子转运蛋白基因jen1和ady2,可将阴离子输送到细胞外,从而增加S.cerevisiae中乳酸的产量[47](图2-c)。最后,通过加强细胞壁结构以减少孔隙度和改变质膜的脂质组成以增加膜刚性来实现细胞包膜重排,也可以增加酸胁迫耐受性[48](图2-d)。
除了前面提到的方法,低pH值适应性实验室进化(adaptive laboratory evolution,ALE)被应用于平衡细胞的耐酸能力与乳酸产量。在ALE获得目标菌株后,通过系统生物学分析阐明有助于酸耐受的遗传机制或遗传靶标,并进行反向代谢工程将有益突变重新引入野生型或其他工程菌株,能进一步提高菌株耐酸能力和乳酸产量(图2-e)。此外,基于随机突变的基因组进化和可示踪基因组进化技术已经被广泛应用于提高酸耐受性(图2-f)。SI等[49]开发了一种自动化多重基因组进化方法,通过结合RNA干扰和CRISPR/Cas9技术,进行多轮迭代基因组进化提高S.cerevisiae乙酸耐受性,将来也可用于提高菌株对其他酸的耐受能力。
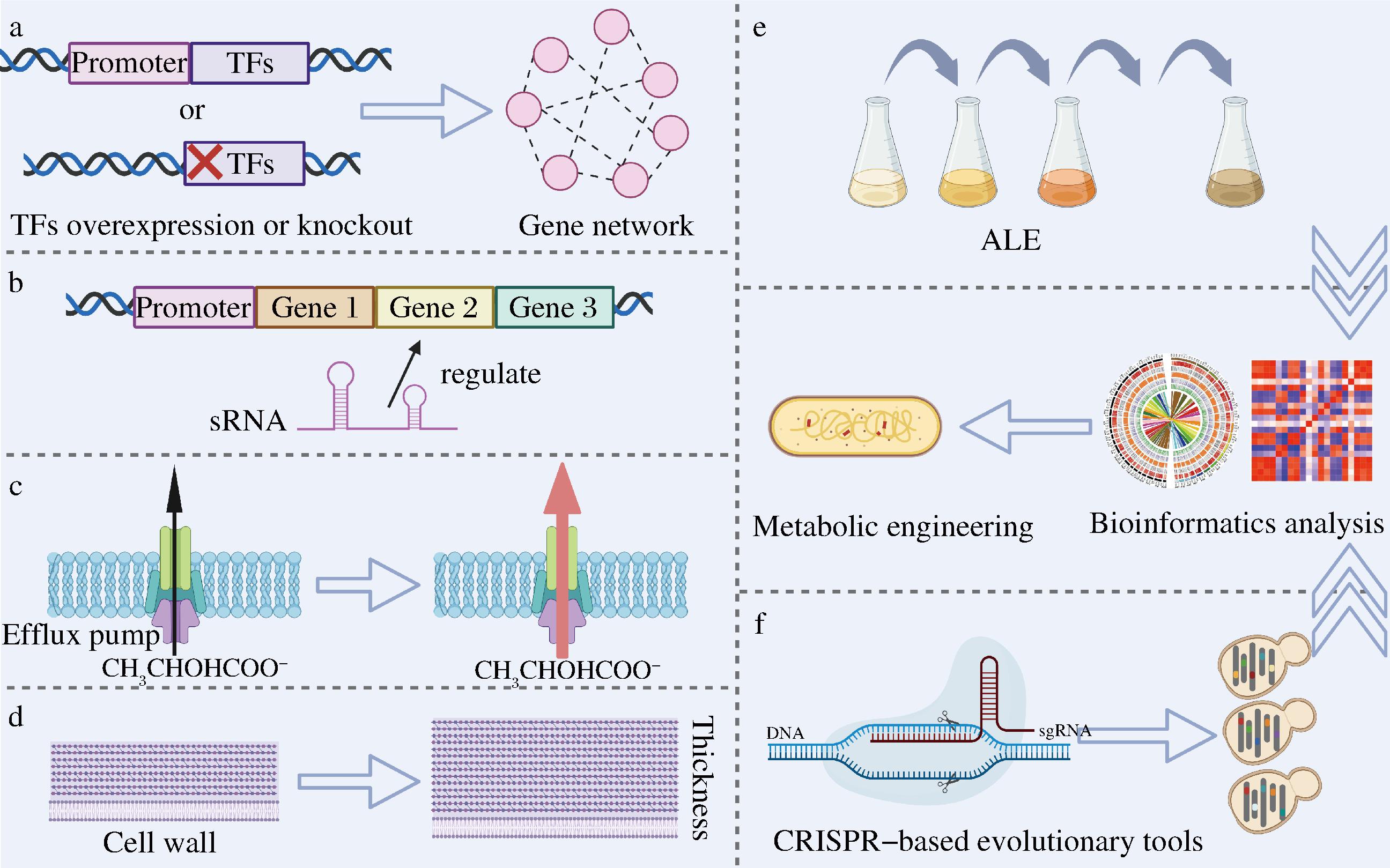
a-应激相关转录因子的表达或敲除;b-sRNA调控基因表达;c-加强乳酸阴离子外排;d-增加细胞壁厚度; e-适应性实验室进化;f-基因组进化
图2 提高菌株酸耐受性的方法(修改自[45, 50])
Fig.2 Methods of improving acid tolerance of strains
4.2 菌株热胁迫耐受性的提高
在工业发酵过程中,微生物的生长代谢和发酵罐的搅拌会产生大量热能。当发酵温度高于某一区间时,菌株会由于新陈代谢速率加快而引起早衰,严重影响乳酸的产率。然而,采用高温发酵可减少副产物的生成、降低噬菌体污染的风险,同时还能降低灭菌成本和冷却费用。从自然界分离的嗜热芽孢杆菌可以在50 ℃时进行乳酸的开放发酵。除此之外,大部分乳酸生产菌株都是嗜温菌。与嗜热菌相比,嗜温菌在分批发酵过程通常具有更好的乳酸生产能力,因此研究其热应激反应的分子基础,特别是热休克蛋白的作用及其调控机制,对提高菌株的热胁迫耐受性具有重要指导意义。当细胞暴露于高温环境中时,DnaK-GrpE-DnaJ和GroES-GroEL伴侣复合物对细胞膜的稳定和DNA复制等生物学过程具有重要作用。在L.lactis中过表达热休克蛋白GroES和DnaK,可以引导蛋白质的正确折叠,从而提高乳酸乳球菌的热胁迫耐受性并得到较高的乳酸生产率[51]。最近,LI等[52]利用CRISPR基因激活文库筛选技术,成功应用于提升K.marxianus的热胁迫耐受性,并阐明了一种新的耐高温机制。研究发现,过表达脂肪酸合成代谢通路的关键基因OLE1可以提高不饱和脂肪酸比例,降低胞内脂质过氧化水平,从而有效提升酵母的热胁迫耐受性。
4.3 菌株抑制剂耐受性的提高
复杂的木质纤维素在预处理过程中会产生抑制剂,如呋喃、酚类化合物、生物醇、无机离子和脂肪酸等。这些化合物能够抑制酶的活性和细胞生长,从而影响乳酸发酵过程。通过生物脱毒可去除一部分抑制剂,但为了减少糖类在脱毒过程中的消耗,仍会残留少量抑制剂。研究表明,L-1,2-丙二醇氧化还原酶FucO可催化糠醛还原为毒性较小的醇,因此可以通过表达天然fucO基因来提高菌株的糠醛耐受性。之后通过引入FucO同源二聚体界面L7F突变,可将酶活力提高10倍,同时糠醛代谢速率增加了一倍[53]。除此之外, LIAN等[54]将可同时进行转录激活、转录抑制和基因敲除的三功能CRISPR体系与寡核苷酸芯片技术结合,成功开发出多功能全基因组进化技术,经过3轮进化,酵母能够在17.5 mmol/L糠醛浓度下消耗大部分的葡萄糖。此外,筛选到多个糠醛耐受性相关的新靶点,如SLX5、NUP133和GPI17等。尽管该技术目前用于乙醇发酵,但未来有望将其应用于乳酸发酵生产。通过短链脱氢酶CGS9114_RS09725的整合可提高P.acidilactici对香草醛的耐受性[55]。表达氧化还原酶基因ZMO1116可提高菌株对苯醌的耐受性[56]。然而,以上研究只针对单一抑制剂,在面对复杂的木质纤维素水解产物中的混合抑制剂时,其耐受性仍然不足。为此,有研究报道,在未脱毒的玉米芯水解液中经过111 d的长期ALE,可同时提高多种醛抑制剂的生物转化能力,从而实现酸预处理玉米芯水解液一锅法生产D-乳酸[57]。
4.4 菌株渗透胁迫耐受性的提高
微生物发酵过程中,高浓度的营养物质会导致细胞内外渗透压产生差异,特别是当发酵罐内初始糖浓度过高时,会导致菌株生长延迟,从而影响乳酸产量和生产率。虽然补料分批发酵可以降低初始糖浓度,但提高菌株的渗透胁迫耐受性可以简化工艺,降低成本。添加渗透保护剂(如甜菜碱和脯氨酸)是提高渗透胁迫耐受性最简单的方法[58]。此外,通过在高浓度的甜菜糖蜜中对菌株进行ALE,能够使菌株的糖耐受性和抗氧化能力增强,最终使乳酸生产率提高31%[58]。QI等[59] 通过对葡萄糖胁迫下L.lactis的代谢组学和蛋白组学进行分析,构建了葡萄糖胁迫反应机制模型,发现L.lactis通过上调氧化还原酶活性来响应葡萄糖胁迫,从而提高葡萄糖到乳酸的代谢通量,为提高乳酸生产菌株的渗透胁迫耐受性提供了新思路。
5 结论与展望
随着市场对乳酸的需求不断增加,亟需开发满足工业要求的高性能乳酸生产菌株。为了应对这一挑战,研究人员采用合成生物学和代谢工程技术对菌株进行改造,有效提高乳酸的产量、产率、生产率以及光学纯度。此外,为应对工业发酵中的极端环境,理性设计与定向进化相结合的耐受性工程已被用来提高菌株鲁棒性。尽管如此,微生物细胞工厂高效合成乳酸仍存在一定挑战,以下问题还有待进一步优化:
1)工业菌株性能的提升往往是通过生物量和目标产物的形成、氧化还原平衡和复杂的耐受调控之间的权衡来实现的。尽管GSMM为系统研究菌株代谢网络提供了有利帮助,但仍缺乏转录调控信息。而全细胞模型能将细胞内所有生命活动模块化(图1-e),有望解决目前代谢工程存在的问题并在未来得以应用。2)大多数基因工程乳酸生产菌株在发酵廉价生物质时仍存在困难。因此,需进一步开发可利用廉价替代底物发酵的菌株以及低毒预处理技术。3)由于菌株中涉及的耐受机制大多是未知的,因此,增强菌株耐受性主要依赖于进化工程。对进化耐受菌株的耐受遗传机制进行系统生物学分析,将有助于加快机制的解析并通过反向代谢工程应用于菌株改造。4)新型基因组进化技术为提高乳酸产量提供了新思路,但如何快速对庞大的突变库进行筛选是目前面临的另一个问题。未来可以结合荧光激活细胞分选技术和液滴微流控分选技术,以大幅提高基因组进化的效率。5)发酵过程中乳酸浓度和pH值对发酵至关重要,但目前还没有准确的实时监测工具。最近,XIAO等[60] 成功研发了基于转录因子LldR的生物传感器BLac-6,可以同时监测Klebsilla oxytoca发酵液中L-乳酸和D-乳酸的浓度,这为未来工业发酵过程中的乳酸浓度实时监测带来了希望。
[1] KLOTZ S, KAUFMANN N, KUENZ A, et al.Biotechnological production of enantiomerically pure D-lactic acid[J].Applied Microbiology and Biotechnology, 2016, 100(22):9423-9437.
[2] TIAN X W, LIU X H, ZHANG Y F, et al.Metabolic engineering coupled with adaptive evolution strategies for the efficient production of high-quality L-lactic acid by Lactobacillus paracasei[J].Bioresource Technology, 2021, 323:124549.
[3] THITIPRASERT S, PILUK J, TOLIENG V, et al.Draft genome sequencing of Sporolactobacillus terrae SBT-1, an efficient bacterium to ferment concentrated sugar to D-lactic acid[J].Archives of Microbiology, 2021, 203(6):3577-3590.
[4] FU X M, WANG Y Z, WANG J H, et al.Semi-industrial scale (30 m3) fed-batch fermentation for the production of D-lactate by Escherichia coli strain HBUT-D15[J].Journal of Industrial Microbiology &Biotechnology, 2017, 44(2):221-228.
[5] WANG Y D, LIAO J Y, CHIANG C J, et al.A simple strategy to effectively produce D-lactate in crude glycerol-utilizing Escherichia coli[J].Biotechnology for Biofuels, 2019, 12:273.
[6] HASEGAWA S, TANAKA Y, SUDA M, et al.Enhanced glucose consumption and organic acid production by engineered Corynebacterium glutamicum based on analysis of a pfkB1 deletion mutant[J].Applied and Environmental Microbiology, 2017, 83(3):e02638-e02616.
[7] TSUGE Y, KATO N, YAMAMOTO S, et al.Metabolic engineering of Corynebacterium glutamicum for hyperproduction of polymer-grade L- and D-lactic acid[J].Applied Microbiology and Biotechnology, 2019, 103(8):3381-3391.
[8] LIU T T, SUN L, ZHANG C, et al.Combinatorial metabolic engineering and process optimization enables highly efficient production of L-lactic acid by acid-tolerant Saccharomyces cerevisiae[J].Bioresource Technology, 2023, 379:129023.
[9] PARK H J, BAE J H, KO H J, et al.Low-pH production of D-lactic acid using newly isolated acid tolerant yeast Pichia kudriavzevii NG7[J].Biotechnology and Bioengineering, 2018, 115(9):2232-2242. [10] WEUSTHUIS R A, MARS A E, SPRINGER J, et al.Monascus ruber as cell factory for lactic acid production at low pH[J].Metabolic Engineering, 2017, 42:66-73.
[11] YAMANE T, TANAKA R.Highly accumulative production of L(+)-lactate from glucose by crystallization fermentation with immobilized Rhizopus oryzae[J].Journal of Bioscience and Bioengineering, 2013, 115(1):90-95.
[12] BI X Y, LIU Y F, LI J H, et al.Construction of multiscale genome-scale metabolic models:Frameworks and challenges[J].Biomolecules, 2022, 12(5):721.
[13] MAZZOLI R, OLSON D G, LYND L R.Construction of lactic acid overproducing Clostridium thermocellum through enhancement of lactate dehydrogenase expression[J].Enzyme and Microbial Technology, 2020, 141:109645.
[14] ZHOU L, SHEN W, NIU D D, et al.Fine tuning the transcription of ldhA for D-lactate production[J].Journal of Industrial Microbiology &Biotechnology, 2012, 39(8):1209-1217.
[15] 田康明, 周丽, 陈献忠, 等.利用温度调节实现新型重组菌高效转化甘油为D-乳酸 [J].生物工程学报, 2013,29(1):111-114. TIAN K M, ZHOU L, CHEN X Z, et al.Temperature-switched high-efficient D-lactate production from glycerol[J].Chinese Journal of Biotechnology, 2013, 29(1):111-114.
[16] HWANG H J, KIM J W, JU S Y, et al.Application of an oxygen-inducible nar promoter system in metabolic engineering for production of biochemicals in Escherichia coli[J].Biotechnology and Bioengineering, 2017, 114(2):468-473.
[17] REIDER APEL A, D’ESPAUX L, WEHRS M, et al.A Cas9-based toolkit to program gene expression in Saccharomyces cerevisiae[J].Nucleic Acids Research, 2017, 45(1):496-508.
[18] YAMADA R, WAKITA K, OGINO H.Global metabolic engineering of glycolytic pathway via multicopy integration in Saccharomyces cerevisiae[J].ACS Synthetic Biology, 2017, 6(4):659-666.
[19] EMMERLING M, BAILEY J E, SAUER U.Glucose catabolism of Escherichia coli strains with increased activity and altered regulation of key glycolytic enzymes[J].Metabolic Engineering, 1999, 1(2):117-127.
[20] TSUGE Y, YAMAMOTO S, SUDA M, et al.Reactions upstream of glycerate-1, 3-bisphosphate drive Corynebacterium glutamicum (D)-lactate productivity under oxygen deprivation[J].Applied Microbiology and Biotechnology, 2013, 97(15):6693-6703.
[21] YAMADA R, WAKITA K, MITSUI R, et al.Enhanced D-lactic acid production by recombinant Saccharomyces cerevisiae following optimization of the global metabolic pathway[J].Biotechnology and Bioengineering, 2017, 114(9):2075-2084.
[22] ZHOU L, CUI W J, LIU Z M, et al.Metabolic engineering strategies for D-lactate over production in Escherichia coli [J].Journal of Chemical Technology &Biotechnology, 2016, 91(3):576-584.
[23] BAEK S H, KWON E Y, KIM Y H, et al.Metabolic engineering and adaptive evolution for efficient production of D-lactic acid in Saccharomyces cerevisiae[J].Applied Microbiology and Biotechnology, 2016, 100(6):2737-2748.
[24] FENG X J, JIANG L Q, HAN X J, et al.Production of D-lactate from glucose using Klebsiella pneumoniae mutants[J].Microbial Cell Factories, 2017, 16(1):209.
[25] OKANO K, UEMATSU G, HAMA S, et al.Metabolic engineering of Lactobacillus plantarum for direct L-lactic acid production from raw corn starch[J].Biotechnology Journal, 2018, 13(5):e1700517.
[26] TIAN K R, HONG X, GUO M M, et al.Development of base editors for simultaneously editing multiple loci in Lactococcus lactis[J].ACS Synthetic Biology, 2022, 11(11):3644-3656.
[27] WANG Z W, SAINI M, LIN L J, et al.Systematic engineering of Escherichia coli for D-lactate production from crude glycerol[J].Journal of Agricultural and Food Chemistry, 2015, 63(43):9583-9589.
[28] OKANO K, SATO Y, HAMA S, et al.L-Lactate oxidase-mediated removal of L-lactic acid derived from fermentation medium for the production of optically pure D-lactic acid[J].Biotechnology Journal, 2022, 17(4):e2100331.
[29] YE C, WEI X Y, SHI T Q, et al.Genome-scale metabolic network models:From first-generation to next-generation[J].Applied Microbiology and Biotechnology, 2022, 106(13-16):4907-4920.
[30] EDWARDS J S, PALSSON B O.The Escherichia coli MG1655 in silico metabolic genotype:Its definition, characteristics, and capabilities[J].Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(10):5528-5533.
[31] BURGARD A P, PHARKYA P, MARANAS C D.Optknock:A bilevel programming framework for identifying gene knockout strategies for microbial strain optimization[J].Biotechnology and Bioengineering, 2003, 84(6):647-657.
[32] RAZAGHI-MOGHADAM Z, NIKOLOSKI Z.GeneReg:A constraint-based approach for design of feasible metabolic engineering strategies at the gene level[J].Bioinformatics, 2021, 37(12):1717-1723.
[33] LIU J H, LI H L, ZHAO G R, et al.Redox cofactor engineering in industrial microorganisms:Strategies, recent applications and future directions[J].Journal of Industrial Microbiology &Biotechnology, 2018, 45(5):313-327.
[34] LIU H M, KANG J H, QI Q S, et al.Production of lactate in Escherichia coli by redox regulation genetically and physiologically[J].Applied Biochemistry and Biotechnology, 2011, 164(2):162-169.
[35] ZHU J, SHIMIZU K.The effect of pfl gene knockout on the metabolism for optically pure D-lactate production by Escherichia coli[J].Applied Microbiology and Biotechnology, 2004, 64(3):367-375.
[36] LEE J Y, KANG C D, LEE S H, et al.Engineering cellular redox balance in Saccharomyces cerevisiae for improved production of L-lactic acid[J].Biotechnology and Bioengineering, 2015, 112(4):751-758.
[37] KIM H J, HOU B K, LEE S G, et al.Genome-wide analysis of redox reactions reveals metabolic engineering targets for D-lactate overproduction in Escherichia coli[J].Metabolic Engineering, 2013, 18:44-52.
[38] ZHENG T Y, LANAHAN A A, LYND L R, et al.The redox-sensing protein Rex modulates ethanol production in Thermoanaerobacterium saccharolyticum[J].PLoS One, 2018, 13(4):e0195143.
[39] SANDER K, CHUNG D, HYATT D, et al.Rex in Caldicellulosiruptor bescii:Novel regulon members and its effect on the production of ethanol and overflow metabolites[J].MicrobiologyOpen, 2019, 8(2):e00639.
[40] YOSHIDA S, OKANO K, TANAKA T, et al.Homo-D-lactic acid production from mixed sugars using xylose-assimilating operon-integrated Lactobacillus plantarum[J].Applied Microbiology and Biotechnology, 2011, 92(1):67-76.
[41] GANDINI C, TARRARAN L, KALEMASI D, et al.Recombinant Lactococcus lactis for efficient conversion of cellodextrins into L-lactic acid[J].Biotechnology and Bioengineering, 2017, 114(12):2807-2817.
[42] LU H Y, ZHAO X, WANG Y Z, et al.Enhancement of D-lactic acid production from a mixed glucose and xylose substrate by the Escherichia coli strain JH15 devoid of the glucose effect[J].BMC Biotechnology, 2016, 16:19.
[43] LI C, GAI Z C, WANG K, et al.Engineering Bacillus licheniformis as a thermophilic platform for the production of l-lactic acid from lignocellulose-derived sugars[J].Biotechnology for Biofuels, 2017, 10:235.
[44] WANG M, LI S J, ZHAO H M.Design and engineering of intracellular-metabolite-sensing/regulation gene circuits in Saccharomyces cerevisiae[J].Biotechnology and Bioengineering, 2016, 113(1):206-215.
[45] 徐美娟, 上官春雨, 陈鑫, 等.谷氨酸棒杆菌耐受胁迫机制及工业鲁棒性合成生物学研究进展[J].生物工程学报, 2021, 37(3):831-845. XU M J, SHANGGUAN C Y, CHEN X, et al.Advances in stress tolerance mechanisms and synthetic biology for the industrial robustness of Corynebacterium glutamicum[J].Chinese Journal of Biotechnology, 2021, 37(3):831-845.
[46] GAIDA S M, AL-HINAI M A, INDURTHI D C, et al.Synthetic tolerance:Three noncoding small RNAs, DsrA, ArcZ and RprA, acting supra-additively against acid stress[J].Nucleic Acids Research, 2013, 41(18):8726-8737.
[47] PACHECO A, TALAIA G, S -PESSOA J, et al.Lactic acid production in Saccharomyces cerevisiae is modulated by expression of the monocarboxylate transporters Jen1 and Ady2[J].FEMS Yeast Research, 2012, 12(3):375-381.
-PESSOA J, et al.Lactic acid production in Saccharomyces cerevisiae is modulated by expression of the monocarboxylate transporters Jen1 and Ady2[J].FEMS Yeast Research, 2012, 12(3):375-381.
[48] ZHU P, LUO R, LI Y Z, et al.Metabolic engineering and adaptive evolution for efficient production of L-lactic acid in Saccharomyces cerevisiae[J].Microbiology Spectrum, 2022, 10(6):e0227722.
[49] SI T, CHAO R, MIN Y H, et al.Automated multiplex genome-scale engineering in yeast[J].Nature Communications, 2017, 8:15187.
[50] LIU H, QI Y L, ZHOU P, et al.Microbial physiological engineering increases the efficiency of microbial cell factories[J].Critical Reviews in Biotechnology, 2021, 41(3):339-354.
[51] ABDULLAH-AL-MAHIN, SUGIMOTO S, HIGASHI C, et al.Improvement of multiple-stress tolerance and lactic acid production in Lactococcus lactis NZ9000 under conditions of thermal stress by heterologous expression of Escherichia coli DnaK[J].Applied and Environmental Microbiology, 2010, 76(13):4277-4285.
[52] LI P S, FU X F, ZHANG L, et al.CRISPR/Cas-based screening of a gene activation library in Saccharomyces cerevisiae identifies a crucial role of OLE1 in thermotolerance[J].Microbial Biotechnology, 2019, 12(6):1154-1163.
[53] ZHENG H B, WANG X, YOMANO L P, et al.Improving Escherichia coli FucO for furfural tolerance by saturation mutagenesis of individual amino acid positions[J].Applied and Environmental Microbiology, 2013, 79(10):3202-3208.
[54] LIAN J Z, SCHULTZ C, CAO M F, et al.Multi-functional genome-wide CRISPR system for high throughput genotype-phenotype mapping[J].Nature Communications, 2019, 10(1):5794.
[55] QIU Z Y, FANG C, GAO Q Q, et al.A short-chain dehydrogenase plays a key role in cellulosic D-lactic acid fermentability of Pediococcus acidilactici[J].Bioresource Technology, 2020, 297:122473.
[56] QIU Z Y, FANG C, HE N L, et al.An oxidoreductase gene ZMO1116 enhances the p-benzoquinone biodegradation and chiral lactic acid fermentability of Pediococcus acidilactici[J].Journal of Biotechnology, 2020, 323:231-237.
[57] QIU Z Y, HAN X S, HE J L, et al.One-pot d-lactic acid production using undetoxified acid-pretreated corncob slurry by an adapted Pediococcus acidilactici[J].Bioresource Technology, 2022, 363:127993.
[58] MLADENOVI D, PEJIN J, KOCI
D, PEJIN J, KOCI -TANACKOV S, et al.Enhanced lactic acid production by adaptive evolution of Lactobacillus paracasei on agro-industrial substrate[J].Applied Biochemistry and Biotechnology, 2019, 187(3):753-769.
-TANACKOV S, et al.Enhanced lactic acid production by adaptive evolution of Lactobacillus paracasei on agro-industrial substrate[J].Applied Biochemistry and Biotechnology, 2019, 187(3):753-769.
[59] QI W, LI X X, GUO Y H, et al.Integrated metabonomic-proteomic analysis reveals the effect of glucose stress on metabolic adaptation of Lactococcus lactis ssp.lactis CICC23200[J].Journal of Dairy Science, 2020, 103(9):7834-7850.
[60] XIAO D, HU C X, XU X Z, et al.A D,L-lactate biosensor based on allosteric transcription factor LldR and amplified luminescent proximity homogeneous assay[J].Biosensors &Bioelectronics, 2022, 211:114378.