肽聚糖(peptidoglycan,PG)广泛存在于原核生物当中,作为细胞的“骨架”,为维持细胞形状和完整性起到关键作用[1]。肽聚糖是革兰氏阴性菌和革兰氏阳性菌细胞壁中唯一共有的成分,且在革兰氏阳性细胞的细胞壁中占有更大比例[2-3]。肽聚糖是一个动态的高分子物质[4],其合成过程十分繁琐,需要多种蛋白质和酶参与调控,不同类型细菌的肽聚糖所涉及的生物合成方式也有所不同,这是肽聚糖结构复杂的直接原因[5-6]。复杂的结构和肽聚糖繁多的功能,在药物研发[7]、生物信号[8]以及生物材料[9]等领域都是经久不衰的研究热点[10]。
课题组前期研究发现,乳酸菌(lactic acid bacteria,LAB)对河豚毒素具有消减作用,发现肽聚糖是乳酸菌消减河豚毒素(tetrodotoxin,TTX)的关键组分,并且不同类型乳酸菌对TTX的消减效果具有差异,推测可能是肽聚糖类型不同的影响[11-12]。由于现有的肽聚糖提取方法时间长,存在一定的安全隐患,并且未考虑不同类型肽聚糖带来的差异性,为后续试验带来一定的困扰。因此,本研究旨在以3种肽聚糖结构不同的菌株[1][粪肠球菌(Enterococcus faecalis,EF):A3α,植物乳杆菌(Lactobacillus plantarum,N115):A1γ,棒状乳杆菌(Lactobacillus coryniformis,I3):A4α]为研究对象,对磷壁酸、脂质和蛋白质的去除方法进行优化和改良,建立高纯度肽聚糖的提取制备方法,并进一步通过定量检测和毒理实验考察了纯化后的肽聚糖对TTX含量和毒性的消减效果,为肽聚糖的工业化制备及其对河豚毒素的毒性消减作用机制研究提供基础数据和科学依据。
1 材料与方法
1.1 材料与设备
EF、N115、I3由上海海洋大学分子生物学实验室保藏;TTX标准品(>99%),上海阿拉丁生化科技股份有限公司;三氯乙酸(trichloroacetic acid,TCA)、十二烷基硫酸钠(sodium dodecyl sulfate,SDS),上海生工生物有限公司;链酶蛋白酶,北京索莱宝科技有限公司;胰蛋白酶,Sigma-Aldrich中国公司。
DF-101S集热式恒温加热磁力搅拌器,上海力辰邦西仪器科技有限公司;CR-GIII高速冷冻离心机, 日本日立公司;HS-3垂直混合器,宁波新芝生物科技股份有限公司;PL2002电子天平,瑞士梅特勒托利多仪器(上海)有限公司;QB-8002微孔板振荡器,海门其林贝尔仪器制造有限公司。
1.2 实验方法
1.2.1 灭活乳酸菌体的制备
选取冻存的EF、N115和I3三种乳酸菌,在MRS培养基中活化3次后,于MRS肉汤中培养至对数期,沸水浴1 h灭活,PBS洗涤3次后离心取沉淀备用(6 000 r/min,10 min)。
1.2.2 肽聚糖纯化方法优化
1.2.2.1 磷壁酸的去除条件优化及含量测定
称取菌泥0.1 g(湿重)分别进行以下处理:加入5 mL 2%(体积分数)曲拉通X-114溶液(50 mmol/L pH 6.5 Tris-HCl),4 ℃搅拌过夜[13];加入5 mL的正丁醇,25 ℃搅拌30 min[14];加入5 mL 100 g/L TCA溶液,沸水浴1 h[15]。经过上述处理后的菌泥用去离子水洗涤3次,离心取沉淀(6 000 r/min,10 min),采用钼酸铵法测定样品中的磷含量,每组平行3次。
1.2.2.2 脂质的去除试剂优化及含量测定
称取菌泥0.1 g(湿重)分别进行以下处理:加入5 mL 50 g/L SDS溶液,混匀后沸水浴25 min,离心后重悬于5 mL 50 g/L SDS溶液,沸水浴15 min,去离子水洗涤至无泡沫[3];加入5 mL V(乙酸钠)∶V(氯仿)∶V(甲醇)=4∶5∶10溶液,37 ℃振荡24 h;加入5 mL 丙酮,37 ℃振荡24 h。采用香草醛法测定样品中的脂质含量,每组平行3次。
1.2.2.3 蛋白质的酶解方法优化及含量测定
称取菌泥0.1 g(湿重)分别进行以下处理:加入5 mL 3 mg/mL胰蛋白酶溶液(0.1 mol/L,pH 7.5,Tris-HCl),37 ℃摇床振荡8 h;加入5 mL 1 mg/mL 链酶蛋白酶溶液(0.1 mol/L,pH 7.5,Tris-HCl,5 g/L SDS),60 ℃孵育2 h;使用两种酶联合处理。采用BCA蛋白定量试剂盒测定样品中的蛋白质含量,每组平行3次。
1.2.3 肽聚糖制备
参照本研究优化后的方法,向灭活菌体中加入100 g/L TCA溶液,沸水浴1 h。取沉淀加入50 g/L SDS溶液,沸水浴25 min,沉淀重悬于50 g/L SDS溶液中,沸水浴15 min,洗涤至无泡沫。加入1 mg/mL 链酶蛋白酶溶液,60 ℃孵育2 h,再向沉淀加入5 mL 3 mg/mL胰蛋白酶溶液,37 ℃摇床振荡8 h。透析72 h,冻干后备用。
1.2.4 肽聚糖表征
1.2.4.1 肽聚糖特异性表征
取肽聚糖标准品、EF(A3α)、N115(A1γ)、I3(A4α)的肽聚糖冻干样,配制成1 mg/mL的溶液,测定200~700 nm处紫外吸收波长。向肽聚糖中加入200 μg/mL溶菌酶溶液(0.01 mol/L,pH 6.2 PBS)至终质量浓度为1 mg/mL,置于37 ℃摇床,间隔1 h测定450 nm处的吸光度,绘制变化曲线。
1.2.4.2 肽聚糖的结构表征
将肽聚糖冻干粉喷金后,置于扫描电子显微镜(scanning electron microscope,SEM)下观察。
1.2.5 肽聚糖对TTX的消减率测定
将TTX标准品用0.1%(体积分数)乙酸溶液配制成1 mg/mL的溶液,将5 mg 灭活菌株和肽聚糖冻干样与20 μg TTX混合至总体积为1 mL,37 ℃恒温摇床(200 r/min)混合消减1 h,离心取上清液(12 000 r/min,3 min),参考国标GB 5009.206—2016《食品安全国家标准 水产品中河豚毒素的测定》进行竞争性酶联免疫吸附实验(enzyme-linked immunosorbent assay, ELISA)实验。
竞争性ELISA样品中TTX的抑制率和消减率按照公式(1)和公式(2)计算:
抑制率![]()
(1)
消减率![]()
(2)
式中:A阳性,阳性孔的OD值;A样品,样品孔的OD值;A阴性,阴性孔的OD值。
1.2.6 肽聚糖对TTX毒性的消减效果
动物实验采用昆明小鼠(19~21 g),购自上海杰思捷实验动物有限公司(中国上海)。所有实验程序均按照《实验动物护理和使用指南》进行。小鼠在实验前喂养3 d,在控制温度(20~25 ℃)下自由饮水。TTX毒性评价采用GB 5009.206—2016《食品安全国家标准 水产品中河豚毒素的测定》内的小鼠生物法。各实验组3只小鼠,后腹腔分别注射灭活菌株和肽聚糖处理TTX后的上清液1 mL。记录注射起始时间和小鼠死亡时间。TTX毒性根据各组平均死亡时间测定,平均死亡时间从注射后至小鼠停止呼吸计算。
1.2.7 数据处理与分析
实验结果采用平均值±标准偏差表示,由SPSS Statistics 23.0软件进行显著性分析,由Graphpad Prism 9.0.0软件进行制图。
2 结果与分析
2.1 肽聚糖的提取纯化工艺优化
2.1.1 磷壁酸的去除条件优化
研究表明,氢氟酸处理是常用的去除细菌中磷壁酸的方法[3],但氢氟酸腐蚀性强且极易挥发,存在安全隐患。为了找到更为安全且适用于多种类型肽聚糖的去除磷壁酸试剂,以3种具有不同类型肽聚糖的乳酸菌(EF:A3α型;N115:A1γ型、I3:A4α型)中磷含量为指标,考察了3种较为温和的试剂(100 g/L TCA,正丁醇,曲拉通X-114)对磷壁酸去除效果的影响。如图1所示,采用100 g/L TCA处理1 h后的3种菌株肽聚糖的磷含量降低最为显著,比对照组降低了93.5%以上;而正丁醇和曲拉通处理组的肽聚糖磷含量残留较多,下降范围为2.18%~45.27%。因此,本实验选取100 g/L TCA沸水浴处理1 h作为磷壁酸去除条件。
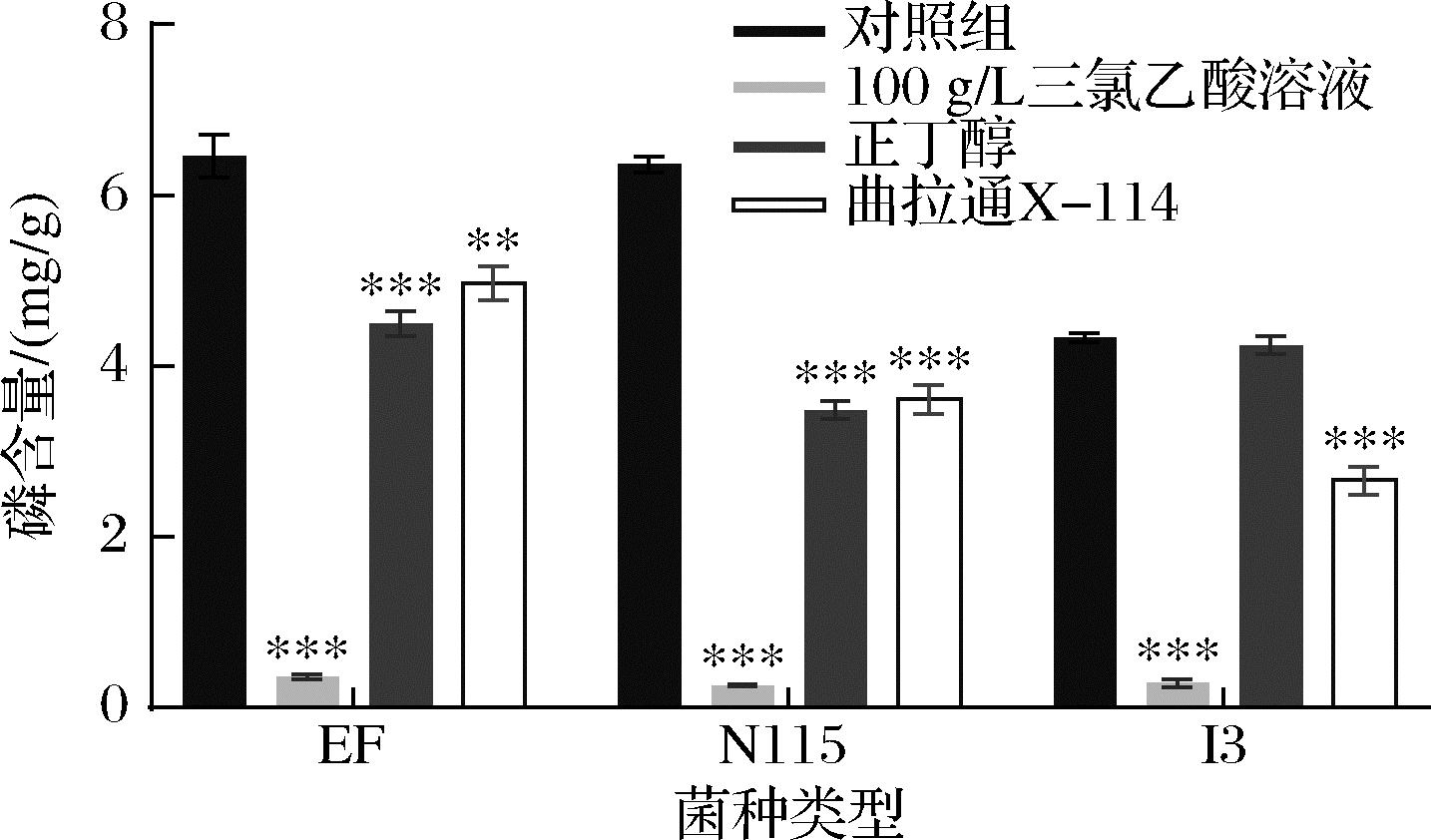
图1 不同处理方式对3种乳酸菌肽聚糖中磷含量的影响
Fig.1 Effect of different treatment methods on phosphorus content in peptidoglycan of three LAB strains
注:所有数据均表示为平均值±SD(n=3),**表示具有显著统计学 差异(P<0.01), ***表示具有极显著统计学差异(P<0.001)(下同)。
2.1.2 脂质的去除试剂优化
研究显示混合有机试剂[V(乙酸钠)∶V(氯仿)∶V(甲醇)=4∶5∶10]可以去除菌体中的脂质,但其处理过程耗时长,且氯仿试剂较为危险[16]。因此,本研究选择了相对安全的50 g/L SDS和在脱脂中广泛使用的丙酮作为实验组进行比较。结果如图2所示,经过50 g/L SDS、丙酮以及混合试剂处理后,3种肽聚糖的脂质含量降至54.56~78.09 mg/g,其中50 g/L SDS处理对菌体的脂质去除率为64.13%~70.68%。表明3种试剂的脂质去除效果无显著差异,且对不同类型肽聚糖中的脂质均有较好的去除效果。但丙酮和混合有机试剂均需处理24 h,而50 g/L SDS仅需处理40 min,故选取50 g/L SDS作为去除脂质的方法。
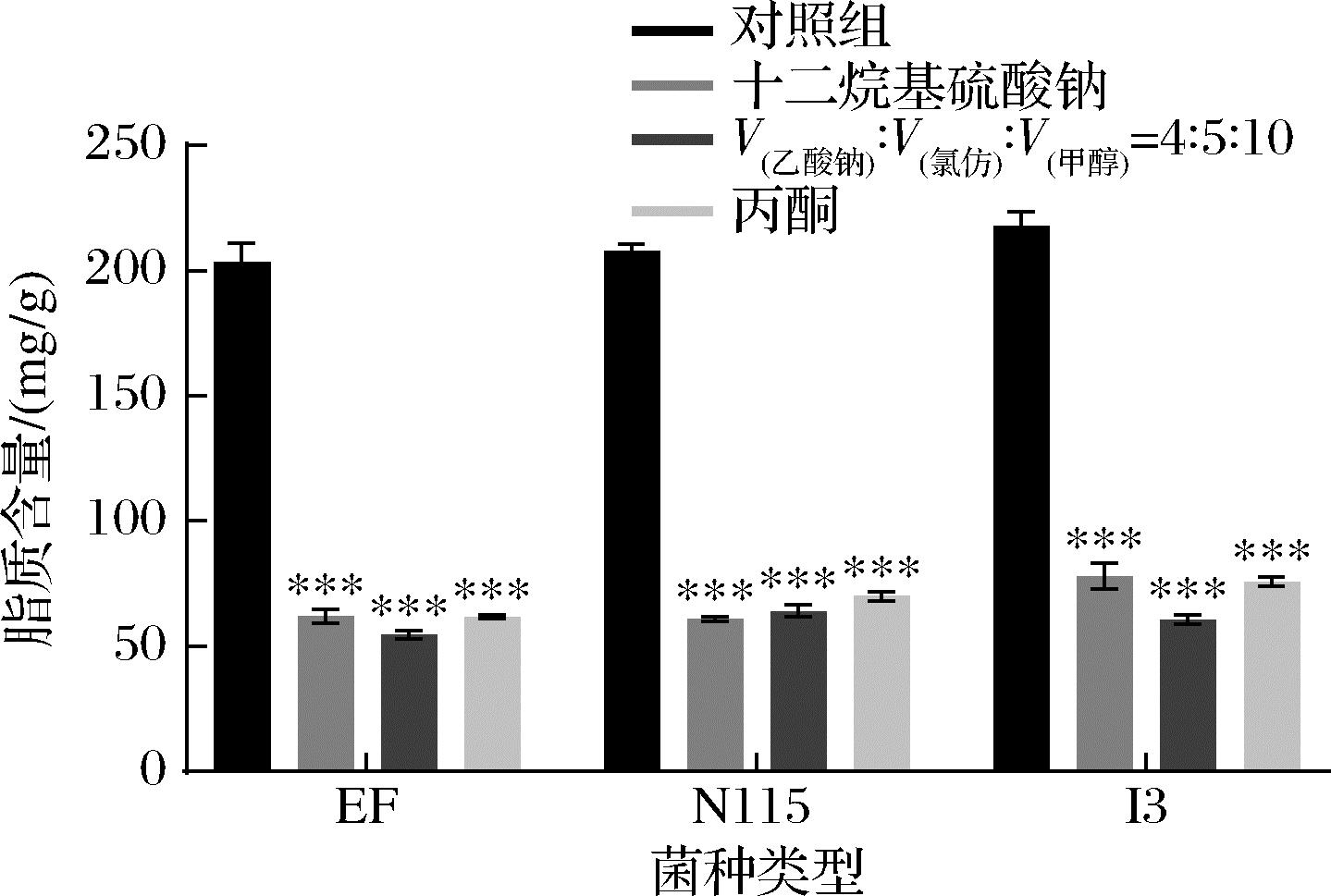
图2 不同试剂对3种乳酸菌肽聚糖中脂质含量的影响
Fig.2 Effects of different reagents on the lipid content of peptidoglycan in three LAB strains
2.1.3 蛋白质的酶解方法优化
胰蛋白酶和链霉蛋白酶是目前常用的去除肽聚糖上共价结合蛋白的2种酶[17]。胰蛋白酶作为一种肽链内切酶,可以特异性的作用于赖氨酸和精氨酸上的羧基,而链霉蛋白酶则作为一种复合酶,非特异性的切断肽键。本研究对比了胰蛋白酶、链霉蛋白酶以及2种酶的联合使用对蛋白质的去除效果结果如图3所示。可以看出对于EF和I3,两种酶联合处理后的蛋白质含量最低,分别为416.72 mg/g和340.29 mg/g,下降了73.28%和67.03%;而N115的联合处理后蛋白质含量(475.87 mg/g)却与单独酶处理后的蛋白质含量接近,无显著性区别;由此可见,在选择酶的种类时需要考虑肽聚糖类型的差异。总体来说,两种蛋白酶的复合处理对肽聚糖中蛋白质的去除率优于单一酶处理方式。
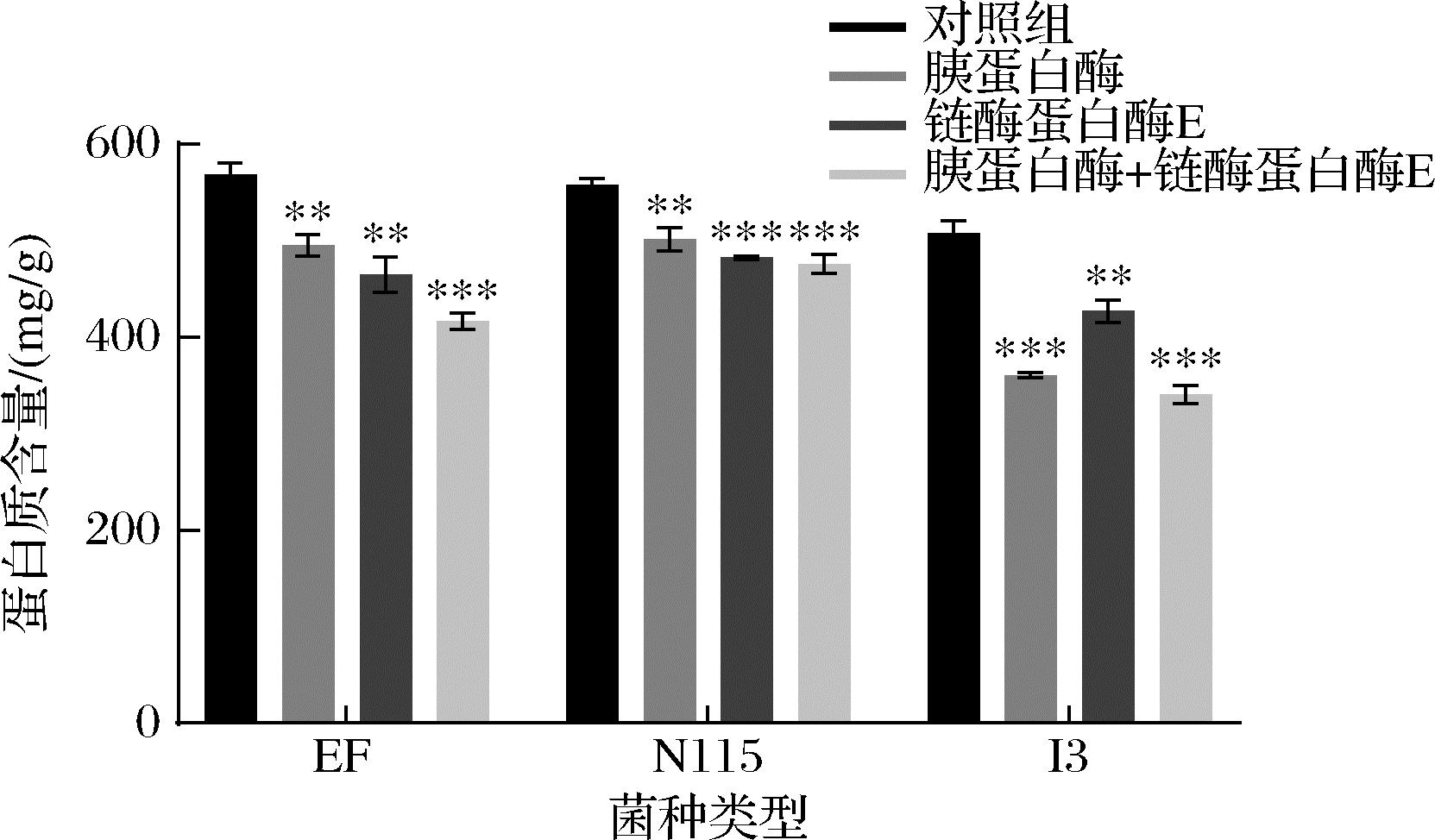
图3 不同酶解方式对3种乳酸菌肽聚糖中 蛋白质含量的影响
Fig.3 Effect of different enzymatic hydrolysis methods on the protein content of peptidoglycan in three LAB strains
2.2 三种肽聚糖的鉴定及其生物学活性分析
2.2.1 肽聚糖的纯度分析与鉴定
为验证去除磷壁酸、脂质和结合蛋白后的肽聚糖纯度,制备的3种肽聚糖样品与肽聚糖标准品的紫外吸收波谱结果如图4-a所示,3种乳酸菌的肽聚糖与肽聚糖标品的峰形在200~700 nm内基本一致,分别在230、260 nm处有特征吸收峰,无其他杂质吸收峰,表明采用优化后制备的肽聚糖纯度较高。
溶菌酶可以断裂肽聚糖糖链上的β-1,4糖苷键,因此可以通过溶菌酶实验确定肽聚糖的糖链在提纯过程中是否被破坏,如图4-b所示,3种肽聚糖在溶菌酶处理后,其在450 nm处的吸光值在30 min内显著下降,在1~4 h后趋于平缓,且溶液逐渐澄清,表明肽聚糖样品已被完全酶解。由此可见,制备所得3种肽聚糖的糖链结构完整。
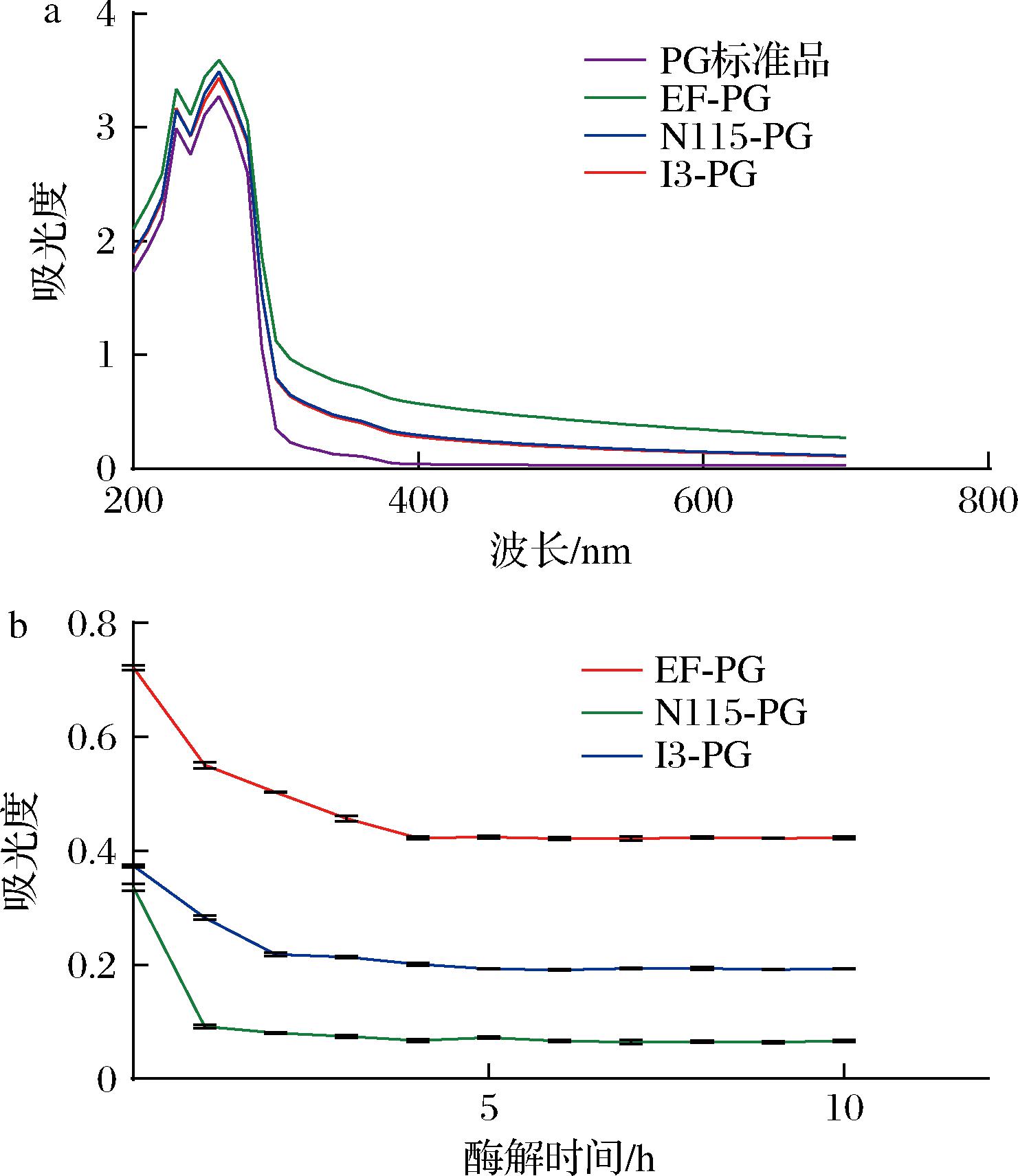
a-紫外全波长扫描图;b-溶菌酶酶解实验图
图4 纯化后肽聚糖的结构分析与鉴定
Fig.4 Structure analysis and identification of purified peptidoglycan
纯化制备的A3α型、A1γ型和A4α型肽聚糖的SEM图像如图5所示,3种肽聚糖均呈现较为统一的球状形态,完整性较好,表明肽聚糖提取时采用的TCA、SDS和复合蛋白酶处理不会对肽聚糖的球壳外形造成明显损伤,推测优化建立的提取纯化方法对肽聚糖的活性影响应该比较小。
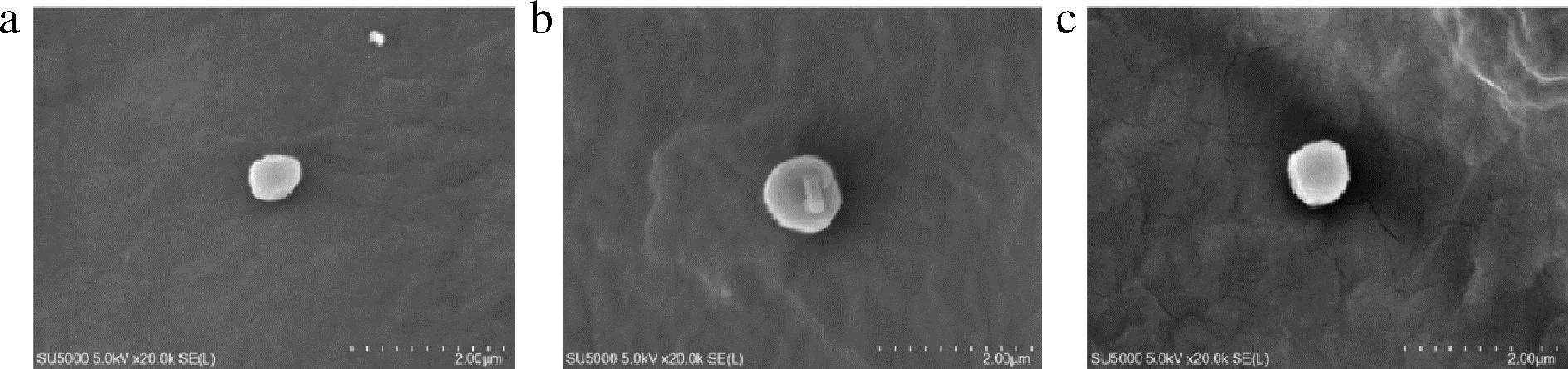
a-EF-PG;b-N115-PG;c-I3-PG
图5 三种乳酸菌肽聚糖的SEM图像
Fig.5 SEM images of three types of LAB peptidoglycans
2.2.2 肽聚糖对TTX含量的影响作用
前期研究发现肽聚糖是乳酸菌消减TTX的关键组分[11],为考察肽聚糖的提取纯化工艺是否会影响乳酸菌对TTX的消减作用,本研究采用ELISA分析了3种灭活菌体和肽聚糖分别处理前后的TTX含量变化。如图6-a所示,TTX经3种灭活菌体处理后,其含量下降幅度为81.20%~85.06%,经3种肽聚糖处理后的下降范围为78.51%~81.88%,由此可见,纯化后的3种肽聚糖对TTX的消减作用与乳酸菌处理效果接近,表明优化后的提取纯化工艺不影响肽聚糖的生物活性。
2.2.3 肽聚糖对TTX毒性的消减效果
经过灭活乳酸菌和肽聚糖处理后TTX的含量明显下降,为进一步评价肽聚糖对TTX毒性的影响作用,本研究利用小鼠生物法分析了3种灭活乳酸菌和肽聚糖对TTX的毒性消减作用,如图6-b所示,灭活后的3株乳酸菌对TTX毒性的消减效果最佳的是I3菌株,消减率为87.65%,其肽聚糖(A4α型)对TTX的毒性消减效果也最好,消减率为93.02%,另外2种肽聚糖(A3α和A1γ型)对TTX毒性的消减效果也较好,分别为87.30%和89.42%。综上,制备所得3种肽聚糖对TTX的毒性和含量的消减效果均较好。
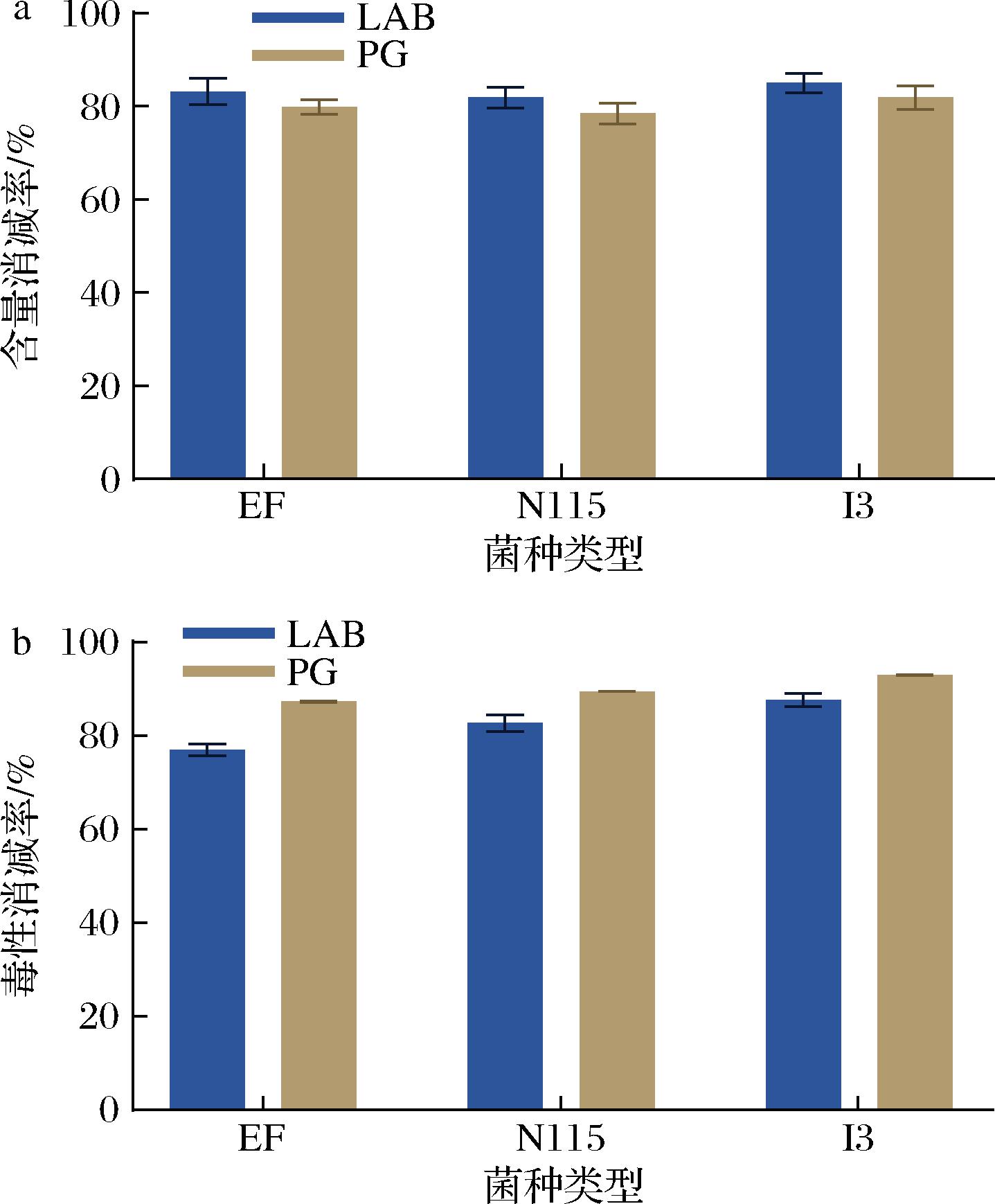
a-含量消减率;b-TTX毒性消减率
图6 灭活乳酸菌和肽聚糖对TTX的消减效果
Fig.6 Removal effect of inactivated LAB and peptidoglycan on TTX
3 讨论与结论
目前已有许多肽聚糖提取相关的研究,PASQUINA-LEMONCHE等[3]使用氢氟酸去除磷壁酸,这种方法对肽聚糖结构的损伤较小,但氢氟酸具有强挥发性和腐蚀性,危险性较大。SCHLEIFER等[1]首先使用TCA去除乳酸菌细胞壁上的磷壁酸,TCA较氢氟酸安全性更高,但长时间的TCA处理会损伤肽聚糖的结构。为减少TCA对肽聚糖结构的破坏,付艳茹等[18]探究了不同质量分数的TCA以及不同处理时间和温度对乳酸菌细胞壁中磷壁酸的去除效果,结果发现100 g/L TCA在70 ℃下处理1 h可以达到最佳的去除效果,在提高TCA对磷壁酸去除效率的同时减少了对肽聚糖结构的破坏。本研究在此基础上用100 g/L TCA对不同类型的肽聚糖进行处理,发现100 g/L TCA处理对不同类型肽聚糖中的磷壁酸均有显著去除效果。除此之外,还有一些新型的去除磷壁酸的试剂[19],如曲拉通X-114和正丁醇,本实验选用了这两种新型试剂与TCA进行对比实验,结果表明在用量相同的情况下,曲拉通X-114和正丁醇并不能达到100 g/L TCA的去除效果,但这两种处理方法更加温和,依然具有一定的应用前景。
SDS、丙酮以及氯仿混合溶剂均为去除脂质的常用试剂,其中SDS[20]和氯仿混合试剂[21]已经在肽聚糖的提取中得到应用,而丙酮还尚未有相关报道。本研究发现3种试剂在相同用量下表现出类似的脂质去除效果,但SDS对比氯仿和丙酮具有较高的安全性,且处理时间最短,因此选择SDS作为肽聚糖提取过程中的脂质去除试剂。不同种类的蛋白酶对肽聚糖上结合蛋白的去除效果已有很多报道,杨媛等[22]使用胰蛋白酶和α-糜蛋白酶对嗜酸乳杆菌细胞壁上的结合蛋白进行酶解来提纯肽聚糖。还有研究比较了胰酶、胰酶加碱性蛋白酶和胰酶加核酶3种处理方法对婴儿双歧杆菌的蛋白去除效果,结果发现胰酶加碱性蛋白酶为最佳处理组合[18]。但这些研究仅使用了一种乳酸菌进行研究,并未考虑肽聚糖种类不同的情况下不同的蛋白酶选择,本研究以3种肽聚糖类型不同的乳酸菌为研究对象,发现肽聚糖类型会显著影响蛋白酶的酶解效果。因此,在肽聚糖提取过程中应充分考虑肽聚糖类型所带来的差异,选用最合适的蛋白酶进行处理,或直接选用复合酶处理。
确定TCA-SDS-复合酶处理工艺后,本研究利用该方法提取制备了3种不同类型的肽聚糖,并分别分析了它们对TTX含量和毒性的消减效果。如图7所示,肽聚糖主要是由糖链和肽尾组成的,部分肽聚糖还会出现肽桥,且不同类型肽聚糖的差异主要体现在肽尾和肽桥的部分;A4α肽聚糖单体的肽桥上有1个天冬氨酸,使该类肽聚糖含有较多的游离氨基[23];而另外两种肽聚糖中,A3α肽聚糖的肽桥上含有5个相连的甘氨酸,无游离活性基团[24];A1γ肽聚糖则没有肽桥[25]。本研究显示不同类型的肽聚糖对TTX的消减效果都较好,但其中I3菌株的A4α型肽聚糖对TTX的含量和毒性均有着最高的消减率(85.06%和93.02%)。因此,推测肽聚糖对TTX的消减效果很可能与其含有的游离基团有关,A4α肽聚糖相比另外两种肽聚糖拥有更多的游离活性氨基,这可能是其对TTX消减效果最好的原因所在。现有研究已经探明TTX是一种两性内盐,其活性基团为胍基和羟基,两者在溶液中分别带正电和负电[11]。3种肽聚糖的肽尾末端丙氨酸(D-Ala)含有带负电的游离羧基、糖链上含有大量的带负电羟基,以及A4α肽聚糖上天冬氨酸含有的带正电氨基,这些基团可能通过静电作用等方式促进肽聚糖与TTX的相互结合,从而使肽聚糖对TTX产生消减作用,目前肽聚糖与TTX的相互作用方式及其作用位点尚不清晰,有待今后深入研究。
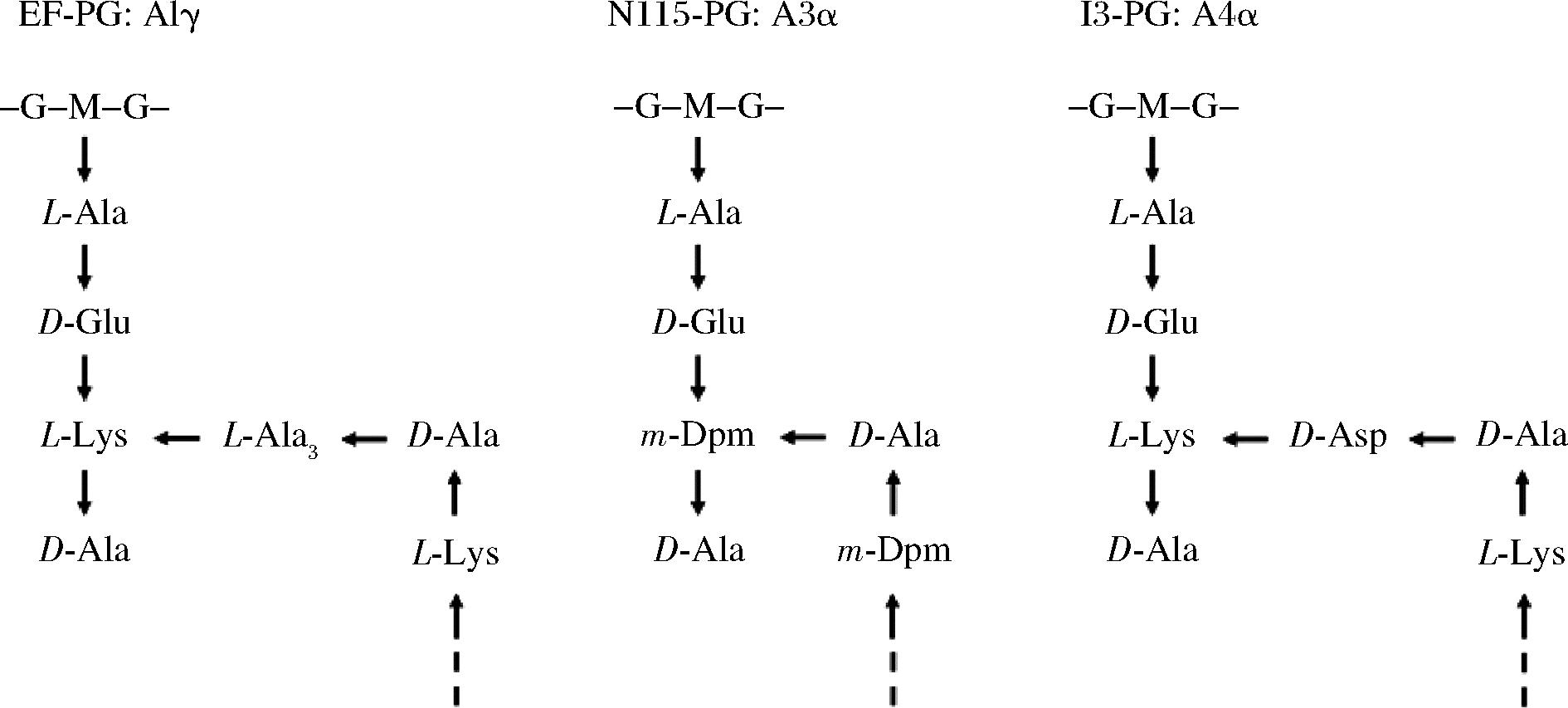
图7 三种肽聚糖的单体分子结构图
Fig.7 Molecular structure diagram of three types of peptidoglycan monomers
综上,本研究通过对肽聚糖提取过程中去除磷壁酸、脂质和蛋白质的工艺优化,选择低毒性的TCA作为去除磷壁酸的试剂,引入胰蛋白酶和链霉蛋白酶的复合酶系统去除肽聚糖上的共价结合蛋白质,并确保整个提取过程的安全性,建立的提取纯化方法可以获得高纯度、结构完整和高活性肽聚糖,并且适用于多种类型的肽聚糖。此外,研究发现制备的3种乳酸菌肽聚糖对TTX的消减效果非常显著,其含量去除率>78%,毒性消减率>87%。本研究为未来肽聚糖的产业化制备及其对TTX的毒素消减作用机制研究提供了方法支撑和科学依据。
[1] SCHLEIFER K H, KANDLER O.Peptidoglycan types of bacterial cell walls and their taxonomic implications [J].Bacteriological Reviews, 1972, 36(4):407-477.
[2] PULS J S, BRAJTENBACH D, SCHNEIDER T, et al.Inhibition of peptidoglycan synthesis is sufficient for total arrest of staphylococcal cell division[J].Science Advances, 2023, 9(12):eade9023.
[3] PASQUINA-LEMONCHE L, BURNS J, TURNER R D, et al.The architecture of the Gram-positive bacterial cell wall [J].Nature, 2020, 582(7811):294-297.
[4] BROGAN A P, RUDNER D Z.Regulation of peptidoglycan hydrolases:Localization, abundance, and activity[J].Current Opinion in Microbiology, 2023, 72:102279.
[5] VAIWALA R, SHARMA P, PURANIK M, et al.Developing a coarse-grained model for bacterial cell walls:Evaluating mechanical properties and free energy barriers[J].Journal of Chemical Theory and Computation, 2020, 16(8):5369-5384.
[6] MASHAYEKH S, BERSCH K L, RAMSEY J, et al.Synthesis of bacterial-derived peptidoglycan cross-linked fragments[J].The Journal of Organic Chemistry, 2020, 85(24):16243-16253.
[7] MORAES G L, GOMES G C, MONTEIRO DE SOUSA P R, et al.Structural and functional features of enzymes of Mycobacterium tuberculosis peptidoglycan biosynthesis as targets for drug development[J].Tuberculosis, 2015, 95(2):95-111.
[8] TOSONI G, CONTI M, DIAZ HEIJTZ R.Bacterial peptidoglycans as novel signaling molecules from microbiota to brain[J].Current Opinion in Pharmacology, 2019, 48:107-113.
[9] ZHANG L Z, LIANG J H, JIANG C Y, et al.Peptidoglycan-inspired autonomous ultrafast self-healing bio-friendly elastomers for bio-integrated electronics[J].National Science Review, 2020, 8(5):nwaa154. [10] 徐艳. 游离放线菌发酵废渣中肽聚糖的提取、纯化与活性研究[D].杭州:浙江工业大学, 2017. XU Y.Studies on the extraction, purification and activities of peptidoglycan from Actinoplanes sp.fermentation residue[D].Hangzhou:Zhejiang University of Technology, 2017.
[11] 叶精勤, 肖叶, 阎俊, 等.鼠李糖乳杆菌对河豚毒素的免疫活性消减作用方式 [J].食品科学, 2021, 42(14):103-109. YE J Q, XIAO Y, YAN J, et al.Immunosuppressive effect and mode of action of Lactobacillus rhamnosus on tetrodotoxin [J].Food Science, 2021, 42(14):103-109.
[12] 丁婕. 河豚毒素毒性消减方法的初步研究[D].上海:上海海洋大学,2019. DING J.Preliminary study on toxicity reduction method of tetrodotoxin[D].Shanghai:Shanghai Ocean University,2019.
[13] SUTCLIFFE I C, HOGG S D.Extraction of lipoteichoic acid from Streptococcus mutants with the non-ionic detergent Triton X-114 [J].Journal of Microbiological Methods, 1993, 17(3):215-225.
[14] CISZEK-LENDA M, NOWAK B, SROTTEK M, et al.Further studies on immunomodulatory effects of exopolysaccharide isolated from Lactobacillus rhamnosus KL37C [J].Central European Journal of Immunology, 2013, 38:289-298.
[15] 李莹. 婴儿双歧杆菌完整肽聚糖提取及其理化特性的研究[D].呼和浩特:内蒙古农业大学, 2009. LI Y.Study on extracting technology and physicochemical characteristics of WPG from Bifidobacterium infantis[D].Hohhot:Inner Mongolia Agricultural University, 2009.
[16] 郑玉寅, 杨永芳, 丁国芳, 等.乌贼墨肽聚糖的制备工艺与体外抗前列腺癌研究[J].时珍国医国药, 2012, 23(1):111-113. ZHENG Y Y, YANG Y F, DING G F, et al.The peptidoglycan extracted from ink of Sepia and the inhibition against the growth of prostatic cancer in vitro[J].Lishizhen Medicine and Materia Medica Research, 2012, 23(1):111-113.
[17] KWAN J M C, QIAO Y A.Mechanistic insights into the activities of major families of enzymes in bacterial peptidoglycan assembly and breakdown[J].Chembiochem,2023,24(7):e202200693.
[18] 付艳茹,靳烨.完整肽聚糖提取工艺的研究[J].中国乳品工业,2009,37(4):12-15. FU Y R, JIN Y.Studies on the optimum extraction process of whole peptidoglycan [J].China Dairy Industry,2009,37(4):12-15.
[19] 关曼缇, 张英春, 卢卫红, 等.乳酸杆菌脂磷壁酸的结构及免疫调控功能研究进展[J].食品科学, 2021, 42(21):278-284. GUAN M T, ZHANG Y C, LU W H, et al.Progress in research on the structure and immune-regulating effect of Lactobacillus lipoteichoic acid[J].Food Science, 2021, 42(21):278-284.
[20] 杨旭, 朱静静, 潘道东, 等.嗜酸乳杆菌肽聚糖的六种提取方法比较研究[J].食品工业科技, 2016, 37(15):66-70;75. YANG X, ZHU J J, PAN D D, et al.Comparison of six extraction methods of peptidoglycan in Lactobacillus acidophilus[J].Science and Technology of Food Industry, 2016, 37(15):66-70;75.
[21] 陈佳妮, 黄占旺, 王素贞, 等.纳豆芽孢杆菌胞壁肽聚糖提取工艺优化及其结构初步鉴定[J].江西农业大学学报, 2019, 41(1):174-185. CHEN J N, HUANG Z W, WANG S Z, et al.Optimization of extraction process of peptidoglycan from cell wall of Bacillus natto and preliminary identification of its structure[J].Acta Agriculturae Universitatis Jiangxiensis, 2019, 41(1):174-185.
[22] 杨媛, 潘道东, 曾小群, 等.嗜酸乳杆菌胞壁肽聚糖的提取及结构分析[J].中国食品学报, 2014, 14(5):202-208. YANG Y, PAN D D, ZENG X Q, et al.Extraction and structure analysis of peptidoglycan in Lactobacillus acidophilus cell wall[J].Journal of Chinese Institute of Food Science and Technology, 2014, 14(5):202-208.
[23] ZHENG J S, WITTOUCK S, SALVETTI E, et al.A taxonomic note on the genus Lactobacillus:Description of 23 novel Genera, emended description of the genus Lactobacillus Beijerinck 1901, and union of Lactobacillaceae and Leuconostocaceae[J].International Journal of Systematic and Evolutionary Microbiology, 2020, 70(4):2782-2858.
[24] SCHLEIFER K H, KILPPER-BALZ R.Transfer of Streptococcus faecalis and Streptococcus faecium to the genus Enterococcus nom.rev.as Enterococcus faecalis comb.nov.and Enterococcus faecium comb.nov[J].International Journal of Systematic Bacteriology, 1984, 34(1):31-34.
[25] MATSUDA T, KOTANI S, KATO K.Structure of the cell walls of Lactobacillus plantarum, ATCC 8014.2.Cross linkage between D-alanine and alpha, alpha’-diaminopimelic acid in the cell wall peptidoglycans studied with an L-11 enzyme from Flavobacterium sp[J].Biken Journal, 1968, 11(2):127-138.