苯乳酸(phenyllactic acid, PLA)又称3-苯基乳酸/β-苯基乳酸,分子式为C9H10O3,是一种天然有机酸,首次于蜂蜜中鉴别出来,后被发现广泛存在于奶酪、泡菜、酸面团等发酵食物中[1]。PLA主要是由乳酸菌合成,如植物乳杆菌[2]、鲁氏乳杆菌[3]、乳酸片球菌[4]和布氏乳杆菌[5]等。此外,一些非乳酸菌菌株也具有合成PLA的能力,如凝结芽孢杆菌[6]、丙酸杆菌[7]和白地霉[8]。在微生物合成PLA途径中,L-苯丙氨酸经脱氨被催化为苯丙酮酸,然后再经乳酸脱氢酶加氢还原,生成PLA[9]。PLA对包括细菌和真菌在内的许多微生物具有广泛的抑菌作用[10],是天然的高效、无毒害作用的生物防腐剂。与以山梨酸钾和苯甲酸钠为代表的传统化学防腐剂相比,PLA具有更安全、更高效的优势,是未来食品工业应用的趋势[11]。PLA由于其亲水性而具有良好的水溶性,使其能够在食物系统中进行均匀扩散[12],而且PLA在酸性和高温环境下性质依然不容易改变[13]。因此,PLA在食品和饲料工业中有广泛的应用。例如,PLA应用于乳制品行业抑制李斯特氏菌的生长,能够延长乳制品的保质期[14]。再者,PLA还可以抑制肠道沙门氏菌的生长,这是许多食源性细菌性疾病的主要病原体,也是许多动物性食品污染的原因,如鸡蛋、牛肉、家禽和猪肉[15]。正因PLA在抑菌方面出色的性能,其抑菌机理引起了学者们的极大关注。
作为一种有潜力的防腐剂和抗菌剂,研究人员已经从细胞和分子水平上研究了PLA的抗菌机理。到目前为止,已发现PLA通过与多种靶点相互作用来抑制有害微生物的生长,主要包括对细胞壁、细胞膜和核酸的破坏作用[16]。黄云坡等[17]通过扫描电子显微镜在微观结构上观察到经PLA胁迫后的单增李斯特菌细胞形态发生变形,且细胞表面出现孔洞,证实了PLA的作用靶点之一是细胞壁。ZHOU等[18]通过凝胶阻滞实验验证了PLA能够通过与DNA结合破环DNA的结构。本研究在此基础上,利用荧光光谱方法,结合分子对接技术,系统分析了PLA与DNA的相互作用模式,并确定了两者之间的相互作用力。此外,为了验证PLA在胞内与DNA结合带来的基因转录变化,通过转录组测序对比了乳酸片球菌DY15在PLA胁迫下基因表达差异,挖掘了PLA作用的相关靶基因,探讨了PLA通过与DNA作用引起基因调控变化的抑菌机制,为PLA作为抗菌剂在食品领域的应用提供了理论支撑。
1 材料与方法
1.1 材料与仪器
乳酸片球菌DY15于37 ℃、180 r/min条件下在MRS培养基中生长。培养基成分为20 g/L葡萄糖、8 g/L牛肉提取物、10 g/L胰蛋白胨、5 g/L CH3COONa、4 g/L酵母提取物、2 g/L柠檬酸三铵、2 g/L K2HPO4、0.58 g/L MgSO4·7H2O、0.19 g/L MnSO4·H2O和1 mL/L吐温80,pH 6.5。加入20 g/L的琼脂粉即为固体培养基。L-PLA,国药沃凯公司;小牛胸腺DNA(ctDNA),Sigma-Aldrich公司,纯度检测符合要求A260/A280>1.8,用Tris-HCl缓冲液(pH 7.4)配制ctDNA母液,根据260 nm处的吸光度测定其终浓度为2.5 mmol/L[ε260nm=6 600 L/(mol·cm)]。L-PLA于双蒸水中溶解,终浓度为1 mmol/L,于4 ℃保存。其他试剂均为分析纯。
F-7000荧光分光光度计,日本日立公司;UV-1800紫外分光光度计,日本岛津公司;迷你金属浴,上海生工生物工程有限公司;ZQZY-CF9.9振荡培养箱,上海知楚仪器有限公司;LightCycler 480 II荧光定量PCR仪,上海罗氏制药有限公司。
1.2 PLA与DNA相互作用的测定
1.2.1 荧光光谱的测定
在7份Tris-HCl缓冲液(pH 7.4)体系中,加入L-PLA母液和ctDNA母液,并定容至1 mL,使PLA终浓度为5×10-5 mol/L,ctDNA终浓度分别为0、4×10-6、8×10-6、12×10-6、16×10-6、20×10-6、24×10-6 mol/L,在298 K恒温水浴中充分混合10 min,得到PLA-ctDNA二元体系。使用荧光分光光度计对二元体系进行荧光光谱扫描,荧光光谱的激发波长为260 nm,发射波长范围为270~400 nm,狭缝宽度为5 nm,扫描速度为100 nm/min,石英电池的路径长度为1 cm。改变水浴锅温度条件分别为310 K和320 K,并重复上述实验,以获得不同温度下的荧光光谱。
1.2.2 KI猝灭实验
取ctDNA(2×10-5 mol/L)和L-PLA(5×10-5 mol/L)-ctDNA(2×10-5 mol/L)溶液,分别加入KI溶液,使KI的终浓度分别为0、0.002、0.006、0.008、0.01 mol/L。充分混合5 min 后,使用1.2.1节中的方法在室温下进行光谱扫描。
1.2.3 DNA熔解曲线的测定
将ctDNA(2×10-5 mol/L)和L-PLA(5×10-5 mol/L)-ctDNA(2×10-5 mol/L)溶液分别置于20~95 ℃的环境中,每5 ℃设为1个梯度。10 min后,快速取出溶液并于紫外分光光度计中测量DNA的吸光度A258nm。根据公式(1)计算。
(1)
式中:A0,20 ℃时的A258nm,Af,95 ℃下的A258nm,A,实时温度下的A258nm。通过计算不同温度下的fss值,得到fss关于温度的曲线,在fss=0.5时对应的温度即为DNA的Tm。
1.3 PLA与DNA的分子对接模拟
使用AutoDock 4进行分子对接模拟,在ZINC小分子化合物数据库中下载L-PLA的晶体结构文件(ZINC ID:388089)。在蛋白质数据库中为ctDNA选择了晶体结构d(CGATCG)2(PDB ID:1nab)。先使用AutoDock 4对二者进行预处理(去水、加氢和添加电荷)。分子对接位置的活性区域包含整个DNA分子,活性区域大小为40 Å×40 Å×160 Å,晶格步长为160 Å,采用拉马克遗传算法(Lamarckian genetic algorithm,LGA)全面搜索可能的构象和结合位点,结合能最低的结构被认为是最佳的相互作用模式。
1.4 转录组测序与实时荧光定量PCR验证
将过夜培养的乳酸片球菌DY15以2%的比例分别接种于10 mL MRS液体培养基和10 mL MRS(含2 g/L L-PLA)液体培养基中,培养8 h后,收集菌体送至苏州金唯智生物技术公司进行转录组测序与基因注释。进行实时荧光定量PCR实验时,取上述菌体,通过Ultrapure RNA试剂盒(康为世纪)提取总RNA。使用HiFiScript gDNA Removal cDNA Synthesis试剂盒(康为世纪)将总共500 ng的总RNA逆转录成cDNA。然后在LightCycler 480 Ⅱ qPCR仪上进行荧光定量PCR实验。选取16S rRNA基因作为内参,并进行3次生物学重复。所有数据均采用2-ΔΔCt方法进行分析。具体引物序列见电子版增强出版附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.035182)。
2 结果与分析
2.1 荧光光谱法研究PLA与DNA的相互作用
2.1.1 ctDNA对PLA的荧光猝灭模型
图1显示了PLA随着ctDNA浓度的增加而产生的荧光发射光谱。PLA在260 nm处被激发后,在290 nm处有一个很强的荧光发射峰,随着DNA浓度的提高,PLA的荧光强度显著降低。结果表明,DNA能与PLA相互作用,猝灭PLA的特征荧光。
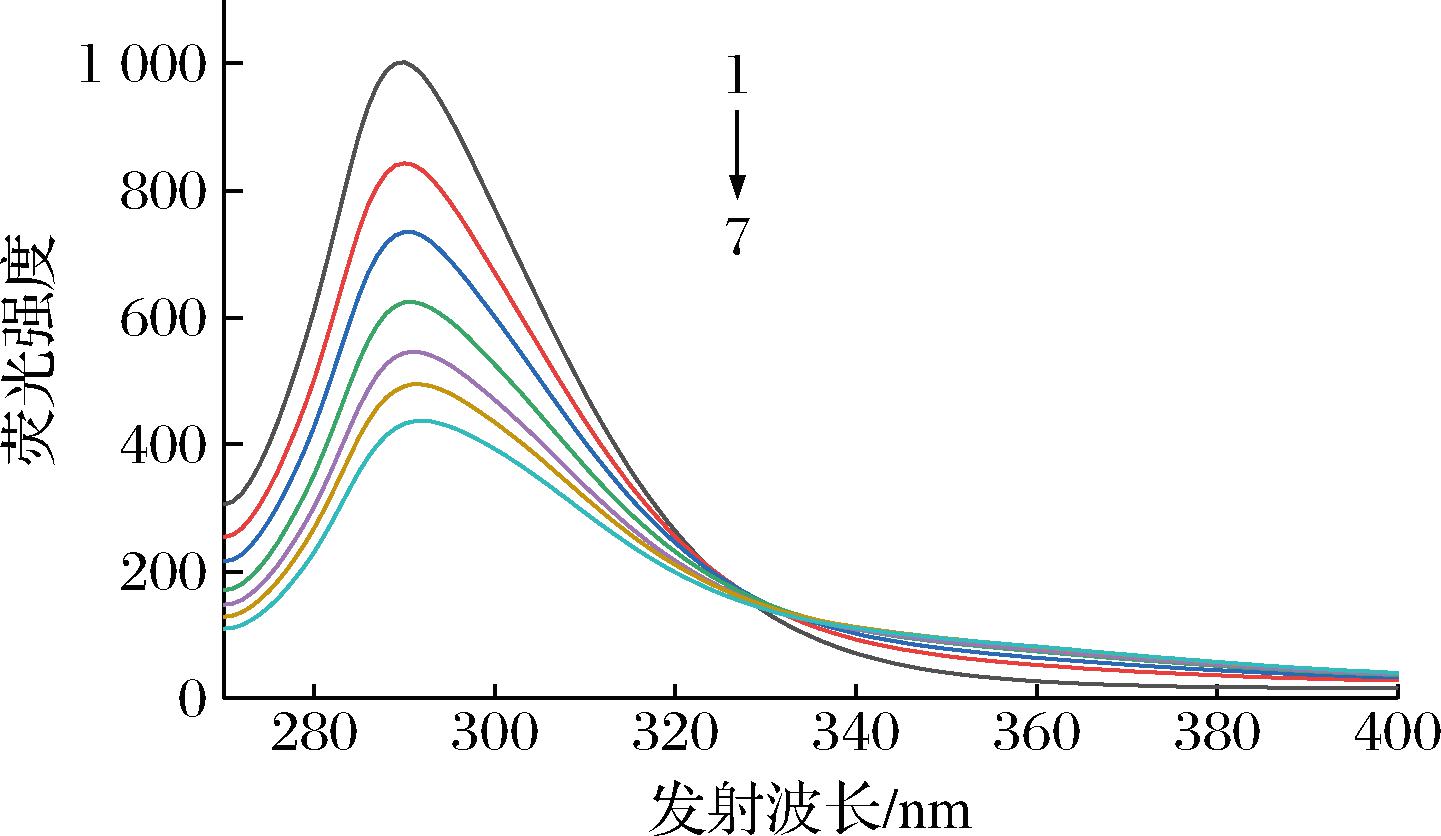
图1 添加ctDNA后PLA的荧光猝灭变化
Fig.1 Changes in fluorescence quenching of PLA after the addition of ctDNA
注:CPLA=5×10-5 mol/L;1-7:CctDNA=0,4,8,12, 16,20,24×10-6 mol/L。
为了研究DNA对PLA的荧光猝灭机理,用Stern-Volmer方程[19]F0/F=1+Kqτ0[DNA]=1+Ksv[DNA]进行数据处理。其中,F0和F为PLA在无猝灭剂和有猝灭剂(DNA)存在时的稳态荧光强度;Kq为生物分子的猝灭速率常数;τ0为没有猝灭剂的荧光团的平均寿命,它的值是10-8 s;[DNA]为猝灭剂的浓度;Ksv为Stern-Volmer猝灭常数。
图2显示了Stern-Volmer方程拟合的F0/F与CctDNA的关系曲线。由该曲线的斜率可以得到PLA在ctDNA体系中的猝灭常数Kq和Ksv。数据表明,Ksv随着温度的升高而降低,这表明猝灭不是由碰撞引起的,而是由络合物的形成引起的。此外,如表1所示,猝灭速率常数Kq远大于2×1010 L/(mol·s),这是各种猝灭剂与生物聚合物的最大散射碰撞猝灭常数。这些证据表明PLA-ctDNA的荧光猝灭机制是静态猝灭而不是动态猝灭。
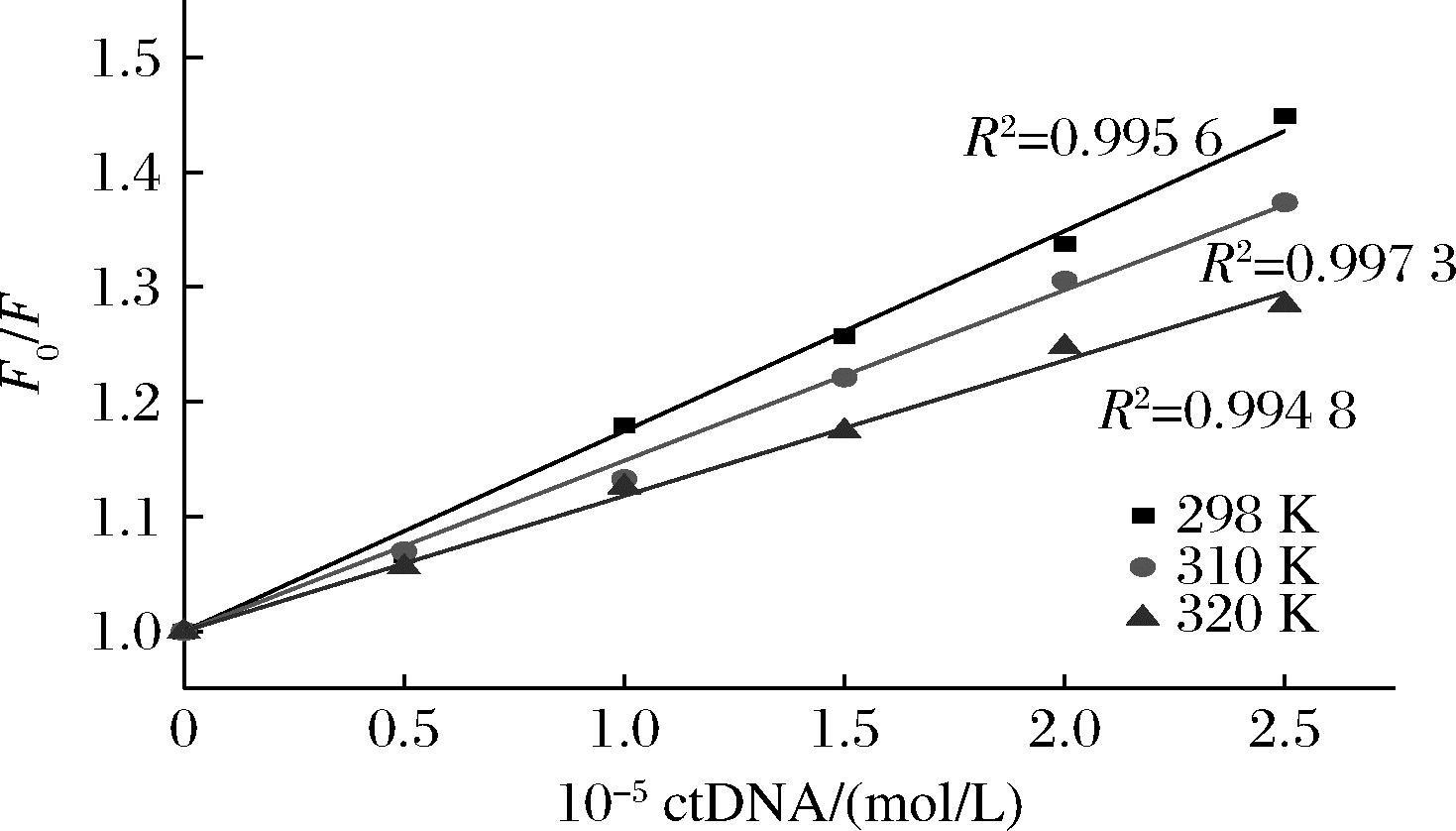
图2 不同温度下ctDNA对PLA荧光猝灭的 Stern-Volmer拟合直线
Fig.2 Stern-Volmer plots for ctDNA quenching of PLA fluorescence at different temperatures
2.1.2 结合常数与结合位点数
根据Lineweaver-Burk对数方程lg[(F0-F)/F]=lgKa+nlg[DNA][20],确定了PLA与ctDNA的结合常数Ka与结合位点数n。
由线性拟合方程式的截距得Ka,斜率为n(图3),不同温度下的Ka与n的计算数值列于表1。在不同温度下,PLA和ctDNA的结合位点数均大于1,表明在ctDNA上至少有一个PLA结合部位。此外,结合位点数随着温度的升高而降低,Ka也随温度的升高而降低,与Ksv对温度的依赖关系一致。这表明,升高温度不利于PLA的结合。由于本文的实验温度远低于ctDNA的Tm值,ctDNA应始终保持双螺旋结构。因此,Ka随温度升高而降低的原因不是由于ctDNA的变性,而是PLA与ctDNA的结合过程是放热反应,温度升高,反应平衡左移,结合常数减小。T=298 K时,Ka值为8.57×104,而经典DNA嵌入剂溴化乙啶的结合常数Ka=1.4×106,证明PLA结合能力低于溴化乙啶。
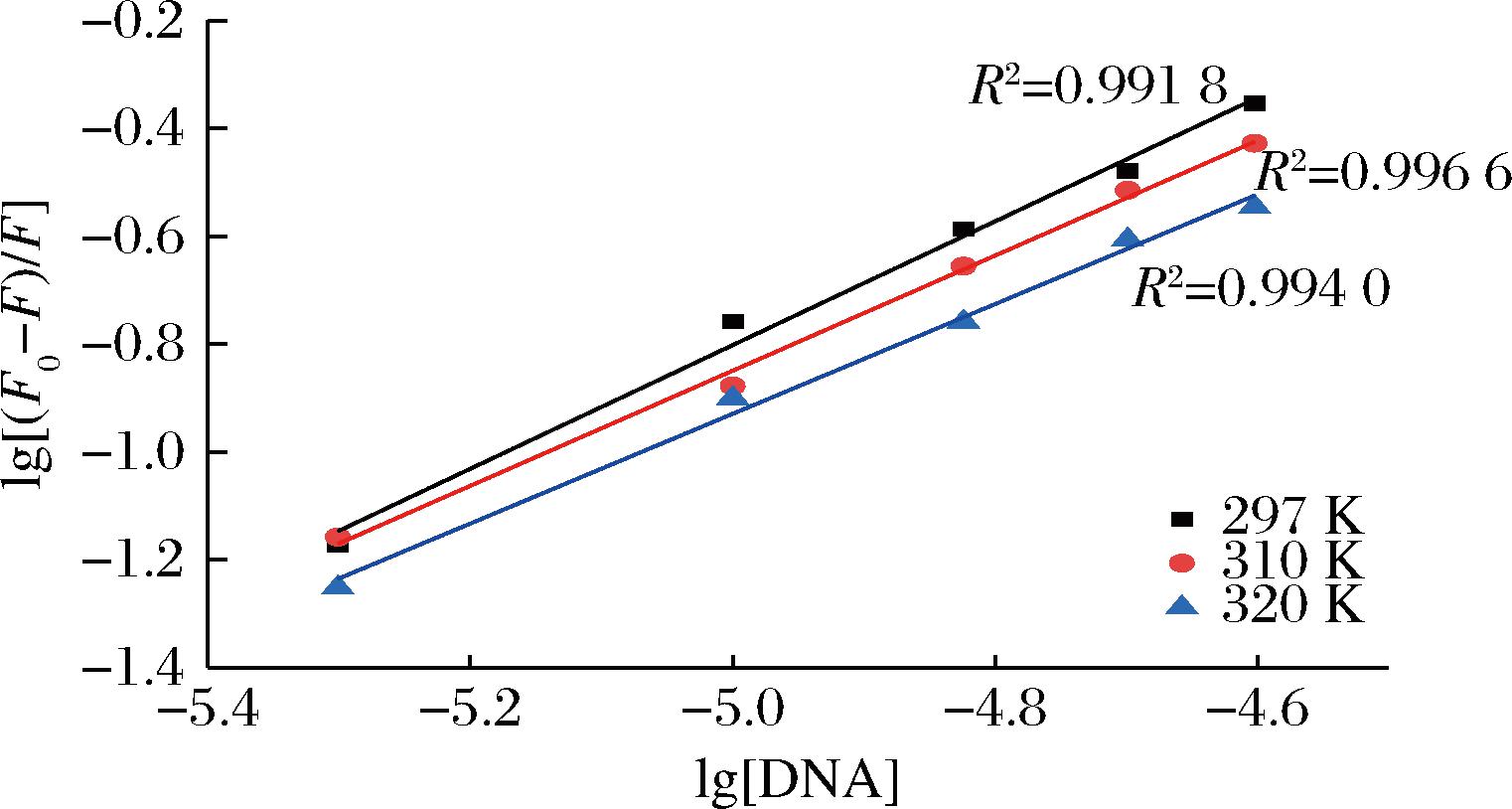
图3 不同温度下ctDNA对PLA荧光猝灭的 Lineweaver-Burk拟合直线
Fig.3 Lineweaver-Burk plots for ctDNA quenching of PLA fluorescence at different temperatures
2.1.3 热力学参数与作用力的测定
配体-DNA相互作用的确切模式通常通过热力学表征来确定。从本质上讲,氢键、范德华力、静电力及疏水作用等作用力在DNA和配体之间的相互作用中发挥着主要作用。PLA与DNA结合反应的热力学参数可以确定主要的结合力与反应的自发性。根据从光谱方法获得的数据,使用公式(2)、公式(3)计算热力学参数(ΔH,ΔS, ΔG):
(2)
ΔG=ΔH-TΔS
(3)
表1总结了ΔH,ΔS和ΔG的值。ΔG<0表明结合过程是自发的。已知当ΔH≈0或ΔH <0且ΔS>0时,静电引力为主要作用力;当ΔH>0且ΔS>0时,疏水作用力发挥主要作用;当ΔH<0且ΔS<0 时,主要的作用力为范德华力和氢键作用[21]。结论表明ΔH<0和ΔS<0,这说明PLA与DNA的结合过程中既有氢键作用,也有范德华力。总之,通过荧光光谱实验对PLA-ctDNA之间相互作用研究发现PLA能够自发通过氢键作用力和范德华力结合到ctDNA上,且至少存在1个结合位点。
表1 PLA-DNA体系在不同温度下的猝灭常数和热力学参数。
Table 1 Quenching constants and thermodynamic parameters for PLA-DNA system at different temperatures.
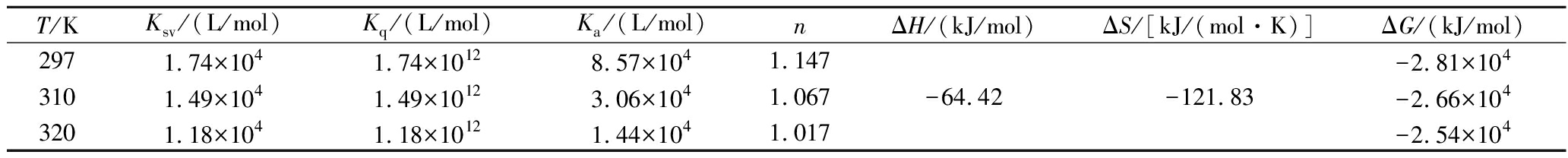
T/KKsv/(L/mol)Kq/(L/mol)Ka/(L/mol)nΔH/(kJ/mol)ΔS/[kJ/(mol·K)]ΔG/(kJ/mol)2971.74×1041.74×10128.57×1041.1473101.49×1041.49×10123.06×1041.0673201.18×1041.18×10121.44×1041.017-64.42-121.83-2.81×104-2.66×104-2.54×104
2.2 PLA与DNA结合方式的测定
DNA双螺旋结构中的大沟、小沟及磷酸骨架、碱基对等部位是配体小分子常见的识别位点。配体与DNA的非共价结合的作用模式主要有3种:静电结合(通过静电作用吸附到DNA外部的磷酸骨架)、沟槽结合(结合到DNA的大沟或小沟的碱基边缘)和插嵌结合(插入DNA内部的碱基对之间)。通过非共价结合往往能够导致许多生物效应,比如抑制DNA的正常复制与转录。
2.2.1 KI猝灭实验
KI是研究小分子与DNA结合方式的常用阴离子猝灭剂。它能有效地猝灭小分子的荧光,根据DNA存在和不存在时荧光的变化,可以推断DNA与小分子的相互作用模式。如果PLA在沟槽中与DNA结合,则结合DNA沟槽中的PLA的Ksv应大于游离PLA的Ksv。这是因为结合的PLA会被暴露在溶剂中,其荧光很容易被阴离子猝灭剂在沟槽中猝灭。相反,插入到DNA碱基对中的PLA的Ksv值应该低于自由小分子的Ksv,因为DNA的双螺旋可以保护结合的分子不受阴离子猝灭剂的影响。图4显示了KI在PLA和PLA-ctDNA体系中的猝灭行为。结果表明,两者的Ksv值分别为154、135 L/mol,说明ctDNA的双螺旋结构对PLA的猝灭起到了保护作用。因此,PLA与ctDNA的相互作用模式应为插嵌结合。
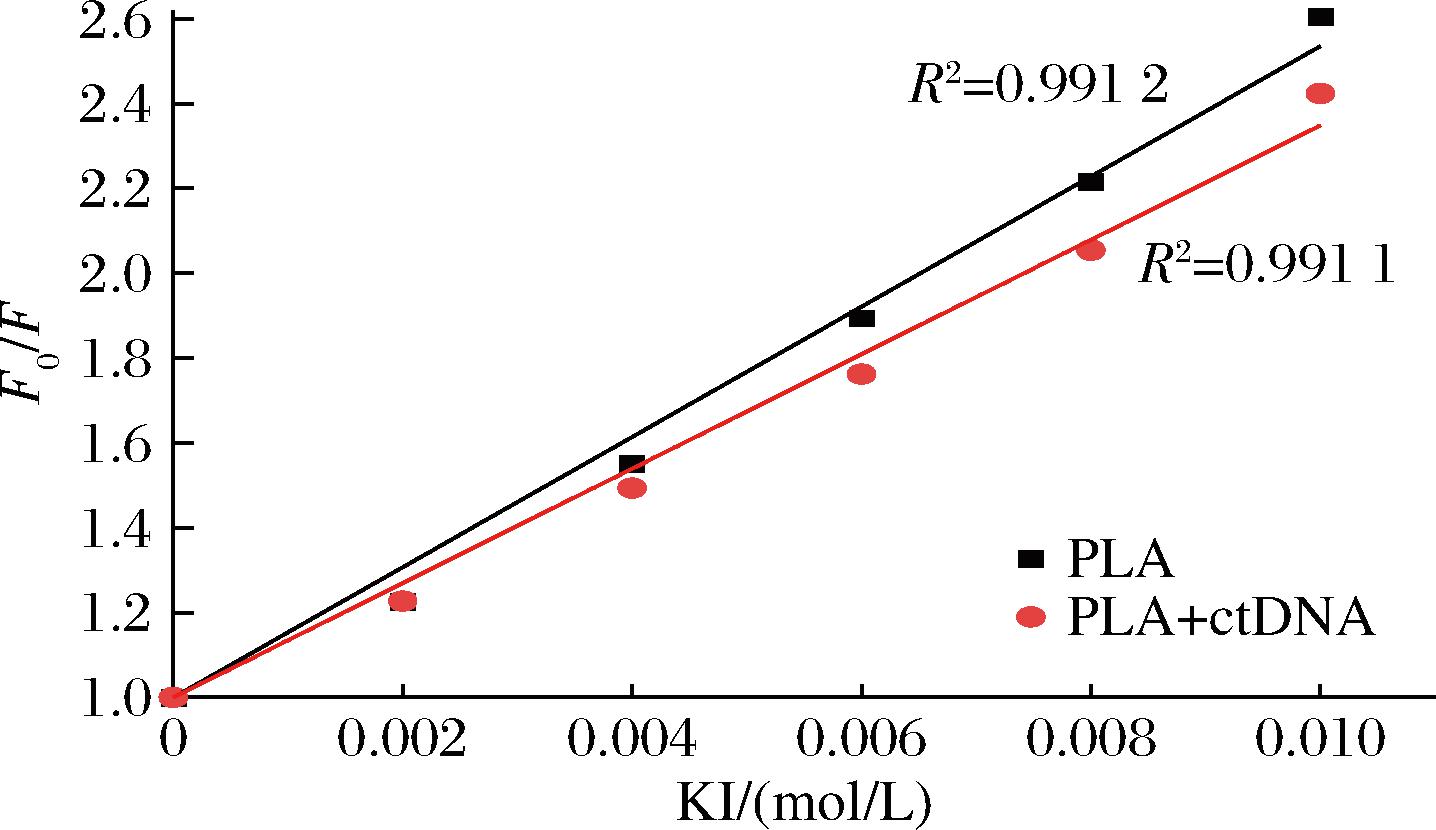
图4 在不存在和存在ctDNA的情况下,KI对PLA的 Stern-Volmer的拟合直线
Fig.4 Stern-Volmer plots of PLA by KI in the absence and presence of ctDNA
2.2.2 PLA对ctDNA熔点的影响
配体小分子以插嵌方式与DNA结合可提高双螺旋结构的稳定性,使DNA的Tm值提高,但非插嵌结合对DNA熔点影响较小。通过绘制熔解曲线能够进一步确定PLA-ctDNA的相互作用模式,根据公式(4)计算不同温度下的fss值,得到fss关于温度的曲线,在fss=0.5时对应的温度即为DNA的Tm。
(4)
结果如图5所示,PLA与DNA结合时,Tm值约为80 ℃,相比于纯ctDNA体系的Tm值提高了约7 ℃,这说明PLA的嵌入增强了双螺旋结构的稳定性。通过上述两个实验验证,证实了PLA是以插嵌的方式与DNA结合。
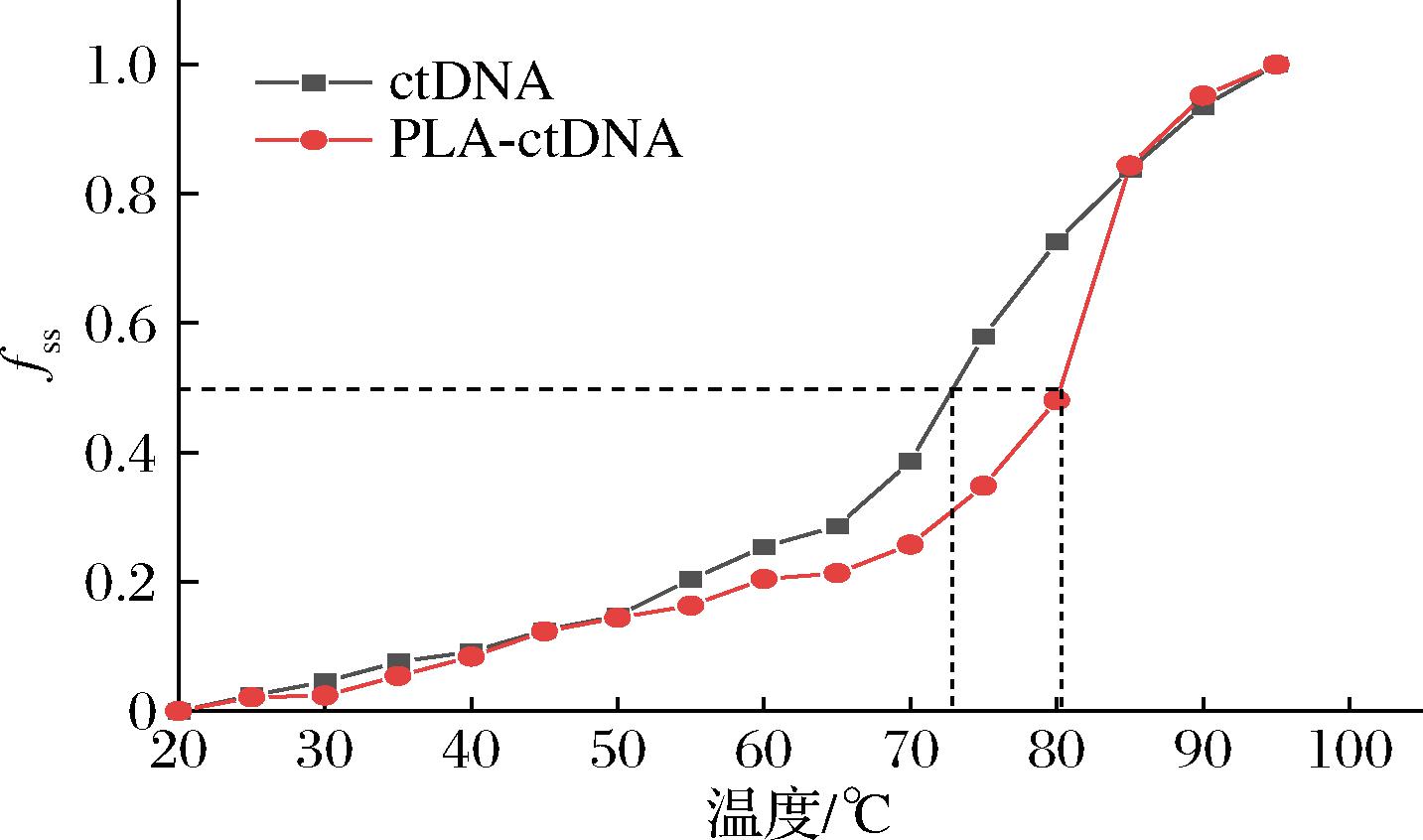
图5 在不存在和存在PLA的情况下,ctDNA的熔解曲线
Fig.5 Melting curve of ctDNA in the absence and presence of PLA
2.3 分子对接模拟PLA与DNA相互作用
分子建模可以更好地理解配体和受体之间的相互作用。在这项研究中,使用AutoDock 4程序,验证PLA与选定的DNA片段的结合模式。由于蛋白质数据库(Protein Data Bank,PDB)中没有ctDNA的结构信息,而B型双链DNA的结构是其主要形式,因此选择d(CGATCG)2序列代替ctDNA。通过LGA遗传算法,在得到的50个对接模拟结果中,选择能量相对较低、结合次数最多的构象作为PLA与DNA结合的最佳构象。图6显示PLA与DNA相互作用模式的构象,PLA滑入碱基对CG之间,与CG碱基对之间的结合能为-20.40 kJ/mol。PLA脂肪链部分的羟基和羧基作为氢键受体(供体)与碱基和脱氧核糖形成氢键,结合距离分别为2.2、2.0、1.7 Å,苯环部分通过堆积作用嵌入碱基对之间,与周围的碱基形成大量π—π作用。分子对接的结果证实了上述光谱结果。因此,本研究为PLA与DNA之间的结合作用提供了进一步的证据,证明PLA与DNA之间的结合是通过插嵌结合发生的。

图6 PLA与B-DNA d(CGATCG)2的分子对接模型
Fig.6 Molecular docking model of PLA with B-DNA of sequence d(CGATCG)2
此外,为了验证L-PLA对于插入不同碱基对的偏好性,选取不同的B-DNA链进行了分子对接模拟。电子版增强出版附图1(https://doi.org/10.13995/j.cnki.11-1802/ts.035182)中展示了PLA分子对于不同嵌入位点(CG, GA, TG)的模拟对接结果。氢键作用与π—π作用依然为主要作用力,当PLA嵌入CG碱基对之间时,苯环能够与碱基平面形成更多的π—π作用力,且氢键键长更短(2.2、2.0、 1.7 Å),说明有较强的氢键作用力。且当插入CG碱基对之间时,配体与受体之间拥有最低的结合能,种种证据表明,PLA更倾向于插入双链DNA的CG碱基对之间。
2.4 转录组学分析PLA与基因组DNA相互作用的影响
先前的实验证明了PLA能够通过插嵌的方式与DNA稳定结合,为了进一步解析PLA通过与DNA结合是如何影响细菌生长,通过转录组测序分析了PLA在与基因组DNA结合下对细菌转录的影响,解析细菌生长受到抑制的关键基因。以受2 g/L D-PLA胁迫(D组),受2 g/L L-PLA胁迫(L组)和未受胁迫(CG组)的乳酸片球菌DY15为样本,在MRS液体培养基中生长到对数中后期时(8 h),收集菌体进行转录组测序(NCBI登录号:PRJNA921976)。对PLA胁迫条件下与未受胁迫的乳酸片球菌DY15的RNA-seq结果进行分析,以log2(Fold Change)>2,Pval≤0.05作为筛选标准,筛选具有显著差异表达的基因。结果,经D-PLA胁迫生长后,共758个基因表达量发生显著变化,261个上调基因,497个下调基因。经L-PLA胁迫生长后,共630个基因表达量发生显著变化,194个上调基因,436个下调基因。
2.4.1 差异表达基因的GO注释分析
为进一步分析PLA胁迫条件下细胞损伤机理,对差异表达基因进行GO富集分析。结果如图7所示,差异表达基因可分为分子功能、细胞成分和生物过程3大类,又分别分为了11、2、12个亚类。细胞组分中涉及生物膜组分和质膜的差异表达基因占比最高,说明PLA作用靶点之一可能在生物膜上。涉及到核酸合成和复制的差异表达基因也较多,可以看出PLA进入细胞内能够破坏基因组的结构。以上结果可以看出两种PLA胁迫下,差异基因富集趋势基本一致,PLA主要影响的是涉及生物膜组分及核酸合成方面的基因。

图7 差异表达基因的GO注释
Fig.7 GO analysis of differentially expressed genes
2.4.2 差异表达基因的KEGG注释分析
为了进一步分析差异表达基因的生物学功能,对差异基因进行了KEGG注释。两种胁迫条件下差异表达基因分别富集到了73、70条代谢通路中,最后选取了28条富集最显著的代谢通路展示在图8中。分析发现,两种胁迫条件中均显著富集的代谢通路有20条,分别为错配修复、氧化磷酸化、氨酰-tRNA合成、核糖体和精氨酸代谢等,其中与核酸合成和修复相关的富集通路最多,侧面印证了PLA与DNA的结合是其攻击细菌的一种方式。

图8 差异表达基因的KEGG注释
Fig.8 KEGG analysis of differentially expressed genes
注:RichFactor指差异表达的基因中位于该通路中的基因数目与所有注释基因中位于该通路的基因总数的比值,值越大,表示富集的程度越大。 Qvalue是经多重假设检验校正之后的Pvalue,Qvalue的取值范围为[0,1],越接近于零,表示富集越显著。
2.4.3 对细胞壁合成和DNA修复关键基因的实时荧光定量PCR验证
通过对差异基因的GO与KEGG注释发现,较多差异基因与生物质膜合成及核酸损失修复相关,因此推测细胞壁与基因组是PLA主要作用靶点。当PLA破环细胞壁或基因组时,胞内的应激反应会使相关基因转录水平上调。选取与细胞壁肽聚糖合成途径中的一系列mur基因进行了实时荧光定量PCR验证,所选的6个基因在L-PLA胁迫下均发生不同程度的转录上调(图9-a)。此外,还分析了DNA损伤修复相关的基因,如:用于DNA损伤修复的DNA聚合酶Ⅳ基因dinB、重组修复基因radA、修复-转录偶联因子mfd、DNA糖基化酶基因mutM和切除修复蛋白基因uvrA,以上代表了细菌几种不同修复机制的基因转录水平也均发生了上调(图9-b)。总之,上述基因转录水平的应激上调间接证实了PLA的主要作用靶点是细胞壁与基因组。
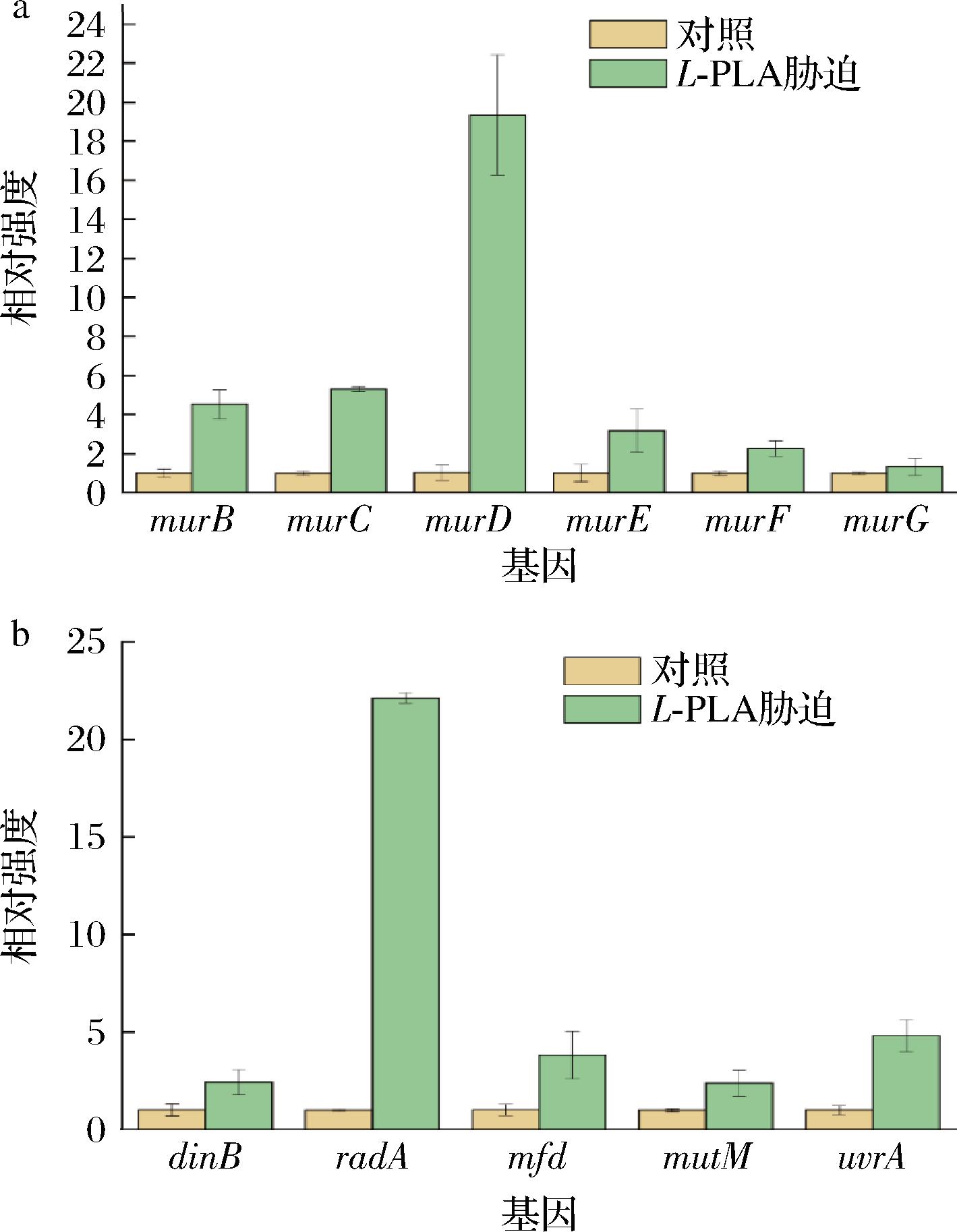
a-实时荧光定量PCR测定受L-PLA胁迫后细胞壁合成 关键基因表达变化;b-实时荧光定量PCR测定受L-PLA 胁迫后DNA损伤修复相关基因表达变化
图9 PLA对细胞壁合成和DNA修复关键基因的表达影响
Fig.9 Effects of phenyllactic acid on the expression of key genes for cell wall synthesis and DNA repair
2.4.4 对精氨酸代谢途径中差异基因的实时荧光定量PCR验证
乳酸菌在生长发酵时会不断产生乳酸,从而降低生长环境中的pH,而乳酸菌之所以能够在酸性环境下生存下来,与其自身的酸胁迫应答机制相关。乳酸菌中常见的耐酸应答机制有质子泵理论、谷氨酸脱羧体系、碱性氨基酸代谢、蛋白修复以及DNA损伤修复机制等[22](图10-a)。
通过对乳酸片球菌DY15的转录组数据分析发现,乳酸片球菌DY15中不存在谷氨酸脱羧酶,在受到PLA胁迫时,与质子泵、蛋白修复和DNA损伤修复相关的基因普遍受到上调,而精氨酸代谢途径受到了极大抑制。在精氨酸代谢途径中,精氨酸经3种酶催化生成2分子NH3从而起到降低pH的作用。通过实时荧光定量PCR对该途径的4个关键蛋白进行基因表达量测定(图10-b),证实经L-PLA胁迫该途径的基因会受到抑制,说明该PLA可能是通过抑制该途径来降低细菌酸耐受性,从而实现抑菌效果。
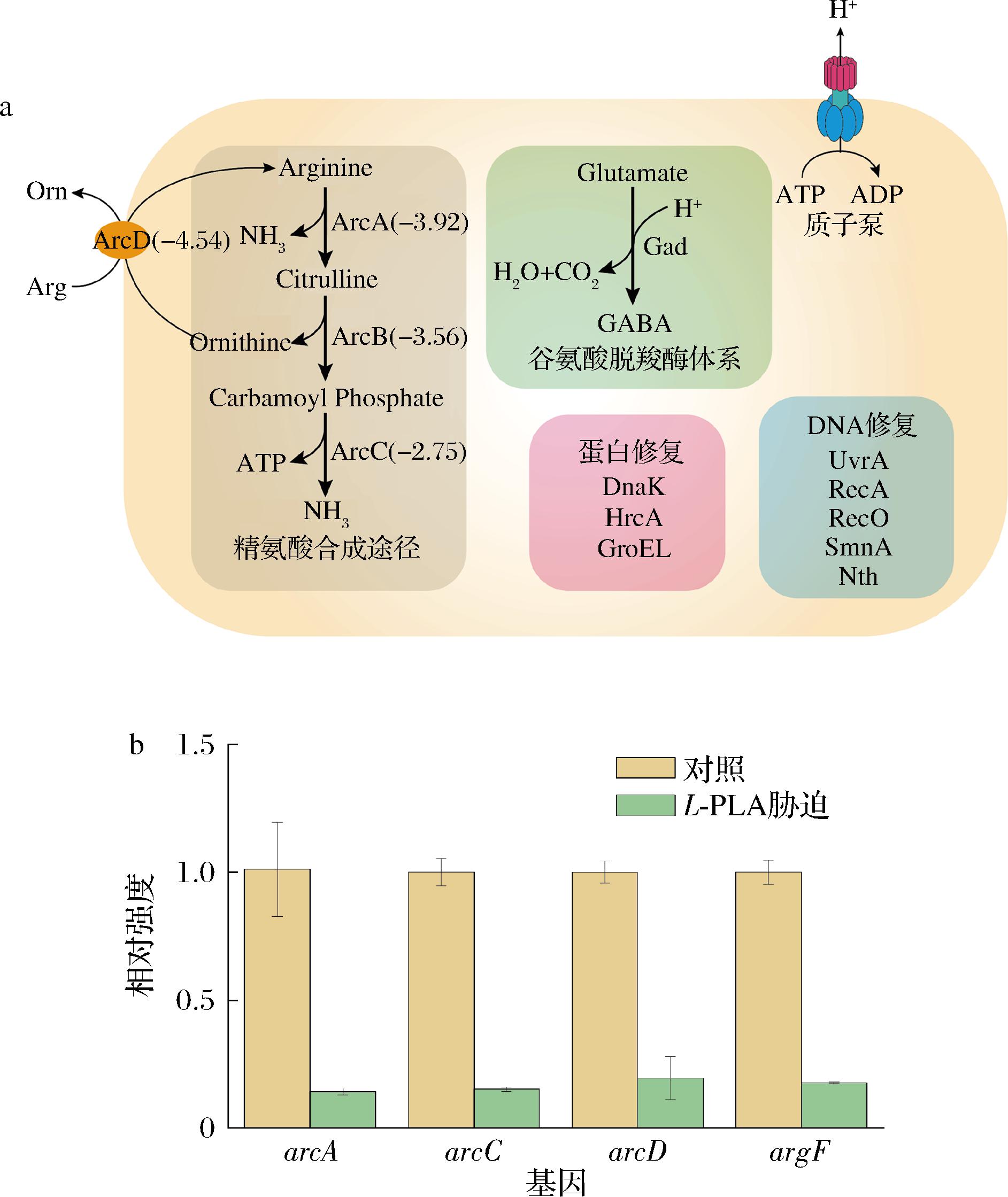
a-乳酸乳球菌DY15的耐酸应答机制,红色代表基因上调,蓝色代 表基因下调,括号里为下调倍数;b-实时荧光定量PCR测定受 L-PLA胁迫后精氨酸代谢途径中关键基因表达变化
图10 PLA对乳酸片球菌耐酸应答机制的影响
Fig.10 Effect of PLA on acid tolerance response mechanism of Pediococcus acidilactici
3 结论与讨论
PLA作为天然抑菌物质,在食品工业、动物饲料方面有着巨大的应用前景。早前关于PLA研究主要集中于对不同微生物的表观抑制效果,如最小抑菌浓度、与其他有机酸的协同抑菌作用[23]等,以及其破坏生物膜的机理研究。本研究集中于研究PLA与DNA的相互作用以及在PLA对于细菌基因表达的影响。通过荧光光谱实验确定了PLA与模式DNA相互作用方式,并通过分子对接模拟获得了PLA-DNA结合模型,说明PLA能够通过插入DNA进而可能会影响复制和转录。进而通过转录组学分析了PLA与DNA结合可能造成转录变化,发现PLA主要影响细胞壁合成、核酸的复制与修复相关的基因,侧面证实了PLA的攻击靶点是细胞壁和基因组的可能性。同时PLA通过抑制精氨酸代谢途径的关键基因表达从而降低了细菌酸耐受性,进而通过生长环境的酸性来抑制细菌生长,这也与PLA和有机酸的协同抑菌能够达到更好效果的结论相印证。综上,本研究对PLA抑菌机理做出了补充,为PLA的应用提供了理论基础。
[1] RAJANIKAR R V, NATARAJ B H, NAITHANI H, et al.Phenyllactic acid:A green compound for food biopreservation[J].Food Control, 2021, 128:108184.
[2] LAVERMICOCCA P, VALERIO F, EVIDENTE A, et al.Purification and characterization of novel antifungal compounds from the sourdough Lactobacillus plantarum strain 21B[J].Applied and Environmental Microbiology, 2000, 66(9):4084-4090.
[3] SCHMIDT M, LYNCH K M, ZANNINI E, et al.Fundamental study on the improvement of the antifungal activity of Lactobacillus reuteri R29 through increased production of phenyllactic acid and reuterin[J].Food Control, 2018, 88:139-148.
[4] MU W M, YU S H, ZHU L J, et al.Production of 3-phenyllactic acid and 4-hydroxyphenyllactic acid by Pediococcus acidilactici DSM 20284 fermentation[J].European Food Research and Technology, 2012, 235(3):581-585.
[5] GUAN J T, HAN C F, GUAN Y X, et al.Optimizational production of phenyllactic acid by a Lactobacillus buchneri strain via uniform design with overlay sampling methodology[J].Chinese Journal of Chemical Engineering, 2019, 27(2):418-425.
[6] ZHENG Z J, MA C Q, GAO C, et al.Efficient conversion of phenylpyruvic acid to phenyllactic acid by using whole cells of Bacillus coagulans SDM[J].PLoS One, 2011, 6(4):e19030.
[7] LIND H, SJÖGREN J, GOHIL S, et al.Antifungal compounds from cultures of dairy propionibacteria type strains[J].FEMS Microbiology Letters, 2007, 271(2):310-315.
[8] DIEULEVEUX V, VAN DER PYL D, CHATAUD J, et al.Purification and characterization of anti-Listeria compounds produced by Geotrichum candidum[J].Applied and Environmental Microbiology, 1998, 64(2):800-803.
[9] LI X F, NING Y W, LIU D, et al.Metabolic mechanism of phenyllactic acid naturally occurring in Chinese pickles[J].Food Chemistry, 2015, 186:265-270. [10] YANG X Y, LI J P, SHI G C, et al.Improving 3-phenyllactic acid production of Lactobacillus plantarum AB-1 by enhancing its quorum-sensing capacity[J].Journal of Food Science and Technology, 2019, 56(5):2605-2610.
[11] LIU F, SUN Z L, WANG F T, et al.Inhibition of biofilm formation and exopolysaccharide synthesis of Enterococcus faecalis by phenyllactic acid[J].Food Microbiology, 2020, 86:103344.
[12] 罗希, 杨泽锋, 臧瑜, 等.乳酸脱氢酶与葡萄糖脱氢酶偶联催化合成D-苯基乳酸[J].食品与发酵工业, 2019, 45(7):22-28. LUO X, YANG Z F, ZANG Y, et al.Synthesis of D-phenyllactic acid catalyzed by coupled lactate dehydrogenase and glucose dehydrogenase[J].Food and Fermentation Industries, 2019, 45(7):22-28.
[13] CORTÉS-ZAVALETA O, L PEZ-MALO A, HERN
PEZ-MALO A, HERN NDEZ-MENDOZA A, et al.Antifungal activity of lactobacilli and its relationship with 3-phenyllactic acid production[J].International Journal of Food Microbiology, 2014, 173:30-35.
NDEZ-MENDOZA A, et al.Antifungal activity of lactobacilli and its relationship with 3-phenyllactic acid production[J].International Journal of Food Microbiology, 2014, 173:30-35.
[14] SORRENTINO E, TREMONTE P, SUCCI M, et al.Detection of antilisterial activity of 3-phenyllactic acid using Listeria innocua as a model[J].Frontiers in Microbiology, 2018, 9:1373.
[15] RODR GUEZ N, SALGADO J M, CORTÉS S, et al.Antimicrobial activity of d-3-phenyllactic acid produced by fed-batch process against Salmonella enterica[J].Food Control, 2012, 25(1):274-284.
GUEZ N, SALGADO J M, CORTÉS S, et al.Antimicrobial activity of d-3-phenyllactic acid produced by fed-batch process against Salmonella enterica[J].Food Control, 2012, 25(1):274-284.
[16] WU H, GUANG C E, ZHANG W L, et al.Recent development of phenyllactic acid:Physicochemical properties, biotechnological production strategies and applications[J].Critical Reviews in Biotechnology, 2023, 43(2):293-308.
[17] 黄云坡, 孙晶, 梅红霞, 等.苯乳酸对单增李斯特菌的细胞膜完整性和通透性的影响[J].食品工业科技, 2019, 40(5):130-135;143. HUANG Y P, SUN J, MEI H X, et al.Effect of D-(+)-3-phenyllactic acid against Listeria monocytogenes 10403 s on cell membrane integrity and permeability[J].Science and Technology of Food Industry, 2019, 40(5):130-135;143.
[18] ZHOU Q Q, GU R C, LI P, et al.Anti-Salmonella mode of action of natural l-phenyl lactic acid purified from Lactobacillus plantarum ZJ316[J].Applied Microbiology and Biotechnology, 2020, 104(12):5283-5292.
[19] LIU J, WANG Z H, LUO G A, et al.The interaction of DNA with dopamine by spectroscopic and electrochemical methods[J].Analytical Sciences, 2002, 18(7):751-755.
[20] YANG H Q, TANG P X, TANG B, et al.Studies of DNA-binding properties of lafutidine as adjuvant anticancer agent to calf thymus DNA using multi-spectroscopic approaches, NMR relaxation data, molecular docking and dynamical simulation[J].International Journal of Biological Macromolecules, 2017, 99:79-87.
[21] ROSS P D, SUBRAMANIAN S.Thermodynamics of protein association reactions:Forces contributing to stability[J].Biochemistry, 1981, 20(11):3096-3102.
[22] 赵小茜, 魏旭丹, 陈戴玲, 等.乳酸菌耐酸耐胆盐机制研究进展[J].乳业科学与技术, 2017, 40(3):33-36. ZHAO X X, WEI X D, CHEN D L, et al.A review on the mechanism of acid and bile salt resistance of lactic acid bacteria[J].Journal of Dairy Science and Technology, 2017, 40(3):33-36.
[23] 宁亚维, 付浴男, 何建卓, 等.苯乳酸和醋酸联用对单核细胞增生李斯特菌的协同抑菌机理[J].食品科学, 2020, 41(23):70-76. NING Y W, FU Y N, HE J Z, et al.Synergistic antibacterial mechanism of phenyllactic acid combined with acetic acid against Listeria monocytogenes[J].Food Science, 2020, 41(23):70-76.