糖基化反应是天然产物结构修饰中最常见和最重要的反应之一[1]。所连接的糖部分对其母体化合物的生物学性质具有深远影响,并可显著调节其生物利用度、生物活性和药物性[2]。糖基化天然产物已成为一种广泛应用于食品工业的天然化合物。例如,红景天活性成分红景天苷因其具有抗氧化等活性广泛应用于食品和保健品领域[3];甜叶菊叶片中提取的甜菊糖苷因其高甜度、零热量可用作甜味剂广泛应用于食品领域[4]。从植物中直接分离提取或使用化学合成的方式获取糖苷具有产率低、成本高等缺点[5]。相比之下,生物催化因其高效温和的反应条件具有广泛的应用空间。
苯乙醇苷类化合物(phenylethanoid glycosides,PhGs)是存在于植物界的具有抗炎、抗氧化和神经保护等多种生理活性的天然产物,广泛应用于医药及保健食品领域[6]。其中松果菊苷和毛蕊花糖苷是最具代表的苯乙醇苷类化合物,在红花钓钟柳(Penstemon barbatus)叶中的含量较为丰富[7]。
糖基化反应通常由UDP-糖基转移酶(UDP-glycosyltransferases,UGTs)催化,该类酶将糖基残基从活化的核苷酸糖转移到受体分子[8]。在过去的研究中,已经从多种植物中鉴定出大量的UGTs可催化黄酮类[9],萜类[10]以及苯丙素类[11]等多种化合物。然而,用于催化PhGs糖基化的酶仅有少量报道。YANG等[12]鉴定了3个来源于粗壮女贞的糖基转移酶,它们参与了紫茎女贞苷B的生物合成。因此,从植物中进一步挖掘用于苯乙醇苷类化合物糖基化的UGTs具有非常大的意义。
为了寻找更多样的催化苯乙醇苷类糖基化的糖基转移酶,对富含该类化合物的红花钓钟柳转录组数据进行不同组织基因差异表达分析,得到了7条糖基转移酶候选基因,经过异源表达及酶活性分析发现了一个具有催化桂叶苷B能力的糖基转移酶。基于氨基酸序列一致性原则,UGT命名委员会(https://labs.wsu.edu/ugt/)将其归为UGT712亚家族,这个新型糖基转移酶可以催化桂叶苷B的苯乙醇部分C-4位置的糖基化形成一种新的糖苷产物。此外,通过底物谱分析发现UGT712A2对黄酮类、香豆素类等多种化合物都具备催化活性。因此,UGT712A2可能是一种潜在的新型多功能糖基转移酶,用于苯乙醇苷类化合物和其他多种化合物的糖基化反应,从而催化生成新的糖苷化合物可应用于食品、医药等领域。
1 材料与方法
1.1 实验材料
1.1.1 分子克隆相关实验材料
高保真DNA聚合酶、一步克隆试剂盒等,南京诺唯赞生物科技有限公司;PCR产物回收试剂盒、质粒小量提取试剂盒、凝胶回收试剂盒等,北京天根生化科技有限公司。
1.1.2 蛋白表达和纯化相关实验材料
用于诱导蛋白表达的异丙基硫代半乳糖(isopropyl-β-D-thiogalactopyranoside,IPTG)母液浓度为1 mol/L;SDS-PAGE凝胶电泳预制胶和缓冲液以及用于蛋白纯化的重力镍柱,天地人和生物科技有限公司。
1.1.3 体外酶反应相关实验材料
利用本实验室大肠杆菌底盘细胞OB-4制备底物桂叶苷B[13];UDP-葡萄糖、胡黄连苷Ⅱ、东莨菪内酯,上海源叶生物科技有限公司;毛蕊花糖苷,四川省维克奇生物科技有限公司;槲皮素天津百倍生物科技有限公司;芦丁,上海吉至生化科技有限公司;7-去甲基软木花椒素,广州市文睿科学仪器有限公司。
1.1.4 仪器与设备
PCR仪,德国Eppendorf公司;核酸/垂直电泳仪,Bio-Red公司;超声波细胞破碎仪,宁波新芝生物科技有限公司;JN3000PLΜS细胞破碎机,Jnbio公司;高效液相色谱仪,岛津仪器有限公司;液质色谱联用仪,安捷伦科技有限公司;600M核磁,Bruker Daltonics公司。
1.2 实验方法
1.2.1 糖基转移酶的筛选
为了筛选编码苯乙醇苷类化合物糖基化的UDP-糖基转移酶的特定基因,首先以拟南芥88条糖基转移酶基因作为检索序列,对红花钓钟柳转录组数据进行本地BLAST,E值设置为1e-5[14]。使用CD-HIT以0.9的阈值对序列进行聚类以去除冗余序列。在去除短序列(<400个氨基酸)后,从转录组数据中挖掘出总共66个糖基转移酶候选序列。
XIE等[7]发现红花钓钟柳叶中松果菊苷含量最多,茎中次之,根中最少。考虑到参与苯乙醇苷类化合物糖基化的基因与苯乙醇苷类化合物含量表现出相似的表达模式,进行了不同组织中基因差异表达分析鉴定与松果菊苷含量共表达的糖基转移酶候选基因。使用TBtools软件进行聚类分析[15],基于皮尔逊相关系数(Pearson′s correlation coefficient,PCC)选取候选基因。
1.2.2 候选糖基转移酶的分子克隆
PCR扩增目的片段,用Snapgene设计扩增引物如表1所示。扩增后的目的片段与载体pET-MBP进行无缝克隆;重组质粒转化到E.coli DH5α感受态细胞中。菌落PCR方法筛选阳性克隆进行DNA测序验证。
表1 本研究所使用的引物
Table 1 Primers used in this study

引物名称 引物序列(5′-3′)MBP_32938_FAATCCAATATTGGAAGTGGAATGACAATACTAGTTGAGAGCAAGAGACMBP_32938_RGGCGCTCGAATTCGGATCCGTCATCCACTCCAAATTTGATCAATAAATGTCMBP_51551_FAATCCAATATTGGAAGTGGAATGGGTTCTATCTCATGCCCACACMBP_51551_RGGCGCTCGAATTCGGATCCGTTAATCTCTATGAGCAATAACCCCTTGTGGMBP_54680_FAATCCAATATTGGAAGTGGAATGGCCACCAACTTCAATTCCGAAGMBP_54680_RGGCGCTCGAATTCGGATCCGTTATCTACAAATATGATGCATGAAAGAATCCATCTCCMBP_55276_FAATCCAATATTGGAAGTGGATGAATTTGATGATGAAGAAGAAGAAGAAAATAATTTTAATACCMBP_55276_RGGCGCTCGAATTCGGATCCGTCAAATAAATTGAGAGAAAGTAGACATTGCTGTAGMBP_56903_FAATCCAATATTGGAAGTGGAATGAATGGGTACATTGGCAAACCCMBP_56903_RGGCGCTCGAATTCGGATCCGTCATGCTGGAAGAACCTCTGCATCMBP_57174_FAATCCAATATTGGAAGTGGAATGAATATGACGAAGGTCACAGGTCGMBP_57174_RGGCGCTCGAATTCGGATCCGTCAGACGAAGAACATGACGTCCTCCMBP_35326_FAATCCAATATTGGAAGTGGAATGGATAAAGAAGGAACTATGTCCAAAATTTTAATGMBP_35326_RGGCGCTCGAATTCGGATCCGTTATTTATTCTTCAAACAAAGCCGCCAC
1.2.3 候选糖基转移酶的异源表达
将测序正确的重组质粒转入E.coli Transetta (DE3)中。转化后的单克隆挑取至含有50 μg/mL卡那霉素和50 μg/mL氯霉素的5 mL LB培养基中,在37 ℃、220 r/min条件下培养过夜;再按照1%的接种量转接至含有50 mL LB培养基中培养至OD值为0.6~0.8,加入10 μL IPTG母液,且在16 ℃下培养18~22 h。收集菌体后用5 mL Buffer C(50 mmol/L Tris-HCl,pH 7.5,100 mmol/L NaCl)重悬,并使用超声破碎仪破碎15 min,离心取上清液可进行体外粗酶反应。并使用SDS-PAGE检测7个重组蛋白的表达情况。
1.2.4 酶活分析
在含有90 μL粗酶上清液、1 mmol/L UDP-葡萄糖、0.1 mmol/L桂叶苷B或者毛蕊花糖苷的100 μL反应体系中进行粗酶反应。将反应物在30 ℃下孵育5 h后加入100 μL甲醇终止。随后,经离心收集上清液,然后通过HPLC和液相色谱高分辨质谱(liquid chromatography high-resolution mass spectrometry,LC-HRMS)进行分析。阴性对照实验用100 ℃煮沸10 min的粗酶蛋白代替活性酶。
1.2.5 糖基化产物制备和纯化
为了获得足够的糖基化产物以鉴定其催化位置和立体化学,首先对重组蛋白使用重力镍柱进行纯化。为了纯化带有6 × His标签的重组蛋白,将细菌细胞重悬于缓冲液A(50 mmol/L Tris-HCl,20 mmol/L咪唑,100 mmol/L NaCl,pH 7.5)中。细胞破碎机3次破碎混悬液,离心后将上清液上样至预平衡好的亲和色谱柱。然后用30 mL Buffer A洗涤具有目的蛋白的色谱柱,并用15 mL Buffer B(50 mmol/L Tris-HCl,500 mmol/L咪唑,100 mmol/L NaCl,pH 7.5)洗脱。通过SDS-PAGE验证重组蛋白纯度。
使用纯化后的MBP-UGT712A2蛋白1 mL、10 mmol UDP-葡萄糖和1 mmol桂叶苷B在30 ℃下孵育5 h,并用1 mL甲醇终止离心后在35 ℃下通过旋转蒸发仪浓缩上清液,并溶于50%甲醇中。随后,使用配有SPD-20 A检测器的Shimadzu LC-6 AD系统和Shim-pack PREP-ODS(H)色谱柱(10 mm×250 mm,5 μm)进行半制备HPLC分离。乙腈(B)和甲酸(0.1%)(A)作为流动相,流速为4 mL/min。梯度条件如下:18% B,5 min;18%~23.5% B,55 min;100% B,8 min;18% B,8 min。321 nm波长下收集新峰产物。
将纯化后的糖苷产物溶于600 μL CD3OD,使用600 MHz Bruker Advance Ⅲ光谱仪进行核磁(nuclear magnetic resonance,NMR)分析,通过1H NMR、13C NMR、异核多键相关谱(heteronuclear multiple bond correlation,HMBC)和异核单量子相关谱(heteronuclear single quantum coherence,HSQC)来判断产物结构,从而确定UGT712A2对于桂叶苷B的催化功能。
1.2.6 系统发育树构建和底物谱分析
将UGT712A2与其他29条已鉴定功能的不同植物来源的糖基转移酶一起构建系统进化树。使用MEGA7软件,ClustalW方法进行序列比对,并使用邻近树法建立系统发育树。
为研究UGT712A2的底物混杂性,使用UDP-葡萄糖作为糖供体检测了不同的糖受体底物,在100 μL含50 mmol/L Tris-HCl、100 mmol/L氯化钠的缓冲液中加入100 μg纯的MBP-UGT712A2,0.5 mmol/L受体底物和5 mmol/L供体底物UDP-葡萄糖进行反应。将所有反应物在30 ℃下孵育5 h,并用100 μL甲醇终止。然后将混合物离心取上清液用于HPLC/LC-HRMS分析。
1.2.7 HPLC和LC-HRMS分析
反应上清液使用配有SPD-M40 PDA检测器的Shimadzu LC-20AD HPLC系统和SilGreen C18 column 色谱柱(4.6 mm×250 mm,5 μm)进行HPLC分析,乙腈(B)和甲酸(0.1%)(A)作为流动相,流速为1 mL/min。其中,桂叶苷B以及毛蕊花糖苷做糖基受体时的分析梯度是:5% B,5 min;5%~13.2% B,13 min;18%~21.6% B,25 min;其余化合物的酶反应分析梯度是:5% B,5 min;5%~25% B,20 min。
对于样品的LC-HRMS分析在Agilent 1200 HPLC系统上进行,该系统与配备有电喷雾电离装置的Bruker-MicrOTOF-II质谱仪偶联。正离子模式下进行检测,其检测条件与HPLC分析相同。
2 结果与分析
2.1 候选基因的筛选结果
结合使用聚类分析和皮尔逊相关系数分析,从红花钓钟柳中提取了7个候选糖基转移酶的开放阅读框,其表达量与相关性如图1所示。

图1 红花钓钟柳中与松果菊苷含量共表达的糖基转移酶候选基因的表达谱
Fig.1 Expression profiles of UGT candidates co-expressed with echinacoside in Penstemon barbatus
2.2 蛋白表达情况
SDS-PAGE检测重组蛋白的表达情况如图2所示,通过分析重组蛋白在全细胞(C),上清液(S)以及沉淀(P)中的表达,可以看出7个候选基因的重组蛋白在Transetta(DE3)均有表达。
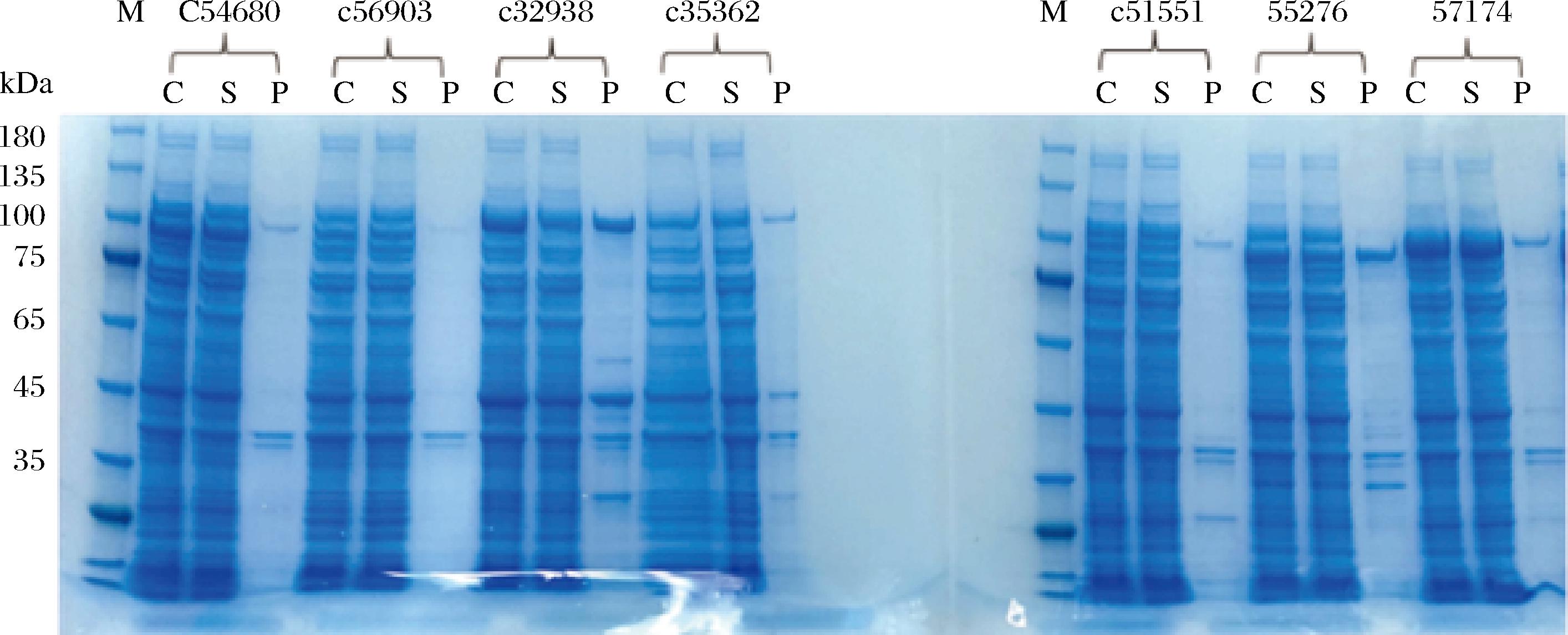
图2 重组蛋白MBP-PbUGTs的SDS-PAGE电泳分析图
Fig.2 SDS-PAGE analysis of expression of MBP-PbUGTs fusion protein
2.3 体外酶促反应结果
以UDP-葡萄糖为糖基供体,以桂叶苷B(1)和毛蕊花糖苷(2)为糖基受体底物,分别与7个候选糖基转移酶的粗酶上清液进行体外酶反应。HPLC和LC-HRMS检测结果发现c32938对桂叶苷B和毛蕊花糖苷有催化活性。本文通过UGT命名委员会将c32938命名为UGT712A2。HPLC检测结果如图3-a所示,UGT712A2的可以催化桂叶苷B和毛蕊花糖苷分别产生新峰1a和2a。LC-HRMS结果如图3-b和图3-c所示,新峰产物1a和2a都是在受体底物分子基础上加了一分子的葡萄糖。表明UGT712A2具有催化其葡萄糖基化反应的能力。

a-酶催化反应HPLC分析;b-糖基化产物1a的LC-HRMS分析;c-糖基化产物2a的LC-HRMS分析
图3 UGT712A2酶催化反应的HPLC和LC-HRMS分析
Fig.3 HPLC and LC-HRMS analysis of the enzymatic reactions catalysed by UGT712A2
2.4 体外酶促反应结果
为进一步确定UGT712A2对苯乙醇苷类化合物的催化位点,首先获得了较纯的重组蛋白MBP-UGT712A2,其SDS-PAGE检测结果如图4-a所示。通过扩大体外酶反应制备产物1a,最终获得1.6 mg 1a(HPLC>98%)。最终获得糖苷产物1a核磁数据:1H NMR (600 MHz,CD3OD) δ 7.68 (1H,d,J=15.9 Hz,H-7″),7.49 (2H,d,J=8.7 Hz,H-2″/6″),7.22 (2H,d,J=8.6 Hz,H-2/6),7.04 (2H,d,J=8.6 Hz,H-3/5),6.83 (2H,d,J=8.5 Hz,H-3″/5″),6.36 (1H,d,J=15.9 Hz,H-8″),5.21 (1H,d,J=1.8 Hz,H-1″′),4.93 (1H,t,J=9.5 Hz,H-4′),4.88 (1H,d,J=7.4 Hz,H-1″″),4.40(1H,d,J=7.9 Hz,H-1′),4.10 (1H,m,H-8α),3.93 (1H,m,H-5′),3.91 (1H,dd,J=12.0,2.1 Hz,H-6″′α),3.83 (1H,t,J=9.2 Hz,H-3′),3.78 (1H,m,H-8β),3.72 (1H,dd,J=12.0,5.4 Hz,H-6″′β),3.64-3.29 (protons in rhamnose and glucoses),2.92 (2H,td,J=7.2,1.7 Hz,H-7),1.10 (3H,d,J=6.2 Hz,H-6″′);13C NMR (100 MHz,CD3OD) δ 168.1 (C-9″),161.3 (C-4″),157.5 (C-4),147.4 (C-7″),133.8 (C-1),131.1 (C-2″,6″),130.8 (C-2,6),126.9 (C-1″),117.6 (C-3,5),116.7 (C-3″,5″),114.6 (C-8″),104.0 (C-1′),102.8 (C-1″′),102.3 (C-1″″),81.4 (C-3′),77.9-70.4 (carbons in rhamnose and glucoses),72.2 (C-5″′),71.8 (C-8),62.3/62.2 (C-6′/6″″),36.2 (C-7),18.2 (C-6″′)。通过HMBC分析发现,UGT712A2催化桂叶苷B形成新的苯乙醇苷类化合物桂叶苷B-4-O-葡萄糖苷。其糖基化反应结构式如图4-b所示。
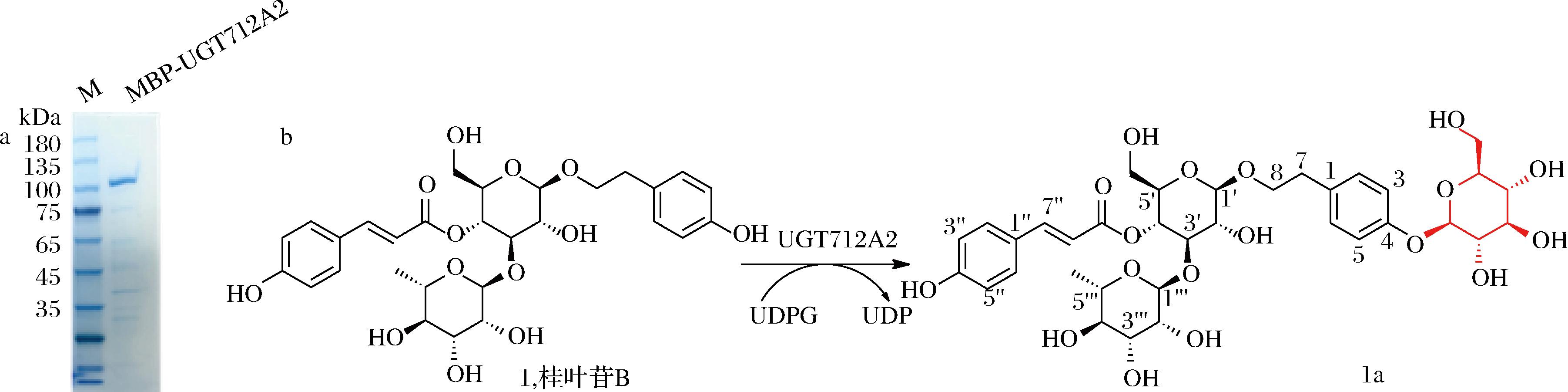
a-重组蛋白MBP-UGT712A2的SDS-PAGE电泳分析图;b-UGT712A2催化桂叶苷B(1)的糖基化反应
图4 糖苷产物1a结构鉴定
Fig.4 Structural identification of glycosylated Product 1a
2.5 系统发育树与底物谱分析
本文将UGT712A2与已鉴定功能的植物UGTs进行系统发育树分析,结果如图5-a所示。UGT712A2和来源于苹果的MdUGT83L3(MD09G1064900)在同一分支上。其可以催化花青素、槲皮素以及山奈酚3种黄酮类化合物的葡萄糖基化反应[16];栀子来源的糖基转移酶UGT85A24(F8 WKW1.1),特异性催化环烯醚萜类糖苷的葡萄糖基化反应[17];与UGT712A2进化关系较近的糖基转移酶,可以催化不同类型化合物的葡萄糖基化反应[18-19]。基于此,本文对UGT712A2的底物谱进行探究。
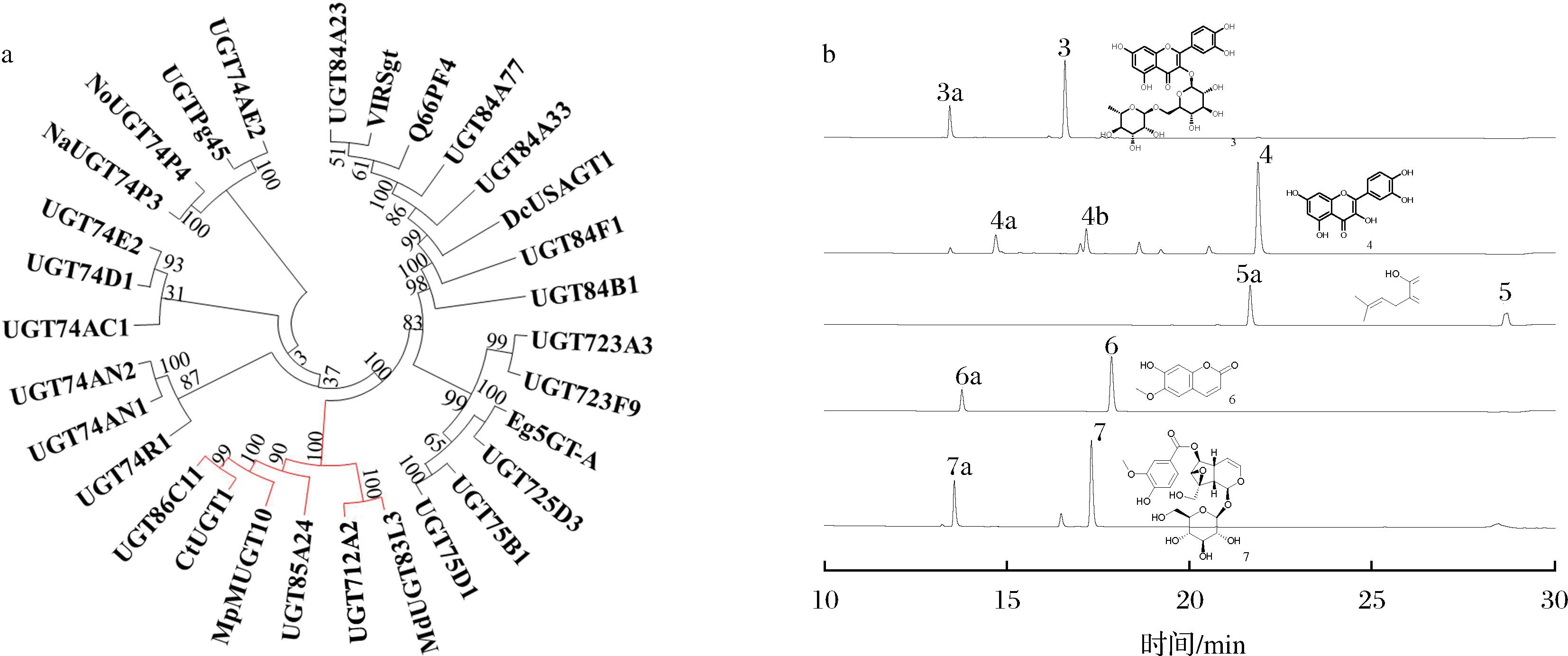
a-系统发育树分析;b-UGT712A2酶催化反应HPLC分析
图5 系统发育树分析与UGT712A2酶催化不同底物HPLC分析
Fig.5 The phylogenetic relationships and HPLC analysis of different substrates catalyzed by UGT712A2 enzyme
本文以UDP-葡萄糖为糖基供体底物,以黄酮类化合物3、4,香豆素类化合物5、6,以及环烯醚萜类化合物7为糖基受体底物,用MBP-UGT712A2进行体外纯酶反应。HPLC结果如图5-b所示,UGT712A2可以催化不同的化合物产生多个新峰。同时,为了确定新峰产物的分子质量,对每个反应进行LC-HRMS检测,结果如图6所示,UGT712A2可以催化不同类型化合物加上一分子或者两分子葡萄糖形成不同类型的糖苷产物3a、4a、4b、5a、6a、7a。

a-3a;b-4a;c-4b;d-5a;e-6a;f-7a
图6 UGT712A2酶催化糖苷产物的LC-HRMS分析
Fig.6 LC-HRMS analysis of the glycosylated glycosides by UGT712A2
3 结论与讨论
本研究以红花钓钟柳转录组为研究对象,基于不同组织中基因差异表达分析筛选到7条糖基转移酶候选基因。通过酶活分析、产物制备、核磁分析等发现糖基转移酶UGT712A2可以催化桂叶苷B苯乙醇部分C-4位置的葡萄糖基化反应生成桂叶苷B-4-葡萄糖苷。同时对UGT712A2进行了底物谱分析发现,它还可以催化毛蕊花糖苷,黄酮类化合物芦丁和槲皮素,香豆素类化合物7-去甲基软木花椒素和东莨菪内酯以及环烯醚萜类化合物胡黄连苷Ⅱ的酚羟基的葡萄糖基化反应,是一种具有功能多样性的糖基转移酶。
随着合成生物学技术以及转录组高通量测序技术的发展,越来越多的糖基转移酶被鉴定出来用于黄酮类、萜类等化合物的糖基化反应,但目前对于催化苯乙醇苷类化合物的糖基转移酶的研究报道较少。YAN等[20]从管花肉苁蓉转录组数据中分析获得一个糖基转移酶UGT71BD1可以催化毛蕊花糖苷等苯乙醇苷类化合物C-3″位的糖基化反应,同时也可以催化芦丁等黄酮类化合物酚羟基的糖基化反应,通过水溶性和药理活性检测发现,其糖苷产物相对于糖基受体底物的水溶性和药理活性都有所增加。体现了糖基转移酶在提高天然产物的生理活性、溶解性以及稳定性等方面的重要的作用。本研究组前期鉴定了来自粗壮女贞的3个参与紫茎女贞苷B生物合成的糖基转移酶,UGT85AF8催化酪醇加上一分子葡萄糖生成红景天苷;UGT79G7催化桂叶苷A加上一分子鼠李糖生成桂叶苷B;UGT79A19催化桂叶苷B加上一分子的鼠李糖生成紫茎女贞苷B。丰富了苯乙醇苷类化合物糖基转移酶类型,并为苯乙醇苷类化合物生物合成途径的解析奠定了基础[12]。
为了丰富苯乙醇苷类化合物糖基转移酶类型,本研究从红花钓钟柳转录组中筛选得到一个新型糖基转移酶UGT712A2,该酶不仅可以催化苯乙醇苷类化合物C-4-OH位的糖基化反应,丰富苯乙醇苷类化合物的糖苷类型,还可以催化其他类型化合物,如芦丁、槲皮素、7-去甲基软木花椒素、东莨菪内酯以及胡黄连苷Ⅱ的葡萄糖基化反应,为后续医药及食品等领域新品研发提供重要候选物,也为植物糖苷类天然产物的异源微生物合成提供基本元件。
[1] LIANG D M,LIU J H,WU H,et al.Glycosyltransferases:Mechanisms and applications in natural product development[J].Chemical Society Reviews,2015,44(22):8350-8374.
[2] AHMED A,PETERS N R,FITZGERALD M K,et al.Colchicine glycorandomization influences cytotoxicity and mechanism of action[J].Journal of the American Chemical Society,2006,128(44):14224-14225.
[3] ZHANG X M,XIE L,LONG J Y,et al.Salidroside:A review of its recent advances in synthetic pathways and pharmacological properties[J].Chemico-Biological Interactions,2021,339:109268.
[4] ARUMUGAM B,SUBRAMANIAM A,ALAGARAJ P.Stevia as a natural sweetener:A review[J].Cardiovascular and Hematological Agents in Medicinal Chemistry,2020,18(2):94-103.
[5] DANBY P M,WITHERS S G.Advances in enzymatic glycoside synthesis[J].ACS Chemical Biology,2016,11(7):1784-1794.
[6] XUE Z Z,YANG B.Phenylethanoid glycosides:Research advances in their phytochemistry,pharmacological activity and pharmacokinetics[J].Molecules,2016,21(8):991.
[7] XIE J,DENG J,TAN F,et al.Separation and purification of echinacoside from Penstemon barbatus (can.) roth by recycling high-speed counter-current chromatography[J].Journal of Chromatography B,Analytical Technologies in the Biomedical and Life Sciences,2010,878(28):2665-2668.
[8] PAQUETTE S,MØLLER B L,BAK S.On the origin of family 1 plant glycosyltransferases[J].Phytochemistry,2003,62(3):399-413.
[9] WANG Z L,WANG S,XU Z,et al.Highly promiscuous flavonoid 3-O-glycosyltransferase from Scutellaria baicalensis[J].Organic Letters,2019,21(7):2241-2245.
[10] HE J B,CHEN K,HU Z M,et al.UGT73F17,a new glycosyltransferase from Glycyrrhiza uralensis,catalyzes the regiospecific glycosylation of pentacyclic triterpenoids[J].Chemical Communications,2018,54(62):8594-8597.
[11] KOEDUKA T,UEYAMA Y,KITAJIMA S,et al.Molecular cloning and characterization of UDP-glucose:Volatile benzenoid/phenylpropanoid glucosyltransferase in Petunia flowers[J].Journal of Plant Physiology,2020,252:153245.
[12] YANG Y H,WU Y N,ZHUANG Y B,et al.Discovery of glycosyltransferases involved in the biosynthesis of ligupurpuroside B[J].Organic Letters,2021,23(20):7851-7854.
[13] YANG Y H,XI D Y,WU Y N,et al.Complete biosynthesis of phenylethanoid glycoside verbascoside[J].Plant Communications,2023:100592.
[14] LI Y,BALDAUF S,LIM E K,et al.Phylogenetic analysis of the UDP-glycosyltransferase multigene family of Arabidopsis thaliana[J].Journal of Biological Chemistry,2001,276(6):4338-4343.
[15] CHEN C J,CHEN H,ZHANG Y,et al.TBtools:An integrative toolkit developed for interactive analyses of big biological data[J].Molecular Plant,2020,13(8):1194-1202.
[16] LI Y J,LI P,ZHANG L,et al.Genome-wide analysis of the apple family 1 glycosyltransferases identified a flavonoid-modifying UGT,MdUGT83L3,which is targeted by MdMYB88 and contributes to stress adaptation[J].Plant Science,2022,321:111314.
[17] NAGATOSHI M,TERASAKA K,NAGATSU A,et al.Iridoid-specific glucosyltransferase from Gardenia jasminoides[J].Journal of Biological Chemistry,2011,286(37):32866-32874.
[18] KARLOVA R,BUSSCHER J,SCHEMPP F M,et al.Detoxification of monoterpenes by a family of plant glycosyltransferases[J].Phytochemistry,2022,203:113371.
[19] XU X P,YAN Y R,HUANG W Q,et al.Molecular cloning and biochemical characterization of a new coumarin glycosyltransferase CtUGT1 from Cistanche tubulosa[J].Fitoterapia,2021,153:104995.
[20] YAN Y R,MO T,HUANG W Q,et al.Glycosylation of aromatic glycosides by a promiscuous glycosyltransferase UGT71BD1 from Cistanche tubulosa[J].Journal of Natural Products,2022,85(7):1826-1836.