质粒是细胞染色体外的双链闭合环状DNA分子,具有独立的自主复制能力,广泛存在于细菌和真菌。质粒本身在生物进化过程中发挥着作用[1]。作为载体,质粒被现代基因工程、合成生物学等领域利用来研究新基因的功能[2-3]。利用质粒作载体携带外源基因,通过转化的方法将其转入微生物细胞,相比将外源基因整合到宿主基因组来说更加便捷方便。大量研究发现,质粒在微生物宿主细胞内拷贝数与所携带基因的表达量关系密切[4-7],优化质粒拷贝数这一方法广泛在生物化工领域用于优化关键基因表达,提高生物催化效率[8]。基于天然质粒构建的优化体系存在诸多局限,近年来已有众多研究,利用前沿合成生物学手段构建出了更先进的质粒调控优化系统。为此,本文对调控质粒拷贝数优化生物催化效率展开综述,并探讨了通过合成生物学手段进一步优化系统的新研究方向。
1 质粒复制及拷贝数简介
质粒的复制大致包括复制起始、延伸和终止。复制的起始从复制起点 (origin of replication,replicon) 开始,涉及复杂的蛋白—蛋白、蛋白—DNA相互作用。不同的复制起点序列具有不同的序列结构特征。质粒复制的分子生物学机理已经研究的较为透彻,详见相关综述总结[9]。
质粒拷贝数是指每个微生物细胞中质粒相对于染色体的个数[10-11]。对于给定宿主、固定的生长条件,具有特定复制起点序列的质粒通常具有特征拷贝数(或一定拷贝数范围),这是特定质粒系统通过调节其复制相关元件来实现的。控制系统通过纠正单个细胞中平均拷贝数的偏差,将复制速率维持在稳定状态,进而决定了质粒拷贝数。相关研究表明,质粒复制起始起作用的负调节因子(抑制剂)参与此调控,主要调控机制包括:a)利用反义RNA (antisense RNA);b)利用转录抑制(transcription repression);c)利用复制起点上的迭代子序列(iterons) [12-13]。
质粒拷贝数可以利用基于荧光定量PCR (qPCR)的方法进行绝对定量[14]。拷贝数测量结果表明,可以将质粒大致划分为高拷贝质粒、低拷贝质粒、单拷贝质粒。a)高拷贝数质粒是指拷贝数较大的质粒,在每个细胞中可达到几十乃至上千份拷贝,其复制与宿主染色体不同步,仅与质粒本身复制子的控制有关,也称为松弛型复制控制的质粒[15]。高拷贝质粒一般用作表达目的基因的载体,因为高拷贝的基因通常能导致获得较高产量的目的基因产物[16];b)低拷贝数质粒是指拷贝数较低的质粒,少到在每个细胞中只有一到几份拷贝[17],与染色体的复制同步进行,受质粒本身复制子和宿主染色体的双重调控,也称为严谨型复制控制的质粒,在宿主细胞中表达效率较低,甚至可能导致质粒分配到子细胞失败[18],因而低拷贝质粒在实际应用中使用较少;c)单拷贝质粒的研究及应用很少。除此之外,宿主的不同、生长条件不同 (培养基成分、温度),也会影响质粒拷贝数[19-21]。
研究者们对模式原核生物大肠杆菌(Escherichia coli)和模式真核微生物酿酒酵母 (Saccharomyces cerevisiae)的质粒系统研究最为透彻。大肠杆菌及酿酒酵母常见的复制起点、质粒拷贝数以及常用质粒分别如表1及表2所示,其他非模式微生物的质粒系统常以它们为参照开发构建。
表1 大肠杆菌常见复制起点及拷贝数
Table 1 Common replication starting point and copy number of Escherichia coli
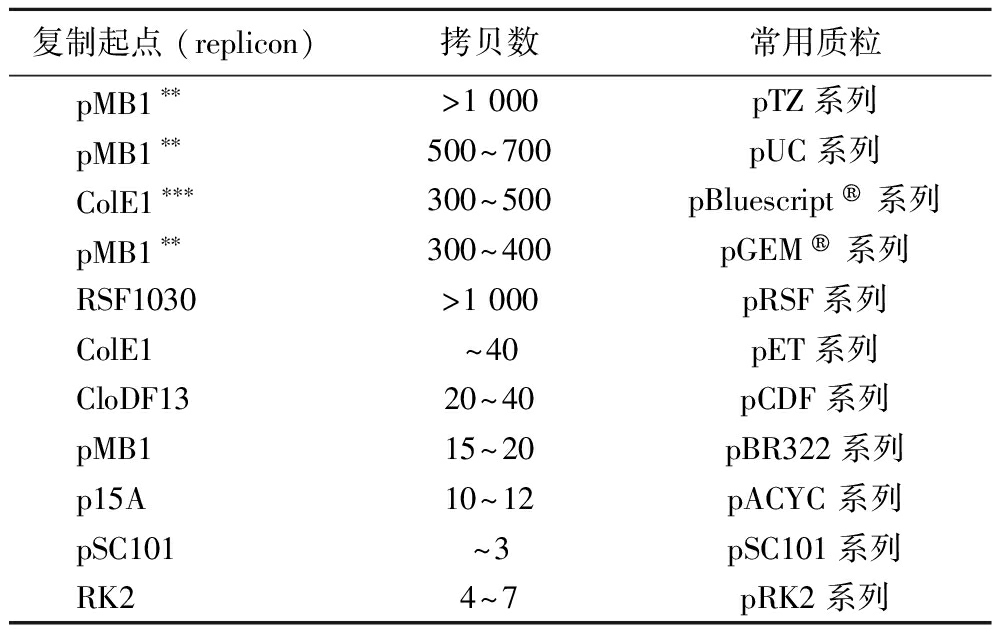
复制起点 (replicon)拷贝数常用质粒pMB1∗∗>1 000pTZ系列pMB1∗∗500~700pUC系列ColE1∗∗∗300~500pBluescript 系列pMB1∗∗300~400pGEM 系列RSF1030>1 000pRSF系列ColE1~40pET系列CloDF1320~40pCDF系列pMB115~20pBR322系列p15A10~12pACYC 系列pSC101~3pSC101系列RK24~7pRK2系列
注:资料来源为Qiagen公司网站;**表示pMB1突变序列;***表示ClolE1突变序列。
表2 酵母常见复制起点及拷贝数
Table 2 Common replication starting point and copy number of Saccharomyces cerevisiae

复制起点拷贝数常用质粒2 μm4~30pRS42XCEN/ARS44~7pRS41X
2 天然质粒系统在优化生物催化的应用
大量研究发现,质粒在宿主细胞内拷贝数与所携带基因的表达量关系密切[4-7]:一般来说,目的基因的表达量随重组质粒在宿主细胞中拷贝数的增加而提高,进而提高了目标蛋白的表达量。这种对应关系甚至在质粒拷贝数>300仍存在[9]。不过,当质粒拷贝数超过某个阈值,会严重影响系统稳定性,反而使得目标蛋白表达量及目标小分子化合物合成效率降低[22]。上述研究也表明,并非质粒(或目标基因)的拷贝数越高,生物催化的效率也越高。测定不同质粒拷贝数的影响(优化质粒拷贝数),是一种简单、有效且已经被广泛应用的优化生物催化效率的策略[8] (表3)。
表3 优化天然质粒系统提高生物催化效率
Table 3 Optimizing natural plasmid systems to improve biocatalytic efficiency
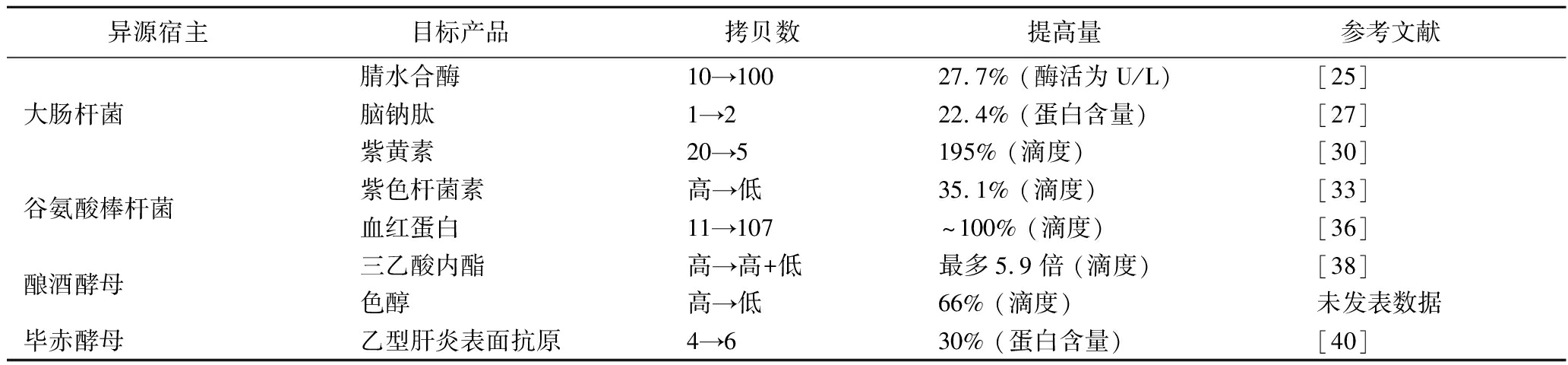
异源宿主目标产品拷贝数提高量参考文献大肠杆菌腈水合酶 10→10027.7% (酶活为U/L)[25]脑钠肽 1→222.4% (蛋白含量)[27]紫黄素 20→5195% (滴度)[30]谷氨酸棒杆菌紫色杆菌素 高→低35.1% (滴度)[33]血红蛋白 11→107~100% (滴度)[36]酿酒酵母三乙酸内酯 高→高+低最多5.9倍 (滴度)[38]色醇 高→低66% (滴度)未发表数据毕赤酵母乙型肝炎表面抗原4→630% (蛋白含量)[40]
2.1 调控质粒拷贝数原核系统生物催化
2.1.1 在大肠杆菌中调控质粒拷贝数的应用
腈水合酶是一类重要的金属酶,可以催化腈类化合物水合生成相应的酰胺,在合成酰胺已广泛应用。但是,目前腈水合酶的数量较少,催化效率低下[23]。为提高腈水合酶的表达量,史悦等[24]在大肠杆菌BL21(DE3)中利用质粒pET28a(+)异源表达诺卡氏菌来源的腈水合酶,比活性可高达450 U/mg,远高于野生诺卡氏菌,说明了异源多拷贝表达相比野生天然宿主表达具有明显优势。王丽燕等[25]在BL21(DE3)中表达Klebsiella oxytoca来源的腈水合酶,考察了不同拷贝数质粒对腈水合酶表达的影响,发现蛋白浓度(g/L)和粗酶活力(U/L)随着拷贝数的提高具有增加。使用pCDFDuet或者pETDuet质粒(拷贝数15~20)比使用pACYCDuet质粒(拷贝数10~12) 单位蛋白的粗酶活力(U/g)也随质粒拷贝数增加而提高,但是使用高拷贝质粒pRSFDuet (拷贝数100~200) 却对提高酶表达效果并不明显。因此,优化质粒拷贝数是提高酶异源表达量的简单可行的办法。
脑钠肽(brain natriuretic peptide,BNP)在诊断心力衰竭、调节血压等方面有很大的医学作用,尤其是在诊断是否为心力衰竭患者方面,已成为公认的诊断心力衰竭的血浆标志物[26]。异源宿主表达是一种简便高效合成BNP的有效方法。易俊波等[27]在大肠杆菌DH5α和BL21(DE3)中利用质粒pCW111异源表达了BNP。作者在pCW111上分别克隆单个和两个BNP的基因表达盒。SDS-PAGE分析发现BNP的表达量随着目标基因拷贝数增加,菌体总蛋白占比从单拷贝的8.12%增加到两拷贝的9.94%,这也预示了利用多拷贝质粒进一步增加BNP的潜力。
紫黄素 (violaxanthin) 是一种存在于高等植物和微藻中重要的天然叶黄素,且被证实具有抗氧性、抗癌等生物活性,因而该物质在化妆品、药品等邻域具有潜在的应用[28]。紫黄素在微藻中的含量可变的,它会受到外部环境(如光照强度)的影响[29]。将合成紫黄素关键合成途径引入工程菌,通过代谢工程改造构建高产菌,则可以克服上述缺点。DONG等[30]发现尝试将催化合成紫黄素的玉米黄质环氧化酶(zeaxanthin epoxidase,ZEP)基因克隆到不同拷贝数的质粒上转入大肠杆菌表达,发现低拷贝质粒(pSC101 ori)效果最好,紫黄素产量相比用高拷贝质粒(pMB1 ori)提高了195%(图1)。该研究说明了并非质粒拷贝数越高目标产物产量越高,这可能跟高拷贝质粒引起的细胞负担有关[31]。
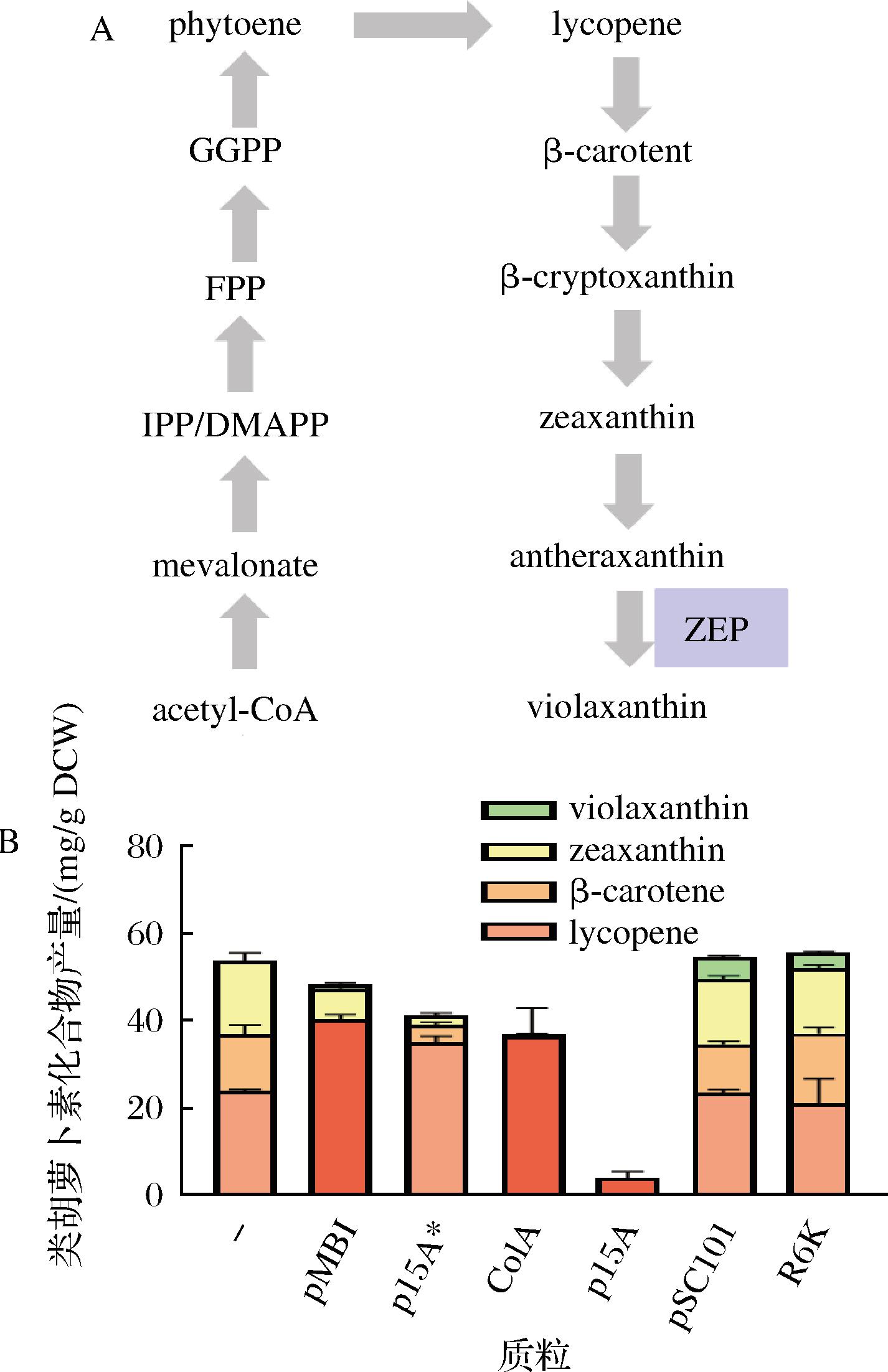
A-紫黄质合成途径;B-使用不同的质粒表达ZEP并应用于类胡萝卜素的合成
图1 利用不同拷贝数质粒携带ZEP基因优化紫黄素产量[30]
Fig.1 Optimizing violacein production using plasmids with different copy numbers carrying ZEP genes[30]
2.1.2 在谷氨酸棒杆菌中调控质粒拷贝数的应用
紫色杆菌素是一种蓝紫天然色素,具有抗肿瘤、抗致病菌等多种活性,在食品、印染、化妆品和医药行业都有重要的潜在应用价值[32]。产生该色素的天然菌种多为条件致病菌且难于培养,由于紫色杆菌素的前体物质为色氨酸,利用高产氨基酸的异源谷氨酸棒杆菌合成该色素成为一种很好的选择。卢丹妮等[33]在谷氨酸棒杆菌中构建了紫色杆菌素的合成途径。研究发现,基于pTET3的低拷贝重组质粒 (p15A复制子,拷贝数~6) 比基于pEC-XK99E的高拷贝重组质粒 (rep/ColE复制子,拷贝数~360)能够使谷氨酸棒杆菌生产紫色杆菌素的产量提高35.1%。该结果也可能跟前述高拷贝质粒引起的细胞负担有关。
血红蛋白广泛存在于大豆、玉米等植物中,该含有血红素的蛋白质可被用作颜色增强剂[34]、色素添加剂[35]等。植物血红蛋白在作为食品添加剂方面具有巨大的价值,且利用微生物可持续生产血红蛋白是一种有效途径。WANG等[36]在谷氨酸棒杆菌中实现了植物来源血红蛋白的高效合成。该研究者们对调控质粒拷贝数相关的起始蛋白编码基因 copA 进行了突变,获得了pXMJ19质粒拷贝数提高的突变体:copAC1033T,copAC1033 del,copAC1032A,copAC1033G,对应测定出的拷贝数分别为107、107、250、318;而表达野生copA时pXMJ19质粒拷贝数仅为11。蛋白表达实验表明,在一定范围内蛋白表达量与拷贝数呈正相关,当质粒拷贝数从11增加107,血红蛋白表达量增加约2倍,但继续增加质粒拷贝数反而导致产量下降。
2.2 调控质粒拷贝数在真核生物合成生物学中的应用
2.2.1 在酿酒酵母中调控质粒拷贝数的应用
三乙酸内酯 (triacetic acid lactone,TAL) 是一种简单的聚酮化合物,可在酵母中通过非洲菊III型2-吡喃酮合酶 (2-PS)从乙酰-CoA和丙二酰-CoA合成,被认为是一种可转化为多种高价值化合物的大宗化学品[37]。YOCUM等[38]通过酵母过氧化物酶体表面展示技术,将TAL合成相关3个酶锚定于细胞质一面,充分利用过氧化酶体流出的乙酰-CoA代谢流合成TAL。研究表明同时利用高拷贝质粒(2 μm)携带的自由酶加上低拷贝质粒(CEN/ARS4)携带的锚定酶,比仅利用高拷贝自由酶或者低拷贝锚定酶,TAL产量分别提高了3.2倍和5.9倍。
色醇是一种源自L-色氨酸的代谢产物,表现出广泛的生物活性,并且作为一种关键的药物前体,色醇及其衍生物化学方式转化为众多具有药用价值的化合物。我们的研究表明色醇可以通过酿酒酵母以葡萄糖为碳源合成。分别用基因组整合、低拷贝质粒(CEN/ARS4)或高拷贝质粒(2 μm)表达限速步骤 (TRP2S65R,S76L),发现低拷贝质粒效果最好,基因组整合次之,高拷贝质粒效果最差。这说明色醇的最终产量是基因表达量和细胞负担两个因素平衡的结果 (未发表数据)。
2.2.2 在毕赤酵母中调控质粒拷贝数的应用
乙型肝炎表面抗原(HBsAg)是HBV的主要特征蛋白,可以作为感染HBV的诊断指标,在减少HBV感染率且消灭乙肝方面作用很大[39]。乙肝疫苗生产成本高,构建多拷贝质粒并在毕赤酵母中高效表达乙型肝炎表面抗原能减低疫苗成本。实验表明,乙型肝炎表面抗原在毕赤酵母中的表达量与质粒拷贝数呈正相关,质粒拷贝数从4增加到6,HBsAg在菌体破碎液的含量增加了30%,且拷贝数的提高并未对毕赤酵母的生长产生影响[40]。
3 通过合成生物学手段改造质粒以精确调控拷贝数范围
综上研究表明,利用天然复制子序列,可以构建具有不同拷贝数的质粒。构建起一系列不同拷贝数的质粒合集,可以广泛用于优化关键基因表达量,进而提高生物催化效率,提高目标产物产量。前述研究进展也表明,并非质粒拷贝数越高,目标产物也越高,这也凸显出优化表达系统质粒拷贝数的必要性。
然而,利用天然复制子构建的质粒,拷贝数并不是均匀分散的,还存在着动态调控范围小等问题。特别是对于真核酵母体系,常用的复制起点只有CEN6/ARS4和2 μm,其拷贝数上限仅为30左右 (表1)。为了克服上述局限,近年来,国内外研究者充分利用合成生物学及分子生物学的前沿技术手段,构建出了质粒拷贝数调控分布更均匀、拷贝数上限更高、可调控范围更大的系统。这些系统应用于优化生物催化,显著提高了生物合成的催化效率 (表4)。
表4 利用合成生物学技术优化质粒拷贝数系统提高生物催化效率
Table 4 Using synthetic biology technology to optimize plasmid copy number systems to improve biocatalytic efficiency
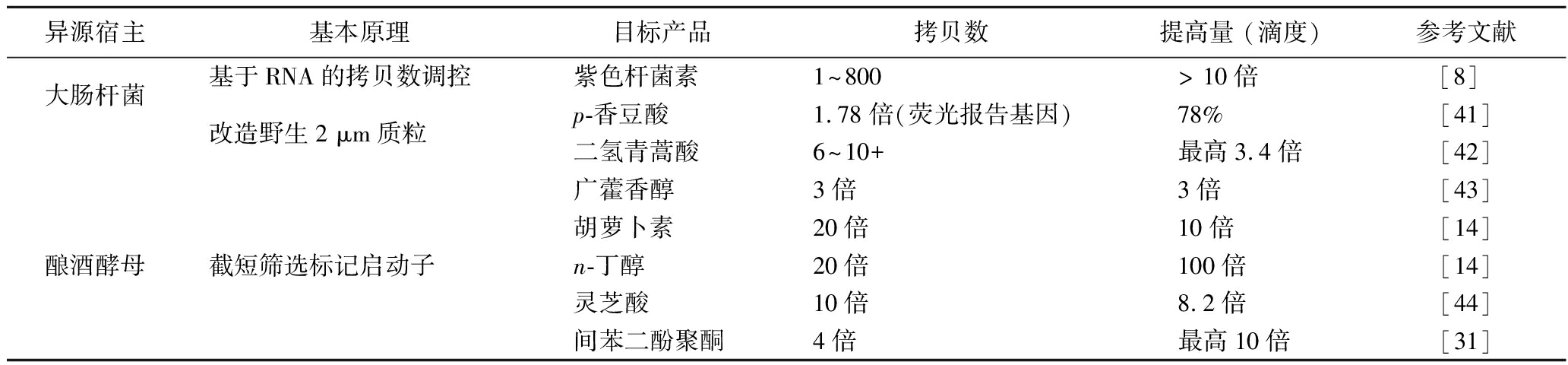
异源宿主基本原理目标产品拷贝数提高量 (滴度)参考文献大肠杆菌基于RNA的拷贝数调控紫色杆菌素1~800> 10倍[8] 改造野生2 μm质粒p-香豆酸1.78倍(荧光报告基因)78%[41]二氢青蒿酸6~10+最高3.4倍[42]广藿香醇3倍3倍[43]胡萝卜素20倍10倍[14]酿酒酵母截短筛选标记启动子n-丁醇20倍100倍[14]灵芝酸10倍8.2倍[44]间苯二酚聚酮4倍最高10倍[31]
3.1 调控质粒拷贝数在真核生物合成生物学中的应用
大肠杆菌中应用广泛的、含有ColE1复制起点的质粒,其拷贝数的控制通过两种RNA实现:a)引发RNA (RNA-p),它以顺式方式充当复制起点附近的引物;b)抑制RNA (RNA-i),其转录为RNA-p的反义序列,并通过在原点附近杂交之前与引发 RNA 结合而发挥反式作用,RNA-p/RNA-i的比例决定着质粒拷贝数[9]。根据上述机制,ROUCHES等[8]通过调控RNA-p或RNA-i的表达量,构建起了拷贝数可变的2个ColE1质粒系统。在第一个系统中,研究者将Ptet启动子设置于RNA-p基因前,通过添加无水四环素 (aTc) 控制RNA-p表达量;而RNA-i的表达受ColE1复制子序列自带启动子Pi控制。实验测定在aTc添加浓度为0~100 ng/mL,该系统质粒拷贝数为1.4~50/细胞。在第二个系统中,研究者以ColE1质粒上控制RNA-p的天然启动子Pp和控制RNA-i的天然启动子Pi的-35和-10盒子上引入定点和随机突变,构建出了1 194个RNA-p/RNA-i比例不同(质粒拷贝数不同)基因文库。序列及拷贝数测定表明质粒拷贝数范围为1~800/细胞,并且此系统还避免了外源添加aTc。该系统被用于测定质粒拷贝数与细胞负担的相关性,表明每个质粒会给细胞带来0.063%的代谢负担。上述2个系统还被应用于了优化生物合成途径提高紫色杆菌素产量。第一个系统因为拷贝数变化范围小、拷贝数较低,紫色杆菌素产量随aTc添加量的增加而增长;第二个系统由于过高质粒拷贝数带来的代谢负担逐渐加重,紫色杆菌素的产量在质粒拷贝数~300时出现峰值。
p15A复制子和ColE1复制子机制类似。LI等根据p15A的复制原理,巧妙设计一套基因回路,构建了受p-香豆酸诱导调控拷贝数的质粒系统。实验数据表明,该系统能够动态调控代谢通路中关键基因拷贝数,平衡细胞生长与合成目标产物p-香豆酸的代谢通量。与质粒拷贝数不变的静态系统相比,该动态调控系统p-香豆酸摇瓶产量提高78%,达到1.69 g/L[42](图2)。
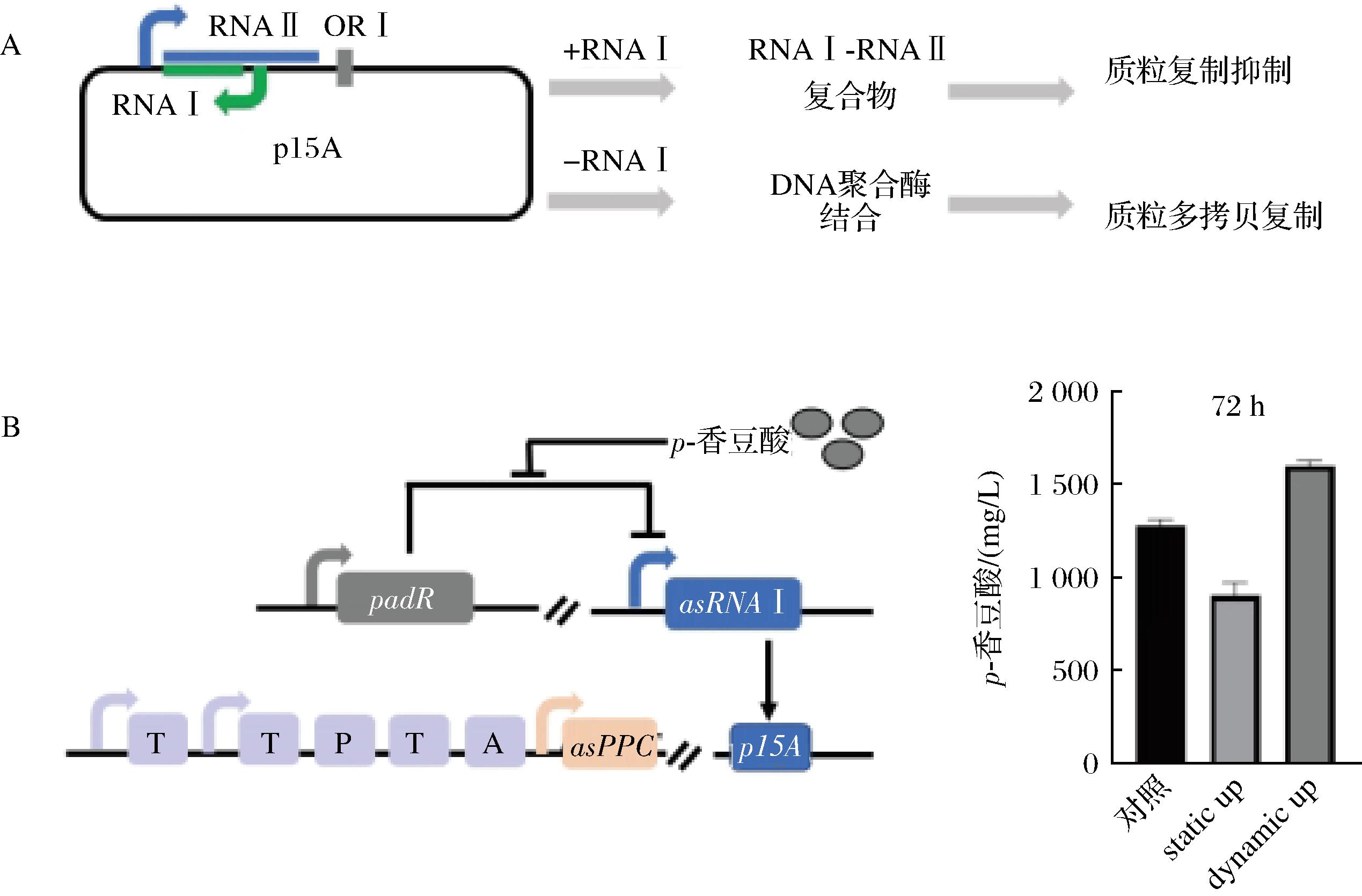
A-RNAI影响质粒复制机理;B-基因拷贝动态控制在p-香豆酸生物合成中的应用
图2 利用p15A质粒复制机理设计基因回路进而动态调控代谢途径提高p-香豆酸产量[41]
Fig.2 Utilizing the p15A plasmid replication mechanism to design gene circuits to dynamically regulate metabolic pathways to increase p-coumaric acid production
JOSHI等[45]近期开发了原核系统中灵活的、能随时间变化的动态质粒拷贝数的新系统TULIP (TUnable Ligand Inducible Plasmid)。TULIP的设计基于pSC101复制起点的自动调节机制设计,与前述ColE1复制机制不同。pSC101复制子通过RepA调控蛋白单体-二聚体的平衡使质粒拷贝数控制在3左右。该研究者们利用RepA突变体,设计了新的基因回路,使pSC101固定拷贝数系统变成了受异丙苯酸诱导调控、拷贝数动态调控范围达到20倍 (3~60拷贝/细胞)的新系统。进一步的表征表明,TULIP在常用的大肠杆菌菌株(NEBStable、DH10B、NEBExpress、BW25113和MG1655) 和生长培养基(M9-葡萄糖、M9-甘油、Lysogeny Broth和Super Optimal Broth)中表现稳定。除了异丙苯酸诱导系统,TULIP也被改造为由其他小分子诱导体系,如N-(3-氧代-己酰基)-L-高丝氨酸内酯[N-(3-oxododecanoyl)-L-homoserine lactone,AHL)],香草酸,异丙基-β-D-硫代半乳糖苷。
3.2 利用合成生物学技术优化真核质粒拷贝数系统
二氢青蒿酸能通过独立且自发的代谢途径形成青蒿素[46],且由于青蒿素的医药价值不断被发现,对于青蒿素合成的研究不断深入。其中利用二氢青蒿酸在酿酒酵母中异源表达,从而进一步合成青蒿素在提高产量方面取得了较好的效果。曾薄轩研究发现,用传统的2 μm多拷贝质粒表达的关键基因ADS、CYP71AV1和DBR2不够稳定高效,进而影响二氢青蒿酸产量[42]。可以复制2 μm多拷贝质粒的酵母自身还含有内源性的野生2 μm质粒 (wt 2 μm质粒),两者相比wt 2 μm质粒拥有更高的拷贝数,且在不含任何酵母生长必须基因的前提下,能随细胞分裂稳定遗传。因此,在不破坏稳定遗传所需必须基因的基础上,作者基于wt 2 μm质粒,设计了利用CRISPR/Cas9基因编辑技术将外源基因插入STB与RAF1基因之间,开发出了pE 2 μm多拷贝质粒系统,可以方便进行外源基因克隆。实验结果表明,利用pE 2 μm多拷贝质粒表达关键基因,相比传统2 μm质粒系统,在有筛选压力和无筛选压力的条件下,二氢青蒿酸产量分别提高到了65%和 3.4倍。类似的,YANG等[47]利用传统克隆方法在wt 2 μm质粒REP1启动子和RAF1启动子之间插入了多克隆位点,方便进行外源基因克隆。研究者们将酪醇合成途径基因引入该系统,酪醇相比基于传统2 μm质粒的体系也有提高。
酿酒酵母常用质粒系统具有拷贝数较低、动态变化范围窄的缺点,很大程度上限制了更多的应用。相关基础研究发现,酵母质粒拷贝数可以调控其筛选标记基因/蛋白的表达量。CHEN等[43]发现通过将泛素/N-降解决定子标签 (ubi-tag)融合到筛选标记N可以破坏标记蛋白的稳定性,进而增加载体的拷贝数和同一载体目标蛋白的表达量。这种增加质粒拷贝数的方法使报告蛋白LacZ的活性最高增加100%,将广藿香醇合成途径构建进该体系,其产量提高了3倍。
LIAN等[14]随后系统性利用截短筛选标记基因启动子的方法来弱化标记蛋白表达量,进而增加质粒拷贝数。他们发现对某些筛选标记 (HIS3,URA3,KanR,HygR),2 μm多拷贝质粒随筛选标记启动子截短而增加;但是对另外一些筛选标记 (TRP1,LEU2)则无此相关性,最高拷贝数达到了100 拷贝/细胞。KanR和HygR的系统随即被用作胡萝卜素及n-丁醇代谢途径的优化,产量较野生型最高增加10倍和100倍。类似通过弱化筛选标记基因表达、进而增加筛选压力来提高质粒拷贝数的方法还用于了重要天然产物灵芝酸[44]。
近期,本课题组将上述拷贝数增加且可调控的方法应用到了双质粒表达真菌I型聚酮合酶的体系中,用于优化产量极低的间苯二酚类聚酮,因为该类化合物极具药用价值[32]。通过组合优化,我们将新发现的化合物产量提高最高10倍(图3)。这加速了此类微量产物的分离纯化和结构鉴定,进而促进了结构新颖的非天然真菌聚酮的合成以及加深了对I型真菌聚酮合酶催化机制的理解。
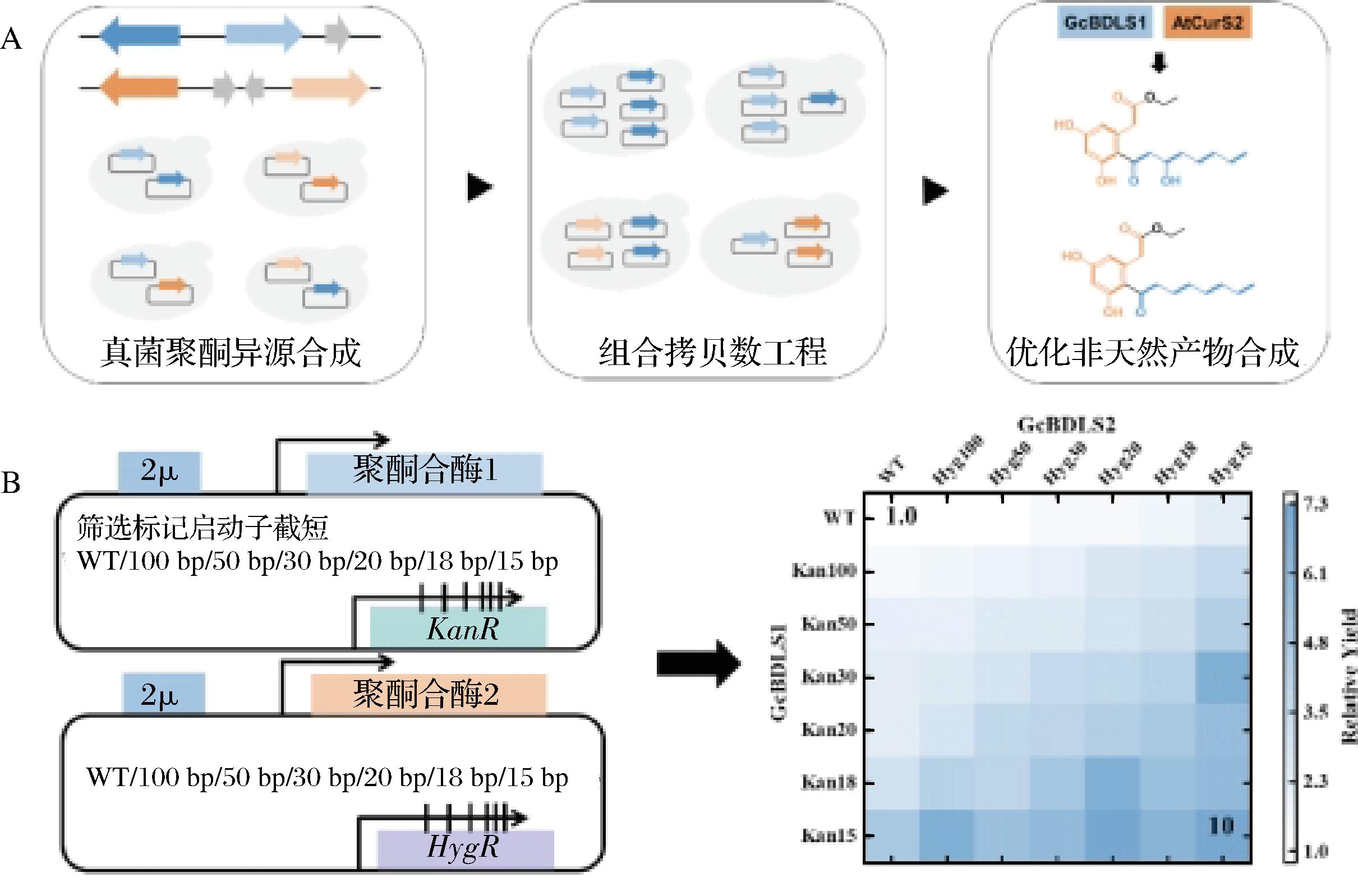
A-组合拷贝数工程优化非天然产物合成;B-通过组合调节还原性聚酮合酶以及非还原性聚酮合酶的质粒拷贝数来优化生产非天然产物
图3 通过截短筛选标记基因的启动子增加质粒拷贝数借以组合优化真菌聚酮合成[31]
Fig.3 Combinatorial optimization of fungal polyketide synthesis via plasmid copy number engineering[31]
同时有实验证明,在截短启动子前提下,不同浓度的抗生素也会影响质粒的拷贝数,且启动子截短程度较高的对抗生素浓度变化更加敏感[14]。结合启动子截短工程和抗生素的不同浓度,可以构建拷贝数变化梯度更大的质粒系统。
4 总结与展望
综上,天然复制子、拷贝数不同的质粒,已经广泛用于优化关键基因表达量,进而提高生物催化效率。然而,天然复制子系统也具有拷贝数分布区间不均匀、动态调控范围小等局限,这些问题对于真核酵母体系尤为突出。近年来,为了克服上述局限,国内外研究者充分利用合成生物学及分子生物学的前沿技术手段,设计并构建出了质粒拷贝数调控分布更均匀、拷贝数上限更高、可调控范围更大的系统。这些系统应用于优化生物催化,显著提高了生物合成的催化效率。但总体而言,目前相关研究不是很多,尤其在真核生物合成生物学领域,这需要对质粒复制机制更深入研究和理解。可以预见,上述领域的进一步进展,可以为合成生物学提供更多解决方案。
[1] RODR GUEZ-BELTR
GUEZ-BELTR N J,DELAFUENTE J,LE
N J,DELAFUENTE J,LE N-SAMPEDRO R,et al.Beyond horizontal gene transfer:The role of plasmids in bacterial evolution[J].Nature Reviews.Microbiology,2021,19(6):347-359.
N-SAMPEDRO R,et al.Beyond horizontal gene transfer:The role of plasmids in bacterial evolution[J].Nature Reviews.Microbiology,2021,19(6):347-359.
[2] 姚庆智.细菌质粒及其在基因工程中的应用[J].内蒙古科技与经济,2000(5):24-26.
YAO Q Z.Bacterial plasmid and its application in genetic engineering[J].Inner Mongolia Science Technolgy and Economy,2000(5):24-26.
[3] SHETTY R P,ENDY D,JR KNIGHT T F.Engineering BioBrick vectors from BioBrick parts[J].Journal of Biological Engineering,2008,2:5.
[4] 王宇鹏.qPCR方法检测不同条件下重组乳杆菌的pLA质粒拷贝数[D].大庆:黑龙江八一农垦大学,2014.
WANG Y P.The pLA plasmid of recombinant Lactobacillus qPCR method for detection of different conditions of PCN[D].Daqing:Heilongjiang Bayi Agricultural University,2014.
[5] KARL F.Plasmid copy number and plasmid stability[J].Advances in Biochemical Engineering/Biotechnology,2004,86:47-82.
[6] 冯尔玲,王恒樑,林云,等.质粒拷贝数对宋内氏菌I相O-抗原表达的影响[J].生物技术通讯,2000,11(3):202-205.
FENG E L,WANG H L,LIN Y,et al.Influence of plasmid copies on the expression of Shigella sonnei form I O-antigen[J].Letters in Biotechnology,2000,11(3):202-205.
[7] 邓明勇,李画敏,魏子贡.lys1-d缺陷型标记介导毕赤酵母超高拷贝质粒整合[J].湖北大学学报(自然科学版),2020,42(5):471-475; 483.
DENG M Y,LI H M,WEI Z G.Superhigh-copy plasmid integration in Komagataella phaffii mediated by lys1-d auxotrophic marker[J].Journal of Hubei University (Natural Science),2020,42(5):471-475; 483.
[8] ROUCHES M V,XU Y S,CORTES L B G,et al.A plasmid system with tunable copy number[J].Nature Communications,2022,13(1):3908.
[9] DEL SOLAR G,GIRALDO R,RUIZ-ECHEVARR A M J,et al.Replication and control of circular bacterial plasmids[J].Microbiology and Molecular Biology Reviews:MMBR,1998,62(2):434-464.
A M J,et al.Replication and control of circular bacterial plasmids[J].Microbiology and Molecular Biology Reviews:MMBR,1998,62(2):434-464.
[10] 付立霞,高雯,韩先干,等.实时定量PCR测定爱德华氏菌隐蔽质粒的拷贝数[J].淡水渔业,2018,48(4):63-70.
FU L X,GAO W,HAN X G,et al.Real time PCR quantification of the copy number of cryptic plasmids in Edwardsiella ictaluri[J].Freshwater Fisheries,2018,48(4):63-70.
[11] SCHMIDT T,FRIEHS K,FLASCHEL E.Rapid determination of plasmid copy number[J].Journal of Biotechnology,1996,49(1-3):219-229.
[12] WATVE M M,DAHANUKAR N,WATVE M G.Sociobiological control of plasmid copy number in bacteria[J].PLoS One,2010,5(2):e9328.
[13] SAN MILLAN A,HEILBRON K,MACLEAN R C.Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations[J].The ISME Journal,2014,8(3):601-612.
[14] LIAN J Z,JIN R,ZHAO H M.Construction of plasmids with tunable copy numbers in Saccharomyces cerevisiae and their applications in pathway optimization and multiplex genome integration[J].Biotechnology and Bioengineering,2016,113(11):2462-2473.
[15] 钟春英.苏云金芽胞杆菌菌株YBT-1520内生质粒生物量的鉴定[D].武汉:华中农业大学,2010.
ZHONG C Y.Evaluation of plasmid DNA amount in Bacillus Thuringiensis strain YBT-1520[D].Wuhan:Huazhong Agricultural University,2010.
[16] JAHN M,GÜNTHER S,MÜLLER S.Non-random distribution of macromolecules as driving forces for phenotypic variation[J].Current Opinion in Microbiology,2015,25:49-55.
[17] PROVIDENTI M A,O′BRIEN J M,EWING R J,et al.The copy-number of plasmids and other genetic elements can be determined by SYBR-Green-based quantitative real-time PCR[J].Journal of Microbiological Methods,2006,65(3):476-487.
[18] SUMMERS D K.The kinetics of plasmid loss[J].Trends in Biotechnology,1991,9(1):273-278.
[19] 周涛.质粒拷贝数的测定方法[J].生物技术通讯,1999,10(1):51-57.
ZHOU T.Determination methods of plasmid copy number[J].Letters in Biotechnology,1999,10(1):51-57.
[20] 王剑,夏杰,陆兵,等.培养基组分对hCG核酸疫苗质粒拷贝数的影响[J].化学与生物工程,2009,26(5):46-49.
WANG J,XIA J,LU B,et al.Effect of medium composition on plasmid amplification of hCG DNA vaccine[J].Chemistry &Bioengineering,2009,26(5):46-49.
[21] 丁满生,马文峰,郭美锦,等.温度诱导模式对重组大肠杆菌质粒拷贝数的影响[J].华东理工大学学报(自然科学版),2006,32(1):33-37.
DING M S,MA W F,GUO M J,et al.Effects of temperature induction modes on the plasmid copy number in Escherichia coli[J].Journal of East China University of Science and Technology,2006,32(1):33-37.
[22] 唐梅,蔡松,杨东成,等.不依赖IPTG诱导产木糖醇大肠杆菌工程菌的构建[J].中国酿造,2021,40(9):173-179.
TANG M,CAI S,YANG D C,et al.Construction of IPTG-inducing independent engineered Escherichia coli for xylitol production[J].China Brewing,2021,40(9):173-179.
[23] 沈瑞华,郭军玲,周哲敏.腈水合酶的稳定性改造研究进展[J].生物加工过程,2020,18(3):269-276.
SHEN R H,GUO J L,ZHOU Z M.Recent advances in stability reconstruction of nitrile hydratase[J].Chinese Journal of Bioprocess Engineering,2020,18(3):269-276.
[24] 史悦,于慧敏,田卓玲,等.产腈水合酶重组大肠杆菌的质粒稳定性研究[J].中国生物工程杂志,2005,25(8):70-75.
SHI Y,YU H M,TIAN Z L,et al.Study on plasmid stability of a recombinant Escherichia coli producing nitrile hydratase[J].China Biotechnology,2005,25(8):70-75.
[25] 王丽燕,王煜,吴坚平,等.腈水合酶NHaseK在大肠杆菌中的功能表达[J].中国生物工程杂志,2016,36(12):42-48.
WANG L Y,WANG Y,WU J P,et al.Functional expression of a nitrile hydratase from Klebsiella oxytoca KCTC 1686 in E.coli[J].China Biotechnology,2016,36(12):42-48.
[26] 李彤,呼海娟,崔炜.慢性心力衰竭患者脑钠肽抵抗机制的研究进展[J].心血管病学进展,2023,44(3):238-242.
LI T,HU H J,CUI W.Mechanism of brain natriuretic peptide tolerance in patients with chronic heart failure[J].Advances in Cardiovascular Diseases,2023,44(3):238-242.
[27] 易俊波,买制刚,卢海蓉,等.多拷贝脑钠肽基因表达质粒的构建及其表达[J].中国生物制品学杂志,2008,21(12):1062-1065.
YI J B,MAI Z G,LU H R,et al.Construction and expression of recombinant expression vector for multi-copy B-type natriuretic peptide gene[J].Chinese Journal of Biologicals,2008,21(12):1062-1065.
[28] TALERO E,GARC A-MAURI
A-MAURI O S,
O S, VILA-ROM
VILA-ROM N J,et al.Bioactive compounds isolated from microalgae in chronic inflammation and cancer[J].Marine Drugs,2015,13(10):6152-6209.
N J,et al.Bioactive compounds isolated from microalgae in chronic inflammation and cancer[J].Marine Drugs,2015,13(10):6152-6209.
[29] WANG F F,HUANG L D,GAO B Y,et al.Optimum production conditions,purification,identification,and antioxidant activity of violaxanthin from microalga Eustigmatos Cf.polyphem (Eustigmatophyceae)[J].Marine Drugs,2018,16(6):190.
[30] DONG X R,LIU B,BAO Y H,et al.Metabolic engineering of Escherichia coli for high-level production of violaxanthin[J].Microbial Cell Factories,2023,22(1):115.
[31] LI Y,LIN P X,LU X,et al.Plasmid copy number engineering accelerates fungal polyketide discovery upon unnatural polyketide biosynthesis[J].ACS Synthetic Biology,2023,12(8):2226-2235.
[32] 郜彦彦,魏明珠,陈晨,等.紫色杆菌素的生物活性及其对草鱼的保鲜效果[J].食品研究与开发,2023,44(4):22-28.
GAO Y Y,WEI M Z,CHEN C,et al.Violacein:Bioactivity and preservation effect on grass carp[J].Food Research and Development,2023,44(4):22-28.
[33] 卢丹妮,洪沛雄,张国超,等.基于低拷贝质粒的高产紫色杆菌素谷氨酸棒杆菌的构建和发酵[J].现代食品科技,2023,39(2):170-179.
LU D N,HONG P X,ZHANG G C,et al.Construction and fermentation of efficient violacein-producing Corynebacterium glutamicum strains using low copy number plasmids[J].Modern Food Science and Technology,2023,39(2):170-179.
[34] SIMSA R,YUEN J,STOUT A,et al.Extracellular heme proteins influence bovine myosatellite cell proliferation and the color of cell-based meat[J].Foods,2019,8(10):521.
[35] SUMAN S P,JOSEPH P.Myoglobin chemistry and meat color[J].Annual Review of Food Science and Technology,2013,4:79-99.
[36] WANG M M,SHI Z,GAO N,et al.Sustainable and high-level microbial production of plant hemoglobin in Corynebacterium glutamicum[J].Biotechnology for Biofuels and Bioproducts,2023,16(1):80.
[37] MEI C A,SCHWARTZ T J,SHANKS B H,et al.Triacetic acid lactone as a potential biorenewable platform chemical[J].Green Chemistry,2012,14(7):1850-1853.
[38] YOCUM H C,BASSETT S,DA SILVA N A.Enhanced production of acetyl-CoA-based products via peroxisomal surface display in Saccharomyces cerevisiae[J].Proceedings of the National Academy of Sciences of the United States of America,2022,119(48):e2214941119.
[39] 谢培琳,张欣欣.乙型肝炎表面抗原生物学功能的研究进展[J].中国病毒病杂志,2020,10(5):333-336.
XIE P L,ZHANG X X.Research progress on hepatitis B virus surface antigen[J].Chinese Journal of Viral Diseases,2020,10(5):333-336.
[40] 何成,蔡蓓蓓,楼觉人.多拷贝重组HBsAg质粒的构建及在毕赤酵母中的高效表达[J].中国生物制品学杂志,2008,21(3):207-211.
HE C,CAI B B,LOU J R.Construction of recombinant plasmid with multi-copy expression cassette for high expression of HBsAg in Pichia pastoris[J].Chinese Journal of Biologicals,2008,21(3):207-211.
[41] LI C Y,ZOU Y S,JIANG T,et al.Harnessing plasmid replication mechanism to enable dynamic control of gene copy in bacteria[J].Metabolic Engineering,2022,70:67-78.
[42] 曾薄轩.在酿酒酵母中高产二氢青蒿酸[D].天津:天津大学,2021.
ZENG B X.High yield of dihydroartemisinin in Saccharomyces cerevisiae[D].Tianjin:Tianjin University,2021.
[43] CHEN Y,PARTOW S,SCALCINATI G,et al.Enhancing the copy number of episomal plasmids in Saccharomyces cerevisiae for improved protein production[J].FEMS Yeast Research,2012,12(5):598-607.
[44] WANG W F,XIAO H,ZHONG J J.Biosynthesis of a novel ganoderic acid by expressing CYP genes from Ganoderma lucidum in Saccharomyces cerevisiae[J].Applied Microbiology and Biotechnology,2022,106(2):523-534.
[45] JOSHI S H N,YONG C T,GYORGY A.Inducible plasmid copy number control for synthetic biology in commonly used E.coli strains[J].Nature Communications,2022,13(1):6691.
[46] 李星,郝鹤,池剑亭,等.利用异源生物生产青蒿素及其前体的研究进展[J].植物学报,2012,47(6):571-580.
LI X,HAO H,CHI J T,et al.An overview of heterologous organisms used to produce artemisinin and its precursors[J].Chinese Bulletin of Botany,2012,47(6):571-580.
[47] YANG J,TIAN Y J,LIU H Y,et al.Harnessing the endogenous 2μ plasmid of Saccharomyces cerevisiae for pathway construction[J].Frontiers in Microbiology,2021,12:679665.