改革开放以来,中国人民的生活水平快速提高,其体现在饮食上也变得丰富多彩,选择多样[1]。但是随之而来的还有肥胖症的高发,也会诱导其他相关疾病发生,如:高血压,高血脂等肥胖类疾病[2]。肥胖症不只在国内,在全世界都是一个严峻的问题,威胁着大众的身心健康[3-4]。相应的药物治疗可能带来代谢紊乱等不可控因素,而随着食疗的发展,研究者发现乳酸菌对肥胖具有很好的预防和治疗效果[5-7]。
近年的研究发现,乳酸菌对肥胖的治疗效果可能归因于胆盐水解酶的活性,胆盐水解酶可以水解胆盐,释放氨基酸和游离胆盐,存在于人体内的肠道微生物中,可以调节人体的胆汁酸代谢[8-10]。ZHU等[11]将威尔逊乳杆菌的胆盐水解酶基因(bsh)在敲除bsh基因的植物乳杆菌WCFS1中异源表达后,通过调节胆汁酸池,改善了小鼠的高胆固醇血症,LIANG等[12]筛选高胆盐水解酶活性的植物乳杆菌H-87通过调节胆汁酸代谢,抑制脂肪的积累,对小鼠高脂饮食诱导的肥胖具有良好的预防效果。
目前已知胆盐水解酶存在于多个种属中,包括乳杆菌、双歧杆菌、肠球菌等,不同来源的胆盐水解酶的酶活性和底物选择性都是显著不同的,不同的菌株在胆盐水解酶在种类和数量上也是不同的[13-14]。同样来自于唾液乳杆菌的BSH1和BSH2就具有不同的酶活性和底物特异性[15],WANG等[16]的研究发现在植物乳杆菌的四个胆盐水解酶基因中BSH1对菌株的胆盐水解酶活性起关键作用。本研究发现实验室保藏的Ligilactobacillus sp.BD7642具有很强的胆盐水解酶活性,尚不明晰该菌株的胆盐水解酶的酶活性特症。本研究的目的是对Ligilactobacillus sp.BD7642胆盐水解酶活性进行特异性分析,研究其对6种胆盐的特异性降解的情况,并对参与其中的胆盐水解酶蛋白进行分析,最终获得高胆盐水解酶蛋白的信息,为Ligilactobacillus sp.BD7642菌株开发提供借鉴,也为未来益生菌的改造提供新的思路和素材。
1 材料与方法
1.1 实验材料
本实验使用的菌株和质粒见表1。PrimeSTAR®HS DNA高保真聚合酶、限制性核酸内切酸(BamH I、EcoR I)、In-Fusion Snap Assembly Master Mix试剂盒、细菌基因组提取试剂盒,大连宝生物有限公司;氨苄青霉素(ampicillin,Amp)、异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactoside,IPTG)、质粒提取试剂盒、DNA片段回收和胶回收试剂盒,上海生工生物工程股份有限公司;SurePAGETM、Bis-Tris,8%、12 wells蛋白预制胶,金斯瑞生物科技有限公司,6种胆盐牛磺胆酸钠盐(taurocholic acid sodium salt,TAC)、甘氨胆酸钠盐(glycocholate sodium salt,GC)、牛磺脱氧胆酸钠盐(sodium taurodeoxycholate,TDC)、甘氨脱氧胆酸钠盐(sodium glycodeoxycholate,GDC)、甘氨鹅脱氧胆酸钠盐(glycochenodeoxycholic acid sodium salt,GCDC)、牛磺鹅脱氧胆酸钠盐(taurochenodeoxycholic acid sodium salt,TCDC),均为色谱纯,美国sigma公司;乙腈、三氟乙酸,上海阿拉丁生化科技股份有限公司。BUGBOX厌氧培养箱,BAKER RUSKINN;HZQ-X500C恒温摇床,上海一恒科学仪器有限公司;超声波细胞粉碎机,宁波新芝生物科技股份有限公司;电泳仪,Bio-Rid;DS-11超微量分光光度计,美国DeNovix;安捷伦1260高效液相色谱仪,安捷伦科技有限公司;T3色谱柱(2.5 μm,2.1 mm×150 mm),沃特世科技(上海)有限公司。
表1 本研究使用的菌株和质粒
Table 1 Strains and plasmids used in this study

菌株(质粒)相关特性来源Ligilactobacillus sp.BD7642基因供体菌株实验室分离保藏E.coli DH5α基因克隆宿主上海生工 E.coli BL21(DE3)基因表达宿主实验室保藏 pET302/NT-HisPT7, TT7, Amp实验室保藏 pET302-g1210pET302-PT7- g1210-TT7本研究 pET302-g1294pET302-PT7- g1294-TT7本研究 pET302-g1443pET302-PT7- g1443-TT7本研究 E.coli BL21(DE3)-g1210E.coli BL21(DE3)包含pET302-g1210本研究 E.coli BL21(DE3)-g1294E.coli BL21(DE3)包含pET302-g1294本研究 E.coli BL21(DE3)-g1443E.coli BL21(DE3)包含pET302-g1443本研究
1.2 引物
本研究使用的引物见表2。
表2 本研究使用的引物
Table 2 Primers used in this study

引物名称引物序列(5′-3′)1210-FCATCATCATCATCACGAATTCATGTGTACAGGTATTA-AGTTTAATGATTCTAATG1210-RGAATATCATCGATCTCGAGCTTAATCAACAGTTATA-ATTTCTGTCCCTTTTG1294-FCATCATCATCATCACGAATTCATGTGTACAGGTT-TACGTTTTAATGAC1294-RGAATATCATCGATCTCGAGCCTAGTATAAAGTTACT-TCTTTAGCGTTCATG1443-FCATCATCATCATCACGAATTCATGAATCAAAAAGAAG-TAAAAGAAATTGC1443-RGAATATCATCGATCTCGAGCTTAATTAACTAATT-TATATTCAGGTGCACG
注:F为游引物;R为下游引物;下划线部分为同源臂序列。
1.3 培养基与培养方法
MRS培养基:德国默克公司;TPY(trypticase phytone yeast)培养基:青岛海博生物公司;胆盐水解酶活筛选平板:TPY+0.3%(质量分数)牛磺脱氧胆酸钠;LB培养基(g/L):酵母提取物5,蛋白胨10,氯化钠10;发酵培养基:LB培养基+Amp (100 μg/mL)。
培养方法:挑取单菌落,接种于10 mL发酵培养基37 ℃,180 r/min培养24 h进行活化;以2%的接种量再转接到10 mL发酵培养基培养24 h作为种子液;将得到的种子液转接到装有50 mL发酵培养基的250 mL锥形瓶中,控制起始生物量OD600值为0.1。待菌株生长OD600值到0.8~1.0时,添加终浓度的500 μmol/L的IPTG在20 ℃诱导,12~16 h后结束诱导收集菌体。菌体置于冰水上经超声波破碎仪处理(功率200 W,工作3 s,间隔2 s,共超声15 min),12 000 r/min离心10 min收集上清液为菌体内容物。
1.4 表达质粒的构建
按照细菌基因组提取试剂盒说明书说明,提取Ligilactobacillus sp.BD7642基因组,基因g1210、g1294、g1443通过Ligilactobacillus sp.BD7642基因组PCR获得,引物见表2。以pET302/NT-His为载体,经BamH I单酶切后,利用同源重组酶进行连接,构建成pET302-g1210、pET302-g1294、pET302-g1443。提取转化子质粒并测序验证。
1.5 蛋白电泳
按照SurePAGETM,Bis-Tris,8%、12 wells蛋白预制胶使用说明书说明将菌体内容物进行SDS-PAGE凝胶电泳。
1.6 测定方法
胆盐的测定通过高效液相色谱测定:6种胆盐标准品混配100 mmol/L混样,跑液相绘制标准曲线,将混配胆盐标准品与菌体内容物1∶1混合,恒温37 ℃反应3 h,对最终的剩余胆盐进行定量;液相色谱程序如下:流动相为A:乙腈+0.09%三氟乙酸与B:水+0.1%三氟乙酸,梯度洗脱,0~2 min:V(A)∶V(B)=25∶75,2~10 min:V(A)∶V(B)=55∶45,10~15 min:V(A)∶V(B)=25∶75。流动相的流速为0.6 mL/min,检测温度为40 ℃,检测波长为205 nm。
2 结果与分析
2.1 Ligilactobacillus sp.BD7642高胆盐水解酶活性的发现
Ligilactobacillus sp.BD7642菌株是光明乳业研究院菌种库实验室保藏菌株,为了获得高胆盐水解酶活性的菌株,对菌种库中的菌株进行筛选。含有牛磺酸脱氧胆酸钠的平板通常作为一个高效筛选胆盐水解酶活性的手段[17],本研究发现该菌株在胆盐水解酶活筛选平板上可以看到非常明显的水解圈,见图1,以该菌株作为研究对象,对其胆盐水解酶的特异性进行分析。
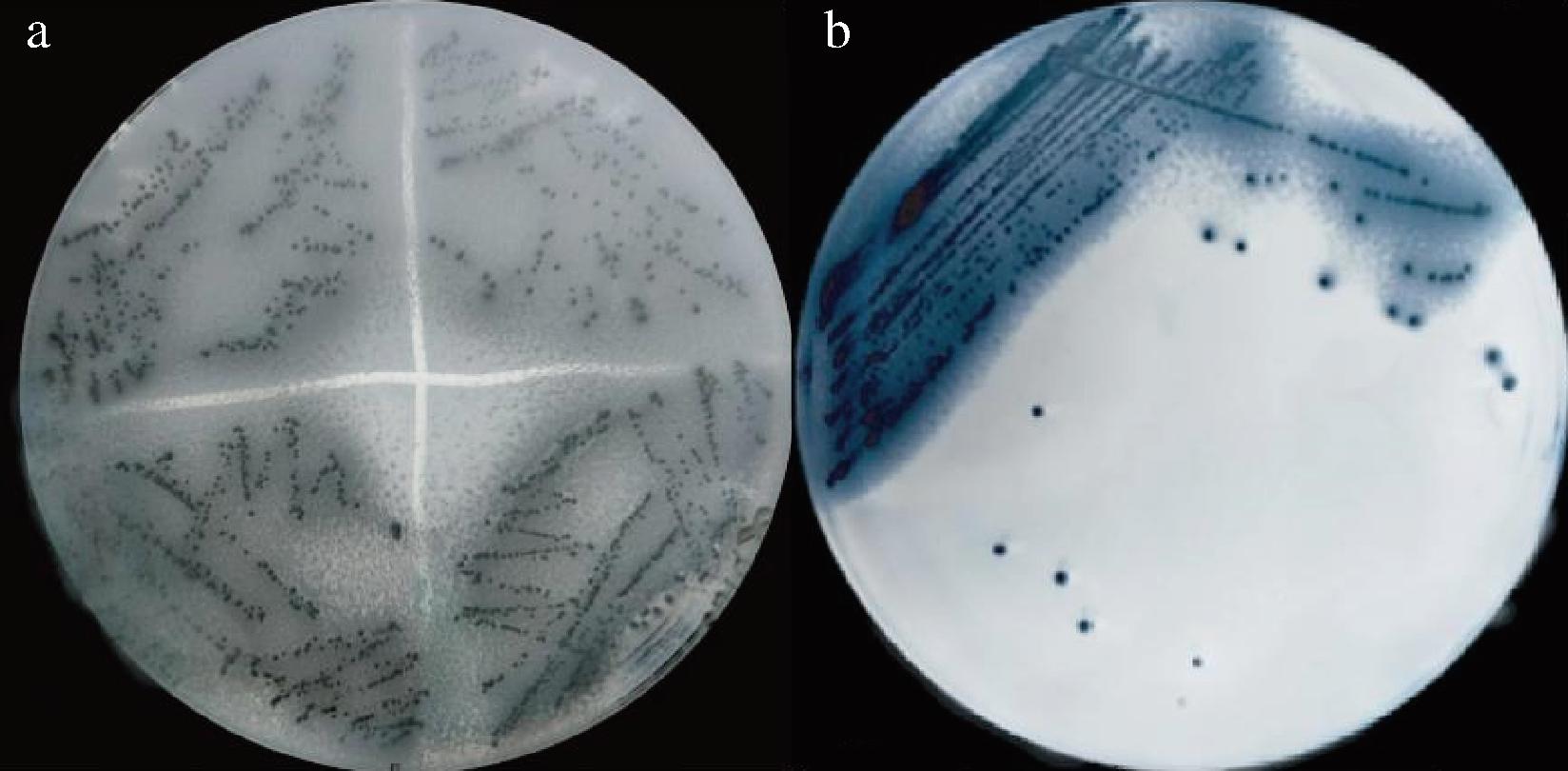
a-胆盐水解酶活筛选平板;b-复筛验证平板
图1 Ligilactobacillus sp.BD7642在平板上生长图
Fig.1 Growth diagram of Ligilactobacillus sp.BD7642 on the medium
2.2 高效液相色谱分析胆盐水解酶活性特异性
HPLC已经广泛应用于化合物的分离鉴定中,具有高效、特异性好等的优点,CORZO等[18]成功利用高效液相色谱法测定了嗜酸乳杆菌的胆盐水解酶活性,夏永军等[14]使用HPLC方法对植物乳杆菌的胆盐水解酶的酶活性特性进行分析,HPLC可以同时检测6种胆盐的降解情况,本研究利用收集到的Ligilactobacillus sp.BD7642菌体以及Ligilactobacillus sp.BD7642的胞内酶与6种胆盐的混合样品进行反应,并利用HPLC检测反应结果,获得的峰图信息见图2。该菌株的胆盐水解酶对牛磺酸胆盐具有极强的降解能力,对3种牛磺酸胆盐的降解率见表3,该菌株几乎可以将牛磺酸胆盐完全降解,具有极强的偏好性。已知胆盐水解酶对甘氨酸胆盐或牛磺酸胆盐的偏好性是普遍存在的[19],来自于植物乳杆菌的胆盐水解酶对甘氨酸胆盐具有偏好性;在ZHU等[11]的研究中也揭示了胆盐水解酶特异性的在胆汁酸调节中重要性,FOLEY等[20]解释胆盐水解酶的特异性对菌株的适应性和定殖具有重要的影响,并且会改变宿主的菌群结构,未来在胆盐水解酶特异性的逐步认知中,其对干预肥胖的影响也将会进一步明晰,对工程设计相关胆盐水解酶特异性来改善人类健康方面具有重要意义。
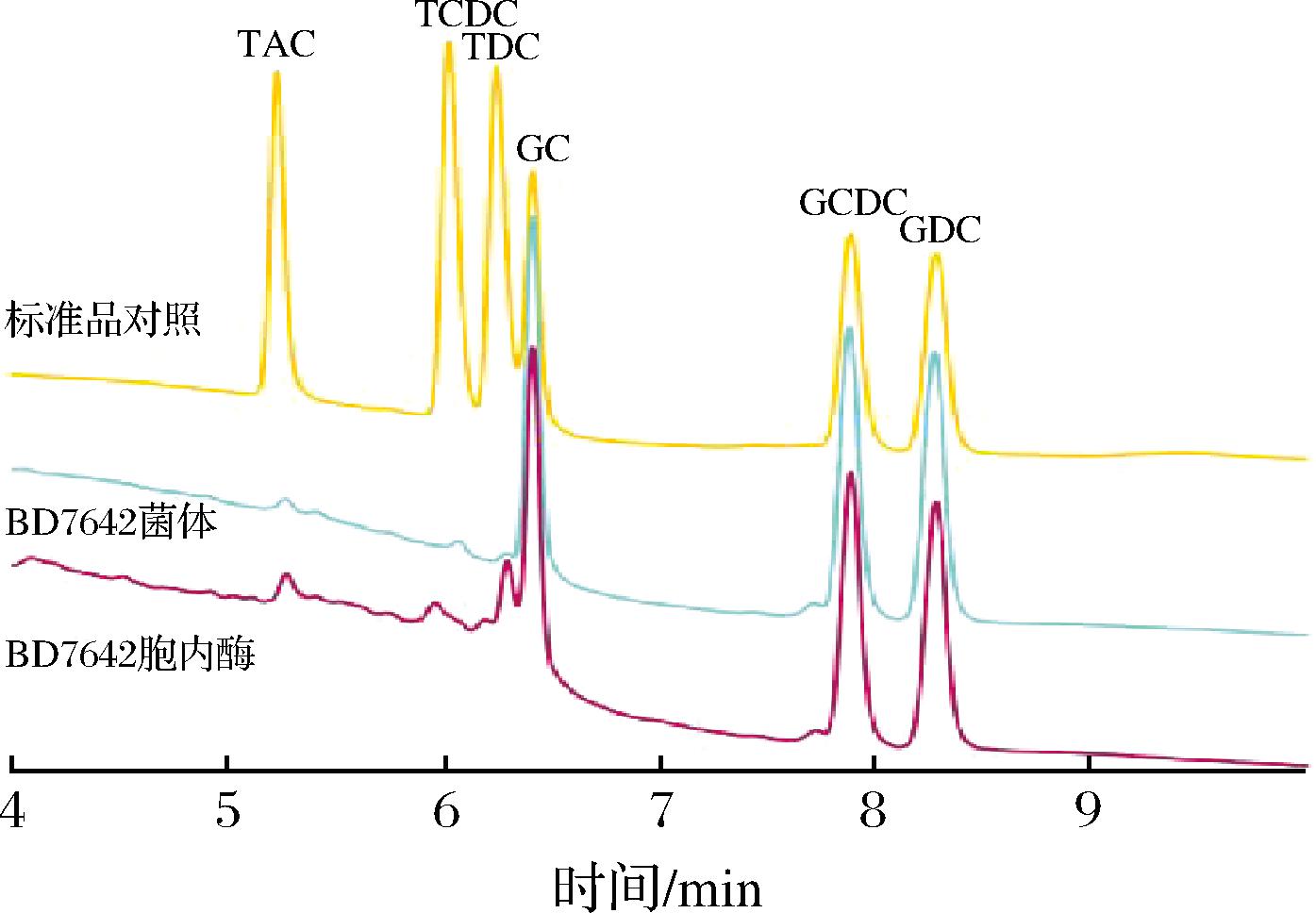
图2 HPLC峰图数据
Fig.2 Peak map data of HPLC
表3 Ligilactobacillus sp.BD7642对牛磺酸3种胆盐的降解率
Table 3 Degradation rate of taurine bile salts by Ligilactobacillus sp.BD7642
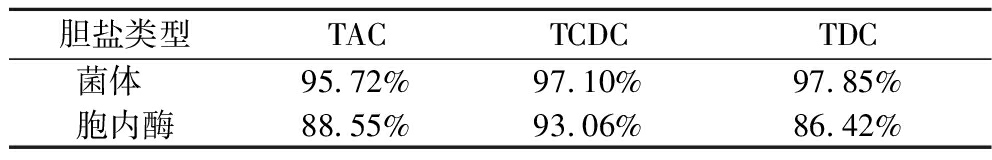
胆盐类型TACTCDCTDC菌体 95.72%97.10%97.85%胞内酶88.55%93.06%86.42%
2.3 胆盐水解酶蛋白的异源表达
基于以上对该菌株胆盐水解酶活性的分析,接下来对该菌株高胆盐水解酶活性的蛋白进行研究。已知为了适应环境不同菌株的胆盐水解酶基因通常会含有多个,如嗜酸乳杆菌NCFM具有2个不同胆盐降解偏好性的胆盐水解酶基因(bshA,bshB)[21],而植物乳杆菌含有4个胆盐水解酶基因(bsh1-4)[22],对Ligilactobacillus sp.BD7642基因组信息进行分析,确定3个疑似胆盐水解酶基因分别为g1210、g1294、g1443,将这3个基因分别构建表达质粒,并转化进E.coli BL21(DE3)中进行异源表达,将异源表达的结果进行SDS-PAGE电泳,结果见图3-a,验证蛋白表达成功,蛋白大小在35 kDa左右,将收获的菌体在胆盐水解酶活筛选平板上进行点板,生长48 h后,结果见图3-b,g1210、g1294蛋白在菌体下方可以看到明显的水解圈,但是g1443蛋白没有观察到。将包括这3个蛋白的的氨基酸序列进行比较,见图4-a,发现g1210、g1294序列相似度更高,均具有更强的牛磺脱氧胆酸钠的水解能力,将不同来源的胆盐水解酶构建进化树分析发现,g1210、g1294、g1443分在了不同的分支上,g1294与鸟乳杆菌(Ligilactobacillus aviarius)来源的胆盐水解酶关系更近,见图4-b。接着利用HPLC的方法对这3个异源表达的蛋白同样进行6种胆盐的特异性降解分析,结果见图5,发现g1294蛋白对3种牛磺酸胆盐的降解能力都很强,g1210对牛磺脱氧胆酸降解能力较弱,对另外2个牛磺酸胆盐降解能力较强,而g1443对牛磺鹅脱氧胆酸的降解能力强,但是对另外2个牛磺酸的降解能力较弱。在植物乳杆菌中发挥主要胆盐水解酶活性的酶为BSH1[22],因此推测在Ligilactobacillus sp.BD7642中g1294蛋白是胆盐水解酶活性的关键蛋白。我们进一步将这3个蛋白与牛磺酸进行分子对接[23-24],结果见图6,预测g1294与牛磺酸的结合更加紧密,可能造成牛磺酸胆盐的水解位点更加暴露,有利于其进行水解反应,我们还将这个酶的活性位点在氨基酸序列上进行标注,见图4-a,这3个酶的活性位点是相似的,未来研究将对这个酶的活性位点进行突变,找到关键位点,并通过定点突变的方法理性改变胆盐水解酶的酶活性与特异性。
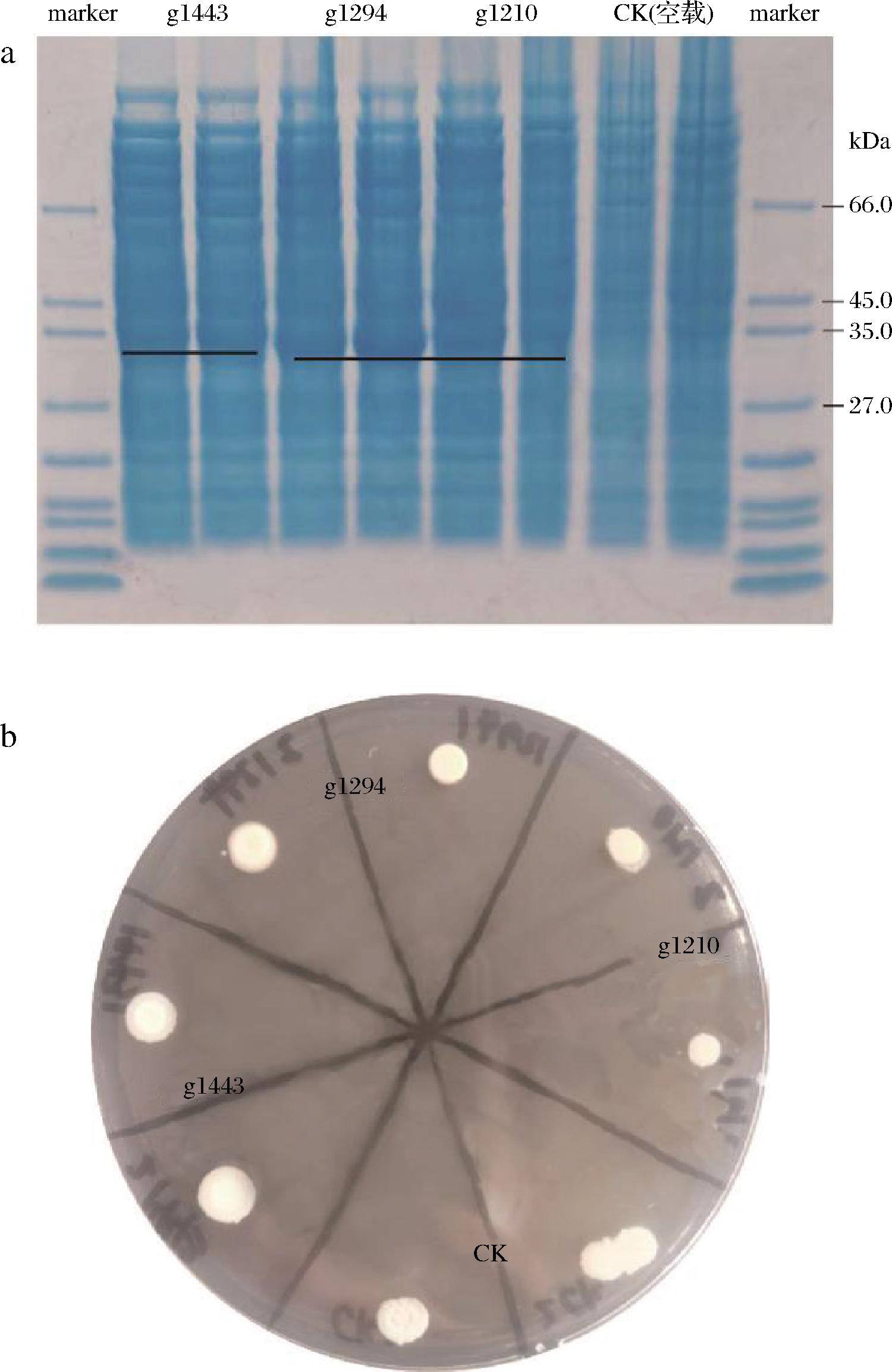
a-SDS-PAGE凝胶电泳图;b-在筛选平板上的点板图
图3 g1210、g1294、g1443异源表达成功验证图
Fig.3 Heterologous expression verification map of g1210,g1294,and g1443

a-g1210、g1294和g1443胆盐水解酶氨基酸序列对比图;b-g1210、g1294和g1443系统发育分析
图4 g1210、g1294和g1443胆盐水解酶氨基酸序列对比图和g1210、g1294和g1443系统发育分析
Fig.4 g1210,g1294,and g1443 comparative diagram of amino acid sequences and phylogenetic analysis of g1210,g1294,and g1443

图5 g1210、g1294、g1443对胆盐降解对比图
Fig.5 Bile salt degradation comparison map of g1210,g1294,and g1443
注:短线为空载对照。
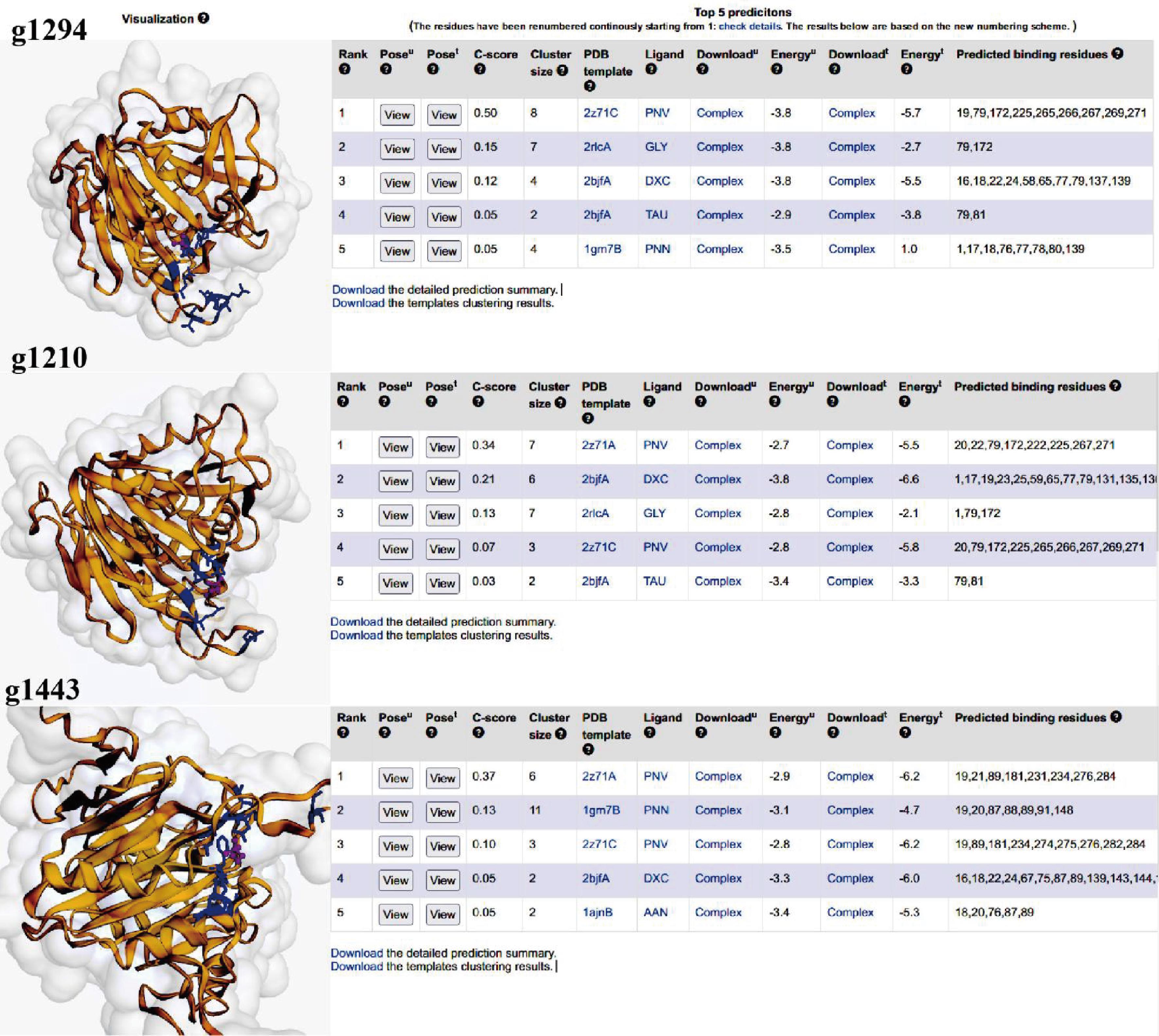
图6 分子对接模型与活性位点预测结果
Fig.6 Molecular docking model and prediction results of active sites
注:紫色为牛磺酸分子。
3 结论
胆盐水解酶可能是一个潜力巨大的针对肥胖症、高胆固醇血症等疾病的治疗方案,本研究从Ligilactobacillus sp.BD7642中发现的高胆盐水解酶活性的蛋白g1294,在该菌的胆盐水解酶活性中发挥着至关重要的作用。该酶对牛磺酸胆盐具有明显的偏好性,且通过分子对接的手段发现其与牛磺酸的连接紧密,这对其偏好性做出了一定的解释,本研究确定g1294蛋白在Ligilactobacillus sp.BD7642菌中胆盐水解酶的作用,并分析这个酶的活性中心位点,未来将对这个酶的活性位点进行研究,确定关键位点,也可以为合成生物学理性改变胆盐水解酶活性提供参考和优秀的供体。
[1] 范晓婧.改革开放后中国民众的饮食生活[J].湘潮(下半月),2016(2):78-82.
FAN X J.The dietetic life of Chinese people after the reform and opening up[J].Xiang Chao,2016(2):78-82.
[2] SAKLAYEN M G.The global epidemic of the metabolic syndrome[J].Current Hypertension Reports,2018,20(2):12.
[3] ALLISON D B,FONTAINE K R,MANSON J E,et al.Annual deaths attributable to obesity in the United States[J].JAMA-Journal of the American Medical Association,1999,282(16):1530-1538.
[4] RENDELL M S.Obesity and diabetes:The final frontier[J].Expert Review of Endocrinology &Metabolism,2023,18(1):81-94.
[5] MOLINA-TIJERAS J A,DIEZ-ECHAVE P,VEZZA T,et al.Lactobacillus fermentum CECT5716 ameliorates high fat diet-induced obesity in mice through modulation of gut microbiota dysbiosis[J].Pharmacological Research,2021,167:105471.
[6] SUN Y N,CHEN S B,REN F Z,et al.Lactobacillus paracasei N1115 attenuates obesity in high-fat diet-induced obese mice[J].Food Science &Nutrition,2022,11(1):418-427.
[7] MAKWANA S,PRAJAPATI J B,PIPALIYA R,et al.Effects of probiotic fermented milk on management of obesity studied in high-fat-diet induced obese rat model[J].Food Production,Processing and Nutrition,2023,5(1):3.
[8] BOURGIN M,KRIAA A,MKAOUAR H,et al.Bile salt hydrolases:At the crossroads of microbiota and human health[J].Microorganisms,2021,9(6):1122.
[9] POLAND J C,FLYNN C R.Bile acids,their receptors,and the gut microbiota[J].Physiology,2021,36(4):235-245.
[10] RIDLON J M,KANG D J,HYLEMON P B,et al.Bile acids and the gut microbiome[J].Current Opinion in Gastroenterology,2014,30(3):332-338.
[11] ZHU H J,ZHAO F,ZHANG W J,et al.Cholesterol-lowering effect of bile salt hydrolase from a Lactobacillus johnsonii strain mediated by FXR pathway regulation[J].Food &Function,2022,13(2):725-736.
[12] LIANG C,ZHOU X H,GONG P M,et al.Lactiplantibacillus plantarum H-87 prevents high-fat diet-induced obesity by regulating bile acid metabolism in C57BL/6 J mice[J].Food &Function,2021,12(10):4315-4324.
[13] SONG Z W,CAI Y Y,LAO X Z,et al.Taxonomic profiling and populational patterns of bacterial bile salt hydrolase (BSH) genes based on worldwide human gut microbiome[J].Microbiome,2019,7(1):9.
[14] 夏永军,王光强,王巧惠,等.HPLC分析植物乳杆菌胆盐水解酶降解特性[J].工业微生物,2015,45(6):16-20.
XIA Y J,WANG G Q,WANG Q H,et al.Degradation characteristics of BSH-1 from Lactobacillus plantarum by HPLC[J].Industrial Microbiology,2015,45(6):16-20.
[15] BI J,FANG F,LU S Y,et al.New insight into the catalytic properties of bile salt hydrolase[J].Journal of Molecular Catalysis B:Enzymatic,2013,96:46-51.
[16] WANG G Q,YU H I,FENG X,et al.Specific bile salt hydrolase genes in Lactobacillus plantarum AR113 and relationship with bile salt resistance[J].LWT,2021,145:111208.
[17] ![]() J,KOS B,
J,KOS B,![]() S,et al.The effect of bile salts on survival and morphology of a potential probiotic strain Lactobacillus acidophilus M92[J].World Journal of Microbiology and Biotechnology,2000,16(7):673-678.
S,et al.The effect of bile salts on survival and morphology of a potential probiotic strain Lactobacillus acidophilus M92[J].World Journal of Microbiology and Biotechnology,2000,16(7):673-678.
[18] CORZO G,GILLILAND S E.Measurement of bile salt hydrolase activity from Lactobacillus acidophilus based on disappearance of conjugated bile Salts1[J].Journal of Dairy Science,1999,82(3):466-471.
[19] BEGLEY M,HILL C,GAHAN C G M.Bile salt hydrolase activity in probiotics[J].Applied and Environmental Microbiology,2006,72(3):1729-1738.
[20] FOLEY M H,O’FLAHERTY S,ALLEN G,et al.Lactobacillus bile salt hydrolase substrate specificity governs bacterial fitness and host colonization[J].Proceedings of the National Academy of Sciences of the United States of America,2021,118(6):e2017709118.
[21] MCAULIFFE O,CANO R J,KLAENHAMMER T R.Genetic analysis of two bile salt hydrolase activities in Lactobacillus acidophilus NCFM[J].Applied and Environmental Microbiology,2005,71(8):4925-4929.
[22] GU X C,LUO X G,WANG C X,et al.Cloning and analysis of bile salt hydrolase genes from Lactobacillus plantarum CGMCC No.8198[J].Biotechnology Letters,2014,36(5):975-983.
[23] YANG J Y,ROY A,ZHANG Y.Protein-ligand binding site recognition using complementary binding-specific substructure comparison and sequence profile alignment[J].Bioinformatics,2013,29(20):2588-2595.
[24] WU Q,PENG Z L,ZHANG Y,et al.COACH-D:Improved protein-ligand binding sites prediction with refined ligand-binding poses through molecular docking[J].Nucleic Acids Research,2018,46(W1):W438-W442.