胰高血糖素样肽1(glucagon-like peptide 1,GLP-1)是一种肠促胰岛素激素,可通过调控胰岛素和胰高血糖素分泌延缓胃排空、降低食欲,发挥调节血糖的作用[1]。GLP-1及其受体已被开发为新型降糖药物(例如利拉鲁肽、艾塞那肽等),在治疗2型糖尿病方面取得良好效果[2-3]。GLP-1主要由远端回肠和结肠中的肠内分泌细胞分泌。肠内分泌细胞属于分化型肠上皮细胞的一种,是肠道中的营养感受器,其细胞表面存在糖、蛋白质和脂肪等各种营养成分的感应受体,当接收到了肠腔中营养物质刺激后,能够释放相应的肠内分泌激素[1, 3-4]。而肠道中,特别是回肠远端和结肠中还存在着大量的肠道微生物。近年来,越来越多的研究指出,肠道菌群在包括糖尿病在内的多种慢性疾病的发展过程中发挥重要作用[5-6],且有研究指出肠道菌群是调节GLP-1等肠促激素产生的重要因素[7-9]。因此,靶向肠道菌群调节肠促激素代谢已成为糖尿病等慢性代谢性疾病防治的研究热点之一。
乳酸菌是肠道微生物的关键组成部分[4, 10]。目前,已有部分学者开展了乳酸菌对GLP-1分泌的影响研究。如SIMON等[11]采用随机临床对照试验研究罗伊氏乳杆菌(Lactobacillus reuteri)对葡萄糖耐受人群GLP-1分泌的影响,发现与安慰剂相比,每天服用L.reuteri会显著增加GLP-1的释放。类似地,WANG等[12]研究表明干酪乳酸菌(L.casei CCFM419)干预会增加糖尿病小鼠GLP-1水平。上述研究均显示LAB干预会增加GLP-1水平,但由于人体和动物试验存在“黑箱效应”,无法准确定位作用途径。因此,部分学者开展了细菌-细胞体外共培养试验以期揭示调控机理。如PANWAR等[4]通过将多种乳杆菌(Lactobacillus)与肠内分泌细胞共培养,证实了乳酸菌可增加肠道内分泌细胞GLP-1分泌。WEI等[9]采用LAB与小鼠肠内分泌细胞STC-1共培养模型筛选出可增强GLP-1分泌的两株乳杆菌L.kefiranofaciens M和L.kefiri K。但细菌直接接触细胞的培养体系无法有效模拟体内环境,因为体内环境中肠内分泌细胞与细菌间存在着肠道黏液等隔离层;此外,高浓度的细菌对细胞存在毒性作用,因此研究结果可能无法直接类推到体内[13]。对此,一些研究人员构建了复杂的体外模型来研究细胞-微生物互作机理,但此类模型对实验室基础设施和人员操作技术要求较高[14]。因此,为规避上述局限性,本研究将采用一种简单易操作、接近体内环境的体外共培养模型分析乳杆菌对肠道内分泌细胞GLP-1分泌的影响,以期更真实的揭示乳杆菌调控糖尿病代谢的作用途径。
考虑到肠内分泌细胞的需氧性、乳杆菌的兼性厌氧性、肠道环境的低氧性,本研究将采用体细胞(有氧)-黏液层-细菌(微需氧)共培养体系分析乳杆菌对细胞GLP-1分泌的影响。选取的pGIP/Neo STC-1细胞系可产生高浓度的肽激素,是研究GLP-1分泌的良好肠内分泌细胞模型[4]。同时考虑到前期直接的共培养,发现鼠李糖乳杆菌(L.rhamnosus)、植物乳杆菌(L.plantarum)和约翰氏乳杆菌(L.johnsonii)可促进肠内分泌细胞GLP-1分泌,此外研究表明上述3种乳杆菌具有调节糖尿病等慢性代谢性疾病和改善肠道稳态的功能[4, 15-18]。因此,研究将选用上述3种菌株,设立不同浓度的乳杆菌菌株共培养体系以分析菌株的剂量效应;设立黏液层缺失组以观察黏液层的缺失对体系的影响;设立共培养管螺旋盖轻旋组和紧闭组以考察不同O2暴露情况对体系的影响。采用酶联免疫吸附试验(enzyme-linked immunosorbent assay,ELISA)分析体系中GLP-1水平的变化,共培养前后体系pH值、细胞活力以及细菌在细胞周围的分布情况。同时,验证了共培养后体系pH值改变对ELISA结果的影响,并结合胞内GLP-1荧光染色确保pH值校正后ELISA结果的可靠性。本研究将为靶向肠道菌群调节宿主GLP-1代谢提供理论依据。
1 材料与方法
1.1 材料与试剂
鼠李糖乳杆菌(L.rhamnosus NCIMB6375)、约翰氏乳杆菌(L.johnsonii NCIMB8795),英国阿伯丁国家工业、食品和海洋细菌收藏中心。植物乳杆菌(L.plantarum KC491380)从健康婴儿粪便中分离得到。肠内分泌细胞系pGIP/Neo STC-1由英国贝尔法斯特女王大学B.Green教授提供,该细胞系是异质多能鼠STC-1细胞的亚克隆,可对多种刺激做出反应分泌GLP-1。
乳杆菌MRS培养基(7406A)、琼脂(7178A),英国Neogen Euope公司;含有4.5 g/L L-谷氨酰胺而不含丙酮酸钠的DMEM培养基,美国生命技术公司;胎牛血清(fetal bovine serum,FBS)、青霉素、链霉素、遗传霉素-G418、Ⅱ型猪黏蛋白、HEPES缓冲盐(NaCl、KCl、CaCl2和MgCl2)、葡萄糖、HEPES、福尔马林、甘氨酸、山羊血清、牛血清白蛋白(bull serum albumin,BSA),德国Sigma公司;GLP-1 ELISA试剂盒(EGLP-35K),德国Merck Millipore公司;2 g/L Trypan blue溶液、Hoechst 33342(20 mmol/L)、透化溶液、吐温20,赛默飞世尔科技公司;GLP-1-FITC抗体,百欧泰生物科技有限公司。其余均为实验室常用试剂,市售分析纯。
1.2 仪器与设备
Hera cells 150 CO2培养箱、Invitrogen细胞计数仪、Savant SPD2010 SpeedVac浓缩仪、Cell Insight NXT High Content筛选平台,赛默飞世尔科技;ROTINA 380 台式离心机,德国Andreas Hettich有限责任公司;FE20/EL20型pH计,瑞士梅特勒-托利多有限公司;AxioCam显微镜,德国ZEISS公司;其余为实验室常用设备。
1.3 试验方法
1.3.1 细菌菌株预培养
将3株乳杆菌菌株接种至MRS液体培养基中,37 ℃过夜(16~18 h)培养进行复苏活化。而后涂布于MRS琼脂平板上,过夜培养后用Parafilm封口膜封口,4 ℃保存备用(限1周内使用)。共培养前一天,挑取单个菌落接种到10 mL MRS培养基中过夜培养。然后使用MRS液体培养基将细菌培养物稀释至OD600=1.5(相当于细菌浓度为1×109 CFU/mL[4])。将稀释的细菌培养物4 ℃保存,将在同一天用于与pGIP/Neo STC-1细胞共培养。
1.3.2 肠内分泌细胞预培养
将pGIP/Neo STC-1细胞从液氮中复苏至补充有4.5 g/L L-谷氨酰胺(不含丙酮酸钠)、100 g/L FBS、100 U/mL青霉素、100 mg/L链霉素和400 g/mL遗传霉素-G418的DMEM培养基中,而后置于37 ℃、5% CO2(体积分数,下同)的培养箱中培养,每周传代两次。将采用第20~50代细胞用于共培养。在共培养前一天,将细胞以2×106个/孔的浓度接种于含有盖玻片(圆形,直径19 mm)的12孔组织培养板中,并在37 ℃孵育过夜(18~24 h),以使细胞黏附于盖玻片上。共培养前1 h,吸弃原含抗生素DMEM培养基,然后向每孔加入不含抗生素的新鲜1 mL预热DMEM培养基。
1.3.3 共培养体系建立
将含有10 g/L琼脂的1 000 mL MRS培养基高压灭菌并冷却至约40 ℃,而后加入1 mL 1.3.1节中已稀释的乳杆菌培养物,摇晃均匀。将该接种物以40 mL/管转移到50 mL无菌管中,静置30 min使其凝固。而后向顶部加入1 mL无菌黏液(50 g/L黏蛋白和8 g/L琼脂,pH 6.8),静置10 min使其凝固。再将1.3.2节中制备的带有pGIP/Neo STC-1细胞的盖玻片倒置放在黏液层顶部(含有细胞面朝向黏液层),然后加入不含抗生素的预热DMEM培养基至总体积为50 mL,盖上试管螺旋盖。最后,将共培养体系置于37 ℃、5% CO2的培养箱中共培养18 h。共培养体系的制备流程见电子版增强出版附图1(https://doi.org/10.13995/j.cnki.11-1802/ts.035891)。
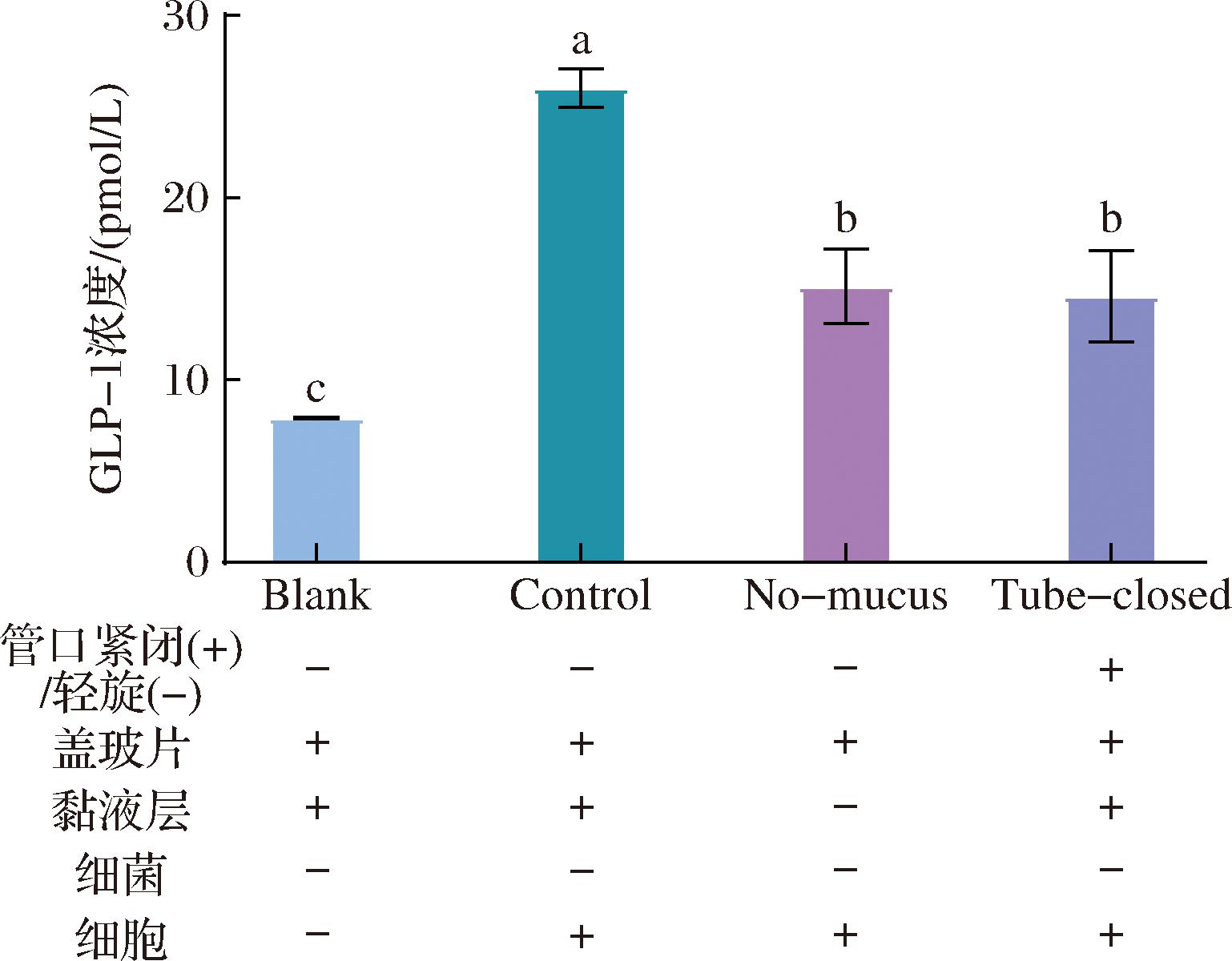
图1 氧气暴露和黏液层缺失对GLP-1分泌的影响
Fig.1 Effect of oxygen exposure and missing mucus layer on the GLP-1 secretion of cells
注:“+”为含有,“-”为不含有。柱状图中不同字母表示存在显著性差异(P<0.05)(下同)。
为研究不同乳杆菌数量对细胞的影响,根据预实验结果,将L.rhamnosus和L.johnsonii的浓度梯度设置为1×109、1×108、1×107 CFU/1 000 mL,将L.plantarum浓度梯度设置为1×109、1×108、1×107、1×106、1×105CFU/1 000 mL。同时,为分析不同氧浓度对共培养体系的影响,分别设置试管螺旋盖轻旋和紧闭体系以测试O2暴露情况对体系的影响。另外还设有无黏液层的体系,以研究黏液存在与否对体系的影响。设置不含乳杆菌以及同时不含乳杆菌和细胞的体系作为对照。每种条件设置3个重复。
1.3.4 顶部液体培养基收集和pH检测
共培养结束后,小心收集体系顶部的液体培养基(约10 mL)至15 mL离心管中(置于冰上),而后4 ℃、5 000×g离心5 min以去除细胞和细菌。收集上清液并测量其pH值,然后将上清液于-80 ℃下储存以备GLP-1含量测定。
1.3.5 GLP-1分泌量检测
采用ELISA试剂盒测定顶部液体培养基中的GLP-1含量。考虑到试剂盒的检测限,将上清液用浓缩器真空浓缩24 h,然后溶解在HEPES缓冲液(pH 7.4)中。本课题组之前的研究经验提示ELISA测试结果可能会受溶液pH值的影响,为了验证这一猜想,测定了浓缩溶液调节pH前后(使用1 mol/L HCl溶液或NaOH溶液调节到7.4)GLP-1浓度的不同。基于此验证试验结果,后续在GLP-1测定前统一将溶解溶液pH值调节至7.4。
1.3.6 细胞活力测试
去除体系顶部液体培养基后,使用细胞刮刀小心地将黏附在盖玻片上的细胞刮下并移至1 mL预热的DMEM培养基中。用2 g/L Trypan blue溶液染色后,使用细胞计数器检查细胞活力。
1.3.7 革兰氏染色和细胞内GLP-1成像
为检查盖玻片上细菌的分布情况,在去除顶部液体培养基后,对盖玻片(带细胞)上的乳杆菌进行革兰氏染色,100倍油镜下观察拍照。
使用Cell Insight NXT High Content筛选平台对细胞内GLP-1进行可视化成像。共培养后,将黏附在盖玻片上的细胞小心地移至12孔组织培养板中。向每孔中加入1 mL 100 g/L福尔马林以固定细胞,之后用1×PBS洗涤细胞4次。加入含有1 g/L Trition X-100的PBS溶液,室温下透化30 min,用1×PBS洗涤细胞4次。在室温下加入封闭溶液(PBS含0.1 g/L吐温20、0.3 mol/L甘氨酸、100 g/L山羊血清和10 g/L BSA)1 h。然后除去封闭溶液,用含有0.1 g/L吐温和10 g/L BSA的PBS稀释GLP-1-FITC抗体至1∶200(U∶mL),并在4 ℃暗处孵育过夜以染色GLP-1。除去上清液,用1×PBS洗涤细胞3次,每次5 min。加入Hoechst溶液,并在室温下在暗处孵育10 min以染色细胞核。用1×PBS洗涤两次,而后重新加入1×PBS,采集细胞在350~461 nm(Hoechst)和485~520 nm(GLP-1-FITC抗体)处的显微图像和荧光强度。
1.4 数据处理
数据采用均值±标准偏差表示,采用SPSS 20.0统计软件组间one-way ANOVA检测,检验的显著性水平设定为P<0.05。
2 结果与分析
2.1 pH值对GLP-1测试结果的影响
由于乳杆菌发酵产酸会改变体系pH,因此首先研究了pH对GLP-1浓度ELISA测定结果的影响,结果如电子版增强出版附图2所示。共测试6组样本(Groups 1~6),每组3份平行样品,其中一份样品的pH值未调节,另外两份调节至pH为7.4(同ELISA试剂盒缓冲液pH值)。共培养后,未调节样品的pH值高于7.4(第1、2、5组)或低于7.4(第3、4、6组)。来自同一组的3个样品具有相同的培养条件。然而,当pH值不同时,与其他两个样品(调节pH)相比,pH值未调节的样品的GLP-1浓度结果呈现不规则的明显变化。而同一组中pH值相同的两个样品则拥有相似的GLP-1结果。这些结果表明溶液pH值会影响GLP-1的ELISA检测结果。
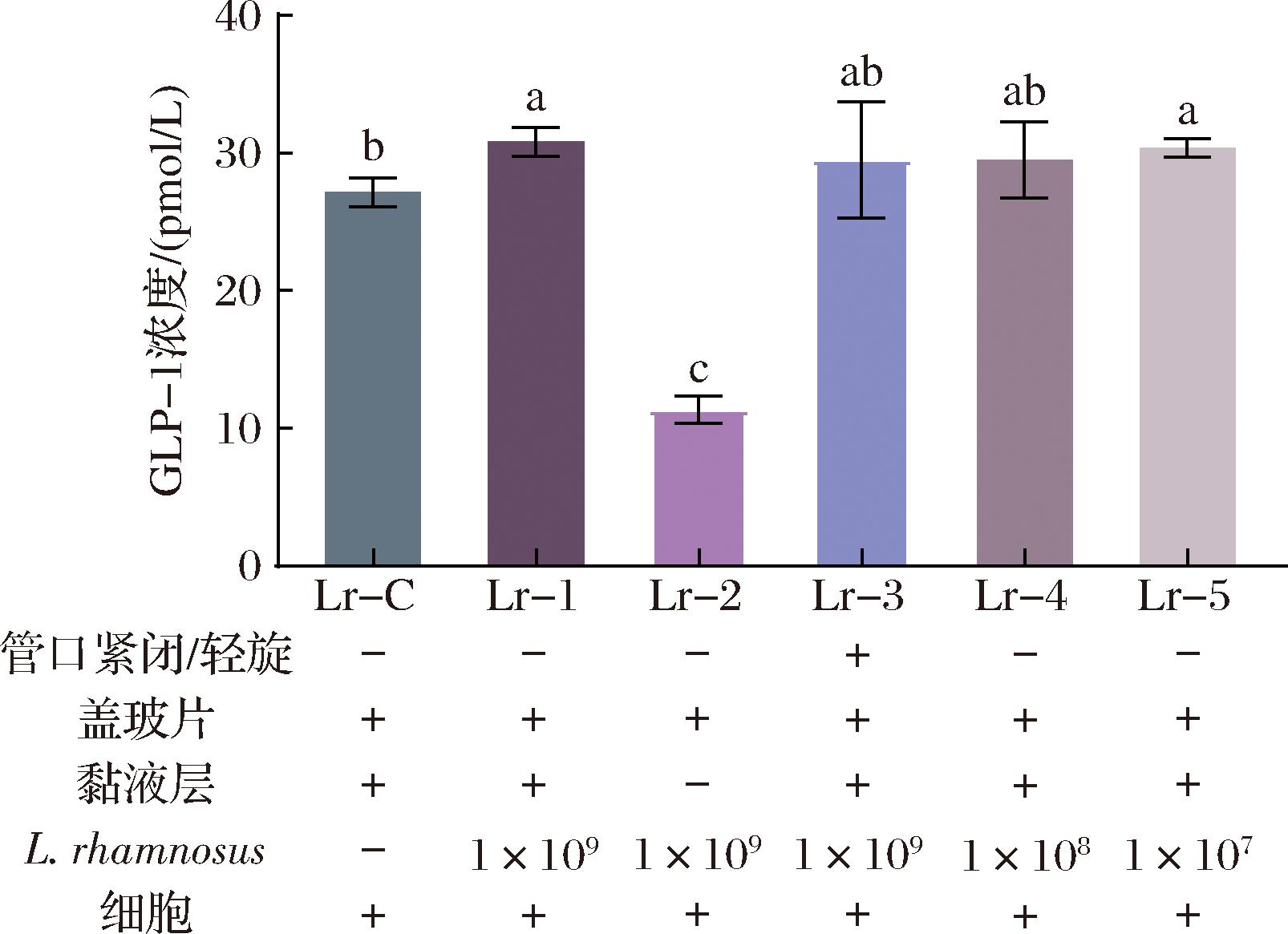
图2 L.rhamnosus对细胞分泌GLP-1的影响
Fig.2 Effect of L.rhamnosus on the GLP-1 secretion of pGIP/Neo STC-1 cells
注:L.rhamnosus行数值为体系中初始细菌浓度,单位为CFU/1 000 mL(下同)。
2.2 O2暴露和黏液层对细胞GLP-1分泌的影响
通过设置试管螺旋盖紧闭(Tube-closed)组和无黏液层(No-mucus)组,经与不含细菌的对照(Control)组和不含乳杆菌和细胞的空白对照(Blank)组比较,分析O2暴露情况和黏液存在与否对体系的影响。共培养体系的实拍图见电子版增强出版附图3。各组GLP-1浓度见图1,结果表明,No-mucus组的GLP-1浓度(15.13 pmol/L)显著低于Control组(26.01 pmol/L)(P<0.05),说明黏液层缺失会降低GLP-1分泌。类似地,Tube-closed组的GLP-1浓度(14.58 pmol/L)明显低于Control组(P<0.05),说明限制O2量会降低GLP-1分泌。
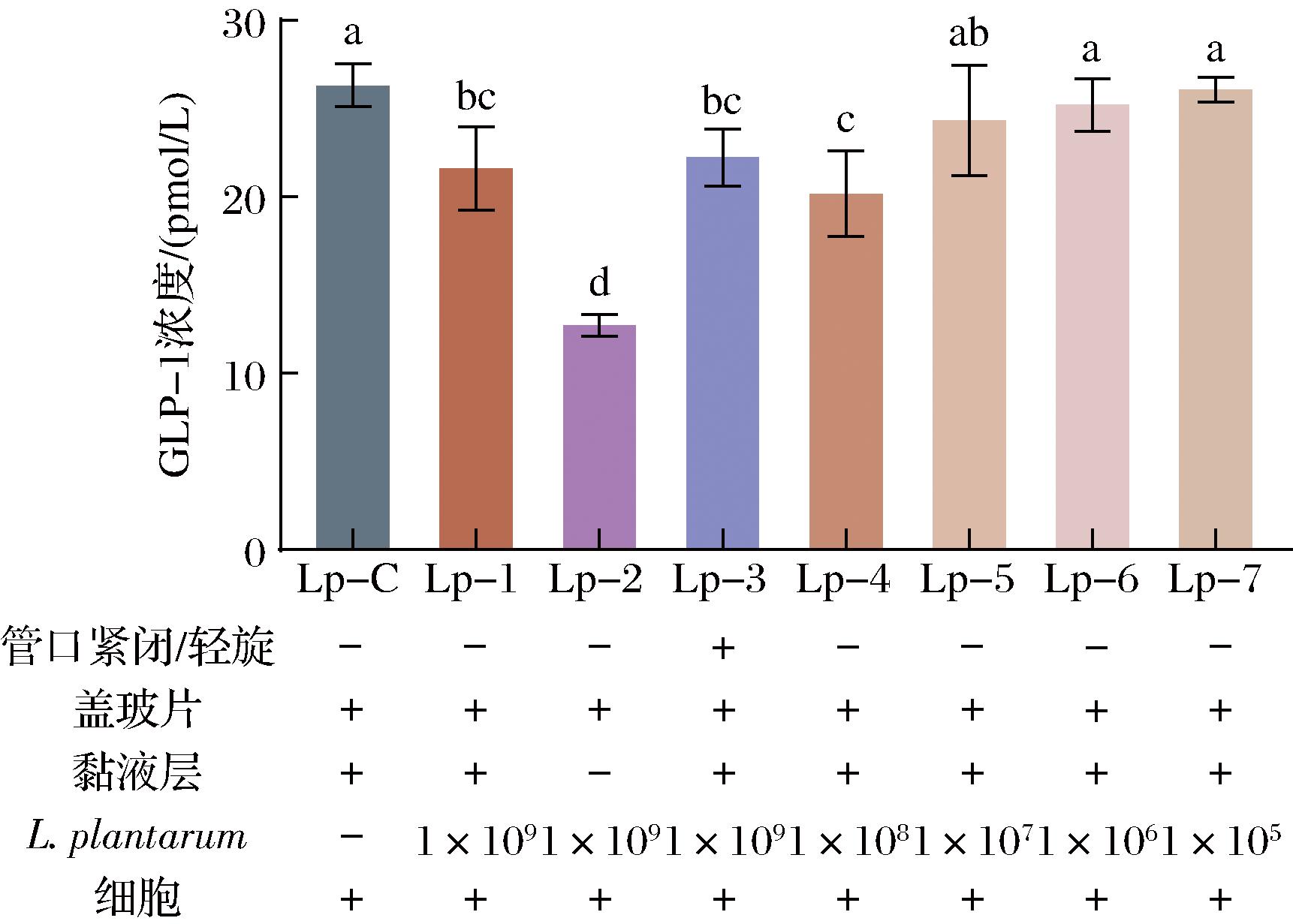
图3 L.plantarum对细胞分泌GLP-1的影响
Fig.3 Effect of L.plantarum on the GLP-1 secretion of pGIP/Neo STC-1 cells
2.3 乳杆菌对pGIP/Neo STC-1细胞GLP-1分泌的影响
2.3.1 L.rhamnosus
图2展示了与L.rhamnosus共同培养后pGIP/Neo STC-1细胞GLP-1的分泌情况。各组的条件设置见图下方标注。Lr-1组的GLP-1分泌明显高于Lr-C组(P<0.05),而Lr-1、Lr-4和Lr-5组间差异无统计学意义。这些结果表明,L.rhamnosus会增加PGIP/Neo STC-1细胞的GLP-1分泌,且菌株初始浓度为1×109、1×108、1×107 CFU/1 000 mL时促进效果是相似的。Lr-2组的GLP-1分泌量明显低于Lr-C组(P<0.05),说明与L.rhamnosus共培养时缺少黏液层会降低细胞GLP-1分泌量。另外,Lr-3组的GLP-1浓度与Lr-1组相比没有显著差异,说明限制O2条件并不影响该体系内GLP-1分泌。
2.3.2 L.plantarum
与L.plantarum共同培养后pGIP/Neo STC-1细胞分泌GLP-1的情况见图3,各组的条件设置见图下方标注。Lp-1和Lp-4组的GLP-1浓度明显低于Lp-C组(P<0.05),Lp-5、Lp-6和Lp-7组间无显著差异,表明L.plantarum初始浓度为1×105、1×106、1×107 CFU/1 000 mL时,不影响细胞GLP-1的分泌。然而当初始浓度为1×109 CFU/1 000 mL或1×108 CFU/1 000 mL,L.plantarum会减少细胞的GLP-1分泌量。Lp-2组的GLP-1浓度显著低于Lp-1组(P<0.05),Lp-1与Lp-3组间差异无统计学意义,说明在没有黏液层的情况下,细胞的GLP-1分泌减少,而O2条件对于与L.plantarum共培养的细胞没有显著影响。
2.3.3 L.johnsonii
图4显示了与L.johnsonii共同培养后pGIP/Neo STC-1细胞GLP-1的分泌情况,各组的条件设置见图下方标注。Lj-1组的GLP-1浓度显著低于Lj-C组(P<0.05),Lj-C、Lj-4和Lj-5组间无显著差异,说明当L.johnsonii的初始浓度为1×107、1×108 CFU/1 000 mL时,与其共培养不影响pGIP/Neo STC-1细胞的GLP-1分泌。而当初始浓度为1×109 CFU/1 000 mL时,会降低细胞的GLP-1分泌。Lj-2和Lj-3组的GLP-1浓度显著低于Lj-1组(P<0.05),说明与L.johnsonii共培养时黏液层缺失或O2受限会减少细胞GLP-1分泌。
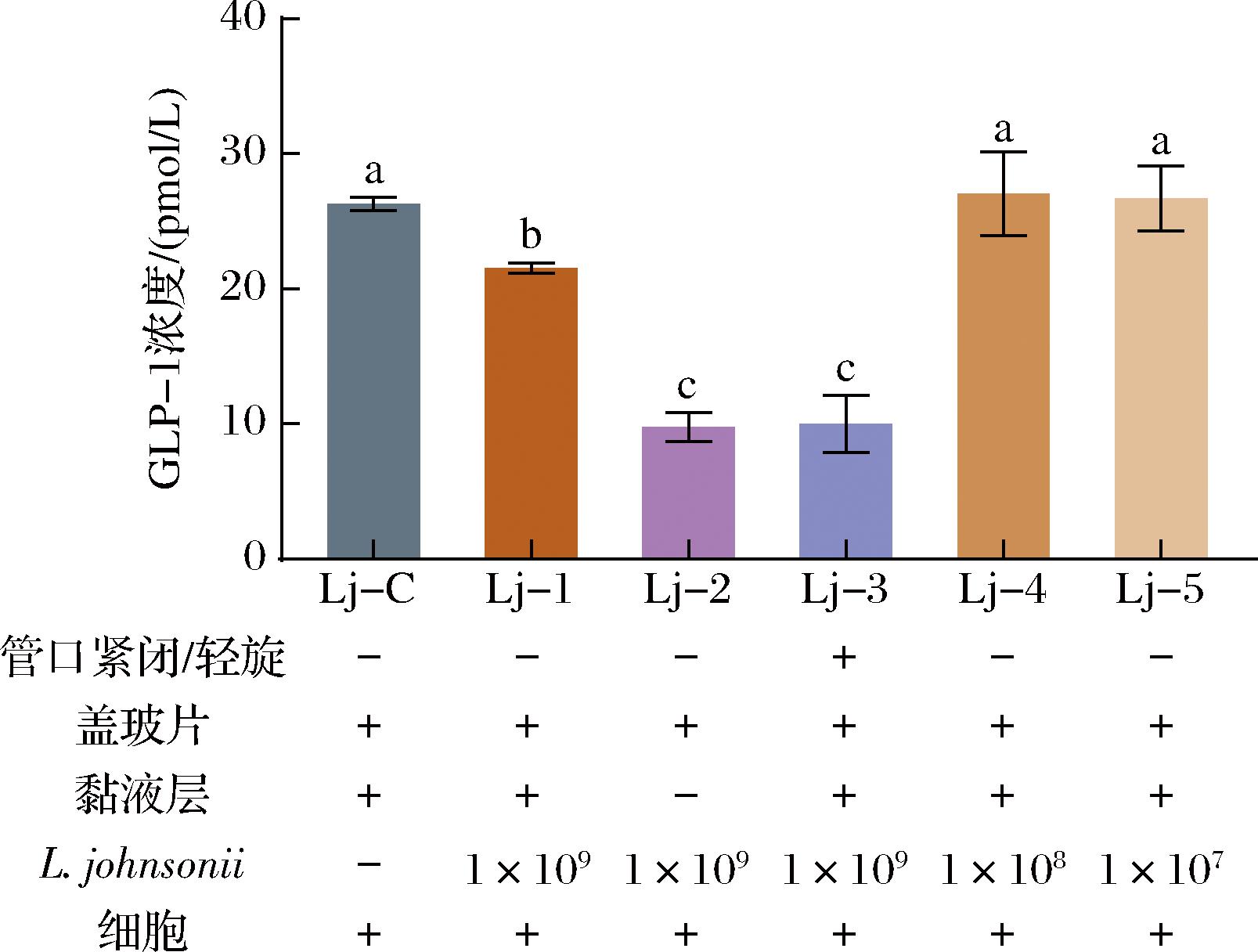
图4 L.johnsonii对细胞分泌GLP-1的影响
Fig.4 Effect of L.johnsonii on the GLP-1 secretion of pGIP/Neo STC-1 cells
2.4 体系pH值和细胞活力
共培养前后各组pH值变化见表1。Blank、Control和No-mucus组的pH值无显著差异,而Tube-closed组的pH显著低于上述3组(P<0.05),但仍高于7.4。Lr-C、Lp-C和Lj-C组的pH值分别显著高于Lr-1~Lr-5、Lp-1~Lp-7和Lj-1~Lj-5组(P<0.05),说明L.rhamnosus、L.plantarum和L.johnsonii的存在降低了共培养体系的pH值。
表1 共培养后顶部液体培养基的pH值和细胞活力
Table 1 pH of the top liquid medium and viability of cells after co-culture
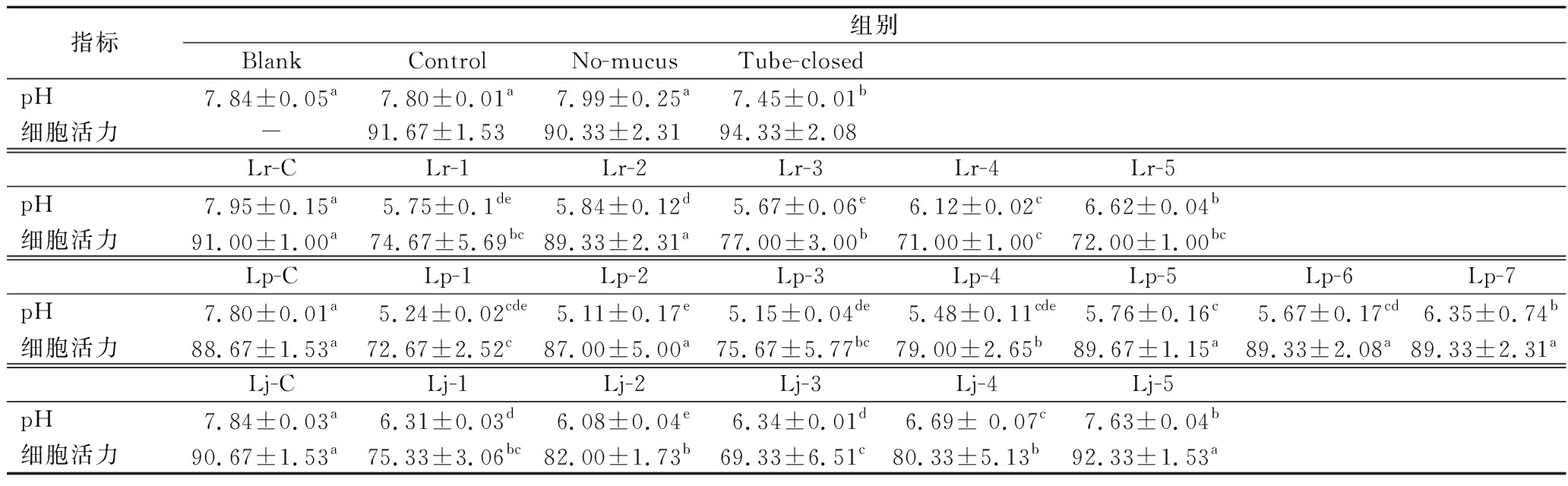
指标组别BlankControlNo-mucusTube-closedpH7.84±0.05a7.80±0.01a7.99±0.25a7.45±0.01b细胞活力-91.67±1.5390.33±2.3194.33±2.08Lr-CLr-1Lr-2Lr-3Lr-4Lr-5pH7.95±0.15a5.75±0.1de5.84±0.12d5.67±0.06e6.12±0.02c6.62±0.04b细胞活力91.00±1.00a74.67±5.69bc89.33±2.31a77.00±3.00b71.00±1.00c72.00±1.00bcLp-CLp-1Lp-2Lp-3Lp-4Lp-5Lp-6Lp-7pH7.80±0.01a5.24±0.02cde5.11±0.17e5.15±0.04de5.48±0.11cde5.76±0.16c5.67±0.17cd6.35±0.74b细胞活力88.67±1.53a72.67±2.52c87.00±5.00a75.67±5.77bc79.00±2.65b89.67±1.15a89.33±2.08a89.33±2.31aLj-CLj-1Lj-2Lj-3Lj-4Lj-5pH7.84±0.03a6.31±0.03d6.08±0.04e6.34±0.01d6.69± 0.07c7.63±0.04b细胞活力90.67±1.53a75.33±3.06bc82.00±1.73b69.33±6.51c80.33±5.13b92.33±1.53a
注:结果以平均值±标准偏差表示,同行数据肩标有不同字母表示差异显著(P<0.05);“-”表示没有细胞。
Control、No-mucus和Tube-closed组的细胞活力无显著差异(表1),表明在没有乳杆菌存在时,黏液层缺失或限氧条件并不会影响pGIP/Neo STC-1细胞的活力。Lr-1、Lr-3、Lr-4和Lr-5组的细胞活力显著低于Lr-C组(P<0.05),表明鼠李糖乳杆菌降低了共培养体系中的细胞活力。Lp-1、Lp-3和Lp-4组的细胞活力显著低于Lp-C组(P<0.05),而Lp-C与Lp-5、Lp-6和Lp-7组差异无统计学意义,说明植物乳杆菌在其起始浓度高于1×108 CFU/1 000 mL时会降低细胞活力,当浓度低于1×107 CFU/1 000 mL时不影响细胞活力。约翰氏乳杆菌组也观察到了相同的趋势,因为Lj-1、Lj-3和Lj-4组的细胞活力显著低于Lj-C和Lj-5组(P<0.05)。这些数据表明不同乳杆菌菌株对pGIP/Neo STC-1细胞活力影响不同,且不同起始浓度的同一种乳酸菌浓度对细胞活力的影响也有差异。
2.5 革兰氏染色和胞内GLP-1荧光成像
革兰氏染色的结果直观显示了盖玻片上乳杆菌和细胞的分布情况(图5)。在不含黏液层时,乳杆菌与细胞直接接触(图5-c),且部分细菌深入细胞内部。在有黏液层的体系中,黏液层隔绝了部分细菌。然而,仍有部分细菌穿过黏液层,迁移至细胞表面(图5-b、图5-d)。说明在该体系中黏液层具有一定的屏障作用,但并不能隔绝所有细菌。为了验证上清液中GLP-1水平是否真实反映GLP-1细胞内生成情况,进行了细胞内GLP-1成像分析(电子版增强出版附图4)。结果显示GLP-1强度与ELISA结果高度一致。说明ELISA结果具有较好的可靠性。

a-无细菌;b-常规共培养体系;c-无黏液层;d-管口紧闭
图5 共培养体系的革兰氏染色结果
Fig.5 Gram staining images of bacteria after co-culture with pGIP/Neo STC-1 cells
3 结论与讨论
目前肠道微生物与宿主间的相互作用分析多依赖于动物模型[19-20],但该方法存在物种差异;此外,部分学者开展了细菌和体细胞直接共培养试验[4, 9],但在体内微生物与细胞之间存在黏液层,因此该方法无法准确反映细菌细胞的互作情况。因此,构建模拟体内环境的体外共培养体系对细菌细胞互作研究具有重要意义。学者们已构建出类器官培养技术[21]、生物反应器模型[22]、微流控器官芯片装置[23]等技术方法体系,但上述方法体系操作复杂,技术和设备要求高。本文采用的共培养体系改良自“Human oxygen-bacteria anaerobic”体系,该体系由SADAGHIAN SADABAD等[24]构建,他们使用该系统研究了粪杆菌(Faecalibacterium prausnitzii,专性厌氧菌)对人体细胞的影响。该方法构建了简单而独特的体细胞-肠道微生物共生体系。在肠道微生物与宿主相互作用关系研究中具有良好的应用潜力。然而,肠腔是低氧环境,细菌在不同的O2条件下的表现可能不同[25-26]。因此,考虑到乳杆菌的生长条件,本研究使用普通MRS培养基代替厌氧培养基,将细菌的厌氧条件改为低氧状态。该体系的建立为研究体细胞与乳杆菌的互作提供了良好的方法模型。
研究分析了不同浓度L.rhamnosus、L.plantarum和L.johnsonii对pGIP/Neo STC-1细胞GLP-1分泌的影响。结果表明,L.rhamnosus会增加细胞的GLP-1分泌,而无浓度效应。然而,当L.plantarum和L.johnsonii的起始浓度分别低于1×107、1×108 CFU/1 000 mL时,GLP-1的分泌不受影响。值得注意的是,当L.plantarum和L.johnsonii的起始浓度分别高于1×108 CFU/1 000 mL和1×109 CFU/1 000 mL时,GLP-1的分泌量降低。与WEI等[9]的结果类似,本研究结果证实了不同的乳酸菌菌株对STC-1细胞具有不同的影响。然而,PANWAR等[4]通过直接将菌株与细胞共培养发现所选的3种菌株都可刺激GLP-1分泌。这说明不同共培养条件会获得不同的研究结果,而本研究的共培养体系更接近于真实的体内条件。
进一步研究了O2暴露对细胞GLP-1分泌的影响。当没有细菌存在时,细胞在低氧条件下表现出显著降低的GLP-1分泌水平(P<0.05)。当与L.rhamnosus和L.plantarum共培养时,有限的O2条件并不影响细胞的GLP-1分泌,而与L.johnsonii共培养时,GLP-1分泌显著降低(P<0.05)。本研究试图通过测量细胞活力来解释这种现象,然而,结果与GLP-1分泌的变化不符,因为有限的O2条件不会影响细胞的活力,无论是否有细菌存在。此外,由于无黏液层存在时细菌直接与细胞接触,但细胞GLP-1分泌量降低,说明细菌是通过产生代谢产物,而非直接接触影响细胞GLP-1代谢。因此,假设GLP-1分泌的不同变化趋势可能是由于菌株的不同代谢物引起的,但该假设有待进一步验证。
综上所述,不同的乳杆菌菌株对pGIP/Neo STC-1细胞GLP-1分泌的影响不同。而且,当浓度不同时,同一菌株表现出不同的效果。在进行体外研究以探讨肠道微生物与宿主之间的相互作用时,应考虑肠腔环境,例如O2浓度和黏液层的存在。在进一步的研究中应收集顶部液体培养基开展代谢组学分析,以筛选L.rhamnosus产生的刺激GLP-1分泌的功能成分。
[1] DRUCKER D J, NAUCK M A.The incretin system:Glucagon-like peptide-1 receptor agonists and dipeptidyl peptidase-4 inhibitors in type 2 diabetes[J].The Lancet, 2006, 368(9548):1696-1705.
[2] GILBERT M P, PRATLEY R E.GLP-1 analogs and DPP-4 inhibitors in type 2 diabetes therapy:Review of head-to-head clinical trials[J].Frontiers in Endocrinology, 2020, 11:178.
[3] BOSE A K, MOCANU M M, CARR R D, et al.Glucagon-like peptide 1 can directly protect the heart against ischemia/reperfusion injury[J].Diabetes, 2005, 54(1):146-151.
[4] PANWAR H, CALDERWOOD D, GILLESPIE A L, et al.Identification of lactic acid bacteria strains modulating incretin hormone secretion and gene expression in enteroendocrine cells[J].Journal of Functional Foods, 2016, 23:348-358.
[5] DE VOS W M, TILG H, VAN HUL M, et al.Gut microbiome and health:Mechanistic insights[J].Gut, 2022, 71(5):1020-1032.
[6] IATCU C O, STEEN A, COVASA M.Gut microbiota and complications of type-2 diabetes[J].Nutrients, 2021, 14(1):166.
[7] GIRDHAR K, SOTO M, HUANG Q, et al.Gut microbiota regulate pancreatic growth, exocrine function, and gut hormones[J].Diabetes, 2022, 71(5):945-960.
[8] LIANG Y Y, LIU L Y, JIA Y, et al.Correlation between gut microbiota and glucagon-like peptide-1 in patients with gestational diabetes mellitus[J].World Journal of Diabetes, 2022, 13(10):861-876.
[9] WEI S H, CHEN Y P, CHEN M J.Selecting probiotics with the abilities of enhancing GLP-1 to mitigate the progression of type 1 diabetes in vitro and in vivo[J].Journal of Functional Foods, 2015, 18:473-486.
[10] SHEN R L, DANG X Y, DONG J L, et al.Effects of oat β-glucan and barley β-glucan on fecal characteristics, intestinal microflora, and intestinal bacterial metabolites in rats[J].Journal of Agricultural and Food Chemistry, 2012, 60(45):11301-11308.
[11] SIMON M C, STRASSBURGER K, NOWOTNY B, et al.Intake of Lactobacillus reuteri improves incretin and insulin secretion in glucose-tolerant humans:A proof of concept[J].Diabetes Care, 2015, 38(10):1827-1834.
[12] WANG G, LI X F, ZHAO J X, et al.Lactobacillus casei CCFM419 attenuates type 2 diabetes via a gut microbiota dependent mechanism[J].Food &Function, 2017, 8(9):3155-3164.
[13] MARZORATI M, VANHOECKE B, DE RYCK T, et al.The HMITM module:A new tool to study the Host-Microbiota Interaction in the human gastrointestinal tract in vitro[J].BMC Microbiology, 2014, 14:133.
[14] LEE K W, SHIN J S, LEE C M, et al.Gut-on-a-chip for the analysis of bacteria-bacteria interactions in gut microbial community:What would be needed for bacterial co-culture study to explore the diet-microbiota relationship?[J].Nutrients, 2023, 15(5):1131.
[15] IQBAL U,WESTFALL S,FARAHDEL L,et al.A novel probiotic blend for potential use in the metabolic syndrome:design and in vitro characterizations[J].International Journal of Probiotics &Prebiotics, 2018, 13(1):25-36.
[16] WANG C H, YEN H R, LU W L, et al.Adjuvant probiotics of Lactobacillus salivarius subsp.salicinius AP-32, L.johnsonii MH-68, and Bifidobacterium animalis subsp.lactis CP-9 attenuate glycemic levels and inflammatory cytokines in patients with type 1 diabetes mellitus[J].Frontiers in Endocrinology, 2022, 13:754401.
[17] TEIXEIRA L D, KLING D N, LORCA G L, et al.Lactobacillus johnsonii N6.2 diminishes caspase-1 maturation in the gastrointestinal system of diabetes prone rats[J].Beneficial Microbes, 2018, 9(3):527-539.
[18] LIU Y W, LIONG M T, TSAI Y C.New perspectives of Lactobacillus plantarum as a probiotic:The gut-heart-brain axis[J].Journal of Microbiology, 2018, 56(9):601-613.
[19] NGUYEN T L A, VIEIRA-SILVA S, LISTON A, et al.How informative is the mouse for human gut microbiota research?[J].Disease Models &Mechanisms, 2015, 8(1):1-16.
[20] FRITZ J V, DESAI M S, SHAH P, et al.From meta-omics to causality:Experimental models for human microbiome research[J].Microbiome, 2013, 1(1):14.
[21] WILLIAMSON I A, ARNOLD J W, SAMSA L A, et al.A high-throughput organoid microinjection platform to study gastrointestinal microbiota and luminal physiology[J].Cellular and Molecular Gastroenterology and Hepatology, 2018, 6(3):301-319.
[22] VAN DEN ABBEELE P, ROOS S, EECKHAUT V, et al.Incorporating a mucosal environment in a dynamic gut model results in a more representative colonization by lactobacilli[J].Microbial Biotechnology, 2012, 5(1):106-115.
[23] JALILI-FIROOZINEZHAD S, GAZZANIGA F S, CALAMARI E L, et al.A complex human gut microbiome cultured in an anaerobic intestine-on-a-chip[J].Nature Biomedical Engineering, 2019, 3:520-531.
[24] SADAGHIAN SADABAD M, VON MARTELS J Z H, KHAN M T, et al.A simple coculture system shows mutualism between anaerobic faecalibacteria and epithelial Caco-2 cells[J].Scientific Reports, 2015, 5:17906.
[25] MILLS D C, GUNDOGDU O, ELMI A, et al.Increase in Campylobacter jejuni invasion of intestinal epithelial cells under low-oxygen coculture conditions that reflect the in vivo environment[J].Infection and Immunity, 2012, 80(5):1690-1698.
[26] SHAH P, FRITZ J V, GLAAB E, et al.A microfluidics-based in vitro model of the gastrointestinal human-microbe interface[J].Nature Communications, 2016, 7:11535.