牦牛被誉为“高原之舟”,长期生活在海拔3 000 m以上的高原地区,是我国高海拔地区特有的珍稀牛种[1]。牦牛肉具有高蛋白、低脂肪、富含多种氨基酸和微量元素等特点,深受消费者的喜爱。风干牦牛肉是藏民族极具特色的传统生食肉制品,是藏民族在长期游牧生活中发现的一种传统牦牛肉贮藏方法[2]。风干牦牛肉是以新鲜牦牛肉为原料,经分割、切条后在低温干燥条件下自然风干而成[3],是当地主要的畜产消费品和接待亲朋好友的上等佳肴。在自然条件下,传统风干肉在内源酶和环境微生物作用下,发生一系列变化而形成具有独特风味的生食肉制品[4]。目前,风干牦牛肉制品主要以牧民家庭式生产、小作坊和企业批量生产3种生产方式生产[5],牧民家庭式生产方式由于传统生活习俗、不规范的制作工艺和自然环境条件下操作等因素,使产品面临环境致病或腐败微生物污染的风险,给产品食用安全性带来潜在风险[6]。因此,探讨传统风干牦牛肉中微生物群落组成及安全性,对改善家庭式风干牦牛肉生产方式,增强农牧民食品安全意识具有重要意义。
食品微生物学研究方法主要包括传统培养法、基于细胞组分或代谢特征差异的非培养生理生化法、基于代谢产物的现代分析技术和基于DNA或RNA等遗传信息的分子生物学方法[7]。高通量测序技术由于其通量高、信息量大、准确性高、能全面反映样品微生物群落结构及其丰度等优点[8],已被广泛应用于食品微生态学领域的研究。王俊钢等[9]利用高通量测序技术分析了新疆传统风干肉中细菌多样性,发现新疆哈萨克族传统风干肉中存在丰富的细菌菌群且含有大量的条件致病菌;文开勇等[10]利用高通量测序技术研究了四川传统腊肉中的微生物群落结构组成,发现不同来源腊肉中微生物群落组成存在一定差异;赵睿等[11]利用高通量测序技术对不同腌制肉品细菌多样性进行了分析,结果表明不同类别的腌制肉中微生物群落结构存在明显差异。目前,西藏传统风干牦牛肉制品的研究主要集中在加工工艺、品质和风味物质等方面[1,3,5],而有关其微生物方面的研究多侧重于益生菌的筛选[12-13],对西藏传统风干牦牛肉中的微生物群落组成研究不够全面。不同的地理环境、加工方式、生活习俗对产品中微生物群落组成及多样性的影响较大[6,9],因此,本研究采用高通量测序技术,分析西藏不同地区传统风干牦牛肉中微生物群落结构组成,明确传统风干牦牛肉中微生物多样性及安全性,旨在为进一步研究微生物菌群与风味品质间的相互关系、改善传统手工加工方式及品质控制提供理论依据。
1 材料与方法
1.1 样品采集与信息
风干牦牛肉样品于2022年3月采集自西藏林芝市巴宜区(BY)、拉萨市曲水县(LS)、山南市桑日县(SN)、日喀则市康马县(RKZ)和那曲市班戈县(NQ),样品信息如表1所示。风干牦牛肉制品均为牧民家庭式生产(新鲜牦牛肉切条后于阴凉处自然风干40 d),无任何添加剂和调味品,每个地区采集4份,共20份样品,装入无菌袋中,于4 ℃条件下带回实验室。
表1 样品信息
Table 1 Sample information
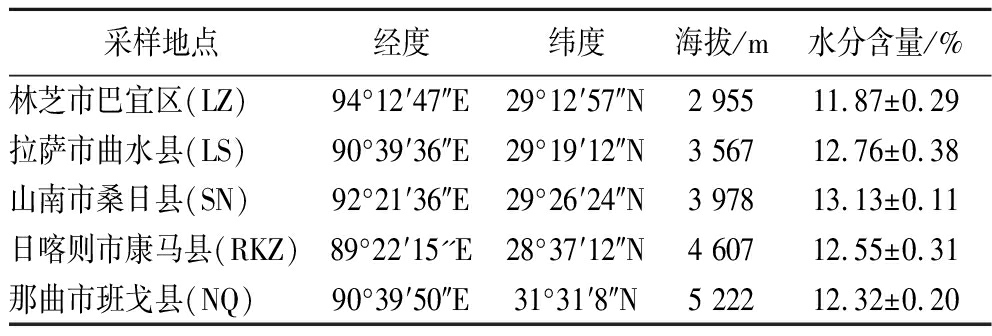
采样地点经度纬度海拔/m水分含量/%林芝市巴宜区(LZ)94°12′47″E29°12′57″N2 95511.87±0.29拉萨市曲水县(LS)90°39′36″E29°19′12″N3 56712.76±0.38山南市桑日县(SN)92°21′36″E29°26′24″N3 97813.13±0.11日喀则市康马县(RKZ)89°22′15''E28°37′12″N4 60712.55±0.31那曲市班戈县(NQ)90°39′50″E31°31′8″N5 22212.32±0.20
1.2 试剂
E.Z.N.A.® Stool DNA Kit M4015-02试剂盒、Gel Extraction Kit D2500-01试剂盒,美国Omega公司;2×Taq PCR Master Mix、D2000 DNA Marker,天根生化科技(北京)有限公司;琼脂糖,美国Invitrogen公司。
1.3 仪器与设备
5418R高速冷冻离心机,德国Eppendorf公司;NanoDrop2000微量分光光度计,美国Thermofisher公司;Tocan 240凝胶成像系统,上海领成生物科技有限公司;T100PCR仪,美国Bio-rad公司;DYY-15D电泳仪,北京六一。
1.4 样品总DNA提取及PCR扩增
按照E.Z.N.A.® Stool DNA Kit试剂盒操作说明提取样品总DNA,1%琼脂糖凝胶电泳检测总DNA质量,用NanoDrop2000微量分光光度计测定DNA浓度和纯度。利用通用引物338F(5′-ACTCCTACGGGAGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)扩增细菌16S rRNA基因V3~V4区,ITS1F(CTTGGTCATTTAGAGGAAGTAA)和ITS2R(GCTGCGTTCTTCATCGATGC)扩增真菌内源转录间隔区(internally transcribed spacer,ITS),胶回收PCR扩增产物并送至上海派森诺生物科技有限公司进行测序。
1.5 数据处理及分析
基于QIIME2 (2019.4)软件和Vsearch (v2.13.4)软件对原始数据进行质量过滤、去噪、拼接、去嵌合体和质控处理;采用QIIME2的classify-sklearn算法,基于细菌Greengenes数据库(Release 13.8, http://greengenes.secondgenome.com/)和真菌UNITE数据库(Release 8.0,https://unite.ut.ee/)进行物种分类学注释;采用QIIME2 (2019.4)和R语言进行alpha多样性分析、绘制稀释曲线、韦恩图和物种组成热图。
2 结果与分析
2.1 高通量测序结果分析
高通量测序结果如表2所示,20个样品共获得1 148 131条16S rRNA有效序列和1 490 806条ITS有效序列,平均序列长度分别为430 bp和254 bp。测序覆盖度和稀释曲线用于验证测序结果是否能真实反映样品微生物多样性[9],20个样品中微生物测序覆盖度为98.01%~100%,随着测序深度的增加,20个样品的稀释曲线均趋于平坦(图1),说明测序结果基本覆盖了样品中所有微生物类群,能真实反映样品微生物多样性。在97%相似度水平下,共获得38 655个细菌OTUs和2 498个真菌OTUs,其中LZ、LS、SN、RKZ和NQ样品中所获得的细菌OTUs数分别为7 464、7 650、8 474、7 759、7 308个,所获得的真菌OTUs数分别为419、657、498、506、418个。OTUs数与样品中微生物丰度密切相关,OTUs数越多,说明样品中微生物丰度越大[14]。SN样品中的细菌OTUs数最多,LS样品中真菌OTUs数最多,NQ样品中的细菌和真菌OTUs数最少,说明SN样品中细菌种类和LS样品中真菌种类最丰富。韦恩图能够直接展示不同样品间微生物群落组成的差异性和相似性[15]。由图2可知,不同地区样品中共有的细菌OTUs数为1 252个,占总OTUs数的3.24%,共有的真菌OTUs数为124个,占总OTUs数的4.96%,说明不同地区风干牦牛肉中微生物种类存在差异,这可能与风干牦牛肉制作环境中的微生物有关[4]。
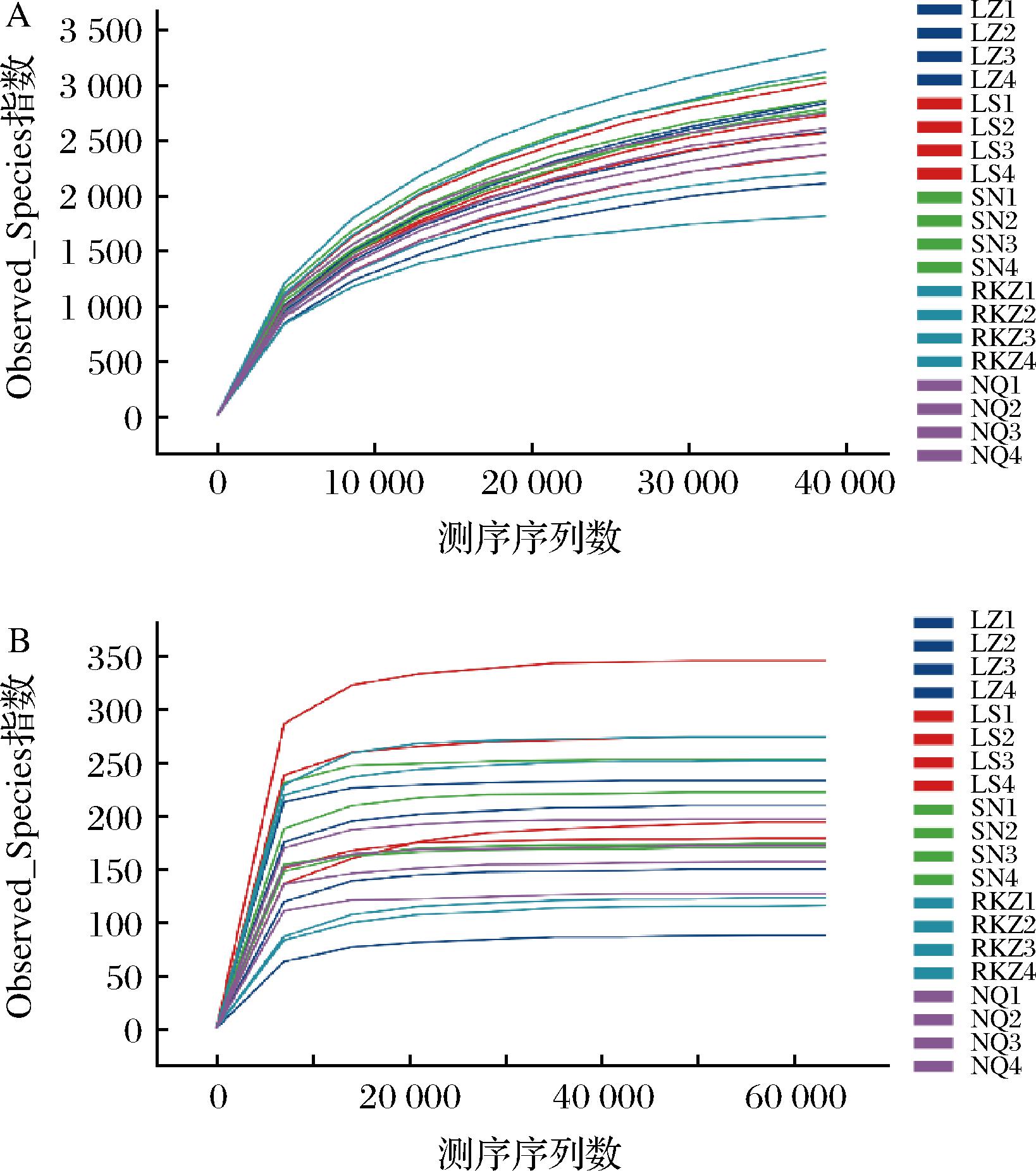
A-细菌;B-真菌
图1 样品细菌和真菌稀释曲线
Fig.1 Rarefaction curves of bacteria and fungi in samples
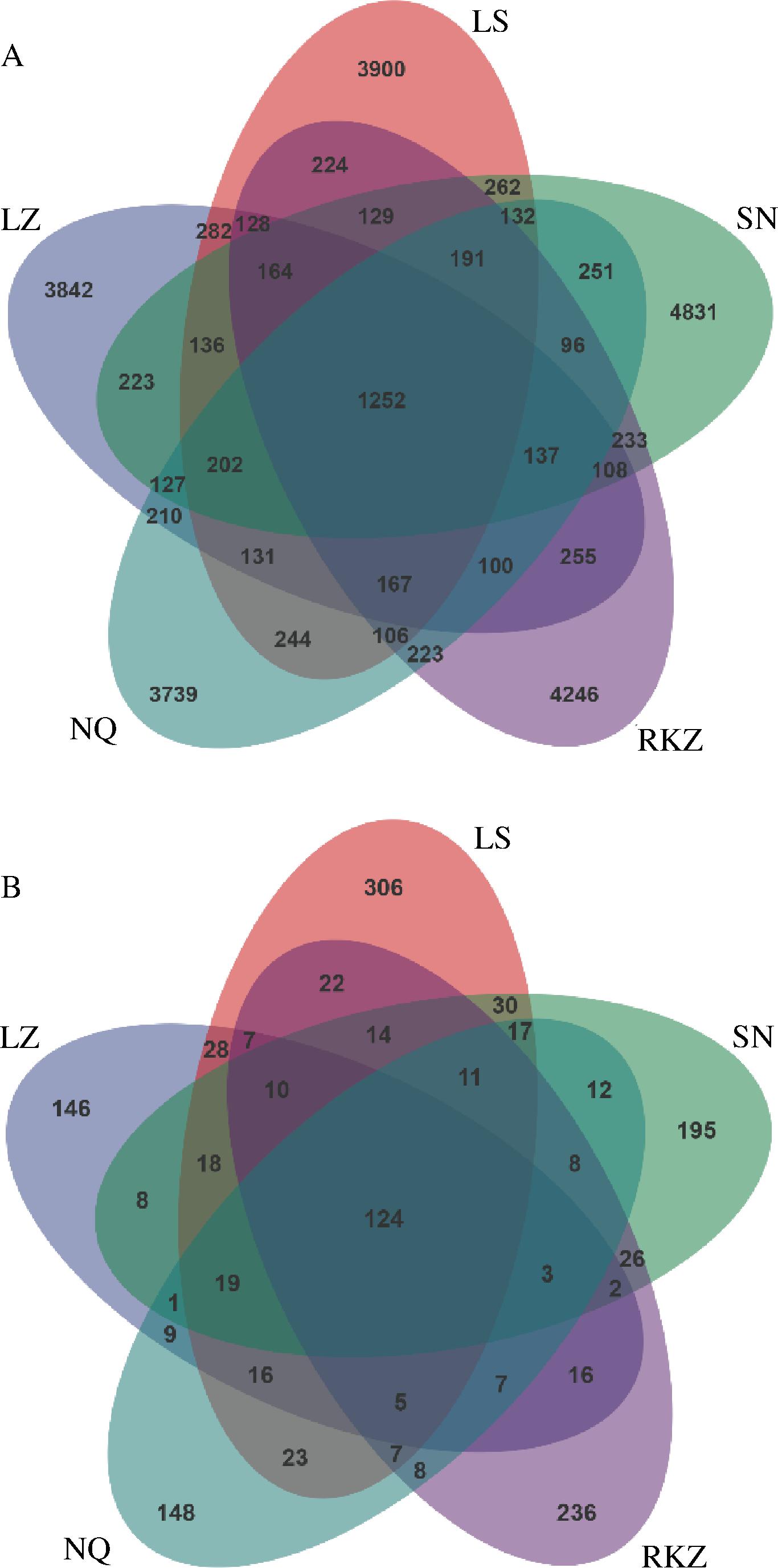
A-细菌;B-真菌
图2 样品细菌和真菌群落OTU韦恩图
Fig.2 Venn diagram of OTU of bacterial and fungal community in samples
表2 样品高通量测序结果
Table 2 The high-throughput sequencing results of the samples
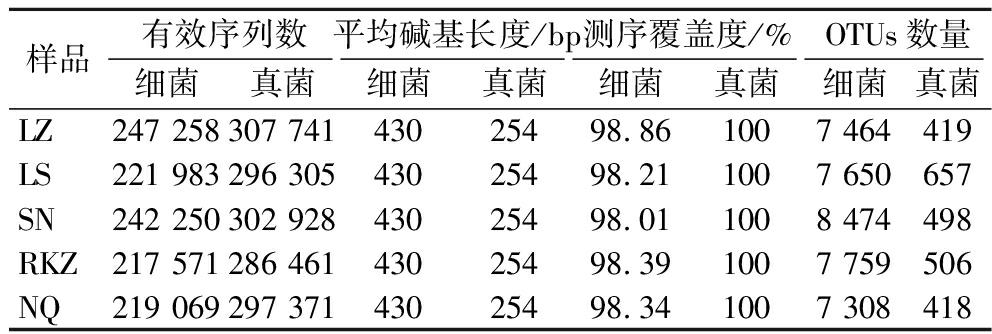
样品有效序列数平均碱基长度/bp测序覆盖度/%OTUs数量细菌 真菌细菌真菌细菌真菌细菌真菌LZ247 258307 74143025498.861007 464419LS221 983296 30543025498.211007 650657SN242 250302 92843025498.011008 474498RKZ217 571286 46143025498.391007 759506NQ219 069297 37143025498.341007 308418
2.2 Alpha多样性分析
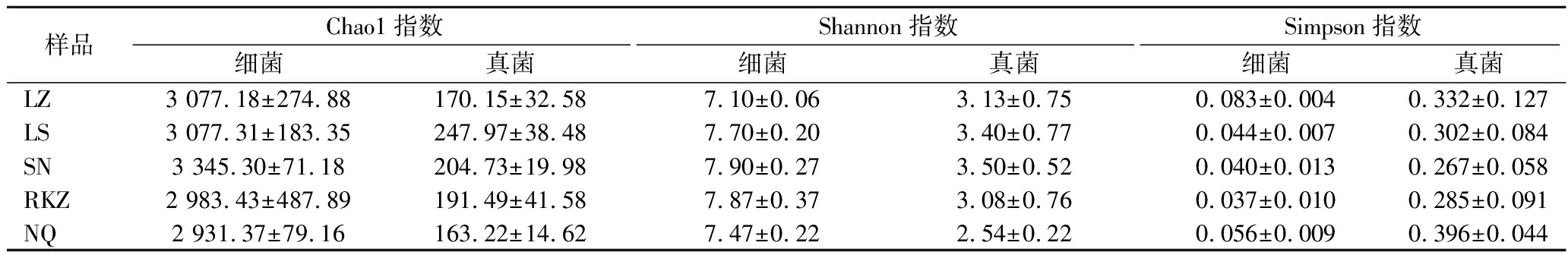
Alpha多样性指数中的Chao1、Shannon和Simpson指数主要用于反映样品微生物丰富度和多样性,Chao1指数值越大说明物种丰富度越高,Shannon指数值越大,Simpson指数值越小说明物种多样性越高[16]。由表3可知,SN样品细菌和LS样品真菌丰富度最高,NQ样品细菌和真菌丰富度最低;SN样品细菌和真菌多样性最高,LZ样品细菌和NQ样品真菌多样性最低。总体而言,随着海拔高度的升高,风干牦牛肉样品中细菌和真菌丰富度和多样性呈先增后降的趋势。高海拔生态系统由于其低降水量、低气温和低气压等特点,影响其环境微生物多样性[17-18],说明海拔对风干牦牛肉中微生物丰度和多样性存在一定的影响。有研究表明,随着海拔高度的升高,曲拉中细菌和真菌群落丰富度和多样性逐渐降低[19],这与本研究结果存在差异,这可能与风干牦牛肉制作环境和安全卫生条件等因素有关。
表3 样品alpha多样性指数
Table 3 Alpha diversity index of samples
样品Chao1指数Shannon指数Simpson指数细菌真菌细菌真菌细菌真菌LZ3 077.18±274.88170.15±32.587.10±0.063.13±0.750.083±0.0040.332±0.127LS3 077.31±183.35247.97±38.487.70±0.203.40±0.770.044±0.0070.302±0.084SN3 345.30±71.18204.73±19.987.90±0.273.50±0.520.040±0.0130.267±0.058RKZ2 983.43±487.89191.49±41.587.87±0.373.08±0.760.037±0.0100.285±0.091NQ2 931.37±79.16163.22±14.627.47±0.222.54±0.220.056±0.0090.396±0.044
2.3 门分类水平样品微生物群落组成
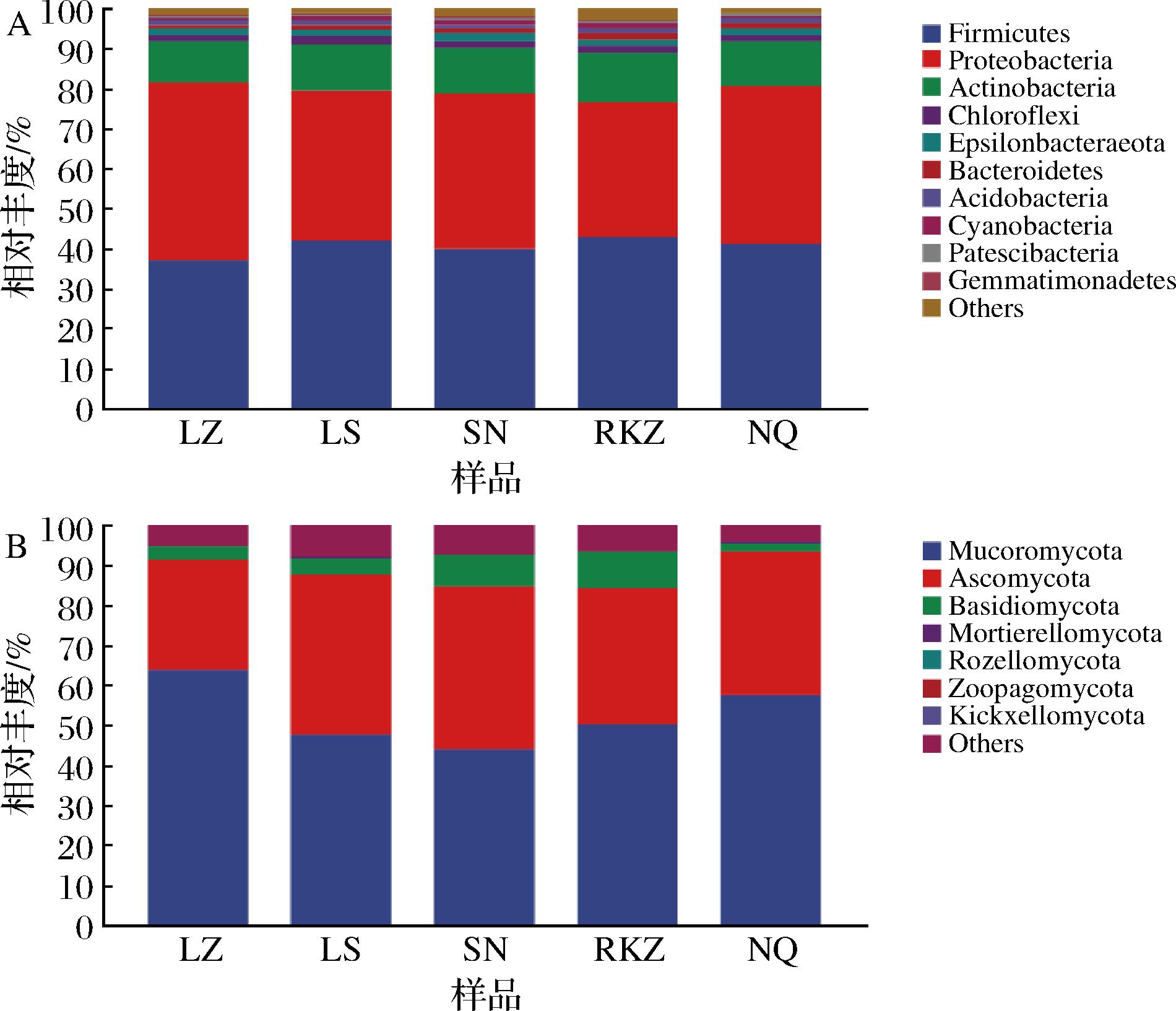
在门分类水平,不同样品微生物群落结构组成见图3。由图3-A可知,不同地区风干牦牛肉样品相对丰度前10的细菌门有厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、埃普西隆杆菌门(Epsilonbacteraeota)、拟杆菌门(Bacteroidetes)、酸杆菌门(Acidobacteria)、蓝菌门(Cyanobacteria)、髌骨细菌门(Patescibacteria)和芽单胞菌门(Gemmatimonadetes),其中厚壁菌门、变形菌门和放线菌门是不同地区风干牦牛肉样品中的主要优势细菌门,其平均相对丰度占比90.79%。变形菌门(44.58%)是LZ样品中的第一优势菌门,其次是厚壁菌门(37.04%)和放线菌门(10.14%),而LS、SN、RKZ和NQ样品中的第一优势菌门均是厚壁菌门,其相对丰度分别为41.84%、39.85%,42.75%和41.10%,其次是变形菌门(相对丰度分别为37.50%,38.73%,33.85%和39.73%)和放线菌门(相对丰度分别为11.80%,11.53%,12.40%和11.12%)。不同样品中的优势菌门基本相同,但相对丰度略有差异。有研究表明,厚壁菌门、变形菌门和放线菌是不同腌制腊肉[11]、传统自然发酵酸肉[20]和风干肉[6]中的优势菌门,这与本研究结果一致。弋凯鸽等[21]研究表明,青藏高原风干牦牛肉中的优势细菌门是厚壁菌门和放线菌门,王俊钢等[9]研究表明,变形菌门、厚壁菌门和蓝细菌门是新疆传统风干肉中的优势细菌门,这与本研究结果存在差异,这可能与采样点地理环境、海拔、加工卫生条件等因素有关。厚壁菌门中的革兰氏阳性菌和变形菌门中的革兰氏阴性菌是人体肠道内的优势益生菌群,如厚壁菌门代谢产生的丁酸和变形菌门发酵产生的丙酸对人体健康十分重要[22-23],同时,厚壁菌门和变形菌门中也存在很多病原菌,如葡萄球菌、沙门氏菌、假单胞菌、霍乱弧菌和大肠杆菌等[9,24],这些病原菌对食品安全性具有潜在隐患,而厚壁菌门和变形菌门是不同样品中的优势菌群,因此,在某种程度上,传统风干牦牛肉可能存在安全隐患。
A-细菌;B-真菌
图3 基于门分类水平样品细菌和真菌群落组成
Fig.3 Bacterial and fungal community composition of samples at phylum level
由图3-B可知,不同地区风干牦牛肉样品中的真菌主要集中在毛霉菌门(Mucoromycota)、子囊菌门(Ascomycota)、担子菌门(Basidiomycota)、被孢霉门(Mortierellomycota)、罗兹菌门(Rozellomycota)、捕虫霉门(Zoopagomycota)和梳霉门(Kickxellomycota)这7个门,平均相对丰度占比93.80%,其中相对丰度﹥1%的真菌门有3个,分别是毛霉菌门、子囊菌门、担子菌门。毛霉菌门是不同样品中的第一优势真菌门,其相对丰度为43.96%~63.61%,其次是子囊菌门(27.9%~41.00%)和担子菌门(2.03%~8.96%)。有研究表明,子囊菌门和担子菌门是四川传统腊肉中的优势菌门[10],被孢霉门、子囊菌门和担子菌门是湘派卤牛肉中的优势菌门[25],新疆传统风干肉中的优势真菌是子囊菌门、担子菌门和被孢霉门[26],这与本研究结果差异较大。地理环境、气候、海拔和原材料等因素可能是导致此差异的关键因素[11,19]。毛霉菌门中存在致腐和致病毛霉门真菌[27-28],这对食品安全存在潜在风险。西藏传统风干牦牛肉中存在大量毛霉门真菌,因此须进一步研究风干牦牛肉中真菌特性以确定其安全性。
2.4 属分类水平样品微生物群落组成
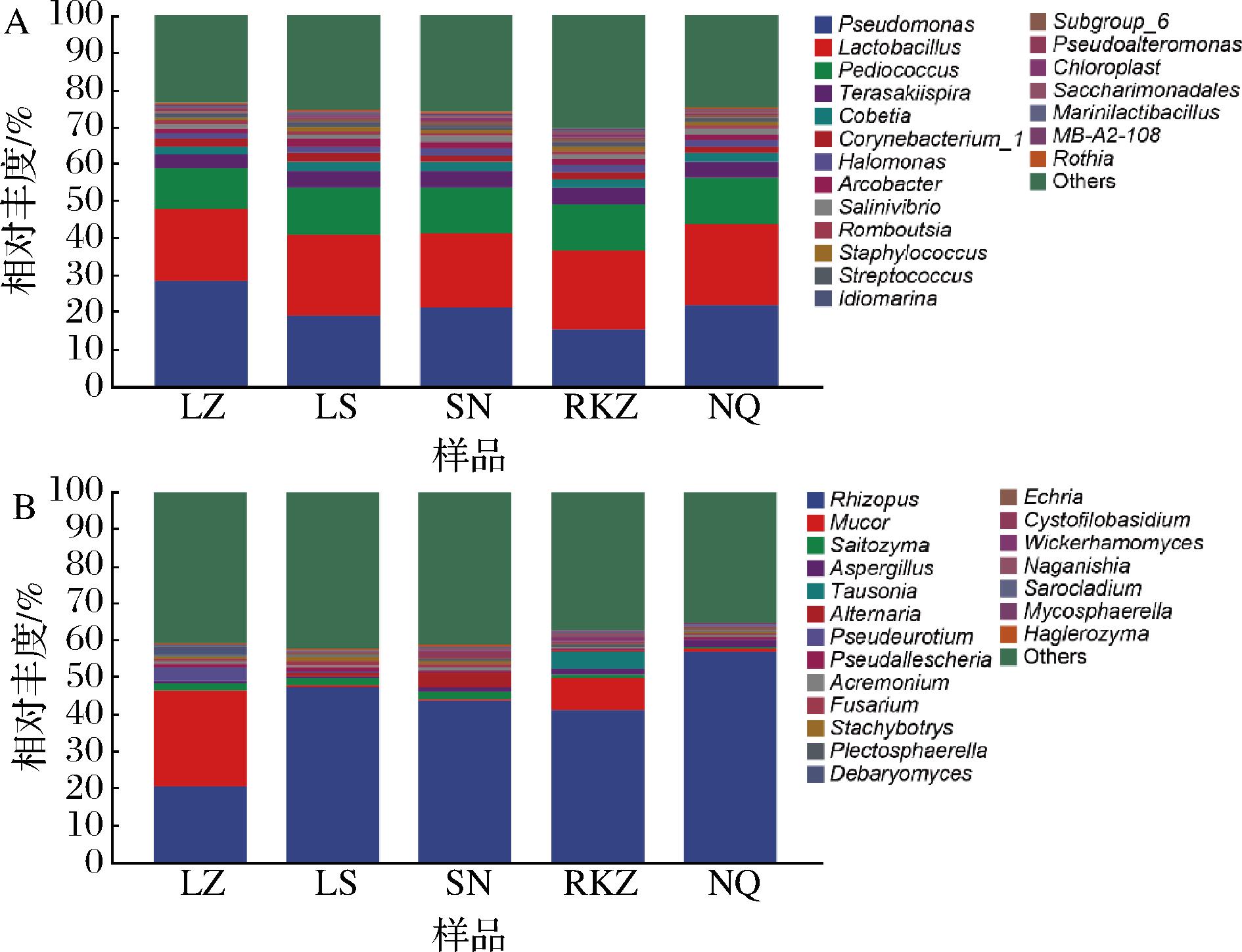
在属分类水平,20个样品共检测出1 151个细菌属,相对丰度前20的细菌属如图4-A所示。不同样品中相对丰度﹥1%的细菌属有9个,分别是假单胞菌属(Pseudomonas)、乳杆菌属(Lactobacillus)、片球菌属(Pediococcus)、Terasakiispira属、Cobetia属、棒状杆菌属(Corynebacterium_1)、盐单胞菌(Halomonas)、弓形杆菌(Arcobacter)和盐弧菌属(Salinivibrio),平均相对丰度占比67.59%。假单胞菌属、乳杆菌属和片球菌属是不同样品中的优势菌属,其相对丰度均在10%以上。虽然不同样品中的主要优势菌属基本相同,但相对丰度存在差异。LZ、SN和NQ样品中的第一优势菌属是假单胞菌属,平均相对丰度占比23.74%,其次是乳杆菌属(20.39%)和片球菌属(12.11%),而LS和RKZ样品中的第一优势菌属是乳杆菌属,平均相对丰度占比21.62%,其次是假单胞菌属(16.87%)和片球菌属(12.65%),说明不同地区风干牦牛肉中细菌群落组成存在差异。有研究表明,发酵时间、地理环境、气候、海拔和原材料等因素影响微生物群落组成[11,19,29],因此,这些因素可能是导致不同地区风干牦牛肉中微生物群落组成差异的原因。葡萄球菌属(Staphylococcus)、嗜冷杆菌属(Psychrobacter)和乳杆菌属是不同腊肉样品中的优势细菌属[11],嗜冷杆菌属和环丝菌属是新疆传统风干肉中的优势细菌属[9],乳酸杆菌属、假单胞菌属、棒状杆菌属和节杆菌属(Arthrobacter)是青藏高原风干牦牛肉中的优势细菌属[21],这与本研究结果差异较大。取样地生态环境、原材料和加工方式等因素可能是导致微生物群落组成差异的关键因素。乳酸菌被认为是食品发酵中的安全菌株,而其他菌属可能对食品安全存在隐患,如假单胞菌、嗜冷杆菌、不动杆菌等属于条件致病菌[6,30-31],而本研究样品中存在大量条件致病菌,严重影响传统风干牦牛肉的食用安全性。
A-细菌;B-真菌
图4 基于属分类水平样品细菌和真菌群落组成
Fig.4 Bacterial and fungal community composition of samples at genus level
20个样品中共检测出306个真菌属,相对丰度前20的真菌属如图4-B所示。LZ样品的优势真菌属(相对丰度>1%)为毛霉属(Mucor)(26.22%)、根霉属(Rhizopus)(20.38%)、假散囊菌属(Pseudeurotium)(3.73%)、德巴利酵母属(Debaryomyces)(2.09%)和沙蜥属(Saitozyma)(1.39%);LS样品中的优势真菌属是根霉属(47.32%)、沙蜥属(2.02%)和镰刀菌属(Fusarium)(1.28%);根霉属(43.77%)和沙蜥属(2.06%)是SN样品中的优势真菌属;根霉属(41.26%)、毛霉属(8.67%)、Tausonia属(4.56%)、曲霉属(Aspergillus)(1.84%)、担子菌酵母属(Naganishia)(1.09%)和威克汉姆酵母属(Wickerhamomyces)(1.08%)是RKZ样品中的优势真菌属;根霉属(56.84%)和曲霉菌属(1.73%)是NQ样品中的优势真菌属。不同样品中主要优势真菌属组成及相对丰度差异较大。有研究表明,新疆风干肉中的优势真菌属是假丝酵母属(Candida)、Mrakia属和德巴利酵母属(Debaryomyces)[26],曲霉属、德巴利酵母属和假丝酵母属是四川传统腊肉中的优势真菌属[10],这与本研究差异较大,说明地理环境、气候、海拔和原材料等因素影响微生物群落组成。根霉属和毛霉属真菌能分泌多种酶类、有机酸和芳香的脂类物质[32],被广泛用于食品发酵工业。霉菌和酵母菌在食品发酵过程中不仅能够提升产品风味,还能抑制腐败微生物的生长和过氧化物的生成,进而提升产品安全性和保质期[26]。不同地区风干牦牛肉中富含丰富的霉菌和一定数量的酵母菌,这可能对风干牦牛肉品质的形成有一定的影响。
2.5 Beta多样性差异分析
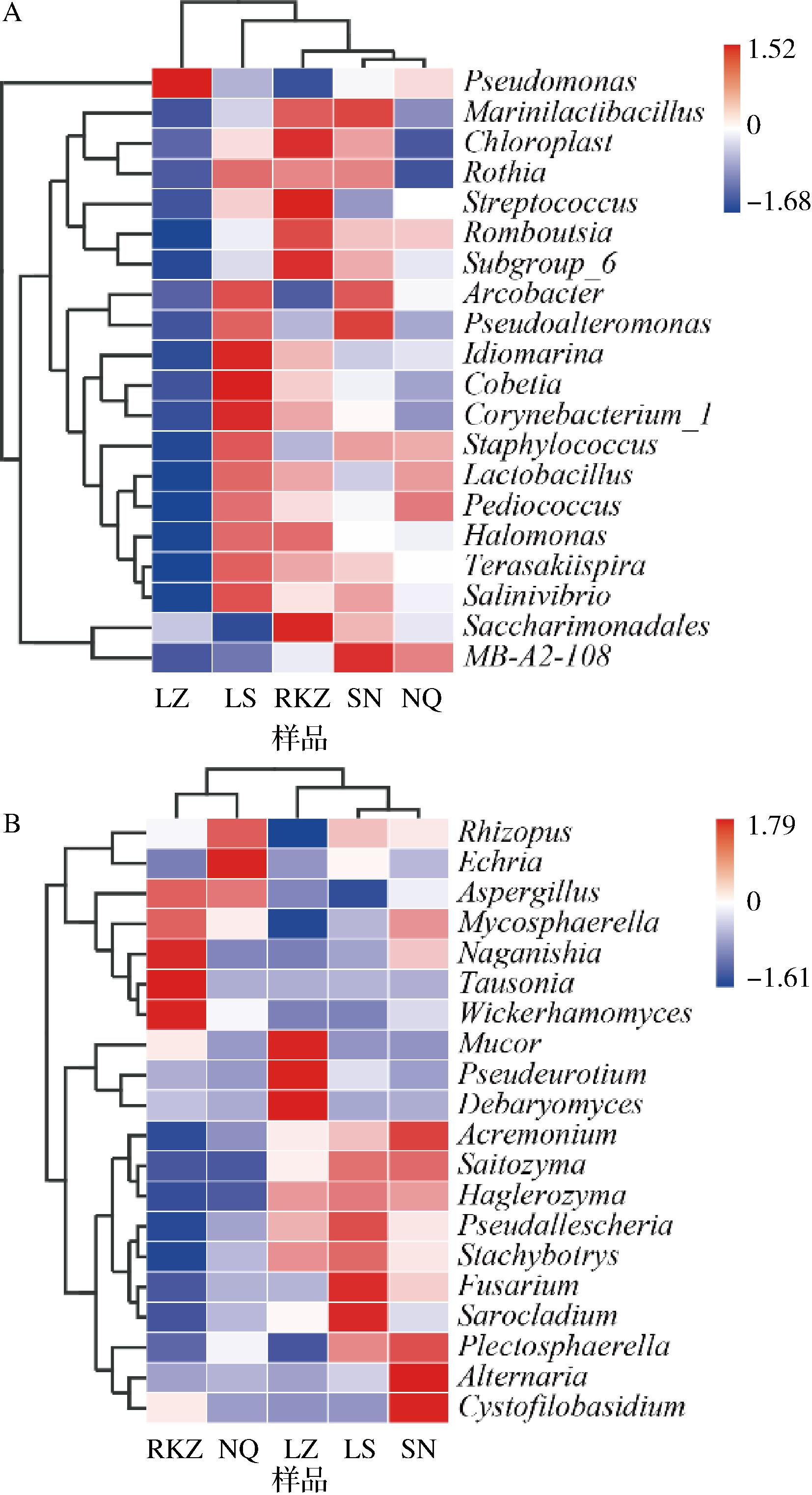
为进一步比较不同样品间微生物群落组成的差异性和相似性,对相对丰度前20的细菌和真菌属进行热图聚类分析。由图5-A可知,SN与NQ样品间距离最近,与LZ样品距离最远,说明SN与NQ样品间细菌群落组成相似,而LZ样品与其他样品细菌群落组成差异较大。不同样品间细菌群落组成差异性与海拔间无明显规律,说明海拔不是影响风干牦牛肉中细菌群落组成的关键因素。弋凯鸽等[21]研究表明,那曲与拉萨地区风干牦牛肉中细菌群落组成相似,而山南与日喀则地区风干牦牛肉中细菌群落组成相似,这与本研究结果存在差异,说明样品采样地环境影响风干牦牛肉中细菌群落结构组成。由图5-B可知,RKZ和NQ样品间距离最近,LS和SN样品间距离最近,说明RKZ和NQ样品间真菌群落组成相似,LS和SN样品间真菌群落组成相似。不同样品间真菌群落组成差异性与海拔间呈一定趋势,说明海拔高度可能影响风干牦牛肉中真菌群落组成。
A-细菌;B-真菌
图5 属分类水平样品中相对丰度前20的细菌和真菌聚类热图
Fig.5 Cluster heat map of relative abundance top 20 bacterial and fungal community at genus level
3 结论
20个样品高通量测序后共获得1 148 131条16S rRNA有效序列和1 490 806条ITS有效序列;在97%相似度水平下,样品共获得38 655个细菌OTUs和2 498个真菌OTUs;不同地区风干牦牛肉中微生物丰富度和多样性随海拔升高呈先增后降的趋势;在门分类水平,不同地区风干牦牛肉样品中的主要优势菌门是厚壁菌门、变形菌门、放线菌门、毛霉菌门、子囊菌门和担子菌门;在属分类水平,假单胞菌属、乳杆菌属、片球菌属、根霉属和毛霉属是不同地区风干牦牛肉中的优势菌属,不同地区风干牦牛肉中细菌和真菌群落组成存在差异;热图聚类分析表明,LZ样品与其他样品微生物群落组成差异较大;不同地区风干牦牛肉中即存在功能性益生微生物菌群,又存在大量条件致病菌,这些致病菌具有一定的安全隐患。
[1] 张丽, 马纪兵, 王妍, 等.甘肃牧区风干牦牛肉加工过程中的品质变化[J].食品工业科技, 2017, 38(21):1-6;11. ZHANG L,MA J B,WANG Y, et al.Quality change during processing of dried yak meat in pastoral areas of Gansu[J].Science and Technology of Food Industry, 2017, 38(21):1-6;11.
[2] 高媛. 风干牦牛肉品质形成规律与工艺改进技术研究[D].北京:中国农业科学院, 2013. GAO Y.Studies on the rule of quality forming and technology improvement of airing yak beef[D].Beijing:Chinese Academy of Agricultural Sciences, 2013.
[3] 王惠惠, 马纪兵, 刘小波, 等.甘肃牧区传统风干牦牛肉加工过程中挥发性风味物质变化分析[J].食品与发酵工业, 2019, 45(4):200-205. WANG H H, MA J B, LIU X B, et al.Analysis of the changes of volatile flavor compounds during processing of traditional air-dried beef in pastoral area in Gansu province[J].Food and Fermentation Industries, 2019, 45(4):200-205.
[4] 党欣. 哈萨克风干牛肉产品特性研究及工艺优化[D].兰州:甘肃农业大学,2014. DANG X.Studies on the rule of quality forming and technology improvement of the Kazakh air-dried beef[D].Lanzhou:Gansu Agricultural University, 2014.
[5] 闫晓晶, 雷元华, 谢鹏, 等.牦牛肉干制品加工研究进展[J].肉类研究, 2019, 33(3):67-71. YAN X J, LEI Y H, XIE P, et al.Recent progress in processing technologies for yak jerky meat[J].Meat Research, 2019, 33(3):67-71.
[6] 田建军, 张开屏, 杨明阳, 等.应用Illumina MiSeq测序技术比较风干肉中细菌多样性和微生物安全性[J].食品科学, 2019, 40(8):33-40. TIAN J J, ZHANG K P, YANG M Y, et al.Comparative bacterial diversity analysis and microbial safety assessment of air-dried meat products by illumina MiSeq sequencing technology[J].Food Science, 2019, 40(8):33-40.
[7] 缪璐欢, 白凤翎, 励建荣.传统发酵食品中乳酸菌生态演替研究进展[J].食品与发酵工业, 2015, 41(1):175-180. MIAO L H,BAI F L, LI J R.Advanced on ecological succession of lactic acid bacteria in traditional fermented food[J].Food and Fermentation Industries, 2015, 41(1):175-180.
[8] KERGOURLAY G, TAMINIAU B, DAUBE G, et al.Metagenomic insights into the dynamics of microbial communities in food [J].International Journal of Food Microbiology, 2015, 213:31-39.
[9] 王俊钢, 李宇辉, 刘成江, 等. 新疆哈萨克族传统风干肉中真菌多样性分析[J]. 食品与发酵工业, 2021, 47(1): 35-42. WANG J G, LI Y H, LIU C J, et al. Diversity of fungus in traditional air-dried meat of Xinjiang Kazakh[J]. Food and Fermentation Industries, 2021, 47(1): 35-42.
[10] 文开勇, 汪月, 文鹏程, 等.四川传统腊肉中微生物群落结构研究[J].食品与发酵工业, 2020, 46(3):36-42. WEN K Y, WANG Y, WEN P C, et al.Study on microbial community structure in Sichuan traditional bacon[J].Food and Fermentation Industries, 2020, 46(3):36-42.
[11] 赵睿, 邵长春, 高世功, 等.高通量测序分析不同腌腊肉制品细菌多样性[J].食品科学, 2020, 41(20):90-96. ZHAO R, SHAO C C, GAO S G, et al.High-throughput sequencing analysis of bacterial diversity of different types of traditional Chinese bacon[J].Food Science, 2020, 41(20):90-96.
[12] 李丹阳, 李宇辉, 高云云, 等.新疆哈萨克族风干肉中产蛋白酶乳酸菌的筛选及酶学特性研究[J].食品与发酵工业, 2020, 46(9):57-63. LI D Y, LI Y H, GAO Y Y, et al.Screening of protease-producing lactic acid bacteria from Xinjiang Kazakh air-dried meat and their enzymatic characteristics [J].Food and Fermentation Industries, 2020, 46(9):57-63.
[13] 田建军, 张开屏, 赵艳红, 等.风干肉中产脂肪酶瑞士乳杆菌TR13全基因组测序及序列分析[J].食品科学, 2020, 41(16):101-109. TIAN J J, ZHANG K P, ZHAO Y H, et al.Whole genome sequencing and sequence analysis of Lactobacillus helveticus TR13 from air-dried mutton [J].Food Science, 2020, 41(16):101-109.
[14] ZHANG M L, ZHANG M H, ZHANG C H, et al.Pattern extraction of structural responses of gut microbiota to rotavirus infection via multivariate statistical analysis of clone library data[J].FEMS Microbiology Ecology, 2009, 70(2):21-29.
[15] EDGAR R C.UPARSE:Highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods, 2013, 10(10):996-998.
[16] QIU Z C, LI N Y, LU X M, et al.Characterization of microbial community structure and metabolic potential using Illumina MiSeq platform during the black garlic processing[J].Food Research International, 2018, 106:428-438.
[17] MOR N-TEJEDA E, L
N-TEJEDA E, L PEZ-MORENO J I, BENISTON M.The changing roles of temperature and precipitation on snowpack variability in Switzerland as a function of altitude[J].Geophysical Research Letters, 2013, 40(10):2131-2136.
PEZ-MORENO J I, BENISTON M.The changing roles of temperature and precipitation on snowpack variability in Switzerland as a function of altitude[J].Geophysical Research Letters, 2013, 40(10):2131-2136.
[18] KUMAR S, SUYAL D C, YADAV A, et al.Microbial diversity and soil physiochemical characteristic of higher altitude[J].PLoS One, 2019, 14(3):e0213844.
[19] 薛蓓, 卢灏泽, 杨帆, 等.高通量测序技术分析西藏不同海拔地区曲拉微生物多样性[J].食品科学, 2022, 43(14):176-182. XUE B, LU H Z, YANG F, et al.High-throughput sequencing technology to analyze microbial community diversity in Qula at different altitudes in Tibet[J].Food Science, 2022, 43(14):176-182.
[20] 米瑞芳, 陈曦, 熊苏玥, 等.传统自然发酵酸肉中细菌群落多样性与风味品质分析[J].食品科学, 2019, 40(2):85-92. MI R F, CHEN X, XIONG S Y, et al.Bacterial community diversity and flavor characteristics of traditional naturally fermented sour meat[J].Food Science, 2019, 40(2):85-92.
[21] 弋凯鸽, 吴寒冰, 李帅康, 等.基于高通量测序技术分析青藏高原风干牦牛肉中细菌多样性[J].食品工业科技, 2022, 43(13):118-124. YI K G, WU H B, LI S K, et al.Analysis of bacterial diversity in Tibetan Plateau dry yak meat based on high-throughput sequencing [J].Science and Technology of Food Industry, 2022, 43(13):118-124.
[22] PRYDE S E, DUNCAN S H, HOLD G L, et al.The microbiology of butyrate formation in the human colon[J].FEMS Microbiology Letters, 2002, 217(2):133-139.
[23] BIANCHI F, DALL’ASTA M, DEL RIO D, et al.Development of a headspace solid-phase microextraction gas chromatography-mass spectrometric method for the determination of short-chain fatty acids from intestinal fermentation[J].Food Chemistry, 2011, 129(1):200-205.
[24] 曲巍, 张智, 马建章, 等.高通量测序研究益生菌对小鼠肠道菌群的影响[J].食品科学, 2017, 38(1):214-219. QU W, ZHANG Z, MA J Z, et al.Effect of probiotics on gut microbiota in mice evaluated by high-throughput sequencing[J].Food Science, 2017, 38(1):214-219.
[25] 车丽娜, 赵良忠, 周晓洁.基于高通量测序分析湘派卤牛肉冷藏过程中真菌多样性[J].食品安全质量检测学报, 2023, 14(4):127-136. CHE L N, ZHAO L Z, ZHOU X J.High-throughput sequencing analysis of fungal diversity in Xiangpai dried beef during cold storage[J].Journal of Food Safety &Quality, 2023, 14(4):127-136.
[26] 王俊钢, 李宇辉, 刘成江, 等.新疆哈萨克族传统风干肉中真菌多样性分析[J].食品与发酵工业, 2021, 47(1):35-42. WANG J G, LI Y H, LIU C J, et al.Diversity of fungus in traditional air-dried meat of Xinjiang Kazakh[J].Food and Fermentation Industries, 2021, 47(1):35-42.
[27] 吕红莉, 李澄, 李燕, 等.中国原发性皮肤毛霉病32例回顾性分析[J].中国真菌学杂志, 2016, 11(2):103-107. LYU H L, LI C, LI Y, et al.Retrospective analysis of primary cutaneous mucormycosis in China:A review of 32 cases[J].Chinese Journal of Mycology, 2016, 11(2):103-107.
[28] 顾沛雯, 张光弟, 王华荣, 等.果蔬采后致腐病菌检测及防治[J].农业科学研究, 2012, 33(1):1-6. GU P W, ZHANG G D, WANG H R, et al.Determination and control of the main pathogens of post-harvest fruit and vegetables rot disease in Ningxia[J].Journal of Agricultural Sciences, 2012, 33(1):1-6.
[29] 李荣源, 卢红梅, 秦兴, 等.高通量测序分析赤水晒醋各生产阶段微生物群落结构变化[J].食品科学, 2020, 41(24):78-86. LI R Y, LU H M, QIN X, et al.High-throughput sequencing study on changes in microbial community structure in different production stages of Chishui sun-dried vinegar[J].Food Science, 2020, 41(24):78-86.
[30] 魏超, 代晓航, 郭灵安, 等.禽肉中铜绿假单胞菌的分离及其耐药性[J].肉类研究, 2017, 31(7):7-10. WEI C, DAI X H, GUO L A, et al.Isolation and drug resistance of Pseudomonas aeruginosa from poultry meat[J].Meat Research, 2017, 31(7):7-10.
[31] 王发祥,王满生,刘永乐,等.低温贮藏下草鱼肉优势腐败菌鉴定及其消长规律[J].食品与发酵工业, 2012, 38(2):66-68. WANG F X, WANG M S, LIU Y L, et al.The dominant spoilage bacteria and their growth and decline law in grass carp during low temperature preservation[J].Food and Fermentation Industries, 2012, 38(2):66-68.
[32] 姚粟, 王鹏辉, 白飞荣, 等.中国传统发酵食品用微生物菌种名单研究(第二版)[J].食品与发酵工业, 2022, 48(1):272-307. YAO S, WANG P H, BAI F R, et al.Research on the inventory of microbial food cultures in Chinese traditional fermented foods (2nd edition) [J].Food and Fermentation Industries, 2022, 48(1):272-307.