随着冷链技术在原料乳运输中的广泛应用,嗜热菌和嗜温菌生长受到抑制,而嗜冷菌在冷藏过程中继续生长,逐渐成为优势菌[1]。常见的嗜冷菌包括假单胞菌属,不动杆菌属(Acinetobacter),肠杆菌属(Enterobacter),芽孢杆菌属(Bacillus),沙雷氏菌属(Serratia),黄杆菌属(Chryseobacterium)等[2-3]。这些菌株在生长对数期会分泌耐热酶,该酶具有较强的热稳定性,即使经过主流加热工艺135 ℃处理,仍有20%~40%的残留[4]。其中,残留的耐热蛋白酶缓慢作用于乳蛋白,使UHT乳在货架期内出现蛋白水解、风味劣化等质量问题[5],给消费者健康和企业声誉造成不良影响。
高温处理可以杀死原料乳中的微生物从而保证乳产品的安全性,但原料乳中原始微生物种类和数量对UHT乳的质量有很大的影响[6]。假单胞菌是主要产蛋白酶嗜冷菌,在生长对数期分泌由aprX基因编码的金属蛋白酶,该酶在不同假单胞菌株中表达量也不同,即使同一属菌株由于分离源不同也有明显的差异性[7]。因此,明确不同牧场嗜冷菌污染分布及假单胞菌蛋白酶活力,对于提高UHT乳的质量,延长货架期,具有重要的指导意义。
然而,中国地区原料乳及牧场环境中嗜冷菌的多样性和蛋白水解活性的分析尚未得到广泛的研究。本研究的目的是调查嗜冷菌在中国不同地区分布情况,分析嗜冷菌产蛋白酶性能以及蛋白酶与是否携带aprX基因的相关性。这是一次大范围对中国地区原料乳及牧场环境中嗜冷菌多样性及蛋白水解活性进行研究。研究结果对中国地区牧场嗜冷菌污染防控及其耐热酶在产品中的残留抑制提供了参考价值。
1 材料与方法
1.1 材料与试剂
营养肉汤培养基、MPC琼脂培养基、蛋白胨盐溶液,北京奥博星生物技术有限责任公司;TAE(50×)缓冲液,北京酷来搏科技有限公司;蛋白酶K,美国默克公司;琼脂糖,北京广达恒益科技有限公司;GeneGreenI核酸染料、细菌基因组DNA提取试剂盒,天根生化科技(北京)有限公司;Trans2K® Plus Ⅱ DNA Marker,北京全式金生物技术股份有限公司;Taq PCR MasterMix,北京金百特生物技术有限公司;偶氮酪蛋白(azocasein),美国Sigma公司;所有分离用有机溶剂均为国产分析纯。
1.2 仪器与设备
全波长酶标仪,瑞士Tecan公司;台式高速离心机,美国Sigma公司;恒温培养箱,上海一恒科学仪器有限公司;微型旋涡振荡器,海门市其林贝尔仪器制造有限公司;pH计,法国梅特勒公司;核酸电泳仪,北京市六一仪器厂;凝胶成像仪,上海勤翔公司;PCR仪 ThermoFisher公司;立式压力蒸汽灭菌锅,UHT杀菌机,上海申安医疗器械厂;无菌试验台,哈尔滨市东联电子技术有限公司;水浴锅,宁波新芝科技有限公司;UHT杀菌机,北京西奥德数控设备有限公司。
1.3 实验方法
1.3.1 样本采集
从中国宁夏回族自治区、黑龙江省、河北省、广东省某规模化牧场,分别在夏季(2022年6月)、冬季(2021年12月)采集原料乳和环境样品8次。将50 mL离心管、采样袋于121 ℃灭菌20 min,TMR饲料、牛舍垫土、青储饲料各取500 g于无菌采样袋,前三把奶、正常乳、运奶车奶样、冷藏罐奶样在挤出30 min之内取50 mL至无菌离心管中,饮用水取50 mL至无菌离心管中,采集后在48 h内冷链运输到实验室进行嗜冷菌的分离。
1.3.2 样本处理
在超净台中将采集的样品用无菌蛋白胨盐溶液按10倍梯度连续稀释5次,不同浓度的样品分别均匀涂布在MPC琼脂培养基上,将培养皿倒置在6.5 ℃恒温培养箱中培养7~10 d[8]。
1.3.3 嗜冷菌的分离纯化
选取嗜冷菌生长比较均匀的平板,将大小形态相异的单个菌落用无菌接种针挑入营养肉汤液体培养基。分离的嗜冷菌于28 ℃恒温培养箱培养24 h,连续传代2次,将生长至对数期的嗜冷菌用25%(体积分数)的甘油保存在-80 ℃冰箱,用于嗜冷菌的鉴定和酶活力研究。
1.3.4 嗜冷菌的鉴定
所有细菌在28 ℃下,于营养肉汤培养基中培养24 h。根据细菌基因组DNA试剂盒说明书提取DNA,采用通用引物27F (5′-AGAGTTTGATCCTGGCTCAG-3′) 和1492R (5′-TACGGCTACCTTGTTACGACTT-3′),在反应体系(DNA模板1 μL;引物27F、1492R各1 μL;2×Taq PCR Master Mix 12.5 μL;dd H2O 9.5 μL),在95 ℃ 5 min预变性,35 Cycles(95 ℃ 30 s,55 ℃ 30 s,72 ℃ 90 s)进行变性、退火、延伸,72 ℃ 10 min进行再延伸,完成细菌的16S rRNA基因序列扩增[9]。扩增产物经1%琼脂糖凝胶电泳验证后,将有扩增条带的进行PCR产物的纯化,扩增产物送六合华大基因科技有限公司(北京,中国)完成测序分析。测序结果在MEGA软件完成拼接,完整的碱基序列提交在National Center for Biotechnology (NCBI)数据库进行序列BLAST比对,确定菌株的种名[10]。菌株的中文名称来源中国典型培养物保藏中心(http://cctcc.whu.edu.cn/portal/dictionary/index)。
1.3.5 假单胞菌蛋白酶活力及热稳定性评价
将1.3.3节中-80 ℃保存的假单胞菌接种到营养肉汤培养基中,28 ℃培养24 h进行活化。采用脱脂奶琼脂平板法,将活化后的菌株进行蛋白酶可视化定性检测[11],具体步骤如下:
(1)脱脂乳平板的配制:脱脂奶粉和琼脂粉分别按照100 g/L,15 g/L用去离子水定容,115 ℃灭菌10 min,倒平板备用。
(2)菌液的接种:将平板用无菌打孔器打孔,吸取200 μL菌液于孔中,将平板正置于28 ℃培养箱中24 h,观察是否有透明水解圈。
将有水解圈的假单胞菌采用偶酪蛋白法进行蛋白酶定量检测[12],具体步骤如下:
(1)取1 mL菌液样品、1 mL磷酸盐缓冲液(pH=7.4)、2 mL底物于10 mL离心管中充分混匀,置于37 ℃恒温培养箱下孵育2 h(空白对照:将样液替换为磷酸盐缓冲液pH=7.4)。
(2)孵育结束后加入4 mL的三氯乙酸(5%)溶液终止反应,室温下放置30 min。
(3)将终止反应液于10 000 r/min,4 ℃条件下离心5 min。
(4)取200 uL上清液于96孔板中,于345 nm处测定吸光度。若离心后上清液中具有悬浮物,可用0.22 um的水系膜进行过滤。
(5)酶活力计算如公式(1)所示:
酶活力/U=(OD 1-OD 2)×100
(1)
式中:OD 1为样品在345 nm处吸光度,OD 2为空白对照在345 nm处吸光度,其中一个酶活力单位(U)以345 nm下,每2 h增加0.01个吸光度值定义。
参照国内UHT乳主流工艺(135 ℃,5 s),计算在该处理条件下假单胞菌蛋白酶残留率,其中残留率计算如公式(2)所示:
残留率![]()
(2)
1.3.6 假单胞菌株aprX基因检测
采用引物aprX-F(5′-AAATCGATAGCTTCAGCCAT-3′)和aprX-R(5′-TTGAGGTTGATCTTCTGGTT-3′)对1.3.4节中保存的假单胞菌基因组进行aprX序列的扩增,PCR体系和循环参数参考1.3.4节,扩增产物经1%琼脂糖凝胶电泳验证是否携带耐热酶基因[13]。
1.3.7 生物信息学分析
采用Excel对文中所需的表格进行制作。使用MEGA软件对16S序列和aprX基因序列进行多序列比较[14]。把两端有差异性序列去除统一化,采用NJ法基于核酸序列进行进化树的绘制,最后将制作的进化树提交至iTOL v6(https://itol.embl.de/)在线软件进行物种标签及物种丰富度的修饰。文中的雷达图使用基因云在线软件(https://www.genescloud.cn/login)进行绘制和修饰[15]。
2 结果与分析
2.1 嗜冷菌的分离鉴定
从中国地区共分离到329株嗜冷菌,经16S rRNA鉴定分为21个属,76个种。其中分离菌株主要包括假单胞菌、嗜冷杆菌(Psychrobacter)、不动杆菌、沙雷氏菌、谷氨酸杆菌(Glutamicibacter)、微小杆菌、金黄杆菌、芽孢杆菌、拉恩氏菌(Rahnella)、葡萄球菌(Staphylococcus)、哺乳动物球菌(Mammaliicoccus)等菌属(图1)。其中假单胞菌189株占比57.45%,P.azotoformans占假单胞菌总数的26.46%, P.lactis占假单胞菌总数的13.76%, 此外还包括P.paralactis、P.gessardii、P.weihenstephanensis、P.fragi、P.fluorescens、P.marginalis、P.fildesensis等33种假单胞菌(图2)。
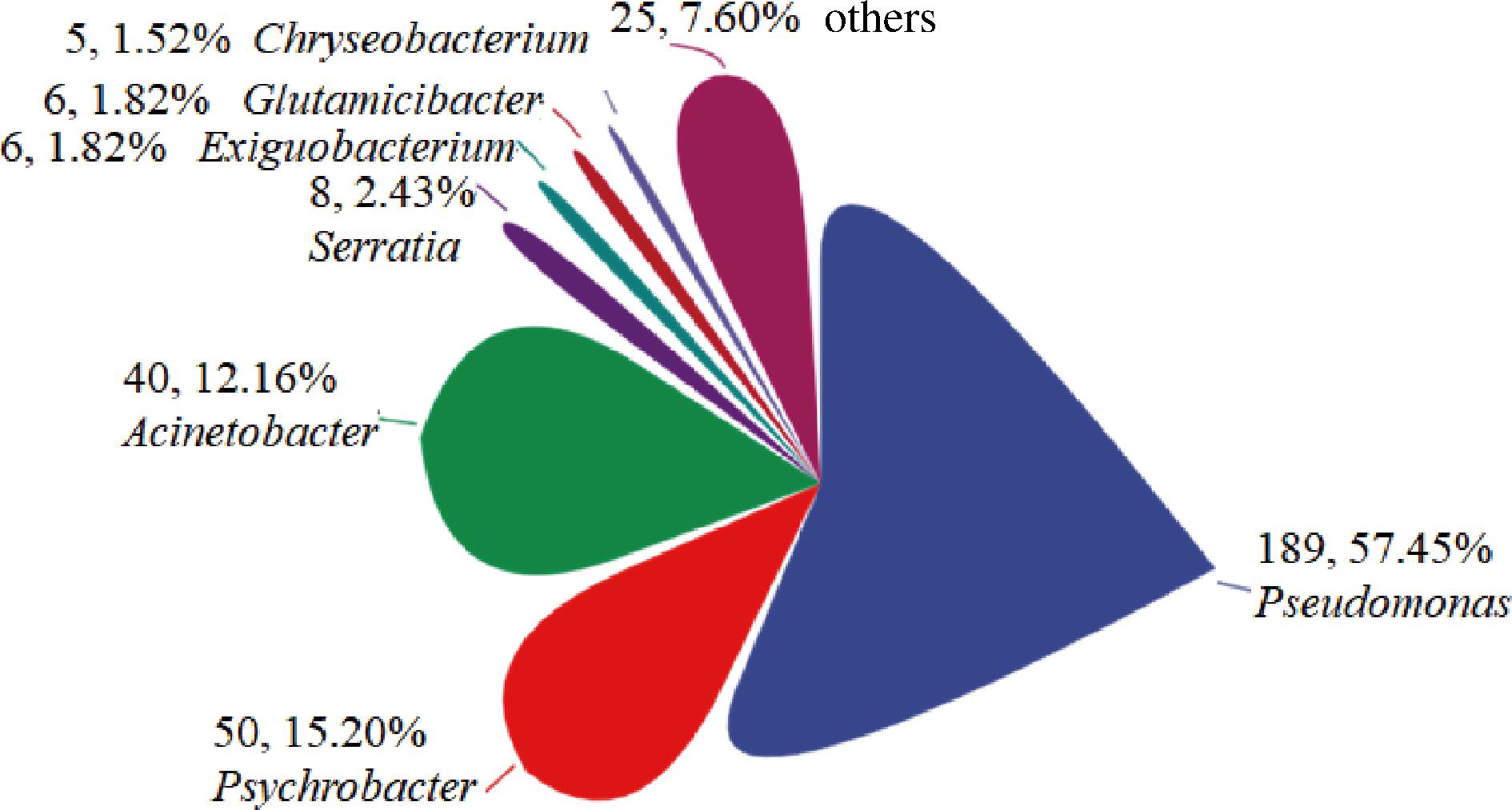
图1 嗜冷菌所占比例
Fig.1 Proportion of psychrophilic
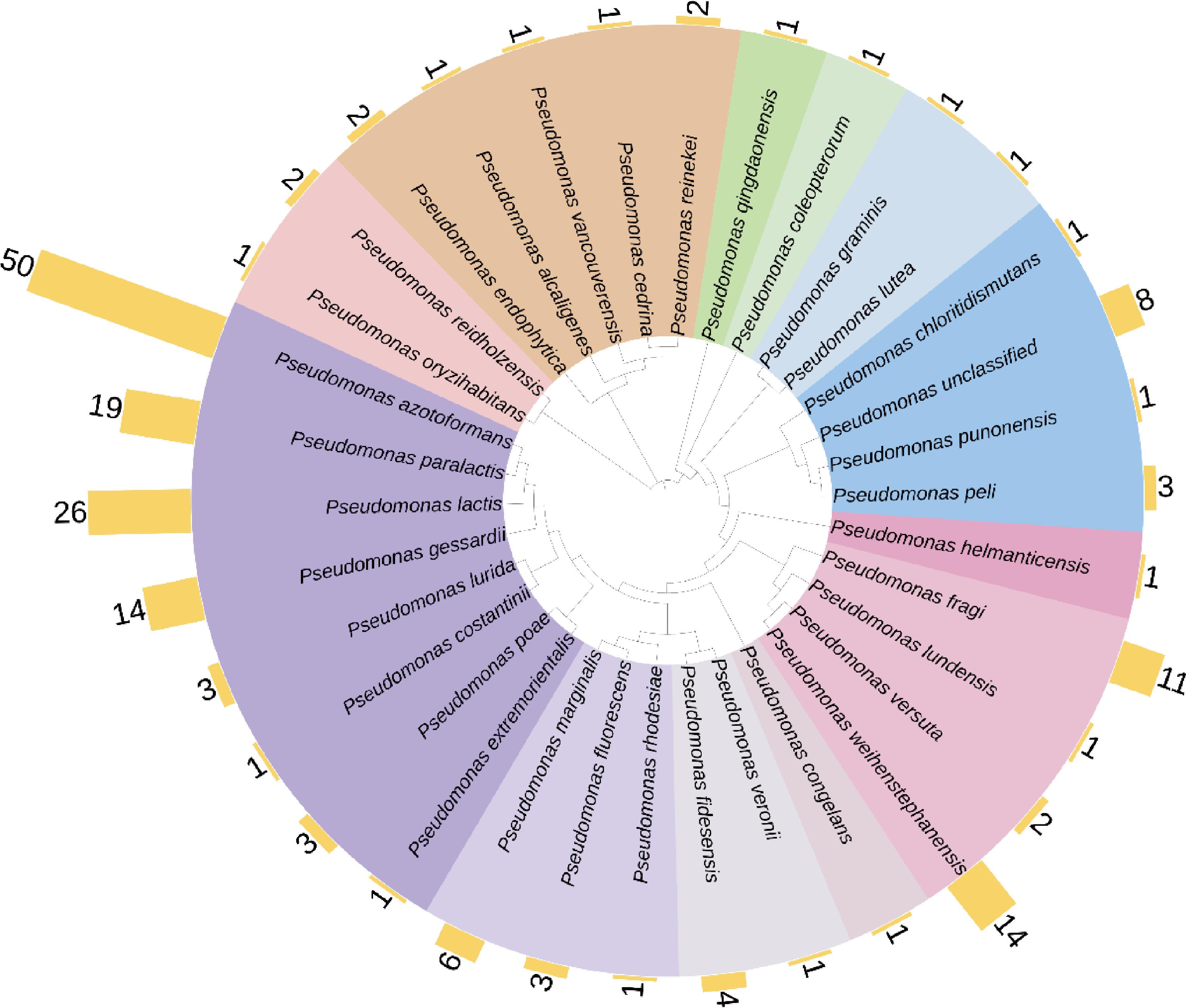
图2 基于16S rRNA假单胞菌的进化树
Fig.2 Pseudomonas evolutionary tree based on 16S rRNA
2.2 嗜冷菌的污染分布
采用雷达图显示嗜冷菌的污染分布,结果如图3所示。从季节上看,4个牧场夏季嗜冷菌丰富度是高于冬季的,可能原因是大部分嗜冷菌的最适生长温度在30 ℃左右,夏季更适合嗜冷菌的生长,导致嗜冷菌的丰富度更高。在嗜冷菌分布上,假单胞菌和不动杆菌分布最广,存在不同牧场的前三把奶、垫土、运奶车、冷藏罐、TMR饲料等各个环节中。前三把奶和垫土嗜冷菌种类最丰富,而青储和饮用水几乎不含有嗜冷菌。其次嗜冷菌种类在夏季和冬季有所差异,金黄杆菌属、索丝菌属(Brochothrix)、耶尔森氏菌属(Yersinia)、莱略特氏菌属(Lelliottia)、拉恩氏菌属(Rahnella)只在冬季牧场分离到,而微小杆菌属、肠球菌属(Enterococcus)、鞘氨醇杆菌属(Sphingobacterium)、节杆菌属(Arthrobacter)、马赛菌属(Massilia)、克吕沃尔氏菌属(Kluyvera)、泛菌属(Pantoea)、葡萄球菌属、哺乳动物球菌属、气单胞菌属(Aeromonas)和沙雷氏菌属更倾向于夏季。
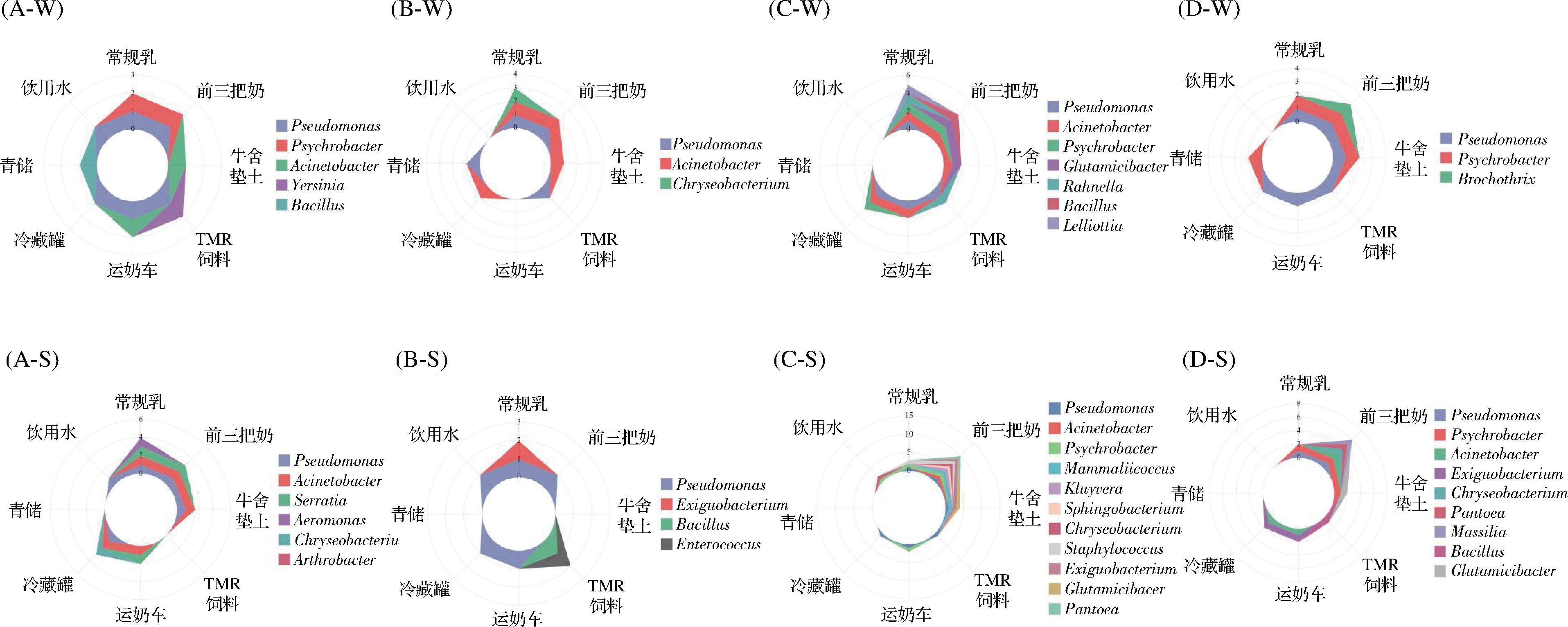
图3 嗜冷菌在不同环节的分布
Fig.3 Distribution of psychrophilic in different links
注:A、B、C、D分别代表宁夏牧场、广东牧场、黑龙江牧场、河北牧场;W、S代表冬季和夏季。
2.3 假单胞菌的蛋白酶活力及耐热性
在28 ℃下,经过脱脂乳平板初步筛选有61.38%的假单胞菌水解脱脂乳平板。在假单胞菌中,分离到的14株的P.gessardii,3株P.fluorescens,3株P.poae,1株P.extremorientalis,1株P.congelans,1株P.coleopterorum,1株P.chloritidismutans,1株P.cedrina在28 ℃下都表现出蛋白水解活性;P.lactis产蛋白酶比例为88.46%,P.paralactis产蛋白酶比例为84.21%,P.azotoformans产蛋白酶比例为84%,P.lurida产蛋白酶比例为66.67%,P.marginalis、P.reinekei和P.endophytica产蛋白酶比例为50%,P.fragi产蛋白酶比例为9.09%;而P.weihenstephanensis、P.fidesensis、P.peli、P.reidholzensis、P.versuta等15种假单胞菌在28 ℃下未产生脱脂乳水解圈(图4)。
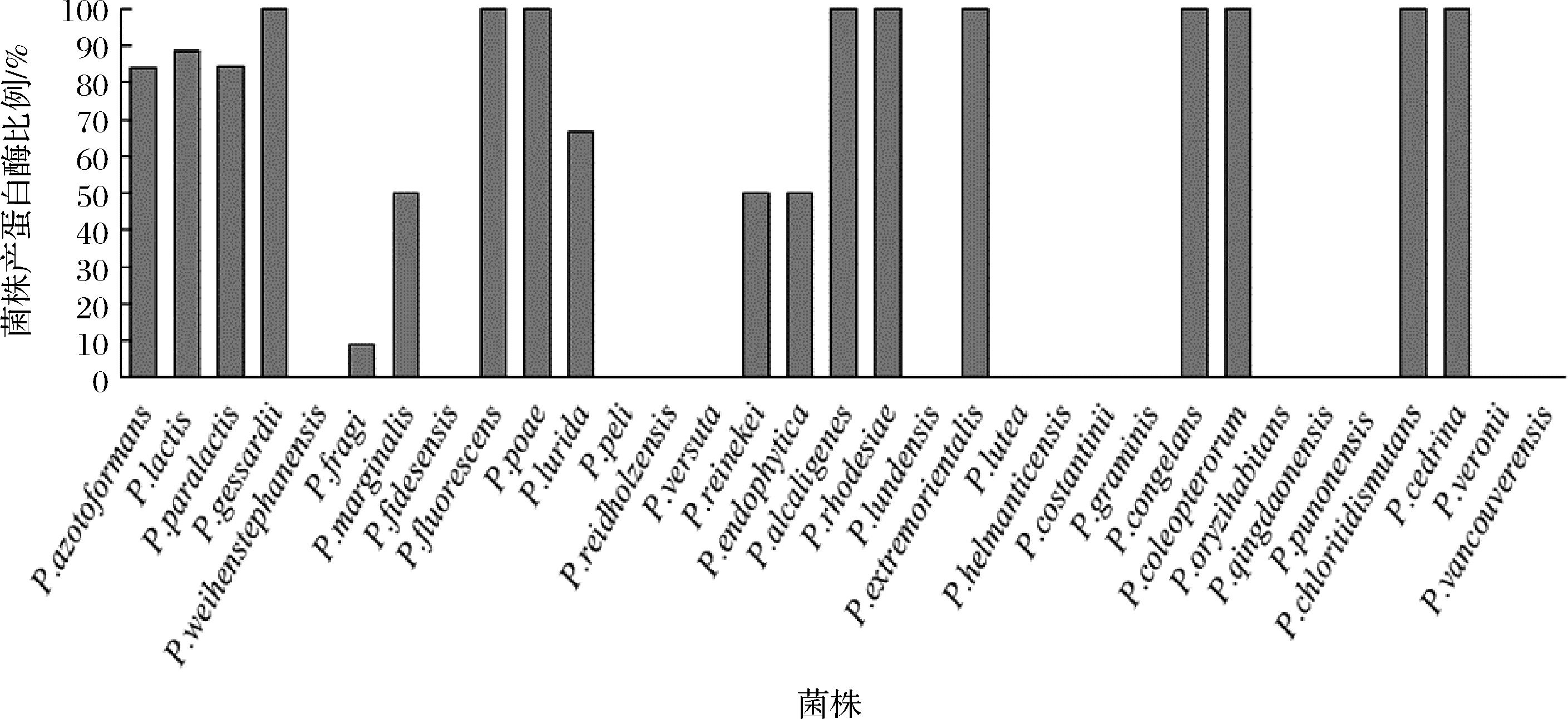
图4 假单胞菌株产蛋白酶比例
Fig.4 Proportion of protease production by Pseudomonas strains
从假单胞菌的蛋白酶水解度来看,63.79%的假单胞菌蛋白酶活力集中在0~10 U/mL,23.28%蛋白酶活力为10~20 U/mL,9.48%蛋白酶活力为20~30 U/mL,3.45%蛋白酶活力超过30 U/mL。假单胞菌分泌的蛋白酶经135 ℃处理5 s后,63.79%的菌株蛋白酶残留率为20%~60%,21.55%的菌株蛋白酶残留率高于60%,有5株P.azotoformans和4株P.gessardii蛋白酶残留率超过80%。此外假单胞的蛋白酶活力和酶残留率分布较分散,即使同一种假单胞菌在蛋白酶活力和酶残留率有较大的差别,对不同来源的嗜冷菌的蛋白酶水解度的研究对牧场管理具有非常重要的意义(图5)。
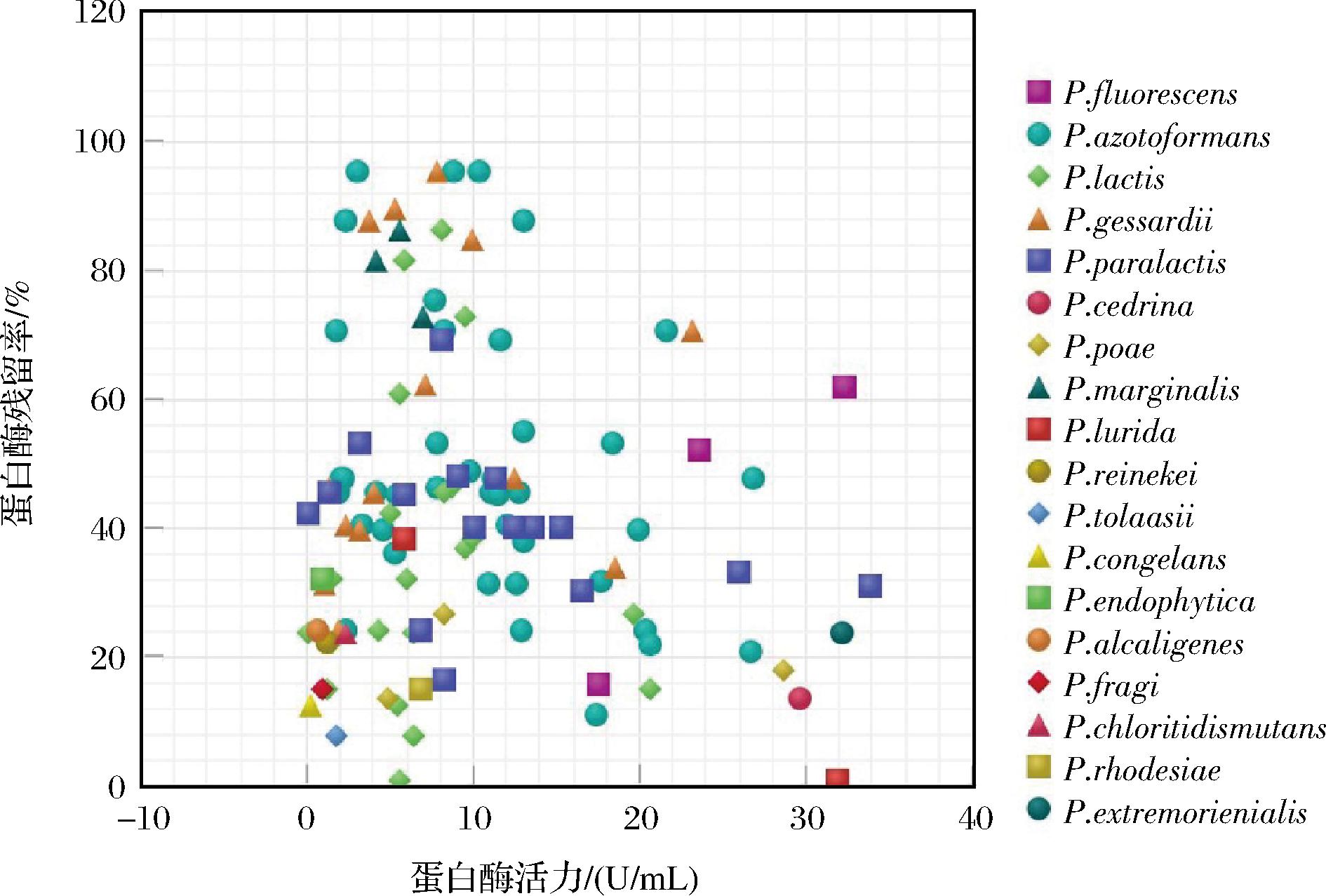
图5 假单胞菌蛋白酶活力及酶残留率
Fig.5 Protease activity and enzyme residue rate for Pseudomonas
注:蛋白酶残留率处理条件为135 ℃ 5 s。
2.4 假单胞菌的aprX基因位点检测结果
此外还对116株产蛋白酶菌株进行aprX基因的检测,发现107株蛋白酶扩增出约800 bp左右条带,说明该基因位点的存在与菌株是否产蛋白酶有较高(92.24%)的相关性。因此,可通过aprX基因位点对产蛋白酶嗜冷菌进行靶向检测,该基因位点是检测假单胞菌最合适的基因序列。
3 讨论
传统的微生物培养比其他研究方法具有诸多优势,是深入探索原料乳微生物资源不可缺少的技术手段,获得纯化的菌株是研究微生物的生长特性、产酶特性的基础。本研究通过传统培养方法,明确中国地区嗜冷菌的分布状况,分析假单胞菌的蛋白水解活性与aprX基因存在的相关性。
通过传统纯培养方法分离出329株嗜冷菌,通过16Sr RNA测序结果,分为21属76种,表明中国原料乳含有复杂的嗜冷细菌群落。在嗜冷菌属水平上,主要包括假单胞菌属、不动杆菌属、嗜冷杆菌属、沙雷氏菌属等。其中假单胞菌属占比为57.45%,主要包括P.azotoformans、P.lactis、P.paralactis、P.gessardii等33种假单胞菌。先前的一项研究发现,假单胞菌是原料乳中最常见的属(74.79%),主要包括25个种,其中最常见的是P.fragi (10.92%),P.lundensis(6.72%)和P.fluorescens (4.48%)[22],该研究结果由于分离源及分离时间不同在菌群丰度上与本研究存在微小的差异性。
从嗜冷菌在各个环节的污染分布来看,前三把奶和牛舍垫土的嗜冷菌种类最多,几乎可以分离到常规乳中所有种类的嗜冷菌,但青储饲料和饮用水几乎不含有嗜冷菌。在之前的研究中,也提出了原料乳在挤出时是无菌的,但原料乳会受到牛舍垫土、榨乳罩杯、头三把乳和贮罐的污染,与本研究是一致的[23-25]。此外发现假单胞菌属和不动杆菌属的分布最广,几乎存在所有环节,是原料乳中的最大污染菌株,对该菌株的控制可以很大程度上减少嗜冷菌的对乳制品的破坏。
耐热蛋白酶的存在是导致UHT乳变质的关键因素之一,本研究评价了假单胞菌的水解蛋白能力,并分析了蛋白酶活力与aprX基因存在的相关性。结果显示,在28 ℃下,61.38%的假单胞菌株都具有水解脱脂乳平板的能力。有研究表明,25 ℃时, 60%的假单胞菌株表现出水解乳琼脂平板,在7 ℃时为30%,在2 ℃时仍有10%水解乳琼脂平板[12],与本研究结果一致。在先前的相关研究中,分析了嗜冷菌中的酪蛋白酶谱,发现不同菌株中含有不同结构类型的蛋白酶(45~130 kDa),蛋白酶结构的多样性可能是菌株蛋白酶活力不同的原因[26]。此外有研究表明菌株产生分子质量为45~48 kDa的蛋白酶是最常见的耐热酶,文献中通常称为AprX,是由aprX基因所编码和表达[27-29]。因此本课题组对分离菌株的aprX基因进行了分析,发现产蛋白酶的嗜冷菌中有92.24%都携带aprX基因,aprX基因的存在与蛋白酶有较大的相关性在先前的研究中也被证实[13,30]。但是本研究中有部分菌株虽然携带aprX基因,但是并未水解乳琼脂板,可能是在28 ℃下,该基因未被表达,也有部分未检测到aprX基因的菌株,却有水解乳琼脂板的能力,可能是由于产生其他分子质量的蛋白酶,而不能产生分子质量为45~48 kDa的蛋白酶(AprX)。
国内外许多研究者对来自不同国家的原料乳样品中嗜冷菌的多样性和蛋白水解活性进行研究(表1)[16-21]。
表1 来自不同国家的原料乳样品中嗜冷菌多样性和蛋白水解活性
Table 1 Psychrophilic diversity and protein hydrolysis activity in raw milk samples from different countries
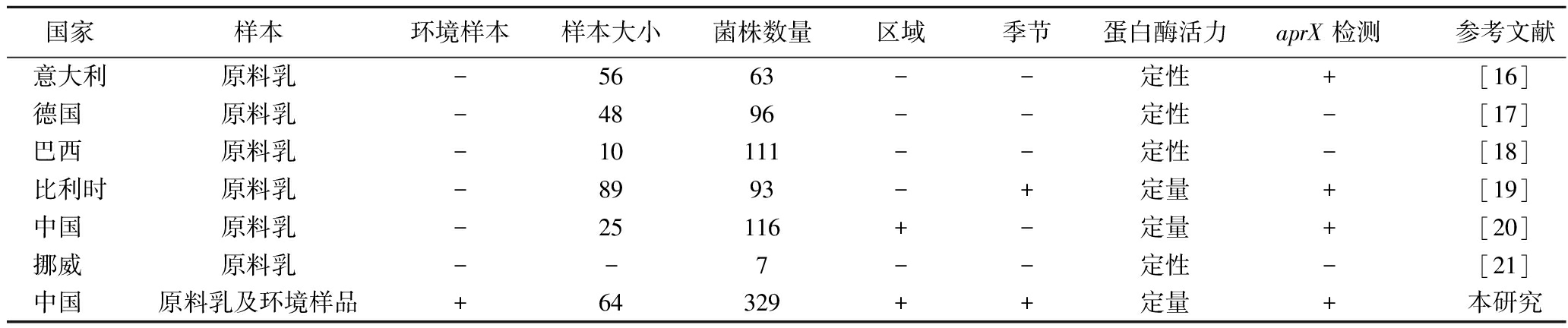
国家样本环境样本样本大小菌株数量区域季节蛋白酶活力aprX 检测参考文献意大利原料乳-5663--定性+[16]德国 原料乳-4896--定性-[17]巴西 原料乳-10111--定性-[18]比利时原料乳-8993-+定量+[19]中国 原料乳-25116+-定量+[20]挪威 原料乳--7--定性-[21]中国 原料乳及环境样品+64329++定量+本研究
注:“+”表示研究者已做相关研究;“-”表示研究者未涉及相关研究。
在本研究中,假单胞菌做为嗜冷菌中的优势菌,占总分离菌株的57.45%,几乎存在牧场环境中所有环节,有61.38%的菌株具有蛋白水解活性。此外不同嗜冷菌,即使是同一种菌株,由于分离源不同产蛋白酶能力存在较大差异。AprX作为嗜冷菌主要蛋白酶,它与aprX基因有较强的相关性,产蛋白酶的嗜冷菌中有92.24%都携带该基因。因此,应针对不同牧场采取不同的嗜冷菌防控措施,并且可通过aprX基因作为目标靶点对产耐热蛋白酶菌株进行研究。
[1] VITHANAGE N R, DISSANAYAKE M, BOLGE G, et al.Biodiversity of culturable psychrotrophic microbiota in raw milk attributable to refrigeration conditions, seasonality and their spoilage potential[J].International Dairy Journal, 2016, 57:80-90.
[2] PAULDAS K, EASOW J, NAIR S, et al.Microbial quality control of raw milk and isolation of spoilage causing microorganisms[J].Advances in Microbiology, 2022, 3:56-64.
[3] JIN S S, WANG Y Z, ZHAO X H.Cold-adaptive mechanism of psychrophilic bacteria in food and its application[J].Microbial Pathogenesis, 2022,169:105652.
[4] ZHANG S, LI H, ULUKO H, et al.Investigation of protease production by Pseudomonas fluorescens BJ-10 and degradation on milk proteins[J].Journal of Food Processing and Preservation, 2015, 39(6):2466-2472.
[5] ZHANG C Y, BIJL E, SVENSSON B, et al.The extracellular protease AprX from Pseudomonas and its spoilage potential for UHT milk:A review[J].Comprehensive Reviews in Food Science and Food Safety, 2019, 18(4):834-852.
[6] FOROUTAN A, GUO A C, VAZQUEZ-FRESNO R, et al.Chemical composition of commercial cow′s milk[J].Journal of Agricultural and Food Chemistry, 2019, 67(17):4897-4914.
[7] TEIDER P I, RIBEIRO J C, OSSUGUI E H, et al.Pseudomonas spp.and other psychrotrophic microorganisms in inspected and non-inspected Brazilian Minas Frescal cheese:Proteolytic, lipolytic and AprX production potential[J].Pesquisa Veterinária Brasileira, 2019, 39(10):807-815.
[8] DU B Y, MENG L, LIU H M, et al.Single molecule real-time sequencing and traditional cultivation techniques reveal complex community structures and regional variations of psychrotrophic bacteria in raw milk[J].Frontiers in Microbiology, 2022, 13:853263.
[9] LIANG L J, WANG P, ZHAO X M, et al.Single-molecule real-time sequencing reveals differences in bacterial diversity in raw milk in different regions and seasons in China[J].Journal of Dairy Science, 2022, 105(7):5669-5684.
[10] KUMAR S, TAMURA K, NEI M.MEGA:Molecular evolutionary genetics analysis software for microcomputers[J].Bioinformatics, 1994, 10(2):189-191.
[11] SCATAMBURLO T M, YAMAZI A K, CAVICCHIOLI V Q, et al.Spoilage potential of Pseudomonas species isolated from goat milk[J].Journal of Dairy Science, 2015, 98(2):759-764.
[12] MENG L, LIU H M, DONG L, et al.Identification and proteolytic activity quantification of Pseudomonas spp.isolated from different raw milks at storage temperatures[J].Journal of Dairy Science, 2018, 101(4):2897-2905.
[13] CALDERA L, FRANZETTI L, VAN COILLIE E, et al.Identification, enzymatic spoilage characterization and proteolytic activity quantification of Pseudomonas spp.isolated from different foods[J].Food Microbiology, 2016, 54:142-153.
[14] TAMURA K, DUDLEY J, NEI M, et al.MEGA4:molecular evolutionary genetics analysis (MEGA) software version 4.0[J].Molecular Biology and Evolution, 2007, 24(8):1596-1599.
[15] BECKER R.The New S Language [M].CRC Press, 2018.
[16] DECIMO M, MORANDI S, SILVETTI T, et al.Characterization of gram-negative psychrotrophic bacteria isolated from italian bulk tank milk[J].Journal of Food Science, 2014, 79(10):M2081-M2090.
[17] HAHNE J, ISELE D, BERNING J, et al.The contribution of fast growing, psychrotrophic microorganisms on biodiversity of refrigerated raw cow′s milk with high bacterial counts and their food spoilage potential[J].Food Microbiology, 2019, 79(JUN.):11-19.
[18] LONGHI R, CORREIA S S, BRUZAROSKI S R, et al.Pseudomonas fluorescens and Pseudomonas putida from refrigerated raw milk:Genetic diversity and lipoproteolytic activity[J].The Journal of Dairy Research, 2022, (89-1):86-89.
[19] MARCHAND S,HEYLEN K,MESSENS W, et al.Seasonal influence on heat-resistant proteolytic capacity of Pseudomonas lundensis and Pseudomonas fragi, predominant milk spoilers isolated from Belgian raw milk samples[J].Environmental Microbiology, 2009, 11(2):467-482.
[20] DU B Y, MENG L, LIU H M, et al.Diversity and proteolytic activity of Pseudomonas species isolated from raw cow milk samples across China[J].The Science of the Total Environment, 2022, 838(Pt 3):156382-156389.
[21] NARVHUS J A, NILSEN B A, EKKELUND O, TIDEMANN E M, et al.Isolates of Pseudomonas spp.from cold-stored raw milk show variation in proteolytic and lipolytic properties[J].International Dairy Journal, 2021, 123:105049.
[22] YANG X Y, GUO X J, LIU W P, et al.The complex community structures and seasonal variations of psychrotrophic bacteria in raw milk in Heilongjiang Province, China[J].LWT- Food Science and Technology, 2020, 134:110218-110229.
[23] 刘潇忆, 张彧, 陈历俊, 等.原料乳生产过程中微生物污染的来源追溯研究[J].中国乳品工业, 2013, 41(6):51-55;64. LIU X Y, ZHANG Y, CHEN L J, et al.Study on the microbial contaminations source traceability of raw milk processing.[J].China Dairy Industry, 2013, 41(6):51-55;64.
[24] BANYK J, VYLETELOV
J, VYLETELOV M.Determining the source of Bacillus cereus and Bacillus licheniformis isolated from raw milk, pasteurized milk and yoghurt[J].Letters in Applied Microbiology, 2009, 48(3):318-323.
M.Determining the source of Bacillus cereus and Bacillus licheniformis isolated from raw milk, pasteurized milk and yoghurt[J].Letters in Applied Microbiology, 2009, 48(3):318-323.
[25] DOYLEAB C J, GLEESONC D, O′TOOLEBD P W, et al.High-throughput sequencing highlights the significant influence of seasonal housing and teat preparation on the raw milk microbiota[J].Applied and Environmental Microbiology, 2016:AEM.02694-02709.
[26] ZAREI M, YOUSEFVAND A, MAKTABI S, et al.Identification, phylogenetic characterisation and proteolytic activity quantification of high biofilm-forming Pseudomonas fluorescens group bacterial strains isolated from cold raw milk[J].International Dairy Journal, 2020, 109:104787-104795.
[27] COLANTUONO A, D′INCECCO P, FORTINA M G, et al.Milk substrates influence proteolytic activity of Pseudomonas fluorescens strains[J].Food Control, 2020, 111:107063-107071.
[28] ZHANG D, LI S Q, PALMER J, et al.The relationship between numbers of Pseudomonas bacteria in milk used to manufacture UHT milk and the effect on product quality[J].International Dairy Journal, 2020, 105:104687.
[29] ZHANG D, PALMER J, TEH K H, et al.Milk fat influences proteolytic enzyme activity of dairy Pseudomonas species[J].International Journal of Food Microbiology, 2020, 320:108543.
[30] MENG L, ZHANG Y D, LIU H M, et al.Characterization of Pseudomonas spp.and associated proteolytic properties in raw milk stored at low temperatures[J].Frontiers in Microbiology, 2017, 8:2158.