在临床医学方面,次生代谢衍生药物已成为治疗感染性疾病的基本药物。其中,非核糖体多肽是新型抗菌剂的来源之一,它们的结构多样性使其具有许多生理活性,典型产品包括万古霉素、环孢菌素A和达托霉素等。非核糖体多肽的生产者大多是土壤微生物,如革兰氏阳性放线菌和杆菌的成员,但也有丝状真菌,最近海洋微生物也成为这类次级代谢物的新的丰富来源。已知负责合成非核糖体多肽的是一种非核糖体肽合成酶(nonribosomal peptide synthetase, NRPS),具有非常大的分子质量和多个模块单元,以重复的、装配线式的方式催化化学反应,产生次生多肽代谢物。NRPS的每个模块可以进一步划分为多个催化域或结构域[1]。由于NRPS模块的顺序主要反映了肽产物中氨基酸的序列,通过重组其组成模块和操纵其特异性来重新编程NRPS,为创造新的生物活性化合物提供了一条有希望的途径。
NRPS系统中模块的数量和顺序决定了形成的肽段的长度和序列,模块本身由保守结构域组成,每个保守结构域在游离氨基酸结合到多肽产物的循环中催化特定的反应步骤。一个典型的非核糖体肽合成酶模块通常含有腺苷化 (adenylation,A)结构域、缩合(condensation,C)结构域和硫酯化(thiolation,T)结构域。终止模块通常为硫酯酶(thioesterase,TE)结构域,负责终止上载氨基酸并且水解释放或者环化多肽链。腺苷化结构域按其选择性识别并激活氨基酸生成氨基酰-AMP,随后结合到硫酯化结构域上,缩合结构域将上游多肽链和下游受体底物缩合生成肽键。该过程按照NRPS模块排列顺序,特异性上载氨基酸使肽链延长,最终由硫酯酶结构域释放链状或环状多肽。非核糖体肽合成酶还含有多种修饰结构域、包括甲基化(methylation,MT)结构域、缩合环化结构域(cyclization,Cy)、差向异构化(epimerization,E)结构域和氧化(oxidation,Ox)结构域等。在大多数真菌中,一个产生目标产物的NRPS既可以是一条多肽链,也可以由几个相互作用的亚基(每个亚基也称为NRPS)组成[2](图1)。
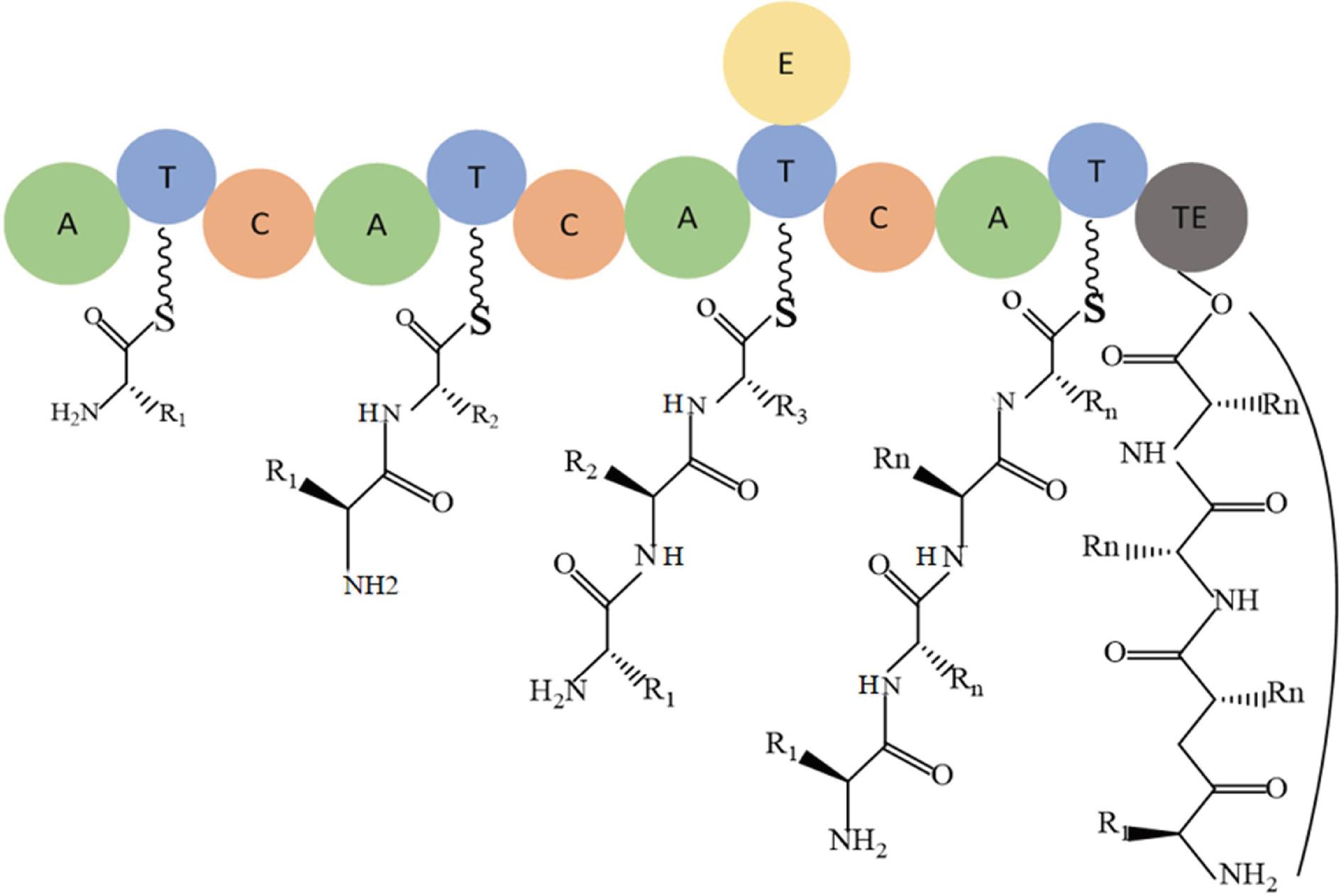
图1 非核糖体肽合成酶组装合成机制
Fig.1 The assembly-line mechanism of NRPS
根据其生物合成逻辑,已知的NRPS系统可分为三类:线性(A型)、迭代(B型)和非线性(C型)[3](图2)。线性NRPS模块的数量与多肽产物中加入的氨基酸单体数量相对应,产物的氨基酸序列与模块上载氨基酸特异性一致[4]。迭代NRPS在单个产物的组装中重复使用它的模块或结构域,这种策略被用来建立由重复的小序列组成的肽链[5]。非线性NRPS模块排列则并不按照熟知的顺序排列,通常伴随着一些不同寻常的内部环化结构域或其他修饰结构域[6]。
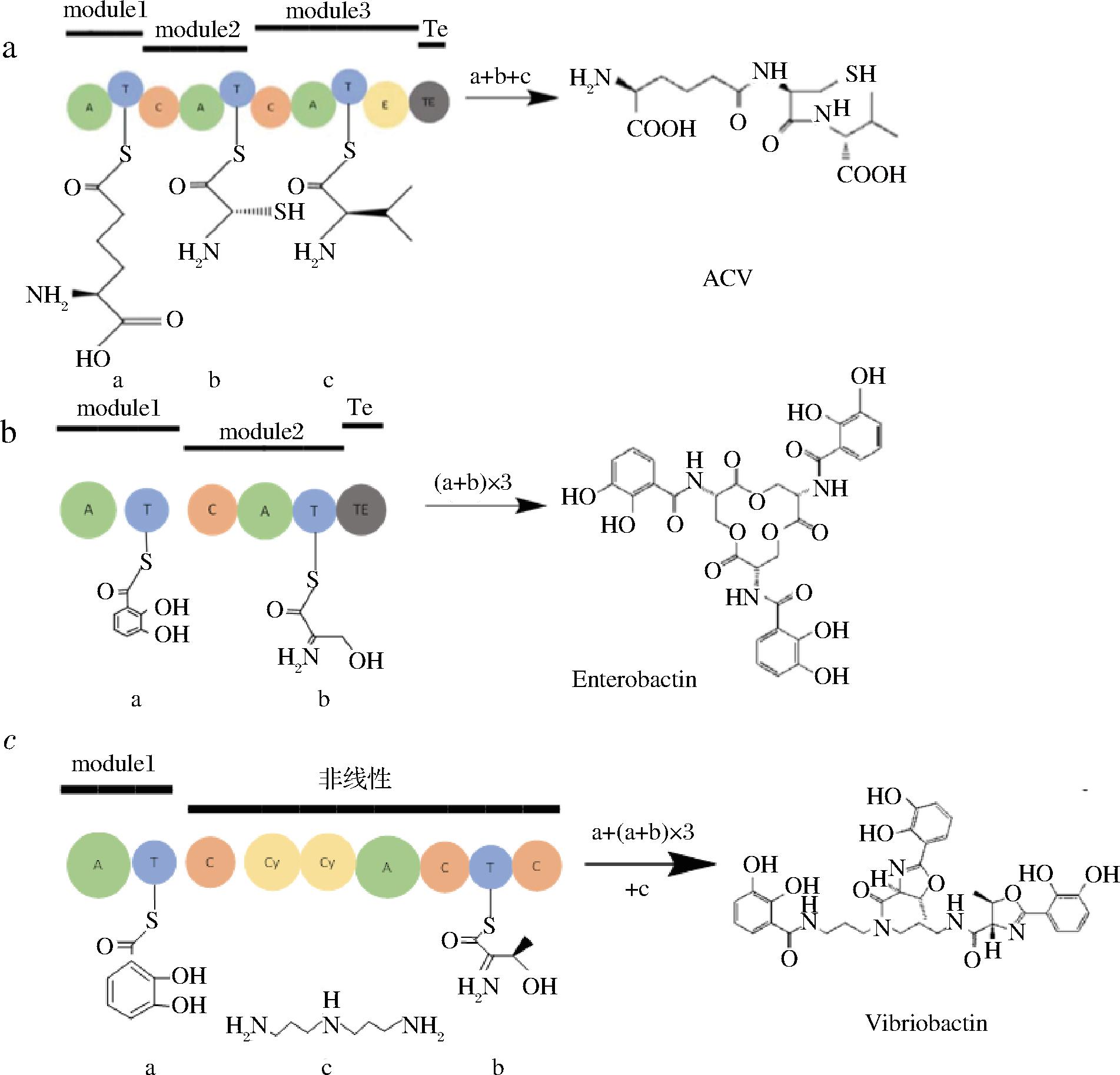
a-线性(A型)NRPS系统;b-迭代(B型)NRPS系统; c-非线性(C型)NRPS系统
图2 三种NRPS系统
Fig.2 Three NRPS systems
1 非核糖体肽合成酶结构域
1.1 腺苷化(adenylation, A)结构域
NRPS模块的腺苷化(A)结构域能特异性识别氨基酸,并将其激活。A结构域高度保守,长度约为500~600个氨基酸残基,负责氨基酸底物的选择和活化,如图3所示,可以将其分为一个大的“核心”结构域(Acore)和一个小的“亚”结构域(Asub)[7],是产物多元化的研究热点。A结构域催化两步反应:在ATP的参与下催化底物氨基酸腺苷化并将其激活,然后将激活的底物转移到硫酯化(thiolation,T)结构域上。
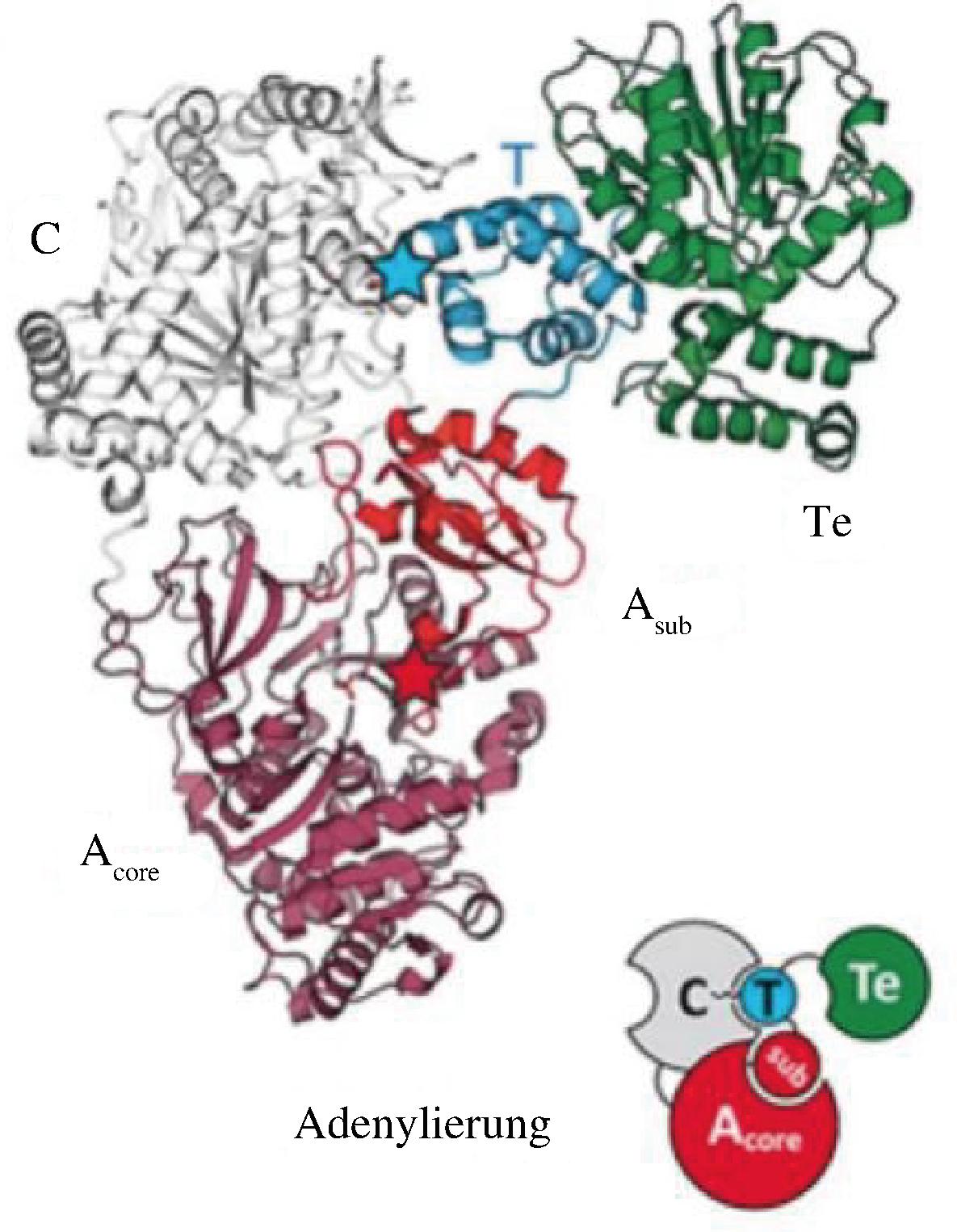
图3 Adenylierung单个NRPS模块立体结构
Fig.3 Stereoscopic structure of Adenylierung single NRPS module
A结构域是NRPS特异选择性上载氨基酸的关键部位,因此成为研究热点。目前科研人员已经开发了许多基于序列相似性来预测腺苷化结构域特异性的方法,BAUNACH等[8]在DNA水平上主要针对A结构域进行分析,比较了序列之间每个位点的平均核苷酸差异数,发现A结构域重组似乎有助于实现产物多样化,在更复杂的重组中,多重重组事件促进了NRPS基因的多样化。有学者将能够合成假单胞铁载体(pyoverdine)的NRPS中原11位特异性为Thr的C-A模块替换为特异性为Lys的C-A模块,产物生成了11位为赖氨酸的假单胞铁载体,因此发现了C-A模块的替换可以改变上载氨基酸的特异性;实验人员发现其NRPS第8个模块(Pa8)和第11个模块(Pa11)缩合(condensation,C)结构域氨基酸序列几乎是平行的(附图1-a,https://doi.org/10.13995/j.cnki.11-1802/ts.035832),随后将C结构域中3个区域进行重组,产生8种可能的组合(附图1-b),然后将每个重组的C结构域引入到pvdD基因结构体中,对8种组合产物进行进化分析,发现除了重组的C结构域KKT外,包含Pa11 3区域的C结构域与Pa11 A结构域的关系更为活跃,包含Pa8 3区域的C结构域与Pa8 A结构域的关系更为活跃,说明区域3是与A结构域连接的关键,为了缩小关键底物的范围,实验人员将Thr特异性模块Pa11 C域内的近端残基簇用Pa8中相应的残基取代,C结构域的修改对pyoverdine的生产几乎没有影响(附图1-c,https://doi.org/10.13995/j.cnki.11-1802/ts.035832),然而,将Pa11连接区域改变为Pa8连接区域,发现原生的PvdD C结构域与Pa8的A结构域一起有效地发挥作用,这个结果表明非核糖体多肽差异主要是由A“核心”结构域或“亚”结构域的重组驱动的,而非C结构域[9]。这展示了直接和自然进化研究的证据,表明新的非核糖体多肽可以仅通过替换A结构域生成。
因此,理论上通过预测NRPS中所有A结构域的底物特异性,可以确定NRPS多肽产物的所有氨基酸构建顺序。在具体的实验操作中,有实验人员通过对A结构域进行基因突变,或将A结构域进行交换成功改变了A结构域特异性[10-11]。LEE等[12]对NRPS的A结构域进行了虚拟筛选,结果表明,A结构域的一个关键的保守芳香残基的构象对于底物富集有显著影响。当前评估NRPS中A结构域底物特异性的标准方法是ATP-[32P]PPi交换实验,但是由于其操作麻烦,HARA等[13]创建了一种新的检测方法比ATP-[32P]PPi交换实验更加方便易操作,该方法利用氨酰-AMP(aminoacyl-AMP)与羟胺(hydroxylamine)形成羟肟酸(hydroxamate),在铁离子存在情况下,羟肟酸和铁离子会形成亮紫色复合物,以此方便进行观察。
研究人员通过在NRPS腺苷化结构域的结合袋中引入单个或联合点突变,可以将非天然氨基酸掺入非核糖体多肽中,或使所需产品成为改造后NRPS系统的主要或唯一产品。有文献报道了第二个A结构域被设计用于引入含氮氨基酸衍生物的例子。在该研究中,A结构域具有同时识别2种不同的氨基酸Arg和Tyr的能力,推测3个位点(E204、S243、A307)的氨基酸突变可导致A结构域对Tyr特异性发生变化,试验中,在307位的19个点突变中,只有4个突变体是有活性的;对243位突变,在19个含有大量非极性侧链氨基酸的突变中产生了15个具有活性的突变,且其A结构域再次选择Tyr而不是Arg[14]。因此,许多研究证据表明,自然界中发现的大量非核糖体多肽生物合成途径是通过点突变、遗传复制、删除和插入事件进化而来的。
通常研究NRPS的A结构域都是在微生物体内进行,但是最近有文献报道,可以通过非核糖体肽合成酶的A结构域可以在体外合成二肽[15]。此外,在一些情况下A结构域可以上载D型氨基酸。有研究人员实验发现以D型氨基酸作为底物,在体外合成二肽时,A结构域也可以顺利上载D型氨基酸[16]。研究人员还发现多数情况下,异源表达A结构域并在胞外使用A结构域合成二肽时,会出现选择性上载结构相似的不同氨基酸的情况,利用这一发现也成功使用A结构域在体外合成了阿斯巴甜[17]。
1.2 T结构域
T结构域在NRPS的功能中起着核心作用,不仅与A结构域相互作用,还与肽键形成、肽修饰或肽释放中涉及的其他催化结构域相互作用。在一个完整的NRPS体系中,T结构域将上游A结构域上载的氨基酸残基或多肽链转运到下游C结构域上,也可以将氨基酸或多肽链呈现给内部修饰酶,如卤素酶、转移酶或单加氧酶[18]。CALCOTT等[19]为研究重组假单胞铁载体非核糖体肽合成酶的T结构域可移植性,如附图2(https://doi.org/10.13995/j.cnki.11-1802/ts.035832)所示,将18例不同T结构域移植到各自C结构域的上游,其中6例的T结构域位于C结构域的上游(记为TC),4例T结构域位于E结构域的上游(记为TE),4例T结构域位于TE结构域上游(记为TTE),4例T结构域位于2个亚型之间(记为TCT)。将18种替换后的菌株在平板上培养,观察荧光可知引入TC的假单胞铁载体的产量与野生型几乎没有差别,其余3种替换均各有一株没有产生荧光;并且通过MALDI-TOF质谱确认了每个菌株产生的假单胞铁载体的同一性。因此可知T结构域通常可以有效地与非相邻的C结构域相互作用。但此前也有其他实验证明,在C域上游的T域,如果置于E域或TE域的上游,通常不能正常工作[20-21]。因此T-C结构域的接头处和T-TE结构域接头处值得深入探究。例如,有研究人员扩增编码Plipastatin合成酶的第7 T结构域、第8 T结构域(含有天然的T-C接头)与TE结构域重组,研究发现产物均与Plipastatin无关,但当保留T-TE的连接部分进行模块拼接时,产物中检测出活性物质,因此在实验中进行模块拼接时,T-TE的连接部分必须保持完整[22]。综上,在T结构域与其他结构域接口处尚需深入研究。
1.3 C结构域
C结构域通常是NRPS模块中的第一个结构域,将供体酰基链与受体氨基酸缩合,起着肽链延伸的作用,而近期有报道发现C结构域可以在NRPS合成多肽时催化内部环化[23]。因此除了少数例外,NRPS的启动模块通常缺乏C结构域。目前在C结构域中发现一段保守的HHXXXDG蛋白质序列[24],对于C结构域的功能研究意义重大。在这个保守基序中第2个氨基酸被认为是催化反应的关键[25]。有实验佐证了这一观点,SAMEL等[26]将Tyrocidine NRPS模块TycC中第5个PCP-C结构域的HHXXXDG序列里第2个氨基酸进行突变,分别突变为HAXXXDG和HVXXXDG,在相同的培养条件下,这2个PCP-C突变体既没有出现肽环化现象,也没有出现水解现象,然后对于该基序的第一个氨基酸进行突变,突变为AHXXXDG却显示出了28%的野生型活性,因此HHxxxDG核心基序中第2个组氨酸(H224)的突变会导致完全失活。最近,在对于NRPS的C结构域活性位点分析方面也取得了成功,发现了在C结构域中2个关键突变点和控制酰基链长度的关键部位,它们改变了底物对脂肪酰基底物的选择性[26-27]。
虽然含有底物结合位点的A结构域是NRPS的主要特异性决定因素,但C结构域也可以表现出特异性,因此在考虑模块重组时,需要同时考虑C、A结构域和C-A连接的完整,BOZHÜYÜK等[28]对于NRPS模块组装形式提出了一个概念:exchange units(XUs),使用这个XU概念(A-T-C或A-T-C/E)得到了一个对NRPS的研究新方法。他们对合成Ambactin、Kolossin、Xenoamicin和GameXPeptide的非核糖体肽合成酶分别进行重新组装,发现当以XU为单位进行完整替换的话,可以产生目的产物并且达到野生型产量的50%及以上。但值得注意的是,在所有的替换中发现替换后的产量与原始相比均会有所下降,而且替换的模块越多产量越低。XU概念还具有一定的使用限制性:在模块替换时必须保证替换模块的氨基酸特异性必须与原模块的氨基酸特异性保持一致,例如,原模块氨基酸特异性为赖氨酸时,替换模块的氨基酸特异性也必须是赖氨酸。
随后,BOZHÜYÜK等[29]在上述实验的基础上又提出了一个新的概念:exchange unit condensation domain (XUC)。研究人员在实验中将C-A或C-A-T作为一个整体进行模块替换时发现了一个新的融合位点,根据实验中拼接的C结构域序列比对,发现HHXXXDG序列,实验中假设该基序附近的4个氨基酸(Gln267-ala270)的Gln267和Ser268可能是一个理想的融合位点,以此位点进行C结构域的融合得以实现多肽的高效生产、甚至建立肽库。此外,研究人员将来自于Photorhabdus luminescens TT01NRPS亚基GxpS的XUC1和亚基XtpS的XUC1进行替换后发现原始GxpS的产物没有出现,而出现了其他的新产物,以此证明XUC概念还可以改变产物特异性。与XU概念相比,XUC概念在不同属的模块之间是不兼容的,即必须来自同一属的XUC模块进行组合。例如文中的GxpS来自于Photorhabdus luminescens TT01,其替换模块均来自于Bacillus 或仅仅来自Photorhabdus/Xenorhabdus。因此,根据XU和XUC概念,实验中可以有效进行模块的同源重组,产生特异的非核糖体多肽。这2个实验也为后续NRPS研究提供重要参考,通过模块拼接产生新的产物,可能会产生对人类健康有益的多肽产品。
1.4 硫酯酶(thioesterase, TE)结构域
TE结构域处于装配过程的末端,负责催化NRPS释放完整的肽链。在一些情况下,硫酯酶结构域也可以作为一个大的环化酶,是这类天然产物实现结构多样性和生物活性的重要手段。在NRPS流水线上,TE结构域被分成两类:TE I和TE Ⅱ,其中I型TE结构域通常是最后一个NRPS模块的最后一个结构域,而Ⅱ型TE结构域是独立的酶,识别错误装载[18]。在TE Ⅱ结构域中存在着一个保守氨基酸序列:GHSXG,有效保证TE Ⅱ结构域具有生物活性[30-32]。短杆菌肽S(GS)的生物合成簇GrsA的A结构域可以上载L-Phe和D-Phe,T结构域上L-Phe和D-Phe均存在,而GrsB1的C结构域对D-Phe具有立体选择性。GrsA中的E结构域可以将A结构域上载的L-Phe异构化为D-Phe,因此当GrsA的E结构域被灭活、L-Phe在T域累积时,GrsB1 的C结构域这种立体选择性阻碍了二肽的形成。
多年来,科学家们对于TE结构域的重组能力进行了深入的研究。SCHWARZER等[33]将杂合NRPS(由短杆菌素NRPS亚基TycA和ProCAT组成)与来源不同且催化终止模式不同的6种TE结构域融合组成双模型重组NRPS,并检测TE结构域在产物释放方面的活性,发现异源NRPS中的TE结构域可用于构建杂合NRPS。TE结构域固有的特异性和区域选择性可能导致其他有助于产生结构多样性的终止模式,并且发现重组来源不同的TE结构域,其活性与原生系统的活性基本一致。可见TE结构域重组可以比较顺利地进行。
GAO等[22]在实验中发现Plipastatin合成酶TE结构域的缺失导致酶的合成被截断,不能产生相应的肽;随后,将Plipastatin合成酶的linker-TE结构域前移,产物中不仅检测到了预期的线性多肽,而且还检测到了线性和环状八肽,并评估这种变化对体内截短的活性类似物产生的影响,发现只有使用完整linker-TE(即天然的T-TE连接子)与之前的模块结合,才能诱导TE结构域催化杂化肽合成酶中成熟的线性多肽的水解释放,并且发现TE结构域原本的功能可能在重组后产生新的变化,例如:原本释放环化产物,在重组后释放线性产物。所以在重组NRPS的时候,T-TE结构域连接处保持完整对于重组NRPS具有重要意义。
2 NRPS改造策略
根据序列分析研究,可知NRPS机制的进化是由常规遗传和水平基因转移共同驱动的。细菌中NRPS基因簇的出现频率更高,它们多见于细菌中的变形菌门、放线菌门、厚壁菌门、蓝藻门和真菌中的子囊菌门[34]。这些NRPS基因簇大多得到了此前未知的最终产物,这凸显了基因组挖掘在识别次级代谢产物生物合成的新遗传机制方面的威力。对NRPS进行了多年的研究后,科学家们也开始尝试研究NRPS整体的合成机制,而不仅仅研究某一个结构域的表现。然而,NRPS复杂的蛋白与蛋白质之间的相互作用给工程设计带来了挑战,因此NRPS系统的改造生产新的衍生物或天然产物,不仅需要有效的修饰催化结构域[28, 35],可能还需要考虑模块与模块之间的相互作用。这些研究通常集中于利用NRPS的模块重组产生新的产物,在模块连接处进行处理,增加或减少模块研究对于产物的影响等。
2.1 NRPS模块替换研究
NRPS模块结构本身和这些重复功能单元中的结构域排列指向了一个产生新脂肽的策略,即如何通过模块或结构域替换来设计现有的NRPS来生产改变的肽,或者如何设计全新的酶系统来合成新的肽。通过与完整模块交换来创建混合NRPS需要在最保守的域序列中选择融合点,因为这些融合点在模块和模块之间高度相似。根据这个思路,研究人员等研究了土曲霉(Aspergillus terreus)几个类似的NRPS模块(ApvA、MelA、BtyA、AtrA、PgnA、AstA、AtqA),其模块组成均为A-T-TE,然后将这些模块中的A结构域,T结构域和TE结构域按照A-T-TE顺序重组,比如将ApvA的A-T和AstA的TE结构域重组,AstA的A结构域和ApvA的T-TE重组,共获得了24种构造,转化进酵母培养表达后,产生了10种活性产物[36]。科研人员替换A54145的E结构域也导致了新的类似物的形成,说明总的模块替换(C-A-T-E)是可行的,但同时这一改变导致产品的产量明显低于原生型,所以保持模块与模块之间形成的连接区域的完整性对产物活性是至关重要的[37]。有实验人员在铜绿假单胞菌体内生产新型铁载体(pyoverdine)衍生物时,仅仅通过替换A结构域或C结构域,用这种结构域替换来产生新的产物[38]。肠杆菌素(enterobactin)和弧菌素(vibriobactin)是由NRPS合成的非核糖体多肽。大肠杆菌中合成肠杆菌素的合成关键模块为entA、entB、entC、entD、entE,霍乱弧菌(Vibrio cholerae)中合成弧菌素的合成关键模块为vibF和vibH。而普城沙雷菌(Serratia plymuthica)V4中用于产生塞拉提奥切林(serratiochelin)的合成关键模块schA、schB、schC、schE,schG与entABCDE具有同源性,schF1F2F3与vibF具有同源性,schH与vibH同源,实验人员将能够合成serratiochelin的schABCEGF1F2F3H克隆到pDSW204质粒中,发现在大肠杆菌中无法表达,为解决这一问题,实验人员利用大肠杆菌和霍乱弧菌中与S.plymuthica V4同源的基因替换产生serratiochelin的基因(entA替换schA,entB替换schB,以此规律一一替换),构建了合成serratiochelin的替代途径,纯化后的上清液中检测到了serratiochelin。在这个路径中,同源基因替换生物合成基因,从而成功地实现了异源NRP的表达[39]。
在模块替换实验中,可以通过模块排列预测产物的氨基酸序列,从而预测产物性能,然而工程天然产物途径往往会产生不佳的结果,部分原因是计算模型预测和实际分子表达功能的途径不能完美对应匹配上。在实验操作中,完全从头设计新NRPS和异源表达培养条件都需要精确地考量。多数情况下,试图改变条件以产生新产物或异源表达条件不够精准都会导致生产完全停止。
2.2 NRPS模块拼接研究
由于非核糖体多肽的氨基酸序列受单个NRPS模块的顺序控制,改变多肽序列的最有效的策略是替换或改变这些NRPS多聚体结构。这种方法遇到的一个问题:即使对模块结构做很小的改变,也会影响其蛋白质的折叠和蛋白质之间的相互作用。直接的模块替换会干扰这些模块之间的连接,阻止域/模块的关联,会导致其失活。因此,模块与模块之间如何拼接显得至关重要。
NRPS亚基之间的非共价相互作用是由特异的N末端和C末端对接结构域(docking domains,DDS)所介导的(附图3-a,https://doi.org/10.13995/j.cnki.11-1802/ts.035832),在合成短杆菌素(tyrocidin)和表面活性剂(surfactin)的NRPS系统中提供肽基的NRPS的C端记为CDD(约25个氨基酸),接受肽基的NRPS的N端记为NDD(约14个氨基酸)[40]。实验人员从Xenorhabdus stockiae KJ12.1中提取横纹肌肽/异质肽样肽NRPS(Kj12A、Kj12B、Kj12C),对NRPS的Kj12A、Kj12B、Kj12C中的3个NDD和2个CDD进行了研究。通过异核单量子相关(heteronuclear singular quantum correlation,HSQC)实验和等温滴定法(isothermal titration colarimetry,ITC)实验滴定发现3个NDD均与CDD相互作用,CDD多肽在滴定逐渐添加的过程中出现不同程度的化学位移和峰增宽现象,其中β1和β2被认为是NDD和CDD对接结构域上的关键氨基酸序列(附图3-b,附图3-c,https://doi.org/10.13995/j.cnki.11-1802/ts.035832),NDD和CDD对接结构域的形成依赖两分子间β1和β2的折叠;实验人员还重组了融合到T域上的Kj12BCDD 和全长Kj12C模块;ITC实验表明,Kj12B T域的存在并不影响Kj12B CDD对Kj12C NDD的亲和力,因此可以通过调节模块两分子之间DDs的形成来影响对接结构域之间的相互作用,进而影响NRPS模块之间的相互作用[36]。研究表明,CDD和NDD通过它们之间的DDs或通信中介域(communication-mediating,COM)进行配对[40]。实验人员通过比较Tyrocidin A和Surfactinlike NRPS的COM结构域,在体外构建了一个“通用COM系统”,使得不同生物合成系统之间的酶相互作用,促进了不同肽的合成[41-42]。对CDD和NDD上关键氨基酸进行突变,可发现产物出现不同的多肽,说明这些DDs突变导致上载氨基酸特异性发生了改变,即蛋白质对底物亲和力改变[36]。除了模块之间的CDD和NDD被报道外,E结构域到C结构域,T结构域到环化(cyclisation,Cyc)结构域的模块域接口也已经被发现[37-38]。
多亚基非核糖体肽合成酶(NRPS)的相互作用是由对接域介导的,以确保亚基与亚基之间正确相互作用。KEGLER等[43]具体研究了嗜线虫致病杆菌(X.bovienii.)的xefoampeptide(XFP)NRPS合成簇XfpS,将其分为XfpS1、XfpS2、XfpS3亚基。他们在XfpS1的E结构域和XfpS2的C结构域中间插入来自Taxlllaik的NRPS的DD对,在XfpS2的T结构域和XfpS3的C结构域中间插入同源基因簇paxABC的DD对,或同时插入2组DD对,将其终产物产量与野生型比较,发现通过DD对连接的基因簇均有活性。但是当进行缺少CDD、缺少NDD和同时缺少CDD和NDD的3组对照实验时,在E结构域和C结构域之间:单独缺少CDD时产量变化不大,但是缺少NDD和同时缺少CDD和NDD时产量急剧下降;在T结构域和C结构域之间:缺少CDD导致DD对功能受损,从而使产量下降;在同时插入2组DD对时产物产量均明显少于野生型[43]。因此可知,DD对在连接T结构域和C结构域时更加重要。
模块与模块间拼接的研究除了依靠N端与C端的对接结构域介导,前文介绍的XU和XUC概念以外,还有其他的拼接方式。合成生物学工具包含了许多用于调节转录和翻译的,但很少有用来控制蛋白质关联的。有科学家报道了22个被叫做SYNZIPs的特异性合成卷曲肽(SZs),对其SZs的寡聚态、螺旋取向和亲和性进行了生物物理分析,认为SZs极大地增加了合成生物学中可用的蛋白质相互作用部件的数量,这将促进分子工程的广泛应用[39]。实验中,研究人员设计了与人类bZIP转录因子区相互作用的合成肽,这些设计的肽段长度为35~54个氨基酸,与bZIP亮氨酸拉链的氨基酸组成特征相同[44]。将这种设计进行应用延伸,SZs是通过前述计算和设计的合成肽,科研人员插入SZs将单个蛋白NRPSs分裂成独立表达和翻译的多肽链,在引入SZs之前,必须满足野生型C-A结构域间连接子设定的距离标准,以确保正确的C-A双结构域接触。实验人员通过重组XtpS、GxpS、RtpS、和Szentiamide合成酶的构建块,生成并共表达了4组合成的NRPS,提供了简单快速的组合不同NRPS亚基构建肽库方法[45]。近年来,人们成功地将SZs应用于PKS中,以取代天然存在的DDs作为工具来创建嵌合的PKS[46]。
2.3 NRPS模块删除研究
实验操作时,有多种基于分子生物学的可用于重塑基因组以促进天然产物生物合成方法,如上调正调控基因的表达、下调负调控基因的表达、删除非靶基因簇等。
丁烯基杀菌素(butenyl-spinosyn)是由须糖多孢菌(Saccharopolyspora pogona)生产的一种很有前途的生物农药,具有更强的杀虫活性和更广泛的杀虫光谱。有学者有效地删除了须糖多孢菌中一个负责phthoxazolin生物合成的NRPS-T1PKS杂合簇(~20 kb),使得突变体对数期延长,生物量增加;缺少phthoxazolin合成基因簇,抑制了除丁烯基杀菌素之外的其他产物的生产路径,在相同条件下,该菌株比野生型菌株需要更多的时间(240 h)来完成葡萄糖消耗,所以导致葡萄糖消耗率降低,因此丁烯基杀菌素产量比野生型菌株提高了4.72倍[47]。因此在进行NRPS产物研究时,可以通过删除其他次生代谢产物合成基因簇,减少葡萄糖等营养成分的消耗,让反应集中于目的产物途径,来进一步促进目的产物的产生。
有实验证明,删除或突变核心结构形成的关键氨基酸会完全破坏酶的活性[48]。TAYLOR等[49]对含有定向突变的非核糖体肽合成酶基因Tex7的青木霉进行定性研究时,观察到一种未知化合物异常大量的出现,并且鉴定出该化合物为七萜酸,研究后发现是Tex7的突变导致了大量具有生物活性的抗癌化合物七萜酸的积累。这为在生产中提高目的产物产量提供了思路,但删除模块后导致NRPS活性的变化,还需要更为深入的研究。
3 NRPS医学价值
从植物、真菌和细菌中获得的天然产物通过治疗诸如癌症和细菌感染之类的疑难疾病延长了人类的预期寿命并提高了生活质量。许多PKs和NRPS合成的聚酮、聚肽和聚酮-聚肽杂合的天然产物已作为临床药物使用。
表1 来自于NRPS用于临床治疗疾病的天然产物
Table 1 Natural products from NRPS for the clinical treatment of diseases

名称用途达托霉素(daptomycin)抑制革兰氏阳性菌,用于治疗复杂的皮肤和软组织感染、右侧感染性心内膜炎和金黄色葡萄球菌菌血症万古霉素(vancomycin)治疗革兰氏阳性细菌感染,用于治疗金黄色葡萄球菌(Staphylococcus aureus,MRSA)感染环孢素(cyclosporin A)阻断细胞壁蛋白质的合成,进而使细菌死亡,用于预防器官或组织移植所发生的排斥反应;预防及治疗骨髓移植时发生的移植物抗宿主反应硫酸博莱霉素(bleomycin)具有抗肿瘤和抗病毒作用,有报告称其可以用于用于治疗不同类型的疣:普通疣、扁平疣、掌跖疣,包括爪周疣和生殖器疣等杆菌肽(bacitracin)对革兰氏阳性菌和革兰氏阴性菌具有抗菌活性,可以用做生物表面活性剂和防腐剂利福霉素(rifamycin)具有广谱抗菌作用,对结核杆菌、麻风杆菌、链球菌、肺炎球菌等革兰氏阳性细菌,特别是耐药性金黄色葡萄球菌的作用都很强。近期有研究表明其对幽门螺旋杆菌(Helicobacter pylori)有杀菌效果多粘菌素(polymyxin)对于革兰氏阴性杆菌 如大肠杆菌、铜绿假单胞菌等的抗菌活性强,用于治疗CRKP以及耐多药鲍曼不动杆菌(multidrug-resistant A.baumannii,MDRAB)和铜绿假单胞菌(MDRPA)[50]替考拉宁(telicoplanin)对革兰阳性菌如葡萄球菌、链球菌、肠球菌和大多厌氧性阳性菌敏感。临床主要用于金葡菌及链球菌属等敏感菌所致的严重感染,如心内膜炎、骨髓炎、败血症及呼吸道、泌尿道、皮肤、软组织等的感染雷帕霉素(rapamycin)临床上用于器官移植的抗排斥反应和自身免疫性疾病的治疗asperphenamate具有抗HIV和抗糖尿病,和抗肿瘤特性
实验中可以通过编码基因的基因工程操纵NRPS,生物合成具有潜在作用的变种的知名药物,甚至完全新的产品[51-52]。托霉素是一种典型的由NRPS合成的环脂肽类抗生素,A54145是一种具有抗革兰氏阳性病原菌活性的环状脂肽抗生素,两者分子结构具有许多相似之处,有实验将用达托霉素的NRPS模块替换了A54145的NRPS模块,产生了新的脂肽,并且发现该新脂肽在牛表面活性剂存在的情况下表现出良好的抑菌活性,并且对肺炎链球菌表现出低抑菌性[53]。
微生物是许多用于癌症治疗的化学疗法的来源,其中放线菌是最有希望的来源生物。DHANEESHA等[54]从北极峡湾沉积物中分离出的海洋放线菌进行了潜在的抗癌活性筛选,扩增的PKs和NRPS参与次生代谢产物的产生,与链霉菌已知生物合成基因的相似性仅为82%,表明该提取物可能产生一种新的次生代谢产物。利用LC-MS/MS分子网络进行化学解离的努力表明存在一系列未描述的四烯多元醇,它对NCIH460细胞具有最强的生长抑制作用。这些结果表明,北极蒿属S.Artemisiae菌株MCCB 248是一个有希望的天然产物药物发现和基因组挖掘潜在抗癌药物的候选株。
asperphenamate是一种小肽类天然产物,因其抗肿瘤活性而备受关注。最近的研究还表明它是一种潜在的神经炎症抑制剂,并具有抗HIV和抗糖尿病的特性。SUBKO等[55]对已知和新的asperphenamate类似物进行解离。结果发现22种新的阿斯奋乃酯用HRMS/MS对类似物进行了表征,其中21个是用蛋白源和非蛋白源氨基酸作为生长培养基的补充设计的。
抗生素耐药性的蔓延对公共健康构成严重威胁,预计到2050年将达到惊人的水平,因此开发过程中新抗菌药物迫在眉睫。非核糖体肽合成酶在底物可用性的基础上具有非凡的灵活性,因此有可能在丝状真菌中操纵和设计新的肽天然抗菌产物。在放线杆菌属中有一个很少被探索的成员,科学家通过对88个本属分离物提取物的分析,确定了一个由C20聚酮链酰化的环肽家族,将其命名为别肽霉素。别肽霉素的生物合成基因簇中含有3个非核糖体肽合成酶模块,由此发现了一种新的抗菌肽是由PKS-NRPS产生的。在寻找具有抗菌活性的新陈代谢物时,必须采用创新的方法来增加从微生物来源发现新化学物质的可能性[56]。
4 展望
目前,NRPS生物合成途径被认为是天然产物新类似物的丰富来源,是获得具有药物活性的天然产物的一种经济可行的方法。已经有许多报道,结构域或模块替换和诱变实验成功开发NRPS功能。然而,这些设计的NRPS往往比它们的野生型效率低得多,因此,这些酶系统的复杂机制需要进一步深入研究。NRPS的高度动态结构和复杂的蛋白与蛋白相互作用给工程设计带来了挑战,另一个障碍是NRPS蛋白的巨大尺寸阻碍了异源表达。由于多种因素的影响,获取NRPS结构域相互作用的结构信息并将其完全应用于NRPS工程仍然具有挑战性。本文通过对NRPS结构域以及NRPS模块改造策略的探讨,将有助于通过合理的工程化努力,实现扩大NRPS生物合成产品的多样性。
实验中,对NRPS的改造工程进展并不迅速,很多实验结果表明相同的方法并不一定适用于所有案例。如何重新编程非核糖体肽合成酶,同时保持与野生型酶相同的活性,似乎还需要深入挖掘。值得期待的是,在过去的几年中合成生物学有了一些显著的发展,这些发展有可能加快NRPS工程化改造的进程,创建和优化多种新的NRPS装配线。不断降低的基因合成成本,新的组装和编辑技术可以比传统基因技术产生和检测更多的突变和重组NRPS结构。如果这些进展被充分利用,NRPS结构的规则将变得更加容易理解,最终可能产生完全人为理性设计的非核糖体多肽。
[1] WALSH C T. Insights into the chemical logic and enzymatic machinery of NRPS assembly lines[J]. Natural Product Reports, 2016, 33(2):127-135.
[2] CACHO R A, TANG Y. Reconstitution of fungal nonribosomal peptide synthetases in yeast and in vitro[J]. Methods in Molecular Biology, 2016, 1401:103-119.
[3] MOOTZ H D, SCHWARZER D, MARAHIEL M A. Ways of assembling complex natural products on modular nonribosomal peptide synthetases[J]. Chembiochem: a European Journal of Chemical Biology, 2002, 3(6):490-504.
[4] BOZHÜYÜK K A, MICKLEFIELD J, WILKINSON B. Engineering enzymatic assembly lines to produce new antibiotics[J]. Current Opinion in Microbiology, 2019, 51:88-96.
[5] YU D Y, XU F C, ZHANG S W, et al. Decoding and reprogramming fungal iterative nonribosomal peptide synthetases[J]. Nature Communications, 2017, 8:15349.
[6] KIM M S, BAE M, JUNG Y E, et al. Unprecedented noncanonical features of the nonlinear nonribosomal peptide synthetase assembly line for WS9326A biosynthesis[J]. Angewandte Chemie (International Ed. in English), 2021, 60(36):19766-19773.
[7] SÜSSMUTH R D, MAINZ A. Nicht-ribosomale peptidsynthese-prinzipien und perspektiven[J]. Angewandte Chemie, 2017, 129(14):3824-3878.
[8] BAUNACH M, CHOWDHURY S, STALLFORTH P,et al.The landscape of recombination events that create nonribosomal peptide diversity[J]. Molecular Biology and Evolution, 2021, 38(5):2116-2130.
[9] CALCOTT M J, OWEN J G, ACKERLEY D F. Efficient rational modification of non-ribosomal peptides by adenylation domain substitution[J]. Nature Communications, 2020, 11(1):4554.
[10] NIQUILLE D L, HANSEN D A, MORI T, et al. Nonribosomal biosynthesis of backbone-modified peptides[J]. Nature Chemistry, 2018, 10(3):282-287.
[11] ![]() A, HÜSKEN A, STEPHAN P, et al. Engineered nonribosomal peptide synthetase shows opposite amino acid loading and condensation specificity[J]. ACS Catalysis, 2021, 11(14):8692-8700.
A, HÜSKEN A, STEPHAN P, et al. Engineered nonribosomal peptide synthetase shows opposite amino acid loading and condensation specificity[J]. ACS Catalysis, 2021, 11(14):8692-8700.
[12] LEE T V, JOHNSON R D, ARCUS V L, et al. Prediction of the substrate for nonribosomal peptide synthetase (NRPS) adenylation domains by virtual screening[J]. Proteins, 2015, 83(11):2052-2066.
[13] HARA R, SUZUKI R, KINO K. Hydroxamate-based colorimetric assay to assess amide bond formation by adenylation domain of nonribosomal peptide synthetases[J]. Analytical Biochemistry, 2015, 477:89-91.
[14] KALJUNEN H, SCHIEFELBEIN S H H, STUMMER D D, et al. Strukturelle Aufklärung der Bispezifität von A-Domänen als Basis für die Aktivierung nicht-natürlicher Aminosäuren[J].Angewandte Chemie, 2015, 127(30):8957-8961.
[15] ABE T, KOBAYASHI K, KAWAMURA S, et al. Dipeptide synthesis by internal adenylation domains of a multidomain enzyme involved in nonribosomal peptide synthesis[J]. The Journal of General and Applied Microbiology, 2019, 65(1):1-10.
[16] KANO S, SUZUKI S, HARA R, et al. Synthesis of d-amino acid-containing dipeptides using the adenylation domains of nonribosomal peptide synthetase[J]. Applied and Environmental Microbiology, 2019, 85(13): e00120-e00119.
[17] WU Z Y, LI Y R, FANG Y K, et al. Adenylation domains of nonribosomal peptide synthetase: A potential biocatalyst for synthesis of dipeptides and their derivatives[J]. Enzyme and Microbial Technology, 2022, 160:110089.
[18] IZORÉ T, CRYLE M J. The many faces and important roles of protein-protein interactions during non-ribosomal peptide synthesis[J]. Natural Product Reports, 2018, 35(11):1120-1139.
[19] CALCOTT M J, ACKERLEY D F. Portability of the thiolation domain in recombinant pyoverdine non-ribosomal peptide synthetases[J]. BMC Microbiology, 2015, 15:162.
[20] LINNE U, DOEKEL S, MARAHIEL M A. Portability of epimerization domain and role of peptidyl carrier protein on epimerization activity in nonribosomal peptide synthetases[J]. Biochemistry, 2001, 40(51):15824-15834.
[21] OWEN J G, ROBINS K J, PARACHIN N S, et al. A functional screen for recovery of 4′-phosphopantetheinyl transferase and associated natural product biosynthesis genes from metagenome libraries[J]. Environmental Microbiology, 2012, 14(5):1198-1209.
[22] GAO L, LIU H X, MA Z, et al. Translocation of the thioesterase domain for the redesign of plipastatin synthetase[J]. Scientific Reports, 2016, 6:38467.
[23] KANIUSAITE M, GOODE R J A, TAILHADES J, et al. Exploring modular reengineering strategies to redesign the teicoplanin non-ribosomal peptide synthetase[J]. Chemical Science, 2020, 11(35):9443-9458.
[24] STACHELHAUS T, MOOTZ H D, MARAHIEL M A. The specificity-conferring code of adenylation domains in nonribosomal peptide synthetases[J]. Chemistry &Biology, 1999, 6(8):493-505.
[25] BERGENDAHL V, LINNE U, MARAHIEL M A. Mutational analysis of the C-domain in nonribosomal peptide synthesis[J]. European Journal of Biochemistry, 2002, 269(2):620-629.
[26] SAMEL S A, SCHOENAFINGER G, KNAPPE T A, et al. Structural and functional insights into a peptide bond-forming bidomain from a nonribosomal peptide synthetase[J]. Structure, 2007, 15(7):781-792.
[27] ZHONG L, DIAO X T, ZHANG N, et al. Engineering and elucidation of the lipoinitiation process in nonribosomal peptide biosynthesis[J]. Nature Communications, 2021, 12(1):296.
[28] BOZHÜYÜK K A J, FLEISCHHACKER F, LINCK A, et al. De novo design and engineering of non-ribosomal peptide synthetases[J]. Nature Chemistry, 2018, 10(3):275-281.
[29] BOZHÜYÜK K A J, LINCK A, TIETZE A, et al. Modification and de novo design of non-ribosomal peptide synthetases using specific assembly points within condensation domains[J]. Nature Chemistry, 2019, 11(7):653-661.
[30] TURGAY K, KRAUSE M, MARAHIEL M A. Four homologous domains in the primary structure of GrsB are related to domains in a superfamily of adenylate-forming enzymes[J]. Molecular Microbiology, 1992, 6(18):2743-2744.
[31] KOHLI R M, TAKAGI J, WALSH C T. The thioesterase domain from a nonribosomal peptide synthetase as a cyclization catalyst for integrin binding peptides[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(3):1247-1252.
[32] KIM B S, CROPP T A, BECK B J, et al. Biochemical evidence for an editing role of thioesterase II in the biosynthesis of the polyketide pikromycin[J]. The Journal of Biological Chemistry, 2002, 277(50):48028-48034.
[33] SCHWARZER D, MOOTZ H D, MARAHIEL M A. Exploring the impact of different thioesterase domains for the design of hybrid peptide synthetases[J]. Chemistry &Biology, 2001, 8(10):997-1010.
[34] WANG H, FEWER D P, HOLM L, et al. Atlas of nonribosomal peptide and polyketide biosynthetic pathways reveals common occurrence of nonmodular enzymes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(25):9259-9264.
[35] KRIES H. Biosynthetic engineering of nonribosomal peptide synthetases[J]. Journal of Peptide Science: an Official Publication of the European Peptide Society, 2016, 22(9):564-570.
[36] HACKER C, CAI X F, KEGLER C, et al. Structure-based redesign of docking domain interactions modulates the product spectrum of a rhabdopeptide-synthesizing NRPS[J]. Nature Communications, 2018, 9(1):4366.
[37] DOWLING D P, KUNG Y, CROFT A K, et al. Structural elements of an NRPS cyclization domain and its intermodule docking domain[J]. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(44):12432-12437.
[38] KOSOL S, GALLO A, GRIFFITHS D, et al. Structural basis for chain release from the enacyloxin polyketide synthase[J]. Nature Chemistry, 2019, 11(10):913-923.
[39] THOMPSON K E, BASHOR C J, LIM W A, et al. SYNZIP protein interaction toolbox: in vitro and in vivo specifications of heterospecific coiled-coil interaction domains[J]. ACS Synthetic Biology, 2012, 1(4):118-129.
[40] HAHN M, STACHELHAUS T. Selective interaction between nonribosomal peptide synthetases is facilitated by short communication-mediating domains[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(44):15585-15590.
[41] CHIOCCHINI C, LINNE U, STACHELHAUS T. In vivo biocombinatorial synthesis of lipopeptides by COM domain-mediated reprogramming of the surfactin biosynthetic complex[J]. Chemistry &Biology, 2006, 13(8):899-908.
[42] HAHN M, STACHELHAUS T. Harnessing the potential of communication-mediating domains for the biocombinatorial synthesis of nonribosomal peptides[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(2):275-280.
[43] KEGLER C, BODE H B. Artificial splitting of a non-ribosomal peptide synthetase by inserting natural docking domains[J]. Angewandte Chemie International Edition, 2020, 59(32):13463-13467.
[44] REINKE A W, GRANT R A, KEATING A E. A synthetic coiled-coil interactome provides heterospecific modules for molecular engineering[J]. Journal of the American Chemical Society, 2010, 132(17):6025-6031.
[45] BOZHUEYUEK K A J, WATZEL J, ABBOOD N, et al. Synthetic zippers as an enabling tool for engineering of non-ribosomal peptide synthetases[J]. Angewandte Chemie International Edition, 2021, 60(32):17531-17538.
[46] KLAUS M, D’SOUZA A D, NIVINA A, et al. Engineering of chimeric polyketide synthases using SYNZIP docking domains[J]. ACS Chemical Biology, 2019, 14(3):426-433.
[47] RANG J, LI Y L, CAO L, et al. Deletion of a hybrid NRPS-T1PKS biosynthetic gene cluster via Latour gene knockout system in Saccharopolyspora pogona and its effect on butenyl-spinosyn biosynthesis and growth development[J]. Microbial Biotechnology, 2021, 14(6):2369-2384.
[48] GEIB E, BALDEWEG F, DOERFER M, et al. Cross-chemistry leads to product diversity from atromentin synthetases in aspergilli from section nigri[J]. Cell Chemical Biology, 2019, 26(2):223-234.e6.
[49] TAYLOR J T, MUKHERJEE P K, PUCKHABER L S, et al. Deletion of the Trichoderma virens NRPS, Tex7, induces accumulation of the anti-cancer compound heptelidic acid[J]. Biochemical and Biophysical Research Communications, 2020, 529(3):672-677.
[50] LIU X F, HUANG C R, BERGEN P J, et al. Chinese consensus guidelines for therapeutic drug monitoring of polymyxin B, endorsed by the Infection and Chemotherapy Committee of the Shanghai Medical Association and the Therapeutic Drug Monitoring Committee of the Chinese Pharmacological Society[J]. Journal of Zhejiang University. Science. B, 2023, 24(2):130-142.
[51] MOOTZ H D, MARAHIEL M A. Biosynthetic systems for nonribosomal peptide antibiotic assembly[J]. Current Opinion in Chemical Biology, 1997, 1(4):543-551.
[52] STACHELHAUS T, SCHNEIDER A, MARAHIEL M A. Rational design of peptide antibiotics by targeted replacement of bacterial and fungal domains[J]. Science, 1995, 269(5220):69-72.
[53] NGUYEN K T, HE X W, ALEXANDER D C, et al. Genetically engineered lipopeptide antibiotics related to A54145 and daptomycin with improved properties[J]. Antimicrobial Agents and Chemotherapy, 2010, 54(4):1404-1413.
[54] DHANEESHA M, BENJAMIN NAMAN C, KRISHNAN K P, et al. Streptomyces artemisiae MCCB 248 isolated from Arctic fjord sediments has unique PKS and NRPS biosynthetic genes and produces potential new anticancer natural products[J]. 3 Biotech, 2017, 7(1):32.
[55] SUBKO K, WANG X H, NIELSEN F H, et al. Mass spectrometry guided discovery and design of novel asperphenamate analogs from Penicillium astrolabium reveals an extraordinary NRPS flexibility[J]. Frontiers in Microbiology, 2021, 11:618730.
[56] IORIO M, GENTILE A, BRUNATI C, et al. Allopeptimicins: Unique antibacterial metabolites generated by hybrid PKS-NRPS, with original self-defense mechanism in Actinoallomurus[J]. RSC Advances, 2022, 12(26):16640-16655.
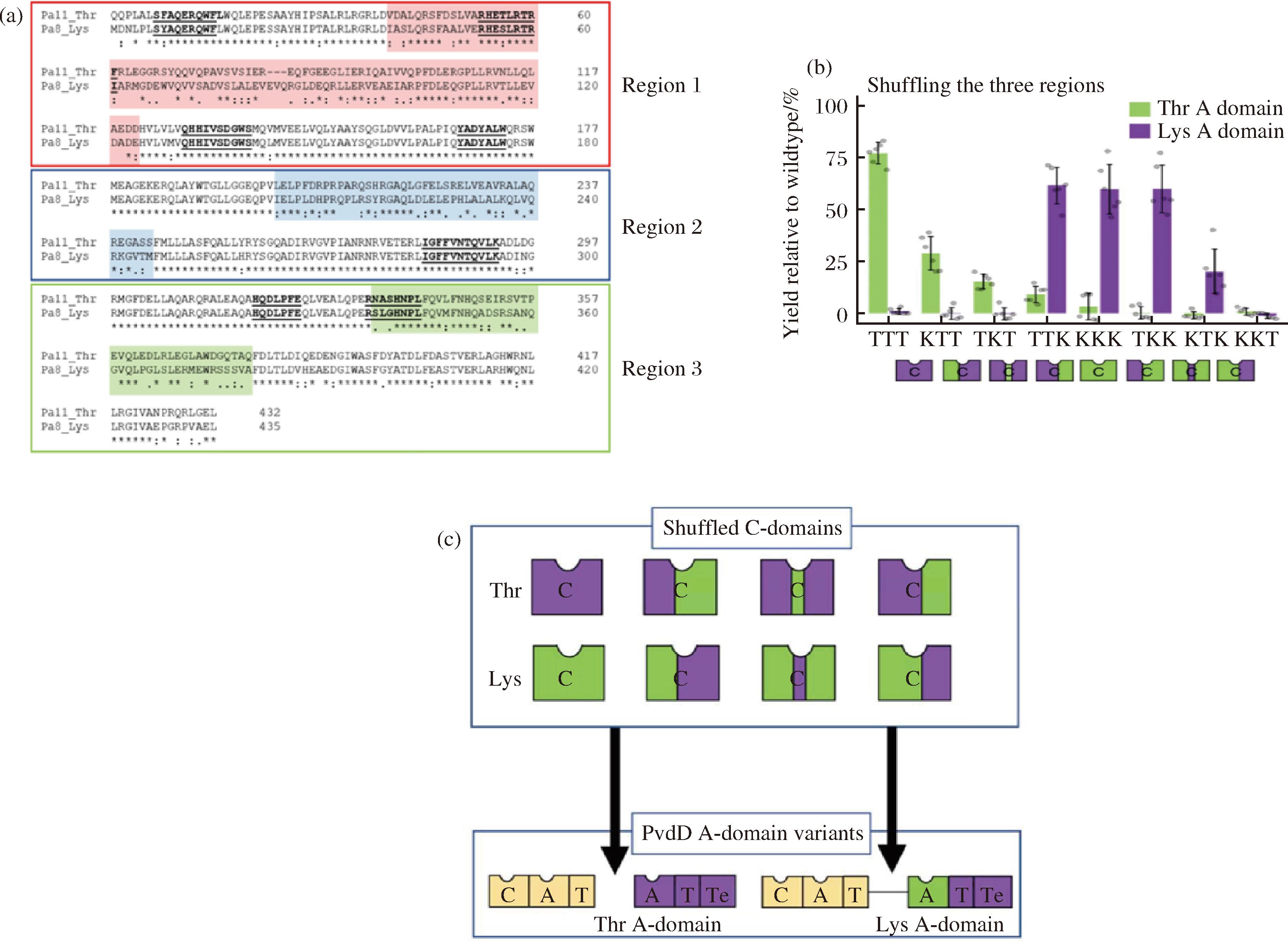
a-Pa11和Pa8的C结构域的氨基酸序列比对;b-8种C结构域重组示意图;c-通过测量相对于野生型绿脓菌在400 nm处的吸光度来评估 由来自b的变体pvdD基因转化的pvdD缺失菌株的绿脓菌荧光素生产。
附图1 结构域交换策略
Fig.S1 Strategies for domain substitution
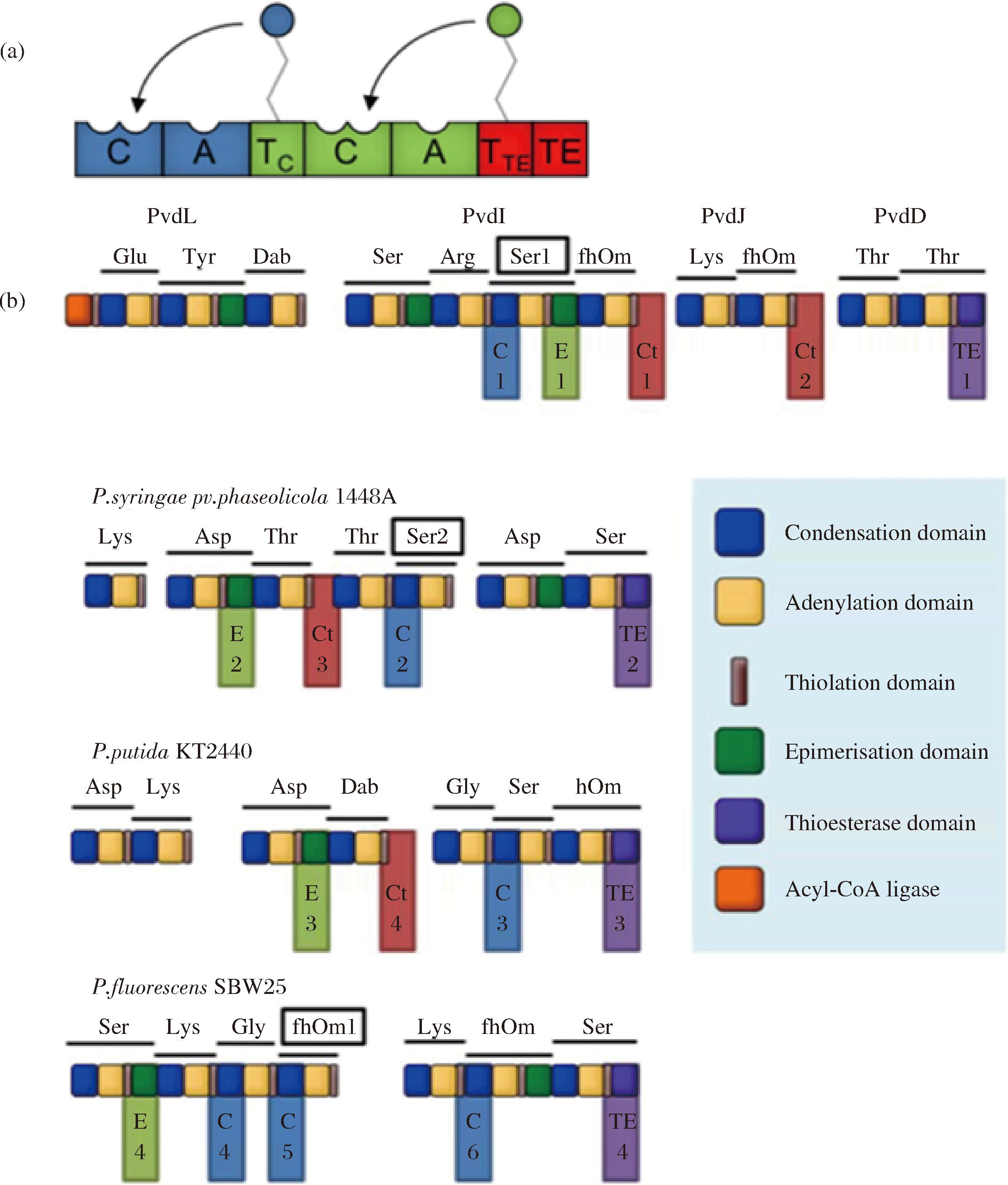
a-将18例不同T结构域移植到各自C结构域的上游;b-6个TC(蓝色)、4个TCT(红色)、4个TE(绿色)和4个TTE(紫色)的 位置和编号由彩色编码的阴影框表示
附图2 结构域交换策略
Fig.S2 Strategies for domain substitution
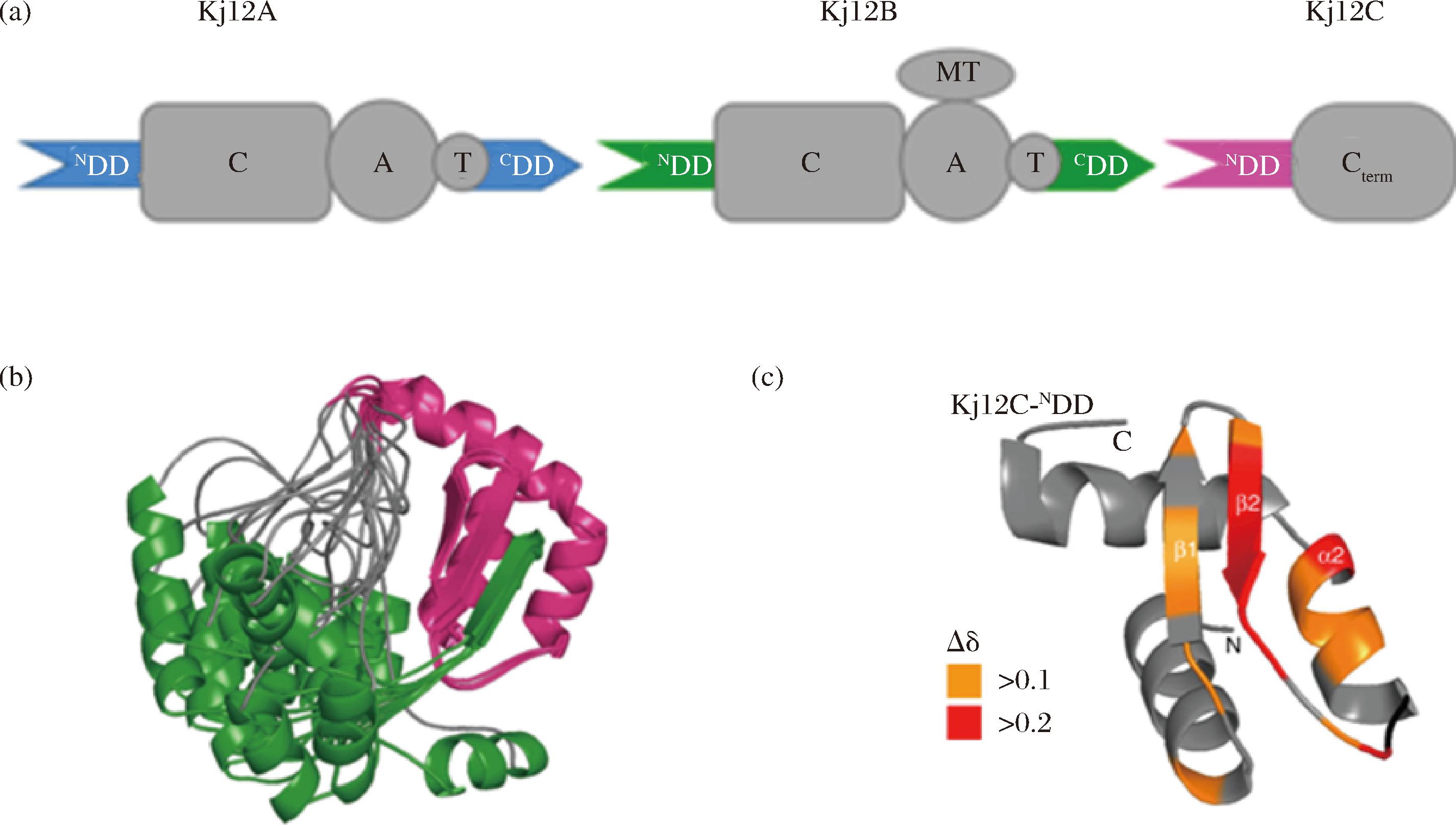
a-从Xenorhabdus stockiae KJ12.1中提取RXP NRPSs (Kj12ABC);b-Kj12C-NDD-Kj12B CDD连接结构的颜色编码; c-添加未标记的Kj12B-CDD后,Kj12C-NDD的化学位移变化映射到Kj12C-NDD的结构上。
附图3 NRPS的对接结构域
Fig.S3 Docking domain of NRPS