多糖类天然产物是自然界中一类重要的生物资源,在生命发展的过程中也发挥了关键作用。木质纤维素作为多糖类天然产物的重要组成部分,主要由纤维素、半纤维素、木质素组成,其在自然界大量存在,也是植物细胞壁的主要组成部分。在全球能源以及资源紧张的情况下,木质纤维素作为可再生资源越来越受到人们的重视,开发利用木质纤维素在降低资源浪费的同时还能减少环境污染。
目前,针对木质纤维素存在2种降解方式,一种是化学水解,另一种是利用酶制剂进行高效酶解[1]。相对于化学方法,酶催化水解具有反应条件温和、能耗低、环境友好等优势,成为近年来的研究热点。通过微生物代谢的糖苷水解酶(glycoside hydrolases,GH)对木质纤维进行降解,是一种高效的生物质资源化方法[2]。但是,植物细胞壁内的纤维素大多为高强度、高密度结晶态,构成了一道天然的“防降解壁垒”,阻止了糖苷水解酶对多糖链结晶区的可及性,限制了其高效降解[3]。同时, GH活性低,不耐高温,不耐酸碱,或有抑制剂干扰,限制了其大规模应用。因此如何高效降解结晶区成为人们的研究动力。近年来,裂解多糖单加氧酶(lytic polysaccharide monooxygenase,LPMO)的发现完全颠覆了对酶解法降解结晶多糖的认知[4]。LPMO是一类铜离子依赖性氧化酶,它可以通过氧化作用切断多糖类中的糖苷键,从而使难于降解的生物质发生解聚并暴露出更多与GH结合的位点,从而加快纤维素和几丁质的水解[5]。
1 裂解多糖单加氧酶的发现
最初人们从粘质沙雷氏菌(Serratia marcescens)中发现一种分子质量很小的碳水化合物结合模块33家族(carbohydrate-binding module family 33,CBM33)蛋白,该蛋白与甲壳素有强烈的结合作用,命名为甲壳素结合蛋白(chitin binding protein,CBP21)[6]。随后的研究表明CBP21可以在有氧和电子供体条件下,氧化裂解几丁质多糖,并且发现酶活性可能与二价铜离子有很强的相关性,因其内切葡聚糖酶活性被归入GH61家族[7]。并且研究发现在太瑞斯梭壳孢霉(Thielavia terrestris)发酵液中加入等量的瑞氏木霉(Trichoderma reesei)纤维素酶,可有效地增加纤维素的降解,显著增加了糖分的转化率。在此基础上,太瑞斯梭壳孢霉菌和瑞氏木霉细胞外纤维素酶系差异分析结果显示,筛选到3种与糖类代谢有关的胞外酶,即GH61B、GH61E、GH61G。这类化合物虽不是GH61家族成员,却能明显增强预处理后的玉米秸秆中纤维素酶的活性[8]。2011 年,这种具有铜离子依赖性并且能够氧化断裂纤维素中的糖苷键的酶被命名为 LPMO,其能够实现对生物质的解聚,将底物的作用位点完全暴露,从而实现对纤维素的高效降解。
2 裂解多糖单加氧酶的分类与来源
最初根据氨基酸序列的相似性,将 LPMO分为CBM33和GH61家族两大类[9]。由于LPMO的氧化作用可以促进糖苷键的断裂,增加了底物和GH结合位点的亲和力,加快了其降解效率。2013年,LPMO被归于碳水化合物活性酶(carbonhydrate-activity enzymes database,CAZy)数据库中的辅助活性(auxiliary activity family,AA)家族。如今,由CAZy数据库中的信息和对LMPO中氨基酸序列与功能之间的分析将LPMO分为8个家族,分别为AA9-11、AA13-17辅助活性家族。它们的来源及底物如表1所示,其中GH61和CBM33分别属于AA9和AA10家族。
表1 LPMO的分类、来源和底物
Table 1 The classification, sources, and substrates of LPMO
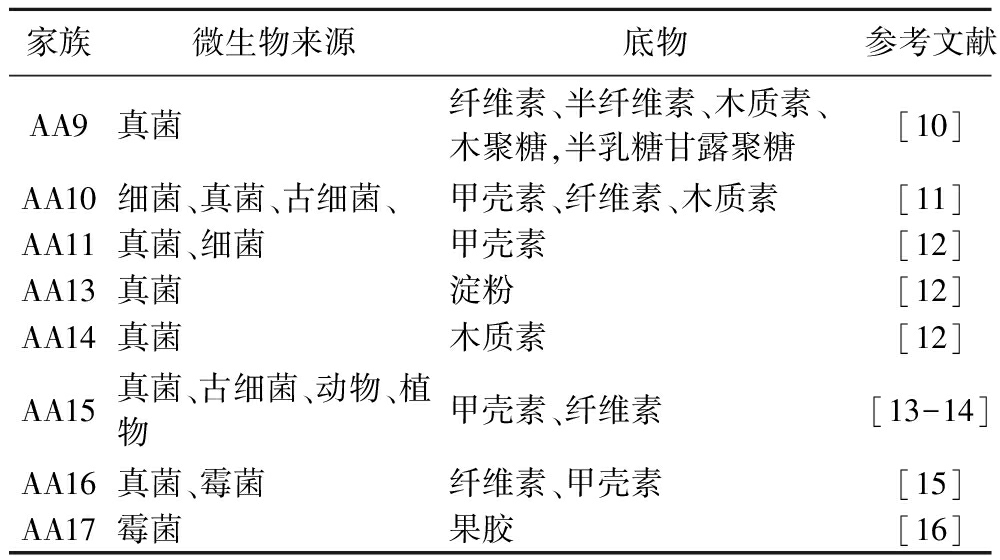
家族微生物来源底物参考文献AA9真菌纤维素、半纤维素、木质素、木聚糖,半乳糖甘露聚糖[10]AA10细菌、真菌、古细菌、甲壳素、纤维素、木质素[11]AA11真菌、细菌甲壳素[12]AA13真菌淀粉[12]AA14真菌木质素[12]AA15真菌、古细菌、动物、植物甲壳素、纤维素[13-14]AA16真菌、霉菌纤维素、甲壳素[15]AA17霉菌果胶[16]
LPMO主要分布在细菌和真菌中[12]。AA9家族来源于真菌,主要降解纤维素、半纤维素、木质素。AA10家族是LPMO中最大的家族,最早发现马氏沙雷氏菌中,其大多来自于细菌也有少量来自于真菌、古生菌,降解底物为纤维素和几丁质。
真菌来源的AA11、AA13、AA14家族LPMO对多糖底物均有降解。细菌来源的AA11家族对几丁质有降解作用,AA13家族的LPMO能在C1位点切断淀粉骨架键释放寡糖从而起到对淀粉的降解作用,AA14家族则可降解木质素。最初, LPMO仅存在于微生物中,在小鱼中AA15LPMO的新发现引发了人们对动物 LPMO的关注[13]。AA15家族LPMO主要来源于真菌、动物、植物等真核生物对虾壳中的几丁质也有降解作用[14]。AA16家族LPMO来源于真菌和部分卵菌中,对纤维素有活性的同时也能协同纤维素酶共同降解纤维素[15]。AA17家族LPMO是目前唯一存在于真菌中具有果胶酶活性的LPMO家族[16]。
3 裂解多糖单加氧酶的结构和功能
3.1 裂解多糖单加氧酶的结构
迄今,已知的LPMO均为β-三明治折叠和组氨酸支架构成的高度保守平面结构[17-19]。以来源于嗜热子囊菌(Thermoascus aurantiacus)的LPMO(TaLPMO)结构为例(图1),β-三明治折叠,由 8~10个反向平行的β-折叠通过α-螺旋相互连接组成;LPMO的活性中心(图1)包括2个保守的组氨酸(His),以及一个酪氨酸(Tyr)或苯丙氨酸(Phe),其分支可以将铜离子固定在其上[20]。除了AA14家族,其他LPMO的结构中均含有铜离子的活性中心。此外,其他的组氨酸骨架在轴位上为酪氨酸(Tyr),而在AA10上为苯丙氨酸(Phe),是其重要的底物结合位点[21]。
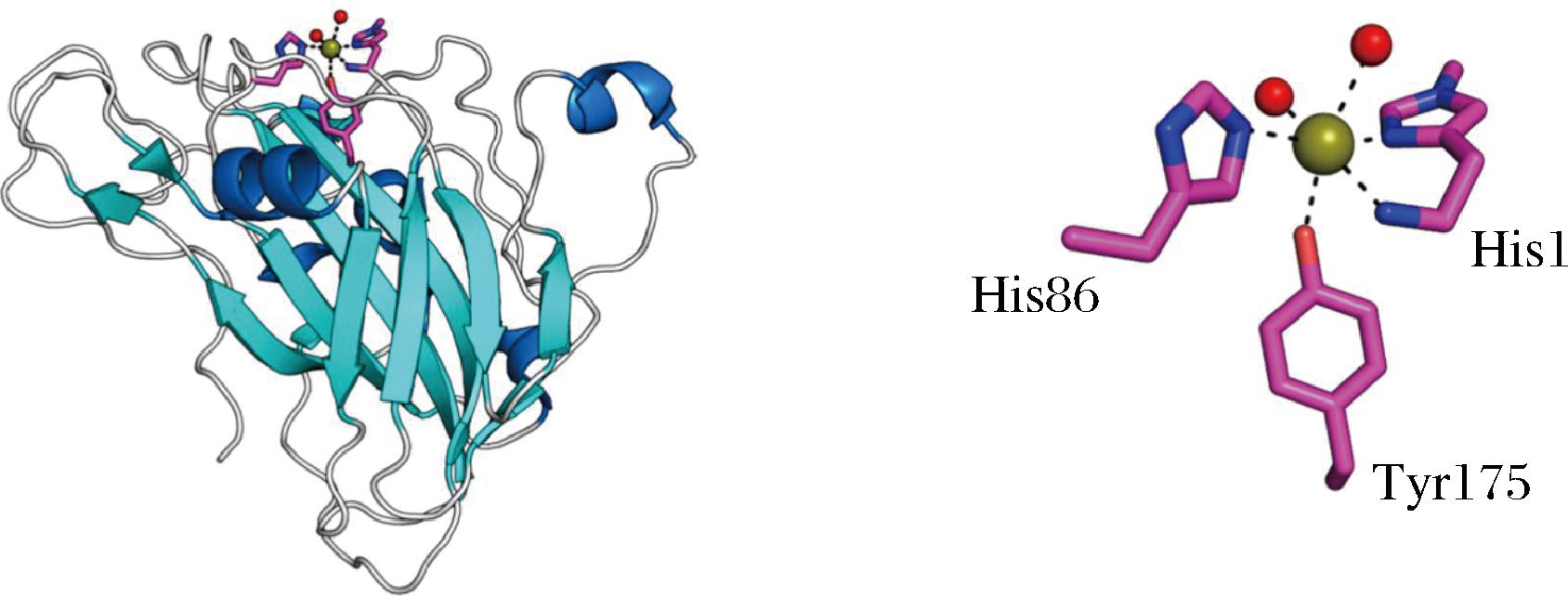
图1 TaLPMO三维结构和活性位点[17]
Fig.1 3D structure and active site diagram of TaLPMO[17]
目前所知的 LPMO一般是一种具有除催化域之外的多个区域的酶,除此之外,还具有CBM。在源于粉红面包霉菌(Neurospora crassa)体内的LPMO(NaLPMO)中就有天然形式的CBM[22],同时LPMO的催化结构域可以与CBM组合从而影响活性。这种模式下不仅可以让酶与底物结合得更好,也使底物的多样性得到极大地加强。
3.2 裂解多糖单加氧酶的催化机制
LPMO在氧分子和电子供体同时存在时,通过氧化的方式断裂多聚糖主链的 β-1,4 糖苷键产生寡糖,电子供体大多为酶类还原剂。后来的研究表明该过程要有铜离子的参与,催化过程中LPMO在氧和铜原子存在条件下生成LPMO-Cu2+复合物,电子供体产生的电子将其转化为LPMO-Cu+复合物。该复合物得到氧分子中的电子产生铜离子过氧化物中间体,有多余电子情况下氧化多糖内C1或C4位的碳原子生成羟基化多糖并产生水[23-25]。还有一种观点认为LPMO在电子和H2O2存在下产生LPMO-Cu2+羟基复合物,从而达到氧化多糖的作用。LPMO作用机制(图2)不同于糖苷水解酶,两者协同作用可以提高对木质纤维素的转化效率,首先LPMO在电子供体和铜离子条件下,切断纤维素结晶区之间的糖苷键,使其出现更多GH的作用位点而增加对纤维素的降解速度。
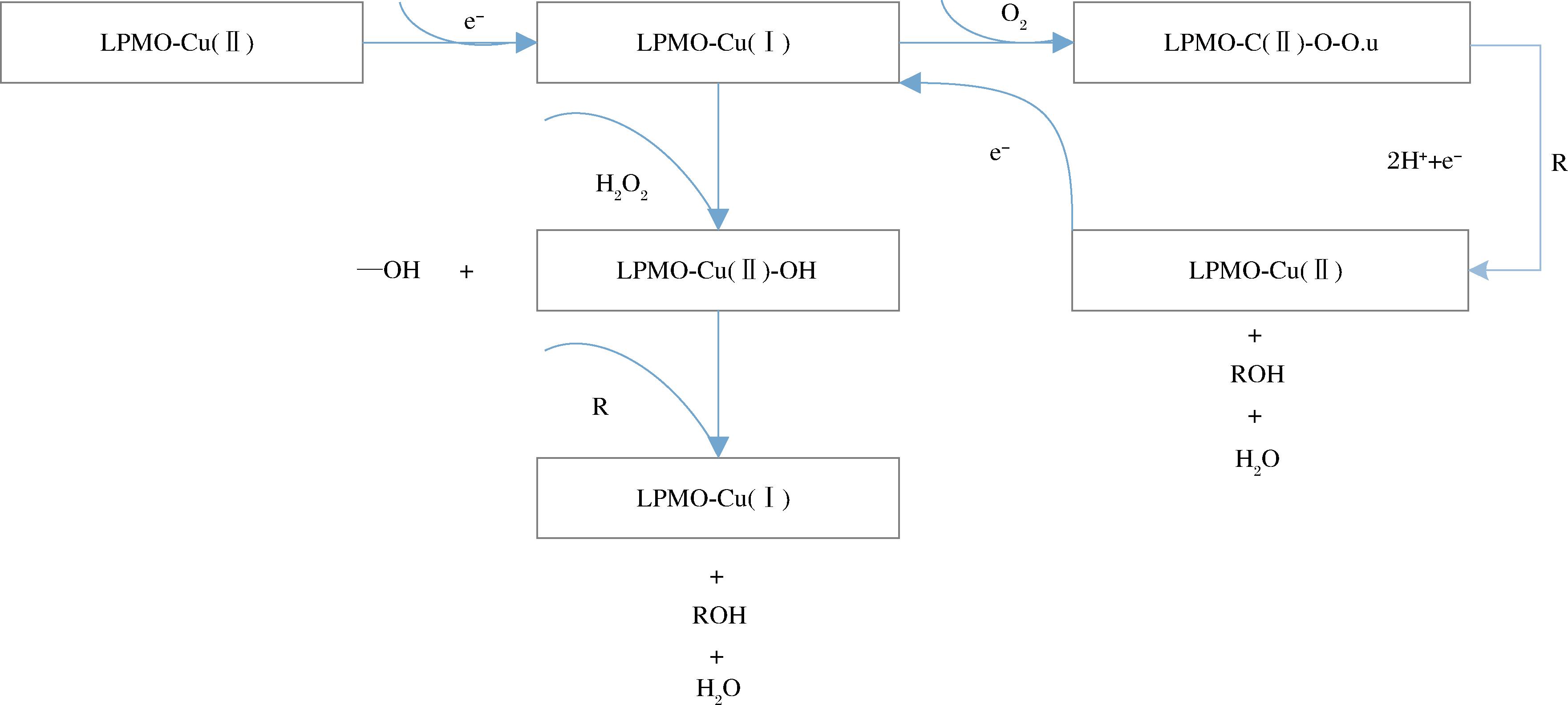
图2 LPMO的催化机制[29]
Fig.2 Catalytic mechanism of LPMO[29]
注:R为多糖底物(下同)。
LPMO根据氧化位点的不同可以分为三类(图3)。第一类只对多糖的C1位进行氧化,产生醛糖酸,第二类仅对多糖的C4位进行氧化,产生C4酮醛糖,最后一类作用C1和C4产生醛糖酸和C4酮醛糖[26-27]。LPMO共底物是O2还是H2O2一直是一个在讨论的话题,现在较多的实验表明H2O2作为共底物的酶活性较高[28]。
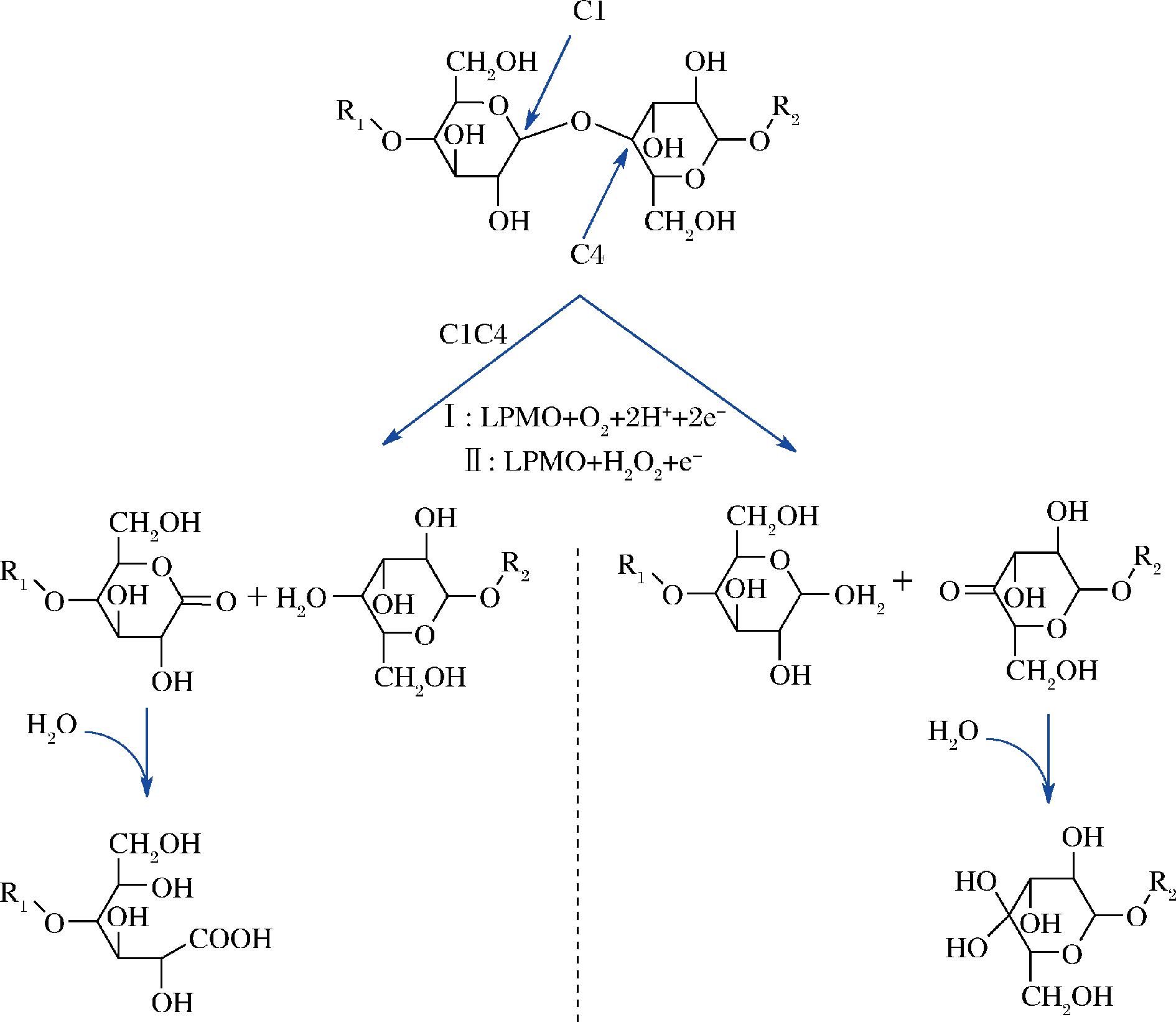
图3 LPMO反应途径
Fig.3 The reaction pathway for LPMO
3.3 裂解多糖单加氧酶的功能
杜文珍等[30]对丝状真菌(Podospora anserina)中AA11家族的5个LPMO 基因进行定点敲除的实验中,5个基因同时被敲除后,菌株对碳源的吸收能力明显降低、生长速度下降、孢子萌发速率降低,子实体数量减少、部分子实体发育不良、寿命缩短以及纤维素降解能力降低。由此表明菌株中的AA11家族基因影响着其生长、生殖、衰老和纤维素降解方面。来源于蝉棒束孢菌(Isaria cicadae)突变体基因IcFBR1其包含糖基磷脂酰肌醇锚定蛋白(glycosylphosphatidylinositol-anchored protein, GPI-AP)同系物的LPMO。敲除IcFBR1后,孢梗维管束不再出现,菌落气生菌丝发育和产孢均不正常。菌体中的多糖含量明显减少,同时还伴随着淀粉酶、蛋白酶活力的降低。其在调控蝉花梗束发育、次生物质代谢以及营养物质的吸收等方面发挥着重要的调控功能[31]。
此外,LPMO在动物植体内也发挥着重要作用。如黑腹果蝇不同的发展阶段中LPMO中有着高水平的表达,同时基因发生改变之后对其致死率有着影响[32]。MoAa91(AA9家族的LPMO)是水稻瘟疫病菌中一个具有诱导黏附作用的蛋白,其敲除会导致黏附能力下降,从而阻碍了水稻的侵染[33]。昆虫以植物为主要食料,其消化道内的酶类及其与共生菌共同完成对植物的高效消化。家蚕无肠道菌群,因此,来自家蚕中肠区的 LPMO(AA15)在其中扮演了重要角色[12]。
4 裂解多糖单加氧酶活性测定方法
LPMO的活性测定影响因素较多。一方面氧化产物多为不溶解性底物,另一方面由于直接作用于底物效率较低。由于LPMO与O2和电子供体(如抗坏血酸)存在的条件下形成H2O2,基于此通过分析H2O2来表示酶活性的测定方法有2,6-二甲基苯酚比色法和Amplex Red辣根过氧化物酶法。通常也可用协同活性来表示表示LPMO的活性,二硝基水杨酸法(3,5-dinitro-2-hydroxybenzoic acid, DNS)便是一种比较普遍的方法。
4.1 二硝基水杨酸法(DNS法)
二硝基水杨酸法是以二硝基水杨酸和还原糖在碱性环境中的氧化-还原反应为基础,以540 nm波长下的吸光值为指标,对还原糖的含量进行了测定。将 LPMO和GH对纤维素底物进行协同作用,将其与 DNS试剂进行混合之后,再进行加热显色,并记录还原糖的 OD540吸光值,通过将单独测活和协同测活以产物的提高量进行比较,来判断 LPMO的活性[34]。此法简单、价格低廉,但其灵敏度不高,仅能对还原性糖类进行测定,而对某些非还原性糖类物质则不能进行测定。此法用于研究LPMO与其他酶的协同作用,但是只能对样品中的单糖进行定量分析。
4.2 2,6-二甲基苯酚比色法(2,6-dimethoxyphenol,2,6-DMP法)
研究表明[35]2分子2,6-DMP和1分子H2O2在LPMO酶的作用下产生2分子2,6-DMP自由基在不稳定的条件下自发二聚产生1分子氢化木醋醌然后与1分子H2O2在LPMO下产生黄色显色产物木醋醌,由木醋醌在469 nm波段具有特征吸收峰即可表现出LPMO活性(图4)。这种方法快速、简单能对浓度0.5~50 mg/L的LPMO进行活性测量,已用于测定纤维素和甲壳素中LPMO的活性,但是该方法在酸性条件下灵敏度较差。氢化木醋醌是比2,6-DMP更好的底物,表观 KM 值低约 30 倍,氧化电位低 160 mV。底物被氢化木醋醌代替之后酶活性显著提高且在酸性pH 值为4~8时也能够检测到,得到了更稳定的检测条件和更高的灵敏度[36]。毛相朝等[37]用该方法检测经过纯化后EbLPMO10A的酶活性,达到了356.36 U/g,同时测得该酶的最适温度与pH值分别为60 ℃和7。
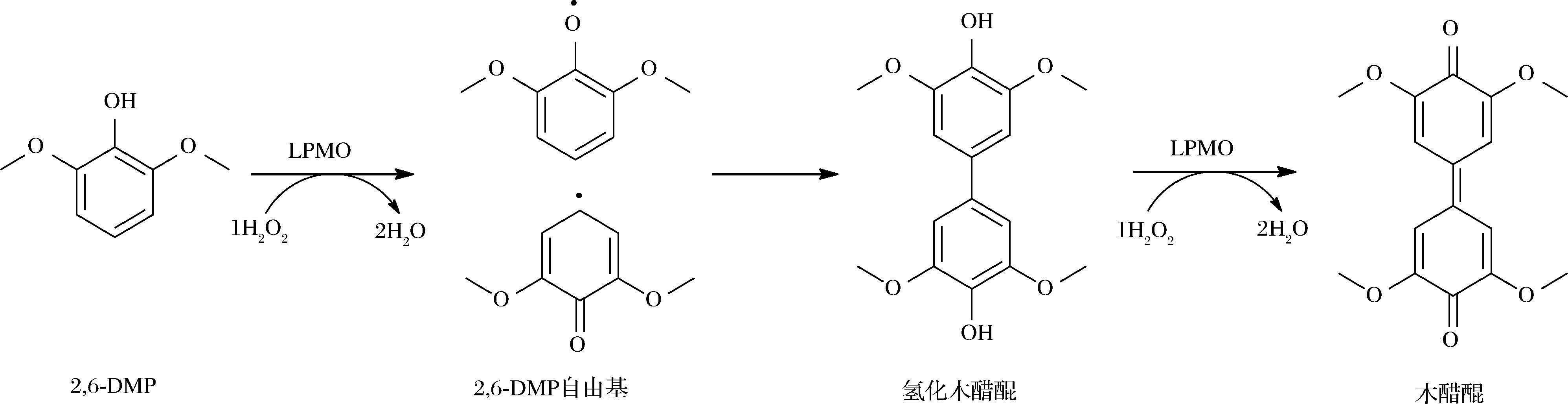
图4 2,6-DMP法测LPMO酶活性原理
Fig.4 Principle of LPMO enzyme activity measurement using 2,6-DMP method
4.3 Amplex Red辣根过氧化物酶法
Amplex Red是一种很好的过氧化物酶底物,在检测H2O2中具有很高的灵敏度和稳定性。在过氧化物酶催化下,Amplex Red试剂与H2O2按1∶1的定量比例发生反应,生成具有红色荧光的氧化产物试卤灵对其进行荧光强度的测定,间接表示LPMO活性。这一方法是以测定H2O2的产量来表征 LPMOs的活力。方法灵敏度很高且操作方便快捷在测定LPMO活性方面有着广泛的应用[38]。用 Amplex Red辣根试剂盒测定 LPMO活性,结果表明[39],H2O2的浓度在0.25~5 μmol/L和5 μmol/L辣根过氧化物酶的条件下,构建H2O2标准曲线且相关性很好(R2=0.99)。后续研究发现[35]测定LPMO为20~574 mg/L时有着比较准确的结果,实验过程中若存在游离态铜离子会对于结果造成影响[40]。
5 裂解多糖单加氧酶的克隆与表达
LPMO资源丰富,但其生产效率低、分离困难。为进一步提高 LPMO的产量,促进其产业化,通过在其他寄主中进行异源表达,是一条行之有效的途径。由于AA9基因来源于真菌,毕赤酵母是应用最为广泛的一种真核表达体系,由于其基因操纵技术的成熟,已经被广泛应用于异源表达AA9[41]。除毕赤酵母外,已有部分AA9基因在其他真菌的表达载体上进行了改造,但仍面临着转化困难、操作难度大等问题。
为进一步提高AA9的产量,通过在真菌表达宿主中构建了一系列的信号肽来实现AA9的高效表达,表达载体为毕赤酵母最常见的信号肽是α-因子信号肽和AA9 的原生信号肽。LADEV ZE等[42]在毕赤酵母中使用2种信号肽表达3种蛋白,结果表明由原始信号肽分泌的LPMO含有更多正确的N端His以及3种采用原生信号肽的酶的比活均大于α-因子信号肽。来自红侧耳Pleurotus djamor的PdLPMO,通过添加组氨酸标签、基因密码子优化、增加基因剂量以及联合共表达分子伴侣蛋白等方法,使构建菌株蛋白含量与酶活性提高75%和66.92%[43]。AA10大部分来源于细菌,目前主要是在芽孢杆菌和大肠杆菌中进行异源表达。有些AA10利用它们自身的信号肽表达,有些则利用其他蛋白的信号肽,AA10对不同信号肽的作用效果存在差异。在对比不同AA10 的信号肽中,SmAA10A的信号肽相比其他蛋白的信号肽有着更强的优势,还能实现多种蛋白的高效表达[44]。徐倩倩[45]在构建 CsLPMO的异源表达过程中,得出pET-22b为最优的表达质粒,后续对其菌株表达条件优化后可溶性蛋白含量与酶活性分别提高 32.1%和45.2%。
ZE等[42]在毕赤酵母中使用2种信号肽表达3种蛋白,结果表明由原始信号肽分泌的LPMO含有更多正确的N端His以及3种采用原生信号肽的酶的比活均大于α-因子信号肽。来自红侧耳Pleurotus djamor的PdLPMO,通过添加组氨酸标签、基因密码子优化、增加基因剂量以及联合共表达分子伴侣蛋白等方法,使构建菌株蛋白含量与酶活性提高75%和66.92%[43]。AA10大部分来源于细菌,目前主要是在芽孢杆菌和大肠杆菌中进行异源表达。有些AA10利用它们自身的信号肽表达,有些则利用其他蛋白的信号肽,AA10对不同信号肽的作用效果存在差异。在对比不同AA10 的信号肽中,SmAA10A的信号肽相比其他蛋白的信号肽有着更强的优势,还能实现多种蛋白的高效表达[44]。徐倩倩[45]在构建 CsLPMO的异源表达过程中,得出pET-22b为最优的表达质粒,后续对其菌株表达条件优化后可溶性蛋白含量与酶活性分别提高 32.1%和45.2%。
LPMO家族的异源表达,主要表达宿主为大肠杆菌和毕赤酵母。从真菌中分离得到的AA9在毕赤酵母中比大肠杆菌更容易得到高产。与此相反,AA10主要来自于细菌,在大肠杆菌中表达较好。
6 裂解多糖单加氧酶的应用
6.1 功能性食品
功能食品作为一种具有保健、防治疾病等功效的食品,近年来日益受到人们的重视。低聚果糖、低聚半乳糖等低聚糖作为益生元添加到食品中开发成功能性食品,不仅可以改善产品口感,而且具有调控肠道菌群和肠道微生态的潜力。LPMO与GH协同,可实现低聚糖的高效合成,在功能性食品领域极具潜力为人类健康提供新的思路。周頔等[46]观察低聚果糖、低聚异麦芽糖、低聚木糖、低聚半乳糖4种低聚糖类益生元分别代替蔗糖对炼乳品质影响过程中,发现糖替代比15%的低聚异麦芽糖炼乳在满足甜味的同时,又实现了减糖的目的,并且益生元的加入使其具有更多的功能性。在酸奶制作中添加乳果糖、菊粉作为益生元能够减少贮存过程中活菌数的下降提升酸奶的品质,同时有效促进嗜热乳杆菌和双歧杆菌等益生菌的增殖,从而改善肠道健康[47]。姜雅杰等[48]开发的功能性壳寡糖复合固体饮料能够改善糖尿病小鼠的肠道微生物群落结构,促进益生菌的增殖,减少病原菌,改善肠道健康,同时还能帮助小鼠降脂和稳定血糖。燕麦奶含有丰富的益生元低聚果糖,摄入后可提高肠道内短链脂肪酸(丁酸盐、乙酸盐、丙酸盐)的生成,降低肠道 pH、氮终产物和粪便酶的量,并提高机体免疫力,促进骨生长[49]。
6.2 饲料添加剂
抗生素在动物饲料中的应用,不仅可以预防动物疾病,提高动物的健康水平,而且可以提高饲料利用率,还可以提高肉、蛋、奶的产量,大大推动了我国畜牧业的迅速发展。与此同时,也引发了许多新的问题,比如在饲料中加入抗生素,所带来的药物残留危害非常明显,因为抗生素的长期使用,会使致病菌的抗药性变得更强,从而形成具有耐药性的超级细菌。与此同时,还会对周围的环境造成污染,进而对人体的健康造成威胁,同时2020年起全面禁止使用抗生素,人们开始寻找安全、有效的无抗饲料[50]。随着人们的生活水平的不断提升,食品安全问题受到了越来越多的重视,众多益生菌、酶制剂如LMPO等添加到饲料中应运而生[51]。
非淀粉多糖(non-starch polysaccharides,NSP)是除淀粉外的一种多糖,其在饲料中具有显著的抗营养作用,其在水中溶解后具有较高的黏度。然而,传统饲料配料中含有的非淀粉多糖等抗营养因子及有毒物质,使其营养价值显著降低,限制了其在生猪生产中的应用。研究发现[52-53],添加非淀粉多糖复合酶能显著地提高了猪对饲料的吸收利用能力,并使其达到了较好的增重效果,同时也使总能量的表观消化率得到了很大的提高。因此,在饲料中添加 LPMO及非淀粉多糖酶,可有效地降解NSP,解除NSP的抗营养作用[54]。同时还可产生功能寡糖类和其他有益物质,纤维寡糖作为纤维素的主要产物,聚合度为2~6的可溶性纤维寡糖可以促进双歧杆菌(Bifidobacteria)、副干酪乳杆菌(Lactobacillusparacei)等肠道内益生菌的生长繁殖,不仅增强人体抵抗力,还改善肠道菌群结构和肠道发育以及使食糜黏度下降从而降低畜禽胃肠疾病的发生,可作为优良的家畜饲料促进畜禽的健康生长[55]。
6.3 生物能源
以木质纤维为原料的生物质资源是继煤炭、石油、天然气之后的一种新的可再生能源,是缓解人类日益严重的环境问题的重要途径。木质纤维素经降解发酵可转化为生物酒精,该过程分为2个步骤:首先将纤维素分解成单糖,再将单糖进行发酵,而这一过程的核心是降解为单糖阶段。因此,开发廉价高效的木质纤维素降解酶,为其高效转化利用提供理论依据和技术支持具有重要意义。R17LPMO和纤维素酶的协同作用对经过蒸汽爆破预处理过的玉米秸秆降解效果显著提高,数据表示200 ℃预处理玉米秸秆3 min效果最好[56]。同样的几丁质在生物能源方面的利用和木质纤维素有着相同的处境,自然界中存在量大但是降解为人们所能利用是一个比较棘手的问题。研究表明具有几丁质降解能力菌株(Chitiniphilus sp. LY72)的裂解多糖单加氧酶(CsLPMO)和几丁质水解酶之间有着协同的辅助性,可以有效地提高对固体几丁质的水解效率,而且CsLPMO用量越大,对几丁质降解的辅助性作用越显著[57]。源于Natrialbaceae archaeon中的NaLPMO10A作为甲壳素的C1氧化剂在温度40 ℃、pH 9.0的条件下对几丁质的糖化效率达到最高,同时也可以在高浓度粒子的极端条件下提升糖化[58]。
7 结论和展望
生物质是继煤炭、石油和天然气之后最重要的一种可再生能源,是解决全球气候变化、环境污染和能源危机的重要途径。在这些物质中,木质纤维素和几丁质的储量都比较大,它们可以被用来转换成绿色的生物燃料和高附加值的化工原料,其高值化和资源化对于维持地球碳循环以及生物炼制行业的发展都有着非常重要的意义。LPMO可通过氧化性断裂破坏纤维素晶体结构,增加糖苷水解酶的结合位点,是实现纤维素高效酶解的关键,是一种很有发展前景的纤维素降解酶。LPMO的发现也让人们对降解结晶多糖有了新的认识,通过其与GH协同作用实现功能性低聚糖的合成,为功能性食品的开发及应用提供帮助。并且在饲料中添加LPMO和相关酶制剂可以降低非淀粉多糖等抗营养因子带来的不利影响,保障畜禽的健康生长。虽然LPMO在降解木质纤维素和几丁质等多糖中表现出较好的效果,但是在工业上产量、高活力以及稳定性等方面还存在不少的挑战,相信在不久的将来可以解决这个问题,进一步推动其在功能性食品、饲料添加剂及生物燃料等方面的应用。
[1] 马钦元, 申雁冰, 丁盼盼, 等. 常压室温等离子诱变与微生物微滴培养选育几丁质脱乙酰基酶高产菌株[J]. 中国酿造, 2020, 39(8):170-174. MA Q Y, SHEN Y B, DING P P, et al. Breeding of strain with high chitin deacetylase yield by atmospheric and room temperature plasma mutagenesis and microbial microdroplet-culture[J]. China Brewing, 2020, 39(8):170-174.
[2] SHIMIZU M, KANEKO Y, ISHIHARA S, et al. Novel β-1, 4-mannanase belonging to a new glycoside hydrolase family in Aspergillus nidulans[J]. The Journal of Biological Chemistry, 2015, 290(46):27914-27927.
[3] HILL J L Jr, HAMMUDI M B, MING T E. The Arabidopsis cellulose synthase complex: A proposed hexamer of CESA trimers in an equimolar stoichiometry[J]. The Plant Cell, 2014, 26(12):4834-4842.
[4] HEMSWORTH G R, JOHNSTON E M, DAVIES G J, et al. Lytic polysaccharide monooxygenases in biomass conversion[J]. Trends in Biotechnology, 2015, 33(12):747-761.
[5] SATO K, CHIBA D, YOSHIDA S, et al. Functional analysis of a novel lytic polysaccharide monooxygenase from Streptomyces griseus on cellulose and chitin[J]. International Journal of Biological Macromolecules, 2020, 164:2085-2091.
[6] SUZUKI K, SUZUKI M, TAIYOJI M, et al. Chitin binding protein (CBP21) in the culture supernatant of Serratia marcescens 2170[J]. Bioscience, Biotechnology, and Biochemistry, 1998, 62(1):128-135.
[7] AACHMANN F L, SØRLIE M, SKJÅK-BR☞K G, et al. NMR structure of a lytic polysaccharide monooxygenase provides insight into copper binding, protein dynamics, and substrate interactions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(46):18779-18784.
[8] HARRIS P V, WELNER D, MCFARLAND K C, et al. Stimulation of lignocellulosic biomass hydrolysis by proteins of glycoside hydrolase family 61: Structure and function of a large, enigmatic family[J]. Biochemistry, 2010, 49(15):3305-3316.
[9] CANTAREL B L, COUTINHO P M, RANCUREL C, et al. The Carbohydrate-Active EnZymes database (CAZy): An expert resource for Glycogenomics[J]. Nucleic Acids Research, 2009, 37(Database issue): D233-D238.
[10] GARCIA-SANTAMARINA S, PROBST C, FESTA R A, et al. A lytic polysaccharide monooxygenase-like protein functions in fungal copper import and meningitis[J]. Nature Chemical Biology, 2020, 16(3):337-344.
[11] PASPALIARI D K, LOOSE J S M, LARSEN M H, et al. Listeria monocytogenes has a functional chitinolytic system and an active lytic polysaccharide monooxygenase[J]. The FEBS Journal, 2015, 282(5):921-936.
[12] VANDHANA T M, REYRE J L, SUSHMAA D, et al. On the expansion of biological functions of lytic polysaccharide monooxygenases[J]. The New Phytologist, 2022, 233(6):2380-2396.
[13] FRANCO CAIRO J P L, CANNELLA D, OLIVEIRA L C, et al. On the roles of AA15 lytic polysaccharide monooxygenases derived from the termite Coptotermes gestroi[J]. Journal of Inorganic Biochemistry, 2021, 216:111316.
[14] SABBADIN F, URRESTI S, HENRISSAT B, et al. Secreted pectin monooxygenases drive plant infection by pathogenic oomycetes[J]. Science, 2021, 373(6556):774-779.
[15] FILIATRAULT-CHASTEL C, NAVARRO D, HAON M, et al. AA16, a new lytic polysaccharide monooxygenase family identified in fungal secretomes[J].Biotechnology for Biofuels, 2019, 12:55.
[16] BHATIA S, YADAV S K. Novel catalytic potential of a hyperthermostable mono-copper oxidase (LPMO-AOAA17) for the oxidation of lignin monomers and depolymerisation of lignin dimer in aqueous media[J]. International Journal of Biological Macromolecules, 2021, 186:563-573.
[17] QUINLAN R J, SWEENEY M D, LEGGIO L L, et al. Insights into the oxidative degradation of cellulose by a copper metalloenzyme that exploits biomass components[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(37):15079-15084.
[18] FRANDSEN K E H, SIMMONS T J, DUPREE P, et al. The molecular basis of polysaccharide cleavage by lytic polysaccharide monooxygenases[J]. Nature Chemical Biology, 2016, 12(4):298-303.
[19] VAAJE-KOLSTAD G, FORSBERG Z, LOOSE J S, et al. Structural diversity of lytic polysaccharide monooxygenases[J]. Current Opinion in Structural Biology, 2017, 44:67-76.
[20] COURTADE G, AACHMANN F L. Chitin-Active Lytic Polysaccharide Monooxygenases[M].Singapore: Springer, 2019:115-129.
[21] GREGORY R C, HEMSWORTH G R, TURKENBURG J P, et al. Activity, stability and 3-D structure of the Cu(ii) form of a chitin-active lytic polysaccharide monooxygenase from Bacillus amyloliquefaciens[J]. Dalton Transactions, 2016, 45(42):16904-16912.
[22] TIAN C G, BEESON W T, IAVARONE A T, et al. Systems analysis of plant cell wall degradation by the model filamentous fungus Neurospora crassa[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(52):22157-22162.
[23] BEESON W T, VU V V, SPAN E A, et al. Cellulose degradation by polysaccharide monooxygenases[J]. Annual Review of Biochemistry, 2015, 84:923-946.
[24] TAN T C, KRACHER D, GANDINI R, et al. Structural basis for cellobiose dehydrogenase action during oxidative cellulose degradation[J]. Nature Communications, 2015, 6:7542.
[25] EIBINGER M, GANNER T, BUBNER P, et al. Cellulose surface degradation by a lytic polysaccharide monooxygenase and its effect on cellulase hydrolytic efficiency[J]. The Journal of Biological Chemistry, 2014, 289(52):35929-35938.
[26] JAGADEESWARAN G, GAINEY L, MORT A J. An AA9-LPMO containing a CBM1 domain in Aspergillus nidulans is active on cellulose and cleaves cello-oligosaccharides[J]. AMB Express, 2018, 8(1):171.
[27] MEIER K K, JONES S M, KAPER T, et al. Oxygen activation by Cu LPMOs in recalcitrant carbohydrate polysaccharide conversion to monomer sugars[J]. Chemical Reviews, 2018, 118(5):2593-2635.
[28] KUUSK S, BISSARO B, KUUSK P, et al. Kinetics of H2O2-driven degradation of chitin by a bacterial lytic polysaccharide monooxygenase[J]. The Journal of Biological Chemistry, 2018, 293(2):523-531.
[29] 宋晓菲, 冯超. 裂解性多糖单加氧酶及其应用研究进展[J]. 微生物学报, 2023, 63(7):2534-2551. SONG X F, FENG C. Lytic polysaccharide monooxygenase and its application[J]. Acta Microbiologica Sinica, 2023, 63(7):2534-2551.
[30] 杜文珍,李元敬,吴佳玲,等.丝状真菌Podospora anserina AA11 家族裂解多糖单加氧酶基因的鉴定和功能研究[J].遗传, 2023, 45(12):1128-1146. DU W Z, LI Y J, WU J L, et al. Identification and functional study of AA11 family polysaccharide monooxygenase genes in filamentous fungus Podospora anserina[J]. Hereditas, 2023, 45(12):1128-1146.
[31] LI D, GAI Y P, MENG J L, et al. GPI-anchored protein homolog IcFBR1 functions directly in morphological development of Isaria cicadae[J]. Journal of Fungi, 2022, 8(11):1152.
[32] MUMMERY-WIDMER J L, YAMAZAKI M, STOEGER T, et al. Genome-wide analysis of Notch signalling in Drosophila by transgenic RNAi[J]. Nature, 2009, 458(7241):987-992.
[33] LI Y, LIU X Y, LIU M X, et al. Magnaporthe oryzae auxiliary activity protein MoAa91 functions as chitin-binding protein to induce appressorium formation on artificial inductive surfaces and suppress plant immunity[J]. mBio, 2020, 11(2): e03304-e03319.
[34] 孙小宝, 万嘉欣, 曹佳雯, 等. 溶解性多糖单加氧酶的研究进展[J]. 生物工程学报, 2018, 34(2):177-187. SUN X B, WAN J X, CAO J W, et al. Progress in lytic polysaccharide monooxygenase[J]. Chinese Journal of Biotechnology, 2018, 34(2):177-187.
[35] BRESLMAYR E, HANŽEK M, HANRAHAN A, et al. A fast and sensitive activity assay for lytic polysaccharide monooxygenase[J]. Biotechnology for Biofuels, 2018, 11:79.
[36] BRESLMAYR E, DALY S, ![]() A, et al. Improved spectrophotometric assay for lytic polysaccharide monooxygenase[J]. Biotechnology for Biofuels, 2019, 12:283.
A, et al. Improved spectrophotometric assay for lytic polysaccharide monooxygenase[J]. Biotechnology for Biofuels, 2019, 12:283.
[37] 毛相朝, 郭超然, 苏海鹏, 等. 裂解性多糖单加氧酶EbLPMO10A及其应用: CN116042549A[P]. 2023-05-02.
[38] 沈洁如, 王素英, 张宏宇, 等. 裂解多糖单加氧酶活性检测方法的研究进展[J]. 分析化学, 2023, 51(9):1391-1402. SHEN J R, WANG S Y, ZHANG H Y, et al. Research progress on detection methods of lytic polysaccharide monooxygenase activities[J]. Chinese Journal of Analytical Chemistry, 2023, 51(9):1391-1402.
[39] LIMSAKUL P, PHITSUWAN P, WAEONUKUL R, et al. A novel AA10 from Paenibacillus curdlanolyticus and its synergistic action on crystalline and complex polysaccharides[J]. Applied Microbiology and Biotechnology, 2020, 104(17):7533-7550.
[40] ![]() D M, V
D M, V RNAI A, DIMAROGONA M, et al. Comparison of three seemingly similar lytic polysaccharide monooxygenases from Neurospora crassa suggests different roles in plant biomass degradation[J]. The Journal of Biological Chemistry, 2019, 294(41):15068-15081.
RNAI A, DIMAROGONA M, et al. Comparison of three seemingly similar lytic polysaccharide monooxygenases from Neurospora crassa suggests different roles in plant biomass degradation[J]. The Journal of Biological Chemistry, 2019, 294(41):15068-15081.
[41] JAGADEESWARAN G, GAINEY L, MORT A J. An AA9-LPMO containing a CBM1 domain in Aspergillus nidulans is active on cellulose and cleaves cello-oligosaccharides[J]. AMB Express, 2018, 8(1):171.
[42] LADEV ZE S, HAON M, VILLARES A, et al. The yeast Geotrichum candidum encodes functional lytic polysaccharide monooxygenases[J]. Biotechnology for Biofuels, 2017, 10:215.
ZE S, HAON M, VILLARES A, et al. The yeast Geotrichum candidum encodes functional lytic polysaccharide monooxygenases[J]. Biotechnology for Biofuels, 2017, 10:215.
[43] 郭俊彤. 裂解多糖单加氧酶基因的高效异源表达研究[D]. 长春: 吉林农业大学, 2023. GUO J T. study on efficient heterologous expression of monooxygenase gene from pyrolysaccharide[D].Changchun: Jilin Agricultural University, 2023.
[44] COURTADE G, LE S B, S☞TROM G I, et al. A novel expression system for lytic polysaccharide monooxygenases[J]. Carbohydrate Research, 2017, 448:212-219.
[45] 徐倩倩. 几丁质高效降解菌株的筛选及裂解性多糖单加氧酶的应用研究[D]. 聊城: 聊城大学, 2022. XU Q Q. Screening of chitin-degrading strains with high efficiency and study on the application of pyrolytic polysaccharide monooxygenase[D].Liaocheng: Liaocheng University, 2022.
[46] 周頔, 徐登薇, 宋锐, 等. 低聚糖类益生元对炼乳品质的影响[J]. 食品安全质量检测学报, 2022, 13(18):5887-5894. ZHOU D, XU D W, SONG R, et al. Effects of oligosaccharide prebiotics on the quality of condensed milk[J]. Journal of Food Safety &Quality, 2022, 13(18):5887-5894.
[47] 何君, 韩育梅, 刘敏, 等. 菊粉和低聚果糖对发酵乳品质的影响[J]. 中国食品学报, 2019, 19(11):116-123. HE J, HAN Y M, LIU M, et al. Effect of inulin and oligofructose on the quality of fermented milk[J]. Journal of Chinese Institute of Food Science and Technology, 2019, 19(11):116-123.
[48] 姜雅杰, 王畅, 席茂盛, 等. 壳寡糖复合固体饮料对Ⅱ型糖尿病小鼠肠道菌群结构的影响[J]. 食品工业科技, 2020, 41(8):301-306. JIANG Y J, WANG C, XI M S, et al. Effect of chitooligosaccharide compound solid beverage on intestinal flora of type II diabetic mice[J]. Science and Technology of Food Industry, 2020, 41(8):301-306.
[49] MARKOWIAK P, ![]() K. Effects of probiotics, prebiotics, and synbiotics on human health[J]. Nutrients, 2017, 9(9):1021.
K. Effects of probiotics, prebiotics, and synbiotics on human health[J]. Nutrients, 2017, 9(9):1021.
[50] 倪学勤. 畜牧养殖开启无抗新时代[J]. 畜牧产业, 2020(8):31. NI X Q. Animal husbandry opens a new era of no resistance[J]. Animal Agriculture, 2020(8):31.
[51] 邓雪娟, 于继英, 刘晶晶, 等. 我国生物发酵饲料研究与应用进展[J]. 动物营养学报, 2019, 31(5):1981-1989. DENG X J, YU J Y, LIU J J, et al. Research and application progress of biological fermented feed in China[J]. Chinese Journal of Animal Nutrition, 2019, 31(5):1981-1989.
[52] 敖翔, 陶璇, 豆松方, 等. 不同抗生素替代组合对川藏黑猪断奶仔猪生长性能和养分消化率的影响[J]. 养猪, 2022(3):34-36. AO X, TAO X, DOU S F, et al. Effects of antibiotic alternatives on growth performance and nutrient digestibility in Chuanzang black weaning pigs[J]. Swine Production, 2022(3):34-36.
[53] 陈铁中. 高产非淀粉多糖酶的菌株选育及应用[D]. 太原: 山西大学, 2023. CHEN T Z. Breeding and application of high-yielding non-starch polysaccharide enzyme strains[D]. Taiyuan: Shanxi University, 2023.
[54] 余建, 范志勇, 王丽, 等. 小麦中抗营养因子及其猪饲用价值改善方法研究进展[J]. 动物营养学报, 2023, 35(8):4777-4794. YU J, FAN Z Y, WANG L, et al. Research progress on anti-nutritional factors in wheat and methods improving its feeding value in pigs[J]. Chinese Journal of Animal Nutrition, 2023, 35(8):4777-4794.
[55] 郑棚, 王雷,胡美荣,等.多酶级联反应合成能够促进肠道益生菌生长的纤维寡糖[J]. 生物工程学报, 2023, 39(8):3406-3420. ZHENG P, WANG L, HU M R, et al. Synthesis of cello-oligosaccharides which promotes the growth of intestinal probiotics by multi-enzyme cascade reaction[J]. Chinese Journal of Biotechnology, 2023, 39(8):3406-3420.
[56] GUO X, AN Y J, LU F P, et al. Optimization of synergistic degradation of steam exploded corn straw by lytic polysaccharide monooxygenase R17L and cellulase[J]. Industrial Crops and Products, 2022, 182:114924.
[57] 徐倩倩, 马春桐, 姚莹莹, 等. 几丁质裂解性多糖单加氧酶的表达及应用研究[J]. 中国酿造, 2022, 41(8):97-104. XU Q Q, MA C T, YAO Y Y, et al. Expression and application of chitinolytic polysaccharide monooxygenase[J]. China Brewing, 2022, 41(8):97-104.
[58] LI F, LIU Y X, LIU Y, et al. Heterologous expression and characterization of a novel lytic polysaccharide monooxygenase from Natrialbaceae archaeon and its application for chitin biodegradation[J]. Bioresource Technology, 2022, 354:127174.