谷氨酰胺转氨酶(transglutaminase, TGase, EC 2.3.2.13)是一种酰基转移酶,可催化蛋白质交联,在肉制品、乳制品和烘焙等食品领域应用广泛[1-2]。随着对TGase性质认识的加深,其应用领域拓展至制药、纺织和皮革等非食品领域,并展现出独特的应用优势[3]。相较于动植物来源的TGase,茂原链霉菌(Streptomyces mobaraensis)来源的TGase具有非Ca2+依赖性、安全性高和生产成本低等明显优势[4],是商品化TGase的主要来源[2]。天然的S.mobaraensis TGase热稳定性较差,其60 ℃半衰期仅为2 min[5]。然而,TGase面临高温应用环境,如牛肉加工中的蛋白交联反应需在60 ℃下进行[6]。因此,TGase热稳定改造一直都是国内外研究的热点。
为提高S.mobaraensis TGase热稳定性,研究者通过饱和突变、基因改组以及组合突变[7-8]得到突变体smTG-S2P-S23V-Y24N-S199A-K294L(TGm1),其60 ℃半衰期和比酶活力分别提升至24.8 min和55.7 U/mg[5]。前期研究中,通过对TGm1进行脯氨酸扫描和活性中心改造构建得到突变体FRAPD-TGm1-E28T-A265P-A287P(FRAPD-TGm2),60 ℃半衰期和比酶活力较TGm1提升2.04倍和43.8%;通过提高酶分子表面静电荷得到FRAPD-TGm2-N96E-S144E-N163D-R183E-R208E-K325E(FRAPD-TGm3),其60 ℃半衰期和比酶活力分别提高至122.91 min和83.74 U/mg[9]。然而,可能由于酶分子表面静电荷的增加也增强其与蛋白类底物斥力,FRAPD-TGm3对蛋白交联效率远低于FRAPD-TGm2[9],不利于其实际应用。因此,需采用新的改造策略提升FRAPD-TGm2热稳定性。
研究表明,酶分子底物结合口袋的刚性对其热稳定性有重要影响[10]。然而,底物结合口袋的柔性与催化活性密切相关,尤其对于大分子底物[11-12]。因此,以底物结合口袋为靶点的热稳定性改造可能使酶催化活性显著下降,通常依赖于大量的饱和突变[10,13]。基于分子动力学模拟的动态互相关性分析已被用于识别蛋白质分子中的远端相互作用[14]。通过改造与底物结合口袋有远端相互作用的位点,可能避免直接对底物口袋改造的不利影响。此外,基于降低折叠自由能的理性设计成为最受关注的酶热稳定性改造策略之一。目前,研究者开发了一系列折叠自由能预测工具(FoldX,Rosetta Cartesian_ddg,IMutant和Eris等[15])用于指导工业酶耐热突变体的筛选[16]。其中,Rosetta Cartesian_ddg预测的折叠自由能变与蛋白质热稳定性的皮尔森相关性系数达到0.743[17]。因此,结合动态互相关性与折叠自由能分析,有望为TGase的热稳定性提升提供新的靶点。
在本研究中,基于动态互相关性和折叠自由能分析对FRAPD-TGm2的热稳定性进行了改造。首先,通过分子动力学模拟和动态互相关性分析确定了FRAPD-TGm2底物口袋柔性残基具有动态互相关性的残基。其次,基于Rosetta Cartesian_ddg对上述残基位点进行虚拟饱和突变,筛选折叠自由能显著下降的FRAPD-TGm2突变体。接下来,对上述突变体进行表达、纯化和酶学性质表征。最后,基于分子动力学模拟解析了突变体热稳定提高的机制。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
大肠杆菌(Escherichia coli)BL21(DE3)用于表达FRAPD-TGm2及其突变体,由本实验保存。pET-22b/TrxA-ProC-TGm2是pET-22b(+)衍生质粒,编码了硫氧还蛋白(TrxA)和融合Streptomyces caniferus来源的TGase酶原(proC)的TGm2[9]。
1.1.2 试剂
MutanBEST Kit试剂盒,Takara公司;中性蛋白酶,Solarbio公司;CBZ-Gln-Gly,Sigma-Aldrich公司;β-酪蛋白,Macklin公司;Bradford蛋白浓度检测试剂盒,Beyotime公司;一步克隆试剂盒(Vazyme ClonExpress Ⅱ One Step Cloning Kit C112),Vazyme公司;5 mL 镍离子亲和层析柱、5 mL G-25凝胶层析柱,GE公司;蛋白电泳Loading buffer、Bis-Tris预制凝胶,Invitrogen公司;其他生物学试剂,国药集团。
1.1.3 培养基与缓冲液
LB培养基(g/L):酵母提取物5,蛋白胨10,NaCl 5,固体培养基添加质量分数2%的琼脂粉。
TB培养基(g/L):酵母提取物24,蛋白胨12,K2HPO4 12.54,KH2PO4 2.31,甘油5。
TGase活化反应液:50 mmol/L Tris-HCl,100 mmol/L NaCl,2 mmol/L CaCl2,1 mmol/L还原型谷胱甘肽,200 g/L中性蛋白酶,调节pH值至7.0。
纯化缓冲液A:50 mmol/L Tris-HCl,200 mmol/L NaCl,25 mmol/L咪唑,调节pH值至7.9。
纯化缓冲液B:50 mmol/L Tris-HCl,200 mmol/L NaCl,500 mmol/L咪唑,调节pH值至7.9。
脱盐缓冲液C:50 mmol /L Tris-HCl,调节pH值至8.0。
TGase活力测定底物:Tris-HCl 200 mmol/L,羟胺100 mmol/L,还原型谷胱甘肽10 mmol/L,CBZ-Gln-Gly 30 mmol/L,调节pH值至6.0。
TGase酶活力测定终止剂:将120 g/L三氯乙酸、3 mmol/L HCl、50 g/L FeCl3·6H2O(溶于0.1 mol/L HCl)等体积混合均匀,避光保存。
酪蛋白溶液:3 mg/mL β-酪蛋白溶于100 mmol/L Tris-醋酸缓冲液,调节pH值至7.0。
1.2 仪器与设备
TC5000 PCR仪,Eppendorf(德国)公司;DYY-6D 核酸电泳仪,北京六一生物科技有限公司;GelDoc凝胶成像仪、蛋白电泳仪,Bio-Rad(美国)公司;LS-35LJ 立式自动压力蒸汽灭菌锅,上天精密仪器(上海)公司;ZQZY-HC恒温摇床,上海知楚仪器公司;实验型高压匀质机,永联生物科技(上海)有限公司;UV-2450 分光光度计,Shimadzu(日本)公司;AKTA 蛋白纯化仪,GE(美国)公司;320-R 冷冻离心机、CT15RE 台式高速冷冻离心机,Hitachi(日本)公司;恒温水浴锅,Thermo Scientific(美国)公司。
1.3 实验方法
1.3.1 FRAPD-TGm2及其突变体质粒的构建
化学合成编码TGm2、TrxA和proC的融合基因片段TrxA-proC-TGm2,并将其克隆到pET-22b(+)(GENWIZ,苏州,中国)的Nde I-Blp I位点上,得到质粒pET-22b/TrxA-proC-TGm2。按照MutanBEST Kit试剂盒生产商的说明,以pET-22b/TrxA-proC-TGm2质粒为模板通过PCR构建了50个FRAPD-TGm2突变体。所用引物见表1。
表1 氨基酸突变相关引物
Table 1 The primers for amino acid mutagenesis

突变引物名称引物序列(5′-3′)Y34WY34W-FgaacaactggattcgcaaatggcagcaagtgtY34W-RcgaatccagttgttcaccacggtggtcgcR215LR215L-FgcggataaactgaaatatggcgatccggatgcgtttR215-RcatatttcagtttatccgcgctactgctgcgatctR98QR98Q-FcggccaaccgcgcagcggcgaaaR98Q-RctgcgcggttggccgtttttcagttcgtttttaaagcgatcttR215MR215M-FgcggataaaatgaaatatggcgatccggatgcgtttR215M-RatttcattttatccgcgctactgctgcgatctS210QS210Q-FatcgcagccaaagcgcggataaacgcaaatatggS210Q-RcgctttggctgcgatcttggccgctcH201LH201L-FcgaaactgttttggagcggccaagatcgcagH201L-RtccaaaacagtttcgcataaatcaccgctttcatgcggR208TR208T-FccaagataccagcagtagcgcggataaacgcaaR208T-RactgctggtatcttggccgctccaaaaatgtttcgcataaaR208QR208Q-FgccaagatcagagcagtagcgcggataaacgcaaaR208Q-RctgctctgatcttggccgctccaaaaatgtttcgcataaaS210DS210D-FcgcagcgatagcgcggataaacgcaaatatggS210D-RcgcgctatcgctgcgatcttggccgctcK216RK216R-FgccgctatggcgatccggatgcgtttK216R-RgatcgccatagcggcgtttatccgcgctactgctG205AG205A-FcgcgcaagatcgcagcagtagcgcggaG205A-RtgcgatcttgcgcgctccaaaaatgtttcgcataaatcaccgcttH201YH201Y-FatgcgaaatatttttggagcggccaagatcgcagH201Y-RctccaaaaatatttcgcataaatcaccgctttcatgcggD213WD213W-FcgcgtggaaacgcaaatatggcgatccggaD213W-RtttgcgtttccacgcgctactgctgcgatcttD213LD213L-FcgcgctgaaacgcaaatatggcgatccggaD213L-RtttgcgtttcagcgcgctactgctgcgatcttgH201FH201-FgcgaaatttttttggagcggccaagatcgcagH201F-RgctccaaaaaaatttcgcataaatcaccgctttcatgcggH201WH201W-FcgaaatggttttggagcggccaagatcgcagH201W-RctccaaaaccatttcgcataaatcaccgctttcatgcggH201IH201I-FcgaaaattttttggagcggccaagatcgcagcagH201I-RcgctccaaaaaattttcgcataaatcaccgctttcatgcggH201MH201M-FcgaaaatgttttggagcggccaagatcgcagH201M-RctccaaaacattttcgcataaatcaccgctttcatgcggN32IN32I-FcgtggtgattaactatattcgcaaatggcagcaagtgtN32I-RtatagttaatcaccacggtggtcgcgcggN32VN32V-FggtggtgaactatattcgcaaatggcagcaagtgtatagccN32V-RatatagttcaccaccacggtggtcgcgcggN32M N32QN32R N32WN32Y N32SN32L N32PN32A N32DN32G N32KN32T32 NNK-F32 NNK-RcgtggtgnnkaactatattcgcaaatggcagcaagtgttatagttmnncaccacggtggtcgcgcggN32CN32C-FccgtggtgtgcaactatattcgcaaatggcagcaagtgtN32C-RtagttgcacaccacggtggtcgcgcggN32EN32E-FccgtggtggaaaactatattcgcaaatggcagcaagtgtN32E-RtagttttccaccacggtggtcgcgcggN32FN32F-FccgtggtgtttaactatattcgcaaatggcagcaagtgtN32F-RtagttaaacaccacggtggtcgcgcggN32HN32H-FccgtggtgcataactatattcgcaaatggcagcaagtgtN32H-RtagttatgcaccacggtggtcgcgcggH201E H201GH201K H201NH201Q H201RH201S H201TH201V H201P 201 NNK-F201 NNK-RgaaannkttttggagcggccaagatcgcagcgctccaaaamnntttcgcataaatcaccgctttcatgcggH201AH201A-FgaaagcgttttggagcggccaagatcgcagH201A-RcgctccaaaacgctttcgcataaatcaccgctttcatgcggH201CH201C-FgcgaaatgcttttggagcggccaagatcgcagH201C-RcgctccaaaagcatttcgcataaatcaccgctttcatgcggH201DH201D-FgcgaaagatttttggagcggccaagatcgcagH201D-Rcgctccaaaaatctttcgcataaatcaccgctttcatgcgg
注:下划线表示突变位点。
1.3.2 蛋白表达
将FRAPD-TGm2及其突变体的表达质粒转化至E.coli BL21(DE3),并通过测序筛选得到对应的表达菌株。将重组E.coli接种于含有50 μg/mL氨苄青霉素的LB培养基,于37 ℃培养10 h。以1.5%的接种量将上述培养物接种至含有50 μg/mL氨苄青霉素的TB培养基,于37 ℃培养至OD600=1后加入异丙基硫代半乳糖苷(终浓度为0.1 mmol/mL),并于20 ℃诱导培养35 h。
1.3.3 蛋白纯化及浓度测定
重组E.coli经离心并弃去上清液后,使用Tris-HCl缓冲液(50 mmol/L,pH=8)将细胞重悬。采用高压均质机(Union-Biotech,上海,中国)在100 MPa的压力下破碎细胞悬液3 min,离心收集上清液。将TGase活化反应液加入上清液中至中性蛋白酶终质量浓度20 mg/mL,并在37 ℃孵育25 min。将活化后的上清液加至经纯化缓冲液A平衡后的亲和柱His-Trap HP纯化,以30%(体积分数)纯化缓冲液B进行洗脱,洗脱液用凝胶柱Superdex 75脱盐(用纯化缓冲液C平衡)。将纯化后的FRAPD-TGm2及其突变体稀释至0.4~0.6 mg/mL用于酶学性质测定。
1.3.4 残余酶活力及半衰期测定
将FRAPD-TGm2及其突变体置于特定温度(60 ℃或65 ℃)孵育,并在特定时间(5、10、15、20、30、60、90、120 min)取出。将不同时间点取出的样品置于冰上冷却后进行活性测定,计算与未经热浴处理的初始酶活力的比值,得出残余酶活力。使用Original 2018-Exponential-ExpDec1非线性拟合不同时间点的残余酶活,计算酶活力下降至50%所对应的时间,即为半衰期。
1.3.5 最适温度测定
测定了FRAPD-TGm2及其突变体在不同温度下(37、45、50、55、60、65、70 ℃)的比酶活以得到其最适反应温度。
1.3.6 动力学参数测定
配制含不同浓度的底物溶液(CBZ-Gln-Gly含量分别为3、6、9、12、15、18、21、24、27、30 mmol/L),测定37 ℃反应10 min后各底物溶液中CBZ-Gln-Gly的转化量。通过Origin 2018-Growth/Sigmodial-Hill非线性拟合获得Km和Vmax值,以酶浓度换算得到kcat值及kcat/Km值。
1.3.7 TGase酶活力测定
TGase酶活力以CBZ-Gln-Gly为底物于37 ℃反应10 min测得[18]。一个TGase酶活力单位被定义为每分钟产生1 μmol的L-谷氨酸-γ-单羟肟酸。
1.3.8 蛋白质分析
蛋白质浓度测定采用Bradford Protein Assay Kit试剂盒。SDS-PAGE检测蛋白质纯度。
1.3.9 蛋白交联活性测定
以β-酪蛋白为底物,检测了FRAPD-TGm2及其突变体对蛋白质的催化活性。在1 mL酪蛋白溶液中加入FRAPD-TGm2或突变体至终质量浓度0.01 mg/mL,于65 ℃反应不同时间(5、10、20、40 min)。SDS-PAGE用于评估交联反应效果。
1.3.10 酶模拟结构构建
采用ColabFold构建FRAPD-TGm2及其突变体模型,并使用Rosetta_Relax进行侧链构象优化[19-20]。使用Discovery Studio-2019(Dassault, Omaha, US)的Flexible Docking模块对接构建FRAPD-TGm2和CBZ-Gln-Gly的复合物构象。选取的复合物构象满足以下条件:酶活性基团Cys64与CBZ-Gln-Gly中Gln距离小于4 Å,且cDOCK Energy最低。
1.3.11 分子动力学模拟
使用GROMACS-2020[21]对酶进行分子动力学分析。主要分析条件如下:力场为FF14 sb[22];将酶分子置于填充SPC/E水模型的立方盒子内,酶与盒子边界距离为15 Å;使用Na+和Cl-平衡体系电荷,并采用最陡下坡法对系统进行能量最小化;等容等温系综和等温等体积系综各进行200 ps后解除限制。在330 K进行分子动力学模拟50 ns,计算模拟过程中均方根偏差(root-mean-square deviation, RMSD)和均方根波动(root mean square fluctuation, RMSF),并选择最终平衡构象。
1.3.12 动态互相关性分析及折叠自由能预测
使用GROMACS-2020对FRAPD-TGm2在370 K进行50 ns的分子动力学模拟3次,每次模拟的最后10 ns坐标被用于动态互相关性分析。应用R语言包Bio3D,依次计算氨基酸残基α-碳原子相对于平均位置的偏移量和不同α-碳原子偏移量之间的点积(协方差),协方差经标准化处理之后获得不同残基之间的互相关性[23]。标准协方差绝对值>0.2的2个残基被认为具有显著的互相关性。使用Rosetta Cartesian_ddG预测FRAPD-TGm2及其饱和突变体折叠自由能。FRAPD-TGm2突变体折叠自由能(ΔG)减去FRAPD-TGm2的ΔG得到折叠自由能变化(ΔΔG)。
1.4 统计学分析
所有实验均独立进行3次重复。数据以平均值±标准差的形式呈现。对样本之间的差异采用Excel(Microsoft Office 365)中的Student’s t-test进行评估。P值小于0.05被认为是具有统计学意义的差异。
2 结果与分析
2.1 与底物口袋柔性区域具有动态互相关性残基的预测及突变体制备
研究表明,通过远端效应调控底物口袋的柔性,可以实现酶的稳定性提升[13-14]。本研究拟通过动态互相关性分析,识别与底物口袋柔性区域具有显著互相关性的远端位点,并基于折叠自由能预测获得潜在的稳定性突变体。使用ColabFold和Rosetta_Relax构建FRAPD-TGm2模拟结构,通过Discovery Studio-2019的Flexible Docking模块获取FRAPD-TGm2与底物CBZ-Gln-Gly的复合物模型。如图1-a所示,其底物口袋由残基1~6、59~69、75~76、238~257、273~291和301~305组成。分子动力学分析显示,底物口袋中残基1~4和244~251为高柔性区(图1-b)。基于分子动力学最后10 ns轨迹用于动态互相关性分析。结果表明,与FRAPD-TGm2底物口袋柔性区域具有动态互相关性的区域有3组(图1-c),且均位于远离于底物口袋的酶分子表面(图1-d)。通过Rosetta Cartesian_ddG对上述3组的氨基酸残基进行虚拟饱和突变,20个突变体ΔΔG<-1 kcal/mol(图2)。基于前期构建的E.coli表达系统[9],表达上述20个突变体(数据未显示)。

a-FRAPD-TGm2与底物CBZ-Gln-Gly复合物结构图;b-FRAPD-TGm2的RMSF值和RMSD值;c-FRAPD-TGm2氨基酸残基动态互相关性热图;d-FRAPD-TGm2分子与底物口袋柔性区的互相关性残基
图1 FRAPD-TGm2结构及动态互相关性分析
Fig.1 Structure and dynamic cross-correlation analysis of FRAPD-TGm2
注:图1-a中底物口袋区域(距离底物12 Å范围的残基)显示为橙色;图1-d中底物口袋柔性区域显示为红色,与其具有动态互相关性的区域显示为青蓝色。FRAPD-TGm2于370 K进行分子动学模拟50 ns。
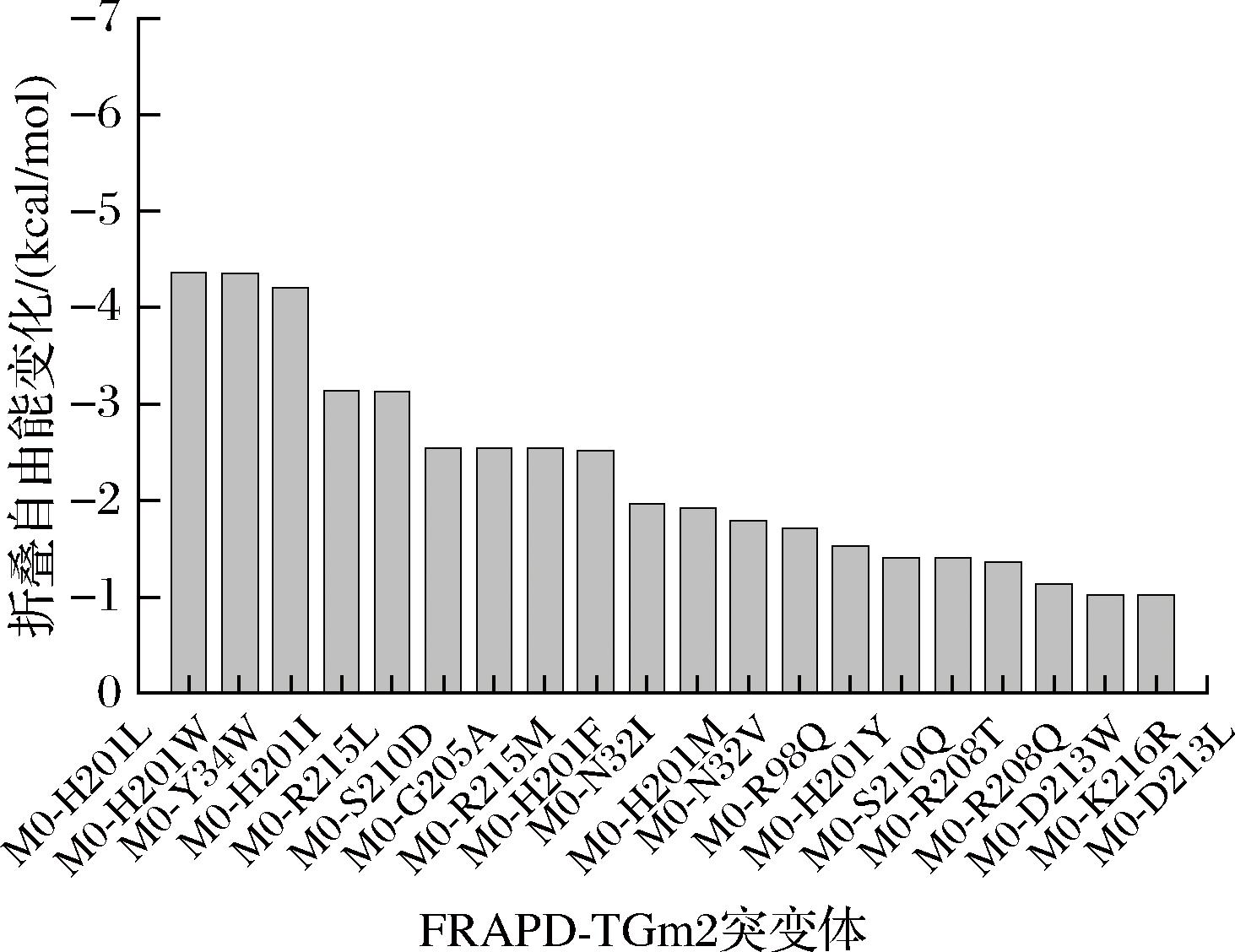
图2 FRAPD-TGm2突变体折叠自由能变化预测
Fig.2 Folding free energy prediction of FRAPD-TGm2 variants
注:M0为FRAPD-TGm2。
2.2 高热稳定型TGase突变体纯化及筛选
除H201M外,其他19个突变体均在E.coli中成功表达(结果未显示)。如图3-a所示,经镍柱亲和纯化和脱盐处理后,上述19个突变体成为对应纯化样品中的主要蛋白条带。对纯化后的FRAPD-TGm2及其突变体进行酶学性质表征,包括60 ℃孵育30 min的残余酶活力和比酶活力测定。相较于FRAPD-TGm2,突变体FRAPD-TGm2-Y34W残余酶活力提升22.2%,比酶活力仅下降4.9%;突变体FRAPD-TGm2-H201L、FRAPD-TGm2-H201Y、FRAPD-TGm2- FRAPD-TGm2-H201F以及FRAPD-TGm2-H201W突变体的比酶活力明显下降,其中FRAPD-TGm2-H201Y、FRAPD-TGm2-H201F以及FRAPD-TGm2-H201W突变体的残余酶活力显著下降;突变体FRAPD-TGm2-N32I和FRAPD-TGm2-N32V的残余酶活力均为0,但比酶活力没有显著变化(图3-b)。其余突变体的比酶活力和残余酶活力没有出现明显变化,仅突变体FRAPD-TGm2-D213W和FRAPD-TGm2-D213L的残余酶活力下降45%左右(图3-b)。因此,Tyr34、His201和Asn32位点对FRAPD-TGm2的热稳定性和催化活性的影响较明显,且Tyr34位的突变能提升其热稳定性。

a-纯化后的FRAPD-TGm2突变体SDS-PAGE电泳分析;b-FRAPD-TGm2突变体比酶活及热稳定性
图3 FRAPD-TGm2突变体纯化及酶学性质表征
Fig.3 Prediction and characterization of FRAPD-TGm2 variants
注:M0为FRAPD-TGm2;残余酶活力为60 ℃孵育30 min测得;*代表差异显著(*P<0.05,**P<0.01,***P<0.001)(下同)。
2.3 饱和突变及组合突变体提高TGase热稳定性
为进一步提高FRAPD-TGm2热稳定性,分别对His201和Asn32位点进行了饱和突变,除H201P外,其余突变体均被成功表达和纯化(图3-a)。相较于FRAPD-TGm2,H201K和H201R突变体60 ℃孵育30 min残余酶活力分别提升21.1%和22.6%,但比酶活力分别下降35.6%和46.6%(图4-a);Asn32位点的突变体均导致残余酶活力下降(图4-b)。在前述的热稳定性突变体FRAPD-TGm2-Y34W中上分别引入突变H201K和H201R,得到突变体FRAPD-TGm2-Y34W-H201R和FRAPD-TGm2-Y34W-H201K。然而,上述2个组合突变体的比酶活力较FRAPD-TGm2-Y34W下降超过50%,且65 ℃孵育20 min残余酶活力变化不明显或下降(图4-c)。尽管如此,上述结果进一步说明了His201和Asn32对FRAPD-TGm2的热稳定性和催化活性的重要影响。
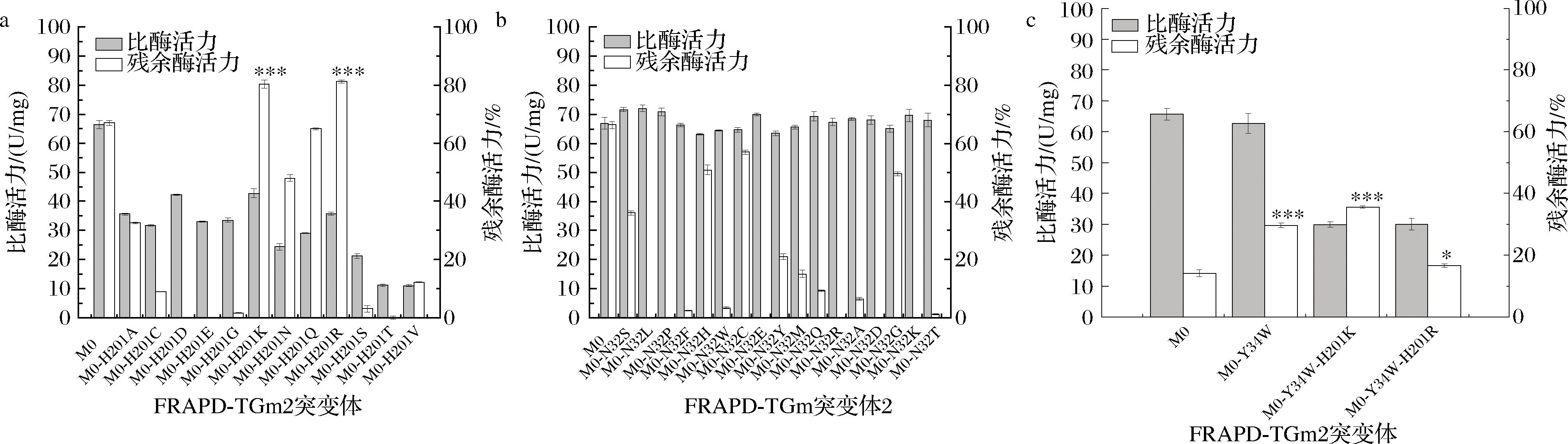
a-H201突变体比酶活力及60 ℃(30 min)残余酶活力;b-N32突变体比酶活力及60 ℃(30 min)残余酶活力;c-组合突变体比酶活力和65 ℃(20 min)残余酶活力
图4 FRAPD-TGm2突变体表征
Fig.4 Characterization of FRAPD-TGm2 variants
2.4 FRAPD-TGm2-Y34W酶学性质及反应动力学
为进一步表征FRAPD-TGm2-Y34W与FRAPD-TGm2的差异,分析了其60 ℃半衰期和最适温度。在60 ℃热处理超过60 min后,FRAPD-TGm2-Y34W的残余酶活力仍保留在60%以上,而FRAPD-TGm2而不足35%(图5-a)。如表2所示,FRAPD-TGm2-Y34W的60 ℃半衰期达到了100.5 min,是FRAPD-TGm2的1.89倍。尽管FRAPD-TGm2-Y34W和FRAPD-TGm2最适反应温度均为60 ℃,但前者在60、65、70 ℃的比酶活力分别比后者高6.8%、28.5%和20.5%(图5-b)。热稳定性的显著提升可能是FRAPD-TGm2-Y34W在高温条件下比酶活力提升的重要原因。
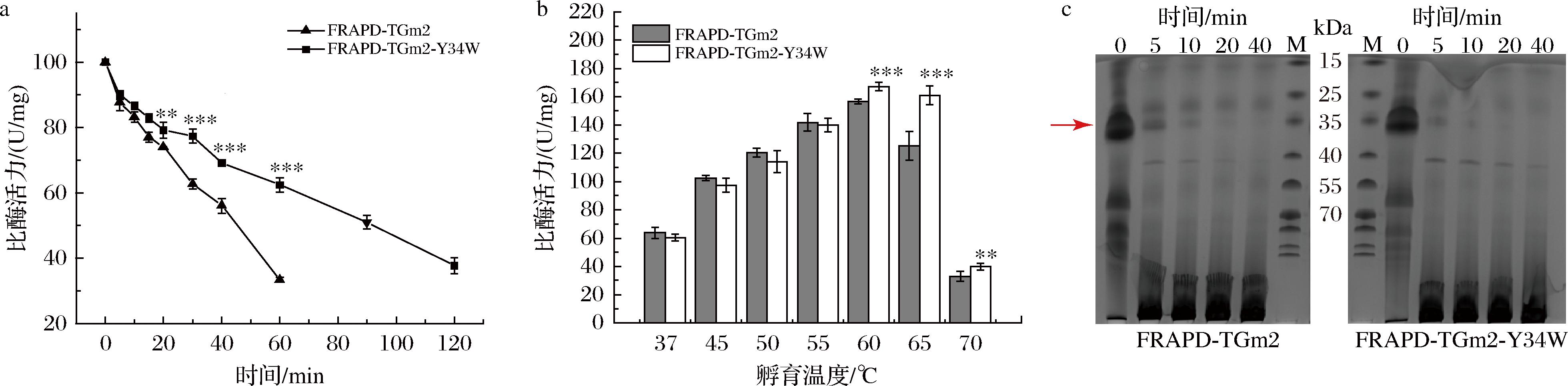
a-60 ℃孵育不同时间残余酶活力;b-不同温度比酶活力;c-酪蛋白交联实验
图5 FRAPD-TGm2-Y34W表征及酪蛋白交联活性测定
Fig.5 Characterization of FRAPD-TGm2-Y34W and determination of casein cross-linking activity
注:图5-c中红色箭头指向β-酪蛋白。
表2 FRAPD-TGm2突变体的半衰期及反应动力学参数
Table 2 Half-life and reaction kinetic parameters of TGase and its variants

组别60 ℃半衰期/minKm/(mmol/L)kcat/s-1kcat/Km/L/(mmol·s)FRAPD-TGm253.15.9±0.639.66.71 FRAPD-TGm2-Y34W100.55.5±0.437.56.82
为评估FRAPD-TGm2-Y34W应用效果,分析了其在65 ℃时对酪蛋白的交联活性。SDS-PAGE分析显示,催化反应在5 min后,FRAPD-TGm2-Y34W反应体系中的β-酪蛋白条带明显变浅,并在靠近加样口处形成大量高分子蛋白条件;在相同反应时间内,FRAPD-TGm2处理样品中仍存在明显的β-酪蛋白条带(图5-c)。研究结果显示,相较于FRAPD-TGm2,FRAPD-TGm2-Y34W在高温反应条件下具有更高的蛋白交联活性。
为进一步表征FRAPD-TGm2及FRAPD-TGm2-Y34W的催化特性,以CBZ-Gln-Gly为底物测了其酶促反应动力学参数。结果显示,FRAPD-TGm2-Y34W和FRAPD-TGm2具有近似的酶反应动力学参数(表2)。
2.5 突变体FRAPD-TGm2-Y34W热稳定性提升机制解析
通过ColabFold和Rosetta_Relax分别构建了FRAPD-TGm2及FRAPD-TGm2-Y34W的结构模型,并在330 K下进行分子动力学模拟50 ns。如图6-a所示,FRAPD-TGm2-Y34W的平均RMSD值(0.191 nm)和最终RMSD值(0.196 nm)均低于FRAPD-TGm2(平均值0.208 nm,最终值0.211 nm)(图6-a),表明FRAPD-TGm2-Y34W结构具有更强的刚性。与FRAPD-TGm2相比,在FRAPD-TGm2-Y34W中Tyr34附近区域(残基34~36)及其动态互相关性的区域(残基243~250与296~303)的RMSF值均较FRAPD-TGm2对应区域下降(图6-b和图1-c)。其中,残基243~250位于底物结合口袋附近。研究显示,底物结合口袋刚性的提升有助于酶稳定性[10]。类似的,突变与活性中心具有动态互相关性的远端残基,使转酮酶口袋区域的柔性下降,实现了55 ℃半衰期的提升10.8倍,且催化活性具有一定提升[13]。因此,通过远端效应降低底物结合口袋的柔性可能是FRAPD-TGm2-Y34W热稳定性提高的重要原因。
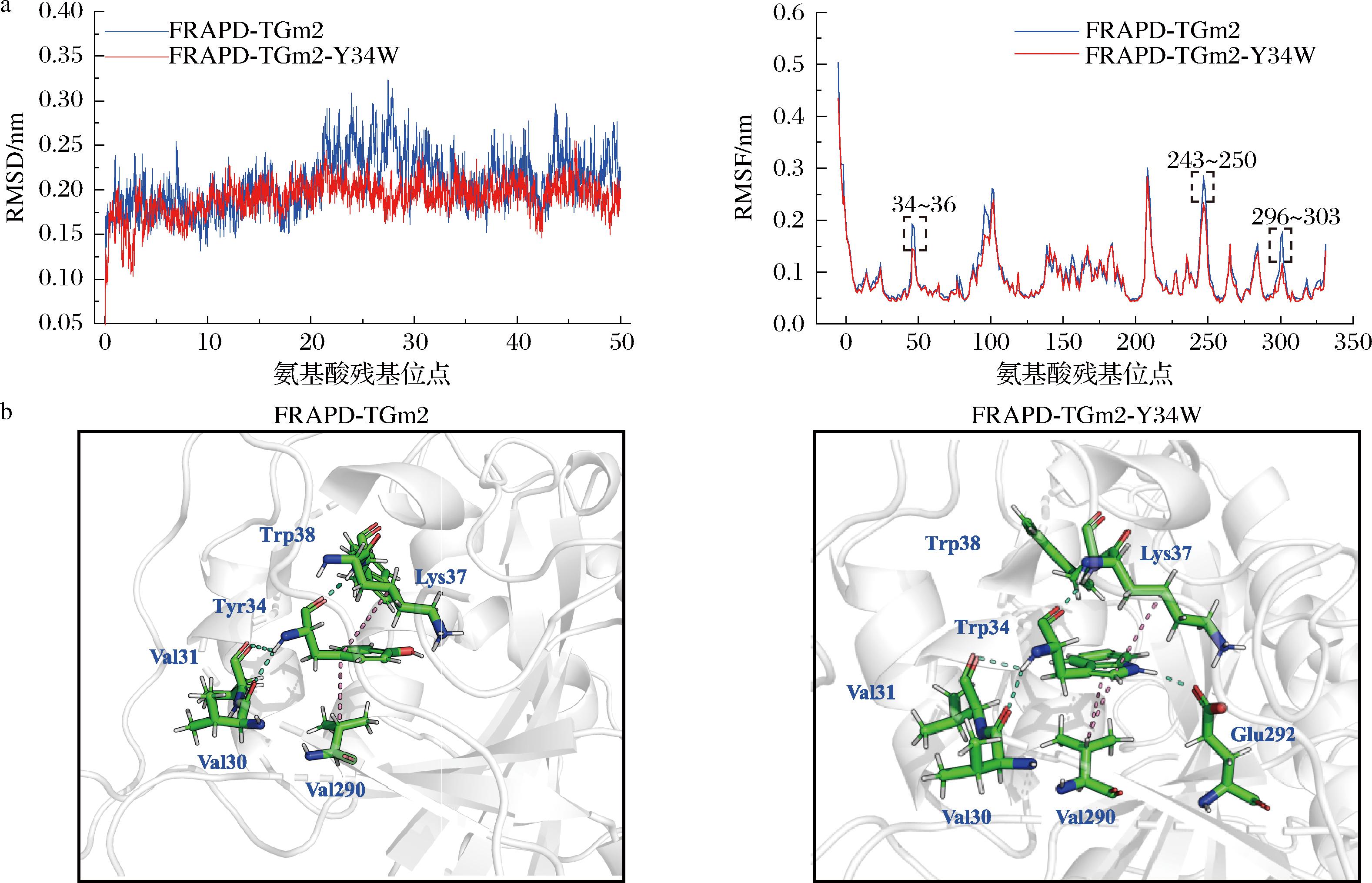
a-RMSD和RMSF值;b-局部作用力
图6 分子动力学模拟及局部非键相互作用力分析
Fig.6 Molecular dynamics simulation and local non-covalent interaction analysis
注:图6-b中氢键和疏水相互作用分别显示为绿色和棕色。
本研究对分子动力学模拟最后的20 ns进行聚类,并提取了占比最高的构象进行局部作用力分析。结果显示,FRAPD-TGm2-Y34W中Tyr34分别与Lys292和Val30形成了新的氢键和疏水相互作用(图6-b)。因此,新的疏水作用力和氢键的产生可能是FRAPD-TGm2-Y34W刚性和蛋白稳定性提升的关键。
3 结论
热稳定性对S.mobaraensis TGase应用有重要影响[24]。为了进一步提高S.mobaraensis TGase热稳定性,本研究基于前期构建的耐热突变体FRAPD-TGm2,通过动态互相关性分析和折叠自由能预测,获得了一种稳定性显著提升的耐热突变体FRAPD-TGm2-Y34W。相较于FRAPD-TGm2,突变体FRAPD-TGm2-Y34W的60 ℃半衰期和65 ℃比酶活力分别提升了89.1%和28.5%,将助于进一步提升TGase在高温条件下的应用效果。
提高结构局部刚性是一种提高酶稳定性的有效策略,而柔性区域和高B-factor的区域一直是热稳定性改造的热点[25]。底物结合口袋是酶分子中重要的柔性区域,是酶热稳定性改造的重要靶点[10]。然而,直接对底物结合口袋进行氨基酸突变可显著提高酶的稳定性,但是会导致催化活性的剧烈下降甚至消失[13]。因此,本研究提出动态互相关性分析和折叠自由能预测相结合的策略,发现了可调控底物结合口袋柔性的“远端位点”。通过对远端位点的突变提升了TGase稳定性,同时避免了对其催化活性的不利影响。相关策略将为其他酶蛋白热稳定性改造提供参考。
[1] AKBARI M, RAZAVI S H, KIELISZEK M.Recent advances in microbial transglutaminase biosynthesis and its application in the food industry[J].Trends in Food Science &Technology, 2021, 110:458-469.
[2] KASHIWAGI T, YOKOYAMA K I, ISHIKAWA K, et al.Crystal structure of microbial transglutaminase from Streptoverticillium mobaraense[J].Journal of Biological Chemistry, 2002, 277(46):44252-44260.
[3] FUCHSBAUER H L.Approaching transglutaminase from Streptomyces bacteria over three decades[J].The FEBS Journal, 2022, 289(16):4680-4703.
[4] KIELISZEK M, MISIEWICZ A.Microbial transglutaminase and its application in the food industry:A review[J].Folia Microbiologica, 2014, 59(3):241-250.
[5] BUETTNER K, HERTEL T C, PIETZSCH M.Increased thermostability of microbial transglutaminase by combination of several hot spots evolved by random and saturation mutagenesis[J].Amino Acids, 2012, 42(2-3):987-996.
[6] SORAPUKDEE S, TANGWATCHARIN P.Quality of steak restructured from beef trimmings containing microbial transglutaminase and impacted by freezing and grading by fat level[J].Asian-Australasian Journal of Animal Sciences, 2018, 31(1):129-137.
[7] MARX C K, HERTEL T C, PIETZSCH M.Random mutagenesis of a recombinant microbial transglutaminase for the generation of thermostable and heat-sensitive variants[J].Journal of Biotechnology, 2008, 136(3-4):156-162.
[8] YOKOYAMA K, UTSUMI H, NAKAMURA T, et al.Screening for improved activity of a transglutaminase from Streptomyces mobaraensis created by a novel rational mutagenesis and random mutagenesis[J].Applied Microbiology and Biotechnology, 2010, 87(6):2087-2096.
[9] WANG X L, DU J H, ZHAO B C, et al.Significantly improving the thermostability and catalytic efficiency of Streptomyces mobaraenesis transglutaminase through combined rational design[J].Journal of Agricultural and Food Chemistry, 2021, 69(50):15268-15278.
[10] XIE Y, AN J, YANG G Y, et al.Enhanced enzyme kinetic stability by increasing rigidity within the active site[J].Journal of Biological Chemistry, 2014, 289(11):7994-8006.
[11] ZENG W, LI X Q, YANG Y Y, et al.Substrate-binding mode of a thermophilic PET hydrolase and engineering the enzyme to enhance the hydrolytic efficacy[J].ACS Catalysis, 2022, 12(5):3033-3040.
[12] JOO S, CHO I J, SEO H, et al.Structural insight into molecular mechanism of poly(ethylene terephthalate) degradation[J].Nature Communications, 2018, 9(1):382.
[13] YU H R, DALBY P A.Exploiting correlated molecular-dynamics networks to counteract enzyme activity-stability trade-off[J].Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(52):E12192-E12200.
[14] GU J, XU Y, NIE Y.Role of distal sites in enzyme engineering[J].Biotechnology Advances, 2023, 63:108094.
[15] THILTGEN G, GOLDSTEIN R A.Assessing predictors of changes in protein stability upon mutation using self-consistency[J].PLoS One, 2012, 7(10):e46084.
[16] DOLGIKH B, WOLDRING D.Site-wise diversification of combinatorial libraries using insights from structure-guided stability calculations[J].Methods in Molecular Biology, 2022, 2491:63-73.
[17] PARK H, BRADLEY P, GREISEN P Jr, et al.Simultaneous optimization of biomolecular energy functions on features from small molecules and macromolecules[J].Journal of Chemical Theory and Computation, 2016, 12(12):6201-6212.
[18] 杜坤, 周丽, 堵国成,等.内含肽介导谷氨酰胺转胺酶酶原的活化[J].食品科学, 2013, 34(9):90-94.DU K, ZHOU L, DU G C, et al.Intein-mediated activation of transglutaminase from Streptomyces hygroscopicus[J].Food Science, 2013, 34(9):90-94.
[19] MIRDITA M, SCHÜTZE K, MORIWAKI Y, et al.ColabFold:Making protein folding accessible to all[J].Nature Methods, 2022, 19(6):679-682.
[20] CONWAY P, TYKA M D, DIMAIO F, et al.Relaxation of backbone bond geometry improves protein energy landscape modeling[J].Protein Science, 2014, 23(1):47-55.
[21] ABRAHAM M J, MURTOLA T, SCHULZ R, et al.GROMACS:High performance molecular simulations through multi-level parallelism from laptops to super computers[J].SoftwareX, 2015, 1-2:19-25.
[22] MAIER J A, MARTINEZ C, KASAVAJHALA K, et al.ff14SB:Improving the accuracy of protein side chain and backbone parameters from ff99SB[J].Journal of Chemical Theory and Computation, 2015, 11(8):3696-3713.
[23] YU H R, DALBY P A.A beginner’s guide to molecular dynamics simulations and the identification of cross-correlation networks for enzyme engineering[J].Methods in Enzymology, 2020, 643:15-49.
[24] DUARTE L, MATTE C R, BIZARRO C V, et al.Review transglutaminases:Part II-industrial applications in food, biotechnology, textiles and leather products[J].World Journal of Microbiology &Biotechnology, 2019, 36(1):11.
[25] BOMMARIUS A S, PAYE M F.Stabilizing biocatalysts[J].Chemical Society Reviews, 2013, 42(15):6534-6565.