β-内酰胺类抗生素是指一类化学结构中含有四元内酰胺环的抗生素,包括青霉素、头孢菌素、头霉素等,其通过抑制参与细胞壁合成的酶来发挥杀菌作用,广泛应用于临床治疗中[1]。青霉素是最早发现和临床使用的第一代β-内酰胺类抗生素,在人类对抗细菌感染上发挥了巨大作用,但随着青霉素的广泛使用,耐青霉素的金黄色葡萄球菌迅速增加。微生物对抗生素具有2种类型的耐药性,即固有耐药和获得性耐药。部分微生物编码的介导耐药性的基因会通过基因水平转移进入其他微生物体内,最早的抗生素耐药性报告是发现大肠杆菌的无细胞提取物能够使青霉素失活。过量使用抗生素所造成的选择性压力会加速耐药基因的进化和传播,导致超级细菌的出现,致病菌的抗生素耐药性对人类健康和全球公共卫生构成了重大威胁。2010年在印度发现了一种新的B类金属β-内酰胺酶NDM-1,能够水解目前绝大部分β-内酰胺类抗生素,感染者死亡率很高[2]。因此,针对微生物耐药基因的发现、溯源、进化以及对超级细菌的防治研究已成为人类健康方面亟待解决的一个关键问题[3]。
微生物通过多种机制实现对β-内酰胺类抗生素耐受,包括表达β-内酰胺酶从而水解药物、诱导蛋白结构变化实现竞争性结合抗生素、激活自身外排泵系统降低胞内浓度等。其中,β-内酰胺酶介导的耐药仍然是最大的临床阻力。β-内酰胺酶按其氨基酸序列及底物专一性可分为A、B、C、D这四类[4]。A、C和D类β-内酰胺酶通过活性位点上的丝氨酸残基与底物形成共价反应中间体催化β-内酰胺类药物的水解开环,因此又被称为丝氨酸β-内酰胺酶。B类β-内酰胺酶与上述3个类型不同,其活性中心含有金属离子,金属配位由水分子完成,从而替代A、C和D类β-内酰胺酶活性部位的丝氨酸残基,通过一种非共价机制催化β-内酰胺类药物的水解,这类酶被称为金属β-内酰胺酶。B类β-内酰胺酶因其具有更加广泛的底物谱,且不会被丝氨酸酶抑制剂所抑制的特点,一直是临床研究的热点。其中,铜绿假单胞菌产生的SPM-1是典型的B类β-内酰胺酶,该酶可以水解几乎所有类别的β-内酰胺类抗生素,且临床上没有可用的抑制剂[5]。青霉素类、头孢菌素类和碳青霉烯类等抗生素的出现及广泛使用促进了β-内酰胺酶的传播和突变,至今,已发现的β-内酰胺酶数量已超8 000 种,给人类健康造成了极大的威胁[6]。
土壤微生物是细菌抗性基因最大的“贮藏库”,大量的临床耐药基因来自于土壤中的微生物[7],然而,土壤中仅有不足1%的微生物可以在现有实验室条件下培养,因此,无法通过微生物分离培养的传统方法对土壤中的抗生素耐药基因进行广泛研究。宏基因组学方法通过直接提取土壤中微生物的总基因组DNA对相关微生物基因进行功能分析,不依赖于微生物的可培养性,为非培养微生物来源的抗生素耐药基因的发现提供了有力的工具[8]。本研究通过功能宏基因组学方法获得1个β-内酰胺耐药基因gob-54,通过生物信息学分析、最小抑菌浓度测定和生化分析,推测gob-54耐药类型,评估其耐药性和酶学性质及底物特异性,为深入研究β-内酰胺类抗生素耐药基因在土壤微生物组中的多样性奠定基础,也为开发新型β-内酰胺类药物提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与载体
Escherichia coli EPI100TM-T1R,美国Epicentre公司;E.coli BL21(DE3),南京擎科生物科技有限公司,标准对照菌株E.coli ATCC25922,本实验室保存;质粒pTG19-T,捷瑞生物工程有限公司;pET-30a(+),德国Novagen公司。ZF700是通过功能宏基因组学方法在珠峰土壤宏基因组文库筛选到的β-内酰胺耐药阳性克隆[9],构建的亚克隆命名为pZF700,将β-内酰胺酶基因gob-54插入到pET-30a(+)表达载体上将其蛋白命名为GOB-54。
1.1.2 主要试剂
头孢西丁、头孢唑啉钠盐、哌拉西林钠、头孢匹罗硫酸盐、头孢哌酮,南京晚晴化玻仪器有限公司;头孢噻肟钠盐,上海阿拉丁生化科技股份有限公司;头孢曲松,北京迈瑞达科技有限公司;头孢氨苄,合肥博美生物科技有限责任公司第二分公司;头孢替安和卡那霉素,上海捷瑞生物工程有限公司;异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)和氨苄西林,北京索莱宝生物有限公司。
1.2 仪器与设备
9600型PCR仪,珠海黑马医学仪器有限公司;CT15RT型低温高速离心机,上海天美仪器有限公司;LDHW-4001D型恒温培养箱,上海申安医疗器械有限公司;DYY-6C型琼脂糖凝胶电泳仪,北京六一生物科技有限公司;TU-1900型双束紫外分光光度计,北京普析公司;GelX1520型凝胶成像仪,欧翔科学仪器有限公司。
1.3 实验方法
1.3.1 溶液配制
Tris-HCl缓冲液:称取1.211 g Tris溶于超纯水中,定容至500 mL,并用浓盐酸调节pH值为7.5。
MH培养基:称取5.25 g MH肉汤粉末,纯水定容至250 mL。
PBS缓冲液:称取16.962 5 g Na2HPO4·12H2O,0.412 5 g NaH2PO4·2H2O,17.532 g NaCl溶于超纯水中,定容至1 L。
最小抑菌浓度(minimum inhibitory concentration,MIC)测定所用抗生素:氨苄西林、哌拉西林、头孢氨苄、头孢西丁、头孢唑啉、头孢替安、头孢哌酮、头孢噻肟和头孢匹罗,配制成质量浓度为5 120 μg/mL的溶液。
动力学参数测定所用抗生素:哌拉西林、头孢氨苄、头孢西丁、头孢唑啉、头孢噻肟和头孢匹罗,配制成质量浓度为1 mg/mL的溶液。
1.3.2 耐头孢噻肟阳性克隆的筛选和亚克隆构建
在含有头孢噻肟(4 μg/mL)、氯霉素(15 μg/mL)和氨苄西林(100 μg/mL)的LB固体培养基上进行文库筛选,文库含有1.04×107克隆,氯霉素和氨苄西林为文库菌载体的抗性。平板在37 ℃下培养24 h,筛选出耐头孢噻肟的阳性克隆。提取阳性克隆的DNA,用Sau3A I进行部分酶切。回收2~4 kbp的DNA片段连接到用BamHⅠ酶切过的pTG19-T载体上,连接产物转化到大肠杆菌E.coli EPI100中。将转化子涂布在含有4 μg/mL头孢噻肟和100 μg/mL氨苄西林的LB固体培养基上。37 ℃下培养24 h后,选择可以在平板上生长的亚克隆进行测序。
1.3.3 pZF700耐药基因生物信息学分析
pZF700为携带头孢噻肟耐药基因的亚克隆,测序由南京擎科生物科技有限公司完成。利用BLASTx根据NCBI的非冗余蛋白质数据库鉴定各开放阅读框(open read frame,ORF)的功能。使用MEGA-X软件和邻接法创建了GOB-54的系统发育树,在线平台esprit3.0 (https://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)进行多重序列比对。使用在线平台SWISS-MODEL基于氨基酸同一性对GOB-54进行同源建模,GOB-18(PDB:5k0w.1)的晶体结构作为模板。
1.3.4 β-内酰胺酶表达和纯化
以pZF700亚克隆质粒为模板,对β-内酰胺酶基因gob-54进行PCR扩增。扩增产物进行NdeⅠ和XhoⅠ酶切后克隆到经过相同酶切割的pET-30a(+)表达载体上,采用电转转化入表达宿主E.coli BL21(DE3)中。基因对应引物对序列:gob-54-F(5′- GGAATTCCATATGAATTCATCGATTCGAAACA-3′);gob-54-R(5′-CCGCTCGAGATCTTCTTTACTTCTTTTGTTATAGT-3′)。挑取单克隆在含有50 μg/mL卡那霉素(载体抗性)的4 mL液体LB培养基中过夜培养。取4 mL过夜培养的菌液加入含有卡那霉素的400 mL液体LB培养基中,摇床培养至OD600值为0.5~0.6。冷却后加入IPTG至终浓度0.2 mmol/L,将培养基置于18 ℃摇床,以160 r/min诱导培养18 h。将诱导表达后的菌液倒入预冷的离心瓶中,在4 ℃离心收集菌体,将菌体重悬于10 mL PBS缓冲液中。对细菌悬浮液进行超声波破碎,破碎1.0 s间歇1.5 s,破碎时间为20 min。将破碎完成后的蛋白于4 ℃离心,去除菌体,得到酶粗液,通过镍离子亲和层析柱进行纯化。用PBS缓冲液和500 mmol/L咪唑配制成10、20、50、100、150、200、250 mmol/L的不同浓度咪唑的洗脱液。先用10个柱体积的超纯水冲洗镍柱,再用5个柱体积的10 mmol/L咪唑对镍柱进行平衡。将粗酶液缓缓加入镍柱中,重复3次,使粗酶液中重组蛋白的组氨酸残基与Ni2+充分结合。再依次用不同浓度的洗脱液洗出目的蛋白。根据目的蛋白的大小,选择阻拦相对分子质量30 kDa的蛋白超滤管进行缓冲液的替换。将蛋白浓缩至1 mL后加入20 mmol/L Tris-HCl溶液,重复3次。
1.3.5 MIC测定
根据美国临床和实验室标准协会(Clinical and Laboratory Standards Institude,CLSI)的文件M100-S28进行药物敏感性试验测定,采用微量肉汤稀释法测定MIC值[10]。抗生素选择常用的9种β-内酰胺类药物,包括青霉素类(氨苄西林、哌拉西林),头霉素类(头孢西丁)、一代头孢菌素(头孢氨苄、头孢唑啉),二代头孢菌素(头孢替安)、三代头孢菌素(头孢噻肟、头孢哌酮)和四代头孢菌素(头孢匹罗),质量控制菌株为E.coli ATCC25922、E.coli EPI100/pET-30a(+)和E.coli EPI100。将菌株接种至4 mL液体LB培养基中,在37 ℃、225 r/min摇床培养16 h。取200 μL菌液至1.8 mL MH培养基中,使得OD600值在0.08~0.12,此时细胞数量大约为1×108~2×108 CFU/mL,在此基础上用MH液体培养基将菌液稀释1 000倍备用。用MH液体培养基将抗生素母液(5 120 μg/mL)稀释10个质量浓度梯度,为512、256、128、64、32、16、8、4、2、1 μg/mL。在96孔板中依次加入浓度由高到低的含抗生素的MH培养基100 μL,再加入100 μL稀释后的菌液,每个浓度设置3个重复。将96孔板置于37 ℃培养箱中培养16~18 h,观察结果。未检测到细菌生长的最低抗生素浓度为MIC值。
1.3.6 β-内酰胺酶动力学参数的测定
纯化的GOB-54在37 ℃下在20 mmol/L Tris-HCl溶液(pH 7.5)中进行动力学参数的测定。用分光光度法测定β-内酰胺抗生素水解引起的吸光度变化,评价β-内酰胺酶的水解活性。用Michaelis-Menten方程对不同浓度的β-内酰胺抗生素测定反应速率进行非线性回归,得到了Michaelis常数Km和催化常数Kcat。测试底物和波长参数如下:头孢氨苄(261 nm)、头孢噻肟(265 nm)、头孢西丁(265 nm)、头孢唑啉(280 nm)、头孢匹罗(270 nm)、哌拉西林(297 nm)[11]。
2 结果与分析
2.1 β-内酰胺酶GOB-54的序列和结构特征分析
通过对一个来源于珠穆朗玛峰土壤的宏基因组文库的筛选,获得了1个头孢噻肟耐药阳性克隆ZF700,对构建的亚克隆pZF700的插入片段进行DNA序列分析,鉴定出1个独特的B类β-内酰胺耐药基因。新的B类β-内酰胺耐药基因ORF为1 026 bp,G+C含量为45%,编码341个氨基酸,被命名为gob-54(GenBank登录号为PP196369),与Elizabethkingia meningoseptica来源的gob-7有最高的氨基酸序列一致性,仅为52.30%。另外,从系统发育树(图1)可以看出,B类β-内酰胺酶在进化关系上有3个分支:B1、B2和B3亚类[12],gob-54与B3亚类的gob家族成员亲缘最近。目前已报道的gob家族成员多来自伊丽莎白菌属,大多从临床分离得来[13],本研究说明了未被抗生素污染的偏远土壤中含有值得挖掘的耐药基因。

图1 B类β-内酰胺酶系统发育树
Fig.1 Phylogenetic tree of the class B β-lactamases
注:分支的数值表示Bootstrap值,标尺表示进化距离。
建模结果显示GOB-54呈现由6个α螺旋,12个β折叠组成的αβ/βα夹层结构,活性位点位于这两半界面的凹槽内。2个核心β折叠片分别由7个β折叠(β1至β7)和5个β折叠(β8至β12)组成,螺旋α1至α4和螺旋α5至α6分布在两端。GOB-54与GOB-18三维结构比较显示(图2),2种蛋白的均方根偏差为0.069,表明两者蛋白结构高度相似。多重序列结果表明,GOB-54拥有GOB家族QXHXD保守基序(图3),该基序对底物结合与水解至关重要。尽管GOB-54与先前报道的家族其他成员显示出较低的氨基酸序列同源性,但多重序列比对及预测的三维结构清楚地表明其重要基序高度保守,三维结构极其相似。
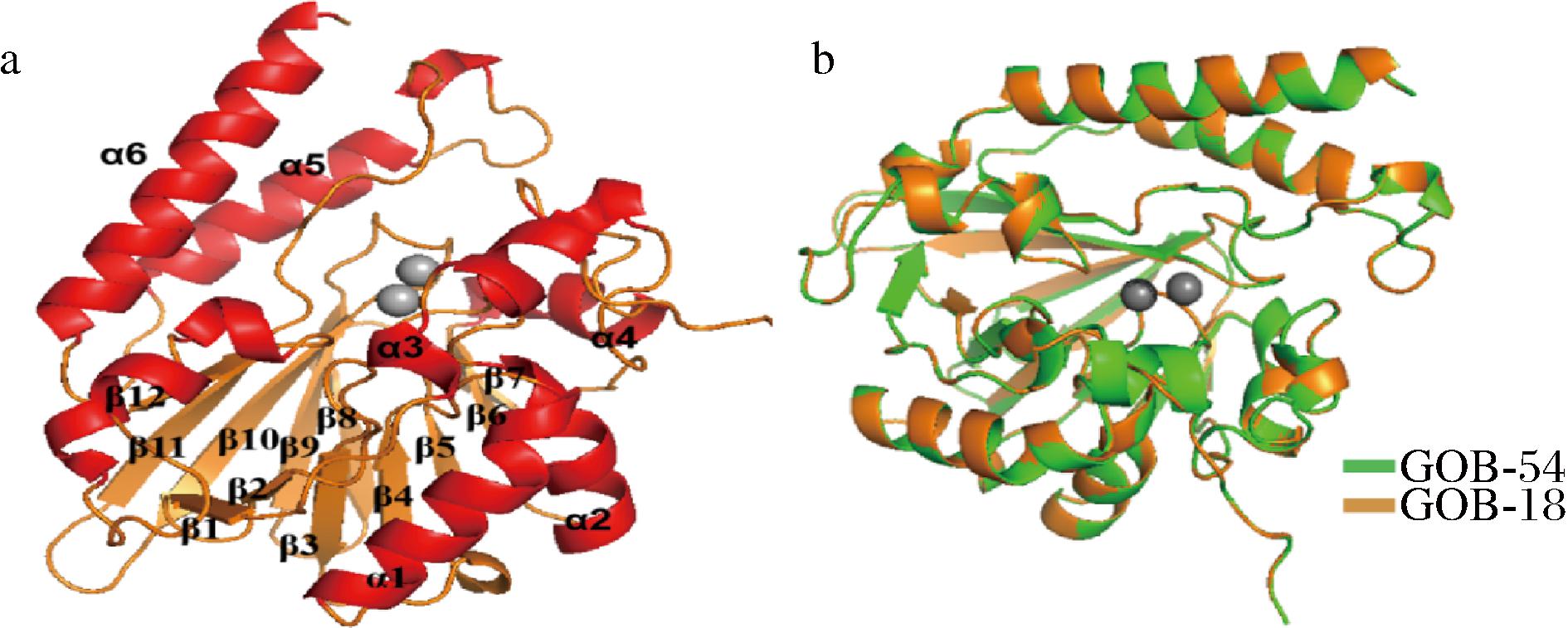
a-GOB-54结构,b-三维结构重叠图
图2 GOB-54和GOB-18的结构重叠图
Fig.2 The structure overlay of the GOB-54 and GOB-18
注:绿色表示GOB-54,橙色表示GOB-18。

图3 GOB-54与同家族酶的多重序列比对
Fig.3 The multiple sequence comparison of GOB-54 with homologous family enzymes
注:深色框表示高度保守。
2.2 最小抑菌浓度分析
采用标准化微量肉汤稀释法测定了GOB-54对9种常用的β-内酰胺类药物的MIC值,测定结果如表1所示。E.coli ATCC25922、E.coli EPI100/pET-30a(+)和E.coli EPI100作为质量控制菌株,对测试抗生素(MICs≤2 μg/mL)均敏感,在CLSI规定范围之内。氨苄西林和哌拉西林为代表性青霉素类抗生素,与质量控制菌株相比,GOB-54对氨苄西林和哌拉西林的耐药值至少提升了256倍(MIC>512 μg/mL),表现出高水平的耐药性;同时,GOB-54对头霉素(头孢西丁)、一代头孢菌素(头孢氨苄和头孢唑啉)、二代头孢菌素(头孢替安)以及三代头孢菌素(头孢哌酮和头孢噻肟)的MIC均高于8 μg/mL,表现了较高的耐药性,这符合B类β-内酰胺酶编码基因表现出的广谱β-内酰胺类药物水解活性;GOB-54对第四代头孢菌素头孢匹罗MIC值也达到了4 μg/mL,均表明GOB-54具有潜在的临床危害性。
表1 最小抑菌浓度 单位:μg/mL Table 1 Minimum inhibitory concentration of GOB-54
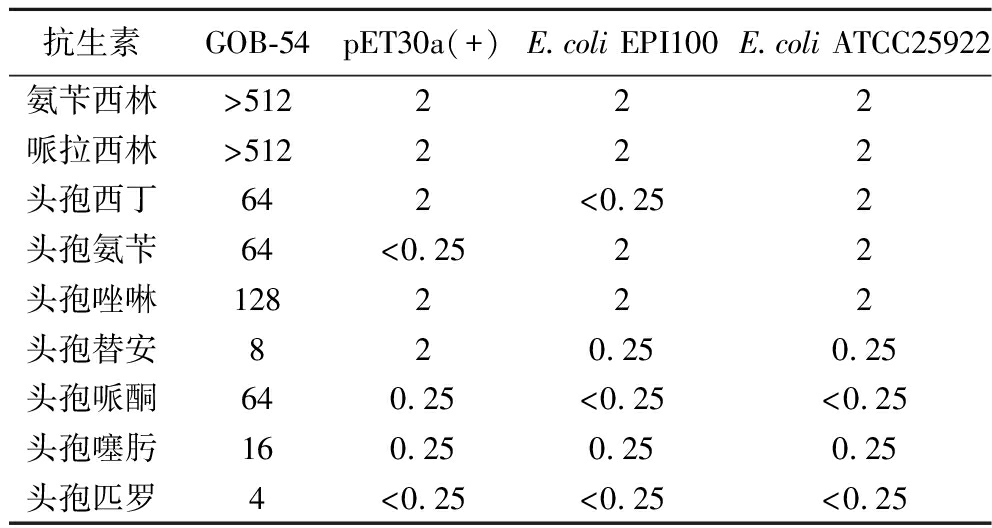
抗生素GOB-54pET30a(+)E.coli EPI100E.coli ATCC25922氨苄西林>512222哌拉西林>512222头孢西丁642<0.252头孢氨苄64<0.2522头孢唑啉128222头孢替安820.250.25头孢哌酮640.25<0.25<0.25头孢噻肟160.250.250.25头孢匹罗4<0.25<0.25<0.25
2.3 GOB-54的表达纯化与生化特性表征
为进一步解析GOB-54在药敏性测定实验中对不同β-内酰胺类药物产生耐药表型差异的机理,我们将基因异源表达后,对纯化后的蛋白生化特性进行表征。将GOB-54诱导表达完成后的菌液经过离心、破碎、上镍柱进行蛋白的纯化。利用SDS-PAGE检测(图4),GOB-54酶的纯化效率高,在SDS-PAGE凝胶上可见单一条带,与预测的目的蛋白分子质量39.1 kDa 一致,根据Brandford法测定纯化后的GOB-54酶蛋白质量浓度为169.36 μg/mL。
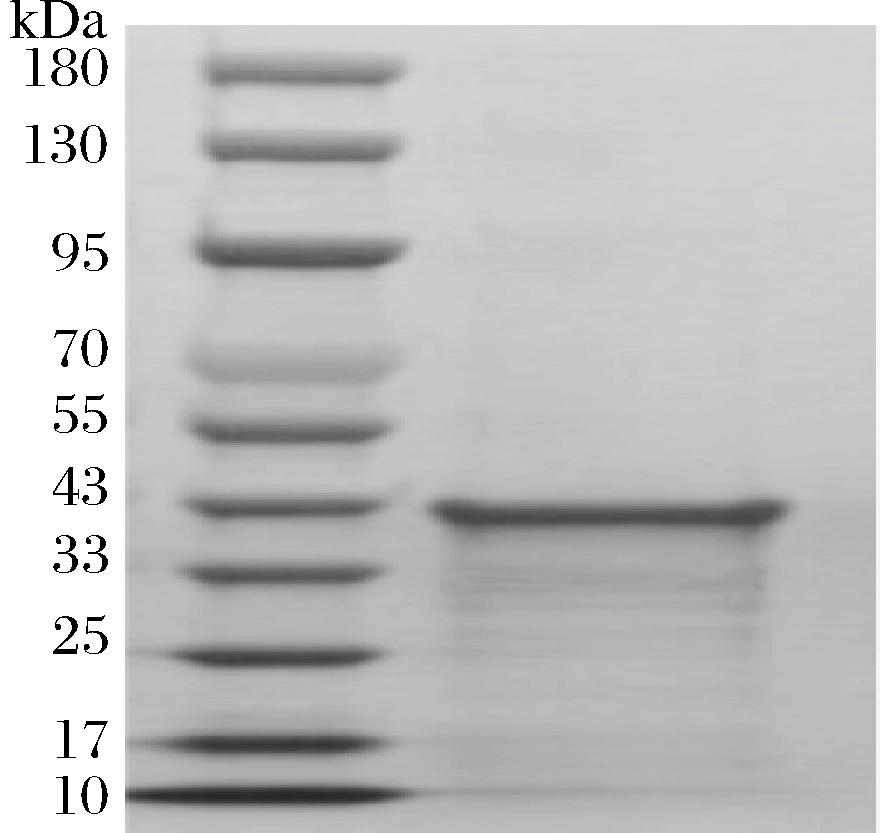
图4 纯化GOB-54的SDS-PAGE分析
Fig.4 SDS-PAGE analysis of the purified GOB-54
通过对GOB-54的动力学参数测定(表2)发现,GOB-54对二代青霉素哌拉西林和一代头孢菌素头孢氨苄显示出较高的亲和力和良好的水解活性,Km<150 μmol/L,Kcat/Km≥20.83 L/(mmol·s);对头霉素头孢西丁、二代头孢菌素头孢唑啉和三代头孢菌素头孢噻肟的Km值相差不大,范围为272.81~468.61 μmol/L,其中头孢唑啉的Kcat/Km值高于头孢西丁和头孢噻肟,为5.19 L/(mmol·s),表明GOB-54对头孢唑啉的转化率和催化效率高,这与MIC结果相吻合。GOB-54对头孢匹罗亲和力最差,催化效率与其他测定底物相比低了100倍以上,Kcat/Km值为0.33 L/(mmol·s)。在以前的研究中也观察到了这种现象,我们推测是由于头孢匹罗的季铵基团,这一结构变化赋予其更高的β-内酰胺酶稳定性[14]。
表2 动力学参数的测定
Table 2 Kinetic parameters for GOB-54
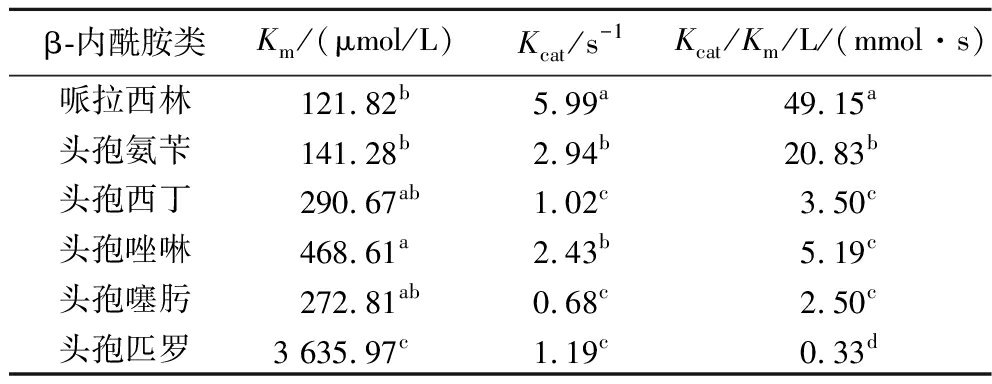
β-内酰胺类Km/(μmol/L)Kcat /s-1Kcat/Km/L/(mmol·s)哌拉西林121.82b5.99a49.15a头孢氨苄141.28b2.94b20.83b头孢西丁290.67ab1.02c3.50c头孢唑啉468.61a2.43b5.19c头孢噻肟272.81ab0.68c2.50c头孢匹罗3 635.97c1.19c0.33d
注:表中同列肩标不同字母表示差异显著(P<0.05)。
3 讨论
β-内酰胺类抗生素作为一种广谱、高效的抗菌药物,在临床上得到了广泛应用,造成的抗生素选择压力可能会导致耐药基因的进化和传播。环境中含有大量不可培养微生物,许多临床相关的耐药基因都起源于环境微生物,功能宏基因组学绕过不可培养的屏障,并且不依靠序列同源性,为新型β-内酰胺类抗生素耐药基因的挖掘提供了可能[15]。本研究鉴定的β-内酰胺类抗生素耐药基因gob-54,属于B3亚类的gob家族,与先前报道的家族其他成员显示出较低的氨基酸序列同源性,仅为52.30%。
本研究药物敏感性实验结果显示,GOB-54对氨苄西林和哌拉西林的MIC>512 μg/mL,表现出高水平的耐药性,与临床来源的GOB-17相比至少高了8倍(MIC≤64 μg/mL)[16]。这可能是由于用于克隆的载体与宿主不同,导致蛋白质产量更高,本研究在药物敏感性实验中使用的载体为pET30a(+),宿主为E.coli EPI100。此外,GOB-54对头霉素(头孢西丁)、一代头孢菌素(头孢氨苄和头孢唑啉)、二代头孢菌素(头孢替安)以及三代头孢菌素(头孢哌酮和头孢噻肟)的MIC≥8 μg/mL,表现出较高的抗性水平。GOB-54对头孢匹罗MIC值为4 μg/mL,与结构相似的GOB-18相比表现出较弱的水解能力[17]。GOB-18空间拓扑等效位置上位于活性口袋的丝氨酸、谷氨酰胺在GOB-54中突变成了109位的半胱氨酸和163位的丙氨酸,影响活性口袋处loop环的波动,使得参与底物分子识别的氨基酸和键能减少,从而导致口袋对底物的催化作用减弱,相关突变导致的水解能力变化的现象在A、B、C类β-内酰胺酶中也已发现[18]。C类β-内酰胺酶CMY-185与CMY-2相比只有4个氨基酸突变,但一级和二级氨基酸取代能够恢复CMY-185对头孢他啶/阿维巴坦耐药性,并进一步赋予其对头孢地考的耐药性[19]。同时,作为第四代头孢菌素的代表性药物,头孢匹罗是在头孢噻肟的基础上引入季铵基团,增加了对部分β-内酰胺酶的稳定性,也导致头孢匹罗对GOB-54的较高耐受性。动力学参数表明GOB-54对哌拉西林和头孢氨苄Km<150 μmol/L,显示出较高的亲和力,对哌拉西林具有最高的催化活性,Kcat/Km值为49.15 L/(mmol·s)。活性测定和生化表征结果均证明gob-54编码的GOB-54是一种典型的广谱β-内酰胺酶。
产生耐药性的病原体永远不会消失,并且会在未来继续增加种类[20]。目前对耐药性及耐药基因的关注多集中在临床,然而,非临床环境中也发现存在β-内酰胺类耐药基因,例如ZHO-1、OXA-48等[21]。微生物生态系统不是孤立的,不同微生物生态系统之间存在基因水平转移的可能性,任何新的耐药基因都可能迅速蔓延到其他地区。例如新德里金属-β-内酰胺酶,该基因在首次发现后不到5年内就开始在全球范围内传播,感染者死亡率很高。如果环境来源的耐药基因通过水平转移被临床相关物种(如肺炎克雷伯菌或铜绿假单胞菌)获得,可能会构成潜在威胁[22]。因此,对土壤中存在的耐药基因进行研究有着不可忽视的意义,可以在其传播到临床前进行早期预警,也能够为研发新的抗菌药物和改良已有的抗生素提供研究依据,从而缓解临床上严重的耐药性威胁[23]。本研究通过功能宏基因组方法,从土壤中鉴定一个新颖的β-内酰胺酶耐药基因,对其生物信息学进行分析,并通过异源表达研究β-内酰胺酶的性质,相关结果有望促进β-内酰胺酶耐药基因理解,为β-内酰胺类抗生素以及其耐药相关研究提供基础。
[1] TOOKE C L, HINCHLIFFE P, BRAGGINTON E C, et al.β-lactamases and β-lactamase inhibitors in the 21 st century[J].Journal of Molecular Biology, 2019, 431(18):3472-3500.
[2] THOMAS C A, CHENG Z S, BETHEL C R, et al.The directed evolution of NDM-1[J].Antimicrobial Agents and Chemotherapy, 2023, 67(11):e0071423.
[3] DONG X T, LIU W L, DONG Y X, et al.Metallo-β-lactamase SMB-1 evolves into a more efficient hydrolase under the selective pressure of meropenem[J].Journal of Inorganic Biochemistry, 2023, 247:112323.
[4] BOYD S E, LIVERMORE D M, HOOPER D C, et al.Metallo-β-lactamases:Structure, function, epidemiology, treatment options, and the development pipeline[J].Antimicrobial Agents and Chemotherapy, 2020, 64(10):e00397-e00320.
[5] DO NASCIMENTO A P B, MEDEIROS FILHO F, PAUER H, et al.Characterization of a SPM-1 metallo-beta-lactamase-producing Pseudomonas aeruginosa by comparative genomics and phenotypic analysis[J].Scientific Reports, 2020, 10(1):13192.
[6] KADERABKOVA N, BHARATHWAJ M, FURNISS R C D, et al.The biogenesis of β-lactamase enzymes[J].Microbiology (Reading), 2022, 168(8):001217.
[7] LAU C H F, VAN ENGELEN K, GORDON S, et al.Novel antibiotic resistance determinants from agricultural soil exposed to antibiotics widely used in human medicine and animal farming[J].Applied and Environmental Microbiology, 2017, 83(16):e00989-e00917.
[8] STREIT W R, DANIEL R.Metagenomics Methods and Protocols[M].New York:Springer Nature, 2023.
[9] WANG S C, GAO X, GAO Y J, et al.Tetracycline resistance genes identified from distinct soil environments in China by functional metagenomics[J].Frontiers in Microbiology, 2017, 8:1406.
[10] JEAN B, BRANDI L, ROBIN P, et al.Performance Standards for Antimicrobial Susceptibility Testing[M].USA:Clinical and Laboratory Standards Institute, 2018.
[11] LUO H Y, LI M R, YANG Z S, et al.Characterization of RASA-1, a novel class A extended-spectrum beta-lactamase from Riemerella anatipestifer[J].Veterinary Microbiology, 2022, 270:109456.
[12] HORSFALL L E, IZOUGARHANE Y, LASSAUX P, et al.Broad antibiotic resistance profile of the subclass B3 metallo-β-lactamase GOB-1, a di-zinc enzyme[J].FEBS Journal, 2011, 278(8):1252-1263.
[13] BURNARD D, GORE L, HENDERSON A, et al.Comparative genomics and antimicrobial resistance profiling of Elizabethkingia isolates reveal nosocomial transmission and in vitro susceptibility to fluoroquinolones, tetracyclines, and trimethoprim-sulfamethoxazole[J].Journal of Clinical Microbiology, 2020, 58(9):e00730-e00720.
[14] DELMAS J, ROBIN F, BITTAR F, et al.Unexpected enzyme TEM-126:role of mutation Asp179Glu[J].Antimicrobial Agents and Chemotherapy, 2005, 49(10):4280-4287.
[15] WANG S C, WEI L, GAO Y J, et al.Novel amikacin resistance genes identified from human gut microbiota by functional metagenomics[J].Journal of Applied Microbiology,2022, 133(2):898-907.
[16] HU R X, ZHANG Q, GU Z M.Whole-genome analysis of the potentially zoonotic Elizabethkingia miricola FL160902 with two new chromosomal MBL gene variants[J].Journal of Antimicrobial Chemotherapy, 2020, 75(3):526-530.
[17] MOR N-BARRIO J, LISA M N, LARRIEUX N, et al.Crystal structure of the metallo-β-lactamase GOB in the periplasmic dizinc form reveals an unusual metal site[J].Antimicrobial Agents and Chemotherapy, 2016, 60(10):6013-6022.
N-BARRIO J, LISA M N, LARRIEUX N, et al.Crystal structure of the metallo-β-lactamase GOB in the periplasmic dizinc form reveals an unusual metal site[J].Antimicrobial Agents and Chemotherapy, 2016, 60(10):6013-6022.
[18] LEIROS H K S, BORRA P S, BRANDSDAL B O, et al.Crystal structure of the mobile metallo-β-lactamase AIM-1 from Pseudomonas aeruginosa:Insights into antibiotic binding and the role of Gln157[J].Antimicrobial Agents and Chemotherapy, 2012, 56(8):4341-4353.
[19] SHROPSHIRE W C, ENDRES B T, BORJAN J, et al.High-level ceftazidime/avibactam resistance in Escherichia coli conferred by the novel plasmid-mediated β-lactamase CMY-185 variant[J].The Journal of Antimicrobial Chemotherapy.2023, 78(10):2442-2450.
[20] FRÖHLICH C, SØRUM V, TOKURIKI N, et al.Evolution of β-lactamase-mediated cefiderocol resistance[J].Journal of Antimicrobial Chemotherapy, 2022, 77(9):2429-2436.
[21] HOOBAN B, JOYCE A, FITZHENRY K, et al.The role of the natural aquatic environment in the dissemination of extended spectrum beta-lactamase and carbapenemase encoding genes:A scoping review[J].Water Research, 2020, 180:115880.
[22] KIEFFER N, GUZM N-PUCHE J, POIREL L, et al.ZHO-1, an intrinsic MBL from the environmental Gram-negative species Zhongshania aliphaticivorans[J].Journal of Antimicrobial Chemotherapy, 2019, 74(6):1568-1571.
N-PUCHE J, POIREL L, et al.ZHO-1, an intrinsic MBL from the environmental Gram-negative species Zhongshania aliphaticivorans[J].Journal of Antimicrobial Chemotherapy, 2019, 74(6):1568-1571.
[23] ZHANG P, SHI Q, HU H, et al.Emergence of ceftazidime/avibactam resistance in carbapenem-resistant Klebsiella pneumoniae in China[J].Clinical Microbiology and Infection, 2020, 26(1):124.e1-124.e4.