γ-谷氨酰转肽酶(γ-glutamyl transpeptidase, GGT, EC:2.3.2.2)是一种N端亲核水解酶,是由大亚基和小亚基组成的异二聚体[1-2]。通过与催化底物谷氨酰胺反应形成γ-谷氨酰-酶复合体,再被受体底物(如氨基酸、短肽)亲核取代,发生转肽反应形成γ-谷氨酰肽[3-5]。近年来,随着减盐政策的提出,γ-谷氨酰肽作为非钠盐替代品,可在不影响食品营养的情况下,提升鲜味强度并增强浓厚味,降低食品中钠盐含量,可解决长期高盐摄入导致的高血压、心血管疾病、脑卒中风和冠心病的问题[6]。此外,γ-谷氨酰肽在医药领域也具有广泛应用,如γ-谷氨酰-L-色氨酸,对肺结核病治疗效果显著[7];γ-L-谷氨酰牛磺酸具有抗癫痫和惊厥的作用[8-9]。因此,利用微生物酶法生产γ-谷氨酰肽已成为研究热点[10-11]。
目前国内外主要采用大肠杆菌(Escherichia coli)、枯草芽孢杆菌(Bacillus subtilis)、谷氨酸棒状杆菌(Corynebacterium glutamicum)等野生型菌株生产γ-谷氨酰转肽酶。然而,这些菌株分泌效率低、表达量不高,且有一部分并不是食品安全菌株。提高酶活力的策略也主要是通过同源过表达,但酶产量依然较低,导致该酶的价格偏高,应用范围和领域受到限制[4,12]。因此提高微生物γ-谷氨酰转肽酶分泌表达能力是提升γ-谷氨酰转肽酶应用的关键问题之一。
根据文献报道,通过信号肽和启动子筛选,构建高分泌表达系统是提升异源蛋白胞外表达量的常用策略。谈沐阳[13]等利用枯草芽孢杆菌内源信号肽替换的方法筛选得到带有SPdacB的重组菌株,其胞外角蛋白酶活力约为出发菌株胞外酶活力的8.1倍。陈龙军等[14]通过构建α-环糊精酶的信号肽文库,筛选获得9条表达α-环糊精酶效率高的信号肽,其中citH信号肽引导分泌效率最高,重组菌株胞外α-环糊精酶活力是野生菌株的14倍。李云菲等[15]通过考察9个单启动子(PamyQ、PamyQ′、PaprE、PgsiB、PHpaII、PnprE、Psrf、Pxyl、Pxyl′)对α/β-环糊精葡萄糖基转移酶表达量的影响,选择α/β CGTase 表达最好的3个单启动子两两组合,得到最优启动子组合PHpaII-PamyQ,重组菌株α/β-环糊精葡萄糖基转移酶活力达到110.4 U/mL。
枯草芽孢杆菌是一种革兰氏阳性好氧细菌,是食品、饲料安全菌株。其具有易培养、高效表达蛋白等特点,是一种优良的工业酶制剂表达宿主。SCK6是枯草芽孢杆菌1A751的衍生菌株,其转化效率较高[16],适用于pDG1730、pHT01等多种芽孢杆菌载体的高效转化。据已有报道,该菌株成功表达过多种外源蛋白,如塔格糖-6-磷酸酶[17-18]。本研究以枯草芽孢杆菌SCK6为表达宿主,选择来源于大肠杆菌的γ-谷氨酰转肽酶基因GGT(NCBI Reference Sequence:WP_218662553.1)、来源于芽胞杆菌的173种信号肽和5组启动子,研究不同启动子和信号肽对γ-谷氨酰转肽酶分泌表达的影响,构建枯草芽孢杆菌高分泌表达系统高产γ-谷氨酰转肽酶,再通过单因素筛选和响应面试验来优化发酵培养基,进一步提高γ-谷氨酰转肽酶的产量,实现生产γ-谷氨酰转肽酶工艺优化,降低生产成本,为γ-谷氨酰转肽酶的产业化生产及开发应用奠定基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
枯草芽孢杆菌B.subtilis SCK6,购于钦诚生物;大肠杆菌E.coli TOP10,生工生物工程(上海)股份有限公司;分泌型蛋白质表达系统B.subtilis Secretory Protein Expression System试剂盒(含有SP DNA mixture和pBE-S DNA质粒),大连宝生物工程(TaKaRa)有限公司;pUC-GGT、pUC-PHpaII-PamyQ、pUC-PaprE、pUC-P43载体委托武汉奥科鼎盛生物科技有限公司合成构建。
1.1.2 试剂
PrimeSTAR® Max DNA Polymerase试剂盒,日本Takara公司;2×Taq PCR Maste rmix,天根生化科技(北京)有限公司;NEBuilder®高保真 DNA 组装预混液试剂盒,NEB公司;卡那霉素,氨苄青霉素,北京索莱宝科技有限公司;DNA回收纯化试剂盒、高纯度质粒DNA小量提取试剂盒,Omega公司;Sure PAGE蛋白预制胶、5×Samper Buffer、Broad Multi Color Pre-Stained Protein Standard、Tris-MOPS-SDS Running Buffer Powder,南京金斯瑞生物科技有限公司;γ-GPNA、Gly-Gly,上海源叶科技有限公司;酵母抽提物FM802、FM902、FP103、FM888、FP101,安琪酵母股份有限公司;其他试剂为国产或进口分析纯;PCR引物合成及测序由武汉奥科鼎盛生物科技有限公司完成。本文中涉及到的引物如表1所示。
表1 本研究使用到的引物
Table 1 Primers used in the study

引物名称引物序列(5′-3′)Z1-FCCGGTGCACATATGATGATCAAACCZ1-RGATAAACTACCGCATTAATAGCCTGCTGTAAGZ2-FCAGGCTATTAATGCGGTAGTTTATCACAGTTAAATTGZ2-RGATCATCATATGTGCACCGGCCGZ3-FGCCGGTGCACATATGATGATCAAACZ3-RCGCGTCCCTCTCCTTTTGCTTAAGTTCAGZ4-FCCTAAACTAGTGGCGGCGTTCZ4-RGCCGATTCTTCATTCTTGACACTCCZ5-FCCTAAACTAGTGTTCGGATTCATCTATGGGZ5-RGATTTTTCATATGTAAATCGCTCCTTTTTAGGTGGZ6-FCCTAAACTAGTTGATAGGTGGTATGTTTTCGCZ6-RCCGATTTTTCATCTATACATTCCTCCAATACCZ7-FGTCAAGAATGAAGAATCGGCTCTTTATTTTGATTTGZ7-RGCCGCCACTAGTTTAGGAACGZ8-FGGAGCGATTTACATATGAAAAATCGGCTCZ8-RGATGAATCCGAACACTAGTTTAGGAACGTACZ9-FGAGGAATGTATAGATGAAAAATCGGCTCTTTATTTTGZ9-RCATACCACCTATCAACTAGTTTAGGAACGTACAG
1.1.3 培养基
LB培养基(g/L):蛋白胨10,酵母粉5,NaCl 10,固体LB培养基添加琼脂粉20。
发酵培养基(g/L):葡萄糖 25,酵母抽提物FM90245,KH2PO4 1.25,K2HPO4 0.75,MgSO4 1.2。
1.2 仪器与设备
C100 Touch PCR仪、Power park Basic 核酸电泳仪、GelDoc GO凝胶成像系统,美国Bio-Rad公司;LRH150生化培养箱,莱特科学仪器;ME204E电子天平,梅特勒-托莱多仪器(上海)有限公司;HH-2恒温水浴锅,常州国华电器有限公司;MX-F小型涡旋器,大龙兴创实验仪器股份公司;BIOSTATB2六联5 L发酵罐,赛多利斯;ACB-4E1-CN超净工作台,ESCO;3-18KS大型冷冻离心机,德国Sigma公司,ZW0615062604紫外可见光分光光度仪,上海光谱仪器有限公司;LG10-2.4A离心机,上海安亭科学仪器厂。
1.3 实验方法
1.3.1 信号肽表达载体文库的构建
以pUC-GGT质粒为模板,使用引物Z1-F/Z1-R PCR扩增获得GGT基因序列;以pBE-S DNA质粒为模板,使用引物Z2-F/Z2-R PCR扩增获得质粒载体骨架基因序列,回收纯化 PCR 产物;利用Gibson组装将2个片段连接后,转化至E.coli TOP10感受态细胞,并将转化菌液涂布于含100 mg/mL氨苄青霉素的LB平板上,37 ℃培养12 h。挑取单菌落进行菌落PCR验证,筛选阳性克隆子。提取质粒进行测序,测序正确的重组质粒命名为pBE-GGT。
以重组质粒pBE-GGT为模板,在信号肽SPaprE上下游分别设计反向引物Z3-F和Z3-R进行PCR扩增,回收纯化PCR产物,将纯化后的PCR产物与SP DNA mixture按比例混合,利用Gibson组装将2个片段进行连接后,转化至E.coli TOP10感受态细胞。并将转化菌液涂布于多个含100 mg/mL氨苄青霉素的LB平板上,37 ℃培养12 h。收集克隆子进行混合培养,提取混合质粒,构建γ-谷氨酰转肽酶信号肽表达载体文库pBE-SP-GGT。
1.3.2 信号肽筛选与鉴定
采用电转化法,将1.3.1节的混合质粒电转化至B.subtilis SCK6感受态细胞中,并将转化菌液涂布于含100 mg/mL卡那霉素的LB平板上,37 ℃培养12 h。挑取单菌落进行菌落PCR验证,筛选阳性克隆子,得到携带不同信号肽的重组菌株。将得到的重组菌株接种至装有100 mg/mL卡那霉素+LB培养基的96孔板中,37 ℃、800 r/min振荡培养24 h获得种子液,再按2%(体积分数)的接种量接种至装有100 mg/mL卡那霉素+发酵培养基的48孔板中,37 ℃振荡培养48 h,发酵液5 000 r/min 离心10 min,收集上清液,测定γ-谷氨酰转肽酶酶活。筛选出γ-谷氨酰转肽酶酶活高的重组菌株,进行摇瓶复筛。再挑取γ-谷氨酰转肽酶活力最高的重组菌株提取质粒并命名为pBE-GGT-M进行测序。
1.3.3 不同启动子表达载体的构建
选用3种单启动子(PHpaII,PamyQ,P43)及1种双启动子(PamyQ-PHpaII)替换原始启动子PaprE探究不同启动子对γ-谷氨酰转肽酶表达量的影响,以pUC-PamyQ-PHpaII、pUC-P43质粒为模板,分别使用引物Z4-F/Z4-R、Z5-F/Z5-R、Z6-F/Z6-R、Z2-F/Z1-R PCR扩增获得相对应的启动子基因序列;以pBE-GGT-M质粒为模板,分别使用引物Z7-F/Z7-R、Z8-F/Z8-R、Z9-F/Z9-R、Z7-F/Z8-R PCR扩增获得相对应的载体骨架基因序列,回收纯化PCR产物;利用Gibson组装将2个片段进行连接后,转化至E.coli TOP10感受态细胞,经含100 mg/mL氨苄青霉素的LB平板和菌落PCR筛选阳性克隆子。提取质粒送测序,测序正确的重组质粒命名为pBE-GGT1、pBE-GGT2、pBE-GGT3、pBE-GGT4。
1.3.4 重组菌株的构建与表达
将上述重组质粒和重组质粒pBE-GGT-M用电转法转化到B.subtilis SCK6,经含100 mg/mL卡那霉素的LB平板和菌落PCR筛选阳性克隆子,分别得到5株重组菌株BS-gts1、BS-gts2、BS-gts3、BS-gts4、BS-gts5。将5株重组菌株各接种至装有2 mL LB培养基+100 mg/L卡那霉素的10 mL摇菌管中,37 ℃、180 r/min过夜振荡培养,得到种子液。将种子液按2%(体积分数)的接种量,接种至装有100 mL发酵培养基+100 mg/L卡那霉素的250 mL三角瓶中,37 ℃、180 r/min振荡培养。每24 h取样,发酵液12 000 r/min 离心2 min,收集上清液,测定γ-谷氨酰转肽酶活力。
1.3.5 发酵培养基优化
1.3.5.1 不同碳源对重组菌株产酶的影响
用蔗糖、乳糖、麦芽糖、麦芽糊精替换原始发酵培养基中的葡萄糖,37 ℃、180 r/min振荡培养6 d。通过测定胞外酶活力筛选出最佳碳源。
1.3.5.2 不同氮源对重组菌株产酶的影响
用FP103、FM802、FM888、FP101替换原始发酵培养基中的FM902,37 ℃、180 r/min振荡培养6 d。通过测定胞外酶活力筛选出最佳氮源。
1.3.5.3 不同无机盐对重组菌株产酶的影响
用CaCl2、KCl、MnCl2·4H2O、NaCl替换原始发酵培养基中的MgSO4,37 ℃、180 r/min振荡培养6 d。通过测定胞外酶活力筛选出最佳无机盐。
1.3.5.4 不同碳源浓度对重组菌株产酶的影响
以重组菌株胞外酶活力为指标,设置20、25、30、35、40 g/L的质量浓度梯度,其他条件不变,筛选出最优碳源浓度。
1.3.5.5 不同氮源浓度对重组菌株产酶的影响
以重组菌株胞外酶活力为指标,设置40、45、50、55、60 g/L的质量浓度梯度,其他条件不变,筛选出最优氮源浓度。
1.3.5.6 不同无机盐浓度对重组菌株产酶的影响
以重组菌株胞外酶活力为指标,设置7.5、10、12.5、15.0、17.5 mmol/L的浓度梯度,其他条件不变,筛选出最优无机盐浓度。
1.3.6 响应面优化
根据单因素试验的结果,选取最优碳源添加量(A)、最优氮源添加量(B)和最优无机盐添加量(C)3个因素,每个因素分别设置高低2个水平(-1,1),以γ-谷氨酰转肽酶活力(Y)为响应值进行实验。
1.3.7 重组菌株5 L发酵罐放大培养
在1 L摇瓶中使用含有100 mg/L卡那霉素的LB培养基制备种子液,将种子液接种至2 L的已灭菌发酵液中(接种量10 %,体积分数)。以氨水和盐酸调控pH,培养温度37 ℃,通过搅拌转速偶联及调节通气量维持溶氧在20%~40%,当溶氧、pH值迅速上升,开始流加质量浓度为500 g/L的葡萄糖补料液,全程控制发酵液葡萄糖含量为10~20 g/L,当γ-谷氨酰转肽酶活力下降时结束培养。
1.3.8 酶活力测定方法
采用分光光度计法检测γ-谷氨酰转肽酶活力[19]。反应体系(1 mL):490 mL 10 mmol γ-GPNA、490 μL 40 mmol Gly-Gly,20 mL适当稀释的酶液,在37 ℃水浴锅中反应10 min,加1 mL 0.1 mol/L盐酸终止反应;在410 nm波长下测定吸光值。该条件下每分钟生成1 μmol对硝基苯胺所需的酶量定义为一个酶活力单位。
1.3.9 SDS-PAGE 分析
取25 mL发酵上清液与5 mL上样电泳缓冲液混合,将混合液在沸水中煮沸10 min,待冷却后离心取上清液,作为电泳样品。采用12 %的分离胶进行SDS-PAGE凝胶电泳。电泳结束后,使用考马斯亮蓝染色液染色1 h,使用脱色液脱色,最后使用凝胶成像系统检测蛋白条带。
2 结果与分析
2.1 信号肽表达载体文库的构建
SP DNA mixture包含能编码173种B.subtilis分泌信号肽的DNA片段,本文运用同源重组的方法将线性载体pBE-GGT与SP DNA mixture进行组装连接,并将连接产物转化至E.coli TOP10感受态细胞,获得1 523个克隆子,随机挑选10个克隆子(对映表2 中菌株编号1~10)提取质粒送测序考察信号肽分布情况,对比结果如表2所示。测序结果经比对检测出6种不同信号肽序列。表明不同信号肽序列在克隆子中有一定的分布,收集克隆子进行混合培养,提取混合质粒,构建γ-谷氨酰转肽酶信号肽表达载体文库pBE-SP-GGT。
表2 信号肽氨基酸序列表
Table 2 Amino acid sequence of signal peptide

菌株信号肽名称信号肽氨基酸序列1、2、3、7、8aprEMRSKKLWISLLFALTLIFTMAFSNMSAQA4YlbLMLRKKHFSWMLVILILIAVLSFIKLPYYITKPGEA5PhrAMKSKWMSGLLLVAVGFSFTQVMVHA6XynAMFKFKKNFLVGLSAALMSISLFSATASA9MreCMPNKRLMLLLLCIILVAMIGFS10YwoFMRLWYFILLAGVLTSVILAFVYDKTKA
2.2 信号肽的筛选与鉴定
用电转法将混合质粒转化到B.subtilis SCK6,经100 mg/L氨苄青霉素抗性平板过夜培养筛选克隆子,获得689个克隆子,通过48孔板(37 ℃,800 r/min,48 h)对689个克隆子进行初步表达分析(图1),将689株重组γ-谷氨酰转肽酶菌株进行48孔板发酵,发酵上清液酶活力检测结果热图如图1所示,其中颜色越深条纹越复杂表明检测酶活力越高,颜色越浅条纹越简单表明酶活力越低。经酶活力检测初筛,其中第176、183、310、396、640株菌株酶活力较高,进行摇瓶复筛,胞外酶活力检测结果如图2所示。其中640号菌株胞外酶活力明显高于其他菌株,提取质粒送测序,鉴定信号肽序列。信号肽序列对比结果为SPcotC(5′-3′MKNRLFILICFCVICLFLSFGQPFFPSMILTVQAAKS)。
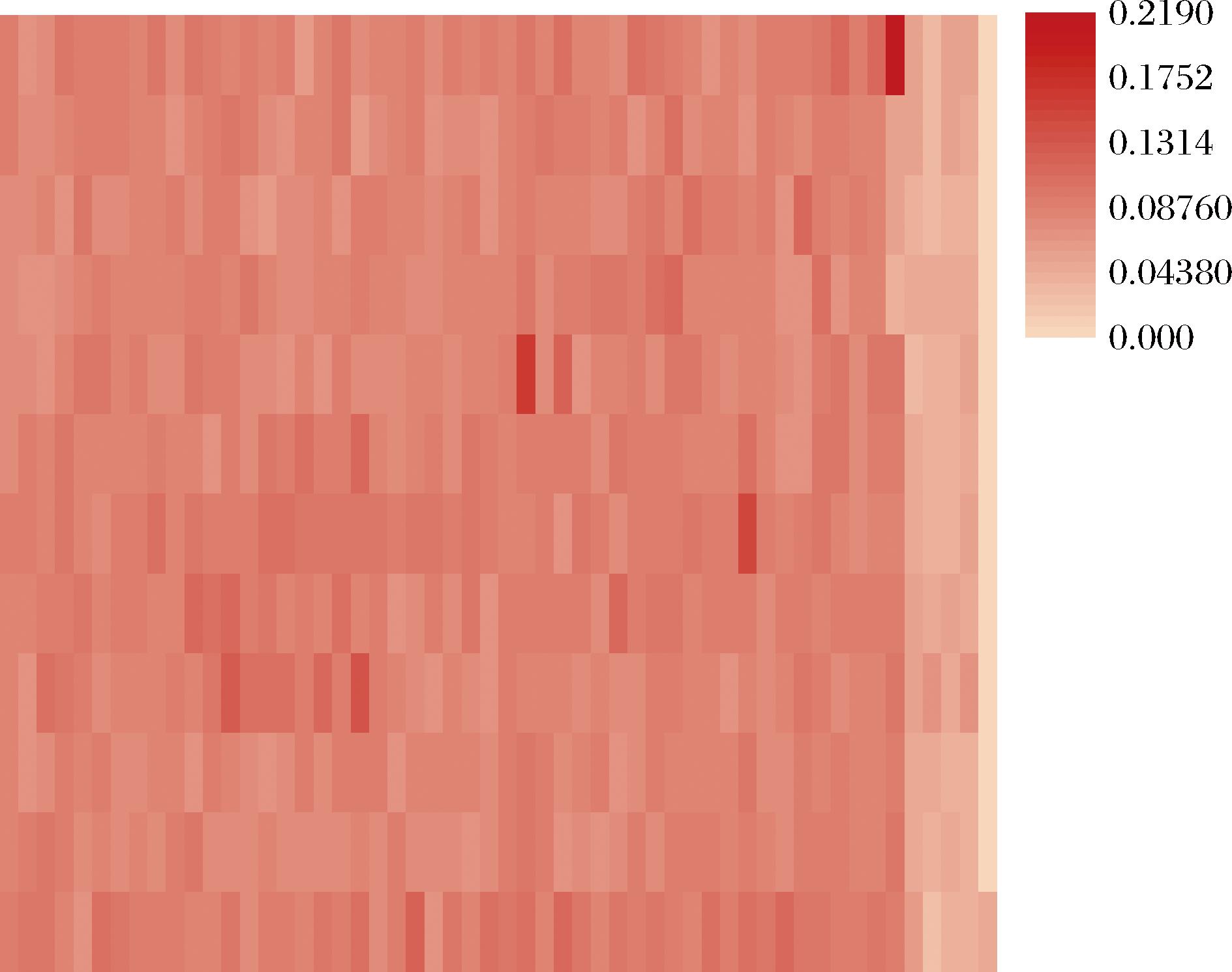
图1 重组菌株γ-谷氨酰转肽酶酶活初筛结果热图
Fig.1 Heat map analysis of γ-glutamyl transpeptidase enzyme activity of recombinant strains
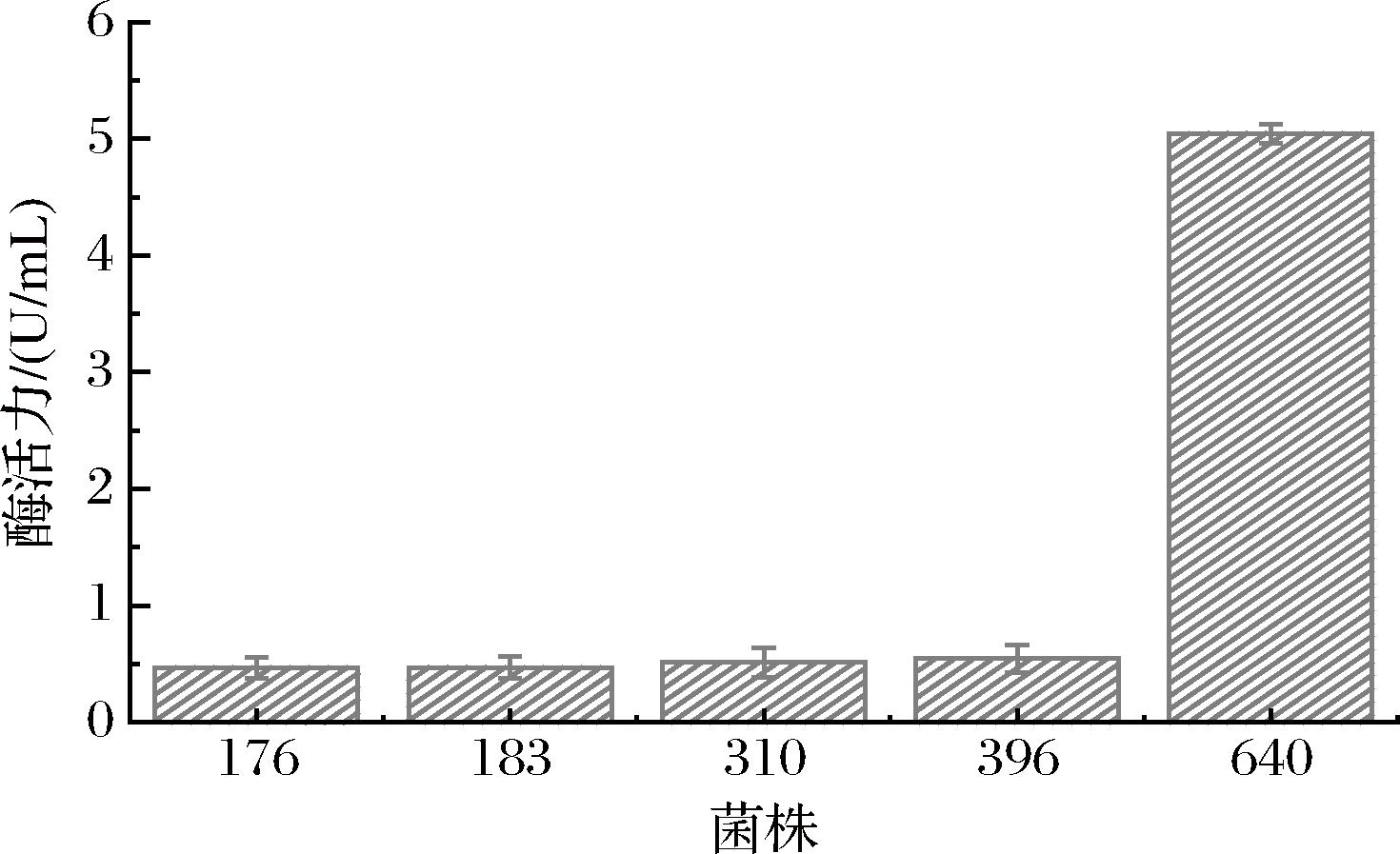
图2 不同重组菌株摇瓶发酵胞外γ-谷氨酰转肽酶酶活力
Fig.2 Extracellular γ-glutamyl transpeptidase enzyme activity of different recombinants in shake flask fermentation
2.3 不同启动子表达载体的构建
选择3种单启动子(PamyQ、PHpaII、P43)和1种双启动子(PHpaII+PamyQ)替换原始启动子PaprE,构建含不同启动子的表达载体。利用不同引物分别PCR扩增启动子PamyQ、PHpaII+PamyQ、P43与pBE载体骨架片段,琼脂糖凝胶电泳检测扩增产物,将扩增产物进行回收,回收产物进行电泳检测,检测结果见图3,利用Gibson组装将启动子片段与骨架进行组装连接。重组产物转化E.coli TOP10感受态细胞,筛选阳性克隆子。提取质粒送测序,获得重组质粒pBE-GGT1、pBE-GGT2、pBE-GGT3、pBE-GGT4。
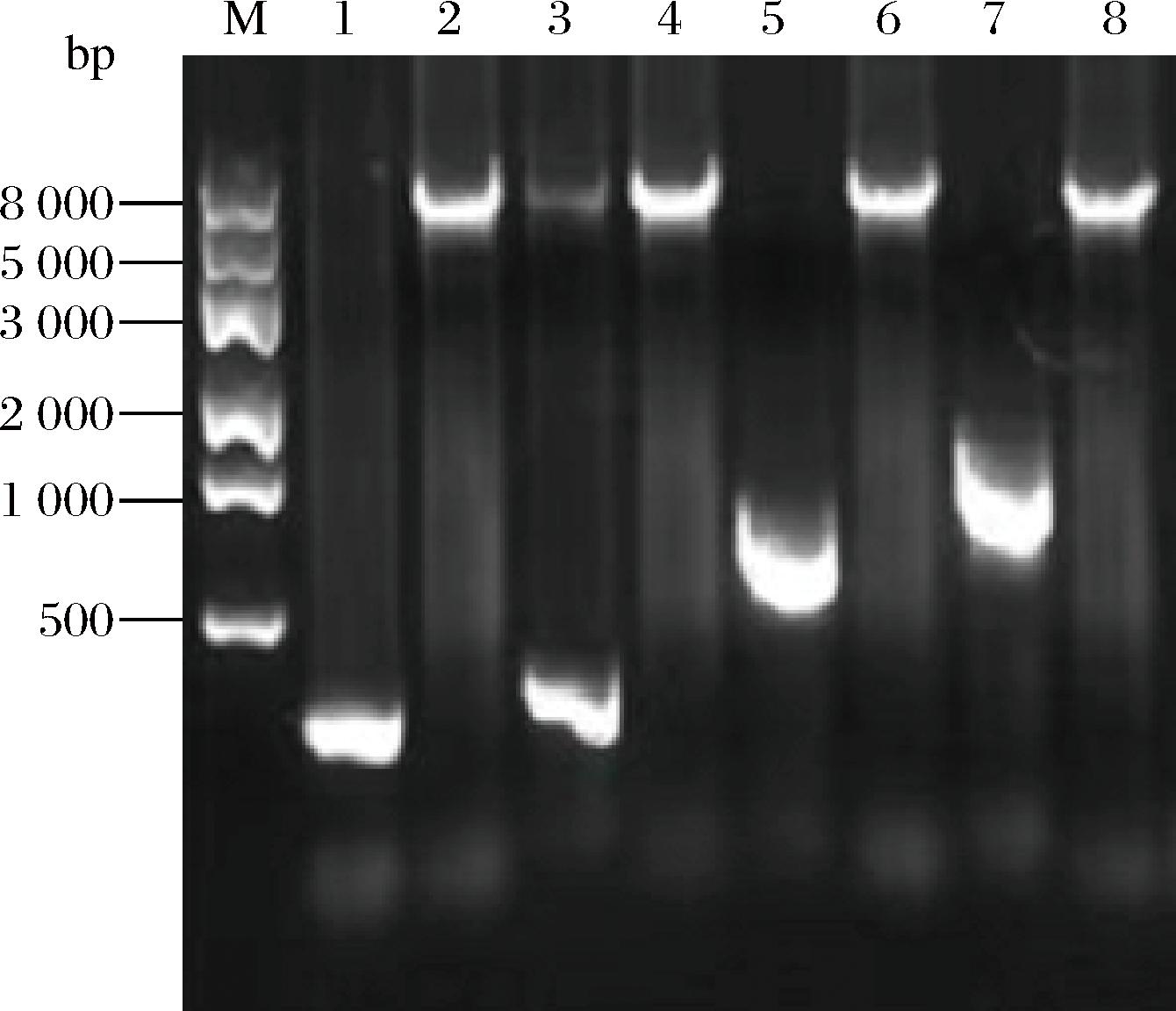
M:DNA Marker;泳道1、2-P43与载体骨架;泳道3、4-PamyQ与载体骨架;泳道5、6-PHpaII与载体骨架;泳道7、8-PHpaII+PamyQ与载体骨架
图3 扩增片段回收产物电泳
Fig.3 Amplified fragment recovery product electropherogram
2.4 重组菌株的构建与表达
本研究在信号肽SPcotC的基础上选用5种不同启动子进行探究。通过电转法重组质粒转Bacillus subtilis SCK6,获得转化子。用相应引物进行PCR验证,均得到相对应大小的条带,PCR产物的琼脂糖凝胶电泳结果见图4,表明含不同启动子序列的重组质粒成功转化入宿主菌中,重组菌株BS-gts1、BS-gts2、BS-gts3、BS-gts4、BS-gts5成功构建。对得到的5株菌株进行摇瓶发酵,并测定酶活力(图5),其中启动子为PaprE的重组菌株,其胞外酶活力为5.04 U/mL,远远高于含有其他启动子的重组菌株。推测启动子PaprE和信号肽SPcotC之间的碱基序列或RBS位点与转录起始位点的距离适宜,匹配性良好,从而提高了转录水平,导致酶的表达量提高。
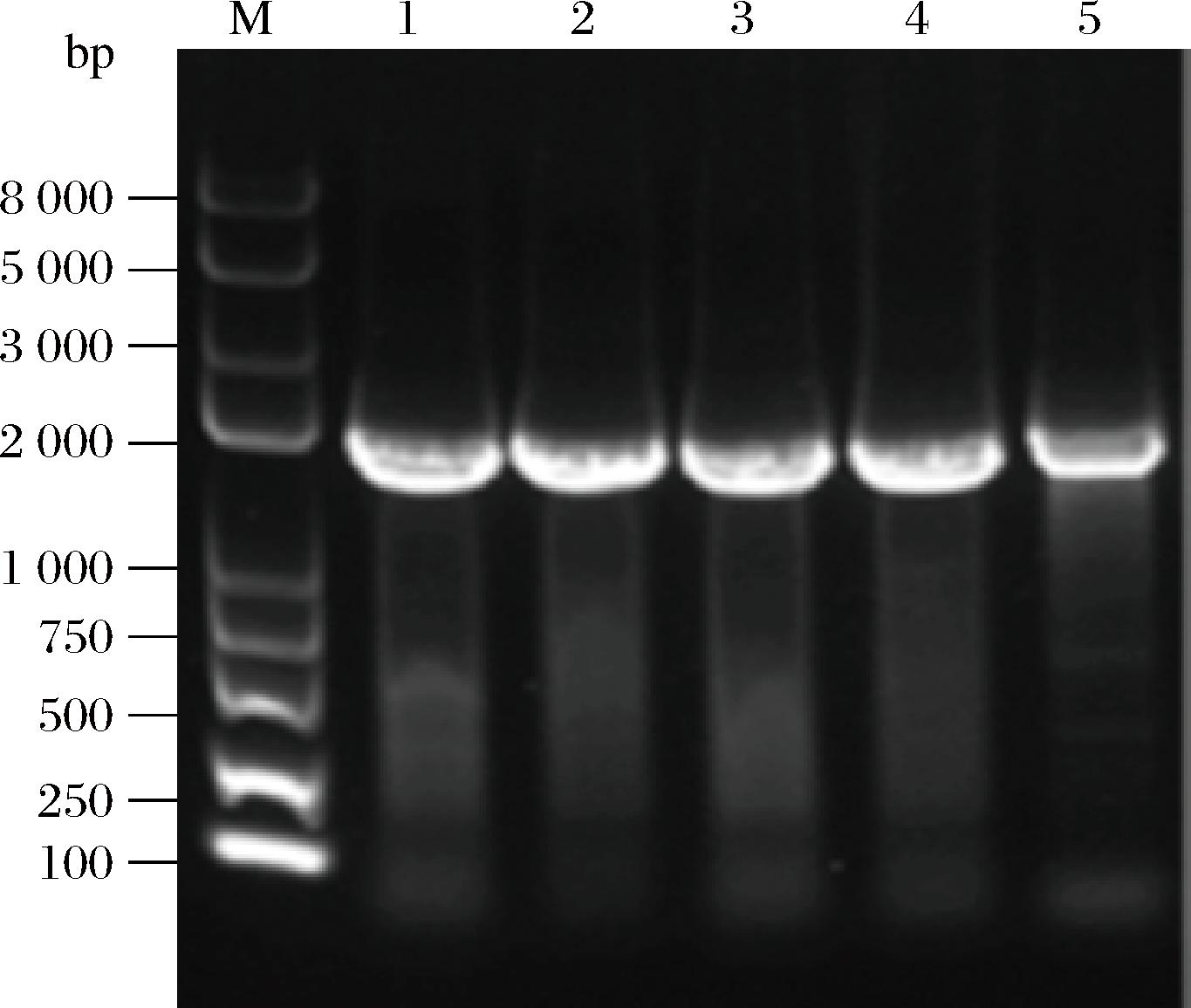
M:DNA Marker;泳道1-PamyQ;泳道2-PHpaII;泳道3-PaprE;泳道4-P43;泳道5-PHpaII+PamyQ
图4 重组菌株PCR验证
Fig.4 PCR confirmation of recombinant strains
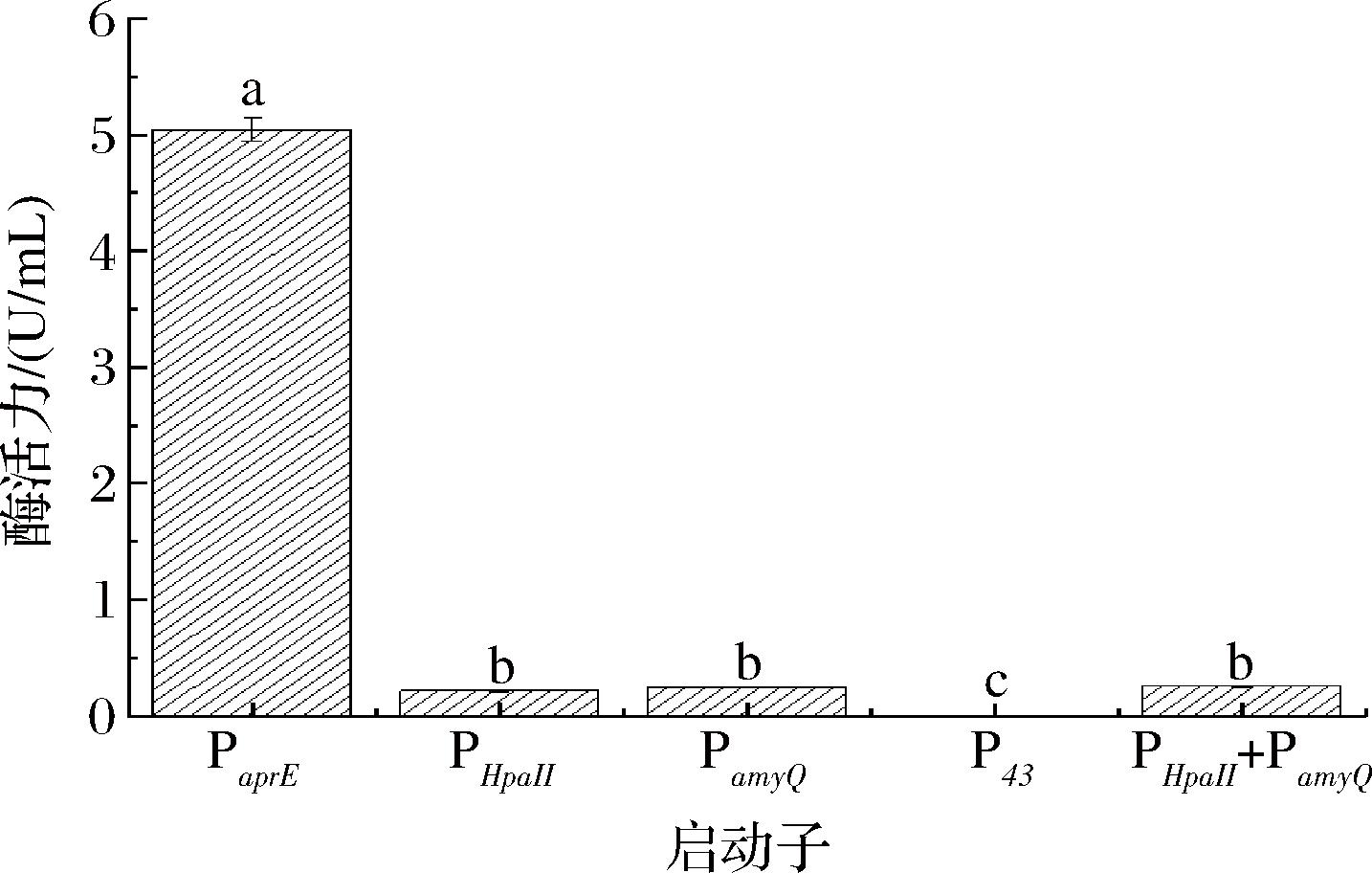
图5 不同启动子对γ-谷氨酰转肽酶表达的影响
Fig.5 Effects of different promoters on γ-glutamyl transpeptidase enzyme expression
注:不同小写字母表示差异显著(P<0.05)(下同)。
2.5 发酵培养基优化
培养基是微生物生存的环境,其营养成分的组成和浓度对微生物的生长和目的代谢产物的产率具有巨大影响。因而,对培养基营养成分的筛选和分析具有重要意义。本研究通过单因素试验对培养基碳源、氮源、无机盐种类及添加量进行筛选。图6结果表明,最优碳源为乳糖,当随着乳糖添加量增加,重组菌株胞外酶活力先增加后降低。当质量浓度为30 g/L时,胞外酶活力最高为5.26 U/mL;最优氮源为酵母粉FM802,当质量浓度为50 g/L时,胞外酶活力最高为5.55 U/mL;最优无机盐为MgSO4,当浓度为10 mmol/L时,胞外酶活力为4.69 U/mL。
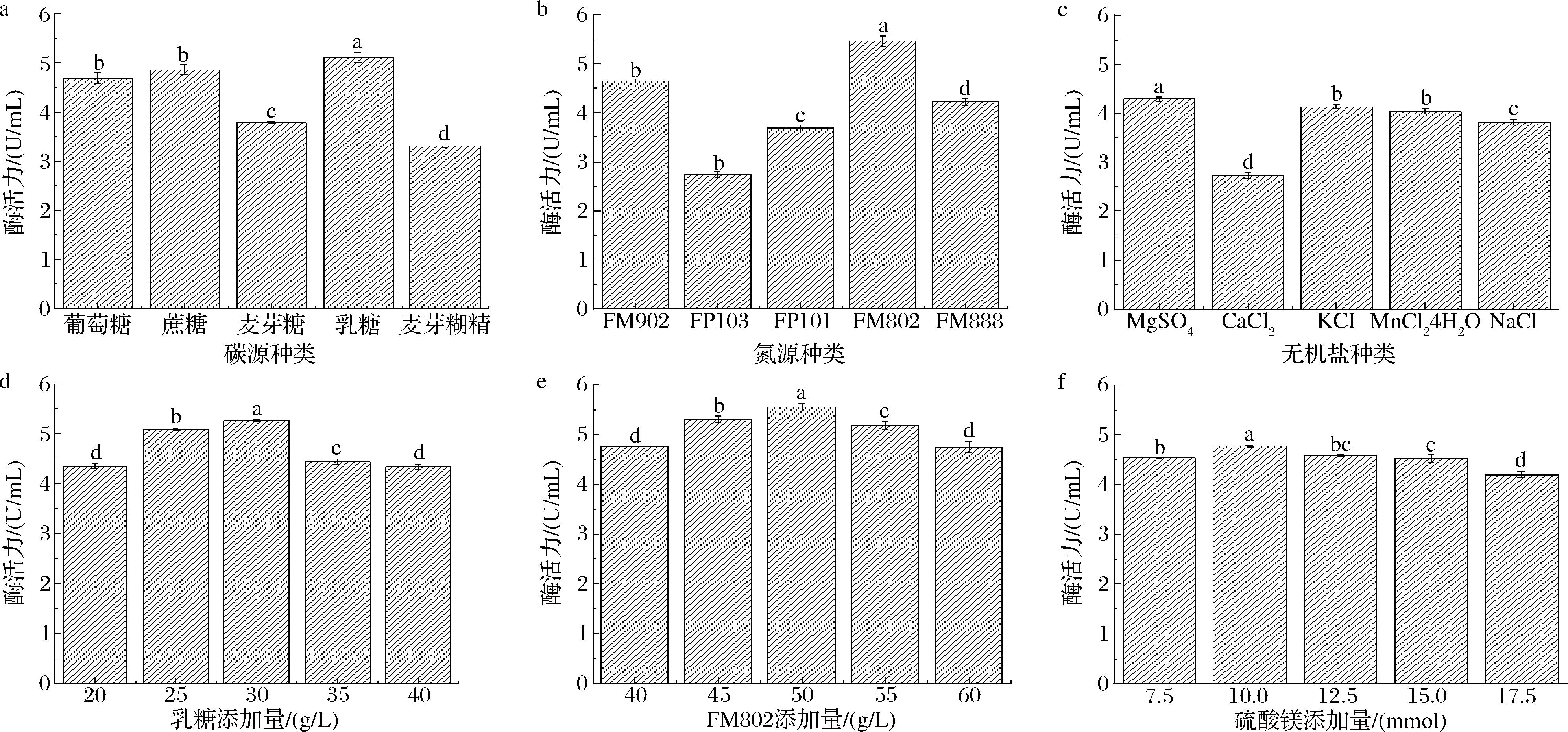
a-碳源种类;b-氮源种类;c-无机盐种类;d-乳糖添加量;e-FM802添加量;f-硫酸镁添加量
图6 培养基组分对重组菌株发酵产γ-谷氨酰转肽酶的影响
Fig.6 Effect of medium components on the production of γ-glutamyl transpeptidase by fermentation of recombinant strain
2.6 响应面优化
根据单因素试验的结果,选取乳糖添加量(A)、FM802添加量(B)和MgSO4添加量(C)3个因素,每个因素分别设置高低2个水平(-1,1),以γ-谷氨酰转肽酶酶活力(Y)为响应值进行实验。结果见表4。
表4 响应面实验结果
Table 4 Response surface experiment results
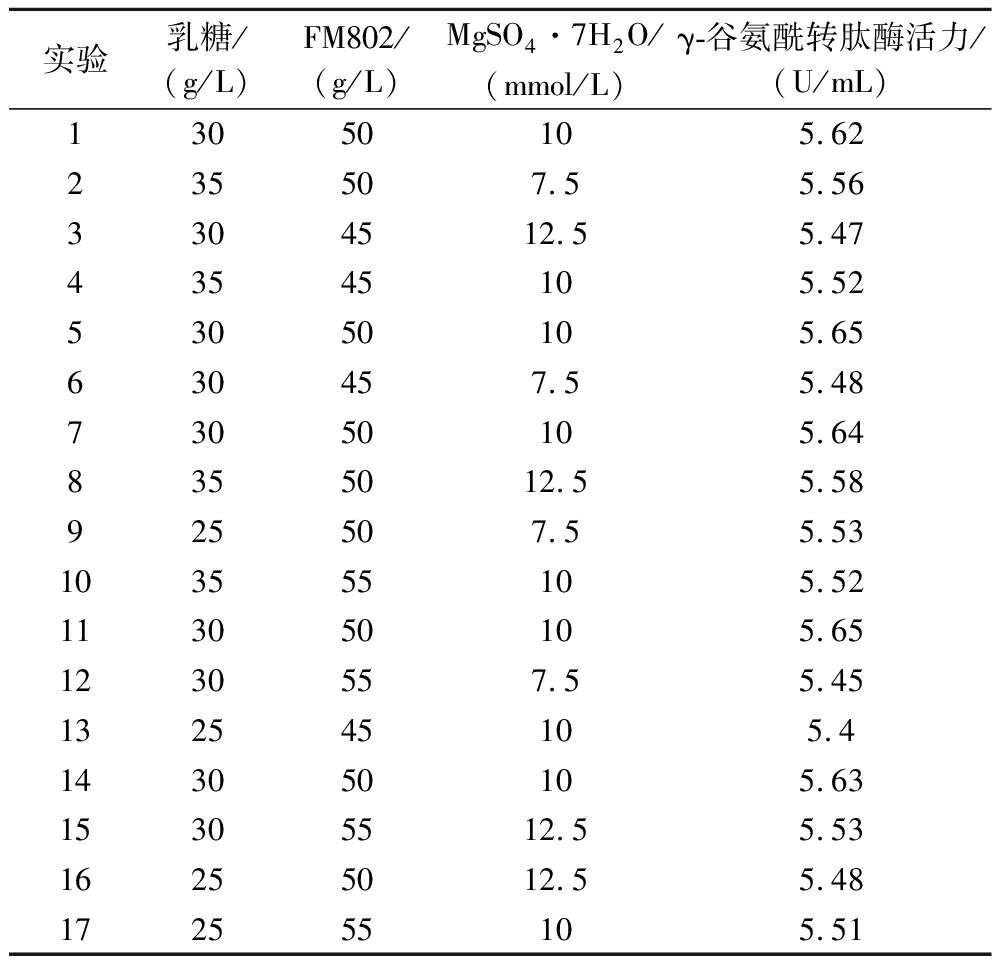
实验乳糖/(g/L)FM802/(g/L)MgSO4·7H2O/(mmol/L)γ-谷氨酰转肽酶活力/(U/mL)13050105.62235507.55.563304512.55.4743545105.5253050105.65630457.55.4873050105.648355012.55.58925507.55.53103555105.52113050105.651230557.55.45132545105.4143050105.6315305512.55.5316255012.55.48172555105.51
利用Design Expert10.0 软件响应面优化实验的设计及结果及方差分析结果见上表,以γ-谷氨酰转肽酶产量为响应值,对数据进行回归分析,可得到如下方程:Y=5.64+0.032 5A+0.0175B+0.005C-0.027 5AB+0.0175AC+0.022 5BC-0.047 8A2-0.102 8B2-0.052 7C2。
对回归方程进行方差分析,结果如表5。由表5可知,模型P=0.000 2<0.05,显著;失拟项P=0.106 2>0.05,不显著;决定系数R2=0.970 4,表明该模型拟合度较好;校正决定系数![]() 说明模型的可行度较高,可用于重组菌株产酶的预测。乳糖和酵母粉FM802都显著影响重组菌株产酶,且对重组菌株产酶的影响大小为乳糖>酵母粉FM802。
说明模型的可行度较高,可用于重组菌株产酶的预测。乳糖和酵母粉FM802都显著影响重组菌株产酶,且对重组菌株产酶的影响大小为乳糖>酵母粉FM802。
表5 响应面实验结果方差分析
Table 5 Variance analysis of response surface experiment results
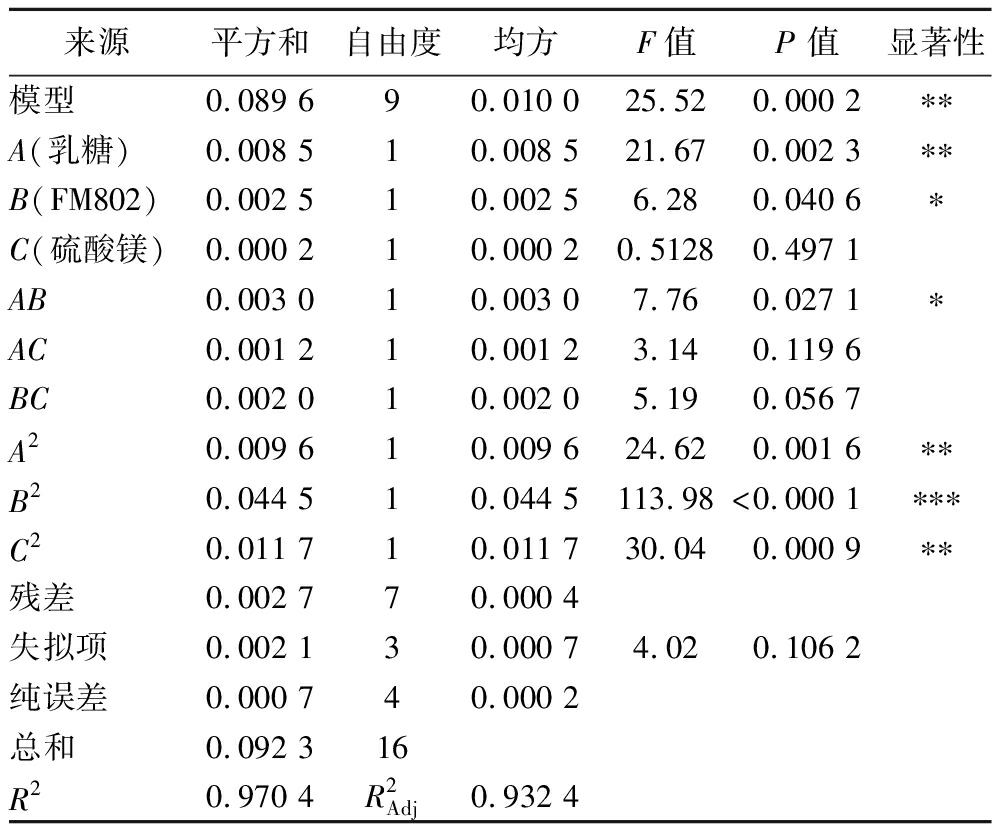
来源平方和自由度均方F值P 值显著性模型0.089 690.010 025.520.000 2∗∗A(乳糖)0.008 510.008 521.670.002 3∗∗B(FM802)0.002 510.002 56.280.040 6∗C(硫酸镁)0.000 210.000 20.51280.497 1AB0.003 010.003 07.760.027 1∗AC0.001 210.001 23.140.119 6BC0.002 010.002 05.190.056 7A20.009 610.009 624.620.001 6∗∗B20.044 510.044 5113.98<0.000 1∗∗∗C20.011 710.011 730.040.000 9∗∗残差0.002 770.000 4失拟项0.002 130.000 74.020.106 2纯误差0.000 740.000 2总和0.092 316R20.970 4R2Adj0.932 4
注:P<0.05,差异显著,标记为*;P<0.01,差异非常显著,标记为**;P<0.000 1,差异极显著,标记为***。
不同因素相互作用对γ-谷氨酰转肽酶活力影响的相应面图结果表明(图7),乳糖与酵母粉FM802有交互作用,硫酸镁与乳糖、酵母粉FM802无交互作用。对得到的回归方程进行求解以得到γ-谷氨酰转肽酶活力的极大值,得出培养基乳糖质量浓度为31.73 g/L、FM802质量浓度为50.26 g/L、硫酸镁质量浓度为1.24 g/L时,预测的重组菌株胞外酶活力为5.644 U/mL。在此条件下进行3组平行实验,得到重组菌胞外酶活力分别为5.623、5.638、5.606 U/mL,平均值为5.623 U/mL,达到预测值的99.61%,较优化前提高了12%。
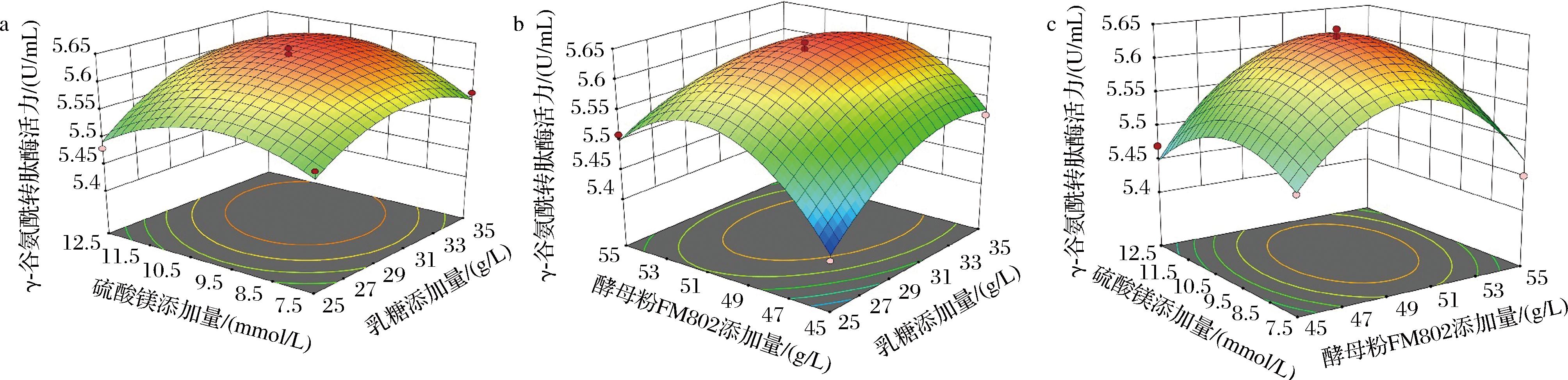
a-硫酸镁和乳糖;b-FM802和乳糖;c-硫酸镁和FM802
图7 各因素交互影响的响应面图
Fig.7 Response surface diagram of interaction of various factors
2.5 重组菌株5 L发酵罐放大培养
以最优培养基为基础,利用5 L发酵罐进行3次平行实验,对重组菌株进行放大培养研究。定期取样以记录重组菌株BS-gts3的生长和酶产生情况。(图8-a),可知前22 h重组菌株生长迅速,γ-谷氨酰转肽酶分泌表达量也随之快速增长,整体趋势为酶活力随着重组菌株生物量的增长而增长。在38 h时,γ-谷氨酰转肽酶活力最高,达到28.18 U/mL。对发酵液上清液进行SDS-PAGE电泳(图8-B),γ-谷氨酰转肽酶分子质量大小为44 kDa,从6 h~40 h,目的蛋白条带随着发酵时间的延长而逐渐变多,表明γ-谷氨酰转肽酶的表达量随发酵时间延长而不断累积。

a-5 L罐发酵曲线;b-SDS-PAGE电泳图
图8 菌株BS-gts3于5 L发酵罐中的发酵曲线及SDS-PAGE分析
Fig.8 Fermentation profile and SDS-PAGE analysis of strain BS-gts3 in a 5 L fermenter
注:M代表Marker。
3 结论
本研究旨在通过优化枯草芽孢杆菌蛋白表达中的关键基因元件以及发酵培养基优化,高效表达来源于大肠杆菌的γ-谷氨酰转肽酶。研究采用启动子和信号肽筛选策略,以来源于枯草芽胞杆菌的173种信号肽为基础构建信号肽文库,筛选出分泌γ-谷氨酰转肽酶效率最高信号肽SPcotC。为了进一步增强γ-谷氨酰转肽酶的表达,筛选了4个单启动子和1个双启动子,得出最优启动子PaprE,最终获得1株表达水平较高的γ-谷氨酰转肽酶重组工程菌株BS-gts3,其摇瓶发酵胞外最高酶活力为5.04 U/mL。通过单因素试验和响应面优化培养基后,胞外酶活力提高了12%,为5.623 U/mL,高于目前报道的2.6 U/mL[20],是已报道同等条件下分泌表达大肠杆菌来源的γ-谷氨酰转肽酶的最高水平。并进行了5 L罐放大培养,在38 h,胞外酶活力达到最高28.18 U/mL。后期将通过优化补料策略、阶段溶氧控制等方式提高重组菌株发酵产酶效率,并研究酶学性质和提取纯化方法,为γ-谷氨酰转肽酶的产业化及酶的后续产品开发奠定基础。
[1] AMINO Y, WAKABAYASHI H, AKASHI S, et al.Structural analysis and taste evaluation of γ-glutamyl peptides comprising sulfur-containing amino acids[J].Bioscience, Biotechnology, and Biochemistry, 2018, 82(3):383-394.
[2] YANG J, BAI W D, ZENG X F, et al.Gamma glutamyl peptides:The food source, enzymatic synthesis, kokumi-active and the potential functional properties-A review[J].Trends in Food Science &Technology, 2019, 91:339-346.
[3] 和斐. 利用重组钝齿棒杆菌高效表达γ-谷氨酰转肽酶及酶催化合成L-茶氨酸研究[D].无锡:江南大学, 2016.HE F.Efficient expression of γ-glutamyl transpeptidase and enzyme-catalysed synthesis of L-theanine using recombinant Corynebacterium obtusum[D].Wuxi:Jiangnan University, 2016.
[4] 熊爱英. C.glutamicum γ-谷氨酰转肽酶的重组表达、酶学性质及其在谷氨酸发酵中应用的研究[D].南京:南京师范大学, 2016.XIONG A Y.Study on recombinant expression, enzymatic properties of C.glutamicum γ-glutamyl transpeptidase and its application in glutamate fermentation[D].Nanjing:Nanjing Normal University, 2016.
[5] 刘栓英.γ-谷氨酰转肽酶分子改造及其应用[D].无锡:江南大学, 2021.LIU S Y.Molecular modification of γ-glutamyl transpeptidase and its application[D].Wuxi:Jiangnan University, 2021.
[6] 辛世华, 韩小珍, 贺晓光.食品中γ-谷氨酰肽的研究进展[J].中国酿造, 2021, 40(12):22-28.XIN S H, HAN X Z, HE X G.Research progress of γ-glutamyl peptides in food[J].China Brewing, 2021, 40(12):22-28.
[7] AMINO Y, NAKAZAWA M, KANEKO M, et al.Structure-CaSR-activity relation of Kokumi γ-glutamyl peptides[J].Chemical &Pharmaceutical Bulletin, 2016, 64(8):1181-1189.
[8] 伍伦杰, 伍圆明, 孙伟峰, 等.Kokumi肽及其受体的研究进展[J].中国调味品, 2019, 44(3):189-197.WU L J, WU Y M, SUN W F, et al. Research progress of kokumi peptides and kokumi receptor[J]. China Condiment, 2019, 44(3):189-197.
[9] 黄煜燃, 杨娟, 汪薇, 等.Kokumi肽的特性及其制备研究进展[J].食品工业科技, 2020, 41(21):339-344.HUANG Y R, YANG J, WANG W, et al.Research progress on characteristics and preparation of kokumi peptides[J].Science and Technology of Food Industry, 2020, 41(21):339-344.
[10] LEE Y C, CHI M C, LIN M G, et al. Biocatalytic Synthesis of γ-glutamyl-L-leucine, a Kokumi-Imparting Dipeptide, by Bacillus licheniformis γ-Glutamyltranspeptidase[J]. Food Biotechnology, 2018, 32(2):130-147.
[11] PICA A, CHI M C, CHEN Y Y, et al.The maturation mechanism of γ-glutamyl transpeptidases:Insights from the crystal structure of a precursor mimic of the enzyme from Bacillus licheniformis and from site-directed mutagenesis studies[J].Biochimica et Biophysica Acta, 2016, 1864(2):195-203.
[12] SAINI M, KASHYAP A, BINDAL S, et al.Bacterial γ-glutamyl transpeptidase, an emerging biocatalyst:Insights into structure-function relationship and its biotechnological applications[J].Frontiers in Microbiology, 2021, 12:641251.
[13] 谈沐阳, 陈希文, 彭政, 等.信号肽筛选和核糖体结合位点优化的组合策略提高角蛋白酶在枯草芽孢杆菌中的胞外表达[J].食品与发酵工业, 2023, 49(4):8-14.TAN M Y, CHEN X W, PENG Z, et al.Enhancing the extracellular expression of Bacillus licheniformis keratinase in Bacillus subtilis by combinatorial strategies of signal peptides screening and ribosome binding site optimization[J].Food and Fermentation Industries, 2023, 49(4):8-14.
[14] 陈龙军, 林陈强, 贾宪波, 等.信号肽筛选优化提高耐热α-环糊精酶在枯草芽胞杆菌中的表达[J].福建农业学报, 2022, 37(3):414-422.CHEN L J, LIN C Q, JIA X B, et al.Enhanced thermophilic α-cyclodextrin glycosyltransferase expression by optimizing target signal peptide in Bacillus subtilis[J].Fujian Journal of Agricultural Sciences, 2022, 37(3):414-422.
[15] 李云菲, 宿玲恰, 吴敬.启动子对枯草芽孢杆菌表达环糊精葡萄糖基转移酶的影响[J].基因组学与应用生物学, 2019, 38(8):3540-3547.LI Y F, SU L Q, WU J.Effect of promoter on the expression of cyclodextrin glucosyltransferase by Bacillus subtilis[J].Genomics and Applied Biology, 2019, 38(8):3540-3547.
[16] 李信志, 卢争辉, 周玉玲, 等.枯草芽孢杆菌 SCK6 超级感受态的制备和转化条件优化[J].生物工程学报, 2017, 33(4):692-698.LI X Z, LU Z H, ZHOU Y L, et al.Preparation and transformation optimization for supercompetent B.subtilis SCK6 cells[J].Chinese Journal of Biotechnology, 2017, 33(4):692-698.
[17] YU X X, XU J T, LIU X Q, et al.Identification of a highly efficient stationary phase promoter in Bacillus subtilis[J].Scientific Reports, 2015, 5:18405.
[18] CAI D, WEI X, QIU Y, et al.High-level expression of nattokinase in Bacillus licheniformis by manipulating signal peptide and signal peptidase[J].Journal of Applied Microbiology, 2016, 121(3):704-712.
[19] BOANCA G, SAND A, OKADA T, et al.Autoprocessing of Helicobacter pylori γ-glutamyltranspeptidase leads to the formation of a threonine-threonine catalytic dyad[J].The Journal of Biological Chemistry, 2007, 282(1):534-541.
[20] 岩崎惠理子. γ-谷氨酰缬氨酰甘氨酸晶体的制备方法[P].日本:CN104540959B,2018-11-13.ERIKO I.Preparation of γ-glutamyl-valyl-glycine crystals [P].Japan:CN104540959B,2018-11-13.