新霉素(neomycin, Nm)是一种氨基糖苷类抗生素,从弗氏链霉菌发酵产物中分离纯化得到[1],新霉素可以通过口服、注射等方式给动物使用,改善肠道健康并提高饲料利用率,从而增加畜禽的生长速度和生产性能[2-4]。新霉素具有良好的抗菌活性,通过与核糖体30S亚基16S rRNA部位结合导致蛋白质错译、抑制核糖体30S亚基的组装、产生羟自由基以及抑制核糖核酸酶活力等方式产生抗菌活性[5-7]。其中,新霉素A又被称为新霉胺,是新霉素生物合成过程的中间产物,其对人体的耳毒性和肾毒性最低,但含量也最少,一般可以忽略不计;新霉素B的抑菌活性最高,含量一般也很高,是新霉素中的主要成分;新霉素C是新霉素生物合成的最终产物,与新霉素B分子式相同但生物活性较低,且对人体毒性较大[8]。近年国内外对提升新霉素产量的研究主要集中在菌种改良以及发酵条件优化上,在分子层面上,研究代谢网络中涉及的关键酶及代谢调控因子对提高菌种合成新霉素能力是一个有效的策略。目前对代谢途径的关键酶已研究的较为透彻,主要位于neo基因簇上,包含新霉素生物合成过程中各个结构基因[9-10]。因此,为了提高新霉素的合成效率,研究者们主要通过分子生物学、酶工程等技术手段对新霉素生物合成途径进行改造以提高新霉素产量次级代谢产物。例如,通过构建带有新霉素生物合成基因簇的质粒pKCZ03并得到弗氏链霉菌工程菌株SF/pKCZ03(CGMCC4.576),使新霉素的产量提升了36%[11]。利用过表达微型基因簇(PkasO*-neoN-metKPkasO*-neoGH-aphA),进而有效地将生物活性组分之外的副产物积累量从19.1%降低到12.7%,同时使新霉素活性组分的含量增加13.1%[12],对蛋白NeoE和NeoN的结构以及催化机理进行了系统研究,并且结合定点突变以及构建NeoE和NeoN过表达工程菌株,促进了新霉素的合成[13-14]。但关于新霉素合成相关转录因子的研究较少,大多数细菌的转录受到严格调控,以促进细菌适应不同环境,转录调控因子在其中起着关键作用。
基因转录调控是基因表达调控的重要机制,而人工转录调控是调节代谢通量的有效策略[15]。当前,越来越多的研究发现并证实转录因子参与微生物代谢合成网络的调控。如弗氏链霉菌中转录因子AfsA-g和NeoR的过表达能够分别使新霉素的产量提高54.9%和21%[16]。转录因子PapR6可以特异性调节普那霉素的生物合成,在Actinoplanes sp.SE50/110中麦芽糖酶基因amlE受到其上游转录因子AmlR的调控,在Streptomyces sp.FR-008中,FscR1、FscR2、FscR3和FscR4四个转录因子对多烯抗生素杀念菌素的生物合成具有重要调控作用[17-19]。Actinomycete中转录因子GlnR通过介导谷氨酰胺合成酶基因转录水平及谷氨酰胺合成酶乙酰化水平,在参与调控放线菌氮代谢调控方面扮演重要角色[20]。Streptomyces coelicolor中的全局转录因子AdpA能够影响放线菌素的产生以及形态分化[21]。上述研究表明,转录因子作为调控细胞代谢的重要元器件,在调控微生物多种细胞进程中发挥着重要作用。
近年来,比较转录组学分析技术已广泛用于微生物新基因的发现、分子标记挖掘、代谢途径的确定等研究,有助于次生代谢相关基因的挖掘与功能研究[22]。例如,比较转录分析表明,转录因子RosR能够通过结合谷氨酸合成代谢途径中的众多启动子区域,从而调控谷氨酸代谢合成网络。过表达转录因子RosR使得谷氨酸的发酵产量提高25.3%[23]。前期通过诱变育种筛选得到的新霉素高产菌株SF-2[24],本研究首先通过比较转录组学分析SF-2和原始菌株SF-1之间的转录水平差异,发现存在5个转录因子转录水平发生了显著变化。分别通过在SF-2中过表达7个转录因子并通过摇瓶发酵验证其新霉素产量,发现转录因子NeyR对于新霉素产量的影响最为显著,启动子优化后的工程菌株SF-2/pP-NeyR能够积累新霉素B达到10 902 U/mL,较原始菌株SF-2提高了19.3%。随后对发酵培养基组分继续正交优化,得出最佳添加量(质量分数)为可溶性淀粉8%,L-谷氨酰胺0.3%,胰蛋白胨1.2%,摇瓶发酵7 d后,新霉素产量可达到11 843 U /mL,相对于出发菌株SF-2在未优化培养基条件下提高了29.6%。为后续代谢机理研究以及进一步提高新霉素产量奠定了基础。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
大肠埃希菌(Escherichia coli)ET12567(pUZ8002),楚灵生物工程有限公司;弗氏链霉菌野生型(S.fradiae GC6010)菌株SF-1、诱变得到的高产弗氏链霉菌SF-2均为实验室保藏菌株;质粒pPR(pSET152-PermE*-RBS)和强启动子Pkoasp*114均为本实验室保存。
1.1.2 培养基
LB培养基(g/L):胰蛋白胨10,酵母粉5,NaCl 10,pH 7.0。
YEME培养基(g/L):酵母粉3,葡萄糖10,蛋白胨5,蔗糖103,麦芽粉3,pH 7.0。
发酵培养基(g/L):葡萄糖20,NaCl 4.5,中温豆饼粉5,高温淀粉酶0.3,磷酸氢二钠0.4,花生饼粉28,酵母粉6,胰蛋白胨9,碳酸钙4,玉米浆2.5,豆油3,可溶性淀粉70,硫酸铵6,pH 6.8~7.3。
MS培养基(g/L):葡萄糖10,牛肉膏1,胰蛋白3,玉米浆3,NaCl 5,琼脂条20,pH 7.3~7.8。
1.1.3 引物
表1 本研究所用引物
Table 1 Primers used in this study
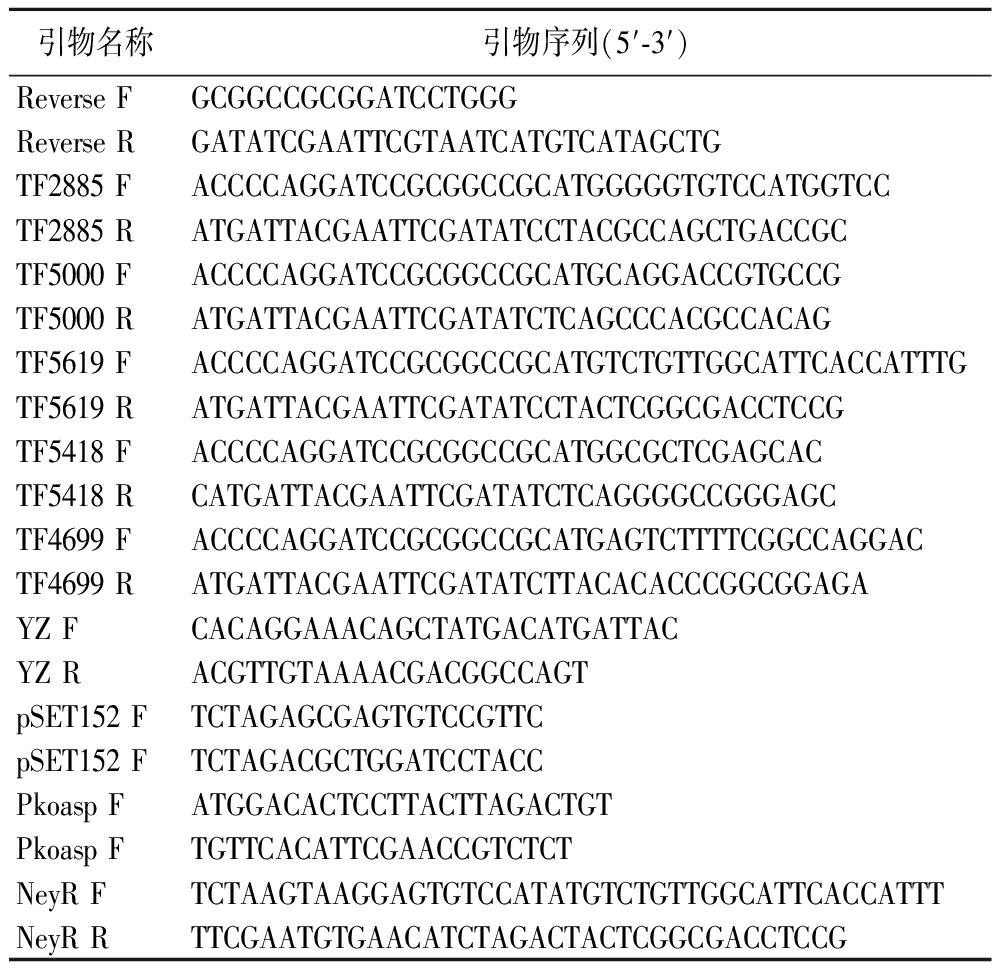
引物名称引物序列(5′-3′)Reverse FGCGGCCGCGGATCCTGGGReverse RGATATCGAATTCGTAATCATGTCATAGCTGTF2885 FACCCCAGGATCCGCGGCCGCATGGGGGTGTCCATGGTCCTF2885 RATGATTACGAATTCGATATCCTACGCCAGCTGACCGCTF5000 FACCCCAGGATCCGCGGCCGCATGCAGGACCGTGCCGTF5000 RATGATTACGAATTCGATATCTCAGCCCACGCCACAGTF5619 FACCCCAGGATCCGCGGCCGCATGTCTGTTGGCATTCACCATTTGTF5619 RATGATTACGAATTCGATATCCTACTCGGCGACCTCCGTF5418 FACCCCAGGATCCGCGGCCGCATGGCGCTCGAGCACTF5418 RCATGATTACGAATTCGATATCTCAGGGGCCGGGAGCTF4699 FACCCCAGGATCCGCGGCCGCATGAGTCTTTTCGGCCAGGACTF4699 RATGATTACGAATTCGATATCTTACACACCCGGCGGAGAYZ FCACAGGAAACAGCTATGACATGATTACYZ RACGTTGTAAAACGACGGCCAGTpSET152 FTCTAGAGCGAGTGTCCGTTCpSET152 FTCTAGACGCTGGATCCTACCPkoasp FATGGACACTCCTTACTTAGACTGTPkoasp FTGTTCACATTCGAACCGTCTCTNeyR FTCTAAGTAAGGAGTGTCCATATGTCTGTTGGCATTCACCATTTNeyR RTTCGAATGTGAACATCTAGACTACTCGGCGACCTCCG
1.1.4 其他
高保真酶2×Phanta Max Master Mix,南京诺唯赞生物科技股份有限公司;柱式DNA胶回收试剂盒,生工生物有限公司。
1.2 RNA-seq测序
在MS固体培养基上将弗氏链霉菌SF-1和SF-2活化,分别接种于YEME液体生长培养基中培养至对数期;按1%的接种量分别转接发酵培养基中培养48 h,发酵结束后4 ℃、8 000 r/min离心10 min收集菌体,使用冰盒保存,并送至上海派森诺生物科技股份有限公司提取RNA进行转录组学分析。
1.3 重组质粒的构建
1.3.1 转录因子基因片段与pPR线性化质粒的PCR扩增
分别使用引物对TF2885 F/TF2885 R、TF5000 F/TF5000 R、TF5619 F/TF5619 R、TF5418 F/TF5418 R、TF4699 F/TF4699 R以弗氏链霉菌SF-2全基因组为模板,利用高保真酶2×Phanta Max Master Mix进行PCR扩增并回收得到线性化片段TF2885、TF5000、TF5619、TF5418、TF1643。使用引物对Reverse F/Reverse R以质粒pPR为模板PCR扩增出线性化pPR质粒片段并回收。
1.3.2 表达载体的构建
根据ClonExpressMultiS One Step Cloning Kit C115试剂盒,将线性化载体片段pPR分别与待插入的转录因子片段同源重组连接,然后转化至E.coli ET12567感受态中。
1.4 基因重组菌株的构建
1.4.1 供体的制备
将含有重组质粒的E.coli ET12567接入LB液体培养基(含50 μg/mL安普霉素、25 μg/mL卡那霉素和25 μg/mL氯霉素)于37 ℃摇床培养OD600值为0.8~1.0。吸取1 mL菌液离心弃上清,再用无抗的LB液体培养基吹洗3次,除去菌液中的抗生素。最后用1 mL无抗LB液体培养基重悬菌体,稀释10倍后备用。
1.4.2 受体的制备
将弗氏链霉菌SF-2在MS平板上活化5~7 d,待孢子成熟用5 mL的YEME培养基进行冲洗,振荡打散后用纱布过滤菌丝,得到孢子悬浮液,离心重悬于0.5 mL的YEME培养基中,50 ℃热激10 min,35 ℃摇床预培养。
1.4.3 接合转移
取1 mL稀释处理好的供体与0.5 mL受体混匀,涂布于含有10 mmol/L MgCl2的MS固体平板上,30 ℃ 倒置培养18 h,用1 mL含1 mg/mL安普霉素和0.5 mg/mL甲氧苄啶的无菌水覆盖平板,吹干后培养7~9 d长出结合子。
1.4.4 结合子的PCR验证
刮取少许结合子进入2 mL EP管,加入100 μL无菌水并放入金属浴煮沸10 min,然后采用高速离心机12 000 r/min条件下离心5 min。离心后以上清液当模板,使用引物对YZ F和YZ R进行PCR验证。
1.5 启动子优化
使用引物对pSET152 F/ pSET152 R以质粒pPR为模板PCR扩增并回收出线性化片段pSET152,同时使用引物对Pkoasp F/ Pkoasp R以启动子Pkasop*114为模板进行PCR扩增并回收出线性化片段Pkasop*114,对2个片段进行同源重组连接,得到新的表达载体pP(pSET152-Pkoasp*114),针对筛选出能显著提高新霉素产量的转录因子NeyR,使用引物对NeyR F和NeyR R以弗氏链霉菌SF-2全基因组为模板进行PCR扩增得到基因片段,按照1.4节方法构建经新的重组菌株。
1.6 摇瓶发酵培养基优化
根据前期研究以及新霉素相关研究报道[11],对发酵培养基成分进行整合分析和正交优化,得出更佳摇瓶发酵条件。
1.7 重组菌株的摇瓶发酵
挑取单菌落接种于YEME培养基至对数生长期,按1%的接种量接种于含有30 mL发酵培养基的250 mL锥形瓶中,在恒温恒湿摇床中发酵7 d,培养条件为35 ℃、220 r/min、75%相对湿度。发酵结束取500 μL发酵液高速离心5 min,将上清液稀释100倍后进行HPLC检测。采用柱前衍生化HPLC法测定新霉素效价,详细方法见参考文献[25]。
2 结果与分析
2.1 比较转录组学挖掘转录因子
转录因子能够响应效应小分子从而调节细胞代谢的关键转录调控元器件,其中转录因子的功能取决于识别目标启动子处特定DNA序列的能力,然后通过上调或下调该启动子处的转录起始,从而影响RNA聚合酶(RNA polymerase, RNAP)发挥正常的转录起始功能,实现对下游基因转录激活或者转录抑制[26-27]。实验室前期以新霉素高产菌株SF-1为出发菌株,经诱变育种获得了更加高产的突变菌株SF-2。将SF-1和SF-2进行转录组测序,通过比较转录组分析发现,相对于SF-1,突变菌株SF-2存在651个显著上调的基因和229个显著下调的基因(图1-a)。通过对这些差异表达基因进行KEGG富集分析(图1-b),发现主要分布在三羧酸循环、氧化磷酸化、氨基酸代谢、丙酸酯代谢和羧酸代谢。差异基因主要是与氧化还原酶活力、NADH脱氢酶活力、细胞氨基酸分解代谢、氮化合物转运、呼吸电子传递链、ATP合成偶联电子传递和碳水化合物分解代谢有关的基因。鉴于转录因子在新霉素发酵调控中的关键作用及其在弗氏链霉菌中的相对稀缺性,本研究进一步分析了比较转录数据。与SF-1相比,在SF-2中约有317个转录因子表达水平表现出差异(图1-c),筛选出5个有显著变化的转录因子(图1-d)。通过序列比对和结构预测,转录因子2885、转录因子5418和转录因子4699属于LuxR家族,转录因子5000和转录因子5619属于LysR家族。LuxR家族转录因子和LysR家族转录因子均具有调节微生物次级代谢产物的功能。因此,推测这5种转录因子可能在调节新霉素合成和发酵控制中发挥重要作用,从而进一步深入研究。

a-差异基因数目统计图;b-基因集代谢途径注释分类统计柱状图;c-调控因子火山图;d-不同转录因子转录差异指数统计图
图1 SF-1和SF-2两种菌株对新霉素合成差异的比较转录组学分析
Fig.1 Comparative transcriptomic analysis of neomycin synthesis differences between SF-1 and SF-2 strains
2.2 重组质粒的构建及验证
2.2.1 PCR扩增转录因子基因片段与pPR线性化质粒
为了进一步确定比较转录组学分析得到的5个转录因子对于弗氏链霉菌发酵新霉素的影响,通过在弗氏链霉菌SF-2中分别过表达经比较转录组学分析筛选得到的5个转录因子,并通过摇瓶发酵研究其对于新霉素产量的影响。首先使用对应的扩增引物,利用高保真酶2×Phanta Max Master Mix获得所需转录因子的目标DNA片段。而后利用柱式DNA胶回收试剂盒回收得到纯化的基因片段,-20 ℃保存备用。
2.2.2 重组质粒的构建
首先根据材料和方法中1.3.1节所述方法,分别构建重组质粒pPR-TF2885、pPR-TF5000、pPR-TF5619、pPR-TF5418、pPR-TF4699,并通过化学转化至接合转移供体菌株E.coli ET12567中。将转化后的菌落接入含安普霉素抗性的LB液体培养基培养过夜然后提取质粒送往测序。将测序结果与实验预测结果进行序列比对,两者完全一致,表明5个重组质粒构建成功。
2.3 过表达菌株构建及验证
将含有过表达转录因子的重组质粒的供体菌株大肠杆菌ET12567(pUZ8002)与受体菌株弗氏链霉菌SF-2进行结合转移,并涂布于安普霉素和甲氧苄啶抗性平板上进行筛选得到结合子。将结合子按照方法1.4.4节处理,使用验证引物对YZ F和YZ R进行PCR验证,验证结果与预期结果一致,表明重组质粒均已成功导入弗氏链霉菌SF-2中(图2)。
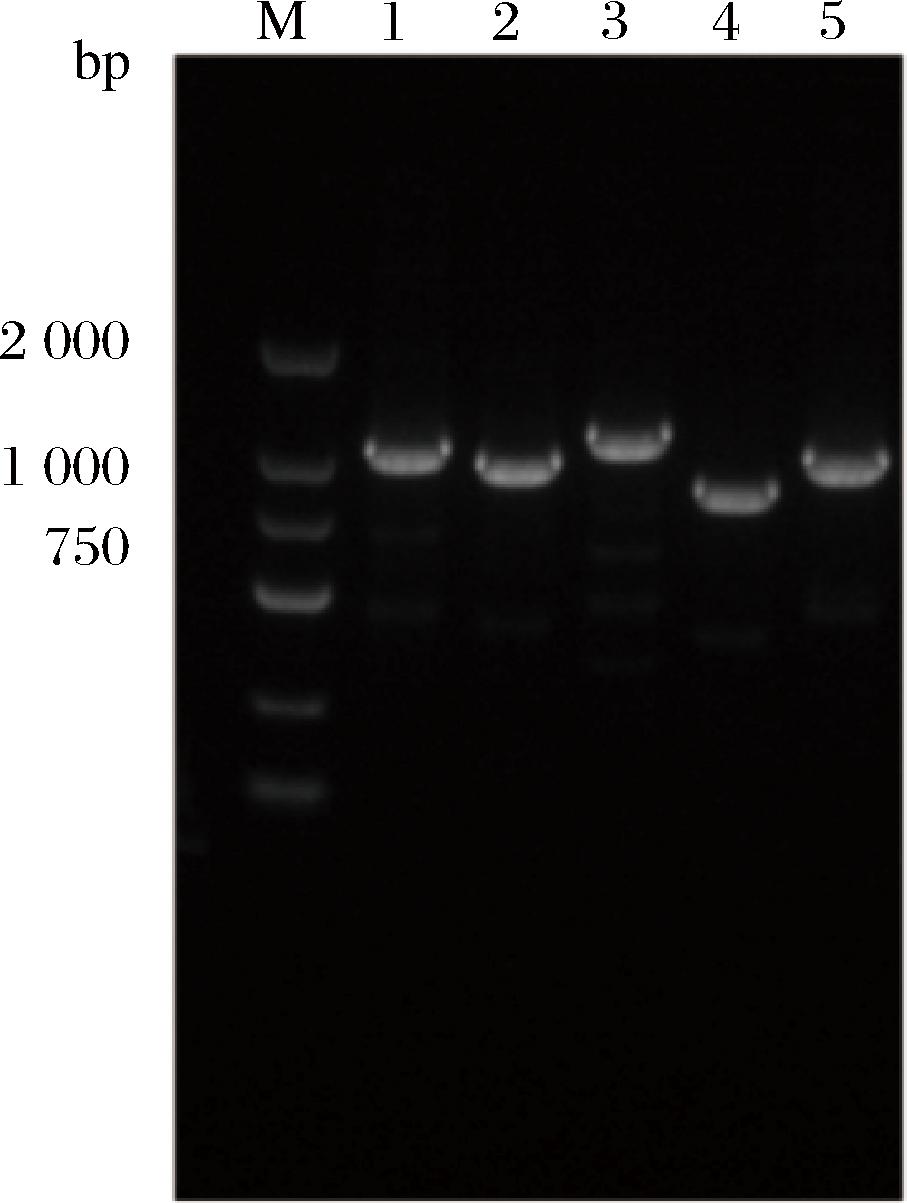
图2 结合子PCR阳性克隆验证
Fig.2 Verification of PCR positive clones of zygotes
注:泳道1~5分别为SF-2/pPR-TF2885、SF-2/pPR-TF5000、SF-2/pPR-TF5619、SF-2/pPR-TF5418、SF-2/pPR-TF4699。
2.4 重组菌株的摇瓶发酵
通过将上述获得的5个转录因子过表达菌株进行摇瓶发酵并测定新霉素的产量发现,相对于出发菌株SF-2,转录因子TF5619摇瓶发酵7 d之后,测定的新霉素产量达到了10 085 U/mL,相对于SF-2的效价9 134 U/mL提高了10.4%(图3)。将影响效果最显著的转录因子命名为NeyR。这部分结果进一步说明,通过比较转录组学分析得到的转录因子NeyR对于新霉素的发酵调控可能发挥着重要的作用,但是其调控新霉素的分子机制尚不清晰,有待进一步的研究。
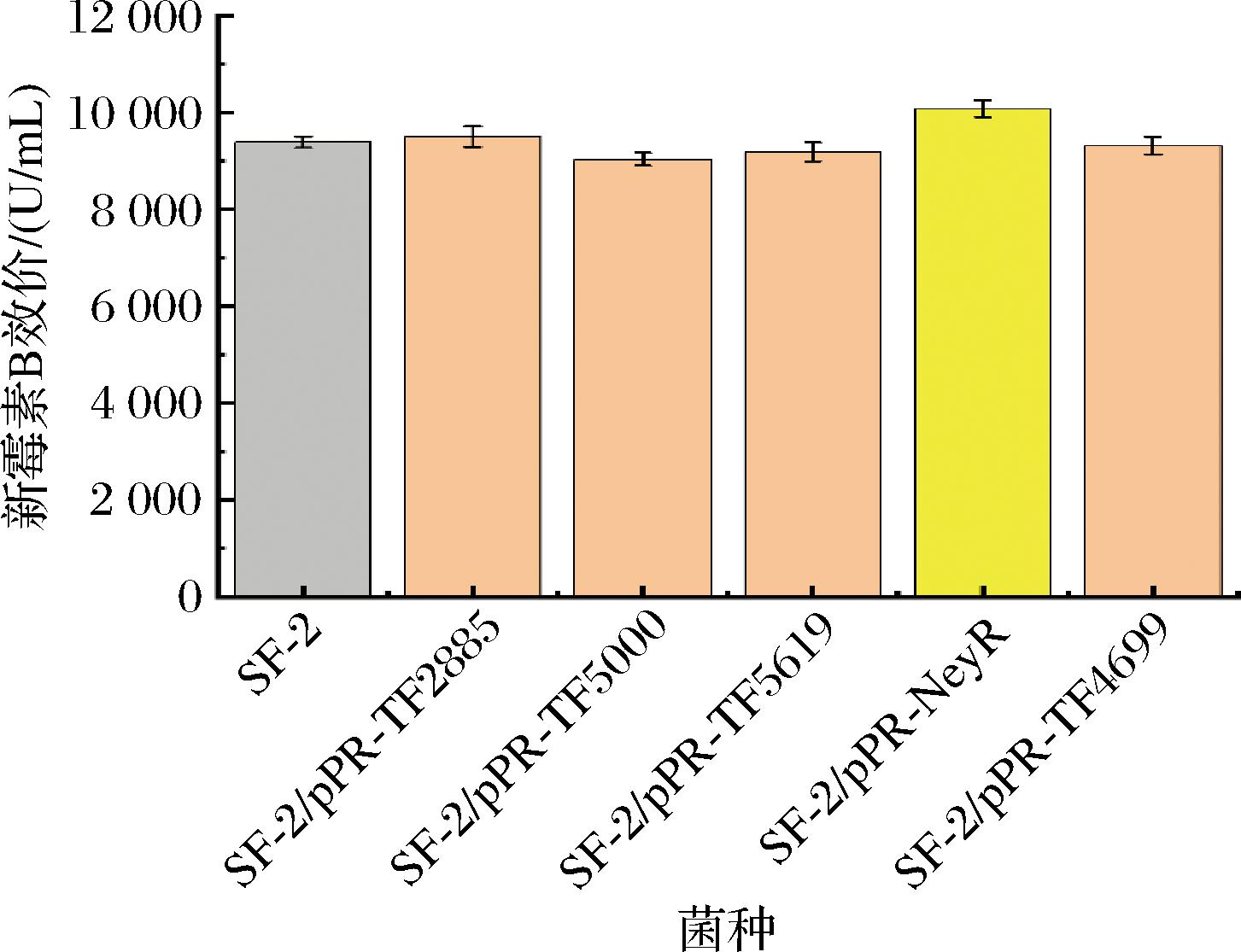
图3 在弗氏链霉菌 SF-2中分别过表达各转录因子对新霉素B生物合成的影响
Fig.3 The effects of overexpression of various transcription factors in Streptomyces fradiae SF-2 on the biosynthesis of neomycin B
2.5 启动子优化强化新霉素合成
上述结果表明转录因子NeyR的过表达能够有效促进弗氏链霉菌中新霉素的发酵合成,为了进一步增强NeyR的表达量并促进新霉素的合成,基于合成生物学策略,通过启动子优化技术,利用实验室前期进化得到的更高强度的启动子Pkasop*114替换了pPR上的启动子PemrE*,并通过结合转移转化到弗氏链霉菌SF-2中命名为SF-2/pP-NeyR。发酵结果表明,启动子优化后的菌株新霉素产量达到了10 902 U/mL,与未进行启动优化的SF-2/pPR-NeyR比较,产量提高了8.1%,与原始菌株SF-2相比,产量提高了19.3%(图4)。上述结果表明,更换更高强度的启动子能够有效促进新霉素的发酵生产,进一步表明转录因子NeyR对于弗氏链霉菌合成新霉素有着重要的正调控作用。
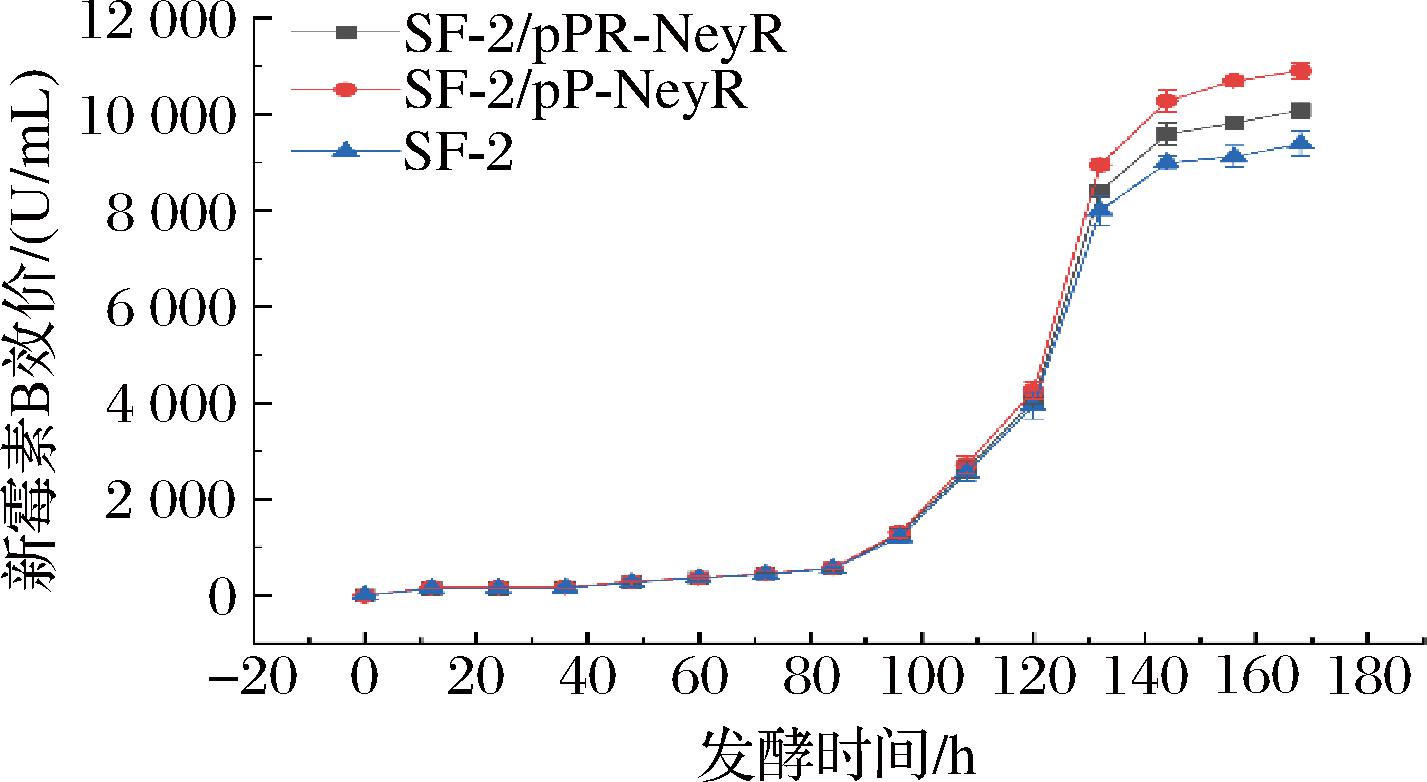
图4 不同菌株发酵的新霉素B效价
Fig.4 The titer of neomycin B fermented by different strains
2.6 新霉素摇瓶发酵培养基优化
培养基的组成成分及比例对于弗氏链霉菌发酵生产新霉素有着重要的影响。为了进一步增强新霉素的发酵水平,首先基于已经报道的L-谷氨酰胺可以有效促进新霉素合成[11],以及整合实验室之前的发酵培养基组分,本试验选择可溶性淀粉、L-谷氨酰胺、胰蛋白胨3种考察因素并选择3个水平,选取L9(34)正交实验设计方法,表2为因素水平表。
表2 因素水平表
Table 2 Factor level table
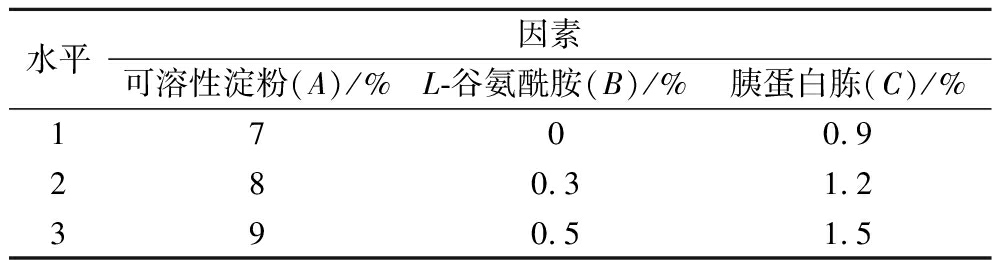
水平因素可溶性淀粉(A)/%L-谷氨酰胺(B)/%胰蛋白胨(C)/%1700.9280.31.2390.51.5
根据表3试验结果进行极差分析,得出可溶性淀粉对新霉素产量的影响大于L-谷氨酰胺大于胰蛋白胨。正交试验产量最高的为试验组5,而分析得出的最优组为A2B2C2。按照试验组5、A2B2C2与原发酵条件对照组进行多次同期摇瓶发酵试验,测定的结果为试验组A2B2C2效价>试验组5>对照组,试验组A2B2C2平均效价为11 843 U/mL,比对照组(105 52 U/mL)增加了12.23%,相对于出发菌株SF-2在未优化培养基条件下提高了29.6%。上述结果进一步说明培养基的组成和比例是影响新霉素发酵水平的重要因素之一,同时上述结果也表明,针对于重组菌株SF-2/pP-NeyR,其最佳培养基比例是可溶性淀粉8%,L-谷氨酰胺0.3%,胰蛋白胨1.2%。
表3 摇瓶发酵优化结果与分析
Table 3 Optimization results and analysis of shaking bottle fermentation
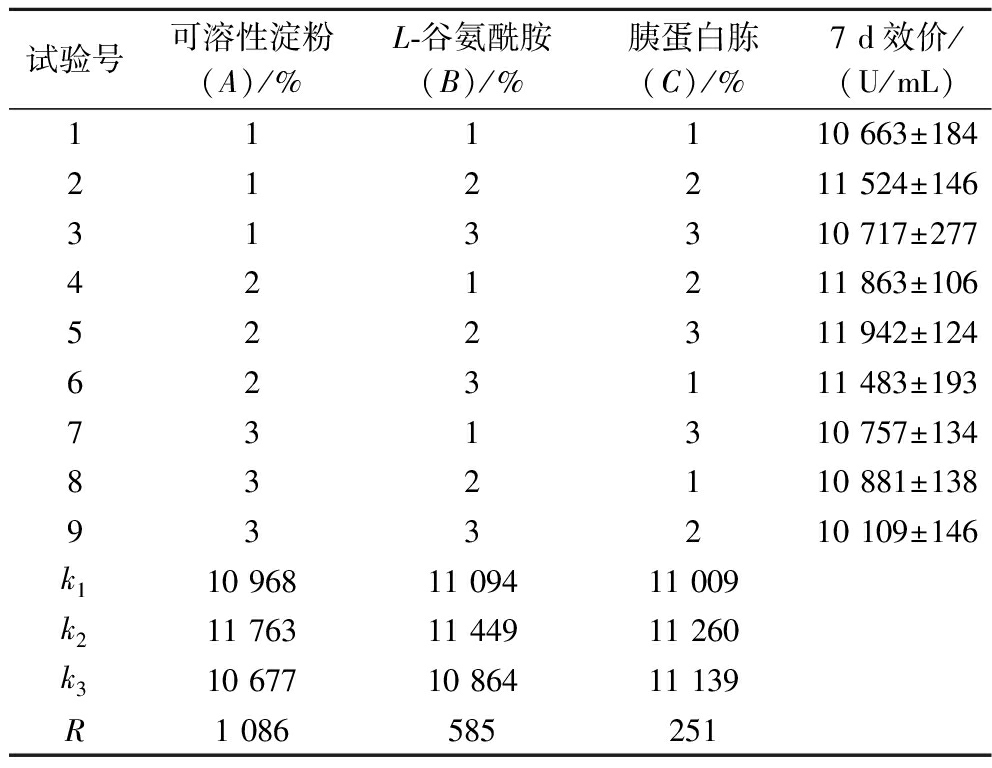
试验号可溶性淀粉(A)/%L-谷氨酰胺(B)/%胰蛋白胨(C)/%7 d效价/(U/mL)111110 663±184212211 524±146313310 717±277421211 863±106522311 942±124623111 483±193731310 757±134832110 881±138933210 109±146k110 96811 09411 009k211 76311 44911 260k310 67710 86411 139R1 086585251
3 结论
新霉素是一种具有良好药理活性的氨基糖苷类抗生素。它在多方面显示出良好的应用前景,具有巨大的市场需求前景。作为一种微生物发酵产品,其生产菌株的性能是决定产品产量和质量的关键因素。在这项研究中,采用了综合的方法来增加弗氏链霉菌中新霉素的产量。比较转录组学分析确定了表现出显著差异表达的转录因子。利用过表达系统,构建过表达这些转录因子的重组菌株,然后摇瓶发酵根据它们对新霉素产量的影响进行筛选。随后对合成新霉素能力增强的工程菌株进行启动子优化,引入了一种更高强度的启动子Pkasop*114增强转录因子的特异性表达,最后根据报道和实验室之前的研究,对发酵培养基的部分组分进行正交优化。上述研究表明转录因子NeyR可以增强新霉素的产生,转录因子通常与受调控基因的上游启动子序列结合,从而调节下游基因转录。猜测转录因子NeyR可能与新霉素代谢途径中关键基因的启动子区域相互作用,从而调节下游基因的表达并最终促进新霉素的合成。后续将根据该假设,诱导纯化NeyR并与新霉素代谢途径中关键基因进行体外凝胶迁移实验和体内荧光报告基因表征实验来探寻NeyR的作用靶点以及其具体作用机理,从而为进一步揭示新霉素生物合成的调控机制奠定基础。
本研究得到了合成新霉素更强的工程菌株SF-2/pP-NeyR,确定摇瓶发酵法采用的培养基中的可溶性淀粉、L-谷氨酰胺、胰蛋白胨3种成分的添加量(质量分数)分别为8%、0.3%和1.2%。最终全部优化后新霉素生产效价11 843 U/mL相对于出发菌株SF-2在原始培养基条件下发酵的新霉素效价9 134 U/mL提高了29.6%。
[1] 胡永彬, 徐礼生.硫酸新霉素的研究[J].广州化工, 2022, 50(12):10-11;19.HU Y B, XU L S.Study on neomycin sulfate[J].Guangzhou Chemical Industry, 2022, 50(12):10-11;19.
[2] SUBRAMANI P, PIROLO M, HAUGEGAARD S, et al.Neomycin resistance in clinical Escherichia coli from Danish weaner pigs is associated with recent neomycin use and presence of F4 or F18 fimbriaes[J].Preventive Veterinary Medicine, 2023, 212:105852.
[3] XIAO C, LIU J F, YANG A K, et al.Colorimetric determination of neomycin using melamine modified gold nanoparticles[J].Microchimica Acta, 2015, 182(7-8):1501-1507.
[4] PAN D, YU Z T.Intestinal microbiome of poultry and its interaction with host and diet[J].Gut Microbes, 2014, 5(1):108-119.
[5] 刘海燕, 殷瑜.氨基糖苷类抗生素作用机制研究进展[J].中国抗生素杂志, 2019, 44(11):1283-1287.LIU H Y, YIN Y.Advances in research on the mechanism of aminoglycoside antibiotics[J].Chinese Journal of Antibiotics, 2019, 44(11):1283-1287.
[6] WAKSMAN S A, LECHEVALIER H A.Neomycin, a new antibiotic active against streptomycin-resistant bacteria, including tuberculosis organisms[J].Science, 1949, 109(2830):305-307.
[7] VADASSERY J, REICHELT M, JIMENEZ-ALEMAN G H, et al.Neomycin inhibition of (+)-7-iso-jasmonoyl-L-isoleucine accumulation and signaling[J].Journal of Chemical Ecology, 2014, 40(7):676-686.
[8] YUAN L L, WEI H P, FENG H T, et al.Rapid analysis of native neomycin components on a portable capillary electrophoresis system with potential gradient detection[J].Analytical and Bioanalytical Chemistry, 2006, 385(8):1575-1579.
[9] KUDO F, HOSHI S, KAWASHIMA T, et al.Characterization of a radical S-adenosyl-L-methionine epimerase, NeoN, in the last step of neomycin B biosynthesis[J].Journal of the American Chemical Society, 2014, 136(39):13909-13915.
[10] KUDO F, EGUCHI T.Biosynthetic genes for aminoglycoside antibiotics[J].The Journal of Antibiotics, 2009, 62(9):471-481.
[11] ZHENG J Z, LI Y, GUAN H Y, et al.Enhancement of neomycin production by engineering the entire biosynthetic gene cluster and feeding key precursors in Streptomyces fradiae CGMCC 4.576[J].Applied Microbiology and Biotechnology, 2019, 103(5):2263-2275.
[12] ZHENG J Z, LI Y, GUAN H Y, et al.Component optimization of neomycin biosynthesis via the reconstitution of a combinatorial mini-gene-cluster in Streptomyces fradiae[J].ACS Synthetic Biology, 2020, 9(9):2493-2501.
[13] LI X F, YU F, LIU K, et al.Uncovering the effects of ammonium sulfate on neomycin B biosynthesis in Streptomyces fradiae SF-2[J].Fermentation, 2022,8(12):678.
[14] LI X F, YU F, WANG F, et al.Point mutation of V252 in neomycin C epimerase enlarges substrate-binding pocket and improves neomycin B accumulation in Streptomyces fradiae[J].Bioresources and Bioprocessing, 2022, 9(1):123.
[15] PAN X W, YOU J J, TANG M, et al.Improving prodigiosin production by transcription factor engineering and promoter engineering in Serratia marcescens[J].Frontiers in Microbiology, 2022, 13:977377.
[16] MENG X X, WANG W Z, XIE Z J, et al.Neomycin biosynthesis is regulated positively by AfsA-g and NeoR in Streptomyces fradiae CGMCC 4.7387[J].Science China Life Sciences, 2017, 60(9):980-991.
[17] ZHANG Y Y, ZOU Z Z, NIU G Q, et al.JadR and jadR2 act synergistically to repress jadomycin biosynthesis[J].Science China Life Sciences, 2013, 56(7):584-590.
[18] SCHAFFERT L, SCHNEIKER-BEKEL S, DYMEK S, et al.Essentiality of the maltase AmlE in maltose utilization and its transcriptional regulation by the repressor AmlR in the acarbose-producing bacterium Actinoplanes sp.SE50/110[J].Frontiers in Microbiology, 2019, 10:2448.
[19] ZHU Y P, XU W H, ZHANG J, et al.A hierarchical network of four regulatory genes controlling production of the polyene antibiotic candicidin in Streptomyces sp.strain FR-008[J].Applied and Environmental Microbiology, 2020, 86(9):e00055-20.
[20] LIAO C H, YAO L L, XU Y, et al.Nitrogen regulator GlnR controls uptake and utilization of non-phosphotransferase-system carbon sources in actinomycetes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(51):15630-15635.
[21] LU T, WU X H, CAO Q, et al.Sulfane sulfur posttranslationally modifies the global regulator AdpA to influence actinorhodin production and morphological differentiation of Streptomyces coelicolor[J].mBio, 2022, 13(3):e0386221.
[22] XU G B, LI J G, LIU Q, et al.Transcriptional analysis of Myceliophthora thermophila on soluble starch and role of regulator AmyR on polysaccharide degradation[J].Bioresource Technology, 2018, 265:558-562.
[23] LI X F, BAO T, OSIRE T, et al.MarR-type transcription factor RosR regulates glutamate metabolism network and promotes accumulation of L-glutamate in Corynebacterium glutamicum G01[J].Bioresource Technology, 2021, 342:125945.
[24] YU F, ZHANG M, SUN J F, et al.Improved neomycin sulfate potency in Streptomyces fradiae using atmospheric and room temperature plasma (ARTP) mutagenesis and fermentation medium optimization[J].Microorganisms, 2022, 10(1):94.
[25] 余飞, 孙俊峰, 刘鹏飞, 等.弗氏链霉菌产硫酸新霉素高通量选育模型的建立及优化[J].食品与发酵工业, 2019, 45(8):162-167;177.YU F, SUN J F, LIU P F, et al.A high-throughput screening method for selecting Streptomyces fradiae mutants with improved neomycin yield and its optimization[J].Food and Fermentation Industries, 2019, 45(8):162-167;177.
[26] PLUTA R, ARAG N E, PRESCOTT N A, et al.Molecular basis for DNA recognition by the maternal pioneer transcription factor FoxH1[J].Nature Communications, 2022, 13(1):7279-7293.
N E, PRESCOTT N A, et al.Molecular basis for DNA recognition by the maternal pioneer transcription factor FoxH1[J].Nature Communications, 2022, 13(1):7279-7293.
[27] KANG Z Q, ZHANG M M, GAO K Y, et al.An L-2-hydroxyglutarate biosensor based on specific transcriptional regulator LhgR[J].Nature Communications, 2021, 12(1):3619-3631.