鞣花酸(ellagic acid,EA)是一类广泛分布于石榴、蓝莓、核桃等水果和坚果中的酚类化合物和天然抗氧化物[1],因为其水溶性极低,导致人体吸收利用度低。人体摄入EA后,首先会在内酯酶和脱羧酶的作用下反应生成尿石素M5,然后尿石素M5在邻苯二酚脱羟酶的作用下,逐步脱羧生成尿石素D、尿石素C、尿石素A或异尿石素A、尿石素B等一系列尿石素物质[2],其中,尿石素A的生物活性最多也最好,具有诱导线粒体自噬、改善肌肉质量[3]、提高运动表现和促进线粒体健康[4],抗菌、抗癌、抗炎及抗氧化[2]、预防帕金森病[5]和阿尔兹海默症[6]等多种生物学活性。然而不同人群转化鞣花酸生成尿石素的能力各不相同[7]。基于个体肠道微生物代谢EA形成尿石素的不同,可将个体分为3种不同的尿石素代谢型,尿石素代谢A型:终产物只有尿石素A;尿石素代谢B型:终产物产生异尿石素A和尿石素B,还会产生低浓度尿石素A;尿石素代谢0型:终产物无尿石素产生或产生量未达到检测限[8]。30岁以上的人群中,仅有约40%的人群为尿石素代谢A型,且随着年龄的增长,尿石素代谢A型所占比率呈下降趋势[9]。因此,2018年美国食品药品监督管理局批准尿石素A作为营养补充剂,用于延缓中老年人的肌肉衰退,受到消费者的青睐。
目前工业制备尿石素A的方法只有化学合成法,造成环境污染,且在产品中有副产物残留[10]。如果能筛选获得发酵转化EA生成尿石素A的菌株,则可以利用微生物发酵制备尿石素A。和化学合成法相比,微生物发酵法具有绿色环保、生产工艺简单、反应温和、生产过程更安全等优势。另外,可利用微生物发酵富含EA的食品制备富含尿石素A的食品,让人们在享受美食的同时,补充尿石素A。目前报道的能转化EA为尿石素A的单菌较少,仅有Bifidobacterium pseudoatenulatum INIA P81535[11]、Streptococcus thermophilus FUA329[12]、Lactococcus garvieae FUA009[13]、Enterococcus faecium FUA027[14]、Lactobacillus plantarum CCFM1286、Lactobacillus plantarum CCFM1290、Lactobacillus plantarum CCFM1291[15]等菌株可以单独发酵转化EA生成尿石素A。
本研究组从尿石素代谢A型人群肠道中筛选得到产尿石素A可应用于婴幼儿食品的公认安全(Generally Recognized as Safe,GRAS)菌株发酵粘液乳杆菌 (Limosilactobacillus fermentum) FUA033。L.fermentum可以通过降解尿酸来调节高尿酸血症、用作食品防腐剂或抗生素的替代品[16]、抗氧化、抗炎、预防心脏代谢紊乱等功效,是新一代益生菌候选菌。菌株的安全性和益生性是其开发和应用的前提条件。随着高通量测序技术的快速发展,菌株基因组测序和生物信息学分析方法已成为研究菌株安全性和益生性的重要方法之一。本研究对L.fermentum FUA033进行全基因组测序,对基因序列进行基因预测分析和功能注释,并结合表型实验结果综合对菌株的安全性和益生特性进行分析和评估。
1 材料与方法
1.1 实验材料
L.fermentum FUA033,分离自健康人体肠道,保存于中国普通微生物菌种保藏管理中心(CGMCC No.28447)。WAM培养基(Wilkins-Chalgren Anaerobe),北京索莱宝科技有限公司。
1.2 仪器与设备
DG250厌氧工作站,上海阔增生物科技有限公司;YXQ-75G立式压力蒸汽灭菌器、SPX-250B-Z生化培养箱,上海博迅实业有限公司医疗设备厂;SW-CJ-1FD超净工作台,上海谱振生物科技;测序技术平台(Pacbio和Illumina),北京诺禾致源科技股份有限公司;M220 Covaris超声波破碎仪,基因有限公司。
1.3 实验方法
1.3.1 菌株的活化及基因组DNA的提取
将-80 ℃甘油管保存的L.fermentum FUA033,三区划线接种于WAM固体培养基上,37 ℃培养24 h后,挑取单菌落接种至WAM液体肉汤培养基,37 ℃静置厌氧培养24 h,8 000×g离心5 min收集菌体。采用STE的方法对L.fermentum FUA033的基因组DNA进行提取[17]。
1.3.2 DNA测序、质控与组装
采用SMRT bell TM Template kit(版本:2.0)试剂盒构建SMRT Bell文库,将经电泳检测合格的DNA样品用超声波破碎仪随机打断成长度约为350 bp的片段,片段经过修复处理后进行纯化和浓度定量,库检合格后,使用PacBio Sequel II/PacBio Sequel Iie平台和Illumina NovaSeq PE150平台进行测序。对原始下机数据(Raw Date)进行过滤处理,保留高质量reads,得到有效数据(Clean Date)。基于测序数据,使用Canu软件(https://github.com/marbl/canu/,版本:2.0)对测序片段进行基因组组装,得到能反映样品基因组基本情况的初步组装结果,然后使用Racon(版本:1.4.13)软件基于三代测序数据对拼接结果进行3轮纠错后,再进行3轮二代测序片段的Pilon软件(版本:1.22)纠错,得到最终的组装结果。
1.3.3 基因预测与功能注释
采用GeneMarkS软件(verison4.17)(http://topaz.gatech.edu/)进行编码基因预测及过滤,将预测基因的蛋白序列与相关功能数据库:COG(Cluster of Orthologous Groups of proteins)[18]、CAZy(Carbohydrate-Active enZYmes Database)[19]、Swiss-Prot[20]进行Diamond比对(E值≤1e-5),将每一条序列的比对结果进行过滤,选取质量值最高的比对结果(默认基因一致性≥40%,覆盖度≥40%)进行功能注释。
1.3.4 L.fermentum FUA033安全性基因分析
使用Diamond软件,把目标菌株的氨基酸序列分别与VFDB(Virulence Factors of Pathogenic Bacteria)数据库[21]和ARDB(Antibiotic Resistance Genes Database)数据库[22]进行比对,把目标菌株的基因和其相对应的毒力因子功能注释信息、耐药功能注释信息结合起来,得到注释结果。
1.3.5 L.fermentum FUA033益生性基因分析
使用Diamond软件,把目标菌株的氨基酸序列与Swiss-Prot等数据库进行比对,通过搜索比对与耐酸、耐胆盐、抗氧化、细胞黏附性有关基因,并与表型实验结果相结合分析,综合评估L.fermentum FUA033益生性。
1.3.6 L.fermentum FUA033表型实验
耐酸耐胆盐测定:使用活菌平板计数法测定菌株在模拟胃肠道环境中的存活率,将L.fermentum FUA033种子液调节至OD600值为1.0±0.05后以2%的接种量分别接种于不同pH(2.0、2.5、3.0、3.5)和不同胆盐质量分数(0.1%、0.2%、0.3%、0.4%)的培养基中,以正常培养基作为对照,37 ℃培养,分别在0、2、4、6 h进行采样,样品适当稀释后进行平板活菌计数,结果以lg CFU/mL计,以Nt表示处理t小时后的活菌数,N0表示起始活菌数,如公式(1)所示:
存活率![]()
(1)
自由基清除率采用南京建成生物工程研究所检测试剂盒进行检测。
疏水性测定:将L.fermentum FUA033发酵液离心(20 ℃,3 000 r/min,10 min)后用PBS缓冲液洗涤,悬浮液调整至OD600值为0.5±0.02(A0),然后将等体积的二甲苯溶液加入到细菌悬浮液中,并在37 ℃下涡旋20 s,37 ℃培养1 h,测定上清液在600 nm处的吸光度(A1),重复3次,如公式(2)所示:
疏水率![]()
(2)
自凝聚率测定:制备菌体悬浮液,步骤同疏水性测定,调整至OD600值为0.5 ± 0.02(A0),37 ℃静置4 h后,测定上清液在600 nm处的吸光度(A2),重复3次,如公式(3)所示:
自凝聚率![]()
(3)
2 结果与分析
2.1 L.fermentum FUA033全基因组概况
2.1.1 全基因组基本特征
通过PacBio Sequel Ⅱ/PacBio Sequel Ⅱe和Illumina NovaSeq PE150相结合的方法对L.fermentum FUA033进行测序,L.fermentum FUA033过滤后的有效测序片段有321 032个,测序片段平均长度5 954 bp;组装后基因组全长2 150 545 bp,不含质粒,GC含量51.34%。编码基因2 215个,基因平均长度为831 bp,编码基因总长为1 841 256 bp,占全基因组的85.62%。基因长度分布如图1所示,基因组长度在100~1 000 bp的数量最多,长度在1 600~2 000 bp的数量最少,长度>2 000 bp的基因数量是100个。
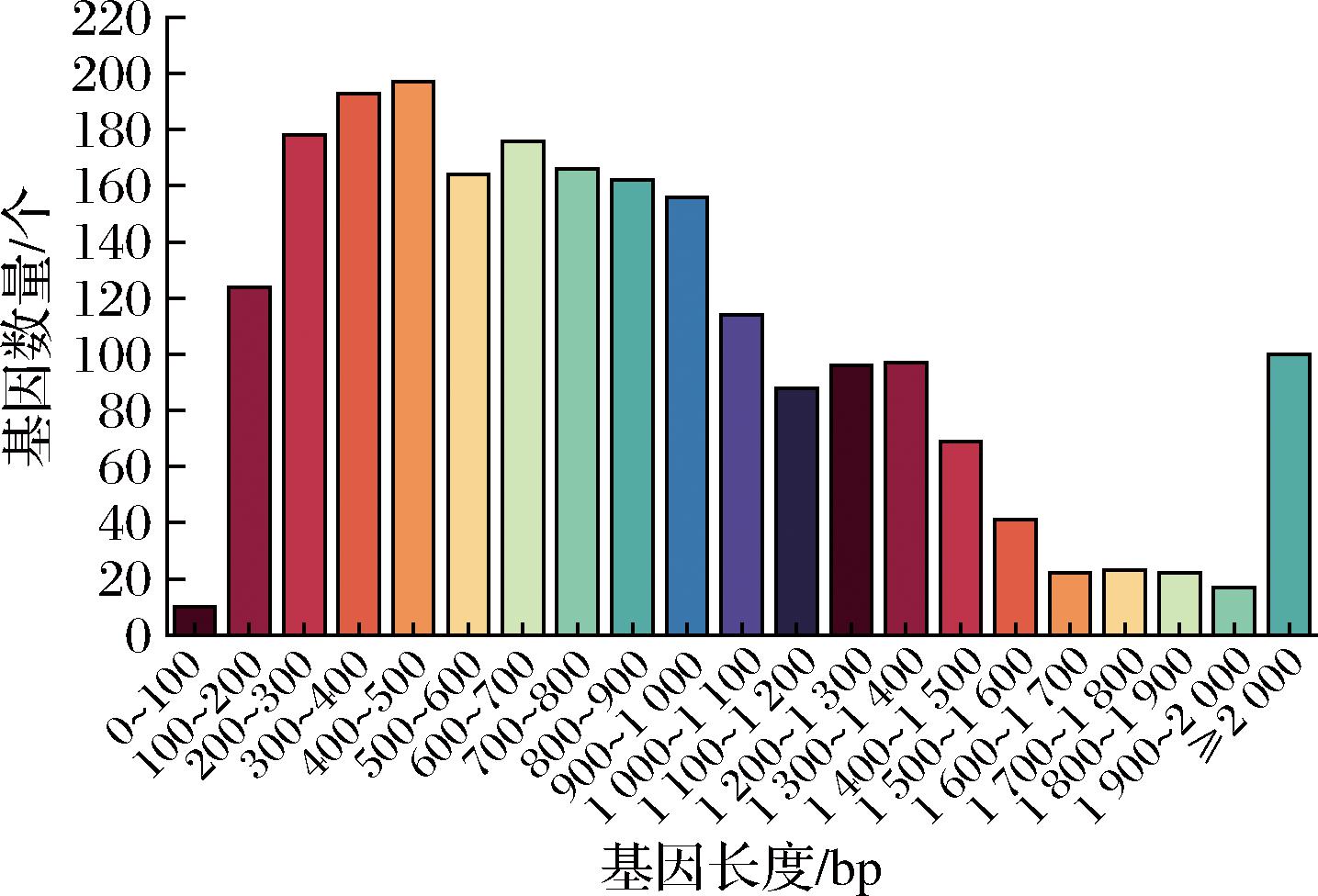
图1 基因长度分布图
Fig.1 Gene length distribution chart
2.1.2 全基因组图谱分析
基因组圈图能将多种信息整合,更全面更直观的了解目标菌株的基因特性。如图2所示,全基因组图谱最外圈是基因组序列位置坐标,由外到里,第1圈是COG基因功能注释结果,根据基因功能分为23类;第2圈是ncRNA,其中包含有58个tRNA,15个rRNA(5S rRNA、16S rRNA、23S rRNA各5个);第3圈是基因组GC含量,向内的蓝色部分表示该区域GC含量低于全基因组平均GC含量,向外的红色部分与之相反,且峰值越高表示与平均GC含量差值越大;第4圈表示基因组GC转移值分布,向内的绿色部分表示该区域G的含量低于C的含量,向外的橘色部分与之相反。
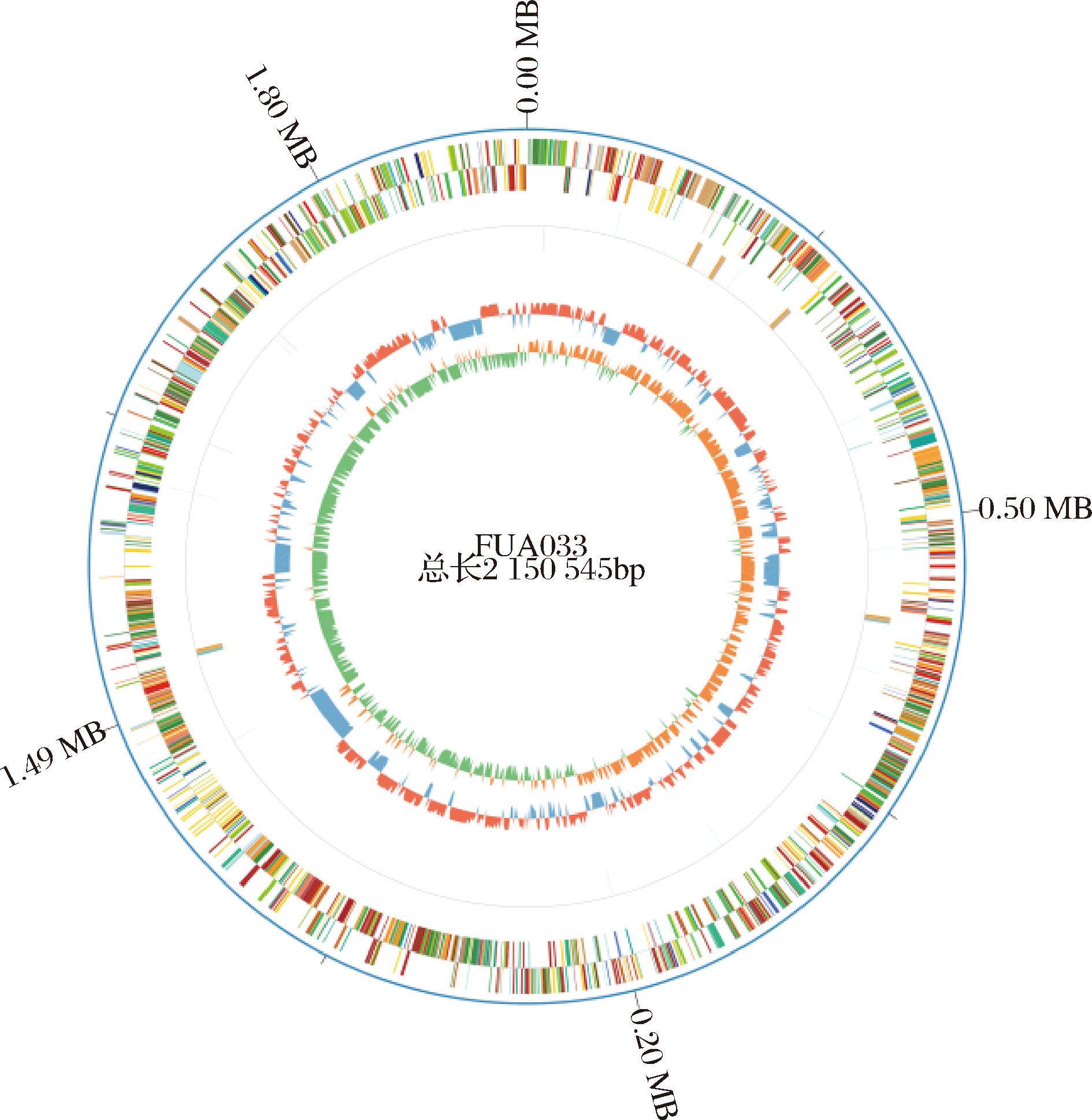
图2 L.fermentum FUA033全基因组图谱
Fig.2 Whole genome map of L.fermentum FUA033
通过COG注释可以推测L.fermentum FUA033基因组中未知蛋白序列的功能,L.fermentum FUA033基因组中详细COG注释结果如图3所示,包括能量产生和转换、细胞周期控制、氨基酸转运和代谢、核苷酸转运和融合等23个功能,共有1 769个编码基因被注释到上述23个功能类别上,占总编码基因的79.86%。
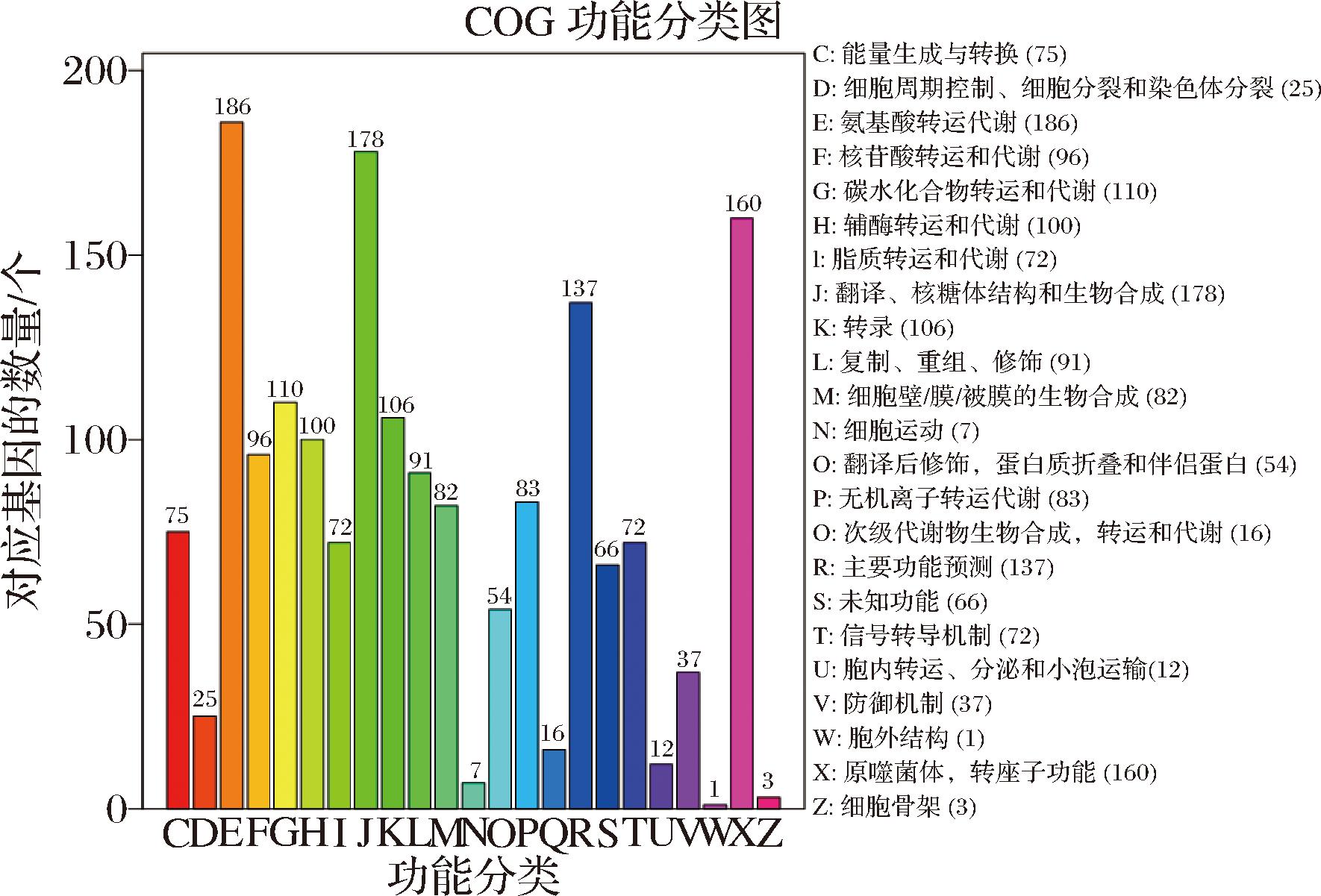
图3 L.fermentum FUA033 COG功能分类图
Fig.3 COG functional classification map of L.fermentum FUA033
其中,A类(RNA加工与修饰)、B类(染色体结构与动力学)、Y类(核酸结构)在L.fermentum FUA033基因组中未被检测出,W类(胞外结构,1个)Z类(细胞骨架,3个)检出数量也非常少,与其他L.fermentum或植物乳杆菌类检测结果类似,推测是因为此类细菌作为原核细胞生物无细胞核结构,染色体结构和胞外结构比较简单,因此W类和Z类基因数量较少。在不考虑未知功能的情况下,根据编码蛋白的基因序列数量排序,L.fermentum FUA033基因组注释排名前3的分别是E类(氨基酸转运和代谢功能,186个)、J类(翻译核糖体结构和生物合成功能,178个)、X类(动员组:原噬菌体,转座子功能,160个),其余类型的基因也不在少数,这也侧面反映出L.fermentum FUA033在执行相关功能的复杂性和进化的生物灵活性。
2.2 L.fermentum FUA033安全性评估
2.2.1 毒力基因分析
VFDB数据库是用于专门研究致病细菌、衣原体和支原体致病因子的数据库,除收录毒力基因的物种信息、基本特征描述外,还提供毒力基因功能和致病机制的详细描述。毒力因子是微生物能够在特定物种的宿主体内建立自身并增强其引起疾病潜力的性质,L.fermentum FUA033的基因组序列通过VFDB数据库注释分析,共预测到可能的毒力基因81个,序列一致性均小于72.2%,这些基因主要参与细菌黏附、定植、分泌系统蛋白、免疫调节等功能,但L.fermentum FUA033中的这些毒力基因是否会表达,还需进一步验证[23]。
2.2.2 耐药性分析
ARDB数据库的注释可以找到耐药性相关基因的名称,所耐受的抗生素种类等信息,L.fermentum FUA033基因组在ARDB数据库耐药性基因注释结果如表1所示,结果中共注释有9个耐药基因,除杆菌肽同源性高达99.6%,其余几种同源性均小于47%。研究表明,乳酸菌中大多数抗生素抗性基因是不会转移的,但与结合质粒、转座子相关的抗性基因会发生转移,从而引起机体耐药性。基因组测定结果表明L.fermentum FUA033中不具有质粒和转座子,从分子水平上初步断定L.fermentum FUA033不具备传递和接收抗生素耐药相关基因的能力。
表1 L.fermentum FUA033基因组耐药基因预测结果
Table 1 Prediction result of drug resistance genes inL.fermentum FUA033 genome
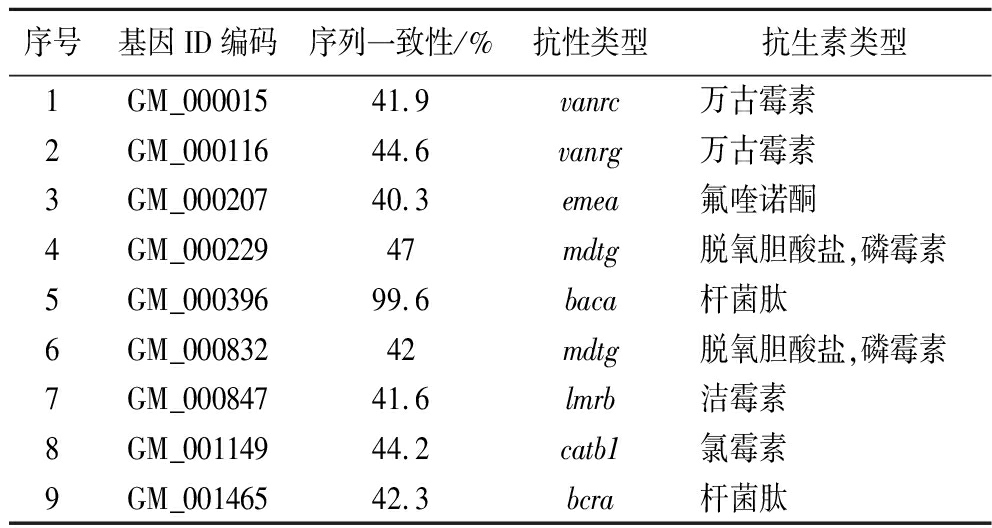
序号基因ID编码序列一致性/%抗性类型抗生素类型1GM_00001541.9vanrc万古霉素2GM_00011644.6vanrg万古霉素3GM_00020740.3emea氟喹诺酮4GM_00022947mdtg脱氧胆酸盐,磷霉素5GM_00039699.6baca杆菌肽6GM_00083242mdtg脱氧胆酸盐,磷霉素7GM_00084741.6lmrb洁霉素8GM_00114944.2catb1氯霉素9GM_00146542.3bcra杆菌肽
2.3 L.fermentum FUA033益生性评估
乳酸菌是能发酵糖类产生大量乳酸的一类细菌,在人体肠道、动物肠道和自然界中都有分布。随着食品发酵工业的发展,关于开发利用乳酸菌的经济效益也在不断增长。L.fermentum FUA033作为一种可用于婴幼儿食品的GRAS益生菌,人体食用后就必须能够在酸度极高的胃酸中和高浓度胆盐中生存,才能顺利到达小肠定殖并发挥益生作用。因此,良好的耐酸、耐胆盐、抗氧化和细胞黏附性是理想益生菌的重要特征之一,解析L.fermentum FUA033中与之有关的基因便很有意义。
2.3.1 L.fermentum FUA033耐酸能力评估
乳酸菌可利用的耐酸性机制大致可分为八类:质子泵、合成碱性化合物、合成生物膜、蛋白质修复、DNA修复、调节因子、细胞密度的改变、代谢方式的改变,其中前2种机制研究的最多。质子泵:通过ATP的合成促使细胞质中质子外排,从而升高细胞内的pH值,或通过谷氨酸脱羧酶体系(glutamic acid decarboxylase,GAD)将谷氨酸进行脱羧达到消耗细胞中质子的目的,再将产物运送至细胞外,从而解除酸胁迫。合成碱性物质:通过脲酶体系催化尿素产生氨,或通过精氨酸脱亚氨酸酶体系(argininedeiminase,ADI)催化精氨酸生成氨,中和细胞内多余的质子,从而升高pH值。如表2所示,通过搜索与比对,在L.fermentum FUA033基因组序列中找到了19个可能与耐酸性有关的基因,这些基因中一部分或全部对L.fermentum FUA033耐受酸性环境有重要的贡献。耐酸性表型实验结果如图4所示,相比较于已报道相似能力的菌株Streptococcus thermophilus FUA329和Enterococcus faecium FUA027,L.fermentum FUA033的耐酸能力更优,在pH 2.0条件下处理6 h后,存活率仍高达62%以上。
表2 L.fermentum FUA033基因组中与耐酸性有关基因的预测
Table 2 Prediction of genes related to acid tolerance inL.fermentum FUA033

序号基因ID编码序列一致性/%基因名称编码蛋白名称1GM_00028799.6argH精氨酸琥珀酸裂解酶2GM_00028683.7argG精氨酸琥珀酸合成酶3GM_001883100arcA精氨酸脱亚胺酶4GM_00188591.3arcB鸟氨酸氨甲酰转移酶5GM_00096275.1gadB谷氨酸脱羧酶6GM_00060155.1ProB谷氨酸5-激酶7GM_00118599.7dapA4-羟基四氢吡啶二羧酸合酶8GM_00212456.2dapA9GM_00118799.6dapH2,3,4,5-四氢吡啶-2,6-二羧酸N-乙酰转移酶10GM_00134399.1argCN-乙酰-γ谷氨酰磷酸还原酶11GM_00020149.2napANa+/H+逆向转运蛋白12GM_001741100atpE13GM_001742100atpB14GM_00173899.6atpA15GM_00173557.1atpCATP合成酶16GM_00173692atpD17GM_001740100atpF18GM_001737100atpG19GM_00173999.4atpH
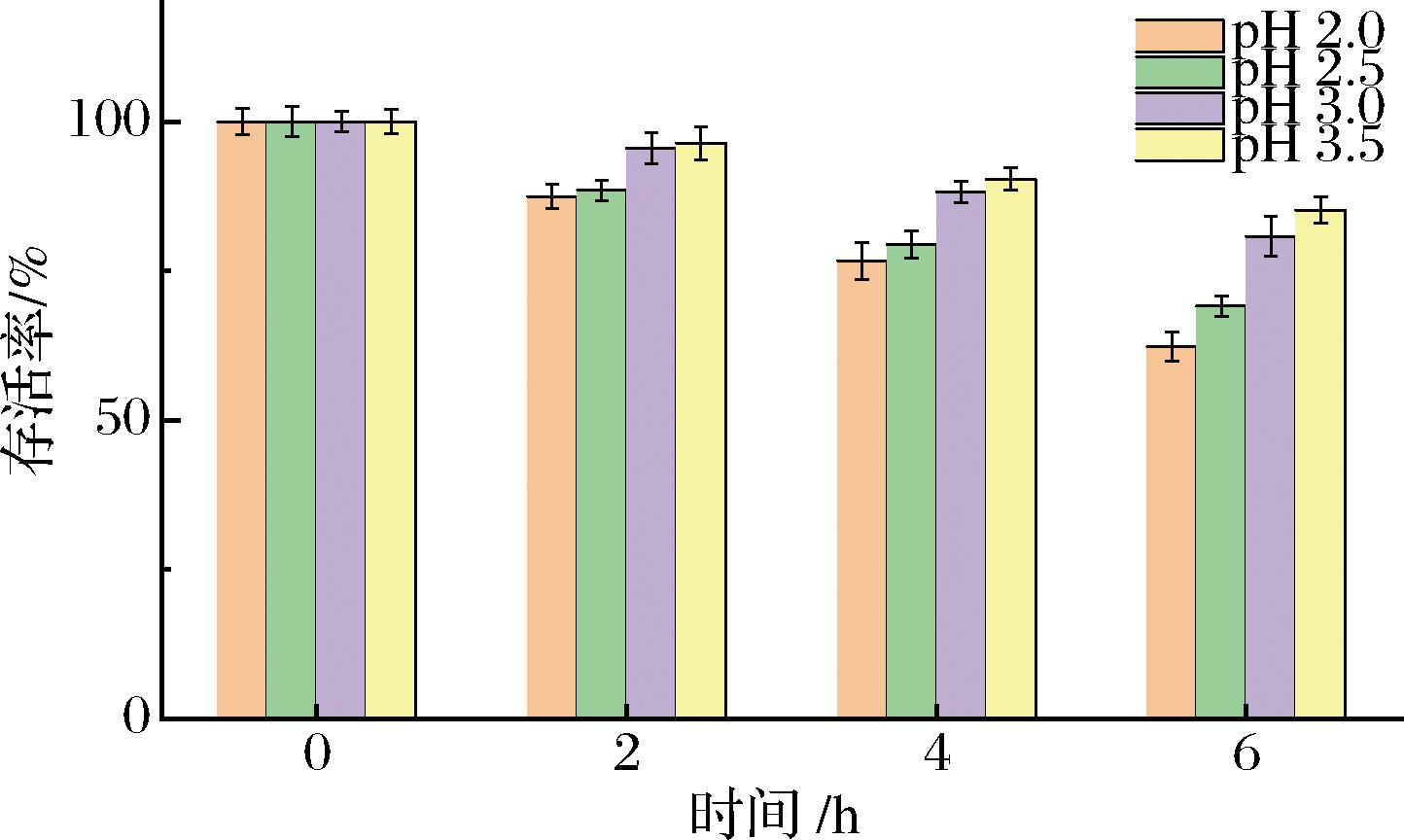
图4 L.fermentum FUA033耐酸性实验结果
Fig.4 Results of acid resistance test of L.fermentum FUA033
2.3.2 L.fermentum FUA033耐胆盐能力评估
菌株在胆盐浓度过高环境中时,细胞膜的通透性和完整性会被破坏,使菌株无法正常存活生长。胆盐耐受能力作为益生菌的另一个重要特性,有助于益生菌通过胃肠道环境并在胃肠道中定殖发挥有益作用。如表3所示,通过搜索与比对,在L.fermentum FUA033基因组序列中找到了16个可能与耐胆盐有关的基因。研究表明[24],ABC转运蛋白编码的外排泵不仅能够有利于抗生素的排出,而且对菌株适应胆盐胁迫也具有重要意义;ABC转运蛋白复合物PstSACB的一部分参与磷酸盐输入,其编码的磷酸盐特异性转运蛋白参与磷酸盐的转运,通过调节碳代谢和氮代谢来应对胆盐引起的能量损失;PTS相关基因(ldhA、sfcA、mtlR)参与着菌株在胆盐胁迫下对碳源的摄取[25],使菌株在胆盐胁迫下具有一定抵抗能力。耐胆盐表型实验结果如图5所示,人体肠道胆盐质量分数一般在0.3%左右,L.fermentum FUA033在胆盐质量分数0.4%条件下处理6 h,存活率仍高达65%以上,优于所报道可以转化EA生成尿石素A的Enterococcus faecium FUA027[26],这与文献报道一致[27],乳酸菌具有一定的胆盐耐受能力。
表3 L.fermentum FUA033基因组中与耐胆盐有关基因的预测
Table 3 Prediction of genes related to bile resistance inL.fermentum FUA033
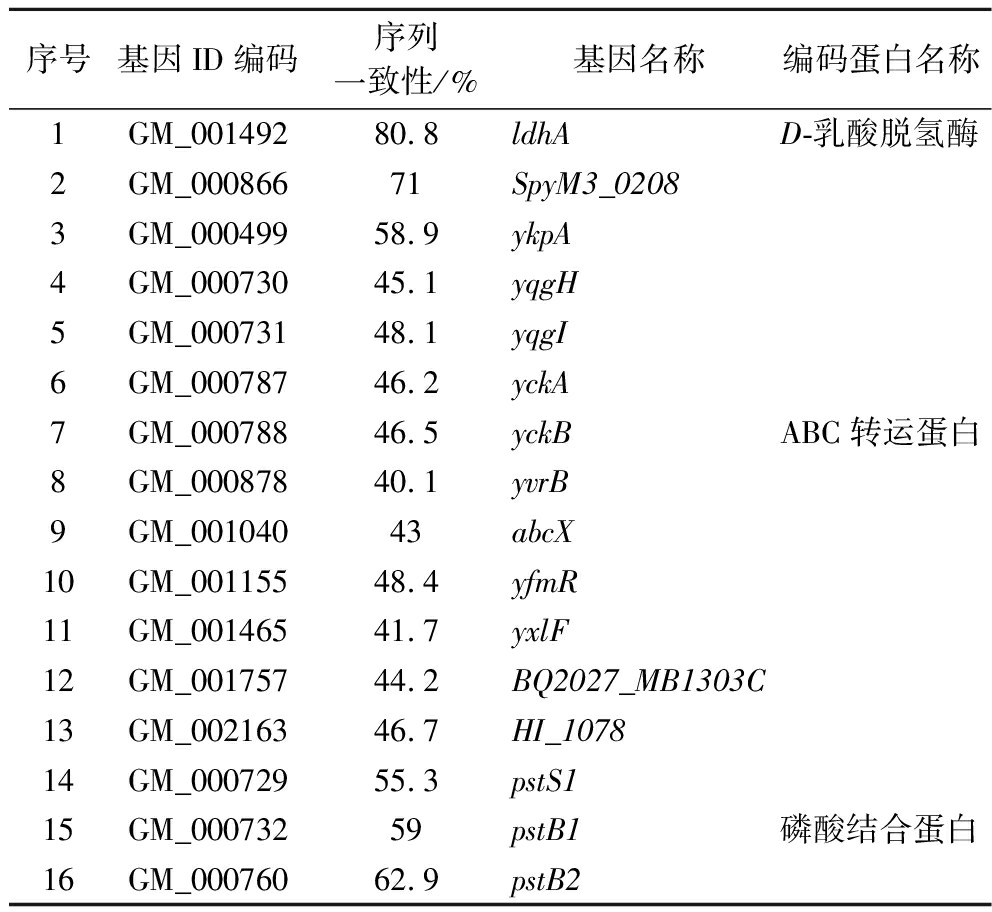
序号基因ID编码序列一致性/%基因名称编码蛋白名称1GM_00149280.8ldhAD-乳酸脱氢酶2GM_00086671SpyM3_02083GM_00049958.9ykpA4GM_00073045.1yqgH5GM_00073148.1yqgI6GM_00078746.2yckA7GM_00078846.5yckBABC转运蛋白8GM_00087840.1yvrB9GM_00104043abcX10GM_00115548.4yfmR11GM_00146541.7yxlF12GM_00175744.2BQ2027_MB1303C13GM_00216346.7HI_107814GM_00072955.3pstS115GM_00073259pstB1磷酸结合蛋白16GM_00076062.9pstB2
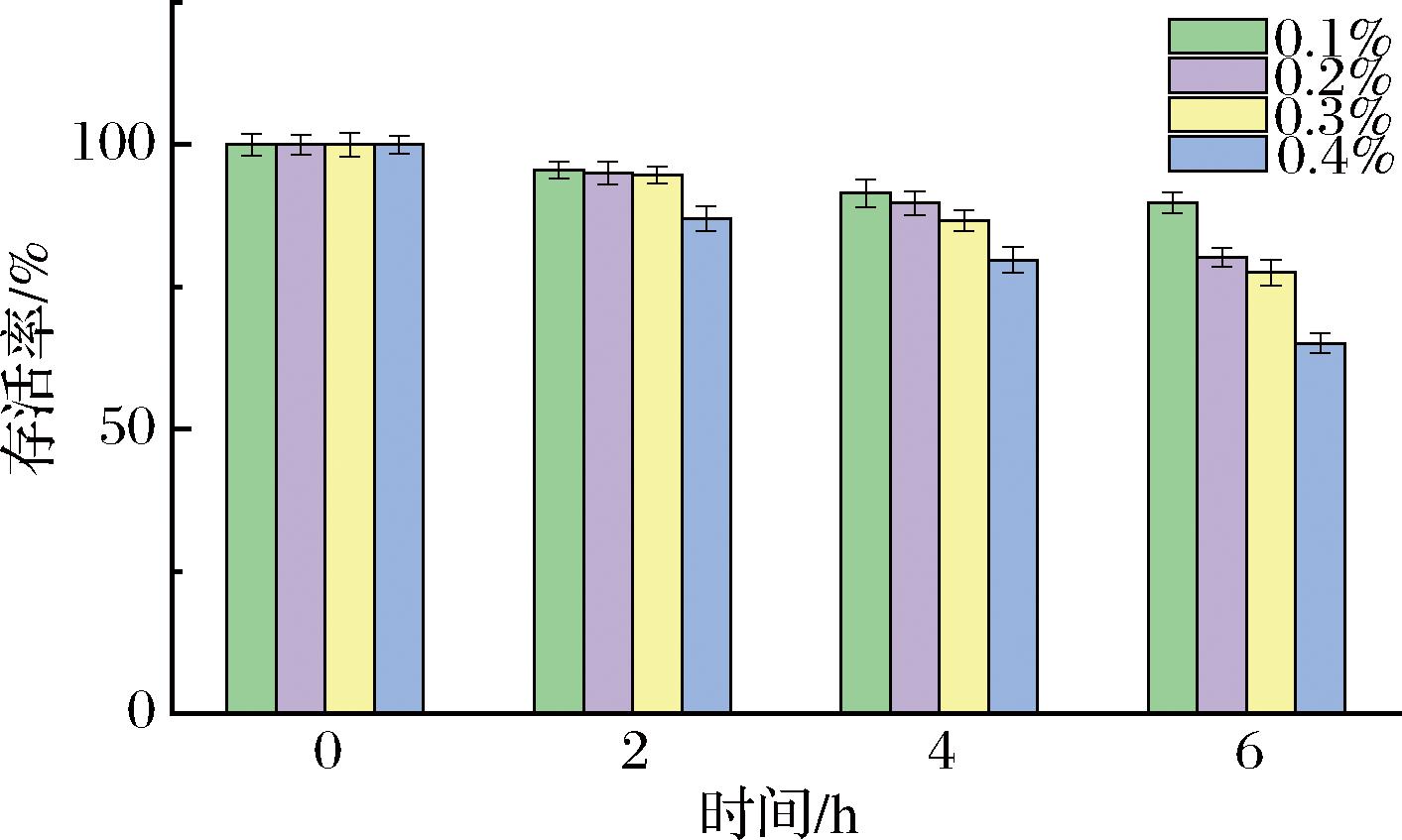
图5 L.fermentum FUA033耐胆盐实验结果
Fig.5 Results of bile salt tolerance test on L.fermentum FUA033
2.3.3 L.fermentum FUA033抗氧化能力评估
菌株良好的抗氧化能力不仅可以提高人体对氧化应激反应的抵抗能力,还可抑制与氧化应激病理条件的形成,如表4所示,通过对比搜索,在L.fermentum FUA033测序结果中共找到11种与抗氧化能力有关基因,其中编码蛋氨酸硫氧化物还原酶(msrA和msrB)的基因有3个;编码硫氧还蛋白家族(Trxs)的基因5个,此类蛋白与抗氧化应激有关;ahpC、tpx、ytpP基因各一个。
表4 L.fermentum FUA033基因组中与抗氧化有关基因的预测
Table 4 Prediction of genes related to antioxidant activity inL.fermentum FUA033

序号基因ID编码序列一致性/%基因名称编码蛋白名称1GM_00177155.3ahpC烷基氢过氧化物还原酶C2GM_00177045.2trxB3GM_00183054.5trxB4GM_00029955.9trxA硫氧还蛋白5GM_00160052.9trxA6GM_00202146.3trxA7GM_00163342.6tpx硫醇过氧化物酶8GM_00063651.6ytpP硫氧还蛋白样蛋白9GM_00126178.5msrA10GM_00126376.2msrA蛋氨酸硫氧化物还原酶11GM_00113373.6msrB
使用抗氧化试剂盒测定L.fermentum FUA033对DPPH自由基、羟自由基、超氧阴离子自由基的清除率,菌体均未表现出对自由基的清除能力,而发酵上清液对自由基有清除能力,对3种自由基的清除率分别为(65.44±0.29)%,(27.39±0.23)%和(26.63±0.47)%,对比所报道的相同功能菌株Lactococcus garvieae FUA009的(31.29±1.57)%,(28.46±1.69)%,(22.98±2.03)%[13],L.fermentum FUA033表现出更好的自由基清除能力。
2.3.4 L.fermentum FUA033定殖能力评估
良好的细胞黏附功能是乳酸菌发挥生物学效应的重要基础,也是作为新一代益生菌的必检项目之一。如表5所示,在L.fermentum FUA033中共找到scpA、scpB、luxS 3个与细胞黏附作用相关的基因。菌株表面的疏水性与自身凝聚性在一定程度上与菌株在体内的定殖能力呈正相关,L.fermentum FUA033的疏水率和自凝聚率分别为(33.2±1.5)%和(10.4±0.5)%,高于所报道的商业长双歧杆菌BB536的(16.8±0.8)%和(6.8±0.7)%[28]。
表5 L.fermentum FUA033基因组中与细胞黏附有关基因的预测
Table 5 Prediction of genes related to cell adhesion inL.fermentum FUA033

序号基因ID编码序列一致性/%基因名称编码蛋白名称100117045.1scpA分离凝集蛋白A200116957.8scpB分离凝集蛋白B3000317100luxSS-核糖基同型半胱氨酸裂解酶
3 结论
本研究通过对L.fermentum FUA033进行全基因组测序和相关功能基因的注释,并结合表型实验结果对该菌株潜在的安全性和益生特性进行了分析和评估。L.fermentum FUA033不含质粒和转座子等可移动元件,含有多个耐酸耐胆盐相关基因和与抗氧化、细胞黏附作用相关基因,保证了菌株进入人体胃肠道后能够更好的存活和发挥益生作用,初步表型实验结果也表明该菌株具有良好的耐酸耐胆盐、抗氧化特性和良好的细胞黏附作用。
[1] BAKKALBA I E, MENTE
I E, MENTE O, ARTIK N. Food ellagitannins-occurrence, effects of processing and storage[J]. Critical Reviews in Food Science and Nutrition, 2009, 49(3):283-298.
O, ARTIK N. Food ellagitannins-occurrence, effects of processing and storage[J]. Critical Reviews in Food Science and Nutrition, 2009, 49(3):283-298.
[2] GONZ LEZ-SARR
LEZ-SARR AS A, LARROSA M, TOM
AS A, LARROSA M, TOM S-BARBER
S-BARBER N F A, et al. NF-kappaB-dependent anti-inflammatory activity of urolithins, gut microbiota ellagic acid-derived metabolites, in human colonic fibroblasts[J]. British Journal of Nutrition, 2010, 104(4):503-512.
N F A, et al. NF-kappaB-dependent anti-inflammatory activity of urolithins, gut microbiota ellagic acid-derived metabolites, in human colonic fibroblasts[J]. British Journal of Nutrition, 2010, 104(4):503-512.
[3] LIU S, D’AMICO D, SHANKLAND E, et al.Effect of urolithin A supplementation on muscle endurance and mitochondrial health in older adults:A randomized clinical trial[J].JAMA Network Open, 2022, 5(1):e2144279.
[4] D’AMICO D, OLMER M, FOUASSIER A M, et al.Urolithin A improves mitochondrial health, reduces cartilage degeneration, and alleviates pain in osteoarthritis[J].Aging Cell, 2022, 21(8):e13662.
[5] WOJCIECHOWSKA O, KUJAWSKA M.Urolithin A in health and diseases:Prospects for Parkinson’s disease management[J].Antioxidants, 2023, 12(7):1479.
[6] JAYATUNGA D P W, HONE E, KHAIRA H, et al.Therapeutic potential of mitophagy-inducing microflora metabolite, urolithin A for Alzheimer′s disease[J].Nutrients, 2021, 13(11):3744.
[7] TOM S-BARBER
S-BARBER N F A, GARC
N F A, GARC A-VILLALBA R, GONZ
A-VILLALBA R, GONZ LEZ-SARR
LEZ-SARR AS A, et al. Ellagic acid metabolism by human gut microbiota: Consistent observation of three urolithin phenotypes in intervention trials, independent of food source, age, and health status[J]. Journal of Agricultural and Food Chemistry, 2014, 62(28):6535-6538.
AS A, et al. Ellagic acid metabolism by human gut microbiota: Consistent observation of three urolithin phenotypes in intervention trials, independent of food source, age, and health status[J]. Journal of Agricultural and Food Chemistry, 2014, 62(28):6535-6538.
[8] XIAN W Y, YANG S Y, DENG Y, et al.Distribution of urolithins metabotypes in healthy Chinese youth:Difference in gut microbiota and predicted metabolic pathways[J].Journal of Agricultural and Food Chemistry, 2021, 69(44):13055-13065.
[9] 肖玉欣, 王楠, 王婧, 等.鞣花酸和尿石素类代谢产物的生物活性及其对肠道健康的作用研究进展[J].食品科学, 2022, 43(9):275-284.XIAO Y X, WANG N, WANG J, et al.A review of the bioactivity and gut health benefits of ellagic acid and its metabolites, urolithins[J].Food Science, 2022, 43(9):275-284.
[10] EVTYUGIN D D, MAGINA S, EVTUGUIN D V.Recent advances in the production and applications of ellagic acid and its derivatives.A review[J].Molecules, 2020, 25(12):2745.
[11] SELMA M V, TOM S-BARBER
S-BARBER N F A, BELTR
N F A, BELTR N D, et al. Gordonibacter urolithinfaciens sp. nov., a urolithin-producing bacterium isolated from the human gut[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 7):2346-2352.
N D, et al. Gordonibacter urolithinfaciens sp. nov., a urolithin-producing bacterium isolated from the human gut[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 7):2346-2352.
[12] LIU Q T, LIU S, YE Q W, et al.A novel Streptococcus thermophilus FUA329 isolated from human breast milk capable of producing urolithin A from ellagic acid[J].Foods, 2022, 11(20):3280.
[13] MI H Y, LIU S, HAI Y, et al.Lactococcus garvieae FUA009, a novel intestinal bacterium capable of producing the bioactive metabolite urolithin A from ellagic acid[J].Foods, 2022, 11(17):2621.
[14] ZHANG X M, FANG Y W, YANG G, et al.Isolation and characterization of a novel human intestinal Enterococcus faecium FUA027 capable of producing urolithin A from ellagic acid[J].Frontiers in Nutrition, 2022, 9:1039697.
[15] ZHANG M W, CUI S M, MAO B Y, et al.Urolithin A produced by novel microbial fermentation possesses anti-aging effects by improving mitophagy and reducing reactive oxygen species in Caenorhabditis elegans [J].Journal of Agricultural and Food Chemistry, 2023, 71(16):6348-6357.
[16] 刘银辉, 余萍, 闵祥博, 等.具有分解嘌呤核苷能力的益生菌菌株筛选[J].中国微生态学杂志, 2023, 35(7):772-777.LIU Y H, YU P, MIN X B, et al.Isolation of probiotic strains capable of decomposing purine nucleosides[J].Chinese Journal of Microecology, 2023, 35(7):772-777.
[17] LIM H J, LEE E H, YOON Y, et al.Portable Lysis apparatus for rapid single-step DNA extraction of Bacillus subtilis[J].Journal of Applied Microbiology, 2016, 120(2):379-387.
[18] GALPERIN M Y, MAKAROVA K S, WOLF Y I, et al.Expanded microbial genome coverage and improved protein family annotation in the COG database[J].Nucleic Acids Research, 2015, 43(Database issue):D261-D269.
[19] CANTAREL B L, COUTINHO P M, RANCUREL C, et al.The Carbohydrate-Active EnZymes database (CAZy):An expert resource for Glycogenomics[J].Nucleic Acids Research, 2009, 37(Database issue):D233-D238.
[20] BAIROCH A, APWEILER R.The SWISS-PROT protein sequence database and its supplement TrEMBL in 2000[J].Nucleic Acids Research, 2000, 28(1):45-48.
[21] CHEN L H, XIONG Z H, SUN L L, et al.VFDB 2012 update:Toward the genetic diversity and molecular evolution of bacterial virulence factors[J].Nucleic Acids Research, 2012, 40(Database issue):D641-D645.
[22] LIU B, POP M.ARDB antibiotic resistance genes database[J].Nucleic Acids Research, 2009, 37(Database issue):D443-D447.
[23] DENG L Q, LIU L M, FU T Y, et al.Genome sequence and evaluation of safety and probiotic potential of Lactiplantibacillus plantarum LPJZ-658[J].Microorganisms, 2023, 11(6):1620.
[24] 刘少文, 何光华, 王玲, 等.乳酸菌的应用及其安全性评价[J].中国畜牧业, 2018(11):53-56.LIU S W, HE G H, WANG L, et al.Application of lactic acid bacteria and its safety evaluation[J].China Animal Industry, 2018(11):53-56.
[25] FRANCL A L, THONGARAM T, MILLER M J.The PTS transporters of Lactobacillus gasseri ATCC 33323[J].BMC Microbiology, 2010, 10:77.
[26] XIA M J, MU S T, FANG Y W, et al. Genetic and probiotic characteristics of urolithin A producing Enterococcus faecium FUA027[J]. Foods, 2023, 12(5):1021.
[27] 胡爱华, 敖晓琳, 陈岑, 等. 乳酸菌耐酸耐胆盐机制的研究进展[J]. 食品工业科技, 2015, 36(8):380-383; 389.HU A H, AO X L, CHEN C, et al. Research progress on mechanism of lactic acid bacteria acid and bile salt resistance[J]. Science and Technology of Food Industry, 2015, 36(8):380-383; 389.
[28] 王崇民, 王翠竹. 可用于婴幼儿食品的“新食品原料”再添新成员: 长双歧杆菌长亚种BB536[J]. 食品安全导刊, 2022(22):8-9.