人体独特的肠道环境为微生物的生存提供了良好的栖息处所[1]。研究表明人体肠道内微生物数量高达1014个,与胃肠道疾病密切相关[2],同时与心血管系统、内分泌系统和神经系统疾病也有着密切的联系[3]。采用基因测序方法对比疾病人群及健康人群的肠道菌群,分析菌群构成及功能差异,寻找与疾病存在密切联系的微生物,从而分析微生物参与人体代谢的关键作用可为疾病的诊断和治疗提供帮助[4]。基于基因组技术的微生物组研究被《科学》杂志评为2010年度十大科学卓见。探寻微生物与健康之间的关系被《科学》杂志列为2013年度十大科学突破。尽管基于非培养手段的基因测序技术极大的方便了肠道微生物的鉴定与分析,但该技术存在一定的局限性,如不能检测到低丰度(<104 /g粪便)的微生物,存在DNA提取误差、序列读取错误、重复拷贝确定错误、质量值误差、算法误差等[5]。更为主要的是测序只是在基因水平上的分析,无法真正地获得目标微生物。这些局限性促使培养组学技术得到发展。
虽然培养法是分离和培育微生物时使用的历史最为悠久的技术,但培养组学方法是由LAGIER等[6]在2012年提出的一种全新方法,指通过不同的培养条件结合原位培养或使用选择性和差异生长培养基,以及向培养基中添加特定生长因子等多种培养方法,高通量地分离复杂环境样品中微生物。该方法极大的提升了发现新种及研究微生物互作的能力[6-8]。虽然通过在培养基中添加微生物生长所需的底物可用于富集和分离微生物,但想要在培养基中实现良好的适合多数微生物生长的条件还非常困难。因此,富含营养的培养基在肠道菌群研究中受到广泛的关注,因其有助于提高微生物的培养效率以及发现新的微生物种类的概率。富含营养的培养基是一类含有多种营养成分的培养基,能提供丰富、多样化的营养物质,以满足微生物在培养过程中的各种生长需求[9]。较常用的包括YCFA培养基(yeast extract-casein hydrolysate-fatty acid medium)和GAM培养基(gifu anaerobic medium)等。其中YCFA培养基是一种富含生长因子、抗氧化剂、挥发性脂肪酸和维生素的富集培养基。BROWNE等[10]采用YCFA培养基成功从人类肠道微生物群中分离出137个物种,其中包括68个新物种。类似地,FORSTER等[11]采用将样本直接接种到YCFA培养基中的方法,成功分离出273种不同的细菌物种,其中包括105个新的细菌物种。GAM培养基是一种用于培养厌氧微生物的培养基,能够为在厌氧条件下生长的微生物提供较理想环境。GOTOH等[12]采用GAM培养基成功分离出32种人类肠道优势细菌物种,包括17种厚壁菌门、14种拟杆菌门和1种放线菌门细菌。但由于不同人群、不同环境、不同个体间肠道菌群存在差异,因此,在开展特定研究时,应筛选适用于相应研究人群的培养基以获得更多的可培养菌株[9]。
本研究将分离人肠道菌群,利用菌落总数测定法结合宏基因组测序技术对比YCFA、GAM、ZJ(一种具有专利保护的适合肠道菌群体外培养的培养基)3种培养基对肠道菌群的培养效果,以评价3种培养基的优劣,为后续人类肠道微生物群体外培养的基质选择提供理论参考。
1 材料与方法
1.1 材料与试剂
300目尼龙纱布,常州市赛辉物资有限公司;纳米胶,深圳市创诚胶带有限公司;粪便收集盒(40 mL),四川日日生金商贸有限公司;HB8518-1 GAM培养基、HB9212 YCFA培养基、HB0101 PCA培养基、HBB274 细菌琼脂粉,青岛高科技工业园海博生物技术有限公司;蛋白胨、酵母粉、牛肉膏、葡萄糖、半胱氨酸、胆盐、纤维二糖、果糖、硫酸镁、磷酸二氢钾、维生素K、硫酸亚铁、血红素、1 mol/L CaCl2溶液(均为分析纯),北京耀友科技有限公司;10019318氯化钠,上海沪试实验室器材股份有限公司;47109凯杰磁珠法试剂盒,德国QIAGEN公司;其余均为实验室常用试剂,市售分析纯。
1.2 仪器与设备
NDF-86V188E医用低温保存箱,安徽中科都菱商用电器股份有限公司;YP10002电子天平,上海越平科学仪器(苏州)制造有限公司;FA2204B电子天平,上海精科天美科学仪器有限公司;HVE-50灭菌器,广东华粤企业集团有限公司;BBS-SDC洁净工作台,济南鑫贝西生物技术有限公司;MGL-16M台式高速冷冻离心机,美瑞克仪器(上海)有限公司;厌氧培养盒、厌氧产气袋,青岛高科技工业园海博生物技术有限公司;7.0 L密封培养罐,三菱瓦斯化学株式会社;PHS-3E pH计,上海蚁霖科学仪器有限公司。其余为实验室常用设备。
1.3 实验方法
1.3.1 培养基的配制
ZJ培养基根据专利[13]要求配制,每1 000 mL中含有蛋白胨2.5 g、酵母粉1.5 g、牛肉膏3 g、葡萄糖0.8 g、半胱氨酸0.8 g、胆盐1.2 g、纤维二糖1.5 g、果糖1.5 g、硫酸镁0.03 g、磷酸二氢钾2.5 g、维生素K1 0.003 g、硫酸亚铁0.001 g、血红素0.004 g、1 mol/L CaCl2溶液0.4 mL、余量为蒸馏水。使用1 mol/L NaOH溶液调节pH值至7.0。配制完成并灭菌后的液体培养基分装在15 mL的无菌离心管中,置于4 ℃冰箱保存。GAM培养基每1 000 mL蒸馏水中含有49.0 g培养基,并于121 ℃高压灭菌15 min,冷却至50 ℃左右时,每1 000 mL培养基中加入无菌0.1%(质量分数)维生素K1溶液1 mL和氯化血红素(5 mg/mL)1 mL,混匀备用。YCFA培养基每1 000 mL蒸馏水中含有18.44 g培养基,加热溶解并于121 ℃高压灭菌15 min。GAM培养基和YCFA培养基按照ZJ培养基的分装方法进行分装。
1.3.2 粪便样本的采集
本实验不涉及临床干预试验,仅收集志愿者粪便样本和基础临床指标,严格遵循伦理原则,已通过上海市杨浦区市东医院伦理委员会授权(审批号:2022-038-01),志愿者均已签署知情同意书。
4份20 g新鲜粪便分别收集自4位健康志愿者(年龄23~30岁)。志愿者在收集前30 d内无抗生素使用史。将尼龙纱布裁剪成合适的尺寸,用纳米胶将尼龙纱布固定在马桶中间偏后的位置,用尼龙纱布取样并用粪便收集盒转移至粪便收集器中,低温快速转移至实验室。从4份样本中分别称取1 g试样,混合均匀后用于后续实验。剩余样本被冻存于-80 ℃保藏[14]。
1.3.3 粪便菌群的获取
首先对比2种分离方法对粪便菌群的提取效果(图1)。
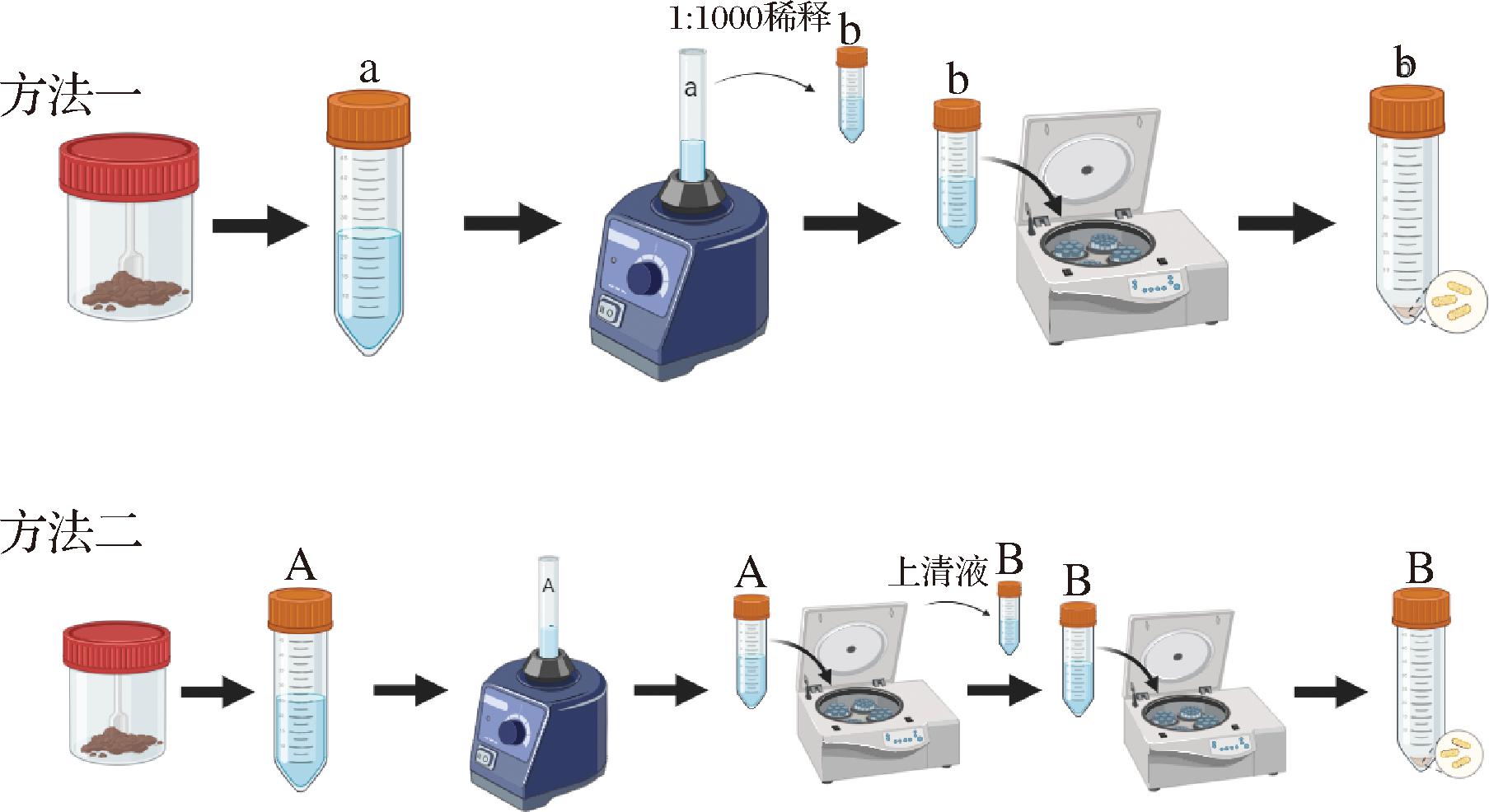
图1 获取粪便菌群的2种方法
Fig.1 Two methods for obtaining fecal microbiota
方法一[15]:称取1 g混合均匀的新鲜粪便于50 mL无菌离心管中(编号a)中,加入9 mL无菌生理盐水,并立即置于涡旋仪上涡旋4 min,根据十倍梯度稀释法吸取1 mL混合溶液,置于9 mL无菌生理盐水中,重复上述操作直至稀释体积比为1∶1 000。将a中所有样品全部稀释完毕后收集到同一无菌离心管中(编号b),4 ℃、10 842×g离心4 min。弃去上清液,收集底部沉淀即为菌群样本。
方法二[13]:取混合均匀的新鲜粪便1.0 g置于50 mL无菌离心管(编号A)中,加入40 mL无菌生理盐水,并立即置于涡旋仪上涡旋3 min,将A管在243×g、4 ℃条件下离心4 min,将上清液置于无菌离心管B中,在10 842×g、4 ℃离心4 min,弃去上清液。重新取40 mL无菌生理盐水置于A中,重复以上操作3次。在最后一次弃去B管中上清液,收集底部沉淀。
经过对比评估,方法一中虽能提取出粪便菌体,但仍有较多粪便杂质影响进一步实验,且稀释步骤较为繁琐。而方法二提取出的菌体纯度相对较高,且步骤更为简便。因此将采取方法二进行粪便菌群的提取。
1.3.4 粪便菌群的培养
实验分别采用YCFA、GAM、ZJ 3种培养基进行粪便菌群的培养,分别命名为YCFA、GAM、ZJ组。不同培养基的配方见表1。向含有菌群样本的50 mL离心管中加入40 mL无菌生理盐水,并立即置于涡旋仪上涡旋3 min制成菌液。而后取300 μL菌液至含有10 mL培养基的15 mL无菌离心管中,将离心管放入装有厌氧包的厌氧盒中37 ℃厌氧培养6、12、24、36、72 h、5、7 d后,测定菌落总数,并采用高通量测序技术分析培养36 h后菌群组成情况。实验同时设立未经培养的粪便分离菌样本(UC组)和未经提取步骤的粪便样本(UI组)低温送至测序公司进行细菌基因组DNA提取和测序。
表1 不同试验组培养基配方
Table 1 Formulation of culture media for different experimental groups
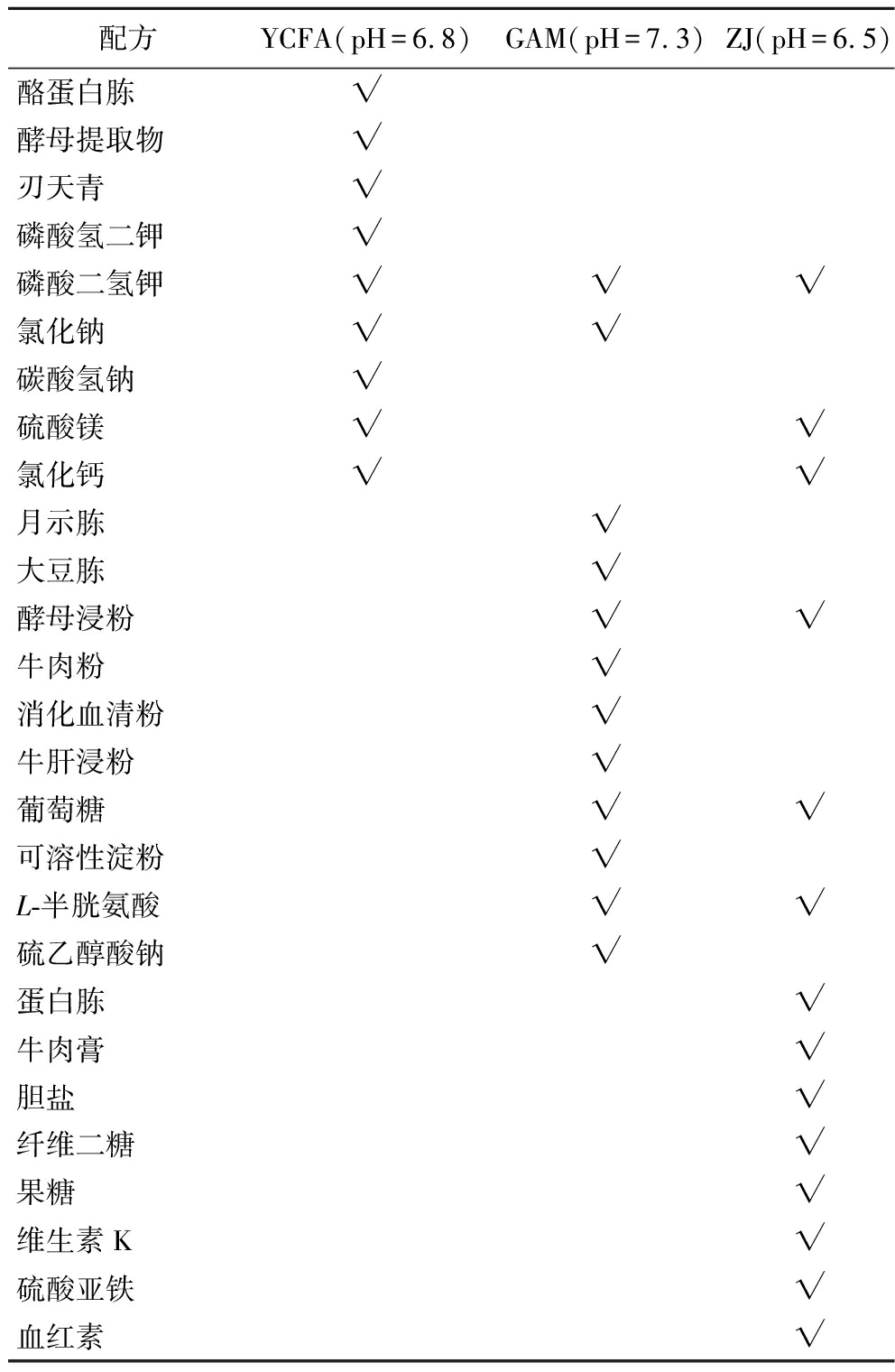
配方YCFA(pH=6.8)GAM(pH=7.3)ZJ(pH=6.5)酪蛋白胨√酵母提取物√刃天青√磷酸氢二钾√磷酸二氢钾√√√氯化钠√√碳酸氢钠√硫酸镁√√氯化钙√√月示胨√大豆胨√酵母浸粉√√牛肉粉√消化血清粉√牛肝浸粉√葡萄糖√√可溶性淀粉√L-半胱氨酸√√硫乙醇酸钠√蛋白胨√牛肉膏√胆盐√纤维二糖√果糖√维生素K√硫酸亚铁√血红素√
1.3.5 菌落总数测定
为了对比GAM、YCFA、ZJ培养基的计数结果,根据标准GB 4789.2—2010《食品安全国家标准 食品微生物学检验 菌落总数测定》采用改进后的平板计数法分别用GAM、YCFA、ZJ、平板计数琼脂(plate count agar, PCA)培养基测定培养后的菌落总数。PCA培养基一般用于食品微生物学检验中菌落总数的测定,通过PCA培养基对肠道菌的计数来为其他3种 固体培养基的结果进行对照。准备96孔深孔板,吸取100 μL不同培养时间的菌液,加入1 350 μL生理盐水进行梯度稀释。而后分别吸取100 μL稀释度为10-5~10-7的菌液于无菌平板中,分别倒入GAM、YCFA、ZJ、PCA的琼脂培养基(琼脂质量分数1.2%)中,待平板凝固后倒置于37 ℃恒温培养箱中培养24~48 h并进行菌落计数。每种固体培养基每个稀释度下重复2组。
1.3.6 菌群高通量测序分析
1.3.6.1 细菌基因组DNA提取
基于菌落总数测定结果,取经不同培养基培养36 h后的样本提取细菌基因组DNA,每个样品设置3个生物学重复,共15个样本。按照试剂盒说明书进行DNA抽提,部分步骤略有修改:a)取适量(0.25~0.5 g)样品加入到提前准备的加样管;b)加入800 μL CD1,5 μL Rnase A溶液,6 m/s匀浆研磨40 s,14 000×g离心5 min;c)转移上清液600 μL至准备好的CD2离心管中,颠倒混匀;d)转入离心机14 000×g 离心3 min;e)取450 μL上清液至试剂盒自带的Qsb1孔板中;f)按工作站提取要求进行后续实验操作,等待收集DNA;g)工作站停止运行并提示运行结束后,取出孔板DNA,定浓度,转到新的1.5 mL收集管,质量分数为1%琼脂糖凝胶电泳检测DNA纯度。
1.3.6.2 PCR及建库
采用通用引物338F(5′-ACT CCT ACGGGA GGC AGC AG-3′)和806R(5′-GGA CTA CHVGGG TWT CTA AT-3′)对细菌16S rDNA进行扩增。扩增体系为:5×FastPfu Buffer 4 μL,2.5 mM dNTPs 2 μL,Forward Primer(5 μmol/L) 0.8 μL,Reverse Primer(5 μmol/L) 0.8 μL,FastPfu Polymerase 0.4 μL,BSA 0.2 μL,Template DNA 10 ng,使用ddH2O补足至20 μL。扩增程序为:95 ℃ 3 min,95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,27个循环;72 ℃延伸10 min,10 ℃保温。PCR结束后,对产物进行琼脂糖电泳和凝胶回收,并送至上海美吉生物医药科技有限公司采用Illumina Hiseq 2000平台进行高通量测序分析。
1.3.6.3 测序结果分析
测序完成后采用FLASH(Version 1.2.11,http://ccb.jhu.edu/software/FLASH/index.shtml)软件将pair-end双端序列拼接。采用Uparse(Version 11,http://www.drive5.com/uparse/)进行OTU聚类算法。采用RDP Classifier(Version 2.13,https://sourceforge.net/projects/)进行序列分类注释。采用Usearch(Version 11,http://www.drive5.com/usearch/)进行OTU统计。采用Mothur(Version 1.30.2,https://www.mothur.org/wiki/)进行α-多样性分析。采用Mega(Version 7.0,https://www.megasoftware.net/)进行进化树分析。实验中所涉及的序列数据已提交至NCBI序列读取存档(sequence read archive,SRA)数据库,登录号为PRJNA 1050141。
1.4 数据处理
数据采用均值±标准偏差(standard deviation,SD)表示,采用IBM SPSS Statistics 25统计软件(IBM, New York, USA)进行单因素方差分析(analysis of variance,ANOVA)检验,检验的显著性水平设定为P<0.05。
2 结果与分析
2.1 不同培养基菌落总数变化趋势
2.1.1 YCFA培养基
不同固体培养基菌落计数条件下YCFA液体培养基中菌落总数变化趋势见图2。经YCFA液体培养基培养后,采用YCFA、PCA、GAM固体培养基进行计数均显示菌落总数的变化均呈先上升后下降的趋势,并且在24 h时达到最高值,分别为9.38、9.51、9.63 lg CFU/mL,三者中经GAM固体培养基计数时菌落总数值最高。采用ZJ固体培养基计数时,菌落总数呈下降趋势,6 h时为最高值(9.68 lg CFU/mL)。综合分析,在24 h时菌落活菌总数普遍达到最高值,在36 h开始呈现下降趋势。
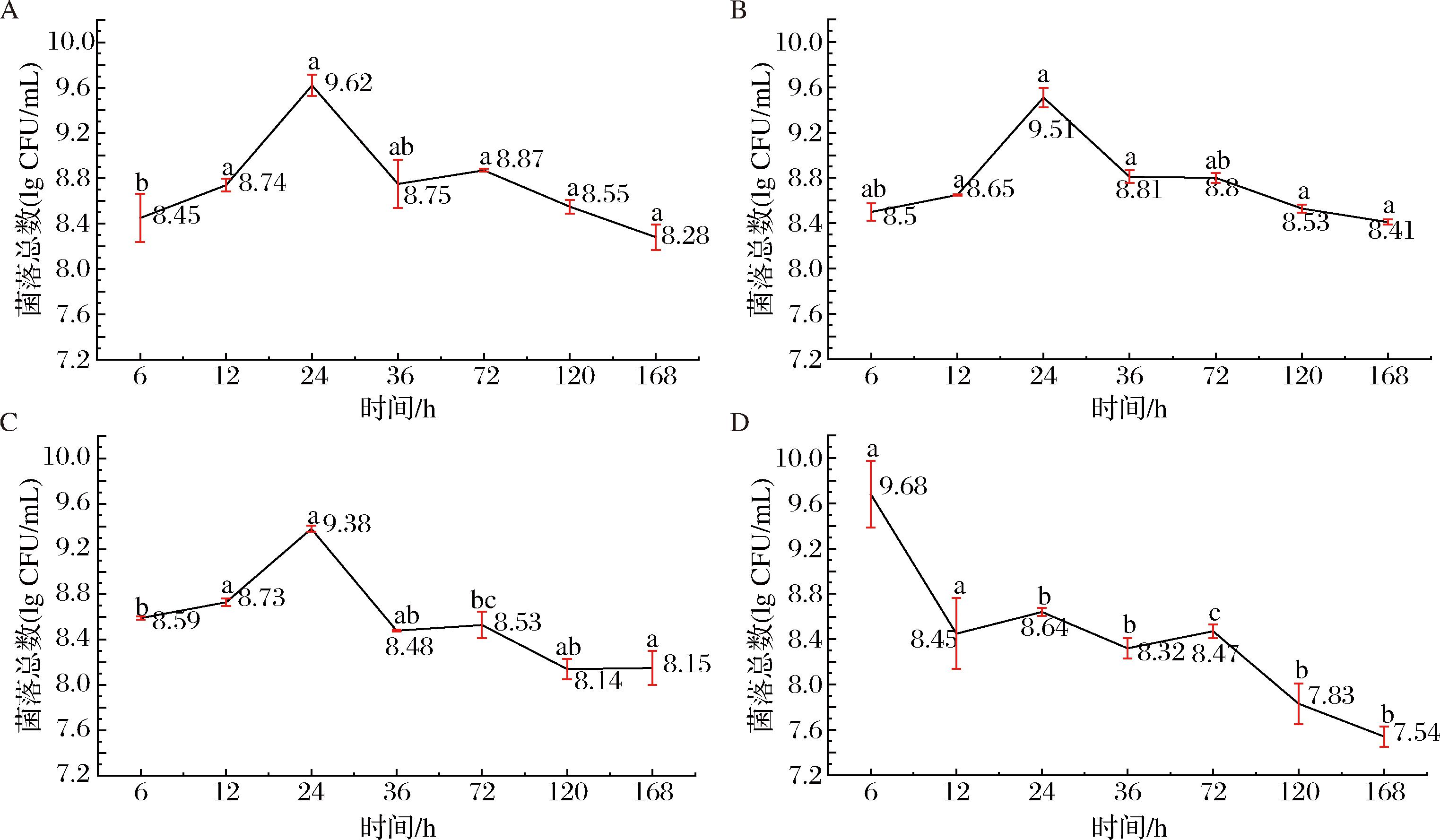
a-GAM组;b-PCA组;c-YCFA组;d-ZJ组
图2 不同固体培养基菌落计数条件下YCFA液体培养基中菌落总数变化趋势
Fig.2 Changes in the total colony counts in YCFA liquid medium under different solid culture conditions
注:不同小写字母代表差异显著(P<0.05)(下同)。
对不同固体培养基菌落计数条件下YCFA液体培养基中菌落总数进行了显著性分析,不同字母表示相同时间下不同固体培养基测得的菌落总数具有显著性差异(P<0.05)。结合显著性分析,在6 h时,ZJ培养基的菌落总数显著(P<0.05)高于其他3种培养基。然而在培养24 h后,YCFA、GAM与PCA培养基之间无显著差异,但三者均显著(P<0.05)高于ZJ培养基。
2.1.2 GAM培养基中菌落总数变化趋势
如图3所示,经过GAM液体培养基培养后,采用PCA、GAM、ZJ固体培养基进行计数均显示菌落总数呈先上升后下降趋势,其中PCA和GAM显示在培养24 h时菌落总数值最高,分别为9.51 lg CFU/mL和9.61 lg CFU/mL。ZJ固体培养基则显示在培养12 h时菌落总数达到最高值,为9.88 lg CFU/mL,而YCFA培养基计数显示菌落总数呈下降趋势,在6 h时达到最高值9.69 lg CFU/mL。
对不同固体培养基菌落计数条件下GAM液体培养基中菌落总数进行了显著性分析,不同字母表示相同时间下不同固体培养基测得的菌落总数具有显著性差异(P<0.05)。在6 h时,YCFA培养基和ZJ培养基的菌落总数显著(P<0.05)高于GAM和PCA培养基。在12 h时,ZJ培养基的菌落总数显著(P<0.05)高于其他3种培养基。但在24 h后,YCFA和ZJ培养基中菌落总数开始显著(P<0.05)低于GAM和PCA培养基。

a-GAM组;b-PCA组;c-YCFA组;d-ZJ组
图3 不同固体培养基菌落计数条件下GAM液体培养基中菌落总数变化趋势
Fig.3 Trend of total colony count in GAM liquid medium under different solid medium colony counting conditions
2.1.3 ZJ培养基中菌落总数变化趋势
如图4所示,经过ZJ液体培养基培养后,利用4种固体培养基计数时菌落总数均显示随着培养时间的增加,菌落总数会呈现先上升后下降的趋势。在12 h时,GAM和ZJ培养基菌落总数达到最高点,分别为8.15 lg CFU/mL和9.79 lg CFU/mL。在24 h时,PCA和YCFA培养基达到最高点,分别为9 lg CFU/mL和7.83 lg CFU/mL。
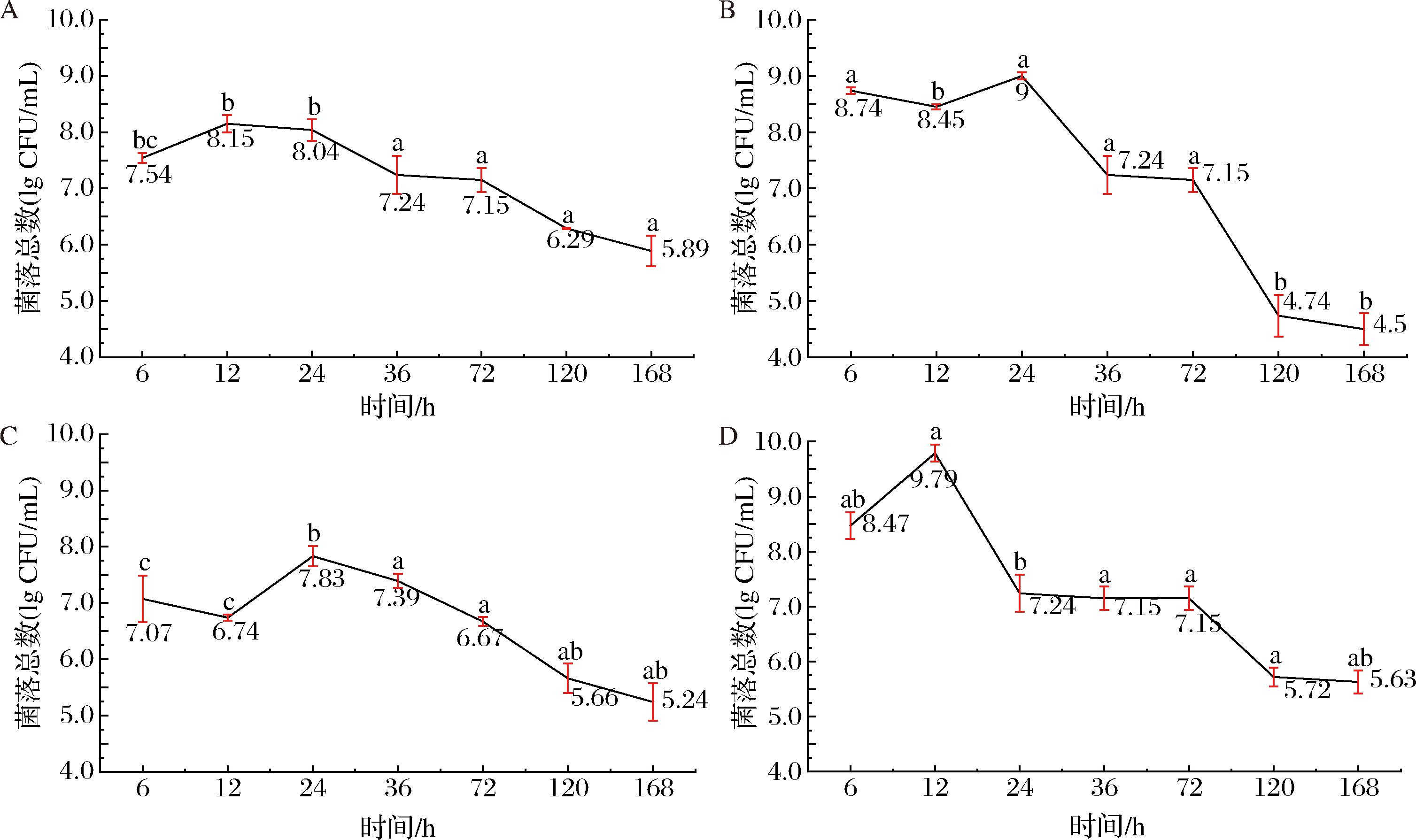
a-GAM组;b-PCA组;c-YCFA组;d-ZJ组
图4 不同固体培养基菌落计数条件下ZJ液体培养基中菌落总数变化趋势
Fig.4 Trend of total colony count in ZJ liquid medium under different solid medium colony counting conditions
对不同固体培养基菌落计数条件下ZJ液体培养基中菌落总数进行了显著性分析,不同字母表示相同时间下不同固体培养基测得的菌落总数具有显著性差异(P<0.05)。培养6 h后,ZJ培养基的菌落总数显著(P<0.05)高于YCFA和GAM,在12 h时该趋势更为明显。
综上,不同培养基的培养效果存在明显差异,经过3种液体培养基培养后的肠道菌群在4种不同固体培养基的计数结果分析可知,经ZJ液体培养基短期培养(6~12 h)后肠道菌群的菌落总数最高。而经YCFA和GAM液体培养基培养时,菌落总数多在24 h时达到最高值,并在36 h开始下降。同时,GAM培养后的菌落总数普遍高于YCFA培养基。
2.2 菌群α-多样性分析
经样品基因组DNA提取、扩增及测序分析后,15个样本共得到130.8万条测序片段,经拼接和质量控制过滤共得到128.14万条序列,序列长度约417 bp。物种Veen图分析如图5所示。不同的颜色代表不同的分组,重叠部分的数字代表多个分组中共有的物种数目,非重叠部分的数字代表对应分组所特有的物种数目,中心是所有分组共有的物种数目。经分析可知,除共有OTU数外,YCFA组和GAM组OTU数显著高于ZJ组,表明相较于ZJ组,YCFA组和GAM组的物种数量更为丰富。
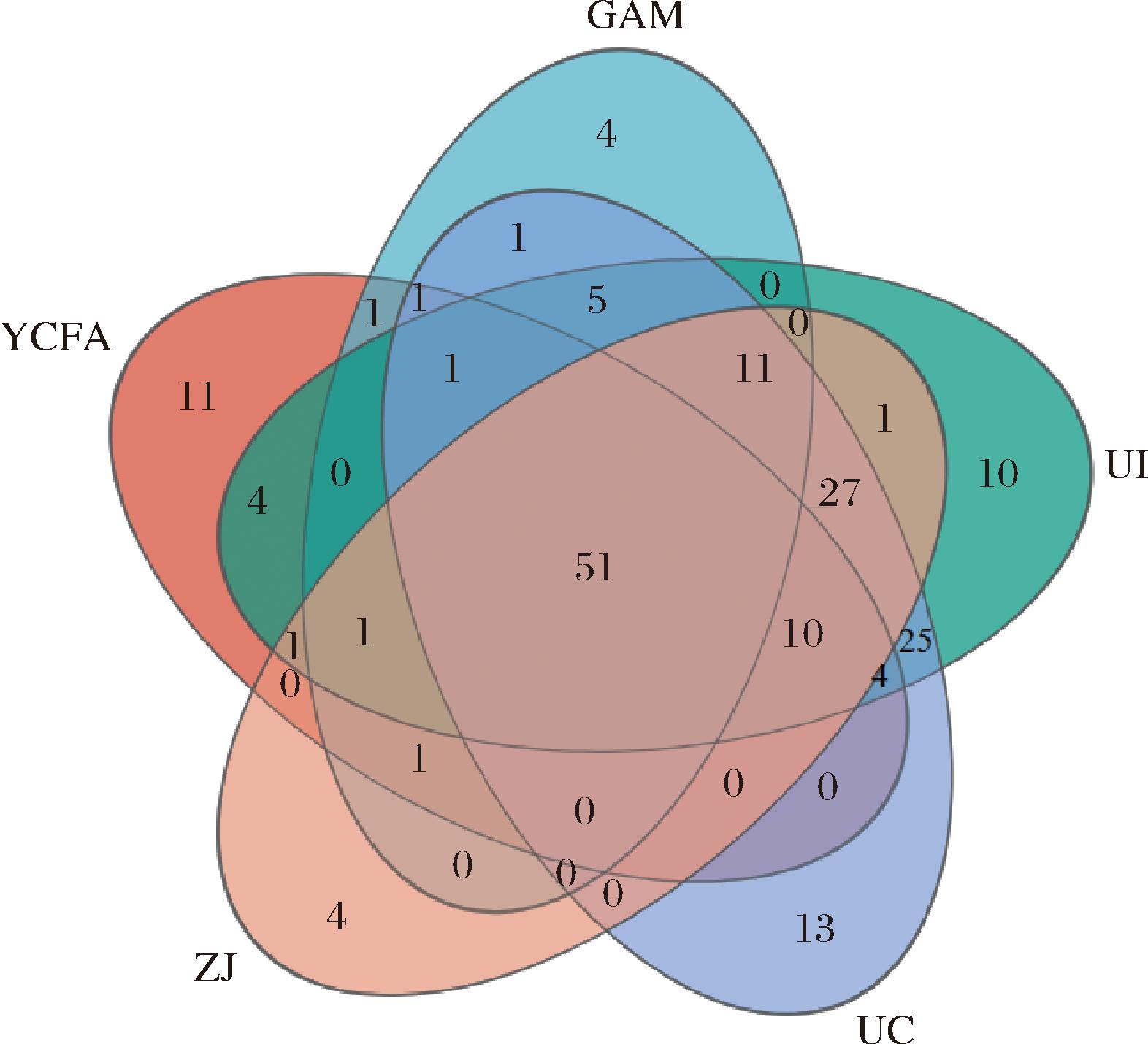
图5 物种Veen图
Fig.5 Species Veen Chart
各样本Shannon指数、Coverage指数、Simpson指数、Ace指数以及Chao1指数见表2。Shannon指数和Simpson指数常用来估算样本中微生物多样性。Shannon值越大,说明群落多样性越高。Simpson指数值越大,说明群落多样性越低。比较各培养组可知,ZJ组的Shannon指数显著(P<0.05)低于YCFA和GAM组,而其Simpson指数显著(P<0.05)高于YCFA和GAM组。表明经ZJ培养基培养后菌群的群落多样性显著下降。Coverage指数反映了各样本文库的覆盖率,数值越高表示样本中的序列被测出的概率越大。在本研究中,各组的Coverage指数均高于0.99,说明测序深度较好。Ace指数和Chao1指数在生态学中常用来估计物种总数。与UI和UC组相比,经培养后的各组肠道菌群的Shannon指数、Ace指数、Chao1指数均显著(P<0.05)降低,Simpson指数显著(P<0.05)升高,说明经体外培养后细菌多样性降低。
表2 不同培养基对菌群α-多样性指数的影响
Table 2 The impact of different culture media on the α-diversity index of microbial communities

组别Shannon指数Coverage指数Simpson指数Ace指数Chao1指数UI3.83±0.24a0.998 7±0.000 11b0.06±0.02d746.20±45.67a722.00±47.60aUC3.73±0.05a0.998 9±0.000 05b0.06±0.01d672.00±19.01b654.10±19.93aYCFA2.26±0.15b0.999 6±0.000 10a0.18±0.03c163.10±10.25c156.10±10.93bGAM1.91±0.05b0.999 6±0.000 02b0.27±0.02b151.20±9.19c147.10±10.58bZJ0.40±0.03c0.999 4±0.000 13a0.89±0.01a178.60±17.84c168.40±25.03b
2.3 三种培养基中菌群组成变化
经物种分类,15个样本中微生物归属于10门、16纲、42目、76科和187属。选取各样本在门水平和属水平的前10个优势菌群构建堆积柱状图(图6)。由图6可知,在门水平上,厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteriota)、拟杆菌门(Bacteroidota)、梭杆菌门(Fusobacteriota)为主要优势门。在UI组和UC组中占主要比重的厚壁菌门经不同培养基培养后丰度均下降,其中ZJ培养基培养后下降最明显。而变形菌门经培养后物种丰度上升,其中ZJ培养基培养后上升最为显著。此外,经YCFA、ZJ培养基培养后放线菌门细菌数量显著降低。梭杆菌门经YCFA培养后丰度显著上升。因此,从门水平细菌组成分析可知,厚壁菌门细菌对体外培养条件较为敏感,ZJ培养基会选择性富集变形菌门细菌,而YCFA培养基对梭杆菌门细菌具有富集作用,相比而言GAM培养基的菌群组成较接近于UI组和UC组。

图6 门水平和属水平菌群组成分析
Fig.6 Microbial community composition at the phylum and genus levels.
在属分类水平上,志贺氏菌属(Escherichia-Shigella)、双歧杆菌属(Bifidobacterium)、梭杆菌属(Fusobacterium)、肠球菌属(Enterococcus)和经黏液真杆菌属(Blautia)为主要优势属。在UI组和UC组中占比较小的志贺氏菌属经3种培养基培养后占比均上升,其中ZJ培养基培养后上升极明显(培养后丰度占比超93%)。肠球菌属(Enterococcus)在经GAM和YCFA培养基培养后占比上升。而梭杆菌属(Fusobacterium)经YCFA培养基培养后占比上升。原样本中占比较高的双歧杆菌属仅在GAM培养基培养后仍有较高占比。因此,志贺氏菌对体外培养环境较为耐受,不同培养基富集的菌种类不同。对比不同培养基可知与门水平相似,在属水平上经GAM培养基培养后的群落组成与体外培养前的原样品最为相似。
2.4 三种培养基菌群相似度分析
3种培养基线性判别分析(linear discriminant analysis,LDA)判别结果如图7所示。LEfSe多级物种差异判别分析(LDA effect size analysis)能够在多个层级(门,纲,目,科,属,种)进行差异检验,分析多层级的差异物种,并用LDA值来衡量物种对差异效果影响大小,能够说明某些物种可能在环境变化过程中起到关键的作用。LEfSe分析可显示在属水平上YCFA组、GAM组和ZJ组之间存在显著差异的细菌分类群。YCFA组的优势菌为副梭状芽孢杆菌(Paraclostridium)、气单胞菌(Aeromonas)和柠檬酸杆菌(Citrobacter)等,GAM组的优势菌为双歧杆菌(Bifidobacterium)、肠球菌(Enterococcus)、乳酸菌(Lactococcus)等,ZJ组的优势菌为志贺氏菌(Escherichia-Shigella)、克里斯滕森菌(Christensenellaceae_R-7_group)和毛螺菌(norank f-lachnospiraceae)等。

图7 属水平下3种培养基LDA判别结果表
Fig.7 Table of LDA discrimination results of three culture media
各组样本在属分类水平上的主坐标分析(principal coordinates analysis,PCoA)图见图8。图中不同颜色或形状的点代表不同分组的样本,两样本点越接近,表明两样本相似度越高。结果显示UC组和UI组的相似度较高,说明本实验肠道菌群分离方法较为成功。3种培养基组培养后的样本与UC组和UI组之间有显著差距,表明经培养后细菌组成和丰度发生明显变化。对比而言GAM组在主成分2(方差贡献率29.35%)方向上与UC组和UI组之间的距离为3种培养基之间最短,表明与YCFA和ZJ培养基相比,经GAM培养基培养后的样本物种组成与未经培养的样本之间物种组成最为相似。
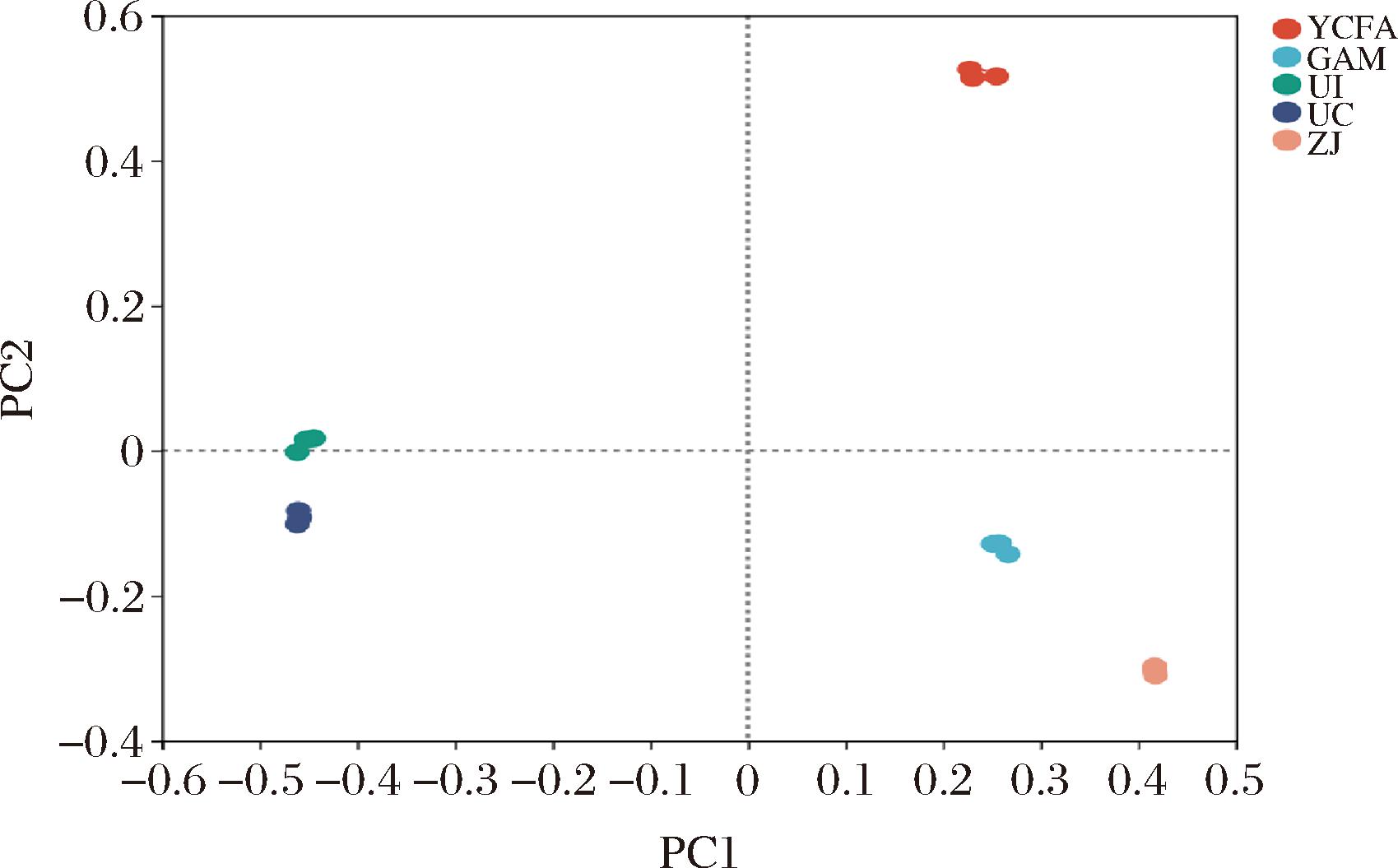
图8 PCoA分析
Fig.8 PCoA analysis
3 结论与讨论
肠道菌群与宿主健康密切相关。为了深入理解肠道菌群的功能,筛选并优化适合肠道菌群生长的培养基显得尤为关键[16]。但由于肠道菌群数量庞大、组成丰富,因此,找到满足多数肠道菌群生长条件的特定培养基仍然较为困难。本研究分析了不同富含营养的培养基对人肠道菌群的培养效果,利用平板计数法分析不同培养基培养不同时间下肠道菌群的菌落总数,结合高通量测序技术分析肠道菌群的多样性差异,筛选出最适培养基,为肠道菌群体外培养的合适培养基选择提供了科学依据。
平板计数法结果显示不同培养液对菌群的培养效果不同,同时相同菌液在不同固体培养基中的培养效果也存在明显差异。对比3种液体培养基的菌落总数可知经ZJ液体培养基短期培养后肠道菌群的菌落总数最高。但高通量测序结果显示ZJ培养基培养后菌群的群落多样性显著下降,变形菌门下的志贺氏菌属丰度明显上升。伍灵怡等[17]通过收集胆汁淤积患者的粪便分析其肠道菌群的结构特征发现,患者体内志贺氏菌属出现了明显的富集现象。推断可能是由于ZJ培养基中含有独特的胆盐成分导致肠道菌群中的志贺氏菌属被选择性富集。志贺氏菌属被认为是致病菌,为革兰氏阴性的促炎症病原体,能通过释放脂多糖引起急性肠道损伤,增加血脑屏障通透性,并激活神经炎症。目前已发现其与抑郁、帕金森病、自闭症等神经疾病相关[18-19]。
经YCFA和GAM液体培养基培养时,菌落总数随时间的变化趋势相似,而GAM培养后的菌落总数普遍高于YCFA培养基。GAM培养基能够选择性富集双歧杆菌属,该菌属为专性厌氧菌[20]。GAM含有半胱氨酸等成分,有助于营造厌氧环境。同时GAM培养基中含有能够满足厌氧菌所需的特定营养物质以及营养元素等,其pH范围(pH=7.3,高于YCFA和GAM培养基)适合厌氧菌的生长条件,常被作为厌氧菌培养的基质[9,21]。双歧杆菌是一种厌氧的革兰氏阳性菌,包含10个系统发育群集,广泛存在于哺乳动物的胃肠道中。来自双歧杆菌属的菌株也是最常用的益生菌补充剂。目前已有大量文献对各种双歧杆菌的益生特性进行了表征,例如其具有免疫调节和抗炎特性,可上调调节性T细胞,改善肠道屏障功能,调节树突状细胞和巨噬细胞活性等[22]。此外肠球菌属在GAM培养基中也得到了一定程度上的富集。类似地,MABWI等[23]采用GAM培养基对肠道菌群进行体外培养后发现肠球菌属被明显富集。肠球菌是革兰氏阳性、过氧化氢酶阴性的细菌,通常被归类为乳酸菌群,可发酵产生乳酸,同时可产生多种有益物质(如细菌素和胞外多糖等),在食品与医药等行业中发挥着重要的角色[24]。β-多样性分析结果显示经GAM培养基培养后的菌群组成更接近未培养时的UI组和UC组。MABWI等[23]同样发现GAM培养基能够培养出物种更加丰富的肠道菌群,并指出可能是由于培养基中的酵母浸粉、牛肉粉、肝脏提取物、蛋白胨、无机盐等能够满足大多数细菌的营养要求,甚至是一些难以培养的肠道菌。GOTOH等[12]以GAM作为标准培养基,从《人类肠道微生物基因目录》列出的前56种物种中,选择了44种在GAM培养基中进行培养,发现有32种(72%)物种可被GAM培养基成功培养。尽管YCFA与GAM培养基均为当前厌氧微生物培养的普遍选择,但我们的结果显示GAM培养基更适合肠道菌群的生长。与我们的研究类似,TIDJANI等[9]发现相较于YCFA培养基,GAM培养基能够培养更多种类的厌氧菌。
本研究比较了3种富营养培养基对人肠道菌群的培养效果,对后续肠道菌群的体外培养研究具有较好的参考意义。但研究也存在一定的局限性。首先,研究采用的样本数量较少,而肠道菌群的个体差异较大,且个体的生活方式以及饮食习惯等均会影响肠道菌群的组成[25-26],因此研究结果无法较为全面地代表人体肠道菌群的特征;其次,研究仅对比了3种富营养培养基,但目前已有多种富营养培养基被用于肠道菌群的体外培养,如brain heart infusion、tryptone- yeast extract-glucose、luria-bertani broth、mueller-hinton broth、anaerobe asal broth以及anaerobic medium base等[27-28];同时由于不同培养基的营养组成不同,特定培养基可能仅选择性富集特定的肠道菌群,因此,培养基种类较少可能导致某些菌群生长的营养缺乏,使得某些菌群被忽略[29],因此研究结果无法完整反映肠道菌群的多样性;再者,由于体外培养条件与人体肠道环境存在较大差异,仅采用富营养培养基结合厌氧环境无法准确反映肠道内的复杂环境[30]。因此,未来将通过增加样本数量、选择更多的富营养培养基、采用更接近体内环境的肠道环境模拟系统开展进一步的研究,以期为肠道菌群的体外培养方法选择提供更高效的数据参考。
综上所述,本研究显示虽然经ZJ培养基短期培养后可获得大量的肠道菌株,但菌群的多样性与未培养时差异较大。相较于ZJ和YCFA培养基,GAM培养基能够培养出较多数量、组成最为接近未培养肠道菌群的菌株。因此,我们推荐选择GAM培养基开展人肠道菌群的体外培养研究。但由于不同研究中采用的培养基营养成分、培养条件、受试者不同,得到的实验结果也可能存在差异。因此,为获得更优的培养效果,推荐筛选适用于相应研究人群和研究环境的培养基。
[1] ABDUL-HUSSEIN Z R, ATIA S S.Antimicrobial effect of pyocyanin extracted from Pseudomonas aeroginosa[J].European Journal of Experimental Biology, 2016, 6(6):1-4.
[2] SAUS E, IRAOLA-GUZM N S, WILLIS J R, et al.Microbiome and colorectal cancer:Roles in carcinogenesis and clinical potential[J].Molecular Aspects of Medicine, 2019, 69:93-106.
N S, WILLIS J R, et al.Microbiome and colorectal cancer:Roles in carcinogenesis and clinical potential[J].Molecular Aspects of Medicine, 2019, 69:93-106.
[3] LIU R X, HONG J, XU X Q, et al.Gut microbiome and serum metabolome alterations in obesity and after weight-loss intervention[J].Nature Medicine, 2017, 23(7):859-868.
[4] NAKATSU G, LI X C, ZHOU H K, et al.Gut mucosal microbiome across stages of colorectal carcinogenesis[J].Nature Communications, 2015, 6:8727.
[5] NAYFACH S, POLLARD K S.Toward accurate and quantitative comparative metagenomics[J].Cell, 2016, 166(5):1103-1116.
[6] LAGIER J C, ARMOUGOM F, MILLION M, et al.Microbial culturomics:Paradigm shift in the human gut microbiome study[J].Clinical Microbiology and Infection, 2012, 18(12):1185-1193.
[7] LAGIER J C, DUBOURG G, MILLION M, et al.Culturing the human microbiota and culturomics[J].Nature Reviews Microbiology, 2018, 16:540-550.
[8] 余中节, 赵洁, 孙志宏.多组学技术在自然发酵乳中的应用[J].生物产业技术, 2019(4):63-68.YU Z J, ZHAO J, SUN Z H.Application of multi-omics technology in naturally fermented milk[J].Biotechnology &Business, 2019(4):63-68.
[9] TIDJANI ALOU M, NAUD S, KHELAIFIA S, et al.State of the art in the culture of the human microbiota:New interests and strategies[J].Clinical Microbiology Reviews, 2020, 34(1):e00 129-19.
[10] BROWNE H P, FORSTER S C, ANONYE B O, et al.Culturing of ‘unculturable’ human microbiota reveals novel taxa and extensive sporulation[J].Nature, 2016, 533(7604):543-546.
[11] FORSTER S C, KUMAR N, ANONYE B O, et al.A human gut bacterial genome and culture collection for improved metagenomic analyses[J].Nature Biotechnology, 2019, 37(2):186-192.
[12] GOTOH A, NARA M, SUGIYAMA Y, et al.Use of Gifu Anaerobic Medium for culturing 32 dominant species of human gut microbes and its evaluation based on short-chain fatty acids fermentation profiles[J].Bioscience, Biotechnology, and Biochemistry,2017, 81(10):2009-2017.
[13] 曾令聪, 胡犁涛.一种从人体粪便中分离肠道菌群的方法技术:中国, CN201510840354.1[P].2015-11-27.ZENG L C, HU L T.A method technology for separating intestinal flora from human feces:China, CN201510840354.1[P].2015-11-27.
[14] CHEN C C, WU W K, CHANG C M, et al.Comparison of DNA stabilizers and storage conditions on preserving fecal microbiota profiles[J].Journal of the Formosan Medical Association, 2020, 119(12):1791-1798.
[15] 常宇骁. 人类肠道微生物培养组优化及肠道菌库构建与应用[D].北京:军事科学院, 2020.CHANG Y X.Optimization of human intestinal microbial culture group and construction and application of intestinal bacterial library[D].Beijing:Academy of Military Science, 2020.
[16] LIU C, DU M X, ABUDUAINI R, et al.Enlightening the taxonomy darkness of human gut microbiomes with a cultured biobank[J].Microbiome, 2021, 9(1):119.
[17] 伍灵怡, 唐黎, 张静, 等.胆汁淤积性肝病患者肠道菌群特征及其临床相关性研究[J].第三军医大学学报, 2020, 42(22):2251-2258.WU L Y, TANG L, ZHANG J, et al.Characteristics of gut microbiota and their correlation with clinical parameters in patients with cholestatic liver disease[J].Journal of Third Military Medical University, 2020, 42(22):2251-2258.
[18] LIU L X, WANG H Y, ZHANG H P, et al.Toward a deeper understanding of gut microbiome in depression:The promise of clinical applicability[J].Advanced Science, 2022, 9(35):e2203707.
[19] LI Z, LIANG H F, HU Y Y, et al.Gut bacterial profiles in Parkinson′s disease:A systematic review[J].CNS Neuroscience &Therapeutics, 2023, 29(1):140-157.
[20] MILLION M, DIALLO A, RAOULT D.Gut microbiota and malnutrition[J].Microbial Pathogenesis, 2017, 106:127-138.
[21] CHE Q M, AKAO T, HATTORI M, et al.Isolation of a human intestinal bacterium capable of transforming barbaloin to Aloe-emodin anthrone[J].Planta Medica, 1991, 57(1):15-19.
[22] GAVZY S J, KENSISKI A, LEE Z L, et al.Bifidobacterium mechanisms of immune modulation and tolerance[J].Gut Microbes, 2023, 15(2):2291164.
[23] MABWI H A, HITAYEZU E, MAULIASARI I R, et al.Simulation of the mucosal environment in the re-construction of the synthetic gut microbial ecosystem[J].Journal of Microbiological Methods, 2021, 191:106351.
[24] KAVITAKE D, DEVI P B, DELATTRE C, et al.Exopolysaccharides produced by Enterococcus genus-An overview[J].International Journal of Biological Macromolecules, 2023, 226:111-120.
[25] SISTI D, PAZIENZA V, PICCINI F, et al.A proposal for the reference intervals of the Italian microbiota “scaffold” in healthy adults[J].Scientific Reports, 2022, 12:3952.
[26] RESENDE A S, LEITE G S F, LANCHA JUNIOR A H.Changes in the gut bacteria composition of healthy men with the same nutritional profile undergoing 10-week aerobic exercise training:A randomized controlled trial[J].Nutrients, 2021, 13(8):2839.
[27] ARANDA-D AZ A, NG K M, THOMSEN T, et al.Establishment and characterization of stable, diverse, fecal-derived in vitro microbial communities that model the intestinal microbiota[J].Cell Host &Microbe, 2022, 30(2):260-272.e5.
AZ A, NG K M, THOMSEN T, et al.Establishment and characterization of stable, diverse, fecal-derived in vitro microbial communities that model the intestinal microbiota[J].Cell Host &Microbe, 2022, 30(2):260-272.e5.
[28] WAN C, WU K Z, LU X Y, et al.Integrative analysis of the gut microbiota and metabolome for in vitro human gut fermentation modeling[J].Journal of Agricultural and Food Chemistry, 2021, 69(50):15414-15424.
[29] ZHANG N, JIN M L, WANG K M, et al.Functional oligosaccharide fermentation in the gut:Improving intestinal health and its determinant factors-a review[J].Carbohydrate Polymers, 2022, 284:119043.
[30] 刘莎, 陈从英.肠道微生物培养的研究进展及应用[J].微生物学报,2023, 63(3):881-899.LIU S, CHEN C Y.Research progress and application of gut microorganism culture[J].Acta Microbiologica Sinica, 2023, 63(3):881-899.