近年来,随着食品发酵技术应用需求的持续增长,为了弥补单一菌种发酵功能的局限性,利用多种具有不同功能的菌种来构建合成微生物群落已成为发酵食品领域的一个研究热点[1-3]。合成微生物群落的目的是利用单一微生物的代谢功能和微生物间的相互作用,实现代谢任务的分工与功能互补,从而增强群落的生产力、稳定性及代谢功能等[4]。合成微生物群落的合理构建需要考虑参与菌种的功能、生长特性、生长环境以及菌种间的相互作用等多种因素,因此分析合成微生物群落中各个菌种的物种丰度是相关研究的一项重要指标[5-6]。
在以往的研究中,分析合成微生物群落的主要手段是非培养方法,如16S rRNA、18S rRNA和转录间隔区(internal transcribed spacer, ITS)扩增子测序[7-8]。尽管这些方法在解析群落结构方面具有显著优势,但在准确量化合成微生物群落中每个物种的绝对丰度方面,其能力仍受到一定限制。此外,利用定量PCR技术对合成微生物群落中每个物种进行绝对定量也是一种有效的方法[6],但也存在很大的局限性,如操作繁复、工作量大、无法评估微生物活性,以及引物设计的挑战。另一种量化合成微生物群落物种绝对丰度的手段是基于选择性培养基的培养法,适用于成员数量较少的合成群落。这种基于培养的检测方法被认为具有成本效益和快速性优势,能够精准测定简单合成微生物群落中各物种的绝对丰度[9-10]。然而,在合成微生物群落研究中,建立基于培养的方法以量化菌群结构是一项具有挑战性的任务,原因在于需要投入大量精力研究如何通过培养将每个物种区分开来[10]。
在先前的研究中,发现鲁氏接合酵母(Zygosaccharomyces rouxii)、肉葡萄球菌(Staphylococcus carnosus)、枯草芽孢杆菌(Bacillus subtilis)、解淀粉芽孢杆菌(Bacillus amyloliquefaciens)、嗜盐四联球菌(Tetragenococcus halophilus)和融合魏斯氏菌(Weissella confusa)对豆瓣酱发酵的强化及品质提升具有重要作用[1,11]。为了精确地评估由上述菌株构成的合成微生物群落中各物种的绝对丰度,本研究将通过筛选对各物种具有针对性的抗生素组合以研制选择性培养基,从而构建基于培养技术的菌群结构检测方法。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
本研究使用的菌株包括鲁氏接合酵母Y-8、肉葡萄球菌M43、枯草芽孢杆菌R1、解淀粉芽孢杆菌M27、嗜盐四联球菌YM26以及融合魏斯氏菌YM34,均筛选自豆瓣酱酱醅并保藏于本实验室。
1.1.2 试剂
胰蛋白胨、蛋白胨、酵母粉、葡萄糖、牛肉膏、氯化钠、乙酸钠、柠檬酸氢二铵、磷酸氢二钾(三水)、硫酸镁、硫酸锰、吐温-80、结冷胶、聚醚F-68,上海国药集团化学试剂有限公司。
用于药敏试验的42种抗生素,MedChemExpress(中国上海)的抗菌化合物库,以溶液形式低温贮存;用于研制选择性培养基的6种抗生素(氨苄西林钠、萘夫西林钠、盐酸金霉素、恶喹酸、林可霉素和制霉菌素),上海阿拉丁试剂有限公司,以固体粉末形式低温贮存,所有抗生素的CAS号见附表1( https://doi.org/10.13995/j.cnki.11-1802/ts.038844)。
表1 五株豆瓣酱源细菌对不同抗生素的敏感性
Table 1 Drug susceptibility of five bacteria derived from broad bean paste to different antibiotics

抗生素类型抗生素对以下菌株抑菌率>90%的最小质量浓度/(μg/mL)SBsBaTW氨苄西林钠10.0000.40010.000N/D10.000萘夫西林钠1.4540.2910.29136.3581.454氯唑西林钠一水合物1.5230.3050.3057.6147.614头孢噻吩0.7930.1590.1590.7930.793阿洛西林钠1.9340.077N/D1.9341.934β-内酰胺类头孢呋辛钠4.4640.0360.8934.4640.893青霉素G普鲁卡因水合物0.2350.047N/D1.1771.177苯唑西林钠一水合物0.8830.1770.1774.4144.414头孢美唑钠4.9350.9870.987N/DN/D头孢克洛N/D0.1470.736N/DN/D卡茚西林钠5.1651.0335.1655.1655.165头孢唑林0.9090.9090.1824.5454.545盐酸金霉素2.5770.1032.577N/DN/D四环素类盐酸米诺环素4.9390.9880.988N/DN/D盐酸四环素N/D0.240N/DN/DN/D恶喹酸N/D0.5222.612N/DN/D环丙沙星N/D0.1330.663N/DN/D氧氟沙星N/D0.7230.723N/DN/D喹诺酮类左氧氟沙星N/D0.7230.723N/DN/D盐酸洛美沙星N/D0.7763.878N/DN/D依诺沙星倍半水合物N/D2.0842.084N/DN/D萘啶酸N/DN/DN/DN/DN/D林可霉素类林可霉素7.376N/DN/D7.3767.376林可酰胺类盐酸克林霉素0.73818.4583.6920.1480.738大环内酯类红霉素14.6790.5870.5872.9362.936螺旋霉素N/D33.72233.72233.72233.722万古霉素5.7971.1591.159N/DN/D多肽类硫酸卷曲霉素N/DN/DN/DN/DN/D乳酸链球菌素N/DN/DN/DN/DN/D盐酸大观霉素N/DN/DN/DN/DN/D硫酸西索米星N/DN/DN/DN/DN/D氨基糖苷类硫酸庆大霉素N/DN/DN/DN/DN/D硫酸卡那霉素N/DN/DN/DN/DN/D硫酸链霉素N/DN/DN/DN/DN/D利福霉素类利福平0.1320.0260.6583.2923.292氯霉素类氯霉素N/D1.0001.000N/DN/D呋喃西林N/D5.9445.944N/DN/D硝酸咪康唑N/DN/DN/DN/DN/D其他类型磺胺嘧啶N/DN/DN/DN/DN/D氯福克酚N/DN/DN/DN/DN/D特比萘芬N/DN/DN/DN/DN/D替硝唑N/DN/DN/DN/DN/D
注:N/D(not detected)表示未检测到抑菌率>90%的抗生素浓度。
1.1.3 培养基
改良LB培养基(g/L):胰蛋白胨10,酵母粉5,氯化钠60,121 ℃灭菌20 min。用于培养肉葡萄球菌、枯草芽孢杆菌和解淀粉芽孢杆菌。
改良MRS培养基(g/L):胰蛋白胨10,酵母粉5,葡萄糖20,氯化钠60,牛肉膏10,乙酸钠5,柠檬酸氢二铵2,磷酸氢二钾(三水)2.62,硫酸镁0.6,硫酸锰0.3,吐温-80 1 mL,115 ℃灭菌15 min。用于培养嗜盐四联球菌和融合魏斯氏菌。
改良YPD培养基(g/L):蛋白胨20,酵母粉10,葡萄糖20,氯化钠60,115 ℃灭菌15 min。用于培养鲁氏接合酵母。
配制固体培养基时,加入琼脂20 g/L。
1.2 仪器与设备
超净工作台,苏州净化设备有限公司;全自动高压蒸汽灭菌锅,日本HIRAYAMA株式会社;恒温恒湿培养箱,上海博迅实业有限公司;紫外可见分光光度计,UNICO(上海)仪器有限公司;Synergy H4微孔板读取器,美国BioTek Instruments股份有限公司;电子天平,METTLER TOLEDO公司;AnaeroPack-Anaero厌氧培养系统,日本三菱瓦斯化学株式会社。
1.3 实验方法
1.3.1 药敏试验
药敏试验的培养体系配方如下:
接种液(g/L):结冷胶0.2,聚醚F-68 0.3,氯化钠60。制备时,将0.2 g结冷胶加入1 L去离子水中,煮沸并持续搅拌,直至结冷胶完全溶解;停止加热,继续搅拌;加入0.3 g聚醚F-68与60 g氯化钠,搅拌至完全溶解。经121 ℃灭菌20 min后获得无菌接种液,用于后续制备菌悬液。该接种液配方参考张朝正等[12]。
菌悬液的制备参考BOCHNER等[13]提出的表型微阵列技术并稍作调整。从琼脂平板培养基的表面采集单个菌落,并将其悬浮在含有接种液的试管中,以获得在600 nm处吸光度为0.07的菌悬液。其中,琼脂平板培养基的配方参见1.1.3节。
培养液:参见1.1.3节中改良的YPD、LB和MRS培养基配方,并将(胰)蛋白胨、酵母粉、葡萄糖和牛肉膏的浓度增加一倍。
所有5株细菌的药敏试验在96深孔板中执行。每个孔含有120 μL菌悬液、60 μL培养液和20 μL抗生素溶液。总共使用42种对革兰氏阳性菌具有抗菌活性的抗生素,每种抗生素设置4个浓度梯度。接种了肉葡萄球菌、枯草芽孢杆菌和解淀粉芽孢杆菌的深孔板培养体系在37 ℃下以180 r/min孵育2 d。接种了嗜盐四联球菌和融合魏斯氏菌的深孔板培养体系在37 ℃下静置孵育4 d。通过Synergy H4微孔板读取器在600 nm处测量每个孔的吸光度,以分析42种抗生素对5种细菌的抑菌活性。
抑菌率的计算如公式(1)所示:
抑菌率![]()
(1)
对照菌液指未添加抗生素时所培养的菌液,试验菌液即添加了对应抗生素所培养的菌液,空白培养体系即未接种菌株的培养体系。
1.3.2 抗生素的筛选
根据药敏试验的结果筛选出5种具有选择性潜力的抗细菌抗生素,包括氨苄西林钠、萘夫西林钠、盐酸金霉素、恶喹酸和林可霉素。随后,测定以上5种抗生素和制霉菌素(一种抗真菌抗生素)在5个浓度梯度下对鲁氏接合酵母、肉葡萄球菌、枯草芽孢杆菌、解淀粉芽孢杆菌、嗜盐四联球菌和融合魏斯氏菌的抑菌活性,试验方法参见1.3.1节。其中,接种了鲁氏接合酵母的深孔板培养体系在28 ℃下以180 r/min孵育2 d。通过这种方法,初步确定了用于选择性培养每个菌株的潜在抗生素组合以及相应抗生素的浓度。
1.3.3 选择性培养基的设计与验证
将上述筛选出的6种抗生素进行组合搭配,并检验各抗生素组合的选择性。在倾倒平板之前,将具有选择性潜力的抗生素组合添加到改良YPD(用于选择鲁氏接合酵母)、改良LB(用于选择肉葡萄球菌、枯草芽孢杆菌或解淀粉芽孢杆菌)或改良MRS(用于选择嗜盐四联球菌或融合魏斯氏菌)琼脂培养基中,制成选择性平板。
为了优化每个组合中的抗生素浓度,将100 μL目标物种菌悬液(将OD600值=1的对应种子液用生理盐水稀释10倍制得)和100 μL包含6个物种的全群落混合菌悬液(将6个物种分别培养的OD600值=1的种子液各取1 mL并用生理盐水定容至10 mL制得)经梯度稀释后分别涂布在相应的选择性平板培养基上。涂布接种后的改良YPD平板在28 ℃下培养3 d,改良LB平板则在37 ℃下培养2 d。涂布接种后的改良MRS平板使用AnaeroPack-Anaero厌氧培养系统在37 ℃下厌氧培养4 d。读取并对比接种了目标菌株的选择性平板和接种了混合菌悬液(包含所有6株菌)的选择性平板上的菌落总数,从而判断选择性培养基是否能够选择性地从混菌体系中培养出目标菌株。实验进行4次重复。
1.3.4 合成微生物群落的培养与群落结构检测
为了制备模拟豆瓣酱发酵的快速发酵培养基,将发酵一个月的豆瓣酱样品与去离子水混合并煮沸5 min 后,用纱布过滤。收集滤液,并添加氯化钠,在115 ℃下灭菌15 min,制成豆瓣酱模拟培养基,最终含有200 g/L豆瓣酱的浸提物,以及100 g/L氯化钠。
将鲁氏接合酵母接种于改良YPD培养基中预培养18 h,将肉葡萄球菌、枯草芽孢杆菌和解淀粉芽孢杆菌分别接种于改良LB培养基中预培养18 h,将嗜盐四联球菌和融合魏斯氏菌分别接种于改良MRS培养基中预培养36 h。上述培养基均添加100 g/L氯化钠,培养条件为30 ℃、100 r/min。使用含有100 g/L氯化钠的无菌盐水洗涤细胞2次,制成种子液。
将对应的种子液按0.5%(体积分数,OD600值=1)的比例接种于100 mL豆瓣酱模拟培养基中,以容量为250 mL的锥形瓶盛装,并在瓶口安装组培封口膜(微孔滤膜孔径为30 mm,比克曼生物),于30 ℃、100 r/min条件下培养4 d。Z、S、Bs、Ba、T和W分别表示鲁氏接合酵母、肉葡萄球菌、枯草芽孢杆菌、解淀粉芽孢杆菌、嗜盐四联球菌和融合魏斯氏菌。本研究培养的物种组合包括:Z+S+Bs+Ba+T+W、S、Bs、Ba、S+Z、S+Bs、S+Ba、S+T、S+W、Bs+Z、Bs+Ba、Bs+T、Bs+W、Ba+Z、Ba+T、Ba+W。每个组合的培养实验均设置3个平行。
在培养2 d和4 d时,充分摇匀合成微生物群落培养物,并将1 mL培养物样品液转移到无菌管中待用。将连续稀释后的样品液涂布在对应的选择性平板培养基上,以检测在第2天和第4天时各物种的绝对丰度。物种丰度单位表示为CFU/mL。每个组合的培养物在每个时间点至少被计数4个菌落数在30~300的平板。
1.4 数据分析
数据统计分析和图形绘制采用Excel、Origin 2017和MATLAB R2023a软件。显著性分析由SPSS 25.0软件完成。
2 结果与分析
2.1 五株豆瓣酱源细菌的药敏试验
不同β-内酰胺类抗生素对5株豆瓣酱源细菌的抑菌率如图1所示。β-内酰胺类抗生素在低浓度下对5株豆瓣酱源细菌的抑菌率具有差异性,在高浓度下对多数菌株具有较强的抑菌效果(抑菌率>90%)。仅有氨苄西林、阿洛西林、普鲁卡因青霉素G、头孢美唑和头孢克洛在高浓度下对个别菌株的抑菌率仍未达到90%(表1)。当添加量为1.454 μg/mL时,萘夫西林钠对嗜盐四联球菌的抑菌率仅为13.5%,而对其余4株细菌的抑菌率均已超过90%,展现出了明显的抑菌能力差异(图1)。由此可见,通过添加适当浓度的萘夫西林钠有望制备针对该嗜盐四联球菌的选择性培养基,从而能抑制其余4株细菌的生长。

a-氨苄西林钠;b-萘夫西林钠;c-氯唑西林钠一水合物;d-头孢噻吩;e-阿洛西林钠;f-头孢呋辛钠;g-青霉素G普鲁卡因水合物;h-苯唑西林钠一水合物;i-头孢美唑钠;j-头孢克洛;k-卡茚西林钠;l-头孢唑林
图1 β-内酰胺类抗生素对5株豆瓣酱源细菌的抑菌率
Fig.1 Antibacterial rate of β-lactam antibiotics against five bacteria derived from broad bean paste
图2展示了四环素类、喹诺酮类、林可霉素类和林可酰胺类抗生素对5株豆瓣酱源细菌的抑菌率。枯草芽孢杆菌相较于其他菌株对四环素类抗生素的耐受性最差。0.103 μg/mL的盐酸金霉素对枯草芽孢杆菌抑菌率>90%,而0.515 μg/mL的盐酸金霉素对解淀粉芽孢杆菌的抑菌率仅为7.7%,表明适当浓度的盐酸金霉素能够在杀灭枯草芽孢杆菌的同时保证解淀粉芽孢杆菌的良好生长,具有制备选择性培养基的潜力。2株乳酸菌对四环素类和喹诺酮类抗生素的耐受性较强,仅有盐酸金霉素(2.577 μg/mL)能够对嗜盐四联球菌达到87.7%的抑菌率(图2)。

a-盐酸金霉素;b-盐酸米诺环素;c-盐酸四环素;d-恶喹酸;e-环丙沙星;f-氧氟沙星;g-左氧氟沙星;h-盐酸洛美沙星;i-依诺沙星倍半水合物;j-萘啶酸;k-林可霉素;l-盐酸克林霉素
图2 四环素类、喹诺酮类、林可霉素类和林可酰胺类抗生素对5株豆瓣酱源细菌的抑菌率
Fig.2 Antibacterial rate of tetracyclines, quinolones, lincomycins, and lincoamides antibiotics against five bacteria derived from broad bean paste
枯草芽孢杆菌和解淀粉芽孢杆菌相较于其他菌株对喹诺酮类抗生素的耐受性最差(图2)。肉葡萄球菌对喹诺酮类抗生素具有较强的耐受性,在本研究中没有检测到抑菌率大于90%的药物浓度(表1)。2.612 μg/mL的恶喹酸对2株芽孢杆菌的抑菌率均大于99%,而对肉葡萄球菌的抑菌率仅为0.9%,说明恶喹酸同样具有制备选择性培养基的潜力,即能够在肉葡萄球菌维持良好生长的同时杀灭枯草芽孢杆菌和解淀粉芽孢杆菌。
7.376 μg/mL的林可霉素对枯草芽孢杆菌和解淀粉芽孢杆菌的抑菌率均小于50%,而对其余菌株的抑菌率均大于90%(图2),说明以上2株芽孢杆菌对林可霉素具有较强的耐受性。因此,可以通过减小梯度稀释的倍数进一步分析林可霉素的有效抑菌浓度,从而获得能够有效抑制其余菌株却不影响芽孢杆菌生长的药物浓度。
大环内酯类、多肽类、氨基糖苷类、利福霉素类和氯霉素类抗生素对5株豆瓣酱源细菌的抑菌率如图3所示。
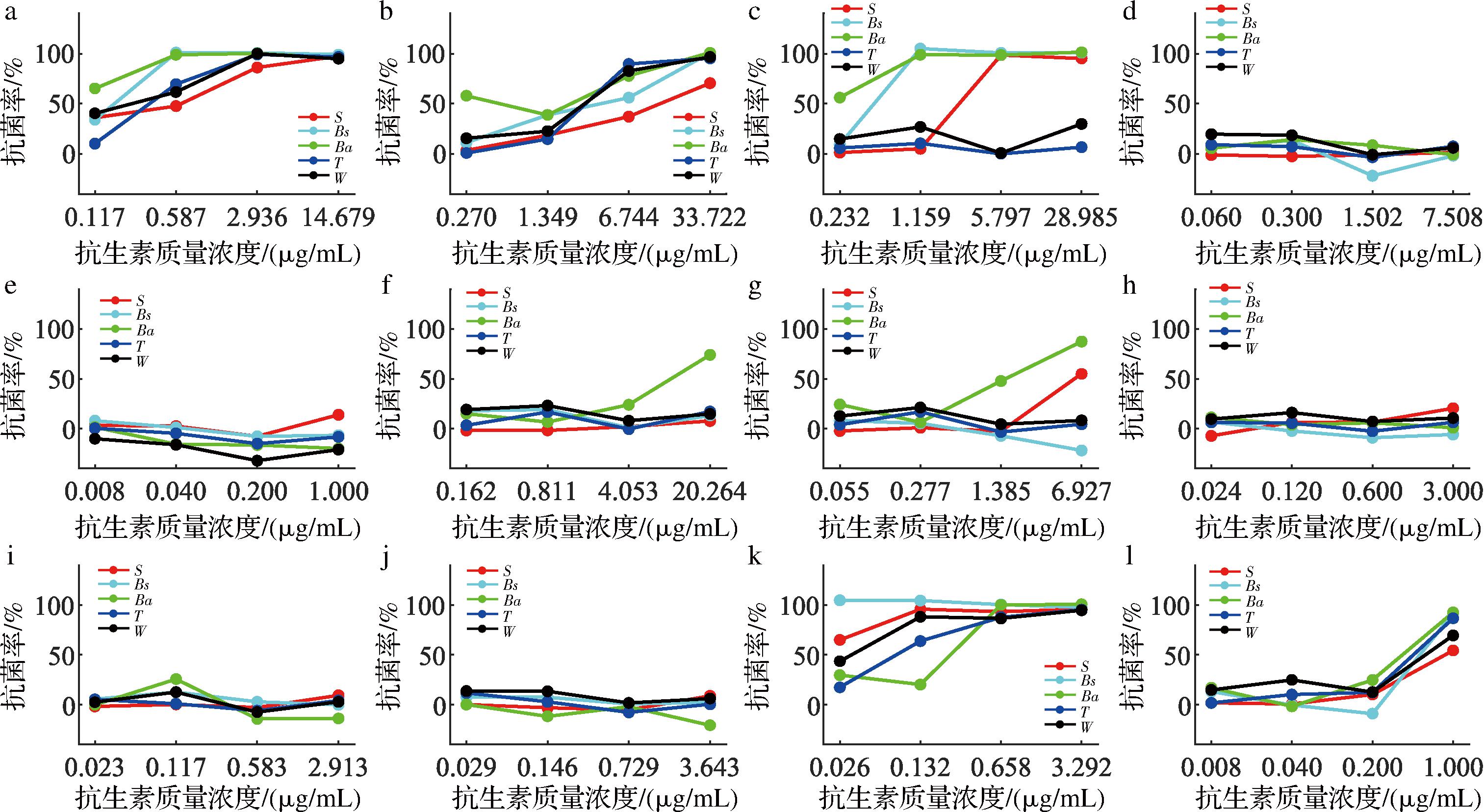
a-红霉素;b-螺旋霉素;c-万古霉素;d-硫酸卷曲霉素;e-乳酸链球菌素;f-盐酸大观霉素;g-硫酸西索米星;h-硫酸庆大霉素;i-硫酸卡那霉素;j-硫酸链霉素;k-利福平;l-氯霉素
图3 大环内酯类、多肽类、氨基糖苷类、利福霉素类和氯霉素类抗生素对5株豆瓣酱源细菌的抑菌率
Fig.3 Antibacterial rate of macrolides, polypeptide, rifamycins, and chloramphenicols antibiotics against five bacteria derived from broad bean paste
盐酸大观霉素、硫酸西索米星、硫酸庆大霉素、硫酸卡那霉素和硫酸链霉素的最大添加质量浓度分别为20.264、6.927、3.000、2.913、3.643 μg/mL,但并没有对任何一株菌的抑菌率达到90%(图3),说明该5株豆瓣酱源细菌对上述氨基糖苷类抗生素具有较强的耐受性。28.985 μg/mL万古霉素、7.508 μg/mL硫酸卷曲霉素和1.000 μg/mL乳酸链球菌素对嗜盐四联球菌和融合魏斯氏菌的抑菌率均小于50%(图3),说明以上2株乳酸菌对上述3种多肽类抗生素具有较强的耐受性。JEONG等[14]报道了大酱中分离出的嗜盐四联球菌对环丙沙星、链霉素、庆大霉素、红霉素的耐药率分别为100%、100%、11.54%、3.85%,与本研究的结果相似。
图4展示了其他类型抗生素对5株豆瓣酱源细菌的抑菌率。总体上,该5株豆瓣酱源细菌对呋喃西林、硝酸咪康唑、磺胺嘧啶、氯福克酚、特比萘芬和替硝唑的耐受性较强。其中,仅有呋喃西林(5.944 μg/mL)对枯草芽孢杆菌和解淀粉芽孢杆菌抑菌率>90%。
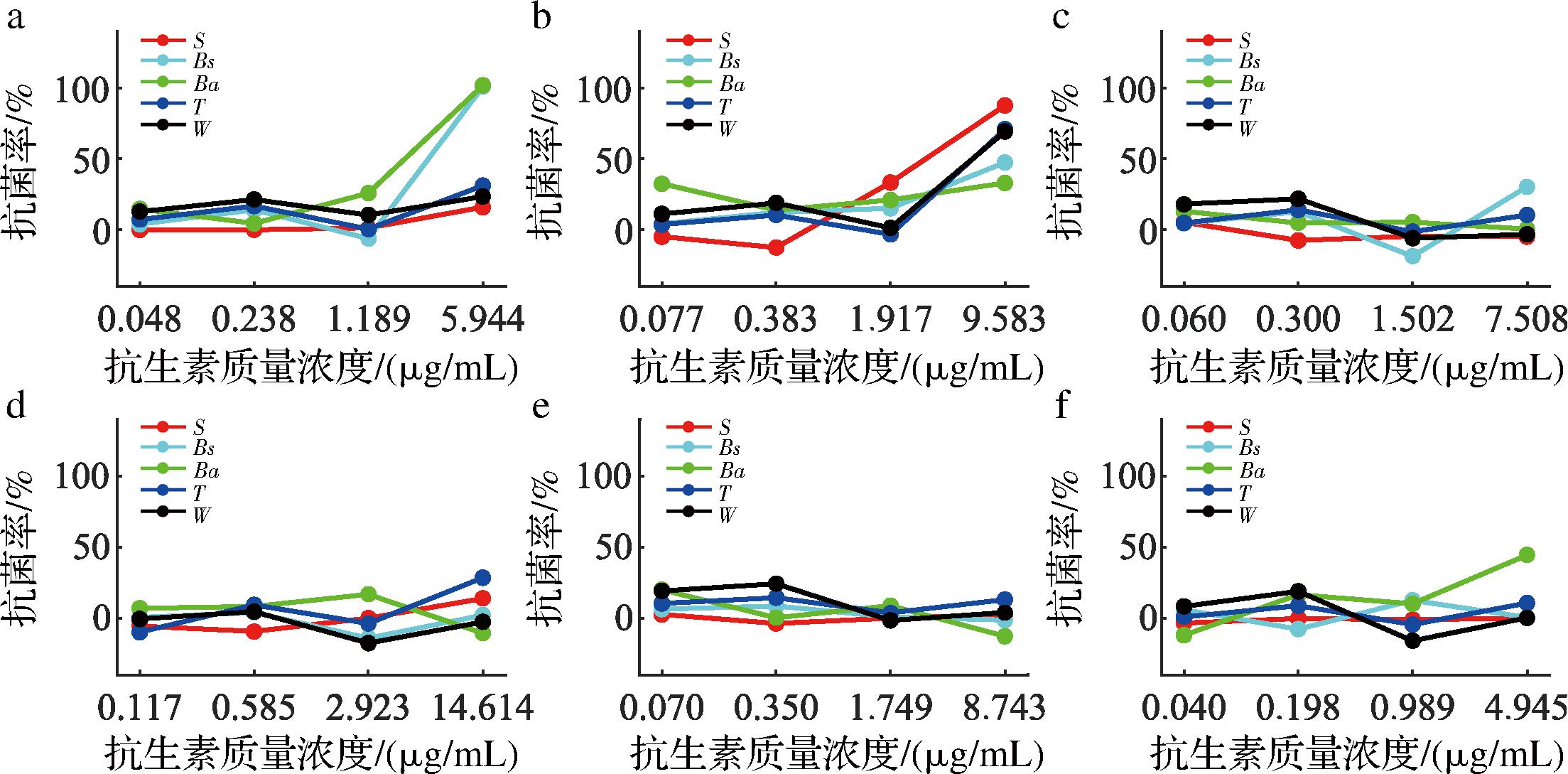
a-呋喃西林;b-硝酸咪康唑;c-磺胺嘧啶;d-氯福克酚;e-特比萘芬;f-替硝唑
图4 其他抗生素对5株豆瓣酱源细菌的抑菌率
Fig.4 Antibacterial rate of other antibiotics against five bacteria derived from broad bean paste
表1展示了5株豆瓣酱源细菌对不同抗生素的敏感性。需要注意的是,为了探究抗生素对各菌株抑菌活性的更大浓度范围,本研究并未采用标准的二倍稀释法测定最小抑菌浓度。因此,本研究的结果与标准的耐药性评估结果可能存在一定的差异。
2.2 选择性培养基的设计
2.2.1 选择性的抗生素组合的筛选
图5展示了鲁氏接合酵母、肉葡萄球菌、枯草芽孢杆菌、解淀粉芽孢杆菌、嗜盐四联球菌和融合魏斯氏菌分别在添加了氨苄西林钠、萘夫西林钠、盐酸金霉素、恶喹酸、林可霉素或制霉菌素的培养体系下的生长活性,每种抗生素设置了5个浓度梯度,每个梯度稀释4倍。根据实验结果,可以从中筛选出对目的菌株具有选择性的抗生素组合。例如,含有100 μg/mL氨苄西林钠的培养基,在含有多种微生物(包括鲁氏接合酵母、肉葡萄球菌、枯草芽孢杆菌、解淀粉芽孢杆菌、嗜盐四联球菌和融合魏斯氏菌)的环境中,将具有潜力实现鲁氏接合酵母的选择性培养;添加了1 μg/mL制霉菌素与0.422 μg/mL恶喹酸的培养基,将具有潜力实现肉葡萄球菌的选择性培养。
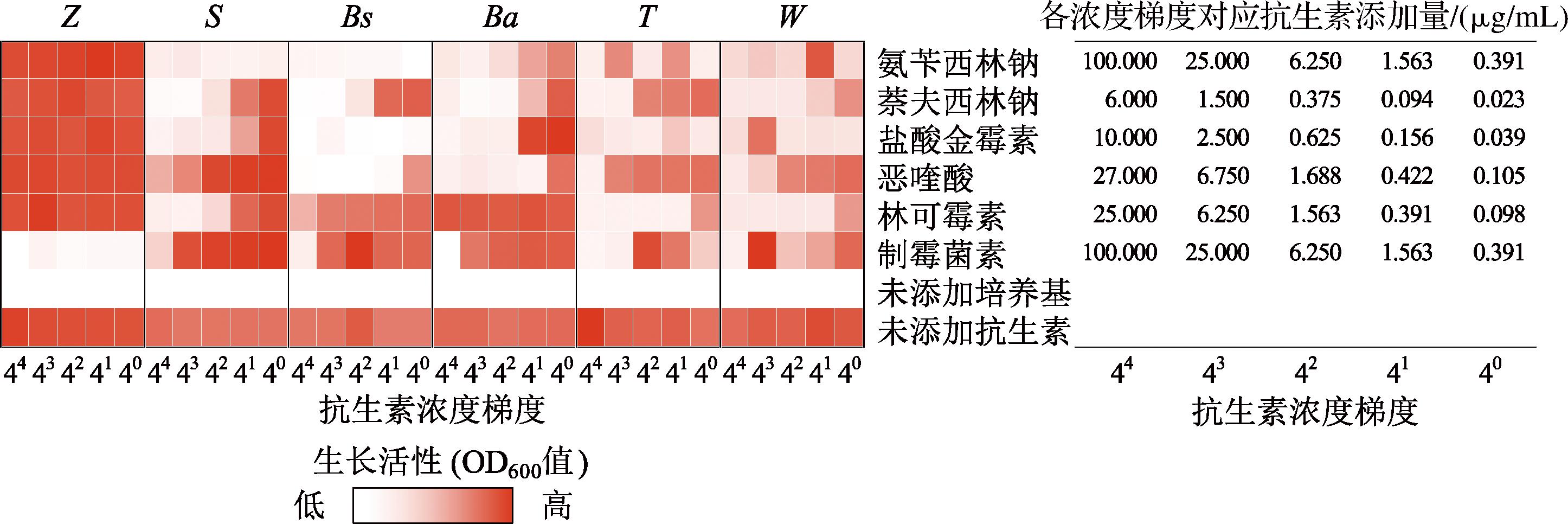
图5 六株豆瓣酱源微生物对不同抗生素的耐受性
Fig.5 Tolerance of six microorganisms derived from broad bean paste to different antibiotics
2.2.2 选择性培养基的设计与验证
基于上述方法,设计了6种抗生素组合(表2)以区分鲁氏接合酵母、肉葡萄球菌、枯草芽孢杆菌、解淀粉芽孢杆菌、嗜盐四联球菌和融合魏斯氏菌。图6展示了选择性培养基的选择性验证结果,其中,每个子图的左侧条形表示仅接种目标菌株的培养基上目标菌株的菌落总数,而右侧条形则代表在接种了混合菌悬液(包含6株菌)的培养基上目标菌株的菌落总数。从图6可以观察到,在接种了鲁氏接合酵母菌悬液和混合菌悬液的选择性培养基上,鲁氏接合酵母的数量分别为4.79×106和4.45×106 CFU/mL;在接种了肉葡萄球菌菌悬液和混合菌悬液的选择性培养基上,肉葡萄球菌的数量分别为4.20×108和4.13×108 CFU/mL;在接种了枯草芽孢杆菌菌悬液和混合菌悬液的选择性培养基上,枯草芽孢杆菌的数量分别为1.00×105和0.97×105 CFU/mL;在接种了解淀粉芽孢杆菌菌悬液和混合菌悬液的选择性培养基上,解淀粉芽孢杆菌的数量分别为6.33×105和6.00×105 CFU/mL;在接种了嗜盐四联球菌菌悬液和混合菌悬液的选择性培养基上,嗜盐四联球菌的数量分别为3.87×109和3.76×109 CFU/mL;在接种了融合魏斯氏菌菌悬液和混合菌悬液的选择性培养基上,融合魏斯氏菌的数量分别为7.17×108和7.23×108 CFU/mL。可以看出,在仅接种目标菌株与接种混合菌悬液的选择性平板上,目标菌株的菌落总数趋于相同,且差异不显著(P>0.05)。此外,由系列稀释涂布结果可知,在所有6组接种了混合菌悬液的选择性培养基上,均只有对应的目标菌株正常生长,其余菌株均未生长(即非目标菌株的菌落总数为0)。这表明所设计的选择性培养基对于目标菌株具有良好的选择性,从而确保了利用培养方法能精确测定由这6株菌构成的微生物群落中任一菌种的物种丰度。
表2 选择性培养基的抗生素组合
Table 2 Antibiotic combinations for selective media
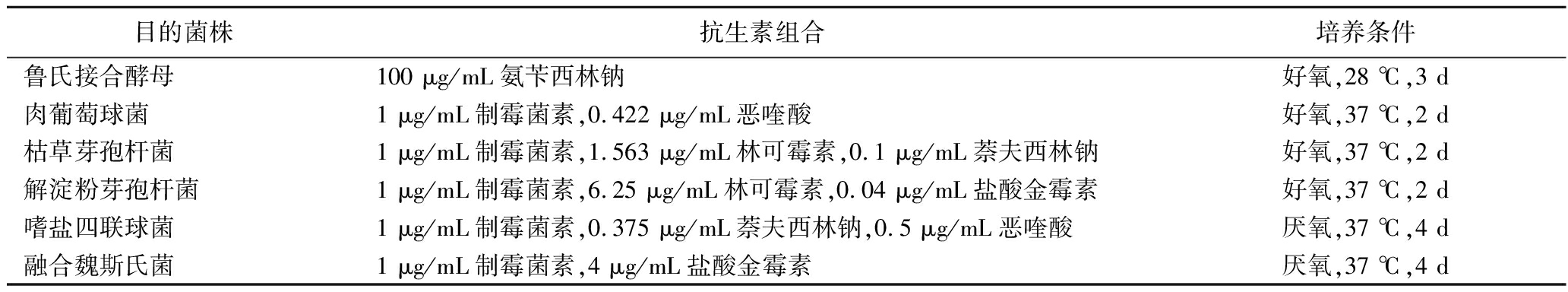
目的菌株抗生素组合培养条件鲁氏接合酵母100 μg/mL氨苄西林钠好氧,28 ℃,3 d肉葡萄球菌1 μg/mL制霉菌素,0.422 μg/mL恶喹酸好氧,37 ℃,2 d枯草芽孢杆菌1 μg/mL制霉菌素,1.563 μg/mL林可霉素,0.1 μg/mL萘夫西林钠好氧,37 ℃,2 d解淀粉芽孢杆菌1 μg/mL制霉菌素,6.25 μg/mL林可霉素,0.04 μg/mL盐酸金霉素好氧,37 ℃,2 d嗜盐四联球菌1 μg/mL制霉菌素,0.375 μg/mL萘夫西林钠,0.5 μg/mL恶喹酸厌氧,37 ℃,4 d融合魏斯氏菌1 μg/mL制霉菌素,4 μg/mL盐酸金霉素厌氧,37 ℃,4 d

A-鲁氏接合酵母菌落数;B-肉葡萄球菌菌落数;C-枯草芽孢杆菌菌落数;D-解淀粉芽孢杆菌菌落数
图6 选择性培养基对目标菌株的选择性
Fig.6 Selectivity of selective plate media for target strains
注:数据以平均值±SE表示;ns表示无显著性差异(P>0.05)(下同)。
随着生物技术的飞速进步,扩增子测序在探究微生物群落结构方面获得了广泛的应用[7-8]。然而,此类方法通常仅能提供每个物种的相对丰度数据。为了对合成微生物群落结构进行有效的定量分析,一种可行的方法是利用补充测量手段(如吸光度或qPCR)来估算绝对丰度,进而将相对丰度数据转化为绝对丰度数据。这种方法能够提供更为精确地定量数据,有助于深入分析合成微生物群落的组成和功能。例如,VENTURELLI等[15]采用了16S rRNA基因测序的相对丰度与总生物量(OD600值)的乘积来估算人类肠道合成微生物群落中每个物种的绝对丰度。他们利用这些估算的绝对丰度数据训练广义Lotka-Volterra模型,并成功地预测了微生物之间的相互作用。然而,在微生物组研究中,收集估计的绝对丰度数据往往面临较高的误差风险[16]。在合成微生物群落中,若群落成员数量较少,基于培养的检测方法似乎是更加行之有效的量化各物种绝对丰度的方式。MOUNIER等[9]通过使用含有不同选择性药剂组合的选择性培养基,精确描述了奶酪微生物群落的生长动态,并证明了酵母与细菌的相互作用对奶酪表面微生态系统建立的贡献。NIU等[10]采用表型微阵列技术,对玉米胚根合成微生物群落中的7株细菌在288种不同培养条件下的生长活性进行了评估。基于这7株细菌的化学敏感性和渗透压响应,他们成功构建了7种选择性培养基,从而能够高效地检测合成微生物群落的菌群结构,并准确量化各菌株的绝对丰度。研究表明,通过微量稀释法探究不同抗生素对各菌株的抑菌活性,能够有针对性地设计出适合各菌株的选择性培养基,进而构建一套基于培养技术的合成微生物群落菌群结构检测方法。该方法具有简便、准确、低成本等优点,能够量化合成微生物群落中任一菌株的绝对丰度。
2.3 选择性培养基的应用
图7-A显示了选择性培养基对豆瓣酱合成微生物群落结构的检测结果。在模拟体系中,合成微生物群落结构以鲁氏接合酵母和肉葡萄球菌为主,其次是嗜盐四联球菌和融合魏斯氏菌,而芽孢杆菌几乎没有生长(图7-A)。这与贾云等[17]报道的菌群结构相似,即在豆瓣酱发酵中后期,接合酵母属和葡萄球菌属分别成为优势酵母和优势细菌,而魏斯氏菌属和芽孢杆菌属则成为劣势细菌。图7-B~图7-D展示了选择性培养基对成对组合中目标物种绝对丰度的检测结果。肉葡萄球菌的生长受鲁氏接合酵母的促进,并且不受乳酸菌和芽孢杆菌属的影响(图7-B),这种成对相互作用关系解释了肉葡萄球菌在群落中表现出的竞争优势。这与ZHAO等[11]的研究相似,他们认为肉葡萄球菌与鲁氏接合酵母之间存在积极的相互作用,有利于豆瓣酱的成熟以及风味物质的产生。然而,芽孢杆菌属的生长受到鲁氏接合酵母、肉葡萄球菌、嗜盐四联球菌和融合魏斯氏菌不同程度的抑制,其中鲁氏接合酵母和肉葡萄球菌在第2天就将2株芽孢杆菌的丰度下降至0 CFU/mL(图7-C和图7-D)。由上述成对相互作用关系可知,芽孢杆菌属的生长受到其他物种的显著抑制,导致芽孢杆菌属在群落中表现出竞争劣势。上述结果表明,应用选择性培养基不仅能检测合成微生物群落的结构组成,同时也有助于分析群落中各物种间的相互作用关系。由此可见,选择性培养基在分析合成微生物群落结构方面展现出良好的应用前景。
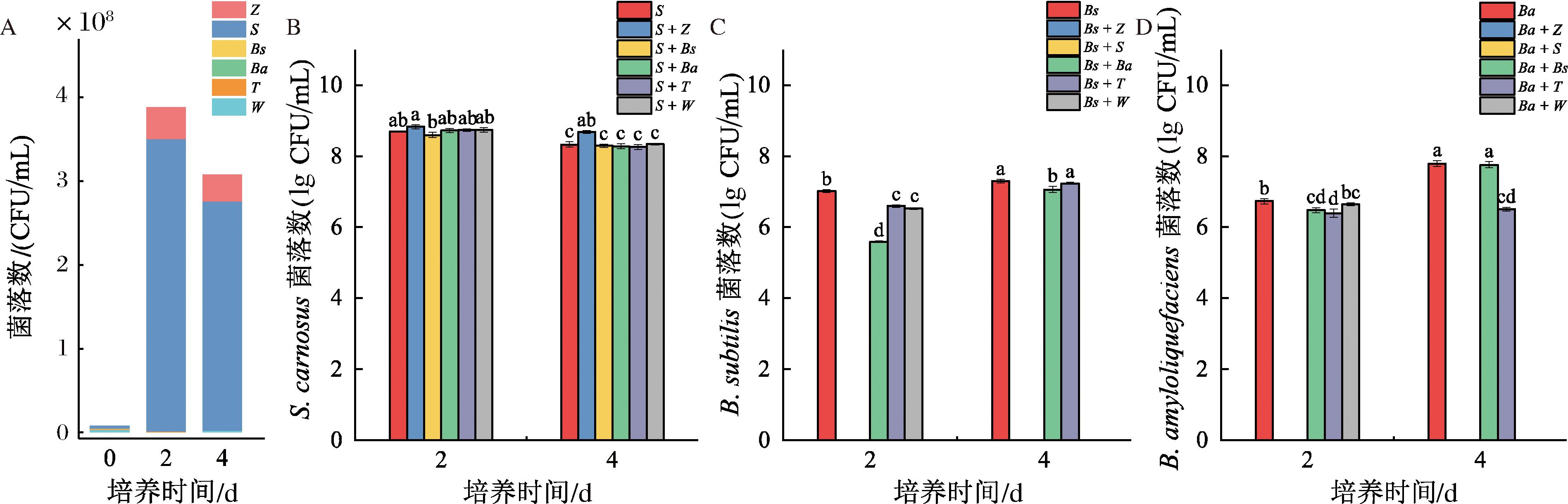
A-鲁氏接合酵母;B-肉葡萄球菌;C-枯草芽孢杆菌;D-解淀粉芽孢杆菌;E-嗜盐四联球菌;F-融合魏斯氏菌
图7 应用选择性培养基检测物种丰度
Fig.7 Application of selective plate media for detecting species abundance
注:不同小写字母代表差异显著(P<0.05)。
3 结论
药敏试验结果表明,通过分析42种抗生素对肉葡萄球菌、枯草芽孢杆菌、解淀粉芽孢杆菌、嗜盐四联球菌和融合魏斯氏菌的抑菌活性,能够有效筛选出能将这些特定细菌区分开的抗生素。通过组合搭配不同浓度的氨苄西林钠、萘夫西林钠、盐酸金霉素、恶喹酸、林可霉素和制霉菌素,设计了一系列选择性培养基,从而构建了一种基于培养技术的菌群结构检测方法。实验结果证明了该方法能够精确量化由鲁氏接合酵母、肉葡萄球菌、枯草芽孢杆菌、解淀粉芽孢杆菌、嗜盐四联球菌和融合魏斯氏菌组成的合成微生物群落中任一菌株的绝对丰度。通过应用该方法,简便、准确、低成本地分析了豆瓣酱合成微生物群落的结构以及群落成员间的相互作用。本研究为成员数量较少的合成微生物群落菌群结构检测方法的建立提供了一种解决方案。
[1] JIA Y, NIU C T, LU Z M, et al.A bottom-up approach to develop a synthetic microbial community model:Application for efficient reduced-salt broad bean paste fermentation[J].Applied and Environmental Microbiology, 2020, 86(12):e00306-20.
[2] 郑鹏飞, 张丽杰, 王栋, 等.一种自下而上的合成微生物组理性构建策略, 用于郫县豆瓣发酵剂设计[J].微生物学报, 2022, 62(10):3913-3931.ZHENG P F, ZHANG L J, WANG D, et al.A bottom-up strategy for constructing a synthetic microbiome and its application to the design of Pixian Douban starter[J].Acta Microbiologica Sinica, 2022, 62(10):3913-3931.
[3] 邱婷. 产乙偶姻微生物群落的合成及其在传统食醋酿造过程的应用[D].无锡:江南大学, 2019.QIU T.Synthesis of acetoin-producing microbial community and its application in traditional vinegar brewing process[D].Wuxi:Jiangnan University, 2019.
[4] BERNSTEIN H C, CARLSON R P.Microbial consortia engineering for cellular factories:In vitro to in silico systems[J].Computational and Structural Biotechnology Journal, 2012, 3(4):e201210017.
[5] 张小龙, 王嘉瑞, 李青卓, 等.合成微生物群落及在发酵食品中应用研究进展[J].中国酿造, 2021, 40(3):17-21.ZHANG X L, WANG J R, LI Q Z, et al.Research progress of synthetic microbial community and its application in fermented food[J].China Brewing, 2021, 40(3):17-21.
[6] WEISS A S, BURRICHTER A G, DURAI RAJ A C, et al.In vitro interaction network of a synthetic gut bacterial community[J].The ISME Journal, 2022, 16(4):1095-1109.
[7] SHETTY S A, KOSTOPOULOS I, GEERLINGS S Y, et al.Dynamic metabolic interactions and trophic roles of human gut microbes identified using a minimal microbiome exhibiting ecological properties[J].The ISME Journal, 2022, 16(9):2144-2159.
[8] LI Z F, BAI X L, JIAO S, et al.A simplified synthetic community rescues Astragalus mongholicus from root rot disease by activating plant-induced systemic resistance[J].Microbiome, 2021, 9(1):217.
[9] MOUNIER J, MONNET C, VALLAEYS T, et al.Microbial interactions within a cheese microbial community[J].Applied and Environmental Microbiology, 2008, 74(1):172-181.
[10] NIU B, PAULSON J N, ZHENG X Q, et al.Simplified and representative bacterial community of maize roots[J].Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(12):E2450-E2459.
[11] ZHAO S, NIU C T, WANG Y H, et al.The facilitation between Staphylococcus carnosus M43 and Zygosaccharomyces rouxii Y-8, and as starter on the quality of broad bean paste[J].Food Bioscience, 2023, 55:103019.
[12] 张朝正, 郭兰珍, 黎明, 等.Biolog微生物鉴定系统中接种液的替代[J].生物技术通讯, 2009, 20(6):836-838.ZHANG C Z, GUO L Z, LI M, et al.Substitution of inoculating fluid of biolog microstation system[J].Letters in Biotechnology, 2009, 20(6):836-838.
[13] BOCHNER B R, GADZINSKI P, PANOMITROS E.Phenotype microarrays for high-throughput phenotypic testing and assay of gene function[J].Genome Research, 2001, 11(7):1246-1255.
[14] JEONG D W, HEO S, LEE J H.Safety assessment of Tetragenococcus halophilus isolates from doenjang, a Korean high-salt-fermented soybean paste[J].Food Microbiology, 2017, 62:92-98.
[15] VENTURELLI O S, CARR A C, FISHER G, et al.Deciphering microbial interactions in synthetic human gut microbiome communities[J].Molecular Systems Biology, 2018, 14(6):e8157.
[16] REMIEN C H, ECKWRIGHT M J, RIDENHOUR B J.Structural identifiability of the generalized Lotka-Volterra model for microbiome studies[J].Royal Society Open Science, 2021, 8(7):201378.
[17] 贾云, 钮成拓, 郑飞云, 等.传统豆瓣酱微生物群落发酵演替规律及其功能分析[J].微生物学报, 2021, 61(9):2749-2764.JIA Y, NIU C T, ZHENG F Y, et al.Succession and function analysis of microbial community during traditional broad bean paste fermentation[J].Acta Microbiologica Sinica, 2021, 61(9):2749-2764.