硫酸软骨素(chondroitin sulfate, CS)是一种广泛存在于动物组织细胞外基质和细胞表面的糖胺聚糖(glycosaminoglycan, GAG)[1]。其糖链由D-葡萄糖醛酸(D-glucuronic acid,GlcA)和N-乙酰-D-半乳糖胺(N-acetyl-β-D-galactosamine,GalNAc)组成的重复二糖单元通过β1→3或β1→4糖苷键连接,并在GalNAc残基上带有不同数量和位置的O-硫酸基团[2]。CS具有多种重要生物学功能,如维持关节软骨的弹性和润滑性、减轻关节炎症[3]以及促进软骨修复[4],在医药、美容和健康领域有广泛应用[5]。
目前,CS的主要生产方法是通过从动物组织中提取获得[6],但这种方法存在产量有限、原料供应不稳定及潜在的安全隐患等问题。因此,研究人员开发了酶催化法和微生物直接发酵法。酶催化法利用磺基转移酶催化软骨素或果糖软骨素合成CS[7]。微生物直接发酵法则通过微生物细胞工厂直接合成CS[8-9],但该方法仍面临产率低、磺酸化程度低且不可控等问题,因此目前主要期望通过酶催化法实现CS更安全、绿色的生物合成。
软骨素和果糖软骨素与CS具备相似的碳骨架结构,是CS酶法合成必需的前体物质[10]。为了提高这些前体物质的合成效率,研究人员在大肠杆菌K4[11-12]、大肠杆菌BL21[13]、枯草芽孢杆菌[14]、谷氨酸棒杆菌[15]和毕赤酵母[8]等菌株中构建了软骨素合成途径,强化前体尿苷二磷酸(uridine diphosphate,UDP)-GlcA和UDP-GalNAc的合成,并阻断竞争性代谢途径,促进软骨素的合成。然而,无论是大肠杆菌BL21、枯草芽孢杆菌、谷氨酸棒杆菌还是毕赤酵母,这些菌株因自身缺乏软骨素转运系统,无法实现软骨素的高效转运,从而限制了软骨素合成效率,并导致提取难度增加[16]。相比之下,大肠杆菌K4天然具备果糖软骨素生物合成途径及其转运系统(图1),具有高产果糖软骨素的潜力[17]。然而,在重组大肠杆菌K4发酵过程中,常需要添加抗生素维持质粒稳定[18]。抗生素抗性基因通过水平基因转移在不同细菌间传播,细菌因此对抗生素产生耐药性[19],耐药性在环境中迅速扩散[20],对生态系统的平衡易构成威胁。因此,开发大肠杆菌K4不依赖抗生素的质粒表达系统对果糖软骨素的生物合成至关重要。
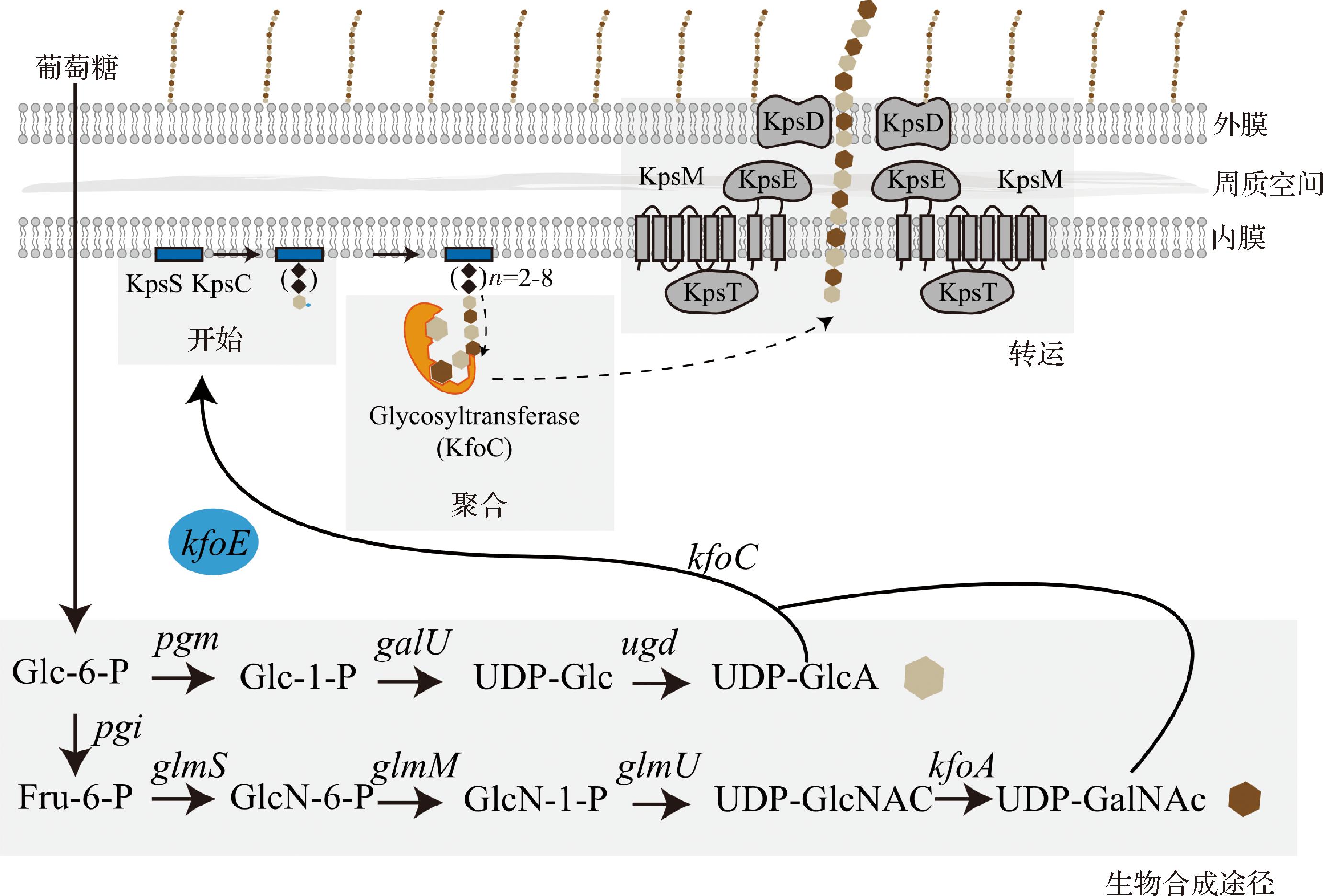
图1 果糖软骨素生物合成与转运示意图
Fig.1 Schematic illustration of fructosylated chondroitin biosynthesis and transport pathways
注:KpsS和KpsC为3-脱氧-D-甘露辛酮糖酸(Kdo)转移酶;KfoC为软骨素合酶;KpsT为ABC转运蛋白中的ATP转运亚基;KpsE为ABC转运蛋白的周质亚基;KpsM为ABC转运蛋白的跨膜蛋白;KpsD为外膜上的转运蛋白;kfoE为果糖基转移酶基因;kfoC为软骨素合酶基因;pgm为磷酸葡萄糖变位酶基因;galU为葡萄糖-1-磷酸尿酰胺转移酶基因;ugd为UDP-葡萄糖脱氢酶基因;pgi为葡萄糖-6-磷酸异构酶基因;glmS为氨基转移酶基因;glmM为磷酸葡萄糖胺变位酶基因;glmU为UDP-N-乙酰氨基葡萄糖焦磷酸化酶基因;kfoA为UDP-N-乙酰氨基葡萄糖-4-差向异构酶基因;Glc-6-P为葡萄糖-6-磷酸;Glc-1-P为葡萄糖-1-磷酸;UDP-Glc为UDP-葡萄糖;UDP-GlcA为UDP-葡萄糖醛酸;Fru-6-P为果糖-6-磷酸;GlcN-6-P为葡萄糖胺-6-磷酸;GlcN-1-P为葡萄糖胺-1-磷酸;UDP-GlcNAC为UDP-乙酰氨基葡萄糖;UDP-GalNAc为UDP-乙酰半乳糖胺葡萄糖(下同)。
本文首先基于大肠杆菌Nissle 1917(EcN)的内源性质粒pMUT2[21]开发了一套大肠杆菌K4的无抗生素表达系统,获得质粒pMUT2 ΔHyp3,该质粒在无抗生素压力下可维持9 d的遗传稳定性。在此基础上,表达了来自5个不同菌种的果糖软骨素合成途径同工酶,并对其进行了比较研究和优化,以促进前体物质UDP-GlcA和UDP-GlcNAc的合成,最终发现表达谷氨酸棒状杆菌来源的UDP-葡萄糖脱氢酶编码基因ugdA2与枯草芽孢杆菌来源的UDP-N-乙酰氨基葡萄糖焦磷酸化酶编码基因glmU后,在不添加抗生素的情况下,果糖软骨素摇瓶产量与大肠杆菌K4野生型相比,由0.1 g/L提高至0.95 g/L。最终,改良培养基组分后在3 L发酵罐中通过分批补料发酵生产了7.5 g/L果糖软骨素,大大提高了果糖软骨素的合成效率,为CS的酶法高效合成奠定了基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
大肠杆菌K4和大肠杆菌Top10为本实验室保藏;实验中所用质粒如附表1(https://doi.org/10.13995/j.cnki.11-1802/ts.042436,下同)所示,实验中所用菌株如附表2所示。
1.1.2 引物
实验中所用引物如附表3所示。
1.1.3 试剂
琼脂糖凝胶DNA回收试剂盒、卡那霉素、异丙基-β-D-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG),上海生工股份有限公司;DNA连接酶、Taq DNA聚合酶,Takara公司;酵母提取物、胰蛋白胨,OXOID公司;除特别说明外其余试剂均购于上海国药试剂公司。
1.1.4 培养基
LB培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10(固体培养基在此基础上加琼脂粉15~20)。
发酵培养基(g/L):KH2PO4 13.5,(NH4)2HPO4 4,柠檬酸1.7,葡萄糖20,NaOH 4,维生素B1 0.1,微量元素10。
发酵培养基-酵母(g/L):KH2PO4 13.5,(NH4)2HPO4 4,柠檬酸1.7,酵母提取物6,葡萄糖20,NaOH 4,维生素B1 0.1,微量元素10。
发酵培养基-蛋白胨(g/L):KH2PO4 13.5,(NH4)2HPO4 4,柠檬酸1.7,胰蛋白胨4,葡萄糖20,NaOH 4,维生素B1 0.1,微量元素10。
发酵培养基-双组份(g/L):KH2PO4 13.5,(NH4)2HPO4 4,柠檬酸1.7,酵母提取物6,胰蛋白胨4,葡萄糖20,NaOH 4,维生素B1 0.1,微量元素10。
LB-营养肉汤培养基(g/L):胰蛋白胨10,酵母提取物5,NaCl 10,营养肉汤20。
M9培养基(g/L):葡萄糖2,MgSO4 0.12,CaCl20.011,盐酸硫胺素0.337,Na2HPO4 3,KH2PO4 3,NaCl 0.5,NH4Cl 1。
甘油培养基(g/L):甘油20,硫胺素10,KH2PO4 13.5,(NH4)2HPO4 4,MgSO4·7H2O 0.12,柠檬酸1.7,微量元素10。
微量元素(g/L):FeSO4·7H2O 10、MnSO4·4H2O 0.5、ZnSO4·7H2O 2.2、CuSO4·5H2O 1、CaCl2 2、Na2B4O7·10H2O 0.02、(NH4)6Mo7O24·4H2O 0.1。
1.2 仪器与设备
DYY-60D电泳仪,北京六一电泳仪厂;GelDocXR凝胶成像系统、T100thermalcycler PCR仪、MicroPulser电穿孔仪,美国Bio-Rad公司;5424R高速离心机,德国Eppendorf公司;AllegraX-15R台式冷冻离心机,美国Beckman公司;905-ULTS超低温冰箱,美国ThermoFisherScientific公司;DKB-600A电热恒温水槽,上海森信实验仪器有限公司;HDPN-Y-50恒温培养箱,上海恒跃医疗器械有限公司;ZQWY-200震荡培养箱,上海知楚仪器有限公司;TB-214分析天平,DENVER公司(美国);UV2450紫外可见分光光度计,日本岛津公司;Infinite200PRO酶标仪,瑞士帝肯(Tecan);SCIENTZ-Nano高压均质机,宁波新芝生物科技股份有限公司;T&J-Atype台式玻璃生物反应器,迪比尔生物工程有限公司。
1.3 实验方法
1.3.1 质粒的构建
从NCBI网站(https://www.ncbi.nlm.nih.gov/)查找质粒载体pMUT1和pMUT2核苷酸序列信息,同时查找软骨素合成途径中所涉及途径酶的氨基酸序列与核苷酸序列。设计引物CH-ecgalU-F:GTATAAGAAGGAGATATACATATGGCTGCCATTAATACGA-AAGTC和CH-ecgalU-R:ACCAGACTCGAGTTCTTAATGCCCATCTCTTCTTC,以大肠杆菌MG1655基因组作为模板,扩增出含ecgalU的线性化片段;设计引物ZAI-F:CTCGAGTCTGGTTAATTAACCTAGGCTGCTGC和ZAI-R:TATGTATATCTCCTTCTTATACTTAACTAATATACT,以质粒pMUT2 ΔHyp3作为模板,扩增出线性化载体;利用一步克隆酶连接线性化载体和片段,其余质粒构建与上述步骤相同,之后对构建好的质粒进行测序确认。
1.3.2 培养条件
种子培养:平板划线培养,37 ℃,培养24 h,刮取菌苔接种至摇菌管,37 ℃,200 r/min过夜培养。
摇瓶发酵培养:取一定量的种子液接种至装有25 mL培养基的250 mL摇瓶,控制初始细胞密度(OD600)为0.1,37 ℃,200 r/min,培养24 h。
3 L发酵罐培养:种子在250 mL摇瓶中培养10 h,以10%的接种量接种至3 L发酵罐中,37 ℃,300 r/min,通气量为3 vvm的条件培养,通过补充14%(体积分数)的氨水控制pH值为7.0左右,流加75%(质量分数)的葡萄糖,葡萄糖含量维持在10 g/L左右。
1.3.3 质粒稳定性测试
将种子液分别接入含有特定抗生素和无抗生素的LB培养基内,每隔12 h进行1次传代培养,并进行一次取样,利用酶标仪对样本进行检测,记录490~530 nm的绿色荧光信号强度,同时测定OD600值。质粒稳定性的计算方式为:无抗生素条件下单位OD600的荧光蛋白信号强度与含抗生素条件下单位OD600的荧光蛋白信号强度之比[21]。
1.3.4 果糖软骨素检测方法
样品纯化:取发酵液,高压均质破碎细胞,离心取上清液。加4倍乙醇,-30 ℃醇沉12 h。离心取沉淀,水溶至原体积,再次离心。重复纯化,得果糖软骨素溶液。
定量分析:采用Bitte-Muir法。配制硼砂-硫酸溶液(4.77 g四硼酸钠/500 mL浓硫酸)和咔唑溶液(1.25 g咔唑/500 mL乙醇)。制备0~50 mg/L葡萄糖醛酸标准溶液(10 mg/L梯度),取200 μL标准液与1 mL硼砂-硫酸溶液高温反应15 min,冷却至室温,加入50 μL咔唑溶液显色20 min后于530 nm测吸光度,绘制标准曲线。发酵样品稀释后同法测定,计算含量[2]。
2 结果与分析
2.1 无抗生素质粒表达系统的构建
质粒由于其高转化率,高表达量等特点,常被用于改造高效表达目的基因以合成目标产物[22]。本研究为在大肠杆菌K4中构建无抗生素质粒表达系统,选择EcN中的2个隐性质粒作为改造对象。EcN中的2个隐性质粒已被改造用作自身不依赖抗生素质粒表达载体。研究表明,隐性质粒pMUT1和pMUT2可以在EcN中稳定存在6.5 d和8.5 d。这2个质粒的稳定性依赖于其携带的毒素-抗毒素系统,当破坏了毒素与抗毒素的平衡后,质粒的稳定性会急剧下降[21]。
为了方便质粒载体的构建与稳定性验证,本研究在质粒上引入绿色荧光蛋白基因(gfp)与抗生素抗性基因作为筛选标记;并通过连续传代实验测定pMUT1和pMUT2质粒在大肠杆菌K4中不依赖抗生素筛选的稳定性,结果表明,质粒pMUT1在第2天便大幅丢失,质粒pMUT2在第7天才开始丢失(图2-a),这一结果表明pMUT1缺乏在大肠杆菌K4中的稳定维持能力,而pMUT2具备稳定的遗传特性,适合作为无抗生素表达载体。为了进一步提高质粒载体的稳定性并缩小质粒表达载体,本研究对质粒pMUT2上的各个元件进行敲除,包括未知蛋白Hyp3,参与质粒接合的解旋酶MobC、移动蛋白MobB与MobD以及毒素蛋白RelE,获得质粒pMUT2 ΔHyp3、pMUT2 ΔMobC、pMUT2 ΔMobB、pMUT2 ΔMobD和pMUT2 ΔRelE。对上述质粒进行稳定性测试发现,pMUT2 ΔHyp3第9天开始丢失(图2-b),pMUT2 ΔMobC、pMUT2 ΔMobB和pMUT2 ΔMobD这3个质粒第8天开始丢失(图2-c~图2-e),而pMUT2 ΔRelE这一质粒第2天便大幅度丢失(图2-f)。这一结果说明了pMUT2质粒在大肠杆菌K4中的稳定性依赖于RelE-RelB毒素抗毒素元件,当敲除RelE毒素元件后,质粒稳定性也急剧下降。未知蛋白Hyp3功能尚不明确,但敲除该元件后,质粒稳定性由7 d提高至9 d,因此,本研究后续选择稳定性更高的pMUT2 ΔHyp3作为质粒表达载体。

a-EcN来源的隐性质粒pMUT1和pMUT2在大肠杆菌K4中的稳定性;b-敲除Hyp3元件后的质粒pMUT2 ΔHyp3在大肠杆菌K4中的稳定性;c-敲除MobC元件后的质粒pMUT2 ΔMobC在大肠杆菌K4中的稳定性;d-敲除MobB元件后的质粒pMUT2 ΔMobB在大肠杆菌K4中的稳定性;e-敲除MobD元件后的质粒pMUT2 ΔMobD在大肠杆菌K4中的稳定性;f-敲除RelE元件后的质粒pMUT2 ΔRelE在大肠杆菌K4中的稳定性
图2 质粒在大肠杆菌K4中的稳定性
Fig.2 Stability of the plasmid in E.coli K4
为得到无抗性基因质粒表达载体,本研究将质粒pMUT2 ΔHyp3中的抗性基因利用FLP重组酶靶位点/FLP重组酶(FRT/FLP)系统进行删除[23],首先在质粒抗性基因前后添加FRT位点,并利用温敏型质粒pCP20表达FLP重组酶,将2个质粒转入大肠杆菌K4后,利用FLP重组酶引导FRT位点进行重组,促进抗性基因片段的缺失(图3-a),温敏型质粒pCP20可通过将菌株于40 ℃过夜培养消除。抗性基因的删除结果如图3-b所示,将携带该质粒的菌株在LB平板上划线验证,发现无抗生素存在的情况下,菌株可以正常生长,在添加卡那霉素的LB平板上,菌株无法生长,结果表明抗性基因被成功删除(图3-b)。
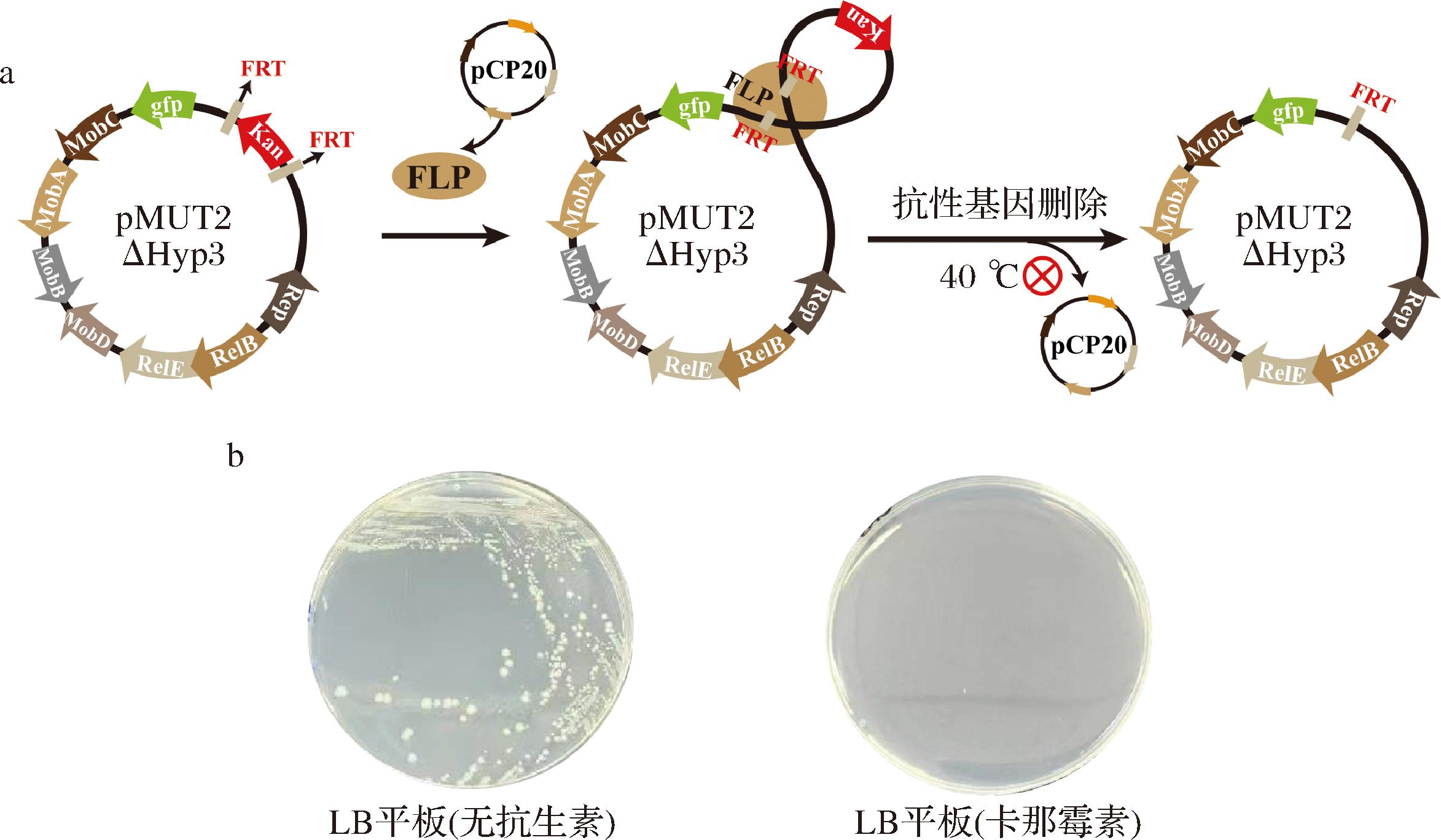
a-流程示意图;b-LB平板划线验证
图3 质粒抗性基因的删除
Fig.3 Deletion of the antibiotic resistance gene on the plasmid
注:基于FRT/FLP系统删除质粒抗性基因,FLP重组酶促进FRT位点重组,从而获得抗性基因缺失的质粒;无抗生素平板上菌株可正常生长,添加卡那霉素的平板上菌株无法生长。
2.2 优化前体UDP-GlcA和UDP-GalNAc的供应提高果糖软骨素的产量
大肠杆菌K4其自身具备果糖软骨素合成途径与转运系统[24](图1)。对大肠杆菌K4野生型摇瓶培养后测定其胞外果糖软骨素的含量,结果显示果糖软骨素产量约为0.10 g/L(图4-b)。为建立高效果糖软骨素生物合成细胞工厂,首先研究了前体UDP-GlcA的生物合成途径[25]。葡萄糖-1-磷酸尿酰胺转移酶(glucose-1-phosphate uridylyltransferase,GalU)和UDP-葡萄糖脱氢酶(UDP-glucose dehydrogenase,Ugd)是该途径上2个重要途径酶。为提高UDP-GlcA的表达通量,选取了来自谷氨酸棒杆菌、枯草芽孢杆菌、兽疫链球菌、恶臭假单胞菌、大肠杆菌5种不同来源的葡萄糖-1-磷酸尿酰胺转移酶基因galU将其构建在载体pMUT2 ΔHyp3上进行比较分析(图4-a),发现无抗生素条件下,过表达兽疫链球菌来源的galU1基因效果最佳,产量可达到0.31 g/L,较大肠杆菌K4野生型产量提高了210%(图4-b)。同样的,对不同来源的UDP-葡萄糖脱氢酶基因ugd进行表达验证,果糖软骨素产量如图4-c所示,过表达谷氨酸棒杆菌来源的ugdA2基因,果糖软骨素产量提升最为显著,达0.57 g/L,较大肠杆菌K4野生型产量提升了470%。该结果表明Ugd催化的UDP-Glc到UDP-GlcA途径是前体UDP-GlcA合成途径中的关键限速步骤,过表达谷氨酸棒杆菌来源基因ugdA对提高果糖软骨素产量的效果最好。
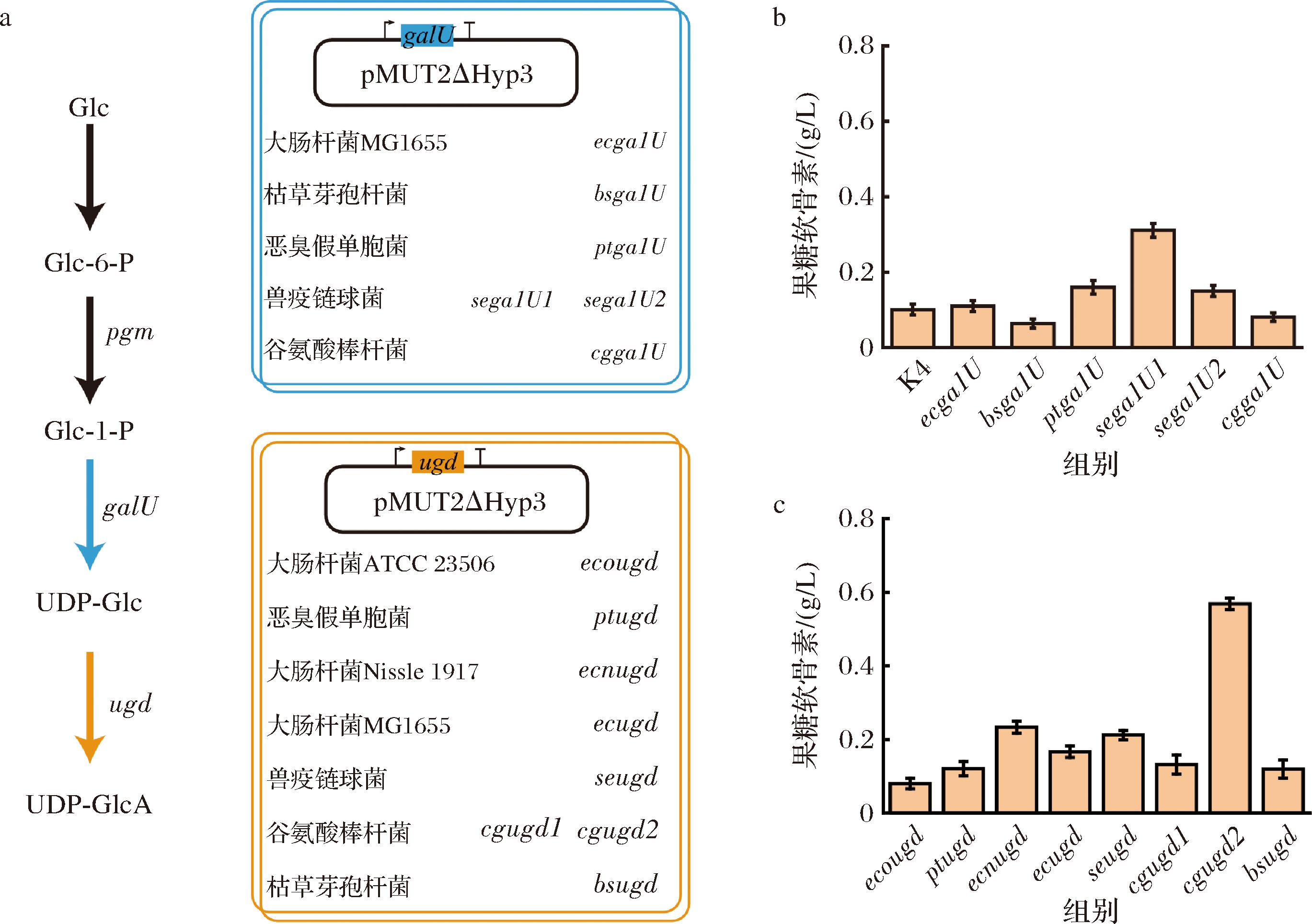
a-UDP-GlcA合成途径示意图及不同来源的途径基因;b-不同来源galU基因对果糖软骨素产量的影响;c-不同来源ugd基因对果糖软骨素产量的影响
图4 果糖软骨素前体UDP-GlcA合成途径强化
Fig.4 Enhancement of the UDP-GlcA biosynthetic pathway for fructochondroitin precursor production
注:K4为大肠杆菌K4野生型的果糖软骨素产量。
参与果糖软骨素合成的另一个前体为UDP-GalNAc,其合成途径从果糖-6-磷酸开始,涉及glmS基因编码的葡萄糖胺-6-磷酸合成酶(glucosamine-6-phosphate synthase,GlmS)、glmM基因编码的磷酸葡萄糖胺变位酶(phosphoglucosamine mutase,GlmM)和glmU基因编码的UDP-N-乙酰氨基葡萄糖焦磷酸化酶(UDP-N-acetylglucosamine pyrophosphorylase,GlmU)等。将来自大肠杆菌MG1655,枯草芽孢杆菌,恶臭假单胞菌,兽疫链球菌和谷氨酸棒杆菌5种不同来源上述相关酶的基因构建至pMUT2 ΔHyp3上进行比较分析(图5-a)。结果如图5-b~图5-d所示,单独过表达枯草芽孢杆菌来源氨基转移酶基因glmS1,大肠杆菌MG1655来源磷酸葡萄糖胺变位酶基因glmM,及枯草芽孢杆菌来源UDP-N-乙酰氨基葡萄糖焦磷酸化酶基因glmU较有效,果糖软骨素产量分别达到0.21、0.20、0.54 g/L,较大肠杆菌K4野生型产量提高了110%、100%及440%。该结果表明GlmU催化的步骤为UDP-GalNAc合成的关键限速步骤,枯草芽孢杆菌来源UDP-N-乙酰氨基葡萄糖焦磷酸化酶基因glmU的过表达强化,果糖软骨素产量提升最为显著。
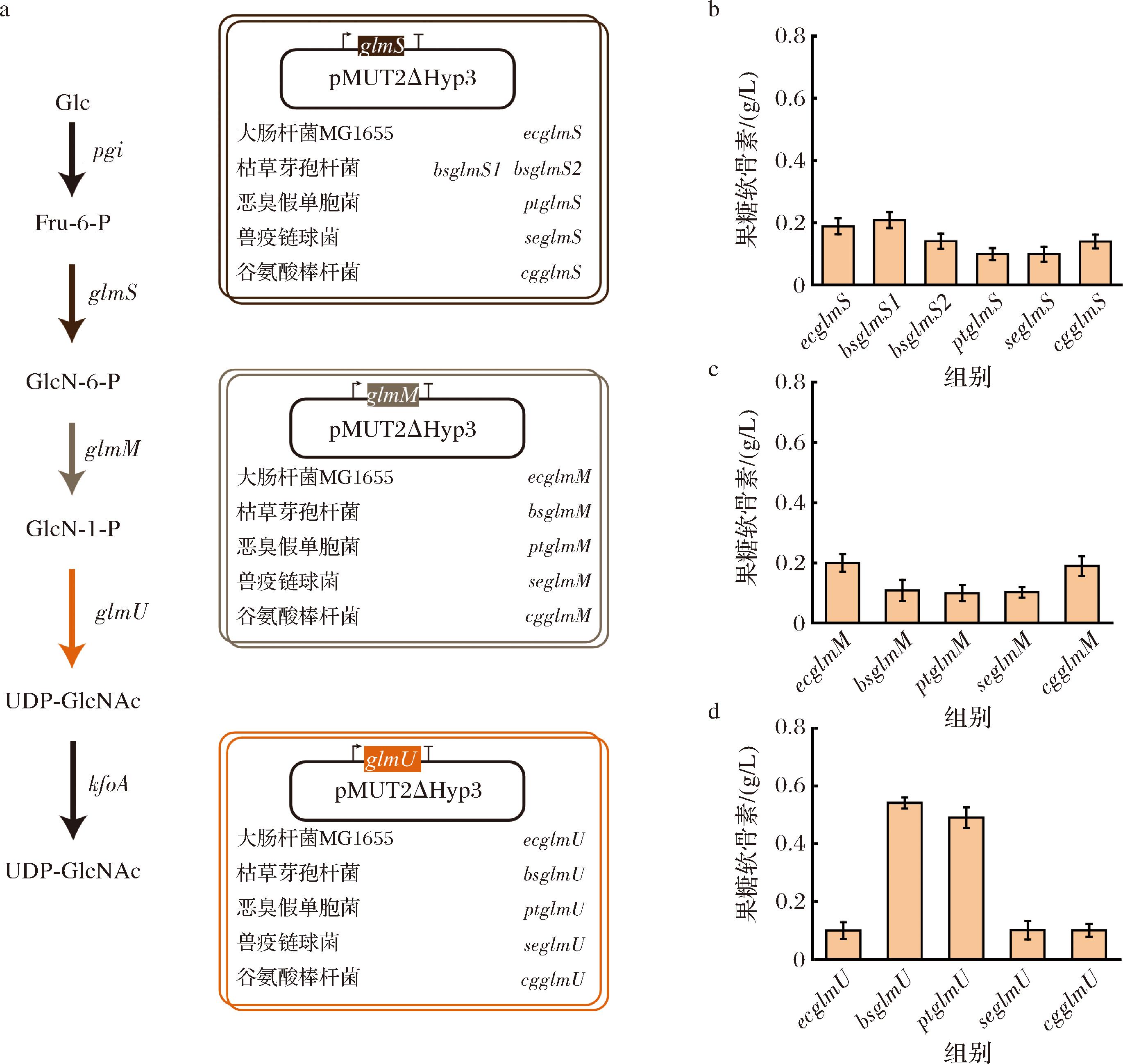
a-UDP-GalNAc合成途径示意图及不同来源的途径基因;b-不同来源glmS基因对果糖软骨素产量的影响;c-不同来源glmM基因对果糖软骨素产量的影响;d-不同来源glmU基因对果糖软骨素产量的影响
图5 果糖软骨素前体UDP-GalNAc合成途径强化
Fig.5 Enhancement of the UDP-GalNAc biosynthetic pathway for fructochondroitin precursor production
2.3 组合优化前体UDP-GlcA和UDP-GalNAc的供应提高果糖软骨素的产量
在分别研究UDP-GlcA和UDP-GalNAc的生物合成对果糖软骨素产量的影响后,本研究又对其进行了组合优化。在过表达谷氨酸棒杆菌来源ugdA2的基础上分别对兽疫链球菌来源的galU1、枯草芽孢杆菌来源的glmS1、大肠杆菌MG1655来源的glmM和枯草芽孢杆菌来源的glmU进行双基因串联组合表达,获得重组菌株K4-CH-1、K4-CH-2、K4-CH-3、K4-CH-4。重组菌株摇瓶无抗生素发酵结果如图6-a所示,K4-CH-1果糖软骨素产量较大肠杆菌K4野生型下降了约50%,仅为0.05 g/L,K4-CH-2、K4-CH-3及K4-CH-4的果糖软骨素产量分别达到了0.29、0.66、0.95 g/L,较大肠杆菌K4野生型产量提高了190%、560%及850%。其中重组菌株K4-CH-1仅提高了前体UDP-GlcA的合成能力,可能造成2个前体代谢通量不平衡,从而导致果糖软骨素产量下降。K4-CH-4菌株果糖软骨素产量提升最为明显,过表达谷氨酸棒杆菌来源的ugdA2与枯草芽孢杆菌来源的glmU基因后产量达到0.95 g/L,该结果说明2个前体的通量平衡影响果糖软骨素的合成,当2个前体的通量大且平衡时可能达到最佳效果,果糖软骨素的产量也达到最高。

a-UDP-GlcA和UDP-GalNAc合成途径基因组合优化;b-发酵培养基组分优化;c-3 L发酵罐分批补料发酵
图6 UDP-GlcA和UDP-GalNAc合成途径基因组合优化及发酵优化
Fig.6 Combinatorial optimization of UDP-GlcA and UDP-GalNAc biosynthetic pathways and fermentation process enhancement
2.4 重组大肠杆菌K4的发酵优化
为进一步提高果糖软骨素产量,对菌株发酵培养基的成分进行了优化。首先选择了LB-营养肉汤、M9基本培养基与甘油培养基3种常用培养基进行发酵,实验结果表明3种培养基较先前的培养基,果糖软骨素产量显著下降。为此在原先发酵培养基的基础上进一步优化碳氮源添加。实验结果表明,组合添加6 g/L酵母提取物和4 g/L胰蛋白胨时,果糖软骨素产量提升最明显,达1.35 g/L(图6-b)。选用发酵培养基-双组分,在3 L发酵罐上对其进行分批补料发酵(图6-c),4~24 h菌株快速生长,随后进入生长稳定期,OD600值维持在100左右。12~28 h发酵液中葡萄糖快速消耗,在这期间果糖软骨素快速合成。发酵40~48 h菌株活力下降,48 h结束发酵,最终果糖软骨素产量达到7.5 g/L,收率为4.4%(质量分数)。
3 结论与讨论
果糖软骨素/软骨素作为酶法合成CS的前体,具有广阔的市场需求。国内外研究人员已在多种宿主中实现了软骨素的生物合成(表1)。HU等[2]对途径基因ugd和glmS过表达,并结合“胞内积累”与“合成解聚耦联”策略,在谷氨酸棒杆菌中实现了软骨素寡聚糖产量达10.9 g/L。JIN等[14]在枯草芽孢杆菌中过表达内源性基因tuaD,使软骨素产量达到5.22 g/L。虽然在谷氨酸棒杆菌,枯草芽孢杆菌中实现了软骨素的生物合成,但由于这些菌株中缺乏软骨素的转运系统,导致软骨素提取成本大大增加。ZHANG等[16]在大肠杆菌K4中敲除pfkA,并过表达pgm、galU、glmS、glmM和kfoC基因,最终果糖软骨素产量达8.43 g/L。D’AMBROSIO等[1]以大肠杆菌K4为宿主,并过表达基因kfoA、kfoF、pgm和galU,使果糖软骨素达2.09 g/L。但上述重组菌株发酵生产果糖软骨素/软骨素都依赖抗生素的添加维持质粒稳定,从而易造成抗生素污染和抗性基因水平转移等问题。
表1 国内外软骨素合成现状
Table 1 Current status of chondroitin synthesis domestically and internationally

软骨素类型菌株方法发酵周期/h发酵罐体积/L产量/(g/L)果糖软骨素大肠杆菌K4利用无抗生素表达系统过表达基因ugdA2和glmU4837.5(本研究)果糖软骨素大肠杆菌K4敲除pfkA,过表达pgm、galU、glmS、glmM和kfoC基因40308.43[16]果糖软骨素大肠杆菌K4微过滤生物反应器分批发酵条件优化402.54.73[25]果糖软骨素大肠杆菌K4过表达基因kfoA、kfoF、pgm和galU23222.09[1]软骨素大肠杆菌BL21 STAR (DE3)调节碳氮池通量大小、通量方向和通量率以重编程软骨素合成代谢通路7259.2[12]软骨素兽疫链球菌过表达大肠杆菌K4来源的基因kfoA和kfoC1830.3[10]软骨素枯草芽孢杆菌过表达内源性基因tuaD8035.22[14]软骨素寡聚糖谷氨酸棒杆菌过表达基因ugd和glmS,并开发“胞内积累”和“合成解聚耦联”策略70310.9[2]
针对生物合成果糖软骨素发酵过程依赖抗生素添加的问题,本研究致力于构建无抗生素表达系统并用于果糖软骨素合成。首先选择可天然生产与转运果糖软骨素的大肠杆菌K4作为底盘细胞,筛选获得来源于EcN的隐性质粒pMUT2,并进一步优化获得稳定性更高的pMUT2 ΔHyp3质粒表达载体,用于构建大肠杆菌K4的无抗生素表达系统,该质粒在无抗生素条件下具有9 d的遗传稳定性。利用该质粒作为表达载体,分别过表达果糖软骨素合成途径中5种不同来源的途径基因,在前体UDP-GlcA的合成路径上,发现单独过表达谷氨酸棒杆菌来源的Ugd编码基因ugdA2,果糖软骨素产量提升最为显著,产量可达到0.57 g/L,较大肠杆菌K4野生型提高了470%。在UDP-GalNAc的合成路径中,确认了过表达枯草芽孢杆菌来源的GlmU编码基因glmU,果糖软骨素产量提升明显,可达到0.54 g/L,较大肠杆菌K4野生型提高了440%。在这基础上组合表达谷氨酸棒杆菌来源的基因ugdA2和枯草芽孢杆菌来源的基因glmU,果糖软骨素产量达到0.95 g/L。对该菌株培养基碳氮源进行优化,产量可进一步提升至1.35 g/L。最终3 L发酵罐不添加抗生素发酵生产果糖软骨素,48 h产量可达到7.5 g/L,收率为4.4%(质量分数)。本研究构建了一套无抗生素表达系统并实现了果糖软骨素的合成,降低了生产中对抗生素的依赖,为绿色酶法合成CS奠定了良好基础,后续可进一步对果糖软骨素产量优化提升,并敲除大肠杆菌K4中的果糖基转移酶(fructosyltransferase,KfoE),直接合成软骨素,从而降低工业生产的成本。
[1] D’AMBROSIO S, ALFANO A, CASSESE E, et al.Production and purification of higher molecular weight chondroitin by metabolically engineered Escherichia coli K4 strains[J].Scientific Reports, 2020, 10:13200.
[2] HU L T, WANG Y, HU Y X, et al.Biosynthesis of non-sulfated high-molecular-weight glycosaminoglycans and specific-sized oligosaccharides[J].Carbohydrate Polymers, 2022, 295:119829.
[3] VOLPI N.Anti-inflammatory activity of chondroitin sulphate:New functions from an old natural macromolecule[J].Inflammopharmacology, 2011, 19(6):299-306.
[4] HENROTIN Y, MARTY M, MOBASHERI A.What is the current status of chondroitin sulfate and glucosamine for the treatment of knee osteoarthritis?[J].Maturitas, 2014, 78(3):184-187.
[5] ZHANG W J, XU R R, CHEN J M, et al.Advances and challenges in biotechnological production of chondroitin sulfate and its oligosaccharides[J].International Journal of Biological Macromolecules, 2023, 253:126551.
[6] SHEN Q S,GUO Y J,WANG K Y, et al.A review of chondroitin sulfate’s preparation, properties, functions, and applications[J].Molecules, 2023, 28(20):7093.
[7] ZHANG W J, XU R R, JIN X R, et al.Enzymatic production of chondroitin oligosaccharides and its sulfate derivatives[J].Frontiers in Bioengineering and Biotechnology, 2022, 10:951740.
[8] SHENG J Y, JIN X R, XU R R, et al.One-pot synthesis of chondroitin sulfate A by engineered Pichia pastoris[J].Sheng Wu Gong Cheng Xue Bao, 2022, 38(7):2594-2605.
[9] WANG Z H, SONG W, WEI W Q, et al.Structural and mechanism-based engineering of sulfotransferase CHST15 for the efficient synthesis of chondroitin sulfate E[J].Applied and Environmental Microbiology, 2025, 91:e01573-24.
[10] CIMINI D, IACONO I.D, CARLINO E, et al.Engineering S.equi subsp. zooepidemicus towards concurrent production of hyaluronic acid and chondroitin biopolymers of biomedical interest[J].AMB Express, 2017, 7(1):61.
[11] CIMINI D, CARLINO E, GIOVANE A, et al.Engineering a branch of the UDP-precursor biosynthesis pathway enhances the production of capsular polysaccharide in Escherichia coli O5:K4:H4[J].Biotechnology Journal, 2015, 10(8):1307-1315.
[12] ZHAO C L, LI X M, GUO L, et al.Reprogramming metabolic flux in Escherichia coli to enhance chondroitin production[J].Advanced Science, 2024, 11(10):2307351.
[13] 赵春雷, 郭亮, 高聪, 等.代谢工程改造大肠杆菌生产软骨素[J].化工学报, 2023,74(5):2111-2122.ZHAO C L, GUO L, GAO C, et al.Metabolic engineering of Escherichia coli for chondroitin production[J].CIESC Journal, 2023, 74(5):2111-2122.
[14] JIN P, ZHANG L P, YUAN P H, et al.Efficient biosynthesis of polysaccharides chondroitin and heparosan by metabolically engineered Bacillus subtilis[J].Carbohydrate Polymers, 2016, 140:424-432.
[15] CHENG F Y, LUOZHONG S, YU H M, et al.Biosynthesis of chondroitin in engineered Corynebacterium glutamicum[J].Journal of Microbiology and Biotechnology, 2019, 29(3):392-400.
[16] ZHANG Q, YAO R, CHEN X L, et al.Enhancing fructosylated chondroitin production in Escherichia coli K4 by balancing the UDP-precursors[J].Metabolic Engineering, 2018, 47:314-322.
[17] CIMINI D, RUSSO R, D’AMBROSIO S, et al.Physiological characterization and quantitative proteomic analyses of metabolically engineered E.coli K4 strains with improved pathways for capsular polysaccharide biosynthesis[J].Biotechnology and Bioengineering, 2018, 115(7):1801-1814.
[18] CARATTOLI A.Plasmids and the spread of resistance[J].International Journal of Medical Microbiology, 2013, 303(6-7):298-304.
[19] 刘欢, 刘畅, 雷化雨, 等.双组分调控系统对革兰氏阴性菌抗生素耐药性的调控研究进展[J].食品与发酵工业, 2025,51(1):381-389.LIU H, LIU C, LEI H Y, et al.Advancements in antibiotic resistance regulation by two-component regulatory systems in Gram-negative bacteria[J].Food and Fermentation Industries, 2025,51(1):381-389.
[20] YANG Y, JIANG X T, CHAI B L, et al.ARGs-OAP:online analysis pipeline for antibiotic resistance genes detection from metagenomic data using an integrated structured ARG-database[J].Bioinformatics, 2016, 32(15):2346-2351.
[21] ZHOU S Y, ZHAO L L, ZUO W J, et al.Minimizing endogenous cryptic plasmids to construct antibiotic-free expression systems for Escherichia coli Nissle 1917[J].Synthetic and Systems Biotechnology, 2024, 9(1):165-175.
[22] KEASLING J D.Manufacturing molecules through metabolic engineering[J].Science, 2010, 330(6009):1355-1358.
[23] DATSENKO K A, WANNER B L.One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products[J].Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(12):6640-6645.
[24] LEROUX M, MICHAUD J, BAYMA E, et al.Misincorporation of galactose by chondroitin synthase of Escherichia coli K4:From traces to synthesis of chondbiuronan, a novel chondroitin-like polysaccharide[J].Biomolecules, 2020, 10(12):1667.
[25] RESTAINO O F, CIMINI D, DE ROSA M, et al.High cell density cultivation of Escherichia coli K4 in a microfiltration bioreactor:A step towards improvement of chondroitin precursor production[J].Microbial Cell Factories, 2011, 10(1):10.