格氏乳杆菌(Lactobacillus gasseri)是一种表面粗糙,颜色呈灰白色的革兰氏阳性菌,不生成孢子且无运动性。在显微镜下观察时,该菌呈现棒状结构,末端呈现圆形,属于同型发酵乳酸菌[1]。它能在厌氧或有氧环境下良好生长,最适生长温度为37 ℃、最适生长pH值为6.0。此外,它还对人工胃肠液及胆盐表现出较强的耐受能力[2-4],广泛存在于人体胃肠道和阴道中,是肠道微生物早期定植的优势物种之一。格氏乳杆菌不仅有助于维持消化道健康[5-6],还能通过产生抗菌细菌素来维持阴道微生态平衡[7],同时,在控制肥胖[8]、治疗糖尿病[9]、调节免疫[10-12]、抗氧化[13-15]等方面都发挥积极作用。
近年来,随着高通量测序技术的不断进步,研究人员已经成功对多种乳酸菌进行了全基因组的解析,并识别出了一系列与抗菌能力、黏附能力、耐酸能力等重要生理功能相关的基因或基因簇[16-17],还将基因组学与体外表型实验联合用来评价益生菌的安全性,进一步挖掘了它们在食品工业及其他领域应用的可能性[18]。培养基优化是通过实验对培养基中的营养成分(碳源、氮源、无机盐等)的含量及比例进行调整以达到菌体利用最大化的一种方法,通过这种方法,可以显著提高菌株的生长速度和产量,是目前最直接也是最常用的提高菌株产量的方法之一。
格氏乳杆菌G098最早从中国西藏自治区拉萨市一个健康婴儿的肠道中分离出来,是一株具有巨大发展潜力的益生菌。本项研究旨在利用先进的全基因组测序技术解析格氏乳杆菌G098的生理特征及益生特性,并对其培养基中营养成分的含量及比例进行优化,以期能够在有限的培养基中大量富集菌体,为格氏乳杆菌的产业化应用和微生物制剂产品的研发提供理论基础。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
本实验所用菌株——格氏乳杆菌G098分离自中国西藏自治区拉萨市一名健康婴儿肠道,由内蒙古农业大学乳酸菌菌种资源库提供。
1.1.2 试剂
葡萄糖、蜜二糖、蔗糖、果糖、阿拉伯糖、可溶性淀粉、乳糖、甘露糖、棉籽糖、纤维二糖、牛肝浸粉、酵母浸粉、鱼蛋白胨、酵母蛋白胨、胰蛋白胨、酪蛋白胨、眎蛋白胨、酵母粉、牛肉膏、大豆蛋白、柠檬酸、柠檬酸钠、磷酸氢二钠、磷酸二氢钠、磷酸二氢钾、磷酸氢二钾、乙酸钠、MnSO4·H2O、FeSO4·7H2O、ZnSO4·7H2O、CuSO4·5H2O、维生素B1、维生素B2、维生素B5、维生素B6、维生素B7、腺嘌呤、尿嘧啶、胸腺嘧啶、亮氨酸、异亮氨酸、赖氨酸、半胱氨酸、丝氨酸、缬氨酸、L-半胱氨酸盐酸盐、甘氨酸、精氨酸、天冬氨酸、谷氨酸、丙氨酸,本实验所用药品均为分析纯。
1.2 仪器与设备
SX-500高压蒸汽灭菌锅,日本Tomy Digital Biology;LRH-250生化培养箱,上海一恒科学仪器有限公司;ZHH-C智能安全型超净工作台,上海智城分析仪器有限公司;5424R台式高速冷冻离心机,德国Eppendorf AG;BX50光学显微镜,日本OLYMPUS;A35厌氧工作站,英国Don Whitley Scientific;PE20精密pH计,梅特勒-托利多仪器(上海)有限公司;UV-1700紫外可见分光光度计,日本岛津株式会社分析仪器部;Bioscreen C全自动生长曲线生长测定仪,芬兰Oy Growth Curves。
1.3 实验与方法
1.3.1 菌株的活化
将保藏于冻存管中的格氏乳杆菌G098接种于4.5 mL MRS液体培养基中进行活化,37 ℃恒温培养24 h,传至第2代,进行后续实验操作。
1.3.2 基因组测序
1.3.2.1 总DNA的提取
将活化好的菌株传代至2代,37 ℃恒温培养至对数生长期对菌液进行4 ℃,8 000 r/min离心,收集菌泥后,使用TIANamp Bacteria DNA Kit细菌基因组DNA提取试剂盒并按照说明书进行菌株总DNA的提取,使用Nanodrop检测基因组DNA的纯度、浓度、核酸吸收峰是否正常,然后使用Qubit精确检测基因组DNA的浓度,同时将Qubit浓度与Nanodrop浓度进行对比,判断样本的纯度,纯度合格后使用凝胶电泳实验检测基因组DNA的完整性。检查电泳条带合格后,进行下一步操作。
1.3.2.2 文库的构建与质控
样品质量检测合格后,方可进行文库构建,文库的构建使用PacBio SMRTbell Template Prep Kit 1.0文库构建试剂盒,具体步骤如下:首先使用gTube将基因组DNA打断,去除3’端的单链末端,然后对DNA链上的单链断裂或碱基缺失、氧化等进行损伤修复并对DNA链进行末端修复,连接barcode接头后进行核酸外切酶消化,去除两端未连接上SMRT哑铃型接头的片段,最后使用磁珠进行2次筛选纯化,获得测序文库。文库构建完成后,使用Qubit 3.0对文库质量进行准确定量,使用Agilent 2100对文库大小进行检测,文库大小符合预期后进行上机测序。
1.3.2.3 上机测序
文库质检合格后,按照目标下机数据量,使用PacBio Sequel Ⅱ进行文库测序,使用SMRT LINK 10.1.0软件进行数据处理。
1.3.2.4 数据的处理
使用hifiasm-0.16.1软件对基因组数据进行组装,使用R Studio软件将获得的完整基因组序列分别与KEGG、GO、COG数据库进行对比分析和功能注释。
1.3.3 各指标的测定
1.3.3.1 菌体密度的测定
将200 μL的待测样液加入96孔板,每孔3个平行对照,然后使用紫外可见分光光度计测量其OD值,波长设置为600 nm。
1.3.3.2 最大比生长率的测定
采用全自动生长曲线生长测定仪,在加样孔中添加200 μL菌液,设置波长600 nm,37 ℃培养24 h,每隔1 h对发酵液的菌体密度进行测定,并计算菌株的最大比生长率。
1.3.3.3 pH的测定
利用精密pH计于室温中测定发酵液的pH。
1.3.3.4 活菌数的测定
将发酵液用灭过菌的生理盐水稀释至所需倍数后,将1 mL稀释液加入无菌的一次性平皿中,再倒入10~15 mL灭过菌的MRS固体培养基,小心摇匀,待培养基凝固后放入恒温培养箱中,37 ℃培养48 h后,采用平板计数法计数。
1.3.4 培养基优化
1.3.4.1 碳源优化
采用单因素试验方法,分别将不同种类的碳源加入培养基中,按2%的接种量接种活化后的格氏乳杆菌G098,培养后测定菌体密度,活菌数和最大比生长率,以确定优化培养基中的碳源种类。
1.3.4.2 氮源优化
同样方法,在碳源优化结果的基础上分别选取不同种类的氮源加入MRS培养基中,按2%的接种量接种活化后的格氏乳杆菌G098,培养后测定菌体密度,活菌数和最大比生长率,对氮源进行筛选。在工业生产运用中,由于单一氮源生产成本较高,往往不使用单一氮源,而是将不同氮源适量复配使用,因此,本实验也将对氮源进行复配实验,以确定最佳氮源复配比。
1.3.4.3 碳氮源总含量及比例优化
在碳源、氮源优化结果的基础上,改变碳氮源总含量及比例,按2%的接种量接种活化后的格氏乳杆菌G098,培养后测定菌体密度,活菌数和最大比生长率,以确定最佳碳氮源总含量及比例。
1.3.4.4 缓冲盐体系优化
在碳氮源总含量及比例的基础上,设计添加不同种类、不同浓度的缓冲盐体系取代MRS培养基中的缓冲盐体系,按2%的接种量接种格氏乳杆菌G098,37 ℃培养24 h后,测定菌株的菌体密度,活菌数和最大比生长率,以确定优化培养基中的缓冲盐体系。
1.3.4.5 无机盐优化
基于上述优化结果,本实验选取不同种类、不同浓度的无机盐,按2%的接种量接种格氏乳杆菌G098,37 ℃培养24 h后,测定菌株的菌体密度,活菌数和最大比生长率,以确定优化培养基中的无机盐种类及浓度。
1.3.4.6 生长因子优化
生长因子是指微生物生长过程中必需的,自身不能合成必须由外界直接提供的某些成分,主要包括氨基酸、嘌呤、维生素等[3]。本研究借助全基因组测序技术对格氏乳杆菌G098的编码基因进行测定,通过与代谢通路综合分析,确定菌株自身不能合成的生长因子种类,进而选取适当的生长因子对培养基进行优化。然后再对上述生长因子设置不同浓度,以未添加生长因子的MRS培养基作为对照组,按2%的接种量接种格氏乳杆菌G098,37 ℃厌氧培养24 h,测定菌体密度,活菌数和最大比生长率,以确定优化培养基中的生长因子种类及浓度。
1.4 数据分析
每个实验设置3次重复,结果以“平均值±标准差”进行展示。数据分析使用SPSS 23.0软件,绘图使用Origin 2021软件,显著性检验采用Duncan’s多重比较方法,当P<0.05时,认为数据在统计学意义上存在显著差异。
2 结果与分析
2.1 基因组分析结果
2.1.1 全基因组测序与分析
格氏乳杆菌G098的基因组大小为1 991 131 bp,GC含量为35.30%,编码基因1 982个,编码基因的总长度为1 791 657 bp,占基因组大小的89.98%。格氏乳杆菌G098的基因组圈图如图1-a所示。全基因组数据已上传至NCBI(BioProject:PRJNA1217619;BioSample:SAMN46496436)。
2.1.2 KEGG数据库注释
通过与KEGG数据库进行比对共注释到了876个功能基因,这些基因一共参与了39条不同的通路,这些通路被分为6类,分别是细胞过程(cellular processes)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)、人类疾病(human diseases)、代谢(metabolism)和生物系统(organismal systems)等通路(图1-b)。其中,细胞过程中包含基因数量最多的是原核生物细胞群体(cellular community-prokaryotes),环境信息处理中包含基因数量最多的是膜转运(membrane transport);遗传信息处理中包含基因数量最多的是翻译(translation);人类疾病中包含基因数量最多的是耐药性:抗微生物(drug resistance: antimicrobial);代谢中包含基因数量最多的是碳水化合物代谢(carbohydrate metabolism);生物系统中包含基因数量最多的是内分泌系统(endocrine system)。
2.1.3 GO数据库注释
将格氏乳杆菌G098基因组序列与GO数据库进行比对,结果如图1-c所示,在生物过程(biological process)中,共有24类功能得到注释,其中,细胞过程和代谢过程(metabolic process)的功能基因最多,分别为744个和658个。在细胞组分(cellular component)中共有3类功能基因得到注释,包括含蛋白质复合物(protein-containing complex)、病毒粒子成分(virion component)和细胞解剖实体(cellular anatomical entity)。在分子功能(molecular function)中共有14类功能得到注释,其中催化活性(catalytic activity)和结合(binding)的功能基因最多,分别为764和730个。
2.1.4 COG数据库注释
将格氏乳杆菌G098的基因组序列在COG数据库进行注释,结果如图1-d所示。格氏乳杆菌G098中共有1 683个基因得到注释,分布在23个COG条目中。其中,翻译、核糖体结构与生物合成(translation, ribosomal structure and biogenesis)中包含的基因数量最多,共193个,占注释到的基因总数的11.47%,其次是碳水化合物转运与代谢(carbohydrate transport and metabolism),共173个,占注释到的基因总数的10.29%。

a-格氏乳杆菌G098基因组圈图;b-KEGG数据库;c-GO数据库;d-COG数据库
图1 格氏乳杆菌G098基因组圈图及KEGG、GO、COG数据库功能注释情况
Fig.1 Genome circle of L.gasseri G098 and functional annotation in KEGG, GO, COG databases
注:图1-a中从外到内依次是基因组位置、限制修饰酶、位于正链的碱基修饰位点、位于负链的碱基修饰位点、位于正链的编码基因、位于负链的编码基因、rRNA和tRNA分布、基因组GC偏斜情况值、基因组GC含量。
2.2 培养基优化结果
2.2.1 碳源优化结果
在前期培养基优化研究基础上[19],本实验分别选取葡萄糖、蜜二糖、蔗糖、果糖、阿拉伯糖、可溶性淀粉、乳糖、甘露糖、棉籽糖、纤维二糖为碳源,分析格氏乳杆菌G098的生长情况,从而筛选出最适合格氏乳杆菌G098生长的碳源,结果如表1所示。
表1 不同碳源对格氏乳杆菌G098增殖情况的影响
Table 1 The effect of different carbon sources on the proliferation of L.gasseri G098
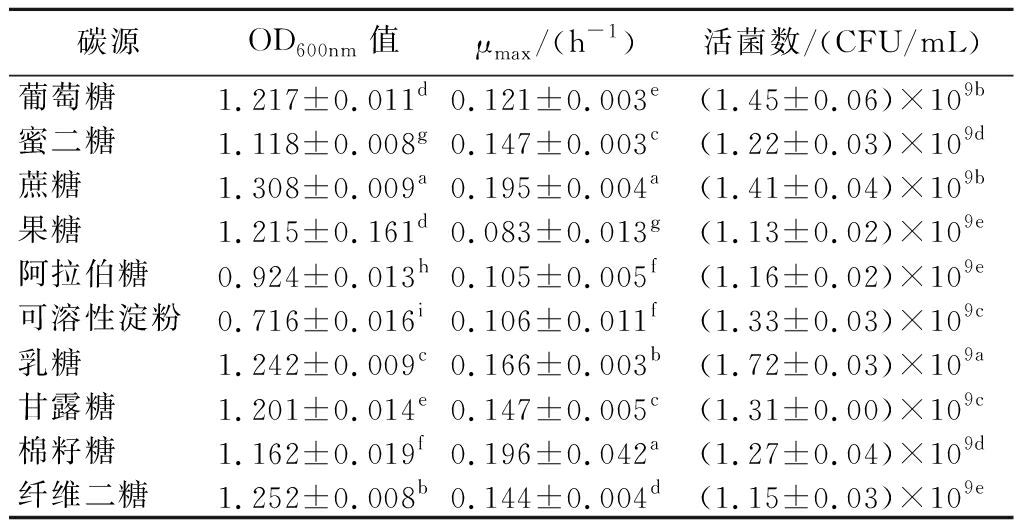
碳源OD600nm值μmax/(h-1)活菌数/(CFU/mL)葡萄糖1.217±0.011d0.121±0.003e(1.45±0.06)×109b蜜二糖1.118±0.008g0.147±0.003c(1.22±0.03)×109d蔗糖1.308±0.009a0.195±0.004a(1.41±0.04)×109b果糖1.215±0.161d0.083±0.013g(1.13±0.02)×109e阿拉伯糖0.924±0.013h0.105±0.005f(1.16±0.02)×109e可溶性淀粉0.716±0.016i0.106±0.011f(1.33±0.03)×109c乳糖1.242±0.009c0.166±0.003b(1.72±0.03)×109a甘露糖1.201±0.014e0.147±0.005c(1.31±0.00)×109c棉籽糖1.162±0.019f0.196±0.042a(1.27±0.04)×109d纤维二糖1.252±0.008b0.144±0.004d(1.15±0.03)×109e
注:不同字母表示数据间差异显著(P<0.05)(下同)。
从菌体密度来看,格氏乳杆菌G098对蔗糖,乳糖的利用能力最佳,从最大比生长率来看,格氏乳杆菌G098对蔗糖,棉籽糖的利用能力最佳,但计数结果显示,当以乳糖为碳源时,格氏乳杆菌G098的生长情况最好,且明显高于其他碳源。综合上述情况,选择乳糖作为格氏乳杆菌G098培养基中的碳源进行下一步实验。
2.2.2 氮源优化结果
本实验分别选取牛肝浸粉、酵母浸粉、鱼蛋白胨、酵母蛋白胨、胰蛋白胨、酪蛋白胨、眎蛋白胨、酵母粉、牛肉膏和大豆蛋白胨为氮源,对格氏乳杆菌G098的培养基进行优化,从中筛选出最适合格氏乳杆菌G098生长的氮源,结果如表2。
表2 不同氮源对格氏乳杆菌G098增殖情况的影响
Table 2 The effect of different nitrogen sources on the proliferation of L.gasseri G098
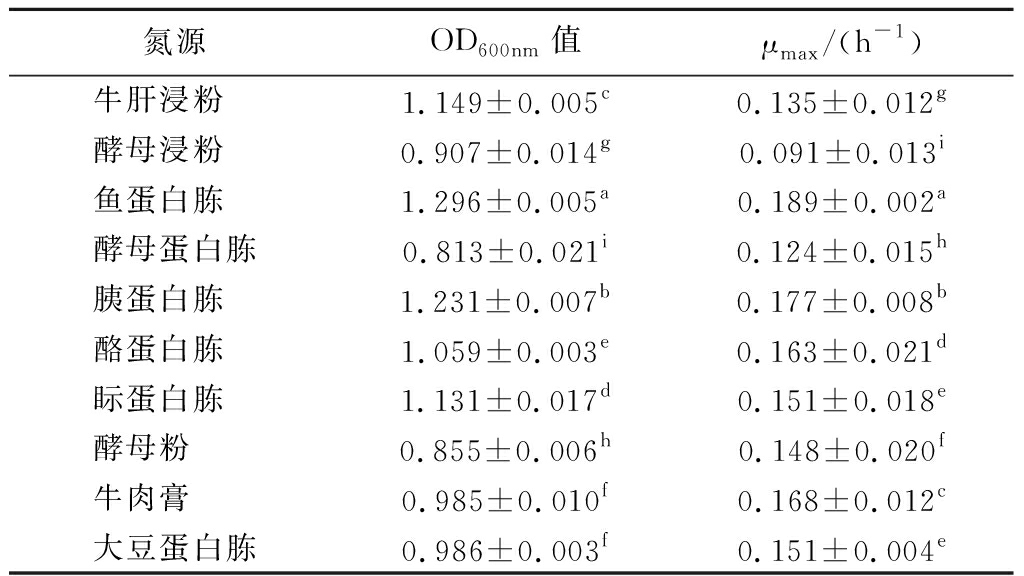
氮源OD600nm值μmax/(h-1)牛肝浸粉 1.149±0.005c0.135±0.012g酵母浸粉 0.907±0.014g0.091±0.013i鱼蛋白胨 1.296±0.005a0.189±0.002a酵母蛋白胨0.813±0.021i0.124±0.015h胰蛋白胨 1.231±0.007b0.177±0.008b酪蛋白胨 1.059±0.003e0.163±0.021d眎蛋白胨 1.131±0.017d0.151±0.018e酵母粉 0.855±0.006h0.148±0.020f牛肉膏 0.985±0.010f0.168±0.012c大豆蛋白胨0.986±0.003f0.151±0.004e
从菌体密度来看,格氏乳杆菌G098对鱼蛋白胨、胰蛋白胨的利用能力最佳,从最大比生长率来看,格氏乳杆菌G098亦对鱼蛋白胨、胰蛋白胨的利用能力最佳。先前的研究表明,当几种氮源组合使用时,菌株的产量往往比使用单一氮源的产量更高,也更符合实际生产运用中节约成本的需求[20-21]。因此对氮源的使用进行复配实验,结合上述结果,选择鱼蛋白胨和胰蛋白胨进行复配。
由表2、表3结果可知,鱼蛋白胨添加量大于胰蛋白胨添加量时,格氏乳杆菌G098的菌体密度和最大比生长率高于其他实验组。接着,对不同氮源比实验组进行活菌计数,结果如表4所示。
表3 不同氮源复配比对格氏乳杆菌G098增殖情况的影响
Table 3 The effect of different nitrogen source combinations on the proliferation of L.gasseri G098
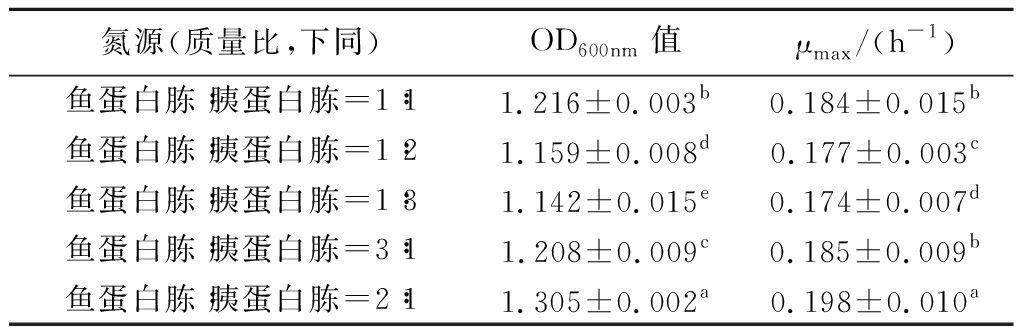
氮源(质量比,下同)OD600nm值μmax/(h-1)鱼蛋白胨∶胰蛋白胨=1∶11.216±0.003b0.184±0.015b鱼蛋白胨∶胰蛋白胨=1∶21.159±0.008d0.177±0.003c鱼蛋白胨∶胰蛋白胨=1∶31.142±0.015e0.174±0.007d鱼蛋白胨∶胰蛋白胨=3∶11.208±0.009c0.185±0.009b鱼蛋白胨∶胰蛋白胨=2∶11.305±0.002a0.198±0.010a
表4 不同氮源复配比对格氏乳杆菌G098活菌数的影响
Table 4 The effect of different nitrogen source compound ratios on the viable count of L.gasseri G098
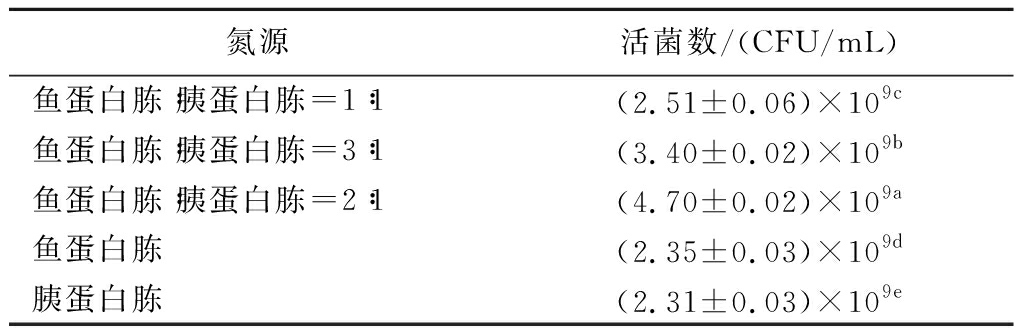
氮源活菌数/(CFU/mL)鱼蛋白胨∶胰蛋白胨=1∶1(2.51±0.06)×109c鱼蛋白胨∶胰蛋白胨=3∶1(3.40±0.02)×109b鱼蛋白胨∶胰蛋白胨=2∶1(4.70±0.02)×109a鱼蛋白胨(2.35±0.03)×109d胰蛋白胨(2.31±0.03)×109e
活菌计数结果显示,鱼蛋白胨∶胰蛋白胨=2∶1(均为质量比)时,格氏乳杆菌G098的活菌数最大。综合菌体密度、最大比生长率和活菌数的结果,选择鱼蛋白胨∶胰蛋白胨=2∶1的复配比作为格氏乳杆菌G098培养基中的氮源进行下一步实验。
2.2.3 碳氮源总含量及比例优化
碳源和氮源都是乳酸菌培养基中必不可少的成分,但其含量并非多多益善,碳氮源之间比例的协调与平衡也十分重要。若培养基中的碳氮总含量过低,会使菌株因缺少生长所需的营养物质而抑制其生长发育,若培养基中的碳氮含量过高,会导致生长过程中渗透压的改变和代谢产物的积累,反而抑制了菌株的生长发育[22]。因此,确定培养基中碳氮源总含量及比例十分重要。设置不同的碳氮源总含量及比例,对格氏乳杆菌G098的碳氮源总含量及比例进行优化,不同碳氮源总含量及比例对菌株增殖情况的影响见图2-a和图2-b。在碳氮源总含量为12%(质量分数),碳源∶氮源=1∶3时菌体密度达到最大值,在碳氮源总含量为6%,碳源∶氮源=1∶3(质量比,下同)时最大菌生产率达到最大值。对这2个实验组以及MRS培养基进行活菌计数,结果显示,当碳∶氮=1∶3,碳氮源总含量为12%时,格氏乳杆菌G098的活菌数(5.21×109 CFU/mL)显著高于碳∶氮=1∶3,碳氮源总含量为6%的实验组(4.60×109 CFU/mL)以及MRS培养基(1.07×109 CFU/mL)的活菌数(P<0.05)。所以选择碳∶氮=1∶3,碳氮总量为12%为格氏乳杆菌G098的碳氮源总含量及比例优化结果。
2.2.4 缓冲盐的优化
缓冲盐可以调节培养基的pH值,中和酸性产物,参与营养物质的主动运输,并促进菌株的生长[23-24]。在前面优化实验所得结果的基础上,设置柠檬酸-柠檬酸钠、磷酸氢二钠-磷酸二氢钠、磷酸氢二钠-柠檬酸、磷酸氢二钠-磷酸二氢钾以及磷酸氢二钾-乙酸钠-柠檬酸钠5种缓冲盐体系,对格氏乳杆菌G098培养基中的缓冲盐体系进行优化。不同缓冲盐体系对菌株增殖情况的影响见图2-c和图2-d。当向培养基中添加0.04 mol/L的磷酸氢二钾-乙酸钠-柠檬酸钠时,格氏乳杆菌G098的菌体密度存在最大值。当向培养基中添加0.08 mol/L的磷酸氢二钾-乙酸钠-柠檬酸钠时,格氏乳杆菌G098的最大比生长率存在最大值。且磷酸氢二钾-乙酸钠-柠檬酸钠体系处于任一浓度下均优于其他缓冲盐实验组。分别对0.04、0.08 mol/L的磷酸氢二钾-乙酸钠-柠檬酸钠缓冲盐体系实验组以及MRS培养基进行活菌计数,结果显示当磷酸氢二钾-乙酸钠-柠檬酸钠体系浓度为0.04 mol/L时活菌数最大(5.38×109 CFU/mL),且显著高于另外2组(P<0.05)。
3.2.5 无机盐的优化
本实验在培养基中添加不同量的4种无机盐MnSO4·H2O、FeSO4·7H2O、ZnSO4·7H2O、CuSO4·5H2O,对菌体增殖情况进行测定,结果如图2-e和图2-f。当向培养基中添加0.10 g/L的FeSO4·7H2O时,格氏乳杆菌G098的菌体密度达到最大(1.64),且在任一添加量,FeSO4·7H2O组结果都明显优于其他组。当向培养基中添加0.08 g/L的ZnSO4·7H2O时,格氏乳杆菌G098的最大比生长率达到最大值。因此,对0.10 g/L的FeSO4·7H2O组,0.08 g/L的ZnSO4·7H2O组和MRS培养基进行活菌计数,结果显示,当添加0.10 g/L的FeSO4·7H2O时,格氏乳杆菌G098的活菌数为5.48×109 CFU/mL,显著高于其他2组的活菌数(P<0.05),故而选择0.10 g/L的FeSO4·7H2O作为优化培养基的缓冲盐体系。
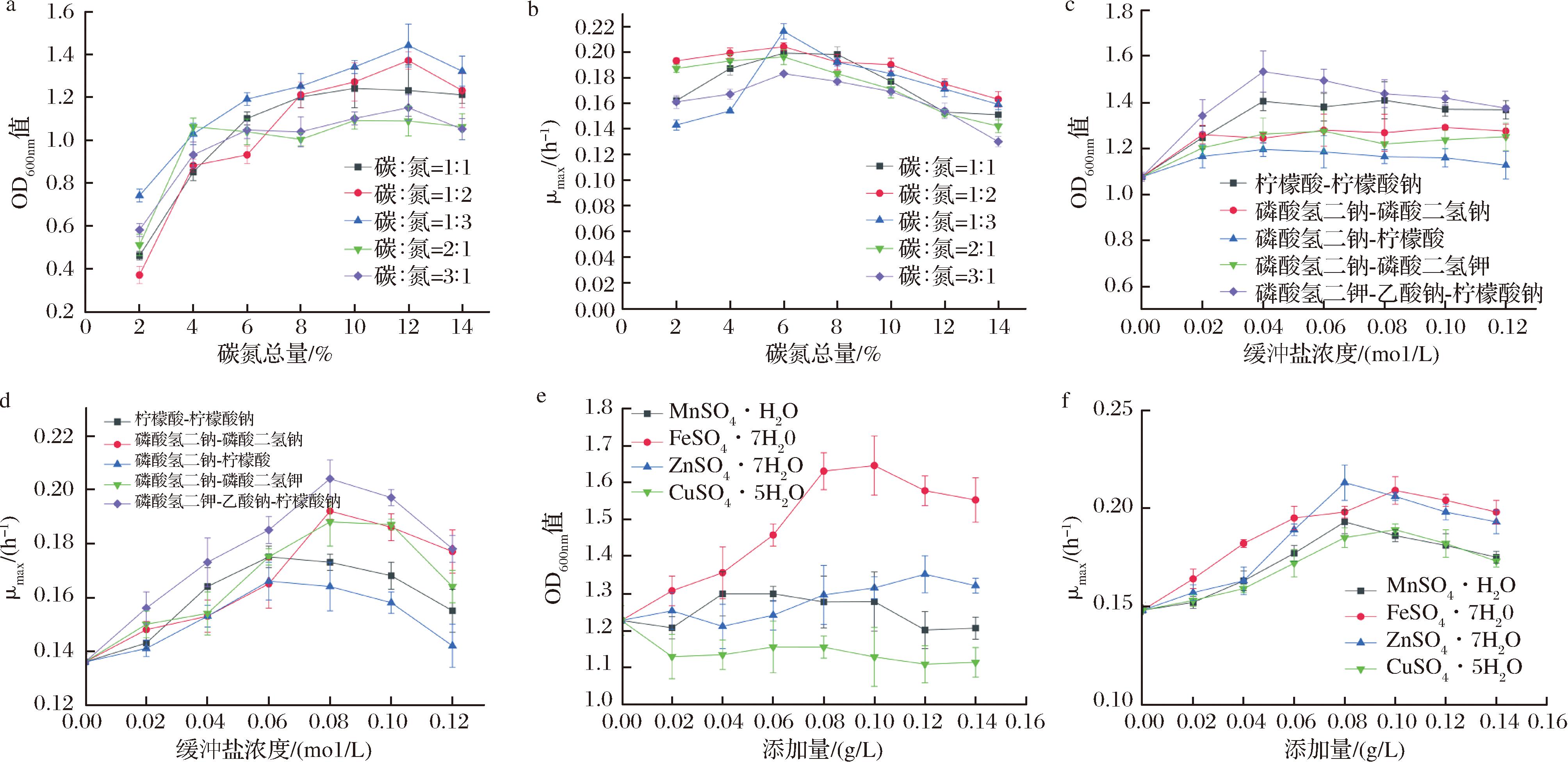
a-不同碳氮源总含量及比例对菌株菌体密度的影响;b-不同碳氮源总含量及比例对菌株最大比生长率的影响;c-不同缓冲盐体系对菌株菌体密度的影响;d-不同缓冲盐体系对菌株最大比生长率的影响;e-不同无机盐对菌株菌体密度的影响;f-不同无机盐对菌株最大比生长率的影响
图2 不同条件下格氏乳杆菌G098的增殖情况
Fig.2 Proliferation of L.gasseri G098 under different conditions
2.2.6 生长因子的优化
以氨基酸的选择为例,目前已知自然界中的氨基酸种类大约有100种,其中在生物体中组成蛋白质的氨基酸约有20多种。由于氨基酸的种类繁多,若对所有种类逐一进行实验,不仅耗时费力,而且效率极低。借助全基因组测序技术能够对格氏乳杆菌G098的编码基因进行测定,再与代谢通路进行综合分析,进而确定菌株自身缺失或不能合成的氨基酸种类。研究结果显示,格氏乳杆菌G098虽然具备多种氨基酸的代谢途径,但却缺少编码这些氨基酸的基因,这些氨基酸包括缬氨酸、亮氨酸、异亮氨酸、赖氨酸、半胱氨酸、甘氨酸、丝氨酸、精氨酸、丙氨酸、天冬氨酸、谷氨酸等。
因此,本研究选择上述氨基酸作为优化培养基的实验材料。其他生长因子的选择同样使用了此方法。
由图3可知,在添加了维生素、嘌呤和氨基酸的培养基中培养菌株时,菌体密度均小于之前的菌体密度最大值1.64,当在培养基中添加0.2 g/L的维生素B2,0.3 g/L的赖氨酸、0.3 g/L的L-半胱氨酸盐酸盐时,最大比生长率显著高于其他实验组(P<0.05)。

a-不同维生素对菌株菌体密度的影响;b-不同维生素对菌株最大比生长率的影响;c-不同和核苷酸对菌株菌体密度的影响;d-不同核苷酸对菌株最大比生长率的影响;e, g-不同氨基酸对菌株菌体密度的影响;f, h-不同氨基酸对菌株最大比生长率的影响
图3 不同生长因子对格氏乳杆菌G098增殖的影响
Fig.3 The effect of different growth factors on the proliferation of L. gasseri G098
注:折线图中虚线表示前面优化后的培养基中的菌体密度和最大比生长率。
为验证实验的可靠性,对添加了0.2 g/L的维生素B2,0.3 g/L的赖氨酸、0.3 g/L的L-半胱氨酸盐酸盐的实验组进行了活菌计数,计数结果在图4中进行展示,这3组实验组的活菌数始终显著低于前面优化后的培养基中的活菌数(P<0.05)。这说明,格氏乳杆菌G098的培养基中维生素、嘌呤和氨基酸的含量已能够满足菌体的生长需求,无需另外补充生长因子。
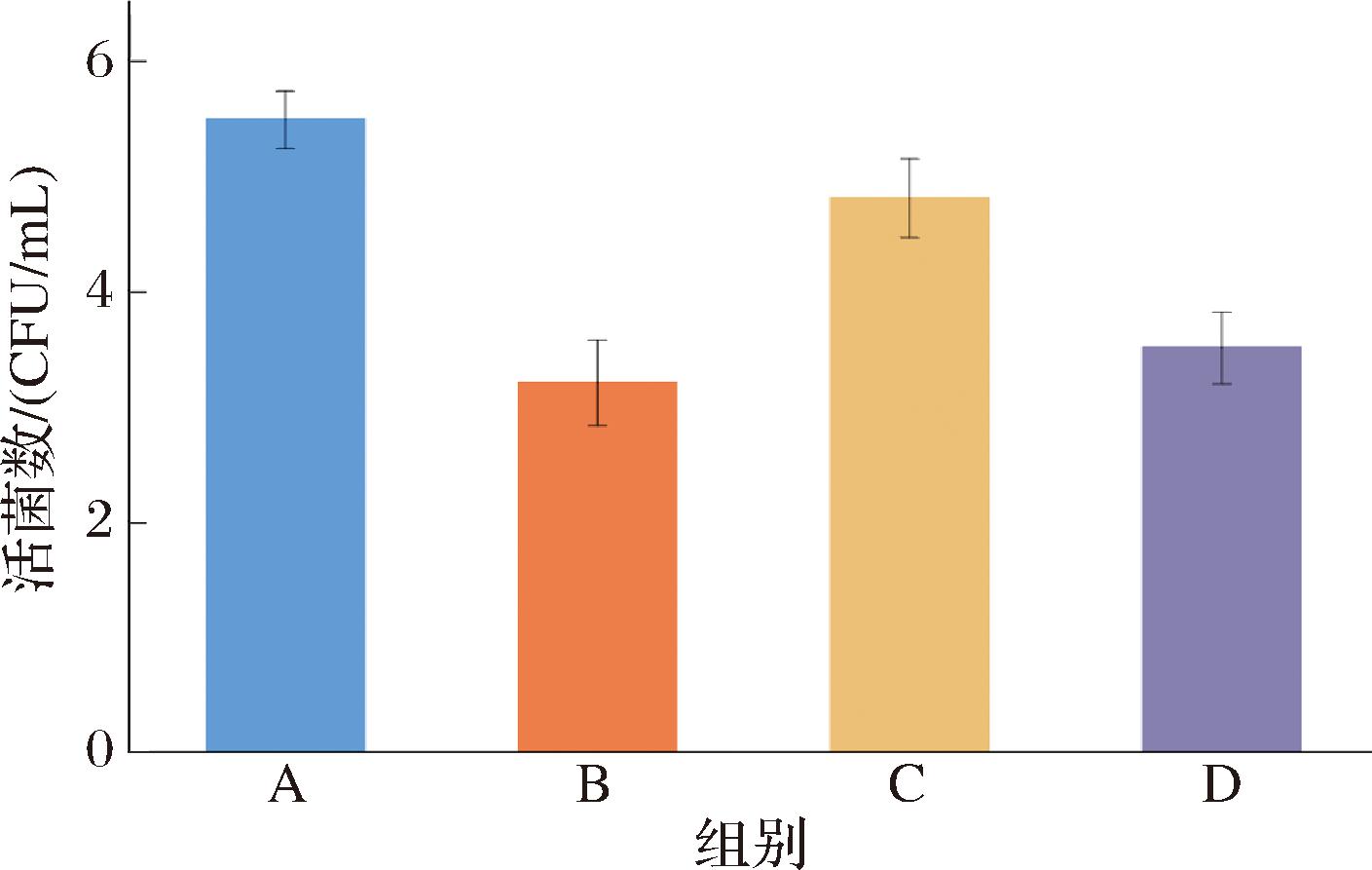
图4 生长因子对格氏乳杆菌G098活菌数的影响
Fig.4 The effect of growth factors on the viable count of L.gasseri G098
注:图中横坐标中A表示前面优化后的培养基,B表示0.2 g/L的维生素B2,C表示0.3 g/L的赖氨酸,D表示0.3 g/L的L-半胱氨酸盐酸盐。
3 结论
本研究通过对格氏乳杆菌G098进行全基因组测序分析,确定了其基因组大小为1 991 131 bp,GC含量为35.30%,识别出了1 982个编码基因,这些编码基因的总长度为1 791 657 bp,占基因组大小的89.98%;结合多个数据库的注释结果进行培养基优化实验,确定了格氏乳杆菌G098的优化培养基配方为:38.00 g乳糖,62.66 g鱼蛋白胨,31.33 g胰蛋白胨,5.75 g磷酸氢二钾,14.27 g无水乙酸钠,5.75 g柠檬酸钠,0.01 g FeSO4·7H2O,1.00 g吐温80,以及1 000 mL蒸馏水。在这种优化的培养条件下,格氏乳杆菌G098的活菌数达到了5.48×109 CFU/mL,约为优化前的5.1倍,大大提高了菌株的生长效率。传统的微生物培养基优化方法通常依赖于大量实验的反复尝试,不仅耗时费力且资源消耗巨大。本研究提出一种基于全基因组测序分析的培养基优化方法,通过该方法快速且精确地确定了格氏乳杆菌G098的优化培养基配方,实现了培养基的精准优化。此外,该方法的成功应用也证明了全基因组测序分析在微生物学研究中的巨大潜力,随着测序技术的不断发展和成本的进一步降低,基于全基因组测序的分析方法有望在更多微生物菌株的培养基优化中得到广泛应用,为微生物资源的开发和利用提供更加高效、精准的技术支持。
[1] FRANCL A L, THONGARAM T, MILLER M J..The PTS transporters of Lactobacillus gasseri ATCC 33323[J].BMC Microbiology, 2010, 10(1):77.
[2] 孙琳琳. 一株格氏乳杆菌免疫调节作用及其调控途径的研究[D].哈尔滨:东北农业大学, 2020.SUN L L.Research on the immunoregulation effects and the mechanism of Lactobacillus gasseri JM1[D].Harbin:Northeast Agricultural University, 2020.
[3] 郭艳荣. 格氏乳杆菌体外筛选及高密度培养工艺研究[D].呼和浩特:内蒙古农业大学, 2020.GUO Y R.The in Vitro screening of Lactobacillus gasseri and the research of high cell density culture[D].Hohhot:Inner Mongolia Agricultural University, 2020.
[4] 蒋晓晗, 阳田恬, 李金转, 等.加氏乳杆菌的特性与生物学功能及其生产应用[J].饲料工业, 2023, 44(6):96-100.JIANG X H, YANG T T, LI J Z, et al.Properties of Lactobacillus gasseri and its biological functions[J].Feed Industry, 2023, 44(6):96-100.
[5] 田亚针. 格氏乳杆菌JM1对结肠炎小鼠肠道屏障的调节作用研究[D].哈尔滨:东北农业大学, 2021.TIAN Y Z.Regulatory effect of Lactobacillus gasseri JM1 on the intestinal barrier in colitis mice[D].Harbin:Northeast Agricultural University, 2021.
[6] LIANG L Z, MENG Z H, ZHANG F, et al.Lactobacillus gasseri LG08 and Leuconostoc mesenteroides LM58 exert preventive effect on the development of hyperuricemia by repairing antioxidant system and intestinal flora balance[J].Frontiers in Microbiology, 2023, 14:1211831.
[7] MALDONADO-BARRAG N A, CABALLERO-GUERRERO B, MART
N A, CABALLERO-GUERRERO B, MART N V, et al.Purification and genetic characterization of gassericin E, a novel co-culture inducible bacteriocin from Lactobacillus gasseri EV1461 isolated from the vagina of a healthy woman[J].BMC Microbiology, 2016, 16(1):37.
N V, et al.Purification and genetic characterization of gassericin E, a novel co-culture inducible bacteriocin from Lactobacillus gasseri EV1461 isolated from the vagina of a healthy woman[J].BMC Microbiology, 2016, 16(1):37.
[8] 杨洋, 高航, 李中柱.益生菌减肥功能的研究进展[J].中国奶牛, 2023(4):39-45.YANG Y, GAO H, LI Z Z.Research progress of probiotics in the treatment of obesity[J].China Dairy Cattle, 2023(4):39-45.
[9] JIANG S M, LIU A J, MA W Y, et al.Lactobacillus gasseri CKCC1913 mediated modulation of the gut-liver axis alleviated insulin resistance and liver damage induced by type 2 diabetes[J].Food &Function, 2023, 14(18):8504-8520.
[10] HSIEH M H, JAN R L, WU L S, et al.Lactobacillus gasseri attenuates allergic airway inflammation through PPARγ activation in dendritic cells[J].Journal of Molecular Medicine, 2018, 96(1):39-51.
[11] OH N S, JOUNG J Y, LEE J Y, et al.Probiotic and anti-inflammatory potential of Lactobacillus rhamnosus 4B15 and Lactobacillus gasseri 4M13 isolated from infant feces[J].PLoS One, 2018, 13(2):e0192021.
[12] AOKI-YOSHIDA A, YAMADA K, HACHIMURA S, et al.Enhancement of oral tolerance induction in DO11.10 mice by Lactobacillus gasseri OLL2809 via increase of effector regulatory T cells[J].PLoS One, 2016, 11(7):e0158643.
[13] DI LUCCIA B, MAZZOLI A, CANCELLIERE R, et al.Lactobacillus gasseri SF1183 protects the intestinal epithelium and prevents colitis symptoms in vivo[J].Journal of Functional Foods, 2018, 42:195-202.
[14] KOBATAKE E, NAKAGAWA H, SEKI T, et al.Protective effects and functional mechanisms of Lactobacillus gasseri SBT2055 against oxidative stress[J].PLoS One, 2017, 12(5):e0177106.
[15] HU J, MA L B, ZHENG W Y, et al.Lactobacillus gasseri LA39 activates the oxidative phosphorylation pathway in porcine intestinal epithelial cells[J].Frontiers in Microbiology, 2018, 9:3025.
[16] GUHANRAJ R, DHANASEKARAN D.Probiotic functional gene explorations in the genome of Limosilactobacillus fermentum GD5MG[J].Microbial Pathogenesis, 2024, 192:106686.
[17] YETIMAN A E, KESKIN A, DARENDELI B N, et al.Characterization of genomic, physiological, and probiotic features Lactiplantibacillus plantarum DY46 strain isolated from traditional lactic acid fermented shalgam beverage[J].Food Bioscience, 2022, 46:101499.
[18] 刘文俊, 张和平.益生菌基因组学在乳酸菌筛选和功能评价中的应用[J].中国食品学报, 2024, 24(1):1-11.LIU W J, ZHANG H P.Application of probiogenomics in screening and functional evaluation of probiotics lactic acid bacteria[J].Journal of Chinese Institute of Food Science and Technology, 2024, 24(1):1-11.
[19] 周亮. 格氏乳杆菌G098的培养基优化及高密度发酵工艺研究[D].呼和浩特:内蒙古农业大学, 2023.ZHOU L.Optimization of medium and high-density fermentation process of Lactobacillus gasseri G098[D].Hohhot:Inner Mongolia Agricultural University, 2023.
[20] 刘香英, 孙洪蕊, 范海茹, 等.益生性植物乳杆菌K25高密度培养工艺优化[J].食品科技, 2018, 43(3):20-25.LIU X Y, SUN H R, FAN H R, et al.The optimization of high cell density culturing of probiotic Lactobacillus plantarum K25[J].Food Science and Technology, 2018, 43(3):20-25.
[21] 田佳雪. 东北酸菜复合发酵剂的制备及其对酸菜品质影响探究[D].长春:吉林大学, 2024.TIAN J X.Preparation of northeast sauerkraut mixed starter cultures and its effect on the quality of sauerkraut[D].Changchun:Jilin University, 2024.
[22] 杨加怀. 植物乳杆菌299高密度培养及冷冻干燥保护的研究[D].天津:天津科技大学, 2015.YANG J H.Study on high cell density culture and freeze-drying proteetion of Lactobacillus plantarum 299[D].Tianjin:Tianjin University of Science and Technology, 2015.
[23] JINGJING E, MA R Z, CHEN Z C, et al.Improving the freeze-drying survival rate of Lactobacillus plantarum LIP-1 by increasing biofilm formation based on adjusting the composition of buffer salts in medium[J].Food Chemistry, 2021, 338:128134.
[24] IIJIMA H, WATANABE A, SUKIGARA H, et al.Simultaneous increases in the levels of compatible solutes by cost-effective cultivation of Synechocystis sp.PCC 6803[J].Biotechnology and Bioengineering, 2020,.117(6):1649-1660.