β-榄香烯(β-elemene)是抗肿瘤药物榄香烯的主要活性成分,来源于温郁金成熟块茎中的倍半萜,可由吉玛烯A(germacrene A)在高温条件下自发重排形成[1]。相关研究表明,β-榄香烯具有广谱抗肿瘤活性和免疫保护特性,被广泛用于肺癌等多种恶性肿瘤的治疗中[2-3]。此外,β-榄香烯还具有抗菌、抗病毒、抗氧化等药理活性[4]。最初,研究者通过非对映异构的内烷基化化学反应合成β-榄香烯,但该方法需高温高压环境且用有毒试剂,操作复杂,在研究和应用中受限[5]。也有报道通过蒸汽蒸馏的方法从药用植物如温郁金的挥发油中提取和分离β-榄香烯[6-7]。然而,从植物中提取β-榄香烯的方法有很多弊端,如产量低、纯化困难、环境污染等[8]。合成生物学与分子生物学技术的快速发展,使得构建微生物细胞工厂以实现天然萜类化合物的高效合成成为领域的研究热点。与化学合成法和植物提取法相比,微生物细胞工厂具有安全、效率高、环境友好等优势,可用于大规模工业化的生产[9-10]。
解脂耶氏酵母(Yarrowia lipolytica)具有利用多种碳源的优势,并且能够适应一些复杂环境等特点,是合成天然产物的优质底盘细胞[11-12]。其次,其具有高通量的三羧酸(tricarboxylic acid cycle, TCA)循环,为合成萜类化合物提供了丰富的前体[13]。此外,日益成熟的遗传分子工具使得解脂耶氏酵母成为一种优秀的代谢工程底盘菌株[14]。目前,已有一些利用微生物细胞工厂合成β-榄香烯的研究,如大肠杆菌(Escherichia coli)、酿酒酵母(Saccharomyces cerevisiae)、汉逊酵母(Ogataea polymorpha)、毕赤酵母(Pichia pastoris)以及解脂耶氏酵母(表1)。相较大肠杆菌而言,酵母因具有完整的内膜系统在表达异源基因及合成生物活性物质生产方面具有优势[14]。
表1 微生物合成β-榄香烯的研究进展
Table 1 Progress in the microbial synthesis of β-elemene
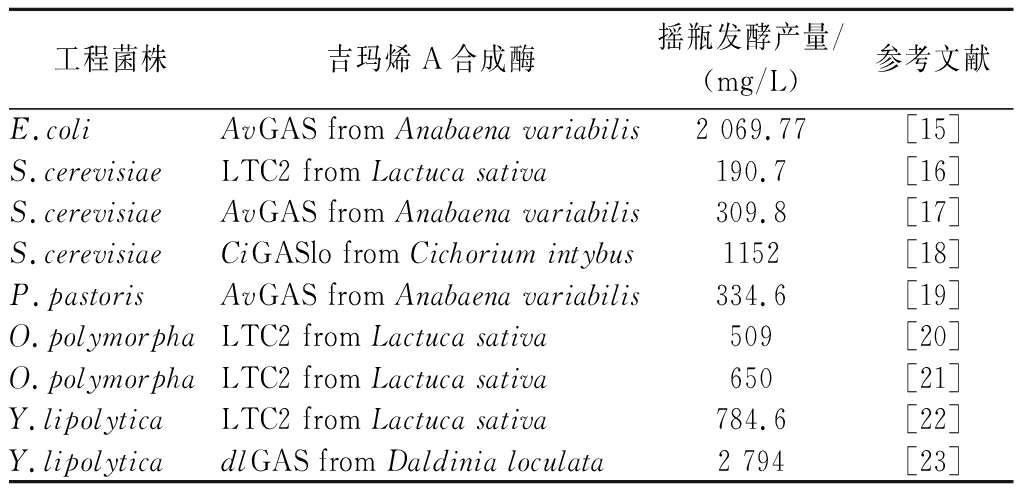
工程菌株吉玛烯A合成酶摇瓶发酵产量/(mg/L)参考文献E.coliAvGAS from Anabaena variabilis2 069.77[15]S.cerevisiaeLTC2 from Lactuca sativa190.7[16]S.cerevisiaeAvGAS from Anabaena variabilis309.8[17]S.cerevisiaeCiGASlo from Cichorium intybus1152[18]P.pastorisAvGAS from Anabaena variabilis334.6[19]O.polymorphaLTC2 from Lactuca sativa509[20]O.polymorphaLTC2 from Lactuca sativa650[21]Y.lipolyticaLTC2 from Lactuca sativa784.6[22]Y.lipolyticadlGAS from Daldinia loculata2 794[23]
CHENG等[19]在毕赤酵母中比较了法尼基焦磷酸合成酶(farnesyl diphosphate synthase, ERG20)与吉玛烯A合成酶(germacrene A synthase, GAS)融合表达连接序列对β-榄香烯产量的影响,其结果表明采用GGGGS和(PT)4P的连接序列均能显著提高β-榄香烯产量。YE等[20]在汉逊酵母中比较了基因融合表达的相对位置,发现ERG20与莴苣来源的吉玛烯A合成酶(LTC2 from Lactuca sativa, LTC2)融合表达ERG20-LTC2是LTC2-ERG20产量的10倍;进一步增加甲羟戊酸(mevalonate, MVA)途径基因拷贝数使β-榄香烯产量提高了76%。LI等[22]在解脂耶氏酵母中表达LTC2,并增加LTC2拷贝数并优化MVA途径,摇瓶产量达到784.6 mg/L。另一研究中经过筛选出高效dlGAS并优化前体供应改造解脂耶氏酵母,在摇瓶产量达到了2 794 mg/L[23]。
本研究首先引入LTC2、过表达MVA途径系列基因以及探讨基因融合表达对β-榄香烯产量的影响(图1)。同时,通过增加融合基因的拷贝数以及通过随机整合的方式增加MVA途径基因的代谢通量提高β-榄香烯,产量达到(1 326.78±62.42)mg/L。进一步通过培养基优化提高β-榄香烯的产量达到(1 668.13±41.01)mg/L。
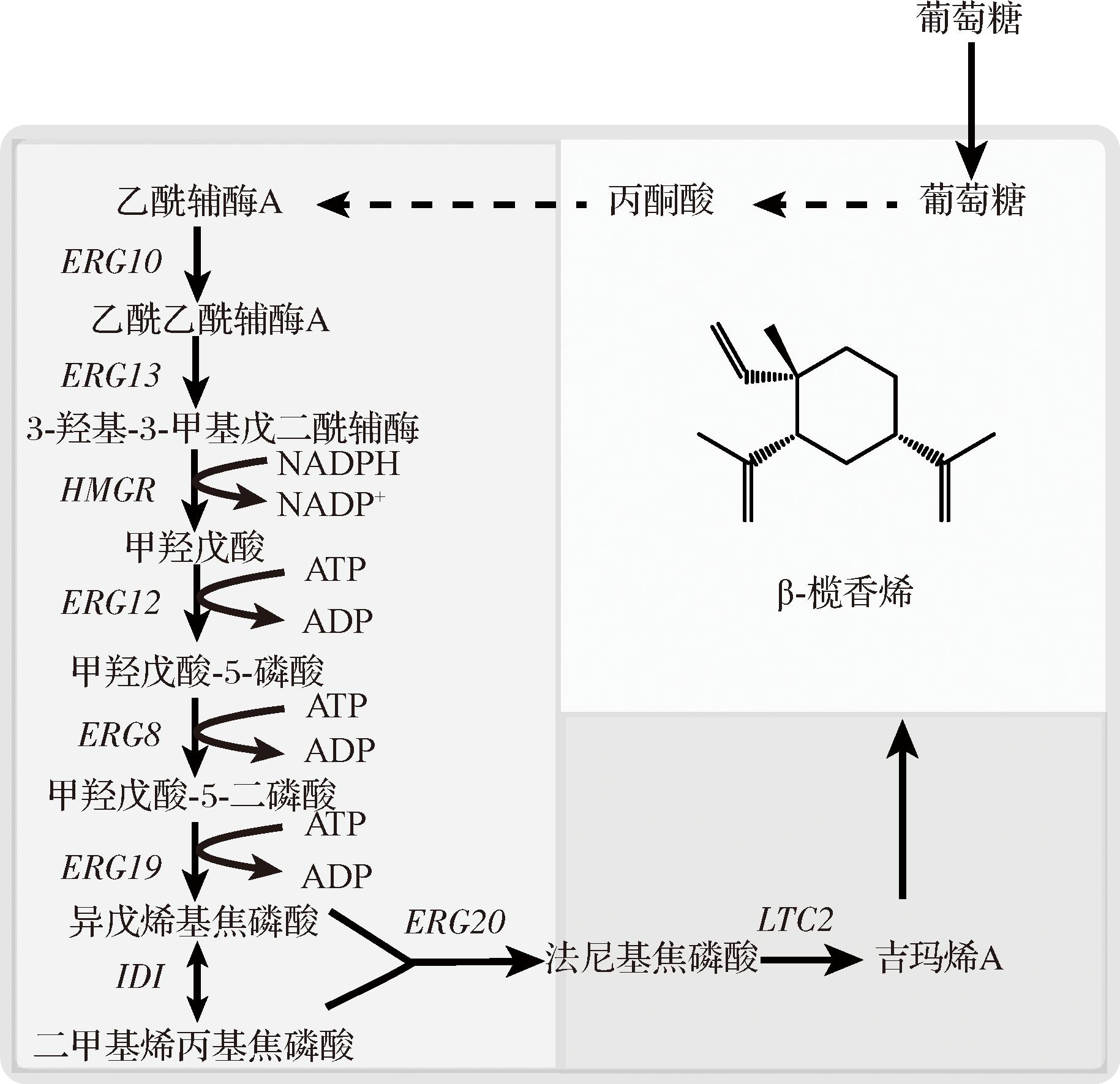
图1 在解脂耶氏酵母中合成β-榄香烯的示意图
Fig.1 Schematic illustration of the synthesis of β-elemene in Y.lipolytica
注:红色字体表示过表达的基因;ERG10:乙酰辅酶A乙酰基转移酶(acetyl-CoA acetyltransferase,ERG10);ERG13:羟甲基戊二酰辅酶A合成酶(hydroxymethylglutaryl-CoA synthase,ERG13);HMGR:羟甲基戊二酰辅酶A还原酶(hydroxymethylglutaryl-CoA reductase,HMGR);ERG12:甲羟戊酸激酶(mevalonate kinase,ERG12);ERG8:磷酸甲羟戊酸激酶(phosphomevalonate kinase,ERG8);ERG19,焦磷酸甲羟戊酸脱羧酶(mevalonate pyrophosphate decarboxylase,ERG19);IDI:异戊烯焦磷酸异构酶(isopentenyl-diphosphate,IDI);ERG20:法尼基焦磷酸合酶(farnesyl diphosphate synthase,ERG20);LTC2:莴苣来源的吉玛烯A合成酶(germacrene A synthase from Lactuca sativa,LTC2)。
1 材料与方法
1.1 实验材料
1.1.1 菌株与质粒
Y.lipolytica(ATCC MYA-2613)、E.coli(JM109)以及部分质粒由本实验室保存;来源于莴苣的吉玛烯A合成酶基因(LTC2)质粒pUC57-LTC2以及引物由北京擎科生物科技有限公司合成。研究中使用及构建的质粒和菌株(表2),基因克隆和质粒构建所用引物(表3)。
表2 本文涉及的质粒和菌株
Table 2 Plasmids and strains used in this study

质粒和菌株 描述来源PUC57-LTC2LTC2, AmpR 实验室保存84609pCRISPRyl with XPR2 targeting sgRNA实验室保存84610pCRISPRyl with A08 targeting sgRNA实验室保存84614HR donor to integrate PUAS1B8TEF (136)-hrGFP-TCYC1into XPR2 locus实验室保存84615HR donor to integrate PUAS1B8TEF (136)-hrGFP-TCYC1into A08 locus实验室保存pINA1312Php4 d, TXPR2, Ura3, KmR实验室保存pINA1269Php4 d, TXPR2, Leu2, AmpR实验室保存T1Ylt1::PTMAL, TCYC1, PFBA, TPEX20t, HisG-Ura3-HisG实验室保存T2Ylt2::PFBA, TPEX20t, PGPD, TCYC1, HisG-Ura3-HisG实验室保存T3Ylt3::PACBP, TXPR2, PTMAL, TCYC1, HisG-Ura3-HisG实验室保存84615-HMGR PTEFin-HMGR-TCYC1本研究84614-LTC2PTEFin-LTC2-TCYC1本研究84614-LTC2-ERG20PTEFin-LTC2-ERG20-TCYC1本研究84614-ERG20-LTC2PTEFin-ERG20-LTC2-TCYC1本研究T1-ERG10-ERG8Ylt1::PTMA-ERG10-TCYC1, PFBA-ERG8-TPEX20t, HisG-Ura3-HisG本研究T2-ERG13-ERG19Ylt2::PFBA-ERG13-TPEX20t, PGPD-ERG19-TCYC1, HisG-Ura3-HisG本研究T3-ERG12-IDIYlt3::PACBP-ERG12-TXPR2, PTMAL-IDI-TCYC1, HisG-Ura3-HisG本研究1312-ERG20-LTC2Php4 d-ERG20-LTC2-TXPR2, Ura3本研究1269-ERG20-LTC2Php4 d-ERG20-LTC2-TXPR2, Leu2本研究L-0ATCCMYA-2613, MATa, leu2-270, ura3-302, xpr2-322, axp-2, (leu2-, ura3-)实验室保存L-1L-0, ΔXPR2::PTEFin-LTC2-TCYC1本研究L-2L-1, ΔA08::PTEFin-HMGR-TCYC1本研究L-3L-0, ΔA08::PTEFin-HMGR-TCYC1本研究L-4L-3, ΔXPR2::PTEFin-LTC2-ERG20-TCYC1本研究L-5L-3, ΔXPR2::PTEFin-ERG20-LTC2-TCYC1本研究L-6L-5, Ylt1::PTMA-ERG10-TCYC1, PFBA-ERG8-TPEX20t本研究L-7L-6, Ylt2::PFBA-ERG13-TPEX20t, PGPD-ERG19-TCYC1本研究L-8L-7, Ylt3::PACBP-ERG12-TXPR2, PTMAL-IDI-TCYC1本研究L-9L-8, Php4 d-ERG20-LTC2-TXPR2, Ura3本研究L-10L-9, Php4 d-ERG20-LTC2-TXPR2, Leu2本研究
表3 本文涉及的引物序列
Table 3 Primer sequences used in this study

引物序列(5'-3')LTC2-FAAGAATCATTCAAAGGCGCGCCATGGCTGCTGTCGACACCAACLTC2-RATAACTAATTACATGAGGCTAGCTTACATGGAGACAGATCCGACHMGR-FTCTGAGTATAAGAATCATTCAAAGCATGCTACAAGCAGCTATTGGAAAGAHMGR-RAGCGTGACATAACTAATTACATGAGCTATGACCGTATGCAAATATTCGAAERG10-FACTCCCCACCACCACTACTCCAACCATGCGACTCACTCTGCCCCGACTTAERG10-RAAGCGTGACATAACTAATTACATGACTCGACAGAAGAGACCTTCTTGATGERG8-FTCTACACAAACTAACCCAGCTCTCCATGACCACCTATTCGGCTCCGGGAAERG8-RAGTTGCGTCAATCGTCAAATATAGCCTACTTGAACCCCTTCTCGAGCCGCERG13-FTCTACACAAACTAACCCAGCTCTCCATGTCGCAACCCCAGAACGTTGGAAERG13-RAGTTGCGTCAATCGTCAAATATAGCCTACTGCTTGATCTCGTACTTTCGTERG19-FTTGAATTAAACACACATCAACAATGATGATCCACCAGGCCTCCACCACCGERG19-RAAGCGTGACATAACTAATTACATGACTACTTGCTGTTCTTCAGAGAACCAERG12-FTCCACACAACACCCACATTACCACCATGGACTACATCATTTCGGCGCCAGERG12-RCGGCAACGTGGGGACAGGCCATGGACTAATGGGTCCAGGGACCGATGTTTIDI-FACTCCCCACCACCACTACTCCAACCATGACGACGTCTTACAGCGACAAAAIDI-RAAGCGTGACATAACTAATTACATGACTACTTGATCCACCGCCGAATCTCGERG20-FTTCTGAGTATAAGAATCATTCAAAGATGTCCAAGGCGAAATTCGAAAGCGlinker-20RTGGCGTTGGTGTCGACAGCAGCCATAGATCCTCCTCCTCCCTTCTGTCGCTTGTAAATCTTGGCGlinker-20FGCTGTTTGTCGGATCTGTCTCCATGGGAGGAGGAGGATCTATGTCCAAGGCGAAATTCGAAAGCGERG20-RAGCGTGACATAACTAATTACATGAGTTACTTCTGTCGCTTGTAAATCTTG20-LTC2-FCCACATCACACATACAACCACACACATCATGTCCAAGGCGAAATTCGAAAGCGTGT20-LTC2-RCGTAGTTGGATCCTTAGTTTCGGGTTCCTTACATGGAGACAGATCCGACAAACAGCA08yz-FATAACGATAGCATATAAACTGCACGA08yz-RCTCTAGTATCTTCTCTGACCCTGTGXPR2yz-FCAGGTGAAGCCCTGGTTGATXPR2yz-RCTGGCCCTGGTCTAACTTCG
1.1.2 主要试剂和酶类
本研究中所使用的限制性内切酶,Thermo Fisher Scientific公司;Phanta Super-Fidelity DNA Polymerase、2×Taq Master Mix(Dye Plus)聚合酶、ClonExpress Ⅱ One Step Cloning Kit,南京诺唯赞生物科技有限公司;胰蛋白胨、酵母提取物、琼脂粉,Biosharp生物;无氨基酸酵母氮源(yeast nitrogen base, YNB),青岛海博生物技术有限公司;氨苄青霉素与卡那霉素,生工生物工程(上海)股份有限公司;十二烷,上海阿拉丁生化科技股份有限公司;Frozen-EZ Yeast Transformation Ⅱ Kit,ZYMO RESEARCH;酵母基因组DNA提取试剂盒,北京索莱宝科技有限公司;质粒提取试剂盒,北京金沙生物科技有限公司;胶回收试剂盒,上海迈跟生物科技有限公司;常规化学试剂(如五氟乳清酸,亮氨酸,尿嘧啶)等,上海麦克林生化科技股份有限公司。
1.1.3 培养基
LB培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠5,超纯水定容至1 L。根据需要加入氨苄青霉素或卡那霉素至终质量浓度为100 μg/mL或50 μg/mL。
酵母浸出粉胨葡萄糖培养基(yeast extract peptone dextrose medium,YPD)(g/L):胰蛋白胨20,酵母提取物10,超纯水定容至900 mL。冷却后添加100 mL 200 g/L葡萄糖。
酵母浸出粉胨葡萄糖-五氟乳清酸培养基(yeast extract peptone dextrose medium-5-fluorooroticacid,YPD-5-FOA)(g/L):胰蛋白胨20,酵母提取物10,超纯水定容至900 mL。冷却后添加100 mL 200 g/L葡萄糖以及五氟乳清酸。
YNB培养基(g/L):无氨基酸酵母氮源6.7,超纯水定容至900 mL。冷却后添加100 mL 200 g/L葡萄糖,根据需要添加终浓度为100 mg/L亮氨酸和100 mg/L尿嘧啶。
根据需要固体培养基添加质量浓度20 g/L琼脂粉,培养基均经121 ℃灭菌处理20 min;葡萄糖经115 ℃灭菌处理20 min;抗生素经0.22 μm水相滤膜过滤除菌。
1.2 实验方法
1.2.1 质粒与菌株的构建
以PUC57-LTC2质粒为模板,使用引物LTC2-F/R扩增LTC2基因,使用一步克隆试剂盒连在经过BssHII和NheI双酶切84614载体上构建84614-LTC2质粒。以ATCCMYA-2613酵母基因组为模板,使用引物ERG20-F/linker-20-R或linker-20-F/ERG20-R扩增ERG20基因,与经过BssHⅡ或NheⅠ单酶切84614-LTC2质粒为载体连接,分别构建出84614-LTC2-ERG20和84614-ERG20-LTC2质粒。以84614-ERG20-LTC2质粒为模板,使用引物20-LTC2-F/R扩增ERG20-LTC2基因,与经pmlⅠ酶切的1312和1269质粒载体连接,分别构建出1312-ERG20-LTC2质粒、1269-ERG20-LTC2质粒。
以ATCCMYA-2613酵母基因组为模板,使用引物HMGR-F/R、ERG12-F/R、ERG8-F/R、ERG13-F/R、ERG19-F/R、ERG10-F/R、IDI-F/R扩增HMGR、ERG12、ERG8、ERG13、ERG19、ERG10、IDI基因,其中HMGR与经过BssHⅡ和NheⅠ双酶切84615质粒载体连接,构建84615-HMGR质粒。ERG10基因与经EcoRI酶切后的T1质粒载体连接,构建T1-ERG10质粒;ERG8基因与经SpeⅠ酶切后的T1-ERG10质粒载体连接,构建T1-ERG10-ERG8质粒。ERG13基因与经EcoRⅠ酶切后的T2质粒载体连接,构建T2-ERG13质粒;ERG19基因与经AvrⅡ酶切后的T2-ERG13质粒载体连接,构建T2-ERG13-ERG19质粒。ERG12与经SpeⅠ酶切后的T3质粒载体连接,构建出T3-ERG12质粒;IDI基因与经NotⅠ酶切后的T3-ERG12质粒载体连接,构建出T3-ERG12-IDI质粒。所有质粒通过测序验证质粒是否构建成功。
通过CRISPR(clustered regularly interspaced short palindromic repeats)/Cas9技术将ERG20-LTC2、HMGR整合到Y.lipolytica基因组XPR2位点和A08位点,设计位于同源臂序列上的引物XPR2yz-F/R、A08yz-F/R,通过PCR验证酵母基因组并送测序验证整合是否成功。待单菌落酵母基因组验证成功后,待在YNB液体培养基中生长后涂至YPD-5-FOA平板上去掉Ura筛选标记。
利用基因组多拷贝位点Ylt将MVA途径进行过表达;利用zeta位点将营养标记和ERG20-LTC2进行整合[24]。将质粒T1-ERG10-ERG8、T2-ERG13-ERG19、T3-ERG12-IDI经SwaⅠ酶切后回收含目的基因表达盒片段,并依次转化酵母涂至YNB+Leu平板;经过3轮转化依次得到菌株L-6、L-7、L-8。将基因片段ERG20-LTC2与营养标记Ura和Leu整合在基因组zeta位点。将质粒1312-ERG20-LTC2经NotⅠ酶切后回收含目的基因表达盒片段,并转化酵母涂至YNB+Leu平板后经筛选得到菌株L-9。将质粒1269-ERG20-LTC2经ApaⅠ酶切后回收含目的基因表达盒片段,并转化至YNB平板后经筛选得到菌株L-10。从转化平板随机挑取30个单菌落进行发酵,并筛选出产量最优菌株去筛选标记作为下一阶段实验出发菌株。
1.2.2 解脂耶氏酵母转化
取解脂耶氏酵母种子在10 mL YPD液体培养基中30 ℃培养20~24 h,取0.5~1.0 mL至50 mL YPD液体培养基中,培养至OD600=0.6~0.8。取1 mL于1.5 mL离心管中,经离心去除培养基。按以下顺序加入Frozen-EZ Yeast Transformation Ⅱ Kit转化试剂:向离心管中加入500 μL EZ Solution Ⅰ并混匀,5 000 r/min离心4 min去掉上清液;向离心管中加入50 μL EZ Solution Ⅱ并混匀;向离心管中加入1 μg sgrna质粒(如84609)、1 μg donor重组质粒(如84614-LTC2)、或者加入5 μg线性化片段(如1312-ERG20-LTC2)并混匀;向离心管中加入500 μL EZ Solution Ⅲ,涡旋震荡混匀。于30 ℃水浴锅中水浴120 min,期间每15~20 min取出颠倒混匀数次。在生物安全柜中将酵母转化体系涂至YNB平板或根据需要补充亮氨酸、尿嘧啶平板上。待平板表面干燥后,倒置放30 ℃培养箱2~3 d。
1.2.3 培养方法
大肠杆菌培养:从甘油保藏管中挑取菌液在LB加相应抗性的固体培养基上划线活化,置于37 ℃培养箱中培养12~18 h;待长出单菌落后,挑取单菌落接种至10 mL LB加相应抗性的液体培养基中,37 ℃、220 r/min条件下培养12~18 h。
酵母种子液培养:从甘油保藏管中挑取菌液在YPD固体培养基上划线活化,置于30 ℃培养箱中培养48~72 h;待长出单菌落后,挑取单菌落接种至10 mL的YPD液体培养基中,30 ℃、220 r/min条件下培养24 h。
酵母摇瓶发酵培养:将种子液转接到50 mL YPD培养基中,使其初始OD600值为0.1,并加入体积分数为4%的十二烷作为有机萃取相,开始摇瓶发酵;30 ℃、220 r/min条件下培养72 h。每组做3个平行并进行重复实验。
1.2.4 β-榄香烯的检测
将发酵液以4 000 r/min离心10 min获取有机层,菌体在100 ℃烘箱烘至恒重。吸取上层十二烷有机相液体为发酵后样本,并用注射器吸取有机相经滤膜过滤后用于GC-MS分析。GC-MS检测条件为:气相色谱柱为HP-5 ms柱,氦气作为载气。采用检测程序:起始温度在80 ℃保持1 min,以30 ℃/min的速率升至140 ℃,保持5 min,以10 ℃/min的速率升至170 ℃,保持1 min,以30 ℃/min的速率升至290 ℃,保持5 min。运行总时长21 min。以十二烷为溶剂,配制β-榄香烯标准品溶液,进行GC-MS分析并绘制标准曲线用于样品中β-榄香烯浓度的定量。
1.3 数据处理与分析
采用Snap Gene 6.0.2软件进行引物设计;所有菌株均做3个平行发酵,采用GraphPad Prism 8软件进行数据分析,数据采用“平均值±标准差”表示。
2 结果与分析
2.1 解脂耶氏酵母中β-榄香烯合成途径构建
本研究使用CRISPR/Cas9方式在Y.lipolytica的基因组中整合LTC2,菌株L-1在发酵72 h后β-榄香烯产量为(23.19±1.48) mg/L(图2),而出发菌株L-0中未检测出β-榄香烯。在菌株L1基础上增加HMGR基因的拷贝,菌株L-2生成了(109.28±5.89) mg/L β-榄香烯,相较于L-1提高了4.71倍。因此,基于菌株L-0中增加HMGR的拷贝得到菌株L-3进行后续实验研究。
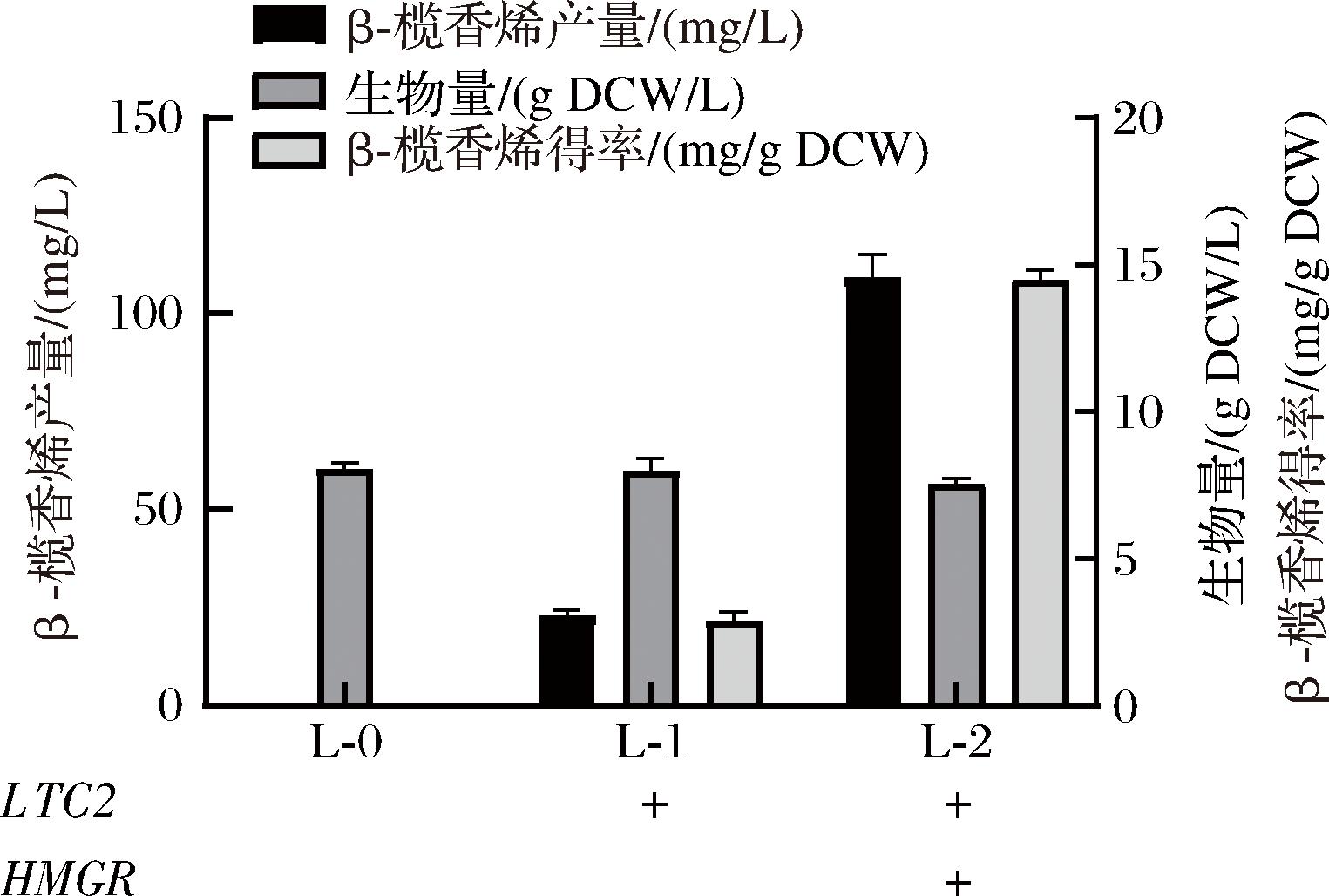
图2 在解脂耶氏酵母中表达关键基因对β-榄香烯产量的影响
Fig.2 Effect of expression of key genes on β-elemene production in Y.lipolytica
注:+:表示整合基因表达(下同)。
2.2 LTC2与ERG20的融合表达
相关研究报道,蛋白质融合是一种提高酶促反应效率的策略,它能缩短底物间的通道,减少反应中间体的损失[25]。倍半萜合成酶基因与ERG20基因融合表达可以增加产物的合成[23]。本研究采用柔性linker GGGGS将LTC2与ERG20融合表达,并且对两者相对位置差异影响β-榄香烯产量进行分析。将其分别整合到菌株L-3的基因组中构建菌株L-4、L-5,发酵72 h后分别生成(217.63±22.52)mg/L、(545.86±16.3)mg/L的β-榄香烯。菌株L-5产量是菌株L-2产量的4.99倍(图3)。上述结果表明,ERG20-LTC2融合表达可以促进法尼基焦磷酸(farnesyl pyrophosphate,FPP)向β-榄香烯转化。
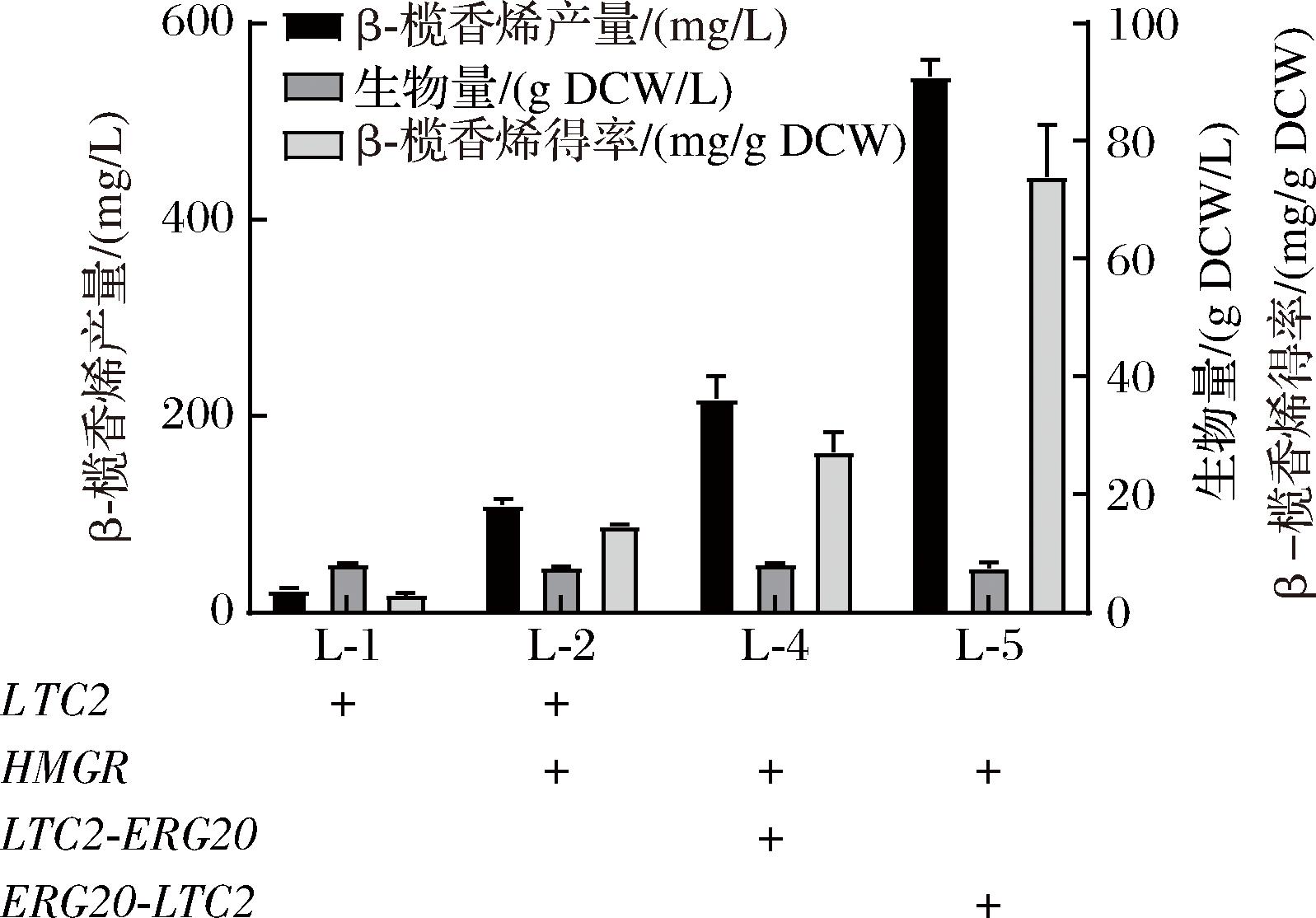
图3 基因ERG20与LTC2融合表达对β-榄香烯产量的影响
Fig.3 Effect of fusion expression of genes ERG20 and LTC2 on β-elemene production
2.3 强化MVA途径基因表达
在解脂耶氏酵母中,其内在的MVA途径为β-榄香烯的合成途径提供FPP前体(图1)。强化前体供应是一种常用且高效的促进产物合成的代谢工程策略[14]。本研究中,依次将MVA基因进行过表达并构建菌株L-6、L-7、L-8,发酵72 h后分别生成(627.68±13.11)、(803.96±20.83)、(881.33±19.27) mg/L的β-榄香烯,菌株L-8与L-5相比产量提高了61.4%(图4)。
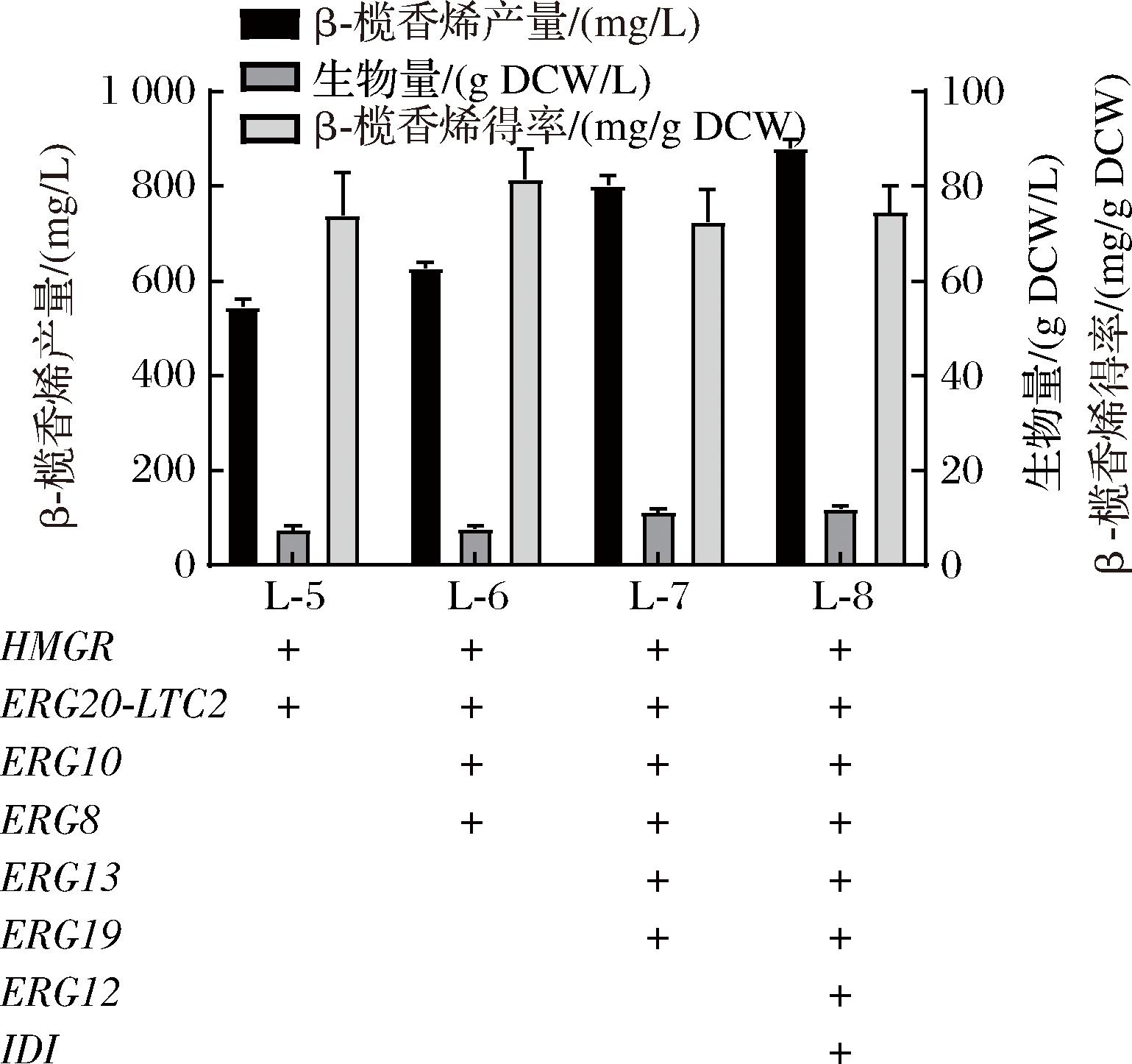
图4 强化MVA途径基因表达对β-榄香烯产量的影响
Fig.4 Effect of enhanced MVA pathway gene expression on β-elemene production
2.4 增加ERG20-LTC2拷贝数
研究表明,增加萜类合酶基因的拷贝数可增加前体的利用效率[23]。本研究在菌株L-8上增加ERG20-LTC2基因拷贝数构建了菌株L-9。发酵72 h后,菌株L-9的β-榄香烯产量达到了(1 019.26±34.45) mg/L,较菌株L-8产量提高了15.6 %。在菌株L-9的基础上增加ERG20-LTC2拷贝得到菌株L-10,其β-榄香烯产量达到了(1 326.78±64.42) mg/L(图5)。
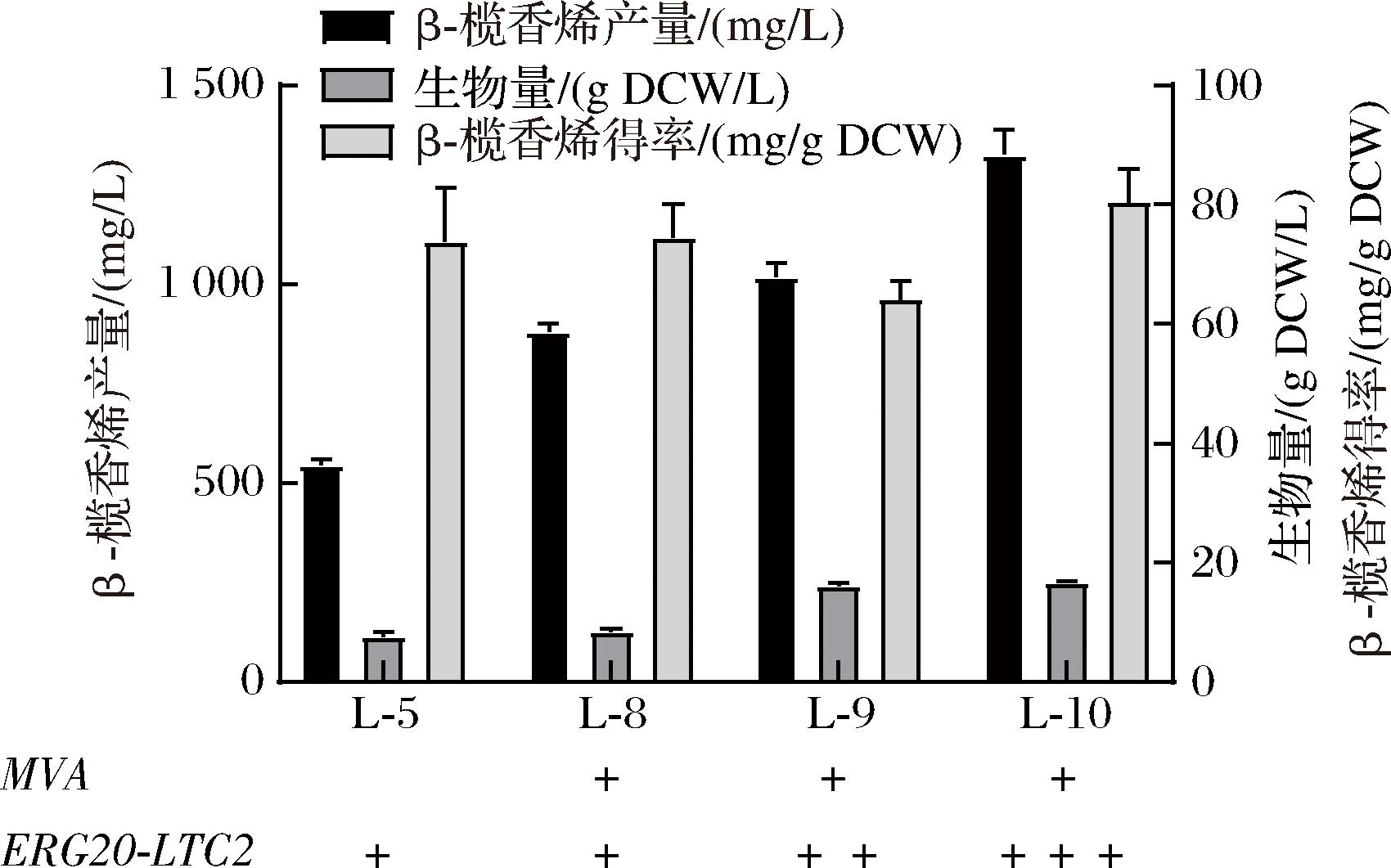
图5 增加基因融合的拷贝数对β-榄香烯产量的影响
Fig.5 Effect of increasing the copy number of gene fusions on β-elemene production
2.5 发酵培养基优化
为了进一步提高菌株L-10生成β-榄香烯产量,本研究通过单因素调整葡萄糖、胰蛋白胨和酵母提取物三者比例对发酵结果的影响。以YPD培养基作为基础培养基,以β-榄香烯产量作为评价标准,依次对培养基中的葡萄糖、胰蛋白胨和酵母提取物进行优化(图6)。进一步采取葡萄糖、酵母提取物和胰蛋白胨质量浓度分别为40、30、10 g/L的优化培养基,β-榄香烯最高产量为(1 668.13±41.01) mg/L。
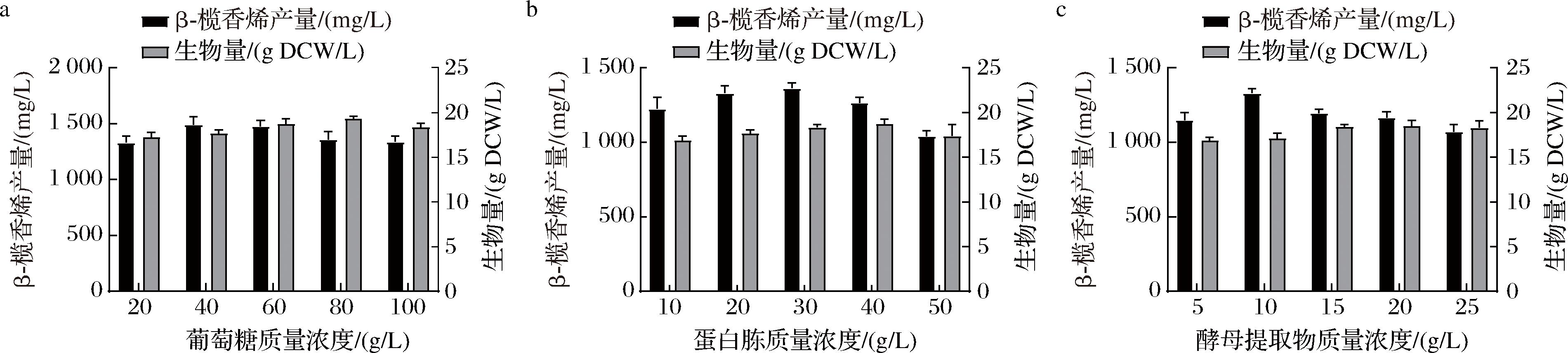
a-发酵初始葡萄糖质量浓度;b-发酵初始蛋白胨质量浓度;c-发酵初始酵母提取物质量浓度
图6 培养基中不同因素对β-榄香烯产量的影响
Fig.6 Effect of different factors in the culture medium on β-elemene production
3 结论与讨论
β-榄香烯具有广谱抗肿瘤活性和免疫保护特性,并且有广阔的应用前景。本研究中,采用基因融合表达可使β-榄香烯的产量升高,并且位置的差异对产量有较大影响,ERG20-LTC2相对位置对β-榄香烯的合成最有利,使FPP流量更多流入β-榄香烯的合成。进一步随机整合过表达MVA途径使β-榄香烯的合成更高效,产量达到(881.33±19.27) mg/L。同时,过表达ERG20-LTC2的拷贝数并回补氨基酸编码基因,使β-榄香烯的产量达到(1 326.78±64.42) mg/L。通过优化培养基,β-榄香烯产量达到(1 668.13±41.01) mg/L,是优化前的1.25倍。后续通过发酵罐扩大培养并动态调控培养过程,有望进一步提高β-榄香烯产量。本研究对解脂耶氏酵母生产β-榄香烯的工业化应用具有一定的参考价值。
[1] GOORÉ S G, OUATTARA Z A, YAPI T A, et al.Chemical composition of Ivorian Artabotrys insignis leaf oil.Combined analysis including 13C NMR, to quantify germacrene A and β-elemene[J].Natural Product Research, 2017, 31(15):1836-1839.
[2] ZHAI B T, ZHANG N N, HAN X M, et al.Molecular targets of β-elemene, a herbal extract used in traditional Chinese medicine, and its potential role in cancer therapy:A review[J].Biomedicine &Pharmacotherapy, 2019, 114:108812.
[3] GONG Z, LIU Z G, DU K Y, et al.RETRACTED:Potential of β-elemene induced ferroptosis through Pole2-mediated p53 and PI3K/AKT signaling in lung cancer cells[J].Chemico-Biological Interactions, 2022, 365:110088.
[4] XIE Q, LI F Z, FANG L, et al.The antitumor efficacy of β-elemene by changing tumor inflammatory environment and tumor microenvironment[J].BioMed Research International, 2020, 2020(1):6892961.
[5] KIM D, LEE J, CHANG J, et al.Stereoselective synthesis of (±)-β-elemene by a doubly diastereodifferentiating internal alkylation:A remarkable difference in the rate of enolization between syn and anti esters[J].Tetrahedron, 2001, 57(7):1247-1252.
[6] CHEN M W, ZHANG J M, YU S Q, et al.Anti-lung-cancer activity and liposome-based delivery systems of β-elemene[J].Evidence-Based Complementary and Alternative Medicine, 2012, 2012(1):259523.
[7] EDRIS A E.Anti-cancer properties of Nigella spp.essential oils and their major constituents, thymoquinone and beta-elemene[J].Current Clinical Pharmacology, 2009, 4(1):43-46.
[8] CHEN X F, HUANG C, LI K L, et al.Recent advances in biosynthesis and pharmacology of β-elemene[J].Phytochemistry Reviews,2023, 22(1):169-186.
[9] SCHEMPP F, DRUMMOND L, BUCHHAUPT M, et al.Microbial cell factories for the production of terpenoid flavor and fragrance compounds[J].Journal of Agricultural and Food Chemistry, 2018, 66(10):2247-2258.
[10] BUTION M L, MOLINA G, ABRAH O M R E, et al.Genetic and metabolic engineering of microorganisms for the development of new flavor compounds from terpenic substrates[J].Critical Reviews in Biotechnology, 2015, 35(3):313-325.
O M R E, et al.Genetic and metabolic engineering of microorganisms for the development of new flavor compounds from terpenic substrates[J].Critical Reviews in Biotechnology, 2015, 35(3):313-325.
[11] MUHAMMAD A, FENG X D, RASOOL A, et al.Production of plant natural products through engineered Yarrowia lipolytica[J].Biotechnology Advances, 2020, 43:107555.
[12] LI Z J, WANG Y Z, WANG L R, et al.Advanced strategies for the synthesis of terpenoids in Yarrowia lipolytica[J].Journal of Agricultural and Food Chemistry, 2021, 69(8):2367-2381.
[13] XU X H, LIU Y F, DU G C, et al.Microbial chassis development for natural product biosynthesis[J].Trends in Biotechnology, 2020, 38(7):779-796.
[14] LIU S C, XU L X, SUN Y J, et al.Progress in the metabolic engineering of Yarrowia lipolytica for the synthesis of terpenes[J].Biodesign Research, 2024, 6:0051.
[15] LU Y H, ZHANG X Y, WANG F, et al.Optimization of germacrene a synthase for efficient production of β-elemene in Escherichia coli[J].ACS Sustainable Chemistry &Engineering, 2024, 12(24):9018-9026.
[16] HU Y T, ZHOU Y J, BAO J C, et al.Metabolic engineering of Saccharomyces cerevisiae for production of germacrene A, a precursor of beta-elemene[J].Journal of Industrial Microbiology &Biotechnology, 2017, 44(7):1065-1072.
[17] ZHANG W X, GUO J Q, WANG Z, et al.Improved production of germacrene A, a direct precursor of β-elemene, in engineered Saccharomyces cerevisiae by expressing a cyanobacterial germacrene A synthase[J].Microbial Cell Factories, 2021, 20(1):7.
[18] HU Y H, ZHANG Q, BAI X, et al.Screening and modification of (+)-germacrene A synthase for the production of the anti-tumor drug (-)-β-elemene in engineered Saccharomyces cerevisiae[J].International Journal of Biological Macromolecules, 2024, 279(Pt 4):135455.
[19] CHENG J T, ZUO Y M, LIU G F, et al.Development of a Pichia pastoris cell factory for efficient production of germacrene A:A precursor of β-elemene[J].Bioresources and Bioprocessing, 2023, 10(1):38.
[20] YE M, GAO J Q, ZHOU Y J.Global metabolic rewiring of the nonconventional yeast Ogataea polymorpha for biosynthesis of the sesquiterpenoid β-elemene[J].Metabolic Engineering, 2023, 76:225-231.
[21] YE M, GAO J Q, LI J J, et al.Promoter engineering enables precise metabolic regulation towards efficient β-elemene production in Ogataea polymorpha[J].Synthetic and Systems Biotechnology, 2024, 9(2):234-241.
[22] LI W J, MAI J, LIN L, et al.Combination of microbial and chemical synthesis for the sustainable production of β‐elemene, a promising plant-extracted anticancer compound[J].Biotechnology and Bioengineering, 2023, 120(12):3612-3621.
[23] LIU Q, ZHANG G, SU L Q, et al.Reprogramming the metabolism of oleaginous yeast for sustainably biosynthesizing the anticarcinogen precursor germacrene A[J].Green Chemistry, 2023, 25(20):7988-7997.
[24] LIU M S, WU J J, YUE M Y, et al.YaliCMulti and YaliHMulti:Stable, efficient multi-copy integration tools for engineering Yarrowia lipolytica[J].Metabolic Engineering, 2024, 82:29-40.
[25] KANG W, MA T, LIU M, et al.Modular enzyme assembly for enhanced cascade biocatalysis and metabolic flux[J].Nature Communications, 2019, 10:4248.