益生菌是指一类摄入足够数量时能够对宿主产生有益作用的活的微生物[1],因其能够调节人体肠道菌群平衡、提高免疫力等功能特性而被添加到食品中[2]。益生菌产品的活菌数是衡量其功效的重要参数。为满足消费者不同的健康需求,市场上广泛推出添加多菌种的复合益生菌产品。然而,对复合益生菌产品中的特定菌种进行靶向活菌定量是当前行业亟待解决的技术难题。因此,开发新的检测技术,实现复合益生菌产品中目标微生物的快速靶向活菌定量,已成为行业内的迫切需求。叠氮溴化丙锭(propidium monoazide,PMA),在强光照射下,可穿过受损的细胞膜与胞内的DNA发生不可逆的共价交联反应,抑制死细胞的DNA在荧光定量PCR(quantitative PCR,qPCR)中的扩增。活细胞具有完整的细胞膜,PMA无法进入胞内与DNA结合,将PMA与qPCR结合,可有效区分死活菌[3-5]。PMA-qPCR方法能够快速且特异性地检测目标菌种,实现活菌的定量分析;此外,该方法还能够识别处于活性但不可培养状态(viable but non-culturable,VBNC)的菌株,提供更准确的活菌数检测结果[6]。
在发酵乳制品领域中,PMA-qPCR方法能够快速、特异性地对商业发酵乳中的活乳酸菌数(植物乳植杆菌、瑞士乳杆菌、乳酸乳球菌等)进行定量检测[7]。PMA-qPCR方法也被应用于检测不同类型奶酪成熟过程中益生菌的存活情况,以揭示奶酪商业化生产中菌种的存活率和优势细菌种类[8]。GUO等[9]经过PMA处理条件的确认、系统的方法学验证和适用性研究,发现PMA-qPCR方法能够快速且特异性的检测复合益生菌固体饮料中的活性鼠李糖乳酪杆菌(Lacticaseibacillus rhamnosus)活菌数,为复合益生菌固体饮料中活性L.rhamnosus的数量的检测提供新的技术支撑。
副干酪乳酪杆菌(Lacticaseibacillus paracasei)广泛存在于人体的口腔、肠道之中,被收录于中国《可用于食品的菌种名单》、欧盟QPS(qualified presumption of safety)名单和美国GRAS(generally recognized as safe)中,具有较大的市场应用价值[10-11]。目前有多株商业化菌株如K56、ET-22、Zhang等广泛应用于乳制品、固体饮料、保健食品等产品[10]。L.paracasei能够促进人体肠道微生态系统的平衡稳定;调节宿主免疫系统平衡、提高宿主免疫力;且具有抗氧化作用和抑菌特性[12-16]。建立益生菌产品中L.paracasei的活菌定量检测方法,对食品行业产品质量控制、市场监管以及消费者权益保护具有重要意义。
DNA提取的效率是影响定量检测准确性的关键因素。不同的DNA提取方法所提取的DNA浓度、纯度、完整性不同,这些差异会影响定量检测的准确性。不同菌种的细胞膜和结构存在差异,导致PMA的渗透能力不同。为了确保PMA-qPCR方法的准确性和可靠性,需要针对目标菌种优化PMA前处理条件。本研究以L.paracasei为研究对象,系统比较了常用的DNA提取方法提取的DNA的效率,并优化了PMA前处理条件等关键参数,建立了L.paracasei活菌定量的PMA-qPCR方法;通过收集代表性益生菌产品进行方法适用性研究,结果表明该方法用于检测复杂菌种体系中目标菌种活菌数具有显著优势,为此方法应用于复合益生菌产品靶向活菌数定量检测奠定研究基础。
1 材料与方法
1.1 材料与试剂
L.paracasei CICC 6263T、L.rhamnosus CICC 6224T、干酪乳酪杆菌(Lacticaseibacillus casei)CICC 6117T,中国工业微生物菌种保藏中心;MRS琼脂培养基,北京陆桥技术股份有限公司;DNA提取试剂盒,天根生化科技有限公司;PMA,北京百瑞极生物科技有限公司;TB GreenTM Premix Ex TaqTM,宝日医生物技术(北京)有限公司。
1.2 仪器与设备
LX-165T2R台式冷冻离心机,海尔集团公司;Thermo scientific Multiskan FC酶标仪,Thermo Fisher Scientific公司;PT-H18A LED光敏仪,Biotium公司;Bag mixer 400cc均质器,interscience公司;Bead Ruptor 12破碎仪,OMNI公司;Votex 2000涡旋振荡仪,Votex公司;7500定量PCR仪,ABI公司。
1.3 实验方法
1.3.1 特异性引物的合成
L.paracasei的qPCR特异性引物由生工生物工程(上海)股份有限公司合成。引物的正向和反向序列分别为:5′-ACGCTGGCATCAATAAGGAATT-3′;5′-CATCGCTCAGGTCTACATCCA-3′[17];产物大小为180 bp。
1.3.2 基因组DNA提取方法效率对比
DNA提取效率是影响qPCR实验定量准确性的关键。常用的DNA提取方法主要有试剂盒提取法[18]、珠式研磨仪破碎法[7-8]、液氮-沸水浴反复冻融法[19]、涡旋振荡法[20]。本研究通过比较不同方法提取的DNA的质量与提取效率,选取DNA提取质量和效率较高的方法用于后续实验,确保定量实验检测的准确性。取500 μL菌液(菌液浓度为108 CFU/mL)于12 000 r/min离心10 min。弃上清液,加入200 μL的ddH2O重悬。将重悬后的菌液采用表1中7种方法进行DNA提取,所得DNA样本用于琼脂糖凝胶电泳实验和qPCR实验,同时测量DNA的浓度和纯度。
表1 7种常用的DNA提取方法
Table 1 7 common DNA extraction methods

方法编号菌体DNA提取方法1珠式研磨仪破碎法(于盛装有0.25 g粒径0.1 mm玻璃珠的无菌研磨管中破碎,速度6 m/s,时间12 s)2珠式研磨仪破碎后(同方法1),利用试剂盒提取DNA3珠式研磨仪破碎后(同方法1),利用液氮-沸水浴(沸水浴1~3 min)反复冻融3个循环4涡旋振荡仪振荡2 min5涡旋振荡仪振荡2 min,液氮-沸水浴(沸水浴1~3 min)反复冻融3个循环6液氮-沸水浴(沸水浴1~3 min)反复冻融3个循环7溶菌酶处理后,利用试剂盒提取DNA
1.3.3 PMA作用条件优化
PMA作用条件是影响活菌定量准确性的关键因素。为有效区分死活菌,精准检测L.paracasei的活菌数,避免出现假阳性、假阴性结果,本实验通过优化PMA浓度、暗孵育时间以及曝光时长,确定L.paracasei的最佳PMA处理条件。
1.3.3.1 最佳热致死时间确认
为设立死菌对照组,建立有效区分死活菌的PMA最佳处理条件,设计本实验确认L.paracasei的最佳热致死时间。
挑取37 ℃培养48 h的L.paracasei于0.85%(质量分数)的无菌生理盐水中,调节菌液的OD620值为0.3~0.5。参考HU等[21]和GOBERT等[22]的方法,在90 ℃下,制备L.paracasei死菌菌悬液。将上述活菌菌液置于90 ℃的水浴锅中,分别加热5、10、15、20 min;未经加热的菌液作为对照组。将加热后的菌液分为2组,一组加入PMA,另一组加入等体积生理盐水;并采用平板计数法测定加热后的菌液的活菌数。提取2组菌液的DNA,进行qPCR实验,确定最佳热致死时间。后续实验以最佳热致死时间对菌液进行热处理,制备死菌菌悬液。
1.3.3.2 PMA浓度、暗孵育时间、曝光时间优化
将98 μL的无菌ddH2O加入1 mg的PMA粉末中,配制为20 mmol/L的PMA原液,置于-20 ℃避光保存。
按照1.3.3.1节制备活菌菌悬液和死菌菌悬液体系。取500 μL活菌和死菌菌悬液,加入PMA,使PMA的终浓度为10、30、50、70、90 μmol/L,置于黑暗避光条件下孵育0、5、10、15、20 min,每1 min混匀一次。孵育完成后,置于LED光敏仪下曝光0、5、10、15、20 min。采用1.3.2节所确认的最佳DNA提取方法,提取DNA,进行qPCR实验,根据qPCR实验结果,确定最佳PMA处理条件。
1.3.4 qPCR反应体系及条件
qPCR反应体系:SYBR qPCR mix 10 μL,正、反向引物各0.4 μL,50×Rox 0.08 μL,DNA模板2 μL,加无菌ddH2O补足体积至20 μL。
qPCR反应条件:第一阶段:95 ℃/30 s,1个循环;第二阶段:95 ℃/5 s,60 ℃/34 s,40个循环。
1.3.5 qPCR标准曲线的建立及定量限确认
qPCR实验获取的直接数据Ct值,无法直观的表明样品中的活菌数,因此需建立活菌数与Ct值的标准曲线。通过标准曲线的建立,qPCR实验所得Ct值可通过标准曲线方程转化为活菌数,准确表征样品中的活菌数。
按1.3.3.1节,制备活菌菌悬液。将菌液分为2组,一组采用平板计数法进行活菌计数,另一组菌液进行PMA前处理。前处理完成后,按照1.3.2节的所确认的最佳DNA提取方法提取DNA。将获得的DNA粗提液进行10倍梯度稀释,稀释至10-5。通过qPCR实验测定每个稀释度的DNA所对应的Ct值。以菌液浓度的对数值为x轴,Ct值为y轴,建立标准曲线。qPCR实验的扩增效率的计算如公式(1)所示:
E=10-1/s-1
(1)
式中:E,扩增效率,%;s,标准曲线的斜率。
PMA-qPCR方法是一种应用于微生物检测领域的活菌定量检测方法。因此,确立该方法的定量限,对确保其在实际检测中的准确性具有重要意义。本研究配制了含有L.paracasei和其他非目标菌的混合菌液,其中L.paracasei的理论添加量分别为1.46×104、7.30×103、3.65×103 CFU/mL,非目标菌的浓度约为108 CFU/mL。通过PMA-qPCR方法对菌液中的目标菌种活菌数进行检测,确认方法定量限。
1.3.6 方法特异性验证
为进一步验证所建立的方法能够在非目标菌种的干扰下,特异性检出目标菌种并对其准确定量,本研究选取了与L.paracasei分类学地位较近的L.rhamnosus和L.casei,配制了3种类型的菌液。第1种类型菌液:将浓度均为108 CFU/mL的L.paracasei、L.rhamnosus、L.casei菌液等比例混合。第2种类型的菌液:将浓度为108 CFU/mL的L.paracasei菌悬液与生理盐水混合,使此菌液的L.paracasei的浓度与第1种类型菌液的L.paracasei浓度相同。第3种类型菌液:将浓度均为108 CFU/mL的L.rhamnosus、L.casei菌液等比例混合。采用PMA-qPCR方法检测3种类型的菌液中L.paracasei的活菌数。
1.3.7 PMA-qPCR方法适用性
本研究共收集了11种代表性益生菌产品用于方法适用性研究,其中包括3种仅添加L.paracasei的菌粉原料,4种标示L.paracasei添加量的复合型益生菌产品,以及4种未标示L.paracasei添加量的复合型益生菌产品。在复合型益生菌产品中,部分样品添加了与L.paracasei分类学地位较为接近的L.rhamnosus和L.casei。此外,相较于单一菌粉原料,复合型产品另外添加了低聚果糖、聚葡萄糖、山楂粉、牛乳粉等辅料。所选样品包括单一菌种和复合菌种,并涵盖了市场上不同标签标识的益生菌产品,具有较强的代表性,能够全面反映当前市场上益生菌产品的多样性及标签标识现状。
将菌粉原料和无标示总菌体量的复合益生菌产品梯度稀释,调节菌液的OD620值为0.3~0.5(菌液浓度为108 CFU/mL),将标示总菌体添加量的复合益生菌产品按照标签标识梯度稀释至总菌体浓度为108 CFU/mL。对于菌粉原料样品,分别采用平板计数法和PMA-qPCR方法检测L.paracasei活菌数。对于复合型益生菌产品,采用所建立的PMA-qPCR方法检测活性L.paracasei的数量。参考ISO 16140 《Microbiology of the food chain》第3部分,比较PMA-qPCR检测结果与平板计数结果或标签声称量的差异是否在可接受的限度(±0.5 lg CFU/g)范围内。
2 结果与分析
2.1 DNA提取方法确定
通过对7种不同方法提取得到的DNA的浓度和纯度进行测量(表2)并结合凝胶电泳实验结果(图1),本研究发现珠式研磨仪破碎结合试剂盒法以及液氮-沸水浴反复冻融法提取的DNA浓度较低、完整性较差,不适用于后续的实验检测;试剂盒(溶菌酶前处理)法提取的DNA完整性较好,但浓度较低,可能是在DNA纯化步骤中造成了DNA的损失[23-25];涡旋振荡法提取的DNA浓度较高,纯度相对较好,但在实验过程中,需要人为对菌液进行涡旋振荡,实验过程中操作的不稳定性可能会影响DNA提取效率;珠式研磨仪破碎法以及珠式研磨仪破碎结合液氮-沸水浴反复冻融法提取DNA的浓度较高,纯度较好,通过qPCR实验的进一步研究表明(图2),珠式研磨仪破碎法的Ct值最低,引物可特异性结合目的基因片段,在qPCR实验中进行有效的扩增。基于此实验结果,选取珠式研磨仪破碎法作为本研究的DNA提取方法。
表2 7种方法提取的DNA样品浓度与纯度对比
Table 2 Comparison of concentration and purity of DNA samples extracted by 7 methods

方法编号菌体提取DNA方法DNA质量浓度/(ng/mL)A260/A230A260/A2801珠式研磨仪破碎102.00±2.831.10±0.021.94±0.022珠式研磨仪破碎后(同方法1),利用试剂盒提取DNA61.50±14.852.86±0.762.02±0.033珠式研磨仪破碎后(同方法1),利用液氮-沸水浴反复冻融3个循环144.00±4.241.26±0.032.04±0.004涡旋振荡仪振荡119.50±0.711.13±0.011.96±0.015涡旋振荡仪振荡,液氮-沸水浴反复冻融3个循环127.00±11.311.23±0.032.02±0.006液氮-沸水浴反复冻融3个循环45.50±0.711.69±0.032.22±0.047溶菌酶处理后,利用试剂盒提取DNA24.50±3.548.20±3.961.88±0.07
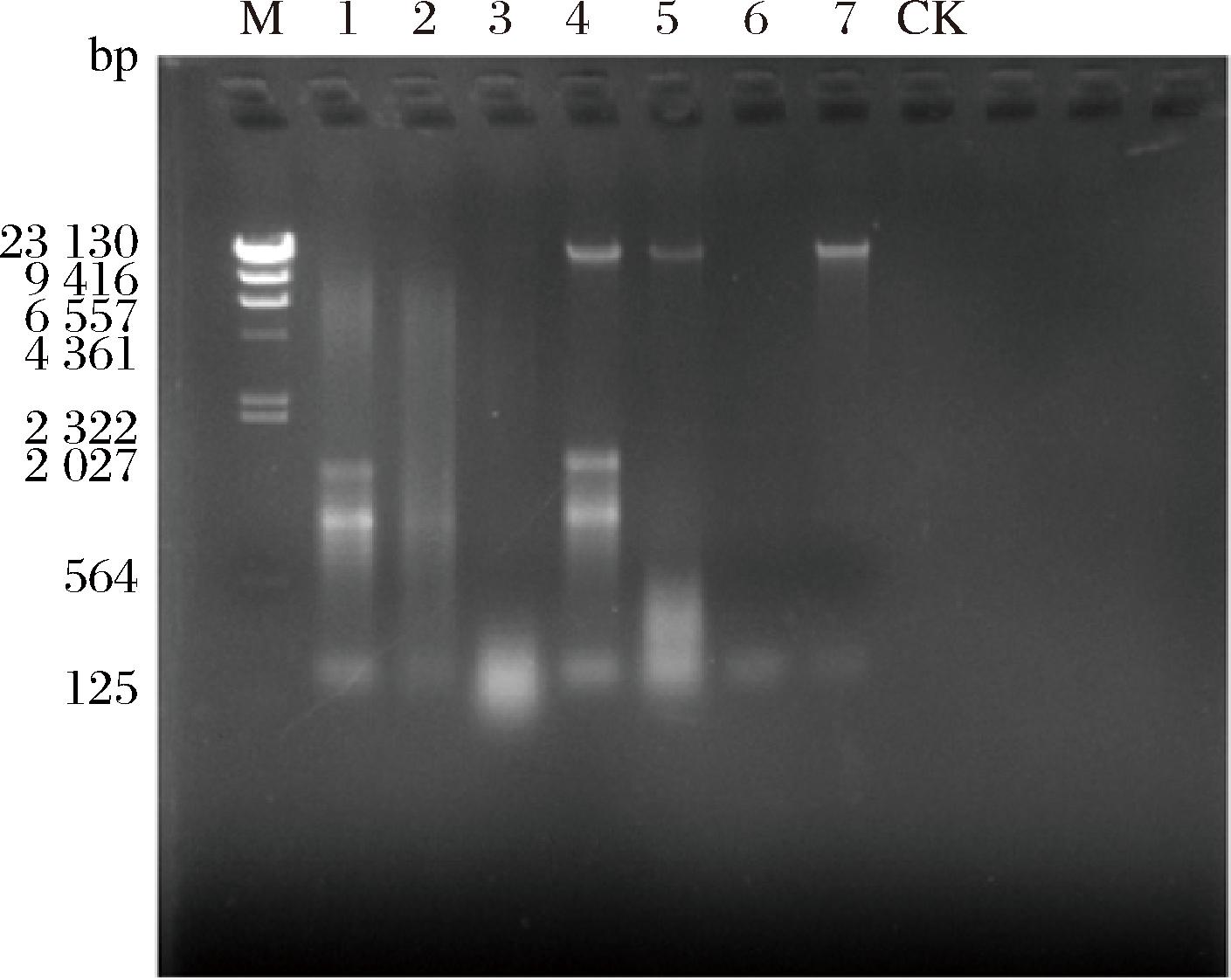
图1 7种方法提取的DNA样品的琼脂糖凝胶电泳图
Fig.1 Agarose gel electrophoresis of DNA samples extracted by 7 methods
注:M:DNA Marker;1:珠式研磨仪破碎法;2:珠式研磨仪破碎结合试剂盒法;3:珠式研磨仪破碎结合液氮-沸水浴反复冻融法;4:涡旋振荡法;5:涡旋振荡结合液氮-沸水浴反复冻融法;6:液氮-沸水浴反复冻融法;7:试剂盒提取法(溶菌酶前处理);CK:阴性对照。
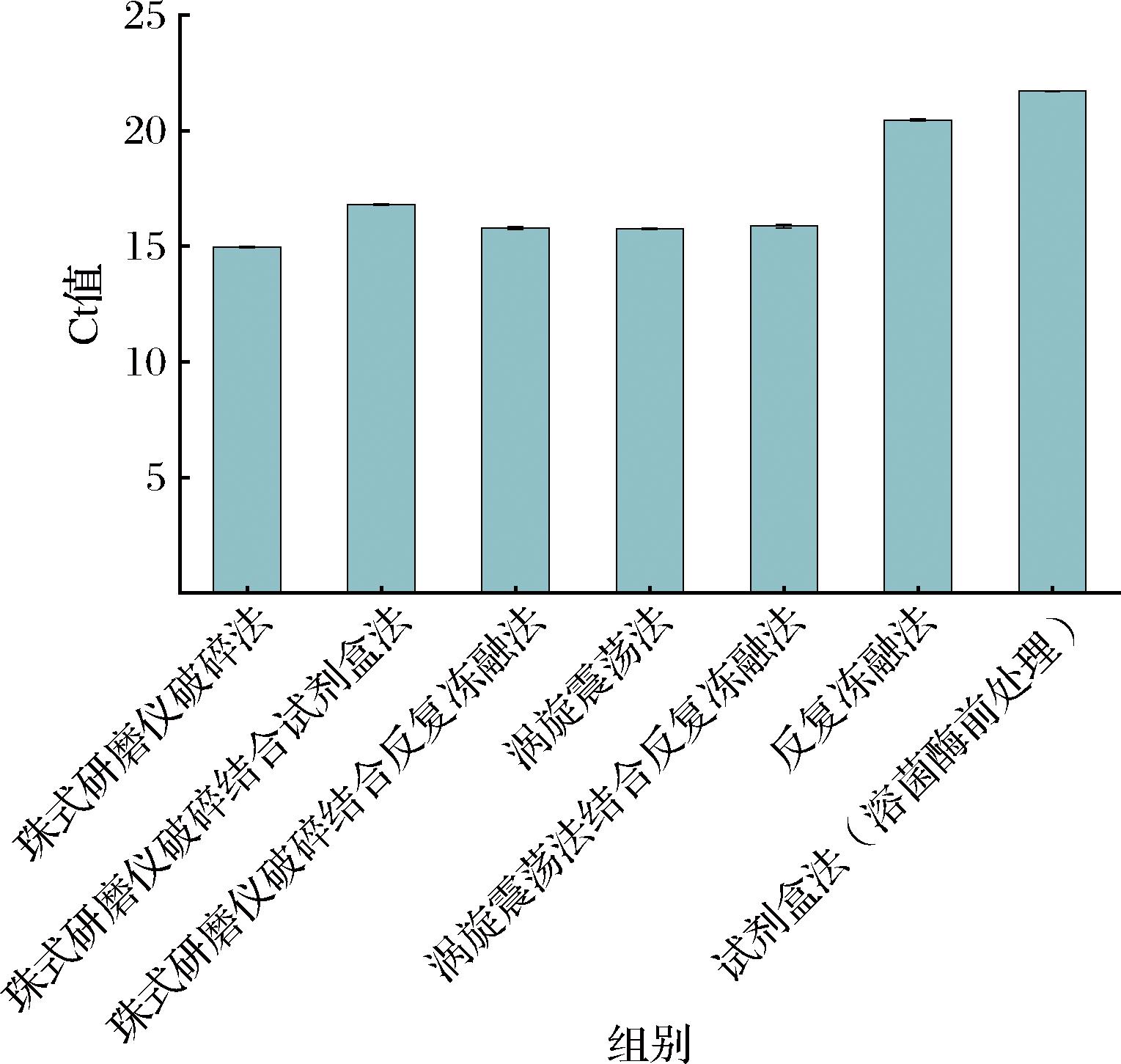
图2 7种方法提取的DNA样品的qPCR扩增结果
Fig.2 The qPCR amplification results of DNA samples extracted by 7 methods
为进一步提升珠式研磨仪破碎法的准确性,本研究对破碎时间进行优化,结果表明当破碎时间为8、10、12、14、16 s时,qPCR实验的Ct值分别为16.04±0.166、15.88±0.067、15.56±0.190、15.62±0.226、15.63±0.277。12~16 s的破碎时间下,qPCR的Ct值无显著性差异(P>0.05),说明破碎时间的微小变化并不会显著影响珠式研磨仪破碎法提取DNA的效率。当破碎时间为12 s时,qPCR实验的Ct值相对较小,DNA的提取效率相对较高,因此选择12 s为最佳破碎时间。采用珠式研磨仪,通过设置固定的速度和破碎时间提取DNA,确保了DNA提取过程的稳定性,珠式研磨仪破碎法相较于其他方法耗时短、易于操作,DNA提取效率较高。
2.2 PMA最佳作用条件确认
2.2.1 最佳致死时间
将热处理后的菌液涂布于平板上37 ℃厌氧培养48 h后,平板上均未有菌体长出,说明经过5、10、15、20 min的热处理菌体被灭活。如图3所示,经过5、10 min热处理后且不加PMA染料的菌液与未经热处理的菌液的Ct值无显著性差异(P>0.05);经过15、20 min热处理且不加PMA染料的菌液与未经热处理的菌液的Ct值有显著性差异(P<0.05),说明较长时间的热处理会影响DNA在qPCR实验中的扩增。经过5、10、15、20 min热处理,加入PMA染料的菌液的Ct值均大于30,与空白对照组Ct值大小相近,说明经过5、10、15、20 min的热处理,菌体均已死亡,DNA在qPCR实验的扩增被PMA有效抑制。后续实验中所检测的样品中有添加复杂基质(低聚果糖、全脂乳粉、山楂粉等)的复合型产品,为了后续实验更完全地致死菌液,建立有效的PMA前处理条件,选取10 min为最佳致死时间。
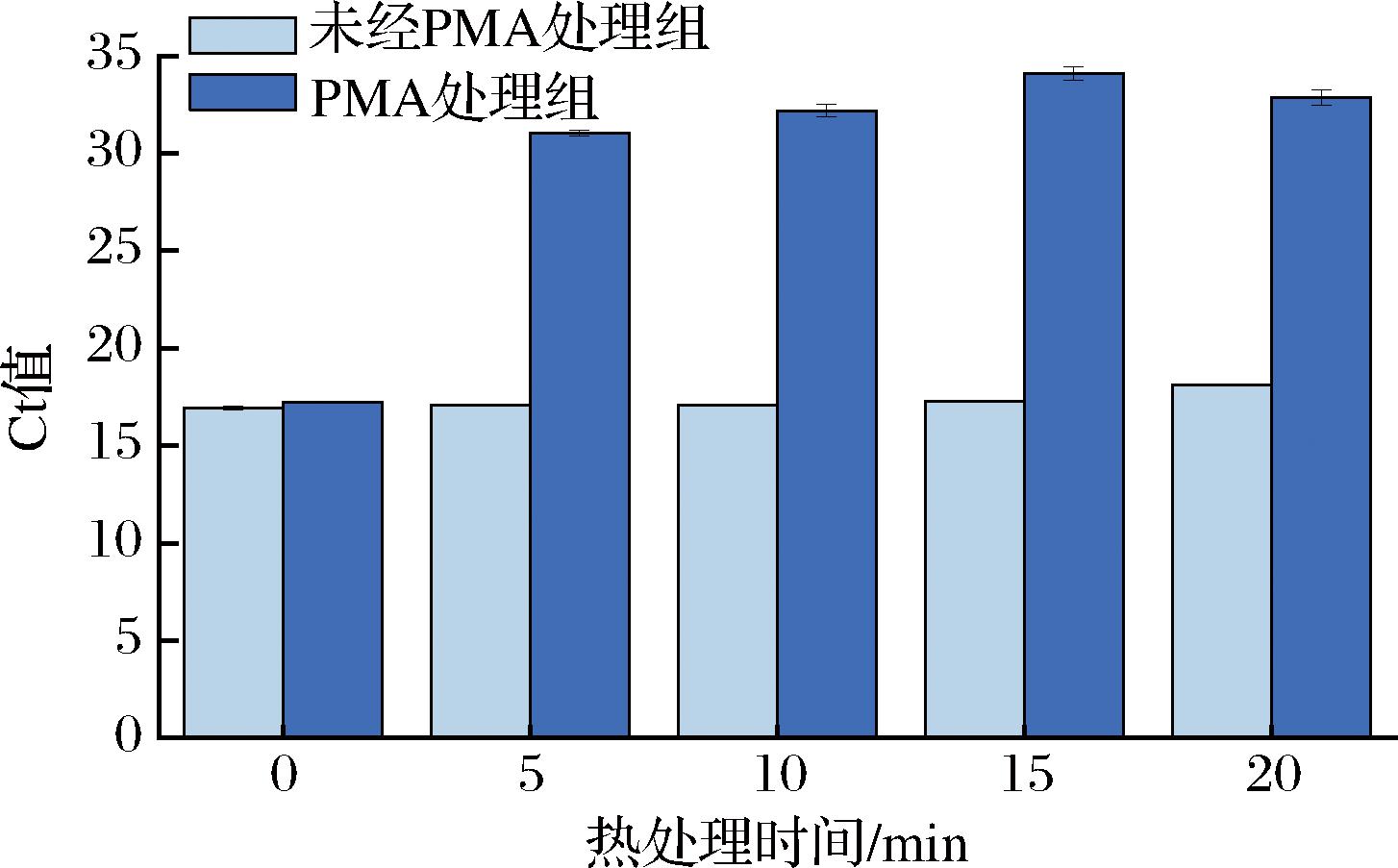
图3 热处理时间对qPCR扩增结果的影响
Fig.3 Effect of heat treatment time on the amplification results of qPCR
2.2.2 PMA浓度
图4结果表明,当PMA终浓度为10~90 μmol/L时,活菌菌液的Ct值无显著性差异(P>0.05),说明在此浓度范围内,PMA对于活菌DNA的扩增没有抑制作用;当PMA浓度为50 μmol/L时,热致死菌组与活菌组qPCR实验的Ct值的差值最大,此时PMA对死菌的DNA扩增的抑制作用最大;因此,选择50 μmol/L作为PMA添加至菌液中的最佳浓度。
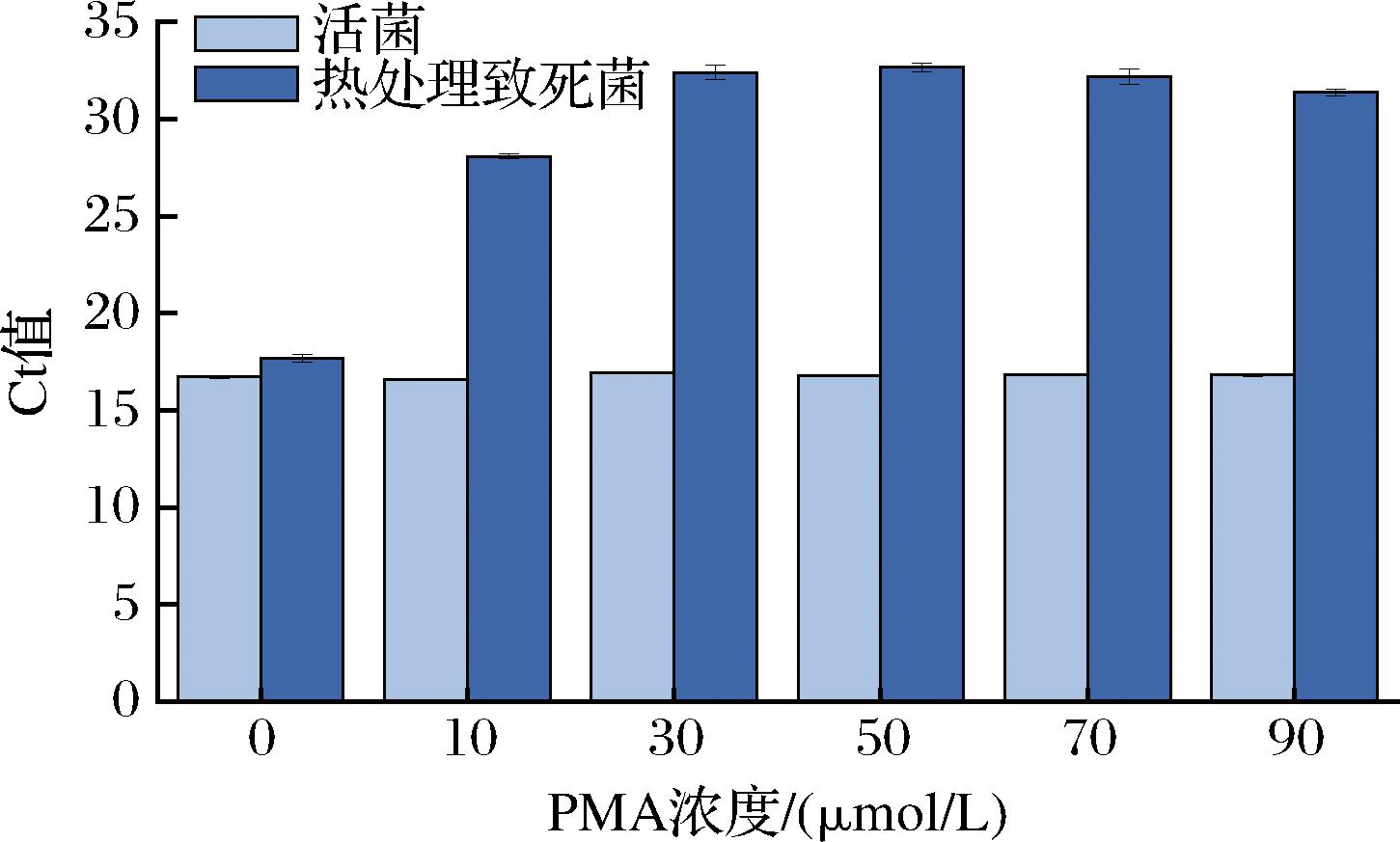
图4 PMA浓度对qPCR扩增结果的影响
Fig.4 Effect of PMA concentration on the amplification results of qPCR
2.2.3 暗孵育时间
添加PMA溶液至菌液中,使PMA的终浓度为50 μmol/L。经过不同时间暗孵育后,活菌组经过0~20 min的暗孵育后,qPCR实验的Ct值无显著性差异(P>0.05)(图5)。当死菌组加入PMA后,经过5、10、15、20 min的暗孵育,qPCR实验的Ct值均大于30(与空白对照组Ct值大小相近)。此外,暗孵育时间为5 min与暗孵育时间为10、15、20 min相比,死菌组与活菌组qPCR实验的Ct值的差值无显著性差异(P>0.05)。因此,暗孵育时间为5 min时,死菌 DNA的扩增已被有效抑制,选取5 min为最佳暗孵育时间。
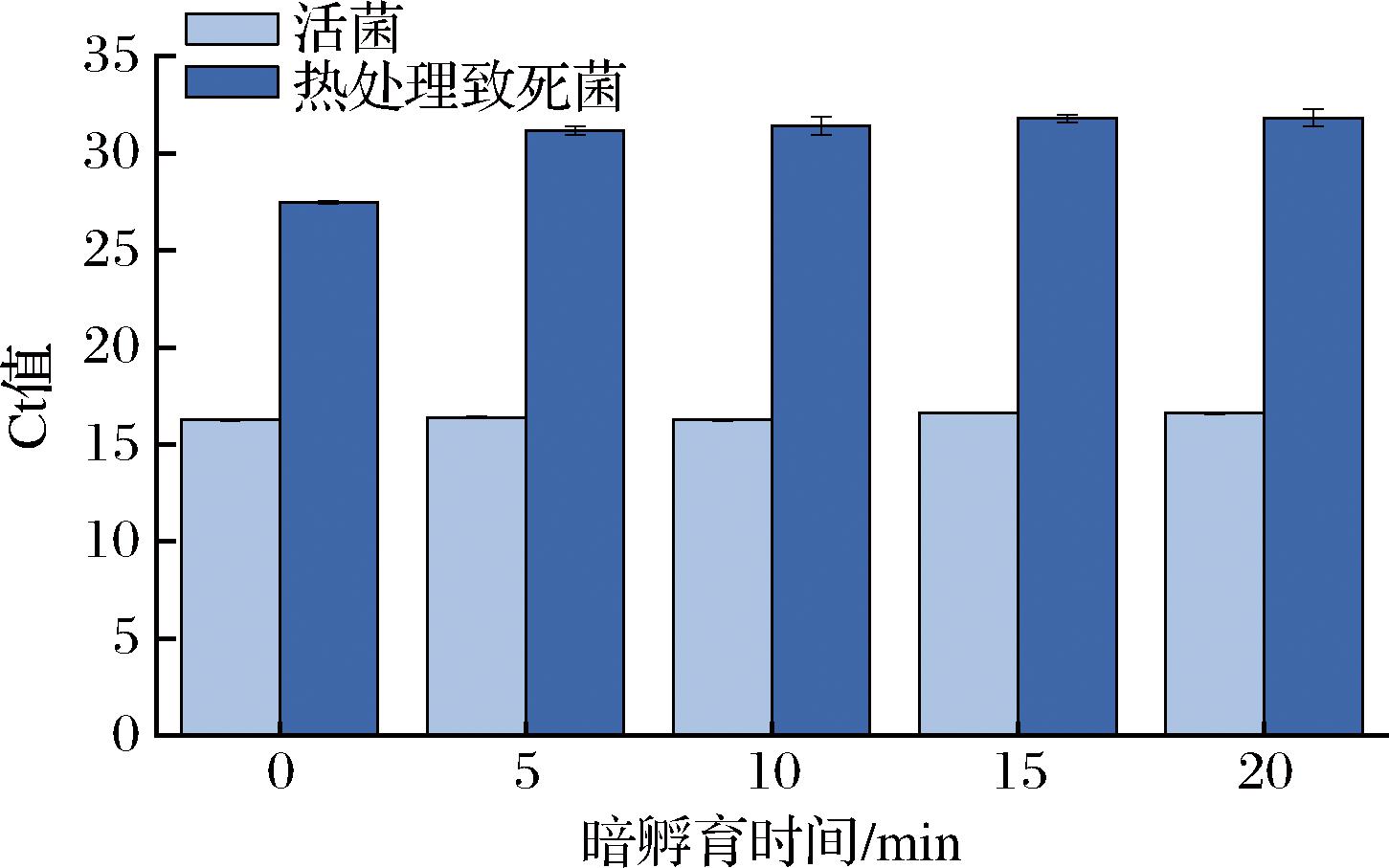
图5 暗孵育时间对qPCR扩增结果的影响
Fig.5 Effect of dark incubation time on the amplification results of qPCR
2.2.4 曝光时间
添加PMA溶液至菌液中,使PMA的终浓度为50 μmol/L,置于黑暗避光条件下孵育5 min,并将处理后的菌液置于LED光源上照射。图6结果表明,无LED光源照射时,PMA不能充分与死菌的DNA发生反应,无法有效抑制其扩增。当曝光时间为15、20 min时,死菌组和活菌组的Ct值的差值无显著性差异(P>0.05)。因此,当曝光时间为15 min时,进入死菌中的PMA能够充分与DNA发生共价交联反应,抑制死菌DNA的扩增。
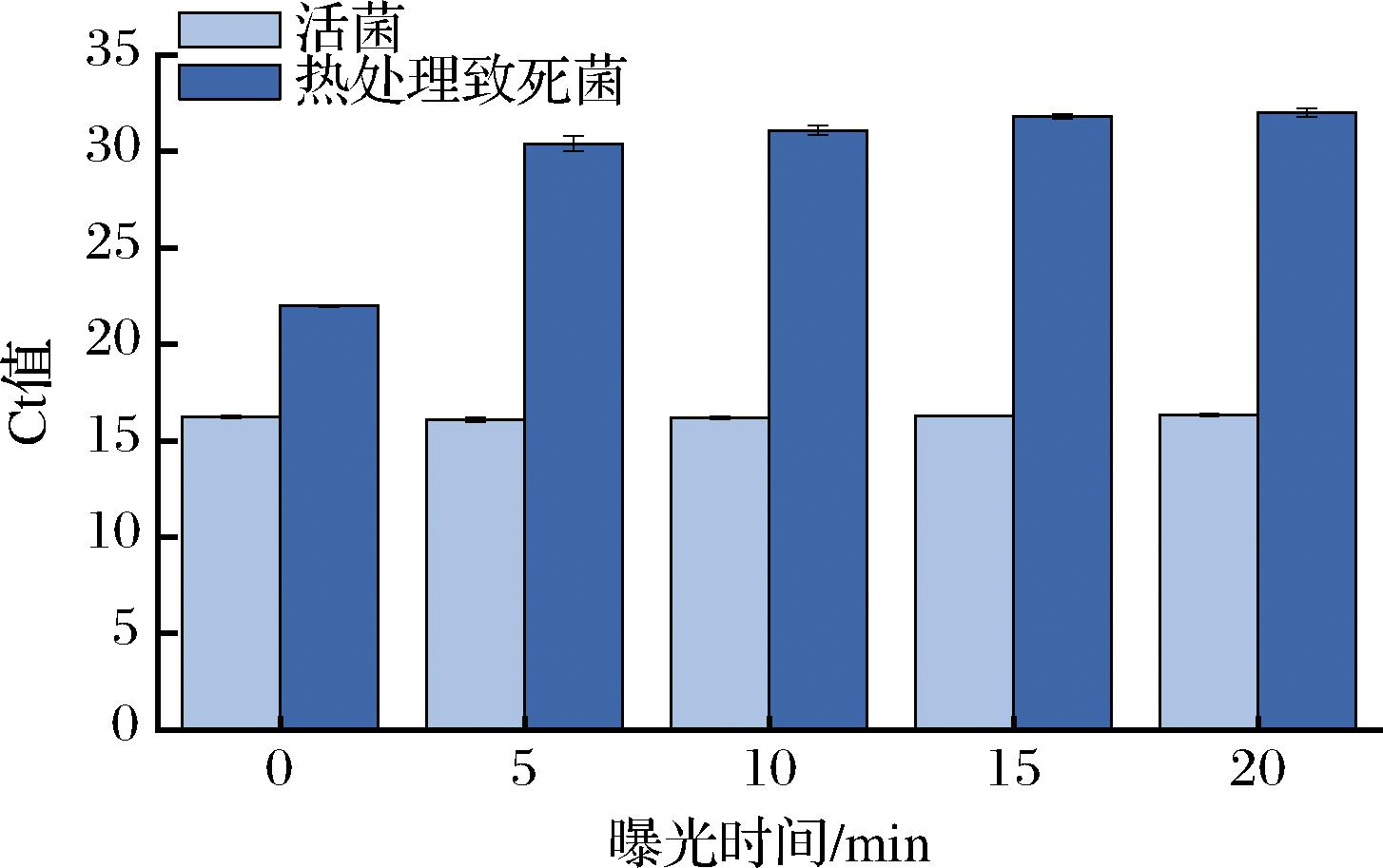
图6 曝光时间对qPCR扩增结果的影响
Fig.6 Effect of exposure time on the amplification results of qPCR
综上,确认本实验的最佳PMA作用条件为:当菌液中PMA终浓度为50 μmol/L,暗孵育5 min,曝光15 min时,可有效区分菌液中的死活菌,实现样品中活性L.paracasei的准确定量。PMA作用条件是基于菌体浓度为108 CFU/mL建立的,因此实验前需将样品稀释至此浓度范围。
2.3 qPCR标准曲线的建立及方法定量限确认
初始菌液的平板计数结果为6.75×108 CFU/mL。以菌落浓度的对数值为x轴,以qPCR实验获得的Ct值为y轴,建立标准曲线(图7)。菌落浓度与Ct值的回归方程为y=-3.403x+42.426,R2=0.998,通过公式计算得,引物的扩增效率为96.72%。所建立的标准曲线中引物的扩增效率在90%~110%之间,且熔解曲线为单一峰,符合qPCR实验要求;标准曲线的R2>0.98,表明菌落浓度与DNA扩增的Ct值之间具有良好的线性关系。利用此曲线,能够对产品中L.paracasei活菌数进行准确定量。
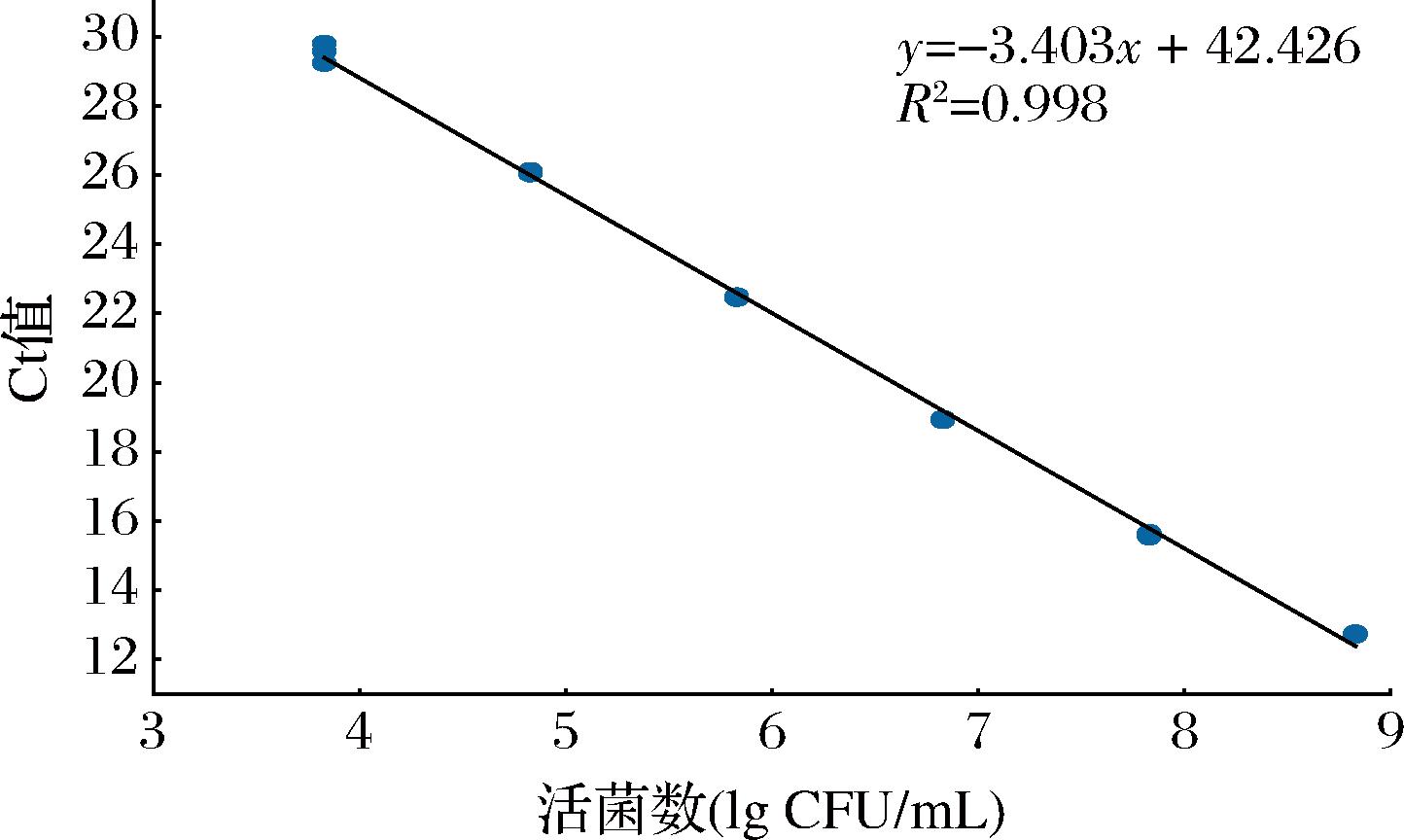
图7 标准曲线
Fig.7 Standard curve
如表3结果所示,混合菌悬液中L.paracasei的理论添加量为3.65×103 CFU/mL时,qPCR实验的Ct值>30,与空白对照的Ct值大小相近,会造成定量结果的不准确;当L.paracasei在菌液中的理论添加量>7.30×103 CFU/mL时,qPCR实验的Ct值<30。当混合菌悬液中L.paracasei的理论添加量为7.30×103 CFU/mL时,采用PMA-qPCR方法测定菌悬液中L.paracasei 的浓度为8.07×103 CFU/mL。因此确认该方法的定量限为8.07×103 CFU/mL。
表3 定量限结果
Table 3 The results of limit of quantification
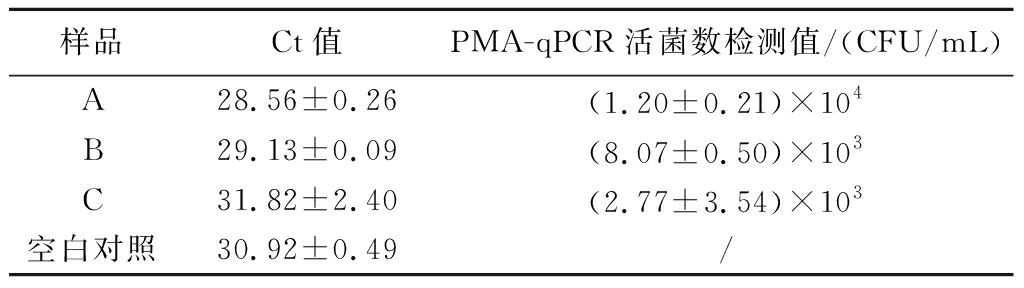
样品Ct值PMA-qPCR活菌数检测值/(CFU/mL)A28.56±0.26(1.20±0.21)×104B29.13±0.09(8.07±0.50)×103C31.82±2.40(2.77±3.54)×103空白对照30.92±0.49/
注:样品A、B、C分别代表L.paracasei的理论添加量为1.46×104、7.30×103、3.65×103 CFU/mL的混合菌悬液;/代表无数据(下同)。
2.4 方法特异性验证
PMA-qPCR方法对含有L.paracasei、L.rhamnosus和L.casei的混合菌液与仅含有L.paracasei单一菌种菌液的L.paracasei活菌数检测结果无显著性差异(P>0.05),样品L.rhamnosus、L.casei的混合菌液的qPCR试验的Ct值均大于30,与空白对照Ct值大小相近,表明菌液中未检出活性L.paracasei。上述实验结果表明该方法具有较好的L.paracasei特异性。在实际检测中,该方法可有效排除非目标菌的干扰,对活性L.paracasei准确定量。
表4 不同类型菌液的计数结果
Table 4 The counting results of different types of bacterial suspensions

菌液类型PMA-qPCR活菌计数结果/(CFU/mL)PMA-qPCR活菌计数结果(lg CFU/mL)P值L.paracasei、L.rhamnosus、L.casei的混合菌液(4.27±0.23)×1088.63±0.020.44L.paracasei单一菌种菌液(4.35±0.06)×1088.64±0.01L.rhamnosus、L.casei的混合菌液///
2.5 PMA-qPCR方法对益生菌原料及固体饮料产品的适用性
对添加了L.paracasei的3种菌粉原料(仅添加L.paracasei单一菌种)和8种复合益生菌产品采用所建立的PMA-qPCR方法进行计数,结果如表5和表6所示。
表5 菌粉原料中L.paracasei的活菌数检测结果
Table 5 Detection results of viable bacteria count of L.paracasei in probiotic powder

样品名称PMA-qPCR计数结果/(CFU/g)平板计数结果/(CFU/g)差值(lg CFU/g测定值-lg CFU/g平板计数结果)菌粉原料A5.92×10114.80×10110.09菌粉原料B3.67×10113.35×10110.04菌粉原料C3.27×10112.32×10110.15
表6 复合益生菌产品中L.paracasei的活菌数检测结果
Table 6 Detection results of viable bacteria count of L.paracasei in compound probiotic products
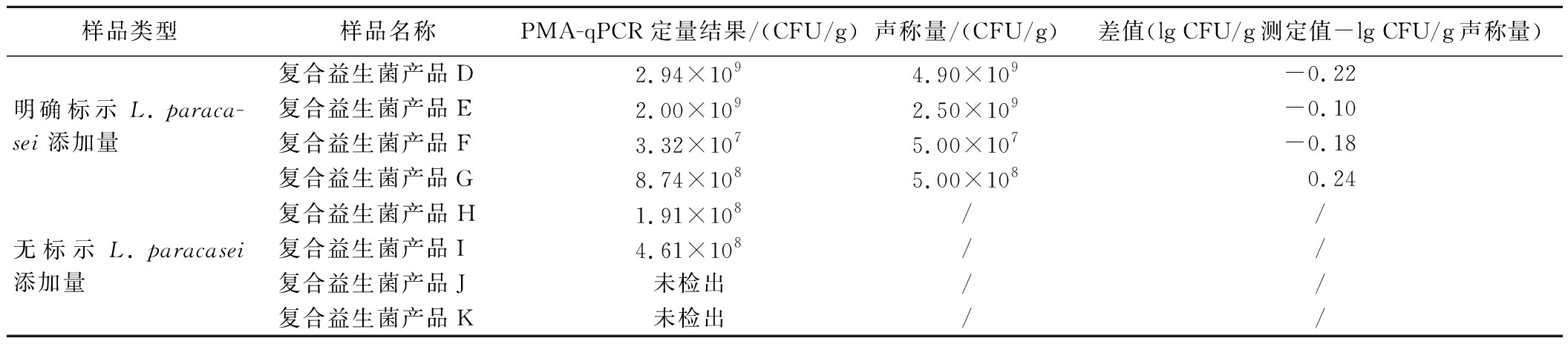
样品类型样品名称PMA-qPCR定量结果/(CFU/g)声称量/(CFU/g)差值(lg CFU/g测定值-lg CFU/g声称量)明确标示L.paraca-sei添加量复合益生菌产品D2.94×1094.90×109-0.22复合益生菌产品E2.00×1092.50×109-0.10 复合益生菌产品F3.32×1075.00×107-0.18复合益生菌产品G8.74×1085.00×1080.24无标示L.paracasei添加量复合益生菌产品H1.91×108//复合益生菌产品I4.61×108//复合益生菌产品J未检出//复合益生菌产品K未检出//
表5结果表明,对于菌粉原料,2种方法检测值的差值在±0.5 lg CFU/g内,检测结果的一致性较高。所建立的PMA-qPCR方法有较好的准确性和可靠性,可作为一种替代方法对样品中的L.paracasei活菌进行精准定量。
复合益生菌产品活菌数检测结果表明(表6),产品D、E、F、G的声称量与PMA-qPCR计数结果的差值在±0.5 lg CFU/g的范围内,检测结果与声称量的一致性较高,说明该方法的检测准确度较高,适用于复合型产品中L.paracasei的活菌数检测;产品H、I、J、K标签上均声称添加了L.paracasei但均未明确标示所添加的活性L.paracasei数量,其中产品H、I均检出了活性L.paracasei,且检出活性L.paracasei的数量>108 CFU/mL;产品J、K的菌液经过PMA前处理,qPCR实验的Ct值均大于30,与空白对照组的Ct值相近,推测其添加的L.paracasei在产品中处于非活性状态。
为进一步探究产品J、K中L.paracasei是否处于活性状态,将每个产品稀释后的菌液分为2组,一组进行PMA处理,另一组不进行PMA处理。随后进行qPCR实验,比较2组样品的Ct值。图8结果表明,2种 产品不经过PMA前处理时,其qPCR实验的Ct值均小于30,说明产品J、K中均存在少量的L.paracasei;2种产品经过PMA处理后,其qPCR实验的Ct值>30,说明2种产品中均未检出活性L.paracasei;通过对比2组实验结果,进一步表明2种产品中添加了微量L.paracasei,但添加的L.paracasei在产品中处于非活性状态。
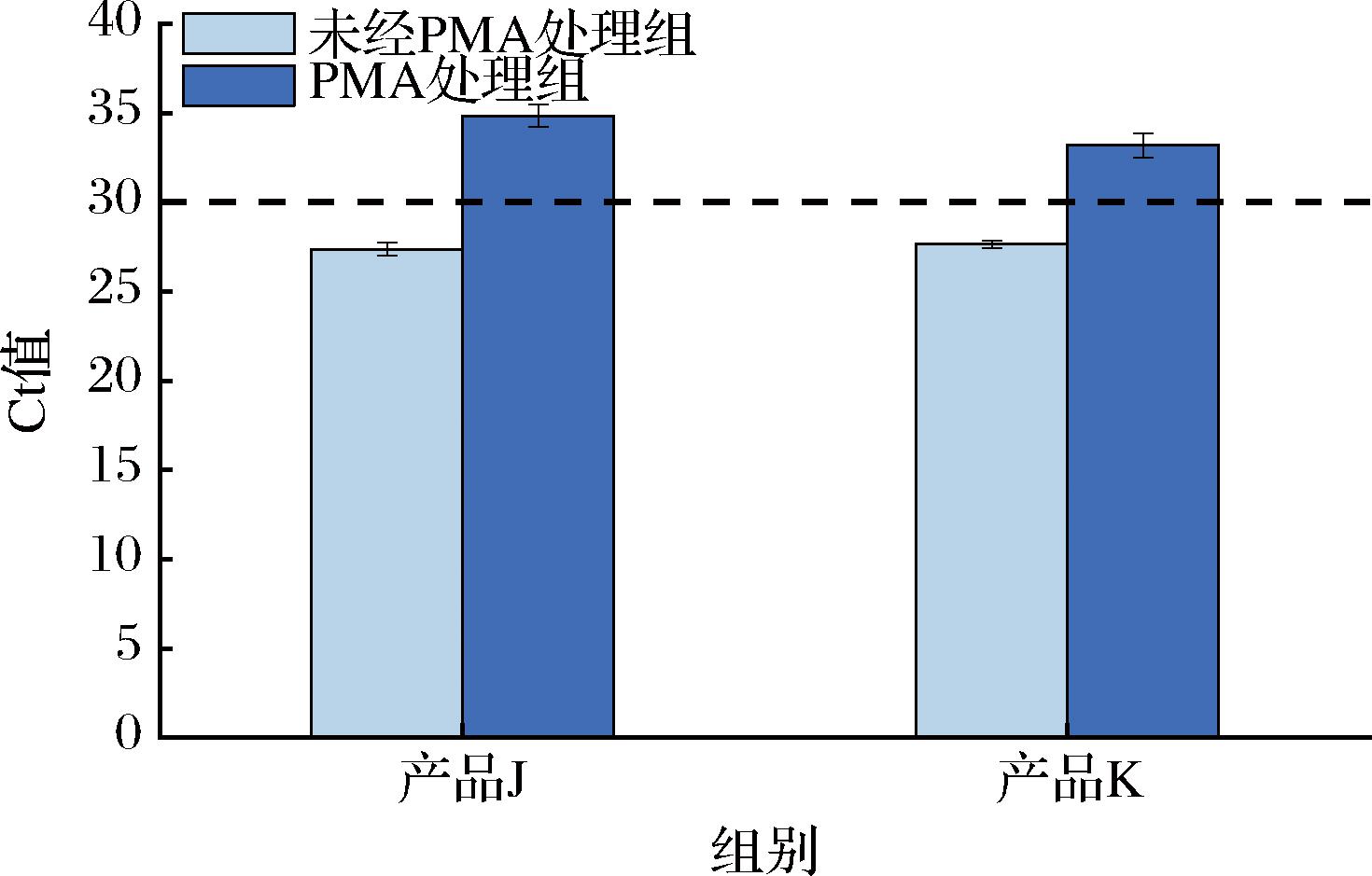
图8 产品J、产品K PMA处理组和未处理组qPCR实验结果
Fig.8 qPCR results of product J, product K PMA treated group and untreated group
与菌粉原料相比,复合型益生菌产品中的多菌种、多基质混合体系对定量检测方法的灵敏度和准确性提出了更高要求。通过对复合型产品的定量检测,本研究表明所建立的PMA-qPCR方法能够有效耐受复杂基质的干扰,特异性地检出活性L.paracasei,并进行精准定量。对于标示L.paracasei活菌添加量的产品,PMA-qPCR方法的测定值与声称量之间的差异位于可接受范围内,表明该方法具有较高的准确性;对于未标示添加量的复合型益生菌产品,PMA-qPCR方法能够有效检测产品中是否存在活性L.paracasei,并精准定量其活菌数,表明该方法在产品质量评估中具有重要应用价值。综上所述,PMA-qPCR方法在复合益生菌产品中进行靶向微生物活菌定量检测方面表现出显著的技术优势,包括高灵敏度、准确性和稳定性,能够满足益生菌行业对检测技术的严格要求。
3 结论
本研究通过优化DNA提取方法和PMA前处理条件关键参数,成功建立了用于复合益生菌产品中L.paracasei活菌定量检测的PMA-qPCR方法。通过对11种代表性益生菌产品的适用性研究,结果表明,该方法在菌粉原料和标示L.paracasei添加量的样品中,测定值与平板计数结果/声称量具有较高的一致性;在无标示添加量的样品中,该方法能够有效地鉴定样品中是否存在活性L.paracasei。PMA-qPCR方法在不同类型复合益生菌产品中对L.paracasei的活菌定量具有良好的适用性,能够满足益生菌行业对准确、灵敏检测的需求。该方法显示出在复合益生菌产品中进行靶向微生物活菌定量检测方面的显著优势。其技术优势使其成为益生菌类产品质量监管和行业标准化发展的有力工具。随着PMA-qPCR方法的进一步优化,预计其将在更为复杂的基质中(如乳制品、糖果压片等)得到广泛应用,实现对目标菌种的高效活菌定量检测。这将为益生菌类食品行业提供坚实的技术支持,推动行业的高质量发展。
[1] 中国食品科学技术学会益生菌分会.益生菌的科学共识 (2020 年版).中国食品学报,2020,20(5):303-307.Probiotics Society of the Chinese Institute of Food Science and Technology. Scientific consensus on probiotics(2020) . Journal of Chinese Institute of Food Science and Technology, 2020,20(5):303-307.
[2] 国慧琴. 复合益生菌中单菌株的生物量定量方法研究[D].呼和浩特:内蒙古农业大学, 2023.GUO H Q.Biomass quantification method of single strain in compound probiotics[D].Hohhot:Inner Mongolia Agricultural University, 2023.
[3] SOEJIMA T, IIDA K I, QIN T, et al.Method to detect only live bacteria during PCR amplification[J].Journal of Clinical Microbiology, 2008, 46(7):2305-2313.
[4] STINSON L F, KEELAN J A, PAYNE M S.Characterization of the bacterial microbiome in first-pass meconium using propidium monoazide (PMA) to exclude non viable bacterial DNA[J].Letters in Applied Microbiology, 2019, 68(5):378-385.
[5] DESHMUKH R, BHAND S, ROY U.BCIG-SMAC medium and PMA-qPCR for differential detection of viable Escherichia coli in potable water[J].Iranian Journal of Microbiology, 2021, 13(5):624-631.
[6] 彭琳媛, 林洪, 王静雪.PMA-qPCR 定量检测VBNC副溶血弧菌方法的建立与优化[J].食品与发酵工业, 2022, 48(1):247-252.PENG L Y, LIN H, WANG J X.Development and optimization of PMA-qPCR for the quantitative detection of viable but non-culturable Vibrio parahaemolyticus[J].Food and Fermentation Industries, 2022, 48(1):247-252.
[7] YANG Y, LIU Y R, SHU Y H, et al.Modified PMA-qPCR method for rapid quantification of viable Lactobacillus spp.in fermented dairy products[J].Food Analytical Methods, 2021, 14(9):1908-1918.
[8] GANESAN B, WEIMER B C, PINZON J, et al.Probiotic bacteria survive in Cheddar cheese and modify populations of other lactic acid bacteria[J].Journal of Applied Microbiology, 2014, 116(6):1642-1656.
[9] GUO L Z, ZE X L, FENG H F, et al.Identification and quantification of viable Lacticaseibacillus rhamnosus in probiotics using validated PMA-qPCR method[J].Frontiers in Microbiology, 2024, 15:1341884.
[10] 刘艺茹, 马霞, 赵婷, 等.分离自人体肠道的副干酪乳杆菌K56和ET-22的菌株鉴定[J].食品与发酵工业, 2022, 48(1):62-69.LIU Y R, MA X, ZHAO T, et al.Identification of Lactobacillus paracasei K56 and ET-22 isolated from human intestine[J].Food and Fermentation Industries, 2022, 48(1):62-69.
[11] 王一帆, 薛云德, 张宝平, 等.副干酪乳杆菌的益生特性及其机制研究进展[J].食品研究与开发, 2024, 45(6):211-217.WANG Y F, XUE Y D, ZHANG B P, et al.Research progress on probiotic properties and mechanism of Lactobacillus paracasei[J].Food Research and Development, 2024, 45(6):211-217.
[12] CHONDROU P, KARAPETSAS A, KIOUSI D E, et al.Assessment of the immunomodulatory properties of the probiotic strain Lactobacillus paracasei K5 in vitro and in vivo[J].Microorganisms, 2020, 8(5):709.
[13] ACURCIO L B, WUYTS S, DE CICCO SANDES S H, et al.Milk fermented by Lactobacillus paracasei NCC 2461 (ST11) modulates the immune response and microbiota to exert its protective effects against Salmonella typhimurium infection in mice[J].Probiotics and Antimicrobial Proteins, 2020, 12(4):1398-1408.
[14] CHEN L H, HUANG S Y, HUANG K C, et al.Lactobacillus paracasei PS23 decelerated age-related muscle loss by ensuring mitochondrial function in SAMP8 mice[J].Aging, 2019, 11(2):756-770.
[15] LEE H A, KIM J Y, KIM J, et al.Anti-Helicobacter pylori activity of acomplex mixture of Lactobacillus paracasei HP7 including the extract of Perilla frutescens var.Acuta and Glycyrrhiza glabra[J].Laboratory Animal Research, 2020, 36:40.
[16] YE P X, WANG J W, LIU M M, et al.Purification and characterization of a novel bacteriocin from Lactobacillus paracasei ZFM54[J].LWT, 2021, 143:111125.
[17] GUO L Z, ZE X L, JIAO Y X, et al.Development and validation of a PMA-qPCR method for accurate quantification of viable Lacticaseibacillus paracasei in probiotics[J].Frontiers in Microbiology, 2024, 15:1456274.
[18] 杨金玉, 杨焕蝶, 陈相艳, 等.食品检测用4种病原菌基因组试剂盒提取方法的比较与优化[J].中国食品学报, 2020, 20(4):246-253.YANG J Y, YANG H D, CHEN X Y, et al.Comparison and optimization of genomic DNAs extraction of four pathogenic bacteria by genomic DNA extraction kit for foodborne microbial detection[J].Journal of Chinese Institute of Food Science and Technology, 2020, 20(4):246-253.
[19] 冯广达, 陈美标, 羊宋贞, 等.用于PCR扩增的细菌DNA提取方法比较[J].华南农业大学学报, 2013, 34(3):439-442.FENG G D, CHEN M B, YANG S Z, et al.A comparative study on bacteria DNA extraction methods used for PCR amplification[J].Journal of South China Agricultural University, 2013, 34(3):439-442.
[20] 蔡杰, 王博, 胡孙林, 等.玻璃珠-涡旋振荡改良法用于硅藻DNA提取[J].法医学杂志, 2022, 38(1):119-126.CAI J, WANG B, HU S L, et al.Improved glass bead-vortex oscillation method for DNA extraction from diatom[J].Journal of Forensic Medicine, 2022, 38(1):119-126.
[21] HU L X, XUE Y L, CUI L R, et al.Detection of viable Lacticaseibacillus paracasei in fermented milk using propidium monoazide combined with quantitative loop-mediated isothermal amplification[J].FEMS Microbiology Letters, 2021, 368(20):fnab148.
[22] GOBERT G, COTILLARD A, FOURMESTRAUX C, et al.Droplet digital PCR improves absolute quantification of viable lactic acid bacteria in faecal samples[J].Journal of Microbiological Methods, 2018, 148:64-73.
[23] PLOTKA M, WOZNIAK M, KACZOROWSKI T.Quantification of plasmid copy number with single colour droplet digital PCR[J].PLoS ONE, 2017, 12(1):e0169846.
[24] LLOYD K G, MACGREGOR B J, TESKE A.Quantitative PCR methods for RNA and DNA in marine sediments:Maximizing yield while overcoming inhibition[J].FEMS Microbiology Ecology, 2010, 72(1):143-151.
[25] NKUIPOU-KENFACK E, ENGEL H, FAKIH S, et al.Improving efficiency of viability-PCR for selective detection of live cells[J].Journal of Microbiological Methods, 2013, 93(1):20-24.