随着消费者对驼奶制品丰富营养价值和潜在健康益处的认识日益增加,这类产品正逐渐受到更多的青睐[1]。2023年,全球骆驼乳制品的市场销售额达到了37亿元[2]。在中国,驼奶制品主要以驼奶粉的形式存在,因为驼奶粉可以保存驼奶的营养特性,并且便于贮存和运输[3]。在营养方面,驼奶比其他反刍动物奶更接近母乳[4],具有多种生物活性,如抗糖尿病、抗过敏、抗菌、抗病毒和抑制肿瘤生长等[5]。然而,因驼奶产量有限,价格高昂有沙漠黄金之称[6],为了获得更高的商业利润,一些供应商在驼奶粉中掺入许多外观类似的廉价原料,如牛奶粉、蛋白粉和淀粉。这些添加物不仅可能严重损害消费者的经济利益,还可能对他们的健康造成重大影响,特别是对牛奶过敏的人群。因此,迫切需要能检测驼奶粉真伪的方法,以确保驼奶粉的质量和消费者的利益。
近年来,低场核磁共振(low-field nuclear magnetic resonance,LF-NMR)作为食品质量评估和掺假检测的关键工具,在食品科学领域获得了广泛应用[7]。LF-NMR技术具有分析速度快、样品制备简单、样品消耗量低、重现性高等优点。T2是重要的LF-NMR参数之一,可提供整个样品的质子弛豫信号(如水和脂质的氢质子)。结合化学计量学方法,T2数据已成功用于食品掺假检测。HU等[8]利用LF-NMR结合偏最小二乘判别分析(partial least squares discriminant analysis,PLS-DA),正确区分了山茶油和掺杂各种油的山茶油,掺杂比例从0%到100%不等,当掺杂率超过40%时,鉴别准确率达到100%。XING等[9]的研究表明,在基于T2光谱的正交偏最小二乘判别分析(orthogonal partial least squares discriminant analysis,OPLS-DA)中,明胶、琼脂和淀粉在正品燕窝中的掺假率分别被准确区分出40%、20%和20%。相比之下,Fisher判别模型对这3种成分的掺假率准确区分分别为20%、20%和40%,SANTOS等[7]发现,当三七掺假率超过30%时,利用三七T2数据建立的模式识别模型对真假三七的分类准确率达到100%。
本文将采用LF-NMR技术获取掺入不同比例牛奶粉、蛋白粉和淀粉的驼奶粉的T2分布,研究拟针对驼奶粉掺假问题,进行系统的定性和定量分析。同时,计划采用5种机器学习方法和主成分分析(principal component analysis,PCA)对掺入不同浓度牛奶粉、蛋白粉和淀粉的驼奶粉进行定性分析。此外,还将运用偏最小二乘回归(partial least squares regression,PLSR)来定量测定驼奶粉样品的掺假量。本研究将为定性和定量检测驼奶粉掺假提供一种新的方法。
1 材料与方法
1.1 材料与试剂
驼奶粉,内蒙古沙漠之神科技有限公司和新疆旺源驼奶实业有限公司;牛奶粉,阿勒泰哈纳斯乳业有限公司和光明乳业股份有限公司;蛋白粉,汤臣倍健股份有限公司和华昱健康大药房有限公司;淀粉,河北古松农副产品有限公司和海天调味食品有限公司。以上产品均从天猫旗舰店获得,原产地和真实性由供应商保证。关于产品来源、产品类型和化学成分(脂肪和蛋白质百分比等)的更多信息由供应商提供,如表1所示。
表1 驼奶粉、牛奶粉、蛋白粉和淀粉的主要成分 单位:%
Table 1 Major compositions of camel milk powder, cow milk powder, protein powder, and starch
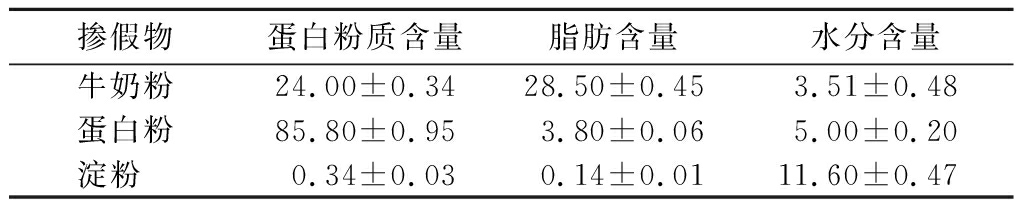
掺假物 蛋白粉质含量脂肪含量水分含量牛奶粉 24.00±0.3428.50±0.453.51±0.48蛋白粉 85.80±0.953.80±0.065.00±0.20淀粉 0.34±0.030.14±0.0111.60±0.47
1.2 仪器与设备
NM120-015V-1低场核磁共振分析仪,上海纽迈电子科技有限公司。LF-NMR弛豫评估使用低场核磁共振分析仪进行,采用Carr-Purcell-Meiboom-Gill(CPMG)脉冲序列测量T2。磁场强度为0.5 T,对应于18 MHz的质子共振频率。脉冲参数设置如下:采样频率为250 kHz,等待时间为1 500 ms,回波时间为0.2 ms,共有8 000个回波,积累次数设置为32。脉冲角设置为90°和180°,脉冲持续时间分别为7、15.4 μs,扫描之间的重复时间为2 μs。测量是在32 ℃进行的。
1.3 实验方法
将牛奶粉、蛋白粉和淀粉这3种常见的掺杂剂分别以6种不同质量分数(10%、20%、30%、40%、50%和100%)添加到驼奶粉中,制备掺假样品,以纯驼奶粉为对照。每1个梯度样品制备一式三份,用于平行实验;将所有制备好的样本分别放入7 mL的色谱瓶内,并置于40 ℃的烘干箱中烘干至恒重。在实验之前,所有样品恒温贮存在烘干炉中。
单个掺假物每个梯度3个重复样品,每个样品测定3次,每个梯度获得9个数据,3种掺假物和纯驼奶粉共获得189个数据。
1.4 数据分析
1.4.1 低场核磁测量数据处理
NMR弛豫测量得到的图为自由诱导指数衰减曲线,横向松弛曲线可用公式(1)进行拟合:
(1)
式中:M(t)是时间t的剩余磁化强度;M0,n是第n次指数的浓度或幅度参数;T2,n是相应的横向弛豫时间常数;e(t)是剩余误差。
通过T-invfit软件,对LF-NMR弛豫测量中获得的自由感应衰减曲线进行了反演拟合处理,可得到样品的T2数据图谱并获知样品的T21、T22、T23峰起始时间及其相应的峰面积比例P21、P22、P23。
1.4.2 分类模型的建立
在本研究中,基于支持向量机(support vector machine,SVM)、k-近邻(k-nearest neighbors,KNN)、随机森林(random forest,RF)、多层感知器(multilayer perceptron,MLP)和极限梯度提升(extreme gradient boosting,XGBoost)开发了不同的分类模型。对所有模型,通过随机抽样,将每个数据集分割为70%的训练集和30%的测试集。训练集用于模型选择和参数调整,测试集则用于评估模型性能。SVM作为一种强大的监督学习算法,通过确定最优超平面来最大化不同类别间的间隔,从而实现分类任务[10]。本研究为了优化SVM的性能探索了多个关键超参数,最终确定了最优组合,C设置为100,γ设置为0.001,研究中选用径向基函数担任核函数。KNN同样是一种经典的监督分类建模方法,它利用距离方法来生成一个简单且灵活的决策边界,原理是测试集中的未知样本使用训练集中最近邻域的大多数来设置类别[11]。本研究探索了不同的k值范围(1~10),并通过比较预测精度来优化k的选择,经过调优后的最佳k值为7。RF通过集成多个独立决策树的预测结果来进行最终分类[12]。为了优化模型性能,本研究采用了网格搜索和交叉验证的方法来确定最佳超参数组合。优化后的最终参数包括:200棵决策树,最大深度30,内部节点分裂至少需要5个样本,叶节点最少包含2个样本。MLP是一种全连接的前馈神经网络,由输入层、多个隐藏层和输出层构成[12]。本研究中,MLP配置了2个隐藏层:第1层有64个神经元,第2层有32个神经元。此外,为了防止过拟合,设置了L2正则化项的系数为0.000 1。这种结构设计使MLP能够在处理驼奶粉掺假数据时保持良好的泛化能力。XGBoost通过迭代训练和优化弱分类器来逐步改进其模型,从而实现更好的数据拟合和复杂问题的解决[13]。经参数优化,本研究决策树数量设为200,最大深度设为6,学习率设为0.1,子样本比例设为0.8,特征比例设为0.8。这些参数配置旨在确保模型能够在处理低场核磁数据时达到最佳性能。
使用PyCharm 2022.2.1 IDE在Python中实现了模型。对每种算法所建立的模型进行了性能测试,精确度(Pr)和召回率(RC)由公式(2)和公式(3)定义,用预测精度和F1评分来评价定性模型对不同掺假物分类的效果,由公式(4)和公式(5)定义:
(2)
(3)
预测精度![]()
(4)
F1评分![]()
(5)
式中:TP是真阳性数;TN是真阴性数;FP是误报数;FN是假阴性数。
1.4.3 PCA
PCA用于区分纯驼奶粉样品和不同掺假程度的驼奶粉样品。PCA通过将相关变量转化为一组主成分来降低维度,从而有效地突出了掺假驼奶粉样品的差异和聚类趋势[14]。数据预处理涉及均值中心化和帕累托缩放以确定代表样本总体的最佳方法[15]。
1.4.4 PLSR
在本研究中,采用了PLSR的线性方法,用于预测驼奶粉中3种不同掺假物的掺假水平。PLSR是一种常用的化学计量技术,它集成了PCA和多变量回归,以简化回归建模并降低数据复杂性[16]。它擅长解决多重共线性问题,并适用于处理自变量数量多于样本数量的数据集。由于NMR参数的过度共线性,PLSR适合用于分析LF-NMR数据[17]。将掺假驼奶粉样品的LF-NMR数据随机分类,2/3用于校准集和验证集,1/3用于预测集,并基于训练集数据建立PLSR模型。模型的准确性通过相关系数(R2)和预测均方根误差(root mean square error,RMSE)来表示[7],由公式(6)和公式(7)定义:
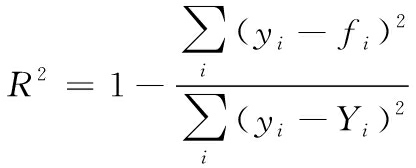
(6)
(7)
式中:fi是模型的预测值;yi是实际测量值。
为了验证PLSR模型的预测能力,本研究准备了一组独立的掺假样品作为验证集,驼奶粉及3种掺假物的来源均与校准集不同。把独立验证集的LF-NMR数据输入到校准好的PLSR模型中进行预测。模型的预测性能通过R2和RMSE进行评估。
另外,本研究选择了10%、30%和50%不同质量分数梯度的掺假样品,每个样品在同一天内进行6次重复测量,计算每个样品的相对标准偏差(relative standard deviation,RSD),来评判方法的日内精密度。选择相同质量分数梯度的掺假样品,由不同的实验人员在连续3 d内进行测量,每天测量2次。同样计算每个样品的日间RSD。
1.5 统计
使用SPSS 21.0统计分析程序对数据进行方差分析。所有测试均一式三份进行,每份样品测量3次,数据表示为“平均值±标准差”。
2 结果与讨论
2.1 LF-NMR弛豫信号
横向弛豫时间T2测量能够反映驼奶粉掺假样品中氢质子所在磁场的均匀性[18]。如图1所示,通过CPMG序列测量获得了纯驼奶粉和3种掺假驼奶粉的LF-NMR横向磁化衰减曲线。随着掺假水平的增加,所有样品的回波幅度均呈下降趋势。这可能与不同掺假水平的驼奶粉样品中氢质子成分的差异有关[19]。为了研究不同样品之间的差异,对不同比例掺假驼奶粉获得的T2分布进行分析。
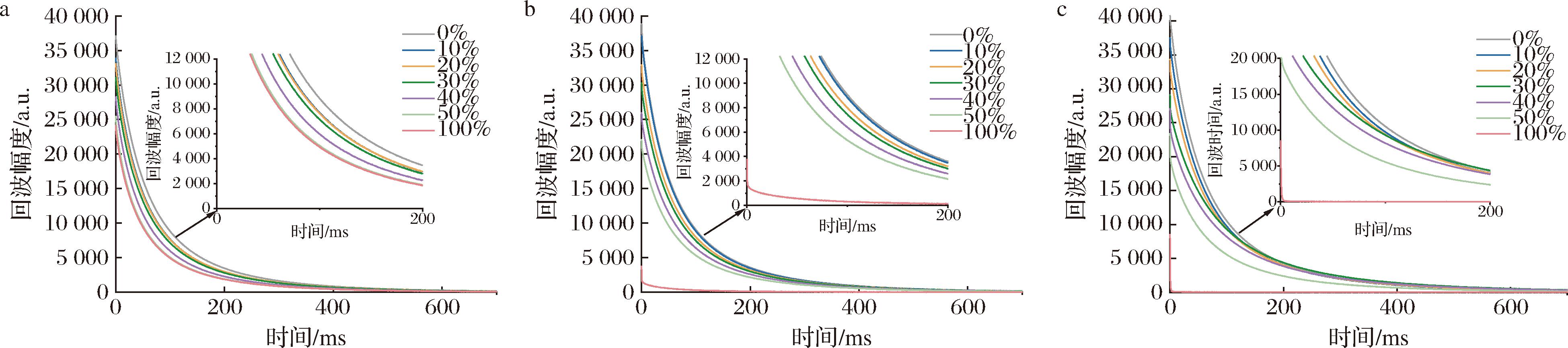
a-掺有牛奶粉的样品;b-掺有蛋白粉的样品;c-掺有淀粉的样品
图1 不同掺假水平下驼奶粉的横向弛豫衰减曲线
Fig.1 The transverse relaxation decay curves of camel milk powder with different adulteration levels
图2显示了纯驼奶粉和掺假驼奶粉的T2弛豫光谱中有3个峰。T21代表结合水,T22代表不可动水,T23可能代表样品中脂质的信号,因为前人研究表明脂质的T2大约为100 ms[20]。对应的峰面积比例分别命名为P21、P22、P23,主要代表氢质子的数量。对于牛奶粉掺假的情况(图2-a),随着牛奶粉掺假量增加,T21峰值的振幅呈现逐渐增加的趋势,T22峰值的振幅几乎无变化,T23峰值的振幅逐渐减小。对于蛋白粉掺假的情况(图2-b),随着蛋白粉掺假量增加,T21峰值的振幅和P21逐渐增加,而T23峰值的振幅和P23逐渐减小。对于淀粉掺假的情况(图2-c),随着淀粉掺假量增加,T21峰值的振幅逐渐增加,T22峰值的振幅在掺假量为100%时显著降低,T23峰值的振幅逐渐减小。
纯驼奶粉因脂肪含量较高(36.6%),T23峰振幅显著,而掺假物的引入改变了脂质分布。掺入牛奶粉(脂肪含量28.5%)时,T23峰振幅随掺假量增加逐渐降低(图2-a),掺入蛋白粉(脂肪含量3.8%)和淀粉(脂肪含量0.14%)时,T23峰振幅进一步减弱(图2-b、2-c),尤其淀粉掺假样品中T23峰几乎消失,与其极低脂肪含量的特性一致(表1)。此外,掺假物对脂质氢质子的影响可通过P23的变化量化。如表2所示,掺假量增加时,P23值显著下降,尤其在淀粉掺假样品中P23从纯驼奶粉的96.22%降至0.25%。因此,LF-NMR技术能有效区分不同掺假程度的驼奶粉,且真实与掺假样品间的T2值存在显著差异,为化学计量学分析提供了可靠基础。

a-掺有牛奶粉的样品;b-掺有蛋白粉的样品;c-掺有淀粉的样品
图2 不同掺假水平下驼奶粉的横向弛豫时间(T2)曲线
Fig.2 The transverse relaxation time (T2) curves of of camel milk powder with different adulteration levels
表2 掺有牛奶粉、蛋白粉和淀粉的驼奶粉的横向弛豫时间(T2)及其对应的峰面积比例(P2)
Table 2 The transverse relaxation time (T2) and the corresponding peak area proportions (P2) of camel milk powder adulterated with cow milk powder, protein powder, and starch

掺假物掺假量/%T21/msT22/msT23/msP21/%P22/%P23/%00.22±0.02ab3.23±0.18e61.07±2.23d1.52±0.23e1.88±0.05g96.22±0.32a100.23±0.02a3.58±0.22d61.61±3.77cd1.96±0.23d2.03±0.06f96.01±0.19a200.22±0.02ab4.04±0.20c68.73±5.53b2.64±0.32c2.14±0.05e95.22±0.29b牛奶粉300.22±0.02ab4.25±0.40bc68.81±6.62b2.68±0.33c2.35±0.06d94.96±0.29b400.24±0.02a4.20±0.15bc65.46±2.38bc2.39±0.18c2.53±0.08c95.08±0.18b500.21±0.01bc4.48±0.41b67.25±6.61bc4.36±0.53b3.25±0.09a92.39±0.47d1000.20±0.01c5.11±0.42a74.64±10.09a9.51±0.84a2.91±0.20b87.59±0.69e00.22±0.02a3.23±0.18c61.07±2.23c1.52±0.23f1.88±0.05d96.60±0.20a100.21±0.00ab4.48±0.41b85.49±8.93a3.62±0.48e1.96±0.06cd94.42±0.46b200.21±0.00c4.18±0.33b78.02±9.13ab4.90±0.62e1.98±0.06cd93.12±0.63c蛋白粉300.21±0.01cd4.59±0.42b83.63±9.79a7.05±0.90d2.07±0.07bcd90.88±0.87d400.20±0.01cd4.51±0.34b85.76±11.59a9.33±1.13c2.17±0.03bc88.50±1.11e500.20±0.01d4.56±0.52b85.81±12.02a12.27±1.75b2.30±0.06b85.43±1.70f1000.19±0.00e5.27±0.45a68.92±11.48bc75.50±2.69a4.97±0.63a19.52±2.11g00.22±0.02e3.23±0.18e61.07±2.23b1.52±0.23g1.88±0.05a96.22±0.32a100.24±0.01d3.61±0.24de63.52±3.43b5.76±0.32f1.66±0.06c92.06±0.25b200.28±0.01c3.93±0.27de67.32±7.33b9.83±0.48e1.50±0.04e88.21±0.37c淀粉 300.32±0.01b5.83±0.52b69.10±9.38b28.88±0.52b1.75±0.15b69.00±0.61f400.28±0.01c4.52±0.40cd74.07±10.05b15.13±0.77d1.47±0.04d83.01±0.68d500.31±0.02b5.45±0.65bc81.11±13.88b21.56±0.59c1.67±0.11c76.49±0.71e1000.43±0.03a16.61±2.57a170.99±78.63a99.17±0.02a0.53±0.04e0.25±0.07g
注:在同一行内,用不同小写字母标记的数据表示具有显著性差异(P<0.05)。
2.2 分类模型的性能
基于3种掺假驼奶粉的LF-NMR弛豫光谱峰数据,本文利用SVM、KNN、RF、MLP和XGBoost这5种机器学习算法进行了分类建模。表3总结了各模型的性能指标。精度和F1评分越接近100%说明分类模型的可靠性越高。SVM的训练和测试精度分别为90.91%和89.47%,训练和测试的F1评分均为90.38%。KNN表现较差,测试准确率为84.21%,测试F1评分为85.41%。RF是最佳的分类模型,准确率和F1评分为96.35%和97.53%。MLP的训练准确率和测试准确率分别为100%和94.74%。相应的F1评分分别为100%和94.74%。XGBoost的训练精度和F1评分与MLP相同,但测试精度和F1评分低于MLP,分别为89.47%和89.54%。总之,RF算法在训练集和测试集上都表现出了良好的分类性能,因为它能够捕获数据特征之间的交互作用和非线性关系[21]。
表3 不同机器学习算法在分类不同掺假驼奶粉样本中的性能表现 单位:%
Table 3 The performance of different machine learning algorithms to classify different adulterated camel milk powder samples

机器算法训练准确率测试准确率训练F1评分测试F1评分SVM90.9189.4790.3889.69KNN90.9184.2190.3885.41RF10096.3510097.53MLP10094.7410094.74XGBoost10089.4710089.54
2.3 PCA
采用横向弛豫参数T2进行PCA,对3种不同掺假剂量的掺假驼奶粉样品进行分类。掺入牛奶粉、蛋白粉和淀粉的驼奶粉样品的PCA结果分别如图3所示。如图3-a所示,前2个主成分的累计贡献率为96%(PC1=87.5%,PC2=8.5%)。PCA在第一主成分轴上成功地区分了纯驼奶粉和掺牛奶粉的样品,且两者之间没有重叠。随着掺假量的增加,样品组在PC1轴上的分布呈现出从左至右的趋势。对于掺有蛋白粉的驼奶粉(图3-b),其变化趋势与掺有牛奶粉的驼奶粉相似。随着蛋白粉量的增加,掺假驼奶粉样品在PC1的正向区域上分布得更加集中。在此情况下,PC1解释了81.7%的方差,PC2解释了16.6%的方差,两者合计解释了98.3%的总方差。图3-c展示了纯驼奶粉与不同量掺假淀粉的驼奶粉样品之间的明显区别。前2个主成分占总方差的91.9%,其中PC1占63.0%,PC2占28.9%。综上所述,PC1和PC2的组合能够有效区分纯驼奶粉和不同量的掺假驼奶粉样品,且变异性的分布主要由第一主成分造成。
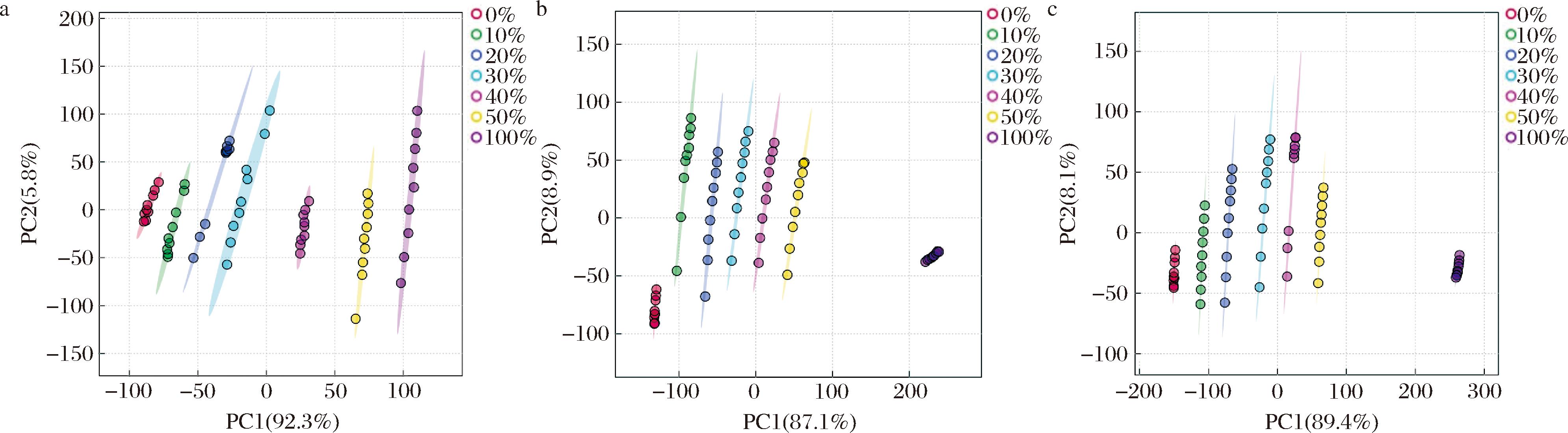
a-掺有牛奶粉的样品;b-掺有蛋白粉的样品;c-掺有淀粉的样品
图3 不同掺假量(0%、10%、20%、30%、40%、50%和100%)的驼奶粉的PCA得分图
Fig.3 The PCA score plots of camel milk powder adulterated with different concentrations of 0%, 10%, 20%, 30%, 40%, 50%, and 100%
2.4 PLSR模型的分析
本文使用留一法交叉验证技术来防止PLSR模型中出现过拟合和欠拟合问题。最佳的潜在变量(latent variables,LVs)由交叉验证均方根误差的最小值来确定。本研究通过使用2个潜在变量构建PLSR模型,以预测驼奶粉中牛奶粉的掺假量。由表4可知,PLSR模型取得了良好的性能,校准决定系数![]() 和校准均方根误差(root mean square error of calibration,RMSEC)分别为0.967 5和0.085 2,交叉验证决定系数
和校准均方根误差(root mean square error of calibration,RMSEC)分别为0.967 5和0.085 2,交叉验证决定系数![]() 和交叉验证均方根误差(root mean square error of cross-validation,RMSECV)分别为0.949 8和0.068 6,预测决定系数
和交叉验证均方根误差(root mean square error of cross-validation,RMSECV)分别为0.949 8和0.068 6,预测决定系数![]() 和预测均方根误差(root mean square error of prediction,RMSEP)分别为0.978 0和0.045 4。对于蛋白粉掺假的驼奶粉样本,使用3个LVs的PLSR模型的预测结果略优于牛奶粉掺假的预测结果
和预测均方根误差(root mean square error of prediction,RMSEP)分别为0.978 0和0.045 4。对于蛋白粉掺假的驼奶粉样本,使用3个LVs的PLSR模型的预测结果略优于牛奶粉掺假的预测结果![]() 7和RMSEP=0.024 3)。构建的驼奶粉中掺假淀粉的PLSR模型表现最佳,该模型使用了3个
7和RMSEP=0.024 3)。构建的驼奶粉中掺假淀粉的PLSR模型表现最佳,该模型使用了3个![]() 和RMSEC分别为0.999 6和0.006
和RMSEC分别为0.999 6和0.006 ![]() 和RMSECV分别为0.982 4和0.040
和RMSECV分别为0.982 4和0.040 ![]() 和RMSEP分别为0.999 4和0.007 6。当R2≥0.90时,PLSR模型的预测精度被认为较好。所有预测3种掺假物质含量的PLSR模型的
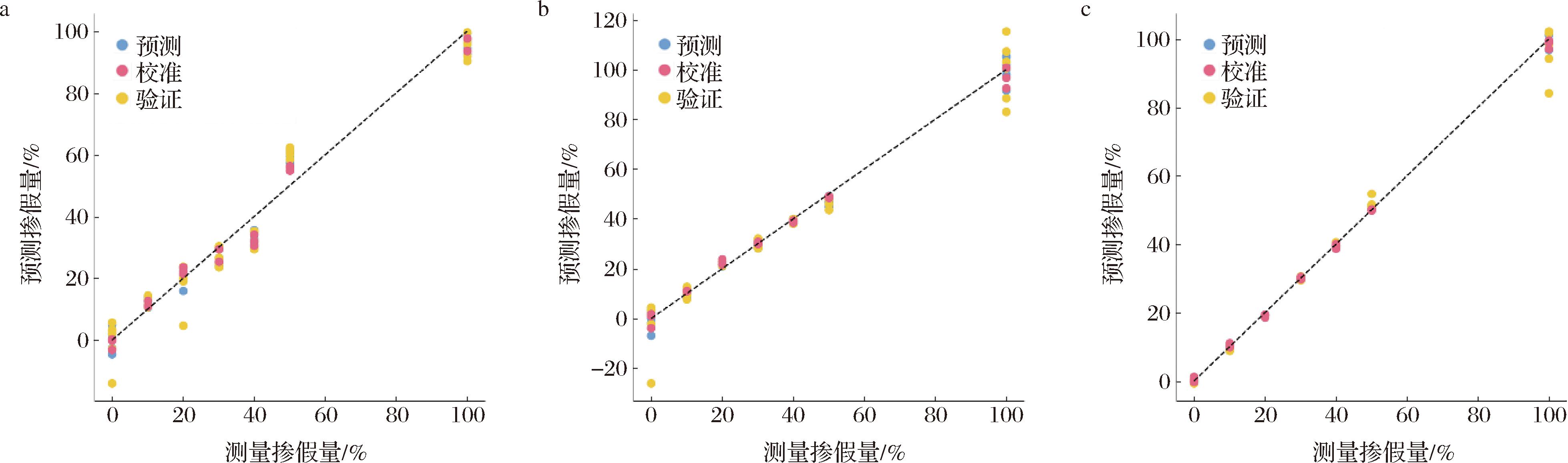
和RMSEP分别为0.999 4和0.007 6。当R2≥0.90时,PLSR模型的预测精度被认为较好。所有预测3种掺假物质含量的PLSR模型的![]() 都大于等于0.978 0,这意味着所建立的模型具有出色的预测准确性。PLSR模型的预测掺假量值与测量掺假量值的散点图如图4所示,预测值与测量值相关性较高。
都大于等于0.978 0,这意味着所建立的模型具有出色的预测准确性。PLSR模型的预测掺假量值与测量掺假量值的散点图如图4所示,预测值与测量值相关性较高。
表4 PLSR模型预测掺假量结果
Table 4 PLSR model results for predicting adulteration concentrations

掺假物LVsR2CRMSECR2CVRMSECVR2PRMSEP牛奶粉20.967 50.085 20.948 90.068 60.978 00.045 4蛋白粉30.999 20.027 40.959 90.061 40.993 70.024 3淀粉 30.999 60.006 00.982 40.040 70.999 40.007 6
a-掺有牛奶粉的样品;b-掺有蛋白粉的样品;c-掺有淀粉的样品
图4 PLSR模型的测量和预测掺假量的散点图
Fig.4 Scatterplot of measured and predicted adulteration concentrations for the PLSR model
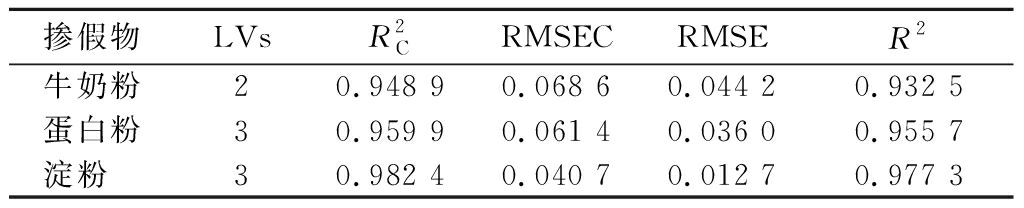
将独立验证集的LF-NMR数据输入到校准好的PLSR模型中进行预测。由表5可知,对于牛奶粉、蛋白粉和淀粉,模型的RMSE分别为0.044 2、0.036 0和0.012 7,R2均超过0.9,显示出模型具有较高的预测精度和稳定性。
表5 PLSR外部验证结果
Table 5 External validation results of PLSR
掺假物LVsR2CRMSECRMSER2牛奶粉20.948 90.068 60.044 20.932 5蛋白粉30.959 90.061 40.036 00.955 7淀粉 30.982 40.040 70.012 70.977 3
本研究基于PLSR模型分析了LF-NMR技术检测驼奶粉掺假的精密度(表6)。结果表明,不同掺假物的日内RSD均低于6.8%,日间RSD为6.1%~10.3%,该模型能够有效区分不同掺假物及比例。
表6 PLSR模型精密度
Table 6 PLSR precision results
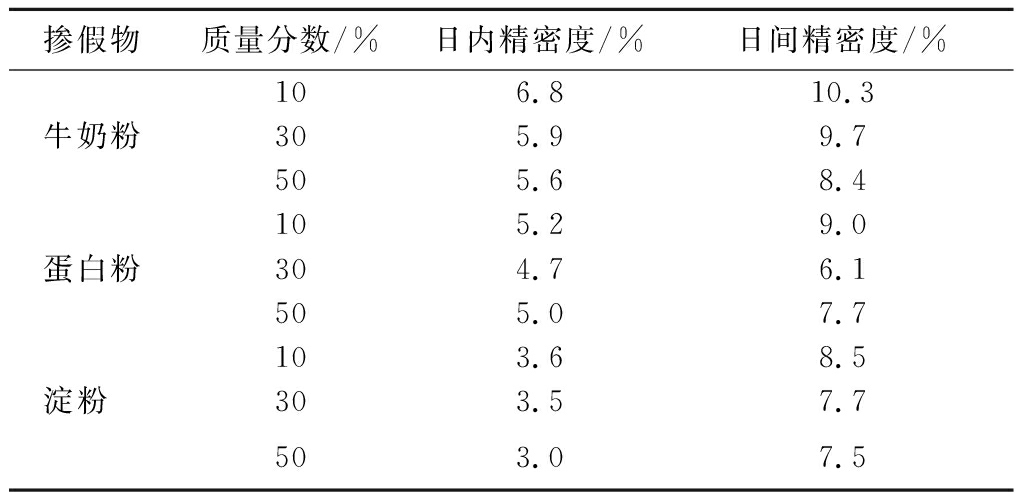
掺假物质量分数/%日内精密度/%日间精密度/%106.810.3牛奶粉305.99.7505.68.4105.29.0蛋白粉304.76.1505.07.7103.68.5淀粉 303.57.7503.07.5
3 结论
本研究基于低场核磁共振技术结合机器学习和化学计量学方法,成功实现了对驼奶粉中牛奶粉、蛋白粉及淀粉掺假的高效检测与定量分析。通过横向弛豫时间谱的解析,发现了不同掺假物对驼奶粉中脂质氢质子分布的差异性影响。在分类模型构建中,SVM、KNN、RF、MLP和XGBoost这5种机器学习算法均展现出对驼奶粉掺假样本的有效区分能力,其中RF算法表现尤为突出,在测试集中的准确率与F1评分分别达到96.35%和97.53%。PLSR模型对3种掺假物的定量预测能力优异,预测决定系数![]() 均高于0.978,其中淀粉掺假的预测精度最高
均高于0.978,其中淀粉掺假的预测精度最高![]() 模型验证结果显示,日内与日间精密度分别低于6.8%和10.3%,表明方法稳定性良好。总的来说,利用LF-NMR技术可以有效地用于驼奶粉掺假类型和掺假量的检测。未来研究可进一步扩大样本量并优化模型泛化能力,以提升复杂掺假场景下的适用性。
模型验证结果显示,日内与日间精密度分别低于6.8%和10.3%,表明方法稳定性良好。总的来说,利用LF-NMR技术可以有效地用于驼奶粉掺假类型和掺假量的检测。未来研究可进一步扩大样本量并优化模型泛化能力,以提升复杂掺假场景下的适用性。
[1] WU X Y, NA Q, HAO S Q, et al.Detection of ovine or bovine milk components in commercial camel milk powder using a PCR-based method[J].Molecules, 2022, 27(9):3017.
[2] QYResearch.2024年全球骆驼乳制品行业总体规模、主要企业国内外市场占有率及排名[EB/OL].(2023-12-22) [2024-11-07].https://cn.qyresearch.com/reports/3087211/camel-dairy.QYResearch.Camel dairy report 2024, global revenue, key companies market shatr &rank [EB/OL].(2023-12-22) [2024-11-07].https://cn.qyresearch.com/reports/3087211/camel-dairy.
[3] LI L Y, WANG J, LI M J, et al.Detection of the adulteration of camel milk powder with cow milk by ultra-high performance liquid chromatography (UPLC)[J].International Dairy Journal, 2021, 121:105117.
[4] MORSI R, GHOUDI K, AYYASH M M, et al.Detection of 11 carbamate pesticide residues in raw and pasteurized camel milk samples using liquid chromatography tandem mass spectrometry:Method development, method validation, and health risk assessment[J].Journal of Dairy Science, 2024, 107(4):1916-1927.
[5] CHI S X, LIU B H, ZHANG B, et al.Development of an ELISA method to determine adulterated cow milk in camel milk[J].International Dairy Journal, 2024, 155:105953.
[6] 李玲玉, 王俊, 李敏婧, 等.基于乳清蛋白的骆驼乳中掺假牛乳的检测及热处理对方法的影响[J].食品科学, 2022, 43(10):329-335.LI L Y, WANG J, LI M J, et al.A method for detection of cow milk in adulterated camel milk based on whey protein and effect of heat treatment on it[J].Food Science, 2022, 43(10):329-335.
[7] SANTOS P M, PEREIRA-FILHO E R, COLNAGO L A.Detection and quantification of milk adulteration using time domain nuclear magnetic resonance (TD-NMR)[J].Microchemical Journal, 2016, 124:15-19.
[8] HU B K, ZHANG D Y, GENG Y Y, et al.Chemometrics analysis of Camellia oil authenticity using LF NMR and fatty acid GC fingerprints[J].Journal of Food Composition and Analysis, 2024, 133:106447.
[9] XING M J, LIU F J, LIN J Z, et al.Origin tracing and adulteration identification of bird’s nest by high- and low-field NMR combined with pattern recognition[J].Food Research International, 2024, 175:113780.
[10] WU M F, LI M M, FAN B, et al.A rapid and low-cost method for detection of nine kinds of vegetable oil adulteration based on 3-D fluorescence spectroscopy[J].LWT, 2023, 188:115419.
[11] YOKOYAMA D, SUZUKI S, ASAKURA T, et al.Chemometric analysis of NMR spectra and machine learning to investigate membrane fouling[J].ACS Omega, 2022, 7(15):12654-12660.
[12] MENÉNDEZ-GARC A L A, GARC
A L A, GARC A-NIETO P J, GARC
A-NIETO P J, GARC A-GONZALO E, et al.Time series analysis for COMEX platinum spot price forecasting using SVM, MARS, MLP, VARMA and ARIMA models:A case study[J].Resources Policy, 2024, 95:105148.
A-GONZALO E, et al.Time series analysis for COMEX platinum spot price forecasting using SVM, MARS, MLP, VARMA and ARIMA models:A case study[J].Resources Policy, 2024, 95:105148.
[13] NAKHAEI-KOHANI R, AMIRI-RAMSHEH B, POURMAHDI M, et al.Extensive data analysis and modelling of carbon dioxide solubility in ionic liquids using chemical structure-based ensemble learning approaches[J].Fluid Phase Equilibria, 2024, 585:114166.
[14] FU X X, MA W T, ZUO Q, et al.Application of machine learning for high-throughput tumor marker screening[J].Life Sciences, 2024, 348:122634.
[15] CUI C J, XIA M Y, CHEN J L, et al.1H NMR-based metabolomics combined with chemometrics to detect edible oil adulteration in Huajiao (Zanthoxylum bungeanum Maxim.)[J].Food Chemistry, 2023, 423:136305.
[16] LI Z M, SONG J H, MA Y X, et al.Identification of aged-rice adulteration based on near-infrared spectroscopy combined with partial least squares regression and characteristic wavelength variables[J].Food Chemistry:X, 2023, 17:100539.
[17] PANDISELVAM R, MAHANTI N K, MANIKANTAN M R, et al.Rapid detection of adulteration in desiccated coconut powder:Vis-NIR spectroscopy and chemometric approach[J].Food Control, 2022, 133:108588.
[18] PENG D, ZHOU Q, SU M, et al.Quantitative determination of the carbonyl value in frying oils based on LF-NMR combined with chemometrics[J].LWT, 2024, 198:116067.
[19] MUNIZ R O, GONZALEZ J L, TOCI A T, et al.Using 1H low-field NMR relaxometry to detect the amounts of Robusta and Arabica varieties in coffee blends[J].Food Research International, 2023, 174:113610.
[20] LI Y T, OBADI M, SHI J C, et al.Determination of moisture, total lipid, and bound lipid contents in oats using low-field nuclear magnetic resonance[J].Journal of Food Composition and Analysis, 2020, 87:103401.
[21] QIU Z J, BIAN Y L, WANG F Y, et al.A novel method for detection of internal quality of walnut kernels using low-field magnetic resonance imaging[J].Computers and Electronics in Agriculture, 2024, 217:108546.