烟酰胺单核苷酸(nicotinamide mononucleotide,NMN)是一种天然存在的维生素B族衍生物,广泛存在于动物、植物、虾类等[1],它存在α与β两种同分异构体,但只有β异构体是具有生物活性,也称为β-烟酰胺单核苷酸(β-NMN)[2]。NMN作为烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NAD+)和烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate,NADP+)的前体之一,参与并调节多种生化反应,在细胞能量代谢中起到关键的作用[3]。研究表明,NMN补充剂可以弥补NAD+或NADP+的缺失以起到多种治疗作用,如治疗神经系统疾病、Ⅱ型糖尿病、肥胖等退行性疾病[4]。NMN还可以恢复阿尔茨海默病大鼠的认知功能,并对神经元有明显的保护作用[5]。除此之外,还发现NMN具有增加抗氧化性,提高免疫力,预防心血管疾病等医疗功效[6]。
目前,NMN的合成法包括化学合成法与生物合成法。在早期的研究当中,NMN大多通过化学合成法获得,以烟酰胺及其衍生物为底物经过多步化学反应来制备,但是化学合成法存在反应条件苛刻、终产物产率低、纯度低等问题[7]。与化学合成法相比,NMN的生物合成法具有高立体结构选择性、反应条件温和、副产物少、产品纯度高等优点,成为目前NMN绿色环保的制备方法[3, 8-10]。在自然界中,存在NMN的从头合成途径和补救合成途径,途径中包括同为烟酰胺衍生物的烟酸(niacin,NA)、烟酸单核苷酸(NaMN)、烟酰胺核糖(nicotinamide riboside,NR)等。其中,烟酰胺(nicotinamide,NAM)与5-磷酸核糖焦磷酸(phosphoribosyl pyrophosphate,PRPP)缩合生成NMN的补救途径因底物经济、转化率较高等优势,已成为NMN生物合成的主要研究途径,该途径中的限速酶为烟酰胺磷酸核糖基转移酶(nicotinamide phosphoribosyltransferase,NAMPT)[11]。同时NAMPT也是哺乳动物体内NAD+生物合成途径中的限速酶[12]。
对于NAMPT的研究,前期主要是从不同宿主中筛选得到酶学性质优良的NAMPT。SHOJI等[13]选择来自哺乳动物和细菌10种NAMPT,分别在大肠杆菌(Escherichia coli)中异源表达,发现来自松树噬几丁质菌(Chitinophaga pinensis)的NAMPT(即CP-NAMPT)的表达效果最佳,其酶活力是来源希瓦氏菌(Shewanella oneidensis)的2.4倍。MARINESCU等[14]报道了在大肠杆菌中过表达来自杜克雷嗜血杆菌(Haemophilus ducreyi)的NAMPT(即Hd-NAMPT),可以将10 g/L的NAM催化生成15.42 mg/L的NMN。除直接筛选外,对NAMPT进行晶体结构分析,从而确定酶工程改造策略也是一种途径。另外,BURGOS等[15]证明了ATP水解与NMN合成相关,NAMPT催化反应的副产物PPi可促进ATP水解,通过增强底物亲和力NAMPT催化效率提高约1 100倍。本研究团队前期挖掘到一种来自弧菌噬菌体(Vibrio bacteriophage)的NAMPT(即VpNadV),发现该酶具有较好的催化活性,在摇瓶中NMN产量达到2.6 g/L,5 L发酵罐中NMN的产量达到16.7 g/L[16]。
研究表明稳定高效的NAMPT对高效生物合成NMN是非常重要的。因此,本研究针对前期筛选得到的野生型VpNadV开展研究工作。首先,在大肠杆菌中通过敲除pncA,阻断NMN降解为NA途径,优化并多拷贝整合表达蕈状芽孢杆菌(Bacillus mycoides)来源的NMN转运蛋白PnuC,获得了用于NMN合成的底盘菌株。然后,通过引入NMN荧光反应体系,构建了一种高通量筛选系统,构建易错PCR突变文库和饱和突变文库进行筛选,迭代突变后,最终确定了最佳催化活性的VpNadV阳性突变体(VpNadVL139V+R382G),其酶活为野生型的1.46倍。在摇瓶发酵中NMN的产量最高达到3.68 g/L,经发酵优化后5 L发酵罐中NMN最高产量达到24.4 g/L。本研究建立的高通量筛选系统以及确定的VpNadV高催化活性突变体,为NMN及其相关衍生物的合成提供了参考。
1 材料与方法
1.1 材料与试剂
1.1.1 实验材料
本研究所使用的实验材料包括涉及的菌株、质粒。其中,E.coli JM109作为克隆宿主用于质粒保存、构建与扩增,E.coli BL21(DE3)作为表达宿主进行蛋白表达、菌株改造和NMN的生产。质粒pET-28a (+)用于重组质粒构建,质粒p-Target和p-Cas9用于基因组编辑。本研究使用的菌株如表1所示,所使用的质粒如表2所示。
表1 本研究使用的菌株
Table 1 Strains used in this study
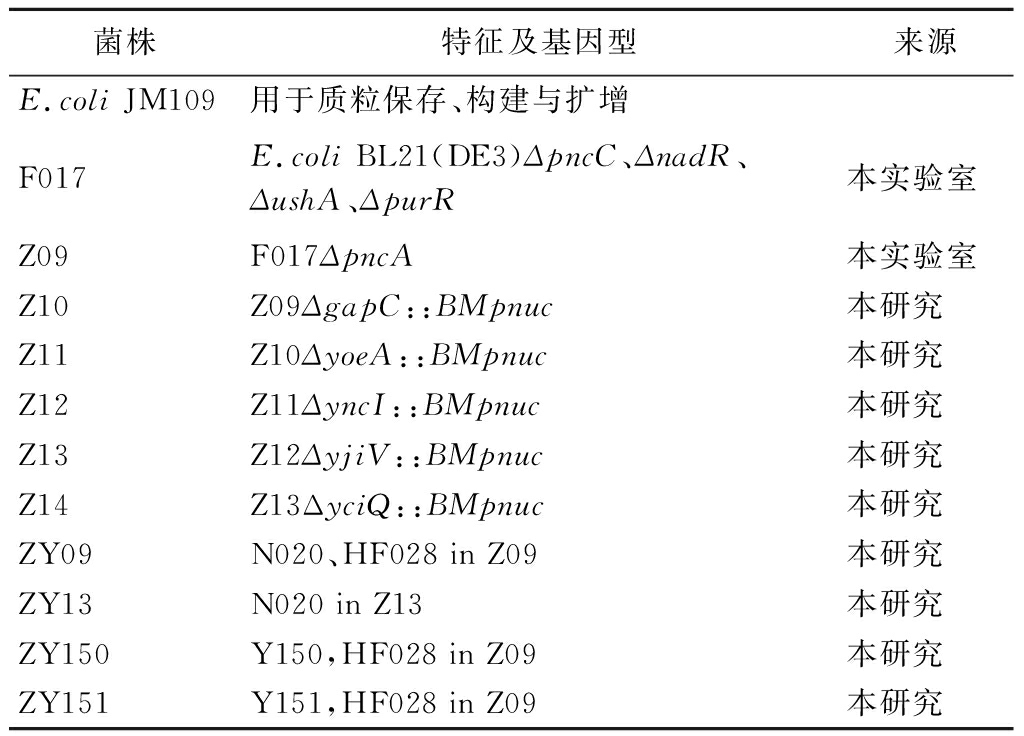
菌株特征及基因型来源E.coli JM109用于质粒保存、构建与扩增F017E.coli BL21(DE3)ΔpncC、ΔnadR、ΔushA、ΔpurR本实验室Z09F017ΔpncA本实验室Z10Z09ΔgapC::BMpnuc本研究Z11Z10ΔyoeA::BMpnuc本研究Z12Z11ΔyncI::BMpnuc本研究Z13Z12ΔyjiV::BMpnuc本研究Z14Z13ΔyciQ::BMpnuc本研究ZY09N020、HF028 in Z09本研究ZY13N020 in Z13本研究ZY150Y150,HF028 in Z09本研究ZY151Y151,HF028 in Z09本研究
表2 本研究使用的质粒
Table 2 Plasmids used in this study

质粒特征抗性来源p-Cas9p-Tar-getN020HF028Y011Y012Y013Y014Y015Y016Y111Y112Y113Y114Y115Y116Y117Y118Y119Y120Y121Y122Y123Y124Y125Y126Y141Y142Y143Y150Y151Y152Y153Y154Y155Y156Y157Y158提高基因敲除与整合需要的Cas9蛋白提供基因敲除与整合需要的sgRNApET28a(+)-BaPRS-VpNadVpCDFDuet(+)-BMpnucpET28a (+)-BaPRS-VpNadVR26EpET28a (+)-BaPRS-VpNadVL139VpET28a (+)-BaPRS-VpNadVD383GpET28a (+)-BaPRS-VpNadVF387SpET28a (+)-BaPRS-VpNadVK390PpET28a (+)-BaPRS-VpNadV473VpET28a (+)-BaPRS-VpNadVR26E+L139VpET28a (+)-BaPRS-VpNadVR26E+D383GpET28a (+)-BaPRS-VpNadVR26E+F387SpET28a (+)-BaPRS-VpNadVR26E+K390PpET28a (+)-BaPRS-VpNadVR26E+A473VpET28a (+)-BaPRS-VpNadVL139V+D383GpET28a (+)-BaPRS-VpNadVL139V+F387SpET28a (+)-BaPRS-VpNadVL139V+K390PpET28a (+)-BaPRS-VpNadVL139V+A473VpET28a (+)-BaPRS-VpNadVD383G+F387SpET28a (+)-BaPRS-VpNadVD383G+F387SpET28a (+)-BaPRS-VpNadVD383G+K390pET28a (+)-BaPRS-VpNadVD383G+A473VpET28a (+)-BaPRS-VpNadVF387S+K390pET28a (+)-BaPRS-VpNadVF387S+A473VpET28a (+)-BaPRS-VpNadVK390P+A473VpET28a (+)-BaPRS-VpNadVL139HpET28a (+)-BaPRS-VpNadVR382GpET28a (+)-BaPRS-VpNadVK390HpET28a (+)-BaPRS-VpNadVL139V+R382GpET28a (+)-BaPRS-VpNadVL139H+R382GpET28a (+)-BaPRS-VpNadVL139V+K390PpET28a (+)-BaPRS-VpNadVL139H+K390PpET28a (+)-BaPRS-VpNadVL139H+K390HpET28a (+)-BaPRS-VpNadVL139V+F387SpET28a (+)-BaPRS-VpNadVK390P+R382GpET28a (+)-BaPRS-VpNadVK390H+F387SpET28a (+)-BaPRS-VpNadVK390H+R382GKmSmKmSmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKmKm本实验室本实验室本实验室本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究本研究
1.1.2 实验试剂
本研究所使用的实验试剂包括分子实验相关试剂与菌株培养所需培养基。2×Taq PCR Master Mix、Phanta®Max Super-Fidelity DNA polymerase,南京诺唯赞生物科技有限公司;快速质粒小提试剂盒、5 min DNA快速纯化试剂盒,北京全式金生物技术有限公司;基因、引物合成及测序相关技术,生工生物工程(上海)股份有限公司;QuickMutationTM基因随机突变试剂盒,碧云天生物技术有限公司。
大肠杆菌菌株所需要培养基包括常规LB培养基、摇瓶发酵培养基MA1和发酵罐培养基MA2。培养基具体成分如下:a) LB培养基(g/L):胰蛋白胨10,酵母提取物5,氯化钠10(加入质量分数为1.5%琼脂粉用于制备固体培养基);b) 摇瓶发酵培养基MA1(g/L):KH2PO4 6、K2HPO4 16.4、(NH4)2SO4 7.5、一水合柠檬酸1.1、MgSO4 1、酵母粉10、葡萄糖20;c)发酵罐培养基MA2(g/L):KH2PO4 6、K2HPO4 16.4、(NH4)2SO4 7.5、一水合柠檬酸1.1、MgSO4 1、酵母粉10、玉米浆3、烟酸0.1、葡萄糖10(初始)、微量元素溶液2 mL/L。
1.2 仪器与设备
DYY-6D电泳电源、DYCP-32B电泳仪,北京六一电泳仪厂;Mastercycler® nexus X2 PCR仪、Centrifuge 5420台式离心机、5920R 冷冻离心机,艾本德(上海)国际贸易有限公司;HDPF-256恒温培养箱,上海跃进医疗器械厂; GelDoc XR+凝胶成像仪,伯乐生命医学产品(上海)有限公司;NanoDrop One超微量分光光度计与SYNERGY-H1酶标仪,赛默飞世尔科技(中国)有限公司;QPix420微生物筛选系统,美谷分子仪器(上海)有限公司;722N可见分光光度计,上海菁华科技仪器有限公司;SHIMADZU LC-20AT高效液相色谱仪,日本岛津公司。
1.3 实验方法
1.3.1 大肠杆菌基因编辑
利用CRISPR/Cas9基因编辑进行大肠杆菌基因组位点敲除与整合。p-Cas9质粒转化至化学感受态细胞制作成电转化感受态细胞。将模板基因和p-Tatget质粒按浓度比例3∶1加入电转化感受态细胞,冰孵20 min后电击,加入LB培养基,在37 ℃/220 r/min条件下孵育1~2 h,然后涂布在LB平板上,37 ℃过夜培养;挑取单菌落进行菌落PCR;验证成功菌株转接至LB培养基,加入终浓度10 mmol/L的鼠李糖,37 ℃/220 r/min培养12~16 h,转板划线后37 ℃过夜培养,以验证消除p-Target质粒;消除成功菌株转接至LB培养基,加入终浓度10 mmol/L的蔗糖,37 ℃/220 r/min培养12~16 h,转板划线后37 ℃过夜培养,以验证消除p-Cas9质粒;消除成功菌株制备化学感受态细胞以供下一步研究。
1.3.2 Qucik Mutation 随机突变(易错PCR)
QuickMutationTM基因随机突变试剂盒(QuickMutationTM random mutagenesis kit)是一种基于易错PCR(error prone PCR)的方法使目的基因片段通过PCR扩增后发生一定几率的随机突变的试剂盒。反应体系包括:RandomMut buffer (10×),5 μL;Mutation enhancer (10×),5 μL;dNTP (2.5 mmol/L),5 μL;模板DNA,Y μL;引物混合物(10 μmol/L),1 μL;最后用超纯水定容补充至50 μL。
1.3.3 蛋白表达、纯化与酶活性测定
转化后接种至LB培养基37 ℃/220 r/min培养10 h,按照2%的转接量转接至MA1培养基。当OD600值为0.6~1.0时,加入异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside,IPTG)诱导蛋白表达,同时降温至25 ℃继续培养12 h。收集细胞并重悬于10 mmol/L的PBS缓冲液中,超声处理,37 ℃条件下向混悬液中加入NAM,用HPLC检测NMN合成情况。利用Ni+与附着在蛋白质上的His标签的高亲和力来纯化目标蛋白质。用75 mmol/L咪唑溶液洗涤镍柱,用105 mmol/L咪唑溶液洗脱蛋白质。随后,使用脱盐柱除去蛋白质溶液中的咪唑,最后将蛋白质加入到PBS溶液(pH=7.0)中。一个单位的酶活性定义为每分钟合成1 μmol NMN所需的酶量。
1.3.4 NMN的荧光反应
ZHANG等[17]开发的NMN荧光衍生法适用于多孔板筛选,14 000 r/min离心,收集全细胞催化后的反应液。每孔最终体积为250 μL,由69 μL上清液样品、27.7 μL溶于二甲基亚砜(dimethyl sulfoxide,DMSO)的体积分数为20%的苯乙酮溶液和27.7 μL的2 mol/L KOH组成。冰孵2 min后,加入125 μL体积分数为88%的甲酸,37 ℃孵育10 min。
1.3.5 同源建模与分子对接
基于人源NAMPT(PDB ID:8 dsc)的晶体结构,构建VpNadV的结构模型。使用Swiss-Model Web服务器预测VpNadV及其突变体的三维结构,其中模型的序列一致性为51.43%、全局质量指标GMQE为0.73。将活性位点视为Caver计算的起始点,并使用PRPP、NAM对接至底物口袋,Mg2+作为配体,将最小探针半径的大小设置为0.9 μm,壳深度设置为4 μm,壳半径设置为3 μm,聚类阈值为3.5 μm,最大距离为3 μm,期望半径为5 μm。使用PyMOL程序可视化所得结构和氢键网络图。
1.3.6 孔板/摇瓶发酵
质粒转化至感受态细胞LB平板37 ℃过夜培养,多菌落接种至LB培养基,37 ℃/220 r/min培养10~12 h。以2%的转接量接种至MA1培养基,37 ℃/220 r/min培养。当OD600值达到0.6~1.0时,加入0.5 mmol/L IPTG以诱导蛋白质表达,同时加入适量底物NAM。
1.3.7 5 L发酵罐发酵
质粒转化至感受态细胞LB平板37 ℃过夜培养,单菌落转板划线,37 ℃培养10 h,挑取5~6个划线菌落接种至LB培养基,37 ℃/220 r/min培养12 h作为种子液。以2%的转接量接种至MA2培养基。通过自动流加NH4OH(50%,体积分数)将pH值维持在6.9。通过溶氧(dissolved oxygen,DO)与搅拌转速关联,将DO保持在30%。当OD600值达到10时,加入终浓度0.5 mmol/L的IPTG诱导蛋白表达,同时流加底物NAM。当葡萄糖耗尽至5 g/L,以适当的速率流加葡萄糖,并维持葡萄糖质量浓度在3 g/L[18]。
1.4 数据处理
1.4.1 荧光强度检测
酶标仪在发射光445 nm及激发光382 nm条件下测定荧光强度。荧光强度与样品中荧光物质的浓度成正比,可用于定量分析。
1.4.2 发酵产物的提取和HPLC检测
全细胞催化样品14 000 r/min离心5 min取上清液,使用高效液相色谱系统进行分析,该系统配备了可变紫外波长检测器和Agilent C18柱(250 mm×4.6 mm,5 μm)。流动相为纯甲醇和1 g/L的庚烷磺酸钠盐溶液,并添加30 μL磷酸,流速0.8 mL/min,进样量10 μL,等度梯度洗脱20 min,检测波长为259 nm。出峰时间:NMN在3~4 min,烟酰胺在6~7 min,烟酸在4~5 min。
2 结果与分析
2.1 积累NMN底盘菌株的优化
2.1.1 消除烟酰胺向烟酸的降解
通过KEGG代谢网络分析及文献调研发现,烟酰胺酶(pncA基因编码)在生物体内催化NAM向NA的转化。这一反应不仅消耗NAM,还可能通过下游代谢途径影响NAD+的稳态平衡。在大肠杆菌中,NAM过度降解会导致NMN合成量降低,尤其是在全细胞催化体系中。本研究敲除pncA基因阻断了NAM向NA的降解,从而减少前体流失并提高NMN的合成量。基于上述发现,本研究在前期构建的NMN合成菌株F017基础上,采用CRISPR-Cas9基因编辑敲除pncA,构建pncA缺陷型菌株Z09(图1-A)。
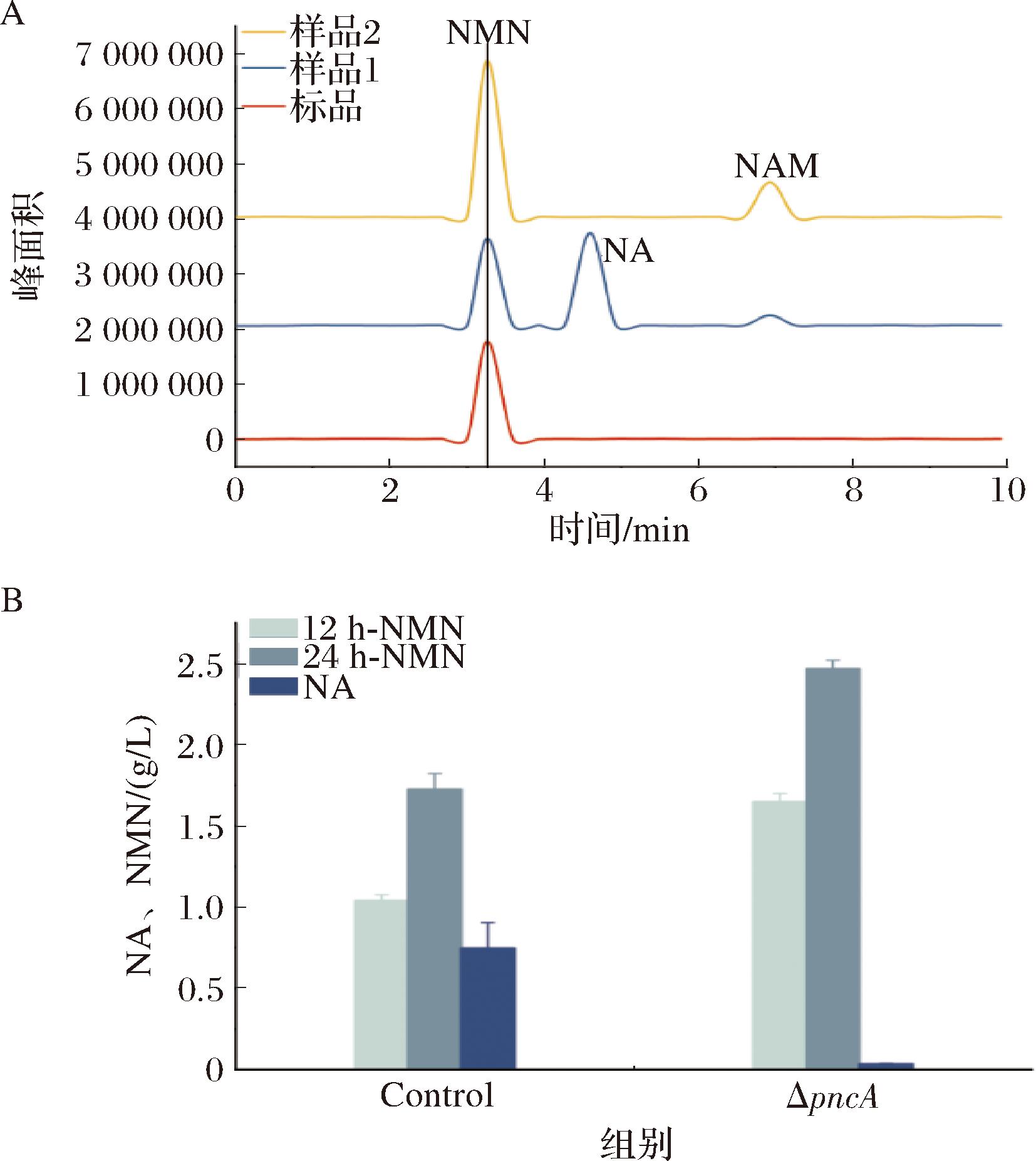
A-pncA成功敲除的HPLC验证(样品1:未ΔpncA,样品2:ΔpncA);B-未敲除与敲除pncA菌株的摇瓶发酵
图1 敲除pncA以抑制NA的生成
Fig.1 Knocking out pncA to inhibit NA generation.
摇瓶发酵实验表明,出发菌株在发酵24 h时,NA浓度累积达0.8 g/L,严重影响NMN合成量;而菌株Z09中,NA几乎完全被抑制,生物量较优于出发菌株,NMN产量也达到了2.48 g/L,表明pncA敲除未引入额外的代谢负担且有益于菌株的生长。(图1-B)。
2.1.2 NMN转运蛋白基因组多拷贝位点的整合
在NMN生物合成体系中,源自Bacillus mycoides的转运蛋白PnuC对NMN的外排具有关键调控作用。当前NMN高产菌株普遍采用多质粒共表达系统,同时携带NMN合成模块(如NAMPT、NMNAT等)和BMpnuc过表达质粒。然而,多质粒系统导致的代谢负担,会显著影响宿主细胞的生长状态。本研究将BMpnuc基因通过基因组多位点整合实现多拷贝表达,旨在构建代谢负荷较低的NMN合成菌株。选取E.coli BL21(DE3)中5个已验证的非必需假基因位点作为整合靶点:gapC、yoeA、yncI、yjiV和yciQ。这些位点经证实,敲除后对细胞生长速率、中心碳代谢及能量状态无显著影响。采用CRISPR-Cas9基因编辑构建不同拷贝数的整合菌株。
摇瓶发酵实验表明,基因组整合菌株的生长性能显著改善。在添加1 g/L NAM的条件下,质粒菌株的OD600最大值为6.7,而基因组菌株为8.5~10.1。值得注意的是,菌株Z13的生长情况最优,发酵24 h后NMN积累量达到2.51 g/L,与对照菌株提升较少(图2)。而菌株Z14 NMN产量降低原因可能为过度表达导致的膜电位失衡。证实基因组多拷贝策略可在降低代谢负荷的同时维持关键转运蛋白的功能表达。
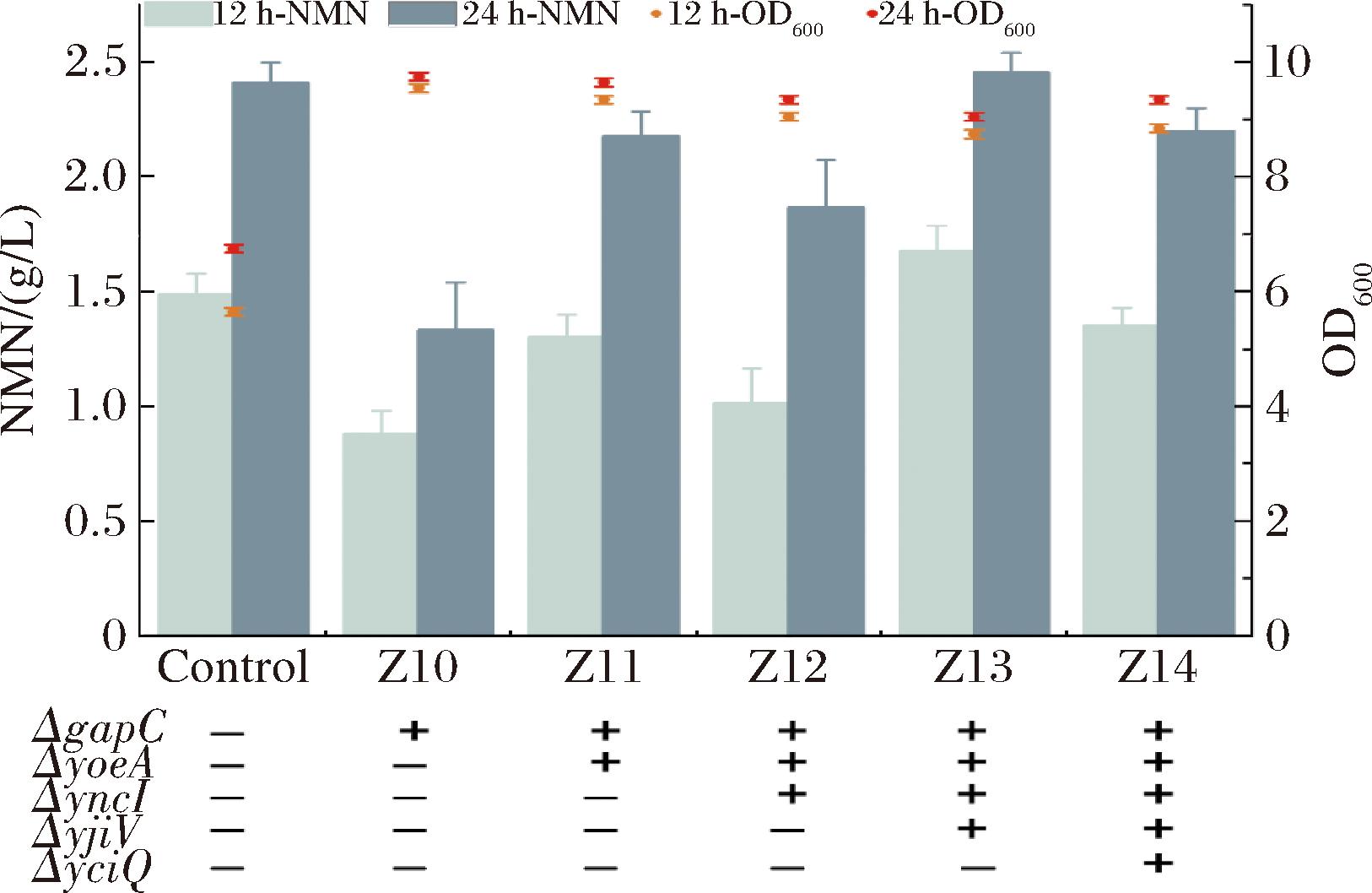
图2 NMN转运蛋白PnuC多拷贝菌株在摇瓶发酵中NMN合成与菌株的生长情况
Fig.2 The synthesis of NMN in the NMN transporter PnuC multi-copy strains in shake flask fermentation and the growth of strains
2.2 高通量筛选系统的建立与优化
本研究开发了基于NMN的荧光偶联筛选系统。首先,核心酶元件选择了弧菌噬菌体(Vibrio bacteriophage)来源的烟酰胺核糖基转移酶VpNadV作为进化模板,该酶具有热稳定性优异、底物特异性强等优势。然后,构建突变文库以重组质粒N020(携带VpNadV基因)为模板,采用易错PCR获得突变基因,将突变基因克隆至载体,最终构建的突变文库覆盖单点突变和三点以下组合突变。最后设计开发了多模块偶联检测体系:a)催化模块,酶催化NAM和PRPP生成NMN;b)报告模块,配置荧光反应体系,检测荧光强度;c)验证筛选模块,测序确定突变位点后进行不同级别生物反应器的发酵筛选(图3)。
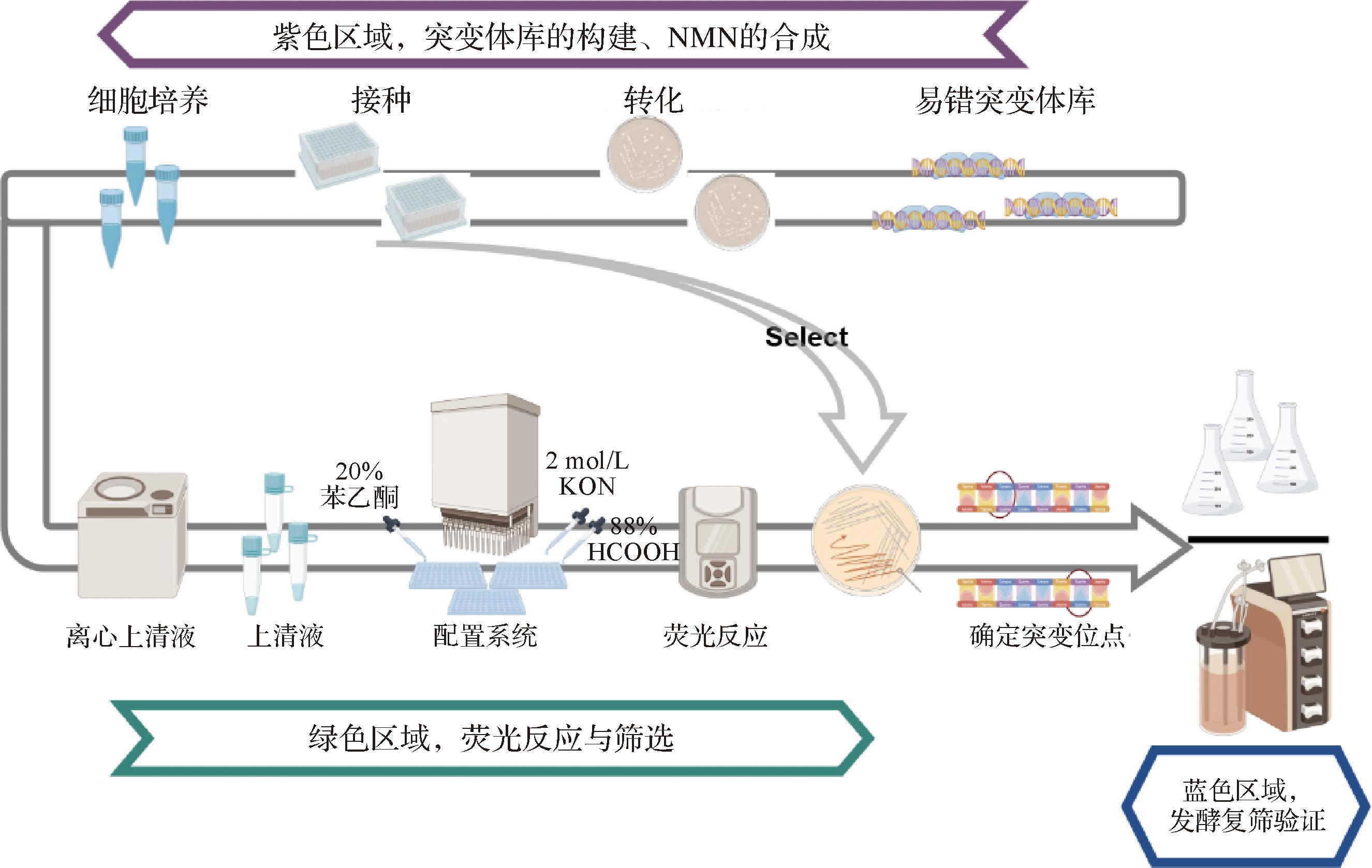
图3 基于NMN荧光反应的高通量筛选系统流程
Fig.3 High-throughput screening system process based on NMN fluorescence reaction
该荧光反应体系包含2个步骤:a)NMN与KOH反应生成荧光物质1;b)荧光物质1与甲酸反应生成荧光物质2(图4-A)。针对步骤1,优化了0 ℃与4 ℃下的不同反应时长,结果显示,在4 ℃下反应所产生的荧光强度比0 ℃下更强。而且在4 ℃条件下,当反应延长至3 min时,荧光强度达到最高,为0 ℃条件下的1.15倍,此后继续延长反应时间,荧光强度不再受到影响。因此,确定了步骤1的最佳反应条件为4 ℃、反应时长3 min(图4-B)。基于步骤1的最优条件,又优化了步骤2,研究显示该反应在1 min时可完成约90%,至10 min时完全反应,此后荧光强度仍在一段时间保持稳定,常将10 min作为该步骤的最佳反应时间。然而,对于发酵上清液,相较于纯酶体系,其中存在更多杂蛋白或杂质。将该步骤的反应时间设定为20 min,即荧光强度达到完全稳定。随后,通过配制不同浓度梯度的NMN标准液,由此获得了质量浓度为10 ~110 mg/L且相关系数为0.97的校准曲线(图4-C)。为使发酵液中的NMN合成量处于线性范围,还确定了诱导后的最佳发酵时间为10 h。
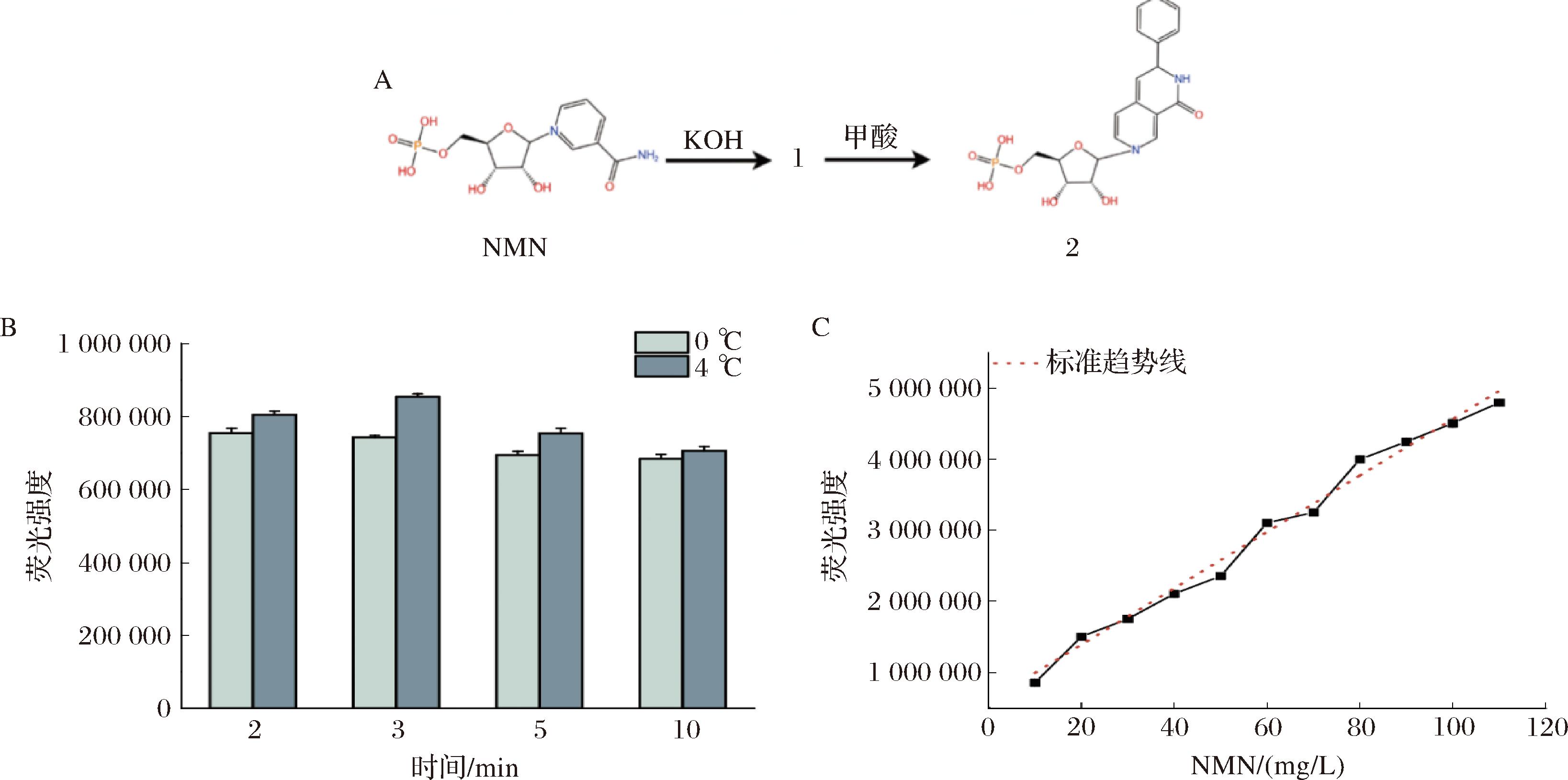
A-荧光反应;B-步骤1中不同反应时间和温度对NMN最终荧光的影响;C-荧光与NMN的标准曲线
图4 反应条件的优化
Fig.4 Optimization of reaction conditions
2.3 最佳VpNadV 突变体的发掘
2.3.1 高通量筛选获得阳性突变体
本研究基于定向进化策略,采用易错PCR技术成功构建了包含13 000个随机突变体的VpNadV酶突变文库。通过前期构建的高通量筛选平台,实现了对大规模突变体的快速筛选。在初筛阶段发现95个突变体表现出显著增强的催化活性,其荧光强度较野生型提高30%以上。经测序确定突变位点后,通过摇瓶发酵复筛后最终获得6个能够使NMN合成量显著提升的阳性突变体(VpNadVR26E、VpNadVL139V、VpNadVD383G、VpNadVF387S、VpNadVK390P、VpNadVA473V)。其中,突变体VpNadVR26E在添加1.25 g/LNAM时,NMN的最高产量为2.98 g/L,相比对照组提升32.9%,底物转化率为88.5%;突变体VpNadVL139V在添加1.5 g/LNAM时,NMN的最高产量3.24 g/L,相比对照组提升43.1%,底物转化率为79.8 %(图5)。
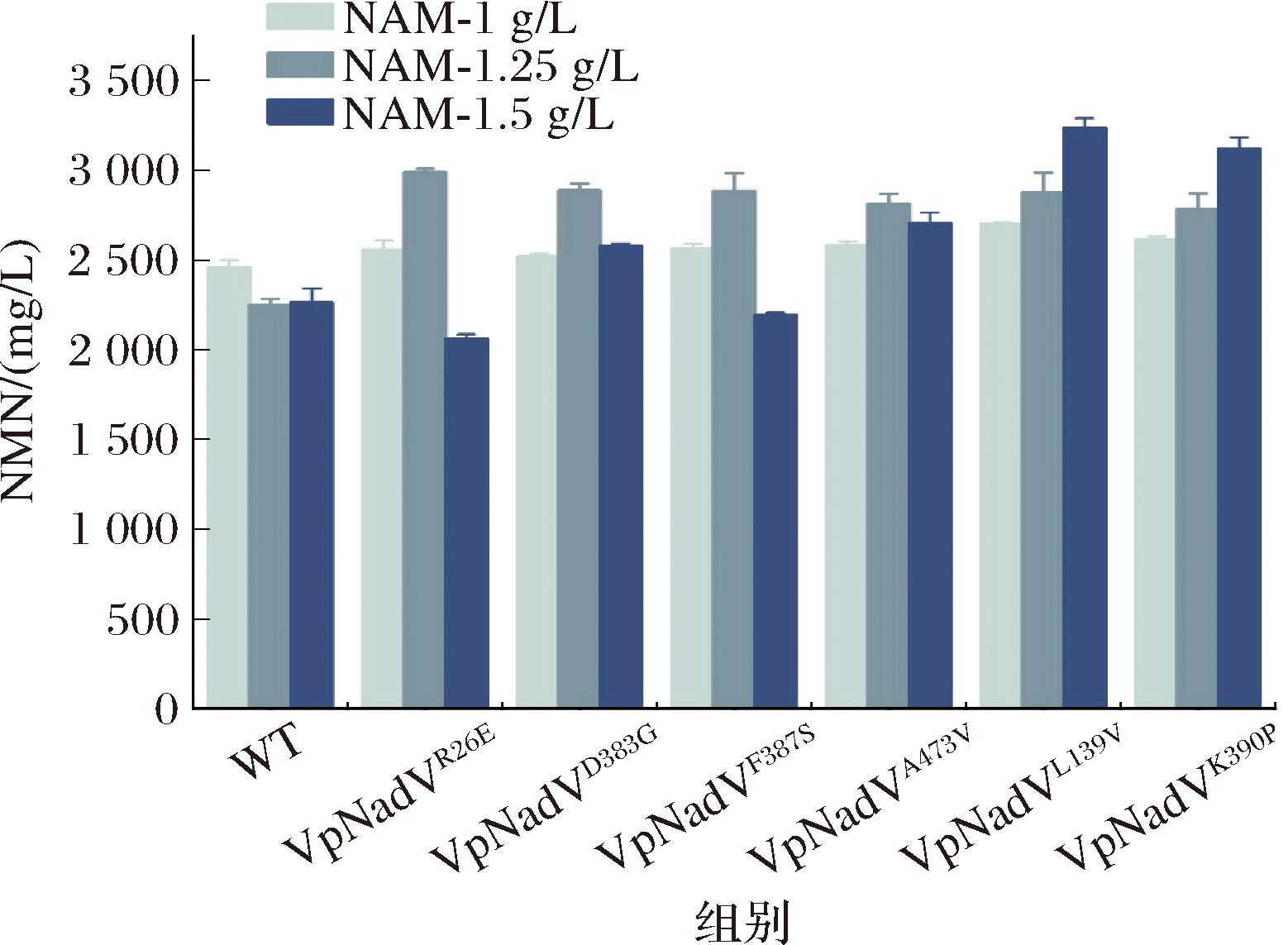
图5 突变菌株摇瓶发酵结果
Fig.5 Fermentation results of mutant strains shake flasks
2.3.2 酶工程发掘阳性突变体
酶的单底物催化反应定向进化策略主要针对酶活性中心位点或酶通道附近的氨基酸残基的进化[19-20],目前关于大体积的酶结构如VpNadV,且为双底物取代反应的进化策略的报道很少。为了发掘具有潜力的VpNadV突变体,首先对VpNadV进行数据库的序列比对和同源建模,分子对接模拟后得到VpNadV酶结构的初步模型,发现该酶以二聚体的形式存在(图6-A)。
NAMPT催化过程是一个取代反应[21]。通过模型分析发现,NAM作为较小分子率先进入底物口袋,而PRPP在ATP的激活下与Mg2+形成配位键之后,进入底物口袋作用于NAM吡啶环上的氮原子,导致NMN的合成。通过对活性中心区域氨基酸残基进行丙氨酸扫描后,确定了10个关键氨基酸残基(图6-B)。以这些位点为基础,建立饱和突变文库,结合所建立的高通量筛选系统进行筛选。摇瓶复筛后,得到了另外的3株阳性突变体VpNadVL139H、VpNadVR382G、VpNadVK390H,其中突变体VpNadVL139H在添加1.25 g/L NAM时,NMN的最高产量为3.13 g/L,相比于对照组提升39.4%,底物转化率为92.8%(图6-C)。对突变体VpNadVF387S、VpNadVL139V、VpNadVK390P、VpNadVL139H、VpNadVR382G、VpNadVK380H进行叠加突变,摇瓶发酵结果分析,突变体VpNadVL139V+R382G在添加1.5 g/L NAM时,NMN的最高产量为3.68 g/L,相比于对照组提升63.18%,底物转化率为91%(图6-D)。
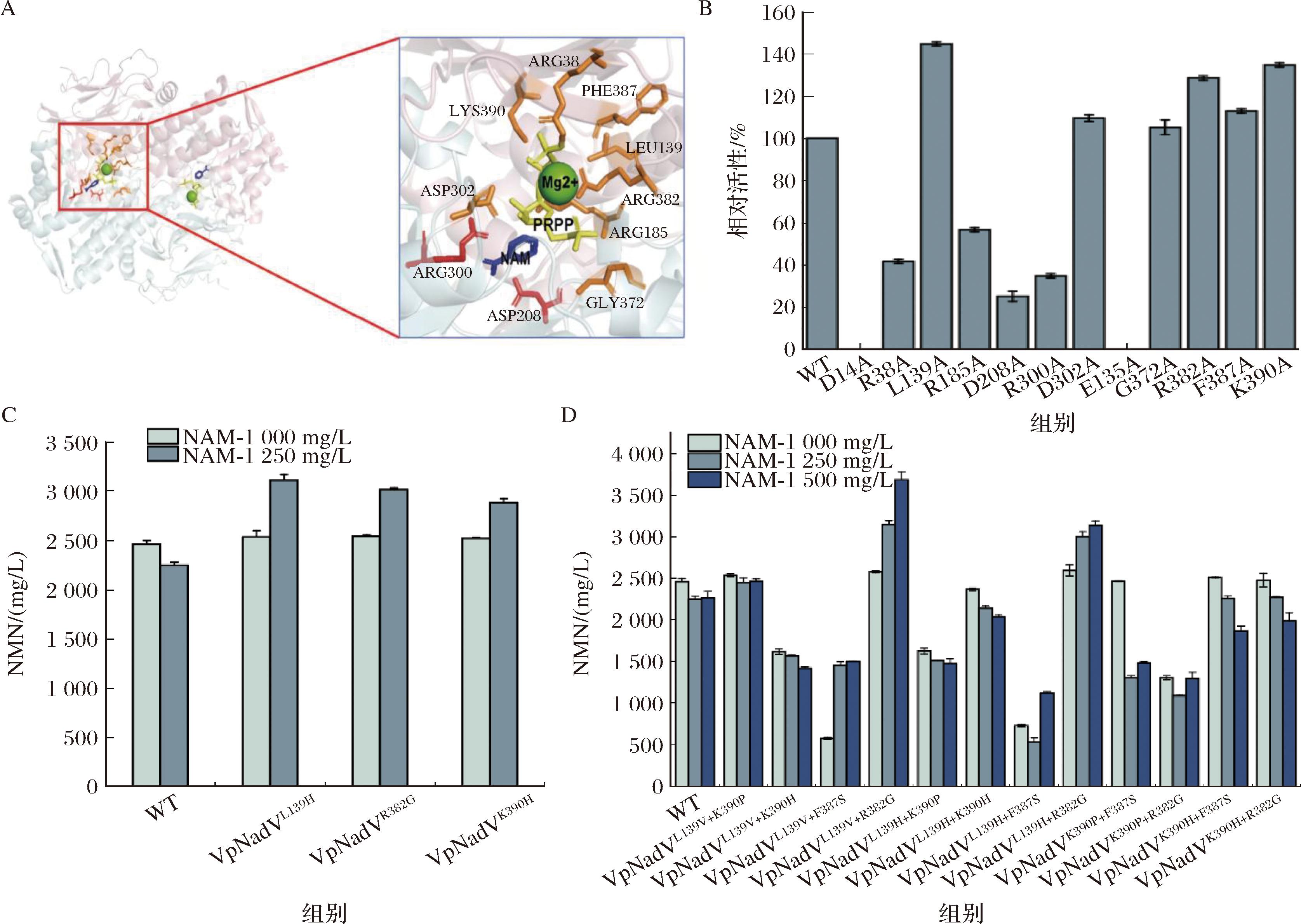
A-催化口袋示意图;B-关键氨基酸残基的丙氨酸扫描;C、D-不同突变体的摇瓶发酵验证
图6 酶工程改造发掘阳性突变体
Fig.6 Enzyme engineering to unearth positive mutants
2.4 5 L发酵罐中生物合成NMN的发酵优化
对所得阳性突变体进行酶活性验证,其中突变体VpNadVL139V+R382G、VpNadVL139H+R382G有着较高活性,相比于野生型VpNadV分别提高46.1%和41.2%(图7-A)。首先为确定5 L发酵罐培养的最适底盘菌株,分别观察底盘菌株Z09与Z13在5 L发酵罐中的生长与NMN合成情况。发现菌株Z13虽然在孔板、摇瓶等小型生物反应器中的OD600值得到了提高,但是在放大培养过程当中,可能由于多条假基因位点的敲除导致发酵过程中其他有害副产物的积累,从而导致了其在发酵罐中的生长情况较差,NMN的合成情况也就较差。因此,为观察突变体在5 L发酵罐放大培养时对NMN合成的影响,选择底盘菌株Z09作为底盘菌株进行发酵验证。分别构建重组菌株ZY150和ZY151,在发酵过程中将DO水平控制在30%,OD600值为10时加入IPTG诱导,同时以适当的流速流加底物NAM,发酵36 h后NMN的产量分别为22.46 g/L和20.23 g/L(图7-B、图7-C),所以选择重组菌株ZY150进行发酵优化。为了进一步提高发酵过程的生物量,尝试在OD600值达到20时再加入IPTG诱导蛋白表达,同时控制底物NAM的流速(前期高流速后期低流速),发酵36 h后NMN产量达到24.4 g/L(图7-D)。结果表明,适当提高发酵过程诱导时的OD600值提高菌体量有利于NMN的积累。
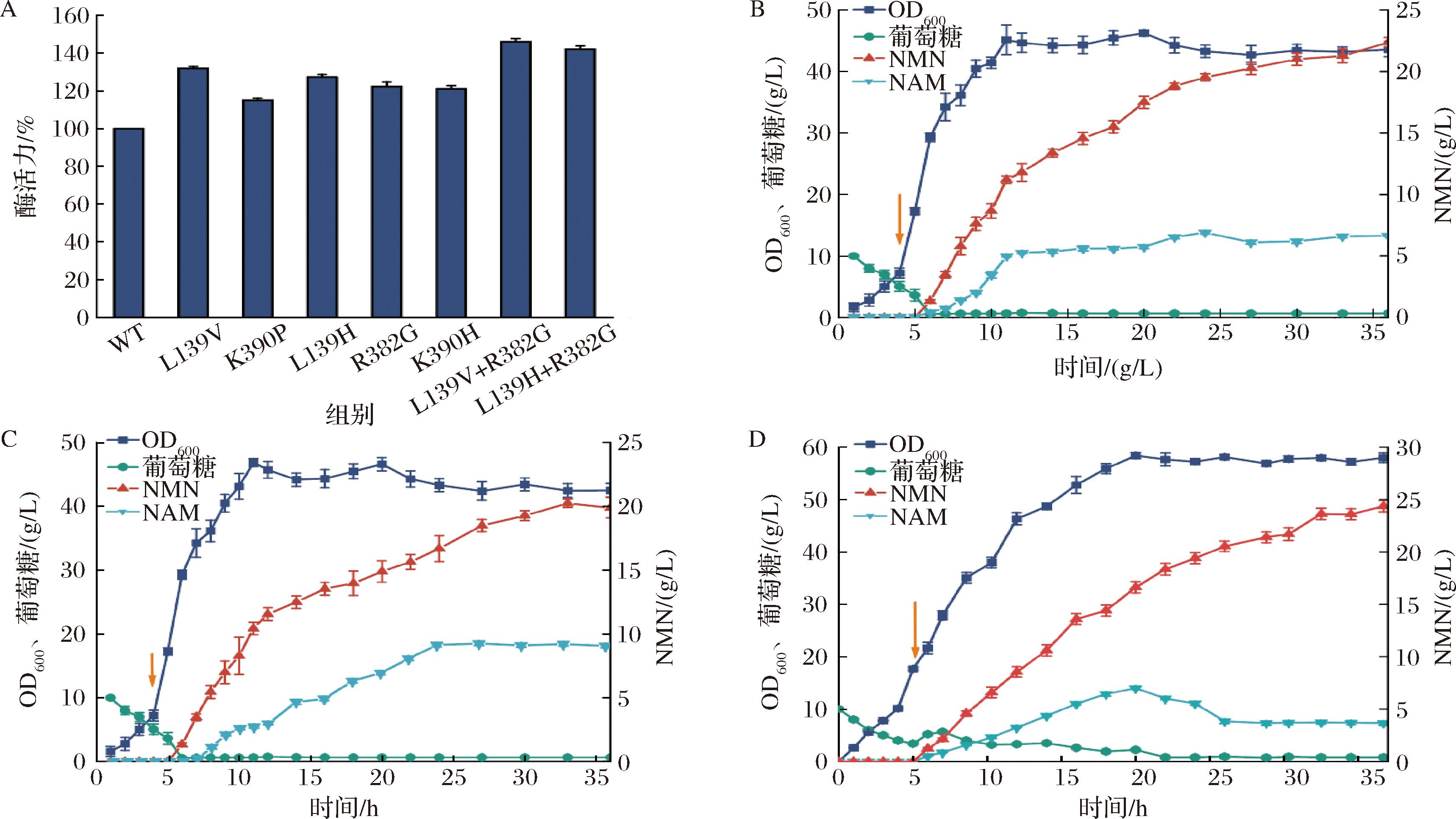
A-阳性突变体的酶活验证;B-重组菌株ZY150的5 L发酵罐培养;C-重组菌株ZY151的5 L发酵罐培养;D-OD600=20诱导的重组菌株ZY150的5 L发酵罐培养
图7 突变体菌株的5 L发酵罐优化与发酵条件优化
Fig.7 Optimization of 5 L fermentation tank and fermentation conditions of mutant strains
3 讨论
NMN作为治疗衰老以及退行性疾病的关键NAM类衍生物,其具有深远的研究意义。本研究立足于高通量筛选与酶工程发掘VpNadV的高效突变体,从而提高NMN的合成量。首先对现有NMN合成菌株进行代谢改造,在一定程度上改善了菌株在生物反应器中的生长情况。然后,根据NMN荧光反应构建新型高通量筛选系统,通过构建突变文库,筛选得到VpNadV阳性突变体,提高了NMN在摇瓶中的积累量。随后,利用酶工程改造结合迭代突变确定最佳突变体VpNadVL139V+R382G,其活性为野生型的1.46倍。通过5 L发酵罐的验证与优化,最终突变体VpNadVL139V+R382G能够显著提高NMN的合成量。所建立的高通量筛选系统为NAMPT的研究和NMN等相关衍生物的合成提供了新的研究方向。
NMN的生物合成仍存在研究潜力:a)本研究中突变体活性提升的机制尚不明确,突变体的完整晶体结构与分子动力学特征尚需解析;b)现有研究表明,NMN的转运是提高NMN胞外产量的关键,进一步挖掘更高效的转运蛋白或许是提高NMN产量的重要因素,此外,NMN转运蛋白PnuC的转运机制仍不明确,可进一步通过蛋白质工程改造实现更有效的NMN转运;c)筛选得到的突变体VpNadVR26E、VpNadVD383G、VpNadVA473V,其位点离活性中心相对较远,却在一定程度上提高了NMN的合成量,其机制有待明确分析;d)通过代谢改造后改善了底盘菌株在孔板、摇瓶中的生长状况,但在5 L发酵罐中菌株自身的生长情况却不能得到同等程度的改善,可能是多基因的敲除导致了其他有害物质的积累,后续需进一步优化;e)从头合成会避免产生NAM的高浓度底物抑制,但目前尚无关于大肠杆菌中NMN从头合成的相关报告,实际上,在大肠杆菌中烟酸单核苷酸(NaMN)是重要的中间体之一[22]。SORCI等[23]在F.tularensis中发现了具有催化NaMN合成NMN的NMN 合酶(FtNadE),这给NMN的从头合成提供了基础。综上,烟酰胺磷酸核糖基转移酶NAMPT的酶工程改造是提高NMN及其衍生物合成量的重要途经,本研究构建的高通量筛选系统和高催化活性的NAMPT突变体为NMN及其衍生物的研究提供了新思路。
[1] MILLS K F, YOSHIDA S, STEIN L R,et al.Long-term administration of nicotinamide mononucleotide mitigates age-associated physiological decline in mice[J].Cell Metabolism, 2016, 24(6):795-806.
[2] REVOLLO J R, GRIMM A A, IMAI S I.The regulation of nicotinamide adenine dinucleotide biosynthesis by Nampt/PBEF/visfatin in mammals[J].Current Opinion in Gastroenterology, 2007, 23(2):164-170.
[3] YOSHINO J, BAUR J A, IMAI S I.NAD+ intermediates:The biology and therapeutic potential of NMN and NR[J].Cell Metabolism, 2018, 27(3):513-528.
[4] XIE N, ZHANG L, GAO W,et al.NAD+metabolism:Pathophysiologic mechanisms and therapeutic potential[J].Signal Transduction and Targeted Therapy, 2020, 5:227.
[5] CAMPBELL J M.Supplementation with NAD+ and its precursors to prevent cognitive decline across disease contexts[J].Nutrients, 2022, 14(15):3231.
[6] FENG Z, QIN Y F, HUO F, et al.NMN recruits GSH to enhance GPX4-mediated ferroptosis defense in UV irradiation induced skin injury[J].Biochimica et Biophysica Acta(BBA)-Molecular Basis of Disease, 2022, 1868(1):166287.
[7] SHEN Q, ZHANG S J, XUE Y Z, et al.Biological synthesis of nicotinamide mononucleotide[J].Biotechnology Letters, 2021, 43(12):2199-2208.
[8] POLLARD C L, YOUNAN A, SWEGEN A,et al.Insights into the NAD+biosynthesis pathways involved during meiotic maturation and spindle formation in porcine oocytes[J].The Journal of Reproduction and Development, 2022, 68(3):216-224.
[9] GROTH B, VENKATAKRISHNAN P, LIN S J.NAD+ metabolism, metabolic stress, and infection[J].Frontiers in Molecular Biosciences, 2021, 8:686412.
[10] SHARMA S, HSIEH Y C, DIETZE J, et al.Early evolutionary selection of NAD biosynthesis pathway in bacteria[J].Metabolites, 2022, 12(7):569.
[11] ROGOZKIN V A, FEDOROVA G P, MASHANSKII V F.Enzymatic synthesis of nicotinamide dinucleotide in isolated skeletal muscle nuclei[J].Voprosy Meditsinskoi Khimii, 1964, 10:546-547.
[12] KONG J Y, DU C M, JIANG L H,et al.Nicotinamide phosphoribosyltransferase regulates cocaine reward through Sirtuin 1[J].Experimental Neurology, 2018, 307:52-61.
[13] SHOJI S, YAMAJI T, MAKINO H, et al.Metabolic design for selective production of nicotinamide mononucleotide from glucose and nicotinamide[J].Metabolic Engineering, 2021, 65:167-177.
[14] MARINESCU G C, POPESCU R G, STOIAN G, et al.β-nicotinamide mononucleotide (NMN) production in Escherichia coli [J].Scientific Reports, 2018, 8:12278.
[15] BURGOS E S, SCHRAMM V L.Weak coupling of ATP hydrolysis to the chemical equilibrium of human nicotinamide phosphoribosyltransferase[J].Biochemistry, 2008, 47(42):11086-11096.
[16] HUANG Z S, LI N, YU S Q, et al.Systematic engineering of Escherichia coli for efficient production of nicotinamide mononucleotide from nicotinamide[J].ACS Synthetic Biology, 2022, 11(9):2979-2988.
[17] ZHANG R Y, QIN Y, LV X Q, et al.A fluorometric assay for high-throughput screening targeting nicotinamide phosphoribosyltransferase[J].Analytical Biochemistry, 2011, 412(1):18-25.
[18] HUANG Z S, WANG X L, LI N, et al.Systematic engineering of Escherichia coli for efficient production of nicotinamide riboside from nicotinamide and 3-cyanopyridine[J].Bioresource Technology, 2023, 377:128953.
[19] LI Z Y, MENG S Q, NIE K L, et al.Flexibility regulation of loops surrounding the tunnel entrance in cytochrome P450 enhanced substrate access substantially[J].ACS Catalysis, 2022, 12(20):12800-12808.
[20] CHEN J L, WANG X, YANG F, et al.3D structure of the transient intermediate of the enzyme-substrate complex of sortase a reveals how calcium binding and substrate recognition cooperate in substrate activation[J].ACS Catalysis, 2023, 13(17):11610-11624.
[21] HOURY D, RAASAKKA A, FERRARIO E, et al.Identification of structural determinants of nicotinamide phosphoribosyl transferase (NAMPT) activity and substrate selectivity[J].Journal of Structural Biology, 2023, 215(3):108004.
[22] WANG X Y, ZHOU Y J, WANG L, et al.Engineering Escherichia coli nicotinic acid mononucleotide adenylyltransferase for fully active amidated NAD biosynthesis[J].Applied and Environmental Microbiology, 2017, 83(13):e00692-17.
[23] SORCI L, MARTYNOWSKI D, RODIONOV D A, et al.Nicotinamide mononucleotide synthetase is the key enzyme for an alternative route of NAD biosynthesis in Francisella tularensis[J].PNAS, 2009, 106(9):3083-3088.