肺炎链球菌(Streptococcus pneumoniae)是引起呼吸道感染的主要致病菌之一[1]。全球每年因肺炎链球菌感染导致肺炎、脑膜炎和中耳炎死亡的人数超过170万,其中70~100万为婴幼儿[2]。尽管肺炎链球菌疫苗已应用于临床,但由于血清型种类有限且疫苗成本较高[3],由肺炎链球菌引起的发病率依然居高不下。
3型荚膜多糖(type 3 capsular polysaccharide,Type 3 CPS)是肺炎链球菌血清型特异性荚膜多糖,不仅在保护肺炎链球菌免受宿主免疫系统攻击中发挥作用,而且对其毒力至关重要[4]。Type 3 CPS是肺炎链球菌的一个特定荚膜类型,能够增强其附着力和定殖能力,促进肺炎链球菌的呼吸道感染[5]。Type 3 CPS作为肺炎链球菌荚膜的重要组成成分,在疫苗开发、免疫逃逸机制研究、抗菌药物研发、快速诊断及致病机制的研究[6]中具有重要作用,并可能成为新的治疗靶点,有助于解决细菌耐药性和免疫逃逸问题[7]。因此,合成Type 3 CPS在肺炎链球菌特异性疫苗的开发中具有重要的应用价值。
Type 3 CPS是由属于糖基转移酶家族2的Cps3B合酶通过交替连接葡萄糖醛酸(glucuronic acid,GlcA)与葡萄糖(glucose,Glc)为二糖单元形成的酸性直链多糖[8],其结构为[3)-β-D-GlcUA-β(1-4)-Glc-β(1-]n。Type 3 CPS与另一类更为常见的链球菌荚膜多糖透明质酸(hyaluronic acid,HA)具有一定的结构相似性,HA的糖链结构为[4)-β-D-GlcA-β(1-3)-GlcNAc-β(1-]n,其中GlcNAc为N-乙酰氨基葡萄糖(N-acetylglucosamine)[9]。HA作为人体细胞外基质的重要组成成分,已广泛应用于医学、化妆品、营养补充和食品等多个领域[10]。因具有卓越的保湿、润滑和修复功能,成为抗衰老、皮肤护理和关节保健等各种产品的重要组分[11]。当前已经报道了以多种微生物(如谷氨酸棒杆菌[12]、枯草芽孢杆菌[13]、大肠杆菌[14])为底盘构建HA细胞工厂,其中以谷氨酸棒杆菌为底盘构建的HA细胞工厂生成效率最高,产量已经达到74.2 g/L[15]。
尽管Type 3 CPS具有重要的研究与应用价值,但目前缺乏Type 3 CPS细胞工厂开发与生物合成相关的报道。本研究目的是借鉴HA细胞工厂构建的成功经验[15-16],探索Type 3 CPS细胞工厂的构建策略,以推动这一类重要多糖的代谢工程与发酵合成研究。本研究通过基因簇分析、蛋白质结构模拟等技术明确合成Type 3 CPS合成所需要的关键酶元件。通过基因表达调控,构建以谷氨酸棒杆菌和大肠杆菌为底盘细胞的Type 3 CPS微生物细胞工厂,并通过分批发酵分析测试微生物细胞工厂以廉价碳源为底物发酵合成Type 3 CPS的可行性和合成效率。
1 材料与方法
1.1 材料与试剂
1.1.1 质粒与菌株
本文使用的质粒详见表1,菌株详见表2。
表1 质粒基本信息
Table 1 Basic information of the plasmids

质粒特征来源pEC-XK99E大肠杆菌-谷氨酸棒状杆菌穿梭表达载体,Ptrc启动子,卡那霉素抗性,pGA1谷氨酸棒状杆菌复制子实验室保存pEC-XK99E-Cps3BpEC-XK99E质粒表达肺炎链球菌Type 3 CPS合成酶基因cps3B本研究构建pEC-XK99E-Cps3B-GalUpEC-XK99E质粒表达肺炎链球菌Type 3 CPS合成酶基因cps3B、UTP-葡萄糖-1-磷酸尿苷基转移酶基因galU本研究构建pEC-XK99E-Cps3B-GalU-UgdpEC-XK99E质粒表达肺炎链球菌Type 3 CPS合成酶基因cps3B、UTP-葡萄糖-1-磷酸尿苷基转移酶基因ga-lU、UDP-葡萄糖-6-脱氢酶基因ugd本研究构建pEC-XK99E-Cps3B-CpsC-GalUpEC-XK99E质粒表达肺炎链球菌Type 3 CPS合成酶基因cps3B、辅助亚基基因cpsC、UTP-葡萄糖-1-磷酸尿苷基转移酶基因galU本研究构建pEC-XK99E-Cps3B-CpsC-GalU-UgdpEC-XK99E质粒表达肺炎链球菌Type 3 CPS合成酶基因cps3B、辅助亚基基因cpsC、UTP-葡萄糖-1-磷酸尿苷基转移酶基因galU、UDP-葡萄糖-6-脱氢酶基因ugd本研究构建
表2 菌种基本信息
Table 2 Basic information of the bacterial strains
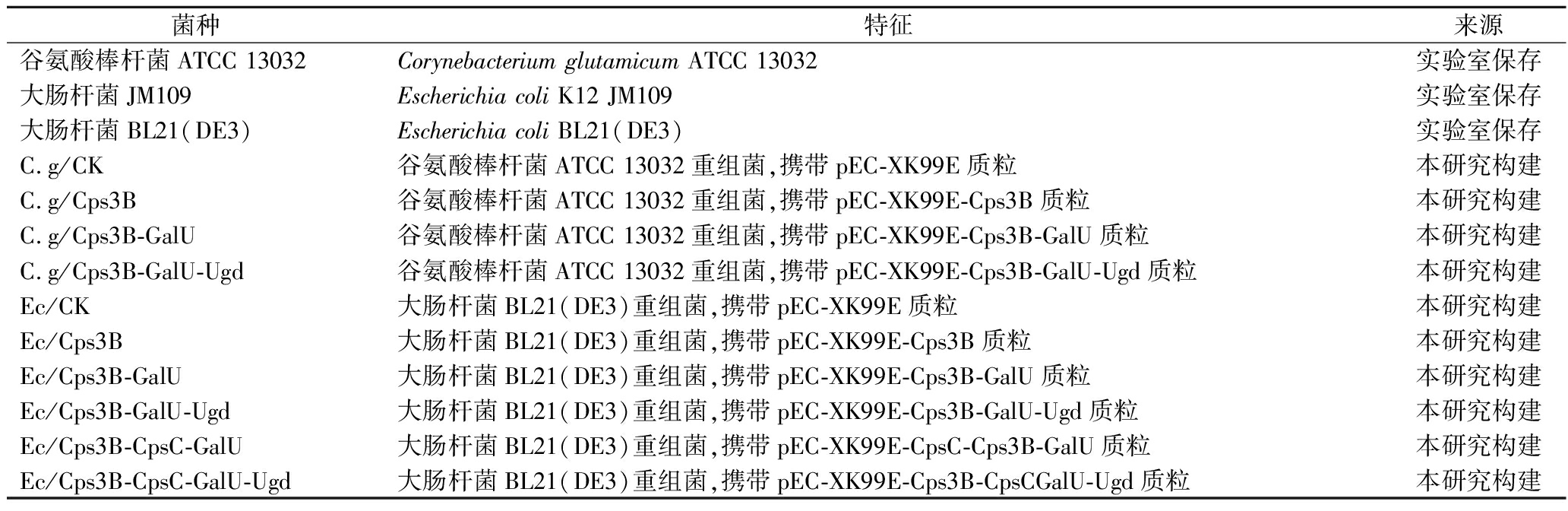
菌种特征来源谷氨酸棒杆菌ATCC 13032Corynebacterium glutamicum ATCC 13032实验室保存大肠杆菌JM109Escherichia coli K12 JM109实验室保存大肠杆菌BL21(DE3)Escherichia coli BL21(DE3)实验室保存C.g/CK谷氨酸棒杆菌ATCC 13032重组菌,携带pEC-XK99E质粒本研究构建C.g/Cps3B谷氨酸棒杆菌ATCC 13032重组菌,携带pEC-XK99E-Cps3B质粒本研究构建C.g/Cps3B-GalU谷氨酸棒杆菌ATCC 13032重组菌,携带pEC-XK99E-Cps3B-GalU质粒本研究构建C.g/Cps3B-GalU-Ugd谷氨酸棒杆菌ATCC 13032重组菌,携带pEC-XK99E-Cps3B-GalU-Ugd质粒本研究构建Ec/CK大肠杆菌BL21(DE3)重组菌,携带pEC-XK99E质粒本研究构建Ec/Cps3B大肠杆菌BL21(DE3)重组菌,携带pEC-XK99E-Cps3B质粒本研究构建Ec/Cps3B-GalU大肠杆菌BL21(DE3)重组菌,携带pEC-XK99E-Cps3B-GalU质粒本研究构建Ec/Cps3B-GalU-Ugd大肠杆菌BL21(DE3)重组菌,携带pEC-XK99E-Cps3B-GalU-Ugd质粒本研究构建Ec/Cps3B-CpsC-GalU大肠杆菌BL21(DE3)重组菌,携带pEC-XK99E-CpsC-Cps3B-GalU质粒本研究构建Ec/Cps3B-CpsC-GalU-Ugd大肠杆菌BL21(DE3)重组菌,携带pEC-XK99E-Cps3B-CpsCGalU-Ugd质粒本研究构建
1.1.2 试剂
葡萄糖醛酸标准样品(分析纯),上海麦克林生化科技有限公司;异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)、D-山梨醇、丙烷磺酸[3-(N-morpholino),MOPS]、玉米浆粉、硫酸卡那霉素(Kanamvcin,Kana)(分析纯),上海生工生物工程公司;2×Taq PCR Master Mix,南京诺唯赞生物科技有限公司;PrimeSTAR® Max DNA聚合酶,宝日医生物技术(北京)有限公司;DNA纯化试剂盒,Thermo Fisher Scientific公司;DNA连接/一步克隆试剂盒,翌圣生物科技(上海)股份有限公司。其余试剂均为国药集团分析纯试剂。
1.1.3 培养基
脑心浸液(brain heart infusion supplemented, BHIS)培养基(g/L):BHI 37、D-山梨醇91,pH 7.0。
谷氨酸棒杆菌发酵培养基(g/L):葡萄糖 40、玉米浆粉 20、(NH4)2SO4 20 、K2HPO4 1、1 KH2PO4 1、MgSO4 0.25和MOPS[3-(N-morpholino)丙烷磺酸]42,pH 7.0。
LB培养基(g/L): NaCl 10、酵母提取物 5、蛋白胨 10,pH 7.0。
大肠杆菌种子培养基(g/L):色氨酸 12、酵母浸提液 24、甘油 5、KH2PO4 2.3、K2HPO4 12.5,pH 7.0。
大肠杆菌发酵培养基(g/L)[17]:葡萄糖 20、硫胺素(维生素B1) 0.1、KH2PO4 13.5、(NH4)2HPO4 4、MgSO4·7H2O 1.4、柠檬酸 1.7、10 mL微量金属溶液、酵母提取物 6、色氨酸4,pH 7.0;
其中,微量金属溶液(5 mol/L HCl、10 g/L FeSO4·7H2O、2 g/L CaCl2、2.2 g/L ZnSO4·7H2O、0.5 g/L MnSO4·4H2O、1 g/L CuSO4·5H2O、0.1 g/L (NH4)6Mo7O24·4H2O、0.02 g/L Na2B4O7·10H2O)。
1.2 仪器设备
SW-CJ-2F双人双面净化工作台,苏州安泰空气技术有限公司;DHP-9272电热恒温培养箱,北京市永光明医疗仪器有限公司;ZQTY-70台式振荡培养箱,海知楚仪器有限公司;H1650-W医用离心机,湖南湘仪实验室仪器开发公司;T-100型PCR核酸扩增仪,伯乐生命医学产品有限公司;UV-1800紫外分光光度计,日本岛津公司;G180DWS高压蒸汽灭菌锅,致徽(厦门)仪器有限公司;M-100葡萄糖分析仪,深圳西尔曼科技有限公司;EclipseNi-E光学显微镜,日本东京尼康公司。
1.3 实验方法
1.3.1 质粒与重组菌株的构建
本研究利用大肠杆菌JM109进行质粒扩增和重组质粒构建。以大肠杆菌BL21(DE3)和谷氨酸棒杆菌ATCC 13032为底盘细胞,选育所有大肠杆菌与谷氨酸棒杆菌工程化菌株。以肺炎链球菌(Streptococcus pneumoniae)str.WU2的cps3B基因为模板在TWIST网站以大肠杆菌为表达宿主进行密码子优化,通过基因合成获得目的片段。将穿梭表达质粒pEC-XK99E用EcoR I/Kpn I进行双酶切,用Gibson Assembly试剂盒将cps3B基因片段一步连接到线性化的质粒pEC-XK99E上,将连接产物转化到大肠杆菌JM109中,用内源DNA重组酶环化,获得重组质粒pEC-XK99E-Cps3B。以恶臭假单胞菌(Pseudomonas putida)KT2440基因组为模板,扩增galU(编码葡萄糖-1-磷酸尿苷酰转移酶)、ugd(编码UDP-葡萄糖6-脱氢酶)。使用PrimeSTAR® Max DNA聚合酶将质粒pEC-XK99E-Cps3B质粒酶切线性化,并与galU、ugd PCR片段进行Gibson组装,随后转化JM109感受态细胞,获得重组质粒pEC-XK99E-Cps3B-GalU-Ugd和pEC-XK99E-Cps3B-GalU。后续在此基础上通过基因合成获得cpsC辅酶基因目的片段,以同样的方法连接到线性化的质粒pEC-XK99E-Cps3B-GalU-Ugd和pEC-XK99E-Cps3B-GalU中,获得pEC-XK99E-Cps3B-CpsC-GalU-Ugd和pEC-XK99E-Cps3B-CpsC-GalU。
采用电转化方法将质粒pEC-XK99E、pEC-XK99E-Cps3B、pEC-XK99E-Cps3B-GalU-Ugd和pEC-XK99E-Cps3B-GalU通过电转化转入到谷氨酸棒杆菌ATCC 13032感受态细胞中,电击条件:15 kV/cm,5 ms,随后46 ℃热处理6 min。恢复培养后在含有对应抗生素(25 mg/L的卡那霉素)BHIS平板上进行重组菌的筛选。采用化学方法将质粒pEC-XK99E、pEC-XK99E-Cps3B、pEC-XK99E-Cps3B-GalU-Ugd、pEC-XK99E-Cps3B-GalU、pEC-XK99E-Cps3B-CpsC-GalU-Ugd和pEC-XK99E-Cps3B-CpsC-GalU转入到大肠杆菌BL21(DE3)中,在含50 mg/L的卡那霉素LB平板上进行重组菌的筛选。
1.3.2 培养条件
大肠杆菌在LB培养基恒温37 ℃培养。谷氨酸棒杆菌在BHIS培养基恒温30 ℃培养。
谷氨酸棒杆菌的电转感受态细胞的制备。在BHIS平板上挑单克隆于BHIS液体培养基中过夜培养,以2%接种量转接到摇瓶中(添加25 mg/L卡那霉素),培养至OD600值为0.2时用体积分数10%的甘油洗涤3次,随后分装存放于-80 ℃。
在大肠杆菌BL21(DE3)中合成Type 3 CPS。在大肠杆菌发酵培养基中,30 ℃、200 r/min培养工程大肠杆菌:在LB平板上挑单克隆于大肠杆菌种子培养基中过夜培养,以在600 nm处初始光密度为0.2转接到摇瓶中(添加50 mg/L卡那霉素),在时间点1.5 h时加入0.1 mmol/L IPTG诱导目的基因表达。
在谷氨酸棒杆菌中合成Type 3 CPS。在谷氨酸棒杆菌发酵培养基中,30 ℃、200 r/min培养工程谷氨酸棒杆菌:在BHIS平板上挑单克隆于BHIS液体培养基中过夜培养,培养至OD600值为0.2转接到摇瓶中(添加25 mg/L卡那霉素),在时间点2.5 h时加入0.1 mmol/L IPTG诱导目的基因表达。
1.3.3 细胞生长和葡萄糖浓度测定
细胞生长通过动态测定OD600的变化进行监测。取对应时间菌液使用培养基进行稀释,稀释倍数根据菌体浓度而定,OD600测定值维持在0.5~1.0,测定值乘稀释倍数得到最终OD600值。
采用葡萄糖分析仪实时测定葡萄糖浓度。
1.3.4 Type 3 CPS浓度的测定
测量谷氨酸棒杆菌胞外总Type 3 CPS。发酵液16 000×g离心15 min除去菌体,将上清液与4倍体积的预冷乙醇混合,-30 ℃下过夜沉淀Type 3 CPS。
测量大肠杆菌胞内总Type 3 CPS。发酵液16 000×g离心15 min收集菌体,加入5 mL P1溶液(pH 8.0,25 mmol/L Tris-HCl、10 mmol/L EDTA、20 g/L溶菌酶)重悬细胞后37 ℃水浴1 h,加入5 mL P2溶液(0.2 mol/L NaOH溶液、质量分数1% SDS)溶解菌体,颠倒至液体澄清。最后加入7.5 mL P3(pH 6.0,3 mol/L 醋酸钾)使蛋白与基因组共沉淀并16 000×g离心15 min去除沉淀。上清液与4倍体积的预冷乙醇混合,-30 ℃下过夜沉淀Type 3 CPS。
将谷氨酸棒杆菌与大肠杆菌过夜沉淀的Type 3 CPS通过在16 000×g下离心10 min收集沉淀。在室温下蒸发残留的乙醇,并将半干燥的不溶性部分溶解在去离子水中,而通过离心除去水不溶性部分。为了去除尽可能多的杂质,将上述步骤重复3次。适当稀释(5~10倍,取决于Type 3 CPS浓度),使用葡萄糖醛酸作为标准物质,通过咔唑测定法测定Type 3 CPS含量[18]。由于谷氨酸棒杆菌天然可以合成葡萄糖醛酸,对产量测定存在干扰性,于是测量产量时生产菌株测得的葡萄糖醛酸含量减去对应野生型菌株测得的葡萄糖醛酸的含量,得到最终的数据作为生产菌株的产量,途径强化生产菌株的葡萄糖醛酸含量减去途径强化野生型菌株葡萄糖醛酸含量,得到最终的数据作为途径强化生产菌株的产量。
1.3.5 细胞形态分析
配制30 g/L的琼脂糖,加热溶解后均匀涂在载玻片上,将细胞滴加在琼脂糖垫的薄层上固定,使用配备有相差显微镜模块的Eclipse NiE显微镜进行相差显微镜观察。
1.3.6 蛋白质结构建模与分子动力学模拟
使用AlphaFold 3模拟蛋白质单体和复合体结构[19]模拟的蛋白质结构通过CHARMM-GUI嵌入磷脂双分子层结构。模拟系统的脂质双层膜由以下成分组成:50%(体积分数)1-棕榈酰-2-油酰-sn-甘油-3-磷酸甘油(1-palmitoyl-2-oleoyl-sn-glycero-3-phosphatidylglycerol,POPG), 25%(体积分数)1,2-二棕榈酰-磷脂酰肌醇(1,2-dipalmitoyl-phosphatidylinositol,DPPI), 12.5%(体积分数)1-棕榈酰-2-油酰-甘油二酯(1-palmitoyl-2-oleoyl diacylglycerol,POGL),以及12.5%(体积分数)1-棕榈酰-2-油酰-单半乳糖基二酰基甘油(1-palmitoyl-2-oleoyl-monogalactosyldiacylglycerol,PO-MGDG)糖脂。为确保适当的离子强度,系统中还包含了150 mmol/L KCl溶液。随后采用GROMACS 2020.6在30 ℃恒粒子数-恒压-恒温(number of particles-pressure-temperature,NPT)条件下进行分子动力学模拟:首先进行能量最小化(50 000步),以消除系统中的原子空间冲突,并优化其几何结构;随后进行平衡阶段(1 750 000步),使系统在温度、压力和体积方面稳定;最后,进行模拟阶段(25 000 000步)进行数据收集。
1.4 数据处理
蛋白质结构数据通过AlphaFold 3进行模拟并通过PyMOL进行可视化分析和图片导出。显微镜照片通过ImageJ进行裁剪和对比度处理[20]。OD600、葡萄糖浓度、Type 3 CPS产量平行测定3次,随后用Excel处理数据获得“平均值与标准差”,产量数据均采用双因素方差分析进行统计学显著性检验,并利用GraphPad Prism软件绘制曲线图和柱状图。
2 结果与分析
2.1 Type 3 CPS与HA合成途径的比较
通过GenBank获取肺炎链球菌与HA天然合成菌(酿脓链球菌)的基因组,分析发现cps3B基因上下游分别与cps3A和cps3C基因相邻(图1-a)。Type 3 CPS合酶与HA合酶都属于糖基转移酶2家族(glycosyltransferase family-2, GT-2)的蛋白质。HA是由GlcNAc和GlcA交替构成的二糖单位所组成的线性多糖,Type 3 CPS是由葡萄糖和葡萄糖醛酸交替连接而成的二糖单位所组成线性多糖(图1-b)。对Type 3 CPS合酶与HA合酶的AlphaFold 3模拟的蛋白质结构进行比较,发现两者具有高度相似性,2种合酶都包括1个跨膜螺旋和1个胞内亲水性区域,其中亲水性区域负责底物的结合与催化反应,而跨膜螺旋则负责将多糖输送至胞外。Cps3B合酶的胞内亲水性区域与HA合酶相似,具有相同的功能,而其跨膜区域比HA合酶的第1个跨膜螺旋TM1短(图1-c)。对Type 3 CPS与HA的合成途径进行比较,Type 3 CPS合成所需的UDP-葡萄糖和UDP-葡萄糖醛酸仅需依赖一条代谢途径,HA的合成除了UDP-葡萄糖醛酸的合成途径外,还需要通过氨糖代谢途径合成UDP-N-乙酰氨基葡萄糖(图1-d)。前期研究发现,在谷氨酸棒杆菌中表达HasA合酶和HasB(UDP-葡萄糖脱氢酶)即可合成HA,因此推断谷氨酸棒杆菌表达Cps3B合酶即可合成Type 3 CPS。
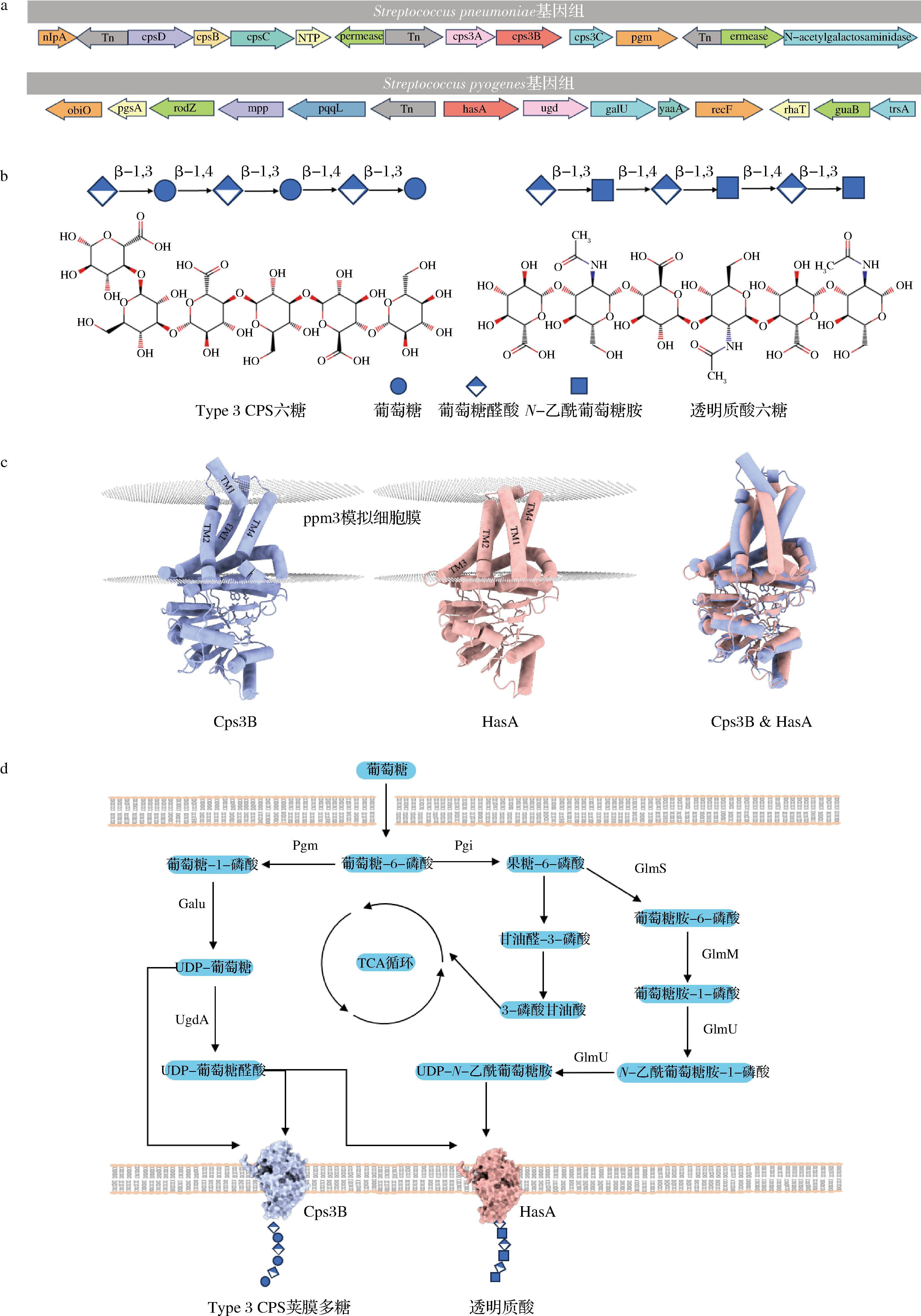
a-Type 3 CPS与HA的基因簇分析;b-Type 3 CPS与HA糖链结构比较;c-Type 3 CPS与HA合酶结构比较;d-Type 3 CPS与HA合成途径的比较
图1 Type 3 CPS与HA合成基因簇、糖链结构、合酶结构和单糖单元合成途径的比较
Fig.1 Comparison of Type 3 CPS and HA synthesis gene clusters, polysaccharide chain structure, synthase structures, and monosaccharide precursor synthesis pathways

a-谷氨酸棒杆菌重组菌发酵过程中的菌体形态变化;b-谷氨酸棒杆菌重组菌发酵过程中的OD600与葡萄糖消耗曲线;c-谷氨酸棒杆菌重组菌合成Type 3 CPS的水平
图2 重组谷氨酸棒杆菌发酵合成Type 3 CPS
Fig.2 Bioproduction of Type 3 CPS by recombinant C. glutamicum
注:误差取3个独立实验的“平均值±标准差”表示,采用双因素方差分析进行统计学显著性检验,ns>0.01;*P<0.01;**P<0.005;***P<0.001;****P<0.000 1(下同)。
2.2 构建合成Type 3 CPS谷氨酸棒杆菌细胞工厂
本研究根据谷氨酸棒杆菌高效合成HA[12,15]的经验构建合成Type 3 CPS的谷氨酸棒杆菌细胞工厂。Type 3 CPS的合成仅需通过一条代谢途径供给UDP-葡萄糖和UDP-葡萄糖醛酸(图1-d),推测UDP-葡萄糖与UDP-葡萄糖醛酸的供应量平衡对Type 3 CPS的合成至关重要。当galU与ugd基因同时过表达时,可能导致代谢途径偏向UDP-葡萄糖醛酸的合成,进而减少UDP-葡萄糖的供应量。为了研究代谢基因对Type 3 CPS合成的影响,首先在谷氨酸棒杆菌中分别表达了cps3B与galU基因及组合共表达cps3B、galU与ugd基因。显微镜观察发现,发酵过程中谷氨酸棒杆菌菌体形态未发生显著变化(图2-a)。通过硫酸咔唑法测定葡萄糖醛酸的含量,分析重组菌胞外Type 3 CPS的合成情况。结果表明,随着发酵的进行,发酵上清液中Type 3 CPS的积累量逐渐增加(图2-c)。在48 h C.g/Cps3B的Type 3 CPS合成最高水平为15 mg/L,进一步过表达galU基因后C.g/Cps3B-GalU提高至58 mg/L;当基因galU和ugd同时过表达时,C.g/Cps3B-GalU-Ugd达到了最高值69 mg/L。上述结果表明,在谷氨酸棒杆菌中表达途径基因能够促进Type 3 CPS的合成,且当galU和ugd基因同时过表达时,促进作用更加显著。
2.3 构建合成Type 3 CPS大肠杆菌细胞工厂
由于谷氨酸棒杆菌合成Type 3 CPS与前期研究报道合成HA的能力相差甚远[12],因此下一步尝试替换大肠杆菌为底盘细胞合成Type 3 CPS。由于大肠杆菌有内膜和外膜之分,而Type 3 CPS和HA合酶仅能将糖链运出内膜,因此如果以大肠杆菌为底盘合成Type 3 CPS,可以预见的是其糖链应该被分泌到周质空间,不能直接进入胞外环境(图3-a)。根据前期研究经验,胞内积累的多糖会形成聚集区域,造成折光率的差异,进而可以通过相差显微镜观察到[21]。

a-谷氨酸棒杆菌与大肠杆菌的膜结构示意图;b-大肠杆菌重组菌发酵过程中的菌体形态变化;c-大肠杆菌重组菌发酵过程中的OD600与葡萄糖消耗曲线;d-大肠杆菌重组菌合成Type 3 CPS的水平
图3 重组大肠杆菌发酵合成Type 3 CPS
Fig.3 Bioproduction of Type 3 CPS by recombinant E. coli
在大肠杆菌BL21(DE3)中,为了探究代谢基因对Type 3 CPS合成的影响,本研究分别表达了galU基因,并同时表达了galU和ugd基因。结果表明Ec/Cps3B的菌体形态未发生显著变化,共表达galU基因后,Ec/Cps3B-GalU菌体在6 h内相较于野生型(Ec/CK)显著变长,12 h时菌体形态变化更为明显,胞内开始出现大量多糖聚集区,表明Type 3 CPS在细胞内发生积累(图3-b)。18 h后,Ec/Cps3B-GalU胞内多糖聚集区逐渐消退,菌体形态持续变化,24 h时菌体形态恢复正常。与此同时,当galU和ugd基因同时过表达时,Ec/Cps3B-GalU-Ugd菌体在整个发酵过程中未出现显著的形态变化,12 h时Type 3 CPS在胞内积累,18 h多糖聚集区消失,24 h菌体形态恢复正常。
在整个发酵过程中,3株重组菌的OD600值和糖耗在发酵初期与Ec/CK相比没有显著差异。然而,在后期重组菌的OD600值开始下降(图3-c)。6 h时3株重组菌均合成了Type 3 CPS,其中Ec/Cps3B-GalU合成水平为39 mg/L,显著高于其他2株。12 h Ec/Cps3B-GalU达到最高79 mg/L,而Ec/Cps3B和Ec/Cps3B-GalU-Ugd合成水平分别为52 mg/L和64 mg/L。18 h Ec/Cps3B-GalU合成水平为56 mg/L,高于其他2株菌。与显微镜观察到的现象一致,12 h时重组菌的形态变化和胞内积累比18 h更为明显,12 h的Type 3 CPS合成水平也明显高于18 h(图3-d)。
2.4 CpsC结构与功能的预测
尽管在大肠杆菌中Type 3 CPS的产量高于谷氨酸棒杆菌,但合成量仍然较低,推测Type 3 CPS的合成可能比HA的合成更加复杂,尤其是其合酶Cps3B与HA合酶HasA相比存在1个更短的跨膜螺旋(图1-c),这可能造成Type 3 CPS的分泌比HA的分泌过程更加困难。通过对Cps3B基因簇的分析,发现合酶Cps3B可能存在一种辅助亚基CpsC(图1-a)。有研究认为CpsC的拓扑结构在调控酪氨酸激酶CpsD的活性中可能发挥关键作用[22],也有研究认为CpsC参与调控荚膜多糖的生物合成,还可能协助多糖将其输出到细胞表面[23]。在肺炎链球菌和金黄色葡萄球菌中,酪氨酸激酶通常由2个独立的蛋白质组成,肺炎链球菌中的酪氨酸激酶为CpsC与CpsD,金黄色葡萄球菌中的酪氨酸激酶则由CpaA与CpaB组成[24],在大肠杆菌中,酪氨酸激酶为单一蛋白质Wzc[25]。基于上述对CpsC功能的报道,推测CpsC 会影响Type 3 CPS的合成。
首先,比对了CpsC与CpaA的蛋白结构,发现它们的结构高度相似(图4-a)。随后将CpsC、CpsD与大肠杆菌可拉酸胞外多糖合成相关酪氨酸激酶Wzc结构模型进行比对,发现Wzc结构与CpsC、CpsD组合结构总体相似;Wzc相比于CpsC在胞外多了4个螺旋结构(图4-b)。由于Cps3B的跨膜螺旋较短,CpsC是穿膜蛋白,有2个跨膜螺旋,所以推测CpsC可能作为Cps3B的跨膜螺旋促进Type 3 CPS的合成。
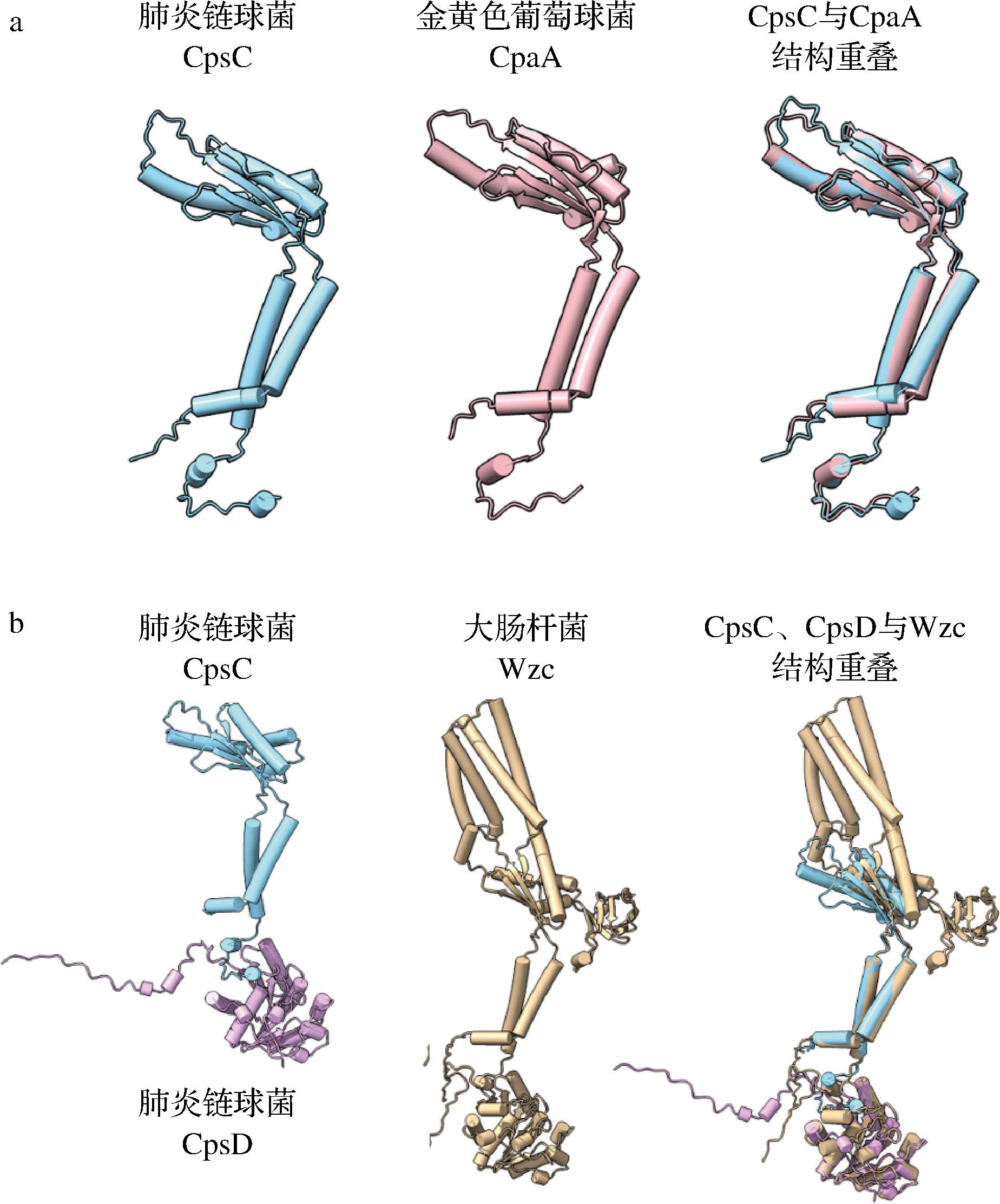
a-肺炎链球菌CpsC与金黄色葡萄球菌CpaA蛋白结构的比较;b-肺炎链球菌CpsC、CpsD与大肠杆菌Wzc蛋白结构的比较
图4 不同物种CpsC同工酶的结构比较
Fig.4 Structural comparison of CpsC isoenzymes from different species
2.5 CpsC对Type 3 CPS合成的影响及CpsC与Cps3B的互作分析
为了验证CpsC对Type 3 CPS合成是否有促进作用,在Ec/Cps3B-GalU与Ec/Cps3B-GalU-Ugd的基础上共表达了CpsC蛋白,并分析了其对大肠杆菌菌体形态及Type 3 CPS合成水平的影响。发酵6 h Ec/Cps3B-CpsC-GalU与Ec/Cps3B-CpsC-GalU-Ugd的形态发生了显著变化,12 h菌体内开始出现大量多糖聚集区,这在Cps3B-CpsC-GalU菌株中尤为明显(图5-a),这一结果表明CpsC对Type 3 CPS合成有促进作用。

a-Ec/Cps3B-CpsC-GalU、Ec/Cps3B-CpsC-GalU-Ugd发酵过程中的菌体形态变化;b-Ec/Cps3B-CpsC-GalU、Ec/Cps3B-CpsC-GalU-Ugd发酵过程中的OD600与葡萄糖消耗曲线;c-Ec/Cps3B-CpsC-GalU、Ec/Cps3B-CpsC-GalU-Ugd合成Type 3 CPS的水平;d-CpsC与Cps3B的位置结构分析;e-CpsC与Cps3B的氢键作用分析
图5 CpsC对Type 3 CPS合成的影响及CpsC与Cps3B的互作分析
Fig.5 Effect of CpsC on Type 3 CPS synthesis and interaction analysis between CpsC and Cps3B
表达CpsC蛋白后,OD600和葡萄糖消耗量未表现出明显差异(图5-b)。Ec/Cps3B-CpsC-GalU菌在整个发酵过程中合成水平维持在60 mg/L左右。Ec/Cps3B-CpsC-GalU-Ugd在6 h和18 h时的产量低于Cps3B-CpsC-GalU,但在12 h,Cps3B-CpsC-GalU-Ugd的合成水平达到105 mg/L(图5-c),产量结果进一步表明表达CpsC对Type 3 CPS合成有促进作用。
为探究辅助亚基CpsC影响Type 3 CPS合成的机制,通过AlphaFold 3模拟了CpsC与Cps3B复合体结构并通过分子动力学计算两者之间的相互作用。结果表明,CpsC具有2个跨膜结构域,位于Cps3B跨膜区域的上方,其N端和C端均位于细胞质内(图5-d)。Cps3B的第1个螺旋位于细胞膜上方,由于其长度较短,可能导致稳定性不足。通过氢键作用分析发现CpsC中的跨膜螺旋位点V116、P117和V118氨基酸残基与Cps3B中的F12、Q13和N14氨基酸残基通过动态氢键作用相互结合,能够稳定Cps3B的TM1跨膜区域,进而调控Type 3 CPS的跨膜转运过程(图5-e)。
3 结论与讨论
本研究首次开展了Type 3 CPS多糖细胞工厂构建,Type 3 CPS在谷氨酸棒杆菌中合成水平已达到69 mg/L,在大肠杆菌中的合成水平达到105 mg/L。并且发现膜蛋白CpsC在促进Type 3 CPS合成中起关键作用,分子动力学模拟表明其与Cps3B之间存在的氢键相互作用可能调控糖链的合成和跨膜转运过程。为了更全面地理解CpsC的作用机制,未来需要进一步研究CpsC如何影响Type 3 CPS的生产。对CpsC的结构和功能的深入研究将对揭示其在生物合成通路中的精确作用至关重要,并且评估通过工程化CpsC或类似蛋白质来进一步提高产量的可能性。
尽管本研究初步实现了在谷氨酸棒杆菌和大肠杆菌表达平台上成功生产Type3 CPS,但合成水平有限。未来的研究可以集中于提升代谢通路的效率,改善发酵条件,优化酶的表达水平,以进一步提高Type 3 CPS的产量。此外,探索表达系统和更多的代谢工程策略也可能帮助提高Type 3 CPS生物合成的效率。最后,虽然目前的研究主要集中在大肠杆菌平台,但将其他微生物如酿酒酵母、枯草芽孢杆菌等作为表达平台进行比较研究,可能会为Type 3 CPS的合成提供新的思路和解决方案。
[1] HARE K M, LEACH A J, SMITH-VAUGHAN H C, et al.Streptococcus pneumoniae and chronic endobronchial infections in childhood[J].Pediatric Pulmonology, 2017, 52(12):1532-1545.
[2] SMITH C M, FRY S C, GOUGH K C, et al.Recombinant plants provide a new approach to the production of bacterial polysaccharide for vaccines[J].PLoS One, 2014, 9(2):e88144.
[3] 田雨, 马可, 苏晓叶.多价肺炎链球菌荚膜多糖-蛋白结合疫苗的研究进展[J].中国生物制品学杂志, 2022, 35(10):1268-1273.TIAN Y, MA K, SU X Y.Progress in research on multivalent Streptococcus pneumoniae capsular polysaccharide-protein conjugate vaccine[J].Chinese Journal of Biologicals, 2022, 35(10):1268-1273.
[4] DILLARD J P, VANDERSEA M W, YOTHER J.Characterization of the cassette containing genes for type 3 capsular polysaccharide biosynthesis in Streptococcus pneumoniae[J].Journal of Experimental Medicine, 1995, 181(3):973-983.
[5] 赵文旭, 杨帆, 黄蕾, 等.肺炎链球菌荚膜多糖结构与合成的研究进展[J].微生物学报, 2024, 64(9):3168-3199.ZHAO W X, YANG F, HUANG L, et al.Advances in the chemical structures and biosynthesis of capsular polysaccharides of Streptococcus pneumoniae[J].Acta Microbiologica Sinica, 2024, 64(9):3168-3199.
[6] DENAPAITE D, HAKENBECK R.A new variant of the capsule 3 cluster occurs in Streptococcus pneumoniae from deceased wild chimpanzees[J].PLoS One, 2011, 6(9):e25119.
[7] LUCK J N, TETTELIN H, ORIHUELA C J.Sugar-coated killer:Serotype 3 pneumococcal disease[J].Frontiers in Cellular and Infection Microbiology, 2020, 10:613287.
[8] THOMAS FORSEE W, CARTEE R T, YOTHER J.Role of the carbohydrate binding site of the Streptococcus pneumoniae capsular polysaccharide type 3 synthase in the transition from oligosaccharide to polysaccharide synthesis[J].Journal of Biological Chemistry, 2006, 281(10):6283-6289.
[9] 肖森, 胡立涛, 石智诚, 等.可控分子量透明质酸的生物合成研究进展[J].合成生物学, 2025, 1:1-16.XIAO S, HU L T, SHI Z C, et al.Research progress on biosynthesis of hyaluronic acid with controllable molecular weight[J].Synthetic Biology, 2025, 1:1-16.
[10] IACONISI G N, LUNETTI P, GALLO N, et al.Hyaluronic acid:A powerful biomolecule with wide-ranging applications-a comprehensive review[J].International Journal of Molecular Sciences, 2023, 24(12):10296.
[11] BURDICK J A, PRESTWICH G D.Hyaluronic acid hydrogels for biomedical applications[J].Advanced Materials, 2011, 23(12):H41-H56.
[12] CHENG F Y, YU H M, STEPHANOPOULOS G.Engineering Corynebacterium glutamicum for high-titer biosynthesis of hyaluronic acid[J].Metabolic Engineering, 2019, 55:276-289.
[13] PARK S A, BHATIA S K, PARK H A, et al.Bacillus subtilis as a robust host for biochemical production utilizing biomass[J].Critical Reviews in Biotechnology, 2021, 41(6):827-848.
[14] SZE J H, BROWNLIE J C, LOVE C A.Biotechnological production of hyaluronic acid:A mini review[J].3 Biotech, 2016, 6(1):67.
[15] WANG Y, HU L T, HUANG H, et al.Eliminating the capsule-like layer to promote glucose uptake for hyaluronan production by engineered Corynebacterium glutamicum[J].Nature Communications, 2020, 11(1):3120.
[16] HU L T, WANG Y, HU Y X, et al.Biosynthesis of non-sulfated high-molecular-weight glycosaminoglycans and specific-sized oligosaccharides[J].Carbohydrate Polymers, 2022, 295:119829.
[17] WANG F, LEE S Y.High cell density culture of metabolically engineered Escherichia coli for the production of poly(3-hydroxybutyrate) in a defined medium[J].Biotechnology and Bioengineering, 1998, 58(2-3):325-328.
[18] BITTER T, MUIR H M.A modified uronic acid carbazole reaction[J].Analytical Biochemistry, 1962, 4(4):330-334.
[19] ABRAMSON J, ADLER J, DUNGER J, et al.Accurate structure prediction of biomolecular interactions with AlphaFold 3[J].Nature, 2024, 630(8016):493-500.
[20] SCHINDELIN J, ARGANDA-CARRERAS I, FRISE E, et al.Fiji:An open-source platform for biological-image analysis[J].Nature Methods, 2012, 9(7):676-682.
[21] HU L T, WANG Y, WANG L L, et al.Construction of osmotic pressure responsive vacuole-like bacterial organelles with capsular polysaccharides as building blocks[J].ACS Synthetic Biology, 2023, 12(3):750-760.
[22] WHITTALL J J, MORONA R, STANDISH A J.Topology of Streptococcus pneumoniae CpsC, a polysaccharide copolymerase and bacterial protein tyrosine kinase adaptor protein[J].Journal of Bacteriology, 2015, 197(1):120-127.
[23] MORONA J K, PATON J C, MILLER D C, et al.Tyrosine phosphorylation of CpsD negatively regulates capsular polysaccharide biosynthesis in Streptococcus pneumoniae[J].Molecular Microbiology, 2000, 35(6):1431-1442.
[24] RAUSCH M, DEISINGER J P, ULM H, et al.Coordination of capsule assembly and cell wall biosynthesis in Staphylococcus aureus[J].Nature Communications, 2019, 10(1):1404.
[25] GRANGEASSE C, OBADIA B, MIJAKOVIC I, et al.Autophosphorylation of the Escherichia coli protein kinase Wzc regulates tyrosine phosphorylation of Ugd, a UDP-glucose dehydrogenase[J].Journal of Biological Chemistry, 2003, 278(41):39323-39329.