植物乳植杆菌是一种可以发酵糖类,产乳酸,厌氧或兼性厌氧的革兰氏阳性菌,为杆状的同型发酵乳酸菌。植物乳植杆菌广泛存在于植物以及人类和动物胃肠道等多种生态环境中。大量研究证明,植物乳植杆菌有较强的肠道黏附能力[1],能够耐酸[2-3]、耐胆盐[4]、耐渗透压[5]、耐饥饿胁迫[6-7]、耐温度变化[8-9]以及耐氧化[10-11],是人类重要的肠道正常菌群。
近年来,全基因组测序技术高速发展。借助该技术,人们得以获取菌株的全部遗传信息,从而对编码基因进行功能分类便于挖掘重要功能基因[12]。目前对植物乳植杆菌潜在益生特性的研究受到现有生理生化方法的限制,因此通过全基因组测序评估益生菌的益生特性、安全性、代谢能力等,是一条筛选优质益生菌菌株的有效途径[13]。
本课题组前期从豆豉中筛选出一株植物乳植杆菌HQ-3(专利号:ZL2019 1 1136050.1),为了进一步挖掘该菌株的潜在益生特性,通过对植物乳植杆菌HQ-3进行全基因组测序,并分析基因组信息,从而预测发现其潜在的益生功能,为该菌株的进一步研究和应用提供理论参考。
1 材料与方法
1.1 材料
1.1.1 菌株
植物乳植杆菌(Lactiplantibacillus plantarum)HQ-3分离自豆豉,由四川农业大学食品学院畜产与发酵食品加工及安全团队实验室分离保存。
1.1.2 培养基
MRS肉汤培养基(g/L):蛋白胨10,牛肉膏10,酵母粉5,K2HPO4 2,柠檬酸二铵2,乙酸钠5,葡萄糖20,吐温-80 1 mL,MgSO4 0.58,MnSO4 0.25,蒸馏水1 L,121 ℃灭菌15 min。
MRS固体培养基:在MRS肉汤培养基的基础上加入1.5%的琼脂。
1.1.3 仪器与设备
ZWY-2102C恒温振荡培养箱,上海智城分析仪器制造有限公司;1300 Series A2生物安全柜、Sorvall ST 16R冷冻离心机,美国Thermo Fisher Scientific公司;LDZF-75L-I立式制动压力蒸汽灭菌器,上海申安医疗器械厂;DHG-9245A电热恒温鼓风干燥箱,上海一恒科技有限公司;C1000 Thermal Cycler PCR仪、Gel Doc XR+凝胶成像系统,美国Bio-Rad公司。
1.2 方法
1.2.1 菌株活化及培养
将保存于-80 ℃的植物乳植杆菌HQ-3划线接种于MRS固体培养基中,37 ℃培养24 h,挑取单菌落接种于MRS液体培养基中,37 ℃培养24 h,转接3代,备用。
1.2.2 基因组DNA提取及测序
对HQ-3进行基因组DNA提取,挑选合格的样品构建文库:DNA样品首先经过随机打断,产生不同长度的DNA片段;对产生的黏性末端进行末端修复转化为平末端,使其具有一致的结构;在DNA片段的3′末端加入腺嘌呤碱基,使其与带有胸苷碱基的3′端特殊接头稳定连接;使用PCR扩增DNA片段,以产生足够数量的DNA片段,整个文库的构建工作完成。构建好的合格文库进行cluster制备,并利用二代Illumina和三代Nanopore技术相结合的方式进行测序。
1.2.3 基因组装
对二代测序所获取的原始数据进行数据过滤去除低质量数据。对过滤后保留的数据进行测序质量值、长度、GC等统计。使用Mecat2对三代测序原始数据进行处理,得到有效数据。选择SMRT Link v5.1.0和Unicycle对样品质控之后的有效数据进行基因组组装。利用arrow软件进行组装结果的优化和校正,得到最终数据。
1.2.4 基因组组分分析
使用Glimmer3/Prodigal(GC含量>70%)进行编码基因预测。非编码RNA中tRNA的预测使用tRNAscan-SE(版本1.3.1),rRNA的预测使用rRNAmmer(版本1.2),sRNA 的预测首先比对Rfam database,然后使用cmsearch程序筛选比对结果。散在重复序列和串联重复序列的预测分别使用RepeatMasker(版本open-4.0.5)和 TRF(tandem repeats finder,版本4.07b)进行分析。利用CRISPR digger对样品基因组进行CRISPR预测。
1.2.5 基因组功能注释
对获得的氨基酸序列,利用直系同源基因数据库(Cluster of Orthologous Groups of proteins,COG)、KEGG、GO、碳水化合物相关酶数据库(Carbohydrate-Active EnZYmes Database, CAZy)、综合抗生素研究数据库(Comprehensive Antibiotic Resistance Database,CARD)进行比对分析,获得基因功能注释结果。
2 结果与分析
2.1 植物乳植杆菌HQ-3全基因组信息分析
2.1.1 HQ-3基因组组装结果
对HQ-3全基因组测序数据进行筛选评估以及组装,得到基因组组装结果如表1所示,HQ-3全基因组包含1个环状染色体,其长度为3 206 835 bp,GC含量为44.64%;含有5个完整质粒,质粒序列长度分别为72 235、64 612、46 953、17 279、14 706 bp,GC含量分别为44.64%、41.53%、39.18%、40.84%、38.78%。
表1 植物乳植杆菌HQ-3基因组组装结果统计
Table 1 Statistics of assembly results in L. plantarumHQ-3 genome

特征L.plantarum HQ-3重叠群总数目6重叠群总长度/bp3 422 620重叠群平均长度/bp570 436.67最长的重叠群长度/bp3 206 835最短的重叠群长度/bp14 706N50 长度/bp3 206 835N90 长度/bp3 206 835GC/%44.41
2.1.2 HQ-3基因组基本特征
基因组不仅包含编码区域,还有非编码区域实现转录调控、转录后调控、翻译调控、翻译调控、表观遗传等功能,通过多种方法,对编码基因、重复序列、非编码 RNA 等组分进行分析,获得样本中的基因组组成情况,结果如表2所示。HQ-3基因组共有3 283个编码基因,总序列长度为2 870 940 bp,占基因组全长的83.88%。sRNA的数量为9个;16S rRNA、5S rRNA、23S rRNA的数量分别为5、6、5个;tRNA数量为74个。HQ-3基因组中存在3个反转录转座子序列和1个DNA转座子序列,78个串联重复序列,3个前噬菌体序列。
表2 植物乳植杆菌HQ-3基因组基本特征
Table 2 General features of L. plantarum HQ-3 genomes
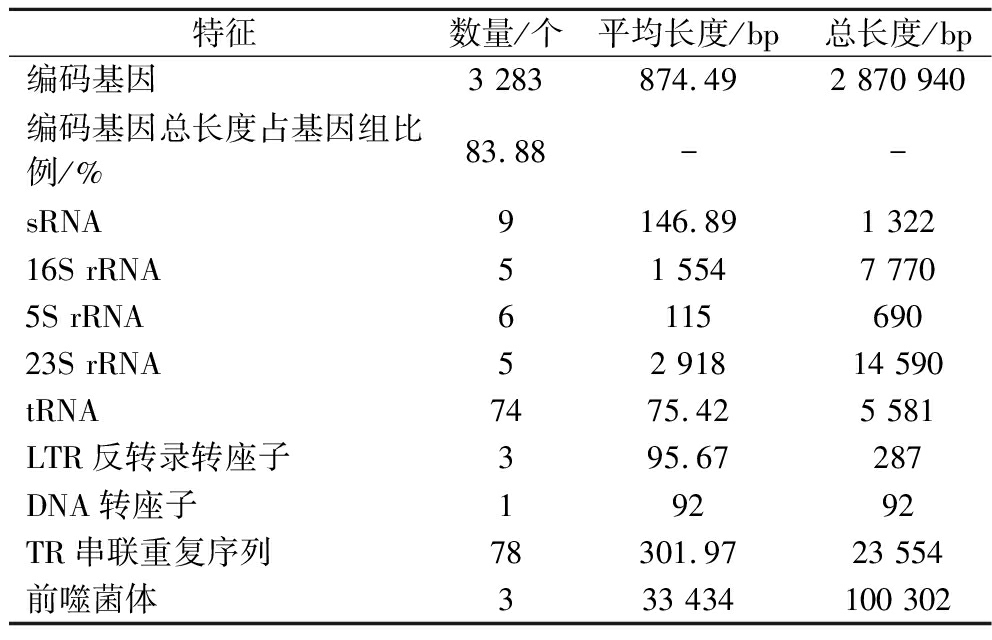
特征数量/个平均长度/bp总长度/bp编码基因3 283874.492 870 940编码基因总长度占基因组比例/%83.88--sRNA9146.891 32216S rRNA51 5547 7705S rRNA611569023S rRNA52 91814 590tRNA7475.425 581LTR反转录转座子395.67287DNA转座子19292TR串联重复序列78301.9723 554前噬菌体333 434100 302
基于组装结果及编码基因的预测结果结合非编码 RNA、基因功能注释分析结果,使用Circos软件进行可视化展示,得到全基因组图谱如图1所示。HQ-3基因组圈图自外向内分别是基因组的刻度标记、正向链上的蛋白质编码基因、反向链上的蛋白质编码基因、正向链上的tRNA和rRNA基因、反向链上的tRNA和rRNA基因、GC内容、GC偏斜。
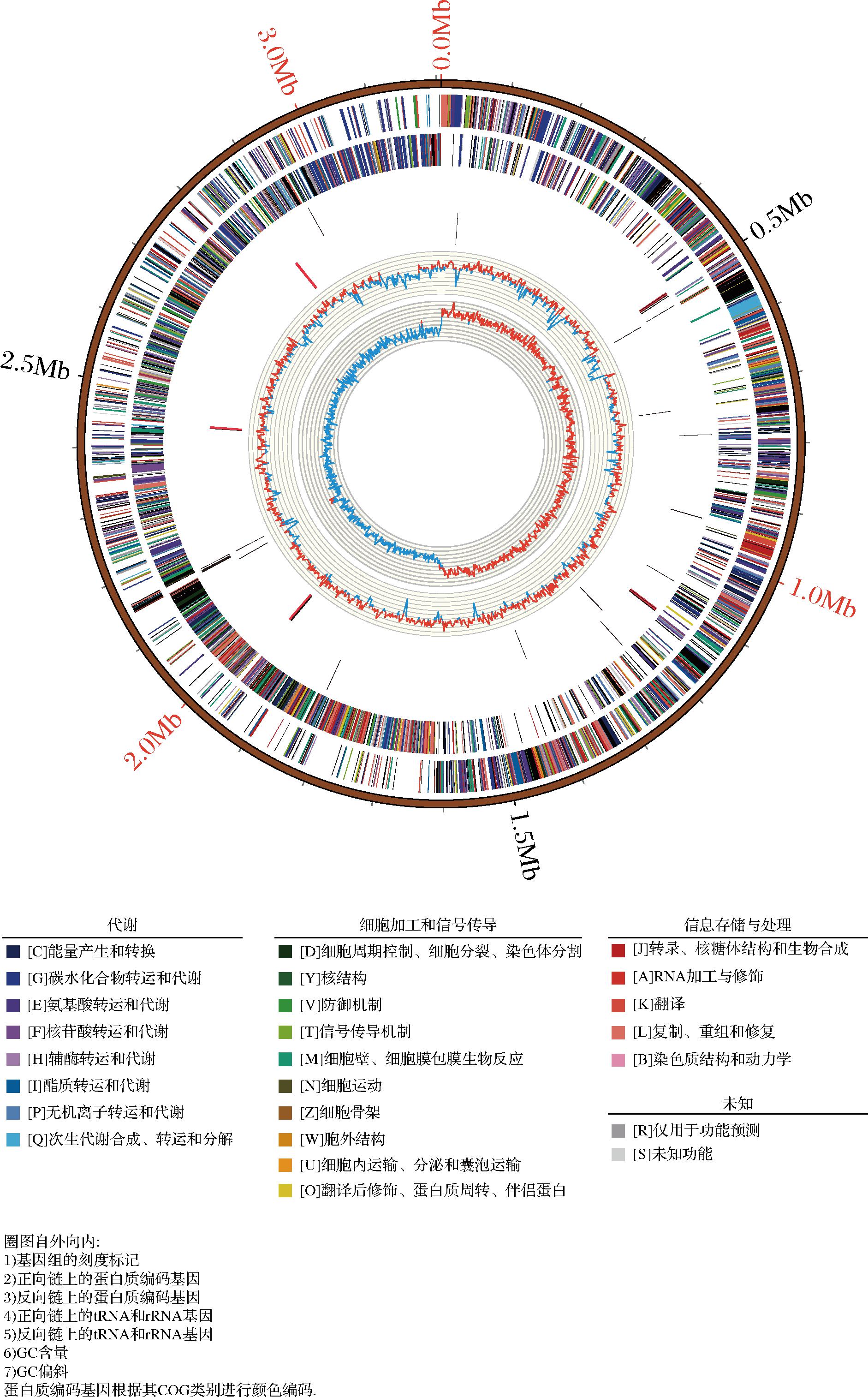
图1 植物乳植杆菌HQ-3基因组图谱
Fig.1 Genome mapping of L. plantarum HQ-3
2.2 HQ-3基因组功能注释与分析
2.2.1 HQ-3基因组GO数据库注释与分析
GO数据库中基因分为三大类[14]:生物过程(biological process);细胞组分(cellular component);分子功能(molecular function)。植物乳植杆菌HQ-3共有3 968个基因得到GO数据库的功能注释,注释结果如图2所示。
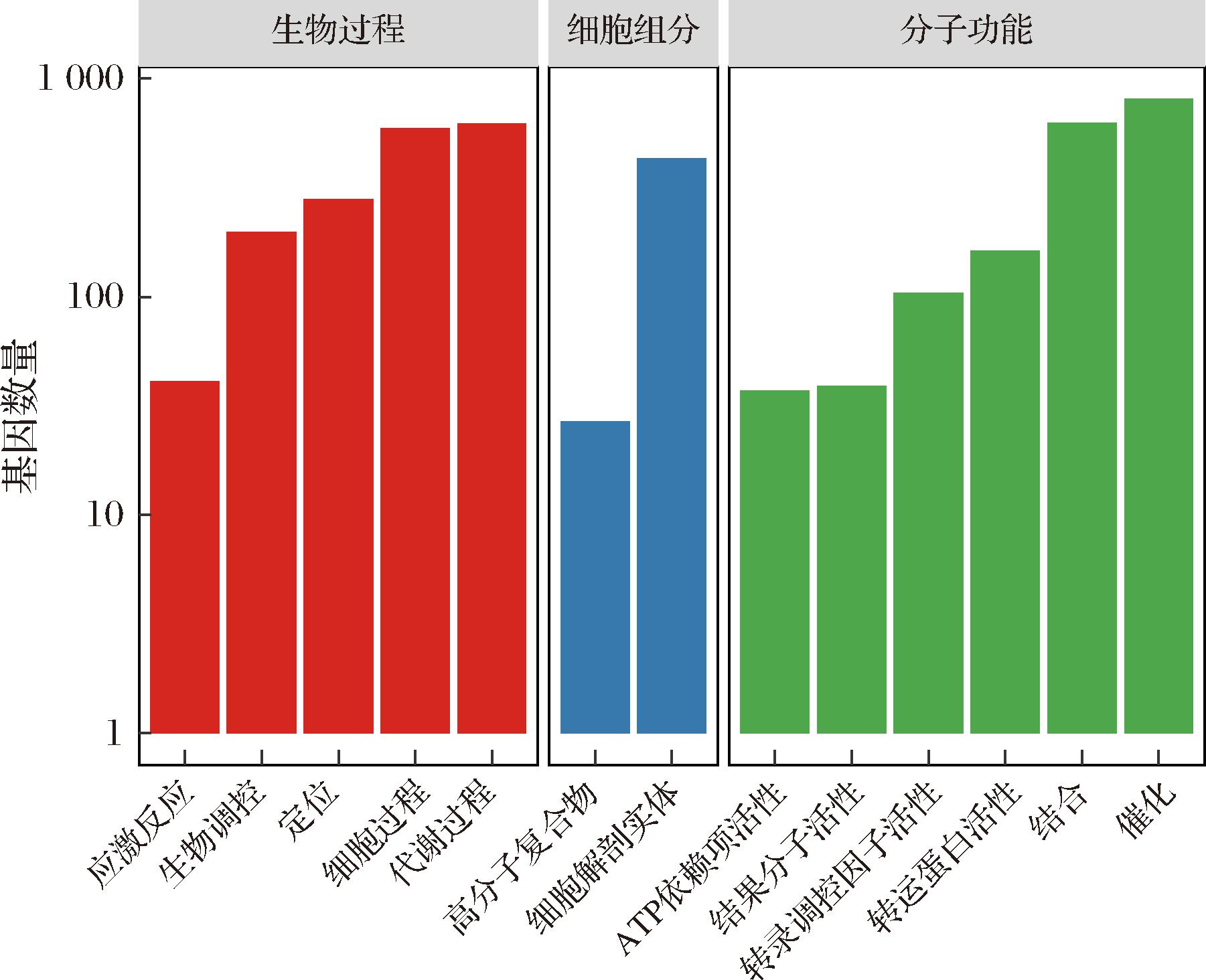
图2 植物乳植杆菌HQ-3 GO数据库注释结果
Fig.2 Result of L. plantarum HQ-3 GO annotations
HQ-3基因组中共有1 731个基因注释与生物过程相关。其中注释量最高的为代谢过程,如碳水化合物代谢、甘油代谢、半乳糖代谢等,共有621个相关基因;其次为细胞过程,基因数量共有592个;与定位相关的基因有279个;与生物调控相关的基因有198个;与应激反应相关的基因有41个。
HQ-3基因组中关于细胞组分的基因注释量为458个。其中以细胞解剖实体相关基因为主,共有431个;高分子复合物相关基因有27个。
HQ-3基因组中关于分子功能的基因注释量为1 676个。其中与催化相关的基因数量最多,为811个,这些基因可参与糖代谢、脂代谢并指导众多功能性蛋白的合成,是植物乳植杆菌HQ-3生长繁殖的基础;其次是与结合相关的基因,数量为626个,这类基因调控着生物反应过程中生物分子之间的附着;与转运蛋白活动相关的基因数量为162个。
2.2.2 HQ-3基因组KEGG数据库注释与分析
KEGG数据库整合了基因组、化学分子和生化系统等方面的数据,其中核心的代谢通路数据库将生物代谢通路分为7个大类:细胞过程(cellular processes)、药物开发(drug development)、环境信息处理(environmental information processing)、遗传信息处理(genetic information processing)、人类疾病(human diseases)、新陈代谢(metabolism)和有机体系统(organismal systems)[15]。
植物乳植杆菌HQ-3共有3 238个基因得到KEGG数据库注释,其中有1 578个基因分别在新陈代谢、有机体系统、环境信息处理、人类疾病、遗传信息处理和细胞过程六大功能共35个通路上得到功能注释,注释结果如图3所示。

图3 植物乳植杆菌HQ-3 KEGG注释结果
Fig.3 Result of L. plantarum HQ-3 KEGG annotations
分析表明HQ-3基因功能注释主要集中在新陈代谢、环境信息处理以及遗传信息处理3个方面。新陈代谢通路共有590个得到注释的基因,其中与碳水化合物代谢相关的基因数量最多,为229个,氨基酸代谢相关基因也有137个,说明HQ-3可能具有较强的糖类代谢和氨基酸代谢能力。环境信息处理中共有248个得到注释的基因,与膜传输相关的基因最多,为181个,主要包括与各类跨膜运输蛋白如ABC转运蛋白、PTS转运蛋白相关的基因,推测植物乳植杆菌HQ-3可能对外界环境的适应能力较强,能够一定程度上抵抗环境变化的压力。遗传信息处理中共有174个得到注释的基因,与翻译相关的基因最多,为82个。此外,植物乳植杆菌HQ-3在人类疾病方面的基因注释量极少,主要是抗药相关基因,推测该菌对人类几乎无潜在危害,安全性较高。
2.2.3 HQ-3基因组COG数据库注释与分析
COG数据库是由NCBI创立并维护的蛋白数据库,该数据库整合细菌、藻类和其他真核生物的基因组的蛋白质编码系统,并按照功能分类分为26个类别[16]。
植物乳植杆菌HQ-3共有2 888个基因在COG数据库中得到了功能注释。注释结果显示,植物乳植杆菌HQ-3在碳水化合物转运和代谢(G)方面的基因注释量最多,为305个,其次与转录(K)相关的基因注释量也达到了291个;另外,在一般功能预测(R)和氨基酸运输和代谢(E)以及翻译、核糖体结构与生物发生(J)方面也获得较多的基因注释,分别为240、226、221个,说明植物乳植杆菌HQ-3的碳水化合物及氨基酸的利用能力较强,这与KEGG数据库注释结果一致。此外,在细胞壁、生物膜和包膜生物发生(M)中获得了180个基因注释量,推测HQ-3可能具有较强的生物膜形成能力。而防御机制(V)的注释量为85个,表明HQ-3可能具有一定的抵抗外界环境的能力,可能与抵抗消化道环境,稳定定植于肠道上皮细胞有关。
WCFS1是目前研究得最深入的植物乳植杆菌之一,具有较强的胆盐耐受能力,可存活于人体肠道、提高人体免疫力,将HQ-3基因组 COG数据库注释结果与WCFS1基因组COG数据库注释结果进行对比,得到的结果如表3所示。
表3 HQ-3与WCFS1 COG分类比较
Table 3 Comparison of the COG classification of HQ-3 and WCFS1

代码COG注释WCFS1HQ-3ARNA加工和修饰00B染色质结构和动力学00C能源生产和转换114119D细胞周期控制、细胞分裂、染色体分割2649E氨基酸转运和代谢267226F核苷酸转运和代谢87115G碳水化合物转运和代谢313305H辅酶运输和代谢91154I脂质转运和代谢75106J翻译、核糖体结构与生物发生155221K转录305291L复制、重组与修复157131M细胞壁、生物膜和包膜生物发生156180N细胞运动性1118O翻译后修饰、蛋白质周转、伴侣62113P无机离子运输和代谢169138Q次生代谢物的生物合成、运输和分解代谢4222R仅通用功能预测421240S未知功能224131T信号转导机制100148U细胞内运输、分泌和膀胱运输2625V防御机制6085W细胞外结构04X动员组:噬菌体、转座子064Y核结构00Z细胞骨架03
比较发现,HQ-3在细胞周期控制、细胞分裂、染色体分割(D)、核苷酸转运和代谢(F)、辅酶运输和代谢(H)、脂质转运和代谢(I)、翻译、核糖体结构与生物发生(J)、细胞壁、生物膜和包膜生物发生(M)、翻译后修饰、蛋白质周转、伴侣(O)、信号转导机制(T)、防御机制(V)、细胞外结构(W)、动员组:噬菌体、转座子(X)、细胞骨架(Z)12个方面的基因注释量均高于WCFS1,说明HQ-3在相关方面的能力可能强于WCFS1。在能源生产和转换(C)、碳水化合物转运和代谢(G)、转录(K)、细胞运动性(N)、细胞内运输、分泌和膀胱运输(U)5个方面的基因注释量与WCFS1差别不大,说明二者可能在相关方面的能力相似。
2.2.4 CAZy数据库注释与分析
CAZy是专门用于研究碳水化合物活性酶(CAZymes)的专业级数据库,其收录的CAZymes主要分为4类:糖苷水解酶(glycoside hydrolases,GHs)、糖基转移酶(glycosyl transferases,GTs)、多糖裂解酶(polysaccharide lyases,PLs)和糖类酯解酶(carbohydrate esterases,CEs),此外,还包含与碳水化合物相关的模块(carbohydrate-binding modules,CBMs)[17]。植物乳植杆菌HQ-3全基因组信息经CAZy数据库分类注释统计结果如图4所示。
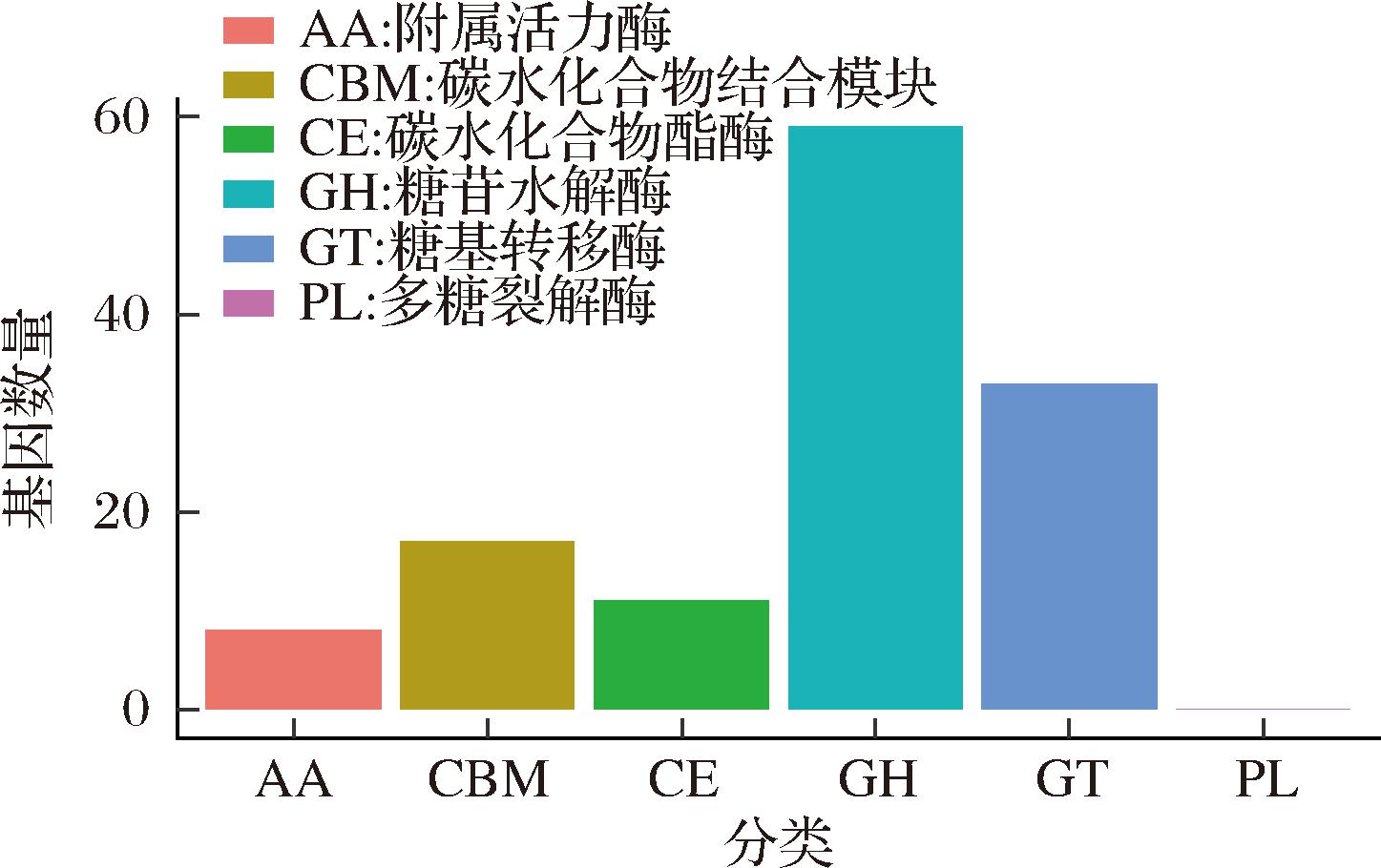
图4 植物乳植杆菌HQ-3 CAZy注释结果
Fig.4 Result of L. plantarum HQ-3 CAZy annotations
植物乳植杆菌HQ-3基因组信息在CAZy数据库中共注释到128个基因。其中GHs的注释量最多,为59个,占总注释46.1%。GHs作用于糖苷键,使多糖水解,从而释放大量能量以支撑细菌的各种生命代谢活动。课题组前期实验证据显示,HQ-3能分解利用乳糖、半乳糖、甘露醇[18]。益生元筛选实验结果表明HQ-3能利用多种常见益生元进行生长,结果如图5所示。前期实验结果和益生元筛选结果与GHs注释结果一致。
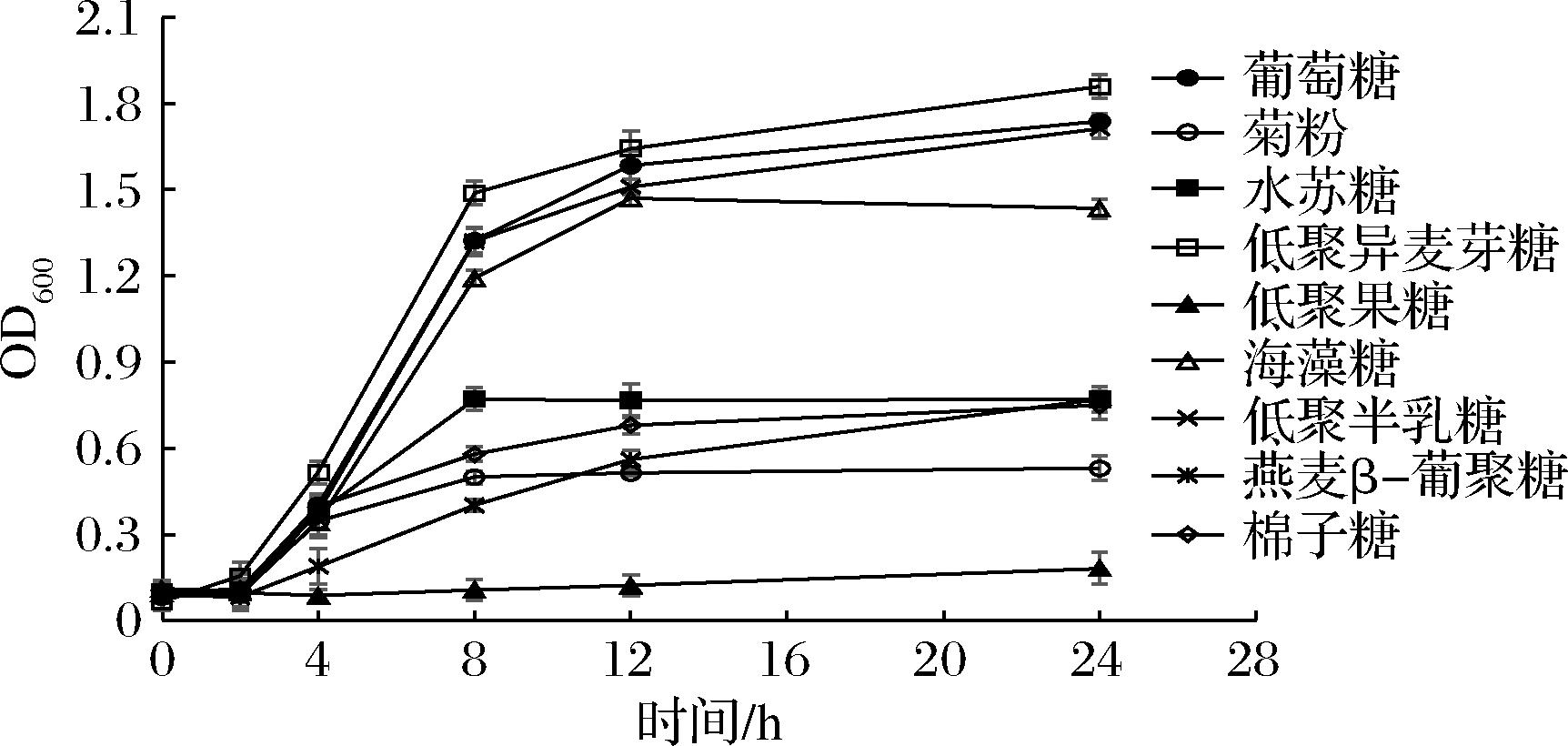
图5 植物乳植杆菌HQ-3利用不同益生元生长情况
Fig.5 Growth conditions of L. plantarum HQ-3 using different prebiotics
另外,在GTs方面也得到了较多的注释量,为33个,占比25.8%。GTs催化糖基与其他物质如蛋白质、脂质或聚糖的基团进行转移或交换,将糖基转移到特定的受体上,形成新的蛋白聚糖或糖蛋白等,从而参与各种生理过程。CBMs、CEs、辅助活性酶(auxiliary activity enzymes,AA)得到的基因注释量分别为17、11、8个,占比分别为13.3%、8.6%、6.3%。CEs能水解糖类酯,将其分解为糖和相应的酸或醇。AA是一类辅助于其他酶或催化反应的辅酶,对于维持生物体内代谢平衡和生理功能具有重要意义。
2.2.5 CARD数据库注释与分析
CARD数据库(The Comprehensive Antibiotic Resistance Database)是一个用于研究抗生素抗性基因和抗性突变的综合性数据库。CARD数据库注释结果显示,植物乳植杆菌HQ-3基因组中仅有2个抗性基因,分别为抗利福霉类抗生素基因和抗β-内酰胺类抗生素基因。课题组前期实验证明HQ-3只对青霉素耐受,对阿莫西林及头孢噻吩中度敏感,对其余利福霉素等11种常见抗生素均敏感[18],与基因组信息CARD数据库分析结果一致。因此HQ-3具有良好的药敏性。
2.3 HQ-3基因组胃肠道存活能力相关基因分析
益生菌要在人体内发挥作用必须具备在胃肠道中存活的能力。研究表明乳酸菌产胆盐水解酶(bile salt hydrolase,BSH)能力与菌株肠道耐受能力相关[19];Atg22家族的MFS型转运蛋白与胆汁耐受基因合成相关[20];黏附蛋白的表达能增强菌株肠道黏附能力,有助于菌株在肠道内的定植[21]。
在对HQ-3的全基因组信息进行分析比对后发现,HQ-3存在4个编码BSH的基因,1个编码MFS型转运蛋白的基因,还含有9个与酸耐受性相关的基因。这与课题组前期对HQ-3进行的胃肠液耐受实验结果一致。实验结果显示,HQ-3在人工模拟胃液中培养存活率为98.75%,再经过人工肠液处理后存活率为72.92%,表明HQ-3对模拟胃肠液具有良好的耐受性[18]。此外,根据基因组信息分析显示,在HQ-3基因组中含有1个黏附蛋白基因slp2588,与课题组前期黏附蛋白基因扩增结果一致[18]。
2.4 HQ-3其他潜在益生特性相关基因分析
细菌素是一类由细菌核糖体合成的具有较强抑菌活性的多肽物质。植物乳植杆菌所产细菌素主要由pln基因簇调控,以II类细菌素为主[22]。植物乳植杆菌素B、植物乳植杆菌素C、植物乳植杆菌素D与3组分调节系统相关,参与生物膜成熟过程,植物乳植杆菌素G、植物乳植杆菌素H与ABC转运系统相关,参与细菌素的胞外分泌[23-24]。
利用全基因组信息分析对HQ-3基因组中潜在的细菌素基因进行挖掘。分析发现HQ-3基因组中含有6个细菌素编码相关基因,其中基因PlnB、PlnC、PlnD、PlnG各1个,PlnH 2个。课题组前期对HQ-3进行了抑菌实验,当把该菌株发酵上清液调节至pH=7.0时,其对大肠杆菌、金黄色葡萄球菌、沙门氏菌的抑菌活性完全消失[25],表明HQ-3在MRS培养基培养下抑菌物质可能为有机酸。抑菌实验结果显示,在此条件下,HQ-3基因组的细菌素基因并未表达。在后续实验中,需要进一步确认HQ-3基因组中编码细菌素相关基因的表达条件。
益生元的利用能力也是益生菌益生特性的重要方面。CAZy数据库注释结果显示HQ-3基因组中GHs的注释量最多。但是乳酸菌对益生元的分解和利用能力还与有关的转运系统相关。细菌细胞对糖类物质的转运主要依靠磷酸烯醇式丙酮酸-糖磷酸转移酶系[26](phosphoenolpyruvate-phosphotransferase system,PEP-PTS)。对HQ-3全基因组信息中参与糖转运系统的相关基因进行挖掘,结果显示,HQ-3基因组中含有PTS纤维二糖转运体亚基EII、PTS β-葡萄糖苷转运体亚基EII、PTS半乳糖转运体亚基EII等3种糖特异性通透酶相关基因20个。益生菌筛选实验已经证实HQ-3具有分解利用低聚半乳糖和β-葡聚糖的能力[18],在后续研究中,需进一步确认HQ-3是否具有利用膳食纤维的能力。
此外HQ-3全基因组信息中还具有3个β-半乳糖苷酶基因以及2个PTS乳糖转运体亚基EII的基因。前期实验证明HQ-3能分解利用乳糖[18],与基因组信息分析一致,据此可以推测HQ-3可应用于食品工业,在降解乳糖,为乳糖不耐受人群提供相关食品产品方面具有一定发展潜力。
2.5 与生产性能相关的基因分析
益生菌活菌制剂的稳定性是支撑益生菌产业发展的重要基础,除了益生菌在宿主体内的耐受能力,还涉及到益生菌在生产环节中的耐受能力。
研究表明热休克蛋白(heat shock proteins, HSPs)是一类热应激蛋白,当益生菌暴露于高温环境时可被诱导表达,从而增加益生菌的抗热能力[27]。根据HQ-3基因组信息,在HQ-3基因组中存在8个编码热休克蛋白相关基因,其编码蛋白包括HtpX、CtsR、GrpE、DnaK、DnaJ、HSP33、HSP20,后续研究需进一步验证热休克蛋白基因表达与HQ-3在加工工序中的耐热能力。此外,冷休克蛋白(cold shock proteins, CSPs)是微生物细胞适应环境温度应激蛋白[28],低温刺激能提高植物乳植杆菌菌体cspL基因表达量[9]。基因组信息分析结果显示HQ-3基因组中含有3个CSPs编码基因,前期实验结果也表明HQ-3在10 ℃条件[18]能正常生长。后续研究需进一步确认CSPs编码基因表达与HQ-3低温适应能力之间的关系。
3 结论
本研究对豆豉来源的植物乳植杆菌HQ-3进行了全基因组测序和潜在益生功能挖掘。HQ-3含有1个环状染色体,5个完整质粒,3 283个编码基因。在GO、KEGG、COG、CAZy、CARD 5个数据库中得到基因注释量分别为3 968、3 238、2 888、128、2个。含有多个耐酸、耐胆盐、细胞黏附作用等胃肠道存活相关基因,以及细菌素、糖类物质转运、热休克蛋白、冷休克蛋白等益生功能相关基因。通过基因组信息分析推测HQ-3具有较强糖类代谢能力,外界环境抵抗能力和胃肠道存活能力,具有良好抗生素敏感性和抗菌能力。
综上所述,HQ-3是具有良好潜在益生特性的菌株,具有研究和应用价值。
[1] ZHANG L H, MA H C, KULYAR M F, et al.Complete genome analysis of Lactobacillus fermentum YLF016 and its probiotic characteristics[J].Microbial Pathogenesis, 2022, 162:105212.
[2] MILLER B J, FRANZ C M A P, CHO G S, et al.Expression of the malolactic enzyme gene (mle) from Lactobacillus plantarum under winemaking conditions[J].Current Microbiology, 2011, 62(6):1682-1688.
[3] DE ANGELIS M, GOBBETTI M.Environmental stress responses in Lactobacillus:A review[J].Proteomics, 2004, 4(1):106-122.
[4] 胡斌. 植物乳杆菌胆盐耐受评价及内在影响因素分析[D].无锡:江南大学, 2015.HU B.Evaluation of bile salt resistance and analysis of internal affecting factors in Lactobacillus plantarum[D].Wuxi:Jiangnan University, 2015.
[5] GLAASKER E, KONINGS W N, POOLMAN B.Osmotic regulation of intracellular solute pools in Lactobacillus plantarum[J].Journal of Bacteriology, 1996, 178(3):575-582.
[6] ERCAN O, WELS M, SMID E J, et al.Genome-wide transcriptional responses to carbon starvation in nongrowing Lactococcus lactis[J].Applied and Environmental Microbiology, 2015, 81(7):2554-2561.
[7] OLIVER J D.Recent findings on the viable but nonculturable state in pathogenic bacteria[J].FEMS Microbiology Reviews, 2010, 34(4):415-425.
[8] SCHOUG Å, FISCHER J, HEIPIEPER H J, et al.Impact of fermentation pH and temperature on freeze-drying survival and membrane lipid composition of Lactobacillus coryniformis Si3[J].Journal of Industrial Microbiology and Biotechnology, 2008, 35(3):175-181.
[9] 李宝坤. 乳酸杆菌冷冻干燥生理损伤机制及保护策略的研究[D].无锡:江南大学, 2011.LI B K.Study on the physiology mechanism of cell damage caused by freeze-drying and protection strategies of Lactobacillus[D].Wuxi:Jiangnan University, 2011.
[10] 杨扬. 植物乳杆菌CAUH2氧化胁迫反应机制及转录因子CopR在氧化胁迫应答中的作用[D].北京:中国农业大学, 2018.YUNG Y.The oxidative stress response mechanism and functional role of transcription factor CopR in oxidative stress in Lactobacillus plantarum CAUH2[D].Beijing:China Agricultural University, 2018.
Lactobacillus plantarum CAUH2[D].Beijing:China Agricultural University, 2018.
[11] MAYHEW M, DA SILVA A C R, MARTIN J, et al.Protein folding in the central cavity of the GroEL-GroES chaperonin complex[J].Nature, 1996, 379(6564):420-426.
[12] 刘超楠, 蔡熙姮, 张新宇, 等.植物乳杆菌基因组学研究进展[J].食品科技, 2020, 45(11):1-7.LIU C N, CAI X H, ZHANG X Y, et al.Progress in genomics of Lactobacillus plantarum[J].Food Science and Technology, 2020, 45(11):1-7.
[13] EVANOVICH E, DE SOUZA MENDONÇA MATTOS P J, GUERREIRO J F.Comparative genomic analysis of Lactobacillus plantarum:An overview[J].International Journal of Genomics, 2019, 2019(1):4973214.
[14] HARRIS M A, CLARK J, IRELAND A, et al.The Gene Ontology (GO) database and informatics resource[J].Nucleic Acids Research, 2004, 32(Database issue):D258-D261.
[15] KANEHISA M, GOTO S.KEGG Kyoto encyclopedia of genes and genomes[J].Nucleic Acids Research, 2000, 28(1):27-30.
[16] TATUSOV R L, FEDOROVA N D, JACKSON J D, et al.The COG database:An updated version includes eukaryotes[J].BMC Bioinformatics, 2003, 4:41.
[17] CANTAREL B L, COUTINHO P M, RANCUREL C, et al.The carbohydrate-active EnZymes database (CAZy):An expert resource for glycogenomics[J].Nucleic Acids Research, 2009, 37(suppl_1):D233-D238.
[18] 朱琦琦, 何维, 余东梅, 等.产纤溶酶的乳酸菌筛选及其潜在益生特性[J].基因组学与应用生物学, 2021, 40(S2):2626-2634.ZHU Q Q, HE W, YU D M, et al.Screening of lactic acid bacteria producing fibrinolytic enzyme and its potential probiotic characteristics[J].Genomics and Applied Biology, 2021, 40(S2):2626-2634.
[19] FOLEY M H, O’FLAHERTY S, ALLEN G, et al.Lactobacillus bile salt hydrolase substrate specificity governs bacterial fitness and host colonization[J].Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(6):e2017709118.
[20] 邓东, 颜宁.MFS超家族转运蛋白结构基础及转运机制[J].科学通报, 2015, 60(8):720-728.DENG D, YAN N.Structural basis and transport mechanism of the major facility superfamily(MFS) transporter[J].Chinese Science Bulletin, 2015, 60(8):720-728.
[21] YU Y X, ZONG M L, LAO L F, et al.Adhesion properties of cell surface proteins in Lactobacillus strains in the GIT environment[J].Food &Function, 2022, 13(6):3098-3109.
[22] S ENZ Y, ROJO-BEZARES B, NAVARRO L, et al.Genetic diversity of the pln locus among oenological Lactobacillus plantarum strains[J].International Journal of Food Microbiology, 2009, 134(3):176-183.
ENZ Y, ROJO-BEZARES B, NAVARRO L, et al.Genetic diversity of the pln locus among oenological Lactobacillus plantarum strains[J].International Journal of Food Microbiology, 2009, 134(3):176-183.
[23] DIEP D B, MYHRE R, JOHNSBORG O, et al.Inducible bacteriocin production in Lactobacillus is regulated by differential expression of the pln operons and by two antagonizing response regulators, the activity of which is enhanced upon phosphorylation[J].Molecular Microbiology, 2003, 47(2):483-494.
[24] NAVARRO L, ROJO-BEZARES B, S ENZ Y, et al.Comparative study of the pln locus of the quorum-sensing regulated bacteriocin-producing L.plantarum J51 strain[J].International Journal of Food Microbiology, 2008, 128(2):390-394.
ENZ Y, et al.Comparative study of the pln locus of the quorum-sensing regulated bacteriocin-producing L.plantarum J51 strain[J].International Journal of Food Microbiology, 2008, 128(2):390-394.
[25] 朱琦琦. 产纤溶酶植物乳杆菌的筛选及其纤溶酶酶学性质研究[D].雅安:四川农业大学, 2020.ZHU Q Q.Screening of Lactobacillus plantarum producing fibrinolytic enzyme and study on the characterizations of fibrinolytic enzyme[D].Ya’an:Sichuan Agricultural University, 2020.
[26] 李敏, 李伟程, 刘亚华, 等.乳酸乳球菌乳酸亚种IMAU11823全基因组测序及胞外多糖基因簇分析[J].中国食品学报, 2021, 21(4):256-266.LI M, LI W C, LIU Y H, et al.Complete genome sequence of Lactococcus lactis subsp.lactis IMAU11823 and analysis of the exopolysaccharide biosynthesis gene cluster[J].Journal of Chinese Institute of Food Science and Technology, 2021, 21(4):256-266.
[27] 李庆伟, 徐静, 李瑶.热休克蛋白进化及生物学功能研究进展[J].辽宁师范大学学报(自然科学版), 2024, 47(1):78-85.LI Q W, XU J, LI Y.Research progress on function and evolution of heat shock protein[J].Journal of Liaoning Normal University (Natural Science Edition), 2024, 47(1):78-85.
[28] KIM W S, KHUNAJAKR N, REN J, et al.Conservation of the major cold shock protein in lactic acid bacteria[J].Current Microbiology, 1998, 37(5):333-336.