酵母菌是一种单细胞真菌,广泛分布于自然界中。在现代生物技术中,酵母菌因其生长速度快、营养需求简单以及遗传操作便捷等优点而被广泛研究应用于食品、医药、工农业和环保等领域[1]。工程酵母细胞工厂已被利用来合成各种功能成分和天然产物,如生物燃料[2]、功能蛋白[3]、药品[4]和微生物肥料[5]等,当前主要通过代谢工程改造以及合成生物学构建底盘酵母细胞工厂来实现对这些产物的可持续生产。随着基因编辑技术的快速发展,对酵母的遗传改造技术和方法不断创新,促进了对酵母细胞生理生化代谢研究的不断加深,推动了酵母底盘细胞的不断改造和升级,从而拓宽了其利用领域。
基因编辑技术是指对目标基因进行的精准修饰,包括基因敲除、敲入、替换和有义的点突变[6],用以改变基因的结构,从而改变特定基因的功能,进而改变某些特性或表型。传统的基因编辑技术是利用DNA同源重组(homologous recombination,HR)介导的基因打靶技术对基因组进行编辑,但这种方法效率较低,严重限制了该技术的应用[7]。近年来,科研工作者已经开发出了一些高效的基因组编辑工具,例如锌指核酸酶系统(zinc finger nucleases,ZFNs)、转录激活因子样效应物核酸酶系统(transcription activator-like effector nucleases,TALENs)以及成簇规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)相关系统[6]。CRISPR是一种存在于细菌和古细菌中的天然免疫系统,能够识别和剪切外源DNA序列,以抵御病毒的入侵。目前,CRISPR/Cas9作为Ⅱ型CRISPR/Cas系统中最常见的系统,因其设计简单、操作方便、效率高等优点,迅速在生物医药、动植物和微生物等领域实现了广泛的应用,并已被应用于各种酵母菌的基因组编辑[8]。本文综述了CRISPR/Cas系统的发展历史、分类情况,CRISPR/Cas9系统的作用机理及其在模式酵母和非模式酵母中的应用,并概述了CRISPRi/a技术在酵母中的应用,以期为酵母细胞的基因编辑研究提供参考。
1 CRISPR/Cas概述及分类
1.1 CRISPR/Cas系统的历史
1987年,日本科学家在大肠杆菌的基因组中首次发现了CRISPR序列[9],研究者在大肠杆菌的碱性磷酸酶基因iap附近发现5个29 bp的重复序列以32 bp的非重复序列为间隔排列。随后,在其他的细菌和古菌中也发现了这一特殊序列。2002年,该重复序列被JANSEN等[10]命名为CRISPR,即成簇规律间隔短回文重复序列。2005年,研究者发现CRISPR的间隔序列与外源噬菌体或质粒的DNA序列一致[11],并推测该系统与免疫机制有关。2007年,BARRANGOU等[12]通过实验发现去除或添加特定的间隔序列会改变细菌细胞对噬菌体的抵抗力,证实了CRISPR系统的获得性免疫作用,发现CRISPR序列与相关的cas基因一起为细菌提供了对噬菌体的抗性,这说明免疫特异性取决于间隔序列。2008年,BROUNS等[13]发现CRISPR可以转录出一些特殊的RNA,即CRISPR RNA(crRNA),这些RNA与Cas蛋白结合成“核酸-蛋白复合体”,以此来发挥功能干扰噬菌体。同年,MARRAFFINI等[14]证明了CRISPR/Cas系统以基因组DNA为靶点而不是以RNA为靶点。2010年,GARNEAU等[15]证实了Cas9可以在crRNA的指导下快速地切割特定序列的DNA。2011年,DELTCHEVA等[16]在研究酿脓链球菌(Streptococcus pyogenes)时发现了一种反式编码的RNA,即tracrRNA(trans-activating CRISPR RNA),并发现其通过广泛保守的内源性RnaseⅢ和CRISPR相关的Csn1蛋白的活性来指导crRNA的生成。同年,SAPRANAUSKAS等[17]发现可以将CRISPR/Cas系统从嗜热链球菌(Streptococcus thermophilus)转移至大肠杆菌(Escherichia coli)并使其发挥免疫活性。
随着CRISPR/Cas系统相关的元件被不断发现,以及对结构功能的持续研究,CRISPR/Cas系统的成员也逐渐丰富,科学家们开始研究其具体的应用。2012年,JINEK等[18]首次提出可以用CRISPR进行体外切割,同年GASIUNAS等[19]发现了Cas9-crRNA复合体是一种RNA引导的内切酶,具有RNA导向的靶序列识别作用和蛋白质介导的DNA切割作用。2013年,多个研究组相继证明了可以利用CRISPR/Cas9系统在哺乳动物中进行基因编辑[20-21],这一发现在2020年为EMMANUELLE CHARPENTIER和JENNIFER DOUDNA两位科学家赢得了诺贝尔化学奖。2014年,研究人员进行了基于Cas9的全基因组功能筛选,并确定Cas9-sgRNA复合物的晶体结构[22]。2014年至今,关于CRISPR/Cas技术的相关应用越来越多,研究员们实现了CRISPR/Cas技术在植物、动物、真菌和细菌等多种生物中的应用[8]。CRISPR/Cas技术的发展历程如图1所示。
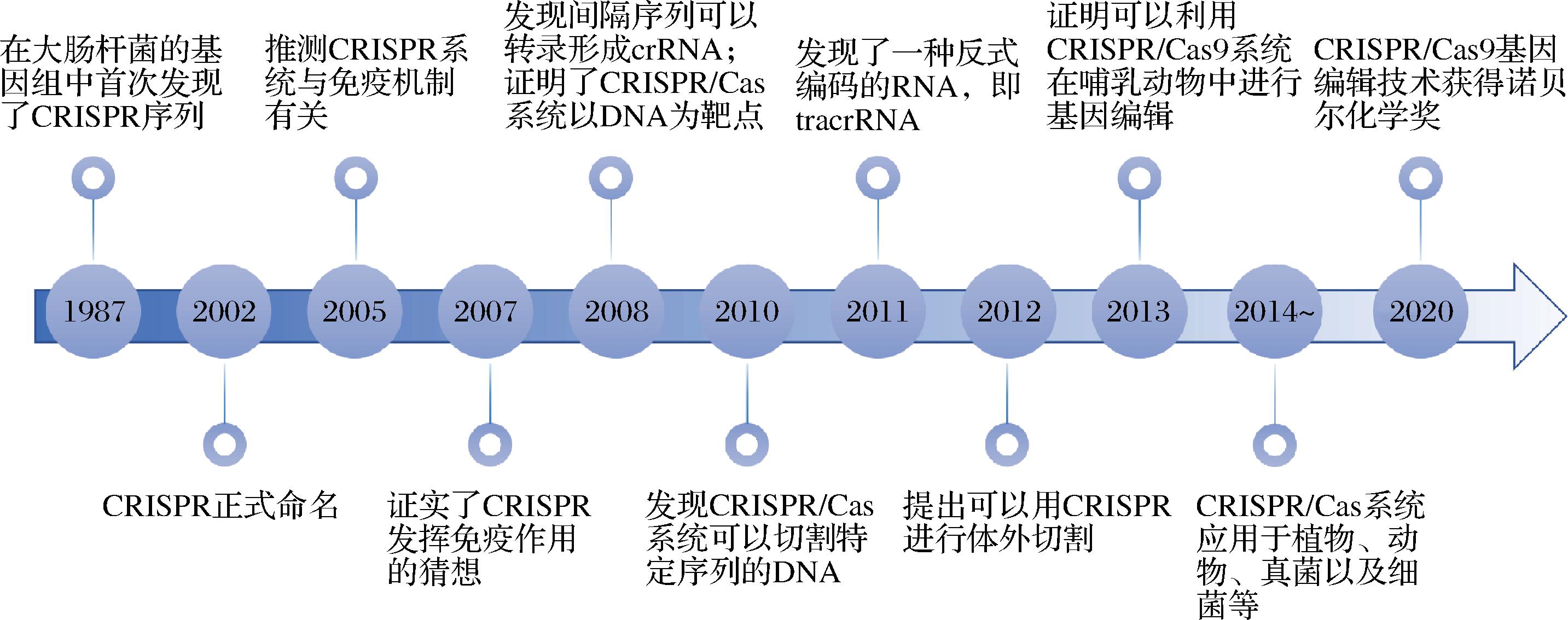
图1 CRISPR/Cas技术的发展历程
Fig.1 Development of CRISPR/Cas technology
1.2 CRISPR/Cas系统的结构及其分类
CRISPR/Cas系统主要包括CRISPR基因座和CRISPR关联(CRISPR associated,Cas)基因两部分(图2)。CRISPR基因座主要由前导区(leader)、重复序列(repeats)和间隔序列(spacers)构成。其中,前导区位于CRISPR基因上游,被认为是CRISPR序列的启动子;重复序列长度为20~50 bp,含有回文序列,可以形成发卡结构;间隔序列是被细菌俘获的外源DNA序列。Cas基因位于CRISPR基因附近或分散于基因组其他地方,编码的蛋白包括Cas1~Cas10,可以与CRISPR序列区域共同发生作用。
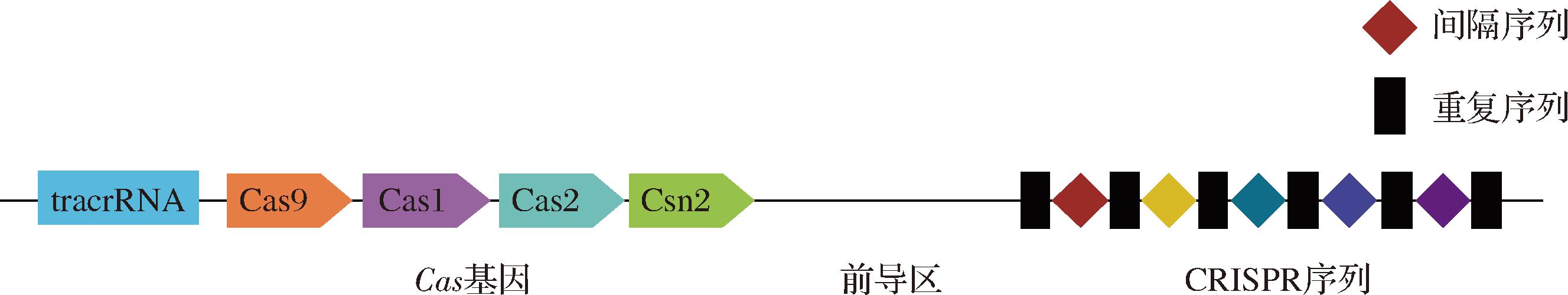
图2 CRISPR/Cas系统的结构
Fig.2 Structure of CRISPR/Cas
CRISPR/Cas系统存在多种类型(图3),依据Cas效应蛋白组成的不同,将其分为两大类:Class Ⅰ和Class Ⅱ[23]。第1大类(Class Ⅰ)的Cas效应蛋白复合物是由4~7个Cas蛋白亚基组成,多发现于细菌和古细菌中,该类别的共同特征是利用多个Cas蛋白效应复合物,实现对靶标核酸的干扰。第2大类(Class Ⅱ)的Cas效应蛋白是一个单一的、多结构域的、分子质量较大的Cas蛋白,目前只在细菌中有发现。根据CRISPR/Cas系统作用机制的适应、表达、干扰3个阶段不同,又可以将两大类系统细分为6种类型,其中Class Ⅰ系统包含类型Ⅰ、Ⅲ和Ⅳ,Class Ⅱ系统包含类型Ⅱ、V和Ⅵ。在适应阶段,通过Cas1和Cas2蛋白的参与俘获外源DNA;表达阶段,ClassI系统中由Cas6同源蛋白加工crRNA,而Class Ⅱ系统中Ⅱ型CRISPR/Cas系统中则由Cas9和RNase Ⅲ负责加工crRNA,V型和Ⅵ型CRISPR/Cas系统中分别由Cas12和Cas13加工crRNA;在干扰阶段,Class I系统中crRNA与多个蛋白组成复合物与靶DNA相结合,而Class Ⅱ系统中crRNA与单个蛋白组成复合物发挥作用。因为效应复合物结构简单,Class Ⅱ系统常被改造成为基因编辑工具,目前研究最热门的2种CRISPR/Cas系统,即CRISPR/Cas9和CRISPR/Cas12a系统都属于Class Ⅱ系统。CRISPR/Cas9系统包含HNH和Ruvc 2个核酸酶结构域,这2个结构域分别负责切割与crRNA互补的DNA序列和非互补的DNA序列[18];CRISPR/Cas12a系统只含有1个类似Ruvc的核酸酶结构域来切割靶标DNA[24]。
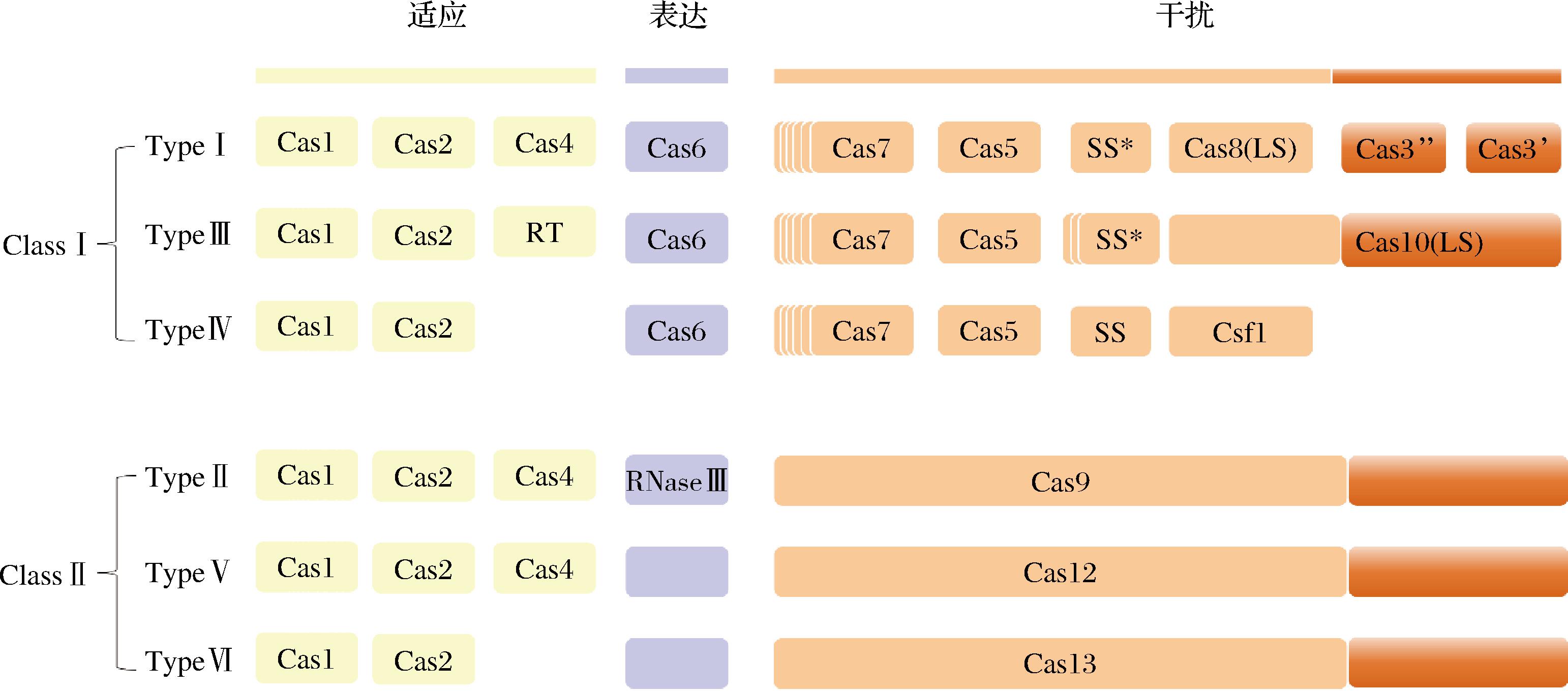
图3 CRISPR/Cas系统的分类
Fig.3 Classification of CRISPR/Cas
注:*表示在几个Ⅰ型亚型中,推定的小亚基(SS)蛋白被融合到了Cas8[Ⅰ型系统的大亚基(LS)]上,而不是作为一个独立的蛋白存在。
1.3 CRISPR/Cas9技术及其工作原理
CRISPR/Cas9系统以其高效性、广泛适应性、操作简便性和巨大的应用潜力等优势,在基因编辑领域脱颖而出,成为当前最受欢迎的基因编辑工具之一。CRISPR/Cas9系统进行免疫防御的过程如图4所示,大致包括3个阶段[8]:适应、表达、干扰。在适应阶段,Cas1和Cas2编码的蛋白将扫描入侵的DNA,并识别出原型间隔区相邻基序(protospacer adjacent motif,PAM),然后将临近PAM的DNA序列作为候选的原间隔序列,接着将原间隔序列从外源DNA剪切出来,并在其他酶的协助下将原间隔序列插入CRISPR位点的前导序列与第一段重复序列之间,形成一个新的重复序列。在表达阶段,CRISPR序列转录出pre-crRNA,pre-crRNA在RNaseⅢ的协助下被加工形成crRNA,这些crRNA与tracrRNA结合形成tracrRNA/crRNA复合物。在干扰阶段,tracrRNA,crRNA与Cas9形成最终复合物,识别并切割能与crRNA互补配对的外源DNA,产生双链断裂的DNA(DNA double-strand break,DSB),从而使入侵DNA降解,达到免疫防御的作用。Cas9需要crRNA和tracrRNA与之结合才能起到靶向切割的作用,为了方便使用,JINEK等[18]将两部分进行简化,组成了single-guide RNA(gRNA或sgRNA)。发生DSB后,宿主细胞通过非同源末端连接修复机制(non-homologous end joining,NHEJ)或同源重组修复(homology-directed repair,HDR)作用对断裂的DNA进行修复,NHEJ途径直接连接断裂末端,可在切割位点随机插入或删除碱基对,导致插入或缺失等突变;而HDR途径则通过同源DNA模板进行精确修复或插入,进而实现基因敲入和碱基编辑等基因组遗传修饰[25]。
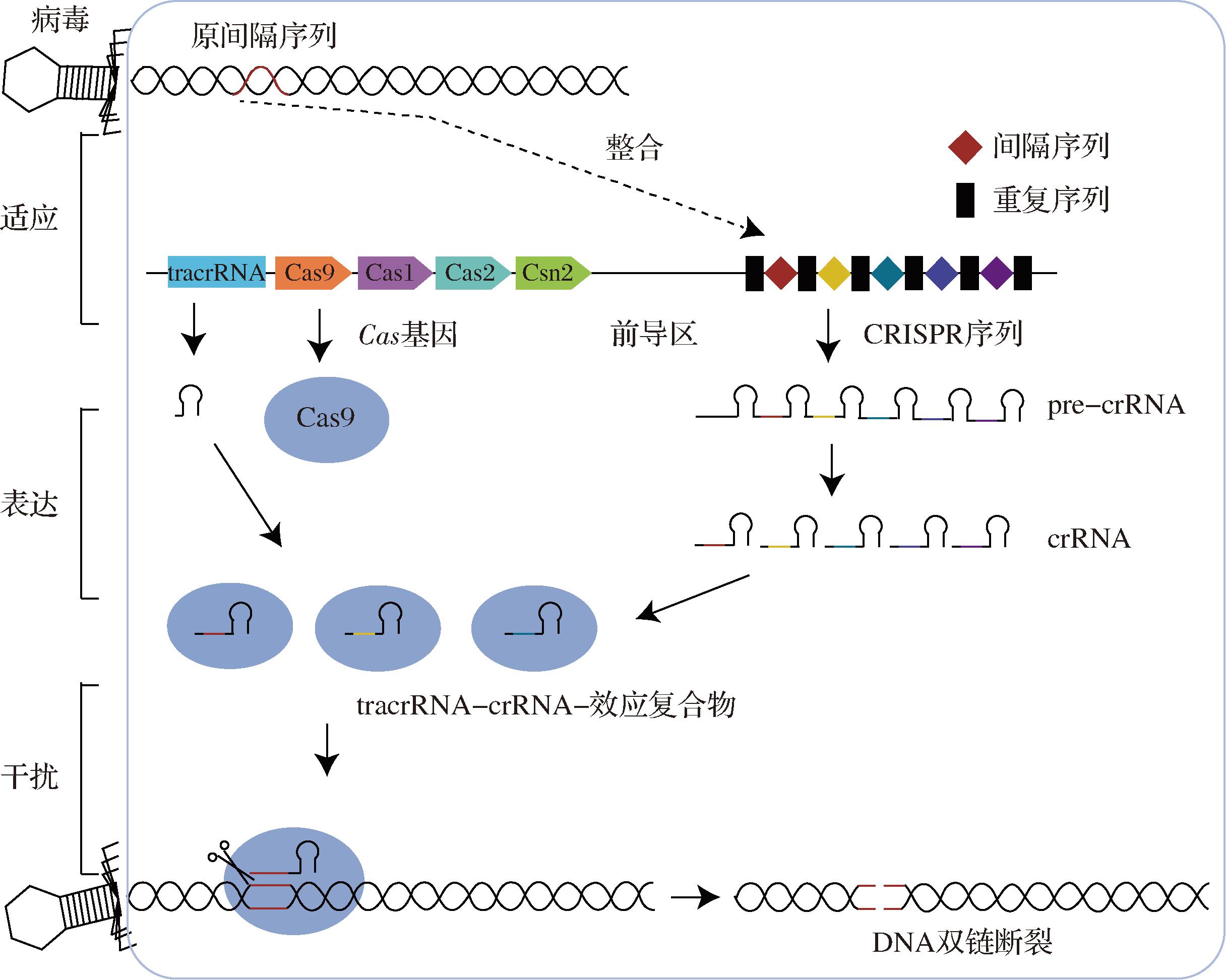
图4 CRISPR/Cas9系统的作用机制
Fig.4 Mechanism of CRISPR/Cas9 system
2 CRISPR/Cas9技术在模式酵母菌中的应用
2.1 CRISPR/Cas9技术在酿酒酵母中的应用
作为模式生物的酵母有2种,主要是酿酒酵母(Saccharomyces cerevisiae)和粟酒裂殖酵母(Schizosaccharomycs pombe)。S.cerevisiae因具有生长周期短、对不良环境耐受性强、遗传操作工具多、安全等优点,已被广泛地应用于食品、医药以及化工等领域。随着生物学的迅速发展,基因编辑技术在S.cerevisiae中实现了有效的应用,其中CRISPR/Cas9已被广泛的应用于S.cerevisiae的基因编辑。DICARLO等[26]在2013年首次利用CRISPR/Cas9系统在S.cerevisiae中敲除精氨酸通透酶编码基因CAN1,并且相较于传统敲除基因的同源重组法其效率提高了130倍。XUE等[27]使用CRISPR/Cas9技术敲除了乙醇脱氢酶编码基因ADH2,结果表明与使用天然菌株获得的乙醇产量相比,突变菌株获得的乙醇产量提高了74.7%。JUNG等[28]通过CRISPR/Cas9系统对S.cerevisiae的精氨酸酶编码基因CAR1进行敲除,从而显著降低了发酵培养基中氨基甲酸乙酯的产量。
除了对S.cerevisiae的单个基因进行编辑,利用CRISPR/Cas9系统对S.cerevisiae进行多基因编辑在提高酵母代谢产物路径的重构效率上更具优势。当CRISPR/Cas9系统要对体内多个靶点进行基因编辑时,需要产生多个gRNA与目的序列位点结合。目前,已有几种产生多个gRNA的策略:1)通过单个或多个表达盒同时表达多个gRNA的策略。例如JAKO I
I NAS等[29]通过构建含多个gRNA表达盒的质粒,实现了对甲羟戊酸合成途径中5个相关基因的编辑,即对YJL064、WYPL062W、BTS1和ROX1的敲除以及对ERG9基因转录水平的下调,从而显著提高了甲羟戊酸的产量。RONDA等[30]开发了一种新的方法CrEdit(CRISPR/Case9 mediated genome Editing),将CRISPR/Cas9的高特异性与EasyClone载体的稳定性相结合,对3个β-胡萝卜素合成基因BTS1、CRTYB和CRTI同时进行整合,极大提高了整合效率。2)通过3种RNA剪切过程实现多个gRNA的产生的策略,即利用来源于绿脓杆菌(Pseudomonas aeruginosa)的内切核糖核酸酶Csy4剪切系统、tRNA序列介导的内源RNA酶剪切系统和来源于病毒的核酶剪切系统产生多个gRNA。例如:FERREIRA等[31]利用Csy4对RNA的加工能力,从单个转录本生成多个gRNA,对FAA1、FAA4、POX1和TES1进行了四重缺失,达到96%的编辑效率;ZHANG等[32]报道了一个针对CRISPR-Cas9的gRNA-tRNA阵列系统:GTR-CRISPR,该系统能够以87%的效率同时破坏8个基因,大大提高了对S.cerevisiae基因组的编辑效率;RYAN等[33]将gRNA与自裂解丁型肝炎病毒(hepatitis delta virus,HDV)核酸酶融合,实现对S.cerevisiae中3个基因座的同时敲除。
NAS等[29]通过构建含多个gRNA表达盒的质粒,实现了对甲羟戊酸合成途径中5个相关基因的编辑,即对YJL064、WYPL062W、BTS1和ROX1的敲除以及对ERG9基因转录水平的下调,从而显著提高了甲羟戊酸的产量。RONDA等[30]开发了一种新的方法CrEdit(CRISPR/Case9 mediated genome Editing),将CRISPR/Cas9的高特异性与EasyClone载体的稳定性相结合,对3个β-胡萝卜素合成基因BTS1、CRTYB和CRTI同时进行整合,极大提高了整合效率。2)通过3种RNA剪切过程实现多个gRNA的产生的策略,即利用来源于绿脓杆菌(Pseudomonas aeruginosa)的内切核糖核酸酶Csy4剪切系统、tRNA序列介导的内源RNA酶剪切系统和来源于病毒的核酶剪切系统产生多个gRNA。例如:FERREIRA等[31]利用Csy4对RNA的加工能力,从单个转录本生成多个gRNA,对FAA1、FAA4、POX1和TES1进行了四重缺失,达到96%的编辑效率;ZHANG等[32]报道了一个针对CRISPR-Cas9的gRNA-tRNA阵列系统:GTR-CRISPR,该系统能够以87%的效率同时破坏8个基因,大大提高了对S.cerevisiae基因组的编辑效率;RYAN等[33]将gRNA与自裂解丁型肝炎病毒(hepatitis delta virus,HDV)核酸酶融合,实现对S.cerevisiae中3个基因座的同时敲除。
多倍体酵母含有2个或多个染色体组,存在多个等位基因,并且缺乏筛选标记,因此增加了对其遗传改造的复杂性和不确定性。而随着基因编辑技术的不断发展,利用CRISPR/Cas9系统在多倍体S.cerevisiae中进行基因编辑也显示了良好的应用潜力。LEE等[34]利用CRISPR/Cas9技术,在工业多倍体S.cerevisiae中,敲除了丙酮酸脱羧酶编码基因PDC和乙醇脱氢酶编码基因ADH,从而提高了2,3-丁二醇的产量。LIAN等[35]通过增加表达gRNA质粒的拷贝数,对二倍体S.cerevisiae(共8个等位基因)和三倍体S.cerevisiae(共12个等位基因)中的4个基因进行敲除,将敲除效率提高至100%。
2.2 CRISPR/Cas9技术在粟酒裂殖酵母中的应用
粟酒裂殖酵母S.pombe作为一种重要的模式生物,在遗传学和细胞生物学领域备受瞩目。由于缺乏用于表达gRNA的RNA聚合酶Ⅲ启动子,CRISPR-Cas9基因组编辑系统在S.pombe中的应用进展缓慢。2014年,JACOBS等[36]利用rrk1的RNA启动子以及锤头状核酶(hammer head ribozyme,HHR)构建了sgRNA表达系统,从而在S.pombe中实现了无筛选标记的特异性诱变。该体系能在S.pombe中进行快速高效的基因组操作,CRISPR/Cas9技术在S.pombe中的应用由此开启。2016年,FERNANDEZ等[37]使用Fex1p作为选择标记,对PIL1基因实现了靶向编辑,编辑效率为33%。由于将sgRNA目标序列引入质粒的克隆步骤最为繁琐和耗时,为了提高在S.pombe中应用CRISPR/Cas9系统的效率,ZHANG等[38]开发了一种无克隆程序,该程序利用S.pombe细胞中的间隙修复将2个线性DNA片段组装成环状质粒,这种基于间隙修复且无克隆的CRISPR/Cas9程序能在S.pombe中快速有效地进行点突变敲入、内源性N端标记和基因组序列删除。
3 CRISPR/Cas9技术在非模式酵母中的应用
比较具有代表性的非模式酵母有巴斯德毕赤酵母(Pichia pastoris)、解脂耶氏酵母(Yarrowia lipolytica)和乳酸克鲁维酵母(Kluyveromyces lactis)等,均已被美国食品药品监督管理局(Food and Drug Administration,FDA)认证为一般认可安全的微生物(generally recognized as safe,GRAS)[39]。近年来,已有越来越多的非模式酵母完成了全基因组的测序,CRISPR/Cas9基因编辑技术也在这些酵母中实现了大力发展。
3.1 CRISPR/Cas9技术在毕赤酵母中的应用
巴斯德毕赤酵母P.pastoris是一种甲醇营养型酵母,能利用甲醇作为唯一碳源和能源,其具有易于高密度培养、对蛋白修饰能力强、表达强度高等优点,是最常用的真核表达宿主之一[40],因此对P.pastoris建立一个高效且便捷的基因编辑技术至关重要。目前在酵母中最常使用的Cas9蛋白是来自于化脓性链球菌(Streptococcus pyogenes)的SpCas9。WENINGER等[41]在2016年通过对Cas9的密码子和sgRNA表达方式进行优化,初次在P.pastoris中建立了可高效打靶的CRISPR/Cas9系统,并发现经过人源密码子优化的HsCas9相较于SpCas9和经过P.pastoris表达密码子优化的PpCas9在P.pastoris中的编辑效率更高。此外,在sgRNA启动子选择方面,该课题组通过测试发现,在sgRNA两端接连HHR/HDV核酶,能显著提高切割效率。
利用CRISPR/Cas9基因编辑技术不仅可以实现对基因的敲除,也可以实现目的基因的定点整合。在P.pastoris中,当Cas9切割基因组DNA产生DSB后,主要依靠NHEJ机制进行修复,这个过程更容易导致基因的插入、缺失和点突变等错误修复,而HDR是一种更精确的修复机制,因此通常会通过抑制NHEJ或增强HDR的方法来建立高效的CRISPR/Cas9系统介导的定点整合技术,例如,WENINGER等[42]通过敲除参与NHEJ的关键基因Ku70,进一步提高了P.pastoris的同源重组能力。GAO等[43]通过过表达HDR相关基因(RAD52、RAD59、MRE11和SAE2)提高了P.pastoris同源重组的能力,实现了单位点、双位点和三位点的整合,效率高达100%、98%和81%。ZHANG等[44]通过在Cas9蛋白的C端融合表达MRE11同时过表达RAD52,使双基因编辑效率提升至86.7%,三基因编辑效率提升至16.7%。此外,还可以通过基因组整合实现P.pastoris细胞工厂的构建,LIU等[45]建立了一种以高效gRNA为靶点的CRISPR/Cas9介导的多位点基因整合方法,实现了双位点整合效率达到70%,三位点共整合效率达到32%,并通过功能基因的一步整合成功组装了6-甲基水杨酸和3-甲基邻苯二酚的生物合成途径。GAO等[46]建立了基于CRISPR的合成生物学工具包,实现了单位点、双位点和三位点的整合,效率高达100%、93%和75%,并证明了利用该工具包可以一步构建P.pastoris细胞工厂用于天然产物微生物(如β-胡萝卜素、玉米黄质和虾青素)的合成。
3.2 CRISPR/Cas9技术在解脂耶氏酵母中的应用
解脂耶氏酵母Y.lipolytica是一种重要的产油微生物,因具有多种独特的生理代谢特征,已成为了当今代谢工程及合成生物学领域中研究和应用最为广泛的工业微生物底盘细胞之一[47]。2016年,SCHWARTZ等[48]首次将CRISPR/Cas9技术应用到了解脂耶氏酵母中,通过将密码子优化的Cas9蛋白与用甘氨酸tRNA(tRNAGly)与RNA聚合酶Ⅲ启动子融合产生的杂交启动子(SCR1′-tRNAGly)表达的sgRNA转化到宿主菌株中,实现了单基因的敲除,效率达到92%。GAO等[49]在sgRNA两端接连HHR/HDV核酶,由内源性的强启动子TEFin控制sgRNA表达,再将Cas9和sgRNA表达盒插入到同一个质粒,并转化到野生型Y.lipolytica中,使单基因编辑效率达到85.6%。进一步通过串联多个sgRNA的表达盒,实现了双基因和三基因的敲除,效率分别达到36.7%和19.3%。HOLKENBRINK等[50]又构建了针对解脂耶氏酵母底盘的EasyCloneYAL1系统,使双基因敲除的效率从6%到66%不等。
在基因整合方面,SCHWARTZ等[48]在Y.lipolytica中通过敲除NHFJ途径中的关键基因KU70,提高了其同源重组的能力,使得基因编辑效率达到94.1%。JI等[51]通过在Y.lipolytica中过表达与HDR机制相关的RAD52基因,提高了其同源重组的效率,是野生型菌株的6.5倍,KU70敲除菌株的1.6倍。
3.3 CRISPR/Cas9技术在克鲁维酵母中的应用
克鲁维酵母属下的酵母中,研究较为广泛的为乳酸克鲁维酵母(Kluyveromyces lactis)和马克思克鲁维酵母(Kluyveromyces marxianus)。乳酸克鲁维酵母K.lactis是一种可以利用乳糖为唯一碳源的酵母,具有生长速率快、蛋白分泌能力强和碳源利用范围广等优点;马克思克鲁维酵母K.marxianus是一种广泛存在于发酵乳中的酵母,具有生长速率快、耐高温、底物范围宽广等优点[52]。LIAO等[53]通过敲除NHEJ相关基因KU80并对一系列流程进行优化,构建了用于K.lactis的CRISPR/Cas9系统,使单基因编辑效率提高至80%。BURGHARDT等[54]敲除了K.lactis的内源果糖转化酶编码基因INV,使合成低聚果糖的果糖转移酶活性提高了66.9%。在K.marxianus中,LÖBS等[55]通过将tRNAGly与RNA聚合酶Ⅲ启动子rpr1融合表达gRNA,得到了对XYL2的最高编辑效率,为(66±8)%。LIU等[56]在K.marxianus中利用CRISPR/Cas9技术对MTC6p基因进行了敲除,提高了异源蛋白的分泌表达。
3.4 CRISPR/Cas9技术在其他非模式酵母中的应用
由于缺乏合适的遗传操作工具,对非模式酵母菌的遗传改良一直是一个难题。随着CRISPR/Cas9基因编辑系统的不断发展,使得对非模式酵母菌进行基因编辑成为可能。热带假丝酵母(Candida tropicalis)是一种二倍体非模式酵母,广泛存在于自然界中,ZHANG等[57]建立了适用于C.tropicalis的CRISPR/Cas9系统,实现了对单基因和双基因的敲除,对2个位点的单基因编辑效率为57%~100%,对双基因(4个位点)同时编辑的效率可达32%,并基于该系统建立了适用于C.tropicalis的β-胡萝卜素及其衍生物的生物合成平台。圆红冬孢酵母(Rhodotorula toruloides)是一种能够天然合成多种类胡萝卜素和油脂的非模式酵母,OTOUPAL等[58]通过CRISPR/Cas9基因编辑技术,使得在R.toruloides中对URA3的敲除效率为0.001%,后续通过优化sgRNA序列,使对URA3的编辑效率提升至0.62%。应用这些优化的设计条件,使得对另一种与类胡萝卜素生物合成有关的基因CAR2的编辑效率达到了50%。多形汉逊酵母(Ogataea polymorpha)因其能利用甲醇、生长快、耐高温、异源蛋白表达能力强、底物谱广泛等优良的生物学特性,被广泛应用于生产蛋白药物、酶制剂及各类天然产物等,GAO等[59]在O.polymorpha中建立了有效的CRISPR/Cas9系统,通过过表达HDR相关基因并下调NHEJ相关基因,使同源重组效率从20%~30%提高至60%~70%。除此之外,CRISPR/Cas9基因编辑技术在其他酵母种属中也实现了应用,例如雄风生假丝酵母(Starmerella bombicola)[60]、白色念珠菌(Candida albicans)[61]以及树干毕赤酵母(Scheffersomyces stipitis)[62]。
4 CRISPR/Cas9基因编辑体系的构建及优化
CRISPR/Case9基因编辑体系由sgRNA和Cas9构成,sgRNA可以指导Cas9蛋白对靶基因进行敲除、插入和突变修饰。因此在CRISPR/Cas9基因编辑体系的构建中,对Cas9和sgRNA的选择及表达尤为重要。
4.1 Cas9的优化
CRISPR/Cas9应用于基因编辑技术时,Cas基因编码的Cas蛋白在防御过程中至关重要,目前在酵母菌中最常用的Cas9是来自S.pyogenes的SpCas9,其PAM序列为5′-NGG-3′。Cas9的表达与启动子以及密码子优化有关:在启动子的选择方面,发现使用强启动子对Cas9进行表达会对菌株的生长和适应性会产生负面影响,例如HXT7、TDH3。使用诱导型启动子会使靶向效率降低,例如CUP1。而使用弱或者中等强度的启动子足以使Cas9表达并对菌株的生长速率没有影响,例如ROX3、FBA1和RNR2[63];其次表达的Cas9基因,既可以是天然的,也可以是经过密码子优化的,例如WENINGER等[41]发现经过人源密码子优化的HsCas9相较于SpCas9在毕赤酵母中的编辑效率更高。
4.2 sgRNA的优化
sgRNA是将crRNA和tracRNA结合后得到的单个RNA复合物,使得CRISPR/Cas9体系的构建和应用更容易。在CRISPR/Cas9介导的基因编辑中,由sgRNA指导Cas9对特定的基因进行编辑,因此需要人工设计sgRNA并在体内表达,sgRNA的5′端1~20个核苷酸对应目的DNA序列不同而做改变,使Cas9蛋白在预期位点发生切割作用。为了确保sgRNA发挥正常的功能,选择合适的启动子和终止子至关重要。酵母中常用的启动子有RNA聚合酶Ⅲ启动子(SNR52)[64]、tRNA启动子[65]和U6启动子[66],常用的终止子是SUP4[63]。对于sgRNA的长度,FU等[67]发现在人类细胞中使sgRNA的长度截短至17或18 nt可以减少脱靶效率,然而在酿酒酵母中,相较于全长的sgRNA,截短的sgRNA在编辑效率上并未展现出显著的优越性或改进。
4.3 Cas9和sgRNA的表达
Cas9和sgRNA需要在功能上共表达,才能在基因组中产生DSB。使用CRISPR/Cas9进行酵母基因编辑,既可以使用2个载体分别表达sgRNA和Cas9,也可以将Cas9整合到基因组中,而sgRNA由质粒表达。除此之外,还可以应用1个质粒同时表达Cas9和sgRNA,并且被表达的sgRNA可以是1个或者多个。例如GENEROSO等[68]在酿酒酵母构建了Cas9和gRNA于一体的载体pRCC-K(表达1个sgRNA)和pDuRCCK(表达2个sgRNA),大大提高了敲除效率。
4.4 CRISPR/Cas9针对脱靶效应的优化
在CRISPR/Cas9介导的基因编辑技术过程中,由于sgRNA可能会与非靶点DNA序列错配,导致在实验过程中发生脱靶效应。目前,已经报道了一些针对脱靶效应的CRISPR/Cas9基因编辑体系优化的方法,例如:1)提高sgRNA的特异性,可以通过设计合理的sgRNA并利用评估脱靶效应的相关软件进行计算机验证,例如使用sgRNAcas9、CasO T和Off-Spotter等软件[69];2)选择优化的Cas9[41];3)调整Cas9和sgRNA的剂量;4)优化递送系统,使用病毒载体或电穿孔等方法,精确递送基因编辑工具,提高特异性。尽管CRISPR/Cas9基因编辑技术展现出了巨大的潜力,其脱靶效应仍是限制其广泛应用的一个重要因素,因此需要进一步开发新的优化策略以有效减轻脱靶效应的影响。
5 CRISPR衍生技术在酵母中的应用
随着对CRISPR/Cas9系统研究的日益深化,一系列衍生技术也相继涌现并在酵母基因编辑中发挥强大的作用。Cas9是一种能够靶向切割DNA序列的核酸酶,而dCas9(dead Cas9)是对Cas9的核酸酶结构域Ruvc1和HNH进行突变后而形成的一种失去了切割DNA功能的Cas9。dCas9蛋白失去内切核酸酶活性,但保留了结合特定DNA序列的能力,将dCas9与不同效应物融合进行工程化改造,便可以实现序列特异性基因组的转录调控,典型应用就是CRISPR干扰(CRISPR interference,CRISPRi)和CRISPR激活(CRISPR activation,CRISPRa)[70],CRISPRa/i技术在酵母中的应用案例如表1所示。
表1 CRISPRa/i技术在酵母中的应用案例
Table 1 Examples of CRISPRa/i technology in yeast

菌株技术应用参考文献Saccharomyces cerevisiaeCRISPRi通过抑制竞争通路的7个基因,提升β-香树脂醇的产量[71]Saccharomyces cerevisiaeCRISPRi通过抑制竞争通路的ERG7基因,提升原人参二醇的产量[72]Saccharomyces cerevisiaeCRISPRi通过抑制竞争通路的基因PDC1,提升2,3-丁二醇的产量[73]Saccharomyces cerevisiaeCRISPRi通过抑制竞争通路的基因ERG6,提升7-脱氢胆固醇的产量[74]Saccharomyces cerevisiaeCRISPRi通过三重CRISPRi系统同时抑制竞争通路的4个基因TYR1、ARO8、AAT2和ALD3,提升2-苯乙醇的产量[82]Saccharomyces cerevisiaeCRISPRi通过CRISPRi筛选揭示了木质素纤维素水解产物中能酿酒酵母生长的基因[83]Pichia pastorisCRISPRi降低醇氧化酶AOX1酶活性[75]Yarrowia lipolyticaCRISPRi抑制NHFJ相关的9个基因,从而增强HDR能力[76]Yarrowia lipolyticaCRISPRi通过抑制竞争通路的基因FAS1、FAS2,提高了柚皮素的产量[77]Kluyveromyces marxianusCRISPRi通过抑制TCA和ETC相关基因,提高了乙酸乙酯的生产率[78]Kluyveromyces marxianusCRISPRi通过抑制乙酸异戊酯酶编码基因IAH1,使乙酸乙酯的产量减少[84]
续表1

菌株技术应用参考文献Saccharomyces cerevisiaeCRISPRadCas9与3种激活剂VP64-P65-RTA(VPR)融合,靶向酿酒酵母中的168个基因,提高了3-羟基丙酸的产量[79]Yarrowia lipolyticaCRISPRadCas9与激活因子VPR融合,提高了β-葡萄糖苷酶编码基因BGL1和BGL2的表达水平,从而使解脂耶氏酵母能够利用纤维二糖[80]Yarrowia lipolyticaCRISPRadCas9与激活因子VPR融合,对FAR基因进行调控,提高了脂肪醇的产量[81]
5.1 基于CRISPRi的基因转录调控在酵母中的应用
CRISPRi通过利用sgRNA指导dCas9靶向结合到目标基因的启动子区域,阻碍RNA聚合酶与DNA序列的结合,从而抑制基因的转录。为了提升S.cerevisiae中β-香树脂醇的产量,NI等[71]利用CRISPRi系统构建了一个含有多个sgRNA盒的质粒,同时下调竞争通路中的7个基因,包括ADH1、ADH4、ADH5、ADH6、CIT2、MLS2和ERG7,7个基因的平均转录抑制效率达到75.5%,从而减少前体物质乙酰辅酶A和2,3-氧化鲨烯的竞争性消耗,进一步优化发酵条件并外加甲基β-环糊精促进β-香树脂醇的胞外转运,使得到的工程酿酒酵母菌株能产生高达156.7 mg/L 的β-香树脂醇,与原酵母菌株相比提高了44.3%。原人参二醇由2,3-氧化鲨烯合成,然而在酵母中2,3-氧化鲨烯会在羊毛甾醇合酶的催化下天然地转化为羊毛甾醇,因此LIM等[72]在S.cerevisiae中通过CTRISPRi系统下调了羊毛甾醇合酶编码基因ERG7,使2,3-氧化鲨烯转化为达马烯二醇Ⅱ,并进一步合成原人参二醇,在摇瓶发酵和分批发酵中,使原人参二醇的产量分别增加了14.4倍和5.7倍。MORITA等[73]利用dCas9抑制了乙醇合成途径中丙酮酸脱羧酶编码基因PDC1,从而减少2,3-丁二醇生产过程中乙醇的合成,并提高了2,3-丁二醇的产量。7-脱氢胆固醇(7-dehydrocholesterol,7-DHC)是维生素D3的关键前体,QU等[74]在S.cerevisiae中利用CRISPRi系统对竞争途径的关键基因甾醇C-24甲基转移酶编码基因ERG6进行动态调控,使7-DHC的产量得到了提高。YANG等[75]在P.pastoris中,利用dCas9使醇氧化酶Aox1酶活水平在24 h降至28%~34%。在Y.lipolytica中,SCHWARTZ等[76]利用CRISPRi系统抑制NHEJ相关的9个基因来增强其HDR能力,其中的8个基因被有效地抑制。乙酰辅酶A和丙二酰辅酶A在脂肪酸合成酶Fas1p和Fas2p(由FAS1和FAS2编码)的作用下组装成长链饱和脂肪酸,而柚皮素和脂质竞争相同的前体丙二酰辅酶A,因此LYU等[77]在Y.lipolytica中,利用CRISPRi系统对FAS1、FAS2基因进行调控,减少脂肪酸的合成,从而提高柚皮素的产量。乙酸乙酯可由乙醇和乙酰辅酶A合成,因此LÖBS等[78]开发和应用多重CRISPRi系统,通过同时下调K.marxianus中三羧酸循环(tricarboxylic acid cycle,TCA)和电子传递链(electron transport chain,ETC)相关基因ACO2b、SDH2、RIP1和MSSSI的表达来调节乙醇和乙酰辅酶A的含量,使最终乙酸乙酯的产率提高了3.8倍。
5.2 基于CRISPRa的基因转录调控在酵母中的应用
CRISPRa通过将dCas9与转录激活因子融合,并在sgRNA指导下结合到目标基因的启动子区域,从而增强特定基因的转录活性。FERREIRA等[79]将dCas9与3种激活剂VP64-P65-RTA(VPR)融合,构建了一个包含3 195个gRNA的文库,靶向S.cerevisiae中的168个基因,并通过多轮荧光激活细胞分选和文库测序筛选出丙二酰辅酶A过度生产者,成功提高了丙二酰辅酶A衍生产物3-羟基丙酸(3-hydroxypropionic acid,3-HP)的产量。SCHWARTZ等[80]在将dCas9与外源的转录激活因子VPR融合,提高了Y.lipolytica内源的β-葡萄糖苷酶编码基因BGL1和BGL2的表达水平,从而使Y.lipolytica能够利用纤维二糖。FAR基因编码的脂酰辅酶A还原酶,可以催化脂酰辅酶A转化为脂肪醇,李孟旭[81]将dCas9与VPR融合建立了CRISPRa系统,并将其应用于脂肪醇代谢通路,对FAR基因进行调控,将脂肪醇产量提高至2.8倍。
5.3 CRISPRa/i组合调控在酵母中的应用
在同一细胞中结合多个CRISPR调控系统可以实现对通路基因多重调控。LIAN等[85]创建了包括CRISPRa、CRISPRi和基因缺失(CRISPR gene deletion,CRISPRd)的正交三功能CRISPR系统(CRISPR system that combines transcriptional activation,transcriptional interference,and gene deletion,CRISPR-AID),利用该系统同时激活甲羟戊酸途径酶编码基因HMG1、抑制β-胡萝卜素合成和内源性甾醇生物合成所必须的酶编码基因ERG9并敲除胁迫响应转录调控酶编码基因ROX1,使S.cerevisiae中β-胡萝卜素的产量增加了2.8倍。翟昊天[86]利用CRISPR a/i系统,在S.cerevisiae中对3-羟基丙酸合成途径中的关键基因ACC1进行激活并对竞争途径中的关键基因FAS1/FAS2/ARG3进行抑制,促进代谢通量更多地流向3-羟基丙酸,使其产量得到提升。李孟旭[81]利用双CRISPR/dCas9 scRNA系统同时实现了基因的转录激活和抑制,并且将此双向调控系统应用于Y.lipolytica中的脂肪醇代谢通路,通过对合成通路相关基因FAR进行激活,同时对降解途径相关基因PEX10进行抑制,使脂肪醇的产量增加到778.8 mg/L。
6 总结与展望
酵母菌作为人类生活中不可或缺的微生物之一,在食品工业、医药健康、生物技术及环境保护等领域都发挥着重要的作用,而通过基因编辑,科学家们可以深入探究其基因功能、代谢途径等生物学问题。CRISPR/Cas9系统是高效的基因组编辑工具,它的发展推动了经济高效的酵母细胞工厂的建设,但仍存在一些问题。一方面,虽然酿酒酵母已是研究最为深入的酵母,但对其完整的代谢途径和调节网络的认知仍然缺乏清晰而透彻的理解。并且利用CRISPR/Cas9技术对酿酒酵母进行改造还存在改变野生种群基因、发生脱靶效应以及多基因编辑效率低等问题。另一方面,与酿酒酵母相比,非模式酵母种类繁多,遗传背景不明,基因操作工具及外源基因导入策略研究较少,此外也受限于适用于非模式酵母的CRISPR/Cas9系统的研究开发较少,因此利用基因编辑技术对非模式酵母进行遗传操作或性能改良的进展缓慢。
但与传统的基因改造技术相比,CRISPR/Cas9系统仍具有显著优势,例如:应用广泛、组成简单、可以对多个基因进行编辑等,因此它依旧是改造酵母的强大工具。随着研究的不断深入,以及计算机技术、AI与生物学的交叉融合,靶序列设计工具将不断涌现,CRISPR/Cas9技术将越来越成熟。因此,随着CRISPR/Cas9基因编辑技术的不断改善和发展,科研工作者得以充分利用CRISPR/Cas9系统对酵母基因功能、代谢调控及其在环境适应性方面的机制等进行广泛而深入的研究,从而为合成生物学、工业生物技术以及新药开发等领域带来新的机遇。
[1] TAO Z K, YUAN H B, LIU M, et al.Yeast extract:Characteristics, production, applications and future perspectives[J].Journal of Microbiology and Biotechnology, 2023, 33(2):151-166.
[2] SUN T, YU Y Z, WANG K F, et al.Engineering Yarrowia lipolytica to produce fuels and chemicals from xylose:A review[J].Bioresource Technology, 2021, 337:125484.
[3] PAN Y J, YANG J, WU J P, et al.Current advances of Pichia pastoris as cell factories for production of recombinant proteins[J].Frontiers in Microbiology, 2022, 13:1059777.
[4] CARSANBA E, PINTADO M, OLⅣEIRA C.Fermentation strategies for production of pharmaceutical terpenoids in engineered yeast[J].Pharmaceuticals, 2021, 14(4):295.
[5] MUKHERJEE A, VERMA J P, GAURAV A K, et al.Yeast a potential bio-agent:Future for plant growth and postharvest disease management for sustainable agriculture[J].Applied Microbiology and Biotechnology, 2020, 104(4):1497-1510.
[6] YANG Z L, BLENNER M.Genome editing systems across yeast species[J].Current Opinion in Biotechnology, 2020, 66:255-266.
[7] HSU P D, LANDER E S, ZHANG F.Development and applications of CRISPR-Cas9 for genome engineering[J].Cell, 2014, 157(6):1262-1278.
[8] WANG J Y, DOUDNA J A.CRISPR technology:A decade of genome editing is only the beginning[J].Science, 2023, 379(6629):eadd8643.
[9] ISHINO Y, SHINAGAWA H, MAKINO K, et al.Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J].Journal of Bacteriology, 1987, 169(12):5429-5433.
[10] JANSEN R, VAN EMBDEN J D A, GAASTRA W, et al.Identification of genes that are associated with DNA repeats in prokaryotes[J].Molecular Microbiology, 2002, 43(6):1565-1575.
[11] BOLOTIN A, QUINQUIS B, SOROKIN A, et al.Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin[J].Microbiology, 2005, 151(Pt 8):2551-2561.
[12] BARRANGOU R, FREMAUX C, DEVEAU H, et al.CRISPR provides acquired resistance against viruses in prokaryotes[J].Science, 2007, 315(5819):1709-1712.
[13] BROUNS S J J, JORE M M, LUNDGREN M, et al.Small CRISPR RNAs guide antiviral defense in prokaryotes[J].Science, 2008, 321(5891):960-964.
[14] MARRAFFINI L A, SONTHEIMER E J.CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA[J].Science, 2008, 322(5909):1843-1845.
[15] GARNEAU J E, DUPUIS M  , ⅥLLION M, et al.The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J].Nature, 2010, 468(7320):67-71.
, ⅥLLION M, et al.The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J].Nature, 2010, 468(7320):67-71.
[16] DELTCHEVA E, CHYLINSKI K, SHARMA C M, et al.CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J].Nature, 2011, 471(7340):602-607.
[17] SAPRANAUSKAS R, GASIUNAS G, FREMAUX C, et al.The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli[J].Nucleic Acids Research, 2011, 39(21):9275-9282.
[18] JINEK M, CHYLINSKI K, FONFARA I, et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science, 2012, 337(6096):816-821.
[19] GASIUNAS G, BARRANGOU R, HORVATH P, et al.Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J].Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39):E2579-E2586.
[20] CONG L, ANN RAN F, COX D, et al.Multiplex genome engineering using CRISPR/Cas systems[J].Science, 2013, 339(6121):819-823.
[21] JINEK M, EAST A, CHENG A, et al.RNA-programmed genome editing in human cells[J].eLife, 2013, 2:e00471.
[22] ZHOU Y X, ZHU S Y, CAI C Z, et al.High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells[J].Nature, 2014, 509(7501):487-491.
[23] MAKAROVA K S, WOLF Y I, IRANZO J, et al.Evolutionary classification of CRISPR-cas systems:A burst of class 2 and derived variants[J].Nature Reviews.Microbiology, 2020, 18(2):67-83.
[24] ZETSCHE B, GOOTENBERG J S, ABUDAYYEH O O, et al.Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-cas system[J].Cell, 2015, 163(3):759-771.
[25] MENG J, QIU Y, SHI S B.CRISPR/Cas9 systems for the development of Saccharomyces cerevisiae cell factories[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:594347.
[26] DICARLO J E, NORⅥLLE J E, MALI P, et al.Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems[J].Nucleic Acids Research, 2013, 41(7):4336-4343.
[27] XUE T, LIU K, CHEN D, et al.Improved bioethanol production using CRISPR/Cas9 to disrupt the ADH2 gene in Saccharomyces cerevisiae[J].World Journal of Microbiology &Biotechnology, 2018, 34(10):154.
[28] JUNG J Y, KANG M J, HWANG H S, et al.Reduction of ethyl carbamate in an alcoholic beverage by CRISPR/Cas9-based genome editing of the wild yeast[J].Foods, 2022, 12(1):102.
[29] JAKO I
I NAS T, BONDE I, HERRGÅRD M, et al.Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae[J].Metabolic Engineering, 2015, 28:213-222.
NAS T, BONDE I, HERRGÅRD M, et al.Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae[J].Metabolic Engineering, 2015, 28:213-222.
[30] RONDA C, MAURY J, JAKO IUNAS T, et al.CrEdit:CRISPR mediated multi-loci gene integration in Saccharomyces cerevisiae[J].Microbial Cell Factories, 2015, 14:97.
IUNAS T, et al.CrEdit:CRISPR mediated multi-loci gene integration in Saccharomyces cerevisiae[J].Microbial Cell Factories, 2015, 14:97.
[31] FERREIRA R, SKREKAS C, NIELSEN J, et al.Multiplexed CRISPR/Cas9 genome editing and gene regulation using Csy4 in Saccharomyces cerevisiae[J].ACS Synthetic Biology, 2018, 7(1):10-15.
[32] ZHANG Y P, WANG J, WANG Z B, et al.A gRNA-tRNA array for CRISPR-Cas9 based rapid multiplexed genome editing in Saccharomyces cerevisiae[J].Nature Communications, 2019, 10(1):1053.
[33] RYAN O W, SKERKER J M, MAURER M J, et al.Selection of chromosomal DNA libraries using a multiplex CRISPR system[J].eLife, 2014, 3:e03703.
[34] LEE Y G, SEO J H.Production of 2, 3-butanediol from glucose and cassava hydrolysates by metabolically engineered industrial polyploid Saccharomyces cerevisiae[J].Biotechnology for Biofuels, 2019, 12:204.
[35] LIAN J Z, BAO Z H, HU S M, et al.Engineered CRISPR/Cas9 system for multiplex genome engineering of polyploid industrial yeast strains[J].Biotechnology and Bioengineering, 2018, 115(6):1630-1635.
[36] JACOBS J Z, CICCAGLIONE K M, TOURNIER V, et al.Implementation of the CRISPR-Cas9 system in fission yeast[J].Nature Communications, 2014, 5:5344.
[37] FERNANDEZ R, BERRO J.Use of a fluoride channel as a new selection marker for fission yeast plasmids and application to fast genome editing with CRISPR/Cas9[J].Yeast, 2016, 33(10):549-557.
[38] ZHANG X R, HE J B, WANG Y Z, et al.A cloning-free method for CRISPR/Cas9-mediated genome editing in fission yeast[J].G3, 2018, 8(6):2067-2077.
[39] XIA Y Y, LI Y J, SHEN W, et al.CRISPR-cas technology for bioengineering conventional and non-conventional yeasts:Progress and new challenges[J].International Journal of Molecular Sciences, 2023, 24(20):15310.
[40] ERGÜN B G, BERRIOS J, BINAY B, et al.Recombinant protein production in Pichia pastoris:From transcriptionally redesigned strains to bioprocess optimization and metabolic modelling[J].FEMS Yeast Research, 2021, 21(7):foab057.
[41] WENINGER A, HATZL A M, SCHMID C, et al.Combinatorial optimization of CRISPR/Cas9 expression enables precision genome engineering in the methylotrophic yeast Pichia pastoris[J].Journal of Biotechnology, 2016, 235:139-149.
[42] WENINGER A, FISCHER J E, RASCHMANOV H, et al.Expanding the CRISPR/Cas9 toolkit for Pichia pastoris with efficient donor integration and alternative resistance markers[J].Journal of Cellular Biochemistry, 2018, 119(4):3183-3198.
H, et al.Expanding the CRISPR/Cas9 toolkit for Pichia pastoris with efficient donor integration and alternative resistance markers[J].Journal of Cellular Biochemistry, 2018, 119(4):3183-3198.
[43] GAO J C, YE C F, CHENG J T, et al.Enhancing homologous recombination efficiency in Pichia pastoris for multiplex genome integration using short homology arms[J].ACS Synthetic Biology, 2022, 11(2):547-553.
[44] ZHANG K, DUAN X P, CAI P, et al.Fusing an exonuclease with Cas9 enhances homologous recombination in Pichia pastoris[J].Microbial Cell Factories, 2022, 21(1):182.
[45] LIU Q, SHI X N, SONG L L, et al.CRISPR-Cas9-mediated genomic multiloci integration in Pichia pastoris[J].Microbial Cell Factories, 2019, 18(1):144.
[46] GAO J C, XU J H, ZUO Y M, et al.Synthetic biology toolkit for marker-less integration of multigene pathways into Pichia pastoris via CRISPR/Cas9[J].ACS Synthetic Biology, 2022, 11(2):623-633.
[47] PARK Y K, LEDESMA-AMARO R.What makes Yarrowia lipolytica well suited for industry?[J].Trends in Biotechnology, 2023, 41(2):242-254.
[48] SCHWARTZ C M, HUSSAIN M S, BLENNER M, et al.Synthetic RNA polymerase Ⅲ promoters facilitate high-efficiency CRISPR-Cas9-mediated genome editing in Yarrowia lipolytica[J].ACS Synthetic Biology, 2016, 5(4):356-359.
[49] GAO S L, TONG Y Y, WEN Z Q, et al.Multiplex gene editing of the Yarrowia lipolytica genome using the CRISPR-Cas9 system[J].Journal of Industrial Microbiology &Biotechnology, 2016, 43(8):1085-1093.
[50] HOLKENBRINK C, DAM M I, KILDEGAARD K R, et al.EasyCloneYALI:CRISPR/Cas9-based synthetic toolbox for engineering of the yeast Yarrowia lipolytica[J].Biotechnology Journal, 2018, 13(9):1700543.
[51] JI Q C, MAI J, DING Y, et al.Improving the homologous recombination efficiency of Yarrowia lipolytica by grafting heterologous component from Saccharomyces cerevisiae[J].Metabolic Engineering Communications, 2020, 11:e00152.
[52] QIU Y B, LEI P, WANG R, et al.Kluyveromyces as promising yeast cell factories for industrial bioproduction:From bio-functional design to applications[J].Biotechnology Advances, 2023, 64:108125.
[53] LIAO L T, SHEN X R, SHEN Z Y, et al.CRISPR/Cas9-based genome editing for protein expression and secretion in Kluyveromyces lactis[J].ACS Synthetic Biology, 2024, 13(7):2105-2114.
[54] BURGHARDT J P, FAN R, BAAS M, et al.Enhancing the heterologous fructosyltransferase activity of Kluyveromyces lactis:Developing a scaled-up process and abolishing invertase by CRISPR/Cas9 genome editing[J].Frontiers in Bioengineering and Biotechnology, 2020, 8:607507.
[55] LÖBS A K, ENGEL R, SCHWARTZ C, et al.CRISPR-Cas9-enabled genetic disruptions for understanding ethanol and ethyl acetate biosynthesis in Kluyveromyces marxianus[J].Biotechnology for Biofuels, 2017, 10:164.
[56] LIU Y, MO W J, SHI T F, et al.Mutational Mtc6p attenuates autophagy and improves secretory expression of heterologous proteins in Kluyveromyces marxianus[J].Microbial Cell Factories, 2018, 17(1):144.
[57] ZHANG L H, ZHANG H B, LIU Y F, et al.A CRISPR-Cas9 system for multiple genome editing and pathway assembly in Candida tropicalis[J].Biotechnology and Bioengineering, 2020, 117(2):531-542.
[58] OTOUPAL P B, ITO M, ARKIN A P, et al.Multiplexed CRISPR-Cas9-based genome editing of Rhodosporidium toruloides[J].mSphere, 2019, 4(2):e00099-19.
[59] GAO J Q, GAO N, ZHAI X X, et al.Recombination machinery engineering for precise genome editing in methylotrophic yeast Ogataea polymorpha[J].iScience, 2021, 24(3):102168.
[60] SHI Y B, ZHANG L H, ZHANG M, et al.A CRISPR-Cas9 system-mediated genetic disruption and multi-fragment assembly in Starmerella bombicola[J].ACS Synthetic Biology, 2022, 11(4):1497-1509.
[61] FLETCHER J, O’CONNOR-MONELEY J, FRAWLEY D, et al.Deletion of the Candida albicans TLO gene family using CRISPR-Cas9 mutagenesis allows characterisation of functional differences in α-, β- and γ- TLO gene function[J].PLoS Genetics, 2023, 19(12):e1011082.
[62] PATRA P, DAS M, KUNDU P, et al.Recent advances in systems and synthetic biology approaches for developing novel cell-factories in non-conventional yeasts[J].Biotechnology Advances, 2021, 47:107695.
[63] RASCHMANOV H, WENINGER A, GLIEDER A, et al.Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts:Current state and future prospects[J].Biotechnology Advances, 2018, 36(3):641-665.
H, WENINGER A, GLIEDER A, et al.Implementing CRISPR-Cas technologies in conventional and non-conventional yeasts:Current state and future prospects[J].Biotechnology Advances, 2018, 36(3):641-665.
[64] RAINHA J, RODRIGUES J L, RODRIGUES L R.CRISPR-Cas9:A powerful tool to efficiently engineer Saccharomyces cerevisiae[J].Life, 2020, 11(1):13.
[65] MEFFERD A L, KORNEPATI A V R, BOGERD H P, et al.Expression of CRISPR/Cas single guide RNAs using small tRNA promoters[J].RNA, 2015, 21(9):1683-1689.
[66] XU L, ZHAO L X, GAO Y D, et al.Empower multiplex cell and tissue-specific CRISPR-mediated gene manipulation with self-cleaving ribozymes and tRNA[J].Nucleic Acids Research, 2017, 45(5):e28.
[67] FU Y F, SANDER J D, REYON D, et al.Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J].Nature Biotechnology, 2014, 32(3):279-284.
[68] GENEROSO W C, GOTTARDI M, OREB M, et al.Simplified CRISPR-Cas genome editing for Saccharomyces cerevisiae[J].Journal of Microbiological Methods, 2016, 127:203-205.
[69] CUI Y B, XU J M, CHENG M X, et al.Review of CRISPR/Cas9 sgRNA design tools[J].Interdisciplinary Sciences, Computational Life Sciences, 2018, 10(2):455-465.
[70] XU X B, HULSHOFF M S, TAN X Y, et al.CRISPR/cas derivatives as novel gene modulating tools:Possibilities and in vivo applications[J].International Journal of Molecular Sciences, 2020, 21(9):3038.
[71] NI J P, ZHANG G L, QIN L, et al.Simultaneously down-regulation of multiplex branch pathways using CRISPRi and fermentation optimization for enhancing β-amyrin production in Saccharomyces cerevisiae[J].Synthetic and Systems Biotechnology, 2019, 4(2):79-85.
[72] LIM S H, BAEK J I, JEON B M, et al.CRISPRi-guided metabolic flux engineering for enhanced protopanaxadiol production in Saccharomyces cerevisiae[J].International Journal of Molecular Sciences, 2021, 22(21):11836.
[73] MORITA K, SEIKE T, ISHⅡ J, et al.Improvement of 2, 3-butanediol production by dCas9 gene expression system in Saccharomyces cerevisiae[J].Journal of Bioscience and Bioengineering, 2022, 133(3):208-212.
[74] QU L S, XIU X, SUN G Y, et al.Engineered yeast for efficient de novo synthesis of 7-dehydrocholesterol[J].Biotechnology and Bioengineering, 2022, 119(5):1278-1289.
[75] YANG Y K, LIU G Q, CHEN X, et al.High efficiency CRISPR/Cas9 genome editing system with an eliminable episomal sgRNA plasmid in Pichia pastoris[J].Enzyme and Microbial Technology, 2020, 138:109556.
[76] SCHWARTZ C, FROGUE K, RAMESH A, et al.CRISPRi repression of nonhomologous end-joining for enhanced genome engineering via homologous recombination in Yarrowia lipolytica[J].Biotechnology and Bioengineering, 2017, 114(12):2896-2906.
[77] LYU Y K, GU Y, XU J L, et al.Coupling metabolic addiction with negative autoregulation to improve strain stability and pathway yield[J].Metabolic Engineering, 2020, 61:79-88.
[78] LÖBS A K, SCHWARTZ C, THORWALL S, et al.Highly multiplexed CRISPRi repression of respiratory functions enhances mitochondrial localized ethyl acetate biosynthesis in Kluyveromyces marxianus[J].ACS Synthetic Biology, 2018, 7(11):2647-2655.
[79] FERREIRA R, SKREKAS C, HEDIN A, et al.Model-assisted fine-tuning of central carbon metabolism in yeast through dCas9-based regulation[J].ACS Synthetic Biology, 2019, 8(11):2457-2463.
[80] SCHWARTZ C, WHEELDON I.CRISPR-Cas9-mediated genome editing and transcriptional control in Yarrowia lipolytica[J].Methods in Molecular Biology, 2018, 1772:327-345.
[81] 李孟旭. 解脂耶氏酵母CRISPR/Cas系统的建立及同时实现转录激活和抑制[D].天津:天津大学, 2022.LI M X.Establishment of a CRISPR/Cas system and simultaneous transcriptional activation and repression in Yarrowia lipolytic[D].Tianjin:Tianjin University, 2022.
[82] FANG S H, FAN X H, LI J L, et al.Triple-CRISPRi-mediateddown-regulation of the shikimate pathway branch genes for enhancing 2-PE biosynthesis in Saccharomyces cerevisiae[J].European Food Research and Technology, 2024, 250(7):1881-1890.
[83] GUTMANN F, JANN C, PEREIRA F, et al.CRISPRi screens reveal genes modulating yeast growth in lignocellulose hydrolysate[J].Biotechnology for Biofuels, 2021, 14(1):41.
[84] MU OZ-MIRANDA L A, ZEPEDA-PE
OZ-MIRANDA L A, ZEPEDA-PE A A C, CASAS-GODOY L, et al.CRISPRi-induced transcriptional regulation of IAH1 gene and its influence on volatile compounds profile in Kluyveromyces marxianus DU3[J].World Journal of Microbiology &Biotechnology, 2024, 40(4):121.
A A C, CASAS-GODOY L, et al.CRISPRi-induced transcriptional regulation of IAH1 gene and its influence on volatile compounds profile in Kluyveromyces marxianus DU3[J].World Journal of Microbiology &Biotechnology, 2024, 40(4):121.
[85] LIAN J Z, HAMEDIRAD M, HU S M, et al.Combinatorial metabolic engineering using an orthogonal tri-functional CRISPR system[J].Nature Communications, 2017, 8(1):1688.
[86] 翟昊天. 酿酒酵母中CRISPR介导的转录调控信号扩增系统的设计及应用[D].济南:山东大学, 2022.ZHAI H T.Design and application of CRISPR-mediated transcriptional regulatory signal amplification systems in Saccharomyces cerevisiae[D].Jinan:Shandong University, 2022.