解脂耶氏酵母(Yarrowia lipolytica)是一种半子囊菌纲的非常规酵母[1],被认为是安全的微生物(generally regarded as safe,GRAS),具有高细胞密度培养、代谢灵活、底物利用率高、产物种类广等重要工业应用特性[2]。对高浓度的盐、碳氢化合物和废碳具有相对较高的耐受能力[3],能产生糖醇来适应环境高渗透压[4]。解脂耶氏酵母还具有较高的磷酸戊糖途径(pentose phosphate pathway,PPP)代谢通量,能够为产物合成提供所需辅因子[5]。目前解脂耶氏酵母已完成全基因测序,遗传背景清晰,基因操作技术完善[6-7],该菌种具备丰富的启动子、终止子等遗传调控元件和预表征整合位点,并结合Golden Gate[8-9]、YaliBricks[10]等分子克隆技术开发了CRISPR基因组编辑技术,成为合成高价值化合物的优质底盘细胞。
在解脂耶氏酵母的基因编辑中,DNA双链断裂(double-strand breaks,DSB)主要是通过非同源末端重组(non-homologous end joining,NHEJ)进行修复。但NHEJ是随机修复[11],结果不可控,给菌株改造带来不确定性。解脂耶氏酵母的同源重组(homologous recombination,HR)能力很弱,当同源臂长度为0.5~1.0 kb时,解脂耶氏酵母的HR效率只有0%~36%[12-13],严重影响编辑效率。因此,有必要开发一种不依赖于同源重组机制的精准高效基因编辑技术。
ANZALONE等[14]开发了先导编辑(prime editing,PE)系统。PE系统不依赖供体DNA,不引入DSB,能以对细胞损伤更小的方式实现基因组DNA的插入、缺失和突变。在PE系统中,反转录酶(reverse transcriptase,RT)与具有DNA单链切割功能的Cas9H840A(nick Cas9,nCas9)组成融合蛋白,Cas9H840A在pegRNA(prime editing guide RNA,pegRNA)的介导下识别基因组并在指定DNA位点单链切割后产生切口后,反转录酶在pegRNA末端的结合位点(primer binding site,PBS)和反转录模板(reverse transcription template,RTT)的作用下催化反转录反应,在修复基因组切口的同时将预设在RTT的突变引入基因组,完成精准基因编辑。通过设计不同RTT,PE系统可以实现任意类型的基因编辑,被誉为“万能编辑器”[15],具有极高的应用潜力和价值。
本研究对CRISPR系统进行改造,首次在解脂耶氏酵母中成功构建了先导编辑系统(prime editing in Y.lipolytica,PEyl),并优化了pegRNA的稳定性,能够有效地对基因组进行无痕编辑,实现单碱基精度的删除、插入和置换,该系统为解脂耶氏酵母的遗传改造提供了新的合成生物学使能工具。研究中应用PEyl系统,实现了对解脂耶氏酵母的EYD1基因进行的移码突变失活,展现了PEyl系统在精准基因编辑中的潜力。
1 材料与方法
1.1 菌株、质粒和引物
解脂耶氏酵母Po1f被用于CRISPR和PEyl系统的改造和优化;大肠杆菌DH5α(Escherichia coli DH5α)被用于质粒的构建和扩增,本实验室保藏;质粒pCRISPRyl[16]被用于解脂耶氏酵母中CRISPR/Cas9基因编辑系统。PCR引物由生工生物工程(上海)股份有限公司或北京擎科生物科技股份有限公司合成。本文所用质粒、菌株、启动子和引物分别详见表1、表2、表3。
表1 本研究使用的质粒
Table 1 Plasmids used in this study

质粒相关特性来源pCRISPRyl解脂耶氏酵母中CRISPR/Cas9系统相关质粒实验室保存pPE1pCRISPRyl 携带PEX10的gRNA转录模块并删除Cas9表达模块本研究pPE2pCRISPRyl 突变Cas9H840A本研究pPE3pPE2携带PEX10的gRNA转录模块本研究pPE4pPE2携带PEX10的gRNA转录模块(删除1nt)本研究pPE5pCRISPRyl 携带Cas9H840A-M-MLV-RT表达模块本研究pPE6pPE5携带PEX10的gRNA转录模块本研究pPE7-Cas9nRT-pegRNA1pPE5携带PEX10的gRNA转录模块(删除1nt)本研究pPE8-Cas9nRT-pegRNA2pPE5携带PEX10的gRNA转录模块(插入1nt)本研究pPE9-Cas9nRT-pegRNA3pPE5携带PEX10的gRNA转录模块(置换of 1nt)本研究pPE10-Cas9nRT-pegRNA4pPE5携带EYD1的gRNA转录模块(删除1nt)本研究
注:nt:nucleotide,核苷酸(下同)。
表2 本研究使用的菌株
Table 2 Strains used in this study

菌株相关特性来源Y. lipolytica Po1f (ATCC MYA-2613)MATa, leu2-270, ura3-302, xpr2-322, axp1-2, pXPR2-SUC2 (URA3-, LEU2-)实验室保存Y.lipolytica Po1f ΔKU70Y. lipolytica Po1f 敲除KU70本研究Y.lipolytica PE-PEX10-1Y. lipolytica Po1f ΔKU70 失活PEX10 (删除of 1nt)本研究Y.lipolytica PE-PEX10-2Y. lipolytica Po1f ΔKU70 失活PEX10 (插入of 1nt)本研究Y.lipolytica PE-PEX10-3Y. lipolytica Po1f ΔKU70 失活PEX10 (置换of 1nt)本研究Y.lipolytica PE-EYD1-1Y. lipolytica Po1f ΔKU70 失活EYD1 (删除of 1nt)本研究
表3 本研究使用的引物
Table 3 Primers used in this study

引物 序列 (5'→3') Cas9H840A-FCGACTACGATGTGGACGCAATTGTGCCCCAGTCCTTCCCas9H840A-RTGCGTCCACATCGTAGTCGGAGN20-PEX10-FTCGATTCCGGGTCGGCGCAGTACAAGGAGGAGCTGGAGAN20-PEX10-RTAACTTGCTATTTCTAGCTCTAAAACTCTCCAGCTCCTCCTTGTACPEX10-seq-FATGTGGGGAAGTTCACATGCPEX10-seq-RTTATCTGATAGGCAACAAGTTCTGpNoCas9-FGCCTGGGGTGCCTAATGAGpNoCas9-RCTCATTAGGCACCCCAGGCCTCGAATTCACTGGCCGTCGpCRISPRyl-seq1-FGTTTTAGAGCTAGAAATAGCAAGTTApCRISPRyl-seq2-FCGCTAAGCTGCAGCTGTCTApCRISPRyl-seq2-RTGCGCCGACCCGGAATCGAgRNA-PE-PEX10-F1CGATTCCGGGTCGGCGCAGTACAAGGAGGAGCTGGAGAGTTTTAGAGCTAGAAATAGCAgRNA-PE-PEX10-R1GAGGAGCTGTAGACGGCGTCCAAGCACCGACTCGGTGCCACgRNA-PE-PEX10-F2ACGCCGTCTACAGCTCCTCCTTGTATTTTTTTTACGTCTAAGAAACCATTATTATCgRNA-PE-PEX10-R2TAACTAGATAGAACCGCGTCATGTGTA-CAAGGAGGAGCTGGTAGACGGCGTCCAAGCACCGACTCGGTGCCACTgRNA-PE-PEX10-R3ACTAGATAGAACCGCGTCATGTGTACAAG-GAGGAGCTGTAGACGGCGTCCAAGCACCGACTCGGTGCCACTgRNA-PE-PEX10-F4CGATTCCGGGTCGGCGCAGCCTGCCCCCAAGAAGCTCGTGTTTTAGAGCTAGAAATAGCAgRNA-PE-PEX10-R4AACTAGATAGAACCGCGTCATGTGTTGC-CCCCAAGAAGCCGTTGGACTCAATGGCACCGACTCGGTGCCACTpCRISPR-PE-PEX10-FACACATGACGCGGTTCTATCTAGT-TACGCGTTAAACCAACTAGAATTTTTTTTACGTCTAAGAAACCATTATTATCp-RT-FGCTGATGGTAGCGAATTTGAATCTCGAGCCGACCCCAAGAACas9-seq-RGTCGCCGCCCAGCTGAGACART-Cas9-FGTCTCAGCTGGGCGGCGACGGAGGTGGGGGAAGTTCCGGRT-seq-RTTCAAATTCGCTACCATCAGCEYD1-seq-FATGGTTTCTTCAGCCGCTACEYD1-seq-RAAGGCCCACTGGTTACCAA
1.2 主要试剂
氨苄青霉素(ampicillin,Amp),北京博奥拓达科技有限公司;PCR反应试剂2×Rapid Taq Master Mix、高保真PCR反应试剂2×KeyPo SE Master Mix (Dye Plus)、同源重组酶2×ClonExpress Mix、质粒小提试剂盒、胶回收/DNA纯化试剂盒,南京诺唯赞生物科技股份有限公司;酵母基因提取试剂盒,天根生化科技(北京)有限公司;PCR引物,由北京擎科生物科技股份有限公司与生工生物工程(上海)股份有限公司合成。
1.3 培养基
YPD培养基(g/L):葡萄糖20,蛋白胨20,酵母浸粉10,115 ℃高压蒸汽灭菌30 min。
LB培养基(g/L):胰蛋白胨10,NaCl 10,酵母浸粉5,用NaOH调节pH值至7.0,121 ℃高压蒸汽灭菌30 min。
亮氨酸营养缺陷型(dropout supplement-leucine SD-Leu)培养基(g/L):酵母氮碱基(yeast nitrogen bases,YNB,不含氨基酸,Difco)6.7,葡萄糖20,DO Supplement-Leu 0.7,pH值为6±0.2,115 ℃高压蒸汽灭菌30 min。
油酸筛选培养基(g/L):酵母氮碱基(YNB,不含氨基酸,Difco)6.7,油酸(oleic acid)3,Tween 40 2,DO Supplement-Leu 0.7,亮氨酸(leucine)0.1,pH值为6±0.2,115 ℃高压蒸汽灭菌30 min。
在以上培养基中添加2.1%(质量分数)琼脂即配制成对应的固体培养基。
100 mg/mL Amp母液:称取25 g的氨苄青霉素粉末,加去离子水至250 mL,完全溶解后过滤除菌、分装,-20 ℃保存。
含Amp的LB培养基:待LB培养基灭菌后冷至50 ℃左右加入Amp抗生素母液,至终质量浓度为80~100 μg/mL。
赤藓糖醇发酵培养基(g/L):葡萄糖330,酵母浸粉5,柠檬酸三铵5,硫酸镁0.25,磷酸二氢钾0.25,115 ℃高压蒸汽灭菌30 min。
1.4 质粒及菌株构建
pPE1、pPE2质粒的构建:使用限制性内切酶AvrⅡ切割pCRISPRyl将质粒线性化;以线性化的pCRISPRyl质粒为模板,Cas9H840A-F/Cas9H840A-R为引物,扩增获得Cas9H840A片段,并使用一步克隆试剂盒连接得到重组质粒pPE2。同时,以pCRISPRyl质粒为模板,以pNoCas9-F/pNoCas9-R为引物,扩增获得NoCas9的质粒骨架;以引物N20-PEX10-F/N20-PEX10-seq-R互为模板,扩增得到PEX10的gRNA片段;将质粒骨架与gRNA连接获得pPE1质粒。
pPE3、pPE4质粒构建:使用限制性内切酶Not1切割将pPE2质粒线性化,并与PEX10的gRNA连接得到质粒pPE3。以引物PEX10-seq-F/PEX10-seq-R互为模板,得到PEX10的pegRNA的片段,与pPE2线性化质粒连接,获得pPE4质粒。
pPE6质粒构建:以pCRISPRyl为模板,分别以p-RT-F/Cas9H840A-R、Cas9H840A-F/Cas9-seq-R为引物,扩增得到2个Cas9相关片段;以pCas-MLVRT-lacZ质粒为模板,以RT-Cas9-F/RT-seq-R为引物,扩增得到MLVRT片段经产物纯化回收后,与扩增获得的2个Cas9片段连接得到质粒pPE5。最后,使用限制性内切酶Not1切割pPE5质粒,获得的线性化质粒与PEX10的gRNA片段连接得到pPE6质粒。
pPE7-Cas9nRT-pegRNA1、pPE8-Cas9nRT-pegRNA2、pPE9-Cas9nRT-pegRNA3质粒的构建:以pCRISPRyl为模板,分别以gRNA-PE-PEX10-F1/gRNA-PE-PEX10-R1、gRNA-PE-PEX10-F2/gRNA-PE-PEX10-R2、gRNA-PE-PEX10-F2/gRNA-PE-PEX10-R3为引物,扩增得到3个PE-gRNA片段;以pPE5质粒为模板,分别以pCRISPRyl-PE-PEX10-F/pCRISPRyl-seq1-R和pCRISPRyl-seq2-F/pCRISPRyl-seq2-R为引物,扩增得到2段pCRISPRyl的骨架片段。产物纯化回收后,利用3个PE-gRNA片段分别与pCRISPRyl的骨架片段通过一步克隆试剂盒连接得到质粒pPE7-Cas9nRT-pegRNA1、pPE8-Cas9nRT-pegRNA2、pPE9-Cas9nRT-pegRNA3。
pPE10-Cas9nRT-pegRNA4质粒的构建:以pCRISPRyl为模板,以gRNA-PE-PEX10-F4/ gRNA-PE-PEX10-R4为引物,扩增得到PE-gRNA4片段;引物EYD1-seq-F/EYD1-seq-R互为模板,合成EYD1的pegRNA片段;产物纯化回收后,与2段pCRISPRyl的骨架片段通过一步克隆试剂盒连接得到质粒pPE10-Cas9nRT-pegRNA4。
1.5 实验方法
1.5.1 大肠杆菌感受态细胞的制备
挑取活化后的E.coil DH5α单克隆于LB液体培养基中过夜培养获得种子液,按照1‰接种量转接至100 mL LB液体培养基中,37 ℃、200 r/min条件下培养3~5 h,严格控制OD600值为0.35~0.4。菌液转移到50 mL无菌离心管中,冰浴10 min。于4 ℃、4 100 r/min条件下离心10 min,去上清液。然后将管倒置以除尽痕量培养液。30 mL预冷0.1 mol/L CaCl2-MgCl2溶液(80 mmol/L MgCl2、20 mmol/L CaCl2)重悬细胞。于4 ℃、4 100 r/min条件下离心10 min,弃上清液。2 mL预冷0.1 mol/L CaCl2重悬细胞,分装、保存至-80 ℃。
1.5.2 解脂耶氏酵母感受态细胞的制备
挑取解脂耶氏酵母单菌落于2.5 mL YPD液体培养基中在30 ℃,220 r/min摇床中培养18 h菌株活化;转接到50 mL YPD培养基(初始OD600值为0.1~0.15),30 ℃振荡培养4 h(至OD600值为0.4~0.6);25 ℃、4 000 r/min离心5 min,弃上清液收集菌体,用100 mmol/L乙酸锂(LiAc)溶液重悬,即为酵母感受态细胞,分装、保存至-80 ℃。
1.5.3 聚乙二醇(ethylene glycol,PEG)-LiAc转化法
将解脂耶氏酵母菌株在YPD平板上30 ℃培养24 h,在无菌离心管中加入1 mL无菌水,刮取细胞,吹吸重悬细胞。3 000×g离心5 min,弃上清液,用1 mL无菌水重悬清洗细胞,该步骤重复3次。离心,去上清液,沉淀即为感受态细胞。加入1 000 ng所需的质粒,混匀重悬的细胞沉淀及转化试剂,试剂如表4所示。39 ℃孵育60 min、室温3 000×g离心5 min,弃上清液;将细胞沉淀重悬于500 μL YPD培养基中,30 ℃,摇床孵育2 h。室温3 000×g离心5 min,弃上清液,在100 μL无菌水中重悬细胞并涂布于SD-Leu培养基上,置于30 ℃培养箱静置培养2 d。
表4 PEG-LiAC转化所用试剂
Table 4 Reagents for PEG-LiAC conversion
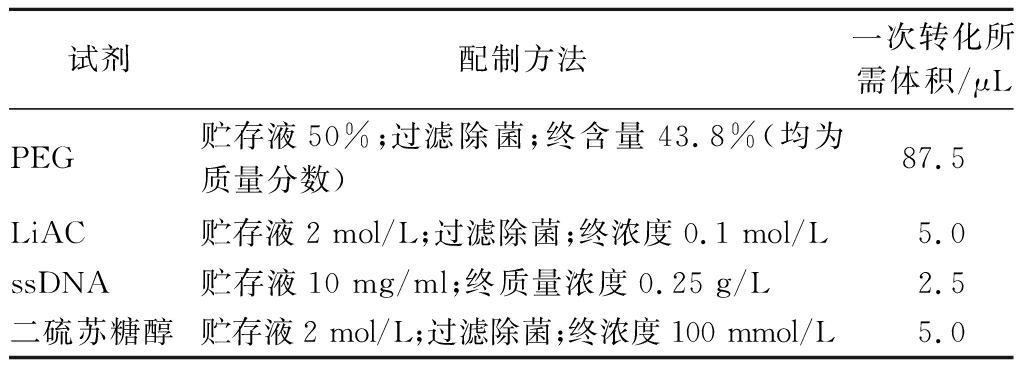
试剂配制方法一次转化所需体积/μLPEG贮存液50%;过滤除菌;终含量43.8%(均为质量分数)87.5LiAC贮存液2 mol/L;过滤除菌;终浓度0.1 mol/L5.0ssDNA贮存液10 mg/ml;终质量浓度0.25 g/L2.5二硫苏糖醇贮存液2 mol/L;过滤除菌;终浓度100 mmol/L5.0
1.5.4 PE系统编辑及效率计算
将转化PE质粒的阳性克隆挑至SD-Leu液体培养基中在30 ℃、220 r/min振荡培养2 d,划线至YPD固体培养基分离单克隆。单克隆在油酸筛选培养基上进行点植,若成功编辑的克隆则PEX10被敲除,该克隆无法利用长链脂肪酸代谢生长,则无法在油酸筛选培养基上生长。基因PEX10被敲除的比例即为PE系统的基因编辑效率。
1.5.5 发酵条件及代谢产物分析
单菌落酵母挑取至5 mL YPD液体培养基中,30 ℃、220 r/min振荡培养过夜,得到种子液;然后,按照2%接种量将种子液接入赤藓糖醇发酵培养基中,30 ℃、220 r/min振荡培养。同时,每3 d取1 mL发酵液离心,取上清液过滤后以HPLC法检测代谢物中葡萄糖、赤藓糖醇以及多元糖醇等成分的含量。
HPLC色谱条件,色谱仪:安捷伦Agilent 1260;检测器:RID-G1362A示差检测器;色谱柱:Carbomix Ca-Np;检测池温度:室温;分离柱温:80 ℃;流动相:超纯水;流速:0.7 mL/min;进样器:G1329A/B自动进样器;进样体积:20 μL。用高效液相色谱法对发酵代谢底物、产物进行检测分析。
2 结果与分析
2.1 解脂耶氏酵母先导编辑系统的建立
本研究在解脂耶氏酵母的CRISPRyl/Cas9系统的基础上改造了PEyl系统。通过对Cas9蛋白的840位组氨酸进行定点突变,获得了单链切割活性的切口酶Cas9H840A。然后,对来源于鼠白血病毒的反转录酶(Moloney murine leukaemia virus reverse transcriptase,M-MLV-RT)进行了适配解脂耶氏酵母的密码子优化并合成了基因M-MLV-RT。再以柔性接头(Gly-Gly-Gly-Ser-Ser)3将M-MLV-RT与Cas9H840A蛋白进行融合表达,获得了融合蛋白Cas9H840A-RT。pegRNA的改造则以sgRNA为基础,在构建质粒的过程中通过添加3′端的PBS和RTT获得。
CRISPR/Cas9H840A系统识别并切割基因组会引发NHEJ机制随机修复,导致基因片段丢失或移码突变,造成功能失活。为了探究Cas9H840A-RT和pegRNA改造是否会对在解脂耶氏酵母中CRISPR/Cas9H840A系统的识别和切割功能产生影响,本研究以PEX10基因为标记基因考察该系统的编辑效率。PEX10基因在解脂耶氏酵母中编码过氧化物酶体合成的相关蛋白,其功能失活会导致菌株对油酸的代谢功能受损,生长过程中呈现出油酸的营养缺陷性状。本研究对PEX10设计了gRNA和靶向单碱基切除的pegRNA与Cas9H840A和Cas9H840A-RT结合,分别构建质粒并转化酵母后将菌落在以油酸为唯一碳源的平板培养基上点植对照,以油酸营养缺陷表型为指标计算PEX10基因的失活概率,即CRISPR/Cas9H840A系统的编辑效率。结果如图1所示,在质粒仅含sgRNA(pPE1)或Cas9H840A(pPE2)的条件下,并不能触发CRISPR/Cas9H840A系统特异性切割并诱发移码突变,即编辑效率为0;当sgRNA和Cas9H840A同时存在时(pPE3)编辑效率为(12.5±4.2)%。与将pPE3分别替换为pegRNA(pPE4)(13.9±2.4)%和Cas9H840A-RT(pPE6)(13.9±4.8)%时,编辑效率相比无显著性差异。说明融合蛋白Cas9H840A-RT和pegRNA的改造并没有影响CRISPR系统的识别和切割功能。
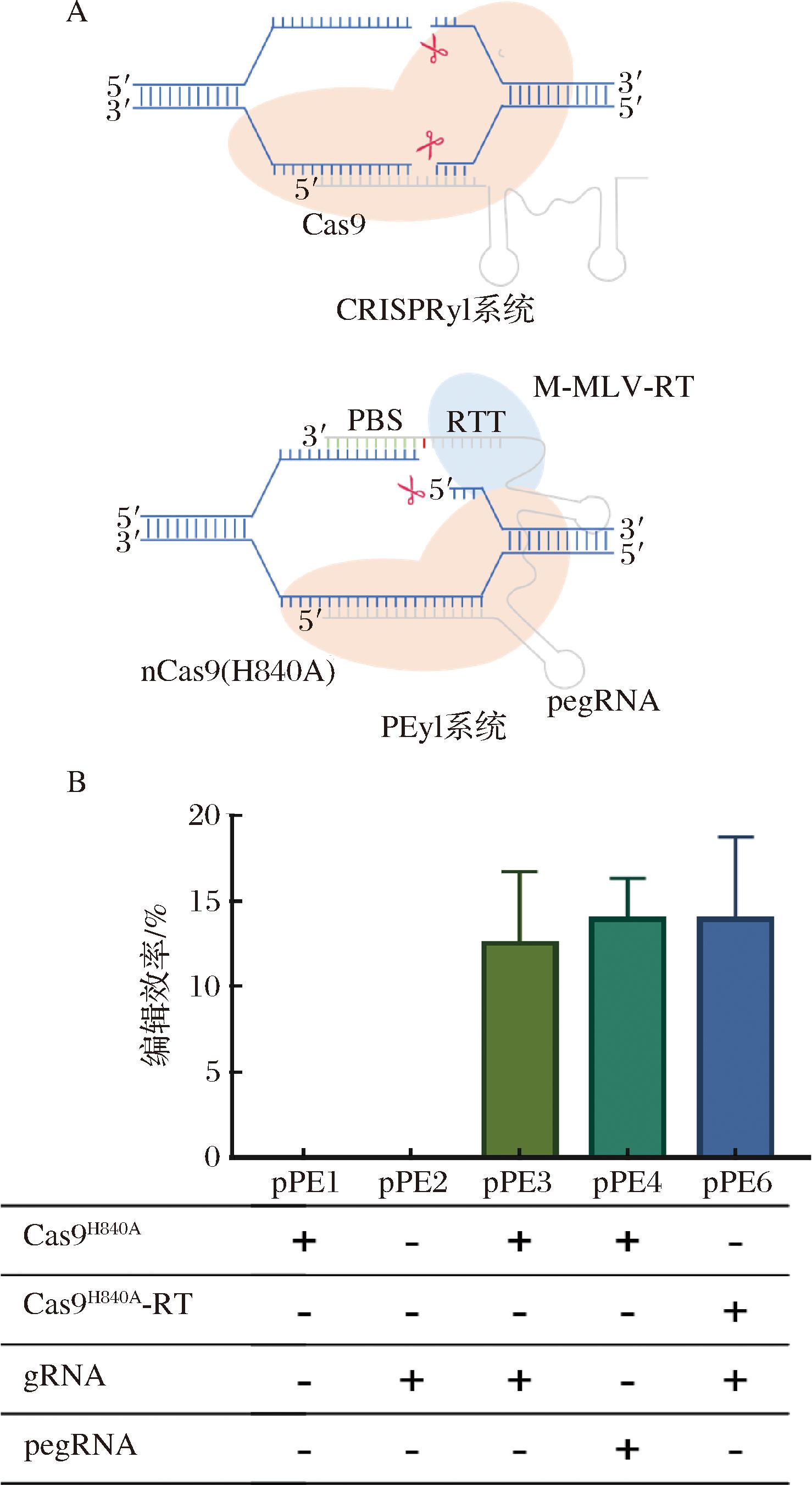
A-CRISPRyl系统与PEyl系统的作用机制;B-改造Cas9H840A-RT和pegRNA对CRISPR系统功能的影响
图1 先导编辑系统在Y.lipolytica中的建立
Fig.1 The establishment of the prime editing system in Y.lipolytica
注:正号(+)和负号(-)分别表示质粒中遗传元件的添加和不添加。
接着,本研究设计pegRNA引入单碱基删除突变,与Cas9H840A-RT相组合,构建质粒并分别在NHEJ机制完整的解脂耶氏酵母Po1f和NHEJ机制受损的Po1f ΔKU70中进行表达,比较PE系统使PEX10基因突变的编辑效率,并通过DNA测序分析突变类型(图2)。结果表明,2个菌株均成功对PEX10基因进行编辑并使基因失活,且Po1f中的总体失活效率略高,但由PE编辑系统引发单碱基删除的目标突变概率仅为(4.3±0.8)%,而由NHEJ引发的随机突变的概率为(11.5±1.3)%;而在Po1f ΔKU70菌株中,PE系统引入目标突变的概率为(10.7±2.1)%,而随机突变概率仅为(1±1.3)%。该结果表明,解脂耶氏酵母自身较强的NHEJ修复机制会对PE系统编辑产生干扰;而Po1f ΔKU70的突变菌株经测序均为预设的单碱基删除突变,说明PE系统成功地在解脂耶氏酵母中发挥了功能,且PE介导的突变修复不依赖于NHEJ机制,在NHEJ受损的菌株中编辑效果更好。
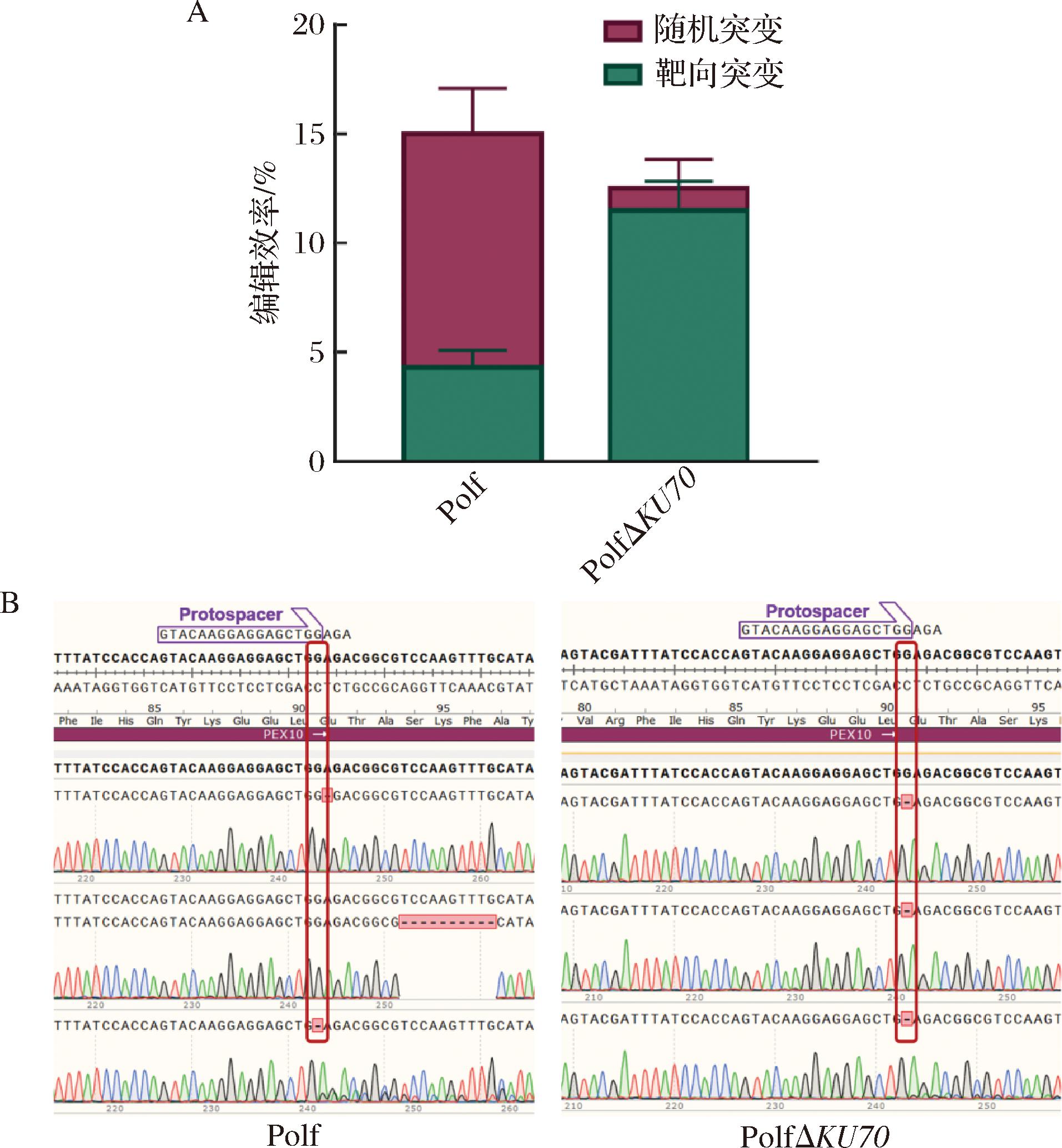
A-PEyl在NHEJ机制完整或受损的解脂耶氏酵母中的编辑效率;B-测序分析突变菌株的序列变化
图2 NHEJ机制对PEyl系统效率的影响
Fig.2 The effect of NHEJ mechanism on the efficiency of PEyl system
注:标红部分为pegRNA预设的单碱基删除突变。
2.2 PE系统的优化和表征
与被Cas9蛋白包裹的sgRNA不同,pegRNA的3′端延伸的PBS与RTT序列在细胞内呈现为裸露的单链RNA状态,极易受到核酸外切酶的识别和切割,因结构不完整而导致功能缺失[17],从而降低了PE系统的编辑效率。核酸“假结”适配体evopreQ1是最小的RNA结构序列之一[18],由一个“茎环”结构和短尾序列组成,且短尾中有部分碱基与茎环互补配对,形成二级结构,从而防止RNA被外切酶降解(图3)。本研究将evopreQ1与pegRNA的3′端连接以增加pegRNA的稳定性,结果如图3-B所示,与原始的pegRNA相比,PEyl系统的编辑效率由(12.5±2.5)%提升了1倍,达到(25.8±3.8)%。表明pegRNA的不稳定是限制PE系统的重要因素之一,采用pegRNA-evopreQ1增加RNA稳定性可以显著提升PE系统的单碱基编辑效率。
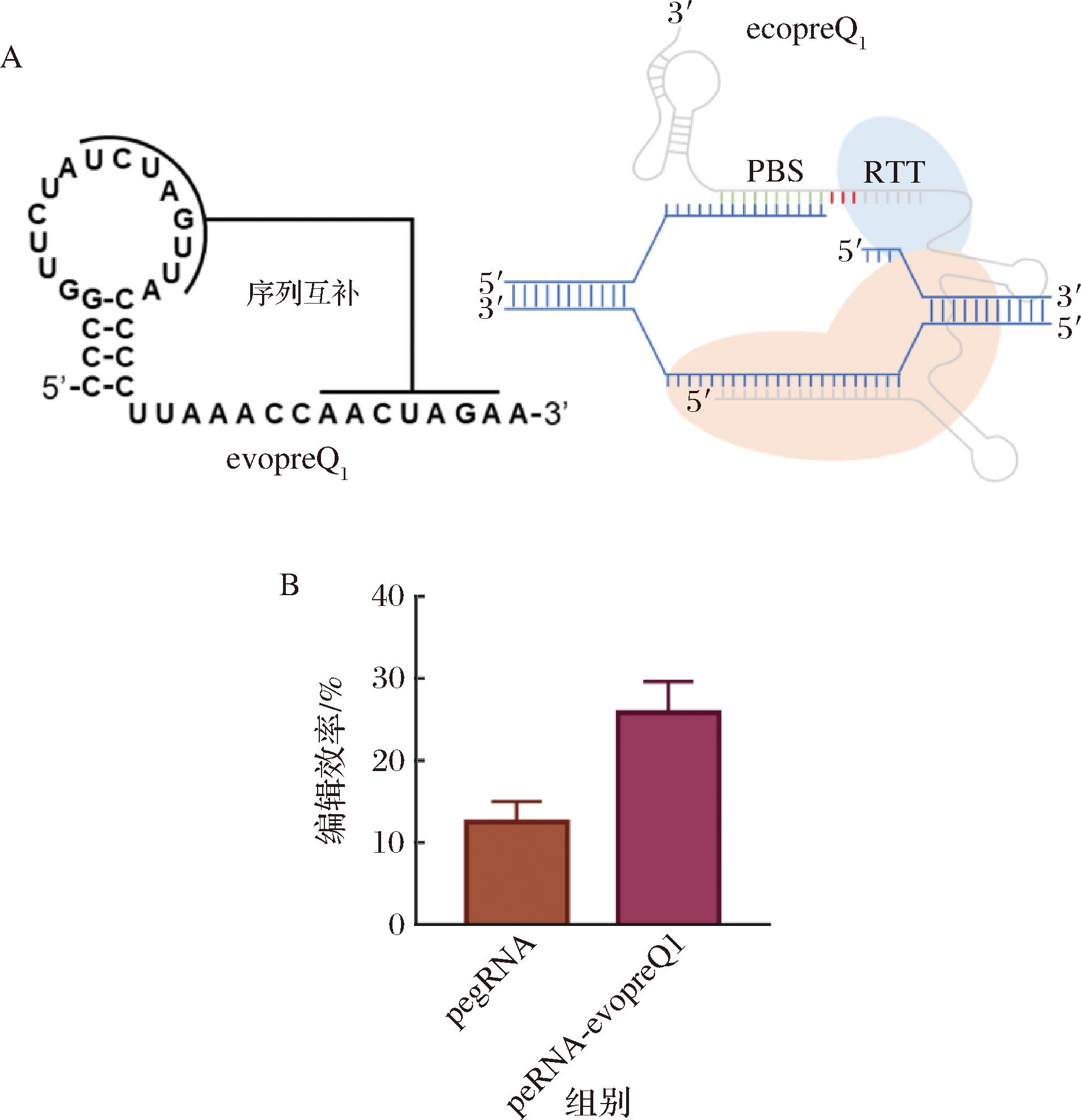
A-evopreQ1的序列和RNA二级结构;B-pegRNA添加evopreQ1对编辑效率的影响
图3 evopreQ1提升PEyl编辑效率
Fig.3 The improvement of evopreQ1 on PEyl editing efficiency
基因组的单碱基突变主要包括删除、插入和替换,为了探究PEyl系统应用于单碱基插入和替换的效果,本研究对PEX10基因分别设计了2种不同方案的pegRNA(图4),并分别构建了质粒转化解脂耶氏酵母Po1f ΔKU70,同时以PEX10基因的失活率比较PEyl系统对3种类型单碱基突变中的编辑效率。油酸营养缺陷平板的点植结果表明,PEyl系统对单碱基插入和替换的编辑效率分别为(13.3±1.4)%和(20±2.5)%。
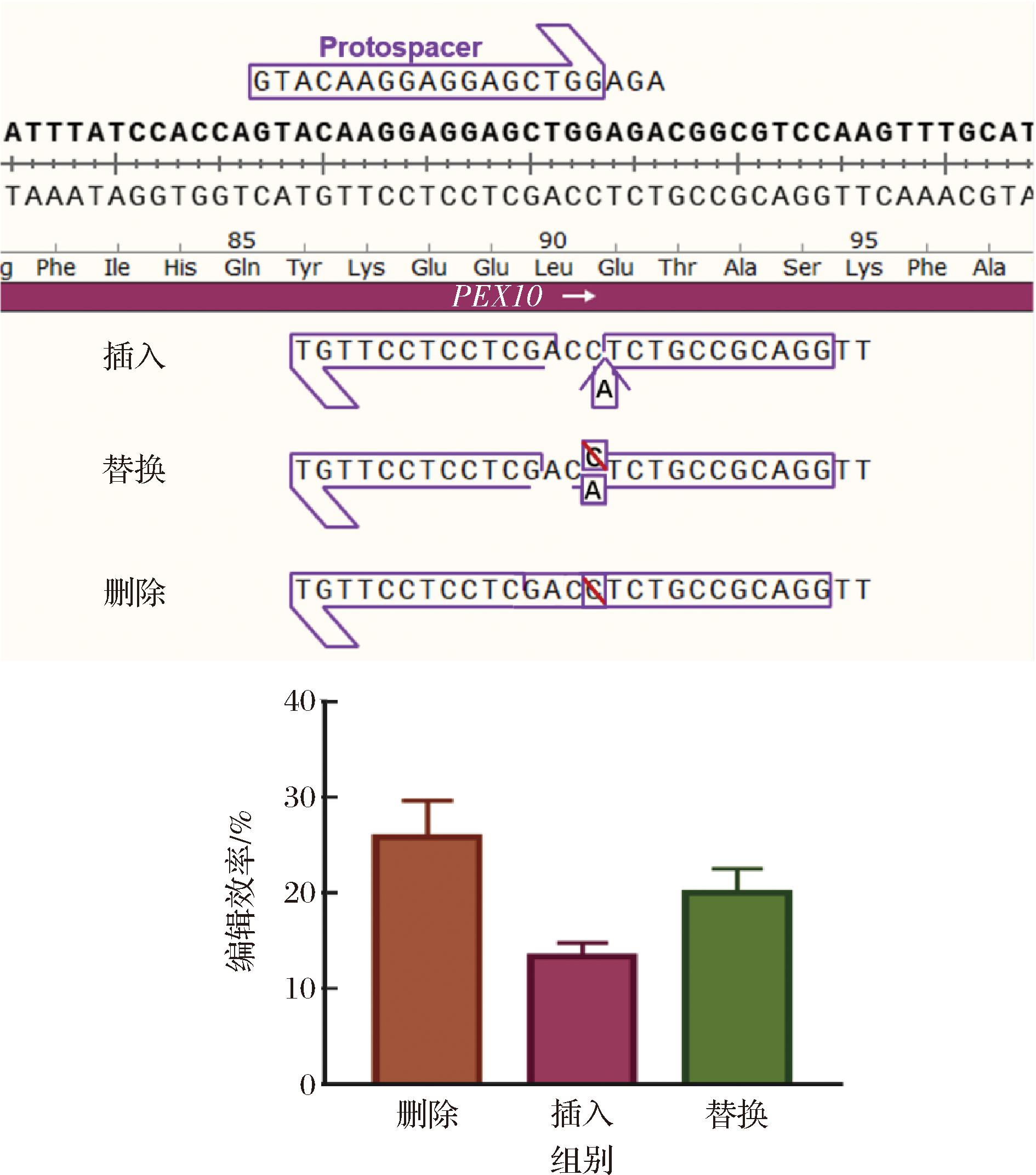
图4 PEyl系统应用于单碱基删除、替换和插入
Fig.4 The application of the PEyl system for single-base deletion, replacement and insertion.
2.3 PEyl系统失活EYD1提升赤藓糖醇产量
为了验证PEyl系统的基因编辑应用效果,本研究选择了解脂耶氏酵母的EYD1基因进行功能失活,提升赤藓糖醇的生物合成量。解脂耶氏酵母是目前以生物法工业生产赤藓糖醇的最优菌种,天然具备代谢合成赤藓糖醇的功能。葡萄糖进入细胞后,经过磷酸戊糖途径转化为赤藓糖,接着生成赤藓糖醇。EYD1基因编码赤藓糖醇脱氢酶,能够催化赤藓糖醇转化为赤藓酮糖进而进入三羧酸循环途径,导致赤藓糖醇的积累量降低。本研究将PEyl系统应用于EYD1基因编辑,选择解脂耶氏酵母Y-31 EYD1基因的高效PAM结合区,并设计了PBS和RTT将删除89位碱基,从而使EYD1基因产生移码突变,导致赤藓糖醇脱氢酶失活。并将获得的工程菌株Y-32于赤藓糖醇发酵培养基培养96 h后通过HPLC检测赤藓糖醇产量。结果如图5所示,与出发菌株相比,菌株Y-32的赤藓糖醇产量显著提升,由(9.29±0.17) g/L达到了(20.63±0.47) g/L。结果表明,PEyl系统可以应用于解脂耶氏酵母的高效编辑,在工程菌株的基因编辑和菌种改造中具有较大的应用潜力。
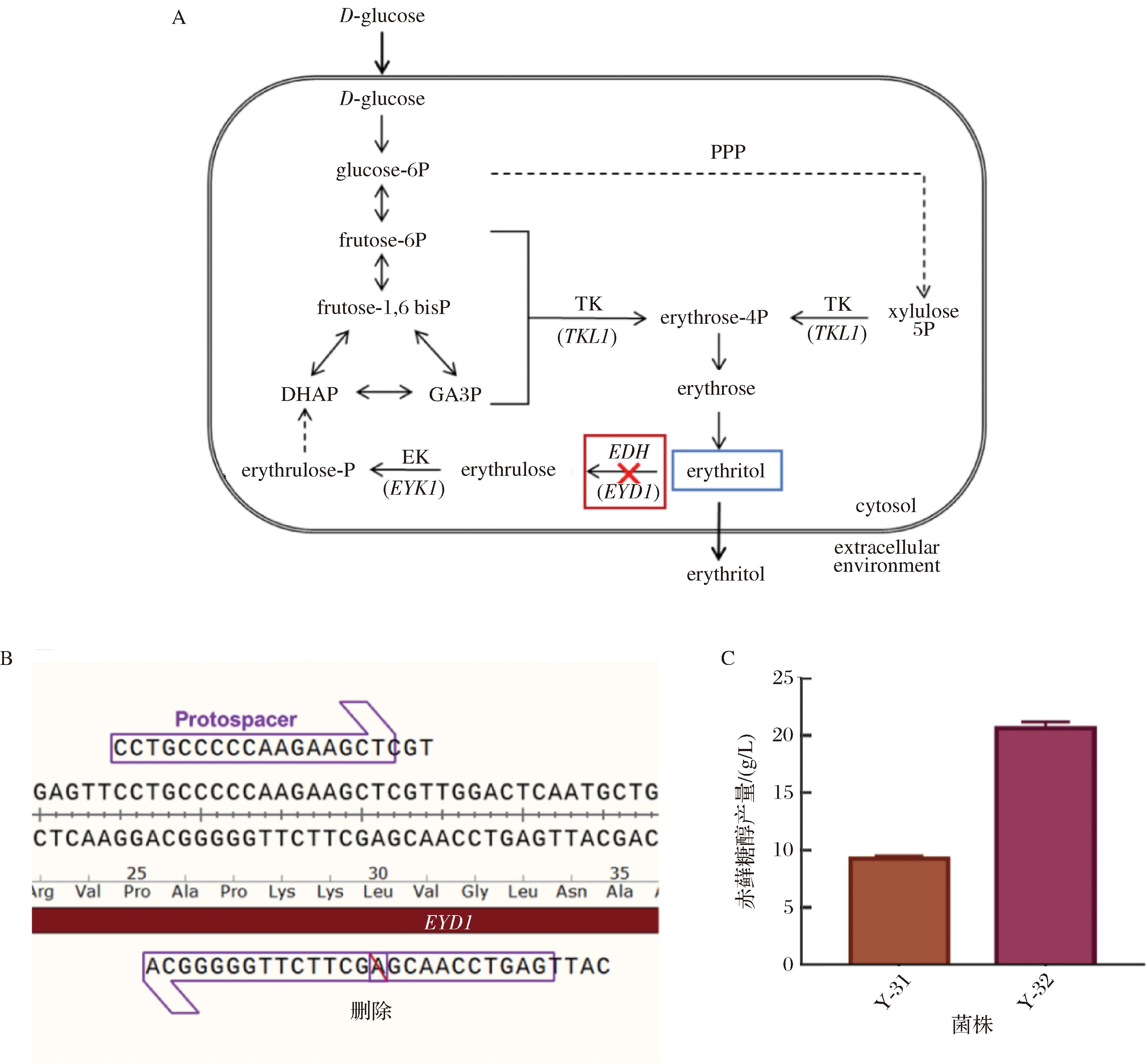
A-解脂耶氏酵母中赤藓糖醇代谢途径;B-EYD1单碱基删除导致移码突变失活;C-失活EYD1对赤藓糖醇产量的影响
图5 PEyl系统失活EYD1提升赤藓糖醇产量
Fig.5 Inactivation of EYD1 by the PEyl system for enhancing erythritol production
注:D-glucose:D-葡萄糖;glucose -6P:6-磷酸葡萄糖;frucose-6P:6-磷酸果糖;frucose-1,6 bisP :1,6-二磷酸果糖;erythrulose:赤藓酮糖;erythrulose-P:磷酸赤藓酮糖;erythrose:赤藓糖;erythrose-4P:4-磷酸赤藓糖;xylulose-5P:5-磷酸木酮糖;DHAP:dihydroxyacetone phosphate,磷酸二羟丙酮;GA3P:glyceraldehyde 3-phosphate,3-磷酸甘油醛;GK:glycerol kinase,甘油激酶;TK:transketolase,转酮醇酶;EDH:赤藓糖醇脱氢酶;erythritol dehydrogenase,EK:erythritol kinase,赤藓糖激酶;erythritol:赤藓糖醇。
3 讨论与结论
解脂耶氏酵母作为极具工业应用优势的非模式菌株,在合成生物学和代谢工程领域发挥了重要的作用[19-20],其高效、多样的基因编辑工具技术的开发一直是研究热点[21]。本研究首次在解脂耶氏酵母中构建了PEyl系统,并利用evopreQ1增加pegRNA的稳定性,成功实现了单碱基层面的删除、插入和替换,能够对解脂耶氏酵母的基因组进行精准编辑。
先导编辑主要包括2个过程:一是通过Cas9H840A蛋白和反转录酶的作用下对基因组进行突变,产生一条含有目的突变的单链DNA(称为编辑链),二是细胞通过自身DNA修复机制,将编辑链和对应的非编辑链之间的错配进行修复[16]。然而,第二步的自发修复及影响其效率的原理尚不清晰,研究发现可能与错配修复[22]、NHEJ修复[23]等机制有关。本研究通过在NHEJ机制完整和受损的2种解脂耶氏酵母中比较PEyl系统的编辑效率并测序分析序列突变,发现在解脂耶氏酵母中,NHEJ的机制受损并不会影响PEyl系统的功能。相反,会因其随机修复的方式干扰基因编辑结果,带来更多假阳性和不确定性,因此更推荐在NHEJ的机制受损的解脂耶氏酵母中应用PEyl系统。此外,本研究对PE系统进行删除、插入和替换3种单碱基编辑方式的效率进行了比较。研究结果表明,与删除和替换相比,插入的效率更低,这与相关研究报道有一定出入[24],推测可能与基因位点、插入长度和核苷酸类型等多种因素有关。本研究还将PEyl系统应用于产赤藓糖醇的解脂耶氏酵母菌株改造中,并成功通过单碱基删除实现了对EYD1基因的功能失活,该结果展现了PEyl系统在精准基因编辑中的应用潜力。
目前,解脂耶氏酵母的基因操作工具依然稀缺,本研究首次将先导编辑系统在解脂耶氏酵母中进行了初步开发和应用,开发了新的基因编辑方法促进菌种的编辑和改造,对拓宽解脂耶氏酵母的遗传操作工具包,进一步开发基因组编辑方法具有重要的意义。
[1] 李运清. 解脂耶氏酵母研究进展[J]. 济宁医学院学报, 2015, 38(1): 8-13.LI Y Q. Advances in studies of Yarrowia lipolytica[J]. Journal of Jining Medical University, 2015, 38(1): 8-13.
[2] LIU H H, JI X J, HUANG H. Biotechnological applications of Yarrowia lipolytica: Past, present and future[J]. Biotechnology Advances, 2015, 33(8):1522-1546.
[3] PAPANIKOLAOU S, AGGELIS G. Biotechnological valorization of biodiesel derived glycerol waste through production of single cell oil and citric acid by Yarrowia lipolytica[J]. Lipid Technology, 2009, 21(4):83-87.
[4] TOMASZEWSKA L, ![]() A, G
A, G ADKOWSKI W. Production of erythritol and mannitol by Yarrowia lipolytica yeast in media containing glycerol[J]. Journal of Industrial Microbiology &Biotechnology, 2012, 39(9):1333-1343.
ADKOWSKI W. Production of erythritol and mannitol by Yarrowia lipolytica yeast in media containing glycerol[J]. Journal of Industrial Microbiology &Biotechnology, 2012, 39(9):1333-1343.
[5] RAFIQUL I S M, MIMI SAKINAH A M. Processes for the production of xylitol: A review[J]. Food Reviews International, 2013, 29(2):127-156.
[6] 赵鹤云, 黄瑛, 杨江科. 解脂耶氏酵母表达系统研究进展[J]. 生物加工过程, 2008, 6(3): 10-16.ZHAO H Y, HUANG Y, YANG J K. Review of Yarrowia lipolytica expression system[J]. Chinese Journal of Bioprocess Engineering, 2008, 6(3): 10-16.
[7] PAL S, MONDAL A K, SAHOO D K. Molecular strategies for enhancing microbial production of xylitol[J]. Process Biochemistry, 2016, 51(7):809-819.
[8] ![]() E, LEDESMA-AMARO R, LARROUDE M, et al. Golden Gate Assembly system dedicated to complex pathway manipulation in Yarrowia lipolytica[J]. Microbial Biotechnology, 2017, 10(2):450-455.
E, LEDESMA-AMARO R, LARROUDE M, et al. Golden Gate Assembly system dedicated to complex pathway manipulation in Yarrowia lipolytica[J]. Microbial Biotechnology, 2017, 10(2):450-455.
[9] LARROUDE M, PARK Y K, SOUDIER P, et al. A modular Golden Gate toolkit for Yarrowia lipolytica synthetic biology[J]. Microbial Biotechnology, 2019, 12(6):1249-1259.
[10] WONG L, ENGEL J, JIN E Q, et al. YaliBricks, a versatile genetic toolkit for streamlined and rapid pathway engineering in Yarrowia lipolytica[J]. Metabolic Engineering Communications, 2017, 5:68-77.
[11] ZHANG T L, YU H W, YE L D. Metabolic engineering of Yarrowia lipolytica for terpenoid production: Tools and strategies[J]. ACS Synthetic Biology, 2023, 12(3):639-656.
[12] JI Q C, MAI J, DING Y, et al. Improving the homologous recombination efficiency of Yarrowia lipolytica by grafting heterologous component from Saccharomyces cerevisiae[J]. Metabolic Engineering Communications, 2020, 11: e00152.
[13] DARVISHI F, ARIANA M, MARELLA E R, et al. Advances in synthetic biology of oleaginous yeast Yarrowia lipolytica for producing non-native chemicals[J]. Applied Microbiology and Biotechnology, 2018, 102(14):5925-5938.
[14] ANZALONE A V, RANDOLPH P B, DAVIS J R, et al. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576(7785):149-157.
[15] YANG L, YANG B, CHEN J. One prime for all editing[J]. Cell, 2019, 179(7): 1448-1450.
[16] SCHWARTZ C M, HUSSAIN M S, BLENNER M, et al. Synthetic RNA polymerase III promoters facilitate high-efficiency CRISPR-Cas9-mediated genome editing in Yarrowia lipolytica[J]. ACS Synthetic Biology, 2016, 5(4):356-359.
[17] HOUSELEY J, TOLLERVEY D. The many pathways of RNA degradation[J]. Cell, 2009, 136(4):763-776.
[18] ROTH A, WINKLER W C, REGULSKI E E, et al. A riboswitch selective for the queuosine precursor preQ1 contains an unusually small aptamer domain[J]. Nature Structural &Molecular Biology, 2007, 14(4):308-317.
[19] ABBASI A R, LIU J L, WANG Z, et al. Recent advances in producing sugar alcohols and functional sugars by engineering Yarrowia lipolytica[J]. Frontiers in Bioengineering and Biotechnology, 2021, 9:648382.
[20] HU M C, GE J Y, JIANG Y R, et al. Advances and perspectives in genetic expression and operation for the oleaginous yeast Yarrowia lipolytica[J]. Synthetic and Systems Biotechnology, 2024, 9(4):618-626.
[21] MA J B, GU Y, MARSAFARI M, et al. Synthetic biology, systems biology, and metabolic engineering of Yarrowia lipolytica toward a sustainable biorefinery platform[J]. Journal of Industrial Microbiology &Biotechnology, 2020, 47(9): 1-18.
[22] DA SILVA J F, OLIVEIRA G P, ARASA-VERGE E A, et al. Prime editing efficiency and fidelity are enhanced in the absence of mismatch repair[J]. Nature Communications, 2022, 13:760.
[23] CHEN P J, LIU D R. Prime editing for precise and highly versatile genome manipulation[J]. Nature Reviews Genetics, 2023, 24(3):161-177.
[24] TONG Y J, JØRGENSEN T S, WHITFORD C M, et al. A versatile genetic engineering toolkit for E. coli based on CRISPR-prime editing[J]. Nature Communications, 2021, 12:5206.