食品安全是全球公共卫生问题之一,它不仅关乎消费者的身体健康和生命安全,还影响国家社会经济的稳定和发展。根据世界卫生组织(World Health Organization, WHO)的数据,每年约有6亿人因食用受污染的食物而患病,其中42万人因食品安全问题死亡。在低收入和中等收入国家,食品安全问题导致的生产力损失和医疗费用高达1 100亿美元[1]。食品安全的风险主要来源于物理、化学和生物污染三大类。目前,常规的食品安全检测技术包括色谱技术、光谱技术和生物技术等。但这些检测方法仍具有一定的局限性,通常需要在专业实验室内由专业人员操作,限制了其在现场检测中的应用[2]。因此,建立简单、快速、便携、小型化和低成本的快检方法尤为重要。常见的快检技术包括免疫分析技术、核酸扩增技术、比色试纸条技术和生物传感器技术,这些技术能够有效缩短检测时间,提高检测特异性和灵敏度,并减少对大型仪器的依赖,它们正逐渐成为食品安全检测的主流技术[3]。特别是生物传感器技术,因其能够实现对目标物的快速鉴定和分析,具有较高的灵敏度和特异性,已广泛应用于临床诊断、环境监测和食品检测等领域。
成簇规律间隔短回文重复序列/相关蛋白(clustered regularly interspaced short palindromic repeats/ CRISPR-associated proteins system, CRISPR/Cas)是细菌和古细菌的适应性免疫系统。能够特异性识别并切割外源序列。近年来,研究者基于CRISPR/Cas系统独特的结合和切割活性,通过与多种检测技术结合开发了CRISPR/Cas生物传感器,这类生物传感器不仅能够检测核酸类靶标,还能通过与功能性DNA结合实现对非核酸类靶标的检测,显著拓宽了CRISPR/Cas系统的应用范围[4]。其中,CRISPR/Cas12a系统在CRISPR RNA(crRNA)的引导下可以特异性识别并切割含有前间隔序列邻近基序(protospacer adjacent motif, PAM)的双链DNA(double-stranded DNA, dsDNA),当Cas12a靶向同源序列时会发生构象变化,并产生对单链DNA(single-stranded DNA, ssDNA)的非特异切割活性,这种切割活性使Cas12a在核酸检测中发挥了巨大作用[5]。基于这一特性,研究者将Cas12a引入生物传感器中,实现了超灵敏的目标物检测,使CRISPR/Cas12a系统成为生物传感器领域中极具前景的检测工具[6]。本文概述了CRISPR/Cas系统的分类和作用机制,介绍了CRISPR/Cas12a生物传感器的工作原理,总结了其在食品安全检测领域的应用进展,旨在为研究人员提供该领域最新的研究动态,并为食品检测方法的开发提供新的思路。
1 CRISPR/Cas生物传感器
1.1 CRISPR/Cas系统作用机制及分类
1.1.1 CRISPR/Cas系统作用机制
CRISPR/Cas系统由CRISPR阵列和Cas蛋白2部分组成。前者用于储存和记录外源核酸入侵的信息,后者负责具体实施免疫防御功能。CRISPR阵列由一系列短的重复序列和间隔序列交替排列构成,其两侧则是编码不同Cas蛋白的基因(图1)[7]。
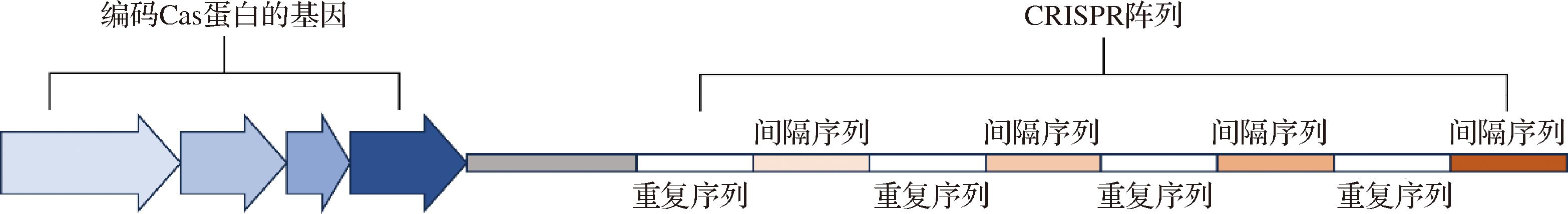
图1 CRISPR/Cas系统组成
Fig.1 CRISPR/Cas system composition
CRISPR/Cas免疫反应包括3个阶段:适应、表达/加工和干扰。在适应阶段,Cas1、Cas2等蛋白将首次入侵的外源基因序列切割并整合到CRISPR阵列中,从而形成免疫记忆。在表达阶段,当外源基因再次入侵时,细菌会转录CRISPR序列,并由核糖核酸酶或Cas蛋白将其加工成成熟的crRNA。最后,在干扰阶段,crRNA与相应的Cas蛋白形成核糖核蛋白复合物,引导Cas蛋白对目标外源基因进行特异性切割,从而阻止其增殖和传播(图2)[8]。
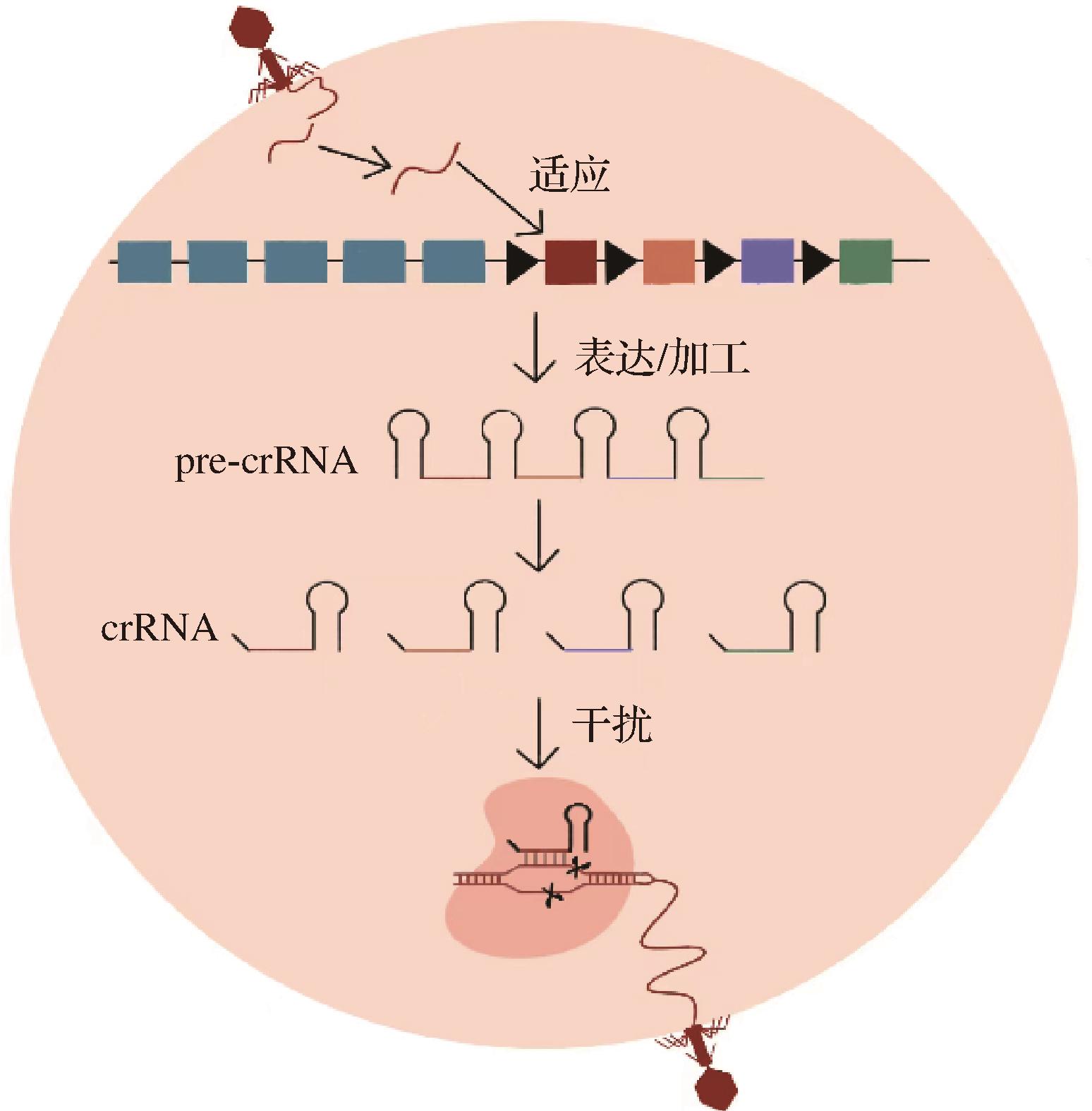
图2 CRISPR/Cas系统作用机制
Fig.2 The mechanism of action of CRISPR/Cas system
1.1.2 CRISPR/Cas系统分类
根据最近新的分类系统,CRISPR/Cas系统分为两大类:Ⅰ类和Ⅱ类。Ⅰ类CRISPR/Cas系统是由多种Cas蛋白组成的多亚基效应复合物构成。Ⅱ类CRISPR/Cas系统则由单个Cas蛋白组成的效应复合物构成,是基因组编辑应用的主要工具[9]。在Ⅱ类CRISPR/Cas系统中,Cas9、Cas12、Cas13和Cas14蛋白常用于核酸检测技术,这些蛋白的功能特征和干扰机制详见表1和图3。
表1 Ⅱ类CRISPR效应蛋白
Table 1 Class Ⅱ CRISPR/Cas system effector proteins
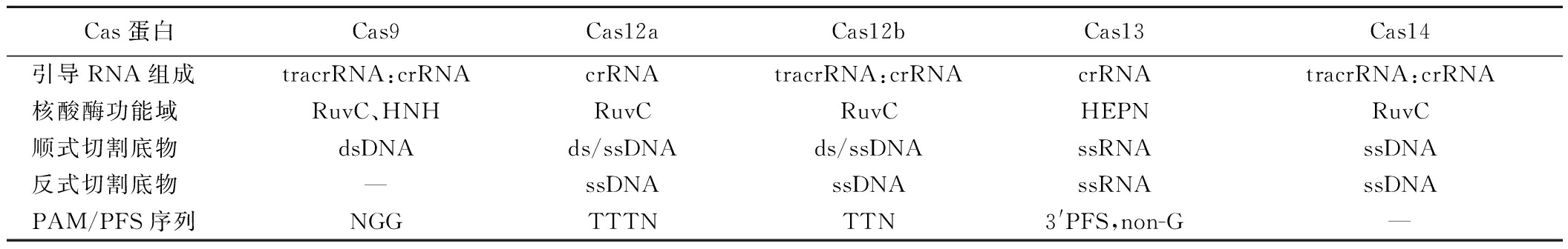
Cas蛋白Cas9Cas12aCas12bCas13Cas14引导RNA组成tracrRNA:crRNAcrRNAtracrRNA:crRNAcrRNAtracrRNA:crRNA核酸酶功能域RuvC、HNHRuvCRuvCHEPNRuvC顺式切割底物dsDNAds/ssDNAds/ssDNAssRNAssDNA反式切割底物—ssDNAssDNAssRNAssDNAPAM/PFS序列NGGTTTNTTN3'PFS,non-G—
注:—表示无数据(下同)。
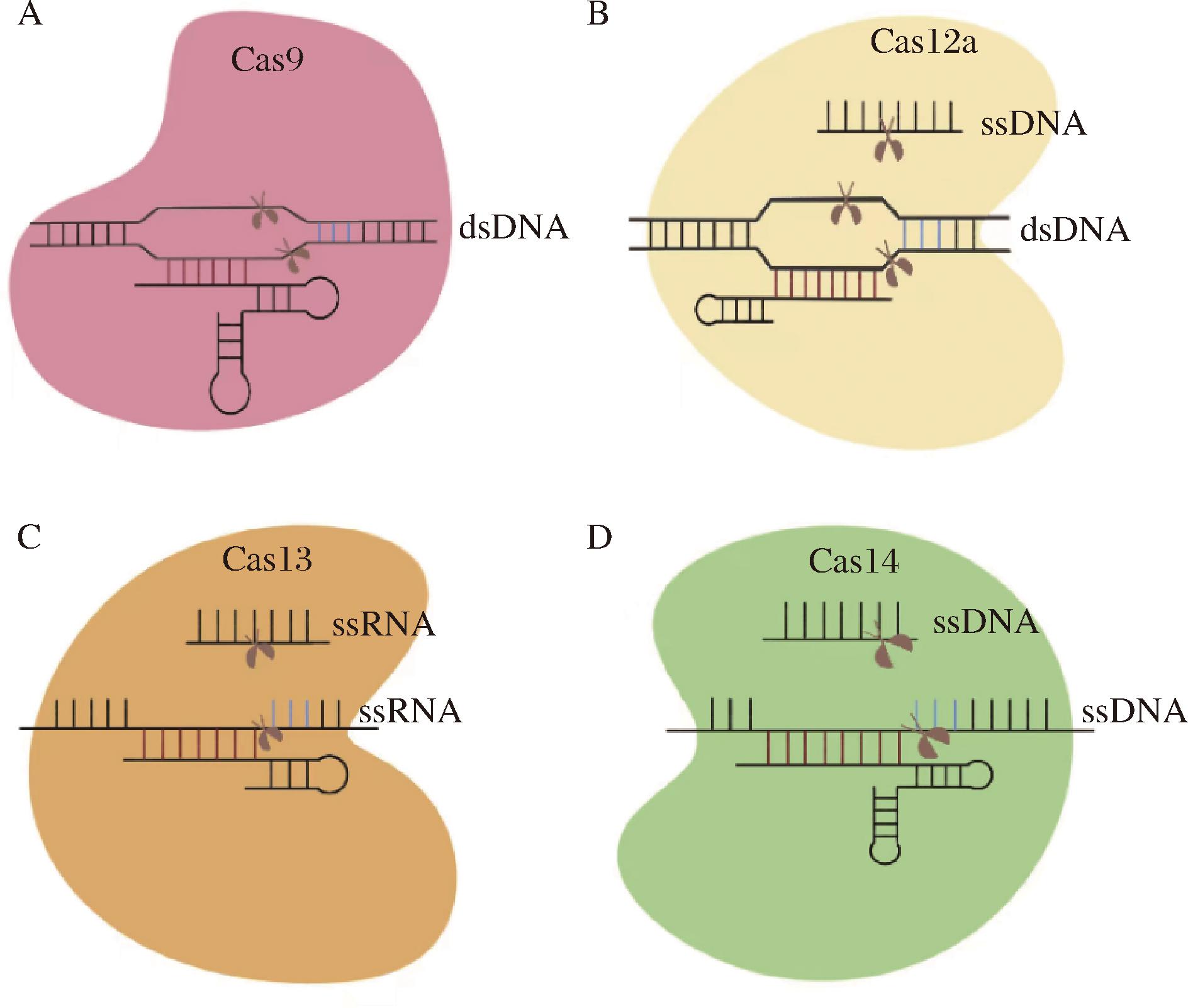
A-Cos9;B-Cas12a;C-Cas13;D-Cas14
图3 Cas9、Cas12a、Cas13和Cas14的干扰机制
Fig.3 Interference mechanisms of Cas9、Cas12a、Cas13 and Cas14
Cas9和Cas12a是Ⅱ类CRISPR/Cas系统的标志性蛋白。Cas9识别底物为dsDNA,当crRNA与反式激活CRISPR RNA(tracrRNA)通过局部碱基配对形成单链引导RNA(single guide RNA, sgRNA)并与Cas9结合后,能够引导Cas9识别并切割靶序列。Cas12a同样识别dsDNA,但其在与crRNA形成复合物后不仅能特异性切割靶序列,还会产生反式切割活性。因此,在检测中常利用这种反式切割活性来切割体系中带有荧光标记的ssDNA,通过检测荧光信号的变化间接反映底物中是否含有靶序列。Cas13和Cas14的工作原理与Cas12类似,但它们识别的底物不同。Cas13的识别底物及非特异性切割底物均为单链RNA(single-stranded RNA, ssRNA),而Cas14的识别底物及非特异性切割底物均为ssDNA[10]。
1.2 基于CRISPR/Cas系统的生物传感器
生物传感器是一种由生物受体(如酶、抗体、DNA等)、传感器(用于将观察到的信号转换为有用的输出信号)以及放大器(负责放大和处理最终信号)组成的分析设备[11]。与传统的生物传感器相比,基于CRISPR/Cas系统的生物传感器具有更高的特异性,且操作简便、快速。CRISPR/Cas系统作为一种可编程的基因编辑工具,能够对特定的核酸序列进行精确切割。例如,Cas12a在识别目标DNA后会激活其反式切割活性,从而非特异性地切割周围的ssDNA。这一特性使得CRISPR/Cas系统成为一种集成了生物识别与信号转导功能的高效检测工具。通过设计简化的接口和信号输出方法,即可实现对目标物的检测。随着生物技术的不断发展,基于CRISPR/Cas12a系统的生物传感器已成功应用于食源性致病菌、转基因靶标、真菌毒素、重金属离子和农药残留等的检测(表2)。
表2 CRISPR/Cas生物传感器与基于不同生物识别元件传感器的比较
Table 2 Comparison of CRISPR/Cas biosensors and biosensors based on different biometric elements
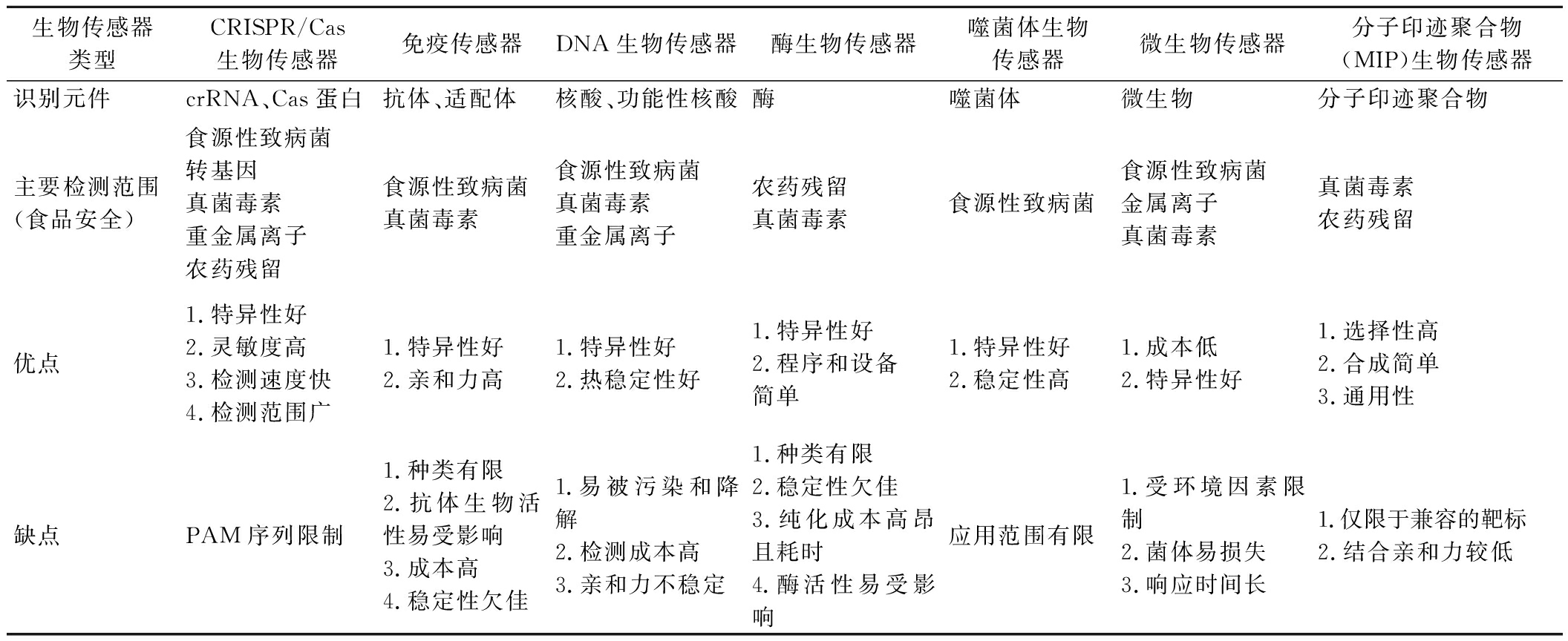
生物传感器类型CRISPR/Cas生物传感器免疫传感器DNA生物传感器酶生物传感器噬菌体生物传感器微生物传感器分子印迹聚合物(MIP)生物传感器识别元件crRNA、Cas蛋白抗体、适配体核酸、功能性核酸酶噬菌体微生物分子印迹聚合物主要检测范围(食品安全)食源性致病菌转基因真菌毒素重金属离子农药残留食源性致病菌真菌毒素食源性致病菌真菌毒素重金属离子农药残留真菌毒素食源性致病菌食源性致病菌金属离子真菌毒素真菌毒素农药残留优点1.特异性好2.灵敏度高3.检测速度快4.检测范围广1.特异性好2.亲和力高1.特异性好2.热稳定性好1.特异性好2.程序和设备简单1.特异性好2.稳定性高1.成本低2.特异性好1.选择性高2.合成简单3.通用性缺点PAM序列限制1.种类有限2.抗体生物活性易受影响3.成本高4.稳定性欠佳1.易被污染和降解2.检测成本高3.亲和力不稳定1.种类有限2.稳定性欠佳3.纯化成本高昂且耗时4.酶活性易受影响应用范围有限1.受环境因素限制2.菌体易损失3.响应时间长1.仅限于兼容的靶标2.结合亲和力较低
2 CRISPR/Cas12a生物传感器
CRISPR/Cas12a生物传感器主要分成3个部分:信号放大(signal amplification)、信号识别(signal transducing)以及信号输出(signal reporting)。
2.1 信号放大
虽然Cas12a对靶序列的亲和性较高,理论上检测灵敏度高,但是Cas12a在完成靶序列切割后,其自身的反式切割活性并不能长久维持,因此在检测低浓度的靶序列过程中,非特异性切割的荧光探针信号不强,所以在使用Cas12a进行检测前,需要对靶序列进行一定程度的富集扩增。
2.1.1 聚合酶链式反应
聚合酶链式反应(polymerase chain reaction, PCR)是一种在食品检测领域使用非常广泛的技术,它最大特点是能将微量的目的基因大幅增加。与其他常见检测方法相比,它具有较高的特异性和灵敏度。2018年,LI等[12]开发出了一种名为HOLMES的检测方法,该方法使用PCR技术扩增目标序列,扩增产物会激活Cas12a的反式切割活性并对带有荧光基团和淬灭基团的ssDNA进行切割,从而产生荧光信号。该方法利用PCR技术的高扩增效率和Cas12a的高特异性,提高了HOLMES方法的灵敏度和准确性。
2.1.2 等温扩增技术
PCR技术通常应用于实验室检测中,为了满足现场检验的需求,操作简单、快捷、灵敏度高的等温扩增技术逐渐被广泛应用。环介导等温扩增(loop-mediated isothermal amplification, LAMP)技术是针对靶序列的6个位置设计4~6条特异性引物,利用链置换DNA聚合酶(Bst DNA polymerase)的活性进行扩增,扩增后首先形成哑铃状结构,哑铃状结构作为起始结构引发循环扩增[13]。该方法具有较高的特异性和灵敏度,但对引物的设计要求比较高,且容易产生假阳性。重组酶聚合酶扩增(recombinase polymerase amplification, RPA)技术是利用重组酶、单链DNA结合蛋白(single-stranded DNA-binding protein, SSB)和链置换DNA聚合酶进行扩增。其中重组酶引导引物结合dsDNA,链置换DNA聚合酶引发扩增,SSB稳定置换的单链。该方法引物设计简单,在37 ℃下就能发生反应,正逐渐成为现场检测的主流方法。其他等温扩增技术大多也是利用常温条件下能够扩增的聚合酶,同时使用有置换功能的重组酶或者形成特殊的结构来实现对靶序列的指数扩增。表3中总结了常见的等温扩增技术,包括链置换扩增(strand displacement amplification, SDA)技术、滚环扩增(rolling circle amplification, RCA)技术、指数式扩增(exponential amplification, EXPAR)技术、重组酶辅助扩增(recombinase aided amplification, RAA)技术、杂交链式反应(hybridization chain reaction, HCR)、催化发卡组装反应(catalytic hairpin assembly, CHA)。
表3 常见等温扩增技术的比较
Table 3 Comparison of common isothermal amplification techniques
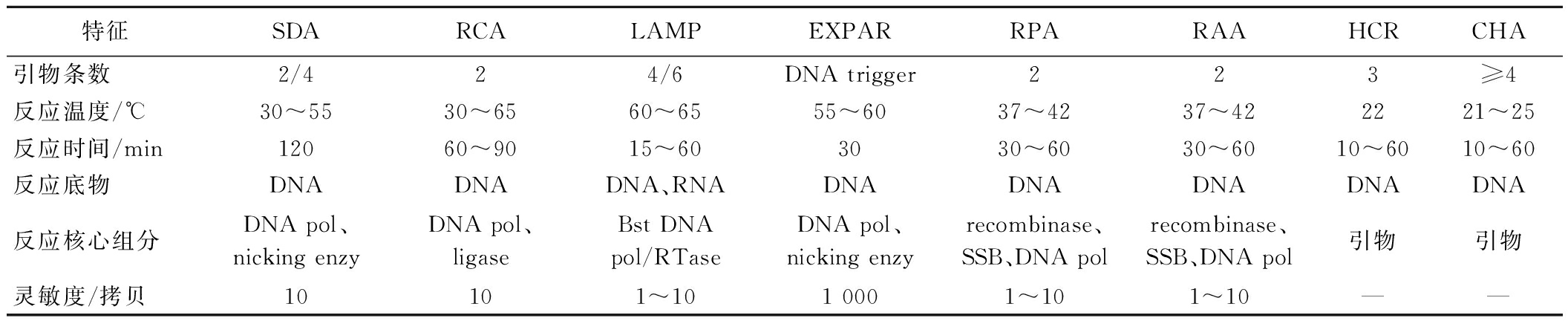
特征SDARCALAMPEXPARRPARAAHCRCHA引物条数2/424/6DNA trigger223≥4反应温度/℃30~5530~6560~6555~6037~4237~422221~25反应时间/min12060~9015~603030~6030~6010~6010~60 反应底物DNADNADNA、RNADNADNADNADNADNA反应核心组分DNA pol、nicking enzyDNA pol、ligaseBst DNA pol/RTaseDNA pol、nicking enzyrecombinase、SSB、DNA polrecombinase、SSB、DNA pol引物引物灵敏度/拷贝10101~101 0001~101~10——
表4 CRISPR/Cas12a生物传感器检测食源性致病菌
Table 4 Detection of foodborne pathogens by CRISPR/Cas12a biosensor

食源性致病菌信号放大信号输出创新检出限/线性范围参考文献Sta、SalRPA荧光信号—1.50(Sta)、24.9(Sal)CFU/mL[25]Sal、E.coli、V.P、L.M、StaRPA比色信号利用AuNPs聚集和分散的状态观察颜色变化100(E.coli)、104(Sal)、102(V.P)、103(Sta)、101(L.M) CFU/mL[26]S.FRPA荧光信号引入LFA,为现场检测提供可能基因组DNA 8.3拷贝/μL[18]B.C、L.M、Sta、P.A、Sal、C.S、V.PRAA荧光信号微流控技术;多重检测小于500 CFU/mL[19]L.MLAMP荧光信号采用四乙烯五胺修饰的磁性纳米颗粒将检测灵敏度提高10倍100 CFU/mL[20]Sal—电化学信号利用纳米复合材料来降低电极的背景信号并提高检测灵敏度160 CFU/mL[21]Sta、L.MERA、bHCR光热信号双等温扩增;银纳米颗粒使DNA金属化,从而检测到光热信号;定量检测1 CFU/mL,100~108 CFU/mL[22]SA、MRSARPA荧光多重检测;利用人工智能技术,实现对结果的快速读取18(SA)、20(MRSA)CFU/mL[23]Sal—全息图像利用人工智能深度学习并辅助处理图像;定量检测;区分活菌与非活菌状态72 CFU/mL[24]
注:Sta、SA,金黄色葡萄球菌;MRSA,抗药性金黄色葡萄球菌;Sal,沙门氏菌;E.coli,大肠杆菌;V.P,副溶血性弧菌;L.M,单核细胞增生李斯特菌;S.F,志贺氏菌;B.C,蜡样芽胞杆菌;P.A,铜绿假单胞菌;C.S,克罗诺杆菌;ERA(enzymatic recombinase amplification),酶促重组等温扩增。
表5 CRISPR/Cas12a生物传感器检测转基因生物
Table 5 Detection of genetically modified organisms by CRISPR/Cas12a biosensor

转基因序列/靶标信号放大信号输出创新检出限/线性范围参考文献p-CaMV35S、t-NOSERA荧光信号一步法检测,避免气溶胶污染10拷贝/反应;0.1% GMO[27]p-CaMV35S、t-NOSRPALFA40 min内完成,无需检测设备1~10拷贝;0.01 ng/μL基因组DNA[32]CP4-EPSPS、Cry1Ab/AcRPALFA多重检测;LFA用不同颜色的微球抗体标记10拷贝/μL[28]华农1号RPA比色信号可视化检测,消除了对实验室条件和专业仪器的依赖20拷贝[33]p-CaMV35S、t-NOSRPA比色信号双重检测;利用人工智能,实现对结果的自动读取检测1 amol/L,1 amol/L~100 fmol/L(p-CaMV35S);1 amol/L~1 pmol/L (t-NOS)[29]p-CaMV35SRCA荧光信号、LFA可利用智能手机读取结果1 fmol/L(荧光信号)、50 fmol/L(LFA)[30]p-CaMV35S—荧光信号可检测至少16 nt核酸片段;无预扩增;3D数字芯片;相对定量 —[31]
表6 CRISPR/Cas12a生物传感器检测真菌毒素
Table 6 Detection of fungal toxins by CRISPR/Cas12a biosensor

真菌毒素信号放大信号输出创新检出限/线性范围参考文献AFB1RCA荧光信号、LFA一步法检测0.016 ng/mL(荧光信号),0.408 ng/mL(LFA)[38]AFB1RCA荧光信号RCA和双酶催化的信号放大5.2 pg/mL,50 pg/mL~100 ng/mL[39]OTAExo Ⅲ电化学信号核酸外切酶Ⅲ(Exo Ⅲ)辅助信号放大0.74 fg/mL[34]ZENMg2+依赖性Dnazyme辅助循环裂解扩增电化学信号NH2-MnO2/Pd@AuNBs纳米复合材料可增强电化学信号6.27×10-6 ng/mL,10-5~10 ng/mL[40]AFB1—荧光信号无需预扩增0.15 ng/ml;0.15~78 ng/ml[41]AFM1—比色信号利用MnO2纳米酶的氧化活性氧化显色底物2.1 ng/L,6~160 ng/L[35]OTA—电化学信号微流控芯片;Pd@PCN-222纳米酶可以催化H2O2产生电化学信号1.21 pg/mL[36]AFB1—比色信号Fe-N-C Sazymes具有过氧化物酶模拟活性,可作为TMB显色催化剂1.5×10-7 ng/μL,10-6~1 ng/μL[42]AFB1、OTA、ZEN、FB1、T-2、DON—荧光信号利用微流控芯片实现多重检测;智能手机检测荧光信号fg/mL级别,10-5~101 ng/mL[37]
注:AFB1,黄曲霉毒素B1;ZEN,玉米赤霉烯酮;FB1,伏马毒素;DON,呕吐毒素。
表7 CRISPR/Cas12a生物传感器检测重金属
Table 7 Detection of heavy metals by CRISPR/Cas12a biosensor

重金属信号放大信号输出创新检出限/线性范围参考文献Pb2+SDA电化学信号合成ZrO2/CeO2纳米棒提高了电化学表面活性0.02 pmol/L[43]Hg2+PCR荧光信号利用智能手机实现荧光信号的可视化检测10 pmol/L[45]Hg2+TMSD血糖仪电极上修饰含有蔗糖酶的探针,蔗糖酶可催化蔗糖转为葡萄糖,通过血糖仪进行定量检测40 fmol/L,100 fmol/L~10 nmol/L[46]Pb2+—比色信号MnO2NRs纳米棒具有氧化酶活性,氧化TMB产生蓝色产物0.54 nmol/L,0.8~2500 nmol/L[47]Pb2+—荧光信号df-DNAzyme探针可作为Cas12a系统的激活剂,也可识别Pb2+1 pg/L,1 pg/L~10 μg/L[50]Pb2+—荧光信号ncCDzy的加入有助于增强DNAzyme与Pb2+的结合能力及其催化效率1.025 nmol/L,10~800 nmol/L[48]Pb2+—荧光信号定量检测;设计CDs@ZIF-8复合纳米材料,可以作为内参信号18 pmol/L[51]Hg2+、Cd2+、Pb2+、Mn2+CHA荧光信号构建逻辑门系统智能检测重金属离子pmol/L级别[49]
表8 CRISPR/Cas12a生物传感器检测农药残留
Table 8 Detection of pesticide residues by CRISPR/Cas12a biosensor
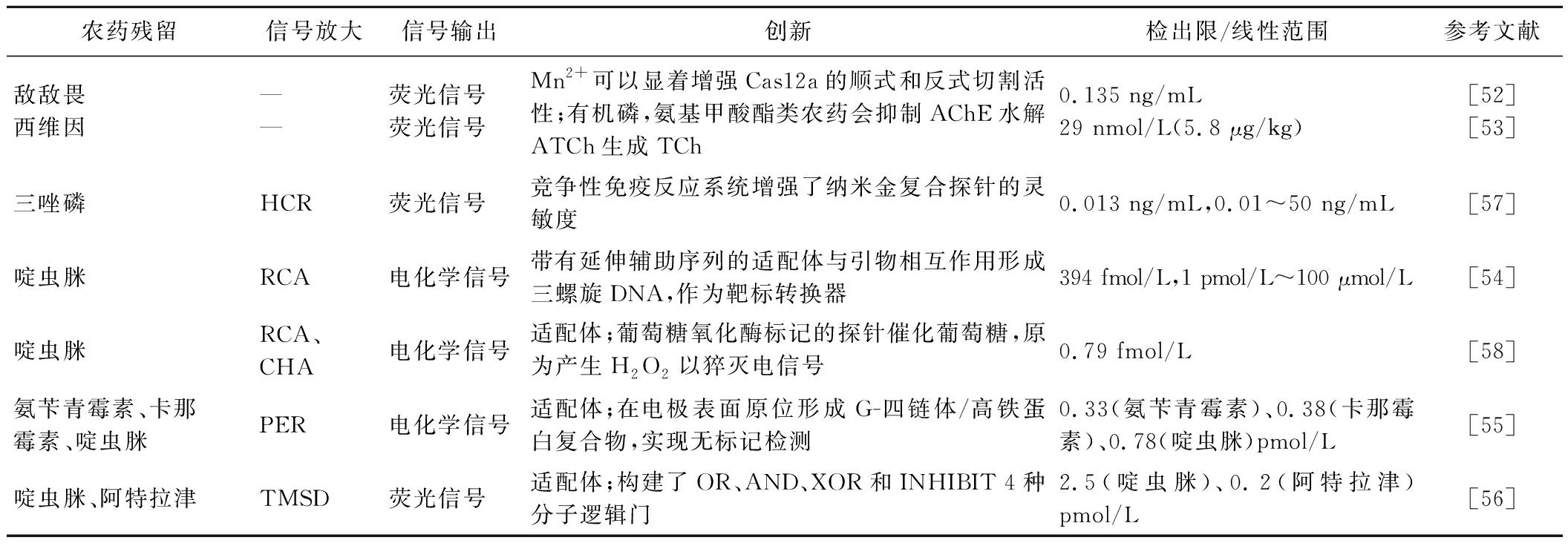
农药残留信号放大信号输出创新检出限/线性范围参考文献敌敌畏西维因——荧光信号 荧光信号 Mn2+可以显着增强Cas12a的顺式和反式切割活性;有机磷,氨基甲酸酯类农药会抑制AChE水解ATCh生成TCh0.135 ng/mL29 nmol/L(5.8 μg/kg)[52][53]三唑磷HCR荧光信号 竞争性免疫反应系统增强了纳米金复合探针的灵敏度0.013 ng/mL,0.01~50 ng/mL[57]啶虫脒RCA电化学信号带有延伸辅助序列的适配体与引物相互作用形成三螺旋DNA,作为靶标转换器394 fmol/L,1 pmol/L~100 μmol/L[54]啶虫脒RCA、CHA电化学信号适配体;葡萄糖氧化酶标记的探针催化葡萄糖,原为产生H2O2以猝灭电信号0.79 fmol/L[58]氨苄青霉素、卡那霉素、啶虫脒PER电化学信号适配体;在电极表面原位形成G-四链体/高铁蛋白复合物,实现无标记检测0.33(氨苄青霉素)、0.38(卡那霉素)、0.78(啶虫脒)pmol/L [55]啶虫脒、阿特拉津TMSD荧光信号 适配体;构建了OR、AND、XOR和INHIBIT 4种分子逻辑门2.5(啶虫脒)、0.2(阿特拉津)pmol/L[56]
2.2 信号输出
基于CRISPR/Cas12a系统的生物传感器常用的信号输出方式包括荧光信号、比色信号和电化学信号。
2.2.1 荧光信号
荧光信号具有高选择性、稳定性和灵敏度等优点,被认为是经典的信号输出方式。大多数CRISPR/Cas12a荧光生物传感器是利用荧光共振能量转移方式实现荧光信号的输出。将携带有荧光基团和淬灭基团的ssDNA作为报告分子,利用Cas12a的反式切割活性,造成报告分子断裂,荧光基团和淬灭基团发生空间分离,从而产生荧光信号。荧光纳米材料辅助信号输出也是荧光输出的方式之一。其中,金纳米颗粒(gold nanoparticles,AuNPs)具有独特的理化特性,已成为各种分析方法中常用的信号放大标记物。AuNPs附近的激发态荧光分子与AuNPs局域表面产生等离子体共振,会造成荧光发射强度显著增加[14]。
2.2.2 比色信号
基于比色信号的生物传感器基本原理是由分析物引起溶液颜色发生变化。它的优点是可以通过肉眼观察颜色的变化从而对被测物进行初步判断,同时操作简单,不需要复杂的仪器,适用于快速判读结果。在CRISPR/Cas12a生物传感器中常用的比色信号有两类,一类是利用AuNPs表面等离子体共振作用,观察AuNPs在聚集和分散状态下呈现的不同颜色变化来确认检测信号[15]。第二类则是利用酶催化或者化学反应使显色底物发生颜色变化,该方法是将ssDNA偶联到一些底物(酶)上并固定到特定基质中,当Cas12a发挥反式切割活性后,底物(酶)被释放到反应溶液中,进而催化显色底物发生颜色变化[16]。
2.2.3 电化学信号
电化学信号主要是根据反应体系中的电化学信号变化来确定是否检测到目标分子,这种电化学信号的变化一般是当探针与目标分子结合时,会发生数量或构象变化,从而使电化学信号发生改变,同时这些改变一般是由Cas12a发挥切割作用后引发[17]。
3 CRISPR/Cas12a生物传感器在食品检测领域的应用
随着CRISPR/Cas12a生物传感技术的发展,该技术已成功实现了针对核酸与非核酸靶标的检测,并逐步拓展至食品安全领域,用于识别食源性致病菌、转基因生物、真菌毒素、重金属离子及其他污染物,从而在维护食品安全方面发挥了关键作用。
3.1 食源性致病菌
食源性致病菌可导致人类出现腹泻等症状,严重时甚至危及生命。传统菌落平板计数法被视为检测食源性致病菌的“金标准”,然而该方法检测周期较长,难以实现高通量筛查和现场检测。因此,为满足快速检测食源性病原菌的需求,需开发具有高特异性、准确度和灵敏度的检测方法。
将CRISPR/Cas12a生物传感器与多种等温扩增方法相结合,能够实现对核酸分子的超灵敏检测。此外,为适应现场快速检测的需求,研究者们进一步整合了侧流层析(lateral flow assay, LFA)及微流控芯片技术,构建了一种便携、高效的CRISPR/Cas12a生物传感平台。XU等[18]建立了RPA-CRISPR/Cas12a-LFA方法,用于志贺氏菌的检测。XING等[19]开发了一种基于RAA-CRISPR/Cas12a的手动驱动微流控荧光生物传感器,能够同时检测7种食源性致病菌。通过手动操作单向控制阀,确保RAA产物顺利转移至CRISPR/Cas12a反应室,同时减少了交叉污染的风险。为了进一步增强CRISPR/Cas12a生物传感器的检测性能,研究者们引入了纳米技术作为辅助手段,有效提升了检测的灵敏度。例如,LEE等[20]采用四乙烯五胺修饰的磁性纳米颗粒与LAMP-CRISPR/Cas12a生物传感器联用,使得单核细胞增生李斯特菌的检测灵敏度提高了10倍。DUAN等[21]则利用CG@MXene纳米复合材料,减少了E-CRISPR生物传感器电极的背景噪声。同时,为了满足定量检测的需求,研究者还探索了基于CRISPR/Cas12a生物传感器的定量检测方法。GU等[22]借助bHCR反应和银纳米颗粒介导的DNA金属化技术,开发了一款便携式的、定量检测金黄色葡萄球菌和单核细胞增生李斯特菌的CRISPR/Cas12a光热生物传感器。此外,随着人工智能技术的发展,AI技术与CRISPR/Cas12a生物传感器的结合不仅提高了检测效率,还实现了精准、灵敏和快速的目标检测。SUN等[23]设计了一种具备多通道功能的便携式等温荧光放大装置,该装置集成了医疗物联网与基于深度学习的数据处理系统,利用YOLOv8模型优化和训练数据集,实现了批量样本检测结果的准确识别,支持远程控制和数据上传。该装置可在不同通道内执行RPA扩增和CRISPR/Cas12a信号识别,可用于金黄色葡萄球菌和抗药性金黄色葡萄球菌的双重检测。ZHOU等[24]提出了一种深度学习辅助的数字单粒子计数生物传感平台,该平台采用无透镜全息显微镜捕捉微球探针的全息图像,并运用基于YOLOv7算法的图像处理技术,以高通量和高分辨率的方式识别微球探针,利用Cas12a的反式切割活性,成功实现了定量检测。
3.2 转基因生物
转基因生物是将目的基因导入到受体生物的基因组中,有效提高农作物的产量。尽管转基因生物产生了许多好处,但它对生态及人类健康的安全问题仍存在争议。因此许多国家针对转基因生物及相关制品进行管控,并制定标识阈值制度。目前PCR技术作为转基因检测的“金标准”,通常适用于实验室检测,对现场检测应用性较差。因此开发用于现场检测的方法将会成为未来研究趋势。
在基于CRISPR/Cas12a生物传感器检测目标物时,通常利用PCR或等温扩增技术来实现信号放大,随后通过专业的荧光检测设备实现信号输出。然而,这种方法存在气溶胶污染和检测场景的限制。因此,为了提高检测特异性,减少气溶胶污染,LIANG等[27]开发了一步法ERA-CRISPR方法,用于检测p-CaMV35S 和t-NOS。该方法采用DSD模型优化ERA扩增和Cas12a的反应体系,确保Cas12a的活性既不会过早触发也不会过度抑制。此外,研究人员通过引入试纸条或可视化检测平台,提高了CRISPR/Cas12a生物传感器在现场检测中的适用性。WANG等[28]基于RPA-CRISPR/Cas12a-LFA策略,开发了一种多色侧向层析试纸条,该试纸条可实现对转基因元件CP4-EPSPS和Cry1Ab/Ac的双重检测。随着智能设备和人工智能等新兴技术的发展,研究人员将其与CRISPR/Cas12a生物传感器深度融合,旨在提高检测效率,实现精准和快速的检测。LI等[29]建立了一个智能Multi-RPA-CRISPR/Cas12a-G4芯片检测平台。该平台利用双重RPA技术同时扩增p-CaMV35S和t-NOS,通过将扩增产物引入带有不同crRNA的检测孔内实现Cas12a的反式切割。通过将YoloV5深度学习算法集成到智能手机APP中,可以实现对结果的自动读取检测。LIU等[30]基于RCA扩增和CRISPR/Cas12a系统建立了一种检测p-CaMV35S的可视化检测平台。该方法通过智能手机内置的图像处理应用,将样本图像的荧光强度转化为数字信息,并根据不同浓度绘制线性范围,从而实现对目标物的定性和定量分析。为了简化检测步骤,提高检测效率,研究人员开发了无预扩增的检测方法,不仅减少了检测时间,还降低了检测成本。PENG等[31]建立了一个无需预扩增的CRISPR/Cas12a荧光数字检测平台,用于检测p-CaMV35S。该方法在不同序列区域设计了多个crRNA,当DNA片段与任意crRNA匹配时,就会激活Cas12a的反式切割活性。该方法将CRISPR反应体系均匀分布至含有20 000个检测孔的QuantStudio 3D数字芯片中,通过检测和计数芯片上阳性孔的数量来实现对p-CaMV35S的相对定量。该方法显示阳性点百分比与靶核酸浓度之间具有良好的线性关系,证明了其潜在的定量能力。未来的研究可以进一步提高灵敏度,以实现对目标物的绝对定量。
3.3 真菌毒素
真菌毒素是霉菌在污染粮油食品后产生的次生代谢产物,这些真菌毒素可通过食物链对人类和其他动物造成危害。目前我国关于真菌毒素的检测方法大多以色谱技术为主,但该技术由于检测步骤繁琐和仪器昂贵等因素不适用于现场检测。因此,开发便捷、灵敏度高、特异性好的真菌毒素检测方法具有很大的发展前景。
基于CRISPR/Cas12a生物传感器检测真菌毒素这类非核酸靶标时,通常需要借助功能性DNA(如适配体和DNAzyme)的辅助,将非核酸靶标信号转化为核酸靶标信号。因此,研究人员开发了一系列基于不同功能性DNA辅助的CRISPR/Cas12a生物传感器,用于检测真菌毒素。WU等[34]将核酸外切酶Ⅲ(exonuclease Ⅲ, Exo Ⅲ)辅助信号放大与CRISPR/Cas12a相结合,开发了一种检测赭曲霉毒素A(ochratoxin A, OTA)的CRISPR/Cas12a电化学适配体传感器。该方法已成功应用于加标大米和花生样品中OTA的测定。此外,为了简化检测步骤,研究人员开发了无预扩增的检测方法,并引入纳米酶等新兴技术以提高真菌毒素的检测性能。MASOOMEH等[35]利用CRISPR/Cas12a的反式切割活性和“花状”MnO2纳米酶的氧化活性,建立了一种用于检测黄曲霉毒素M1(aflatoxin M1, AFM1)的比色适配体传感器。WU等[36]开发了一种基于CRISPR/Cas12a的电化学生物传感器,用于检测OTA。其中,Pd@PCN-222纳米酶的类过氧化物酶催化活性能够催化H2O2还原为H2O,并产生显著的还原峰值电流,从而提高了生物传感器的检测灵敏度。
此外,还有研究人员借助微流控芯片等技术,提高了CRISPR/Cas12a生物传感器的检测效率。XIANG等[37]利用功能性DNA和CRISPR/Cas12a系统,开发了一种能够同时检测6种真菌毒素的超灵敏CRISPR/Cas12a微流控生物传感器。该传感器中,样品溶液进入微流控芯片内,采用矿物油密封反应室,从而避免了交叉污染。
3.4 重金属
由于重金属离子的半衰期长,当它通过食物链进入人体后,会在人体骨骼和脂肪组织中积累,导致关键营养素缺失,并造成中枢神经系统缺陷等疾病,严重危害人类身体健康。因此,快速、高效、灵敏地检测水和食品中的重金属离子,对于保证食品安全具有重要意义。
在CRISPR/Cas12a生物传感器检测重金属离子时,常利用脱氧核糖核酶(DNAzyme)辅助检测。DNAzyme是一种在金属离子协同存在情况下具有催化功能的DNA序列,对金属离子具有极高的选择性。通过成熟的体外筛选技术,可以获得特异性结合特定重金属离子的DNAzyme。YUE等[43]利用GR-5 DNAzyme、SDA反应和CRISPR/Cas12a,设计了一种电化学生物传感器,用于检测Pb2+。同时,有研究发现,Hg2+能够选择性地与DNA双链中的胸腺嘧啶-胸腺嘧啶(T-T)碱基对结合,形成T-Hg2+-T结构[44]。基于这一特性,研究人员开发了基于T-Hg2+-T结构的CRISPR/Cas12a生物传感器。KONG等[45]利用T-Hg2+-T结构,建立了一种CRISPR/Cas12a荧光生物传感器,用于Hg2+的检测。当Hg2+存在时,会形成T-Hg2+-T结构并引发PCR扩增,通过扩增产物激活Cas12a的反式切割活性,同时利用智能手机实现荧光信号的可视化检测。SHI等[46]基于立足点介导的链置换反应(toehold-mediated strand displacement reaction, TMDR)和CRISPR/Cas12a系统,建立了一种现场检测Hg2+的生物传感器,通过血糖仪进行定量分析。此外,为了简化操作流程并降低成本,同时减少样品中的干扰物质对DNAzyme识别效率的影响,一些研究者将纳米酶引入CRISPR/Cas12a生物传感器,开发了无需预扩增的检测方案。例如,XU等[47]提出了一种基于CRISPR/Cas12a的比色生物传感器,用于Pb2+的检测。在此方法中,ssDNA上的带有磁珠的MnO2纳米酶可作为TMB催化剂。YU等[48]通过将DNA纳米笼限制的DNAzyme(ncCDzy)与CRISPR/Cas12a系统结合,建立了一种检测Pb2+的方法。此外,为了满足日益增加的检测需求,PAN等[49]基于CHA扩增和CRISPR/Cas12a系统,构建了基于重金属离子(Hg2+、Cd2+、Pb2+和Mn2+)作为输入的分子逻辑门。当相应的DNAzymes识别重金属离子后,利用逻辑计算激活相应的CHA反应,根据相应荧光信号判断检测结果。该智能传感器成功构建了AND、OR、INHIBIT和级联逻辑门,可以为不同重金属离子的检测提供智能传感系统。
3.5 农药残留
农药及其代谢物可通过富集和生物积累转移到食物链中并在食物链内迁移,从而对农产品的质量和安全产生不利影响,严重时会危害人类身体健康并造成环境污染。目前,色谱技术作为农药及其代谢物的常用检测方法仍局限于实验室中检测。因此,为了满足现场检测的需求,需开发简单、快速和准确度高的快检方法。
在检测食品中的农药残留时,可采用酶抑制法、竞争性免疫反应及适配体辅助的CRISPR/Cas12a生物传感器,以实现对食品中微量农药的快速检测。乙酰胆碱酯酶(acetylcholinesterase, AChE)是生物神经传导过程中的一种关键酶,能够将乙酰硫代胆碱(acetylthiocholine, ATCh)降解为硫胆碱(thiocholine, TCh)。当环境中存在农药时,AChE的催化活性会受到抑制。基于这一原理,研究人员将酶抑制作用与CRISPR/Cas12a生物传感器相结合,开发出了一系列检测有机磷和氨基甲酸酯类农药的方法。例如,YU等[52]利用这一特性,建立了一种检测有机磷农药的CRISPR/Cas12a荧光生物传感器。TIAN等[53]则利用相同原理,建立了一种检测氨基甲酸酯类杀虫剂的荧光生物传感器。此外,在CRISPR/Cas12a生物传感器中,可以通过适配体与农药的特异性结合,将非核酸靶标信号转换为核酸靶标信号,从而激活Cas12a的反式切割活性,实现对农药的快速检测。LI等[54]基于RCA反应和CRISPR/Cas12a系统,构建了一种用于检测啶虫脒的电化学生物传感器。ZHU等[55]则利用引物交换反应(primer exchange reaction, PER)与CRISPR/Cas12a系统相结合,实现了对抗生素、农药的高灵敏度检测。此外,还有研究人员利用分子逻辑门技术,实现了CRISPR/Cas12a生物传感器对农药的智能检测。YAN等[56]基于CRISPR/Cas12a平台和信号放大技术,开发了一种多功能逻辑传感系统,用于同时检测啶虫脒和阿特拉津。
4 结论与展望
CRISPR/Cas12a系统作为新一代食品安全检测工具,在检测方面展现出许多其他技术无法比拟的优势,例如检测时间短、成本低、反应条件温和以及实验条件要求简单等。这些特点使得CRISPR/Cas12a生物传感器检测不受环境和地理因素的限制,能够在现场检测中发挥显著优势,从而提高食品检测效率并促进食品安全监管。然而,基于CRISPR/Cas12a生物传感器的检测技术在实际应用过程中仍面临一些挑战,需要进一步优化和发展。
a)提高检测结果的特异性和准确性。为避免检测结果出现假阳性,研究人员设计了一步法检测,有效防止气溶胶污染。然而,这种方法在检测过程中会导致Cas蛋白同时消耗引物和底物,从而降低扩增效率。因此,未来基于CRISPR/Cas生物传感器的研究应着重优化反应体系和步骤,并利用纳米等新兴技术,提高检测结果的灵敏度和特异性。
b)多重检测。在CRISPR/Cas12a生物传感器检测中,由于大多数检测信号的产生依赖Cas蛋白的非特异性反式切割活性,因此在单个反应中进行多重检测具有挑战性。目前,大多数基于CRISPR/Cas12a生物传感器的多重检测是通过物理分离和加入不同Cas蛋白来实现的,然而这些方法增加了检测成本和难度。因此,未来应继续深入探索Cas蛋白的性能,寻求在切割活性和特异性上的突破,以实现多重检测的简便性。
c)减少PAM序列限制。Cas12a在检测时需要PAM序列来识别靶标。这极大限制了靶标的选择范围。因此,未来应挖掘更多具有识别不同PAM序列的Cas蛋白以扩大靶标的选择范围。另外,也可以通过工程化改造Cas蛋白来改变PAM序列的识别能力[59]。
d)非核酸类靶标检测。目前,基于CRISPR/Cas12a生物传感器在非核酸靶标检测方面仍处于研究的初级阶段。对于非核酸类检测,关键在于将非核酸信号转化为核酸信号,这需要设计合适且兼容的适配体或DNAzyme等功能性DNA来实现。因此,未来针对非核酸类靶标检测方法的开发应重点关注提高适配体与目标分子结合的特异性和亲和力、优化适配体的结合条件、减少非特异性结合以及降低成本等方面。
综上所述,尽管CRISPR/Cas12a生物传感器在实际应用中仍面临一些挑战,但随着该生物传感器的不断进步与完善,其在食品检测等领域将发挥日益重要的作用。
[1] WORLD HEALTH ORGANIZATION.Food safety[EB/OL].(2024-10-04)[2024-10-31].https://www.who.int/news-room/fact-sheets/detail/food-safety.
[2] TANG J, GAO Z Y, XU L F, et al.Smartphone-assisted colorimetric biosensor for the rapid visual detection of natural antioxidants in food samples[J].Food Chemistry, 2025, 462:141026.
[3] SUN Y N, WEN T J, ZHANG P, et al.Recent advances in the CRISPR/Cas-based nucleic acid biosensor for food analysis:A review[J].Foods, 2024, 13(20):3222.
[4] WU H, CHEN X Y, ZHANG M Y, et al.Versatile detection with CRISPR/Cas system from applications to challenges[J].TrAC Trends in Analytical Chemistry, 2021, 135:116150.
[5] CHEN J S, MA E B, HARRINGTON L B, et al.CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity[J].Science, 2018, 360(6387):436-439.
[6] ZAVVAR T S, KHOSHBIN Z, RAMEZANI M, et al.CRISPR/Cas-engineered technology:Innovative approach for biosensor development[J].Biosensors and Bioelectronics, 2022, 214:114501.
[7] CHARPENTIER E, RICHTER H, VAN DER OOST J, et al.Biogenesis pathways of RNA guides in archaeal and bacterial CRISPR-Cas adaptive immunity[J].FEMS Microbiology Reviews, 2015, 39(3):428-441.
[8] GASIUNAS G, SINKUNAS T, SIKSNYS V.Molecular mechanisms of CRISPR-mediated microbial immunity[J].Cellular and Molecular Life Sciences, 2014, 71(3):449-465.
[9] SHMAKOV S, SMARGON A, SCOTT D, et al.Diversity and evolution of class 2 CRISPR-Cas systems[J].Nature Reviews Microbiology, 2017, 15(3):169-182.
[10] HILLARY V E, CEASAR S A.A review on the mechanism and applications of CRISPR/Cas9/Cas12/Cas13/Cas14 proteins utilized for genome engineering[J].Molecular Biotechnology, 2023, 65(3):311-325.
[11] NATH S.Advancements in food quality monitoring:Integrating biosensors for precision detection[J].Sustainable Food Technology, 2024, 2(4):976-992.
[12] LI S Y, CHENG Q X, WANG J M, et al.CRISPR-Cas12a-assisted nucleic acid detection[J].Cell Discovery, 2018, 4:20.
[13] MU OZ H E, RICHE C T, KONG J E, et al.Fractal LAMP:Label-free analysis of fractal precipitate for digital loop-mediated isothermal nucleic acid amplification[J].ACS Sensors, 2020, 5(2):385-394.
OZ H E, RICHE C T, KONG J E, et al.Fractal LAMP:Label-free analysis of fractal precipitate for digital loop-mediated isothermal nucleic acid amplification[J].ACS Sensors, 2020, 5(2):385-394.
[14] LIU S T, XU L, HUANG Z H, et al.Recent advances of nanoparticles-assisted CRISPR/Cas biosensors[J].Microchemical Journal, 2024, 199:109930.
[15] 苏邵, 李晶, 汪联辉.基于纳米材料比色传感器的构建及其应用[J].南京邮电大学学报(自然科学版), 2018, 38(3):98-110.SU S, LI J, WANG L H.Construction and application of nanomaterials-based colorimetric sensors[J].Journal of Nanjing University of Posts and Telecommunications(Natural Science Edition), 2018, 38(3):98-110.
[16] YIN L J, DUAN N H, CHEN S, et al.Ultrasensitive pathogenic bacteria detection by a smartphone-read G-quadruplex-based CRISPR-Cas12a bioassay[J].Sensors and Actuators B:Chemical, 2021, 347:130586.
[17] DAI Y F, SOMOZA R A, WANG L, et al.Exploring the trans-cleavage activity of CRISPR-Cas12a (cpf1) for the development of a universal electrochemical biosensor[J].Angewandte Chemie, 2019, 131(48):17560-17566.
[18] XU J R, ZHANG T X, LV X R, et al.An RPA-based CRISPR/Cas12a assay in combination with a lateral flow assay for the rapid detection of Shigella flexneri in food samples[J].Foods, 2024, 13(19):3200.
[19] XING G W, SHANG Y T, WANG X R, et al.Multiplexed detection of foodborne pathogens using one-pot CRISPR/Cas12a combined with recombinase aided amplification on a finger-actuated microfluidic biosensor[J].Biosensors and Bioelectronics, 2023, 220:114885.
[20] LEE S Y, KIM U, KIM Y, et al.Enhanced detection of Listeria monocytogenes using tetraethylenepentamine-functionalized magnetic nanoparticles and LAMP-CRISPR/Cas12a-based biosensor[J].Analytica Chimica Acta, 2023, 1281:341905.
[21] DUAN M L, LI B Y, HE Y W, et al.A CG@MXene nanocomposite-driven E-CRISPR biosensor for the rapid and sensitive detection of Salmonella Typhimurium in food[J].Talanta, 2024, 266:125011.
[22] GU X J, TANG Q, KANG X X, et al.A portable CRISPR-Cas12a triggered photothermal biosensor for sensitive and visual detection of Staphylococcus aureus and Listeria monocytogenes[J].Talanta, 2024, 271:125678.
[23] SUN X D, SHAN Y J, JIAN M H, et al.A multichannel fluorescence isothermal amplification device with integrated internet of medical things for rapid sensing of pathogens through deep learning[J].Analytical Chemistry, 2023, 95(41):15146-15152.
[24] ZHOU Y, ZHAO J P, CHEN R, et al.A portable deep-learning-assisted digital single-particle counting biosensing platform for amplification-free nucleic acid detection using a lens-free holography microscope[J].Nano Today, 2024, 56:102238.
[25] FU X R, SUN J D, YU B Q, et al.Investigating enzyme kinetics and fluorescence sensing strategy of CRISPR/Cas12a for foodborne pathogenic bacteria[J].Analytica Chimica Acta, 2024, 1290:342203.
[26] SONG Y, XU Y W, WANG R R, et al.CRISPR-Cas12a-based nanoparticle biosensor for detection of pathogenic bacteria in food[J].Microchemical Journal, 2024, 207:111813.
[27] LIANG Q Q, WANG X H, XIE Q Q, et al.Development of highly sensitive one-pot ERA-CRISPR assays for on-site detection of CaMV35S promoter and NOS terminator in genetically modified crops[J].Food Control, 2025, 170:111049.
[28] WANG J B, LUO J W, LIU H, et al.“Blue-red-purple” multicolored lateral flow immunoassay for simultaneous detection of GM crops utilizing RPA and CRISPR/Cas12a[J].Talanta, 2025, 282:127010.
[29] LI X H, LIU M L, XIA X H, et al.Deep learning assisted, smartphone based universal Multi-RPA-CRISPR/Cas12a-G4 portable chip for simultaneous detection of CaMV35S and NOS[J].Food Control, 2025, 168:110947.
[30] LIU M L, LI X H, XU J B, et al.Sensitive detection of CaMV35S based on exponential rolling circle amplification reaction and CRISPR/Cas12a using a portable 3D-printed visualizer[J].Microchemical Journal, 2024, 205:111313.
[31] PENG C, WANG Y L, CHEN X Y, et al.A localized CRISPR assay that detects short nucleic acid fragments in unamplified genetically modified samples[J].ACS Sensors, 2023, 8(3):1054-1063.
[32] WANG J B, WANG Y, LIU H, et al.An ultra-sensitive test strip combining with RPA and CRISPR/Cas12a system for the rapid detection of GM crops[J].Food Control, 2023, 144:109383.
[33] ZHU L L, HE G W, YANG G Q, et al.A rapid on-site visualization platform based on RPA coupled with CRISPR-Cas12a for the detection of genetically modified papaya ‘Huanong No.1’[J].Talanta, 2024, 277:126437.
[34] WU C Y, WANG X J, GUO L, et al.An electrochemical aptasensor based on exonuclease III-assisted signal amplification coupled with CRISPR-Cas12a for ochratoxin A detection[J].Food Control, 2023, 148:109631.
[35] ESMAELPOURFARKHANI M, RAMEZANI M, ALIBOLANDI M, et al.CRISPR-Cas12a-based colorimetric aptasensor for aflatoxin M1 detection based on oxidase-mimicking activity of flower-like MnO2 nanozymes[J].Talanta, 2024, 271:125729.
[36] WU C Y, YUE Y Y, HUANG B C, et al.CRISPR-powered microfluidic biosensor for preamplification-free detection of ochratoxin A[J].Talanta, 2024, 269:125414.
[37] XIANG X R, SONG M H, XU X W, et al.Microfluidic biosensor integrated with signal transduction and enhancement mechanism for ultrasensitive noncompetitive assay of multiple mycotoxins[J].Analytical Chemistry, 2023, 95(20):7993-8001.
[38] ZHAO X X, WANG Z X, ZHANG H, et al.Highly sensitive one-pot isothermal assay combining rolling circle amplification and CRISPR/Cas12a for aflatoxin B1 detection[J].Analytical Chemistry, 2024, 96(45):18070-18078.
[39] WANG Z L, WEI L Y, RUAN S L, et al.CRISPR/Cas12a-assisted chemiluminescence sensor for aflatoxin B1 detection in cereal based on functional nucleic acid and in-pipet rolling circle amplification[J].Journal of Agricultural and Food Chemistry, 2023, 71(10):4417-4425.
[40] YAN H, HE B S, ZHAO R Y, et al.Electrochemical aptasensor based on CRISPR/Cas12a-mediated and DNAzyme-assisted cascade dual-enzyme transformation strategy for zearalenone detection[J].Chemical Engineering Journal, 2024, 493:152431.
[41] ZHU F X, ZHAO Q.Competitive aptamer assay for aflatoxin B1 detection using CRISPR/Cas12a as a signal amplifier[J].Microchemical Journal, 2024, 207:112054.
[42] LIU M L, LI X H, ZHOU S Y, et al.Ultrasensitive detection of mycotoxins using a novel single-Atom, CRISPR/Cas12a-Based nanozymatic colorimetric biosensor[J].Chemical Engineering Journal, 2024, 497:154418.
[43] YUE Y Y, WANG S T, JIN Q, et al.A triple amplification strategy using GR-5 DNAzyme as a signal medium for ultrasensitive detection of trace Pb2+ based on CRISPR/Cas12a empowered electrochemical biosensor[J].Analytica Chimica Acta, 2023, 1263:341241.
[44] ONO A, TOGASHI H.Highly selective oligonucleotide-based sensor for mercury(II) in aqueous solutions[J].Angewandte Chemie International Edition, 2004, 43(33):4300-4302.
[45] KONG F G, WANG C X, PENG S C, et al.CRISPR-Hg:Rapid and visual detection of Hg2+ based on PCR coupled with CRISPR/Cas12a[J].Talanta, 2024, 277:126379.
[46] SHI K, CHEN J X, LI Y H, et al.Hg2+-triggered cascade strand displacement assisted CRISPR-Cas12a for Hg2+ quantitative detection using a portable glucose meter[J].Analytica Chimica Acta, 2023, 1278:341756.
[47] XU S Q, WANG S T, GUO L, et al.Nanozyme-catalysed CRISPR-Cas12a system for the preamplification-free colorimetric detection of lead ion[J].Analytica Chimica Acta, 2023, 1243:340827.
[48] YU Y, ZHANG Y, LI W H, et al.DNA nanocage confined DNAzyme for detection of lead ions coupled with CRISPR-Cas12a system[J].Chemical Engineering Journal, 2024, 480:148177.
[49] PAN J F, DENG F, LIU Z, et al.Construction of molecular logic gates using heavy metal ions as inputs based on catalytic hairpin assembly and CRISPR-Cas12a[J].Talanta, 2023, 255:124210.
[50] WEN J L, DENG H J, HE D G, et al.Dual-functional DNAzyme powered CRISPR-Cas12a sensor for ultrasensitive and high-throughput detection of Pb2+ in freshwater[J].Science of the Total Environment, 2024, 911:168708.
[51] YANG C Y, DU C Y, YUAN F Y, et al.CRISPR/Cas12a-derived ratiometric fluorescence sensor for high-sensitive Pb2+ detection based on CDs@ZIF-8 and DNAzyme[J].Biosensors and Bioelectronics, 2024, 251:116089.
[52] YU H M, LIANG G X, WANG H Y, et al.A MnO2 nanosheet-mediated CRISPR/Cas12a system for the detection of organophosphorus pesticides in environmental water[J].Analyst, 2024, 149(3):729-734.
[53] TIAN F Y, JIANG L, WANG Z Y, et al.Mn2+-activated CRISPR-Cas12a strategy for fluorescence detection of the insecticide carbaryl[J].Sensors and Actuators B:Chemical, 2024, 398:134695.
[54] LI W, LI Y, ZHAO L D, et al.Triple-helix as a target converter for trace pesticide detection based on CRISPR/Cas12a-based ECL biosensor[J].Sensors and Actuators B:Chemical, 2024, 409:135599.
[55] ZHU L, ZHANG X M, YANG L, et al.Primer exchange reaction activation of CRISPR/Cas12a system to construct a versatile and label-free electrochemical sensing platform[J].Sensors and Actuators B:Chemical, 2025, 423:136791.
[56] YAN C, SHI G, CHEN J H.Fluorescent detection of two pesticides based on CRISPR-Cas12a and its application for the construction of four molecular logic gates[J].Journal of Agricultural and Food Chemistry, 2022, 70(39):12700-12707.
[57] WANG Y S, DU P F, WANG X H, et al.A novel HCR-CRISPR/Cas12a immunosensor for the sensitive detection of pesticide residues in animal-derived foods[J].Food Bioscience, 2024, 62:105131.
[58] PENG X G, HE Y, ZHAO J W, et al.CRISPR/Cas12a-mediated aptasensor based on tris-(8-hydroxyquinoline) aluminum microcrystals with crystallization-induced enhanced electrochemiluminescence for acetamiprid analysis[J].Analytical Chemistry, 2023, 95(26):10068-10076.
[59] COLLIAS D, BEISEL C L.CRISPR technologies and the search for the PAM-free nuclease[J].Nature Communications, 2021, 12(1):555.