腊肉是我国的传统特色食品,其历史可追溯至先秦时期的肉脯加工技艺,凭借独特的色泽、风味及工艺多样性在中华饮食文化中占据重要地位。在腊肉加工中,氯化钠不仅承担基础调味功能,更通过形成细胞内外渗透压梯度破坏微生物代谢平衡、降低水分活度抑制胞外酶活性等抑菌机制保障产品的安全。这一双重作用使氯化钠成为传统腊肉不可或缺的防腐剂,但高盐工艺(盐分添加量≥5%)引发的钠摄入过量问题已引起国际关注——世界卫生组织建议每日钠摄入量应控制在2 g(折合食盐5 g)以内,而我国居民实际摄入量已达推荐值的1.8~2.4倍[1]。流行病学研究证实,膳食钠过量与高血压、心血管疾病等慢性病风险呈显著正相关[2],推动低钠腊肉研发成为食品科学领域的重要课题。
微生物群落动态是影响腊肉品质与安全的核心要素。现有研究通过分子生物学技术揭示了不同地域腊肉的特征菌群。范俐等[3]采用PacBio SMRT测序发现闽北腊肉风干阶段以放线菌属、葡萄球菌属等为优势菌群,真菌群落则以德巴利氏酵母属、库兹马尼氏菌属等为主导;左瑶等[4]通过16S rRNA基因测序探究了镇巴腊肉在加工过程中微生物的多样性变化,发现在制作过程中微生物群落存在明显的演替,其中丰度最高的细菌为厚壁菌门和变形菌门;文开勇等[5]基于Illumina MiSeq平台解析四川腊肉菌群结构,发现其细菌多样性显著高于真菌,优势菌属为葡萄球菌属和曲霉属。这些研究为理解传统腊肉微生物生态提供了重要基础,而现有低钠腊肉研究多局限于可培养菌群(如乳酸菌、葡萄球菌等)的定量分析,对未培养微生物及整体微生物组成并没有详细的探究。
本文将宏基因组学方法应用于低钠腊肉风干贮藏过程的微生物分析,通过构建低钠腊肉风干贮藏过程产品的宏基因组测序数据库,系统解析风干贮藏过程菌群的Alpha多样性的变化规律、潜在腐败菌与致病菌的丰度动态以及关键代谢通路的微生物贡献度。研究结果可为建立基于微生物靶向调控的低钠腊肉品质控制体系提供理论支撑,同时为传统肉制品减盐工艺优化提供方法参考。
1 材料与方法
1.1 材料与试剂
猪肉、氯化钠、白砂糖、花椒,市售;氯化钾、亚硝酸钠,分析纯,国药集团化学试剂有限公司;PF Mag-Bind Soil DNA Kit DNA提取试剂盒,美国Omega Bio-Tek公司;Agarose75510019琼脂糖(invitrogen原装),美国Thermo Scientific公司;NEXTFLEX Rapid DNA-Seq Kit建库试剂盒,美国Bioo Scientific公司;NovaSeq 6000 S4 Reagent Kit v1.5(300 cycles)建序试剂盒,美国Illumina公司。
1.2 仪器与设备
Eppendorf N13462C移液器、Eppendorf 5430R小型离心机、Eppendorf 5424R高速台式冷冻离心机,德国Eppendorf公司;ABSON MiFly-6小型离心机,中国合肥艾本森科学仪器有限公司;NanoDrop2000超微量分光光度计、Qubit 4.0荧光定量仪,美国Thermo Fisher Scientific公司;FastPrep-24 5G MP研磨仪,美国MP公司;JY600C双稳定时电泳仪,中国北京君意东方电泳设备有限公司;Novaseq测序仪,美国Illumina公司;Covaris M220台式自动聚焦声波基因组剪切仪,中国基因公司。
1.3 实验方法
1.3.1 低钠腊肉的制作
在生猪宰杀后,取100 kg新鲜后腿肉切割成20 cm长,15 cm宽的块状并用盐腌制。腌盐由氯化钠(2.68%,质量分数,下同)、氯化钾(0.88%)、花椒(0.4%)、白砂糖(0.5%)、亚硝酸钠(0.01%)混合而成,各腌料的添加量以肉重的百分比计[6]。将原料肉擦上盐后入缸,4 ℃持续腌制15 d;腌肉出缸、水洗后,在65 ℃烘烤3 h,然后在50 ℃下烟熏45 h,最后取出,在10 ℃左右通风阴凉的场所晾挂贮藏。在腊肉贮存过程中的第0、1、2、3、4个月(分别标记为XB0、XB1、XB2、XB3、XB4阶段)采样,迅速置于-80 ℃保存,以备检测。
1.3.2 DNA提取与宏基因组测序
5组腊肉共计15个样品由上海美吉生物医学技术有限公司进行DNA提取和Illumina测序。使用DNA提取试剂盒提取样品中的DNA。用琼脂糖电泳检测DNA浓度、纯度及基因组DNA完整性。通过台式自动聚焦声波基因组剪切仪将DNA片段化并筛选约400 bp的片段,用于构建双末端测序文库(paired-end,PE)。使用NEXTFLEX Rapid DNA-Seq建库试剂盒构建PE文库。对DNA进行桥式PCR扩增后使用Illumina NovaSeq测序平台进行宏基因组测序。
1.3.3 宏基因组测序数据分析
使用软件fastp(https://github.com/OpenGene/fastp,version 0.23.0)进行数据质控,对读段 3′端和5′端的接头序列进行质量剪切并保留高质量的双端读段和单端读段。通过软件BWA(http://bio-bwa.sourceforge.net/,version v0.7.17)将读段与宿主DNA序列对比,将相似性高的污染读段去除。使用拼接软件MEGAHIT(https://github.com/voutcn/megahit,version 1.1.2)对优化序列进行拼接组装,筛选≥300 bp的连续序列作为最终的组装结果。使用MetaGene(http://metagene.cb.k.u-tokyo.ac.jp/)对连续系列进行开放阅读框预测(open reading frame prediction,ORF)预测,并使用软件CD-HIT(http://www.bioinformatics.org/cd-hit/,version 4.6.1)对所有样品预测出的基因序列进行聚类,以构建非冗余基因集。使用软件SOAPaligner(http://soap.genomics.org.cn/,version 2.21)分别将每个样品的高质量读段与非冗余基因集进行比对(95%一致性),统计基因在对应样品中的丰度信息。最后使用Diamond(https://github.com/bbuchfink/diamond,version v2.0.13)和hmmscan(http://hmmer.janelia.org/search/hmmscan)等工具将非冗余基因集与非冗余蛋白序列数据库(non-redundant protein sequence database,NR,https://ftp.ncbi.nlm.nih.gov/blast/db/FASTA/)、KEGG(https://www.genome.jp/kegg)和直系同源蛋白分组比对数据库(evolutionary genealogy of genes: non-supervised orthologous groups,eggNOG,http://eggnog5.embl.de/#/app/downloads)等数据库进行比对注释,获得各样本中物种的相对丰度,并对样本进行相似性聚类、排序检验以及差异统计比较等分析。
1.4 数据处理
使用 OriginPro 2025软件绘制低钠腊肉样品中微生物群落的Alpha多样性指数图,物种相对丰度、直系同源基因簇(database of clusters of orthologous genes,COG)、KEGG数据库注释结果均使用美吉生物云平台(https://cloud.majorbio.com/)进行分析和绘制,对数据结果进行算术平均处理。
2 结果与分析
2.1 不同风干贮藏阶段低钠腊肉的宏基因组测序结果与统计
对低钠腊肉风干贮藏过程的5个阶段(共15个样品)开展宏基因组测序,数据统计如表1所示。结果表明,所有样品共获得678 706 034条原始序列,经质控过滤后保留670 744 732条有效序列,平均有效数据率达98.83%。通过NR数据库比对,成功解析样品中微生物的物种分类信息,鉴定微生物涵盖7界、15门、28纲、46目、75科、121属、412种,呈现丰富的群落结构。微生物分类层级分布表明,低钠腊肉中菌群组成复杂,且在门、属、种水平均存在显著多样性。
表1 低钠腊肉的宏基因组测序结果
Table 1 Metagenomic sequencing results of low-sodium cured meat

样品插入片段长度/bp测序读长/bp原始读段数原始碱基数/bp有效读段数有效碱基/bp原始读段保留率/%原始碱基保留率/%XB0_149015044 569 3146 729 966 41444 014 2686 607 792 53298.754 645 4098.184 628 65XB0_247915046 564 3047 031 209 90445 987 4526 903 479 14298.761 171 2298.183 374 36XB0_349615050 907 5507 687 040 05050 322 6347 556 796 41998.851 023 0898.305 672 53XB1_146015043 984 6566 641 683 05643 578 6146 552 594 08799.076 855 3598.658 638 66XB1_246115041 985 7046 339 841 30441 589 3526 253 678 99499.055 983 4498.640 939 01XB1_349215046 092 7446 960 004 34445 530 9346 836 214 22698.781 131 3698.221 407 46XB2_149215046 737 6867 057 390 58646 187 2566 934 297 47698.822 299 4298.255 826 87XB2_247515048 326 9887 297 375 18847 768 3527 173 888 84698.844 049 6298.307 797 82XB2_349015045 337 7186 845 995 41844 817 3106 729 890 75798.852 152 2098.304 049 98XB3_148215045 841 3986 922 051 09845 336 7146 808 716 78198.899 064 9998.362 706 15XB3_248115043 217 9446 525 909 54442 660 5346 404 151 91298.710 234 8098.134 242 73XB3_348815042 173 3686 368 178 56841 662 6986 253 512 99498.789 117 3498.199 397 63XB4_150015042 508 9086 418 845 10841 977 0086 301 555 68898.748 732 8598.172 733 29XB4_248515044 878 6906 776 682 19044 306 9386 649 763 76398.726 005 5998.127 130 31XB4_348915045 579 0626 882 438 36245 004 6686 757 515 31498.739 785 3998.184 901 32
2.2 不同风干贮藏阶段低钠腊肉中微生物Alpha多样性分析
Alpha多样性分析可定量表征低钠腊肉贮藏过程中微生物群落的结构动态,其核心指标包括反映物种丰富度的Chao指数、ACE(abundance-based coverage estimator,基于丰度的覆盖度估计量)指数,以及衡量综合多样性的Shannon指数和Simpson指数[7]。Chao和ACE指数的数值与可检测物种数呈正相关。图1显示,这2个指数在贮藏初期呈下降趋势,于第1个月达到最低值(49.67),表明此阶段微生物种类显著减少;第2个月出现峰值(65.67),提示新物种在快速定殖;随后在第3~4个月再次下降,第4个月(51.67)比初始水平(56.00)低,说明微生物种类再次显著减少,降至初始水平以下。
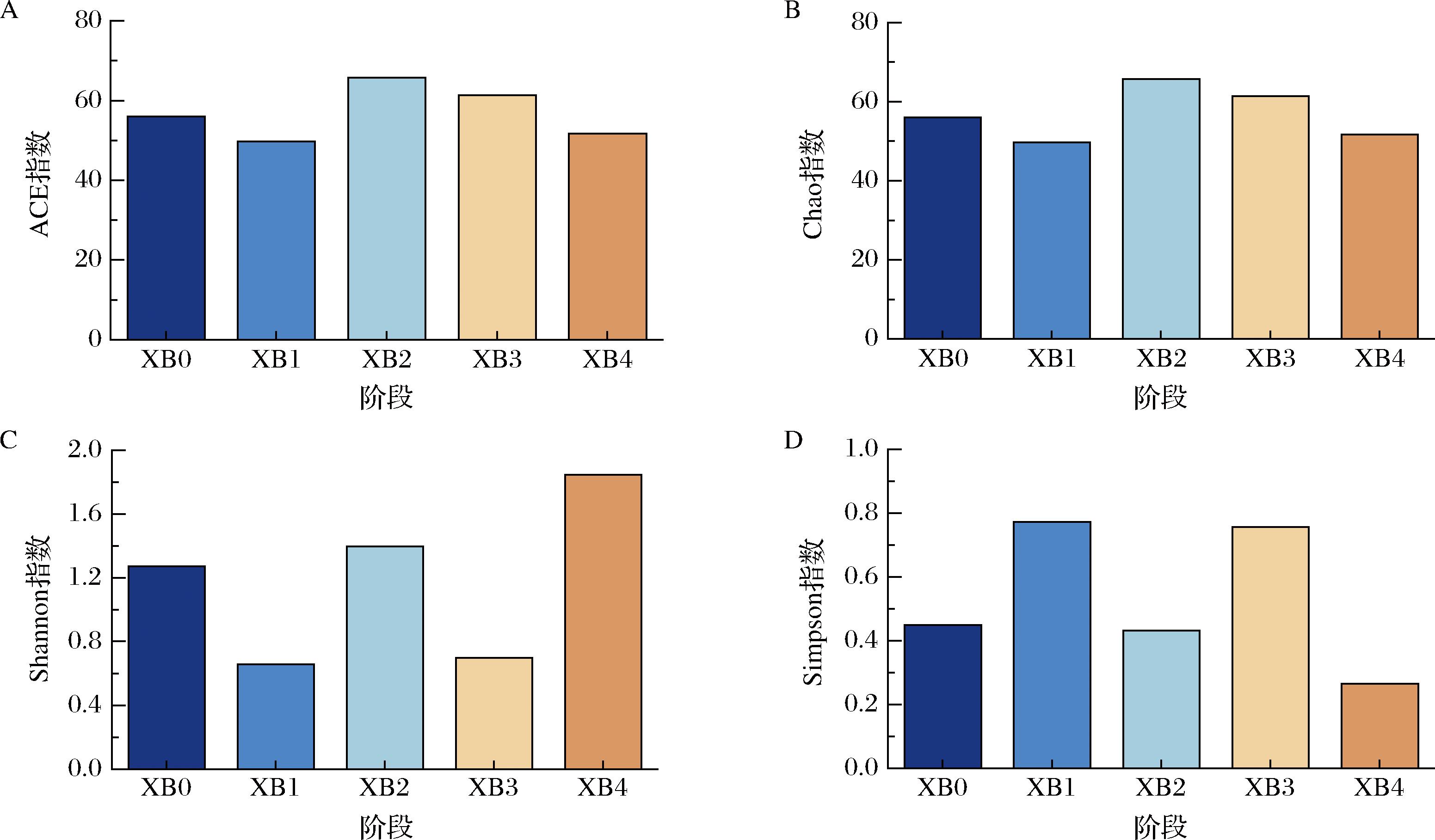
A-ACE指数;B-Chao指数;C-Shannon指数;D-Simpson指数
图1 风干贮藏过程中低钠腊肉的微生物Alpha多样性
Fig.1 Microbial Alpha diversity of low-sodium cured meat during air-dried storage
注:XB0-贮藏第0个月;XB1-贮藏第1个月;XB2-贮藏第2个月;XB3-贮藏第3个月;XB4-贮藏第4个月(下同)。
Shannon指数能同时兼顾物种的丰富度与均匀度,对稀有物种的变化敏感,当少量新物种植入时,Shannon指数提升幅度可能高于Simpson指数,而Simpson指数则对物种均匀度敏感,优势物种占比越高,指数值越高。它们在图1中呈现出镜像变化。贮藏第1个月,Shannon指数最低(0.66),Simpson指数最高(0.77),表明此时微生物丰度低,群落结构单一;贮藏4个月,Shannon指数跃升至最高(1.85),Simpson指数降至最低(0.26),表明物种分布趋于均衡。
以上数据揭示,贮藏1个月后,低钠腊肉中的物种丰富度与多样性同步降至最低,可能源于钠盐抑制效应导致的微生物生长迟滞期;贮藏2个月后,物种丰富度达到峰值,但Shannon指数仅小幅回升,提示此时新定殖菌群尚未形成稳定生态位;贮藏4个月后,物种丰富度回归初期水平,而Shannon指数显著升高,反映群落内物种竞争达到动态平衡,优势菌群与稀有物种形成复杂互作网络。结果表明,贮藏中期(第2个月)和后期(第4个月)是微生物代谢活跃的关键窗口期,此阶段菌群结构变化可能直接影响腊肉风味物质合成与腐败风险。
2.3 不同风干贮藏阶段低钠腊肉中微生物组成分析
2.3.1 门水平微生物组成分析
图2显示了低钠腊肉贮藏过程中门水平微生物群落的动态演替特征。门水平上,微生物群落以芽孢杆菌门(Bacillota,88.28%)为主导,其次为子囊菌门(Ascomycota,5.64%)、假单胞菌门(Pseudomonadota,4.19%)、放线菌门(Actinomycetota,1.27%)及尾噬菌体门(Uroviricota,0.33%)。
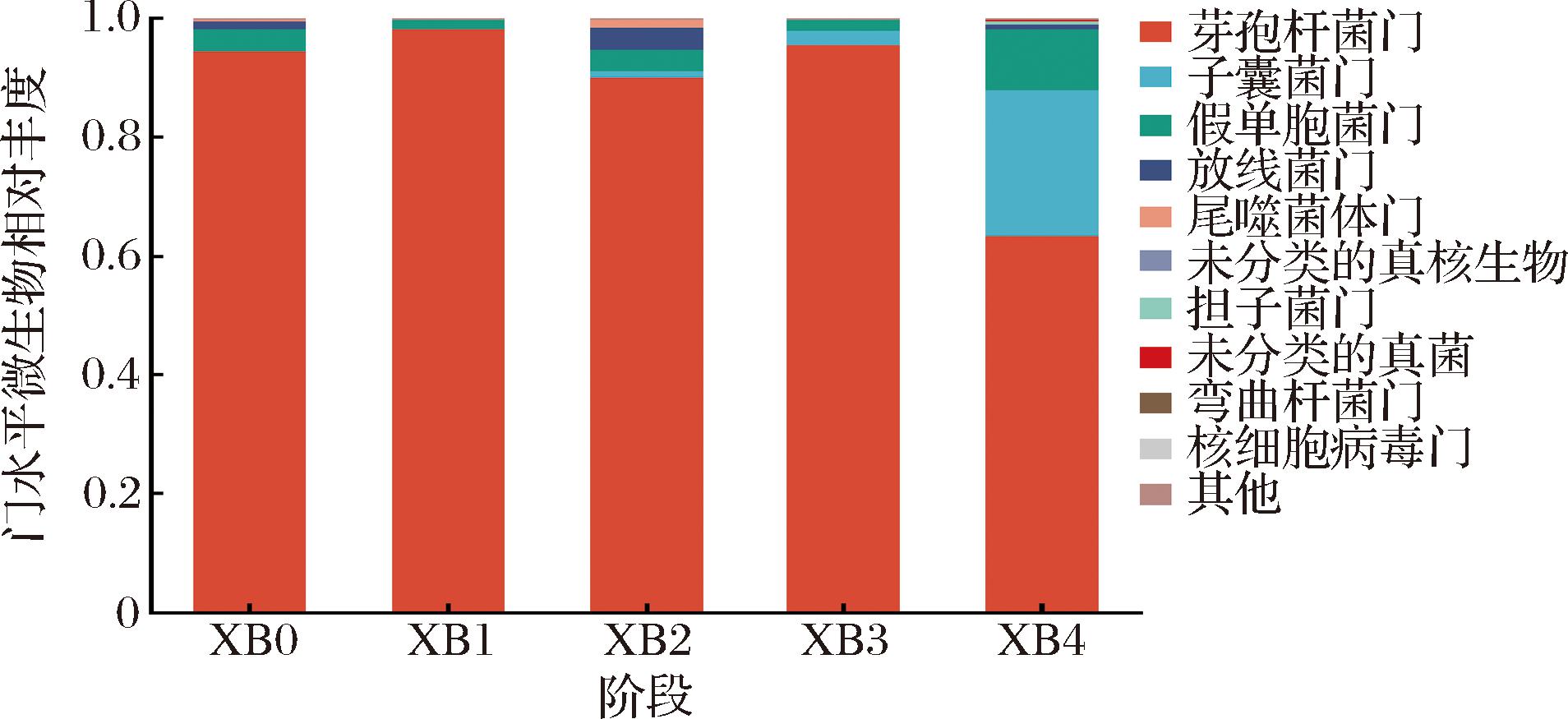
图2 风干贮藏过程中低钠腊肉的门水平微生物相对丰度
Fig.2 Microbial relative abundance on phylum level in low-sodium cured meat during air-dried storage
在贮藏过程中,芽孢杆菌门在贮藏前3个月呈现动态平衡(94.35%→98.13%→89.95%),第1个月相对丰度峰值(98.13%)可能与其耐盐特性相关[8];第4个月骤降至63.54%,推测与脂质氧化产物积累引发的菌群竞争抑制有关[9]。子囊菌门相对丰度从初始0.06%持续递增至第4个月24.43%,其后期优势可能与蛋白酶活性增强促进蛋白质水解相关[10]。假单胞菌门贮藏阶段相对丰度呈现“高-低-高-低-高”波动模式(3.75%→1.62%→3.53%→1.81%→10.21%),第4个月峰值可能源于游离脂肪酸降解产物的代谢促进作用[11]。而放线菌门与尾噬菌体门在第2个月同步达到峰值(3.96%和1.33%)。放线菌门在氨基酸代谢中扮演着重要角色。为适应生存环境,放线菌门通过降解蛋白质生成游离氨基酸,同时能调控氨基酸代谢生成次级代谢物(如抗生素类物质),抑制腐败微生物生长;而尾噬菌体门是噬菌体的一个分类单元,其可以通过裂解宿主细菌释放胞内氨基酸等营养物质,间接支持放线菌门等优势菌群的增殖,形成代谢协同效应。放线菌门和尾噬菌体门的占比同步达到峰值,反映此阶段代谢产物的互作增强,该波动模式与腊肉贮藏中期氨基酸代谢活跃期的菌群响应机制相关[12-13]。低钠腊肉优势菌群组成与信阳黑猪腊肉(芽孢杆菌门平均占比75.21%)[14]、徽派腊肉(芽孢杆菌门峰值52.5%,假单胞菌门峰值52.2%)[15]、川渝腊肉(芽孢杆菌门占比在95.24%~99.20%波动)[16]具有显著一致性,印证了传统腊肉发酵过程中菌群演替的普适性规律。
2.3.2 属水平微生物组成分析
图3显示了低钠腊肉贮藏过程中属水平微生物群落的动态演替特征。在属水平上,按平均相对丰度,优势微生物依次为广布乳杆菌属(Latilactobacillus,45.45%)、葡萄球菌属(Staphylococcus,21.25%)、拟梭菌属(Clostridioides,16.73%)和曲霉属(Aspergillus,4.73%)等。广布乳杆菌属的相对丰度呈现显著波动(49.94%→82.68%→9.39%→81.20%→4.06%)。该波动模式与低盐腊肉中乳酸菌对盐分渗透压的阶段性适应特征吻合。葡萄球菌属的相对丰度变化与广布乳杆菌属呈显著负相关,第2个月达到峰值65.46%。此现象与湘西传统腊肉中葡萄球菌属主导脂质氧化与风味前体物质合成的功能特性一致[17]。
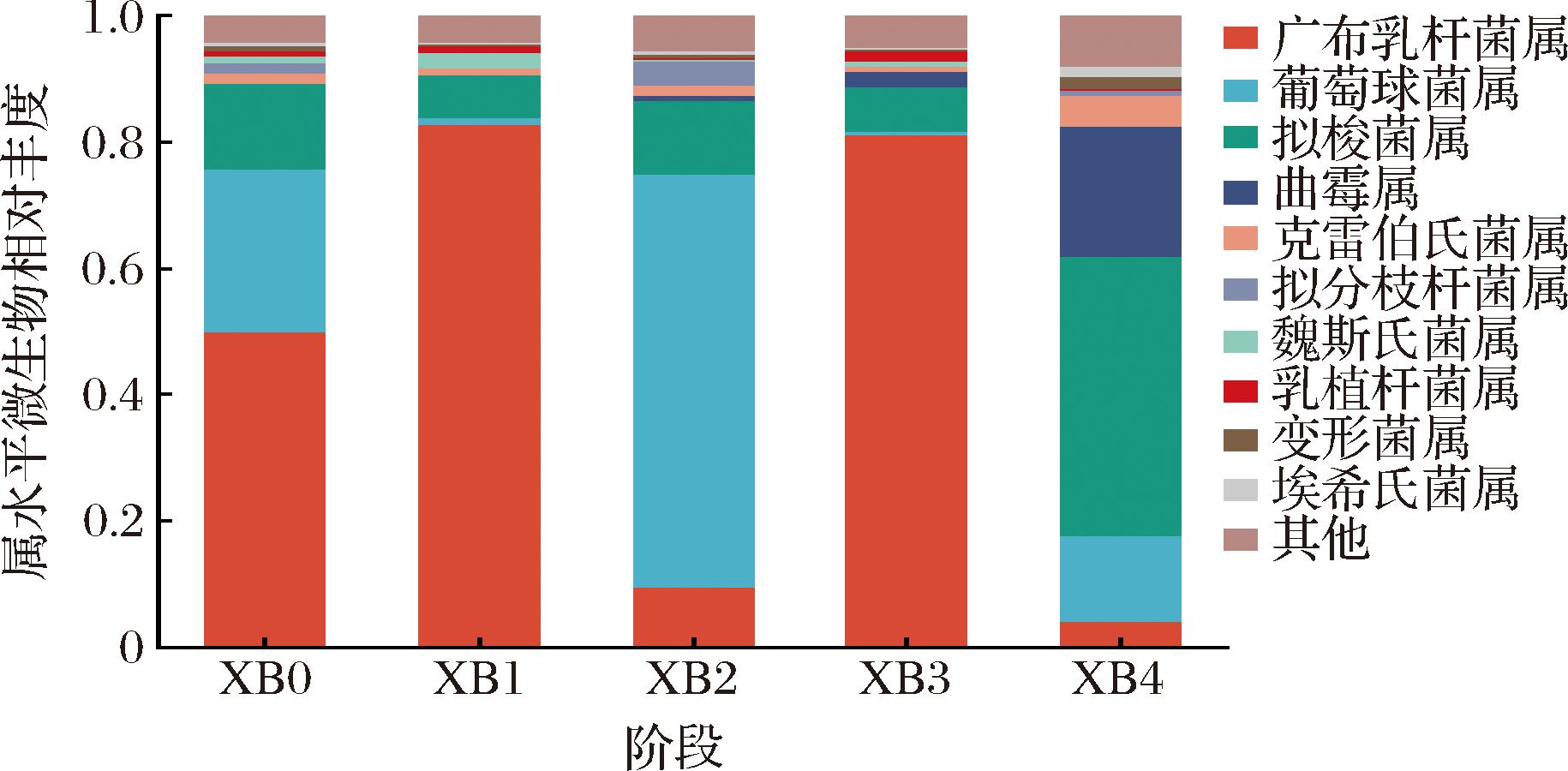
图3 风干贮藏过程中低钠腊肉的属水平微生物相对丰度
Fig.3 Microbial relative abundance on genus level in low-sodium cured meat during air-dried storage
此外,在低钠腊肉中发现了致病菌与条件致病菌。在贮藏前中期(第0~3个月),拟梭菌属的相对丰度为6.90%~13.45%,在第4个月骤增至44.35%,其异常增殖可能源于低钠环境下腐败代谢产物的积累。克雷伯氏菌属(Klebsiella)在第4个月检出(相对丰度4.91%)。曲霉属在腊肉的贮藏前期(第0~1月)并没有被发现,但是在第2个月开始逐渐上升,在第4个月相对丰度达20.68%。
主要优势菌属中,广布乳杆菌属和葡萄球菌属能在酸性和高盐的环境下生长,是腊肉形成风味的关键微生物[14]。拟梭菌属是一种人畜共患病致病菌,代表菌种有艰难拟梭菌,其极容易引起抗生素相关性腹泻和坏死性肠炎等疾病[18]。曲霉属是霉菌的一种,属于好氧微生物,在贮藏过程中会附着在肉制品的表面,具有抑制腐败微生物的繁殖和改善腊肉风味的作用[19]。然而霉菌在改善风味的同时,也会产生对人体有害次生代谢物霉菌毒素,因此需要谨慎对待。曲霉属的出现与腊肉的贮藏方式以及贮藏环境有关,环境的湿度增长与腊肉的水分含量上升会导致曲霉属的增长,贮藏时间过长,会导致曲霉属大量生长成为优势菌属。克雷伯氏菌是能寄生于肠道与呼吸道的条件致病菌,有可能引起人肺部感染[20]。
结果表明,风干贮藏中期(第2~3个月),低钠腊肉的菌群剧烈波动,是风味形成的关键窗口期,而后期(第4个月)的致病菌增殖,需通过控湿(<70%)与防腐剂干预以实现风险管控。
2.3.3 种水平微生物组成分析
宏基因组学方法具有全面解析整个微生物群落的能力,包括可培养和不可培养微生物在内,拥有更广泛的覆盖范围和更深入的分析能力。由于传统生物学方法的限制,目前有关腊肉微生物组成在物种水平上的相关信息较为有限。宏基因组学方法为深度探究腊肉微生物群落结构提供了可能性。
图4展示了不同风干贮藏阶段的低钠腊肉微生物群落在物种水平上的相对丰度变化特征。在种水平上,主要优势微生物按平均相对丰度依次为清酒广布乳杆菌(Latilactobacillus sakei,33.11%)、艰难拟梭菌(Clostridioides difficile,16.73%)、腐生葡萄球菌(Staphylococcus saprophyticus,11.1%)、弯曲广布乳杆菌(Latilactobacillus curvatus,10.46%)、葡萄球菌(Staphylococcus sp.,5.38%)、密歇根克雷伯氏菌(Klebsiella michiganensis,1.96%)、广布乳杆菌(Latilactobacillus sp.,1.77%)、脓肿拟分枝杆菌(Mycobacteroides abscessus,1.21%)、科氏葡萄球菌(Staphylococcus cohnii,1.06%)和鸡葡萄球菌(Staphylococcus gallinarum,1.03%)等。
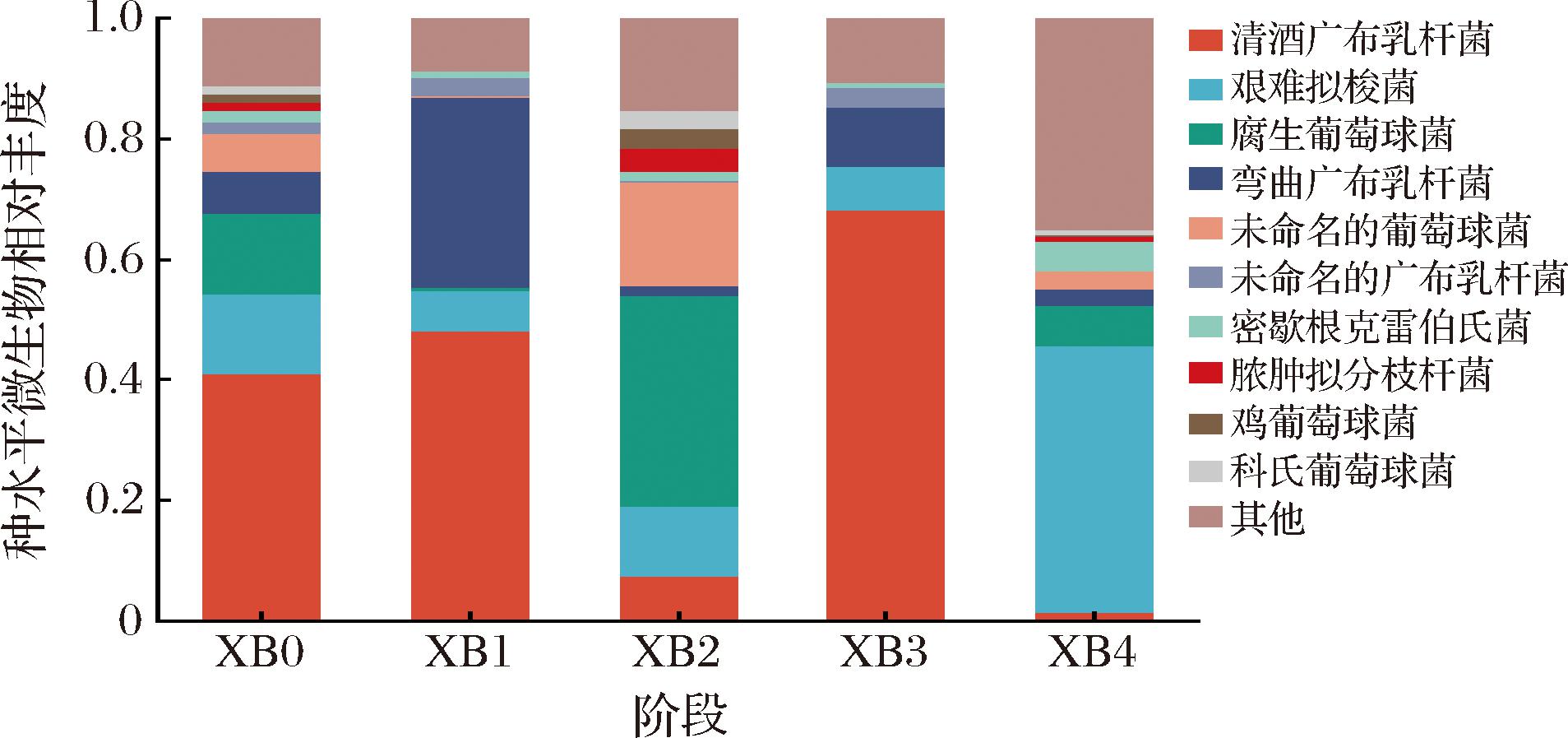
图4 风干贮藏过程中低钠腊肉的种水平微生物相对丰度
Fig.4 Microbial relative abundance on species level in low-sodium cured meat during air-dried storage
贮藏初始时(第0个月),主要优势微生物为清酒广布乳杆菌(40.91%)、艰难拟梭菌(13.44%)和腐生葡萄球菌(13.21%);贮藏中期(第2个月)时,清酒广布乳杆菌迅速减少至7.34%,而腐生葡萄球菌的相对丰度则上升至34.81%;而在贮藏结束时(第4个月),艰难拟梭菌成为主要的优势微生物(44.34%),而清酒广布乳杆菌的相对丰度仅占1.19%。清酒广布乳杆菌是发酵食品中常见的菌种之一,广泛存在于发酵肉制品、清酒、面团、酸菜和泡菜等食品中,通过产乳酸抑制腐败菌增殖[14,21]。腐生葡萄球菌是一种发酵剂,其脂酶活性可能驱动腊肉特征性风味物质生成,产生的硝酸还原酶能促进腊肉特征色泽形成[22]。然而,艰难拟梭菌是一种具有潜在食源传播性的孢子形成病原体,可能导致严重的肠道疾病发作[23]。许多地区存在悬挂腊肉进行长期贮藏的习惯,因此必须严格控制腊肉的保存时间,以确保食用安全。
2.4 低钠腊肉中微生物的功能注释分析
2.4.1 COG功能注释分析
将获得的数据结果与eggNOG数据库进行比对,各基因获得的COG注释情况如图5所示。通过COG注释,将所有基因按照26个功能进行了分类。结果显示,26个COG功能中,核心代谢功能的占比呈现了“三高一低”的格局。其中,高占比功能包括J类(翻译、核糖体结构和生物合成,占比8.14%~10.08%)、G类(碳水化合物转运和代谢,占比6.49%~8.88%)以及E类(氨基酸转运和代谢,占比5.68%~11.18%)。反之,低占比功能是I类(脂质转运和代谢,占比3.5%~5.21%),显示出低钠腊肉中微生物对脂质代谢过程的弱依赖性。
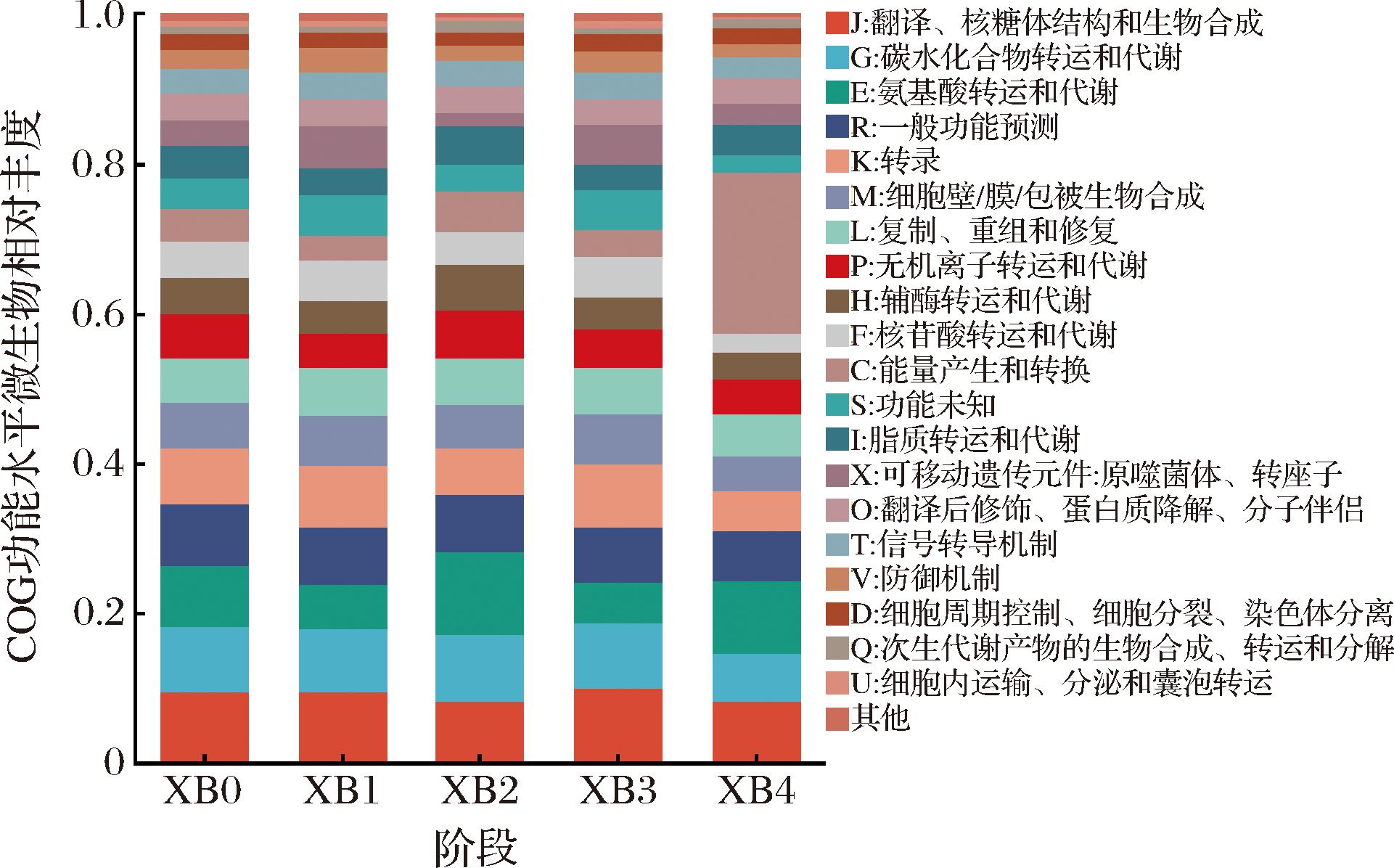
图5 风干贮藏过程中低钠腊肉的COG功能水平微生物相对丰度
Fig.5 Microbial relative abundance on COG function level in low-sodium cured meat during air-dried storage
J类(翻译、核糖体结构和生物合成)代谢通路持续高占比(>8%),这表明微生物通过增强核糖体RNA聚合酶活性维持蛋白质合成的效率,这与腊肉贮藏中微生物应对渗透压应激的适应性进化相关[24]。而G类(碳水化合物转运和代谢)和E类(氨基酸转运和代谢)代谢通路成为主要通路,主要是因为糖类和蛋白质是微生物的关键营养来源。微生物在利用这些营养物质时会引发一系列的转运和代谢反应,这些过程也是腊肉风味物质产生的主要来源。
值得注意的是,在第4个月,能量转换功能的占比骤增至21.49%。这一变化的机制可能涉及电子传递链复合酶活性的激增以及腐败菌群(如艰难拟梭菌)通过底物水平磷酸化的方式获取能量。这些变化表明在腊肉贮藏的过程中,微生物代谢和能量产生的模式发生了重要的变化。
2.4.2 KEGG数据库功能注释分析
将测序数据映射到KEGG数据库,通过逐层解析代谢通路,可以揭示不同风干贮藏阶段低钠腊肉中微生物的功能基因分布差异以及代谢机制。图6展示了1级代谢通路分布情况,在腊肉贮藏的过程中,“新陈代谢”通路是最主要的代谢通路,前4个月的占比稳定为73.96%~79.21%,但在第4个月却急剧下降至33.03%。同时,“人类疾病”通路在贮藏前中期(第0~3个月)的占比并不高(4.14%~5.60%);而在第4个月,占比突然上升至46.77%,与致病菌(如艰难拟梭菌)的异常增殖密切相关,表明贮藏后期存在显著食品安全风险。钠盐可以通过降低食品的水分活度、在微生物细胞内外形成渗透压的方式抑制腐败菌和致病菌的生长。然而,低钠配方导致腊肉基质水分活度升高及渗透压降低,使得致病菌的细胞膜稳定性增强,致病菌更易黏附到腊肉表面形成生物膜,而生物膜可以为致病菌提供物理屏障,保护其免受脱水、pH波动及残留防腐剂的杀伤,致病菌得以繁殖,因此第4个月的“人类疾病”代谢通路占比上升。
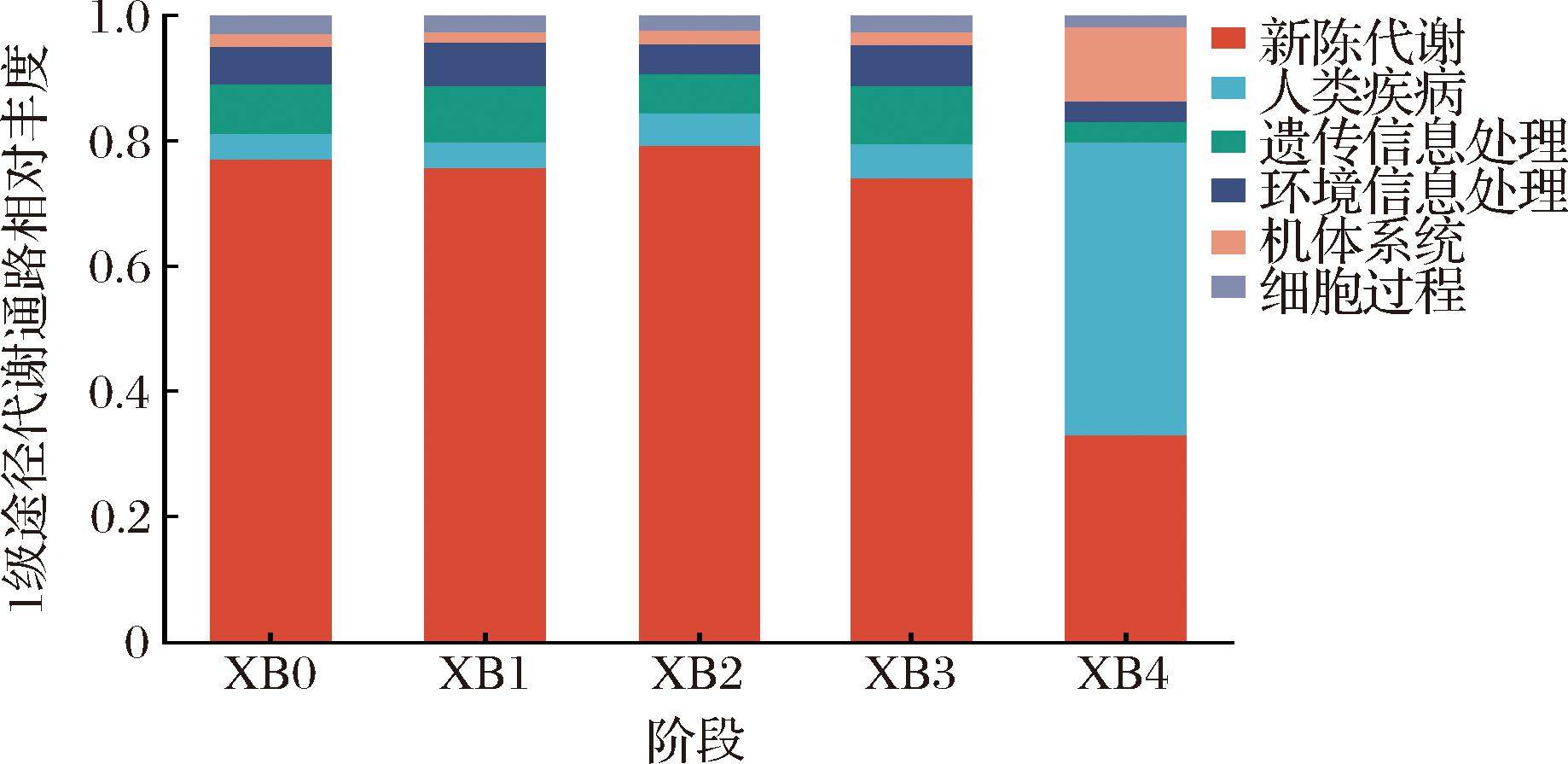
图6 风干贮藏低钠腊肉的1级代谢通路分布
Fig.6 Distribution of primary metabolic pathways in low-sodium cured meat during air-dried storage
进一步细分“新陈代谢”和“人类疾病代谢通路”,2级代谢通路如图7所示。图7-A展示了“新陈代谢”的2级通路,“全局和概览图”占据主导地位(占比51.34~52.93%),反映腊肉微生物群落的代谢网络相对稳定;其次,“碳水化合物代谢”(占比9.98%~14.49%)和“氨基酸代谢”(占比5.38%~9.48%)相对活跃。整个贮藏过程中,2级代谢通路占比相对稳定,没有很明显的变化,只有“能量代谢”在第4个月突然从初始水平的3.93%增加到18.01%,表明腐败菌群通过底物水平磷酸化加速了能量的获取。

A-新陈代谢相关途径;B-人类疾病关联途径
图7 风干贮藏低钠腊肉2级代谢通路分布
Fig.7 Distribution of secondary metabolic pathways in low-sodium cured meat during air-dried storage
图7-B显示,人类疾病的2级代谢通路中“神经退行性疾病”代谢通路在第0~3个月相对稳定(占比8.97%~25.65%),但在第4个月急剧增至64.30%,这可能与黄曲霉毒素合成基因表达增加、β-淀粉样蛋白前体蛋白编码基因丰度增加相关[25]。另外,“药物抗性:抗菌”代谢通路在贮藏前中期(第0~3个月)的占比从16.82%~29.20%急剧下降至第4个月的0.38%,而“药物抗性:抗肿瘤”通路也由贮藏前中期(第0~3个月)的5.52%~3.76%迅速下降至0.45%,表明在这一阶段低钠腊肉食品安全难以保障。
3 结论
本研究分析了低钠腊肉在风干贮藏过程中微生物的多样性和代谢活动。从腊肉样品中共鉴定出7界、15门、28纲、46目、75科、121属和412种的微生物。结果显示,贮藏中期(第2个月)呈现最高物种丰富度指数(Chao指数为65.67),而贮藏末期(第4个月)微生物多样性指数(Shannon指数为1.85)达峰值。低钠腊肉中优势菌属包括广布乳杆菌属、葡萄球菌属、拟梭菌属和曲霉属,主要优势菌种有清酒广布乳杆菌、艰难拟梭菌、腐生葡萄球菌、弯曲广布乳杆菌、葡萄球菌、密歇根克雷伯氏菌、广布乳杆菌、脓肿拟分枝杆菌、科氏葡萄球菌和鸡葡萄球菌。
与eggNOG数据库的比对发现,在低钠腊肉中的主要代谢通路包括J类(翻译、核糖体结构及生物合成)、G类(碳水化合物转运及代谢)以及E类(氨基酸转运及代谢)。与KEGG数据库对比发现,1级途径的主要代谢通路为“新陈代谢”和“人类疾病”。与新陈代谢通路相关的主要2级代谢通路为“全局和概览图”、“碳水化合物代谢”和“氨基酸代谢”;而与人类疾病代谢通路相关的主要2级代谢通路为“神经退行性疾病”。在贮藏期后期,腊肉中微生物群落结构发生明显变化,致病菌开始大量繁殖,人类疾病代谢通路占比迅速上升,而“药物抗性(抗菌)”代谢通路几乎消失,导致腊肉的食用安全受到严重威胁。
本研究基于宏基因组学方法,分析了低钠腊肉在风干贮藏期间的微生物多样性,明确了优势微生物菌群,并揭示了微生物主要的代谢途径。未来的研究可以基于贮藏过程中低钠腊肉微生物群落的组成和功能特征,进一步筛选和培养关键微生物,为今后低钠腊肉配方的设计和贮藏过程中的安全质量控制提供更多的理论支持。
[1] CHEN R X, LIU X C, XIANG J Y, et al. Prospects and challenges for the application of salty and saltiness-enhancing peptides in low-sodium meat products[J]. Meat Science, 2023, 204:109261.
[2] WONG K M, CORRADINI M G, AUTIO W, et al. Sodium reduction strategies through use of meat extenders (white button mushrooms vs. textured soy) in beef patties[J]. Food Science &Nutrition, 2019, 7(2):506-518.
[3] 范俐, 秦鑫宇, 周聪麟, 等. 闽北风吹肉制成中微生物组成及多样性变化[J]. 农产品加工, 2023(21):85-88.FAN L, QIN X Y, ZHOU C L, et al. Microbial composition and diversity in wind meat in northern Fujian[J]. Farm Products Processing, 2023(21):85-88.
[4] 左瑶, 朱联旭, 路宏朝, 等. 镇巴腊肉生产过程中微生物组成及多样性变化[J]. 肉类研究, 2023, 37(5):1-10.ZUO Y, ZHU L X, LU H Z, et al. Changes in microbial composition and diversity during the production of Zhenba bacon[J]. Meat Research, 2023, 37(5):1-10.
[5] 文开勇, 汪月, 文鹏程, 等. 四川传统腊肉中微生物群落结构研究[J]. 食品与发酵工业, 2020, 46(3):36-42.WEN K Y, WANG Y, WEN P C, et al. Study on microbial community structure in Sichuan traditional bacon[J]. Food and Fermentation Industries, 2020, 46(3):36-42.
[6] RUAN J G, WU Z C, XU J B, et al. Effects of replacement partial sodium chloride on characteristic flavor substances of bacon during storage based on GC×GC-MS and non-targeted metabolomics analyses[J]. Food Chemistry, 2023, 428:136805.
[7] 范鹏飞, 冯武, 肖瑶, 等. 基于高通量测序与培养基鉴定法分析不同省份不同养殖模式下小龙虾菌群的多样性[J]. 食品工业科技, 2023, 44(1):181-188.FAN P F, FENG W, XIAO Y, et al. Analysis of microbial diversity of crayfish under different culture modes in different provinces based on high-throughput sequencing and traditional culture methods[J]. Science and Technology of Food Industry, 2023, 44(1):181-188.
[8] DAMODARAN T, MISHRA V K, JHA S K, et al. Identification of rhizosphere bacterial diversity with promising salt tolerance, PGP traits and their exploitation for seed germination enhancement in sodic soil[J]. Agricultural Research, 2019, 8(1):36-43.
[9] ZHOU X Y, LIAN P Q, LIU H, et al. Causal associations between gut microbiota and different types of dyslipidemia: A two-sample mendelian randomization study[J]. Nutrients, 2023, 15(20):4445.
[10] DE SOUZA L M D, OGAKI M B, TEIXEIRA E A A, et al. Communities of culturable freshwater fungi present in Antarctic lakes and detection of their low-temperature-active enzymes[J]. Brazilian Journal of Microbiology, 2023, 54(3):1923-1933.
[11] AL-MADBOLY L A, YAGI A, KABBASH A, et al. Microbiota-derived short chain fatty acids in fermented Kidachi Aloe promote antimicrobial, anticancer, and immunomodulatory activities[J]. BMC Microbiology, 2023, 23(1):240.
[12] LU Y T, ZHANG P J, LI W, et al. Comparison of gut microbial communities, free amino acids or fatty acids contents in the muscle of wild Aristichthys nobilis from Xinlicheng reservoir and Chagan lake[J]. BMC Microbiology, 2022, 22(1):32.
[13] ZHU L, HUANG C H, LI L P, et al. Innovative insights into organic nitrogen degradation through protein family domains analysis in chicken and pig manure composting using metagenomic sequencing[J]. Bioresource Technology, 2024, 406:131048.
[14] 崔文明, 杨书婷, 张秋会, 等. 信阳黑猪腊肉的微生物多样性分析及优势菌分离鉴定[J]. 食品安全质量检测学报, 2023, 14(14):59-67.CUI W M, YANG S T, ZHANG Q H, et al. Microbial diversity analysis and dominant microorganisms identification and isolation of Xinyang black pig bacon[J]. Journal of Food Safety &Quality, 2023, 14(14):59-67.
[15] 周莹, 王兆明, 涂健, 等. 基于16S rRNA的徽派腊肉加工过程中微生物群落结构分析[J]. 肉类研究, 2021, 35(3):1-7.ZHOU Y, WANG Z M, TU J, et al. Analysis of microbial community structure during the processing of Hui-style bacon by 16S rRNA sequencing[J]. Meat Research, 2021, 35(3):1-7.
[16] 赵睿, 邵长春, 高世功, 等. 高通量测序分析不同腌腊肉制品细菌多样性[J]. 食品科学, 2020, 41(20):90-96.ZHAO R, SHAO C C, GAO S G, et al. High-throughput sequencing analysis of bacterial diversity of different types of traditional Chinese bacon[J]. Food Science, 2020, 41(20):90-96.
[17] 熊力, 易浪波, 粟桂蓉, 等. 基于宏基因组学分析湘西腊肉的细菌多样性[J]. 中国微生态学杂志, 2022, 34(1):23-28.XIONG L, YI L B, SU G R, et al. Diversity of bacteria in Western Hu’nan bacon based on metagenomic analysis[J]. Chinese Journal of Microecology, 2022, 34(1):23-28.
[18] 赵敏, 欧阳紫柔, 赵建宏. 艰难拟梭菌毒素致病机制及毒素基因调控的研究进展[J]. 微生物学报, 2023, 63(8):2935-2947. ZHAO M, OUYANG Z R, ZHAO J H. Advances in pathogenesis of Clostridioides difficile and regulation of toxin gene expression[J]. Acta Microbiologica Sinica, 2023, 63(8):2935-2947.
[19] 祁乐乐. 发酵肉制品中的微生物发酵剂及其作用[J]. 现代食品, 2016, 22(7):127-128.QI L L. The function of microorganisms in fermented meat products[J]. Modern Food, 2016, 22(7):127-128.
[20] 王炜强, 孙浩云, 杨志松. 动物园梅花鹿源肺炎克雷伯氏菌的分离鉴定及耐药性[J]. 野生动物学报, 2022, 43(1):220-223. WANG W Q, SUN H Y, YANG Z S. Isolation, identification and drug resistance of Klebsiella pneumoniae isolated from Sika deer (Cervus nippon) in zoo[J]. Chinese Journal of Wildlife, 2022, 43(1):220-223.
[21] LI Z J, LI Y H, XIAO C G, et al. Genomic and metabolic features of the Lactobacillus sakei JD10 revealed potential probiotic traits[J]. Microbiological Research, 2022, 256:126954.
[22] LIU R, MA Y, CHEN L, et al. Effects of the addition of leucine on flavor and quality of sausage fermented by Lactobacillus fermentum YZU-06 and Staphylococcus saprophyticus CGMCC 3475[J]. Frontiers in Microbiology, 2022, 13:1118907.
[23] FLOCK G, YIN H B, CHEN C H, et al. Survivability of Clostridioides difficile spores in fermented pork summer sausage during refrigerated storage[J]. Veterinary World, 2022, 15(1):162-167.
[24] 彭铭烨, 黄婷, 张晓娟, 等. 比较基因组学解析谷物醋醋醅中巴氏醋杆菌和欧洲驹形杆菌的功能差异[J]. 微生物学报, 2023, 63(2):638-655.PENG M Y, HUANG T, ZHANG X J, et al. Comparative genomics reveals the functional differences between Acetobacter pasteurianus and Komagataeibacter europaeus in vinegar Pei of Zhenjiang aromatic vinegar[J]. Acta Microbiologica Sinica, 2023, 63(2):638-655.
[25] MAKHLOUF M M M. Histological and ultrastructural study of AflatoxinB1 induced neurotoxicity in Sciatic nerve of adult male Albino rats[J]. Ultrastructural Pathology, 2020, 44(1):52-60.