健康的饮食和生活方式已成为人们追求的重要目标[1]。然而,摄入过多的高热量糖分,如蔗糖、果糖等,可能会导致诸多健康问题,包括肥胖、糖尿病等[2-3]。因此,寻找一种甜度高、热量低、安全性好的天然甜味剂成为了人们关注的焦点[4-5]。
莱鲍迪苷M(rebaudioside M, Reb M)作为一种天然甜味剂,因其更高的甜度和较少的回苦味以及近似蔗糖的口感等特点,在众多天然甜味剂中脱颖而出[6-9]。莱鲍迪苷M源自植物甜叶菊(Stevia rebaudiana Bertoni),这种南美灌木属于菊科泽兰属,原产于巴拉圭和巴西边境的高山草甸,也被称为“巴拉圭草”。在当地,甜叶菊叶子的使用作为天然甜味剂已有数百年历史[9]。
早期,莱鲍迪苷M主要是从植物甜叶菊中提取,但其在甜叶菊干叶中的占比低于1%,这种方式成本高且难以满足市场需求[10-12]。因此,寻找一种高效、低成本的合成方法成为研究的热点。目前,莱鲍迪苷M的主要合成方法包括化学法、发酵法和生物酶法。例如,GUO等[9]基于 UGT76G1 的蛋白质结构,通过分子对接分析与Reb D可能有相互作用的氨基酸残基,并选取这些氨基酸残基位点以UGT76G1-T284S作为出发酶进行丙氨酸扫描、定点饱和突变和组合突变,得到催化活性是UGT76G1-T284S的2.38倍的突变体UGT76G1-T284S/M88L/L200A,利用突变酶UGT76G1-T284S/M88L/L200A与拟南芥来源的蔗糖合酶AtSuSy 构建级联反应,并通过优化级联反应实现了以90.5%的产率制备出23.37 g/L 莱鲍迪苷M。WANG等[13]研究了在毕赤酵母中进行莱鲍迪苷M全细胞催化合成,探究了酶催化、混合酶反应和混合细胞反应对合成的影响,结果表明混合酶催化效果最好,可以在96 h内以95%的转化率合成0.24 mmol/L的莱鲍迪苷M。然而,化学法步骤繁琐、条件复杂;发酵法存在周期长、底物转化率低及纯化工艺复杂等不足;酶法合成虽然效率较高,但工作量较大,酶分离纯化繁琐[11]。本文利用目前表现最优的突变体UGT76G1(突变T284S M88L L200A)和UGT91C1(突变F208M F379A),采用的全细胞催化法合成莱鲍迪苷M,这是一种介于生物发酵合成和酶法催化之间的生物催化技术。该方法先在底盘细胞内积累酶量,后续收集菌体用于催化,操作简便,省去了繁琐的酶纯化步骤,且相较化学法更为可控[14]。
本研究通过全细胞催化法合成莱鲍迪苷M,利用糖基转移酶 UGT76G1和UGT91C1以及蔗糖合成酶atuSUS1搭建多酶级联反应体系,使价格较为低廉的莱鲍迪苷A先经UGT91C1催化为莱鲍迪苷D,再经UGT76G1催化产生莱鲍迪苷M,避免直接用昂贵且稀有的莱鲍迪苷D作为合成莱鲍迪苷M的底物,使原料更易获取。该过程中atuSUS1介导的二磷酸尿苷葡糖(uridine diphosphate glucose,UDPG)循环反应为反应提供了额外的辅因子UDPG,在一定程度上不仅提高了合成效率,降低了成本,而且为天然甜味剂的生产提供了一种新的思路。此外,通过添加促溶标签来提高蛋白可溶性,增加了表达量。后续通过发酵条件优化,得到的最优菌株在最优发酵条件下催化24 h后莱鲍迪苷M的产量可达到24.318 g/L。这种方法的成功应用,将为人们的健康饮食提供更多选择,也将为天然甜味剂的市场需求提供有力保障。
1 材料与方法
1.1 实验材料
1.1.1 菌株及质粒
本实验所涉及的菌株及质粒信息见表1。
表1 本研究所用菌株及质粒
Table 1 Strains and plasmids used in this study

菌株或质粒描述来源E.coli JM109克隆菌株实验室保藏E.coli BL21(DE3)表达菌株实验室保藏B-01BL21(DE3)-UGT76G1(T284S M88L L200A)-UGT91C1(F208M F379A)+atuSUS1(下面简写为BL21-76G1-91C1+SUS1)本研究构建B-02BL21-XXA-76G1-91C1+SUS1本研究构建B-03BL21-76G1-XXA-91C1+SUS1本研究构建B-04BL21-XXA-76G1-XXA-91C1+SUS1本研究构建B-05BL21(ΔaraA)-XXA-76G1-91C1+SUS1本研究构建B-06BL21(ΔaraAΔushA)-XXA-76G1-91C1+SUS1本研究构建B-07BL21(ΔaraAΔushA)-XXA-76G1-91C1+SUS1-galU本研究构建pET-28af1 ori, lacI, T7 promoter, kanR实验室保藏pBAD-oriP15Ap15A ori, lacI, araBAD, AmpR实验室保藏pKD46λRed homologous recombination method—assistedGenome editing in E.coli实验室保藏pKIKOarsBKmλRed homologous recombination method—assistedGenome editing in E.coli实验室保藏pCP20λRed homologous recombination method—assistedGenome editing in E.coli实验室保藏
1.1.2 培养基
LB培养基(g/L):蛋白胨 10,酵母提取物 5,NaCl 10。
全细胞催化反应液(g/L):柠檬酸三钠 23.5,蔗糖 500,Na2HPO4·12H2O 33.4,NaH2PO4 1.1,EDTA 1.0,AEO9(表面活性剂) 2.0,Reb A(纯度97%) 30.0。
SOB培养基(g/L):蛋白胨 10,酵母粉 5,NaCl 0.58,1 mol/L MgCl2 10 mL,KCl 0.19,1 mol/L MgSO4 10 mL。
M9培养基(g/L):Na2HPO4 6.78,KH2PO4 3,NaCl 0.5,NH4Cl 1,MgSO4 0.241,CaCl2 0.011,葡萄糖 4。
TB培养基(g/L):胰蛋白胨 11.8,酵母提取物 23.6,K2HPO4 9.4, KH2PO4 2.2,甘油 4 mL/L。
复合培养基MT9(g/L):葡萄糖 5,胰蛋白胨 11.8,酵母提取物 23.6,K2HPO4 9.4, KH2PO4 2.2。
1.2 实验方法
1.2.1 本实验引物序列
实验所使用的引物均由苏州金唯智生物科技有限公司合成,见表2。
表2 本实验中所用引物
Table 2 Primers used in this experiment

引物序列(5′-3′)76G1-pET-FCAAATATGTATCCGCTCATGAATTAATTCTTAGAAAAAC76G1-pET-RCAAATAGGGGTTCCGCGCAC91C1-FGTGCGCGGAACCCCTATTTGTAATACGACTCACTATAGGGGAATTGTGAGCG91C1-RCATGAGCGGATACATATTTGCAAAAAACCCCTCAAGACCCGTTTV-91C1-FCGTTCTTTAATAGTGGACTCTTGTTCCAAACV-91C1-RCGGTGAGTTTTCTCCTTCATTACAGAAADaraA-FATCCACGCTTCGGAAGCAGTTGGCAGATCCGGTTGCGCTTTCCACAGCGCTAATCGTAGGCTGGAGCTGCTTDaraA-RGATGAGTACGAAAGCTGCTACACCATGACGCCTGCCACACAAATCCACGGCAATACCGCATGCAATCATGGTCTATATGAATV-araA-FGTGATTTCAATGTCGTGCATCTCGGV-araA-RTGGCGATAACATGCGTGAAGTGDushA-FACAAAATTACAGTTCTGCATACCAATGATCATCATGGGCATTTTTGGCGCTAATCGTAGGCTGGAGCTGCTTDushA-RAAGGTCGTTCAGTTTGCCGTCTTTCGCCACAAAGCTAACGTTGGCAAATTTACCGCATGCAATCATGGTCTATATGAATV-ushA-FATGAAATTATTGCAGCGGGGCV-ushA-RTTACTGCCAGCTCACCTCACC
续表2

引物序列(5′-3′)SUS1-FATAAAACAGAATTTGCCTGGCGGCSUS1-RCAGACCGCTTCTGCGTTCTGgalU-FCAGAACGCAGAAGCGGTCTGAGGAGGAATTAACCATGGCTGCCATTAATACGAAAGTCAAAAAAgalU-RCCAGGCAAATTCTGTTTTATTTACTTCTTAATGCCCATCTCTTCTTCAAGCCV-galU-FACGCATTGAAGTACCGCCCV-galU-RTCTTTCGACTGAGCCTTTCGTTTTATTTGUGT76G1-FATCTCGATCCCGCGAAATTAATACGUGT76G1-RCTCGATCCTCTACGCCGGACXXA-76G1-FGTCCGGCGTAGAGGATCGAGGGATCCCCTAAATTACGCGACGXXA-76G1-RTAATTTCGCGGGATCGAGATTCGAGTGCGGCCGCAAV-XXA-76G1-FATCTTCCCCATCGGTGATGTCGV-XXA-76G1-RATTATTTCTAGAGGGGAATTGTTATCCGCUGT91C1-FTATACCATGGATTCTGGATACAGCAGTAGTTUGT91C1-RATCTCGATCCCGCGAAATTAATACGXXA-91C1-FgccgcgcggcagccatatggctagcATGCCTAAATTACGCGACGCCGCXXA-91C1-RCATTTGGTCTTCACTTAATGCGTCCTCATTTGGTCTTCACTTAATGCGTCCTV-XXA-91C1-FACAACACTCAACCCTATCTCGGTCTATV-XXA-91C1-RACGAGGGGAAATTAATAGGTTGTATTGATG
1.2.2 菌株及质粒构建
以pET-28a-UGT76G1(T284S M88L L200A)为模板,设计引物76G1-pET-F、76G1-pET-R扩增模板作为载体。以pET-28a-UGT91C1(F208M F379A)为模板,设计引物91C1-F、91C1-R,引物两端分别引入pET-28a-UGT76G1(T284S M88L L200A)模板扩增缺口上下游20 bp的碱基,进行PCR扩增,得到含有模板同源臂的目的基因。将模板与目的基因进行同源重组得到质粒pET-28a-UGT76G1(T284S M88L L200A)-UGT91C1(F208M F379A)。引物V-91C1-F、V-91C1-R用于验证同源重组是否成功。
以pKIKO为模板,设计引物DaraA-F、DaraA-R,引物两端分别引入araA基因上下游50 bp的碱基,进行PCR扩增,得到含有目的基因同源臂和卡那霉素抗性基因的敲除片段。同理,以pKIKO为模板,设计引物DushA-F、DushA-R,引物两端分别引入ushA基因上下游50 bp的碱基,进行PCR扩增,得到含有目的基因同源臂和卡那霉素抗性基因的敲除片段。将片段电转至底盘菌株BL21(DE3)进行基因敲除得到BL21(DE3)ΔaraA和BL21(DE3)ΔaraA ΔushA。V-araA-F、V-araA-R用于验证araA基因的敲除情况,V-ushA-F、V-ushA-R用于验证ushA基因的敲除情况。
以pBAD-oriP15A-atuSUS1为模板,设计引物SUS1-F、SUS1-R,进行PCR扩增,得到载体片段。以大肠杆菌基因组为模板,设计引物galU-F、galU-R,引物两端分别引入载体pBAD-oriP15A-atuSUS1基因上下游20 bp的碱基,进行PCR扩增,得到含有模板同源臂的目的基因。将模板与目的基因进行同源重组得到质粒pBAD-oriP15A-atuSUS1-galU。引物V-galU-F、V-galU-R用于验证同源重组是否成功。
以pET-28a-UGT76G1(T284S M88L L200A)-UGT91C1(F208M F379A)为模板,设计引物UGT76G1-F、UGT76G1-R,进行PCR扩增,得到载体片段。以pET-28a-XXA为模板,设计引物XXA-76G1-F、XXA-76G1-R,引物两端分别引入载体pET-28a-UGT76G1(T284S M88L L200A)-UGT91C1(F208M F379A)基因上下游20 bp的碱基,进行PCR扩增,得到含有模板同源臂的目的基因。将模板与目的基因进行同源重组得到质粒pET-28a-XXA-UGT76G1(T284S M88L L200A)-UGT91C1(F208M F379A)。引物V-XXA-76G1-F、V-XXA-76G1-R用于验证同源重组是否成功。同理以pET-28a-UGT76G1(T284S M88L L200A)-UGT91C1(F208M F379A)为模板,设计引物UGT91C11-F、UGT91C1-R,进行PCR扩增,得到载体片段。以pET-28a-XXA为模板,设计引物XXA-91C1-F、XXA-91C1-R,引物两端分别引入载体pET-28a-UGT76G1 (T284S M88L L200A)-UGT91C1(F208M F379A)基因上下游20 bp的碱基,进行PCR扩增,得到含有模板同源臂的目的基因。将模板与目的基因进行同源重组得到质粒pET-28a-UGT76G1(T284S M88L L200A)-XXA-UGT91C1(F208M F379A)。引物V-XXA-91C1-F、V-XXA-91C1-R用于验证同源重组是否成功。
1.2.3 摇瓶培养
大肠杆菌重组菌株在37 ℃下以200 r/min的转速在LB培养基中生长12 h,培养基中根据需要补充相关抗生素(100 mg/L卡那霉素或100 mg/L氨苄青霉素)。后续按培养基体积1%的接种量至LB培养基,在37 ℃下以200 r/min培养至对数生长期,加入终质量浓度15 g/L的L-阿拉伯糖和终浓度0.1 mmol/L的IPTG诱导后,在18 ℃下诱导14 h后,10 000 r/min离心收菌,随后用全细胞催化反应液重悬菌体至OD600=30.0,后续在42 ℃下反应,取样检测。
1.2.4 电转感受态制备
菌株接种5 mL LB在30 ℃下过夜培养,以OD600=0.05接种20 mL SOB培养基,培养至OD600=0.15,添加200 μL的L-阿拉伯糖(1 mol/L储存液)以诱导pKD46 λ-red表达,继续培养细胞直到OD600达到大约0.5开始制备电转化感受态细胞。离心,弃去上清液,用10 mL预冷的无菌ddH2O重悬,离心,弃上清液,用40 mL预冷的无菌体积分数为10%的甘油重悬,重复2次,弃去上清液,加入200 μL预冷的无菌体积分数为10%甘油溶液,重悬菌体。
1.2.5 电转化
取出感受态细胞,在冰浴中融化。加入待转化的DNA 1~2 μL,冰浴3 min;将上述混合物转入预冷的0.2 cm电转杯中,冰浴中预冷10 min;打开电转仪,将参数设定为2.5 kV,200/25 μF,电击时间约为5 ms;从冰中取出电转杯,用纸巾吸去表面的水分,放入样品槽中;电击后立即加入 1 mL SOB培养基,30 ℃、200 r/min 培养1.5 h后将转化后的感受态细胞涂布在含有相应抗生素的LB平板上于30 ℃培养。扩增片段转化含有pKD46 的感受态细胞时,电转化后的培养条件为30 ℃培养1.5 h,然后升温至42 ℃培养12~16 h以去除pKD46;利用pCP20消除转化子中卡那霉素片段时,电转化后的培养条件为30 ℃培养2 h,然后升温至42 ℃培养12~16 h以去除pCP20[15]。
1.2.6 全细胞催化
将发酵后的发酵液收集离心弃去上清液,菌体用于全细胞催化反应。基本反应体系为5 mL,菌体富集到OD600=30.0,加入23.5 g/L柠檬酸三钠,500 g/L蔗糖,33.4 g/L Na2HPO4 · 12H2O,1.1 g/L NaH2PO4,1.0 g/L EDTA,2.0 g/L AEO9(表面活性剂),30.0 g/L Reb A(纯度97%)做催化反应。催化反应条件为42 ℃,反应时间为24 h[16]。
1.2.7 发酵工艺优化
初始从甘油管中沾取重组菌液进行划线,挑取单菌落于15 mL LB液体培养基中,37 ℃、200 r/min培养12 h,按1%的接种量接种于复合培养基,于37 ℃、200 r/min 培养2 h(OD600=0.6~0.8),添加0.1 mmol IPTG和15 g/L L-阿拉伯糖,于16 ℃、200 r/min条件下培养14 h。优化条件包括培养基种类(LB、TB、M9、复合培养基MT9)、诱导温度(16、20、25、30、37 ℃)、诱导剂添加时间(2、4、5 h)、诱导时间(12、14、16 h)、IPTG浓度(0.05、0.1、0.2、0.5、1.0 mmol/L)、L-阿拉伯糖质量浓度(7.5、15、30、45、60 g/L)。
1.2.8 产物测定
高效液相色谱检测莱鲍迪苷M的条件如下:色谱柱Diamonsil Plus 5 μm C18-A 250×4.6 mm;柱温40 ℃;流动相:V(乙腈)∶V(10 mmol/L磷酸钠缓冲液)=32∶68 (pH=2.6);流速1.0 mL/min;进样量20 μL;检测波长210 nm[17]。
2 结果与分析
2.1 引入关键酶合成莱鲍迪苷M
本研究中莱鲍迪苷M的合成途径如图1所示[9]。UGT76G1(GenBank 登录号:NW_027069978.1)、UGT91C1(GenBank 登录号:NC_031990.1)和atuSUS1(GenBank 登录号:NC_003076.8)是莱鲍迪苷M生物合成途径中的关键酶。其中UGT76G1和UGT91C1负责将底物莱鲍迪苷A转化为莱鲍迪苷M,atuSUS1负责以蔗糖为底物提供途径所需要的辅因子UDPG[18]。本研究选用了2个常用的表达载体:pET-28a、pBAD-ori p15A,选取了UGT76G1和UGT91C1这2个关键酶基因的最优突变体,分别是T284S M88L L200A和F208M F379A[9]。这些突变体是通过基因工程手段获得的,旨在提高酶的活性、稳定性和特异性。
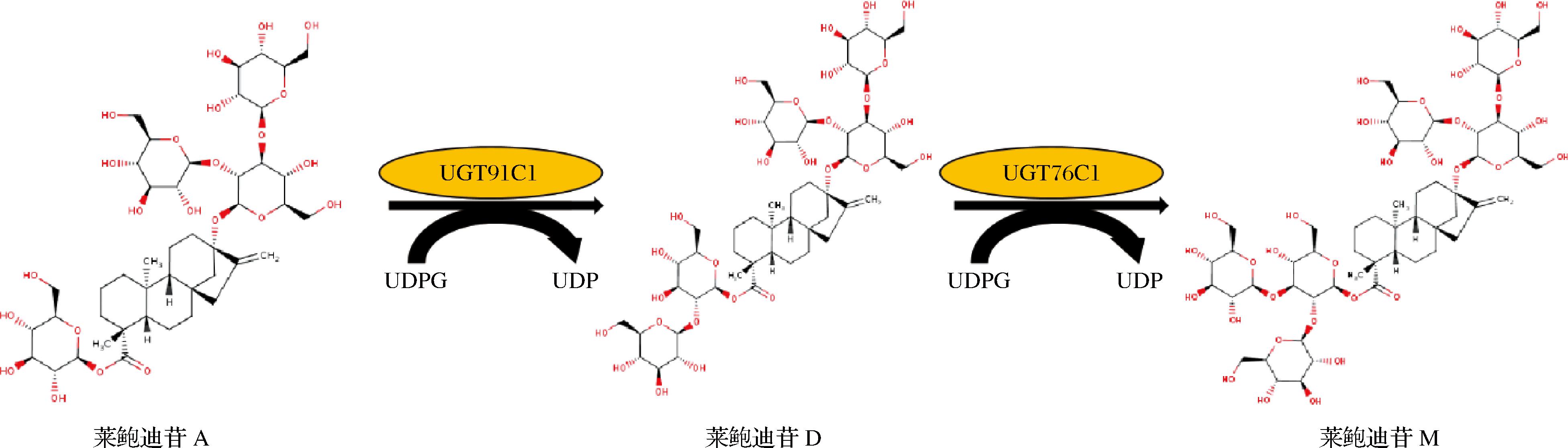
图1 UGT91C1、UGT76G1催化合成莱鲍迪苷M
Fig.1 Synthesis of Reb M catalyzed by UGT91C1 and UGT76G1
将这2个突变体基因分别插入到pET-28a载体中,并在大肠杆菌BL21(DE3)中进行表达。成功表达后,将其与含有atuSUS1基因的pBAD-ori p15A载体同时克隆到BL21(DE3)中得到初始菌株B-01进行诱导表达。atuSUS1基因编码的酶负责合成UDPG,这是莱鲍迪苷M合成途径中的重要辅因子。诱导表达后,收集菌体,将其重悬在Reb A质量浓度为30 g/L的全细胞催化反应液中,至OD600=30.0,42 ℃下进行全细胞催化。结果表明,这一途径在大肠杆菌中成功表达并合成莱鲍迪苷M。然而,检测结果显示关键酶的表达量较低,其可溶性较低,导致无法高效表达从而大量合成莱鲍迪苷M。根据文献报道,抗冻蛋白AXX的逆转录蛋白XXA是一种新型促溶标签。XXA比AXX表现出更好的稳定性且易于表达,XXA与测试的包涵体融合,与常用的可溶性融合标签(如GST、Trx、Sumo、MBP和NusA)相比,可溶性表达显着增加。为了提高蛋白表达量和产物产量,我们添加了促溶标签XXA以提高蛋白的可溶性[19]。其次,我们对表达途径进行了优化,包括调整诱导条件、优化培养基组成和辅助因子优化等,以提高蛋白表达量和酶的活性。
2.2 合成莱鲍迪苷M的整体途径优化
2.2.1 添加促溶标签提高蛋白表达量
根据之前的研究结果,发现关键酶UGT76G1和UGT91C1存在可溶性低的情况,而可溶性融合标签能帮助一些不溶性蛋白正确折叠,增强目的蛋白的溶解度。近期的研究表明,与常用的促溶性融合标签相比,XXA蛋白能显著提高目的蛋白的可溶性表达[19]。所以本研究采用此标签来提高蛋白可溶性。根据蛋白电泳结果(图2),促溶标签XXA可以极大提高蛋白的溶解度。在前一步研究的基础上,通过分别在UGT76G1前添加XXA标签、在UGT91C1前添加XXA标签以及在UGT76G1和UGT91C1前均添加标签并克隆至BL21(DE3),构建的3株菌B-02、B-03、B-04分别诱导表达后,收菌进行全细胞催化并通过高效液相色谱检测产物生成情况,结果如图3所示,B-01菌株产量为2.84 g/L,B-02菌株产量为7.04 g/L,B-03菌株产量为0.14 g/L,B-04菌株产量为4.82 g/L。因此,在UGT76G1前添加促溶标签XXA对产量提高有所帮助,后续在此基础上继续改造。
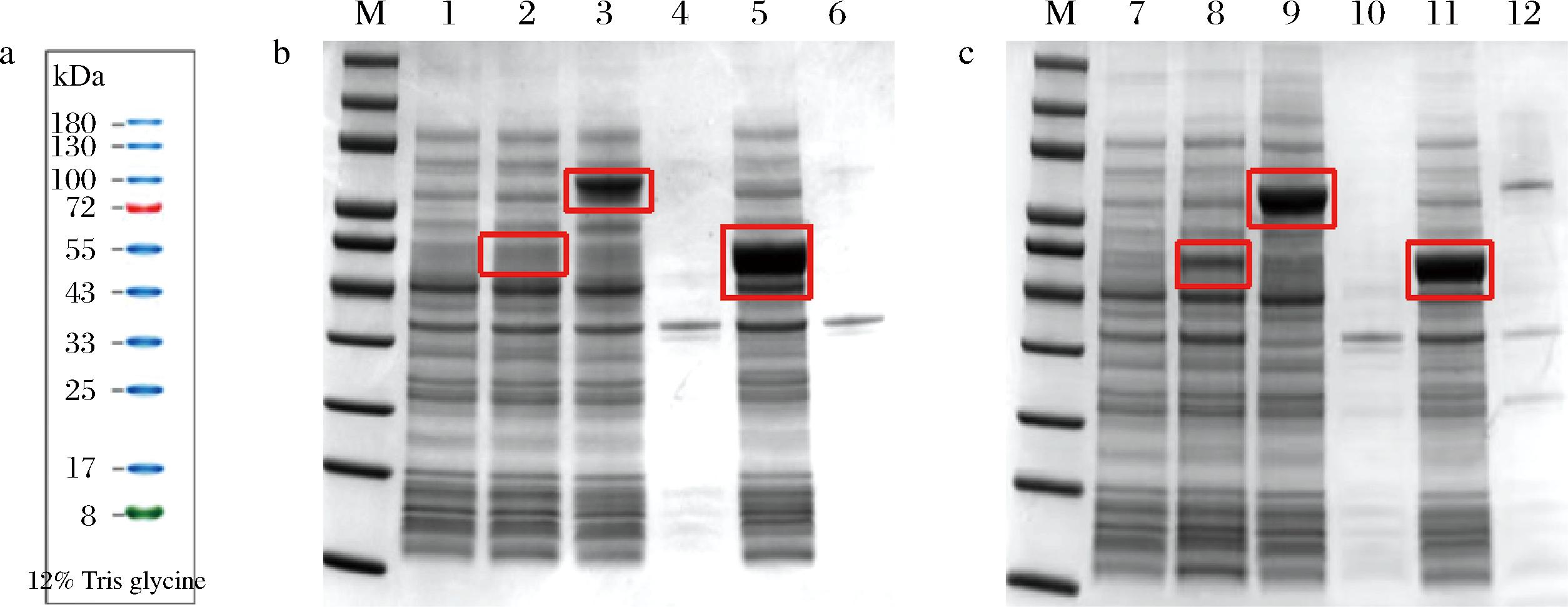
M-Marker;1-pET-28a-BL21破碎液上清液;2-UGT76G1-BL21破碎液上清液;3-XXA-UGT76G1-BL21破碎液上清液;4-pET-28a-BL21破碎液沉淀;5-UGT76G1-BL21破碎液沉淀;6-XXA-UGT76G1-BL21破碎液沉淀;7-pET-28a-BL21破碎液上清液;8-UGT91C1-BL21破碎液上清液;9- XXA-UGT91C1-BL21破碎液上清液;10-pET-28a-BL21破碎液沉淀;11-UGT91C1-BL21破碎液沉淀;12-XXA-UGT91C1-BL21破碎液沉淀。
a-预染蛋白marker;b-UGT6G1蛋白胶图;c-UGT91C1蛋白胶图
图2 重组菌蛋白表达情况
Fig.2 Expression of fusion protein of recombinant bacteria
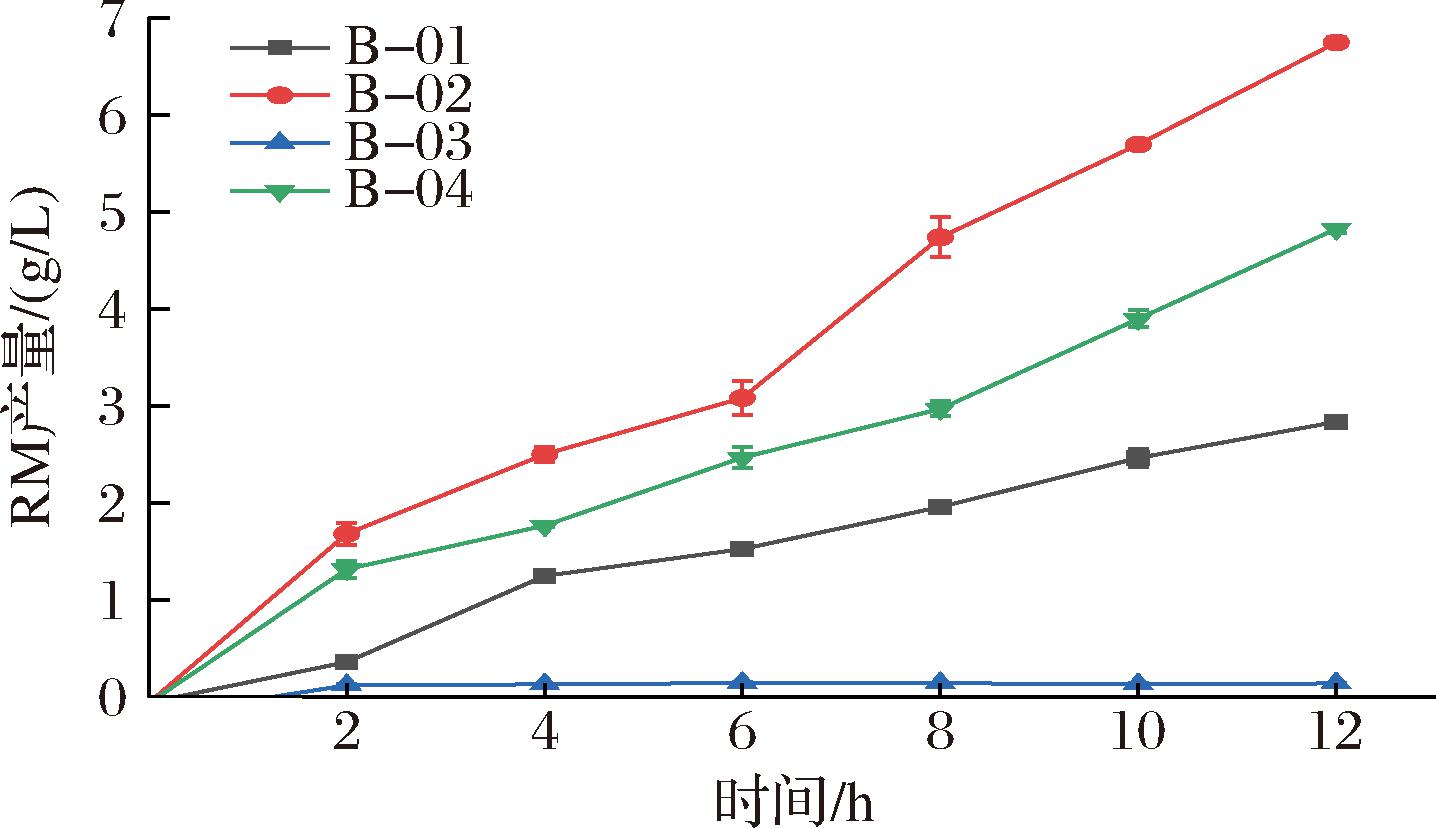
B-01-UGT76G1-UGT91C1+atuSUS1-BL21;B-02-XXA-UGT76G1-UGT91C1+atuSUS1-BL21;B-03-UGT76G1-XXA-UGT91C1+atuSUS1- BL21;B-04-XXA-UGT76G1-XXA-UGT91C1+atuSUS1-BL21。
图3 添加促溶标签XXA
Fig.3 Add the solubility-enhancing tag XXA
2.2.2 增强支路途径代谢流
UDPG是莱鲍迪苷M合成途径中的重要辅因子[20],而本研究中应用的用于合成UDPG的基因atuSUS1-pBAD ori p15A载体利用L-阿拉伯糖诱导(图4),菌体自身可以利用L-阿拉伯糖,所以通过采取敲除L-阿拉伯糖异构酶的编码基因araA得到菌株B-05,阻断阿拉伯糖利用途径,促使阿拉伯糖浓度保持在较高的水平,增强诱导效果。结果如图5所示,将araA基因敲除后,莱鲍迪苷M的产量由7.04 g/L提高到8.18 g/L,提高了16%。
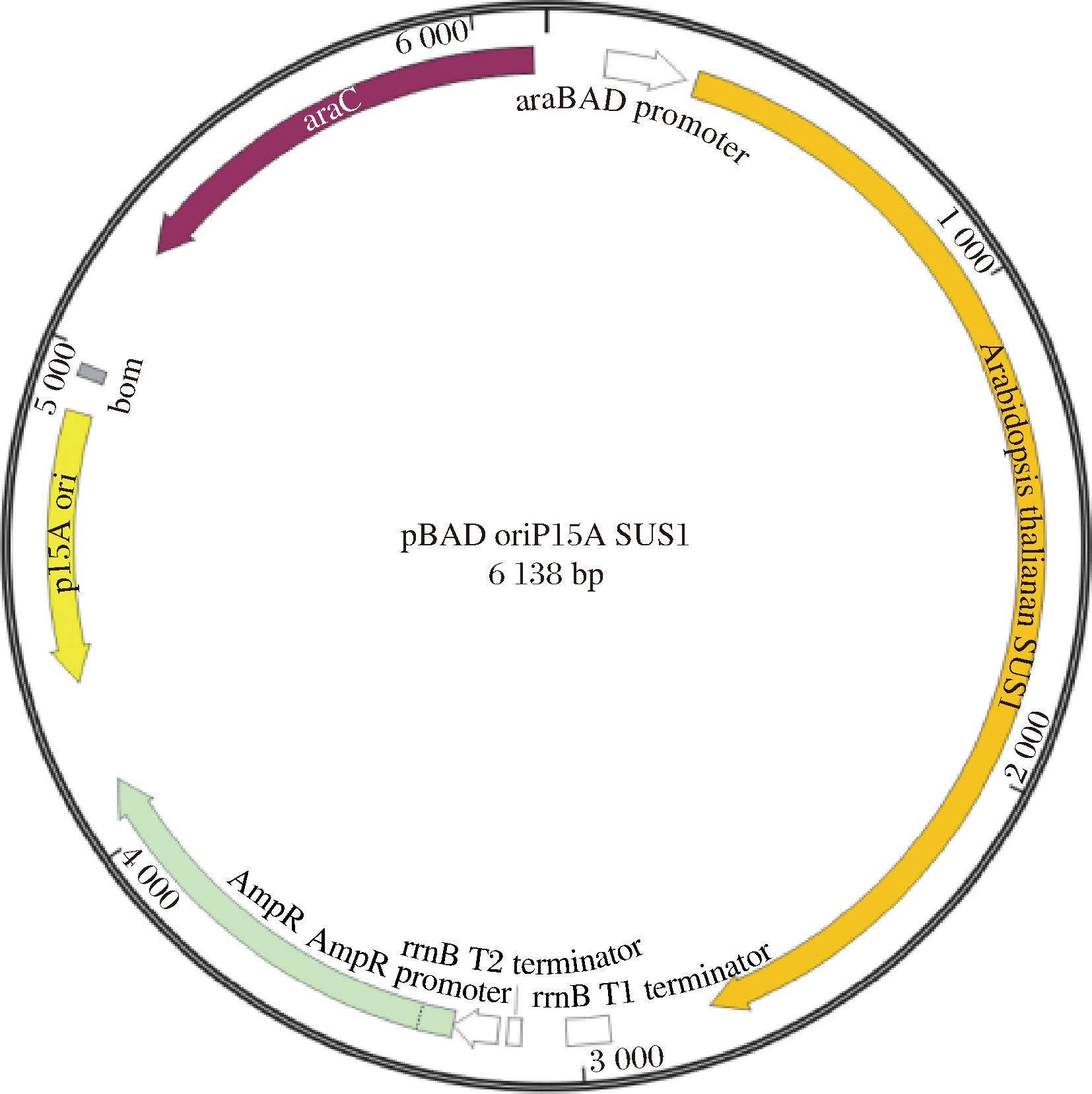
图4 atuSUS1-pBAD ori p15A
Fig.4 atuSUS1-pBAD ori p15A
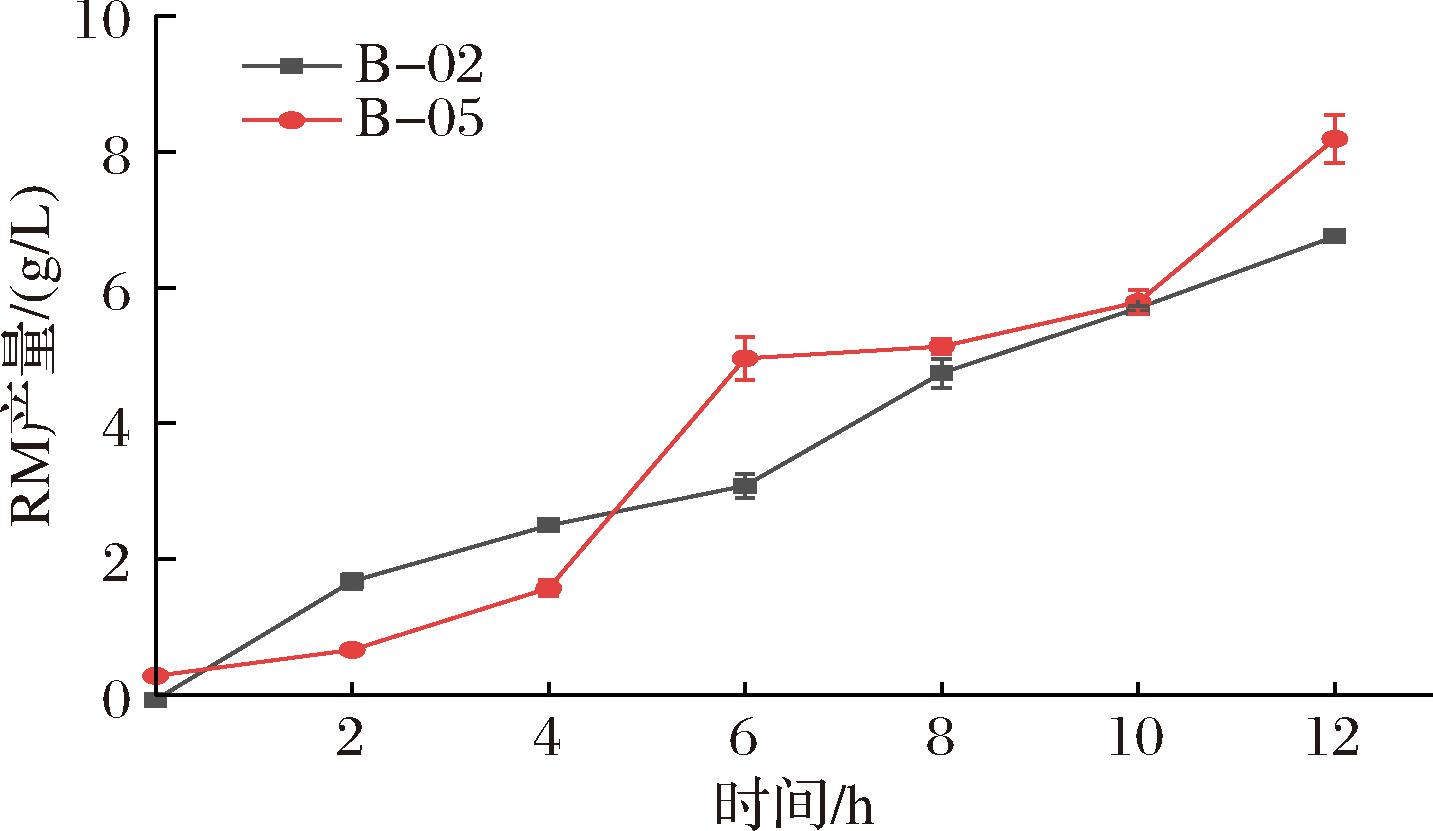
B-02-XXA-UGT76G1-UGT91C1+atuSUS1-BL21;B-05-XXA-UGT76G1-UGT91C1+atuSUS1-BL21(ΔaraA)。
图5 基因敲除araA
Fig.5 Gene knockout of araA
大肠杆菌自身存在UDPG循环(图6),根据代谢途径,可以通过敲除UDP糖水解酶的编码基因ushA得到菌株B-06,减少 UDPG 的消耗。在此基础上通过过表达葡萄糖-1-磷酸尿苷转移酶编码基因galU得到菌株B-07,促进糖酵解所产生的 6-磷酸葡萄糖向 UDPG合成及累积的方向流动[21]。在敲除ushA后,结果如图7所示,莱鲍迪苷M的产量由8.18 g/L提高到8.72 g/L,提高了6.6%。过表达galU后,结果如图8所示,莱鲍迪苷M的产量由8.72 g/L提高到9.09 g/L,提高了4.2%。根据实验结果,采用B-07菌株进行后续实验。
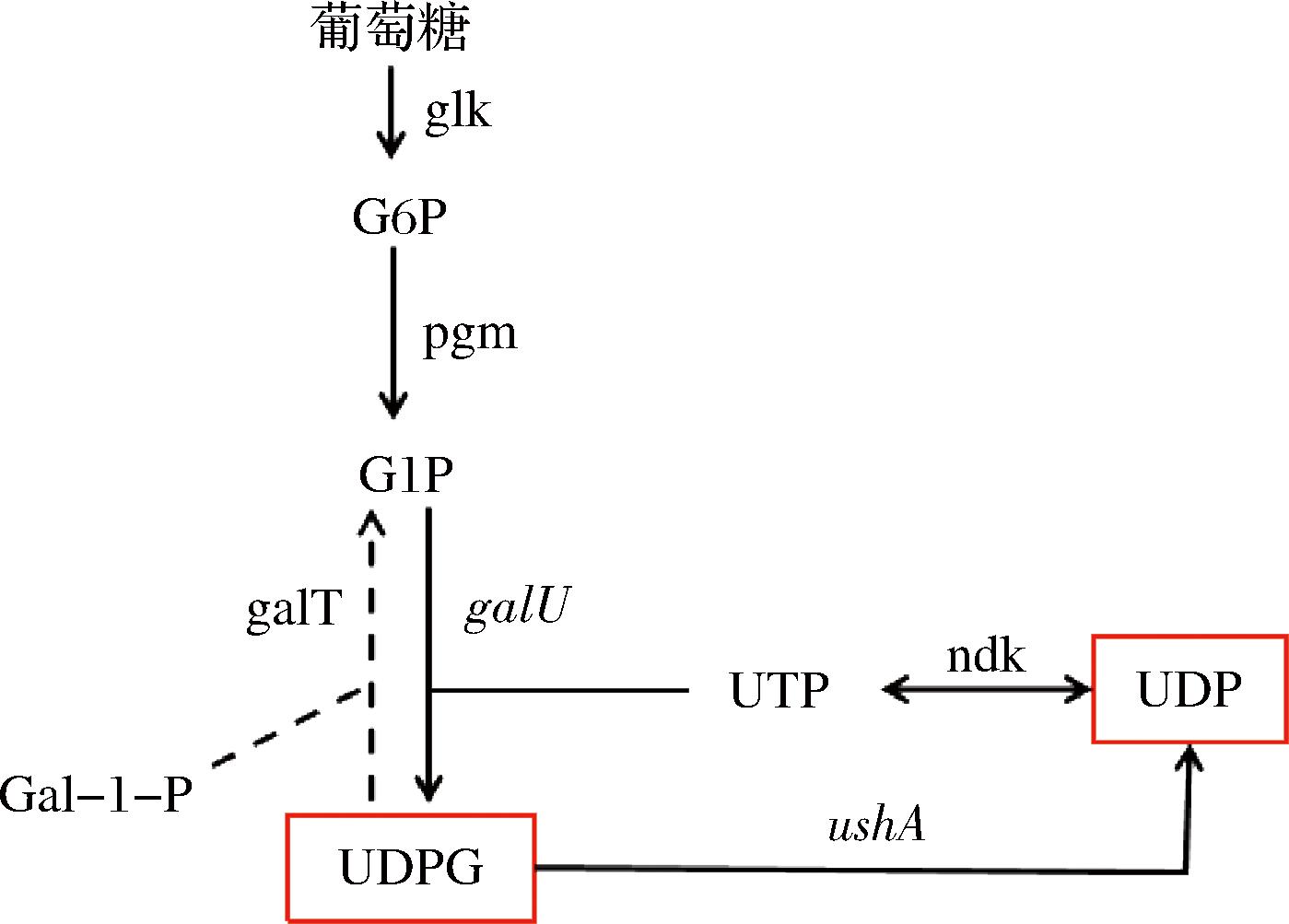
Reb A-莱鲍迪苷A;Reb D-莱鲍迪苷D;Reb M-莱鲍迪苷M;UDP-Gal-尿苷二磷酸半乳糖;UDPG-尿苷二磷酸葡萄糖;UDP-二磷酸尿苷;G1P-葡萄糖-1-磷酸;UTP-三磷酸尿苷;galE-编码UDP-半乳糖-4-差向异构酶;ushA-编码UDP糖水解酶;galU-编码葡萄糖-1-磷酸尿苷转 移酶;ndk-编码核苷二磷酸激酶。
图6 大肠杆菌中的 UDP-葡萄糖代谢途径
Fig.6 UDP-glucose metabolic pathway in E.coli
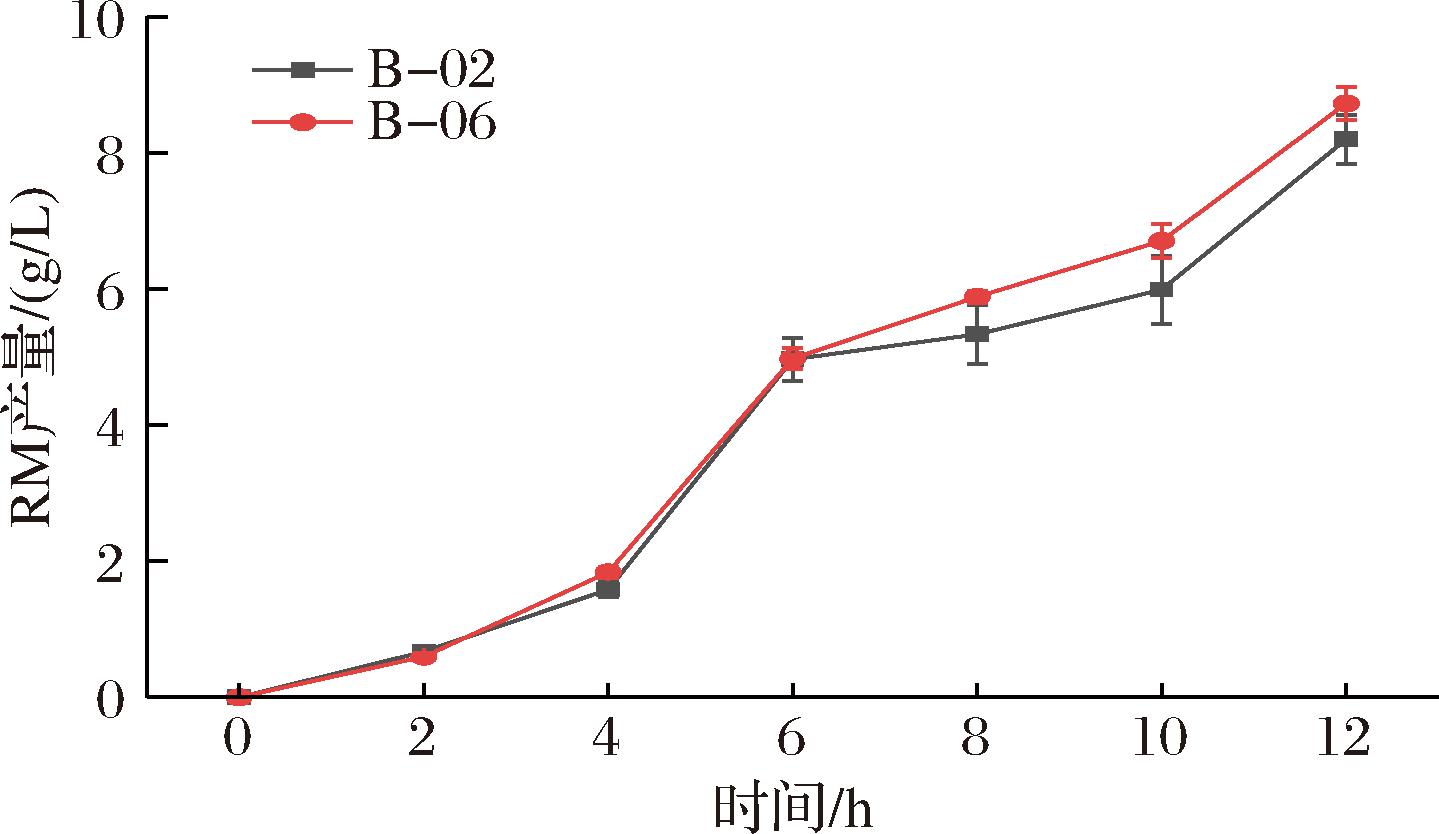
B-02-XXA-UGT76G1-UGT91C1+atuSUS1-BL21;B-06-XXA-UGT76G1-UGT91C1+atuSUS1-BL21(ΔaraA ΔushA)。
图7 基因敲除araA和ushA
Fig.7 Gene knockout of araA and ushA
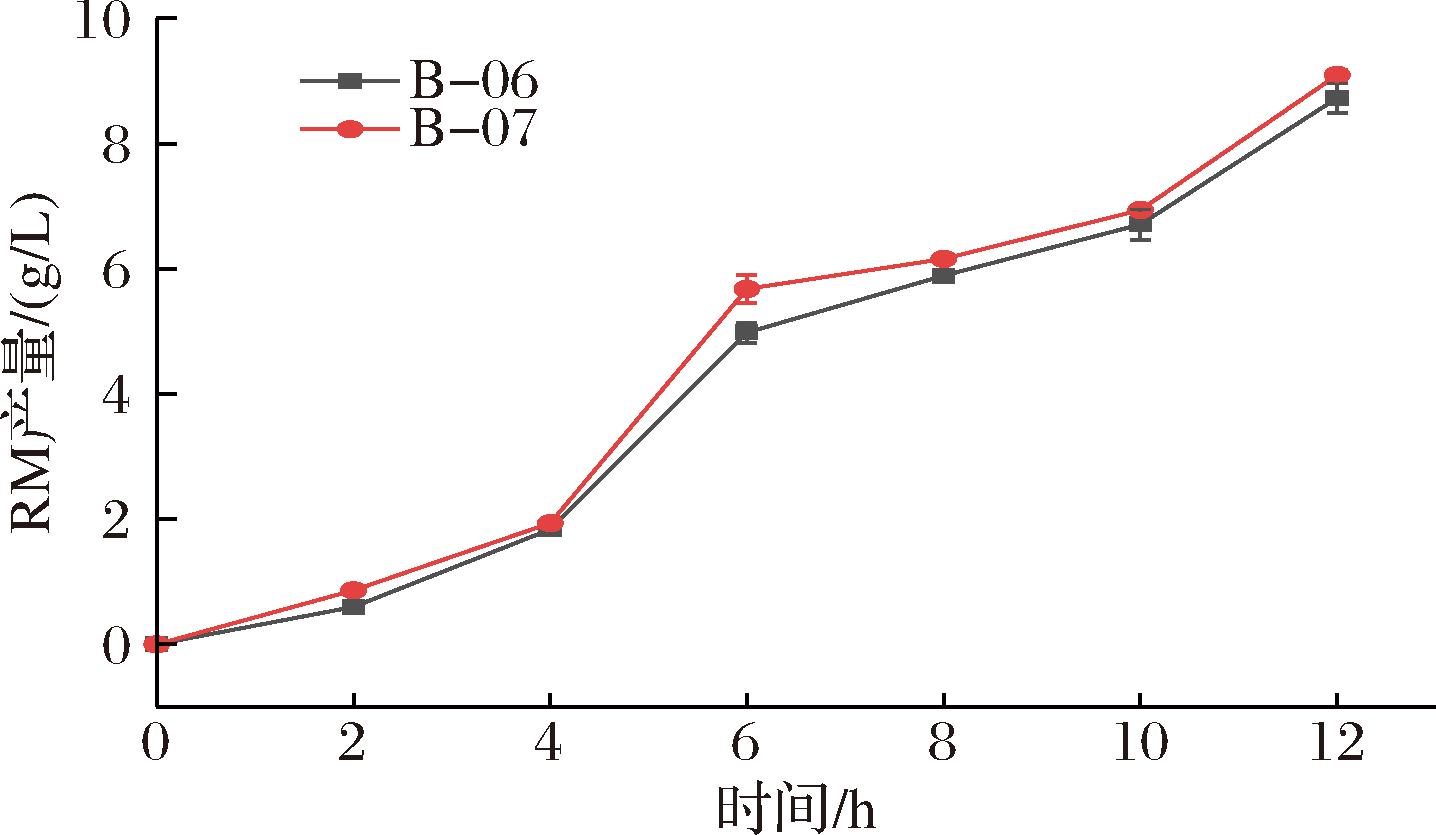
B-06-XXA-UGT76G1-UGT91C1+atuSUS1-BL21(ΔaraA ΔushA);B-07-XXA-UGT76G1-UGT91C1+atuSUS1-galU-BL21(ΔaraA ΔushA)。
图8 过表达galU
Fig.8 Overexpression of galU
2.3 发酵优化
2.3.1 重组菌BL21(ΔaraAΔushA)-XXA-76G1-91C1+pBAD-SUS1-galU生长曲线测定
将BL21(ΔaraAΔushA)-XXA-76G1-91C1+pBAD-SUS1-galU于复合培养基、37 ℃、200 r/min 培养24 h,取样测定OD600值,绘制生长曲线。如图9所示,2~4 h为对数生长前期,4~7 h为对数生长后期,7~24 h为稳定期,根据该生长曲线对重组菌进行诱导条件的优化。

图9 重组菌生长曲线
Fig.9 Growth curve of recombinant bacteria
2.3.2 培养基种类
培养基是微生物生长和代谢的基础,不同的培养基成分和种类对发酵过程有着重要的影响,因此对比了不同培养基对细胞生长和产物产量的影响。通过对比菌株在LB、TB、M9和复合培养基MT9中的产物得率以及总转化率(单位菌体量转化率×总OD600),结果如图10-a所示,在不牺牲菌体量的前提下选择复合培养基作为最适培养基。

a-培养基类型;b-温度;c-诱导时间点;d-诱导时间;e-IPTG浓度;f-L-阿拉伯糖浓度
图10 发酵工艺优化
Fig.10 The optimization of fermentation process
2.3.3 诱导温度
温度是影响细胞生长和代谢的主要因素之一,低温能够促进蛋白正确折叠,高温不仅影响质粒稳定性而且促使蛋白快速合成而发生错误折叠[22]。结果如图10-b所示,随着温度的升高,重组菌的总转化率呈现降低的趋势,莱鲍迪苷M产量也逐渐降低。16 ℃诱导时,总转化率与莱鲍迪苷M产量均最高,所以选择16 ℃为最优诱导温度。
2.3.4 诱导剂添加时间
在大肠杆菌中表达异源蛋白时,通常会影响细胞的正常生长和代谢,过早表达会给重组菌带来代谢负担,太晚诱导强度可能不够,影响细胞生长从而降低目的蛋白的产生[23]。结果如图10-c所示,诱导剂添加过早或者过晚都会影响菌株的生长和代谢,4 h添加诱导剂时重组菌生产莱鲍迪苷M的能力最优,因此选择4 h添加诱导剂。
2.3.5 诱导时间
诱导时间过短无法得到足够多的菌体用于全细胞催化,而诱导时间过长则会导致菌体老化,催化活力下降,并且增加发酵成本。结果如图10-d所示,随着诱导时间的延长,总转化率与莱鲍迪苷M产量都逐渐增多,但诱导时间太长会导致培养基营养成分不足,细胞不能完成正常代谢,结合周期和成本问题,选择诱导时间为16 h。
2.3.6 IPTG质量浓度
重组菌中所含的重组质粒pET28a-UGT76G1-UGT91C1为诱导型的T7启动子,需要IPTG诱导目的蛋白表达。IPTG 诱导浓度太低不足以结合阻遏蛋白而诱导目的蛋白的转录而降低蛋白表达,太高则会影响重组菌的生长和蛋白表达,并且IPTG 比较昂贵[24]。本研究中针对IPTG的质量浓度开展研究。结果如图10-e所示,随着诱导剂IPTG质量浓度的升高,目的蛋白的表达受到影响进而影响产物生成。从结果来看,添加终浓度0.1 mmol/L的IPTG 时莱鲍迪苷M产量可以达到14.38 g/L,并且考虑到过高质量浓度IPTG会抑制菌体生长进而影响蛋白表达,因此选择0.1 mmol/L为IPTG最适浓度。
2.3.7 L-阿拉伯糖质量浓度
重组质粒pBAD ori p15A-SUS1为诱导型的araBAD启动子,需要L-阿拉伯糖诱导目的蛋白表达。结果如图10-f所示,在一定范围内随着L-阿拉伯糖质量浓度提高,诱导效果逐渐提高但并不明显,本着节约成本的原则,选择15 g/L的L-阿拉伯糖为最适质量浓度。
结果显示,采用最优发酵条件,即利用复合培养基,加入0.1 mmol/L IPTG和15 g/L L-阿拉伯糖在16 ℃下诱导16 h,随后收集细胞进行全细胞催化,莱鲍迪苷M产量可达到24.318 g/L,转化率达到91%。
3 结论
本研究通过在大肠杆菌内共表达UGT76G1突变体T284S M88L L200A,UGT91C1突变体F208M F379A和atSUS1构建了莱鲍迪苷M合成途径,初始菌株B-01通过全细胞催化可产生2.84 g/L的莱鲍迪苷M。为了提高其产量,我们采取敲除了基因araA和ushA,过表达galU以及添加促融标签XXA等方式。通过全细胞催化结果得到最优菌株为敲除araA及ushA、过表达galU、单独在UGT76G1前添加XXA标签的BL21(DE3)菌株,莱鲍迪苷M产量为9.09 g/L,较初始菌株提高了2.2倍。同时对培养基种类、温度、诱导时间点、诱导时间、诱导剂浓度进行单因素优化并确定了最优的发酵条件:利用复合培养基,加入0.1 mmol/L IPTG和15 g/L L-阿拉伯糖在16 ℃下诱导16 h。该条件下莱鲍迪苷M产量为24.318 g/L,较初始条件提高了7.56倍。本研究深入探索了大肠杆菌在发酵体系下,积累全细胞催化合成莱鲍迪苷 M 过程中关键限速酶的能力,通过发酵优化以及辅因子优化,为莱鲍迪苷M的大量合成提供了基础,推动全细胞催化合成莱鲍迪苷M的研究进展。
[1] 廖琼. 功能性甜味剂木糖醇在食品加工中的应用[J].食品安全导刊, 2024(31):160-162.LIAO Q.Application of functional sweetener xylitol in food processing[J].China Food Safety Magazine, 2024(31):160-162.
[2] CASTRO-MU OZ R, CORREA-DELGADO M, C
OZ R, CORREA-DELGADO M, C RDOVA-ALMEIDA R, et al.Natural sweeteners:Sources, extraction and current uses in foods and food industries[J].Food Chemistry, 2022, 370:130991.
RDOVA-ALMEIDA R, et al.Natural sweeteners:Sources, extraction and current uses in foods and food industries[J].Food Chemistry, 2022, 370:130991.
[3] 刘梦雨, 何海琳, 马婷, 等.微生物合成甜菊糖的研究进展[J].食品工业科技, 2025, 46(6):387-397.LIU M Y, HE H L, MA T, et al.Progress on synthetic biology for steviol glycosides biosynthesis[J].Science and Technology of Food Industry, 2025, 46(6):387-397.
[4] SALEHI B, L PEZ M D, MART
PEZ M D, MART NEZ-L
NEZ-L PEZ S, et al.Stevia rebaudiana Bertoni bioactive effects:From in vivo to clinical trials towards future therapeutic approaches[J].Phytotherapy Research, 2019, 33(11):2904-2917.
PEZ S, et al.Stevia rebaudiana Bertoni bioactive effects:From in vivo to clinical trials towards future therapeutic approaches[J].Phytotherapy Research, 2019, 33(11):2904-2917.
[5] ANDRADE L, LEE K M, SYLVETSKY A C, et al.Low-calorie sweeteners and human health:A rapid review of systematic reviews[J].Nutrition Reviews, 2021, 79(10):1145-1164.
[6] SARAIVA A, CARRASCOSA C, RAHEEM D, et al.Natural sweeteners:The relevance of food naturalness for consumers, food security aspects, sustainability and health impacts[J].International Journal of Environmental Research and Public Health, 2020, 17(17):6285.
[7] PRAKASH I, MARKOSYAN A, BUNDERS C.Development of next generation Stevia sweetener:Rebaudioside M[J].Foods, 2014, 3(1):162-175.
[8] GOYAL S K, SAMSHER, GOYAL R K.Stevia (Stevia rebaudiana) a bio-sweetener:A review[J].International Journal of Food Sciences and Nutrition, 2010, 61(1):1-10.
[9] GUO B D, DENG Z W, MENG F, et al.Enhancement of rebaudioside M production by structure-guided engineering of glycosyltransferase UGT76G1[J].Journal of Agricultural and Food Chemistry, 2022, 70(16):5088-5094.
[10] CEUNEN S, GEUNS J M C.Steviol glycosides:Chemical diversity, metabolism, and function[J].Journal of Natural Products, 2013, 76(6):1201-1228.
[11] 郭保党, 饶义剑.新型甜味剂莱鲍迪苷D和莱鲍迪苷M的生物转化进展[J].食品与发酵工业, 2023, 49(7):289-296.GUO B D, RAO Y J.Current advances in the biotransformation of new type sweeteners Rebaudioside D and Rebaudioside M[J].Food and Fermentation Industries, 2023, 49(7):289-296.
[12] OLSSON K, CARLSEN S, SEMMLER A, et al.Microbial production of next-generation Stevia sweeteners[J].Microbial Cell Factories, 2016, 15(1):207.
[13] WANG X Q, LI J Q, LI L N, et al.Fusion glycosyltransferase design for enhanced conversion of rebaudioside A into rebaudioside M in cascade[J].Molecular Catalysis, 2023, 547:113317.
[14] RUDROFF F.Whole-cell based synthetic enzyme cascades:Light and shadow of a promising technology[J].Current Opinion in Chemical Biology, 2019, 49:84-90.
[15] 李鑫, 李亚芯, 戴建君.Red两步同源重组法在大肠杆菌基因敲除中的应用[J].中国畜牧兽医, 2017, 44(7):1934-1940.LI X, LI Y X, DAI J J.Usage of two-step red homologous recombination method to knockout the gene of Escherichia coli[J].China Animal Husbandry &Veterinary Medicine, 2017, 44(7):1934-1940.
[16] 费理文, 王勇.甜味剂莱鲍迪苷D的高效生物催化合成[J].食品与发酵工业, 2018, 44(4):1-7.FEI L W, WANG Y.Whole cell catalysis of sweetener rebaudioside D by recombinant E.coli[J].Food and Fermentation Industries, 2018, 44(4):1-7.
[17] KOLATE N S, MISHRA H, KINI S G, et al.A validated RP-HPLC method for quantification of steviol glycoside:Rebaudioside a in extracts of Stevia Rebaudiana leaf[J].Chromatographia, 2021, 84(1):21-26.
[18] GO S R, LEE S J, AHN W C, et al.Enhancing the thermostability and activity of glycosyltransferase UGT76G1 via computational design[J].Communications Chemistry, 2023, 6:265.
[19] XIE X, WU P, HUANG X C, et al.Retro-protein XXA is a remarkable solubilizing fusion tag for inclusion bodies[J].Microbial Cell Factories, 2022, 21(1):51.
[20] MA S Y, MA Y Y.A sustainable strategy for biosynthesis of Rebaudioside D using a novel glycosyltransferase of Solanum tuberosum[J].Biotechnology Journal, 2024, 19(2):2300628.
[21] 许韶华, 吴旭日, 陈依军.糖基转移酶活性供体UDP-糖的生物合成及循环再生[J].药物生物技术, 2019, 26(3):244-249.XU S H, WU X R, CHEN Y J.Biosynthesis and regeneration of UDP-sugar donor in glycosyltransferase-catalyzed glycosylation[J].Pharmaceutical Biotechnology, 2019, 26(3):244-249.
[22] YUAN S L, DUAN H Q, LIU C J, et al.The role of thioredoxin and disulfide isomerase in the expression of the snake venom thrombin-like enzyme calobin in Escherichia coli BL21 (DE3)[J].Protein Expression and Purification, 2004, 38(1):51-60.
[23] ZOU C, DUAN X G, WU J.Enhanced extracellular production of recombinant Bacillus deramificans pullulanase in Escherichia coli through induction mode optimization and a glycine feeding strategy[J].Bioresource Technology, 2014, 172:174-179.
[24] DUAN X G, CHEN J, WU J.Optimization of pullulanase production in Escherichia coli by regulation of process conditions and supplement with natural osmolytes[J].Bioresource Technology, 2013, 146:379-385.