香芹醇,亦称葛缕醇,是一种含有p-薄荷骨架的单环萜醇类化合物,存在于短梗八角、葛缕子等植物精油中。该化合物为无色至浅黄色液体,微溶于水,易溶于氯仿、乙酸乙酯等有机溶剂,具有类似薄荷与香芹的香气,是高级食品和化妆品香精的重要原料[1]。此外,药理学研究表明,香芹醇具有抗肿瘤、抗菌、抗氧化、抗炎、保肝、缓解糖尿病以及降血脂等多重生物活性[2-4],因此在医药领域也展现出广阔的应用前景。
由于其自然来源有限,香芹醇的制备主要依赖化学合成方法,但这种方法存在产物质量差、环境不友好的缺点[5]。相比之下,生物催化是利用微生物或其酶作为催化剂对底物进行催化转化,具有反应条件温和、高效以及针对性强等优势,已受到广泛的关注。据报道,以廉价且丰富的柑橘加工副产物柠檬烯为底物,利用细胞色素P450酶(CYP71、CYP102A7、CYP71D582、CYP71D18、CYP2C11、CYP2C9、P450cam突变体)对其C6位进行氧化,能够生成香芹醇[6-12]。此外,双加氧酶、葡萄糖氧化酶和辣根过氧化物酶也被报道能够催化柠檬烯氧化生成香芹醇[13-15]。然而,这些酶存在活性不高、专一性不足等问题,例如CYP450酶催化效率低,改造难度大,且反应过程中需要昂贵的辅酶参与,这在很大程度上限制了其实际应用以及大规模生产香芹醇。虽然也有利用微生物(如Rhodococcus opacus PWD4)转化柠檬烯生成香芹醇的研究[16],但这些菌株中的酶系通常较为复杂,缺乏区域特异性,导致副产物较多,且后续需要昂贵的纯化过程才能获得较高纯度的香芹醇。因此,需要进一步对柠檬烯氧化生成香芹醇的关键酶进行挖掘,丰富酶资源库,以实现香芹醇的经济高效合成。
本实验室在前期研究中筛选得到一株能够降解柠檬烯生成香芹醇的克雷伯杆菌(Klebsiella sp.O852),并对该菌株进行全基因组测序。基于此,本研究通过本地BLAST和生物信息学分析,结合多序列比对及进化树分析,对Klebsiella sp.O852催化柠檬烯生成香芹醇的关键酶进行挖掘,并以大肠杆菌为表达宿主对酶基因进行异源表达,旨在鉴定酶的功能,为香芹醇的生物合成提供酶资源。
1 材料与方法
1.1 试验材料
1.1.1 基因、引物、菌株和质粒
本实验所用到的基因均来源于Klebsiella sp.O852,经过大肠杆菌(Escherichia coli)密码子优化后,由上海生工生物有限公司合成,本实验所用到的菌株、质粒和引物见表1。
表1 本研究所使用到的菌株、质粒及引物
Table 1 Strains, plasmids, and primers used in this study

注:AmpR表示氨苄青霉素抗性。
菌株/质粒/引物性质来源Klebsiella sp.O852用于所有克隆实验实验室保存E.coli BL21 (DE3)重组蛋白表达宿主实验室保存E.coli DH5α重组蛋白表达宿主实验室保存BL21/pET32a-KlebDOX1重组E. coli BL21 (DE3),携带pET32a-KlebDOX1本研究构建BL21/pET32a-KlebDOX2重组E. coli BL21 (DE3),携带pET32a-KlebDOX2本研究构建BL21/pET32a-KlebFdR1重组E.coli BL21 (DE3),携带pET32a-KlebFdR1本研究构建BL21/pET32a-KlebFdR2重组E.coli BL21 (DE3),携带pET32a-KlebFdR2本研究构建pET32a-KlebDOX1pET32a携带来源于Klebsiella sp.O852经过密码子优化的KlebDOX1基因,AmpR本研究构建pET32a-KlebDOX2pET32a携带来源于Klebsiella sp.O852经过密码子优化的KlebDOX2基因,AmpR本研究构建pET32a-KlebFdR1pET32a携带来源于Klebsiella sp.O852经过密码子优化的KlebFdR1基因,AmpR本研究构建pET32a-KlebFdR2pET32a携带来源于Klebsiella sp.O852经过密码子优化的KlebFdR2基因,AmpR本研究构建KlebDOX1-FGCTACGCATCCGCCAGTATGAAG上海生工生物有限公司合成KlebDOX1-RGGTCGTAACGCTGCTGTTCCTG上海生工生物有限公司合成KlebDOX2-FCGGTTCGCCTGGAAGAGTATGC上海生工生物有限公司合成KlebDOX2-RGTAACGGTCAACCTGTACGGAATCC上海生工生物有限公司合成KlebFdR1-FGGAGGTGCGGCAGGTGAATTG上海生工生物有限公司合成KlebFdR1-RACGCTGGCTACCCTGTGTCTG上海生工生物有限公司合成KlebFdR2-FTCCACGACGCCGACAATAATGC上海生工生物有限公司合成KlebFdR2-RACGCCTCTCCGCCTTTATCCTC上海生工生物有限公司合成
1.1.2 主要试剂
2×Taq PCR Mix with Dye v2,爱博泰克生物科技有限公司;氨苄青霉素(ampicillin, Amp)、5×蛋白上样缓冲液、异丙基-β-D-硫代半乳糖苷(isopropyl-beta-D-thiogalactopyranoside, IPTG)、彩虹180广谱蛋白marker、BCA蛋白定量测定试剂盒,北京索莱宝科技有限公司;柠檬烯,阿法埃莎(中国)化学有限公司;香芹醇,上海麦克林生化科技股份有限公司;其他相关试剂均为市售分析纯。
1.1.3 培养基
LB液体培养基(g/L):NaCl 10,蛋白胨10,酵母浸粉5,pH自然,121 ℃高压蒸汽灭菌30 min。
LB固体培养基:LB液体培养基中添加2%(质量分数)的琼脂粉。
LB-A培养基:LB培养基中添加终质量浓度为100 μg/mL Amp。
1.2 仪器与设备
ZF-258全自动凝胶成像分析系统,上海嘉鹏科技有限公司;T100 PCR仪,Bio-Rad生命医学产品有限公司;Agilent 7890A-5975B气相色谱质谱联用仪(GC-MS),美国Agilent公司;YX280/15高压蒸汽灭菌锅,上海三申医疗器械有限公司;SW-CJ-1D型洁净工作台,上海力辰邦西仪器科技有限公司;固相微萃取纤维头(solid-phase microextraction, SPME,50/30 μm DVB/CAR/PDMS),美国Supeleo公司;BSD-TS270振荡培养箱,上海博迅医疗生物仪器股份有限公司;JY88-IIN超声波细胞破碎仪,宁波新芝生物科技股份有限公司;DZCZ-24DN型电泳仪,北京市六一机械厂;TGL-16B高速冷冻离心机,上海安亭科学仪器厂。
1.3 试验方法
1.3.1 关键酶的挖掘
为了挖掘Klebsiella sp.O852中催化柠檬烯生成香芹醇的关键酶,以Klebsiella sp.O852全基因组文库作为数据库,以已经报道过的具有催化柠檬烯生成香芹醇能力的酶序列(表2)作为对比文件,使用本地BLAST进行比对,筛选出相似度高的蛋白序列。将筛选得到的蛋白序列在NCBI上进行BLAST P分析,利用DNAMAN 9.0和MEGA 7.0进行多序列比对和进化树分析。
表2 已知能够催化柠檬烯生成香芹醇的酶
Table 2 Known enzymes involved in the conversion of limonene to carveol
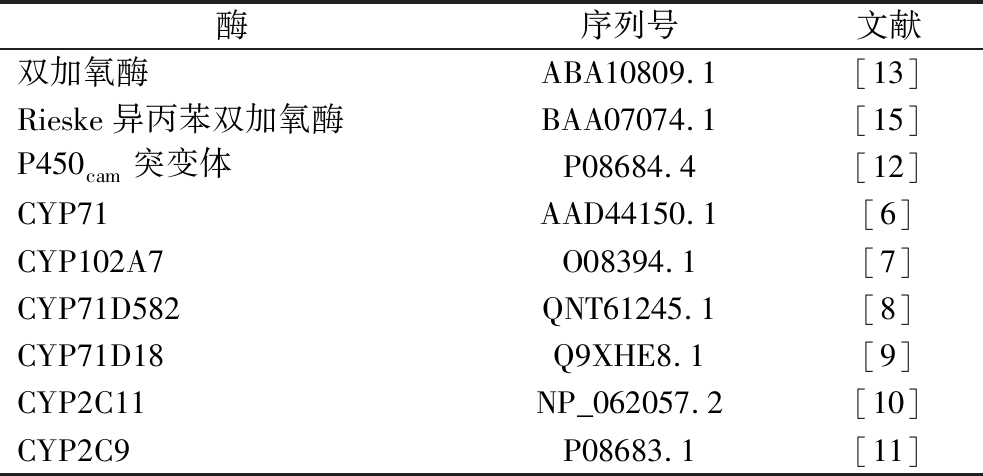
酶序列号文献双加氧酶ABA10809.1[13]Rieske异丙苯双加氧酶BAA07074.1[15]P450cam突变体P08684.4[12]CYP71AAD44150.1[6]CYP102A7O08394.1[7]CYP71D582QNT61245.1[8]CYP71D18Q9XHE8.1[9]CYP2C11NP_062057.2[10]CYP2C9P08683.1[11]
1.3.2 关键酶KlebDOX1、KlebDOX2、KlebFdR1和KlebFdR2生物信息学分析
生物信息学分析内容及网址见表3。
表3 生物信息学工具
Table 3 Bioinformatics tools
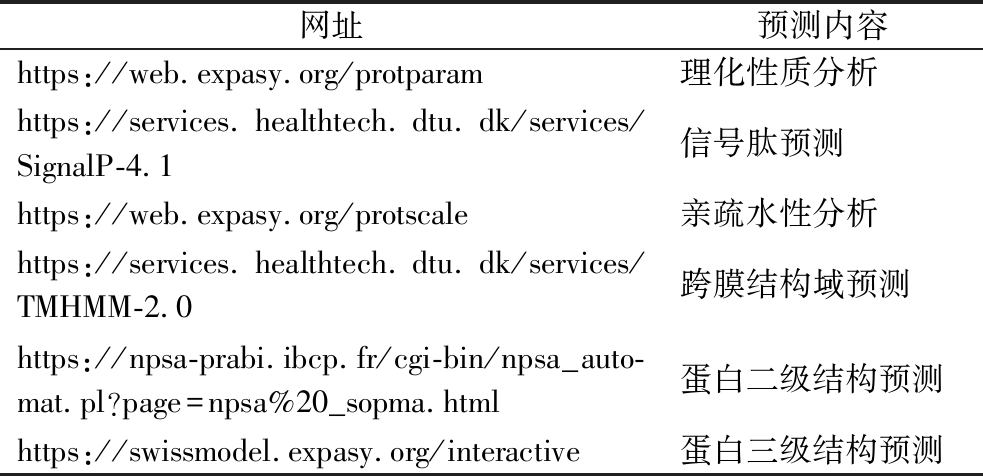
网址预测内容https://web.expasy.org/protparam理化性质分析https://services.healthtech.dtu.dk/services/SignalP-4.1信号肽预测https://web.expasy.org/protscale亲疏水性分析https://services.healthtech.dtu.dk/services/TMHMM-2.0跨膜结构域预测https://npsa-prabi.ibcp.fr/cgi-bin/npsa_auto-mat.pl?page=npsa%20_sopma.html蛋白二级结构预测https://swissmodel.expasy.org/interactive蛋白三级结构预测
1.3.3 关键酶基因克隆与表达
以菌株Klebsiella sp.O852基因组为模板,使用引物KlebDOX1-F/KlebDOX1-R、KlebDOX2-F/KlebDOX2-R、KlebFdR1-F/KlebFdR1-R、KlebFdR2-F/KlebFdR2-R进行PCR扩增,PCR扩增体系:2×Taq PCR Mix with Dye v2(12.5 μL),ddH2O(8.5 μL),上/下游引物(各1.0 μL),DNA模板(1 μL)。PCR扩增条件为:96 ℃、5 min;96 ℃、22 s,60 ℃、22 s,72 ℃、25 s,30个循环;72 ℃、1 min。经1%琼脂糖凝胶电泳后,利用试剂盒回收获取目的基因片段。
将目的基因片段和经过双酶切后的载体pET-32a进行连接,构建重组质粒pET32a-KlebDOX1、pET32a-KlebDOX2、pET32a-KlebFdR1、pET32a-KlebFdR2,并转化为E.coli DH5α感受态,菌液PCR后筛选阳性克隆子进行测序验证。
将测序成功的重组质粒转入E.coli BL21(DE3)中表达,获得重组菌株BL21/pET32a-KlebDOX1、BL21/pET32a-KlebDOX2、BL21/pET32a-KlebFdR1、BL21/pET32a-KlebFdR2。将重组菌株接种于LB-A培养基,37 ℃、200 r/min培养至OD600=0.6~0.8,加入终浓度为0.5 mmol/L的IPTG,20 ℃、100 r/min诱导表达20 h。诱导结束后,4 ℃、5 350 r/min离心20 min收集菌体,向菌体内加入适量的Tris-HCl缓冲液重悬,进行超声波破碎(功率250 W,20 min,工作3 s,间隔5 s),离心收集破碎后的上清液和沉淀。分别取未诱导和诱导后的菌体以及诱导后经过超声波破碎的上清液和沉淀,进行SDS-PAGE,分析重组蛋白的表达情况。
1.3.4 关键酶功能验证
将重组菌株BL21/pET32a-KlebDOX1、BL21/pET32a-KlebDOX2、BL21/pET32a-KlebFdR1、BL21/pET32a-KlebFdR2分别接种于含100 mL的LB-A培养基中,37 ℃、200 r/min培养至OD600=0.6~0.8时,将菌株BL21/pET32a-KlebDOX1和BL21/pET32a-KlebFdR1混合,BL21/pET32a-KlebDOX2和BL21/pET32a-KlebFdR2混合。随后,分别向混菌中添加IPTG(终浓度0.5 mmol/L)和底物柠檬烯(终浓度1 mmol/L),在30 ℃、150 r/min条件下进行转化,分别在不同转化时间点(1、2、3、4、5、6 h)取样,并通过SPME-GC-MS检测产物香芹醇的含量,以验证关键酶的功能[17]。同时设置不添加菌的对照组。
1.3.5 SPME-GC-MS检测转化产物
取5 mL反应后的菌液移入30 mL萃取瓶中,同时添加1.8 g NaCl于磁力搅拌器上40 ℃加热平衡15 min后,插入己活化好的萃取头,推出纤维头,顶空吸附40 min后,插入GC-MS进样口解析5 min。
GC条件:毛细管柱为HP-5(30 m×320 μm×0.25 μm),程序升温,起始温度40 ℃,保持3 min,以3 ℃/min升至160 ℃,保持2 min,再以8 ℃/min升至220 ℃,保持3 min。进样口温度250 ℃,检测器温度:FID 250 ℃。MS条件:离子源温度230 ℃,四极杆温度150 ℃,离子化方式EI,电子能量70 eV,质量范围为45~550 amu/sec。
使用计算机谱和香芹醇标准品进行定性分析,使用外标法进行香芹醇定量分析[18],香芹醇的标准曲线为y=32 432x+10 594,R2=0.996 8[x为香芹醇浓度(mmol/L),y为峰面积]。
1.3.6 数据处理
使用GraphPad Prism 8.0进行数据分析和绘图,使用SPSS 24.0进行单因素方差分析,采用邓肯法进行显著性分析,所有数据均为3次实验重复所得。
2 结果与分析
2.1 关键酶挖掘
利用本地BLAST,以Klebsiella sp.O852全基因组序列作为数据库,以已经报道过的具有催化柠檬烯生成香芹醇能力的酶序列作为对比文件,在Klebsiella sp.O852基因组中鉴定得到2个潜在关键酶,即O852_GE001080和O852_GE002092。其中O852_GE001080与Rieske型芳香环双加氧酶系的末端加氧酶(来自Pseudomonas sp.PWD32,序列号ABA10809.1)在蛋白水平上有30%的相似性[13],O852_GE002092与Rieske型芳香环双加氧酶系的末端加氧酶(来自P.fluorescens IP01,序列号BAA07074.1)在蛋白水平上有28%的相似性[15]。
Rieske型芳香环双加氧酶是含有Rieske [2Fe-2S] 中心的多组分酶系统,能够催化多种反应,是由末端氧化酶、还原酶和铁氧还蛋白3部分组成。其中,末端氧化酶由α亚基组成,含有一个Rieske [2Fe-2S] 中心和一个单核铁活性位点。还原酶组分是一种含有黄素腺嘌呤二核苷酸(flavin adenine dinucleotide,FAD)或黄素单核苷酸(flavin mononucleotide,FMN)的黄素蛋白,一些还原酶组分中同样也含有 [2Fe-2S] 中心。铁氧还原蛋白是含有[Fe-S] 中心的小型酸性蛋白[19]。这3部分通常是一起发挥作用的,且编码还原酶和铁氧还蛋白的基因是位于末端加氧酶α亚基编码基因的下游。在Pseudomonas sp.PWD32和P.fluorescens IP01的基因组中,均发现在编码末端加氧酶α亚基的正下游存在编码铁氧还蛋白和铁氧还蛋白还原酶的基因,且只有当3个组分共同表达时,重组菌株才能转化柠檬烯生成香芹醇。
基于此,利用SnapGene软件分析了基因O852_GE001080和O852_GE002092在Klebsiella sp.O852基因组中的位置,发现在基因O852_GE001080和O852_GE002092的正下游存在编码铁氧还蛋白和铁氧还蛋白还原酶的融合基因O852_GE001082和O852_GE002094(图1)。同时根据基因组数据对这4个基因进行功能注释(表4),结果表明基因O852_GE001080和O852_GE002092均编码双加氧酶α亚基,分别命名为KlebDOX1和KlebDOX2,基因O852_GE001082和O852_GE002094均编码还原酶组分,分别命名为KlebFdR1和KlebFdR2。综上分析,推测由基因KlebDOX1-KlebFdR1和KlebDOX2-KlebFdR2表达的双加氧酶系可能参与调控柠檬烯的氧化过程,催化柠檬烯生成香芹醇。
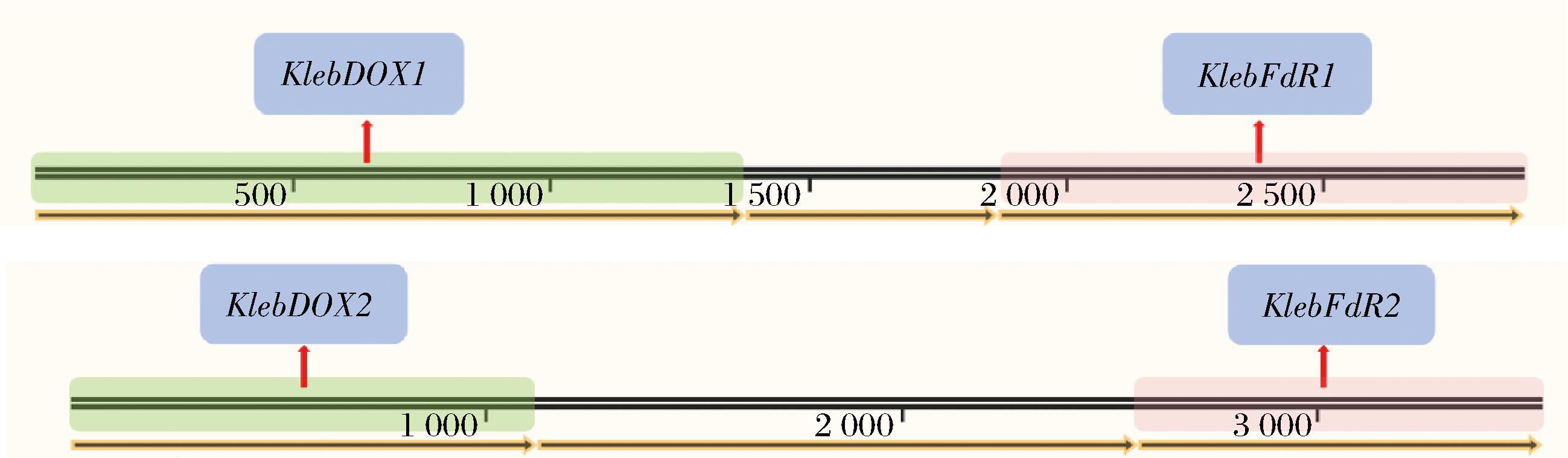
图1 编码末端氧化酶α亚基基因与编码铁氧还蛋白/铁氧还蛋白还原酶融合基因位置示意图
Fig.1 Schematic diagram of the location of the gene encoding the alpha subunit of terminal oxidase and the fusion gene encoding ferredoxin/ferredoxin reductase
表4 关键酶基因功能注释
Table 4 Functional annotation of key enzyme genes

基因 ID蛋白KEGG注释O852_GE001080苯甲酸/甲苯酸1,2-双加氧酶α亚基xylene degradation;metabolic pathways;degradation of aromatic compoundsO852_GE002092芳香环羟化双加氧酶α亚基phthalate 4,5-dioxygenase activity;2-chlorobenzoate 1,2-dioxygenase activity;degradation of aromatic compounds;2 iron, 2 sulfur cluster binding;oxidation-reduction processO852_GE001082苯甲酸/甲苯酸1,2-双加氧酶还原酶组分electron transfer activity;metal ion binding;dioxygenase activity; 2 iron, 2 sulfur cluster binding;oxidation-reduction processO852_GE002094肉碱单加氧酶还原酶electron transfer activity;metal ion binding;dioxygenase activity;2 iron, 2 sulfur cluster binding;oxidation-reduction process
2.2 关键酶多序列比对和进化树分析
从NCBI数据库中分别获得10个与KlebDOX1、KebDOX2序列相似度高的末端氧化酶氨基酸序列,以及10个与KlebFdR1、KebFdR2序列相似度高的铁氧还蛋白/铁氧还蛋白还原酶氨基酸序列。使用MEGA 7.0软件绘制系统发育树,使用邻位连接法构建进化树,自检重复设定为1 000。结果如图2-a所示,基于氨基酸序列的KlebDOX1和KebDOX2进化树分支置信度在58~100,表明该进化树可信。其中KlebDOX1与同Klebsiella属的STW43064.1位于同一分支,进而和同属的KDM20303.1汇聚为一支,这两个蛋白均被注释为双加氧酶α亚基;同理,KlebDOX2与同Klebsiella属的HDK6239874.1为同一分支,进而与同属的STU77376.1和CDL47432.1依次汇聚到同一分支上,这个蛋白同样被注释为双加氧酶,因此推测KlebDOX1和KlebDOX2基因编码的蛋白可能是双加氧酶α亚基,即末端加氧酶。
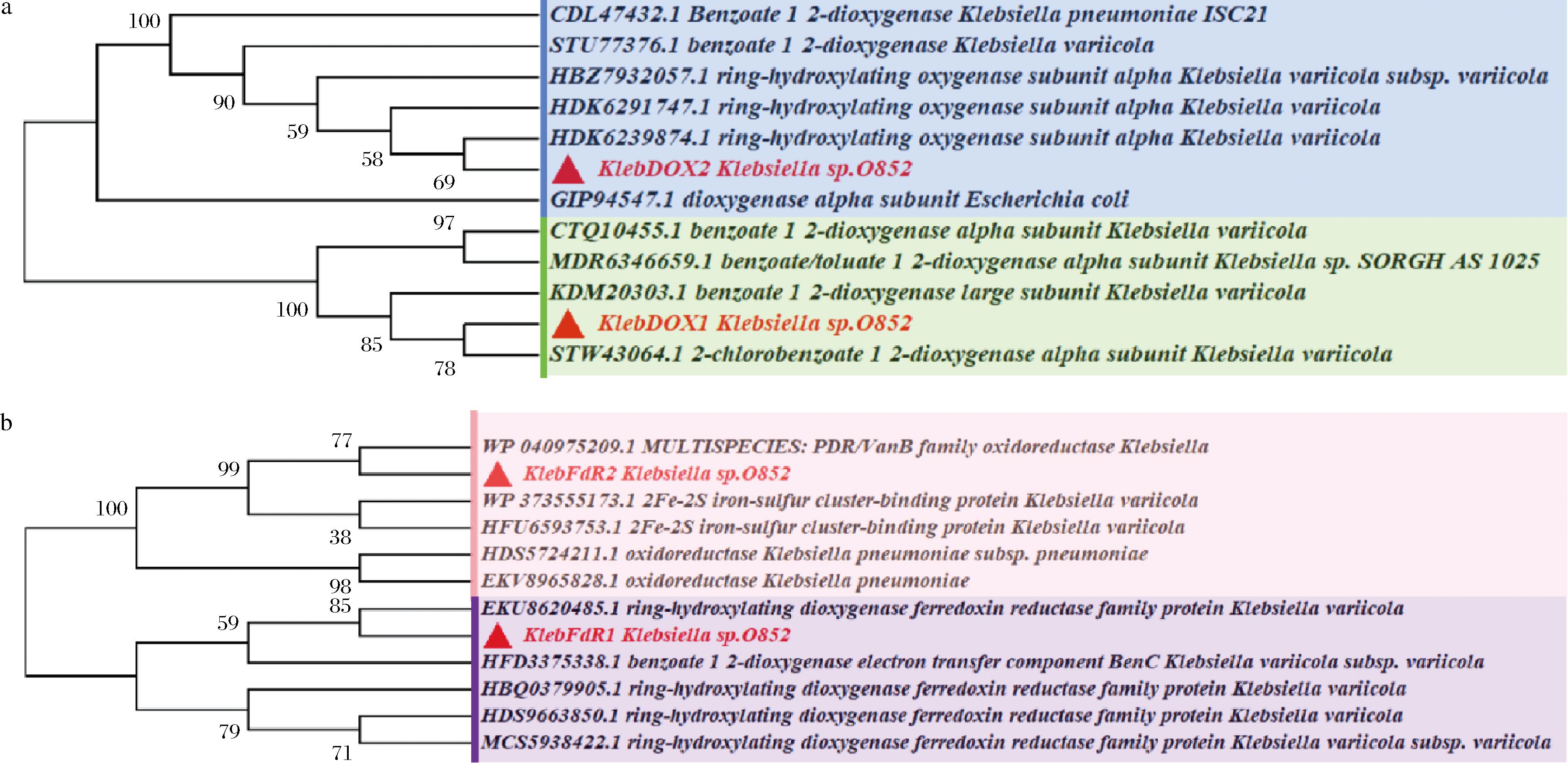
a-KlebDOX1和KebDOX2系统进化树分析;b-KlebFdR1和KebFdR2系统进化树分析
图2 基于氨基酸序列的系统进化树分析
Fig.2 Phylogenetic tree analysis based on amino acid sequences
如图2-b所示,基于氨基酸序列的KlebFdR1和KebFdR2进化树分支置信度在38~100,表明该进化树可信。其中KlebFdR1与同Klebsiella属的EKU8620485.1位于同一分支,这个蛋白被注释为环羟基化双加氧酶铁氧还蛋白还原酶;同理,KlebFdR2与同Klebsiella属的WP_040975209.1为同一分支,这个蛋白被注释为氧化还原酶,因此推测KlebFdR1和KlebFdR2基因编码的蛋白可能是铁氧还蛋白/铁氧还蛋白还原酶。
进一步使用DNAMAN 9.0软件对KlebDOX1和KebDOX2及其同源物、KlebFdR1和KebFdR2及其同源物进行多序列比对和结构域分析。结果如图3-a所示,这些双加氧酶基因都含有Rieske [2Fe-2S] (图中S-S区域,即Cys-X-His区域中的Cys与Cys-X-X-His中的Cys形成二硫键,从而构建铁硫中心)和单核铁结合位点(图中红色箭头所示,即单核铁与2个His和1个Asp结合的活性位点)。铁硫中心接收电子,并将其传递给单核铁活性位点,从而激活酶的催化功能[19]。此外,铁氧还蛋白/铁氧还蛋白还原酶的FAD和NAD结构域亦参与电子传递过程。如图3-b所示,KlebFdR1和KebFdR2及其同源序列均包含FAD结合结构域(图中红色箭头所示)和NAD结合结构域(图中红色方框所示)[20]。

a-KlebDOX1和KebDOX2多序列比对分析;b-KlebFdR1和KebFdR2多序列比对分析
图3 多序列比对
Fig.3 Multiple sequence alignment
注:a中□表示Cys-X-His区域中的Cys与Cys-X-X-His中的Cys形成S-S,↓表示单核铁结合位点;b中→表示FAD结构域;□表示NAD结构域。

a-KlebDOX1;b-KlebDOX2;c-KlebFdR1;d-KlebFdR2
图4 KlebDOX1、KlebDOX2、KlebFdR1和KlebFdR2三维结构预测
Fig.4 Three-dimensional structure prediction of KlebDOX1, KlebDOX2, KlebFdR1, and KlebFdR2
综合上述结果,表明KlebDOX1-KlebFdR1和KlebDOX2-KlebFdR2可能为Rieske型芳香环双加氧酶系。
2.3 KlebDOX1、KlebDOX2、KlebFdR1和KlebFdR2生物信息分析
使用ProtParam在线工具分析KlebDOX1、KlebDOX2、KlebFdR1和KlebFdR2的理化性质,结果表明KlebDOX1、KlebDOX2、KlebFdR1和KlebFdR2分别编码633、547、503和486个氨基酸,分子质量分别为71.29、61.51、54.30和53.24 kDa,等电点(isoelectric point,pI)分别为6.01、5.58、4.92和5.36,均为胞质表达,且为亲水性蛋白,不存在信号肽。这些蛋白的二级结构结果显示分别含有30%、29.68%、28.4%和24.92%的α螺旋,15.65%、14.71%、22.19%和26.17%的延伸链,54.35%、55.61%、49.41%和48.91%的无规则卷曲。图4是KlebDOX1、KlebDOX2、KlebFdR1和KlebFdR2的三维结构预测图,可以看出这些蛋白含有大量的无规则卷曲。
2.4 关键酶基因克隆与表达
以Klebsiella sp.O852基因组为模板扩增目的基因KlebDOX1、KlebDOX2、KlebFdR1和KlebFdR2,并与载体pET-32a(+)连接构建重组质粒,将构建成功的重组质粒分别转化至E.coli BL21(DE3)中,获得重组菌株BL21/pET32a-KlebDOX1、BL21/pET32a-KlebDOX2、BL21/pET32a-KlebFdR1、BL21/pET32a-KlebFdR2。随后,对这些重组菌株进行IPTG低温诱导表达,以未经诱导的重组菌作为对照,分别收集诱导后的菌体以及诱导后经过超声破碎的上清液和沉淀,进行SDS-PAGE分析,结果如图5所示。从泳道1和2可以看出,经过IPTG诱导之后,重组菌株BL21/pET32a-KlebDOX1、BL21/pET32a-KlebDOX2、BL21/pET32a-KlebFdR1、BL21/pET32a-KlebFdR2分别在71、62、54、53 kDa左右出现特异性的条带,这些条带在诱导后明显地变亮、变粗,且分子质量与预期一致。此外,通过观察泳道3和4可以发现,尽管超声波破碎后的沉淀中仍含有重组蛋白,但上清液中亦显著存在重组蛋白,这表明重组蛋白在大肠杆菌中成功表达。

M-蛋白Marker;泳道1~4依次是:未诱导的菌体、诱导后的菌体、诱导后经过超声破碎的上清液和沉淀。
a-KlebDOX1;b-KlebDOX2;c-KlebFdR1;d-KlebFdR2
图5 重组蛋白可溶性表达的SDS-PAGE图
Fig.5 SDS-PAGE analysis of soluble expression of recombinant protein
2.5 关键酶功能验证
为了验证基因KlebDOX1-KlebFdR1和KlebDOX2-KlebFdR2表达的双加氧酶系是否参与调控柠檬烯氧化过程,催化柠檬烯生成香芹醇,将重组菌株BL21/pET32a-KlebDOX1和BL21/pET32a-KlebFdR1(即BL21/pET32a-KlebDOX1-KlebFdR1)、以及BL21/pET32a-KlebDOX2和BL21/pET32a-KlebFdR2(即BL21/pET32a-KlebDOX2-KlebFdR2)进行混菌低温诱导表达,同时在体系中添加底物柠檬烯,检测产物香芹醇随时间的变化曲线。
结果如图6所示,在混菌BL21/pET32a-KlebDOX1-KlebFdR1和BL21/pET32a-KlebDOX2-KlebFdR2的体系中均检测到了香芹醇,并且其含量随着反应时间的延长而逐渐增加,当反应时间为6 h时,混菌BL21/pET32a-KlebDOX1-KlebFdR1体系中香芹醇的含量为0.137 mmol/L,BL21/pET32a-KlebDOX2-KlebFdR2体系中香芹醇的含量为0.154 mmol/L。而在对照组(未添加任何菌株的反应体系)中,未能检测到香芹醇的存在。此外,还观察到在菌株BL21/pET32a-KlebDOX1、BL21/pET32a-KlebDOX2、BL21/pET32a-KlebFdR1、BL21/pET32a-KlebFdR2单独低温诱导表达并添加底物柠檬烯进行发酵时,均未检测到香芹醇的生成,这说明只有末端氧化酶、铁氧还蛋白和铁氧还蛋白还原酶3个组分共同表达时,重组菌株才能转化柠檬烯生成香芹醇,这与先前的研究结果一致[21]。
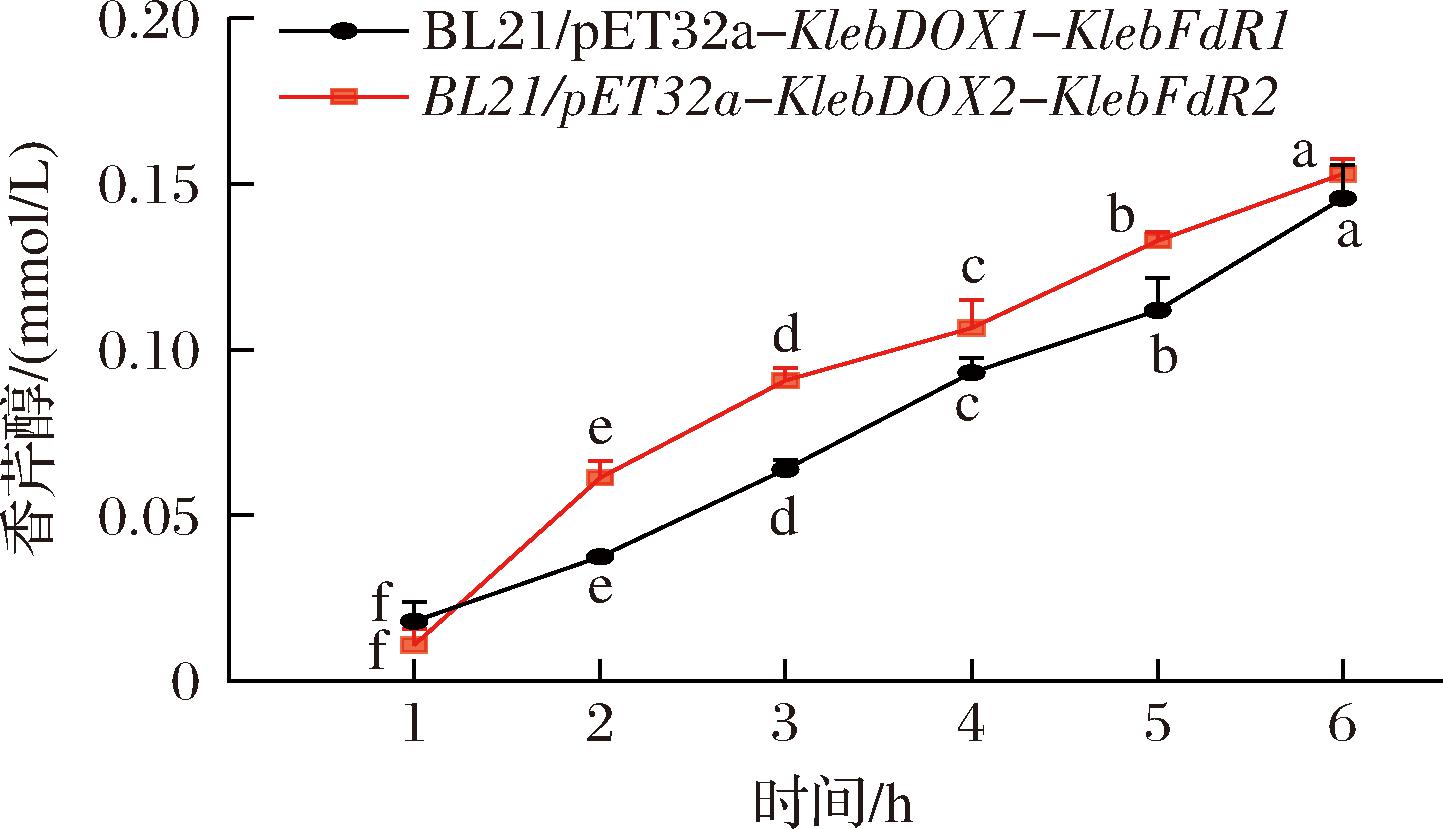
图6 重组菌株转化柠檬烯生成香芹醇
Fig.6 Conversion of limonene to carveol by recombinant strains
注:不同小写字母表示显著性差异 (P<0.05)。
此外,在整个反应过程中,尽管香芹醇的含量随着反应时间的延长而逐渐增加,但总体生成量仍相对较低。据推测,导致这一现象的原因可能包括以下几点:1)柠檬烯在水中的溶解度较低,仅为0.015 mmol/L,并且具有较高的挥发性和较差的化学稳定性。这些特性限制了柠檬烯作为底物在水相中的有效浓度,从而影响了产物的生成[22]。2)作为一种亲脂性单萜,柠檬烯对微生物细胞具有一定的毒性,能够与微生物细胞膜磷脂的疏水端产生亲合作用,引发氧化应激反应。在蛋白诱导表达的过程中,柠檬烯会抑制菌株的生长,进一步影响蛋白的表达和酶的活性,最终影响产物的生成[23]。在后续的研究中,将通过蛋白质工程改造提高末端氧化酶活性、3个组分共表达或融合表达、双向体系或底物分批流加降低柠檬烯毒性、以及发酵优化等策略,提高产物香芹醇的含量。
3 结论
香芹醇作为一种重要的单萜类化合物,在医药、食品和日化等领域具有广泛的应用价值。然而,由于其自然来源有限以及化学合成方法的局限性,利用微生物或其酶催化柠檬烯氧化生成香芹醇已成为研究热点。目前,主要通过CYP450酶催化柠檬烯氧化生成香芹醇,但大多数CYP450蛋白在表达时以包涵体形式存在,导致分离纯化困难并增加成本[24]。因此,有必要进一步挖掘柠檬烯氧化生成香芹醇的关键酶,丰富酶资源库,以实现香芹醇的经济高效合成。
本研究基于前期的Klebsiella sp.O852基因组数据,通过本地BLAST和生物信息学分析,结合多序列比对和进化树分析,筛选出2个Rieske型芳香环双加氧酶系:末端氧化酶KlebDOX1和铁氧还蛋白/铁氧还蛋白还原酶KlebFdR1、以及末端氧化酶KlebDOX2和铁氧还蛋白/铁氧还蛋白还原酶KlebFdR2,并以大肠杆菌为表达宿主对这4个酶基因进行异源表达,以鉴定其功能。结果显示在混菌BL21/pET32a-KlebDOX1-KlebFdR1和BL21/pET32a-KlebDOX2-KlebFdR2的体系中均检测到香芹醇的存在,表明由基因KlebDOX1-KlebFdR1和KlebDOX2-KlebFdR2表达的双加氧酶系具有氧化柠檬烯生成香芹醇的功能。本研究不仅为香芹醇的生物合成提供了重要的酶资源,也为相关酶的功能和机制研究奠定了坚实的基础。未来,我们将进一步分离纯化该双加氧酶系,并对其酶学性质及其他功能进行深入研究。
[1] BHATIA S P, MCGINTY D, LETIZIA C S, et al.Fragrance material review on carveol[J].Food and Chemical Toxicology, 2008, 46(11):S85-S87.
[2] DE LIMA SERAFIM C A, ARARUNA M E C, ALVES J NIOR E B, et al.(-)-carveol prevents gastric ulcers via cytoprotective, antioxidant, antisecretory and immunoregulatory mechanisms in animal models[J].Frontiers in Pharmacology, 2021, 12:736829.
NIOR E B, et al.(-)-carveol prevents gastric ulcers via cytoprotective, antioxidant, antisecretory and immunoregulatory mechanisms in animal models[J].Frontiers in Pharmacology, 2021, 12:736829.
[3] AHMED M S, KHAN A U, AL KURY L T, et al.Computational and pharmacological evaluation of carveol for antidiabetic potential[J].Frontiers in Pharmacology, 2020, 11:919.
[4] KAUR N, CHAHAL K K, KUMAR A, et al.Antioxidant activity of Anethum graveolens L.essential oil constituents and their chemical analogues[J].Journal of Food Biochemistry, 2019, 43(4):e12782.
[5] 徐龙兴, 袁丽杰, 刘顺成.解脂耶氏酵母合成萜类化合物的研究进展[J].微生物学通报, 2024, 51(9):3268-3302.XU L X, YUAN L J, LIU S C.Research progress in the synthesis of terpenoids from Yarrowia lipolytica[J].Microbiology China, 2024, 51(9):3268-3302.
[6] LUPIEN S, KARP F, WILDUNG M, et al.Regiospecific cytochrome P450 limonene hydroxylases from mint (Mentha) species:CDNA isolation, characterization, and functional expression of (-)-4S-limonene-3-hydroxylase and (-)-4S-limonene-6-hydroxylase[J].Archives of Biochemistry and Biophysics, 1999, 368(1):181-192.
[7] DIETRICH M, EIBEN S, ASTA C, et al.Cloning, expression and characterisation of CYP102A7, a self-sufficient P450 monooxygenase from Bacillus licheniformis[J].Applied Microbiology and Biotechnology, 2008, 79(6):931-940.
[8] LING Z Y, LI J R, DONG Y M, et al.Terpene produced by coexpression of the TPS and P450 genes from Lavandula angustifolia protects plants from herbivore attacks during budding stages[J].BMC Plant Biology, 2023, 23(1):477.
[9] WÜST M, CROTEAU R B.Hydroxylation of specifically deuterated limonene enantiomers by cytochrome P450 limonene-6-hydroxylase reveals the mechanism of multiple product formation[J].Biochemistry, 2002, 41(6):1820-1827.
[10] MIYAZAWA M, SHINDO M, SHIMADA T.Sex differences in the metabolism of (+)- and (-)-limonene enantiomers to carveol and perillyl alcohol derivatives by cytochrome p450 enzymes in rat liver microsomes[J].Chemical Research in Toxicology, 2002, 15(1):15-20.
[11] MIYAZAWA M, SHINDO M, SHIMADA T.Metabolism of (+)- and (-)-limonenes to respective carveols and perillyl alcohols by CYP2C9 and CYP2C19 in human liver microsomes[J].Drug Metabolism and Disposition, 2002, 30(5):602-607.
[12] BELL S G, SOWDEN R J, WONG L L.Engineering the haem monooxygenase cytochrome P450 for monoterpene oxidation[J].Chemical Communications, 2001(7):635-636.
[13] GROENEVELD M, VAN BEEK H L, DUETZ W A, et al.Identification of a novel oxygenase capable of regiospecific hydroxylation of D-limonene into (+)-trans-carveol[J].Tetrahedron, 2016, 72(46):7263-7267.
[14] TRYTEK M, FIEDUREK J.Biotransformation of d-limonene to carvone by means of glucose oxidase and peroxidase[J].Acta Microbiologica Polonica, 2002, 51(1):57-62.
[15] FARR T, WISSNER J L, HAUER B.A simple and efficient method for lyophilization of recombinant E.coli JM109 (DE3) whole-cells harboring active Rieske non-heme iron dioxygenases[J].MethodsX, 2021, 8:101323.
[16] DUETZ W A, FJ LLMAN A H, REN S, et al.Biotransformation of D-limonene to (+) trans-carveol by toluene-grown Rhodococcus opacus PWD4 cells[J].Applied and Environmental Microbiology, 2001, 67(6):2829-2832.
LLMAN A H, REN S, et al.Biotransformation of D-limonene to (+) trans-carveol by toluene-grown Rhodococcus opacus PWD4 cells[J].Applied and Environmental Microbiology, 2001, 67(6):2829-2832.
[17] 底心怡, 刘春立, 刘秀霞, 等.恶臭假单胞菌F1表达P450酶催化柠檬烯生物合成紫苏醇[J].食品与发酵工业, 2024, 50(2):34-40.DI X Y, LIU C L, LIU X X, et al.Biosynthesis of perillyl alcohol using limonene catalyzed by cytochrome P450 in Pseudomonas putida F1[J].Food and Fermentation Industries, 2024, 50(2):34-40.
[18] LI Z H, WANG W Y, WANG G G, et al.Heterologous expression and characterization of the carveol dehydrogenase from Klebsiella sp.O852[J].Molecular Catalysis, 2024, 561:114158.
[19] 刘玉, 杨键, 李茹, 等.微生物Rieske型芳香环双加氧酶研究进展[J].广西科学院学报, 2020, 36(3):272-281.LIU Y, YANG J, LI R, et al.Research advances of microbial rieske-type aromatic ring dioxygenases[J].Journal of Guangxi Academy of Sciences, 2020, 36(3):272-281.
[20] KARLSSON A, BEHARRY Z M, MATTHEW EBY D, et al.X-ray crystal structure of benzoate 1, 2-dioxygenase reductase from Acinetobacter sp.strain ADP1[J].Journal of Molecular Biology, 2002, 318(2):261-272.
[21] SAITO A, IWABUCHI T, HARAYAMA S.A novel phenanthrene dioxygenase from Nocardioides sp.Strain KP7:Expression in Escherichia coli[J].Journal of Bacteriology, 2000, 182(8):2134-2141.
[22] 台亚楠, 董曼, 任婧楠, 等.柠檬烯微生物转化的研究进展[J].食品科学, 2014, 35(17):272-277.TAI Y N, DONG M, REN J N, et al.Recent advances in biotransformation of limonene[J].Food Science, 2014, 35(17):272-277.
[23] HAMADA H, KONDO Y, ISHIHARA K, et al.Stereoselective biotransformation of limonene and limonene oxide by Cyanobacterium, Synechococcus sp.PCC 7942[J].Journal of Bioscience and Bioengineering, 2003, 96(6):581-584.
[24] 程婕, 龚毅, 杨凌.细胞色素P450酶系的异源表达研究[J].河北大学学报(自然科学版), 2006, 26(3):331-336.CHENG J, GONG Y, YANG L.Study of the cytochrome P450 s expressed in heterologous systems[J].Journal of Hebei University (Natural Science Edition), 2006, 26(3):331-336.