近年来,随着生活节奏的加快,焦虑已成为一种常见的情绪体验。焦虑是人体在面临压力、威胁等情境时产生的一种紧张、不安的情绪反应,适度的焦虑能激发个人的应对能力,但当这种情绪过度且持续发生,就可能发展为焦虑症[1]。据世界卫生组织数据显示,焦虑症已成为全球范围内最常见的精神障碍疾病之一,影响着数以亿计的人群,严重降低患者的生活质量,带来沉重的社会经济负担[2]。目前,焦虑症的治疗手段主要包括药物治疗和心理治疗。药物治疗多使用抗抑郁药、苯二氮 类药物等,虽能在一定程度上缓解症状,但存在副作用大、易成瘾、治疗周期长等问题[3];心理治疗则依赖专业心理咨询师的指导,且治疗效果受患者个体差异和配合度影响较大[4]。因此,如何有效优化现有治疗手段,解决药物治疗的副作用、成瘾性以及长周期的问题,已成为当下焦虑症治疗领域亟需攻克的难题。
类药物等,虽能在一定程度上缓解症状,但存在副作用大、易成瘾、治疗周期长等问题[3];心理治疗则依赖专业心理咨询师的指导,且治疗效果受患者个体差异和配合度影响较大[4]。因此,如何有效优化现有治疗手段,解决药物治疗的副作用、成瘾性以及长周期的问题,已成为当下焦虑症治疗领域亟需攻克的难题。
γ-氨基丁酸(γ-aminobutyric acid,GABA)作为中枢神经系统的核心抑制性神经递质,在促进人体放松、缓解焦虑方面效果显著[5]。然而,因其无法直接通过血脑屏障进行外源补充,对脑内GABA水平提升有限,因此调控内源性GABA代谢通路就成为抗焦虑药物研发的核心策略。γ-氨基丁酸转氨酶(γ-aminobutyric acid transaminase,GABA-T)作为GABA降解过程中的限速酶,是维持脑内GABA稳态的核心调控靶点,抑制其活性可有效减少GABA的降解[2]。生物活性肽是蛋白质水解后产生的一类具有特殊生理功能的小分子肽。近年来,生物活性肽在辅助治疗各类疾病方面受到了学者们的广泛关注[6-7]。与传统的药物治疗相比,生物活性肽具有来源天然、副作用小、易于吸收、作用靶点多等优势,有望为焦虑症的治疗开辟出新的途径[8]。现阶段,科研人员热衷于从富含生物活性的海洋生物,如鱼类、藻类和海绵中,探索小分子肽的药用效果[9]。螺旋藻作为藻类中极具代表性的一员,蛋白质含量可达细胞干重的70%,且富含藻蓝蛋白[10]。藻蓝蛋白是一种具有独特结构和显著生物活性的天然色素蛋白,具备强大的抗氧化、抗肿瘤等生物活性[11]。然而,目前针对藻蓝蛋白在抗焦虑领域的相关研究却极为缺乏。考虑到焦虑症在社会中的高发性以及现有疗法的局限性,藻蓝蛋白独特的生物活性极有可能为焦虑症的治疗带来新突破[12]。
传统生物活性肽的筛选方法是利用特定酶对蛋白进行水解,随后对产生的肽段进行分离、纯化、鉴定、合成,并开展体外和体内的验证试验[13]。但这种方法不仅耗时费力,还有可能造成微量但活性高的生物活性肽的损失[14]。因此,为克服传统筛选方法的缺陷,本研究采用虚拟酶切、计算机预测和分子模拟技术,对螺旋藻藻蓝蛋白中潜在的GABA-T抑制肽进行筛选。通过科学、系统的筛选流程,实现快速挖掘具有靶向活性的抗焦虑肽,同时为螺旋藻在食药领域的开发利用提供新思路,有望进一步挖掘螺旋藻的潜在价值。
1 材料与方法
1.1 材料与试剂
藻蓝蛋白的氨基酸序列从UniProt在线数据库(https://www.uniprot.org/)中获取;受体蛋白酶的晶体结构从RCSB PDB蛋白质数据库(https://www.rcsb.org/)中获得。
1.2 模拟软件
MOE(Molecular Operating Environment)(Version 2015.10)软件在分子模拟和药物设计领域占据重要地位,由加拿大化学计算集团公司(Chemical Computing Group Inc.)开发并持续维护。GROMACS 2024.1(Groningen Machine for Chemical Simulations)软件是一款广泛应用于生物分子和化学体系模拟的开源软件,由荷兰格罗宁根大学和瑞典皇家理工学院等机构开发维护。Discovery Studio 2021分子模拟软件由美国BIOVIA公司开发和推出,用于生命科学领域的分子设计和仿真研究。Microsoft Origin 2018用于数据分析和绘图。
1.3 实验方法
1.3.1 藻蓝蛋白序列获取及分析
藻蓝蛋白是螺旋藻藻胆蛋白中占比最高的一类蛋白,包括α和β两个亚基,其蛋白序列从UniProt在线数据库(https://www.uniprot.org/)中获取,蛋白质编号及氨基酸数目分别为:C-phycocyanin alpha subunit,P72509,162;C-phycocyanin beta subunit,P72508,172。
1.3.2 计算机虚拟酶切
选择ExPASy Peptide Cutter(https://web.expasy.org/peptide_cutter/)以及BIOPEP-UWM(http://www.uwm.edu.pl/biochemia/index.php/pl/biopep)数据库内的酶对藻蓝蛋白进行虚拟酶切。所使用的水解酶包括:胰蛋白酶(EC 3.4.21.4)、胃蛋白酶(pH 1.3)(EC 3.4.23.1)、胃蛋白酶(pH>2)、胰凝乳蛋白酶(EC 3.4.21.1)、木瓜蛋白酶(EC 3.4.22.2)、枯草杆菌蛋白酶(EC 3.4.21.62)、梭菌蛋白酶(EC 3.4.22.8)。
1.3.3 多肽生物活性预测
利用Peptide Ranker(http://distilldeep.ucd.ie/PeptideRanker/)对酶切获得的肽段进行潜在生物活性预测,并计算不同蛋白酶水解获得高生物活性肽的得率。Peptide Ranker预测结果越高代表具有生物活性的潜力越大,其中超过0.5阈值的肽被认为具有潜在生物活性[15],根据公式(1)计算高活性肽得率。
高生物活性肽得率![]()
(1)
式中:A,生物活性评分>0.5的肽段数量;B,蛋白酶水解获得的肽段总数量。
1.3.4 分子对接
通过MOE(Version 2015.10)软件分析目标活性肽与GABA-T的对接能力。配体准备:首先,通过NovoPro(https://www.novoprolabs.com/tools/convert-peptide-to-smiles-string)网站计算高生物活性肽段的SMILES,随后利用NovoPro(https://novopro.cn/tools/smiles2pdb.html)网站将肽段的SMILES转化成pdb结构,在MOE中通过能量最小化[力场:MMFF94x,梯度阈值:0.05 kcal/(mol·Å)]优化配体构象。受体准备:GABA-T的晶体结构从RCSB蛋白数据库(https://www.rcsb.org/)中下载,PDB ID为1OHW。使用MOE软件提取GABA-T酶的A链,并进行去水加氢处理,随后通过能量最小化[力场:AMBER99,梯度阈值:0.01 kcal/(mol·Å]优化受体结构[2]。受体与配体经能量最小化处理后,以受体自身携带的配体为中心,设置15 Å×15 Å×15 Å的对接盒子,网格点间距为0.375 Å,采用三角形匹配法进行初始对接,结合快速傅里叶变换优化对接构象,每个配体运行30次独立对接计算,输出能量最低的5个构象。从PubChem(https://pubchem.ncbi.nlm.nih.gov/)在线数据库中下载已知的GABA-T抑制剂4-羟基苯甲醛(CID:126),采用同样的方法进行对接作为阳性对照。Discovery Studio 2021用于分子对接结果的可视化。选择对接能高于阳性对照的肽段进行后续分析。
1.3.5 毒性、致敏性及水溶性分析
利用ToxinPred[14](https://webs.iiitd.edu.in/raghava/toxinpred/index.html)网站预测肽的毒性,采用2种方法预测:SVM(Swiss-Prot)+Motif和SVM(TrEMBL)+Motif,选择预测结果均为无毒的肽段;通过AlgPred 2.0[16](https://webs.iiitd.edu.in/raghava/algpred2/batch.html)网站预测肽的致敏性,采用Hybrid (RF+BLAST+MERCI)进行预测,阈值设定为0.5;利用Innovagen(http://www.innovagen.com/proteomics-tools)网站预测肽的水溶性。筛选出无毒、无致敏且水溶性良好的肽段用于后续分析。
1.3.6 药物代谢动力学分析
使用admetSAR 3.0在线网站(https://lmmd.ecust.edu.cn/admetsar3/about.php)进行药物代谢动力学预测,分析目标肽在体内的吸收、分布、代谢、排泄和毒性[17]。并进一步分析目标肽的人体肠道吸收率(human intestinal absorption,HIA)、血脑屏障通透性(blood-brain barrier,BBB)、血浆蛋白结合率(plasma protein binding, PPB)、CYP450酶的抑制性和肝毒性(drug induced liver injury,DILI)以评价目标肽的食药安全性和消化代谢效果。
1.3.7 分子动力学模拟
GROMACS 2024.1软件被用于对候选抗焦虑肽与GABA-T的复合物进行100 ns的分子动力学模拟。4-羟基苯甲醛的分子结构通过PubChem数据库(CID:216)获取SMILES字符串,并利用ACPYPE Server基于CGenFF力场生成拓扑参数。候选肽段与GABA-T形成的复合物置于1 nm3的立方体盒子的中心,内部充满SPC216水模型。模拟采用CHARMM27全原子力场,为中和掉多余的电荷,向体系内部添加Na+和Cl-[18]。分子动力学模拟前采用三步预处理:通过最陡下降法进行能量最小化,步长设置0.01,步数50 000步;在300 K温度下进行100 ps 的NPT平衡;在1.0 atm压力下进行100 ps的NVT平衡。正式模拟阶段采用LINCS算法约束共价键(时间步长2 fs),每2 ps采集一次轨迹数据,通过分析均方根偏差(root mean square deviation,RMSD)及均方根波动(root mean square fluctuation, RMSF)评估复合物稳定性与相互作用机制。
2 结果与分析
2.1 藻蓝蛋白的虚拟酶切
藻蓝蛋白作为一类由辅基蛋白与藻蓝胆素结合形成的天然捕光色素蛋白,因其独特的分子结构和显著的生物活性,目前已在抗氧化、抗肿瘤、抗癌、抗炎等多个领域得到应用[11]。基于其良好的应用前景和经济效益,本研究利用ExPASy Peptide Cutter和BIOPEP-UWM在线网站,选择7种水解能力强的蛋白酶对藻蓝蛋白进行虚拟酶切。研究选用的蛋白酶涵盖了广泛的切割位点,兼具特异性与广谱性,其中胰蛋白酶可特异性靶向识别精氨酸和赖氨酸的羧基端,胃蛋白酶和胰凝乳蛋白酶则分别识别芳香族氨基酸的氨基端和羧基端,而木瓜蛋白酶、枯草杆菌蛋白酶及梭菌蛋白酶具有广谱切割活性,可作用于多种氨基酸残基。这种组合特性可以高效水解藻蓝蛋白中富含的关键氨基酸(如精氨酸、疏水性氨基酸)及其邻近区域(如天冬氨酸、谷氨酸富集区)[19]。此外,所选蛋白酶在食品与生物工业中的成熟应用,也进一步增强了本研究的实际转化潜力。虚拟酶切结果如图1所示。
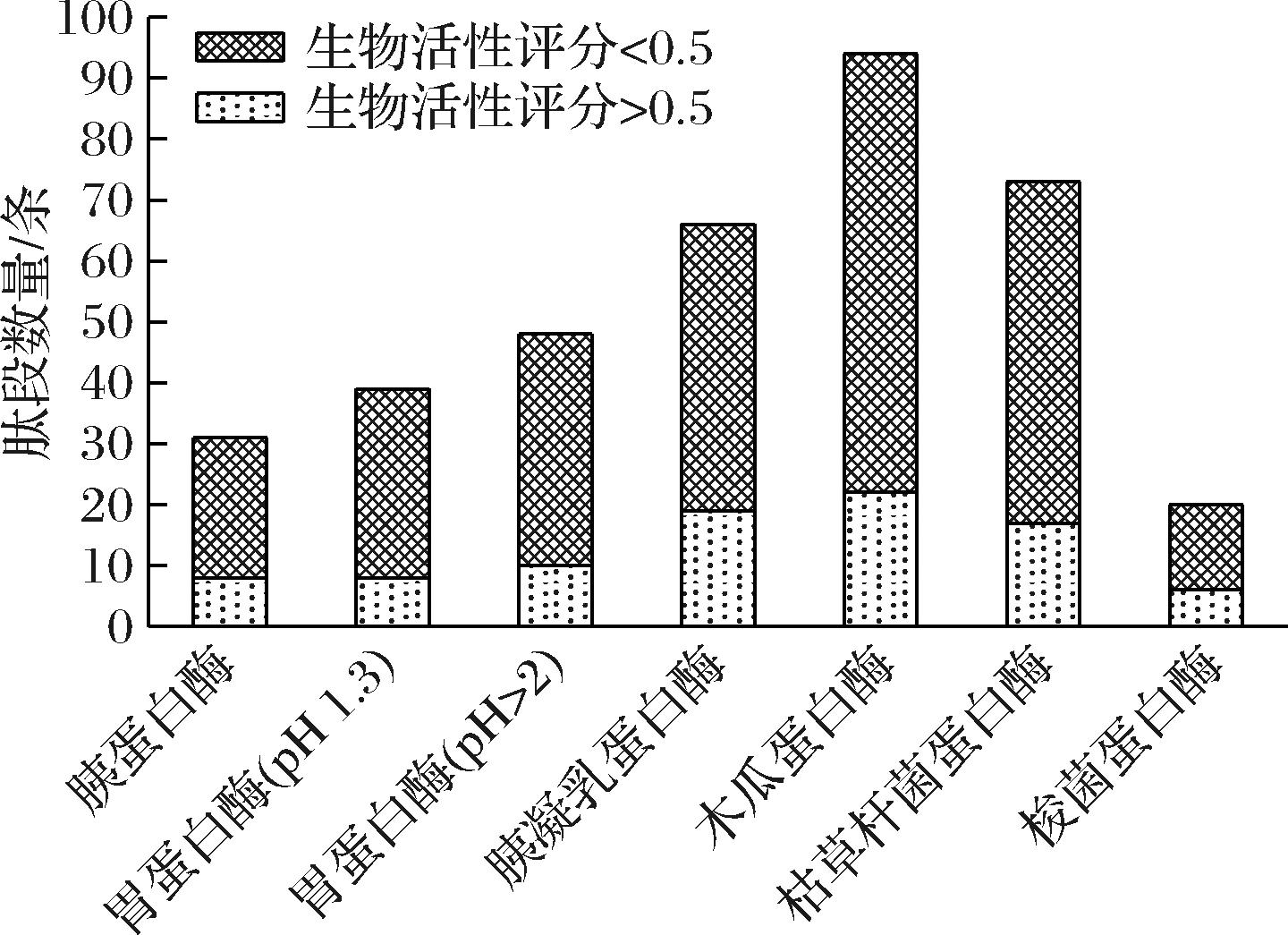
图1 不同酶虚拟酶切的肽段个数统计图
Fig.1 Statistical chart of the number of peptide segments digested by virtual enzymes of different enzymes
本研究的目的是从藻蓝蛋白中筛选出具有抗焦虑作用的小分子肽,进而挖掘其在抗焦虑领域的药用潜力,因此对所有酶解获取的肽段进行潜在生物活性预测。由图1可知,这些蛋白酶均可以水解藻蓝蛋白产生高生物活性肽(生物活性评分>0.5),其中木瓜蛋白酶、枯草杆菌蛋白酶和胰凝乳蛋白酶水解得到的肽段数量最多。7种酶水解出的高生物活性肽段如表1所示。7种蛋白酶水解得到的高生物活性多肽共计有49条,其中,胰蛋白酶、胰凝乳蛋白酶和梭菌蛋白酶高生物活性肽得率达到25%以上,这表明其在水解藻蓝蛋白时具有更高的效率和特异性,能产生大量具有潜在生物活性的肽段。
表1 肽序列及生物活性评分
Table 1 Peptide sequence and bioactivity score

水解酶肽序列生物活性评分水解酶肽序列生物活性评分胰蛋白酶FR0.985 719MFDAFTK0.695 309DIGYYLR0.679 515MAACLR0.677 658CLNGLR0.675 589CAR0.648 416MVTYCLIAGGTGPMDEYLI-AGIDEINR0.520 671胃蛋白酶(pH 1.3)MF0.996 643GRF0.979 857DAF0.804 969SPSWYIEAL0.781 129IAPGGNAYTSRRMAACL0.598 299NGL0.507 526胃蛋白酶(pH>2)LF0.986 934GRF0.979 857LSPSW0.729 126NKF0.623 609FE0.589 707RY0.543 741胰凝乳蛋白酶GRF0.979 857KF0.906 764CL0.879 917RM0.847 822SPSW0.845 37GL0.808 777DAF0.804 969PY0.736 696RTF0.719 957AACL0.673 804DPAGITPGDCSAL0.663 753AVF0.589 919RDM0.571 844RY0.543 741QGPN0.540 248IAGGTGPM0.526 903木瓜蛋白酶MF0.996 643AF0.973 259CL0.879 917PG0.877 086YCL0.831 604PL0.811 148ASYF0.790 284ACL0.746 733APG0.745 179AC0.735 313SPSWYIE0.703 632QM0.607 122YYL0.598 987AVF0.589 919YL0.575 36AG0.546 994PNY0.510 169枯草杆菌蛋白酶MF0.996 643GRF0.979 857QGRF0.921 54CL0.879 917RM0.847 822VF0.815 398DAF0.804 969PY0.736 696NKF0.623 609QM0.607 122VAF0.559 611RRMAACL0.552 524RY0.543 741NGL0.507 526梭菌蛋白酶FR0.985 719DIGYYLR0.679 515MAACLR0.677 658CLNGLR0.675 589MVTYCLIAGGTGPMDEYLI-AGIDEINR0.520 671
2.2 分子对接结果及毒性、致敏性、水溶性分析
分子对接是基于受体与配体间的相互作用,通过计算机模拟预测两者最佳结合模式及亲和力的方法。其核心原理是通过计算配体与受体的空间互补性、能量匹配以及分子间作用力,筛选出最优的结合构象[20]。本研究选用MOE软件,以已知的GABA-T抑制剂4-羟基苯甲醛为阳性对照,对具有潜在生物活性的49条多肽进行分子对接。依据结合自由能越负,化合物与受体结合可能性越高的原则,对数据进行分析。依据分子对接结果选择结合能最低即结合最稳定的前30位多肽进行毒性、致敏性、水溶性检测,进一步验证其作为口服肽的可能性,结果见表2。
表2 肽段的毒性、致敏性、水溶性分析
Table 2 Analysis of toxicity, allergenicity, and water solubility of peptides
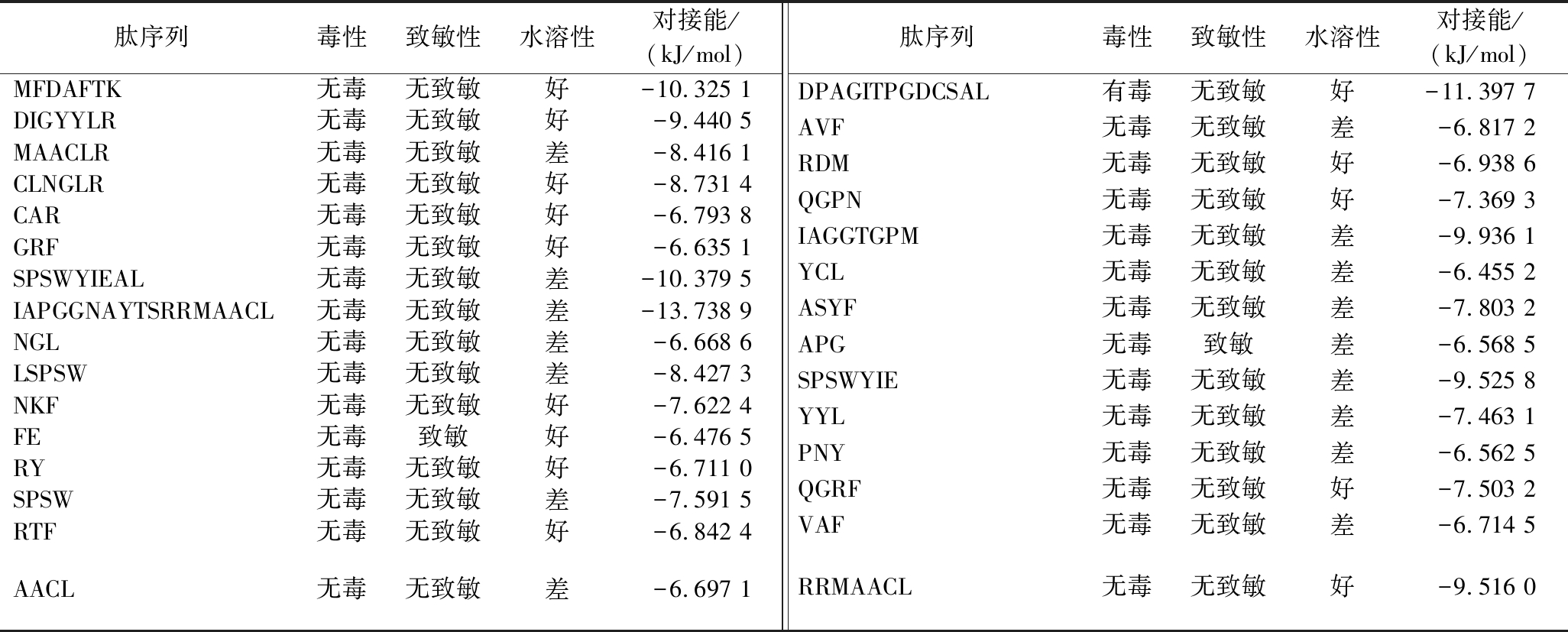
肽序列毒性致敏性水溶性对接能/(kJ/mol)肽序列毒性致敏性水溶性对接能/(kJ/mol)MFDAFTK无毒无致敏好-10.325 1DIGYYLR无毒无致敏好-9.440 5MAACLR无毒无致敏差-8.416 1CLNGLR无毒无致敏好-8.731 4CAR无毒无致敏好-6.793 8GRF无毒无致敏好-6.635 1SPSWYIEAL无毒无致敏差-10.379 5IAPGGNAYTSRRMAACL无毒无致敏差-13.738 9NGL无毒无致敏差-6.668 6LSPSW无毒无致敏差-8.427 3NKF无毒无致敏好-7.622 4FE无毒致敏好-6.476 5RY无毒无致敏好-6.711 0 SPSW无毒无致敏差-7.591 5RTF无毒无致敏好-6.842 4AACL无毒无致敏差-6.697 1DPAGITPGDCSAL有毒无致敏好-11.397 7AVF无毒无致敏差-6.817 2RDM无毒无致敏好-6.938 6QGPN无毒无致敏好-7.369 3IAGGTGPM无毒无致敏差-9.936 1YCL无毒无致敏差-6.455 2ASYF无毒无致敏差-7.803 2APG无毒致敏差-6.568 5SPSWYIE无毒无致敏差-9.525 8YYL无毒无致敏差-7.463 1PNY无毒无致敏差-6.562 5QGRF无毒无致敏好-7.503 2VAF无毒无致敏差-6.714 5RRMAACL无毒无致敏好-9.516 0
在毒性预测方面,ToxinPred在线网站可精准识别可能引发细胞毒性或溶血活性的风险分子。对口服肽而言,毒性预测具有双重意义:一方面,胃肠道吸收要求多肽不能破坏肠上皮细胞完整性;另一方面,血脑屏障穿透需规避对红细胞膜的破坏。通过该分析可有效排除具有潜在组织损伤性的候选肽,降低后续实验的安全隐患,结果显示绝大多数肽序列在2种模型预测下均表现为无毒。致敏性评估则通过AlgPred 2.0完成,可快速筛查多肽中潜在的致敏表位。这一步骤对口服制剂的研发尤为关键,因肠道作为免疫敏感区域,更易因致敏原暴露引发全身性的不良反应[21]。水溶性分析则依托肽溶解度预测工具Innovagen。口服肽类药物需在胃酸环境中保持可溶状态以避免沉淀,同时在中性肠液中维持适度溶解以实现跨膜吸收。低水溶性不仅影响药物生物利用度,还可能导致局部浓度过高引发刺激性,由表2可知,约一半的多肽被预测为水溶性差。最后,综合毒性、致敏性与水溶性检测结果对30条多肽进行分析,挑选同时符合无毒、无致敏、水溶性良好的12条多肽进行后续研究。
2.3 药物代谢动力学
药物代谢动力学是研究药物在体内吸收、分布、代谢、排泄及毒性动态过程的学科,其核心目标是评估药物的生物利用度、作用持续时间及安全性,为候选分子的结构优化与剂量设计提供依据[22]。研究多肽的药物代谢动力学有助于筛选出具有良好成药性的抗焦虑肽,减少药物研发失败的风险。对前期筛选得到的12条无毒、无致敏且水溶性良好的多肽进行ADMET分析,结果如表3所示。血脑屏障是化合物在中枢神经系统产生作用的前提条件,抗焦虑肽需穿透血脑屏障与GABA受体结合,才能起到缓解焦虑的作用,因此BBB是首要筛选标准。表中CLNGLR、GRF、QGRF、RRMAACL肽序列的BBB评分显著优于其他肽段,可能归因于其阳离子特性和氢键供体数符合BBB穿透的经典规则[23]。对于需口服给药的中枢神经系统药物,除BBB外,还需同时满足肠道水平的吸收[24]。HIA是评估口服药物候选物吸收特性的重要指标,HIA越高,意味着口服后通过肠道吸收进入血液的药物比例越高,为后续发挥药效提供的药物储备就越充足,表3中DIGYYLR、CLNGLR、CAR、GRF、NKF、RY、QGRF、RRMAACL肽序列口服吸收潜力较高。从安全性的角度出发,所有多肽的PPB和CYP450抑制性均较低,表明其可以在快速发挥药效的同时规避药物相互作用的风险,适合长期用药;表3中MFDAFTK、DIGYYLR、RRMAACL肽序列的PPB值较高,可能与其他药物发生相互作用而存在安全隐患。而DILI均为无毒也验证了前文ToxinPre在线预测多肽无毒的准确性。综合BBB、HIA和PPB预测,最终选择多肽CLNGLR、GRF、QGRF进行后续分析。
表3 肽段的ADMET分析
Table 3 ADMET analysis of peptide segments
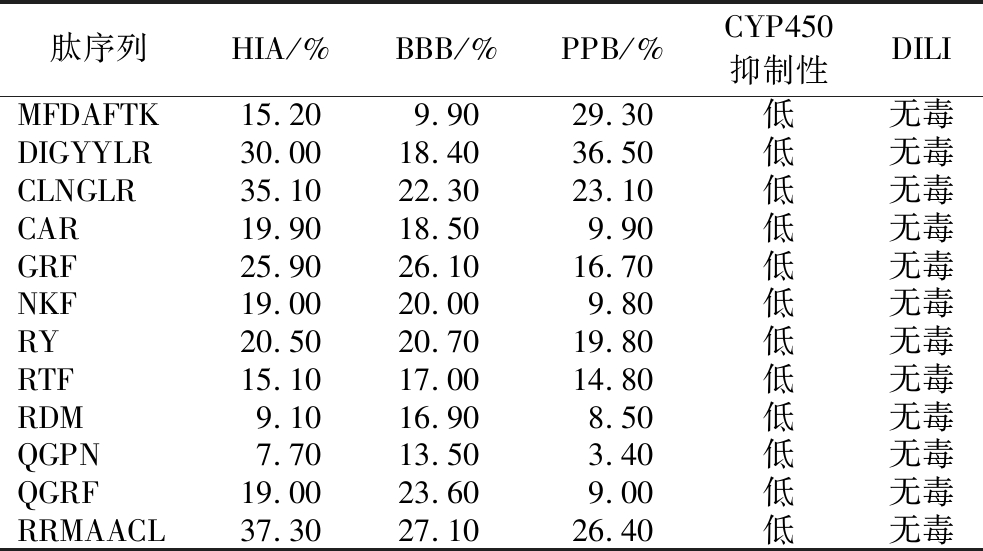
肽序列HIA/%BBB/%PPB/%CYP450抑制性DILIMFDAFTK15.209.9029.30低无毒DIGYYLR30.0018.4036.50低无毒CLNGLR35.1022.3023.10低无毒CAR19.9018.509.90低无毒GRF25.9026.1016.70低无毒NKF19.0020.009.80低无毒RY20.5020.7019.80低无毒RTF15.1017.0014.80低无毒RDM9.1016.908.50低无毒QGPN7.7013.503.40低无毒QGRF19.0023.609.00低无毒RRMAACL37.3027.1026.40低无毒
2.4 分子对接结果可视化
为了深入展示候选肽与GABA-T的结合姿态,本研究以4-羟基苯甲醛为阳性对照,分析CLNGLR、GRF、QGRF在分子对接中与GABA-T的结合模式,具体结果见图2和表4。图2显示出多肽与GABA-T结合的三维结构,其中红色棒状结构为配体,绿色棒状结构为受体的氨基酸残基,绿色虚线为受体与配体结合形成的氢键。
表4 候选肽与GABA-T的分子对接位点
Table 4 Molecular docking sites of the candidate peptide and GABA-T
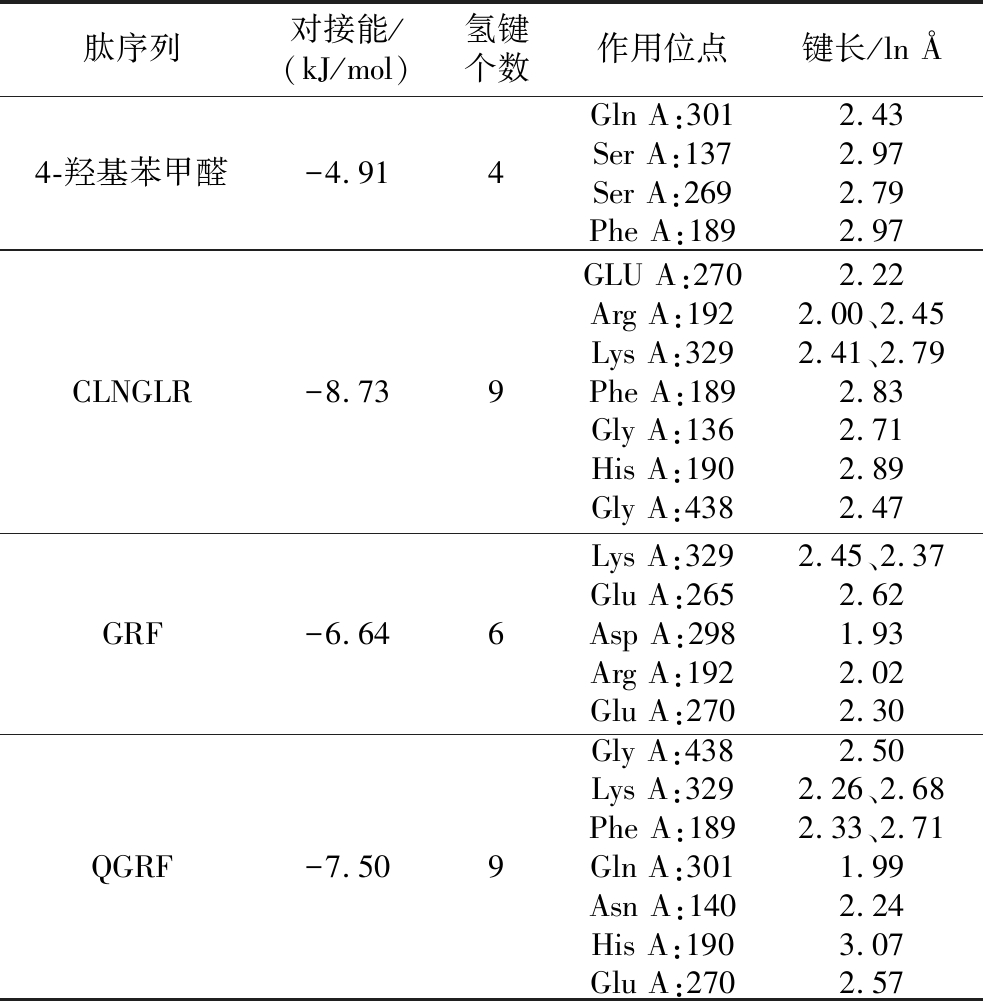
肽序列对接能/(kJ/mol)氢键个数作用位点键长/ln Å4-羟基苯甲醛-4.914Gln A:301Ser A:137Ser A:269Phe A:1892.432.972.792.97CLNGLR-8.739GLU A:270Arg A:192Lys A:329Phe A:189Gly A:136His A:190Gly A:4382.222.00、2.452.41、2.792.832.712.892.47GRF-6.646Lys A:329Glu A:265Asp A:298Arg A:192Glu A:2702.45、2.372.621.932.022.30QGRF-7.509Gly A:438Lys A:329Phe A:189Gln A:301Asn A:140His A:190Glu A:2702.502.26、2.682.33、2.711.992.243.072.57
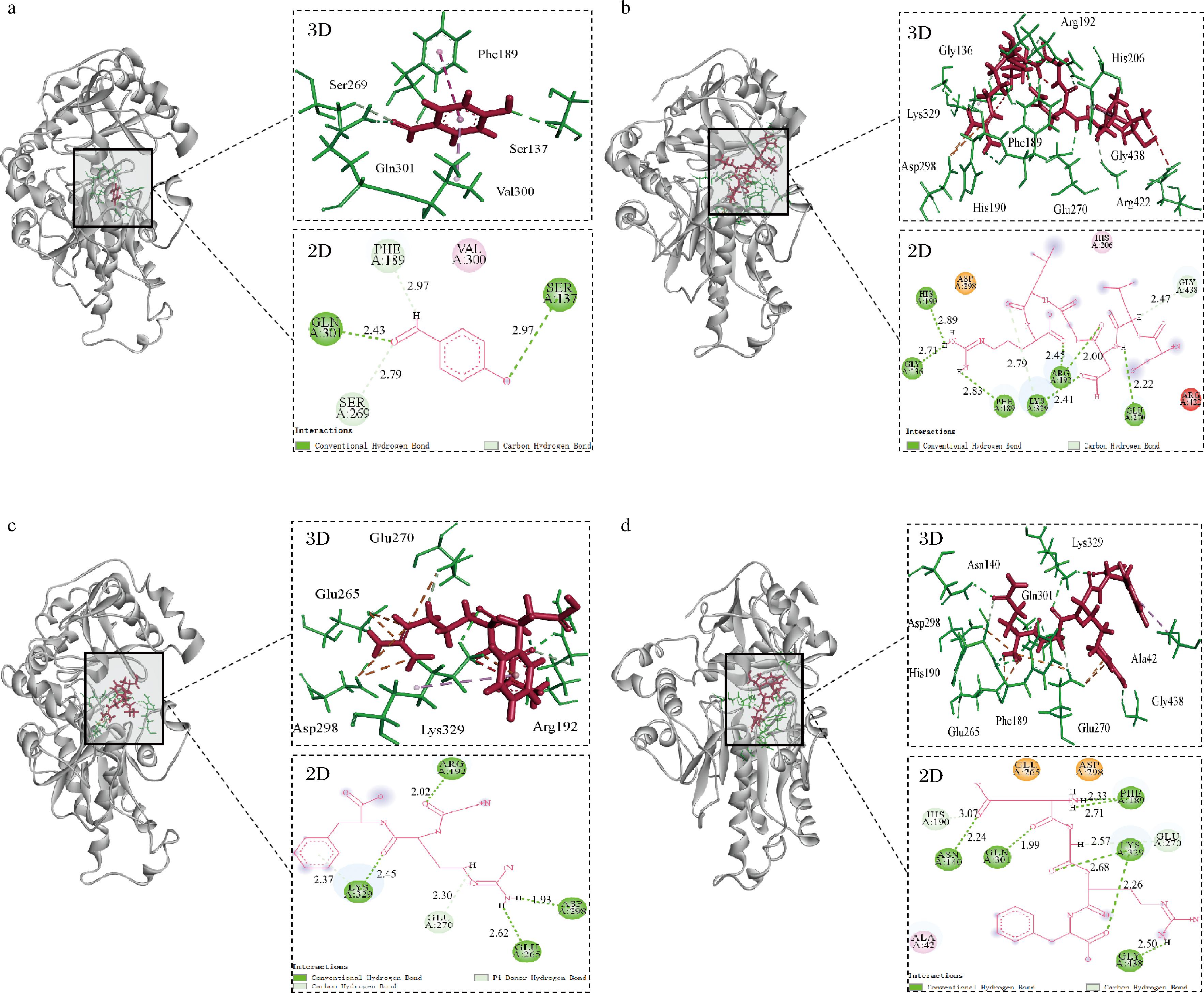
a-阳性对照;b-CLNGLR;c-GRF;d-QGRF
图2 候选肽与GABA-T的分子对接图
Fig.2 Molecular docking diagram of the candidate peptides with GABA-T
表4对多肽与GABA-T对接后最佳构象的作用位点进行分析,得出各自的对接能量、氢键个数、作用位点以及氢键键长等信息。结果显示,阳性对照4-羟基苯甲醛的结合能最低,且氢键个数明显少于目标肽,说明CLNGLR、GRF、QGRF与GABA-T形成的复合体匹配程度更高。从对接位点来看,4-羟基苯甲醛的对接位点是Gln301、Ser137、Ser269和Phe189,其中Gln301和Phe189与目标肽的对接位点相同,推测其可能是对接形成稳定复合体不可或缺的。同时,对接结果显示肽序列CLNGLR、GRF、QGRF结合在GABA-T的Glu270、Arg192、Lys329、His190、Gly438残基附近,具有相似的对接位点,与LIU等[2]对接发现的关键残基位点一致,可以推断出这5个氨基酸位点也是决定对接复合物稳定性的关键残基。Meeta Sahu的研究表明,现有药物Vigabatrin与GABA-T的关键位点Glu270、Gln301和Lys329形成氢键[25]。这也表明CLNGLR、GRF、QGRF可以与GABA-T的关键残基结合形成稳定的复合物,可为开发新型的GABA-T抑制肽提供依据。
2.5 分子动力学模拟
本研究通过100 ns的分子动力学模拟来分析4-羟基苯甲醛、目标肽与GABA-T形成复合物的稳定性和潜在的相互作用。RMSD反映了蛋白质在模拟过程中的构像与初始构像之间的位置偏离变化,是判断蛋白质体系是否稳定的重要指标。研究表明,RMSD越低表征蛋白质越稳定,RMSD越高表示在模拟时间内骨架的结构和构像变化越大[26]。如图3-a所示,经过100 ns的动力学模拟,空载GABA-T、4-羟基苯甲醛-GABA-T以及目标肽-GABA-T均达到了平衡。RMSD值排序为:4-羟基苯甲醛>目标肽>空载GABA-T。结果表明,单独GABA-T蛋白具有较稳定的构像,当4-羟基苯甲醛或多肽加入体系中时会引起蛋白质骨架发生一定改变,且多肽的加入引起GABA-T骨架发生的波动程度与阳性对照4-羟基苯甲醛相比更小,复合物结构更为稳定。这种增强的构象稳定性可能与抑制功能密切相关,稳定的目标肽-GABA-T复合物可能通过锁定酶的活性口袋构象(特别是底物GABA结合域),阻碍底物结合或产物释放通道,从而发挥抑制作用。该结果与分子对接中目标肽结合能更低的结果一致,表明目标肽是具有潜力的GABA-T抑制剂。
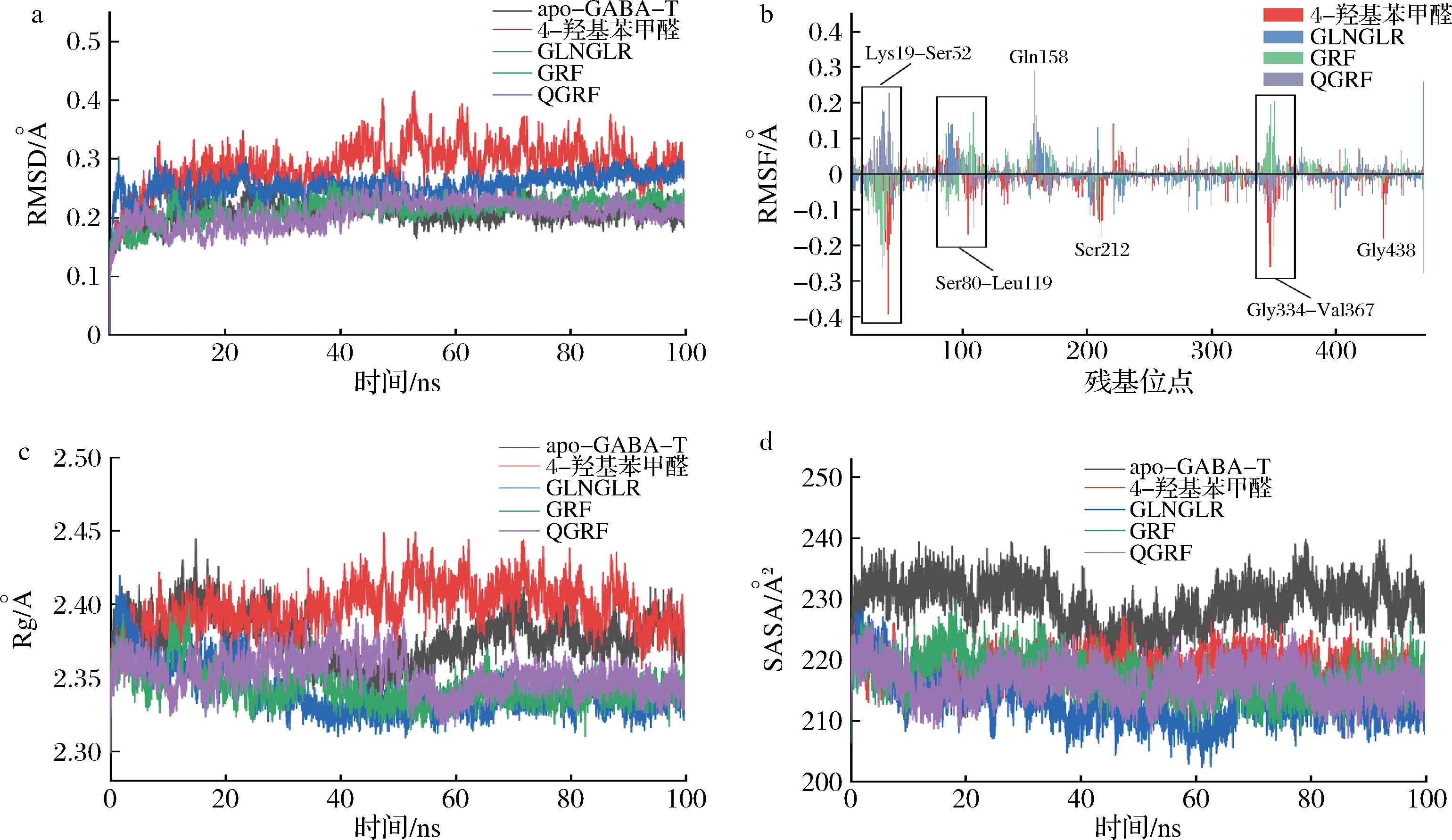
a-RMSD图;b-RMSF图;c-Rg图;d-SASA图
图3 4-羟基苯甲醛、GLNGLR、GRF、QGRF-GABA-T复合物的分子动力学模拟结果
Fig.3 Molecular dynamics simulation results of 4-hydroxybenzaldehyde, GLNGLR, GRF, QGRF-GABA-T complex
RMSF反映了在模拟期间,氨基酸残基的波动情况。如图3-b所示,4-羟基苯甲醛和目标肽的添加使GABA-T的Lys19-Ser52、Ser80-Leu119、Gln158、Ser212、Gly334-Val367和Gly438位残基发生较大波动。推测原因可能是4-羟基苯甲醛或目标肽与GABA-T的部分氨基酸残基形成了氢键,从而引起周围氨基酸残基发生了较大范围的波动。这也进一步验证了分子对接中,4-羟基苯甲醛和目标肽与GABA-T的活性中心紧密相连,引起了结合位点附近氨基酸残基的位置发生变化的现象。回旋半径(radius of qyration,Rg)表征蛋白质结构的紧密性以及在模拟过程中的松散程度。如图3-c所示,与4-羟基苯甲醛相比,GLNGLR、GRF、QGRF的添加均使得GABA-T蛋白的Rg值降低或波动更平稳,证实了多肽的添加可使蛋白质具有更紧密的结构,增强了蛋白结构在模拟过程中的稳定性,减少了其松散程度。图3-d展示了4-羟基苯甲醛和目标肽与GABA-T结合前后,GABA-T的溶剂可及表面积(solvent accessible surface area,SASA)随时间变化情况。可以看出,相较于未结合状态(apo-GABA-T),加入4-羟基苯甲醛和目标肽后,GABA-T的SASA值整体更低。这表明4-羟基苯甲醛和目标肽的添加,促使GABA-T的局部构象趋于紧凑,减少了GABA-T与溶剂分子的接触面积。从酶抑制机制的角度来看,这种靶向活性域的构象收缩可通过双路径干扰GABA-T对底物的催化功能:一方面,收缩的活性口袋可能直接阻碍底物GABA进入通道,另一方面,构象收缩进一步催化核心位点氨基酸残基的SASA值降低,进而削弱GABA-T与底物的动态相互作用效率。目标肽诱导活性口袋SASA值的降低,与更稳定的复合物整体构象(RMSD值最低)以及强结合模式(分子对接结合能<-6.64 kJ/mol)形成协同证据链,共同支持其通过“锁定催化微环境”抑制GABA-T的可行性。该机制与阳性对照4-羟基苯甲醛的作用原理一致,为多肽维持GABA水平、缓解焦虑提供了结构基础。
3 结论与讨论
本文通过计算机模拟技术,构建了一套从螺旋藻藻蓝蛋白中筛选GABA-T抑制肽的系统性策略,成功筛选出具有显著抑制潜力的功能性多肽CLNGLR、GRF和QGRF,为开发天然源抗焦虑药物提供了理论依据。该策略整合了虚拟酶切、PeptideRanker评分、分子对接、毒性、致敏性、水溶性预测以及药代动力学分析,最后对筛选出的多肽进行动力学模拟,各个环节紧密配合,不仅大幅缩小筛选范围,提高筛选效率,还确保了筛选出的多肽具有良好的成药性和安全性。然而,由于螺旋藻C-藻蓝蛋白的α和β亚基在特定区域(如发色团结合域、表面环区)具有独特的氨基酸序列特征,这些特征已被报道与其生物活性和酶切敏感性相关,因此,基于螺旋藻C-藻蓝蛋白特定序列酶解筛选出的多肽,其结构、功能及与GABA-T的结合模式可能具有物种来源依赖性[27]。未来,研究可进一步比较其他藻类(如颤藻、聚球藻)来源藻蓝蛋白的酶解肽谱及生物活性差异,以便深入探究物种特异性问题。本研究展示了计算机模拟技术从天然产物中挖掘生物活性物质的巨大潜力,推动螺旋藻藻蓝蛋白在抗焦虑药物和功能性食品领域开发应用的同时,为海洋生物资源的药物研发提供了新思路。
[1] HALAJ A, KONSTANTAKOPOULOS G, GHAEMI N S, et al.Anxiety disorders:The relationship between insight and metacognition[J].Psychopathology, 2024, 57(5):434-443.
[2] LIU M Q, WANG T, WANG Q L, et al.Structure-guided discovery of food-derived GABA-T inhibitors as hunters for anti-anxiety compounds[J].Food &Function, 2022, 13(24):12674-12685.
[3] GARNER M, MÖHLER H, STEIN D J, et al.Research in anxiety disorders:From the bench to the bedside[J].European Neuropsychopharmacology, 2009, 19(6):381-390.
[4] AMORIM D, BRITO I, CASEIRO A, et al.Electroacupuncture and acupuncture in the treatment of anxiety - A double blinded randomized parallel clinical trial[J].Complementary Therapies in Clinical Practice, 2022, 46:101541.
[5] TUFAIL T, AIN H B U, VIRK M S, et al.GABA (γ-aminobutyric acid) enrichment and detection methods in cereals:Unlocking sustainable health benefits[J].Food Chemistry, 2025, 464:141750.
[6] 刘梦琪, 刘畅, 刘博浩, 等.基于定量构效关系制备火麻仁蛋白ACE抑制肽[J].中国食品学报, 2023, 23(12):143-152.LIU M Q, LIU C, LIU B H, et al.Preparation of ACE inhibitory peptide of hemp seed protein based on quantitative structure-activity relationship[J].Journal of Chinese Institute of Food Science and Technology, 2023, 23(12):143-152.
[7] PATIL S P, GOSWAMI A, KALIA K, et al.Plant-derived bioactive peptides:A treatment to cure diabetes[J].International Journal of Peptide Research and Therapeutics, 2020, 26(2):955-968.
[8] ABEYRATHNE E D N S, NAM K, HUANG X, et al.Plant- and animal-based antioxidants’ structure, efficacy, mechanisms, and applications:A review[J].Antioxidants, 2022, 11(5):1025.
[9] CUNHA S A, PINTADO M E.Bioactive peptides derived from marine sources:Biological and functional properties[J].Trends in Food Science &Technology, 2022, 119:348-370.
[10] 宋盈萱, 尹馨一, 刘盈萱.螺旋藻营养成分及生物活性研究进展[J].食品安全导刊, 2024(27):178-182.SONG Y X, YIN X Y, LIU Y X.Research progress on nutrients and bioactivity of Spirulina[J].China Food Safety Magazine, 2024(27):178-182.
[11] 殷欢. 藻蓝蛋白分离纯化、酶解及对淀粉消化特性的影响研究[D].郑州:河南工业大学, 2023.YIN H.Study on isolation and purification of phycocyanin, enzymatic hydrolysis and its effect on starch digestion characteristics[D].Zhengzhou:Henan University of Technology, 2023.
[12] FEDOCE A D G, FERREIRA F, BOTA R G, et al.The role of oxidative stress in anxiety disorder:Cause or consequence?[J].Free Radical Research, 2018, 52(7):737-750.
[13] 马寅龙, 郭锐斌, 于雁松, 等.基于分子模拟技术筛选牛乳清蛋白源抗氧化肽[J].中国食品学报, 2024, 24(2):11-31.MA Y L, GUO R B, YU Y S, et al.Screening antioxidant peptides from bovine whey protein based on molecular simulation technology[J].Journal of Chinese Institute of Food Science and Technology, 2024, 24(2):11-31.
[14] LE N MADRAZO A, SEGURA CAMPOS M R.In silico prediction of peptide variants from chia (S.hispanica L.) with antimicrobial, antibiofilm, and antioxidant potential[J].Computational Biology and Chemistry, 2022, 98:107695.
N MADRAZO A, SEGURA CAMPOS M R.In silico prediction of peptide variants from chia (S.hispanica L.) with antimicrobial, antibiofilm, and antioxidant potential[J].Computational Biology and Chemistry, 2022, 98:107695.
[15] AHMED A S, EL-BASSIONY T, ELMALT L M, et al.Identification of potent antioxidant bioactive peptides from goat milk proteins[J].Food Research International, 2015, 74:80-88.
[16] SHARMA N, PATIYAL S, DHALL A, et al.AlgPred 2.0:An improved method for predicting allergenic proteins and mapping of IgE epitopes[J].Briefings in Bioinformatics, 2021, 22(4):bbaa294.
[17] JIA C Y, LI J Y, HAO G F, et al.A drug-likeness toolbox facilitates ADMET study in drug discovery[J].Drug Discovery Today, 2020, 25(1):248-258.
[18] LIU M Q, WANG B R, QIU Y C, et al.Regulation and mechanism of enzyme metabolism in germinated hemp seeds by ultrasound combined with exogenous calcium chloride treatment[J].International Journal of Biological Macromolecules, 2024, 276:133732.
[19] NANNI B, BALESTRERI E, DAINESE E, et al.Characterisation of a specific Phycocyanin-hydrolysing protease purified from Spirulina platensis[J].Microbiological Research, 2001, 156(3):259-266.
[20] KUMAR A, TIWARI A, SHARMA A.Changing paradigm from one target one ligand towards multi-target directed ligand design for key drug targets of Alzheimer disease:An important role of in silico methods in multi-target directed ligands design[J].Current Neuropharmacology, 2018, 16(6):726-739.
[21] DE GROOT A S, ROBERTS B J, MATTEI A, et al.Immunogenicity risk assessment of synthetic peptide drugs and their impurities[J].Drug Discovery Today, 2023, 28(10):103714.
[22] LAI Y R, CHU X Y, DI L, et al.Recent advances in the translation of drug metabolism and pharmacokinetics science for drug discovery and development[J].Acta Pharmaceutica Sinica B, 2022, 12(6):2751-2777.
[23] PARDRIDGE W M.Drug transport across the blood-brain barrier[J].Journal of Cerebral Blood Flow and Metabolism, 2012, 32(11):1959-1972.
[24] FEDI A, VITALE C, PONSCHIN G, et al.In vitro models replicating the human intestinal epithelium for absorption and metabolism studies:A systematic review[J].Journal of Controlled Release, 2021, 335:247-268.
[25] SAHU M, SIDDIQUI N, SHARMA V, et al.5, 6-dihydropyrimidine-1(2H)-carbothioamides:Synthesis, in vitro GABA-AT screening, anticonvulsant activity and molecular modelling study[J].Bioorganic Chemistry, 2018, 77:56-67.
[26] VAN DER SPOEL D, LINDAHL E, HESS B, et al.GROMACS:Fast, flexible, and free[J].Journal of Computational Chemistry, 2005, 26(16):1701-1718.
[27] WANG X Q, LI L N, CHANG W R, et al.Structure of C-phycocyanin from Spirulina platensis at 2.2 A resolution:A novel monoclinic crystal form for phycobiliproteins in phycobilisomes[J].Acta Crystallographica.Section D, Biological Crystallography, 2001, 57(Pt 6):784-792.