壳聚糖酶(EC 3.2.1.132,chitosanase,Csn)是一种糖苷水解酶,能够特异性催化壳聚糖中的β-1,4-糖苷键断裂,产生壳寡糖(chitosan oligosaccharide,COS)[1]。因此,Csn最重要的应用之一是制备具有独特生理活性的COS。COS不仅具有水溶性好、易吸收等优点,而且具有抗菌、抗真菌、调节机体免疫、抗癌等多种功能,在功能性食品和保健药品领域具有广泛应用前景[1]。由于虾、蟹、贝类等海产品的壳是壳聚糖的重要来源,Csn也可促进海洋副产品的高值化利用。此外,Csn还被用于植物病害防治[2],基因递送[3]和原质体制备[4]等领域。因此,挖掘高活性Csn及实现其高效生产是相关领域研究的热点。
Csn在自然界中分布十分广泛,在微生物、植物和动物中均有发现。目前已表征的Csn大多来源于芽孢杆菌(Bacillus)与链霉菌属(Streptomyces)。然而,野生型菌株发酵水平较低,多数产量在5 U/mL以下[5]。采用传统诱变选育的周期较长,构建高产的基因工程是迅速提高Csn发酵水平的重要途径。目前,Bacillus和Streptomyces等不同来源的Csn已在大肠杆菌(Escherichia coli)[5]、毕赤酵母(Pichia pastoris)[6]和枯草芽孢杆菌(Bacillus subtilis)[7]中实现了高效表达。然而,E.coli产内毒素,P.pastoris依赖于甲醇诱导,一定程度上限制了重组Csn在食品领域中的应用。尽管研究尝试了在食品级宿主变铅青链霉菌(Streptomyces lividans)和酿酒酵母(Saccharomyces cerevisiae)中表达Csn,但酶活力仅分别为0.461 U/mL[8]和2 U/mL[9]。在食品级宿主B.subtilis中表达Csn,需添加卡那霉素维持游离表达质粒[7],存在成本和安全性问题。因此,有必要寻找新的食品级宿主来表达Csn。
20余年前,由茂原链霉菌(Streptomyces mobaraensis)生产的谷氨酰胺转氨酶(transglutaminase,TGase)通过了美国食品药品监督管理局(Food and Drug Administration,FDA)安全认证[一般公认安全(generally recognized as safe,GRAS)]。由此S.mobaraensis成为世界范围内唯一的食品级TGase生产菌株[10]。前期工作中,通过常压室温等离子体诱变(atmospheric and room temperature plasma,ARTP),成功筛选到了一株高产TGase的S.mobaraensis(smY2022),胞外酶的产量达到1.68 g/L[11]。同时,S.mobaraensis可以使用廉价的鱼粉作为发酵原料[12]。此外,在S.mobaraensis基因编辑方面也取得重要突破,成功构建了TGase基因缺失菌株(smY2022Δtg),为其他酶的重组表达提供了可能性[11]。作为Csn的重组来源,S.mobaraensis具有成为食品级Csn表达宿主的潜力。
S.mobaraensis DSM40587的基因组序列已测定完成(GenBank:GCA_020099395.1)[13]。本研究中,基于基因组注释和结构分析,从S.mobaraensis DSM40587中克隆了Csn基因SmCsn46(GenBank登录号:UBI35095.1),并成功在smY2022Δtg中表达。经蛋白质纯化,对SmCsn46的酶学性质及其对壳聚糖的水解特异性进行了分析。研究结果将有助于进一步推动Csn工业化生产和应用。
1 材料方法
1.1 材料与试剂
1.1.1 菌株与质粒
E.coli DH5α用于基因克隆。E.coli ET12567/pUZ8002(Invitrogen, Carlsbad, 美国)用于Streptomyces菌株的原生质体转化。S.mobaraensis DSM40587诱变株smY2022Δtg用于Csn表达。pSET152-PsmY2022-TGase用于克隆Csn表达元件,质粒pCRISPomyces-2-2[14]用于链霉菌整合表达。以上质粒与菌株均由本实验室保藏。
1.1.2 试剂
壳聚糖[脱乙酰度(degree of deacetylation,DDA) 85%,90%,95%],源叶生物科技有限公司;COS标准品 (GlcN)2、(GlcN)3、(GlcN)4、(GlcN)5、和(GlcN)6,惠诚生物科技有限公司;保真DNA聚合酶(Phanta Max Master Mix)、一步克隆连接试剂盒One Step Cloning Kit(ClonExpressTM Ⅱ),南京诺唯赞生物科技股份有限公司;高保真DNA聚合酶(KOD FX),东洋纺(上海)生物科技有限公司;限制性内切酶、蛋白预制胶、蛋白质Marker、MES蛋白电泳缓冲液,赛默飞世尔科技(中国)有限公司;安普霉素、DNA产物纯化试剂盒、质粒抽提试剂盒,生工生物工程(上海)股份有限公司;Ni-NTA Purose 6 Fast Flow组氨酸亲和层析柱,千纯生物科技有限公司;Bis-Tris预制凝胶、PageRulerTM Plus蛋白质Marker(10~250 kDa),Thermo Fisher公司。
1.1.3 培养基与缓冲液
GYM琼脂培养基(g/L):葡萄糖10,酵母粉4,麦芽粉3,琼脂粉20。
MS琼脂培养基(g/L):甘露醇20,麦芽提取物2.5,大豆粉20,琼脂20,50 mmol/L MgCl2。
种子培养基(g/L):甘油20,酵母粉5,胰蛋白胨20,K2HPO4 4,MgSO4 2,pH 7.2。
发酵培养基(g/L):甘油20,鱼粉蛋白胨25,酵母粉5,玉米浆粉5.5,(NH4)2SO4 5.5,K2HPO4 2, MgSO4 2, pH 7.2~7.4。
1.2 实验方法
1.2.1 质粒构建
通过一步克隆法构建Csn在S.mobaraensis中的整合表达质粒。使用引物对152-Scsn-F/152-csn-R从S.mobaraensis DSM40587基因组中扩增得到包含内源分泌信号肽的smCsn46基因片段。使用引物对152-F/152-S-R从pSET152-PsmY2022-TGase扩增得到包含TGase启动子(PsmY2022)和终止子的基因片段。将上述基因片段通过一步克隆连接,构建得到编码smCsn46基因表达盒的质粒pSET152-Scsn-Csn。类似的,通过引物对152-csn-F/152-csn-R和引物对152-F/152-R分别扩增得到不含内源分泌信号肽的smCsn46基因片段和包含PsmY2022、分泌信号肽和终止子的基因片段,并将2个基因片段连接,得到编码Csn基因表达盒的质粒pSET152-Stg-Csn。使用引物对CSN-F/CSN-R从pSET152-Scsn-Csn和pSET152-Stg-Csn分别扩增得到含有smCsn46分泌信号肽和TGase信号肽的Csn表达盒基因片段。引物对300fwT7 sense/300fwT7antisense于95 ℃引物退火后,用靶向TGase位点sgRNA使用。95 ℃引物退火后形成编码sgRNA(靶向TGase基因位点)基因片段,并克隆至pCRISPomyces-2-2 Bbs I位点,得到pCRISPomyces-2-2-sgRNA。分别使用引物对L1000-F/L1000-R和R1000-F/R1000-R从S.mobaraensis DSM40587基因组中克隆了TGase 的上游(1 000 bp)和下游(1 000 bp)同源臂。最后,通过一步克隆将Csn表达盒(含smCsn46信号肽或TGase信号肽)同源臂和经Xba I切割的pCRISPomyces-2-2-sgRNA进行连接,得到smCsn46的整合表达质粒pCRISPomyces-Scsn-Csn和pCRISPomyces-Stg-Csn。以上涉及引物序列如表1所示。
表1 本研究引物序列
Table 1 Primers used in this study

名称核苷酸序列(5′-3′)300fwT7 senseACGCCGACCCTGCTGCGTCGATGA300fwT7antisenseAAACTCATCGACGCAGCAGGGTCG152-csn-FCGGCCACCGGCAGTGGCAGTGCGTCCGCGGCC-CCCGCCTC152-Scsn-FCAAAGGAGTTGTTGTTCTTCATGCCGCGGCCGC-CGCAG152-csn-RGCTTACACTAGTGGTGGTGGTGGTGGTGCTCGT-CGATGCGGTACGCGTCGCCGTA152-FCCACCACTAGTGTAAGCGGGGAGGGGAGGGGA-GG152-RACTGCCACTGCCGGTGGCCGC152-S-RGGGGAGGGGAGGGGAGGL1000-FGCCGGGCGTTTTTTATCTAGAGTGGTGCACGTA-CAGGTACGACGGL1000-RCTACAGCTCGTGGCCCGTCGTGCTGTCCR1000-FAATCCGTCCGTCAGATGTCCCGGAAGATCTCGR1000-RTTACGGTTCCTGGCCTCTAGCGCATGGACCAGT-TCGGCATCCCGTACSN-FCGACGGGCCACGAGCTGTAGGGTGGGGAGGGG-AGCCGGATGCTCSN-RACATCTGACGGACGGATTGTGGTGTTGCCGGGG-AGTTAACTGGGAGACver-FGCTTCATACAGGTCCGGATGGCGCGAver-RCAACGACGGCAACGACCTGGAGATCATT
1.2.2 S.mobaraensis转化及双交换同源重组菌株筛选
pCRISPomyces-Scsn-Csn和pCRISPomyces-Stg-Csn被分别转化至非甲基化的E.coli菌株ET12567/pUZ8002中。在含有25 μg/mL氯霉素、50 μg/mL卡那霉素和100 μg/mL氨苄青霉素的LB培养基中于37 ℃培养2 h后,收集重组E.coli并与在50 ℃预热10 min的smY2022Δtg孢子悬浮液混合。将悬浮液涂布在MS琼脂培养基上,于30 ℃下培养16 h。最后,使用添加有10 μg/mL的安普霉素溶液琼脂培养基进行筛选。利用引物对Ver-F/Ver-R(表1)进行PCR并测序验证,筛选得到成功整合的S.mobaraensis菌株smY2022-Scsn-Csn和smY2022-Stg-Csn。
1.2.3 蛋白表达与纯化
S.mobaraensis在GYM琼脂培养基上30 ℃培养4~5 d。将孢子接种到含有30 mL种子培养基的摇瓶中,于30 ℃和220 r/min培养24 h。取2.4 mL的种子培养基接种到30 mL的发酵培养基中,于30 ℃和220 r/min培养3 d。每12 h取样,于4 ℃和12 000×g离心10 min,收集含有Csn的发酵上清液。由于重组蛋白含有6xHis标签,使用Ni-NTA Purose 6 Fast Flow进行亲和纯化。用洗脱缓冲液(50 mmol/L 2-(N-吗啉代)乙磺酸[2-(N-morpholino)ethanesulfonic acid,MES],150 mmol/L NaCl,100 mmol/L咪唑,pH 6)洗脱Csn。随后,使用截留分子质量10 kDa的超滤管进行浓缩和脱盐。脱盐后的酶液用于下一步分析。
1.2.4 酶学性质测定
将酶液稀释至0.15~0.2 mg/mL后进行酶学性质分析。为测定最适的反应温度,分别在30~80 ℃和pH 5.5测定酶活力;计算各温度下相对于50 ℃和pH 5.5酶活力的百分比。为测定酶热稳定性,将稀释后的酶液在不同温度下分别孵育0~120 min,每30 min取样测定酶活力;计算不同孵育时间样品的相对酶活力,在50 ℃和pH 5.5孵育0 min时的酶活力为100%。为测定最适反应pH,在50 ℃测定不同pH环境的酶活力;pH 3~6使用乙酸-乙酸钠缓冲液,pH 6~7使用MES缓冲液,pH 7~9使用Tris-HCl缓冲液;缓冲液浓度均为200 mmol/L。计算各pH环境的相对酶活力,以50 ℃和pH 5.5的酶活力为100%。为测定pH稳定性,分别使用上述缓冲液稀释酶液,于50 ℃下孵育120 min,测定孵育后残余酶活力;计算不同孵育时间样品的相对酶活力,以50 ℃和pH 5.5孵育0 min时的酶活力为100%。
1.2.5 Csn水解产物分析
为明确产物特异性,将纯化的Csn按终浓度8 U/mL添加至壳聚糖反应体系[1%(体积分数)壳聚糖(脱乙酰度≥95%),200 mmol/L乙酸钠缓冲液,pH 5.5]。酶促反应在50 ℃恒温条件下进行梯度时间处理(0.5~8 h)。反应结束后,立即通过沸水浴(100 ℃)灭活酶活性10 min。经10 000×g离心(4 ℃)10 min分离沉淀物,取上清液通过0.22 μm聚醚砜滤膜超滤除杂。采用HPLC进行产物分析:色谱柱为Asahipak NH2P-50 4E(4.6 mm×250 mm,10 μm),流动相为V(乙腈)∶V(水)=70∶30,在30 ℃恒温条件下以1 mL/min流速洗脱。示差折光检测器实时监测色谱信号,数据采集速率设为1 Hz。
1.2.6 Csn活力测定
Csn活力测定方法在DNS方法基础上进行改良[15]。将350 μL底物溶液[1%(体积分数)壳聚糖,200 mmol/L乙酸缓冲液,pH 5.5]与50 μL Csn酶液混合,于50 ℃下反应15 min后,立刻加入600 μL终止液(DNS)于沸水浴5 min终止反应。向反应液中加入3 mL ddH2O稀释后,测定其520 nm吸光度。酶活力单位(U)被定义为每分钟内产生1 μmol还原糖所需的酶量[氨基葡萄糖盐酸盐(GlcN)作为标准品]。
1.2.7 蛋白质分析
使用Bradford Protein Assay Kit试剂盒进行蛋白浓度测定。将30 μL待测样品和10 μL 5×上样缓冲液混匀,90 ℃加热5 min使蛋白变性。SDS-PAGE分析参考Bis-Tris预制凝胶说明书。蛋白质Marker为PageRulerTM Plus (10~250 kDa)。
1.2.8 生物信息学分析
蛋白质序列比对通过ClustalW和ESPript(http://espript.ibcp.fr/ESPript/ESPript/)完成。信号肽预测通过SignalP 4.1(https://services.healthtech.dtu.dk/service/SignalP-4.1)完成。酶分子三维结构预测通过SWISS-MODEL(http://swissmodel.expasy.org)完成。使用Pymol将其与来自Streptomyces sp.N174的Csn(PDB编号:1CHK)进行三维结构比对,计算两者结构的均方根偏差(root mean square deviation,RMSD)。CRISPOR(http://crispor.tefor.net/)用于靶向S.mobaraensis基因组的sgRNA设计。
2 结果与分析
2.1 smCsn46序列和结构分析
除了Bacillus外,Streptomyces是Csn另一个重要的微生物来源[1]。基于S.mobaraensis DSM40587基因组注释,鉴定出1个推测的Csn基因,并命名为smCsn46(GenBank:UBI35095.1),其开放阅读框(open reading frame,ORF)长度为882 bp。根据SignalP 4.1的预测,smCsn46的ORF编码的前37个氨基酸构成其信号肽序列,成熟酶部分由256个氨基酸残基组成,分子质量约为32 kDa。序列比对分析表明,smCsn46与Streptomyces sp.N174、Streptomyces coelicolor A3(2)、Bacillus mojavensis SY1和Bacillus subtilis 168等来源GH46家族Csn相序列一致性分别为73%、73.9%、49.3%、48.4%。此外,smCsn46中含有GH46家族Csn的关键催化活性位点[16],Glu40和Asp58(图1)。

图1 smCsn46与其他GH46家族Csn的氨基酸序列比对
Fig.1 Amino acid sequence alignment of smCsn46 with other Csns from the GH46 family
注:保守催化残基Asp58和Glu40用三角形突出显示。
基于SWISS-MODEL对smCsn46的三维结构进行预测。如图2-a所示,smCsn46呈现GH46家族Csn典型三维结构,其由一段α螺旋连接一个较小的N端结构域和一个较大的C端结构域,呈不对称哑铃状[16]。表面电荷分析显示,其结构域之间存在一个带负电荷的裂缝状底物结合口袋(图2-b),这有利于与带负电的壳聚糖结合[17]。此外,smCsn46与Streptomyces sp.N174来源的Csn[18]的RMSD值仅为0.635(图2-a),表明两者具有高度相似的三维结构。研究显示,Streptomyces sp.N174来源的Csn对壳聚糖的水解产物主要集中有重要生物活性的3~5糖[6],可以推测smCsn46可能具有类似的产物特异性。
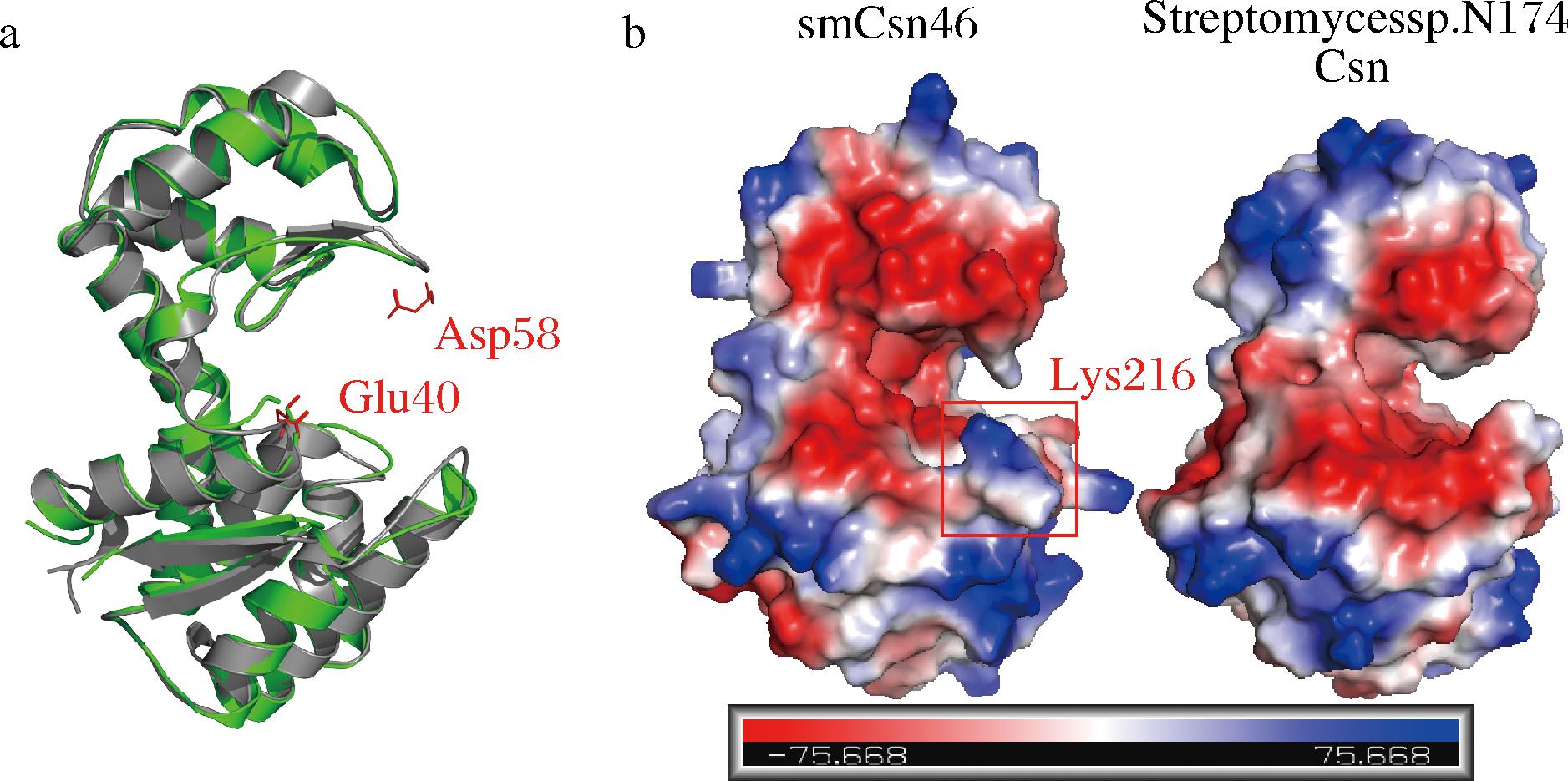
a-SWISS-MODEL根据链霉菌属Csn的晶体结构预测smCsn46的三级结构(绿色),来自Streptomyces sp.N174(PDB ID:1CHK,以灰色显 示);b-smCsn46和来自Streptomyces sp.N174的Csn表面静电荷
图2 smCsn46三级结构及表面静电荷分析
Fig.2 Tertiary structure and electrostatic-potential surfaces analysis of smCsn46
2.2 smCsn46在smY2022Δtg中的过表达
S.mobaraensis长期以来主要用于TGase发酵生产,TGase是其主要的胞外蛋白。前期研究中,通过从S.mobaraensis突变株smY2022中删除TGase基因,构建了一株突变株smY2022Δtg,为外源基因的表达提供了新的宿主[11]。为实现smCsn46在S.mobaraensis中的高效表达,本研究从S.mobaraensis DSM40587基因组中扩增得到包含和不包含内源信号肽的smCsn46基因。基于CRISPR/Cas9系统,将smCsn46基因整合至smY2022Δtg的TGase基因位点,构建了重组菌株smY2022-Scsn-Csn和smY2022-Stg-Csn。这些菌株在TGase启动子和终止子的调控下表达smCsn46(图3-a)。为了评估信号肽对表达的影响,分别使用smCsn46和TGase的信号肽序列表达smCsn46,构建了smY2022-Scsn-Csn和smY2022-Stg-Csn(图3-a)。
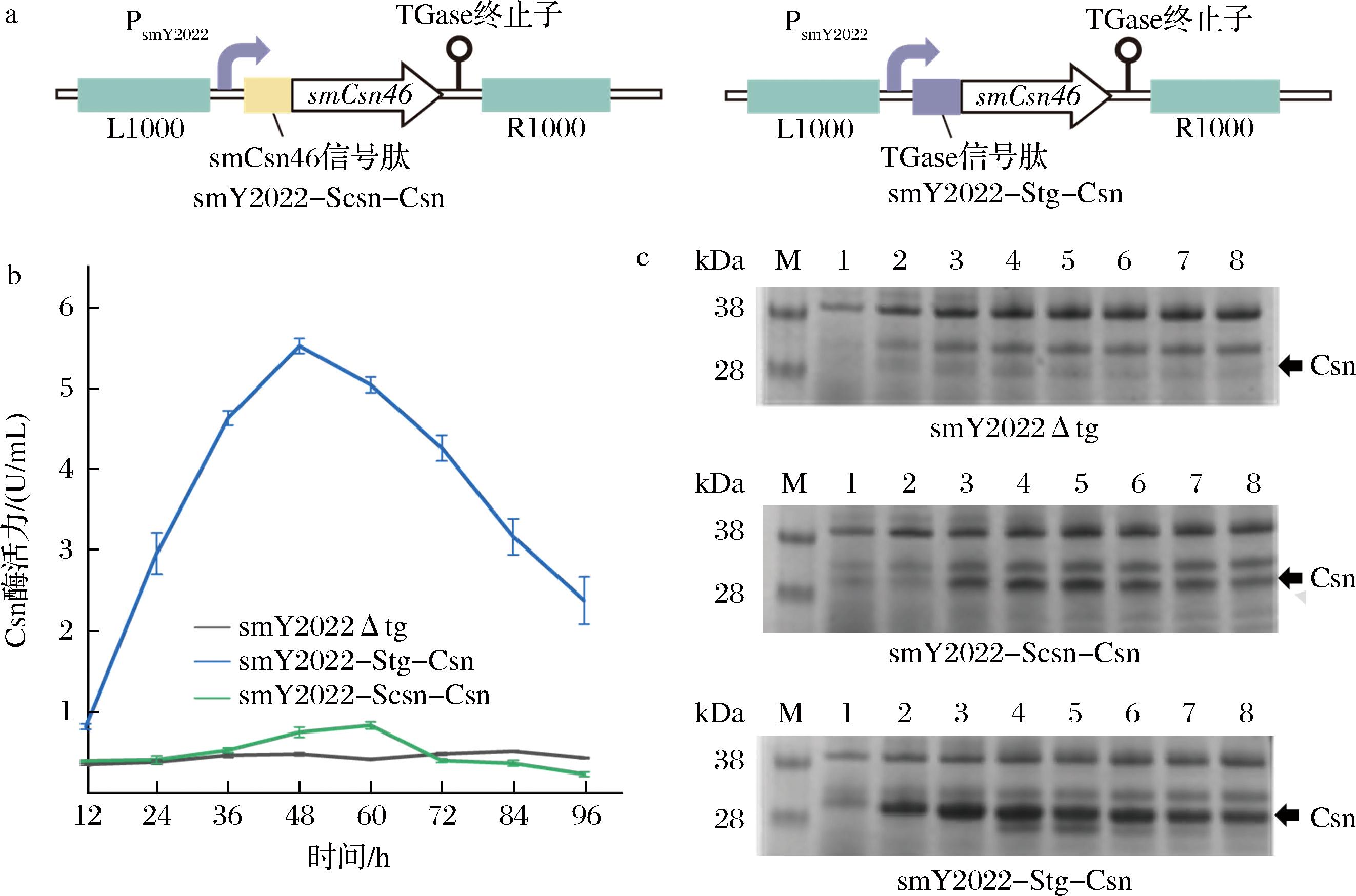
M-蛋白质标准分子质量;泳道1~8-12、24、36、48、60、72、84、96 h。
a-整合到S.mobaraensis菌株基因组中的smY2022-Scsn-Csn和smY2022-Stg-Csn的smCsn46表达框示意图;b-smY2022-Scsn-Csn和smY2022-Stg-Csn的上清液Csn活性;c-smY2022-Scsn-Csn和smY2022-Stg-Csn发酵上清液SDS-PAGE分析
图3 在S.mobaraensis中整合表达smCsn46
Fig.3 Integration and expression of smCsn46 in S.mobaraensis
酶活性测定结果表明,尽管smY2022Δtg基因组中天然存在smCsn46基因,但其胞外Csn活性几乎检测不到(图3-b)。相比之下,smY2022-Scsn-Csn在发酵60 h后,胞外Csn活性为0.83 U/mL(图3-b)。而在smY2022-Stg-Csn中,Csn活性迅速上升,在48 h达到峰值5.52 U/mL。发酵24 h后,smY2022-Stg-Csn上清液具明显的smCsn46蛋白条带(32 kDa),而smY2022-Scsn-Csn和smY2022Δtg上清液的smCsn46蛋白条带则显著较浅(图3-c)。这些结果表明,采用TGase启动子和终止子能实现smCsn46在S.mobaraensis中的分泌表达,且TGase信号肽较Csn信号肽更有利于smCsn46的分泌表达。
本研究构建了能够生产Csn的重组S.mobaraensis菌株,其Csn活性显著高于食品级宿主S.lividans(0.461 U/mL)[8]和S.cerevisiae(2 U/mL)[9]。值得注意的是,CRISPR/Cas9编辑系统未在smY2022-Stg-Csn中引入任何抗性标记。此外,smY2022-Stg-Csn以成本较低的水产副产物鱼粉为主要氮源。因此,smY2022-Stg-Csn在食品级Csn的工业化生产中具有显著的应用潜力。在未来研究中,计划通过优化表达元件和提高基因拷贝数,进一步提高smCsn46在S.mobaraensis中的表达水平。
2.3 smCsn46的生化表征
由于在smCsn46的C端融合了His-tag,利用镍柱从smY2022-Stg-Csn发酵上清液中纯化得到smCsn46,并分析了其最适反应条件和稳定性。结果显示,smCsn46最适温度为50 ℃(图4-a)。值得注意的是,smCsn46热稳定性较好,在50 ℃下孵育2 h后仍能保持95%以上的酶活力,而在60 ℃下其失活速度显著加快(图4-b)。相较而言,来自B.subtilis 168的GH46家族Csn相同条件下孵育30 min完全失活[19]、来自杆状链霉菌(Streptomyces bacillaris)的GH5家族Csn在50 ℃孵育2 h后几乎完全失活[20]、来自草酸青霉菌(Penicillium oxalicum M2)的GH75家族Csn在50 ℃条件下和smCsn46显示出相近的稳定性,但在60 ℃下稳定性很差,孵育2 h后失活[21]。此外,smCsn46最适pH值为5.5(图4-c),并表现出较高的pH稳定性,即使在pH 3条件下处理2 h后仍能保持70%以上的残余活性(图4-d)。这与多数GH46家族的Csn性质一致[22],例如来自B.subtilis 168的Csn,最适pH值为5,且在酸性条件下稳定性良好[19]。鉴于壳聚糖在酸性条件下溶解度增强(pH<6)[23],smCsn46优秀的耐酸性和耐热性使其成为COS生产的潜在应用候选者。
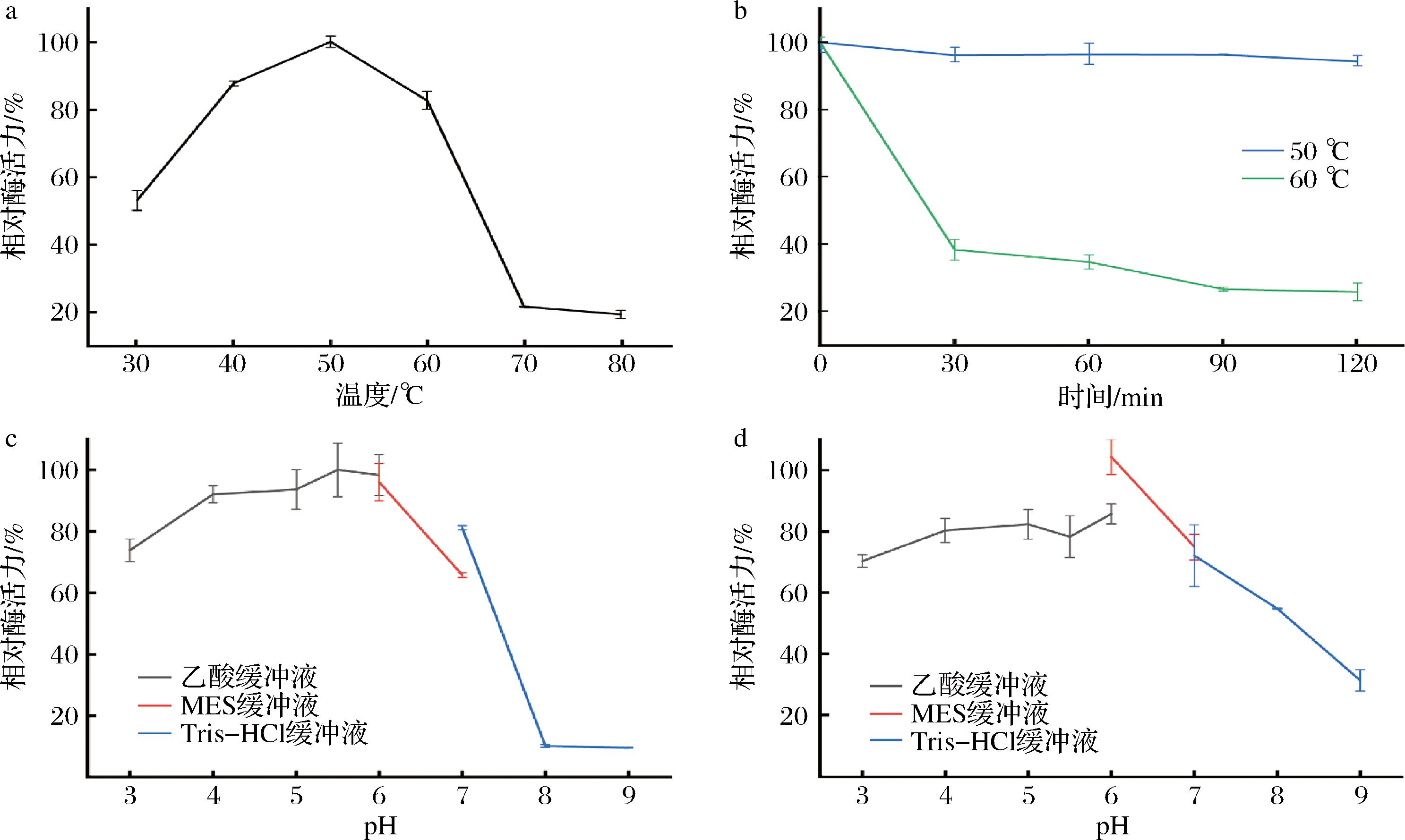
a-最适反应温度;b-温度稳定性;c-最适反应pH;d-pH稳定性
图4 由S.mobaraensis 表达的smCsn46酶学性质
Fig.4 Characterization of smCsn46 expressed by S.mobaraensis
接着,本研究对smCsn46的底物特异性进行了分析。如图5-a所示,smCsn46对85%~95% DDA的壳聚糖均具有催化活性,且随着DDA降低而增加,但对几丁质没有活性。其中,smCsn46对85% DDA的壳聚糖的活性达到44.36 U/mg。这与在Csn中观察到的常见趋势相反。例如,来自Bacillus sp.strain KCTC 0377BP[15]和Streptomyces sp.N174[18]的Csn对高DDA的壳聚糖活性更高。普遍认为,高DDA的壳聚糖由于亲水性氨基的暴露增加,溶解性更好,从而更有利于Csn的催化[24]。有趣的是,smCsn46与来自Streptomyces sp.N174的Csn虽然具有高度相似的整体结构,但来自Streptomyces sp.N174的Csn底物袋主要为带负电荷的氨基酸,而smCsn46具有独特的带正电荷的残基Lys216(图2-b)。推测smCsn46底物袋内的这个带正电的区域可能会与壳聚糖中存在的带正电的氨基产生静电排斥。这种排斥力可能会随着壳聚糖DDA的增加而增强,从而降低酶的催化活性。未来的研究将通过对Lys216定点突变,进一步阐明这一特定残基对smCsn46底物特异性的影响。
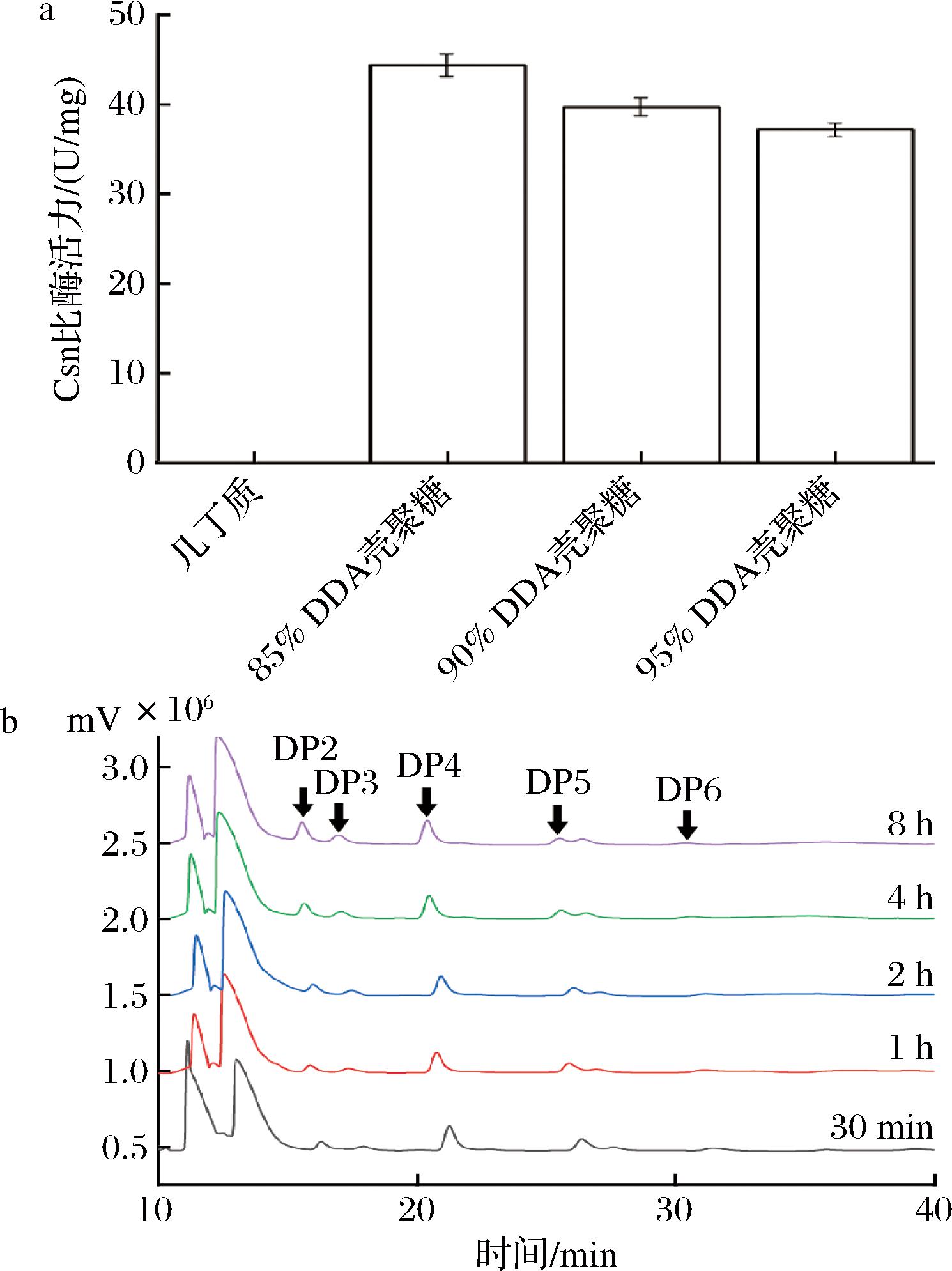
a-smCsn46底物特异性(使用具有不同DD的各种壳聚糖底物测量smCsn46的酶活力,以评估其底物偏好);b-对smCsn46 催化的95% DD的壳聚糖水解产物进行HPLC分析[聚合度(degree of polymeri- zation,DP)范围为 2~6 的产品用箭头表示]
图5 smCsn46底物特异性和水解产物分析
Fig.5 Substrate specificity and hydrolysis products of smCsn46
最后,以DDA为95%的壳聚糖为底物,测定了smCsn46的产物特异性。如图5-b所示,smCsn46催化反应30 min后,壳聚糖的主要水解产物为壳四糖,其次是壳五糖和壳二糖。随着反应进行,壳五糖的含量显著减少,同时壳二糖和壳三糖的含量显著增加(图5-b)。值得注意的是,与之前的报道不同,壳四糖的浓度在整个反应过程中保持相对稳定(图5-b)。这种产物分布模式与之前的研究报道有所不同。例如,来自Streptomyces sp.N174的SsCsn46经改造,主要产物为壳五糖[6],而Gynuella sunshinyii来源的Csn则主要生成壳二糖和壳三糖,两者占总产物的94.6%[25]。目前,COS应用范围广泛,其中壳四糖因具有显著的抗炎活性而备受关注,而本研究中的壳聚糖酶smCsn46能够特异性生成壳四糖作为主要产物,进一步凸显其在抗炎领域的应用潜力。鉴于聚合度在调控COS生物活性中的重要作用,未来的研究工作将重点评估smCsn46催化COS的抗氧化及其他生物活性。
3 结论
本研究鉴定并表征了一种来自于S.mobaraensis DSM40587的GH46家族的Csn。序列分析和结构分析表明,smCsn46与Streptomyces sp.N174和S.coelicolor A3(2)来源的Csn具有高度同源性和相似性。通过在工程菌株smY2022Δtg中利用TGase的启动子、信号肽和终止子进行过表达,实现了高效的分泌表达和高酶活力,显示出其在食品级壳寡糖酶生产中的工业潜力。smCsn46对酸性和高温环境表现出偏好,并主要生成四糖。总体而言,本研究加深了对GH46家族Csn的理解,并为COS的生产提供了新的途径。
[1] THADATHIL N, VELAPPAN S P.Recent developments in chitosanase research and its biotechnological applications:A review[J].Food Chemistry, 2014, 150:392-399.
[2] CUI D D, YANG J, LU B S, et al.Efficient preparation of chitooligosaccharide with a potential chitosanase csn-SH and its application for fungi disease protection[J].Frontiers in Microbiology, 2021, 12:682829.
[3] LIANG D C, LIU W G, ZUO A J, et al.Pre-deliver chitosanase to cells:A novel strategy to improve gene expression by endocellular degradation-induced vector unpacking[J].International Journal of Pharmaceutics, 2006, 314(1):63-71.
[4] CHITNIS M V, DESHPANDE M V.Isolation and regeneration of protoplasts from the yeast and mycelial form of the dimorphic zygomycete Benjaminiella poitrasii:Role of chitin metabolism for morphogenesis during regeneration[J].Microbiological Research, 2002, 157(1):29-37.
[5] ZHOU Z P, ZHAO S Z, WANG S Q, et al.Extracellular overexpression of chitosanase from Bacillus sp.TS in Escherichia coli[J].Applied Biochemistry and Biotechnology, 2015, 175(7):3271-3286.
[6] DING M, ZHANG T, SUN C, et al.A Chitosanase mutant from Streptomyces sp.N174 prefers to produce functional chitopentasaccharide[J].International Journal of Biological Macromolecules, 2020, 151:1091-1098.
[7] JIANG Z Q, MA S A, GUAN L Y, et al.Biochemical characterization of a novel bifunctional chitosanase from Paenibacillus barengoltzii for chitooligosaccharide production[J].World Journal of Microbiology and Biotechnology, 2021, 37(5):83.
[8] GHINET M G, ROY S, POULIN-LAPRADE D, et al.Chitosanase from Streptomyces coelicolor A3(2):Biochemical properties and role in protection against antibacterial effect of chitosan[J].Biochimie et Biologie Cellulaire, 2010, 88(6):907-916.
[9] ZHANG Q S, CAO H.Expression of chitosanase from Aspergillus fumigatus chitosanase in Saccharomyces cerevisiae by CRISPR-Cas9 tools[J].Bioresources and Bioprocessing, 2024, 11(1):20.
[10] STROP P.Versatility of microbial transglutaminase[J].Bioconjugate Chemistry, 2014, 25(5):855-862.
[11] YE J C, YANG P H, ZHOU J W, et al.Efficient production of a thermostable mutant of transglutaminase by Streptomyces mobaraensis[J].Journal of Agricultural and Food Chemistry, 2024, 72(8):4207-4216.
[12] ROMEIH E, WALKER G.Recent advances on microbial transglutaminase and dairy application[J].Trends in Food Science &Technology, 2017, 62:133-140.
[13] YANG H L, HE T S, WU W X, et al.Whole-genome shotgun assembly and analysis of the genome of Streptomyces mobaraensis DSM 40847, a strain for industrial production of microbial transglutaminase[J].Genome Announcements, 2013, 1(2):e0014313.
[14] COBB R E, WANG Y J, ZHAO H M.High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system[J].ACS Synthetic Biology, 2015, 4(6):723-728.
[15] CHOI Y J, KIM E J, PIAO Z, et al.Purification and characterization of chitosanase from Bacillus sp.strain KCTC 0377BP and its application for the production of chitosan oligosaccharides[J].Applied and Environmental Microbiology, 2004, 70(8):4522-4531.
[16] VIENS P, LACOMBE-HARVEY M  , BRZEZINSKI R.Chitosanases from family 46 of glycoside hydrolases:From proteins to phenotypes[J].Marine Drugs, 2015, 13(11):6566-6587.
, BRZEZINSKI R.Chitosanases from family 46 of glycoside hydrolases:From proteins to phenotypes[J].Marine Drugs, 2015, 13(11):6566-6587.
[17] CHEN H, LIN B L, ZHANG R, et al.Controllable preparation of chitosan oligosaccharides via a recombinant chitosanase from marine Streptomyces lydicus S1 and its potential application on preservation of pre-packaged tofu[J].Frontiers in Microbiology, 2022, 13:1007201.
[18] BOUCHER I, DUPUY A, VIDAL P, et al.Purification and characterization of a chitosanase from Streptomyces N174[J].Applied Microbiology and Biotechnology, 1992, 38(2):188-193.
[19] PECHSRICHUANG P, LORENTZEN S B, AAM B B, et al.Bioconversion of chitosan into chito-oligosaccharides (CHOS) using family 46 chitosanase from Bacillus subtilis (BsCsn46A)[J].Carbohydrate Polymers, 2018, 186:420-428.
[20] SU H P, SUN J N, CHU W Q, et al.Biochemical characterization and cleavage pattern analysis of a novel chitosanase with cellulase activity[J].Applied Microbiology and Biotechnology, 2022, 106(5):1979-1990.
[21] CAO S N, GAO P, XIA W S, et al.A novel chitosanase from Penicillium oxalicum M2 for chitooligosaccharide production:Purification, identification and characterization[J].Molecular Biotechnology, 2022, 64(9):947-957.
[22] LIANG J Y, HE S B, SUN J, et al.Secretory production and characterization of a highly effective chitosanase from Streptomyces coelicolor A3(2) M145 in Pichia pastoris[J].Biotechnology Journal, 2024, 19(2):2300402.
[23] SOGIAS I A, KHUTORYANSKIY V V, WILLIAMS A C.Exploring the factors affecting the solubility of chitosan in water[J].Macromolecular Chemistry and Physics, 2010, 211(4):426-433.
[24] YU M, ZHANG K, GUO X, et al.Effects of the degree of deacetylation on the single-molecule mechanics of chitosans[J].The Journal of Physical Chemistry B, 2023, 127(19):4261-4267.
[25] QIN Z, CHEN Q M, LIN S, et al.Expression and characterization of a novel cold-adapted chitosanase suitable for chitooligosaccharides controllable preparation[J].Food Chemistry, 2018, 253:139-147.