几丁二糖[chitobiose, (GlcNAc)2]又被称为N,N′-二乙酰壳二糖,由2个N-乙酰氨基葡萄糖(N-acetyl-D-glucosamine, GlcNAc)通过β-1,4糖苷键聚合而的壳寡糖[1]。壳寡糖具有许多生物活性,包括抗肿瘤、抗氧化、抗炎、抗菌、免疫刺激和改善肠道微生态活性[2]。因此,它们在农业、食品、制药、化妆品等多个领域得到了广泛应用,如作为膳食补充剂、骨关节炎治疗剂、化妆品成分等[3-4]。此外,壳寡糖作为药物生物活性产物,已证明具有良好的降血糖作用、降脂活性和螯合能力[5]。这些功能评估都是针对构成几丁寡糖的不同大小的混合物,因此每种不同聚合度的几丁寡糖的作用有待进一步拓展[6]。
目前主要通过酸或酶处理来将几丁质低分子化得到几丁二糖[7-8],然而强酸处理水解并不环保,并且难以控制分子大小。用酶水解也很难得到相应分子量的几丁寡糖,反应控制非常困难。因此本文采用全细胞催化法的混菌耦合发酵直接用合成壳二糖,减少了酸水解甲壳素法中所用强酸对环境的污染,且不需要添加过多的底物及营养物质,可更容易实现目的产物的分离纯化。
作为能降解高聚物几丁质生成(GlcNAc)2和GlcNAc的水解酶[9],几丁质酶根据其裂解几丁质链的作用方式分为3种类型(内切几丁质酶、外切几丁质酶和N-乙酰氨基葡萄糖苷酶)[10],大部分几丁质酶是在链霉菌属和弧菌科中发现的[11],这些系统通过磷酸烯醇丙酮酸磷酸转移酶系统(phosphotransferase system, PTS)将单糖转入到细胞体内,几丁二糖、三糖和低聚糖是通过ATP结合盒(ATP binding cassette transpertor, ABC)转运蛋白转运进入细胞。霍乱弧菌几丁质酶产生的几丁质低聚糖通过跨膜运输系统转运到细胞周质[12],进一步分解为GlcNAc和(GlcNAc)2[13]。(GlcNAc)2是弗尼斯弧菌(Furnissii)分解代谢几丁质过程中重要的信号传导,在参与信号传导的过程中能诱导产生许多蛋白质参与到几丁质分解代谢过程[14]。
糖基转移酶已用于这些重要碳水化合物的酶促合成,糖基转移酶因其区域和立体选择性优势,在碳水化合物的酶促合成中得到了广泛应用。与传统化学合成和几丁质酶相比,糖基转移酶无需保护官能团即可实现高效合成[11]。N,N′-二乙酰壳二糖磷酸化酶(N,N′-diacetylchitobiose phosphorylase, Chbp)就作为弗尼斯弧菌(Vibrio furnissii)中的一种催化GlcNAc和GlcNAc-1-P转化为(GlcNAc)2的糖基转移酶,首次在2000年被PARK等[15]发现,对1-P-Glucose也有活性,但活性不到对GlcNAc-1-P的10%[16],所以利用双歧杆菌(Bifidobacteria)中N-乙酰己糖胺激酶(N-acetylhexosamine 1-kinase, Nahk)催化GlcNAc为GlcNAc-1-P[17]作为底物,后加入大肠杆菌来源的多聚磷酸酶(polyphosphate kinase, PPK)[18]使ATP与ADP间互相转换,实现能荷再生,形成循环。基于上述背景,本研究提出了利用三菌耦合策略的全细胞催化法来实现(GlcNAc)2的经济高效合成,其具体催化流程如图1所示。该方法不仅提高了几丁二糖的产量,还为大规模工业化生产提供了新的思路和技术支持。

图1 三菌耦合合成(GlcNAc) 2流程图
Fig.1 Flowchart for the coupled synthesis of (GlcNAc)2 by three bacteria
1 材料与方法
1.1 材料与试剂
GlcNAc,国药集团化学试剂有限公司;(GlcNAc)2,上海泰坦科技股份有限公司;腺嘌呤核苷三磷酸(adenosine triphosphate, ATP),杭州凯朋生物技术有限公司;卡那霉素(kanamycin, kan),北京伊诺凯科技有限公司;氨苄青霉素(ampicillin, amp),北京伊诺凯科技有限公司。
1.2 仪器与设备
CFX-96 PCR仪,美国Bio-Rad公司;neofleX 基质辅助激光解析电离串联飞行时间质谱仪,美国布鲁克·道尔顿公司;H2500R 高速冷冻离心机,湖南湘仪实验室仪器开发有限公司;WIX-EP300 蛋白电泳仪,上海达燊实业有限公司;1220 Infinity Ⅱ高效液相色谱仪,安捷伦科技公司;Dionex ICS-6000 高压离子色谱系统,美国赛默飞世尔科技公司。
1.3 实验方法
1.3.1 质粒与菌株
本实验所用的质粒和菌株见表1。
表1 本文所用菌株的来源及构建
Table 1 Source and construction of the strains used in this article
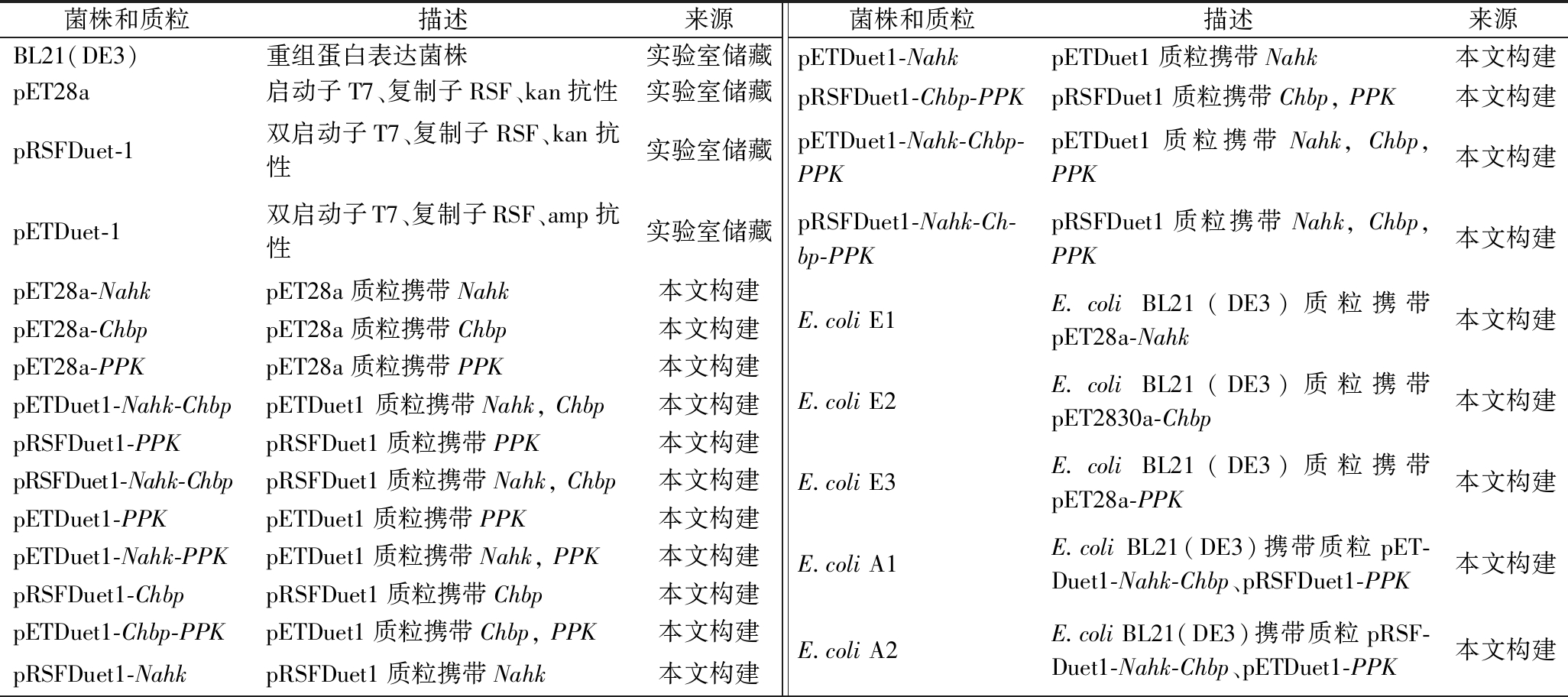
菌株和质粒描述来源菌株和质粒描述来源BL21(DE3)重组蛋白表达菌株实验室储藏pET28a启动子T7、复制子RSF、kan抗性实验室储藏pRSFDuet-1双启动子T7、复制子RSF、kan抗性实验室储藏pETDuet-1双启动子T7、复制子RSF、amp抗性 实验室储藏pET28a-NahkpET28a质粒携带Nahk本文构建pET28a-ChbppET28a质粒携带Chbp本文构建pET28a-PPKpET28a质粒携带PPK本文构建pETDuet1-Nahk-ChbppETDuet1 质粒携带Nahk, Chbp本文构建pRSFDuet1-PPKpRSFDuet1质粒携带PPK本文构建pRSFDuet1-Nahk-ChbppRSFDuet1质粒携带Nahk, Chbp本文构建pETDuet1-PPKpETDuet1质粒携带PPK本文构建pETDuet1-Nahk-PPKpETDuet1质粒携带Nahk, PPK本文构建pRSFDuet1-ChbppRSFDuet1质粒携带Chbp本文构建pETDuet1-Chbp-PPKpETDuet1质粒携带Chbp, PPK本文构建pRSFDuet1-NahkpRSFDuet1质粒携带Nahk本文构建pETDuet1-NahkpETDuet1质粒携带Nahk本文构建pRSFDuet1-Chbp-PPKpRSFDuet1质粒携带Chbp, PPK本文构建pETDuet1-Nahk-Chbp-PPKpETDuet1质粒携带Nahk, Chbp, PPK本文构建pRSFDuet1-Nahk-Ch-bp-PPKpRSFDuet1质粒携带Nahk, Chbp, PPK本文构建E.coli E1E.coli BL21(DE3)质粒携带pET28a-Nahk本文构建E.coli E2E.coli BL21(DE3)质粒携带pET2830a-Chbp本文构建E.coli E3E.coli BL21(DE3)质粒携带pET28a-PPK本文构建E.coli A1E.coli BL21(DE3)携带质粒pET-Duet1-Nahk-Chbp、pRSFDuet1-PPK本文构建E.coli A2E.coli BL21(DE3)携带质粒pRSF-Duet1-Nahk-Chbp、pETDuet1-PPK本文构建
续表1
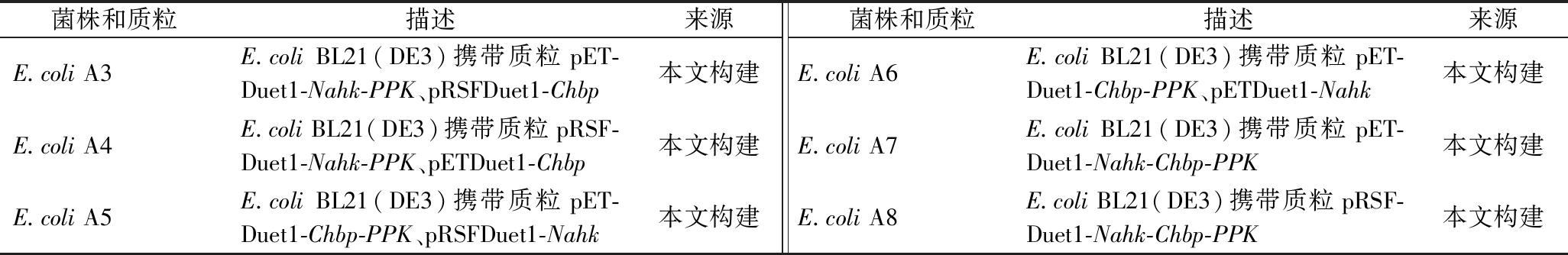
菌株和质粒描述来源菌株和质粒描述来源E.coli A3E.coli BL21(DE3)携带质粒pET-Duet1-Nahk-PPK、pRSFDuet1-Chbp本文构建E.coli A4E.coli BL21(DE3)携带质粒pRSF-Duet1-Nahk-PPK、pETDuet1-Chbp本文构建E.coli A5E.coli BL21(DE3)携带质粒pET-Duet1-Chbp-PPK、pRSFDuet1-Nahk本文构建E.coli A6E.coli BL21(DE3)携带质粒pET-Duet1-Chbp-PPK、pETDuet1-Nahk本文构建E.coli A7E.coli BL21(DE3)携带质粒pET-Duet1-Nahk-Chbp-PPK本文构建E.coli A8E.coli BL21(DE3)携带质粒pRSF-Duet1-Nahk-Chbp-PPK本文构建
1.3.2 培养基和培养条件
LB液体培养基(g/L):酵母提取物5、胰蛋白胨10、氯化钠10,琼脂20,pH 7.0,121 ℃、20 min下灭菌。
TB液体培养基(g/L):胰蛋白胨11.8、酵母提取物23.6、K2HPO4 9.4、KH2PO4 2.2、4 mL甘油,离子与培养基其他组分分开在121 ℃、20 min条件下灭菌。
1.3.3 工程菌的构建
以双歧杆菌中Nahk(GenBank ID:AB303839.1)为模版合成目的基因片段,设计引物Nahk-F(5′-CCATGGGCAAAAAAATCCTGACTGTGCTGTCT-3′)和FnahkR(5′-GGTGGTGCTCGAGTCACTTGGTCGTCTC-3′)对目的基因Nahk进行PCR扩增,设计引物pET-28a-F(5′-CATTGGTGTTGTTCATATGGCTGC-3′)和pET-28a-R(5′-GACGACCAAGTGACTCGAGCACCAC-3′)对载体pET-28a进行PCR扩增,验证回收后,用ClonExpress技术,将PCR后的Nahk基因片段与载体片段纯化连接,得到重组质粒pET28a-Nahk。再将其转化到感受态细胞E.coli BL21(DE3)中,以kan为筛选标记进行平板涂布,经过菌落PCR验证后挑取单菌落进行摇瓶培养,提取菌液中的质粒酶切验证,并送去公司(苏州金唯智生物科技)测序鉴定。同理以弗尼斯弧菌中的Chbp(GenBank ID:AF230379.1)和大肠杆菌来源的PPK(GenBank ID:L03719.1)构建pET28a-Fkp、pET28a-PPK。
1.3.4 重组蛋白的表达与验证
将工程菌株于kan板上划板过夜培养,挑取单菌落于10 mL含有50 μg/mL卡那霉素的LB液体培养基内37 ℃、200 r/min培养12~16 h,再在100 mL含有50 μg/mL卡那霉素的TB液体培养基内37 ℃、200 r/min培养至OD600值在0.6~0.8后加入终浓度为0.3 mmol/L的异丙基-β-D-1-硫代半乳糖苷(IPTG),在16 ℃、200 r/min培养24 h,离心收集菌体。
蛋白质用十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gel electrophoresis, SDS-PAGE)(120 g/L丙烯酰胺)进行分析,用考马斯亮蓝法(0.25 g/L考马斯亮蓝R-250、450 mL/L甲醇、100 mL/L冰乙酸、450 mL/L H2O)染色。
1.3.5 全细胞催化反应体系
E.coli BL21(DE3)/pET28a-Nahk催化合成GlcNAc-1-P:100 mmol/L Tris-HCl(pH 8.0)、50 mmol/L ATP、20 mmol/L MgCl2、50 mmol/L GlcNAc、20 mmol/L Poly-P、20 mL/L酒精、10 mmol/L DTT、10 g/L十八胺聚氧乙烯醚,50 g/L的工程菌(E.coli BL21(DE3)/pET28a-Nahk),37 ℃、200 r/min反应4 h。
E.coli BL21(DE3)/pET28a-Nahk和E.coli BL21(DE3)/pET28a-Chbp耦合合成(GlcNAc)2:100 mmol/L Tris-HCl(pH 8.0)、50 mmol/L ATP、20 mmol/L MgCl2、20 mmol/L Poly-P、20 mL/L酒精、10 mmol/L DTT、10 g/L十八胺聚氧乙烯醚、60 mmol/L GlcNAc、50 g/L的工程菌(E.coli BL21(DE3)/pET28a-Nahk和E.coli BL21(DE3)/pET28a-Chbp),30 ℃、200 r/min反应8 h。
E.coli BL21(DE3)/pET28a-PPK活性验证:100 mmol/L Tris-HCl(pH 8.0);50 mmol/L ADP;20 mmol/L MgCl2;50 mmol/L 六偏磷酸钠、50 g/L的工程菌,在37 ℃、200 r/min反应4 h。
E.coli BL21(DE3)/pET28a-Nahk、E.coli BL21(DE3)/pET28a-Chbp、E.coli BL21(DE3)/pET28a-PPK三菌耦合合成(GlcNAc)2:50 g/L的工程菌,在100 mmol/L KH2PO4/K2HPO4(pH 8.0)、50 mmol/L ATP、20 mmol/L MgCl2、20 mmol/L Poly-P、20 mL/L酒精、10 mmol/L DTT、10 g/L十八胺聚氧乙烯醚、100 mmol/L GlcNAc,37 ℃、200 r/min条件下反应24 h。
双质粒表达菌株全细胞催化合成(GlcNAc)2:50 g/L的工程菌,在100 mmol/L KH2PO4/K2HPO4(pH 7.5)、50 mmol/L ATP、20 mmol/L MgCl2、20 mmol/L Poly-P、20 mL/L酒精、10 mmol/L DTT、10 g/L十八胺聚氧乙烯醚、100 mmol/L GlcNAc,37 ℃、200 r/min条件下反应3 h。
1.3.6 底物的合成验证
GlcNAc-1-P:基质辅助激光电离飞行时间质谱(matrix-assisted laser desorption ionization-time of flight mass spectrometry, MALDI-TOF-MS)对纯化产物进行分子量鉴定。点样基质为2,5-二羟基苯甲酸(Na+);点样量:点样基质与样品各取1 μL后混合阴干;质谱条件:使用反射阳离子模式;扫描分子质量范围:100~500 Da。
(GlcNAc)2的检测:高压离子色谱(high pressure ion chromatography, HPIC)检测,反应结束后,12 000 r/min离心5 min收集上清液,经一定比例稀释后的上清液经0.22 μm水系滤膜过滤,由HPLC检测。色谱柱为Aminex HPX-87H Ion Exclusion Column(7.8 mm×300 mm);检测器为UV检测器,紫外检测波长为210 nm;流动相为5 mmol/L H2SO4;柱温为60 ℃;进样量为10 μL;流速为0.6 mL/min;分析时间为30 min。
薄层层析(thin layer chromatography, TLC)检测。展开剂:V(正丁醇)∶V(乙酸)∶V(水)=2∶1∶1;显色剂:茴香醛染料(900 mL/L体积分数为95%乙醇,30 mL/L对茴香醛溶液,50 mL/L质量分数为97%浓硫酸)。待展开剂向上展开约90%的薄板长度后取出,自然晾干后,使用茴香醛染料浸染,放置160 ℃烘箱2~3 min进行显色。
2 结果与分析
2.1 单质粒菌株重组蛋白的表达与验证
本研究将经过密码子优化后合成得到的目的基因Nahk、Chbp和PPK克隆到质粒pET28a上,并进行测序,测序结果与合成基因序列完全一致,表明重组质粒pET28a-Nahk、pET28a-Chbp、pET28a-PPK、构建成功,3株工程菌表达的目的蛋白分子质量理论值分别为40、80、28 kDa,重组菌株中酶的诱导表达情况通过SDS-PAGE来进行验证,见图2-b所示。从图2可以看出诱导表达蛋白其电泳结果显示目的条带分子质量与报道的一致,这说明所有蛋白在E.coli BL21(DE3)中均成功高效表达。
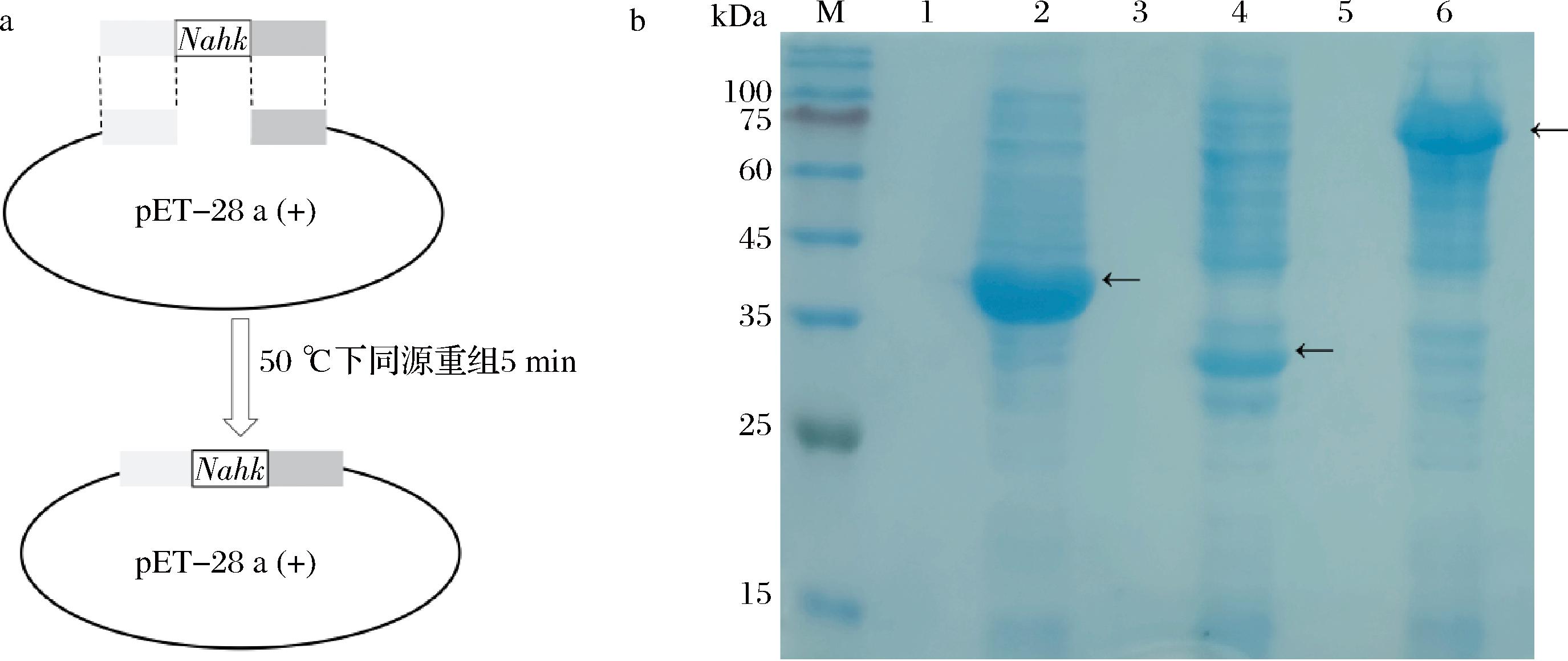
M-蛋白质质量标准溶液;1-Nahk诱导前;2-Nahk诱导后;3-PPK诱导前;4-PPK诱导后;5-Chbp诱导前;6-Chbp诱导后。
a-单表达质粒的构建;b-重组蛋白的SDS-PAGE图
图2 重组蛋白的构建与表达SDS-PAGE图
Fig.2 SDS-PAGE diagram of recombinant protein
2.2 GlcNAc-1-P的合成
在以N-乙酰氨基葡萄糖为唯一底物的情况下判断E.coli BL21(DE3)/pET28a-Nahk菌株的催化能力。如图3所示,因为没有GlcNAc-1-P的标品,利用基质辅助激光电离飞行时间质谱法检测,GlcNAc-1-P的相对分子质量为301,一级质谱图结果显示目的产物在m/z=346.395[M+2 Na-H]+出现离子峰初步判断GlcNAc-1-P的合成。证实了E.coli BL21(DE3)/pET28a-Nahk菌株在以N-乙酰氨基葡萄糖为底物的条件下,能够将GlcNAc转化为GlcNAc-1-P。
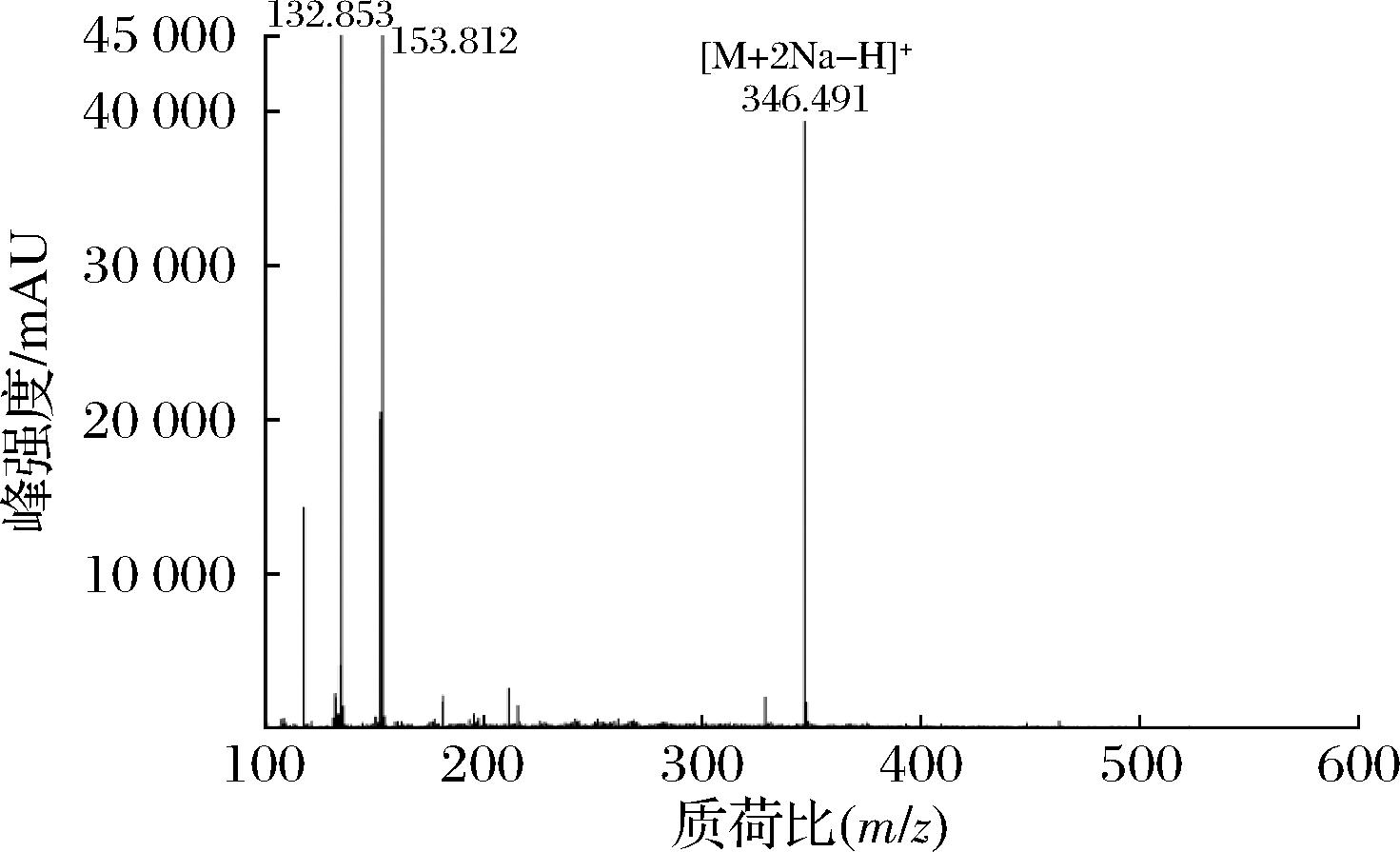
图3 GlcNAc-1-P的质谱分析图
Fig.3 MALDI-TOF MS of GlcNAc-1-P
2.3 双菌耦合合成几丁二糖
为验证Nahk和Chbp酶的活性,使E.coli BL21(DE3)/pET28a-Nahk和E.coli BL21(DE3)/pET28a-Chbp双菌耦合全细胞催化反应,得到反应液。对反应液进行TLC初步验证,如图4-a,反应液中存在与标品几丁二糖相同比移值(retention factor value, Rf)的斑点,初步判断有几丁二糖的合成。为了进一步验证和对几丁二糖的产量进行定量分析,引入HPIC法检测,如图4-b所示,得到双菌耦合几丁二糖的具体产量为1 450 mg/L。发现反应液中除了生成了(GlcNAc)2之外,还残留了一定量的GlcNAc。这一结果表明,尽管Nahk和Chbp的耦合催化体系能够有效地将GlcNAc转化为(GlcNAc)2,但反应并未完全进行到底,仍有部分GlcNAc未能被完全转化。这可能与反应条件的优化不足、酶的活性限制或底物浓度过低等因素有关。
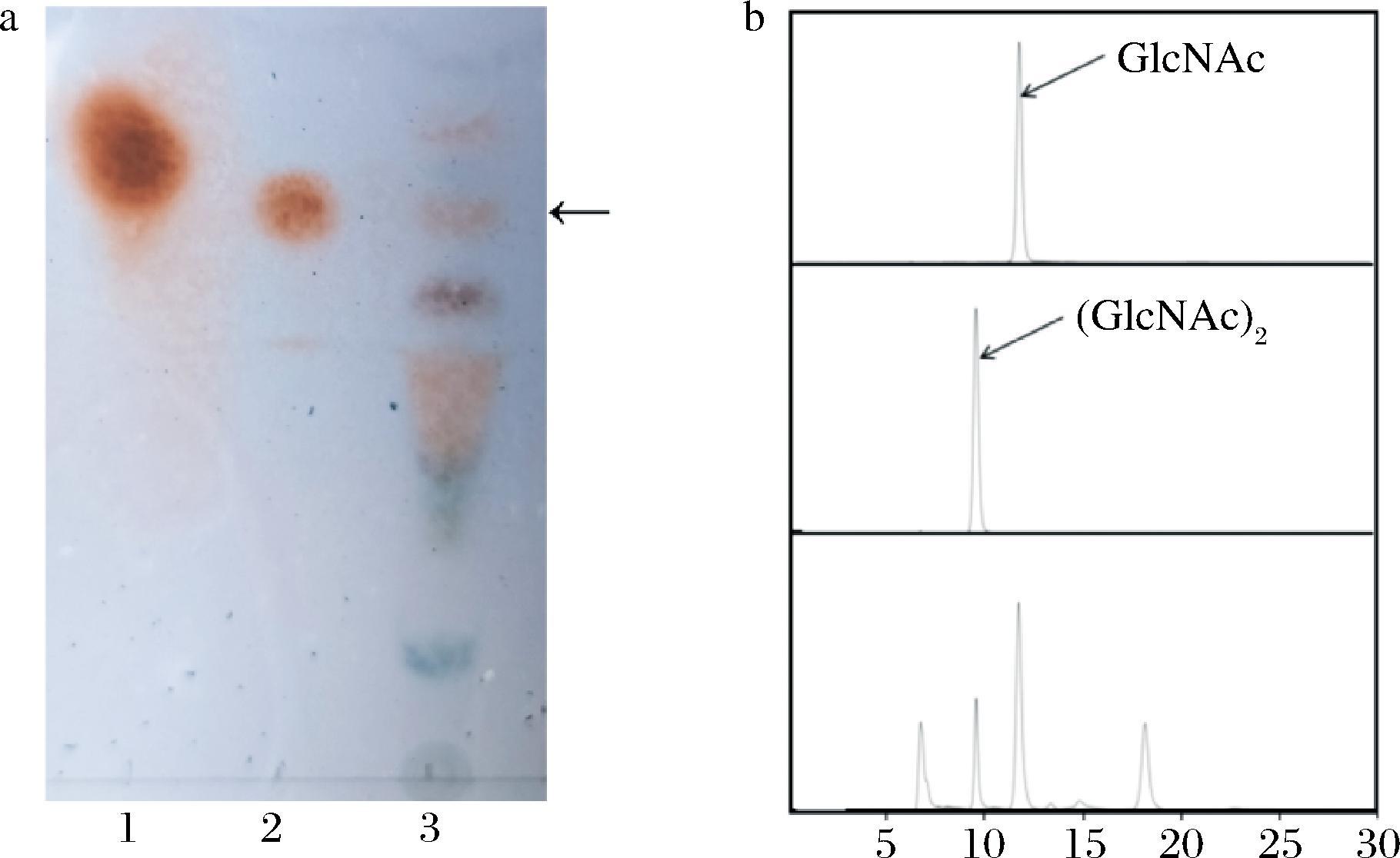
a-验证几丁二糖的合成的TLC图(1-GlcNAc标品,2-(GlcNAc)2标品,3-双菌耦合反应液);b-验证几丁二糖的合成的液相图
图4 验证几丁二糖的合成
Fig.4 Validation of the synthesis of (GlcNAc)2
2.4 三菌耦合合成(GlcNAc)2
为了进一步能核再生,降低反应成本,引入PPK使ATP与ADP间形成循环,使反应更充分进行。先以ADP为底物,验证PPK的活性,后将PPK加入到三菌耦合反应中验证其循环的可行性。通过液相检测,在同浓度GlcNAc反应条件下就算降低ATP的量也不会减少(GlcNAc)2的合成,确定三菌耦合循环的可行性。
为了确定三菌耦合的可行性,加入不同的ATP量比较E.coli E1和E.coli E2双菌耦合与E.coli E1-E.coli E3三菌耦合的全细胞催化下的几丁二糖的产量。分别在反应体系中加入不同浓度的ATP,以考察ATP浓度对几丁二糖合成的影响。如图5-b,在低浓度ATP条件下,几丁二糖的产量较低且双菌耦合的产量远远低于三菌耦合产量,表明ATP的供应不足限制了反应的进行,在一定程度内,随着ATP浓度的增加,在ATP浓度为50 mmol/L时,几丁二糖的产量得到提高,提高到1 675 mg/L。但当ATP浓度进一步增加到100 mmol/L时,产量反而有所下降。推测可能是离子浓度太高,限制了反应的进行。因为ATP为酸性物质,加入过量的ATP会使pH显著下降,为了维持pH的一致性需要加入更多的NaOH进行调节,从而使离子浓度提高,限制了反应。
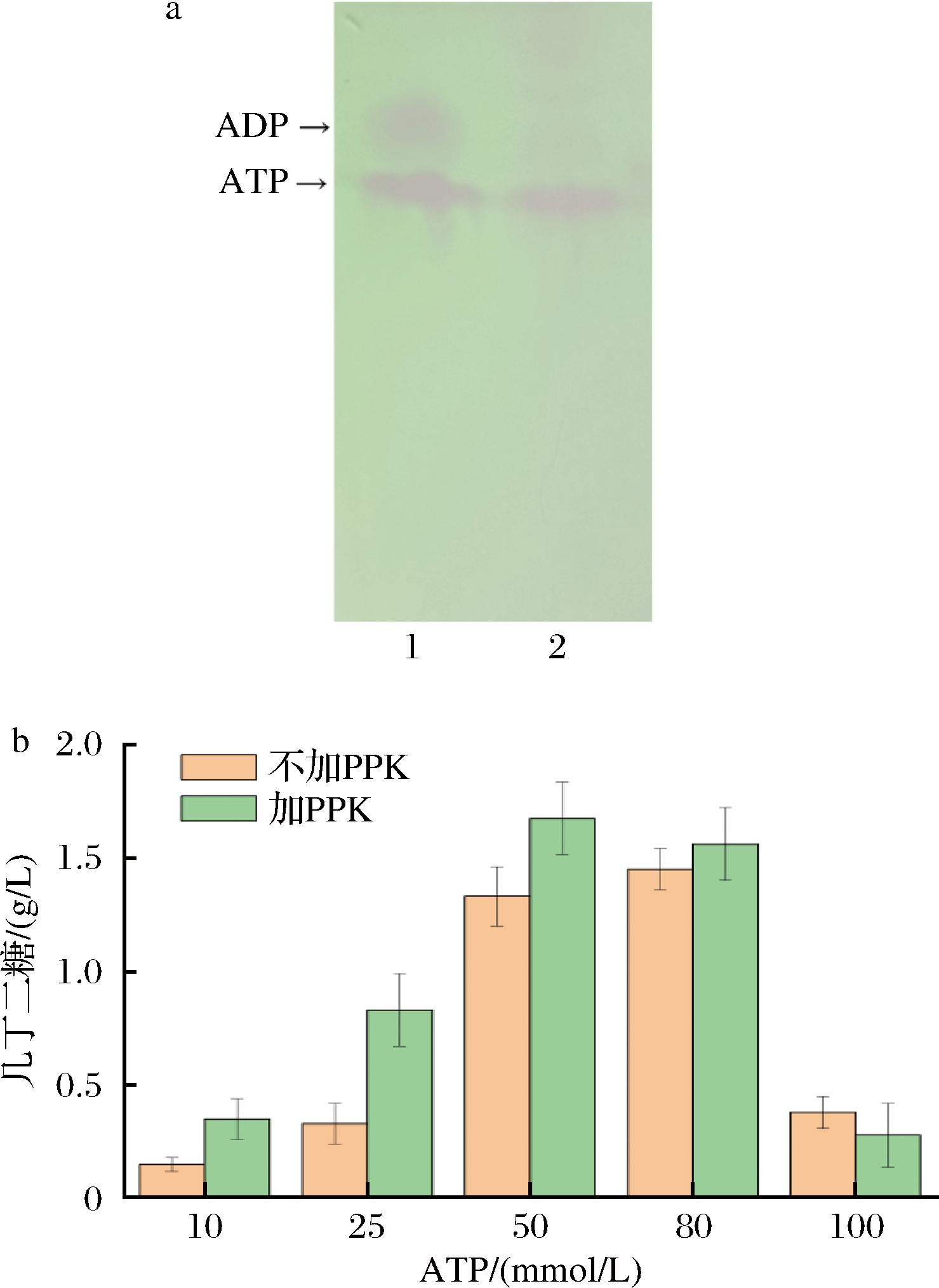
a-验证ATP的合成的TLC图(1-ADP与ATP的混合标品,2-PPK催化反应液);b-不同ATP条件下加入PPK对几丁二糖合成的影响
图5 验证PPK酶的活性
Fig.5 Verify the activity of PPK
2.5 优化三菌耦合合成(GlcNAc)2体系
多酶耦合合成目的产物时酶的适配性极为关键,此外,辅因子、底物浓度和反应时间也对产物的生成有着重要的影响。在确定了三菌耦合体系的可行性后,从十八胺聚氧乙烯醚反应、Mg2+浓度、反应时间、pH浓度4个维度对三菌耦合实现N-乙酰氨基葡萄糖和岩藻糖到三糖的转化进行单因素催化条件的优化,结果见图6所示。得出合成(GlcNAc)2的最佳反应条件为37 ℃下反应3 h,20 mmol/L MgCl2、10 g/L十八胺聚氧乙烯醚,pH为7.5的条件下三糖的产量达到最高,经过上述转化体系的优化,几丁二糖的产量由1 675 mg/L提升到5 214 mg/L。
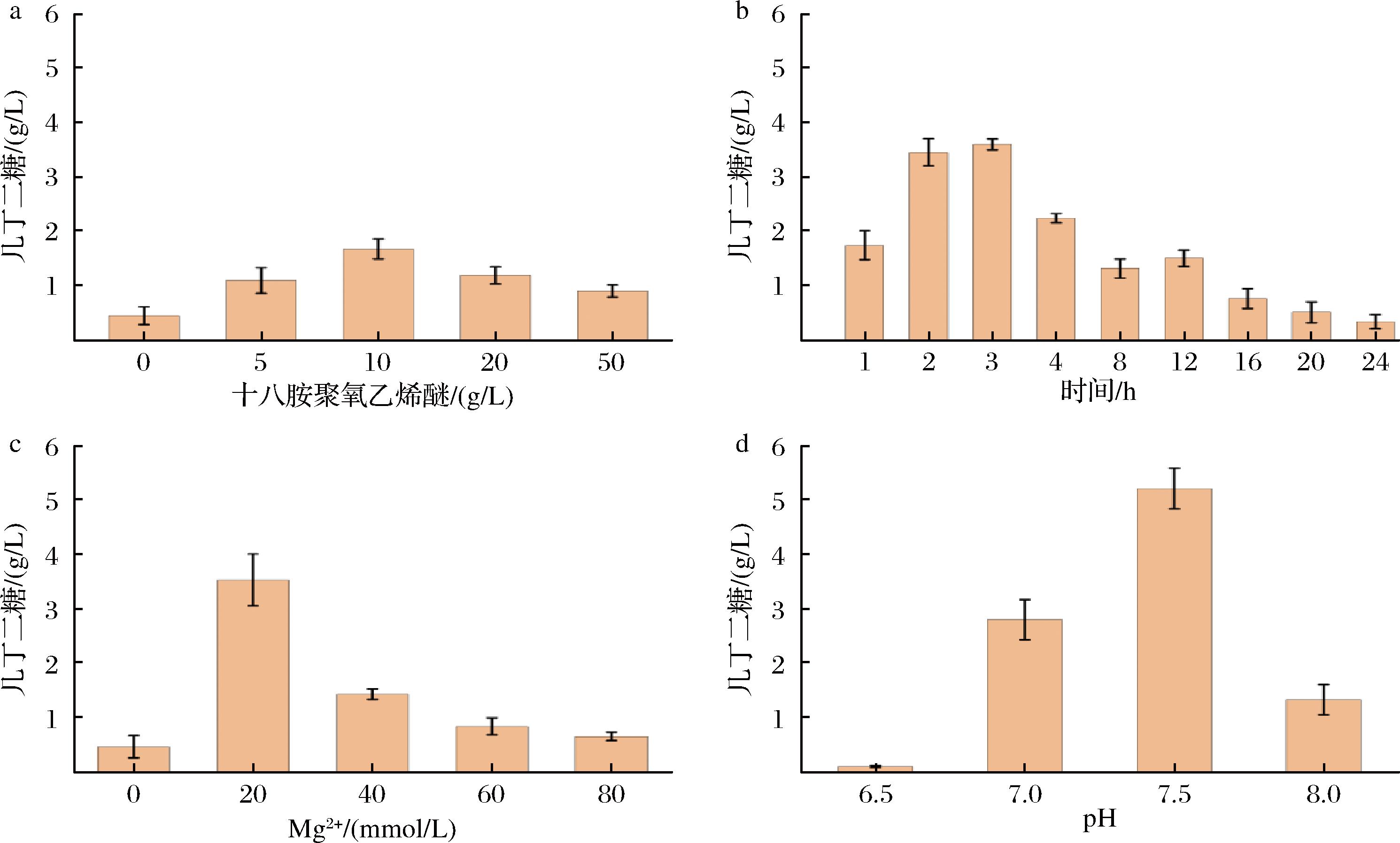
a-十八胺聚氧乙烯醚浓度对三菌耦合合成(GlcNAc)2的影响;b-反应时间对三菌耦合合成(GlcNAc)2的影响;c-Mg2+浓度对三菌耦合合成(GlcNAc)2的影响;d-pH对三菌耦合合成(GlcNAc)2的影响
图6 三菌耦合体系的优化
Fig.6 Optimization of the three-bacteria coupling system
2.6 双质粒菌株的筛选
为了防止中间产物的积累和最大程度生成几丁二糖,构建了几丁二糖生产菌株并通过不同拷贝数的质粒组合平衡途径基因的表达。本文采用pETDuet1(中拷贝数)和pRSFDuet1(高拷贝数)两种有两个表达框的质粒,采用双质粒共表达策略共表达3个基因来构建工程菌,通过双质粒的组合优化或通过柔性linker将两个基因转入到一个表达框的方式分别构建了6株工程菌A1~A8,其构建结构如图7-a所示。如图7,使用SDS-PAGE确定4种酶在一个大肠杆菌内的成功表达后投入反应,通过液相和TLC结果筛选出最佳组合为E.coli A2。
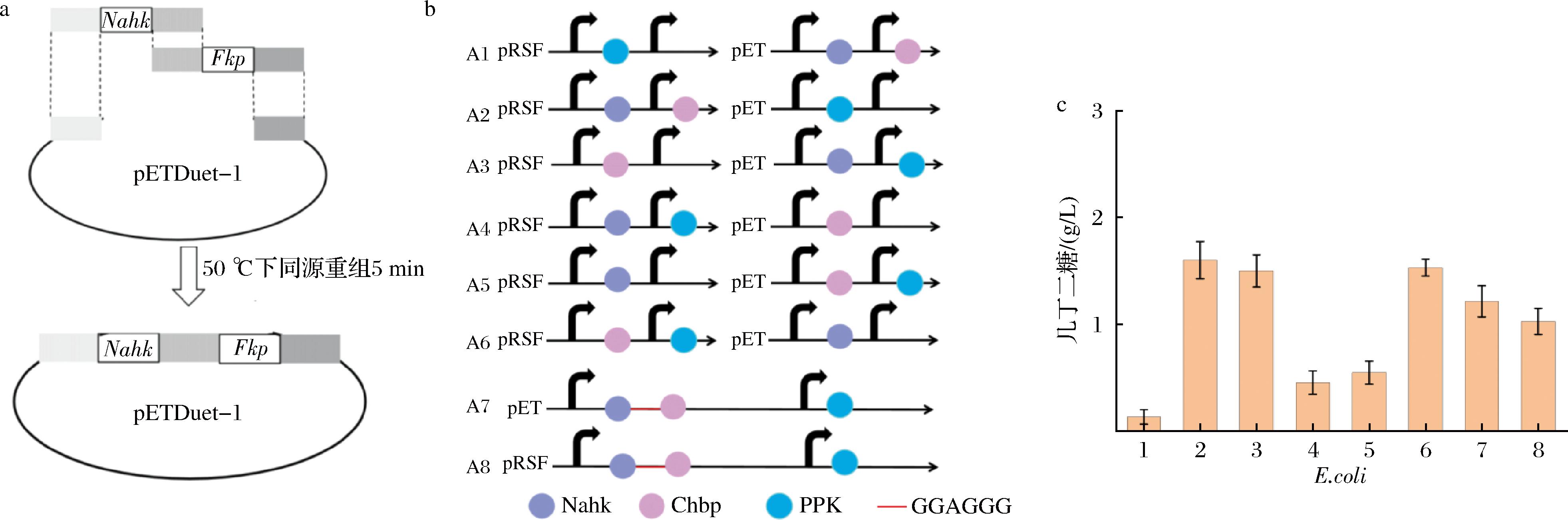
a-质粒pETDuet-Nahk-Fkp构建流程图;b-多蛋白表达菌株A1~A8的构建;c-菌株A1~A8全细胞合成几丁二糖的产量
图7 重组菌株的构建与筛选
Fig.7 Construction and screening of recombinant strains
但最优组合的产量仍比单基因表达菌株的产量低,可能是因为表达蛋白过多,菌株超载,难以负担这么多蛋白的表达。在活化双质粒菌株时就发现,双质粒菌株的生长速度比单质粒菌株的速度要慢,相同2%接种量单表达菌株基本上能在2~3 h内OD600就可以达到0.6左右,但是双表达菌株基本上要3~4 h才能达到相同OD值。所以从时间和原料成本而言最后还是选择三菌耦合体系生产几丁二糖。
3 结论与讨论
以往获取几丁二糖的方法主要是从壳聚糖内提取,目前研究(GlcNAc)2的最高产量为CHAROENPOL等[19]在鱿鱼笔甲壳素(shrimp shells chitin)中提取的,每100 mg鱿鱼笔能提取61.1 mg,但因为是通过酸解的方式得到,产物复杂纯化成本高,且对环境有污染,存在一定缺陷。JUNG等[20]为了解决酸解法对环境的污染问题,提出过酶解法,直接从蟹壳中利用几丁质酶分解得到几丁质,但产物复杂,从1-6单体的几丁质同时存在,从而很难得到单独的几丁二糖。SONG等[21]在JUNG研究员的基础上提出来固定化几丁质酶从而达到可持续利用的酶,尽管对底物的分解效率提高了,但并未解决底物复杂的问题,几丁二糖的分离困难。在本研究尽管产量相对较低,但通过N-乙酰氨基葡萄糖为底物进行生产,能够精确地将底物合成目标产物几丁二糖而不会产生过多的副产物,纯化工艺简单成本低,而且利用全细胞催化不需要对蛋白质进行纯化,更适应大规模工厂化生产。细胞内完整的多酶体系可以实现酶的级联反应,从而弥补酶法催化中级联催化过程不易实现的不足,提高了催化效率,同时,又省去了繁琐的酶纯化过程,制备更加简单,生产成本更低。
但是,目前技术也存在以下两个问题:(1)细胞内合成的(GlcNAc)2不能高效的分泌到细胞外;(2)未对(GlcNAc)2进行纯化分离,后续可以从通道蛋白入手,实现胞内(GlcNAc)2的外排,从而方便获得(GlcNAc)2,从而实现几丁二糖的大规模生产化,为进一步降低几丁二糖生产成本奠定了基础。
[1] WALTER A, FRIZ S, MAYER C.Chitin, chitin oligosaccharide, and chitin disaccharide metabolism of Escherichia coli revisited:Reassignment of the roles of ChiA, ChbR, ChbF, and ChbG[J].Microbial Physiology, 2021, 31(2):178-194.
[2] MUKHERJEE S, BEHERA P K, MADHUPRAKASH J.Efficient conversion of crystalline chitin to N-acetylglucosamine and N, N′-diacetylchitobiose by the enzyme cocktail produced by Paenibacillus sp.LS1[J].Carbohydrate Polymers, 2020, 250:116889.
[3] JI X G, ZHU L L, CHANG K L, et al.Chitooligosaccahrides:Digestion characterization and effect of the degree of polymerization on gut microorganisms to manage the metabolome functional diversity in vitro[J].Carbohydrate Polymers, 2022, 275:118716.
[4] QIN X, XIN Y Z, SU X Y, et al.Heterologous expression and characterization of thermostable chitinase and β-N-acetylhexosaminidase from Caldicellulosiruptor acetigenus and their synergistic action on the bioconversion of chitin into N-acetyl-d[J].International Journal of Biological Macromolecules, 2021, 192:250-257.
[5] LIAQAT F, ELTEM R.Chitooligosaccharides and their biological activities:A comprehensive review[J].Carbohydrate Polymers, 2018, 184:243-259.
[6] JIA L G, QI W, WANG K N, et al.Efficient bioconversion of chitinous waste to N-acetylchitobiose and N-acetylglucosamine using a novel salt-tolerant chitinase from Bacillus clausii[J].ACS Sustainable Chemistry &Engineering, 2023, 11(31):11470-11481.
[7] ZHANG J, FENG M, LU X M, et al.Base-free preparation of low molecular weight chitin from crab shell[J].Carbohydrate Polymers, 2018, 190:148-155.
[8] HONG S, YANG Q R, YUAN Y, et al.Sustainable co-solvent induced one step extraction of low molecular weight chitin with high purity from raw lobster shell[J].Carbohydrate Polymers, 2019, 205:236-243.
[9] ARNOLD N D, BRÜCK W M, GARBE D, et al.Enzymatic modification of native chitin and conversion to specialty chemical products[J].Marine Drugs, 2020, 18(2):93.
[10] BEIER S, BERTILSSON S.Bacterial chitin degradation-mechanisms and ecophysiological strategies[J].Frontiers in Microbiology, 2013, 4:149.
[11] JIAN X, LI C, FENG X D.Strategies for modulating transglycosylation activity, substrate specificity, and product polymerization degree of engineered transglycosylases[J].Critical Reviews in Biotechnology, 2023, 43(8):1284-1298.
[12] HULLINGER A C, GREEN V E, KLANCHER C A, et al.Two transmembrane transcriptional regulators coordinate to activate chitin-induced natural transformation in Vibrio cholerae[J].bioRxiv, 2025:2024.09.30.615920.
[13] ZHOU X L, HUANG Y, LIU Y Y, et al.Efficient production of N-acetyl-β-D-glucosamine from shrimp shell powder using chitinolytic enzyme cocktail with β-N-acetylglucosaminidase from domesticated microbiome metagenomes[J].Fermentation, 2024, 10(12):652.
[14] ITOH T, YAGUCHI M, NAKAICHI A, et al.Structural characterization of two solute-binding proteins for N, N′-diacetylchitobiose/N, N′, N″-triacetylchitotoriose of the gram-positive bacterium, Paenibacillus sp.str.FPU-7[J].Journal of Structural Biology:X, 2021, 5:100049.
[15] PARK J K, KEYHANI N O, ROSEMAN S.Chitin catabolism in the marine bacterium Vibrio furnissii[J].Journal of Biological Chemistry, 2000, 275(42):33077-33083.
[16] ITOH T, KIMOTO H.Bacterial chitinase system as a model of chitin biodegradation[M].Targeting Chitin-containing Organisms.Singapore:Springer Singapore, 2019:131-151.
[17] HONDA Y, KITAOKA M, HAYASHI K.Reaction mechanism of chitobiose phosphorylase from Vibrio proteolyticus:Identification of family 36 glycosyltransferase in Vibrio[J].The Biochemical Journal, 2004, 377(Pt 1):225-232.
[18] GUAN J, JAKOB U.The protein scaffolding functions of polyphosphate[J].Journal of Molecular Biology, 2024, 436(14):168504.
[19] CHAROENPOL A, CRESPY D, SCHULTE A, et al.Immobilized chitinase as effective biocatalytic platform for producing bioactive di-N-acetyl chitobiose from recycled chitin food waste[J].Bioresource Technology, 2024, 406:130945.
[20] JUNG W J, PARK R D.Bioproduction of chitooligosaccharides:Present and perspectives[J].Marine Drugs, 2014, 12(11):5328-5356.
[21] SONG J, TAKE L, HYUN L, et al.Highly efficient production of N, N′-diacetylchitobiose according to substrate modification and changes in enzyme kinetics[J].Process Biochemistry, 2023, 133:179-189.