褐藻是一种产量高、分布广的多功能海藻,是生产生物燃料的重要原料之一。褐藻胶,又称海藻酸盐,是海洋褐藻中含量最丰富的多糖之一。它是由α-L-古罗糖醛酸和β-D-甘露糖醛酸以交替排列的方式聚合而成,两者通过1,4-糖苷键连接。根据2种单糖在褐藻胶内含量的不同可将其分为3种类型:聚甘露糖醛酸(polyM)、聚古罗糖醛酸(polyG)和混合型(polyMG)褐藻胶[1]。嵌段类型和分子质量大小影响海藻酸盐的成胶能力和黏度。单体单元的排列方式也是影响海藻酸盐性能的重要因素[2]。褐藻胶因其天然环保,安全易降解,原材料丰富等特性使其在食品、医药、环境保护等领域具有显著优势,被广泛应用。在食品工业中,褐藻胶具有多方面的功能,它可以用于生产食品水凝胶(如离子凝胶和酸性凝胶)以封装活性物质、缓释营养成分[3],并在低脂乳制品和乳化食品中充当增稠剂和稳定剂[4],在生物燃料开发和生物化学用途等方面也备受关注[5]。但同时褐藻胶显著的高分子特性也限制了其在各个领域的应用潜力。褐藻胶的分子质量大、溶解度低、分子结构复杂,在被作为药物载体应用时,人体不能将其有效分解和吸收,进而影响药物释放。因此,针对这一问题,科研人员采用褐藻胶裂解酶生物解聚策略,将大分子多糖定向酶促分解为具有应用价值的低分子量褐藻寡糖(alginate oligosaccharides,AOS)。为了进一步拓宽褐藻资源的应用途径,开发出更具产业化价值的AOS产品,需要不断发掘优良产酶菌株,开发新型褐藻胶裂解酶。
AOS是海藻酸盐的降解产物,与褐藻胶相比,其分子质量较小,水溶性较高,溶解度和溶液黏度也较低,具有比褐藻胶更好的生物活性,如抑制肿瘤[6]、血压调控[7]、抗氧化[8]、抑菌[9]、保护神经系统[10]等。因此,AOS被广泛地应用于农业,动物营养,食品及药物加工等行业。根据组成AOS的糖醛酸类型的不同又可将其分为3种类型:古罗糖醛酸寡糖(guluronic acid oligosaccharides,GAOS)、甘露糖醛酸寡糖(mannuronic acid oligosaccharides,MAOS)和杂合糖醛酸寡糖(heterozygous acid oligosaccharides,HAOS)[11]。GAOS和MAOS都是由单一糖醛酸组成的寡糖,不同糖醛酸组成的AOS表现出来的活性有所差异[12]。除此之外,不同聚合度(degree of polymerization,DP)的寡糖所具有的活性和功效也不一样,如DP为3~6的AOS可以调控细胞因子的合成和分泌[13],AOS的抗氧化活性也与DP值的大小相关。AOS的制备主要依赖于解聚技术,化学降解法、物理降解法和生物降解法是目前制备AOS的3种主要的方式。化学法和物理降解法是传统的制备方法,它们的成本较高,环保性也较差,且生成的产物具有随机性,难以控制其特定的DP。相比之下,催化效率高,底物专一性强且更为绿色安全环保的生物降解法成为主要的AOS制备方法。
褐藻胶裂解酶可以通过β-消除反应从非还原端C4和C5之间的1,4-糖苷键来降解褐藻胶,从而产生不饱和AOS作为主要产物[14]。目前,褐藻胶裂解酶已从多种生物中分离出来,包括藻类、海洋无脊椎动物以及海洋和陆地微生物[15]。在CAZy数据库(http://www.cazy.org/)中已将目前发现的藻酸裂解酶归类为PL5、6、7、14、15、17、18、32、34、36和39家族[16]。褐藻胶的完全糖化需要内切酶和外切酶的共同作用[17],一些利用海藻酸盐裂解酶协同作用高效降解海藻酸盐的研究被广泛关注[18]。而同时具有内切和外切2种活力的双功能褐藻胶裂解酶在水解褐藻胶制备高附加值AOS产品上更具优势。不同的褐藻胶裂解酶具有不同的底物特异性,主要分3类:polyG特异性裂解酶、polyM特异性裂解酶以及可以同时降解2种片段的双功能裂解酶[19]。迄今为止研究的大多数裂解酶都为polyM特异性裂解酶。GAOS在抗凝血、免疫调节等方面的活性优于MAOS[20]。因此,系统挖掘与功能解析褐藻胶裂解酶资源,筛选具有古罗糖醛酸底物偏好性的新型裂解酶,对褐藻胶资源的进一步利用及精准制备GAOS具有重要科学价值与应用潜力。本研究从一株褐藻胶降解菌Alteromonas sp.A1-6中预测并克隆表达了褐藻胶降解基因alg1264,对重组酶的酶学性质进行了较为系统的研究,发现该酶对polyG具有偏好性,为更好地开发褐藻胶资源提供理论依据也为AOS的制备提供了一种新的酶。
1 材料与方法
1.1 材料
1.1.1 菌株
褐藻胶降解菌Alteromonas sp.A1-6最初以浒苔多糖为唯一碳源分离得到[21],也能以褐藻胶为唯一碳源生长,培养于含4 g/L海藻酸钠的人工海水中。Escherichia coli DH5a,Escherichia coli BL21(DE3)为本实验室保存。
1.1.2 试剂及培养基
褐藻胶,国药集团有限公司;polyM、polyG、褐藻单糖、二糖、三糖,青岛博智汇力有限公司;DNA Marker,Takara公司;Prestained Protein Ladder,Thermo Scientific公司;One Step Seamless Cloning kit,北京艾德莱生物科技有限公司;2×Pro Taq Master Mix、质粒提取试剂盒、琼脂糖凝胶回收试剂盒,江苏康为世纪科技股份有限公司;Ni NTA Beads 6FF,常州天地人和生物科技有限公司;PCR引物,生工生物工程(上海)股份有限公司合成;其他培养基组分等相关试剂均为分析纯产品。
褐藻胶培养基:0.4 g褐藻胶,1×人工海水[22]定容至100 mL。
1.2 仪器与设备
T-GRADIENT PCR仪,德国Biometra公司;BS224S分析天平,北京赛多利斯仪器系统有限公司;SW-CJ-IBM超净工作台,苏州安泰空气有限公司;LDZM-80L-Ⅲ高压自动灭菌锅,上海申安医疗器械厂;荧光定量PCR仪,美国伯乐公司;DYY-6C琼脂糖凝胶电泳系统,北京六一仪器公司。
1.3 实验方法
1.3.1 荧光定量PCR检测基因alg1264表达水平
将Alteromonas sp. A1-6的单菌落接种于10 mL褐藻胶培养基中,28 ℃、200 r/min振荡培养。利用Trizol溶液进行RNA提取,电泳检测RNA提取成功并去除基因组的DNA后进行逆转录反应。1)DNA去除反应体系及反应条件:5×g DNA Eraser缓冲液2 μL,gDNA Eraser 1 μL,RNA 6 μL,RNase Free ddH2O 1 μL,42 ℃反应2 min;2)逆转录反应体系及反应条件:5×PrimeScript Buffer(实时荧光定量PCR) 4 μL,PrimeScriptRT Enzyme MixI 1 μL,Random primer 1 μL,上述去除DNA的RNA提取液8 μL,RNase Free ddH2O 6 μL,37 ℃反应15 min后于85 ℃反应10 s使逆转录酶失活。设计荧光定量PCR的引物和内参16S rRNA基因的引物(如表1所示),以逆转录cDNA为模板进行荧光定量PCR,反应体系:TOROGreen®qPCR Master Mix 12.5 μL,引物F 1 μL,引物R 1 μL,cDNA模板1 μL,无菌水9.5 μL。反应条件:95 ℃ 3 min;95 ℃ 15 s,56 ℃ 20 s,72 ℃ 30 s,循环40次;65 ℃ 5 s,95 ℃ 2 min。根据内参基因进行归一化处理和2-△△CT法[23]计算相对表达量,用琼脂糖凝胶电泳对PCR产物进行分析是否有非特异性扩增。
表1 荧光定量PCR的引物序列
Table 1 Primer sequences used for fluorescence-based quantitative polymerase chain reaction

引物名称引物序列(5′-3′)退火温度/℃Orf1264-F(荧光定量 PCR)tggtgcccatagttccttacg Orf1264-R(荧光定量 PCR)tcaacgcaataccgtcttca 56Eub341FcctacgggaggcaggcagEub534Rattaccgcggctgctggc561264-Faggagatataccatgaagggagaagagcaaacatcc1264-Rtgcggccgcaagcttatgctcgtatttaaggtgatagaaa56pET28a-FcatggtatatctccttcttaaagttaaacaaaattatpET28a-Raagcttgcggccgcactc62
1.3.2 褐藻胶裂解酶Alg1264的生物信息学分析
使用SignalP 6.0预测Alg1264的N端信号肽,利用ExPAsy的在线工具ProtParam(https://web.expasy.org/protparam/)对蛋白质的理论等电点、分子质量进行预测,使用软件MEGA11中的邻接法(neighbour-joining,NJ)构建褐藻胶裂解酶Alg1264的系统发育树,通过NCBI Conserved Domain(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)分析褐藻胶裂解酶Alg1264的结构域,使用DNAMan软件将Alg1264与PL7家族中已经充分解析的3种褐藻胶裂解酶进行多序列分析,这3种酶分别是来自Microbulbifer sp.6532A的AlgMsp(BAJ62034)、Saccharophagus degradans 2-40的Alg7D(ABD81807)、Agarivorans sp.L11的AlyL2(AJO61885)。
1.3.3 褐藻胶裂解酶基因alg1264的克隆
根据褐藻胶裂解酶Alg1264的基因序列利用Primer 5.0设计上游引物1264-F和下游引物1264-R,挑取褐藻胶降解菌A1-6的单菌落悬浮于50 μL无菌水中,沸水浴10 min后置于冰上快速冷却,12 000 r/min离心1 min,取上清为模板进行PCR扩增。PCR的扩增体系(总体积50 μL):5×PCR缓冲液10 μL,基因组 DNA 4 μL,引物-orf1264-F 1 μL,引物-orf1264-R 1 μL,10×mmol/L dNTPs 4 μL,ddH2O 29.5 μL,fast pfu酶 0.5 μL。PCR扩增条件:95 ℃预变性 5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,循环30次;72 ℃终延伸8 min。pET28a质粒的提取和载体的线性化,以pET28a质粒为模板,利用引物pET28a-F和pET28a-R将载体线性化。PCR的扩增体系(总体积50 μL):基因组DNA 1 μL,引物-F 1 μL,引物-R 1 μL,fast pfu酶 0.5 μL,5×PCR Buffer 10 μL,10×mmol/L dNTPs 4 μL,ddH2O 32.5 μL。PCR扩增条件:95 ℃预变性5 min;95 ℃变性30 s,62 ℃退火30 s,72 ℃延伸3 min,循环30次;72 ℃终延伸8 min。将PCR扩增产物以及载体线性化产物进行凝胶电泳检测,确定条带大小正确后进行胶回收并于-20 ℃进行保存。
1.3.4 重组表达载体的构建
利用One Step Seamless Cloning kit将目的基因扩增产物与线性化载体进行连接。酶联反应体系(总体积10 μL):目的基因4 μL,线性载体1 μL,2×One Step Cloning Mix 5 μL。在50 ℃中酶联30 min后利用将重组质粒转入大肠杆菌DH5α感受态细胞中,复苏后将其涂布于LB固体培养基(含有50 μg/mL卡那霉素),37 ℃过夜培养。挑选阳性克隆子进行菌落PCR验证,最后经过测序筛选出正确的阳性克隆子。生工生物技术公司(上海)对克隆目的基因进行序列测定,最后将构建成功的重组质粒pET28a-alg1264转入大肠杆菌BL21(DE3)表达。
1.3.5 重组酶Alg1264的诱导表达及纯化
挑取验证成功的重组菌单菌落接种于5 mL LB培养基中,于37 ℃、200 r/min过夜培养,吸取5 mL菌液转接于500 mL LB培养基(含有50 μg/mL卡那霉素)中,37 ℃、200 r/min培养至OD600为0.4~0.6。加入诱导剂异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)使其终浓度为0.1 mmol/L,16 ℃低温诱导16 h后离心收集菌体沉淀,将菌体清洗2遍后用适量缓冲液(50 mmol/L PBS,pH 7.4)将菌体吹打混匀,超声波破碎(400 W,2 s/3 s间歇循环模式,30 min)后离心收集上清即为粗酶液。通过Ni-NTA亲和层析柱法对粗酶液中的重组酶进行纯化,并用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamidegel electrophoresis,SDS-PAGE)对重组酶的大小和纯化情况进行检测。蛋白质含量采用Bradford方法[24]测定,以牛血清白蛋白为标准。
1.3.6 酶活力及酶动力学参数的测定
采用DNS法[25]测定生成的还原糖的含量。底物为200 μL 4 mg/mL的海藻酸钠溶液(溶解在pH 7.0,50 mmol/L Na2HPO4-NaH2PO4缓冲液中),加入1 μL酶液,在30 ℃水浴中反应5 min。反应后用DNS试剂终止,100 ℃显色5 min,再迅速置于冰上冷却至室温。1 000 r/min离心1 min后取上清测定其OD540。对照组加入的为灭活的粗酶液,在酶活力测定实验过程中对照组与实验组均设置3个重复。1个酶活力单位定义为在30 ℃下每分钟降解褐藻胶生成1 μmol还原糖所需的酶量。
配制不同质量浓度的褐藻胶溶液(0.5、1、1.5、2、2.5、3、3.5、4、5 mg/mL),按照上述酶活力测定方法,测定重组酶Alg1264在不同底物浓度下的酶活力。以底物浓度为横坐标,酶反应速率为纵坐标,使用Origin 2021对底物浓度与反应速率间的关系进行拟合,计算出重组酶Alg1264的最大反应速率(Vmax)、米氏常数(Km)值,催化常数(kcat)值及kcat/Km值。
1.3.7 酶学性质的分析
1.3.7.1 最适pH的测定
使用不同pH的缓冲液(50 mmol/L 醋酸-醋酸钠缓冲液,pH 4.0~6.0;50 mmol/L Na2HPO4-NaH2PO4缓冲液,pH 6.0~8.0;50 mmol/L Tris-HCl缓冲液,pH 8.0~9.0;50 mmol/L甘氨酸-NaOH缓冲液,9.0~10.0)配制不同pH浓度的海藻酸钠底物溶液,依据上面描述的DNS酶活力测定方法测定出褐藻胶裂解酶Alg1264在不同pH下的酶活力,设定测得的最高酶活力为100%,通过比较不同pH下重组酶的相对酶活力,确定该酶的最适pH。
pH稳定性的测定。测定酶液在不同pH的缓冲液(50 mmol/L醋酸-醋酸钠缓冲液,pH 4.0~6.0;50 mmol/L Na2HPO4-NaH2PO4缓冲液,pH 6.0~8.0;50 mmol/L Tris-HCl缓冲液,pH 8.0~9.0;50 mmol/L甘氨酸-NaOH缓冲液,9.0~10.0)于4 ℃下孵育1 h后残留的酶活力,测定未被处理的酶液活力即为100%,计算用不同pH缓冲液处理过的重组酶的相对酶活力,测定重组酶的pH稳定性。
1.3.7.2 最适温度的测定
分别测定重组酶Alg1264在0、10、20、30、40、50、60、70、90 ℃时的酶活力,底物为50 mmol/L Na2HPO4-NaH2PO4缓冲液(pH 7.0)配制的海藻酸钠溶液,设定测得的最高酶活力为100%,计算不同温度下重组酶的相对酶活力,确定该酶的最适温度。
温度稳定性的测定。分别测定酶液在5、10、15、20、25、30、35、40、45,50 ℃下保温30 min后残留的酶活力,测定的未被处理的酶液活力即为100%,计算在不同温度下孵育30 min后重组酶的相对酶活力,测定重组酶的温度稳定性。
1.3.7.3 金属离子对酶活力的影响
在上述酶活力测定反应体系中加入金属离子,设置金属离子终浓度分别为1 mmol/L和5 mmol/L的2组实验组,在最适反应条件下反应5 min后测定酶活力,对照组加入等体积的50 mmol/L Na2HPO4-NaH2PO4缓冲液(pH 7.0),测定的对照组的酶活力即为100%,以此计算加入不同浓度离子后重组酶的相对酶活力,分析金属离子对重组酶Alg1264酶活力的影响。
1.3.7.4 有机试剂对酶活力的影响
在上述酶活力测定反应体系中分别加入低浓度[1 mmol/L或5%(体积分数),其中甲醇、乙醇、异丙醇、二甲基亚砜(dimethyl sulfoxide, DMSO)体积分数为5%,其他试剂浓度为1 mmol/L]和高浓度[5 mmol/L 或20%(体积分数),其中甲醇、乙醇、异丙醇、DMSO体积分数为20%,其他试剂浓度为5 mmol/L]的有机试剂,在最适反应条件下反应5 min后测定酶活力。对照组加入等体积的50 mmol/L Na2HPO4-NaH2PO4缓冲液(pH 7.0),设定对照组的酶活力为100%,计算加入不同浓度有机溶剂后重组酶的比活力和相对酶活力,分析有机试剂对重组酶Alg1264酶活力的影响。
1.3.7.5 底物特异性分析
分别配制4 mg/mL的海藻酸钠、polyG、polyM溶液,加入酶液后将反应体系置于最适反应条件下反应5 min,测定不同底物下重组酶活力,并设定在海藻酸钠为底物时测定的酶活力为100%,分析重组酶对不同底物的偏好性。
1.3.8 褐藻胶裂解酶水解产物的分析
将5 μL重组酶液分别加入200 μL最适pH缓冲液配制的4 mg/mL褐藻胶、polyM和polyG 溶液中,在最适温度下进行反应。设置2组不同的反应时间(30 min和过夜),以进行对比实验。反应完成后,将样品100 ℃水浴加热5 min,随后通过离心去除其中的蛋白质。本实验采用液质联用的方法对重组酶降解不同底物产生的水解产物的组分进行分析。LC-MS分析采用安捷伦LC-MS系统,配备Poroshell 120 EC-C18色谱柱(2.1 mm×150 mm,2.7 μm)。流动相为:流动相A[0.1%(体积分数)甲酸水溶液]和流动相B(乙腈)。梯度洗脱程序如下:流速0.3 mL/min;0~2 min,95% A;2~5 min,95%~50% A;5~12 min,50%~5% A;12~15 min,5%~95% A。柱温40 ℃,进样量3 μL。质谱分析使用SCIEX QTRAP 6500+质谱仪,采用电喷雾负离子模式,质谱参数如下:毛细管电压3.0 kV,入口电压10 V,去聚簇电压100 V,离子源温度550 ℃,气帘气流速35 L/h,离子源气体1和气体2流速50 L/h,碰撞能量35 eV,质量范围(m/z)100~1 200,扫描速率2 000 Da/s。
1.4 数据处理
所有实验数据为3次以上重复实验取均值,采用Origin 2021、SPSS进行实验数据的处理与统计分析。仅2组比较直接用t-test(如独立样本t-test或配对t-test)。若方差不齐,改用Welch’s t-test。3组及以上比较优先用ANOVA,避免多次t-test导致Ⅰ类错误(假阳性)累积。ANOVA显著后,需通过事后检验(Tukey’s HSD)确定具体组间差异。
2 结果与分析
2.1 荧光定量PCR检测基因alg1264的表达水平
Alteromonas sp. A1-6原为浒苔多糖降解菌,对其基因组信息进行分析,预测alg1264为褐藻胶裂解酶基因,为验证该基因是否为褐藻胶降解相关基因,进行荧光定量PCR实验。由图1可知,以琥珀酸培养的基因表达水平为对照进行归一化处理,经褐藻胶诱导以后,基因alg1264的转录水平显著上升(P<0.05),故初步推断基因alg1264为褐藻胶降解相关基因。

图1 荧光定量PCR检测基因alg1264表达量
Fig.1 Analysis of alg1264 mRNA levels via fluorescence-based quantitative PCR
注:*表示差异性显著,P<0.05。
2.2 褐藻胶裂解酶Alg1264的生物信息学分析
基因alg1264的大小为981 bp,编码327个氨基酸,利用在线网站SignalP 6.0 (https://services.healthtech.dtu.dk/services/SignalP-6.0/)推测其N端有一个由23个氨基酸组成的信号肽,利用ExPAsy (https://www.expasy.org/)预测蛋白的分子质量为36 KDa,理论等电点(pI)为5.93。将Alg1264与CAZy 数据库中已注释过的褐藻胶裂解酶基因序列进行比对,发现Alg1264与来自Agarivorans sp.L11菌株的褐藻胶裂解酶AlyL2 (AJO61885)一致性最高(48.6%)。从系统发育树上来看,Alg1264与PL7家族的褐藻胶裂解酶属于同一个分支(图2-a)。NCBI Conserved Domain的结构域分析表明Alg1264存在结构域Algylase super family,与已经报道过的PL7家族褐藻胶裂解酶相符,因此,推断Alg1264属于PL7家族。与已知结构的藻酸盐裂解酶进行多序列比对发现,Alg1264包含3个保守区域“RTELREMLR”,“QIH”和“MYFKAG” (图2-b),这些区域与底物结合及催化活性密切相关。已有研究表明,“QIH”或“QVH”保守区在褐藻胶裂解酶的底物特异性中起关键作用,这2个区域可作为预测酶底物偏好的重要指标,当褐藻胶裂解酶具有“QIH”区域时,表现出对polyG结构单元的降解偏好。

a-Alg1264的系统进化树(Bootstrap 1 000);b-Alg1264的氨基酸序列分析
图2 Alg1264的氨基酸序列比对和系统进化分析
Fig.2 Multiple sequence alignment and phylogenetic analysis of Alg1264
注:图中相同的氨基酸序列用深蓝色表示。
2.3 褐藻胶裂解酶基因的克隆与表达
以Alteromonas sp. A1-6的基因组为模板进行PCR扩增。去除了信号肽基因序列的目的基因为912 bp,通过琼脂糖凝胶电泳图可知PCR产物条带大小正确。将目的基因与载体连接后构建的重组质粒pET28a-alg1264转入大肠杆菌DH5a感受态细胞中。挑选阳性克隆子并提取质粒,以重组质粒为模板进行PCR验证后送往测序,测序结果与目的基因的序列一致,表明重组质粒pET28a-alg1264构建成功。
2.4 重组酶Alg1264的诱导表达及纯化
在低温诱导下,加入IPTG后,重组菌BL21(pET28a-alg1264)成功表达出目的蛋白Alg1264。离心收集菌体破碎后再离心即得到粗酶液,粗酶液经过Ni-NTA亲和层析柱进行目的蛋白的纯化。纯化后的蛋白经过SDS-PAGE电泳检测发现目的蛋白在80 mmol/L咪唑下被洗脱,条带大小与预期目的蛋白分子质量相吻合(图3)。
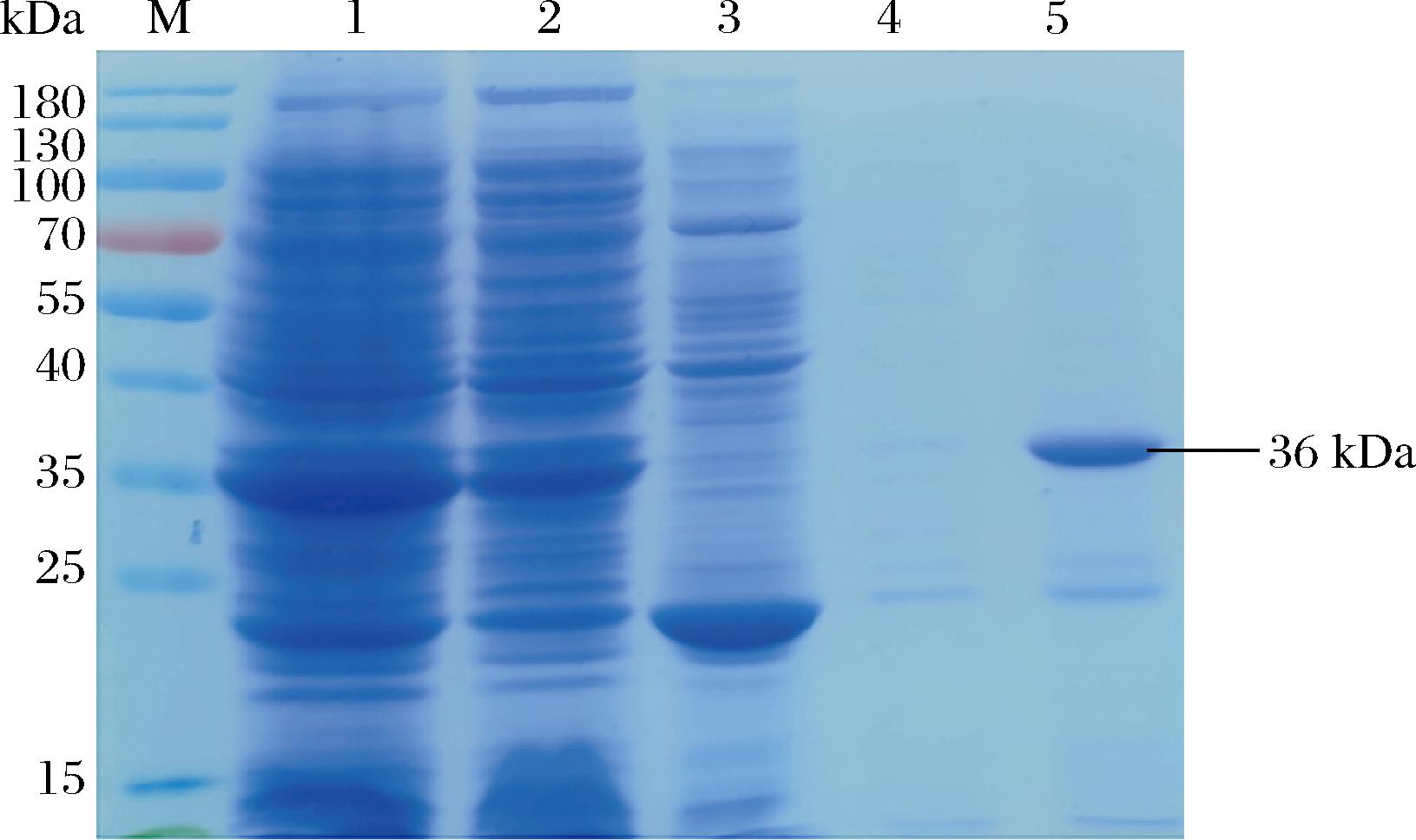
M:蛋白marker;1:粗酶液;2:流穿液;3:20 mmol/L咪唑洗脱蛋白;4:40 mmol/L咪唑洗脱蛋白;5:80 mmol/L咪唑洗脱蛋白。
图3 SDS-PAGE检测重组褐藻胶裂解酶Alg1264
Fig.3 Analysis of recombinant Alg1264 alginate lyase with SDS-PAGE
2.5 酶动力学参数的测定
以不同浓度的褐藻胶为底物,在最适反应条件下测定酶催化反应速度。以反应速率为纵坐标,以底物浓度为横坐标,利用Michaelis Menten模型进行拟合(如图4所示)。可得最大反应速率Vmax为0.130 μmol/min,米氏常数Km为4.034 mg/mL,kcat为52.653 s-1,kcat/Km为13.052 mL/(mg·s)。
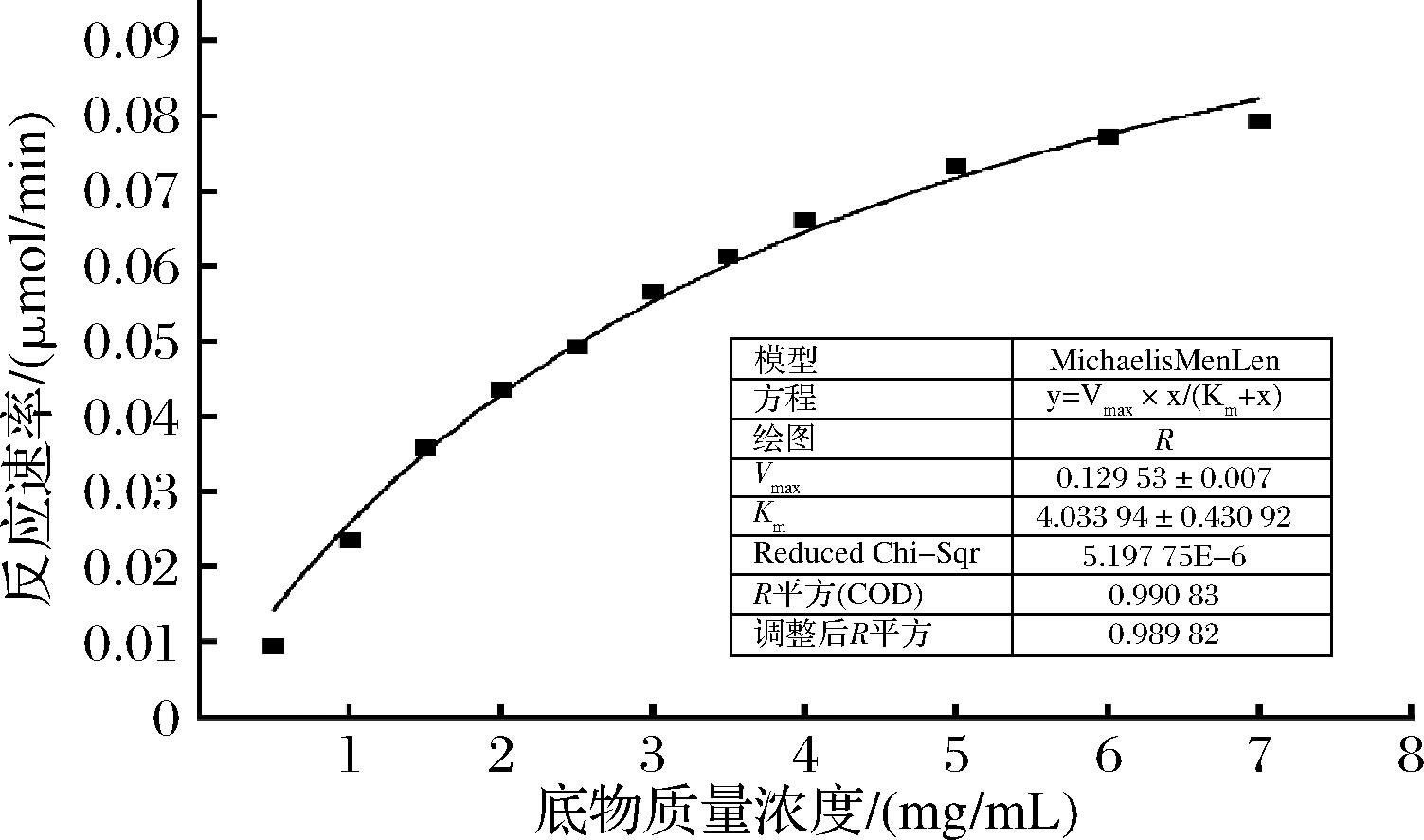
图4 重组褐藻胶裂解酶Alg1264的动力学常数
Fig.4 Determination of kinetic constants of the recombinant alginate lyase Alg1264
2.6 酶学性质的分析
2.6.1 最适pH和pH稳定性
测定重组酶Alg1264在不同pH下的酶活力,结果表明重组酶Alg1264的最适pH值为7.0(图5-a)。在不同pH下孵育1 h后测定重组酶残留的酶活力,结果表明重组酶在pH 5.0~11.0始终维持有77%以上的酶活力(图5-b),表明该酶的pH稳定性良好。
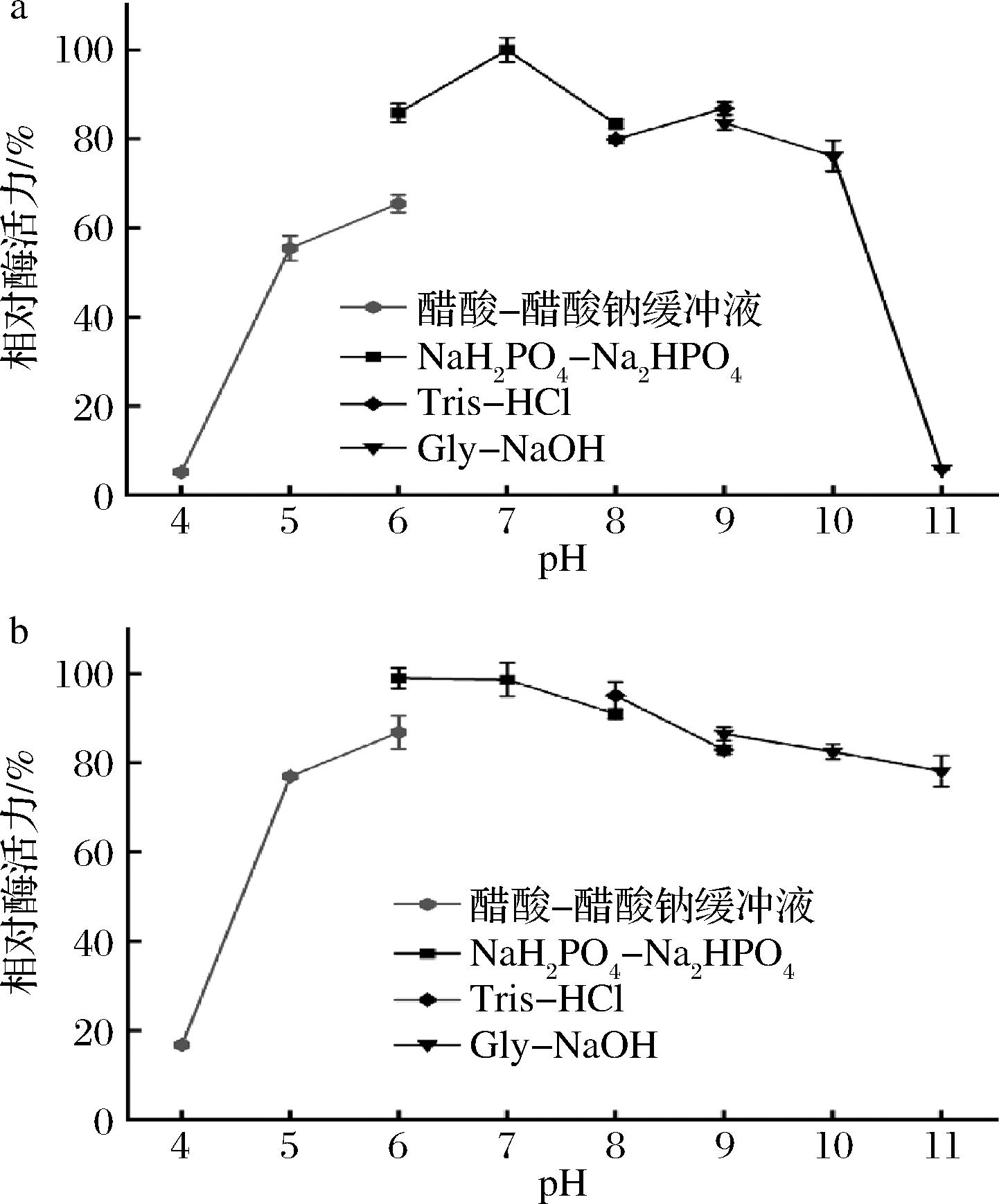
a-Alg1264酶在不同pH下相对酶活力;b-Alg1264酶在不同pH下孵育1 h后相对酶活力
图5 pH对重组酶Alg1264酶活力的影响
Fig.5 Effects of pH on the activity of Alg1264
2.6.2 最适温度和温度稳定性
测定重组酶Alg1264在不同温度下的酶活力,结果表明重组酶Alg1264的最适温度为35 ℃(图6-a)。在不同温度下孵育30 min后测定重组酶残留的酶活力,结果表明重组酶在5~35 ℃相对酶活力基本不受影响,始终能保持在86%以上,当温度提升到40 ℃时酶活力明显下降,在超过50 ℃后重组酶完全失活(图6-b)。
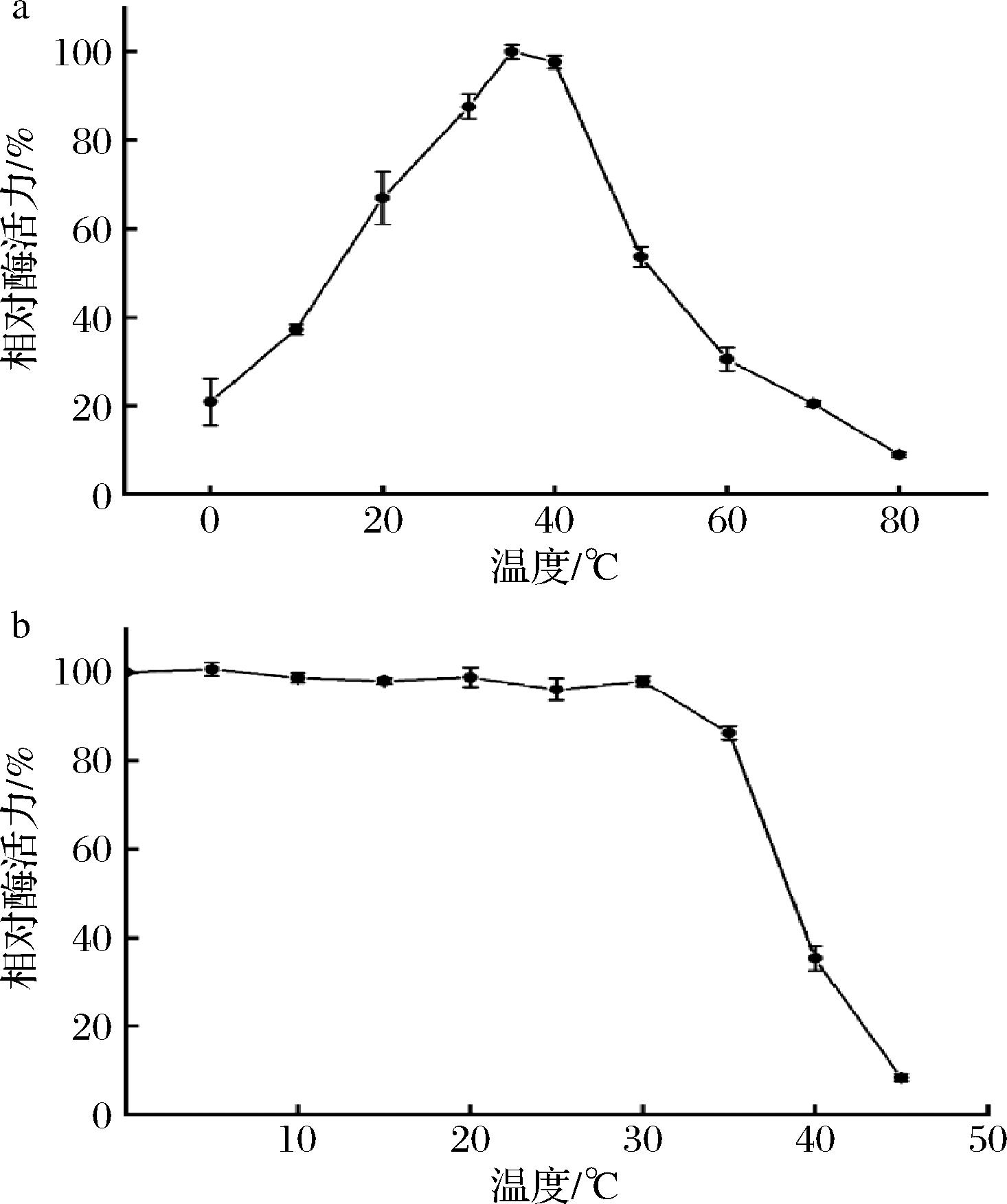
a-Alg1264酶在不同温度下相对酶活力;b-Alg1264酶在不同温度下孵育30 min后相对酶活力
图6 温度对重组酶Alg1264酶活力的影响
Fig.6 Effects of temperature on the activity of Alg1264
2.6.3 金属离子对酶活力的影响
加入不同的金属离子测定其对重组酶酶活力的影响。在1 mmol/L的低浓度条件下,Mn2+、Ca2+、Fe2+、Mg2+、Cu2+、Ni2+对重组酶活力基本无影响,相对酶活力维持在80%以上,Co2+、Zn2+和EDTA对重组酶的影响较小,相对酶活力维持在65%以上;在离子浓度上调为5 mmol/L时,在Mg2+、Cu2+、Ni2+影响下,相对酶活力依旧维持在86%左右,在Co2+、Fe2+影响下,相对酶活力维持在50%以上,而在Mn2+、Zn2+和EDTA的作用下重组酶的相对酶活力都低于40%(图7)。
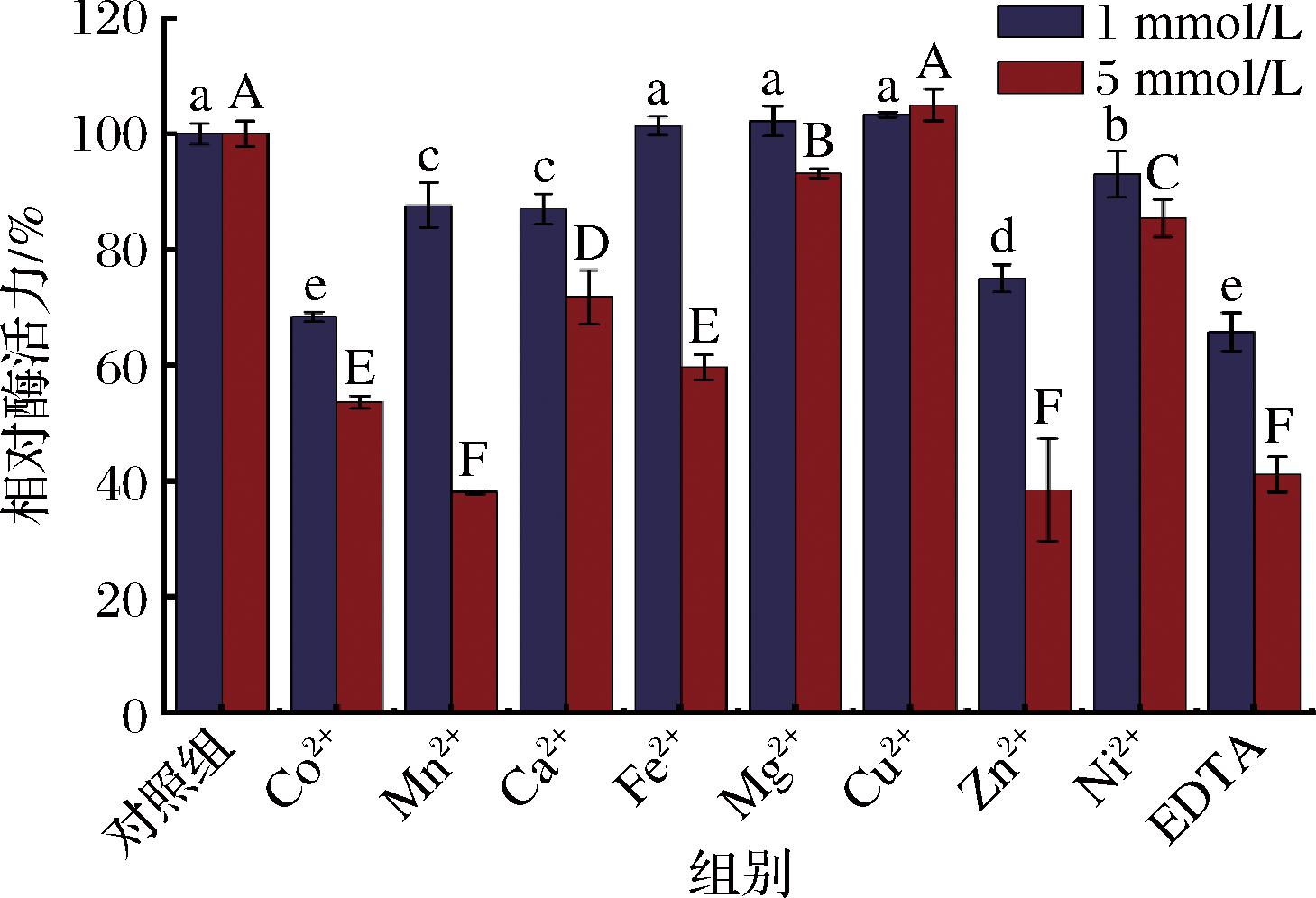
图7 金属离子对重组酶Alg1264活力的影响
Fig.7 Effects of metal ions on the activity of Alg1264
注:不同字母表示不同组别的差异显著(P<0.05),小写字母为低浓度组,大写字母为高浓度组(图8同)。
2.6.4 有机试剂对酶活力的影响
加入不同的有机试剂测定其对重组酶酶活力的影响。分析可知在低浓度和高浓度时,二硫苏糖醇(dithiothreitol,DTT)均对重组酶Alg1264活力有较明显的促进作用,最终使重组酶活力提升了近50%,由此推测重组酶Alg1264的活性中心具有巯基(—SH),DTT作为还原剂能够维持其还原态,进而维持酶的活性构象,最后提升了酶的活力。1 mmol/L盐酸胍、β-巯基乙醇以及体积分数5%的异丙醇、DMSO处理对重组酶活力影响较小,其相对酶活力基本维持在85%以上,在1 mmol/L TritonX-100、体积分数5%的甲醇和乙醇中的重组酶活力次之,但也能维持在70%左右。在5 mmol/L β-巯基乙醇和体积分数20%的DMSO处理下,Alg1264的相对酶活力也能维持在80%以上;在高浓度5 mmol/L盐酸胍、TritonX-100和体积分数20%的甲醇、乙醇以及异丙醇存在下,Alg1264的相对酶活力也能维持在60%以上(图8)。
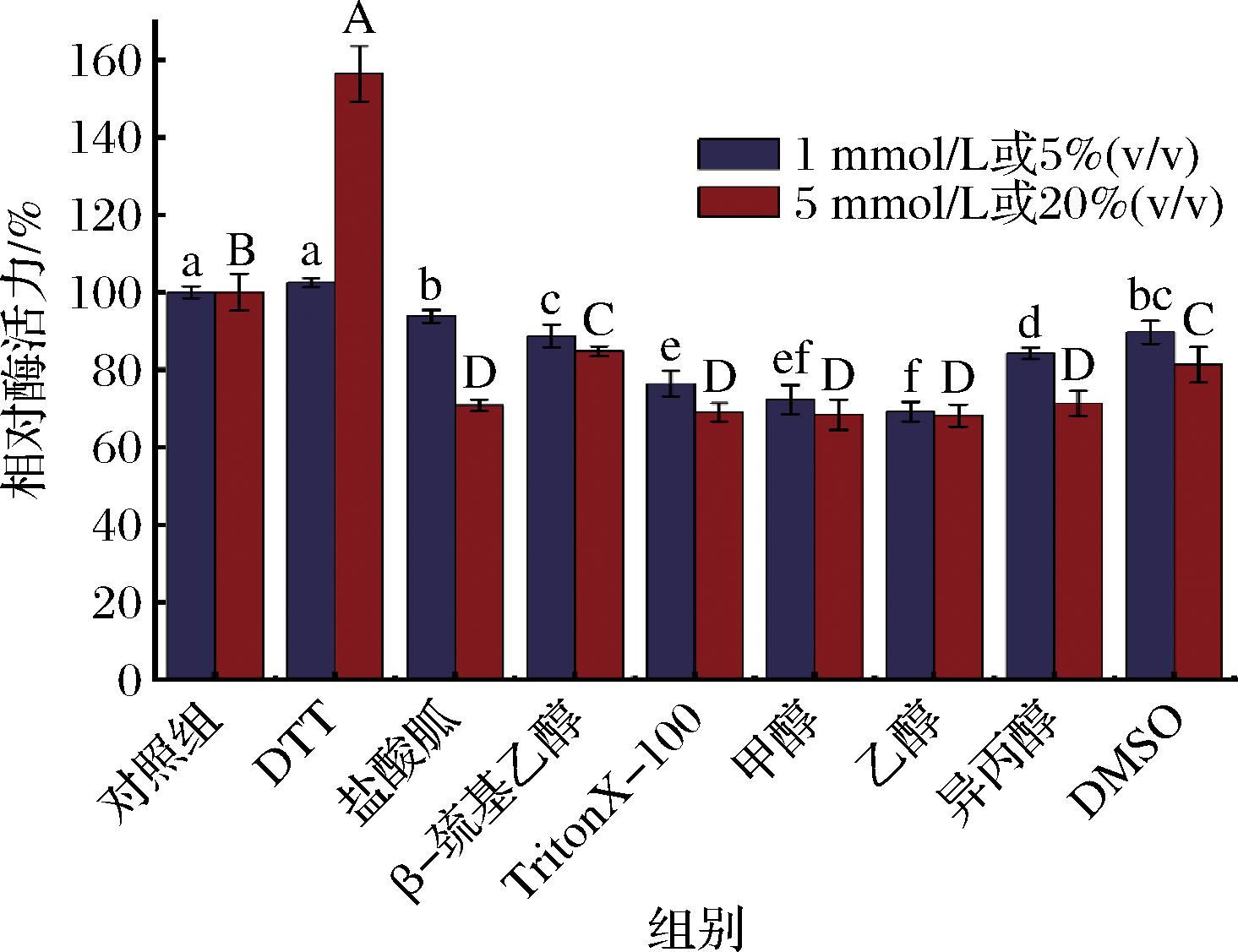
图8 有机试剂对重组酶Alg1264活力的影响
Fig.8 Effects of organic reagents on the activity of the recombinant enzyme Alg1264
2.6.5 底物特异性分析
检测重组酶在不同底物下的酶活力以探究酶的底物偏好性。重组酶对褐藻胶、polyG都有良好的降解活力,对polyG的降解活力最高,比活力为57.45 U/mg;对褐藻胶的降解活力次之,比活力为45.67 U/mg;对polyM的降解活力稍低,比活力为40.19 U/mg(图9),推断该酶是一种对polyG具有偏好的polyG裂解酶。
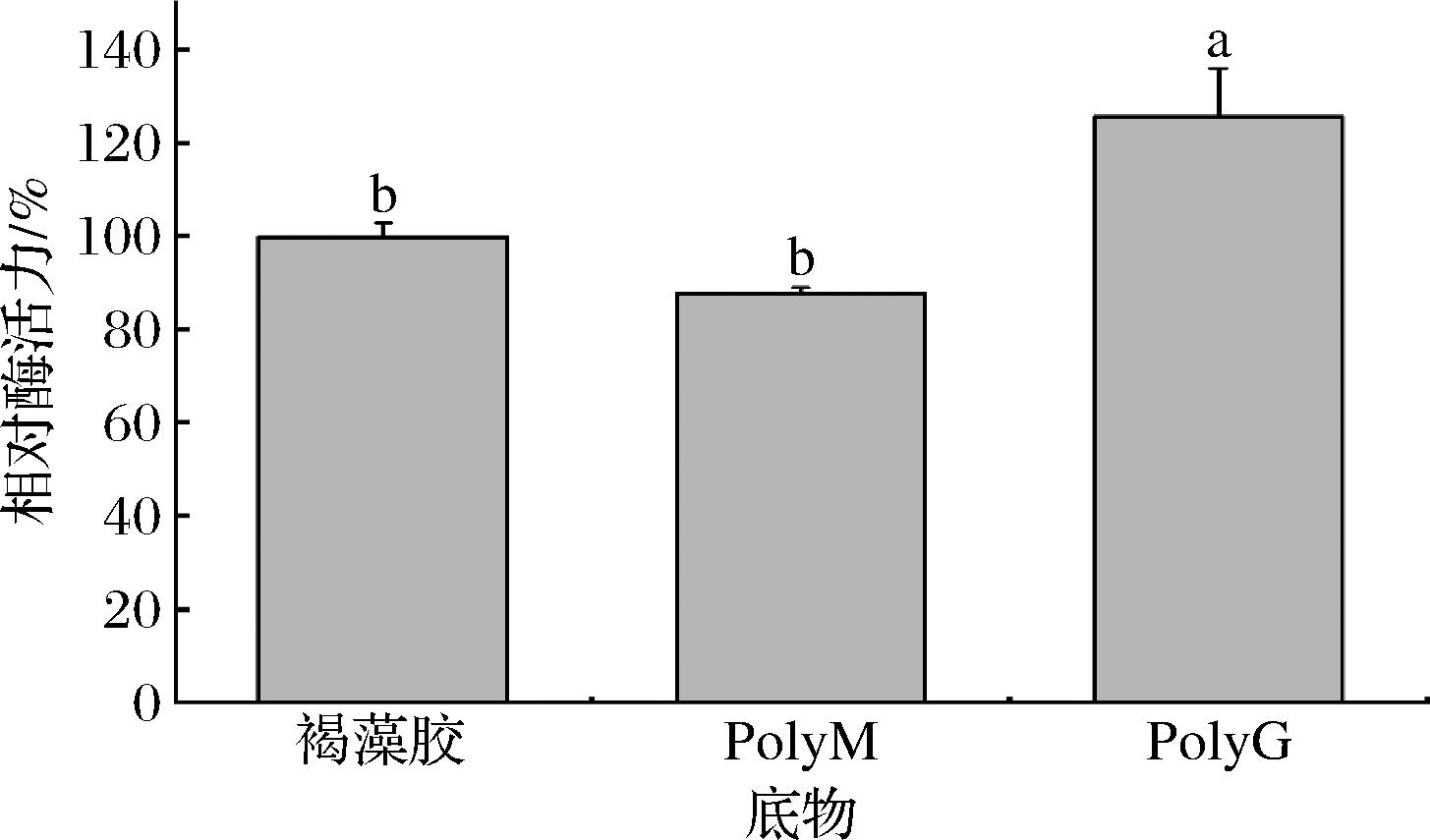
图9 重组酶Alg1264的底物特异性分析
Fig.9 Analysis of substrate specificity of the recombinant Alg1264
注:不同小写字母表示不同组别的差异显著(P<0.05)。
2.7 褐藻胶裂解酶水解产物的分析
从质谱分析结果可以看出,该酶降解褐藻胶、polyG、polyM 3种底物生成的产物均为单糖(194.52 m/z)、二糖(350.84 m/z)、三糖(526.76 m/z)、四糖(702.78 m/z)、五糖(878.70 m/z)。其中单糖含量最高,因此可以推断Alg1264是一种外切酶。但同时这些结果显示降解产物中确实有部分DP为2~5的寡糖生成,这表明重组酶也同时具有内切酶活力。因此,重组酶Alg1264是一种具有内切和外切特性的双功能褐藻胶裂解酶(图10)。
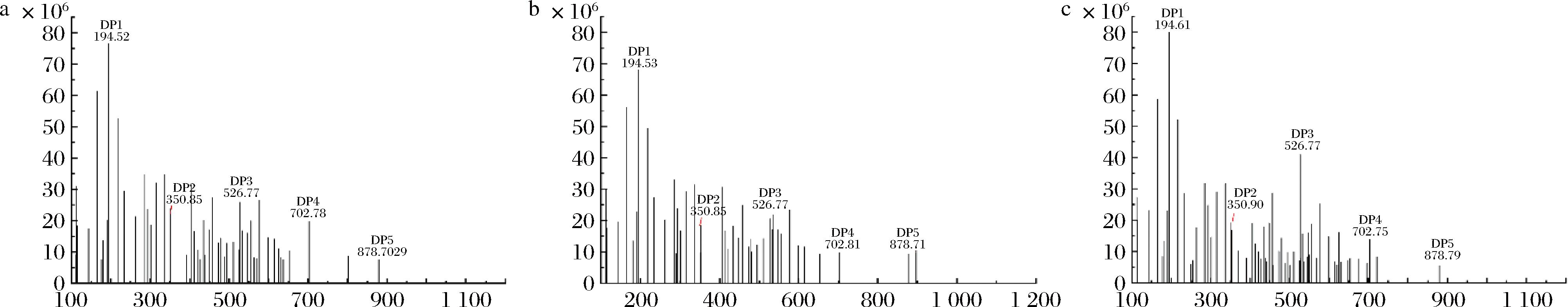
a-褐藻胶过夜反应水解产物的LC-MS分析;b-polyM过夜反应水解产物的LC-MS分析;c-polyG过夜反应水解产物的LC-MS分析
图10 LC-MS检测重组酶Alg1264的酶解产物
Fig.10 Characterization of Alg1264 enzymatic cleavage products with LC-MS
3 讨论
Alteromonas sp.A1-6原为一株浒苔多糖降解菌,对其基因组测序结果进行分析,alg1264被注释为褐藻胶裂解酶基因,因此以褐藻胶为唯一碳源进行诱导培养,检测基因alg1264的转录水平,发现其表达量显著上升。褐藻胶裂解酶能够高效降解褐藻胶并生成具有特定活性的寡糖,这对于满足日益增长的AOS需求至关重要。
本研究从Alteromonas sp. A1-6中鉴定出一种新的褐藻胶裂解酶Alg1264,经分析该酶属于PL7家族,对于polyG具有底物偏好性。GAOS与MAOS相比,在一些生理活性方面展现了更强的效果。而使用对polyG具有底物偏好性的酶,能够高效且精准地降解含有古罗糖醛酸结构单元的多糖,在褐藻胶处理中可以优先降解polyG链,减少副产物的产生。而在已知的褐藻胶裂解酶中,大多数表现出对polyM的底物偏好[26]。经底物特异性分析验证,重组酶Alg1264是一种对polyG具有偏好性的polyG裂解酶,可适用于GAOS的生产。同时重组酶对于polyM和褐藻胶也具有一定的活性,在天然褐藻胶的处理过程中,既能高效解聚目标区域(polyG),又能部分降解其他区域(polyM或MG模块),可减少预处理步骤,提升总糖转化率。
在已报道的褐藻胶裂解酶中,大多是具有内切活力的酶,对外切褐藻胶裂解酶和双功能酶的鉴定仍然有限,具有外切活力的酶大多属于PL15和PL17家族。尽管PL7家族中也有具外切活力的褐藻胶裂解酶,但其中已表征的褐藻胶裂解酶仍以内切酶为主。经分析,重组酶Alg1264是属于PL7家族的褐藻胶裂解酶。在对重组酶的水解产物进行鉴定时,发现重组酶Alg1264水解褐藻胶、polyG、polyM 3种底物产生的产物主要是单糖和DP为2~5的寡糖,因此重组酶Alg1264是一种兼具褐藻胶内切和外切活力的双功能酶。在PL7家族中,同样具有这种特性的有源于Alteromonas portus HB161718[27]的Alg2951和Volcanic metagenomic library[28]的KAlLy。重组酶Alg2951同样具有polyG偏好性,降解褐藻胶的主要产物为不饱和单糖和三糖,重组酶Alg2951仅在pH 7.0~8.5具有相对较高的酶活力,在pH<7.0或pH>8.5的环境中,其相对酶活力都在40%以下。在pH 4.0~6.0的条件下孵育1 h后测定其酶活力,发现酶基本失活。而重组酶Alg1264在pH 6.0~10.0的相对酶活力始终维持在80%以上,在pH 5.0~11.0孵育1 h后测定残留的酶活力,仍然保持77%以上的酶活力。由此可知,重组酶Alg1264的pH作用范围较广,且具有良好的pH稳定性。重组酶KAlLy对polyM具有底物特异性,研究发现,KAlLy易受金属离子的影响,浓度为2 mmol/L的Fe2+、Co2+、Cu2+、Ni2+、Zn2+能够明显抑制酶的活力。重组酶Alg1264在浓度为5 mmol/L 的Cu2+、Ni2+影响下,相对酶活力依旧维持在86%左右,而在5 mmol/L 的Co2+、Fe2+影响下,相对酶活力维持在50%以上。
除此之外,重组酶Alg1264还具有良好的有机溶剂耐受性,已报道的褐藻胶裂解酶中很少有酶具有这种特性。在利用褐藻制备AOS的生产工艺中,预处理阶段常涉及甲醇、乙醇或异丙醇等有机溶剂的使用,以去除杂质或改善褐藻胶的溶解性。然而,这些有机溶剂可能对后续酶解过程中所用的褐藻胶裂解酶的活力和稳定性产生不利影响。因此,筛选或改造具有有机溶剂耐受性的褐藻胶裂解酶,不仅有助于提高酶解效率和产物得率,还能优化工艺流程,增强其在工业生产中的适用性。在PL7家族中,目前已报道的来源于Microbulbifer thermotolerans DAU221的褐藻胶裂解酶Mt Al138[29]在体积分数20%的DMSO或者乙醇存在条件下分别仅剩下9%和25%的酶活力。而重组酶Alg1264在体积分数20%的乙醇或者DMSO中,其酶活力分别为60%和80%。
4 结论
本研究从Alteromonas sp.A1-6中克隆并异源表达同时具有内切和外切酶活力的褐藻胶裂解酶编码基因alg1264,经系统发育树分析可知该酶属于PL7家族。Alg1264的最适pH为7.0,最适温度为35 ℃,重组酶对褐藻胶、polyG都有良好的降解活力,对polyG的降解活力最高,比活力为57.45 U/mg;对褐藻胶的降解活力次之,比活力为45.67 U/mg;对polyM的降解活力稍低,比活力为40.19 U/mg。Mg2+、Ni2+对重组酶的影响比较小,在5 mmol/L的浓度影响下,其活力能维持在85%以上,Cu2+对重组酶活力基本没影响。此外,重组酶Alg1264对有机试剂有良好的耐受性,在5 mmol/L β-巯基乙醇和体积分数20%的DMSO的处理下,Alg1264的相对酶活力可以维持在80%以上;在高浓度5 mmol/L盐酸胍、TritonX-100和体积分数20%的甲醇、乙醇以及异丙醇存在下,Alg1264的相对酶活力也能维持在60%以上,这使得Alg1264能更好地应用于工业生产中。LC-MS分析可知,该酶降解褐藻胶,polyM,polyG生成的终产物主要为单糖、二糖、三糖和少量其他寡糖,故Alg1264是一种同时具有内切和外切酶活力的对polyG具有偏好性的裂解酶,该酶在AOS和GAOS的制备方面具有巨大潜力。
[1] 江君, 刘军, 杨绍青, 等. 黄杆菌褐藻胶裂解酶的高效表达及其在褐藻寡糖制备中的应用[J]. 食品科学, 2021, 42(4):145-152.JIANG J, LIU J, YANG S Q, et al. High level expression of alginate lyase from Flavobacterium sp. and its application in preparation of alginate oligosaccharides[J]. Food Science, 2021, 42(4):145-152.
[2] ZHANG L Z, LI X, ZHANG X Y, et al. Bacterial alginate metabolism: An important pathway for bioconversion of brown algae[J]. Biotechnology for Biofuels, 2021, 14(1):158.
[3] ÖZBILENLER C, ALTUNDA E M, GAZI M. Synthesis of quercetin-encapsulated alginate beads with their antioxidant and release kinetic studies[J]. Journal of Macromolecular Science, Part A, 2020, 58(1):22-31.
E M, GAZI M. Synthesis of quercetin-encapsulated alginate beads with their antioxidant and release kinetic studies[J]. Journal of Macromolecular Science, Part A, 2020, 58(1):22-31.
[4] LI A Q, GONG T, HOU Y J, et al. Alginate-stabilized thixotropic emulsion gels and their applications in fabrication of low-fat mayonnaise alternatives[J]. International Journal of Biological Macromolecules, 2020, 146:821-831.
[5] MARTIN M, PORTETELLE D, MICHEL G, et al. Microorganisms living on macroalgae: Diversity, interactions, and biotechnological applications[J]. Applied Microbiology and Biotechnology, 2014, 98(7):2917-2935.
[6] HAN Y, ZHANG L, YU X, et al. Alginate oligosaccharide attenuates α2,6-sialylation modification to inhibit prostate cancer cell growth via the Hippo/YAP pathway[J]. Cell Death &Disease, 2019, 10(5): 374.
[7] TERAKADO S, UENO M, TAMURA Y, et al. Sodium alginate oligosaccharides attenuate hypertension and associated kidney damage in dahl salt-sensitive rats fed a high-salt diet[J]. Clinical and Experimental Hypertension, 2012, 34(2):99-106.
[8] ZHOU R, SHI X Y, BI D C, et al. Alginate-derived oligosaccharide inhibits neuroinflammation and promotes microglial phagocytosis of β-amyloid[J]. Marine Drugs, 2015, 13(9):5828-5846.
[9] TØNDERVIK A, SLETTA H, KLINKENBERG G, et al. Alginate oligosaccharides inhibit fungal cell growth and potentiate the activity of antifungals against Candida and Aspergillus spp[J]. PLoS One, 2014, 9(11): e112518.
[10] EFTEKHARZADEH B, KHODAGHOLI F, ABDI A, et al. Alginate protects NT2 neurons against H2O2-induced neurotoxicity[J]. Carbohydrate Polymers, 2010, 79(4):1063-1072.
[11] LU S, NA K, WEI J N, et al. Alginate oligosaccharides: The structure-function relationships and the directional preparation for application[J]. Carbohydrate Polymers, 2022, 284:119225.
[12] 黄友涛, 梁青平, 高筱雅, 等. 不同结构褐藻胶寡糖的制备与功效研究进展[J]. 食品科学, 2024, 45(1):313-325.HUANG Y T, LIANG Q P, GAO X Y, et al. Research progress on preparation and functions of alginate oligosaccharides with different structures[J]. Food Science, 2024, 45(1):313-325.
[13] GUO Q, DAN M L, ZHENG Y T, et al. Construction and characterization of a novel fusion alginate lyase with endolytic and exolytic cleavage activity for industrial preparation of alginate oligosaccharides[J]. Food Chemistry, 2024, 453:139695.
[14] SUN H H, GAO L, XUE C H, et al. Marine-polysaccharide degrading enzymes: Status and prospects[J]. Comprehensive Reviews in Food Science and Food Safety, 2020, 19(6):2767-2796.
[15] WONG T Y, PRESTON L A, SCHILLER N L. ALGINATE LYASE: Review of major sources and enzyme characteristics, structure-function analysis, biological roles, and applications[J]. Annual Review of Microbiology, 2000, 54:289-340.
[16] SAWABE T, TAKAHASHI H, EZURA Y, et al. Cloning, sequence analysis and expression of Pseudoalteromonas elyakovii IAM 14594 gene (alyPEEC) encoding the extracellular alginate lyase[J]. Carbohydrate Research, 2001, 335(1):11-21.
[17] ARNTZEN M Ø, PEDERSEN B, KLAU L J, et al. Alginate degradation: Insights obtained through characterization of a thermophilic exolytic alginate lyase[J]. Applied and Environmental Microbiology, 2021, 87(6): e02399-20.
[18] BADUR A H, JAGTAP S S, YALAMANCHILI G, et al. Alginate lyases from alginate-degrading Vibrio splendidus 12B01 are endolytic[J]. Applied and Environmental Microbiology, 2015, 81(5):1865-1873.
[19] ZHU B W, YIN H. Alginate lyase: Review of major sources and classification, properties, structure-function analysis and applications[J]. Bioengineered, 2015, 6(3):125-131.
[20] 崔永燕, 杨敏, 刘楠, 等. 褐藻胶寡糖的酶法定向制备及其结构-功能关系的研究进展[J]. 食品科学, 2024, 45(10):320-329.CUI Y Y, YANG M, LIU N, et al. Research progress on the directed enzymatic preparation and structure-function relationship of alginate oligosaccharides[J]. Food Science, 2024, 45(10):320-329.
[21] 黎芯怡, 王静涵, 甄莉, 等. 浒苔多糖降解菌的筛选及其碳源利用谱分析[J]. 广西科技大学学报, 2023, 34(3):123-131.LI X Y, WANG J H, ZHEN L, et al. Screening of Ulva prolifera polysaccharide-degrading bacteria and analysis of its carbon source utilization spectrum[J]. Journal of Guangxi University of Science and Technology, 2023, 34(3):123-131.
[22] 李慧敏.高效褐藻胶降解菌株的筛选、基因挖掘及异源表达研究[D]. 武汉: 武汉轻工大学, 2023.LI H M. Alginate degrading gene mining from screened high efficiency strains and its heterologous expression[D]. Wuhan: Wuhan Polytechnic University, 2023.
[23] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method[J]. Methods, 2001, 25(4):402-408.
[24] BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72:248-254.
[25] SUMNER J B, GRAHAM V A. Dinitrosalicylic acid: A reagent for the estimation of sugar in normal and diabetic urine[J]. Journal of Biological Chemistry, 1921, 47(1):5-9.
[26] 李慧敏, 邵宗泽, 路瑶, 等. 弧菌DS32褐藻胶裂解酶基因vralg1的异源表达和酶学性质研究[J]. 应用海洋学学报, 2024, 43(2):255-265.LI H M, SHAO Z Z, LU Y, et al. Heterologous expression and enzymatic properties of the alginate lyase gene vralg1 from Vibrio sp. DS32[J]. Journal of Applied Oceanography, 2024, 43(2):255-265.
[27] HUANG H Q, LI S, BAO S X, et al. Expression and characterization of a cold-adapted alginate lyase with exo/endo-type activity from a novel marine bacterium Alteromonas portus HB161718T[J]. Marine Drugs, 2021, 19(3):155.
[28] TSOPANAKIS V, ANASTASIADOU E, MIKKELSEN M D, et al. Identification and characterization of a novel thermostable PL7 alginate lyase from a submarine volcanic metagenomic library[J]. Enzyme and Microbial Technology, 2024, 180:110486.
[29] JEONG H R, YOO J S, CHOI Y L, et al. Characterization of an organic solvent-tolerant polysaccharide lyase from Microbulbifer thermotolerans DAU221[J]. International Journal of Biological Macromolecules, 2021, 169:452-462.