甲萘醌-7(menaquinone-7,MK-7)是维生素K2的亚型之一[1],由2-甲基-1, 4-萘醌母环和位于母环C-3位置上的一个七聚异戊二烯侧链组成,主要贮存在细胞膜中[2]。MK-7具有治疗骨质疏松,保护心血管健康,预防癌症等功效[3],且在人体中比MK-4等其他维生素K2亚型能存在更久的时间,因此具有较高的医用价值[4]。MK-7对人类特别是对儿童、孕妇和中老年人等特殊人群在维持其骨骼和血液健康等方面具有重要作用,并且MK-7在天然食品中的含量较少。随着现代人类饮食习惯的改变,MK-7也常被用作乳制品以及其他食品中的重要营养补剂,因此,MK-7在食品保健领域同样具有广阔的市场前景[5-7]。
MK-7多存在于纳豆和豆豉等天然发酵食品中,可以通过直接提取这类食品中的MK-7进行生产[8-9],但是这类天然提取方法往往面临产量低成本高的问题。另外,也可以直接利用代谢工程改造微生物进行发酵,如纳豆芽孢杆菌[10]、枯草芽孢杆菌[11]和大肠杆菌[12]等。CUI等[11]通过全局调控代谢途径和过表达甲基赤藓糖醇4-磷酸途径(methylerythritol 4-phosphate pathway,MEP)等策略,实现了枯草芽孢杆菌在摇瓶上积累415 mg/L的MK-7。XU等[13]通过级联调控异戊二烯基二磷酸(isopentenyldiphosphate,IPP)代谢,实现利用枯草芽孢杆菌在50 L发酵罐中发酵产生1 549.6 mg/L 的MK-7,这也是目前报道的MK-7生产的最高产量。大肠杆菌作为模式菌株,具有发酵周期短、易于操作等优势,但是大肠杆菌本身只有八聚异戊二烯焦磷酸酶IspB,其内源只能合成MK-8和辅酶Q8等相关代谢产物。GAO等[14]通过引入枯草芽孢杆菌的七聚异戊二烯焦磷酸合成酶HepPPS实现了大肠杆菌中合成七聚异戊二烯基焦磷酸(heptaprenylpyrophosphate,HepPP),这也是大肠杆菌合成MK-7的关键所在,又通过过量表达了1, 4-二羟基-2-萘甲酸七烯基转移酶MenA和甲萘醌生物合成C-甲基转移酶UbiE(MenG),实现了MK-7的合成,其摇瓶产量可达129 mg/L。
本研究以一株引入甲羟戊酸途径(mevalonate pathway,MVA)的大肠杆菌作为底盘菌株,通过DLKcat模拟程序[15]比较不同来源的关键酶HepS、HepT、MenA和MenG,并对其进行组合表达优化,MK-7产量可达到84.18 mg/L。接着,通过引入异戊烯基焦磷酸异构酶EcIdi平衡二甲基烯丙基二磷酸(dimethylallyldiphosphate,DMAPP)和IPP,MK-7积累量可以达到106.54 mg/L。随后,在5 L发酵罐上对得到的菌株进行补料分批发酵的发酵工艺优化,包括延迟诱导时间,提升溶氧量至60,最终,MK-7产量达到929.58 mg/L,但其前体物质去甲基甲萘醌-7(demethylmenaquinone-7,DMK-7)也存在大量堆积,达到1 217.58 mg/L。为降低DMK-7的堆积问题,筛选不同来源的MenG,发现Bs168MenG可以有效降低DMK-7。最终,5 L发酵罐上MK-7的最高产量达到1 090.45 mg/L,DMK-7积累量降低至231.14 mg/L。本研究结果对应用大肠杆菌生产MK-7具有一定的指导意义。
1 材料和方法
1.1 菌株和引物
本研究中采用Escherichia coli JM109用于DNA片段扩增,质粒构建和质粒保存,E.coli BW25113用于改造并生产MK-7,Bacillus subtilis168用于DNA片段扩增。本研究中构建并使用到的菌株如表1所示。
表1 本研究中使用的菌株
Table 1 Strains used in this study

菌株描述来源E.coli JM109General cloning host实验室保存E.coli BW25113BW25113,ParaBAD-menF,ΔpoxB::ParaBAD-mvaE-mvaS-mvk, ParaBAD-pmk-mvd,Eb-01实验室保存pACYCDuetCmR,araBAD promoter,p15A ori本研究pRSFDuetKanR,araBAD promoter,RSF ori本研究pETDuetAmpR,araBAD promoter,f1 ori本研究pCDFDuetSmR,araBAD promoter,CloDF13 ori本研究pSB1aAmpR, araBAD promoter,pSC101 ori本研究Eb-02Eb-01ΔldhA::ParaBAD-Ecidi本研究Eb-03Eb-02ΔispB本研究Eb-04Eb-02ΔubiCA本研究Eb-05Eb-02ΔwrbA本研究Eb-06Eb-02ΔqorB本研究Eb-07Eb-02ΔwrbAΔqorB本研究E1K-1Eb-01 harboring pACYCDuet-RBS2-BshepS-RBS2-BshepT-RBS2-EcmenA-RBS2-BsmenG本研究E1K-2Eb-01 harboring pACYCDuet-RBS3-hepS-RBS3-hepT-RBS3-menA-RBS3-menG本研究E1K-3Eb-01 harboring pACYCDuet-RBS1-hepS-RBS1-hepT-RBS1-menA-RBS1-menG本研究E1K-4Eb-01 harboring pACYCDuet-RBS2-hepS-RBS2-hepT-RBS1-menA-RBS3-menG本研究E1K-11Eb-01 harboring pRSFDuet-BshepS-BshepT-EcmenA-BsmenG本研究E1K-12Eb-01 harboring pETDuet-BshepS-BshepT-EcmenA-BsmenG本研究E1K-13Eb-01 harboring pCDFDuet-BshepS-BshepT-EcmenA-BsmenG本研究E1K-14Eb-01 harboring pSB1a-BshepS-BshepT-EcmenA-BsmenG本研究E1K-15Eb-01 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG本研究E1K-16Eb-01 harboring pSB1a-MihepS-MihepT-MbmenA-BsmenG本研究E1K-17Eb-01 harboring pSB1a-CfhepS-CfhepT-PrmenA-BsXTmenG本研究E2K-15Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG本研究E3K-15Eb-03 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG本研究E4K-15Eb-04 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG本研究E5K-15Eb-05 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG本研究E6K-15Eb-06 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG本研究E7K-15Eb-07 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG本研究E2K-18Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG and pET28a-menD本研究E2K-19Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG and pET28a-dxs本研究E2K-21Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-BsmenG本研究E2K-22Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-BsXTmenG本研究E2K-23Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-EcmenG本研究E2K-24Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-Bs168 menG本研究E2K-26Eb-02 harboring pSB1a-BthepS-BthepT-MnmenA-NtmenG-ParaBAD-Scsam2本研究
本实验中均使用高保真DNA聚合酶扩增DNA片段,由上海生工即用型无缝克隆试剂盒进行质粒构建。质粒构建以pET28a-menD构建为例,以质粒pET28a为模板,以引物pET-F/pET-R扩增获得载体片段,以E.coli JM109作为模板,以引物MenD-F/MenD-R扩增获得menD基因片段,按照Gibson组装质粒。本研究中所有引物均由上海生工生物工程股份有限公司合成,部分引物如表2所示,构建质粒中使用的不同强度的核糖体结合位点(ribosomebinding site,RBS)[16]如表3所示。
表2 本实验中所用引物
Table 2 The primers used in this experiment

引物名称序列(5′-3′)pACYC-FCTGCTGCCACCGCTGAGpACYC-RATTTCCTAATGCAGGAGTCGCATAAGGGpRSF-FGTGTCGGCATCGTTGAATCAGCpRSF-RCGCCTGTGTACATCGCAATACGpET-FCATGCCTGCCTGCTGATGGpET-RCAGGCCTGCCAGTTCCTTCpCDF-FCACGGCGTCACACTTTGCTpCDF-RGAATTCGGTGAGCTCGGTCTGCpSB1a-FGCACGGCGTCACACTTTGCTpSB1a-RGTGAGCTCGGTCTGCAGCTGGParaBAD-FCACGGCGTCACACTTTGCTATGCCATAGCATTTTTATParaBAD-RCTAACAGGAGGAATTAACCATGGGGATGCAAGACATCTACGGAACRBS1-HepS-FAGCGAACCACGACAATGCAAGACATCTACGGAACTTTAGRBS1-HepS-RCCTATCATCAGAAAGTGGAAGAAGGGTAAGAGCTAGCGAACCACGACARBS1-HepT-FGAGCTAGCGAACCACGACAATGTTAAATATCATTCGTTTACTGGCGGAGRBS1-HepT-RGCCATCGCAAAATATATCGGTAAAAGAAAATTTTAAGAATTAGCGAACCACGACARBS1-MenA-FGAATTAGCGAACCACGACAATGACTGAACAACAAATTAGCCGAACRBS1-MenA-RTAAGCCAGTGGGCAGCATAAGAGCTAGCGAACCACGACARBS1-MenG-FGAGCTAGCGAACCACGACAATGCAGGACTCAAAAGAACAGCGRBS1-MenG-RCCACGCATATCGGATGGAAATGAGAATTCGGTGAGCTCGGTCTGRBS2-HepS-FCAGGAGGAATTAACCATGGGGATGCAAGACATCTACGRBS2-HepS-RCCTATCATCAGAAAGTGGAAGAAGGGTAAGAGCTCAGGAGGAATTAACRBS2-HepT-FGAGCTCAGGAGGAATTAACATGTTAAATATCATTCGTTTACTGGCGGRBS2-HepT-RCCATCGCAAAATATATCGGTAAAAGAAAATTTTAAGAATTCAGGAGGAATTAACRBS2-MenA-F GAATTCAGGAGGAATTAACATGACTGAACAACAAATTAGCCGAACRBS2-MenA-RCTAAGCCAGTGGGCAGCATAAGAGCTCAGGAGGAATTAACRBS2-MenG-RGCCACGCATATCGGATGGAAATGAGAATTCGGTGAGCTCGGTCTRBS3-HepS-FGGGGAAGAAGGAGATATAATGCAAGACATCTACGGAACTTTAGRBS3-HepS-RCATCAGAAAGTGGAAGAAGGGTAATAGTCGAAGAAGGAGATATARBS3-HepT-FTAGTCGAAGAAGGAGATATAATGTTAAATATCATTCGTTTACTGGCGGRBS3-HepT-RTATATCTCCTTCTTCGACTATTAAAATTTTCTTTTACCGATATATTTTGCGATGGCRBS3-MenA-FTAGTCGAAGAAGGAGATATAATGACTGAACAACAAATTAGCCGAACRBS3-MenA-RTATATCTCCTTCTTCGACTATTATGCTGCCCACTGGCTTAGRBS3-MenG-FTAGTCGAAGAAGGAGATATAATGCAGGACTCAAAAGAACAGCGRBS3-MenG-RGCCACGCATATCGGATGGAAATGAGAATTCGGTGAGCTCGGTCTBthepS-FAGGAGGAATTAACCATGGGGATGCAGCACATCCACGCGBthepS-RCGTGAAGAAAATCGTTGAAGAAGGCTAAGAGCTCAGGAGGAATTAACBthepT-FGAGCTCAGGAGGAATTAACATGAAACTGAAAATGATGTACAGCTTCCTGBthepT-RCATGTTAATTCCTCCTGAATTCTTAGAATTTACGTTTGCCGATGAATTTCGMnmenA-FGAATTCAGGAGGAATTAACATGGATCAGCAGACCCTGGGMnmenA-RCGGCCTGTTCATCAGCTACTACCTGTAAGAGCTCAGGAGGAATTAACNtmenG-FGAGCTCAGGAGGAATTAACATGCGTGAACACCAGAGCAAAGAAGAAAAAGTTCACCACGNtmenG-RCAGACCGAGCTCACCGAATTCTTATTTTTCTTTGAAGCCGATGTGAACCMihepS-FAGGAGGAATTAACCATGGGGATGATCGTGCAGAACATCTACGACMihepS-RCACTGCAAAAACGTTGTTGGCGAAGGCTAAGAGCTCAGGAGGAATTAACMihepT-FGAGCTCAGGAGGAATTAACATGAAACTGAAAATGATGTACGCGTTCMihepT-RTGTTAATTCCTCCTGAATTCTTAGAATTTACGTTTGCCAACGAATTTCGMbmenA-FAGAATTCAGGAGGAATTAACATGACCAAATCTAACAACGACAGCMbmenA-RCAGCTACGGCCTGCGTTAAGAGCTCAGGAGGAATTAACBsmenG-FGAGCTCAGGAGGAATTAACATGCAGGATAGCAAAGAACAGCGBsmenG-RAGACCGAGCTCACCGAATTCTTATTTCCAGCCGATGTGGGTCCfhepS-FGGAGGAATTAACCATGGGGATGCACGATGTTAAGATCAAGCTGGCfhepS-RGCAAAGACCTTTGTGGAAGAAGGTTAAGAGCTCAGGAGGAATTAACCfhepT-FGAGCTCAGGAGGAATTAACATGAAGCTGAAGATGATGTACTCTTTCCCfhepT-RTGTTAATTCCTCCTGAATTCTTAGAACTTACGTTTGCCAATAAATTTGGCPrmenA-FGAATTCAGGAGGAATTAACATGAGCCAGACCAGTTATGCCPrmenA-RAGCGTGGGTCTGTTCATTAGTTATTATCTGTAAGAGCTCAGGAGGAATTAACBsXTmenG-FGAGCTCAGGAGGAATTAACATGCAGGATAGCAAAGAACAGCBsXTmenG-RCAGACCGAGCTCACCGAATTCTTATTTCCAACCAATATGGGTTGCGGMenD-FGAGCTCAGGAGGAATTAACATGTCAGTAAGCGCATTTAACCGACMenD-RGCTTTGTTAGCAGCCGGATCTCATAAATGGCTTACCTGCGCCDxs-FCAGGAGGAATTAACCATGGGGATGAGTTTTGATATTGCCAAATACCCGAC
续表2

引物名称序列(5′-3′)Dxs-RGCTTTGTTAGCAGCCGGATCTTATGCCAGCCAGGCCTTGBs168menG-FGAGCTCAGGAGGAATTAACATGCAGGBs168menG-RCCACGCATATCGGATGGAAATGAGAATTCGGTGAGCTCEcmenG-FGAGCTCAGGAGGAATTAACATGGTGGATAAGTCACAAGAAACGACEcmenG-RCAGACCGAGCTCACCGAATTCTCAGAACTTATAACCACGATGCAGCGEcidi-FATGCAAACGGAACACGTCATEcidi-RAATGCAGGGGAGCGGCAAGATTATTTAAGCTGGGTAAATGCAGATAATCG
表3 不同强度的RBS
Table 3 RBS of different strength
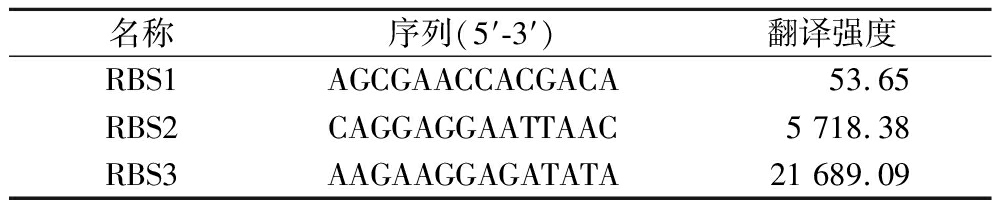
名称序列(5′-3′)翻译强度RBS1AGCGAACCACGACA 53.65RBS2CAGGAGGAATTAAC5 718.38RBS3AAGAAGGAGATATA21 689.09
1.2 培养基
LB培养基(g/L):蛋白胨10,酵母粉5,NaCl 10,在115 ℃条件下灭菌20 min。
TB培养基(g/L):蛋白胨12,酵母粉24,KH2PO4 2.31,K2HPO4 16.14,甘油4,在115 ℃条件下灭菌20 min。
M9培养基:葡萄糖18 g/L,Na2HPO4·7H2O 12.8 g/L,KH2PO43 g/L,NaCl 0.5 g/L,NH4Cl 1 g/L,CaCl2·2H2O 0.1 mmol/L和MgSO4·7H2O 5 mmol/L。
发酵培养基:葡萄糖25 g/L,甘油10 g/L,(NH4)2SO4 7.5 g/L,K2HPO4·3H2O 3 g/L,KH2PO4 2 g/L,MgSO4·7H2O 1 g/L,柠檬酸钠1.0 g/L,维生素B1 0.1 g/L,酵母粉7 g/L,微量元素1 mL/L,依据需求加入适量相应的抗生素。
补料培养基(g/L):葡萄糖700,酵母粉10,MgSO4·7H2O 3。
微量元素(g/L):Al2(SO4)3·18H2O 2.0,CoSO4·7H2O 0.75,CuSO4·5H2O 2.5,H3BO3 0.5,MnSO4·H2O 24,NiSO4·6H2O 2.5,ZnSO4·7H2O 15。
1.3 培养方法
1.3.1 摇瓶培养
大肠杆菌菌株在37 ℃下以220 r/min的转速在LB培养基中生长,培养基中根据需要补充相关抗生素(50 mg/L卡那霉素、50 mg/L链霉素或100 mg/L氨苄青霉素)。按照培养基体积2%的量接种至TB培养基中,经终质量浓度为2 g/L的阿拉伯糖诱导后,在30 ℃,220 r/min条件下培养16 h,随后3 500 r/min离心10 min收集菌体,利用M9培养基重悬再培养10 h,随后取样检测。
1.3.2 5 L发酵罐补料分批发酵
对改造后的发酵菌株活化后,接种至LB液体培养基,在37 ℃,220 r/min条件下培养6~7 h,按照培养基体积8%的量接种至发酵培养基中,在37 ℃条件下培养,在OD600值达40~70时降温至30 ℃,加入终质量浓度为2 g/L的阿拉伯糖诱导8 h,随后恢复至37 ℃继续培养至发酵结束,在此期间每6 h取1次样。根据菌种不同加入相应抗生素(50 mg/L卡那霉素、50 mg/L链霉素或100 mg/L氨苄青霉素)。
1.4 分析方法
将发酵液稀释至合适倍数后,用分光光度计测量600 nm处的吸光度以测定细菌细胞密度。以V(正己烷)∶V(异丙醇)=2∶1的比例混合用作MK-7萃取剂,取1 mL发酵液在室温下以10 000 r/min离心3 min获取大肠杆菌菌体,加入萃取剂振荡混匀10 min,随后离心收集萃取剂进行检测。使用配备有光子二极管阵列紫外检测器和C18色谱柱(4.6 mm×150 mm,3.5 μm)的高效液相色谱仪检测MK-7的含量,检测条件是40 ℃、波长254 nm。流动相为甲醇,流速1 mL/min。
1.5 DLKcat预测并筛选关键酶
将关键酶七聚异戊二烯焦磷酸合成酶Ⅱ(BsHepT)、1, 4-二羟基-2-萘甲酸七烯基转移酶(EcMenA)和甲萘醌生物合成C-甲基转移酶(BsMenG)的基因序列在NCBI数据库(https://www.ncbi.nlm.nih.gov/home/analyze/)中进行比对挖掘,分别将序列相似性排名在前500的基因利用DLKcat程序模拟与各自的底物对接并预测kcat值,然后将其进行排序,分别挑选得分最高的3组基因如表4所示,随后利用NCBI数据库将选出的hepT相同来源的hepS作为一组,具体关键酶基因的来源及其GenBank登录号如表5所示,通过上海生工生物工程股份有限公司优化密码子后进行合成。
表4 DLKcat模拟对接预测分数
Table 4 DLKcat simulated the predicted score of docking
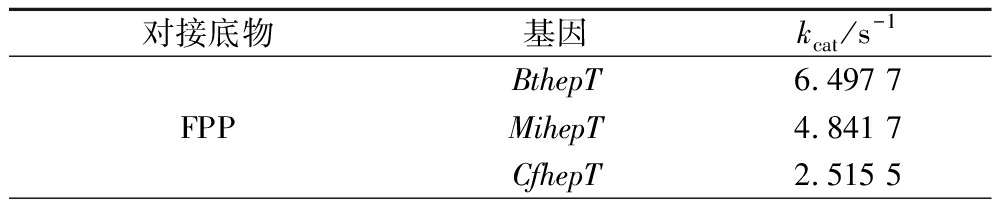
对接底物基因kcat/s-1FPPBthepT6.497 7MihepT4.841 7CfhepT2.515 5
续表4
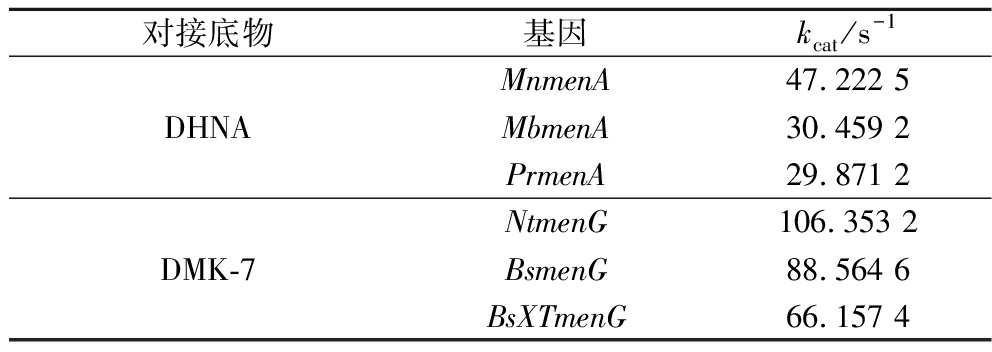
对接底物基因kcat/s-1DHNAMnmenA47.222 5MbmenA30.459 2PrmenA29.871 2DMK-7NtmenG106.353 2BsmenG88.564 6BsXTmenG66.157 4
表5 DLKcat筛选关键基因
Table 5 DLKcatscreened key genes

基因来源GenBank IDBthepSBacillus timonensisMCA1030522.1BthepTBacillus timonensisMCA1030520.1MnmenAMetabacillus niabensisWP_226527137.1NtmenGNiallia taxiWP_144452823.1MihepSMetabacillus iocasaeWP_239583272.1MihepTMetabacillus iocasaeWP_205183397.1MbmenAMetabacillus sp.DBTR6MBZ5749476.1BsmenGBacillus subtilisWP_069149790.1CfhepSCytobacillus firmusWP_061793825.1CfhepTCytobacillus firmusWP_061793827.1PrmenAPriestia flexaWP_263621957.1BsmenGBacillus sp.XT-2WP_263463246.1
2 结果与分析
2.1 引入关键酶合成MK-7
首先选用不同拷贝数的载体,将BshepS(GenBank登录号:NP_390157.1)、BshepT(GenBank登录号:NP_390155.1)、EcmenA(GenBank登录号:AIN34229.1)、BsmenG(GenBank登录号:NP_390156.1)4种关键酶基因进行连接,并进行表达,随后采用不同强度的RBS进行连接。结果表明产量最高的组合是由pSB1a作为载体、RBS2连接基因构建的质粒整合进菌株Eb01的组合E1K-14(图1),MK-7产量可达50.63 mg/L。结合其他组合摇瓶发酵的结果分析,不管是高拷贝还是强RBS,构建的质粒进行发酵时MK-7的产量均低于较低拷贝和中强度RBS组合,这可能是由于MenA是膜蛋白,并且存在多个跨膜结构域,无法过量表达所导致[17-18]。
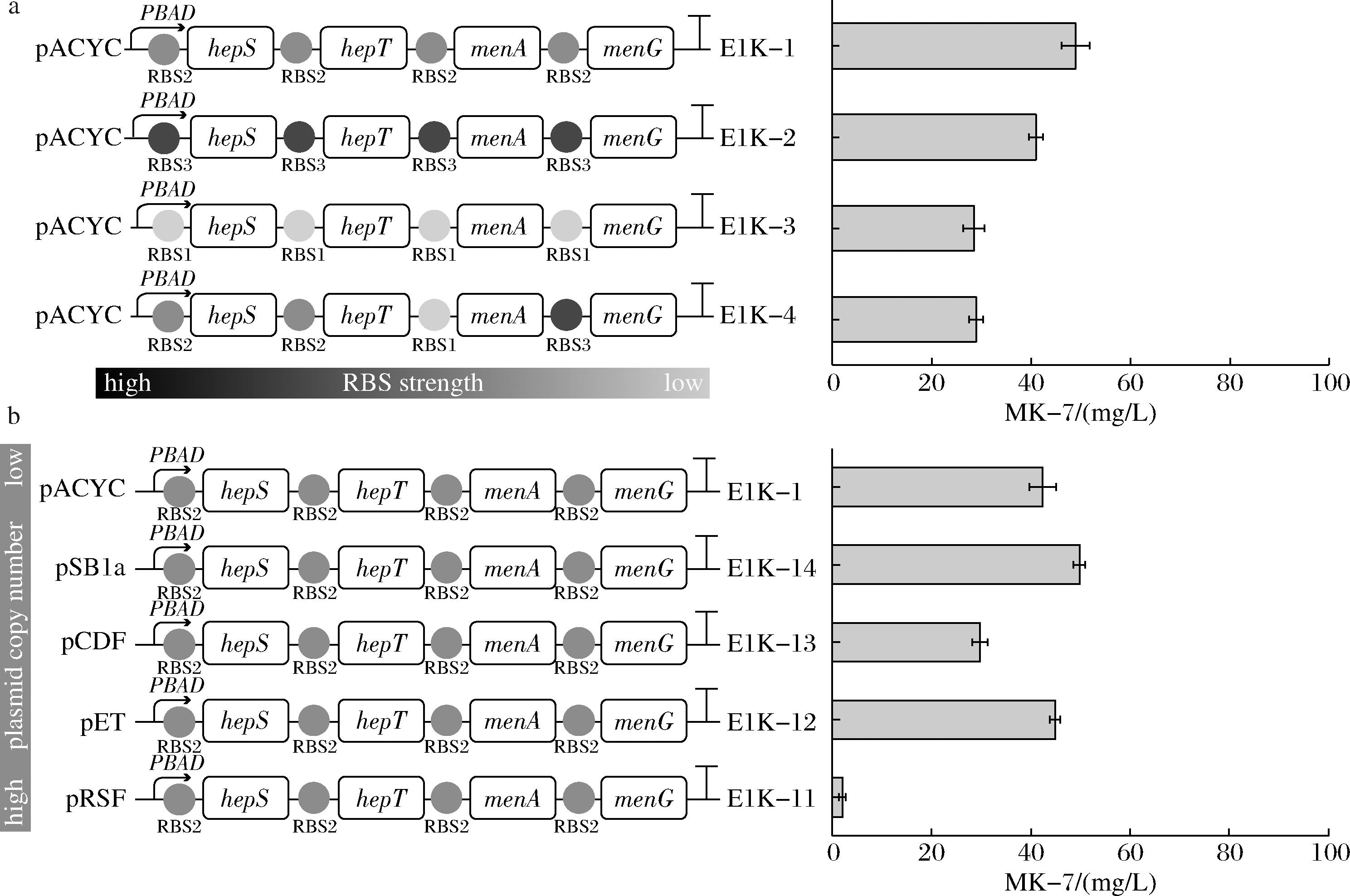
a-不同强度的RBS构建质粒生产MK-7;b-不同载体构建质粒生产MK-7
图1 对关键酶进行表达优化提升MK-7产量
Fig.1 The expression of key enzymes was optimized to increase MK-7 yield
为了获得更加高效的关键酶来提升MK-7的合成能力,利用DLKcat模拟预测不同来源的HepS、HepT、MenA、MenG的kcat值,并分别选择预测kcat值排名在前3位的基因(表4)。将它们分别以质粒pSB1a为载体,以RBS2连接起来组合构建质粒,并转化至Eb01中,得到菌株E1K-15、E1K-16、E1K-17,进行摇瓶发酵验证。结果如图2所示,这3株菌均可以产生MK-7,其中由BthepS(GenBank登录号:MCA1030522.1)、BthepT(GenBank登录号:MCA1030520.1)、MnmenA(GenBank登录号:WP_226527137.1)和NtmenG(GenBank登录号:WP_144452823.1)组合后所得菌株E1K-15效果最好(如图2),摇瓶水平产量达84.18 mg/L。
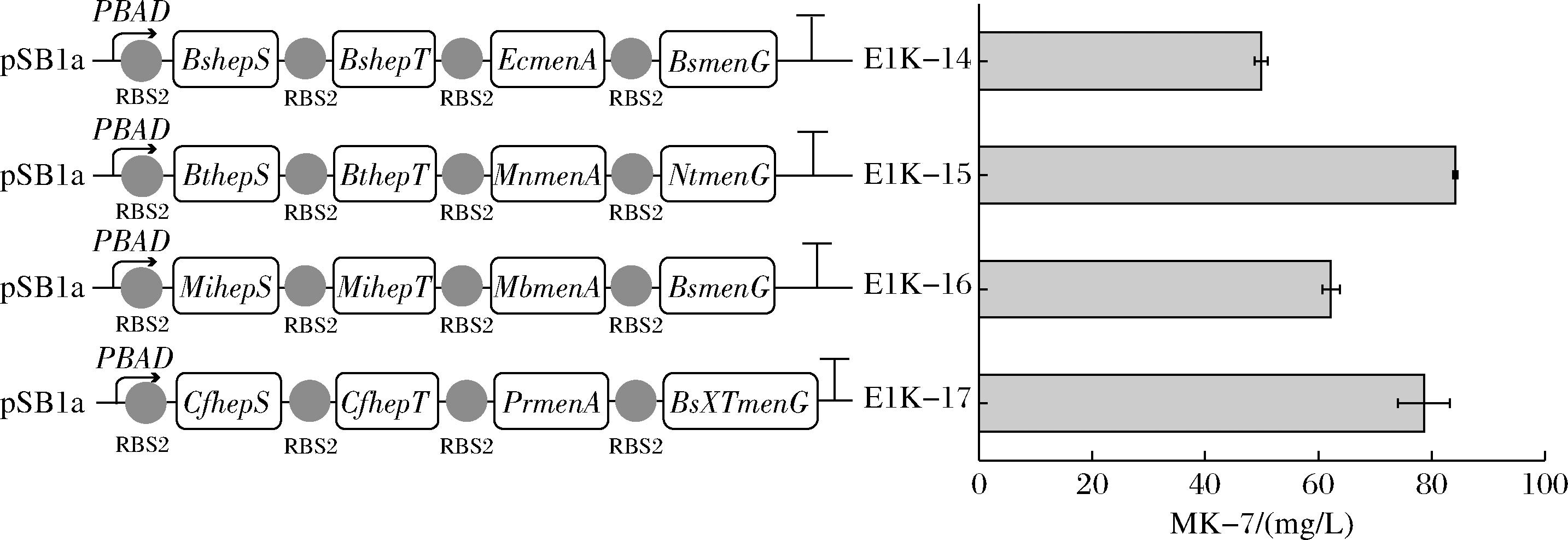
图2 组合不同来源的关键基因生产MK-7
Fig.2 Combination of key genes from different sources to produce MK-7
2.2 合成MK-7的整体途径优化
在前期研究中,通过引入酵母来源的MVA途径[19]和过表达EcMenF[12],增强了MK-7的两大关键前体七聚异戊二烯焦磷酸HepPP和1, 4-二羟基-2-萘甲酸(1,4-dihydroxy-2-naphthoate,DHNA)的合成。经过2.1节引入BthepS、BthepT、MnmenA和NtmenG并优化表达后,继续对前体合成途径上的关键酶进行强化表达以保证前体供应充足,且对其竞争和降解途径进行敲除。KONG等[20]研究表明过表达MenD,Idi,Dxr时可增强MK-8合成。首先,在E1K-15整合了基因Ecidi构成菌株E2K-15,以平衡DMAPP和IPP的量。MK-7达到了106.54 mg/L(图3-b)。通过过表达MenD和Dxs并同时直接外源添加1 mmol/L DHNA,发现MK-7的直接前体去甲基甲萘醌-7(DMK-7)的积累量明显增高,而MK-7的合成量大幅下降,可见前体供应的平衡对DMK-7转化为MK-7有较大影响。
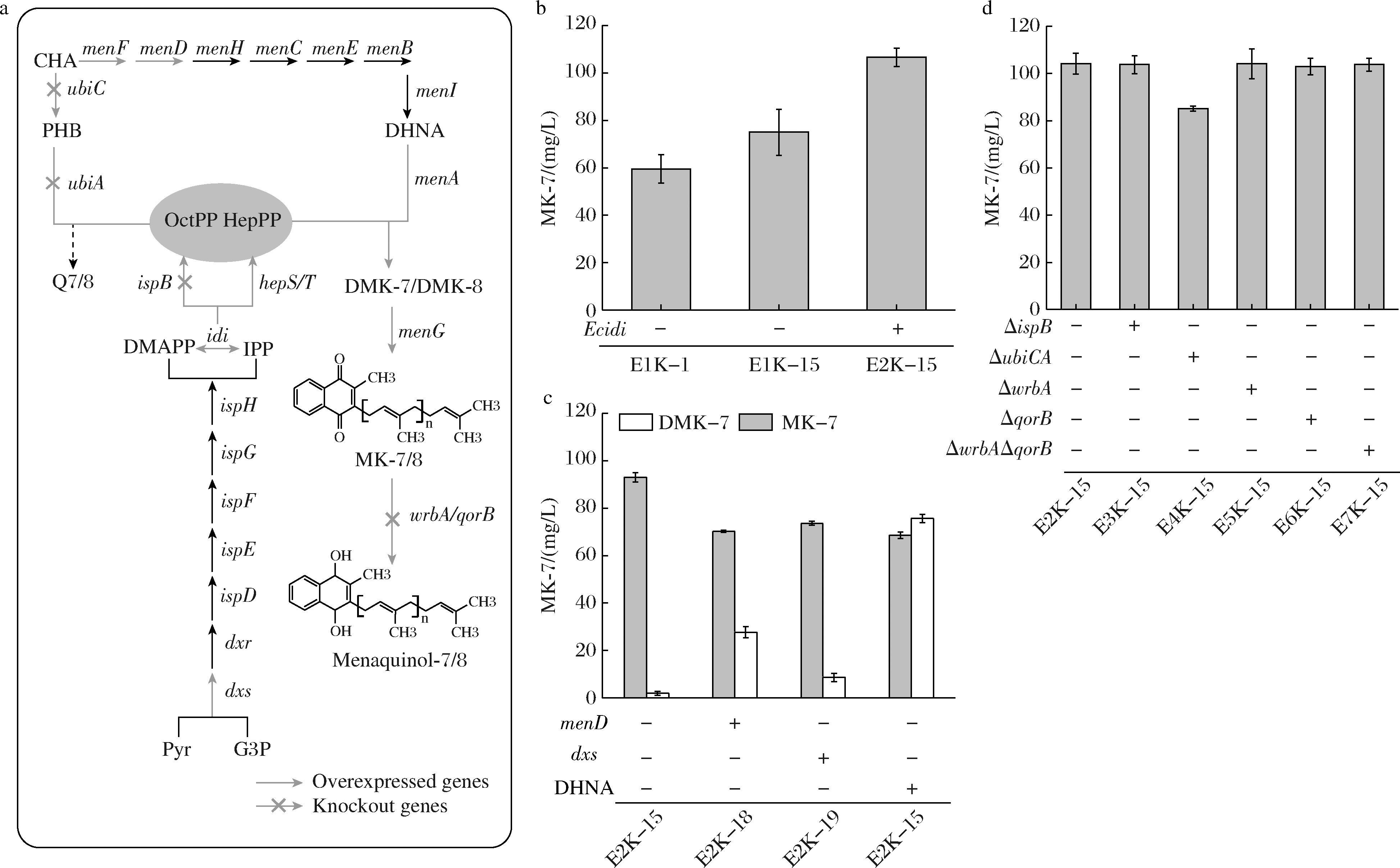
a-改造整体代谢合成MK-7途径;b-整合过表达Idi 提升MK-7合成;c-过表达MenD或Dxs和外源添加DHNA对MK-7合成的影响;d-敲除竞争途径和降解途径对MK-7合成的影响
图3 合成MK-7的整体途径优化以提升产量
Fig.3 The overall pathway for the synthesis of MK-7 was optimized to increase yield
另外,MK-8和辅酶Q8是大肠杆菌内源的甲萘醌和泛醌,ispB[20]和ubiCA[21]作为合成这两者途径上的关键酶基因,将其敲除可能降低前体的竞争消耗。根据KEGG数据库(https://www.kegg.jp/kegg/)查询可知,WrbA[22]和QorB[23]作为醌氧化还原酶,可将甲萘醌-7还原为二氢甲萘醌-7。然而,在E1K-15中分别敲除上述酶的基因后,MK-7的量并无明显提升,甚至在ubiCA敲除后,MK-7的产量出现明显下降(图3-d)。因此,后续研究仍选用E2K-15菌株。
2.3 对MK-7合成的发酵条件优化
对菌株E2K-15在5 L发酵罐进行补料分批发酵。将菌株活化后,依次接种至一级种子液及二级种子液中,二级种子液在37 ℃中生长10 h后按照培养基体积8%的接种量进行罐上接种。在37 ℃下培养至OD600值达40时,添加诱导剂,在30 ℃诱导8 h,随后将温度调至37 ℃,发酵过程溶氧(dissolved oxygen,DO)值保持在30。结果表明,MK-7在62 h时达到最高产量577.32 mg/L,然而,同时出现大量DMK-7的堆积,高达851.91 mg/L,且菌体生长速度缓慢,22 h时OD600值仅达41.37,发酵结束时OD600值仅为89.21(如图4-a)。由于菌株生产MK-7需要足够的关键酶进行催化,而关键酶的合成多集中在添加诱导剂后的8 h诱导阶段,此时也是菌体的快速生长期。因此,为保证在诱导前有足够多的菌体量,先提升E2K-15在罐上的生长速度,对菌株E2K-15的二级种子液进行生长曲线的测定(如图4-b),确定二级种子液在生长7 h后进行发酵罐转种,且在OD600值达到75时添加诱导剂,结果表明MK-7产量在62 h可达741.57 mg/L,OD600值最高达109(如图4-c)。
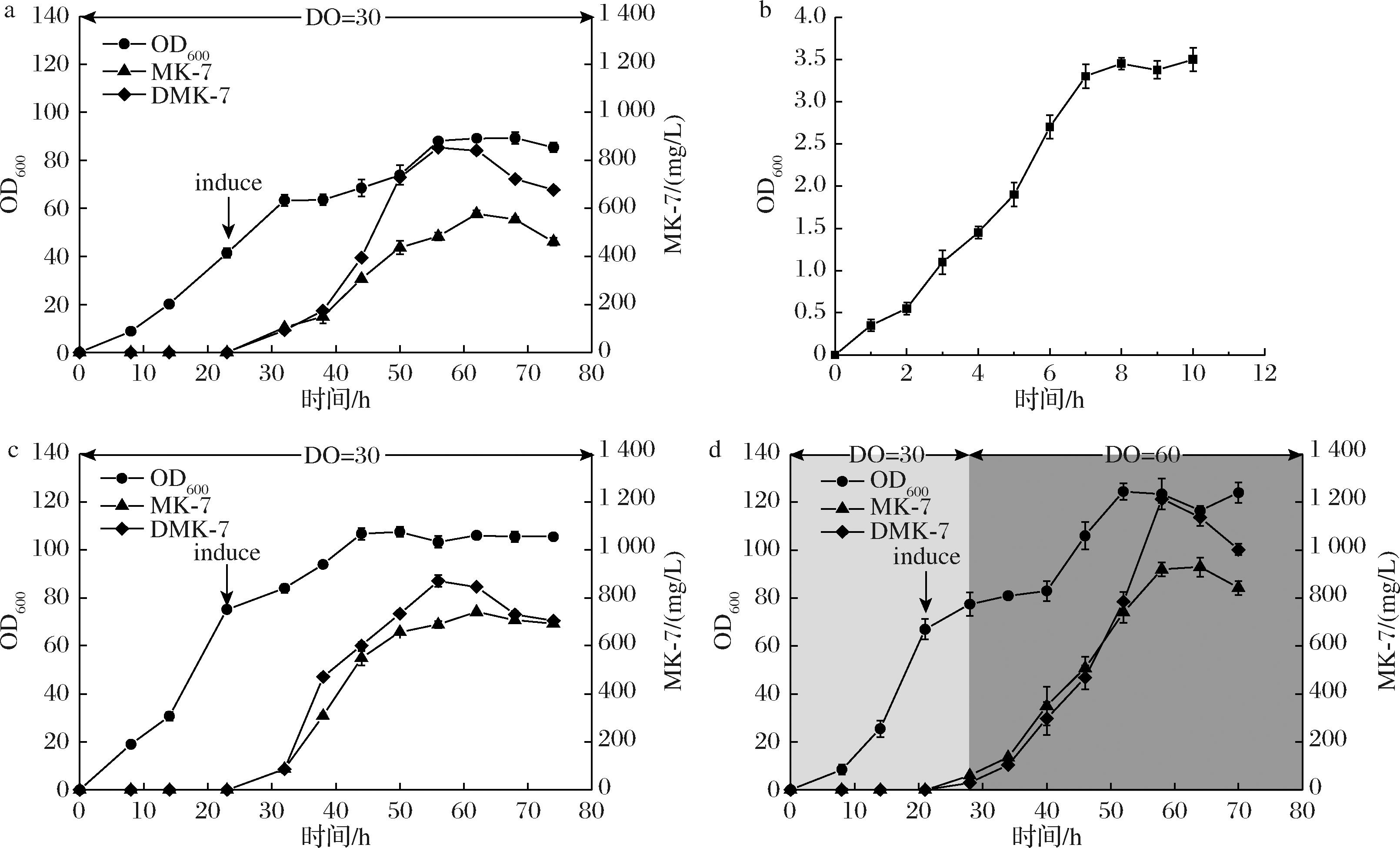
a-菌株E2K-15的分批补料发酵情况;b-菌株E2K-15的生长曲线;c-延长诱导时间对MK-7生产的影响;d-提升溶氧量对MK-7生产的影响
图4 对5 L发酵罐上的MK-7合成进行发酵条件优化
Fig.4 The fermentation conditions of MK-7 synthesis in 5 L fermentor were optimized
甲萘醌和泛醌作为电子传递链组分同时存在于大肠杆菌内,其所占比例也受到含氧量的影响,而溶氧值也是发酵放大过程的一个重要控制参数,因此控制发酵过程中溶氧量高低往往可以影响最终的发酵结果[2, 24]。研究人员对枯草芽孢杆菌和纳豆芽孢杆菌的放大实验的溶氧量进行优化,发现较高的溶氧量有利于MK-7的合成[25-26]。因此,在诱导时间结束后将DO值提高至60后,结果表明在64 h时MK-7达到929.58 mg/L,但同时其直接前体DMK-7的积累量也提高至1 217.58 mg/L。
2.4 降低DMK-7堆积提升MK-7合成
前期5 L发酵上结果显示,菌株E2K-15在合成MK-7的同时会存在大量的DMK-7堆积,表明MK-7的产量存在进一步的提升空间,且过多的DMK-7和MK-7混在一起会增加后期MK-7分离纯化的难度。作为MK-7的直接前体,从DMK-7到MK-7仅需要经过一步由S-腺苷甲硫氨酸(S-adenosyl methionine,SAM)依赖型酶MenG催化的甲基化反应,对此,强化SAM的供应通常可以有效提升此类反应的反应效率[27]。WEI等[28]通过引入酿酒酵母来源Sam2提升大肠杆菌内SAM供应用以促进菌体生产生物素。
本研究对比了不同来源的MenG用于催化DMK-7转化为MK-7,发现由B. subtilis 168来源的MenG(NCBI登录号:NP_390156.1)构建的菌株E2K-24合成的MK-7最高,摇瓶产量可达127.02 mg/L(图5-b)。随后,为了进一步提升SAM的供应以促进MenG的催化,引入ParaBAD-Scsam2连接在质粒pSB1a-BthepS-BthepS-MnmenA-Bs168 menG之后转化构成菌株E2K-26,摇瓶发酵后MK-7产量可达134.59 mg/L。按照2.3节优化后的发酵方式,将E2K-24和E2K-26分别在5 L发酵罐上进行补料分批发酵,结果显示,发酵60 h时,菌株E2K-24的OD600值为105,MK-7最高产量可达1 090.45 mg/L,DMK-7的积累量降低至231.14 mg/L(图5-d)。菌株E2K-26的OD600值与菌株E2K-24相当,为105,但发酵产生的MK-7却并不高,仅有544.65 mg/L,DMK-7的积累量为488.71 mg/L(图5-e)。这或许是由于SAM涉及到大肠杆菌中较多的SAM依赖型酶促反应,过表达后同时影响了其他途径的代谢。
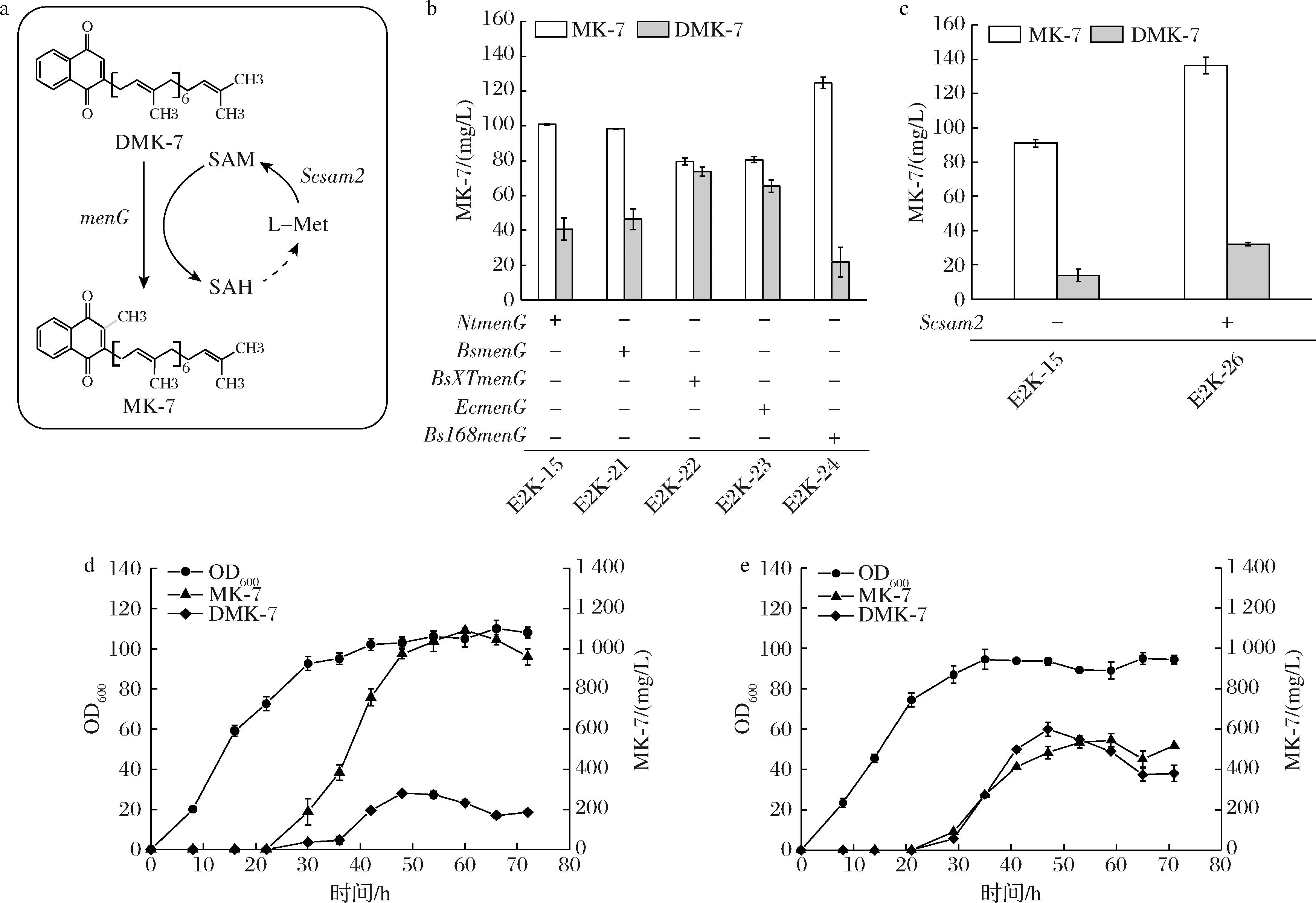
a-DMK-7转化为MK-7的途径示意图;b-不同来源的MenG对合成MK-7的影响;c-过表达ScSam2后对MK-7合成的影响;d-菌株E2K-24发酵结果;e-菌株E2K-26发酵结果
图5 降低DMK-7堆积提升MK-7产量
Fig.5 Reducing the accumulation of DMK-7 increased MK-7 production
3 结论与讨论
甲萘醌-7作为维生素K的重要亚型,由于它在医药保健等领域的应用价值和在人体内的更长半衰期,使得微生物发酵高效合成MK-7具有更重要的现实意义。尽管纳豆芽孢杆菌[29]和枯草芽孢杆菌[30]作为常见MK-7生产菌株,然而由于其存在发酵周期过长,以及放大发酵时产量较低等问题,在大规模发酵生产中优势有限。GAO等[12]在大肠杆菌中引入枯草芽孢杆菌来源的HepPPS,MenA和UbiE,并进行膜工程改造,最终在摇瓶水平上可达129 mg/L。
本研究首先利用DLKcat挖掘筛选出合成MK-7的关键酶BtHepS、BtHepS、MnMenA和NtMenG,采用低拷贝质粒和RBS对其进行表达,实现了大肠杆菌内合成MK-7。其次,对合成MK-7的前体途径强化以及竞争途径和消耗途径的关键酶进行敲除,在过表达EcIdi后MK-7的产量得到了进一步提高,而对ispB、ubiCA、wrbA和qorB进行敲除时,却并未起到效果,在敲除ubiCA后,MK-7产量反而下降。猜测可能是由于ubiCA作为合成MK-7的竞争产物辅酶Q的关键基因,尽管参与了MK-7前体的竞争,然而在有氧条件下大肠杆菌主要以辅酶Q参与电子传递,菌体必须保留合成辅酶Q的途径。WrbA和QorB尽管会消耗部分MK-7,但是同样是由于在有氧条件下大肠杆菌主要以辅酶Q参与电子传递,MK-7参与电子传递的占比较少,因此并未起到提升MK-7产量的作用。随后,将改造后的菌株在5 L罐上发酵进行发酵条件优化,在延迟诱导时间至OD600值达到75,且提升溶氧量至60,MK-7的产量达到929.58 mg/L,但同时存在大量DMK-7的堆积。通过引入枯草芽孢杆菌的MenG后,MK-7的产量提高至1 090.45 mg/L,DMK-7降低至231.14 mg/L。在本研究中,利用DLKcat程序模拟筛选关键酶时,将其进行了组合表达,并未对底物通量的改变情况进行细化研究,考虑到不同来源的关键酶对MK-7的合成影响较大,后续研究可以对由酶的改变导致的底物通量的变化进一步进行测定,从而更具针对性的进行改造。由于直接敲除ubiCA会对菌体的呼吸链造成影响,后续研究可以尝试弱化其表达量,以达到削弱竞争途径的目的。另外,在引入ScSam2后摇瓶水平MK-7产量有所提高,但在发酵罐水平并未达到预期效果,后续可以改造优化最后一步SAM依赖型甲基化反应。总之,本研究对代谢改造大肠杆菌发酵生产MK-7的具有一定的指导意义。
[1] HIROTA Y, TSUGAWA N, NAKAGAWA K, et al.Menadione (vitamin K3) is a catabolic product of oral phylloquinone (vitamin K1) in the intestine and a circulating precursor of tissue menaquinone-4 (vitamin K2) in rats[J].Journal of Biological Chemistry, 2013, 288(46):33071-33080.
[2] BRAASCH-TURI M M, KOEHN J T, CRANS D C.Chemistry of lipoquinones:Properties, synthesis, and membrane location of ubiquinones, plastoquinones, and menaquinones[J].International Journal of Molecular Sciences, 2022, 23(21):12856.
[3] PALMER C R, BLEKKENHORST L C, LEWIS J R, et al.Quantifying dietary vitamin K and its link to cardiovascular health:A narrative review[J].Food &Function, 2020, 11(4):2826-2837.
[4] 董润锜. 维生素K2的生物学效应及临床意义的研究进展[J].河南医学研究, 2021, 30(18):3451-3454.
DONG R Q.Research progress on the biological effects and clinical significance of vitamin K2[J].Henan Medical Research, 2021, 30(18):3451-3454.
[5] KOZIO -KOZAKOWSKA A, MARESZ K.The impact of vitamin K2 (menaquionones) in children’s health and diseases:A review of the literature[J].Children, 2022, 9(1):78.
-KOZAKOWSKA A, MARESZ K.The impact of vitamin K2 (menaquionones) in children’s health and diseases:A review of the literature[J].Children, 2022, 9(1):78.
[6] 刘琳, 吕宁, 林超, 等.乳及乳制品中维生素K2的发展现状及展望[J].中国乳业, 2023(6):79-84.
LIU L, LYU N, LIN C, et al.The development status and prospect of vitamin K2 in milk and dairy products[J].China Dairy, 2023(6):79-84.
[7] KNAPEN M H J, BRAAM L A J L M, TEUNISSEN K J, et al.Yogurt drink fortified with menaquinone-7 improves vitamin K status in a healthy population[J].Journal of Nutritional Science, 2015, 4:e35.
[8] 李保龙, 向雪松, 祁邦国, 等.中国传统发酵豆制品维生素K2含量分布[J].卫生研究, 2023, 52(3):440-444.
LI B L, XIANG X S, QI B G, et al.Distribution of vitamin K2 content in Chinese traditional fermented soybean products[J].Journal of Hygiene Research, 2023, 52(3):440-444.
[9] KANG M J, BAEK K R, LEE Y R, et al.Production of vitamin K by wild-type and engineered microorganisms[J].Microorganisms, 2022, 10(3):554.
[10] WANG H, SUN X J, WANG L, et al.Coproduction of menaquinone-7 and nattokinase by Bacillus subtilis using soybean curd residue as a renewable substrate combined with a dissolved oxygen control strategy[J].Annals of Microbiology, 2018, 68(10):655-665.
[11] CUI S X, LYU X Q, WU Y K, et al.Engineering a bifunctional Phr60-Rap60-Spo0A quorum-sensing molecular switch for dynamic fine-tuning of menaquinone-7 synthesis in Bacillus subtilis[J].ACS Synthetic Biology, 2019, 8(8):1826-1837.
[12] GAO Q X, CHEN H, WANG G Y, et al.Highly efficient production of menaquinone-7 from glucose by metabolically engineered Escherichia coli[J].ACS Synthetic Biology, 2021, 10(4):756-765.
[13] XU X H, LYU X Q, CUI S X, et al.Remodeling isoprene pyrophosphate metabolism for promoting terpenoids bioproduction[J].Engineering, 2023, 28:166-178.
[14] GAO Q X, CHEN H, WANG W Z, et al.Menaquinone-7 production in engineered Escherichia coli[J].World Journal of Microbiology &Biotechnology, 2020, 36(9):132.
[15] LI F R, YUAN L, LU H Z, et al.Deep learning-based kcat prediction enables improved enzyme-constrained model reconstruction[J].Nature Catalysis, 2022, 5:662-672.
[16] WANG D A, CHEN J M, WANG Y, et al.Engineering Escherichia coli for high-yield production of ectoine[J].Green Chemical Engineering, 2023, 4(2):217-223.
[17] DING X M, ZHENG Z M, ZHAO G H, et al.Bottom-up synthetic biology approach for improving the efficiency of menaquinone-7 synthesis in Bacillus subtilis[J].Microbial Cell Factories, 2022, 21(1):101.
[18] 董雅君, 崔世修, 刘延峰, 等.功能膜微域在七烯甲萘醌合成过程中的作用解析[J].生物工程学报, 2023, 39(6):2215-2230.
DONG Y J, CUI S X, LIU Y F, et al.Functional analysis of functional membrane microdomains in the biosynthesis of menaquinone-7[J].Chinese Journal of Biotechnology, 2023, 39(6):2215-2230.
[19] 冯凡, 许杨, 陶勇, 等.提高大肠杆菌通过MVA途径合成异戊二烯[J].生物工程学报, 2015, 31(7):1073-1081.
FENG F, XU Y, TAO Y, et al.Improving isoprene production by engineered heterologous mevalonate pathway in Escherichia coli[J].Chinese Journal of Biotechnology, 2015, 31(7):1073-1081.
[20] KONG M K, LEE P C.Metabolic engineering of menaquinone-8 pathway of Escherichia coli as a microbial platform for vitamin K production[J].Biotechnology and Bioengineering, 2011, 108(8):1997-2002.
[21] BURGARDT A, PELOSI L, CHEHADE M H, et al.Rational engineering of non-ubiquinone containing Corynebacterium glutamicum for enhanced coenzyme Q10 production[J].Metabolites, 2022, 12(5):428.
[22] ROSSI F, CATT C, MUGNAI G, et al.Effects of the quinone oxidoreductase WrbA on Escherichia coli biofilm formation and oxidative stress[J].Antioxidants, 2021, 10(6):919.
C, MUGNAI G, et al.Effects of the quinone oxidoreductase WrbA on Escherichia coli biofilm formation and oxidative stress[J].Antioxidants, 2021, 10(6):919.
[23] KIM I K, YIM H S, KIM M K, et al.Crystal structure of a new type of NADPH-dependent quinone oxidoreductase (QOR2) from Escherichia coli[J].Journal of Molecular Biology, 2008, 379(2):372-384.
[24] LIU Y, YANG Z M, XUE Z L, et al.Influence of site-directed mutagenesis of UbiA, overexpression of dxr, menA and ubiE, and supplementation with precursors on menaquinone production in Elizabethkingia meningoseptica[J].Process Biochemistry, 2018, 68:64-72.
[25] 李楚, 王晗, 王丽, 等.纳豆芽孢杆菌men基因过表达菌株的构建及提高溶氧强化MK-7合成[J].应用与环境生物学报, 2022, 28(6):1393-1399.
LI C, WANG H, WANG L, et al.Enhancement of MK-7 synthesis by engineered strains of Bacillus subtilis natto that overexpress men genes and by increasing available dissolved oxygen[J].Chinese Journal of Applied and Environmental Biology, 2022, 28(6):1393-1399.
[26] 殷峻杰, 刘龙, 李江华, 等.基于CFD模拟的七烯甲萘醌发酵过程优化[J].食品与生物技术学报, 2023, 42(5):78-87.
YIN J J, LIU L, LI J H, et al.Optimization of menaquinone-7 fermentation process based on CFD simulation[J].Journal of Food Science and Biotechnology, 2023, 42(5):78-87.
[27] YU P, ZHU P Z.Improving the production of S-adenosyl-L-methionine in Escherichia coli by overexpressing metk[J].Preparative Biochemistry &Biotechnology, 2017, 47(9):867-873.
[28] WEI P P, ZHU F C, CHEN C W, et al.Engineering a heterologous synthetic pathway in Escherichia coli for efficient production of biotin[J].Biotechnology Letters, 2021, 43(6):1221-1228.
[29] WANG Y D, ZHANG Z Y, ZHOU Y M, et al.High synthetic mechanisms and concentration improvement of menaquinone-7 from Bacillus subtilis subsp.natto[J].Biocatalysis and Agricultural Biotechnology, 2023, 54:102957.
[30] CUI S X, XIA H Z, CHEN T C, et al.Cell membrane and electron transfer engineering for improved synthesis of menaquinone-7 in Bacillus subtilis[J].iScience, 2020, 23(3):100918.