四川泡菜是一种流行于西南地区的传统发酵食品,传统四川泡菜风味的形成依赖于以乳酸菌为主导的自然发酵。其发酵过程中,微生物具有明显的演替特征。对于新制泡菜发酵体系,明串珠菌属(Leuconostoc)和魏斯氏菌属(Weissella)微生物由于繁殖快,微需氧,是乳酸发酵的启始微生物,随着酸度升高,植物乳植物杆菌(Lactiplantibacillus plantarum)和短促生乳杆菌(Levilactobacillus brevis)等耐酸性强的乳酸菌逐渐占据主导并维持稳态[1-2]。
亚硝酸盐广泛存在于各种盐渍蔬菜中[3-4],一直被认为是发酵蔬菜中首要的安全风险因子,但通过乳酸菌接种和泡菜母水发酵能够显著抑制其生成[4-5]。传统泡菜和工业化发酵的泡菜由于清洁程度较好且使用“老母水”或发酵剂进行发酵,亚硝酸盐危害风险一般较低。但对家庭和作坊生产的新制泡菜而言,发酵早期仍可能因污染产生过量的亚硝酸盐。
目前研究普遍认为,盐渍蔬菜发酵早期,体系含有一定氧气,pH值较高,微生物在短时间内将蔬菜原料中的硝酸盐还原成亚硝酸盐,从而在发酵起始后一段时间内形成“亚硝峰”。随着发酵的进行,亚硝酸盐在微生物和酸性环境的作用下被清除,含量逐渐下降。近年来已有部分研究试图揭示腌渍蔬菜中亚硝酸盐的代谢机制。例如,YAO等[6]从泡菜盐渍废液中筛选获得一株蜡状芽孢杆菌(Bacillus cereus),并发现其能通过反硝化作用将硝基氮还原为亚硝基氮。WEI等[3]通过研究高盐萝卜咸菜中微生物基因推测,咸菜内的嗜盐古菌能够通过代谢葡萄糖获得电子,并通过EMP和TCA循环将电子转移给截断反硝化过程,使亚硝酸盐还原成N2。SONG等[7]研究了酸菜发酵中与亚硝酸盐代谢相关的基因,推测含有编码硝基氮还原酶和亚硝基氮还原酶基因的假单胞菌(Pseudomonas)是亚硝酸盐生成和消除的主要微生物。然而,对于四川泡菜中亚硝酸盐生成和转化的研究仍十分缺乏,行业内对泡菜发酵中亚硝酸盐的生成和代谢情况仍普遍缺少进一步认识。
本研究使用常见原料白萝卜,按照传统工艺制作了四川泡菜,并对其发酵早期阶段亚硝酸盐含量变化、微生物群落结构以及与亚硝酸盐生成与转化相关的基因进行了探索和分析,旨在初步探究泡菜发酵早期亚硝酸盐含量与微生物演替的关系,为四川泡菜亚硝酸盐的控制和安全生产提供理论依据。
1 材料与方法
1.1 材料与试剂
白萝卜(Raphanus sativus L.longipinnatus Bailey)、食用盐、500 mL玻璃泡菜坛,采购于当地超市。硼砂、乙酸锌、亚硝酸钠、对氨基苯磺酸、盐酸萘乙二胺(均为分析纯),重庆川东化工集团有限公司;FastDNA SPIN Kit for Soil土壤DNA提取试剂盒,MP生物医药公司。
1.2 仪器与设备
BG-160电热恒温培养箱,上海博讯实业有限公司;H2050R-1离心机,长沙湘仪离心机仪器有限公司;YP20002电子天平,上海越平科学仪器制造有限公司;752G紫外可见分光光度计,上海仪电分析仪器股份有限公司。
1.3 实验方法
1.3.1 传统四川泡菜的制作
将白萝卜去皮后与质量分数3%的食盐水按照质量比1∶1.5混合,装入500 mL玻璃泡菜坛,30 ℃恒温密封发酵,每天取出一坛用于测定分析。
1.3.2 泡菜浸渍液总酸和pH值的测定
泡菜浸渍液中总酸的测定参考GB 12456—2021《食品安全国家标准 食品中总酸的测定》第二法进行,将泡菜充分搅拌均匀,取泡菜浸渍液,适当稀释后用0.02 mol/L NaOH标准溶液滴定至pH值达到8.2,根据所消耗的NaOH标准溶液的量计算浸渍液中总酸的含量,结果用100 g样品中含有的乳酸当量(g/100 g)来表示。泡菜浸渍液的pH值使用pH计进行测定。
1.3.3 泡菜浸渍液中亚硝酸盐含量的测定
取100 mL泡菜浸渍液,参考GB 5009.33—2016《食品安全国家标准 食品中亚硝酸盐与硝酸盐的测定》第二法,对亚硝酸盐含量进行测定。取适量泡菜浸渍液,适当稀释后精确称取5 g于250 mL具塞锥形瓶中,加12.5 mL 50 g/L饱和硼砂溶液和约150 mL 70 ℃左右的水,混匀后沸水浴加热15 min,冷水浴冷却至室温。定量转移上述提取液至200 mL容量瓶中,加入5 mL 106 g/L亚铁氰化钾溶液,摇匀,再加入5 mL 220 g/L乙酸锌溶液,加水至刻度,摇匀,放置30 min,取滤液40 mL于50 mL具塞比色管,加入2 mL 4 g/L对氨基苯磺酸溶液,混匀,静置5 min后各加入1 mL 2 g/L盐酸萘乙二胺溶液,加水至刻度,混匀,静置15 min,于波长538 nm处测吸光度,并根据亚硝酸盐梯度溶液制备的标准曲线计算样品中亚硝酸盐的含量(以亚硝酸钠计,mg/kg)。
1.3.4 宏基因组分析
取300 mL泡菜浸渍液,5 000 r/min离心10 min,取沉淀-80 ℃备用,送至上海美吉生物医药科技有限公司,提取样本中的全部DNA,利用Illumina MiSeq二代测序平台构建基因文库,并对文库中的序列进行测序。使用fastp去除原始测序数据中adapter序列、低质量测序片段和宿主测序片段,在美吉工作平台上对剩余的测序片段进行分析:通过NR202209数据库进行物种注释,使用物种对应的基因的读序数占样品总读序数的百分比表示该物种的相对丰度。
利用KEGG Orthology数据库对测序片段进行注释,获得测序片段的同源基因(KEGG Orthology,KO)信息。选取与氮素代谢相关的通路单元(pathway module):M00175(固氮作用)、M00531(同化硝酸盐还原)、M00530(异化硝酸盐还原)、M00529(反硝化作用)、M00528(硝化作用)、M00804(完全硝化作用,完全氨氧化),对测序片段中的KO信息进行筛选,各样品中KO的相对丰度用其reads数来表示。
1.4 数据处理与分析
采用Spearman相关性分析对与亚硝酸盐代谢相关基因、亚硝酸盐含量等指标与微生物丰度的相关性进行分析,并使用美吉云平台对数据进行可视化处理。
2 结果与分析
2.1 白萝卜泡菜发酵早期总酸和pH值的变化
白萝卜泡菜发酵前5 d pH值和总酸的变化情况如图1所示,泡菜发酵起始时,浸渍液的pH和总酸分别为(5.37±0.10)和(0.03±0.00) g/100 g,随后pH值迅速下降,发酵1 d后达到3.72±0.08,总酸含量稳步上升。发酵1 d后泡菜浸渍液pH下降趋于缓慢,发酵5 d后pH值下降到3.28±0.05,与之对应,泡菜浸渍液的总酸含量不断升高,发酵5 d后到达(0.74±0.04) g/100 g。
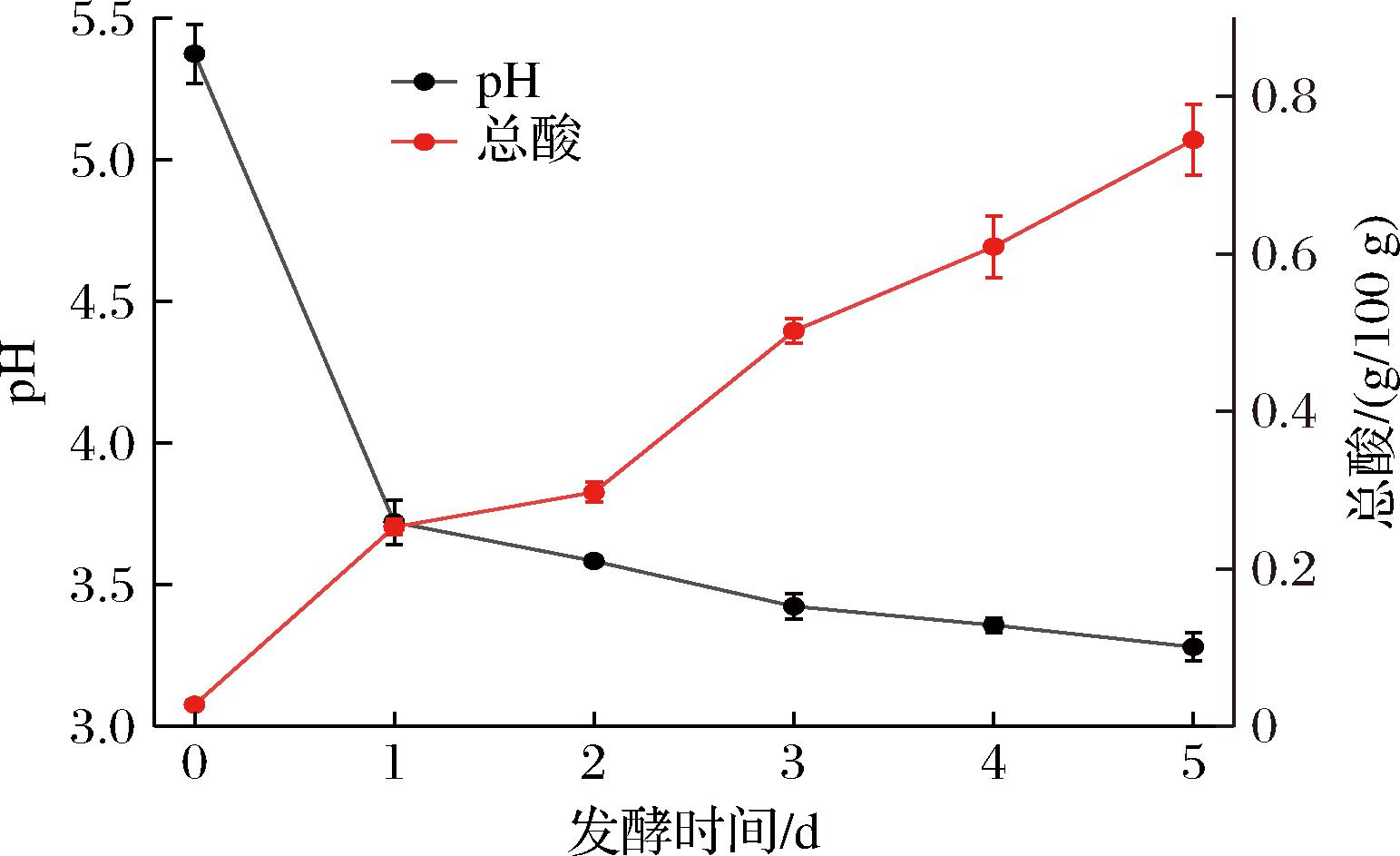
图1 泡菜发酵过程中pH和总酸的变化
Fig.1 Changes of pH value and total acidity during fermentation of paocai
2.2 白萝卜泡菜发酵早期亚硝酸盐含量的变化
由图2可知,在泡菜发酵过程中,亚硝酸盐含量呈现先增后减的趋势。发酵初始浸渍液中的亚硝酸盐含量为(1.16±0.00) mg/L,发酵起始后第1天浸渍液中的亚硝酸盐含量迅速上升,达到(20.68±0.43) mg/L,略微超出我国国标GB 2762—2022《食品安全国家标准 食品中污染物限量》对酱腌菜亚硝酸盐含量的限制,随着发酵的继续,泡菜中的亚硝酸盐含量迅速下降并逐渐趋于稳定,发酵3 d后浸渍液的亚硝酸盐含量已降至1 mg/L以下。CHEN等[2]认为泡菜总酸含量超过0.5 g/100 g时即可视为发酵成熟,本研究中的白萝卜泡菜,发酵3 d时总酸达到(0.50±0.02) g/100 g,同时亚硝酸盐含量迅速降低到(0.44±0.10) mg/kg,说明泡菜在发酵第3天已经成熟,可供食用。
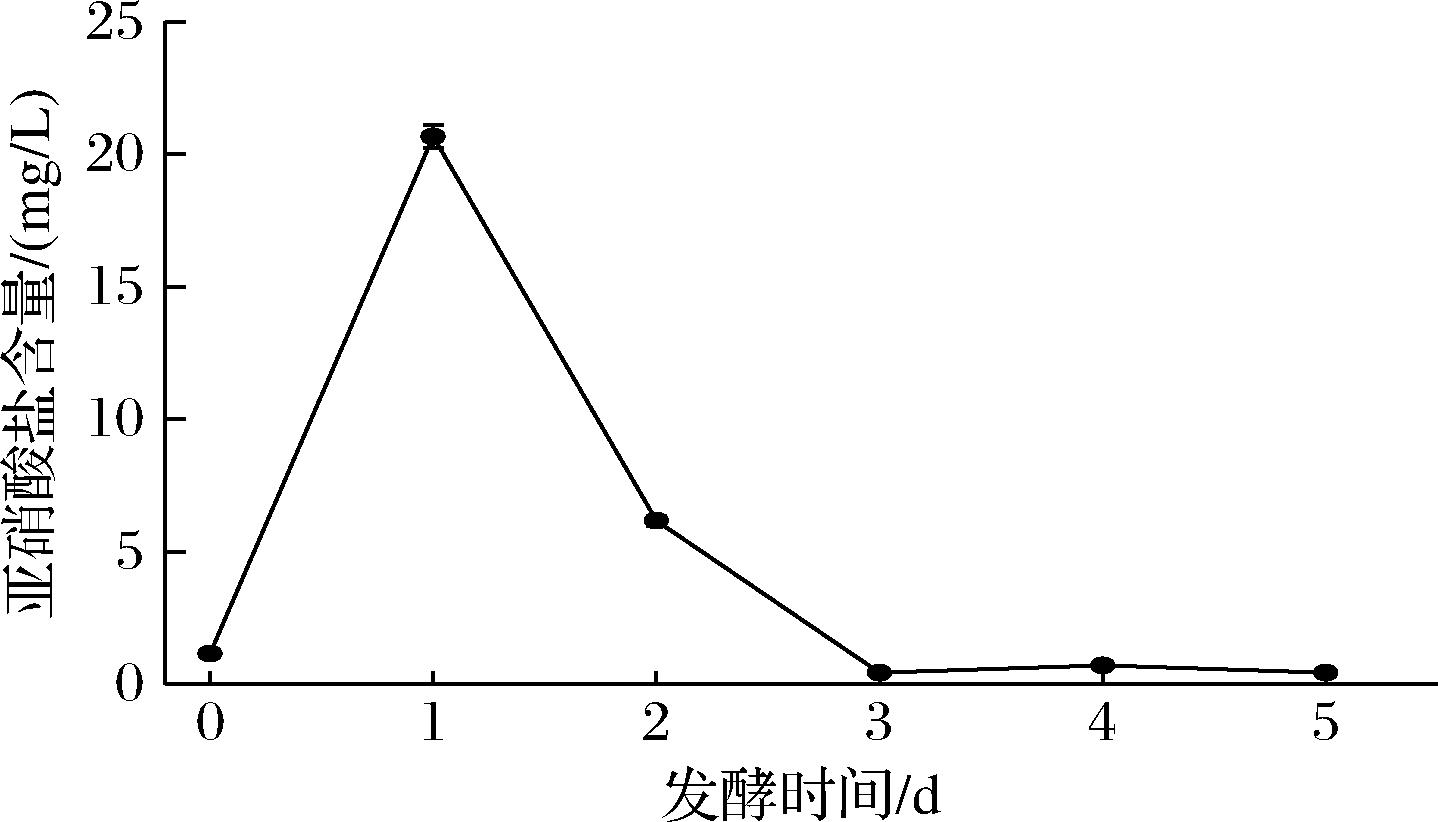
图2 泡菜发酵过程中亚硝酸盐的含量
Fig.2 Nitrite content of paocai during fermentation
2.3 白萝卜泡菜发酵早期微生物多样性的变化
由图3可知,随着泡菜发酵的进行,体系中微生物构成不断变化。总体来看,发酵起始时样本shannon指数为4.70,而发酵5 d的shannon指数仅有2.56(数据未展示),说明样本内的物种丰富度不断下降。发酵起始时,假单胞菌属(Pseudomonas,30.30%)微生物相对丰度最高,黄单胞菌属(Xanthomonas,7.97%)、泛菌属(Pantoea,7.20%)、植物杆菌属(Plantibacter,6.20%)、寡养单胞菌属(Stenotrophomonas,6.26%)和来略特菌属(Lelliottia,4.08%)也占有一定比例,这些微生物属在土壤中和果蔬表面均有报道[8-12],也是泡菜发酵起始阶段的常见微生物[1,13],这说明发酵起始时体系中的微生物主要来源于蔬菜原料。
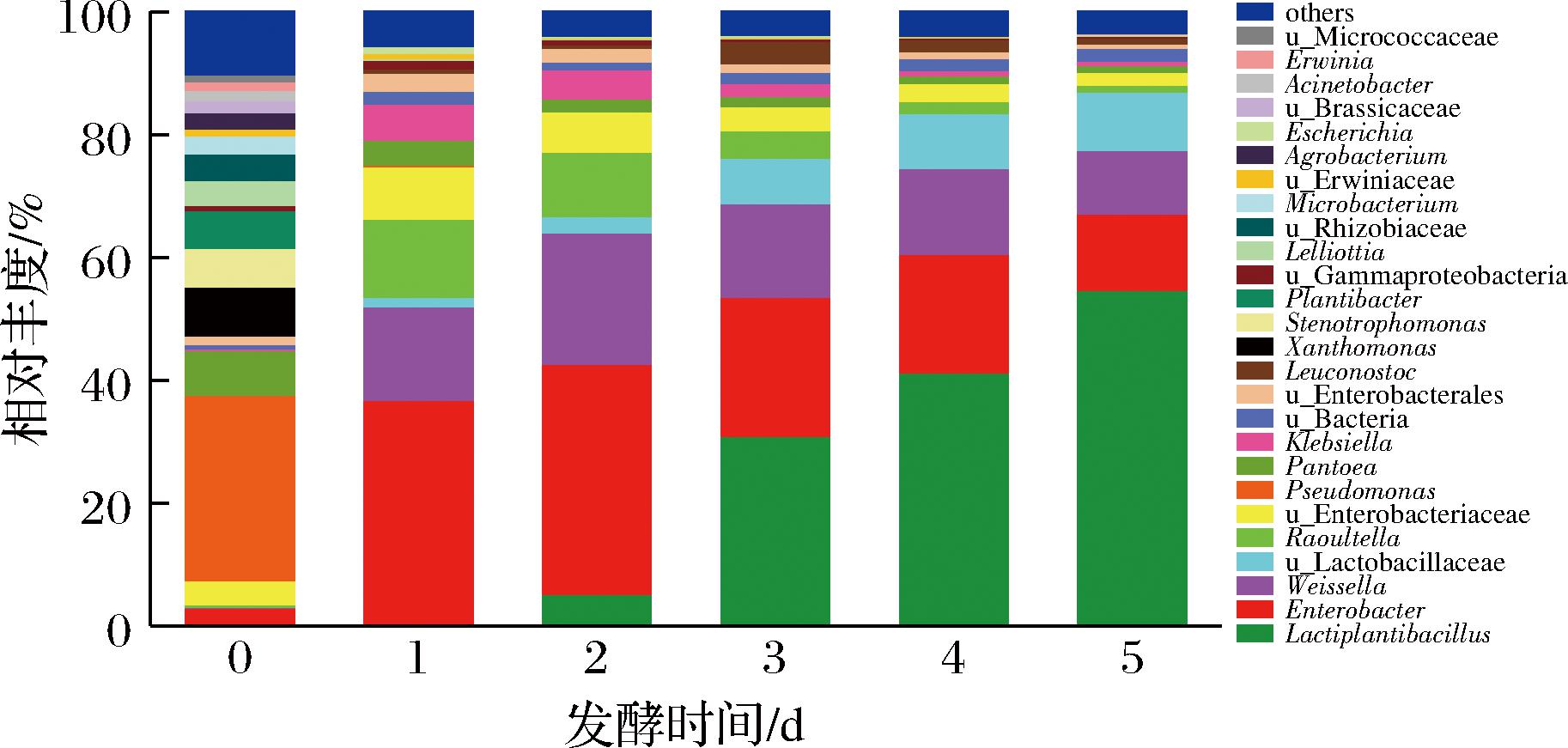
图3 泡菜发酵前5 d的微生物属水平分布
Fig.3 Microbial distribution of paocai during the first five days of fermentation at genus level
注:“u”代表该属在所示分类水平下未知。
经过1 d的密封发酵,泡菜浸渍液中的微生物构成迅速改变,肠杆菌属(Enterobacter)成为体系中的优势微生物,其相对丰度(36.80%)较发酵起始时显著增加。此外,魏斯氏菌属(Weissella,15.23%)、拉乌尔菌属(Raoultella,12.67%)、克雷伯菌属(Klebsiella,5.9%)和泛菌属(Pantoea,4.06%)在发酵1 d时也占有较大的比例。这说明发酵初始时许多微生物无法适应泡菜的发酵环境,已开始消亡,而肠杆菌属、魏斯氏菌属等微生物可在泡菜发酵起始阶段大量增殖。随着发酵的进行,肠杆菌属(Enterobacter)和魏斯氏菌属(Weissella)微生物相对丰度在发酵第2天达到最大(分别为37.44%和21.44%),随后逐渐下降,发酵5 d相对丰度下降到12.46%和10.31%,拉乌尔菌属(Raoultella)、克雷伯菌属(Klebsiella)和泛菌属(Pantoea)微生物在发酵2 d后相对丰度持续下降,而乳植物杆菌属(Lactiplantibacillus)微生物迅速增加并成为主要的微生物,由发酵2 d的5.30%增加到5 d时的54.79%。
目前对发酵蔬菜的研究均表明,泡菜发酵早期的微生物构成差异较大,存在一定的偶然性,而乳植物杆菌属微生物是泡菜发酵后期的主导微生物[1,13],这与本研究的结果相近。泡菜发酵前5 d的微生物群落结构存在明显的演替特征,说明体系内乳酸菌的产酸和氧气、底物的消耗对微生物群落起到了定向筛选的作用。
2.4 亚硝酸盐代谢相关基因的分析
泡菜中亚硝酸盐的生成转化是自然界氮素循环在食品发酵体系中的具体表现,通过研究发酵过程中氮代谢的具体过程,有利于阐明泡菜亚硝酸盐的代谢机制。本研究使用KEGG Orthology数据库对泡菜样本的基因集进行注释,选取与氮代谢相关的基因,其种类、催化反应和相对丰度如图4所示。
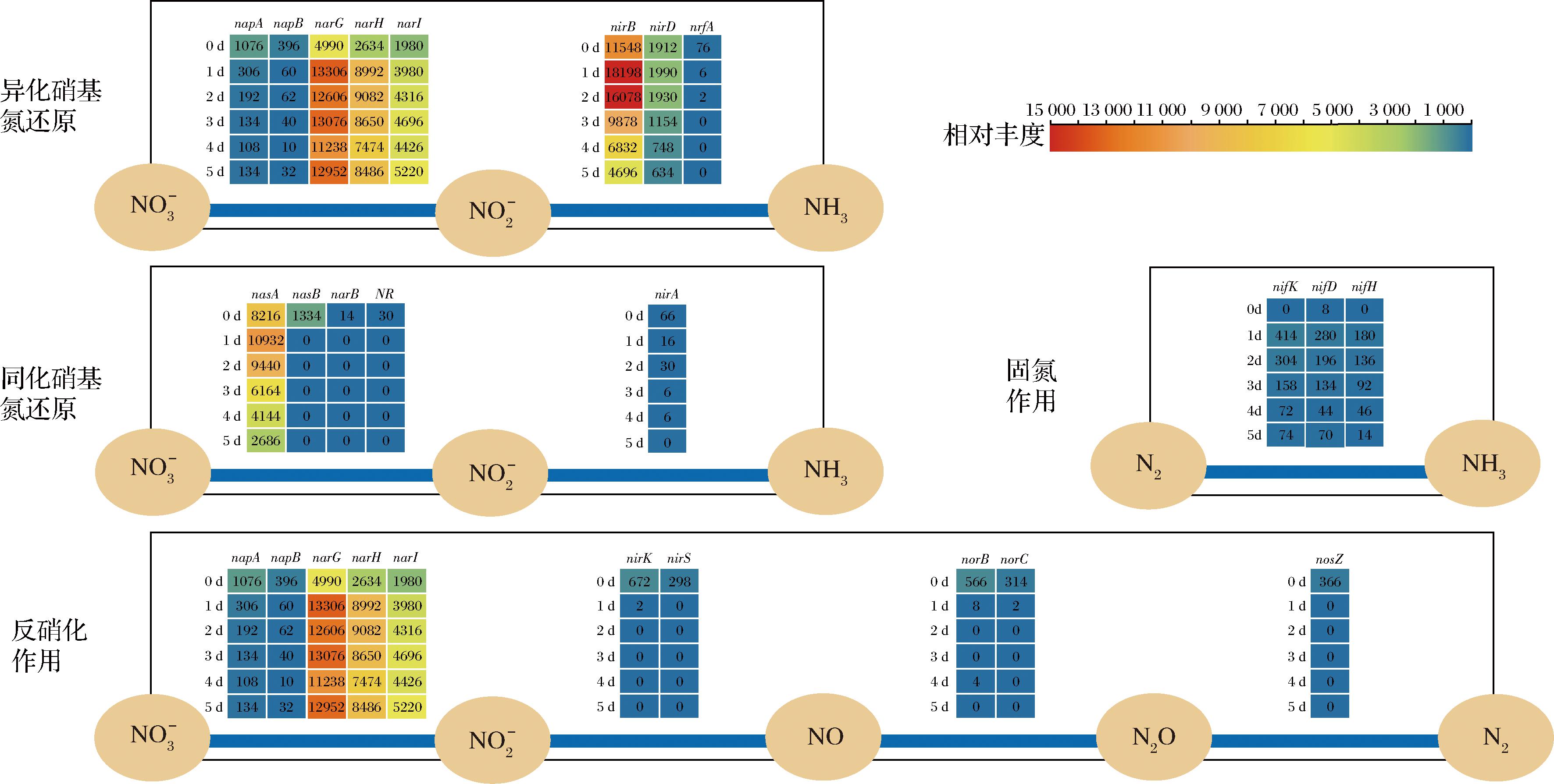
图4 泡菜发酵早期亚硝酸盐代谢相关酶基因及丰度
Fig.4 Genes abundance of enzymes involving in nitrogen metabolism during early stage of paocai fermentation
注:每个热图自左到右分别表示发酵0~5 d样品中的相对丰度。
微生物的固氮作用涉及到nifK、nifD、nifH这3个基因,它们分别编码固氮酶(1.18.6.1)中具有催化功能的钼离子蛋白β链、钼离子蛋白α链和具有电子传递功能的铁蛋白[14]。3个基因在发酵起始丰度很低,但1 d时显著增加,随后逐渐下降。说明泡菜发酵早期存在固氮微生物。但由于固氮作用严格厌氧,推测发酵体系内发生固氮反应的可能性不大。
同化硝基氮还原酶催化硝酸盐向亚硝酸盐还原的过程,主要用于自身含氮大分子物质的合成[15]。基因nasA、nasB负责编码同化硝基氮还原酶(1.7.1.1,NAS)多聚体上的不同亚基[16-17],narB负责编码同化亚硝基氮还原酶(1.7.7.2,NAS)[16],这些酶能够将硝酸盐还原为亚硝酸盐用于微生物的合成代谢[18]。泡菜发酵中,nasA丰度在发酵1 d到达峰值,随后逐渐下降,但总体而言丰度较高,同化硝基氮还原途径中其他酶基因丰度则比较低。
异化硝基氮还原酶将硝酸盐还原为亚硝酸盐,主要用于微生物的能量代谢、解毒和氧化还原调控等异化作用[15]。异化硝基氮还原酶(1.7.5.1,NaR),能够耦合硝基氮呼吸过程,将质子跨越细胞膜,形成电化学梯度,并由此驱动ATP的合成[16,18]。基因narG、narH和narI分别编码NaR异源三聚体的3个亚基[16]。这3个基因在白萝卜泡菜发酵早期始终维持着较高的丰度,这可能与肠杆菌属(Enterobacter)和克雷伯菌属(Klebsiella)等杂菌在泡菜发酵早期持续存在有关。
此外,泡菜发酵早期体系内也存在着少量的napA、napB,该基因编码周质硝基氮还原酶(1.9.6.1,NAP)[16]。可以推测,NAS催化的同化硝基氮还原和NaR催化的异化硝基氮还原大量存在于泡菜发酵早期,是亚硝酸盐生成的主要代谢途径,NAP在亚硝酸盐的生成中也可能起到了一定的作用。基因nirB和nirD分别编码位于亚硝基氮还原酶(1.7.1.15,NiR)上的不同亚基[19],该酶可将亚硝酸盐直接还原为胺[20],它们存在于肠杆菌属(Enterobacter)、克雷伯菌属(Klebsiella)[21]中,在以往研究中,NiR也被认为和东北酸菜中亚硝酸盐的消除直接相关[7]。泡菜发酵早期,这2种基因的丰度均呈现先略微增加后逐渐下降的趋势,可能与这些微生物的大量增殖和消亡有关。
其他编码氮代谢酶的基因,如nirK和nirS(编码亚硝基氮还原酶,催化亚硝酸盐还原为NO)、nrfA(编码亚硝基氮还原酶,催化亚硝酸盐还原为胺)[22]、nor(编码一氧化氮还原酶,催化NO还原为N2O)、nos(编码氧化亚氮还原酶,催化N2O还原为N2),这些基因仅在泡菜发酵初始丰度较高,发酵起始后便消失。说明含有该基因的微生物在泡菜发酵起始便已经消亡,这些反应途径可能不存在于泡菜发酵中。总体来看,泡菜中的亚硝酸盐可能通过NAS、NaR、NAP等酶的催化,由硝酸盐还原生成;并由NiR催化,进一步被还原为胺。
2.5 亚硝酸盐代谢相关基因与微生物丰度的关系
将泡菜发酵中主要理化指标、硝酸盐代谢相关基因的丰度与微生物丰度进行相关性分析,结果如图5所示。与WANG等[23]的研究结果类似,主导发酵的乳植物杆菌属与发酵体系总酸呈现显著的正相关,而与pH值呈现负相关,这与发酵体系中的各种杂菌形成了明显的差异。这说明乳植物杆菌属是泡菜发酵体系产酸的主要微生物。亚硝酸盐含量则与主要乳酸菌的相对丰度呈现负相关,这说明乳酸菌的增殖过程伴随着泡菜亚硝酸盐含量的下降。DODDS等[24]认为,乳酸菌可通过产酸和产酶2种方式清除亚硝酸盐:亚硝根在酸性条件下不稳定,易发生歧化反应生成硝酸根和NO[25],研究发现pH<4,亚硝根便有转化的趋势[3],许多乳酸菌还具有NiR等亚硝基氮还原酶[26],因此乳酸菌的增殖可快速降低泡菜发酵体系内的亚硝酸盐含量。

图5 泡菜发酵早期主要微生物丰度和亚硝酸盐代谢基因的相关性
Fig.5 Correlation between microbial abundance of main microbes and relative genes of nitrite metabolism during the early stage of paocai fermentation
注:“U”代表该属在所示分类水平下未知;“*” “**”代表两变量的相关性在95%、99%的置信区间内显著。
克雷伯菌属(Klebsiella)、埃希氏菌属(Escherichia)、肠杆菌属(Enterobacter)和拉乌尔菌属(Raoultella)等微生物具有编码钼铁固氮酶并固氮的能力[27-29]。与其他微生物不同,这些微生物与编码钼铁固氮酶的nifK、nifD、nifH这3个基因具有较强的正相关性。
与亚硝酸盐生成相关的基因中,nasA、napA、napB与大部分杂菌呈现正相关。之前的研究表明,泡菜中一些微生物,如来略特菌属(Lelliottia)[30]、克雷伯菌属(Klebsiella)[17]微生物中拥有nasA基因,农杆菌属(Agrobacterium)、假单胞菌属(Pseudomonas)、肠杆菌属(Enterobacter)[10,31]中则具有nap基因。可以推测,泡菜发酵体系内的杂菌,可利用NAS、NAP等酶,通过同化或异化亚硝基氮还原途径将泡菜发酵体系中的硝酸盐还原为亚硝酸盐。与之相反,主要乳酸菌的丰度与这些基因则呈现负相关,这说明乳酸菌的生长代谢抑制了产亚硝酸盐微生物的增殖。
与nas、nap基因不同,narG、narH、narI与大部分杂菌呈现负相关,而与乳植物杆菌属、肠杆菌属丰度呈现一定的正相关。这3个基因所编码的硝基氮还原酶(1.7.5.1)广泛分布于肠杆菌属(Enterobacter)[32]、克雷伯菌属(Klebsiella)、埃希氏菌属(Escherichia)[15]、来略特菌属(Lelliottia)[30]、假单胞菌属(Pseudomonas)[33]、寡养单胞菌属(Stenotrophomonas)[34]等常见的泡菜发酵杂菌中,且在厌氧状态下催化硝基氮还原供能的过程[35]。可以推测,随着发酵的进行,体系内O2耗尽,具有该基因的微生物在厌氧条件下继续还原硝酸盐供能并增殖,使该基因丰度不断上升,表现出和乳植物杆菌属、肠杆菌属相似的趋势。
与亚硝酸盐还原相关的基因中,nirB、nirD与大部分杂菌呈现正相关,而与主要乳酸菌呈现负相关,这说明泡菜发酵中亚硝酸盐的酶促还原主要由杂菌完成,乳酸菌可能主要通过产酸降低体系中的亚硝酸盐水平。
3 结论
通过对白萝卜泡菜发酵前5 d的研究,可以发现:a)泡菜发酵早期,浸渍液的总酸迅速升高,pH迅速下降,亚硝酸盐呈现先上升后下降的趋势,在发酵1 d达到峰值(20.68 mg/L),5 d则下降到0.74 mg/L。b)发酵起始时,体系内的微生物以假单胞菌属(Pseudomonas)等杂菌为主,经过1 d发酵,微生物构成迅速改变,肠杆菌属(Enterobacter)成为体系中的优势微生物,随着发酵的进行,乳植物杆菌属(Lactiplantibacillus)微生物迅速增加并占据主导,而肠杆菌属微生物相对丰度在发酵第2天达到最大后逐渐下降。c)泡菜发酵早期,硝酸盐可能通过同化硝基氮还原酶(1.7.1.1/1.7.7.2)、异化硝基氮还原酶(1.7.5.1)被还原为亚硝酸盐,并通过亚硝基氮还原酶(1.7.1.15)将亚硝酸盐还原为胺。d)乳植物杆菌属与发酵体系总酸呈现显著的正相关,与亚硝酸盐含量呈现负相关,亚硝酸盐生成和还原相关酶基因的丰度与大部分杂菌呈现正相关,亚硝酸盐的酶促代谢可能由杂菌主导,产酸可能是乳酸菌清除亚硝酸盐的主要途径。
[1] WANG D D, CHEN G, TANG Y, et al.Effects of temperature on Paocai bacterial succession revealed by culture-dependent and culture-independent methods[J].International Journal of Food Microbiology, 2020, 317:108463.
[2] CHEN A J, LUO W, PENG Y T, et al.Quality and microbial flora changes of radish Paocai during multiple fermentation rounds[J].Food Control, 2019, 106:106733.
[3] WEI W, HU X Y, YANG S, et al.Denitrifying halophilic Archaea derived from salt dominate the degradation of nitrite in salted radish during pickling[J].Food Research International, 2022, 152:110906.
[4] JI F D, JI B P, LI B, et al.Effect of fermentation on nitrate, nitrite and organic acid contents in traditional pickled Chinese cabbage[J].Journal of Food Processing and Preservation, 2009, 33:175-186.
[5] XIA Y J, LIU X F, WANG G Q, et al.Characterization and selection of Lactobacillus brevis starter for nitrite degradation of Chinese pickle[J].Food Control, 2017, 78:126-131.
[6] YAO R L, QIU L N, ZHANG W W, et al.Isolation and characteristics of heterotrophic nitrification-aerobic denitrification bacterium, Bacillus cereus X7 at high salinity[J].Advanced Materials Research, 2013(864-867):111-114.
[7] SONG Q Z, ZHAO F K, WANG B B, et al.Metagenomic insights into Chinese northeast Suancai:Predominance and diversity of genes associated with nitrogen metabolism in traditional household Suancai fermentation[J].Food Research International, 2021, 139:109924.
[8] GERMIDA J J, SICILIANO S D, RENATO DE FREITAS J, et al.Diversity of root-associated bacteria associated with field-grown canola (Brassica napus L.) and wheat (Triticum aestivum L.)[J].FEMS Microbiology Ecology, 1998, 26(1):43-50.
[9] IZUMI H, SERA K.Changes in bacterial flora of Japanese cabbage during growth and potential source of flora[J].Journal of Food Protection, 2011, 74(4):645-650.
[10] OSEI R, YANG C D, CUI L X, et al.Isolation, identification, and pathogenicity of Lelliottia amnigena causing soft rot of potato Tuber in China[J].Microbial Pathogenesis, 2022, 164:105441.
[11] LEFF J W, FIERER N.Bacterial communities associated with the surfaces of fresh fruits and vegetables[J].PLoS One, 2013, 8(3):e59310.
[12] BEHRENDT U, ULRICH A, SCHUMANN P.Plantibacter[M].John Wiley &Sons, Inc., 2015.
[13] LIANG H P, YIN L G, ZHANG Y H, et al.Dynamics and diversity of a microbial community during the fermentation of industrialized Qingcai Paocai, a traditional Chinese fermented vegetable food, as assessed by Illumina MiSeq sequencing, DGGE and qPCR assay[J].Annals of Microbiology, 2018, 68(2):111-122.
[14] PENCE N, LEWIS N, ALLEMAN A B, et al.Revealing a role for the G subunit in mediating interactions between the nitrogenase component proteins[J].Journal of Inorganic Biochemistry, 2021, 214:111273.
[15] DURAND S, GUILLIER M.Transcriptional and post-transcriptional control of the nitrate respiration in bacteria[J].Frontiers in Molecular Biosciences, 2021, 8:667758.
[16] GONZ LEZ P J, CORREIA C, MOURA I, et al.Bacterial nitrate reductases:Molecular and biological aspects of nitrate reduction[J].Journal of Inorganic Biochemistry, 2006, 100(5-6):1015-1023.
LEZ P J, CORREIA C, MOURA I, et al.Bacterial nitrate reductases:Molecular and biological aspects of nitrate reduction[J].Journal of Inorganic Biochemistry, 2006, 100(5-6):1015-1023.
[17] LIN J T, GOLDMAN B S, STEWART V.Structures of genes NasA and nasB, encoding assimilatory nitrate and nitrite reductases in Klebsiella pneumoniae M5al[J].Journal of Bacteriology, 1993, 175(8):2370-2378.
[18] SPARACINO-WATKINS C, STOLZ J F, BASU P.Nitrate and periplasmic nitrate reductases[J].Chemical Society Reviews, 2014, 43(2):676-706.
[19] HARBORNE N R, GRIFFITHS L, BUSBY S J, et al.Transcriptional control, translation and function of the products of the five open reading frames of the Escherichia coli nir operon[J].Molecular Microbiology, 1992, 6(19):2805-2813.
[20] KHLEBODAROVA T M, REE N A, LIKHOSHVAI V A.On the control mechanisms of the nitrite level in Escherichia coli cells:The mathematical model[J].BMC Microbiology, 2016, 16(1):7.
[21] LI R R, ZHENG M L, ZHENG M H, et al.Metagenomic analysis reveals the linkages between bacteria and the functional enzymes responsible for potential ammonia and biogenic amine production in alfalfa silage[J].Journal of Applied Microbiology, 2022, 132(4):2594-2604.
[22] BESSON S, ALMEIDA M G, SILVEIRA C M.Nitrite reduction in bacteria:A comprehensive view of nitrite reductases[J].Coordination Chemistry Reviews, 2022, 464:214560.
[23] WANG Z X, SHAO Y Y.Effects of microbial diversity on nitrite concentration in Pao cai, a naturally fermented cabbage product from China[J].Food Microbiology, 2018, 72:185-192.
[24] DODDS K L, COLLINS-THOMPSON D L.Incidence of nitrite-depleting lactic acid bacteria in cured meats and in meat starter cultures[J].Journal of Food Protection, 1984, 47(1):7-10.
[25] GREENWOOD N N, EARNSHAW A.Chemistry of the Elements (Second Edition).Chapter11 Nitrogen[M].Elsevier Ltd., 1997.
[26] QIU H L, CHANG X Y, LUO Y, et al.Regulation of Nir gene in Lactobacillus plantarum WU14 mediated by GlnR[J].Frontiers in Microbiology, 2022, 13:983485.
[27] BRISSE S, GRIMONT F, GRIMONT P A D.The Prokaryotes.The Genus Klebsiella[M].Springer,2006.
[28] KHALIFA A.Beneficial Microbes in Agro-Ecology Bacteria and Fungi.Chapter 14 Enterobacter[M].Elsevier Inc.,2020.
[29] BERDE C V, BRAMHACHARI P V, BERDE V B.Plant growth-promoting diazotrophs:Current research and advancements[M].Amsterdam:Elsevier, 2022:207-223.
[30] THAKUR P, GAUBA P.Identification and examination of nitrogen metabolic genes in Lelliottia amnigena PTJIIT1005 for their ability to perform nitrate remediation[J].BMC Genomics, 2023, 24(1):104.
[31] WATTS C A, RIDLEY H, CONDIE K L, et al.Selenate reduction by Enterobacter cloacae SLD1a-1 is catalysed by a molybdenum-dependent membrane-bound enzyme that is distinct from the membrane-bound nitrate reductase[J].FEMS Microbiology Letters, 2003, 228(2):273-279.
[32] RAJTA A, BHATIA R, SETIA H, et al.Role of heterotrophic aerobic denitrifying bacteria in nitrate removal from wastewater[J].Journal of Applied Microbiology, 2020, 128(5):1261-1278.
[33] BORRERO-DE ACU A J M, ROHDE M, WISSING J, et al.Protein network of the Pseudomonas aeruginosa denitrification apparatus[J].Journal of Bacteriology, 2016, 198(9):1401-1413.
A J M, ROHDE M, WISSING J, et al.Protein network of the Pseudomonas aeruginosa denitrification apparatus[J].Journal of Bacteriology, 2016, 198(9):1401-1413.
[34] ZHANG Y, LI D H, YAN Q, et al.Genome-wide analysis reveals the emergence of multidrug resistant Stenotrophomonas acidaminiphila strain SINDOREI isolated from a patient with sepsis[J].Frontiers in Microbiology, 2022, 13:989259.
[35] SODERGREN E J, HSU P Y, DEMOSS J A.Roles of the narJ and narI gene products in the expression of nitrate reductase in Escherichia coli[J].Journal of Biological Chemistry, 1988, 263(31):16156-16162.