细菌的抗生素耐药性问题在食品和医疗领域受到越来越多的关注,已经成为人类健康和食品安全的重大威胁。与革兰氏阳性菌相比,革兰氏阴性菌因其独特的双层膜结构,具有对某些抗生素的天然耐药性[1],此外,某些具有多重耐药的革兰氏阴性菌,如碳青霉烯类耐药肠杆菌科,对几乎所有可用的抗生素产生了耐药性[2]。因此,革兰氏阴性菌的耐药性问题亟需解决。在食物流通过程中,食品处理人员、动物、食品以及环境之间存在多重联系[3],这为微生物及其移动遗传元件的传播创造了机会,进一步推动了抗生素耐药性的扩散[4]。在尼泊尔不同零售店收集的样品中,发现来源于鸡肉和水牛肉生肉的大肠杆菌(Escherichia coli)分离株中有52.5%的菌株表现为多重耐药性[5]。在中国江苏4个生猪屠宰场中分离出的105株沙门氏菌(Salmonella)菌株均显示出多重耐药性,其中对四环素的耐药性高达95.4%[6]。对印度北方邦4个区域的淡水养鱼场的抗菌药物耐药性评估表明,气单胞菌(Aeromonas)对β-内酰胺类和氨基糖苷类的抗生素耐药性最高,分别为35.5%和14.5%[7]。从韩国多个养鱼场采集的145个鲻鱼(Mugill cephallus) 样本中鉴定了22个鳗弧菌(Vibrio anguillarum)菌株,所有分离株均表现出多重耐药性,且对苯唑西林、替卡西林、链霉素、环丙沙星100%耐药[8]。从巴基斯坦街头售卖的新鲜果汁中得到的肺炎克雷伯菌(Klebsiella pneumoniae)分离株对头孢吡肟和头孢曲松耐药率分别为72%和64%[9]。
随着对细菌耐药性的深入研究,针对双组分调控系统(two-component regulatory systems,TCSs)的靶向干预已成为一种潜在的抗耐药策略,因此备受关注[10]。首先,TCSs广泛存在于微生物中,但不存在于人体和其他哺乳动物体内,因此开发TCSs小分子抑制剂能避免对人类和其他哺乳动物产生毒性[11]。其次,TCSs与微生物的生存密切相关,它可以调控包括营养物质利用、细胞趋化性、渗透压平衡、呼吸调节等在内的重要生命活动。因此,通过抑制TCSs可以降低细菌自身的存活能力,进而达到抗菌治疗的目的。此外,部分TCSs对微生物的毒力因子表达具有重要影响[12],若针对这些TCSs开发抑制剂,就可以在不影响其生长的情况下有效降低细菌的致病性,减少耐药性的产生。
1 TCSs的组成
双组分调控系统广泛存在于革兰氏阴性菌中。从结构上看,经典的TCSs由两部分组成:感受信号的组氨酸激酶(histidine kinase, HK)和相应的响应调节蛋白(response regulator, RR)。
HK是一种同源二聚体结构的跨膜蛋白,用于感知外界信号的变化,其结构包括N端信号接收区和C端激酶区,两者通过跨膜螺旋连接。N端信号接收区序列高度可变,包括位于细胞外基质或者细胞周质间隙区域的信号受体。C端激酶结构区序列高度保守,位于细胞质基质中,包括组氨酸自磷酸化位点(His)和催化ATP结合结构域。
RR是一种位于细胞质内的同源二聚体蛋白,其主要功能是接收HK的磷酸化信号,并对下游基因的表达进行调控,它包含N端接受器结构域(receiver domain, REC)和C端效应器结构域(effector domain, ED)。N端接受器结构域的序列包括了接受磷酸基团的天冬氨酸位点(Asp)并且具有一定的保守性, C端效应器结构域序列是高度可变的,含有与靶标 DNA 特异性结合的效应器结构域,大部分效应器结构包含一个共同的螺旋-转角-螺旋(helix-turn-helix, HTH)基序。
2 TCSs的作用机理
经典的TCSs作用机理为:外界的信号作用于HK的N端信号接收区,随后激活C端的ATP结合部位,将ATP水解为ADP,并将水解得到的磷酸基团转移到组氨酸残基上,导致自磷酸化。通过磷酸基团的复杂传递过程将组氨酸结合的磷酸基团转移至RR保守N端结构域的天冬氨酸残基,实现天冬氨酸的磷酸化,启动RR可变C端的输出结构域DNA结合活性并产生构象上的改变,从而调节不同靶标DNA表达。
此外,还有一种非经典的TCSs作用机制,由一种杂合的组氨酸激酶(hybrid histidine kinase,HHK)、组氨酸磷酸转移(histidine-containing phosphotransfer, Hpt)蛋白和RR组成,在这种组氨酸磷酸转移蛋白的C端有一个组氨酸磷酸转移结构域。杂合组氨酸激酶自体磷酸化后先将磷酸基团转移到自身的Asp残基上,随后传递到组氨酸磷酸转移蛋白Hpt结构域上的His残基,最后再转移到RR分子的Asp残基,激活其转录活性,即His-Asp-His-Asp途径[13]。经典TCSs及非经典TCSs作用机制如图1所示。
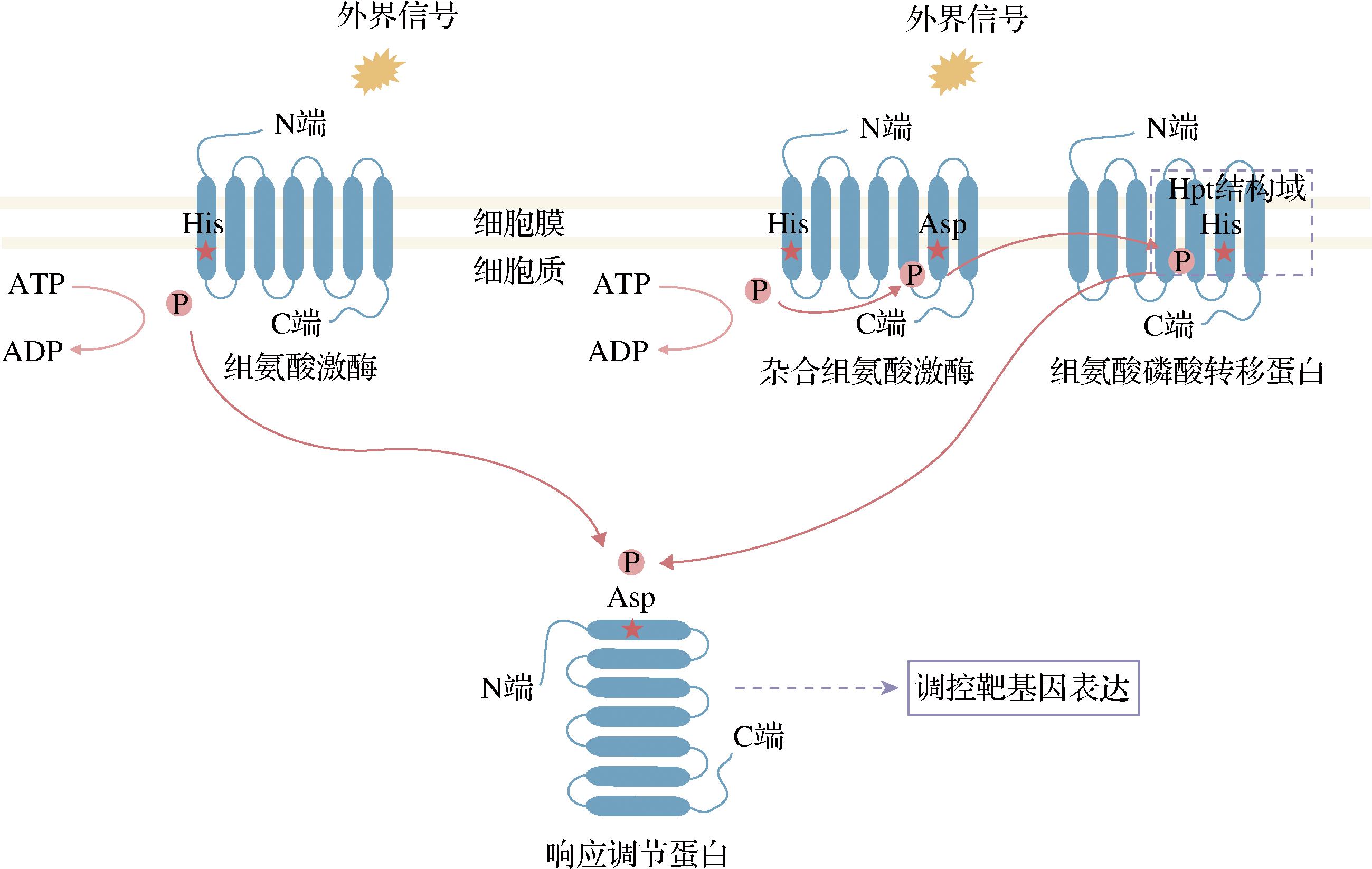
图1 经典与非经典的双组分调控系统作用机制图
Fig.1 Mechanism diagram of the classical and non-classical TCSs
3 TCSs对革兰氏阴性菌抗生素耐药性的调控作用机制
虽然抗生素种类繁多,但TCSs在抗生素耐药过程中发挥作用的调控机制主要可以分为4类:调控抗生素灭活酶的产生,调控细胞表面的修饰,药物流入或流出调节和其他调控机制(如表1所示)。
表1 介导革兰氏阴性菌抗生素耐药性的关键TCSs
Table 1 Key TCSs associated with antibiotics resistance in Gram-negative bacteria

耐药机制双组分调控系统菌种抗生素耐药性的调节抗生素耐药参考文献产生灭活酶CpxA/CpxRE. coli通过感知aD-五肽的浓度激活β-内酰胺酶基因ampC表达β-内酰胺类抗生素[14]BlrA/BlrB嗜水气单胞菌(A. hydrophila)通过感知肽聚糖紊乱从而启动三种β-内酰胺酶(ImiH,CepH,AmpH)的表达β-内酰胺类抗生素[15-16]CreB/CreC铜绿假单胞菌(Pseudomonas aeruginosa)通过上调内膜蛋白CreD的合成,间接影响β-内酰胺酶的表达β-内酰胺类抗生素[17]BesR/BesS泰国伯克霍尔德杆菌(Burkholderia thailandensis)感知包膜紊乱调节A类β-内酰胺酶基因penL的表达β-内酰胺类抗生素[18]PhoP/PhoQ嗜麦芽窄食单胞菌(Stenotrophomonas maltophilia)通过肽聚糖稳态相关基因mltD1和slt增强β-内酰胺酶表达β-内酰胺类抗生素[19]VbrK/VbrR副溶血性弧菌(V. parahaemo-lyticus)感知抗生素并增强β-内酰胺酶表达β-内酰胺类抗生素[20-21]细胞表面的修饰PhoP/PhoQPmrA/PmrBRcsEnvZ/OmpRP. aeruginosa激活arnBCADTEF-pmrE操纵子表达,合成4-氨基-4-脱氧-L-阿拉伯糖(4-amino-4-deoxy-L-arabi-nose, L-Ara4N),对脂质A进行修饰多黏菌素B(polymyx-inB)[22-24]K. pneumoniae受到mgrB负面调节并通过合成L-Ara4N对脂质A进行修饰多黏菌素(polymyxins)和哺乳动物抗菌肽(mammalian antimicrobi-al) peptides[25]阴沟肠杆菌(Enterobacter cloa-cae)促进L-Ara4N修饰脂质A美罗培南(meropenem)[26]肠炎沙门菌(S.enteritidis)促进pmrCAB和arnBCADTEF-pmrE操纵子表达,添加磷酸乙醇胺(phosphoethanolamine, pEtN)和L-Ara4N进行脂质A的修饰多黏菌素B、黏菌素等[24]鲍曼不动杆菌(Acinetobacter baumannii)激活pmrCAB操纵子表达,将pEtN添加到脂质A上黏菌素[27]Salmonella调控ugd基因的表达,合成L-Ara4N修饰脂质A多黏菌素B[28]小肠结肠炎耶尔森氏菌(Yersinia enterocolitica)通过PhoPQ促进pagP基因的表达,增强脂质A的棕榈酰化修饰多黏菌素B[29]A. hydrophila促进arnBCADTEF-pmrE操纵子表达,添加L-Ara4N进行脂质A的修饰黏菌素[30]药物的流入和流出EnvZ/OmpRPhoB/PhoRPhoP/PhoQBaeS/BaeRAdeR/AdeSEvgA/EvgSRstA/RstBPhoP/PhoQCusS/CusRCzcR/CzcSParS/ParRE. coli降低孔蛋白OmpC和OmpF的表达β-内酰胺类抗生素[31-32]肠道沙门氏菌(S. enterica)降低孔蛋白OmpD和OmpW的表达β-内酰胺类抗生素[33]K. pneumoniae降低孔蛋白KpnO的表达头孢他啶(ceftazi-dime)、头孢吡肟、头孢曲松、厄他培南(ertap-enem)、羧苄西林(car-benicillin)和喹诺酮类药物(quinolones)[34]P. aeruginosa降低孔蛋白OprH 表达多黏菌素B[35]A. baumannii激活外排泵MacAB-TolC和AdeIJK表达替加环素(tigecycline)[36]A. baumannii激活外排泵AdeABC表达广谱抗生素耐药性[37]E. coli激活外排泵YhiUV的表达苯唑西林、氯唑西林(cloxacillin)或萘夫西林(nafcillin)[38]荧光假单胞菌(P. fluorescens)激活外排泵EmhABC和MexCD-OprJ表达氯霉素(chlorampheni-col)和庆大霉素(genta-micin)等[39]S. maltophilia激活外排泵SmeZ的表达多黏菌素B[40]K. pneumoniae激活外排泵CusCFBA的表达替加环素[41]P. aeruginosa促进外排泵CzcCBA的表达并抑制孔蛋白OprD的表达碳青霉烯类抗生素(carbapenem)[42]P. aeruginosa激活外排泵MexXY表达并降低孔蛋白OprD的表达阿米卡星(amikacin)和亚胺培南(imipenem)[43]
表1

耐药机制双组分调控系统菌种抗生素耐药性的调节抗生素耐药参考文献药物的流入和流出CpxA/CpxRBaeS/BaeRK. pneumoniae下调孔蛋白OmpC表达,同时上调耐药结节化细胞分化(resistance nodulation cell division, RND)家族外排泵(如AcrB和AcrD)和小多重耐药(small multidrug resistance, SMR)外排泵(如Kp-nEF)表达β-内酰胺类抗生素和氯霉素等[44]E. coli激活外排泵AcrD表达并降低 OmpF 孔蛋白的表达;负向调控外排泵EmrKY表达β-内酰胺类抗生素和氨基糖苷类抗生素;氧氟沙星(ofloxacin)和红霉素(erythromycin)[14, 45]E. coli激活外排泵AcrAD-TolC和MdtABC的表达;同时减少外膜蛋白的表达替莫西林(temocillin)和头孢菌素(cephalospo-rins)[46-47]其他机制SagS/SagRP. aeruginosa调节生物被膜形成固有耐药[ 48]CpxA/CpxR和BasS/BasRE. coli调节生物被膜形成固有耐药[45, 49]AdeR/AdeSA. baumannii调节生物被膜形成固有耐药[50]PhoP/PhoQ阪崎克罗诺杆菌(Cronobacter sakazakii)调节生物被膜形成固有耐药[51]EnvZ/OmpR维氏气单胞菌(A. veronii)调节生物被膜形成固有耐药[52]QseB/QseCE. coli利用群体感应(quorum sensing, QS)机制调节生物被膜形成固有耐药[53]LasI/LasR和RhlI/RhlRP. aeruginosa利用QS机制调节生物被膜形成固有耐药[54]PprA/PprBP. aeruginosa通过调节IVb型菌毛和CupE菌毛影响生物被膜形成固有耐药[55]BfmS/BfmRA. baumannii间接激活菌毛表达增强生物被膜形成固有耐药[56]WigK/WigR霍乱弧菌(V. cholerae)影响细胞壁合成β-内酰胺类抗生素[57]VxrA/VxrBV. cholerae参与细胞壁合成;调节生物被膜形成β-内酰胺类抗生素[58]
3.1 TCSs通过调控革兰氏阴性菌抗生素灭活酶的产生而改变其耐药性
β-内酰胺类抗生素是临床上使用时间最长、范围最广的抗菌药物。青霉素结合蛋白(penicillin binding protein, PBP)在细菌中扮演着关键角色,它是细菌中重要的肽聚糖合成酶,负责肽聚糖的合成与交联。β-内酰胺类抗生素通过与PBP不可逆地结合,阻断肽聚糖的合成,从而导致细菌裂解死亡。为应对这一机制,细菌常常通过产生灭活酶(钝化酶)来对抗β-内酰胺类抗生素的作用。
C型β-内酰胺酶AmpC是一种头孢菌素酶,由ampC基因编码,它可以水解多种β-内酰胺类抗生素,是肠杆菌科细菌和非发酵革兰氏阴性杆菌对β-内酰胺类抗生素产生耐药性的主要原因[16]。AmpC的诱导表达与肽聚糖循环密切相关,例如在E.coli中,CpxAR系统能够感知周质中aD-五肽的浓度增加,从而激活CpxAR响应,导致β-内酰胺酶基因ampC的过度表达[14]。类似地,许多通过双组分调控系统诱导β-内酰胺酶表达的机制也都是通过感知细胞壁损伤来激活的。A.hydrophila中,β-内酰胺类抗生素抑制PBP活性使肽聚糖紊乱受损,激活BlrAB从而启动3种β-内酰胺酶(ImiH,CepH,AmpH)的表达[15-16]。在P. aeruginosa中,CreBC与BlrAB同源,它可以通过上调与BlrD同源的内膜蛋白CreD的合成,间接影响β-内酰胺酶的表达[17]。B. thailandensis中,BesRS系统通过检测由β-内酰胺类、多黏菌素B或其他化学试剂引起的包膜紊乱,调节A类β-内酰胺酶基因penL的表达[18]。S. maltophilia中PhoPQ的缺失下调了与肽聚糖稳态相关基因mltD1和slt的表达,降低了β-内酰胺酶的合成并提高了细胞膜的透过性,进而影响了β-内酰胺类药物抗性[19]。不同于通过破坏细胞壁来激活β-内酰胺酶的机制,V.parahaemolyticus中的VbrK是通过感知β-内酰胺类抗生素后发生空间结构变化(如图2所示),从而诱导A类β-内酰胺酶的表达[20-21]。
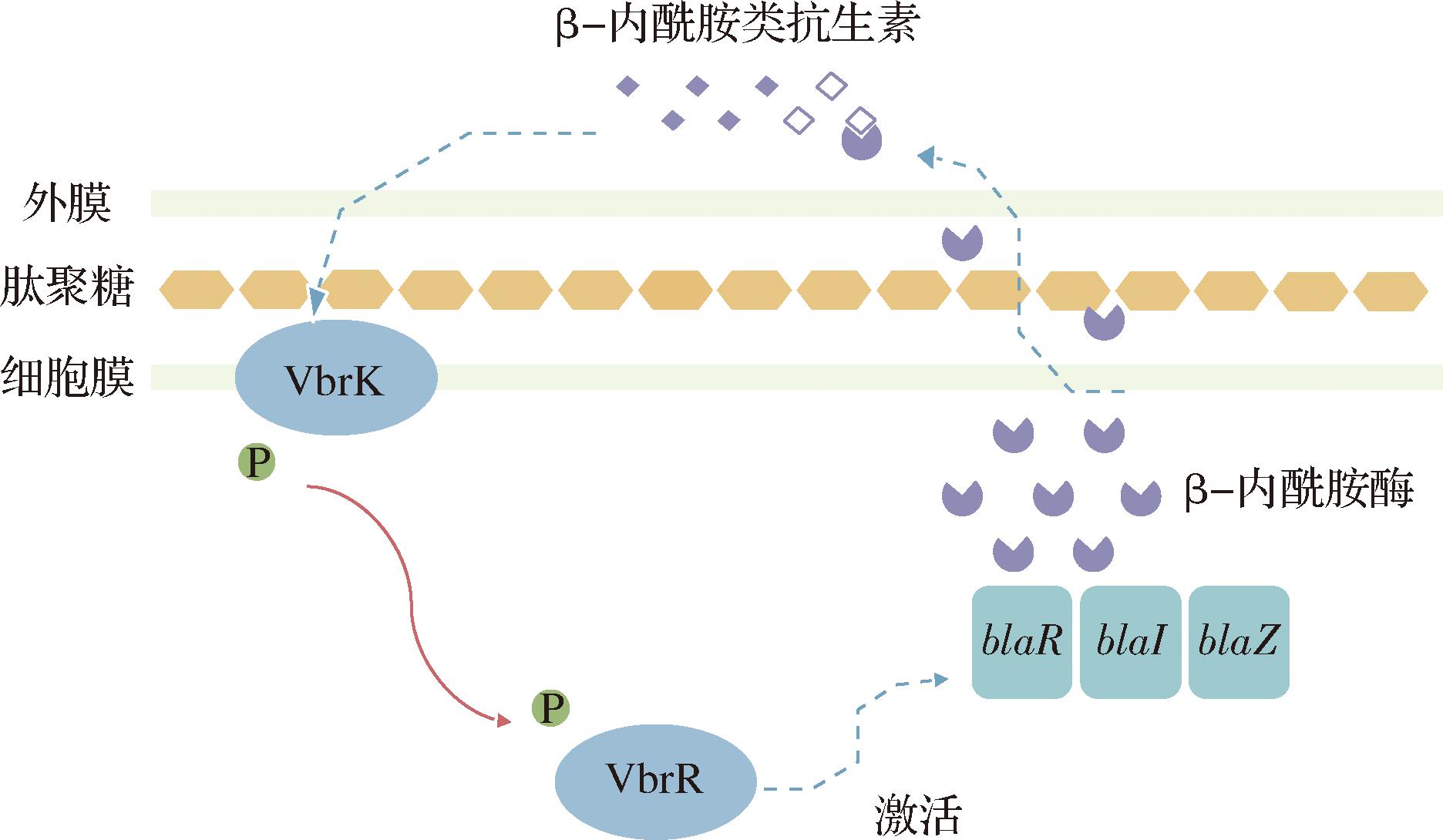
图2 VbrK/VbrR调控副溶血弧菌β-内酰胺类抗生素耐药性示意图
Fig.2 Schematic diagram of β-lactams resistance regulated by VbrK/VbrR in V.parahaemolyticus
由于细菌通过编码灭活酶基因的突变和转移能够迅速适应抗生素的更迭进步,产生更强的耐药性,革兰氏阴性菌对β-内酰胺类抗生素的耐药性日益严峻。早期的β-内酰胺酶已经逐渐被超广谱β-内酰胺酶(extended-spectrum β-lactamases, ESBLs)取代,显著降低了传统抗生素在治疗食源性致病菌感染中的疗效。同时,食品供应链的复杂化扩大了耐药性细菌的传播范围,进一步加深了革兰氏阴性菌的抗生素耐药性问题。因此,β-内酰胺酶的抑制剂研发成为当前的研究热点,应针对β-内酰胺酶诱导表达通路中的靶向分子,增加对新型β-内酰胺酶抑制剂和新型抗生素的研发投入。将新药物与现有抗生素联合使用,从而有效应对革兰氏阴性菌中的β-内酰胺类耐药问题。
3.2 TCSs通过调控革兰氏阴性菌细胞表面修饰而改变其耐药性
多黏菌素B、黏菌素、氨基糖苷类以及宿主的阳离子抗菌肽(cationic antimicrobial peptides, CAMPs)等带有正电荷的抗生素利用革兰氏阴性细菌外膜的净负电荷发挥作用[59]。外膜的负电荷性质来源于脂多糖(lipopolysaccharide, LPS)中带负电荷的脂质A。细菌通过共价修饰脂质A来改变外膜的电荷性质,常见的修饰包括添加L-Ara4N、pEtN或棕榈酸。其中,添加L-Ara4N能将膜的净负电荷降低至零,pEtN修饰是将电荷从-1.5降低至-1,棕榈酸的加入则会降低细胞膜的流动性[60]。
PhoPQ与PmrAB这两种经典的TCSs对革兰氏阴性菌细胞表面的修饰具有重要调控作用,如图3所示。在P.aeruginosa中,低浓度Mg2+的条件下,PhoPQ系统激活编码7种蛋白质的arnBCADTEF-pmrE操纵子,此操作子负责合成L-Ara4N并转移到脂质A上,提高对多黏菌素B的耐药性[22-24]。在K.pneumoniae中,PhoPQ系统的活性受到mgrB基因的负反馈调节。mgrB失活或缺失会导致PhoPQ系统过表达,添加L-Ara4N对脂质A进行修饰[25]。E. cloacae中的PhoPQ系统可促进脂质A的L-Ara4N修饰,从而引发对美罗培南的耐受[26]。PmrAB在E.coli、S.enterica、K.Pneumoniae、鼠疫杆菌(Yersinia pestis)、啮齿枸橼酸杆菌(Citrobacter rodentium)、P.aeruginosa和A. baumannii中均是脂质A修饰的主要调节因子,外部信号如高浓度的Fe3+、Al3+和低pH值能触发 PmrB 的自磷酸化,然后将磷酸基团转移到 PmrA 的保守天冬氨酸残基,激活后的PmrA(PmrA-P)与脂质A修饰基因(如arnBCADTEF-ugd、eptA和naxD)的启动子区域结合,并诱导其转录[61]。在S.enteritidis中,PhoP可以通过PmrD进一步激活PmrAB,促进pmrCAB和arnBCADTEF-pmrE操纵子修饰脂质A[24]。pmrCAB操纵子是通过激活编码磷酸乙醇胺转移酶的pmrC(也称为eptA)表达,将pEtN基团添加到LPS上,来影响多黏菌素与细菌外膜的结合。A.baumannii在酸性条件下通过PmrAB激活pmrCAB操纵子,将pEtN添加到脂质A上,从而保护细胞外膜免受黏菌素的作用[27]。
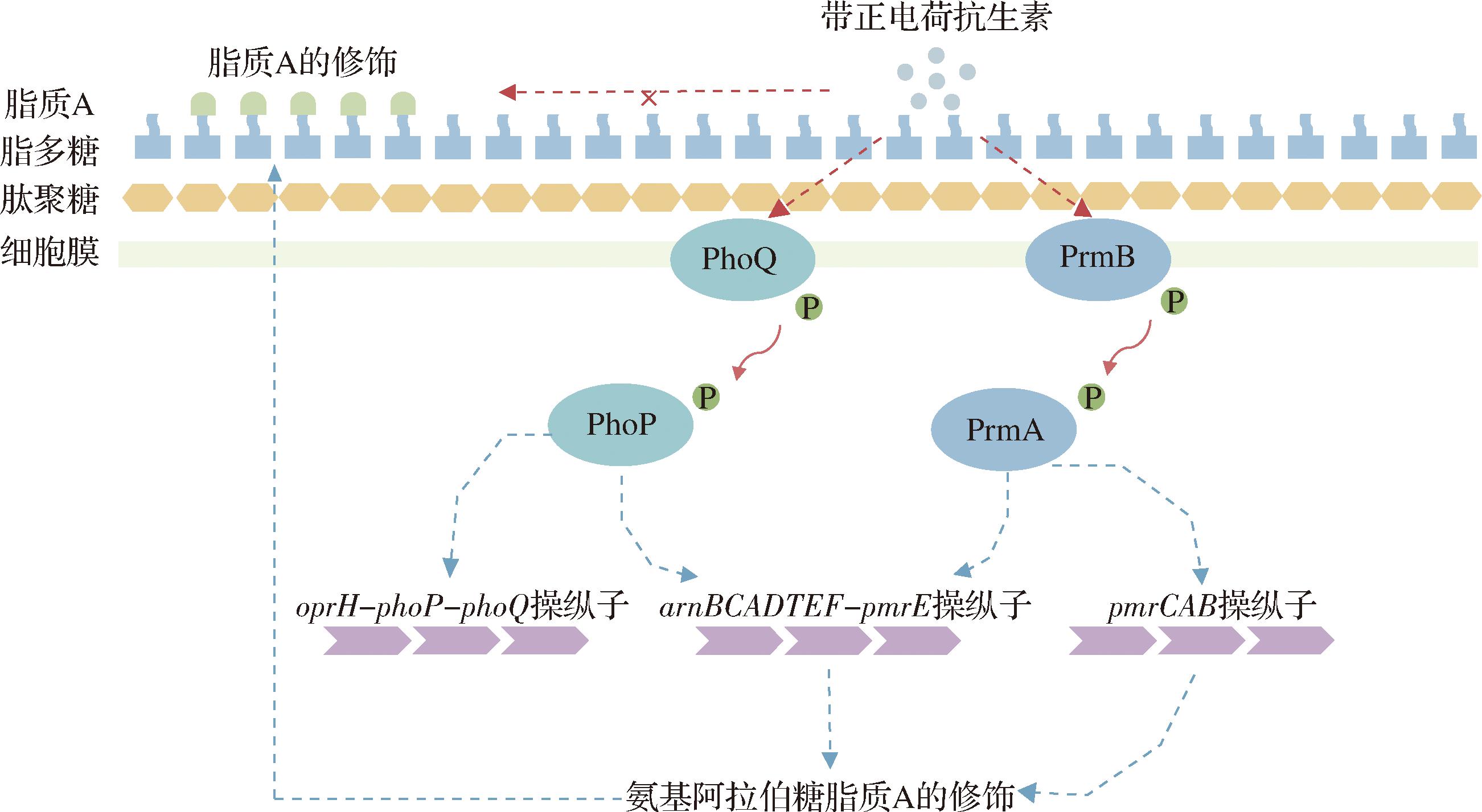
图3 PhoPQ与PmrAB通过细胞表面修饰调控细菌耐药性作用示意图
Fig.3 Regulation diagram of bacterial antibiotic resistance through cell surface modification by PhoPQ and PmrAB
Rcs系统是一种广泛存在于肠杆菌科中非经典的TCS,由跨膜杂交激酶RcsC、跨膜蛋白RcsD和反应调节因子RcsB三部分组成[29]。在Salmonella中,rcsC基因参与调控ugd基因的表达,ugd基因负责合成L-Ara4N,以此修饰LPS,导致对多黏菌素B的耐药性[28]。在Y.enterocolitica中,Rcs系统积极调节PhoPQ系统的活性,促进pagP基因(编码棕榈酰转移酶)的表达,增强脂质A的棕榈酰化修饰,提高对多黏菌素B的耐药性[29]。在A.hydrophila中,EnvZ/OmpR系统能上调arnBCADTEF操纵子的表达而调控LPS的L-Ara4N修饰,此外,当EnvZ/OmpR系统缺失后, PhoPQ可作为其替代系统,通过PmrC调控脂质A的pEtN修饰,从而增强对黏菌素的耐药性[30]。
3.3 TCSs通过调节药物的流入和流出而改变其耐药性
细菌通过孔蛋白和外排泵的表达来调节分子的进出,从而影响对抗生素的耐药性。孔蛋白也称为外膜蛋白(outer membrane protein, OMP),是革兰氏阴性菌外膜中一种允许分子被动扩散的β-barrel蛋白。亲水抗生素如β-内酰胺类、氨基糖苷类和氟喹诺酮类药物可通过孔蛋白进入细胞。因此,抑制孔蛋白的表达可降低抗生素渗透性,产生耐药作用。如在E.coli中,EnvZ-OmpR系统通过降低孔蛋白OmpC和OmpF的表达[31],提高了对β-内酰胺类药物的抗性[32]。在S.enterica中,EnvZ/OmpR 系统通过抑制OmpD和OmpW的表达,降低外膜通透性,从而增强对β-内酰胺类抗生素的耐药性[33]。PhoBR系统中的响应调节蛋白PhoB可与孔蛋白KpnO编码基因启动区结合而促进其表达,进而影响K.pneumoniae对头孢他啶、头孢吡肟、头孢曲松、厄他培南、羧苄西林和喹诺酮类药物的耐药性[34]。低Mg2+浓度下P.aeruginosa 中PhoPQ控制oprH-phoP-phoQ操作子,调节孔蛋白OprH 表达,使其对多黏菌素B的耐药性增强[35]。
细菌外排泵(efflux pumps, EPs)是菌体用来排出药物并维持细胞稳态所必需的活性转运蛋白。根据其结构和功能,这些泵通常被分为5个主要家族:耐药结节化细胞分化RND家族、多药和毒性化合物外排(multidrug and toxic compound extrusion, MATE)家族、主要易化超(major facilitator superfamily, MFS)家族、SMR家族和ATP结合盒(ATP-binding cassette, ABC)家族。在A.baumannii中,BaeSR通过激活MacAB-TolC和AdeIJK外排泵系统来影响其对替加环素的耐药性[36]。AdeRS可以通过激活AdeABC外排泵的表达将四环素、氯霉素、红霉素、甲氧苄啶、替加环素和氨基糖苷类抗生素排出细胞外,使A.baumannii具有广谱抗生素耐药性[37]。E.coli中的EvgAS系统负责诱导RND型外排泵YhiUV的表达,从而增强对苯唑西林、氯唑西林或萘夫西林的耐药性[38]。RstAB通过激活EmhABC和MexCD-OprJ外排泵的表达来增加P.fluorescens对氯霉素和庆大霉素等多种抗生素耐药性[39]。在低浓度的Mg2+下,PhoPQ 通过调控S.maltophili外排转运蛋白SmeZ的表达和细胞膜的透过性而影响细菌对多黏菌素B的耐药性[40]。CusSR可以通过CusCFBA外排泵诱导K.pneumoniae对替加环素的耐药性[41]。
某些双组分调控系统能够同时调控孔蛋白和外排泵的表达。P.aeruginosa中的CzcRS系统可促进外排泵CzcCBA的表达并直接抑制孔蛋白OprD的表达,从而影响对碳青霉烯类抗生素的耐药性[42]。此外,该菌中ParSR激活外排泵基因mexXY并降低孔蛋白OprD的表达,增强阿米卡星和亚胺培南等抗生素耐药性[43]。在K.pneumoniae中,CpxAR通过下调孔蛋白OmpC,同时上调RND 型外排泵(如AcrB和AcrD)和SMR 型外排泵(如KpnEF),从而增强对β-内酰胺类和氯霉素等多种抗生素耐药性[44]。在E.coli中,CpxAR系统则通过调节与抗生素摄取(如ompF)和排出(如acrD)相关基因的表达而影响其对β-内酰胺类和氨基糖苷类抗生素的抗性[14]。此外,该系统还通过负向调控MFS型外排泵EmrKY,影响对氧氟沙星和红霉素的敏感性[45]。在E.coli中,BaeR通过调节外排泵AcrAD-TolC和MdtABC的表达来提高对替莫西林抗生素的耐药性,同时通过减少外膜蛋白的表达而降低对头孢菌素的敏感性[46-47]。
3.4 TCSs改变革兰氏阴性菌抗生素耐药性的其他调控机制
3.4.1 TCSs通过影响革兰氏阴性菌的生物被膜而改变其耐药性
生物被膜在细菌的生存过程中起到不可忽视的作用,它是在生物或非生物表面由细菌分泌的物质包裹形成的一个复杂的微生物聚集体。生物被膜能减弱环境对细菌的不利影响,同时缓解细胞内部的压力,并将外界的信号传递到细菌内部。当生物被膜的形成增多时,驻留于其内的微生物能够通过多种机制(如降低药物的渗透性和处于休眠状态等)增强对抗生素的耐受性[62]。TCSs在生物被膜的形成与抗生素耐药性之间的关系非常复杂,主要通过以下几种机制介导。首先,一些TCSs可以感知到特定的外部刺激信号,并激活与生物被膜生成相关的基因。例如,P.aeruginosa中的SagSR在生物被膜形成的附着阶段发挥着关键作用,SagS的失活导致生物被膜相较于浮游细胞对妥布霉素和诺氟沙星表现出更高的敏感性[48]。在E.coli中,CpxAR[45]和BasSR[49]的缺失减少了生物被膜形成,增加了抗生素敏感性。A.baumannii中受到AdeRS系统调节的AdeB蛋白缺失会导致黏膜组织上生物被膜的形成减少,进而影响其抗生素抗性[50]。PhoPQ系统缺失后C. sakazakii生物被膜含量显著降低[51]。维氏气单胞菌(A.veronii)中EnvZ-OmpR系统可以通过增强生物被膜形成能力提高自身对头孢曲松和新霉素的抗性[52]。其次,TCSs通过QS机制调控生物被膜合成,进而引发抗生素耐药性。例如在E.coli中,群体感应调节因子QseBC可以通过上调生物被膜相关基因的转录来增加生物被膜的形成,以此增强耐药性[53]。LasIR和RhlIR通过催化N-酰基高丝氨酸内酯(N-acylated homoserine lac-tone, AHL)来调节P.aeruginosa的生物被膜形成,进而影响其对多种抗生素的耐药性[54]。其他一些TCSs则是通过调节菌毛表达来间接调控生物被膜合成,如P.aeruginosa中PprAB是通过调控黏附素BapA、IVb型菌毛、CupE菌毛、eDNA等影响生物被膜的形成从而改变细菌的妥布霉素耐药性[55]。A.baumannii中BfmRS可以间接激活菌毛表达增强其被膜形成能力[63],增加A.baumannii对美罗培南和粘菌素的耐药性[56]。生物被膜作为细菌自我保护的一种状态,深刻影响着细菌的存活率和耐药性。TCSs在生物被膜形成中的调控作用使得生物被膜的形成更具适应性,但目前针对细菌生物被膜的药物疗效仍未达到理想水平。
3.4.2 TCSs通过影响革兰氏阴性菌细胞壁而改变其耐药性
细胞壁作为细菌的重要保护结构,其生物合成所需的酶常常成为许多强效抗生素的靶标。然而,对于细胞壁在抗生素攻击下的自我修复机制理解较浅。V.cholerae中WigKR的激活能够引发细胞壁合成相关基因的表达上调,从而促使细菌重新合成细胞壁并恢复其杆状形态,这一过程在细胞壁生物合成和抗生素耐受性方面发挥了重要作用[57]。此外,VxrAB不仅参与霍乱弧菌细胞壁合成,还在生物被膜形成及VI型分泌系统中发挥作用,显著增强了对β-内酰胺类药物的耐受性[58]。
4 展望
TCSs在革兰氏阴性细菌中普遍存在,并在抗生素耐药性方面发挥了至关重要的作用。本文综述了TCSs在革兰氏阴性菌中介导的4种耐药机制,一些TCS在细菌中参与多个截然不同的耐药过程,不同TCS之间存在垂直或水平的调控机制,从而构建了一个错综复杂的调控网络。例如,CpxAR系统在E.coli中不仅调控β-内酰胺酶基因的表达[14],还涉及外排泵和孔蛋白的调节[14, 45],同时影响生物被膜的合成[45]。PmrAB系统在多种革兰氏阴性菌均可以通过对脂质A的修饰增强其对抗生素的耐受性[24, 27]。此外,BaeSR与AdeRS系统的相互作用使得A.baumannii展现出广谱抗生素耐药性[36-37]。这些例证进一步展现出TCSs在细菌耐药调控过程中的核心作用及其作为靶标在开发新型抗耐药菌策略的巨大潜力。然而,目前对TCSs的理解仍有许多空白,如需要进一步鉴定探究新型的未知TCSs,研究其感知和传递信号的机制以及不同TCSs之间的相互作用。现有研究表明,TCSs可以作为抗菌治疗的靶点,且对细胞的潜在伤害较小。目前已经有多种天然和合成的TCSs抑制剂,它们通过抑制细菌的HK或RR活性对抗生素耐药性以及毒力产生一定影响。未来的研究应重点关注革兰氏阴性菌中TCSs在耐药基因表达及其传播中的调控机制,以深入揭示细菌抵御抗生素压力的适应性策略。结合TCSs的功能研究,应开发快速灵敏的检测方法,以识别和监测食品中耐药性革兰氏阴性菌的存在。这将为食品行业提供实时应对措施,确保产品安全,降低耐药性细菌通过食品传播的风险。深入理解双组分调控系统的调控机制,不仅有助于研发新型抗微生物剂和天然防腐剂,还可以有效减缓耐药性传播的速度。
[1] IMAI Y, MEYER K J, IINISHI A, et al.A new antibiotic selectively kills Gram-negative pathogens[J].Nature, 2019, 576(7787):459-464.
[2] SONG M R, LIU Y, HUANG X Y, et al.A broad-spectrum antibiotic adjuvant reverses multidrug-resistant Gram-negative pathogens[J].Nature Microbiology, 2020, 5(8):1040-1050.
[3] GEMEDA B A, ASSEFA A, JALETA M B, et al.Antimicrobial resistance in Ethiopia:A systematic review and meta-analysis of prevalence in foods, food handlers, animals, and the environment[J].One Health, 2021, 13:100286.
[4] HALAWA E M, FADEL M, AL-RABIA M W, et al.Antibiotic action and resistance:Updated review of mechanisms, spread, influencing factors, and alternative approaches for combating resistance[J].Frontiers in Pharmacology, 2024, 14:1305294.
[5] SAUD B, PAUDEL G, KHICHAJU S, et al.Multidrug-resistant bacteria from raw meat of buffalo and chicken, Nepal[J].Veterinary Medicine International, 2019, 2019:7960268.
[6] LIU Q X, CHEN W J, ELBEDIWI M, et al.Characterization of Salmonella resistome and plasmidome in pork production system in Jiangsu, China[J].Frontiers in Veterinary Science, 2020, 7:617.
[7] KUMAR C B, RATHORE G.Assessment of freshwater fish farms for the identification of the geographical areas harbouring antimicrobial resistance[J].Aquaculture, 2024, 586:740808.
[8] KUMARAGE P M, MAJEED S, DE SILVA L A D S, et al.Detection of virulence, antimicrobial resistance, and heavy metal resistance properties in Vibrio anguillarum isolated from mullet (Mugil cephalus) cultured in Korea[J].Brazilian Journal of Microbiology, 2023, 54(1):415-425.
[9] SIDDIQUE M H, QAMAR M U, HAYAT S, et al.Polymicrobial multidrug-resistant bacteria isolated from street vended fresh fruit juices in Pakistan[J].British Food Journal, 2018, 120(6):1358-1365.
[10] DE GAETANO G V, LENTINI G, FAM A, et al.Antimicrobial resistance:Two-component regulatory systems and multidrug efflux pumps[J].Antibiotics, 2023, 12(6):965.
A, et al.Antimicrobial resistance:Two-component regulatory systems and multidrug efflux pumps[J].Antibiotics, 2023, 12(6):965.
[11] PAPON N, STOCK A M.Two-component systems[J].Current Biology:CB, 2019, 29(15):R724-R725.
[12] TIWARI S, JAMAL S B, HASSAN S S, et al.Two-component signal transduction systems of pathogenic bacteria As targets for antimicrobial therapy:An overview[J].Frontiers in Microbiology, 2017, 8:1878.
[13] TIERNEY A R, RATHER P N.Roles of two-component regulatory systems in antibiotic resistance[J].Future Microbiology, 2019, 14(6):533-552.
[14] MASI M, PINET E, PAG S J M.Complex response of the CpxAR two-component system to β-lactams on antibiotic resistance and envelope homeostasis in Enterobacteriaceae[J].Antimicrobial Agents and Chemotherapy, 2020, 64(6):e00291-20.
S J M.Complex response of the CpxAR two-component system to β-lactams on antibiotic resistance and envelope homeostasis in Enterobacteriaceae[J].Antimicrobial Agents and Chemotherapy, 2020, 64(6):e00291-20.
[15] AVISON M B, NIUMSUP P, NURMAHOMED K, et al.Role of the ‘cre/blr-tag’ DNA sequence in regulation of gene expression by the Aeromonas hydrophila beta-lactamase regulator, BlrA[J].The Journal of Antimicrobial Chemotherapy, 2004, 53(2):197-202.
[16] 徐超奕, 张婷, 蔡静晓, 等.革兰氏阴性菌中β-内酰胺酶诱导表达调控机制研究进展[J].生物工程学报, 2018, 34(8):1288-1296.
XU C Y, ZHANG T, CAI J X, et al.Progress in regulatory mechanism for inducing β-lactamase in Gram-negative bacteria[J].Chinese Journal of Biotechnology, 2018, 34(8):1288-1296.
[17] MOYA B, DÖTSCH A, JUAN C, et al.Beta-lactam resistance response triggered by inactivation of a nonessential penicillin-binding protein[J].PLoS Pathogens, 2009, 5(3):e1000353.
[18] LEE D J, PARK J, YI H, et al.A two-component-system-governed regulon that includes a β-lactamase gene is responsive to cell envelope disturbance[J].mBio, 2022, 13(4):e0174922.
[19] HUANG H H, WU B K, LI L H, et al.Role of the PhoPQ two-component regulatory system in the β-lactam resistance of Stenotrophomonas maltophilia[J].Journal of Antimicrobial Chemotherapy, 2021, 76(6):1480-1486.
[20] GOH B C, CHUA Y K, QIAN X L, et al.Crystal structure of the periplasmic sensor domain of histidine kinase VbrK suggests indirect sensing of β-lactam antibiotics[J].Journal of Structural Biology, 2020, 212(2):107610.
[21] LI L, WANG Q Y, ZHANG H, et al.Sensor histidine kinase is a β-lactam receptor and induces resistance to β-lactam antibiotics[J].Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(6):1648-1653.
[22] SCHUREK K N, SAMPAIO J L M, KIFFER C R V, et al.Involvement of pmrAB and phoPQ in polymyxin B adaptation and inducible resistance in non-cystic fibrosis clinical isolates of Pseudomonas aeruginosa[J].Antimicrobial Agents and Chemotherapy, 2009, 53(10):4345-4351.
[23] PUJA H, BOLARD A, NOGU S A, et al.The efflux pump MexXY/OprM contributes to the tolerance and acquired resistance of Pseudomonas aeruginosa to colistin[J].Antimicrobial Agents and Chemotherapy, 2020, 64(4):e02033-19.
S A, et al.The efflux pump MexXY/OprM contributes to the tolerance and acquired resistance of Pseudomonas aeruginosa to colistin[J].Antimicrobial Agents and Chemotherapy, 2020, 64(4):e02033-19.
[24] 于永峰, 权衡, 董文豪, 等.双组分调控系统介导革兰阴性菌耐药的作用机制[J].畜牧兽医学报, 2022, 53(6):1689-1701.
YU Y F, QUAN H, DONG W H, et al.The mechanism of two-component regulatory system mediating drug resistance of gram-negative bacteria[J].Acta Veterinaria et Zootechnica Sinica, 2022, 53(6):1689-1701.
[25] KIDD T J, MILLS G, S -PESSOA J, et al.A Klebsiella pneumoniae antibiotic resistance mechanism that subdues host defences and promotes virulence[J].EMBO Molecular Medicine, 2017, 9(4):430-447.
-PESSOA J, et al.A Klebsiella pneumoniae antibiotic resistance mechanism that subdues host defences and promotes virulence[J].EMBO Molecular Medicine, 2017, 9(4):430-447.
[26] MURTHA A N, KAZI M I, SCHARGEL R D, et al.High-level carbapenem tolerance requires antibiotic-induced outer membrane modifications[J].PLoS Pathogens, 2022, 18(2):e1010307.
[27] KO S Y, KIM N, PARK S Y, et al.Acinetobacter baumannii under acidic conditions induces colistin resistance through PmrAB activation and lipid A modification[J].Antibiotics, 2023, 12(5):813.
[28] MOUSLIM C, GROISMAN E A.Control of the Salmonella ugd gene by three two-component regulatory systems[J].Molecular Microbiology, 2003, 47(2):335-344.
[29] MENG J, YOUNG G, CHEN J Y.The rcs system in Enterobacteriaceae:Envelope stress responses and virulence regulation[J].Frontiers in Microbiology, 2021, 12:627104.
[30] LIU J Q, XIAO G, ZHOU W P, et al.Various novel colistin resistance mechanisms interact to facilitate adaptation of Aeromonas hydrophila to complex colistin environments[J].Antimicrobial Agents and Chemotherapy, 2021, 65(7):e0007121.
[31] CAI S J, INOUYE M.EnvZ-OmpR interaction and osmoregulation in Escherichia coli[J].The Journal of Biological Chemistry, 2002, 277(27):24155-24161.
[32] RODRIGUES I C, RODRIGUES S C, DUARTE F V, et al.The role of outer membrane proteins in UPEC antimicrobial resistance:A systematic review[J].Membranes, 2022, 12(10):981.
[33] KO D, CHOI S H.Mechanistic understanding of antibiotic resistance mediated by EnvZ/OmpR two-component system in Salmonella enterica serovar Enteritidis[J].Journal of Antimicrobial Chemotherapy, 2022, 77(9):2419-2428.
[34] SRINIVASAN V B, VENKATARAMAIAH M, MONDAL A, et al.Functional characterization of a novel outer membrane porin KpnO, regulated by PhoBR two-component system in Klebsiella pneumoniae NTUH-K2044[J].PLoS One, 2012, 7(7):e41505.
[35] MACFARLANE E L A, KWASNICKA A, OCHS M M, et al.PhoP-PhoQ homologues in Pseudomonas aeruginosa regulate expression of the outer-membrane protein OprH and polymyxin B resistance[J].Molecular Microbiology, 1999, 34(2):305-316.
[36] LIN M F, LIN Y Y, LAN C Y.The role of the two-component system BaeSR in disposing chemicals through regulating transporter systems in Acinetobacter baumannii[J].PLoS One, 2015, 10(7):e0132843.
[37] 袁茂冉, 葛宏华, 马金鸣.鲍曼不动杆菌外排泵介导多药耐药性[J].中国生物化学与分子生物学报, 2020, 36(11):1295-1302.
YUAN M R, GE H H, MA J M.Efflux pumps mediate multidrug resistance in Acinetobacter baumannii[J].Chinese Journal of Biochemistry and Molecular Biology, 2020, 36(11):1295-1302.
[38] HIRAKAWA H, NISHINO K, YAMADA J, et al.Beta-lactam resistance modulated by the overexpression of response regulators of two-component signal transduction systems in Escherichia coli[J].The Journal of Antimicrobial Chemotherapy, 2003, 52(4):576-582.
[39] LI D Y, HAN J T, ZHANG M Y, et al.The two-component system RstA/RstB regulates expression of multiple efflux pumps and influences anaerobic nitrate respiration in Pseudomonas fluorescens[J].mSystems, 2021, 6(6):e0091121.
[40] LIU M C, TSAI Y L, HUANG Y W, et al.Stenotrophomonas maltophilia PhoP, a two-component response regulator, involved in antimicrobial susceptibilities[J].PLoS One, 2016, 11(5):e0153753.
[41] CHEN D J, ZHAO Y N, QIU Y Q, et al.CusS-CusR two-component system mediates tigecycline resistance in carbapenem-resistant Klebsiella pneumoniae[J].Frontiers in Microbiology, 2020, 10:3159.
[42] DIEPPOIS G, DUCRET V, CAILLE O, et al.The transcriptional regulator CzcR modulates antibiotic resistance and quorum sensing in Pseudomonas aeruginosa[J].PLoS One, 2012, 7(5):e38148.
[43] MULLER C, PLÉSIAT P, JEANNOT K. A two-component regulatory system interconnects resistance to polymyxins, aminoglycosides, fluoroquinolones, and β-lactams in Pseudomonas aeruginosa[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(3): 1211-1221.
[44] LI L F, MA J Y, CHENG P, et al.Roles of two-component regulatory systems in Klebsiella pneumoniae:Regulation of virulence, antibiotic resistance, and stress responses[J].Microbiological Research, 2023, 272:127374.
[45] MA K, WANG H, LV Z F, et al.The two-component system CpxRA affects antibiotic susceptibility and biofilm formation in avian pathogenic Escherichia coli[J].Animals, 2023, 13(3):383.
[46] PÉREZ-PALACIOS P, RODR GUEZ-OCHOA J L, VEL
GUEZ-OCHOA J L, VEL ZQUEZ-ESCUDERO A, et al.Implications of two-component systems EnvZ/OmpR and BaeS/BaeR in in vitro temocillin resistance in Escherichia coli[J].Journal of Antimicrobial Chemotherapy, 2024, 79(3):641-647.
ZQUEZ-ESCUDERO A, et al.Implications of two-component systems EnvZ/OmpR and BaeS/BaeR in in vitro temocillin resistance in Escherichia coli[J].Journal of Antimicrobial Chemotherapy, 2024, 79(3):641-647.
[47] WANG S, YOU C, MEMON F Q, et al. BaeR participates in cephalosporins susceptibility by regulating the expression level of outer membrane proteins in Escherichia coli[J]. The Journal of Biochemistry, 2021, 169(1): 101-108.
[48] GUPTA K, MARQUES C N H, PETROVA O E, et al.Antimicrobial tolerance of Pseudomonas aeruginosa biofilms is activated during an early developmental stage and requires the two-component hybrid SagS[J].Journal of Bacteriology, 2013, 195(21):4975-4987.
[49] YU L M, WANG H, HAN X G, et al.The two-component system, BasSR, is involved in the regulation of biofilm and virulence in avian pathogenic Escherichia coli[J].Avian Pathology, 2020, 49(6):532-546.
[50] RICHMOND G E, EVANS L P, ANDERSON M J, et al.The Acinetobacter baumannii two-component system AdeRS regulates genes required for multidrug efflux, biofilm formation, and virulence in a strain-specific manner[J].mBio, 2016, 7(2):e00430-16.
[51] MA Y, ZHANG Y Y, SHAN Z G, et al.Involvement of PhoP/PhoQ two-component system in biofilm formation in Cronobacter sakazakii[J].Food Control, 2022, 133:108621.
[52] WANG Y D, GONG J S, GUAN Y C, et al.OmpR (TCS response regulator) of Aeromonas veronii plays a major role in drug resistance, stress resistance and virulence by regulating biofilm formation[J].Microbial Pathogenesis, 2023, 181:106176.
[53] LI W C, XUE M, YU L M, et al.QseBC is involved in the biofilm formation and antibiotic resistance in Escherichia coli isolated from bovine mastitis[J].PeerJ, 2020, 8:e8833.
[54] BA KAN C, Y
KAN C, Y LD
LD R
R M T, BILGIN M, et al.Determination of biofilm formation, antibiotic susceptibility profiles and quorum sensing mediated virulence factors in ceftazidime resistant Pseudomonas aeruginosa[J].Biologia, 2023, 78(10):2881-2893.
M T, BILGIN M, et al.Determination of biofilm formation, antibiotic susceptibility profiles and quorum sensing mediated virulence factors in ceftazidime resistant Pseudomonas aeruginosa[J].Biologia, 2023, 78(10):2881-2893.
[55] DE BENTZMANN S, GIRAUD C, BERNARD C S, et al.Unique biofilm signature, drug susceptibility and decreased virulence in Drosophila through the Pseudomonas aeruginosa two-component system PprAB[J].PLoS Pathogens, 2012, 8(11):e1003052.
[56] RUSSO T A, MANOHAR A, BEANAN J M, et al.The response regulator BfmR is a potential drug target for Acinetobacter baumannii[J].mSphere, 2016, 1(3):e00082-16.
[57] DÖRR T, ALVAREZ L, DELGADO F, et al.A cell wall damage response mediated by a sensor kinase/response regulator pair enables beta-lactam tolerance[J].Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(2):404-409.
[58] SHIN J H, CHOE D, RANSEGNOLA B, et al.A multifaceted cellular damage repair and prevention pathway promotes high-level tolerance to β-lactam antibiotics[J].EMBO Reports, 2021, 22(2):e51790.
[59] SHERMAN M E, SMITH R D, GARDNER F M, et al.A sensitive GC-MS method for quantitation of lipid A backbone components and terminal phosphate modifications[J].Journal of the American Society for Mass Spectrometry, 2022, 33(12):2301-2309.
[60] NIKAIDO H. Molecular basis of bacterial outer membrane permeability revisited[J]. Microbiology and Molecular Biology Reviews, 2003, 67(4): 593-656.
[61] HUANG J Y, LI C, SONG J N, et al.Regulating polymyxin resistance in gram-negative bacteria:Roles of two-component systems PhoPQ and PmrAB[J].Future Microbiology, 2020, 15(6):445-459.
[62] GRANDE R, PUCA V, MURARO R.Antibiotic resistance and bacterial biofilm[J].Expert Opinion on Therapeutic Patents, 2020, 30(12):897-900.
[63] GADDY J A, ACTIS L A.Regulation of Acinetobacter baumannii biofilm formation[J].Future Microbiology, 2009, 4(3):273-278.