纤维素酶是能够水解纤维素的复合酶系(葡聚糖内切酶、葡聚糖外切酶和β-葡萄糖苷酶),3种酶的协同作用下能够将纤维素最终水解为葡萄糖[1-2]。目前,纤维素酶已广泛应用于食品业、医药业、纺织业、畜牧业以及生物质能源等领域[3-4]。提高纤维素酶活性和纤维素酶的产量,同时降低纤维素酶的使用成本,一直是纤维素酶研究的热点。
纤维素酶主要由微生物产生,这些微生物包括细菌、真菌和放线菌。其中细菌有芽孢杆菌属(Bacillus)、纤维杆菌属(Fibrobacter)、梭菌属(Clostridium)、瘤胃球菌属(Ruminococcus)、小双胞菌属(Microbispora)等。真菌有曲霉属(Aspergillus)、木霉属(Trichoderma)、里氏木霉、青霉属(Penicillium)、粗糙脉胞菌(Neurospora crassa)、白腐菌(Laetiporus sp.)、草酸青霉(Penicillium oxalicum)等。真菌产纤维素酶的能力强,产酶量高,酶解效率高[5-9],但真菌生长速度缓慢,pH和温度的耐受性较差[10-11],其应用存在局限性。细菌与真菌相比抗逆性强,生长快速,能够产生特异性更高的多酶复合物等,易于工业化生产[12-13]。虽然目前筛选分离出大量具有产纤维素酶能力的微生物,但所产纤维素酶依然存在降解效率低、酶活性低及受水解底物抑制等问题。因此,筛选分离纤维素降解能力强的细菌,开发高活性、高稳定性的纤维素酶,对纤维素类资源的利用以及相关工业领域的生产具有重要的意义。
本研究以河南南阳宝天曼自然保护区落叶及小麦秸秆的堆体等样本富集筛选获得的混合菌系为菌源,并从中分离得到1株具有滤纸条崩解能力的菌株,经过基因组测序后,对其相关纤维素降解基因的功能进行分析和纤维素酶活性进行验证。
1 材料与方法
1.1 培养基
LB培养基(g/L):NaCl 10.0、蛋白胨10.0、酵母膏5.0、琼脂15.0。
蛋白胨纤维素(peptone cellulose solution,PCS)培养基(g/L):蛋白胨5.0、酵母粉5.0、NaCl 5.0、球磨纤维素5.0、CaCO3 2.0、K2HPO4 0.5,微量元素溶液0.5 mL[微量元素溶液(g/L):柠檬酸5.0、ZnSO4 5.0、硫酸亚铁铵1.0、CuSO4 0.25、MnSO4 0.05、硼酸0.05、钼酸钠0.05]。
TB培养基(g/L):胰蛋白胨12.0、酵母提取物24.0、K2HPO4 12.54、KH2PO4 2.31,甘油4 mL。
富集培养基(g/L):羧甲基纤维素钠(sodium carboxymethyl cellulose-Na,CMC-Na)2.0、NaCl 5.0、NaNO3 3.0、MgSO4·7H2O 3.0、K2HPO4 1.0、FeSO4·7H2O 0.005、琼脂20.0。
滤纸条培养基(g/L):KH2PO4 1.0、NaCl 5.0、FeCl3 0.01、MgSO4 0.3、NaNO3 2.5、CaCl2 0.1,1 cm×1 cm滤纸条。
上述培养基均在121 ℃,101 kPa灭菌20 min。
1.2 工具酶及试剂
细菌基因组DNA快速提取试剂盒、溶液回收试剂盒,OMEGA公司;LA Taq DNA聚合酶、T4 DNA连接酶、RNase A酶、DNA限制性内切酶、5 000 bp DNA Ladder,TaKaRa 宝生物公司;蛋白Marker、一抗(6×His Tag Monoclonal Antibody)、二抗(Goat anti Mouse IgG),赛默飞世尔科技公司;蛋白快速检测试剂盒、蛋白定量检测试剂盒、异丙基-β-D-硫代吡喃半乳糖苷(isopropyl-β-D-thiogalactoside,IPTG)、卡那青霉素(kanamycin,Kana)、Ni-NTA柱(Pre-Packed Gravity Column,5 mL),生工生物工程(上海)股份有限公司;其他试剂均为国产分析纯。
1.3 纤维素降解菌的筛选与分离
a)将取自河南南阳宝天曼自然保护区落叶及小麦秸秆的堆体等环境样本,CMC富集培养基50 ℃培养过夜,将混合菌梯度稀释后涂布于PCS培养基,50 ℃培养过夜。挑选不同形态的单菌落转接至新的PCS固体培养基和CMC固体培养基,50 ℃培养箱培养过夜。挑选不同形态的单菌落转接至滤纸培养基,50 ℃、180 r/min摇床培养,观察滤纸降解情况。
b)刚果红染色:过夜培养后,PCS培养基作为备份,在长出单菌落的CMC培养基中加入质量浓度为1 mg/mL的刚果红溶液,计时10 min,倒去刚果红染液,加入浓度为1 mol/L NaCl溶液,15 min后观察是否有透明圈。
c)芽胞染色:涂片固定单菌落,滴加适量孔雀绿染液,用木夹夹住载玻片,酒精灯加热,从染液有蒸气冒出时开始计时4~5 min,加热过程中不能使染液蒸干,过程中可酌情添加少量染液。加热完成后待载玻片冷却用蒸馏水冲洗至孔雀绿染液不再褪色,加入番红复染1 min,蒸馏水冲洗、干燥,40倍显微镜观察。
1.4 基因组测序与生物信息学分析
挑取单菌落,利用PCS培养基50 ℃、180 r/min摇床培养,培养到OD600为0.6~0.8,收集菌体,提取基因组,送深圳华大基因利用DNBSEQ平台和PacBio平台对基因组进行测序。
a)基因组测序与组装:利用DNBSEQ平台(BGI)和PacBio平台(BGI)对候选菌株进行建库测序。利用Falcon(v0.3.0,https://github.com/PacificBiosciences/falcon)对PacBio数据进行矫正和组装,利用Pilon对基因组进行单碱基矫正。
b)基因注释;利用Glimmer(v3.02)对基因组进行基因结构注释,基因功能注释分别利用Kyoto Encyclopedia of Genes and Genomes(KEGG,版本:89.1)、Swiss-Prot(release-2019-07)、NR(release-2019-07-27)、Carbohydrate-Active enZYmes Database(CAZy,release-2019-06)进行注释。使用SignalP对蛋白的信号肽进行预测(https://services.healthtech.dtu.dk/services/SignalP-6.0)。
1.5 蛋白表达与纯化
a)重组蛋白表达载体的构建:根据大肠杆菌密码子优化基因合成,将验证正确的PCR产物回收,同时与pET30a质粒均用NdeI和XhoI进行双酶切,酶切总体系200 μL,于37 ℃水浴2 h。进行琼脂糖凝胶电泳,回收酶切产物,DNA连接酶连接载体与片段,连接总体系20 μL,于16 ℃金属浴过夜连接。将连接产物转至DH5α感受态细胞中,涂布于TB(含100 μg/mL Kana)平板,37 ℃恒温培养16 h,挑取转化子,进行PCR验证和酶切验证。将验证正确的重组质粒送至生工公司测序,测序正确的菌株提取质粒保藏。
b)重组蛋白的表达:将测序正确的重组质粒按热休克转化法转化至BL21(DE3)感受态细胞中,涂布于TB(含100 μg/mL Kana)平板,37 ℃培养箱培养过夜,将重组菌株接种于10 mL TB(含100 μg/mL Kana)液体培养基中,37 ℃、200 r/min振荡过夜培养。按照2%(体积分数,下同)的接种量转接至100 mL TB(含100 μg/ mL Kana)液体培养基,放入培养箱中培养至OD600为0.4~0.6,加入0.5 mmol/L IPTG溶液,20 ℃诱导6~8 h后,8 000 r/min离心10 min,收集菌体进行超声波破碎。超声波破碎条件为:功率285 W,工作时间30 min,超声波1 s,停3 s。超声波破碎完成后,4 ℃、13 000 r/min离心10 min,上清液即粗酶液。
c)重组蛋白表达条件优化:采取单一变量控制法对蛋白诱导表达条件进行逐一优化。优化条件包括:菌液OD600值分别为0.2、0.4、0.6、0.8、1.0,IPTG诱导浓度分别为0.1、0.2、0.4、0.6、0.8、1.0、1.2 mmol/L,诱导时间分别为2、4、6、8、10、12、22 h,诱导温度分别为20、28、37 ℃,根据蛋白表达情况,筛选出最佳诱导条件。
d)蛋白的纯化:将重组菌株接种于10 mL TB(含100 μg/mL Kana)液体培养基中,37 ℃、200 r/min振荡过夜培养,按照2%的接种量转接于100 mL TB(含100 μg/mL Kana)液体培养基,放入37 ℃、200 r/min摇床培养至OD600为0.4,加入0.1 mmol/L IPTG溶液,20 ℃、200 r/min诱导表达过夜,8 000 r/min离心10 min收集菌体,加入40 mL 50 mmol/L pH 8.0的Tris-HCl缓冲液复溶,冰置超声波破碎,超声波破碎条件为:功率285 W,工作时间30 min,超声波1 s,停3 s。超声波破碎完成后,4 ℃、8 000 r/min离心10 min,取上清液即得到粗酶液。用0.22 μm滤膜过滤粗酶液后,重复上样3次,用20 mmol/L咪唑(20 mmol/L Tris、500 mmol/L NaCl,20 mmol/L咪唑,pH值为8.0)洗去杂蛋白,用100 mmol/L咪唑(20 mmol/L Tris、500 mmol/L NaCl,100 mmol/L咪唑,pH值为8.0)洗脱10倍柱体积,用200 mmol/L咪唑(20 mmol/L Tris、500 mmol/L NaCl,200 mmol/L咪唑,pH值为8.0)洗脱10倍柱体积,进行SDS-PAGE检测和以His为特异性标签的Western blot检测。
1.6 纤维素酶活力的测定
a)纤维素内切酶活性:采用DNS法[14]测定纤维素酶活性,取100 μL配制好的10 g/L CMC-Na溶液和50 μL粗酶液充分混匀,空白组不加粗酶液,37 ℃水浴30 min,在空白管中加入粗酶液,加入150 μL DNS试剂150 μL,沸水浴10 min,冷却,用蒸馏水定容至1 250 μL充分混匀,于波长540 nm处测定吸光值。
b)外切酶活性:取100 μL配制好的10 g/L微晶纤维素溶液和50 μL粗酶液充分混匀,空白组不加粗酶液,37 ℃水浴30 min,在空白管中加入粗酶液,加入150 μL DNS试剂,沸水浴10 min,冷却,用蒸馏水定容至1 250 μL充分混匀,于波长540 nm处测定吸光值。
c)β-葡萄糖苷酶活性:取100 μL配制好的10 g/L水杨苷溶液和50 μL粗酶液充分混匀,空白组不加粗酶液,37 ℃水浴30 min,在空白管中加入粗酶液,加入150 μL DNS试剂,沸水浴10 min,冷却,用蒸馏水定容至1 250 μL充分混匀,于波长540 nm处测定吸光值。
d)酶活力定义:在一定条件下,将在30 min内催化纤维素水解生成1 μmol葡萄糖所使用的酶量称为1个酶活力单位(U)。
1.7 酶学性质研究
a)最适pH测定:分别用pH值为4.0、5.0、6.0、7.0、8.0、9.0、10.0的缓冲液配制10 g/L CMC-Na溶液作为不同pH值条件下的酶作用底物,柠檬酸缓冲液(pH值为4.0、5.0、6.0),Tris-HCl缓冲液(pH值为7.0、8.0、9.0),Gly-NaOH(pH值为10.0),加入纯酶液,于37 ℃水浴保温30 min,测定酶活性,确定最适pH值。
b)最适反应温度测定:用柠檬酸缓冲液(最适pH)配制10 g/L CMC-Na溶液作为酶的反应底物,加入纯酶液,以5 ℃为梯度,分别在20、25、30、35、40、45、50、55、60、65 ℃条件下,水浴保温30 min,测定酶活性,确定最适反应温度。
c)酶的温度稳定性测定:分别在30、35、40、45、50、55、60、65 ℃条件下,将纯酶液水浴保温30 min后,用柠檬酸缓冲液(最适pH值)配制10 g/L CMC-Na溶液作为酶的反应底物,加入纯酶液,55 ℃水浴保温30 min,测定酶活性,以4 ℃保存的酶活性定义为100%,测定酶的温度稳定性。
d)金属离子对酶活性的影响:以分别含有10 mmol/L 的CaCl2、MgCl2、MnSO4、CoCl2、CuSO4、NiSO4、ZnCl2、K2SO4、NH4Cl的10 g/L CMC-Na溶液作为酶的反应底物,在最适pH和最适温度条件下保温30 min后,测定酶活性。以不加任何无机盐的1% CMC-Na溶液作为底物所测出来的酶活力值为100%相对酶活力,测定酶的金属离子对酶活性的影响。
e)Km值测定:以10种不同浓度CMC-Na作为底物,加入相同的Gene2269纯酶进行酶促反应。以底物浓度的倒数1/S为横坐标,以葡萄糖的产生速率的倒数1/V为纵坐标,得到双倒数曲线。
1.8 数据处理
实验数据均平行测定3次,采用Excel 2019软件处理数据,采用SPSS 22.0进行单因素方差分析(ANOVA),采用Origin 2024软件作图。
2 结果与分析
2.1 纤维素降解菌的分离与筛选
将筛选分离得到这株菌株命名为I2。I2经纤维素固体平板刚果红染色法进行初筛,显示具有纤维素酶活性(图1-a),滤纸条培养基降解实验显示I2具有较强降解滤纸的能力,48 h可以降解大部分滤纸(图1-b)。革兰氏染色和芽孢染色制片实验结果表明,I2为革兰氏阳性菌有芽孢(图1-c、图1-d)。
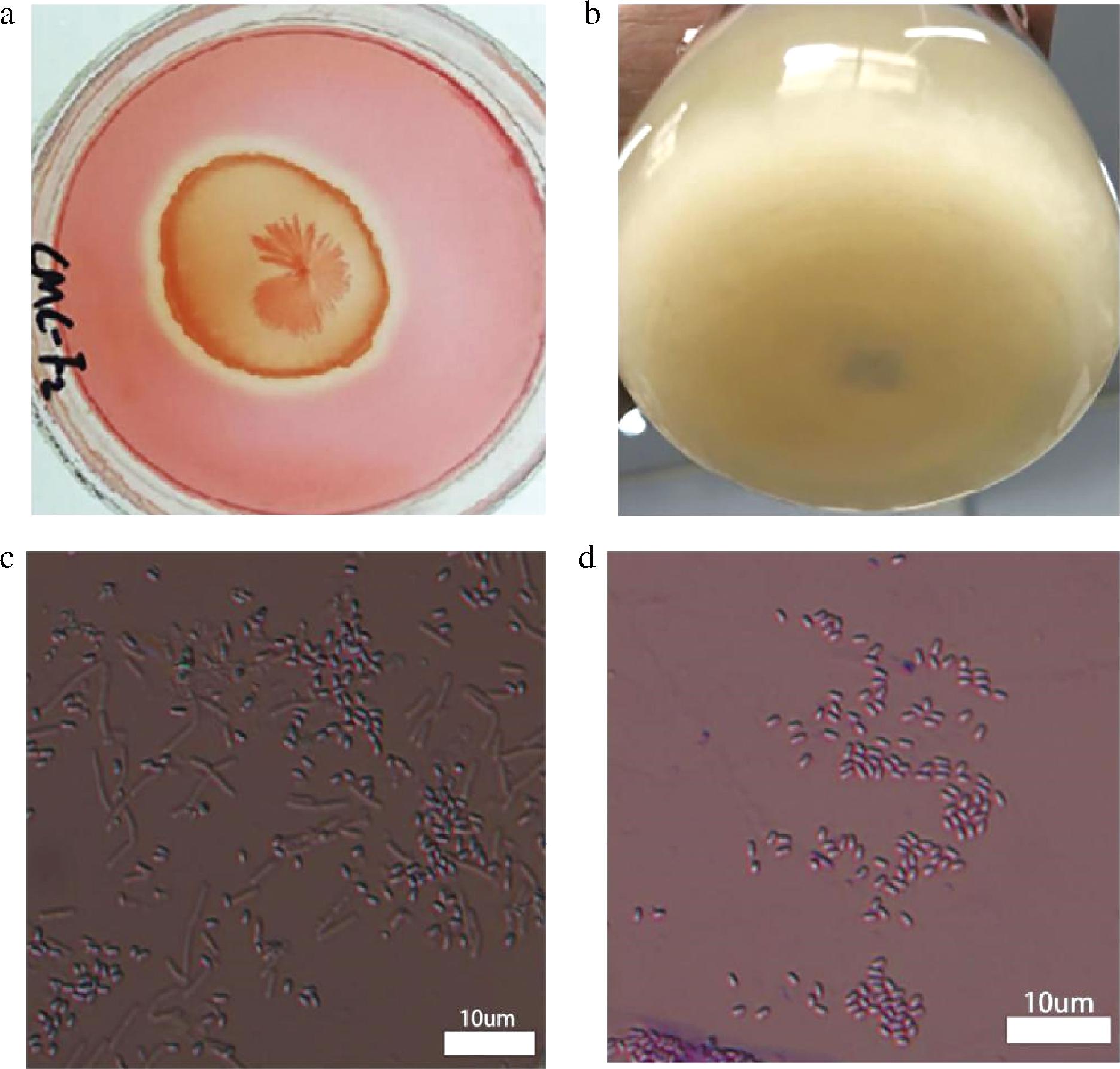
a-I2刚果红染色;b-I2菌株48 h降解滤纸情况;c-I2芽孢染色电镜图;d-I2革兰氏染色电镜图
图1 纤维素降解菌I2的分离与染色
Fig.1 Isolation and staining of cellulose degrading bacterium I2
2.2 I2基因组解析与纤维素降解相关基因预测
对I2基因组进行从头测序、组装,并在组装的基础上,进行基因组组分、功能注释、比较基因组学等分析。I2基因组大小4.3 Mbp,不含有质粒,GC含量为46.84%,含有4 635个基因,基因平均长度813.81 bp,基因长度占基因组总长的比例为87.55%,Blast比对结果为芽孢杆菌属的短小芽孢杆菌。
对I2的蛋白集用CAZy碳水化合物活性相关酶数据库进行注释,得到182个相关基因。这些基因中糖苷水解酶(glycoside hydrolases,GHs)79个,糖基转移酶(glycosyl transferases,GTs)32个,多糖裂解酶(polysaccharide lyases,PLs)9个,糖类酯解酶(carbohydrate esterases,CEs)21个,辅助功能(auxiliary activities,AAs)2个,碳水化合物相关模块(carbohy-drate-binding modules,CBM)39个(表1)。根据这些蛋白功能注释信息,利用关键词glucanase搜索,得到4个可能参与纤维素降解的基因,分别为gene1878、gene1879、gene1880和gene2269,均为糖苷水解酶基因。经与Swissport数据库比对,上述4个基因编码的蛋白可能具有纤维素外切酶、纤维素内切酶活性等,且蛋白功能均未被鉴定。Gene1878、Gene1879、Gene1880和Gene2269(以下简写为G1878、G1879、G1880和G2269)的蛋白信息如表2所示。
表1 I2蛋白集碳水化合物数据库(CAZy)注释结果
Table 1 Annotation results of I2 protein carbohydrate database (CAZy)
基因家族GHsGTsCBMCEsPLsAAs总数基因数量7932392192182
表2 纤维素相关基因注释结果
Table 2 Annotation results of cellulose related genes
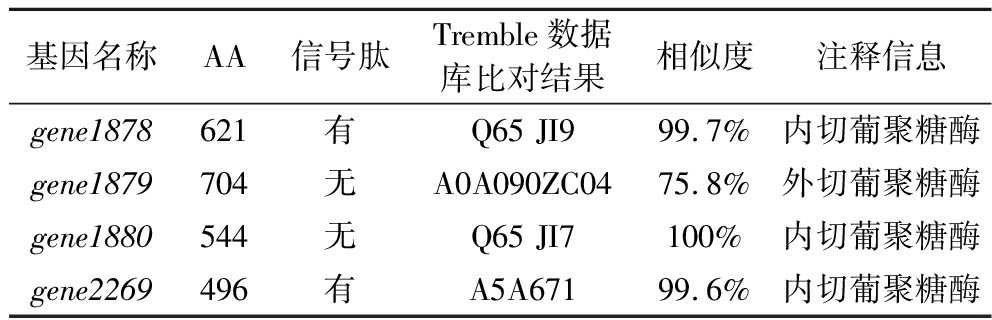
基因名称AA信号肽Tremble数据库比对结果相似度注释信息gene1878621有Q65 JI999.7%内切葡聚糖酶gene1879704无A0A090ZC0475.8%外切葡聚糖酶gene1880544无Q65 JI7100%内切葡聚糖酶gene2269496有A5A67199.6% 内切葡聚糖酶
2.3 纤维素酶的功能验证
根据G1878、G1879、G1880和G2269的蛋白序列,参考大肠杆菌密码子,对gene1878、gene1879、gene1880和gene2269进行去除信号肽后的全基因合成,并克隆至表达载体pET30a。重组质粒转化到大肠杆菌表达菌BL21(DE3)中后,分别获得重组大肠杆菌工程菌。经IPTG诱导,SDS-PAGE检测,上述4个重组蛋白在大肠杆菌中获得表达,在相同培养和诱导条件下,G1878、G1879、G1880的蛋白表达量相对较高,G2269的表达量相对较低(图2)。
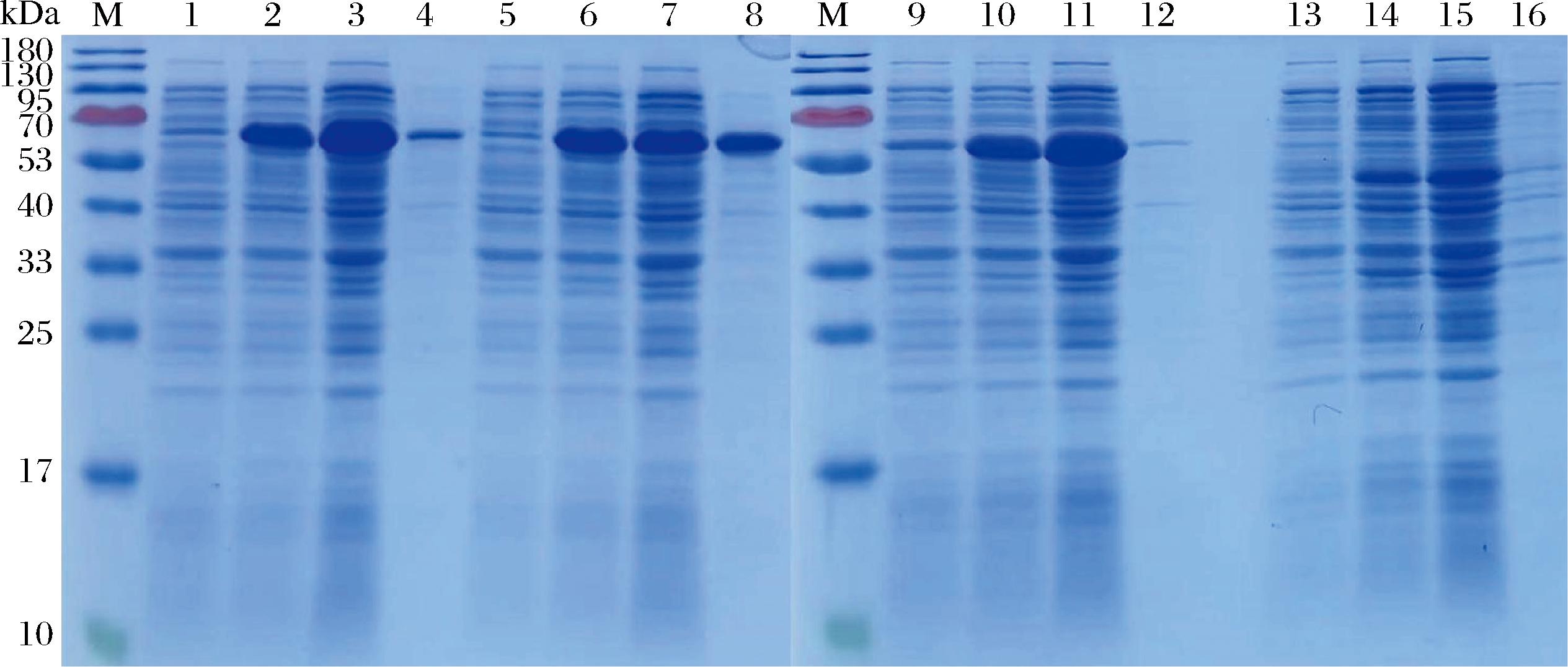
M-蛋白分子标准;1~4-G1878诱导前、诱导后,超声波破碎后的上清液和沉淀;5~8-G1879诱导前、诱导后,超声波破碎后的上清液和沉淀;9~12-G1880诱导前、诱导后,超声波破碎后的上清液和沉淀;13~16-G2269诱导前、诱导后,超声波破碎后的上清液和沉淀
图2 纤维素降解菌I2重组蛋白的诱导表达
Fig.2 Induced expression of recombinant protein from cellulose degrading bacterium I2
将诱导表达的蛋白进行超声波破碎,得到的上清液即为粗酶液,分别以CMC-Na、微晶纤维素、水杨苷为底物,测定粗酶液的纤维素内切酶、外切酶和β-葡萄糖苷酶的活性。如图3所示,4个蛋白均具有纤维素内切酶、外切酶和β-葡萄糖苷酶活性。其中,G2269纤维素内切酶活性最高,G1880纤维素外切酶活性最高,4个蛋白的β-葡萄糖苷酶活性相差不大。根据粗酶液的活性测定,G2269的蛋白表达量最低,而纤维素内切酶活性最高,因此后续将对G2269的表达条件进行优化,并对其酶学性质进行详细研究。
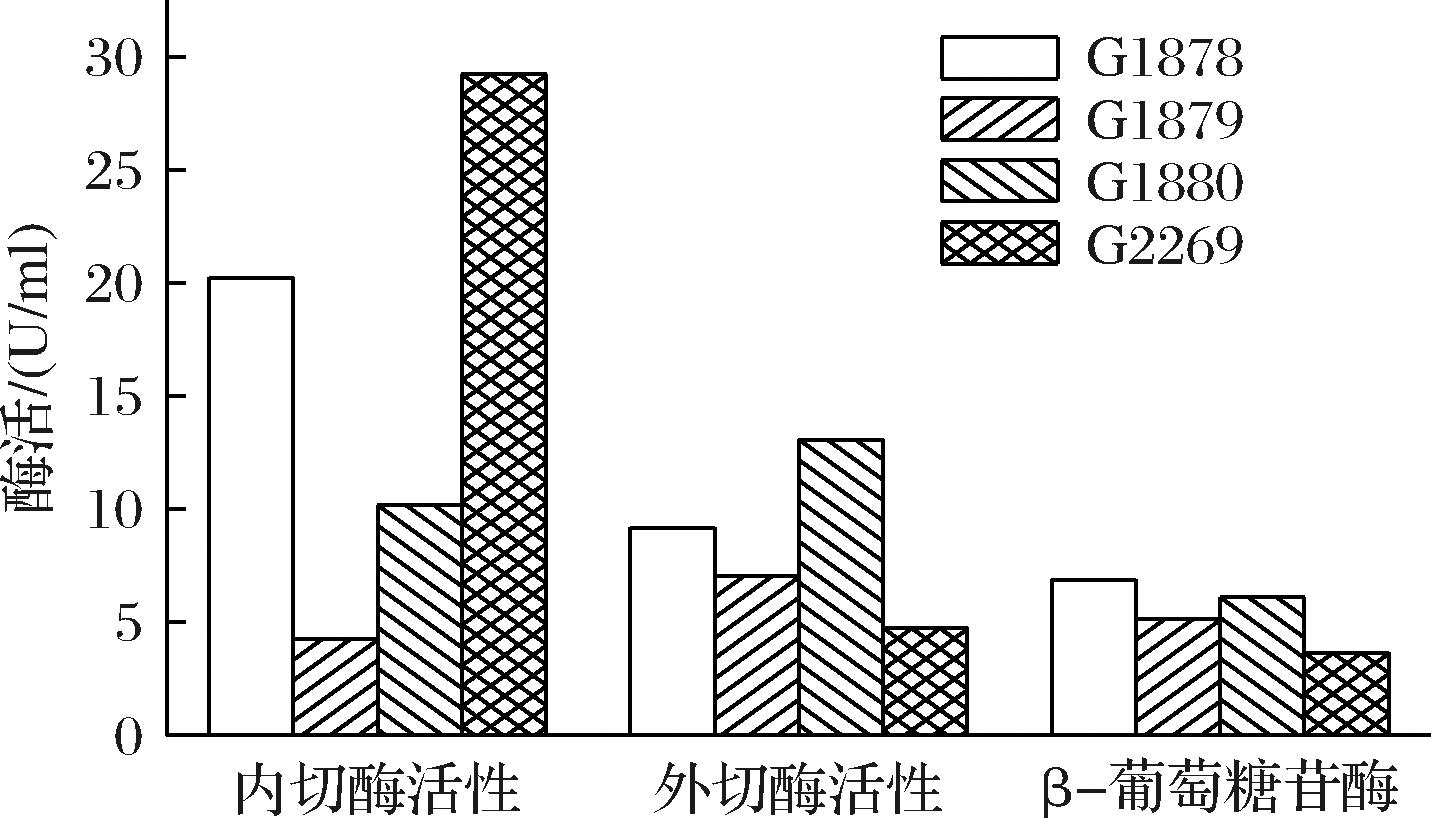
图3 纤维素降解菌I2中纤维素酶活测定
Fig.3 Determination of cellulase activity in cellulose degrading bacterium I2
2.4 纤维素内切酶G2269表达条件优化
表达条件优化是获得高表达产量和可溶性蛋白的关键,采取单一变量控制法对纤维素水解酶G2269的蛋白诱导表达条件(菌液诱导浓度、诱导温度、诱导剂浓度、和诱导时间)进行逐一优化。经SDS-PAGE检测后,实验结果如图4所示,G2269蛋白OD600值的最适菌液浓度是0.4(图4-a);最佳IPTG终浓度为0.1 mmol/L(图4-b);最佳诱导温度20 ℃(图4-c);最佳诱导时间为10 h(图4-d),在此条件下G2269蛋白表达量最高。
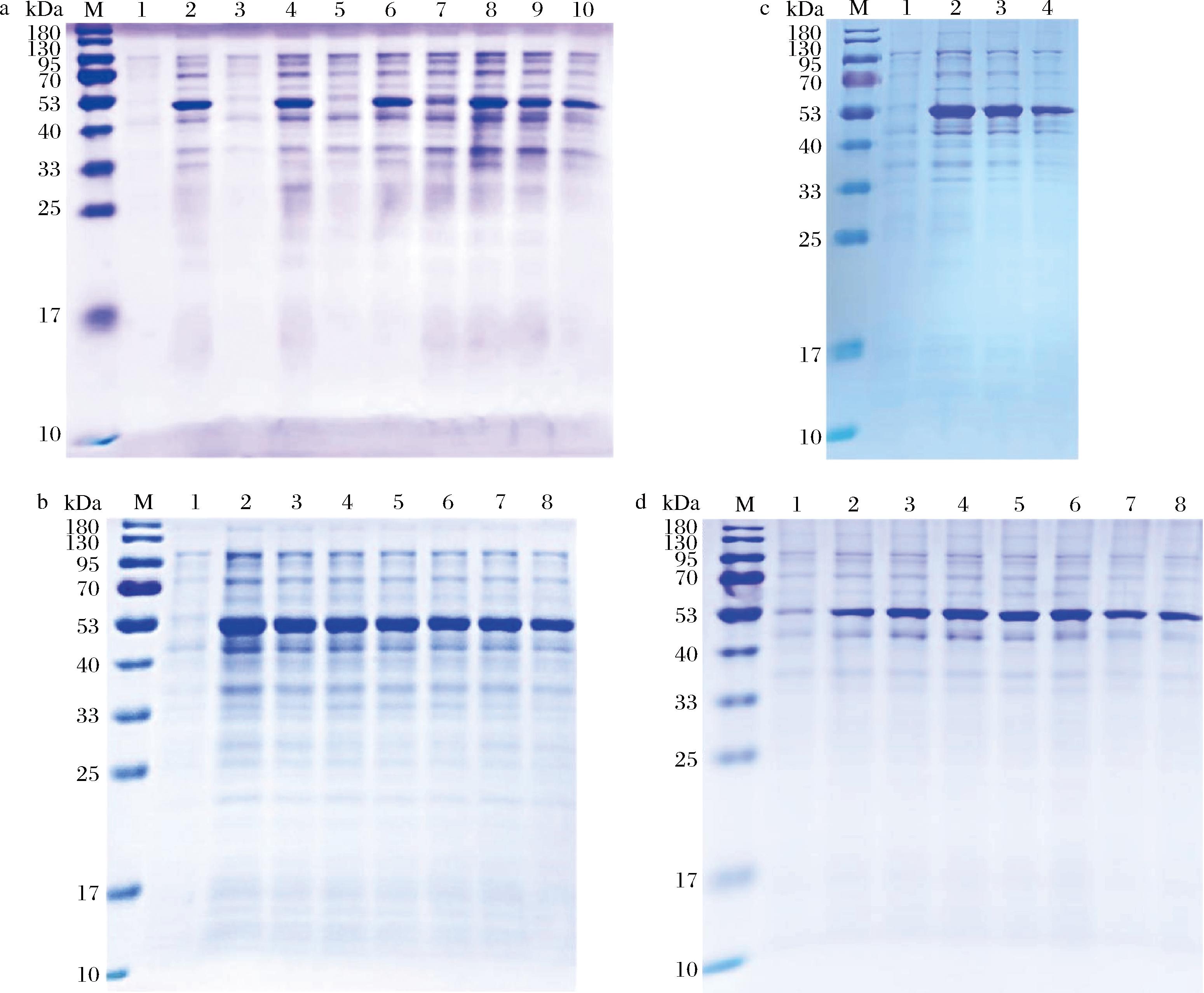
M-蛋白分子标准a-G2269不同菌液浓度的电泳图(1、2-蛋白OD600为0.2时诱导前、诱导后;3、4-蛋白OD600为0.4时诱导前、诱导后;5、6-蛋白OD600为0.6时诱导前、诱导后;7、8-蛋白OD600为0.8时诱导前、诱导后;9、10-蛋白OD600为1.0时诱导前、诱导后);b-G2269不同IPTG浓度电泳图(1-诱导前;2~8-IPTG诱导浓度分别为0.1、0.2、0.4、0.6、0.8、1.0、1.2 mmol/L);c-G2269不同诱导温度的电泳图(1-诱导前; 2、3、4-20、28、37 ℃条件下诱导);d-G2269不同诱导时间的电泳图(1-诱导前;2~8-IPTG诱导时间分别为2、4、6、8、10、12、22 h)
图4 纤维素酶G2269表达条件优化
Fig.4 Optimization of expression conditions for cellulase G2269
2.5 纤维素内切酶G2269的纯化及相关酶学性质研究
利用Ni-NTA柱对G2269蛋白进行纯化,SDS-PAGE检测结果如图5-a所示,目的蛋白经Ni-NTA亲和层析能检测到较高的纯度,实际大小与理论值相符(52 kDa)。以His为特异性抗体的Western blot对G2269蛋白进行鉴定,证实重组融合蛋白得到了适当的纯化(图5-b),表明纤维素酶基因在c端(His)6标签区成功构建。
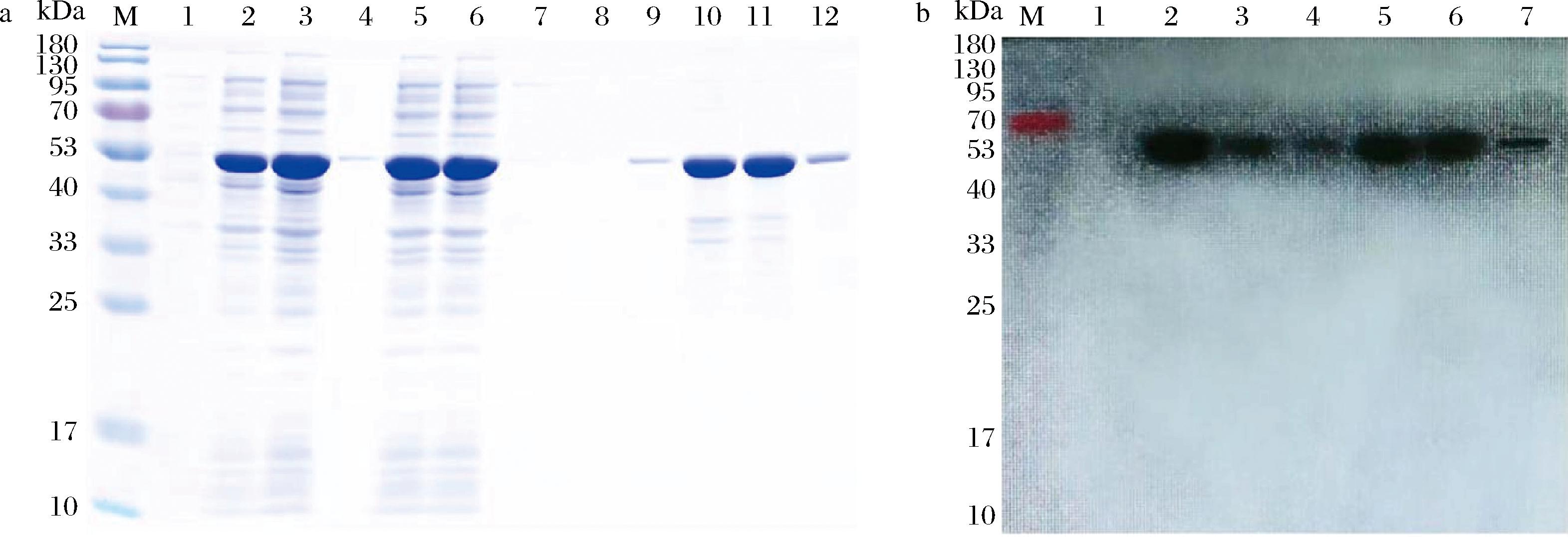
M-蛋白分子标准a-G2269的蛋白纯化电泳图(1~2-诱导前、诱导后;3~4-超声波后的上清液、沉淀;5~6-流穿液;7-洗杂液;8~11-100 mmol/L的4管洗脱液;12-200 mmol/L的洗脱液);b-G2269以His为特异性抗体的WB图(1-诱导前;2-超声波破碎后的上清液;3-流穿液;4-洗 杂液;5~6-100 mmol/L的2管洗脱液;7-200 mmol/L的洗脱液)
图5 纤维素酶G2269蛋白纯化及Western blot鉴定
Fig.5 Cellulase G2269 protein purification and Western blot identification
对纯化后的G2269进行酶学性质研究(图6),G2269纤维素内切酶最适pH值为5.0(图6-a),最适反应温度为55 ℃(图6-b)。添加10 mmol/L金属离子Ca2+、Zn2+,G2269的活性分别提高了1.36、1.41倍;而Mn2+和Cu2+对G2269活性有较高的抑制作用,酶活力分别降低到原有酶活力的15%、23%;Mg2+、Co2+、K+、Ni2+、NH4+对于G2269的活性无显著影响,酶活力分别为原有酶活力的89%、99%、96%、74%、65%(图6-c)。纤维素内切酶G2269温度稳定性较高,在0~65 ℃范围内,随着温度的提高,稳定性逐渐下降,在65 ℃保持0.5 h后依旧保留有70%的相对酶活力(图6-d)。原始的粗酶液的比活力为0.88 U/mg,纯酶的比活力为2.88 U/mg,纯化后酶的比活力提升了3倍,经米氏方程计算得出,以CMC-Na为底物测定G2269纯酶液的纤维素内切酶活性Km值为35.70 mol/L。
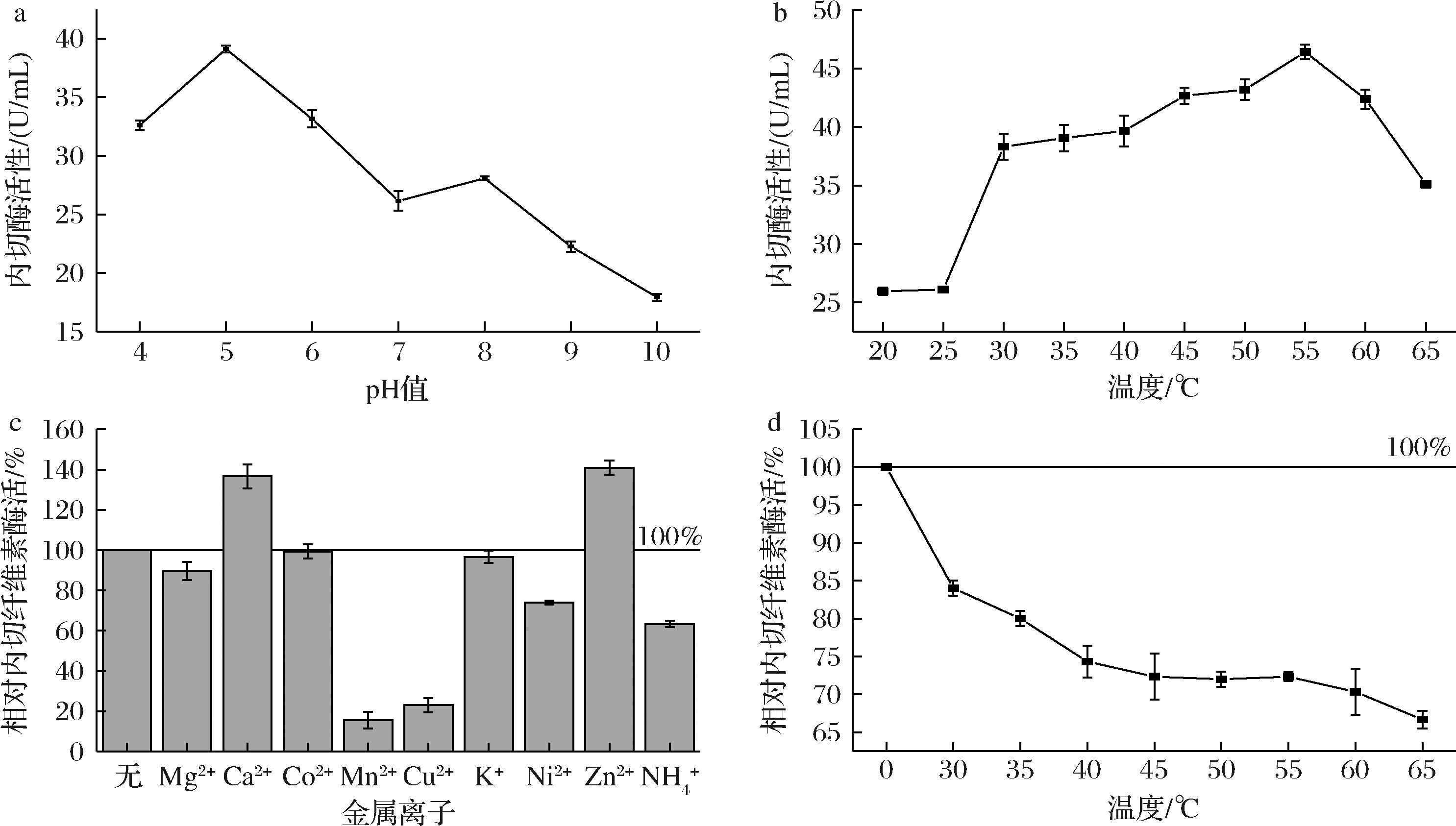
a-pH;b-反应温度;c-金属离子;d-温度
图6 纤维素酶G2269酶活性质研究
Fig.6 The enzyme activity characteristic of cellulase G2269
3 讨论与结论
本研究以长期堆积的秸秆堆积物为样本,分离筛选出1株能快速降解纤维素、有滤纸降解能力的芽孢杆菌I2,通过基因组测序和注释分析获得了该菌的全部纤维素降解相关的基因,通过密码子优化和基因合成,克隆至表达载体pET30a构建重组质粒,转化到大肠杆菌表达菌BL21(DE3)中进行表达,并鉴定了4个纤维素酶基因的功能,获得的4个蛋白均具有纤维素内切酶、外切酶及β-葡萄糖苷酶的活性,其中纤维素酶内切酶G2269与目前报道的纤维素酶相比具有以下特点。一是酶活力高。SONG等[15]在强启动子和合成先导子的控制下将纤维素酶基因进行密码子优化,其中菌株SK10-3纤维素内切酶的比活性最高为2.55 U/g。AMRAINI等[16]利用外源纤维素酶基因构建重组大肠杆菌EgRK2,其纤维素酶活力为1.905 U/mL。相较于其他学者研究获得的纤维素酶,本研究获得的纤维素酶G2269,纯酶的内切纤维素酶活力为49.02 U/mL,纯酶比活力可达2.88 U/mg,高于一般重组纤维素酶,表明其产内切纤维素酶能力较优。二是环境适应性强。KIM等[17]将地衣芽孢杆菌ATCC 14580的纤维素酶基因与冰核蛋白基因进行重组,纤维素酶的最佳条件为50 ℃和pH值为7.0。许颖等[18]将绵羊瘤胃元基因组的内切纤维素酶在毕赤酵母GS115表达优化,内切纤维素酶活力为9.26 U/mL,最适pH值为7.0。本研究获得的纤维素酶G2269纯酶液的最适pH值为5.0,在pH值为4.0~8.0时仍保留70%的相对酶活力,具有更为广泛的环境pH耐受性,产纤维素酶的细菌主要是分离自中性、碱性的土壤表层,以及河流、湖泊和海洋中,酸性纤维素酶较为稀少,研究表明纤维素经过酸性预处理可以提高纤维素的降解效率[19],因此酸性纤维素酶和酸处理协同处理更具有优势,同时纤维素酶G2269最适反应温度为55 ℃,在65 ℃保持0.5 h后依旧保留有60%的相对酶活力,有较高的热稳定性,对环境具有较强的耐受能力和适应性。三是纤维素的降解是在内切酶、外切酶和β-葡萄糖苷酶的多种酶系协同作用下进行分解的[20],仅通过单一菌株很难完成降解过程,本研究纤维素酶G2269的内切酶活性较高为49.02 U/mL,而外切酶和β-葡萄糖苷酶活性均为4~5 U/mL,后期可以研究纤维素酶的协同作用增强纤维素酶G2269的外切酶和β-葡萄糖苷酶活力,进一步提升纤维素酶的降解效果[21]。
纤维素酶作为蛋白质,其良好的热稳定性和贮存稳定性是大规模生产和商品化的关键,本研究获得的纤维素内切酶具有良好热稳定性和酸碱稳定性,具备在食品、纺织、造纸、饲料及能源等行业中的应用前景。
[1] HARVEY D G.On the metabolism of some aromatic nitro compounds by different species of animal.III.The toxicity of the dinitrophenols, with a note on the effects of high environmental temperatures[J].Journal of Pharmacy and Pharmacology, 1959, 11:462-474.
[2] SHARMA A, TEWARI R, RANA S S, et al.Cellulases:Classification, methods of determination and industrial applications[J].Applied Biochemistry and Biotechnology, 2016, 179(8):1346-1380.
[3] AREESHI M Y.Microbial cellulase production using fruit wastes and its applications in biofuels production[J].International Journal of Food Microbiology, 2022, 378:109814.
[4]  UCHOV
UCHOV K, FEHÉR C, RAVN J L, et al.Cellulose- and xylan-degrading yeasts:Enzymes, applications and biotechnological potential[J].Biotechnology Advances, 2022, 59:107981.
K, FEHÉR C, RAVN J L, et al.Cellulose- and xylan-degrading yeasts:Enzymes, applications and biotechnological potential[J].Biotechnology Advances, 2022, 59:107981.
[5] 邹潇潇, 易子霆, 孙前光, 等.纤维素降解真菌DF14101的筛选与鉴定[J].微生物学杂志, 2016, 36(6):68-72.
ZOU X X, YI Z T, SUN Q G, et al.Screening and identification of a cellulose-degrading fungus DF14101[J].Journal of Microbiology, 2016, 36(6):68-72.
[6] KUMARI D, SINGH R.Pretreatment of lignocellulosic wastes for biofuel production:A critical review[J].Renewable and Sustainable Energy Reviews, 2018, 90:877-891.
[7] 张新杰, 周兴瑞, 孙慧颖, 等.拮抗植物病原真菌的纤维素/木质素降解菌株的筛选及鉴定[J].微生物学通报, 2023, 50(1):251-261.
ZHANG X J, ZHOU X R, SUN H Y, et al.Screening and identification of cellulose/lignin-degrading strains against plant pathogenic fungi[J].Microbiology China, 2023, 50(1):251-261.
[8] BEHERA B C, SETHI B K, MISHRA R R, et al.Microbial cellulases-diversity &biotechnology with reference to mangrove environment:A review[J].Journal of Genetic Engineering and Biotechnology, 2017, 15(1):197-210.
[9] AWASTHI M K, PANDEY A K, KHAN J, et al.Evaluation of thermophilic fungal consortium for organic municipal solid waste composting[J].Bioresource Technology, 2014, 168:214-221.
[10] 白长胜, 刘秋瑾, 尹珺伊, 等.产木质纤维素降解酶真菌的筛选及产酶特性[J].微生物学通报, 2023, 50(3):1098-1110.
BAI C S, LIU Q J, YIN J Y, et al.Screening and enzymatic characterization of the fungal strains producing lignocellulose-degrading enzymes[J].Microbiology China, 2023, 50(3):1098-1110.
[11] 李子婧, 刘帆, 汤胜, 等.纤维素降解菌长枝木霉菌(Trichoderma longibrachiatum)ZJ-10的筛选及产酶条件优化[J].浙江大学学报(农业与生命科学版), 2022, 48(5):614-624.
LI Z J, LIU F, TANG S, et al.Screening of cellulose-degrading fungus Trichoderma longibrachiatum ZJ-10 and optimization of enzyme production conditions[J].Journal of Zhejiang University (Agriculture and Life Sciences), 2022, 48(5):614-624.
[12] CAWOY H, MARIUTTO M, HENRY G, et al.Plant defense stimulation by natural isolates of Bacillus depends on efficient surfactin production[J].Molecular Plant-Microbe Interactions, 2014, 27(2):87-100.
[13] JIN Q, JIANG Q Y, ZHAO L, et al.Complete genome sequence of Bacillus velezensis S3-1, a potential biological pesticide with plant pathogen inhibiting and plant promoting capabilities[J].Journal of Biotechnology, 2017, 259:199-203.
[14] 董自星, 李伟国, 佟新新, 等.黑曲霉内切β-1, 3(4)-葡聚糖酶的基因克隆与酶学特性分析[J].食品与发酵工业, 2016, 42(11):58-64.
DONG Z X, LI W G, TONG X X, et al.Gene cloning and biochemical characterization of endo-1, 3 (4)-β-glucanases from Aspergillus niger[J].Food and Fermentation Industries, 2016, 42(11):58-64.
[15] SONG X F, LI Y Z, WU Y Z, et al.Metabolic engineering strategies for improvement of ethanol production in cellulolytic Saccharomyces cerevisiae[J].FEMS Yeast Research, 2018, 18(8):pp620.
[16] AMRAINI S Z, ARIYANI L P, HERMANSYAH H, et al.Production and characterization of cellulase from E.coli EgRK2 recombinant based oil palm empty fruit bunch[J].Biotechnology and Bioprocess Engineering, 2017, 22(3):287-295.
[17] KIM D, KU S.Bacillus cellulase molecular cloning, expression, and surface display on the outer membrane of Escherichia coli[J].Molecules, 2018, 23(2):503.
[18] 许颖, 刘智磊, 詹晓北, 等.毕赤酵母中内切β-1, 4葡聚糖酶的表达及共培养制备低分子质量黄原胶[J].食品与发酵工业, 2023, 49(9):1-8.
XU Y, LIU Z L, ZHAN X B, et al.Endo-β-1, 4-glucanase expression in Pichia pastoris for coupled fermentation with Xanthomonas campestris for low-molecular-weight xanthan gum preparation[J].Food and Fermentation Industries, 2023, 49(9):1-8.
[19] 董茂林, 陈李栋, 黄六莲, 等.酸性助水溶剂制备木质纳米纤维素及功能应用研究进展[J].化工学报, 2023, 74(6):2281-2295.
DONG M L, CHEN L D, HUANG L L, et al.Research progress in preparation of lignonanocellulose by acid hydrotropes and their functional applications[J].CIESC Journal, 2023, 74(6):2281-2295.
[20] KALOGEROPOULOU A, PLIONI I, DIMITRELLOU D, et al.Biosynthesis of fuel-grade ethanol from cellobiose by a cell-factory of non-GMO Saccharomyces cerevisiae/starch-gel-cellulase[J].Fuel, 2022, 313:122986.
[21] WANG L, XIE Y J, CHANG J J, et al.A novel sucrose-inducible expression system and its application for production of biomass-degrading enzymes in Aspergillus niger[J].Biotechnology for Biofuels and Bioproducts, 2023, 16(1):23.