L-高丝氨酸是多种必需氨基酸(特别是L-蛋氨酸和L-苏氨酸)生物合成途径中的重要中间产物,在食品、饲料、医药和农业等领域中具有重要应用前景[1-3]。目前,L-高丝氨酸已成为生产各种重要平台化合物的基本中间体,例如L-高丝氨酸内酯、γ-丁内酯、四氢呋喃和1,3-丙二醇[2]。此外,有研究表明,L-高丝氨酸及其衍生物具有免疫刺激特性,能够有助于改善植物和哺乳动物的生长性能[4]。由于L-高丝氨酸与草铵膦具有相同的化学支架,因此其在光学纯L-草铵膦的合成中具有重要应用价值。草铵膦作为使用最广泛的非选择性除草剂之一,在现代农业和杂草管理实践中备受推崇[5]。因此,L-高丝氨酸的绿色生物制造将为活性L-草铵膦的高效合成提供有前景的替代方案。
由于化学合成过程昂贵且复杂,L-高丝氨酸大规模生产尚未实现商业化[1]。近年来,微生物发酵作为一种经济可行且环境友好的方式,逐渐成为高效生产L-高丝氨酸的关注焦点。目前,大多数研究集中在大肠杆菌底盘细胞上,该菌株被证实是生产L-高丝氨酸的最有效宿主之一[2, 3, 6-10]。VO等[11]研究通过对大肠杆菌 W3110进行迭代遗传改造,成功开发了大肠杆菌高效微生物细胞工厂,最终获得了110.8 g/L L-高丝氨酸滴度,生产强度达到 1.82 g/L/h。LIU等[8]通过实施涉及合成途径工程与细胞分裂调节相结合的策略,在大肠杆菌中实现了最佳的L-高丝氨酸生产强度,最终的工程菌株可积累101.31 g/L L-高丝氨酸,生产强度为 1.91 g/(L·h)。总体而言,许多研究人员仍然致力于改进用于L-高丝氨酸生产的大肠杆菌发酵特性,并不懈地追求提高其整体效率和生产能力。
在工业微生物中,谷氨酸棒杆菌在氨基酸的大规模发酵生产方面拥有悠久而成功的历史。然而,目前改造谷氨酸棒杆菌以高效生产L-高丝氨酸的案例仍相对较少[1]。LI等[12]通过重新分配谷氨酸棒杆菌代谢流,使其成为有前途的生产L-高丝氨酸菌株,工程菌在发酵培养基中积累了8.8 g/L L-高丝氨酸。随后的研究中,作者基于CRISPR基因组编辑系统获得一种高效产L-高丝氨酸的谷氨酸棒杆菌,在5 L生物反应器可积累22.1 g/L L-高丝氨酸[13]。此外,通过基于双通道糖酵解的辅因子供应策略,改造获得的菌株在5 L生物反应器中发酵96 h后可积累63.5 g/L L-高丝氨酸[14]。尽管在谷氨酸棒杆菌中发酵生产L-高丝氨酸方面已经取得了一些进展,但其总体滴度仍远远落后于大肠杆菌中所达到的水平。鉴于谷氨酸棒杆菌具有高效氨基酸发酵的强大潜力,未来仍需继进一步改造提升其发酵生产L-高丝氨酸性能。因此,在本研究中,以野生型谷氨酸棒杆菌为出发菌,通过一系列遗传改造和发酵测试,改善和提升了其发酵生产L-高丝氨酸的能力,可为L-高丝氨酸绿色生物制造提供新的底盘工程菌。
1 材料与方法
1.1 材料
1.1.1 菌株和质粒
本研究中大肠杆菌(Escherichia coli DH5α)和谷氨酸棒杆菌(Corynebacterium glutamicum ATCC13032)由中国科学院天津工业生物技术研究所保藏。大肠杆菌-谷氨酸棒杆菌穿梭质粒pXMJ19用于基因表达,由本实验室保藏。温度敏感敲除质粒pCRD206用于谷氨酸棒杆菌基因敲除和敲入,由本实验室保藏。所用菌株与质粒见表1。
表1 本研究所用菌株与质粒
Table 1 Bacteria and plasmids used in this study

菌株和质粒相关描述来源DH5αE.coli derivative;competent cells for general cloning实验室保藏ATCC 13032C.glutamicum wild-type strain实验室保藏HOM1C.glutamicum derivative;ΔthrB, Psod-lysCC932T, Psod-homG1133A本研究构建HOM2C.glutamicum HOM1 derivative;Psod-ppc本研究构建HOM3C.glutamicum HOM1 derivative;PH7-aceAB本研究构建HOM4C.glutamicum HOM2 derivative;ΔalaT,Δddh本研究构建HOM5C.glutamicum HOM4 derivative;Δpck::Pself-EcPntAB本研究构建HOM6C.glutamicum HOM4 derivative;Δpck::Pgap-EcPntAB本研究构建HOM7C.glutamicum HOM6 derivative;Δldh::Ptac-EcrhtAmut本研究构建HOM8C.glutamicum HOM7 derivative;ΔptsG::Ptuf-Bsglk-Pgap-ppgk本研究构建pXMJ19C.glutamicum-E.coli shuttle expression vector, Cmr实验室保藏pCRD206Temperature-sensitive replicon and Bacillus subtilissacB gene, Kanr实验室保藏pXMJ19-NCgl2732pXMJ19 derivate;containing the NCgl2732-NCgl2733 genes from C.glutamicum本研究构建pXMJ19-NCgl0029pXMJ19 derivate;containing the NCgl0029-NCgl0031 genes from C.glutamicum本研究构建pXMJ19-NCgl0213pXMJ19 derivate;containing the NCgl0213gene from C.glutamicum本研究构建pXMJ19-NCgl0694pXMJ19 derivate;containing the NCgl0694-NCgl0698genes from C.glutamicum本研究构建pXMJ19-NCgl1329pXMJ19 derivate;containing the NCgl1329-NCgl1332 genes from C.glutamicum本研究构建pXMJ19-NCgl2373pXMJ19 derivate;containing the NCgl2373-NCgl2377 genes from C.glutamicum本研究构建pXMJ19-NCgl1862pXMJ19 derivate;containing the NCgl1862 gene from C.glutamicum本研究构建pXMJ19-NCgl1858pXMJ19 derivate;containing the NCgl1858 gene from C.glutamicum本研究构建pXMJ19-NCgl1861pXMJ19 derivate;containing the NCgl1861 gene from C.glutamicum本研究构建pXMJ19-NCgl2553pXMJ19 derivate;containing the NCgl2553 gene from C.glutamicum本研究构建pXMJ19-NCgl2614pXMJ19 derivate;containing the NCgl2614 gene from C.glutamicum本研究构建pXMJ19-NCgl2933pXMJ19 derivate;containing the NCgl2933 gene from C.glutamicum本研究构建pXMJ19-NCgl2934pXMJ19 derivate;containing the NCgl2934 gene from C.glutamicum本研究构建pXMJ19-NCgl0109pXMJ19 derivate;containing the NCgl0109 gene from C.glutamicum m本研究构建pXMJ19-NCgl0178pXMJ19 derivate;containing the NCgl0178 gene from C.glutamicum本研究构建pXMJ19-NCgl0278pXMJ19 derivate;containing the NCgl0278 gene from C.glutamicum本研究构建pXMJ19-NCgl0640pXMJ19 derivate;containing the NCgl0640 gene from C.glutamicum本研究构建pXMJ19-NCgl1411pXMJ19 derivate;containing theNCgl1411 gene from C.glutamicum本研究构建pXMJ19-NCgl2141pXMJ19 derivate;containing theNCgl2141 gene from C.glutamicum本研究构建pXMJ19-NCgl2549pXMJ19 derivate;containing the NCgl2549 gene from C.glutamicum本研究构建pXMJ19-NCgl2876pXMJ19 derivate;containing the NCgl2876 gene from C.glutamicum本研究构建pXMJ19-NCgl2903pXMJ19 derivate;containing the NCgl2903 gene from C.glutamicum本研究构建pXMJ19-NCgl2922pXMJ19 derivate;containing the NCgl2922 gene from C.glutamicum本研究构建pXMJ19-NCgl2953pXMJ19 derivate;containing the NCgl2953 gene from C.glutamicum本研究构建pXMJ19-ptsGpXMJ19 derivate;containing the ptsG gene from C.glutamicum本研究构建pXMJ19-iolT1pXMJ19 derivate;containing the iolT1gene from C.glutamicum本研究构建pXMJ19-iolT2pXMJ19 derivate;containing the iolT2 gene from C.glutamicum本研究构建pXMJ19-EcgalPpXMJ19 derivate;containing tuf promoter and the galP gene from E.coli本研究构建pXMJ19-BsglcPpXMJ19 derivate;containing tuf promoter the glcP gene from B.subtilis本研究构建pXMJ19-BsglcUpXMJ19 derivate;containing tuf promoter the glcU gene from B.subtilis 本研究构建pXMJ19-VpsglSpXMJ19 derivate;containing tuf promoter the sglS gene from Vibrio parahaemolyticus本研究构建pXMJ19-ScglcPpXMJ19 derivate;containing tuf promoter the glcP gene from Staphylococcus carnosus本研究构建pXMJ19-EcrhtApXMJ19 derivative;containing the rhtA gene from E.coli本研究构建pXMJ19-EcrhtBpXMJ19 derivative;containing the rhtB gene from E.coli本研究构建
1.1.2 工具酶及试剂
Phusion DNA聚合酶、限制性内切酶,赛默飞世尔科技公司;2×Rapid Taq Master Mix、ClonExpress MultiS One Step Cloning Kit,南京诺唯赞生物科技有限公司;细菌基因组提取试剂盒、质粒提取试剂盒、胶回收试剂盒,天根生化科技有限公司;NADP+/NADPH检测试剂盒,上海碧云天生物技术有限公司;L-高丝氨酸,天津希恩思奥普德科技有限公司;其他试剂均为国产分析纯。
1.1.3 培养基与培养条件
LB培养基(g/L):氯化钠5,酵母提取物5,胰蛋白胨10,121 ℃灭菌30 min。固体添加20 g/L琼脂粉。
LBHIS培养基(g/L):D-山梨醇91,胰蛋白胨5,酵母提取物2.5,脑心浸液18.5,氯化钠5,121 ℃灭菌30 min。固体添加20 g/L琼脂粉。
CGX Ⅱ 基本培养基(g/L):葡萄糖20,尿素5,磷酸二氢钾1,硫酸铵20,磷酸氢二钾1.3,3-N-吗啉基丙磺酸42,调整pH 7.0,121 ℃ 灭菌20 min。固体添加20 g/L琼脂粉。使用前添加微量元素MgSO4·7H2O 0.25 g/L,ZnSO4·7H2O 10 mg/L,MnSO4·H2O 100 mg/L,FeSO4·7H2O 100 mg/L,CuSO40.2 mg/L,NiCl·6H2O 0.02 mg/L,CaCl20.01 g/L,维生素B1 0.7 mg/L,生物素0.2 mg/L,原儿茶酸0.03 g/L。
种子培养基(g/L):玉米浆30,尿素5,硫酸铵5,磷酸二氢钾1,葡萄糖25,硫酸镁0.5,L-苏氨酸0.4 g/L,调整pH 7.0,115 ℃灭菌30 min。
发酵培养基:葡萄糖50 g/L,玉米浆30 g/L,硫酸铵20 g/L,MgSO4 0.5 g/L,磷酸0.225 mL/L,乙酸钾0.5 g/L,磷酸二氢钾0.5 g/L,维生素溶液7 mL/L(生物素300 mg/L,维生素B1 500 mg/L,泛酸钙盐2 g/L,烟酰胺600 mg/L),FeSO4-柠檬酸0.55 mL/L(FeSO4·7H2O 20 g/L,柠檬酸18.14 g/L),苏氨酸0.4 g/L,调整pH 7.0,115 ℃高温灭菌30 min。摇瓶培养基中添加20 g/L碳酸钙。
大肠杆菌通常在37 ℃的LB培养基中培养,谷氨酸棒杆菌细胞在32 ℃的LBHIS丰富培养基或CGXⅡ基本培养基中培养。在需要时,向培养基中添加抗生素,谷氨酸棒杆菌的抗生素最终质量浓度为15 μg/L氯霉素。目的基因表达时使用200 μmol/L异丙基β-D-1硫代半乳糖苷进行诱导。
1.2 实验方法
1.2.1 重组质粒构建
采用穿梭质粒pXMJ19用于转运蛋白基因的表达。利用Phusion DNA 聚合酶,以各菌株基因组DNA为模板扩增获得目的基因片段。通过基于非连接酶依赖型的无缝克隆方法将目的基因片段与Hind Ⅲ/EcoR Ⅰ双酶切线性化的pXMJ19载体骨架进行连接,获得重组表达质粒。
采用温度敏感穿梭质粒pCRD206构建目的基因敲除或敲入质粒。利用Phusion DNA 聚合酶扩增获得靶基因位点上下游~1 kb 侧翼序列、强启动子序列和目的基因编码区,将其与靶基因位点上下游~1 kb侧翼序列,以及BamH Ⅰ/Xba Ⅰ双酶切线性化的pCRD206载体骨架,通过基于非连接酶依赖型的无缝克隆方法进行多片段连接,构建获得目的质粒。
1.2.2 重组菌株构建
采用基于温度敏感和 SacB 蔗糖致死原理的同源重组技术进行靶基因的框内无痕敲除(In-frame deletion),采用基因敲除的类似策略,利用适用于谷氨酸棒杆菌的强启动子序列,在染色体水平上可实现目的靶基因敲入[15]。将敲除或敲入质粒电转化相应谷氨酸棒杆菌后,通过在含卡那霉素的培养基中正向筛选得到重组载体整合至染色体上的菌落,再通过在含蔗糖的培养基中反向筛选,得到发生第二次同源双交换的菌落,通过平板筛选和PCR验证,获得相应的谷氨酸棒杆菌基因改造工程菌。在谷氨酸棒杆菌工程菌构建中主要采用组成型强启动子Ptuf、Psod、Ptac、Pgap、PH7等进行基因过表达,上述启动子序列已经广泛用于增强合成途径关键基因的表达水平[16-17]。
1.2.3 菌株生长测定
将上述构建成功的表达质粒电转到谷氨酸棒杆菌底盘菌中,挑取单克隆菌落至含15 μg/mL氯霉素的LBHIS培养基中,32 ℃,220 r/min过夜培养。将过夜培养的菌液按照初始OD600=0.1转接至含25 mL CGXⅡ基本培养基的250 mL三角瓶中,32 ℃,220 r/min,摇瓶培养48 h后,测定最终菌体生物量。
1.2.4 胞内NADPH测定
将在种子培养基中过夜培养的重组菌株,按照5%接种量转接到发酵培养基中进行培养,收集生长至对数后期的重组菌,调整OD600值为5.0,利用PBS缓冲液将菌体进行清洗后检测。胞内NADPH含量的测定使用上海碧云天NADP+/NADPH检测试剂盒,具体操作步骤按照说明书进行。
1.2.5 L-高丝氨酸发酵培养及检测
挑取菌落至于5 mL的LBHIS液体培养基中,过夜培养后,以5%(体积分数)的接种量转接至含20 mL种子培养基的250 mL三角瓶中,32 ℃,220 r/min,培养16 h。在摇瓶发酵实验中,按照初始OD600=1转接至含有25 mL发酵培养基的250 mL三角瓶中,在32 ℃,220 r/min发酵培养60 h。在5 L发酵罐实验中,将过夜培养的种子液以5%的接种量转接至含2 L发酵培养基的生物反应器中,控制pH 7.0,温度32 ℃,控制溶氧在20%左右,将搅拌转速与溶氧耦联,确保溶氧稳定,发酵时间60 h,每间隔12 h取样。发酵培养基中,初始葡萄糖质量浓度为50 g/L。采用生物传感分析仪,测定发酵培养基中葡萄糖的浓度,当培养基中葡萄糖质量浓度降至5 g/L时,通过流加80%葡萄糖溶液,控制培养基中葡萄糖质量浓度在5 g/L以下。使用分光光度计测定菌液在OD600的吸收值,用于评估菌株的生长情况。使用装配有ZORBAX Eclipse AAA色谱柱的Agilent 1260高效液相色谱仪,测定发酵液中L-高丝氨酸含量。
2 结果与分析
2.1 生产L-高丝氨酸的谷氨酸棒杆菌底盘菌构建
随着微生物基因组注释的完善,许多细菌中L-高丝氨酸生物合成途径已被解析地较为清楚,目前关于L-高丝氨酸生物合成和代谢调控的研究主要集中于大肠杆菌和谷氨酸棒杆菌等模式微生物中。谷氨酸棒杆菌作为一种重要的食品安全级工业微生物菌种,已被广泛用于多种氨基酸的工业发酵,以及有机酸、核苷酸和维生素等的生物合成,具有重要的经济价值和应用前景[18]。研究表明,谷氨酸棒杆菌中L-高丝氨酸的生物合成是从天冬氨酸支路途径开始的(图1)。首先,葡萄糖经过糖酵解途径、磷酸戊糖途径和三羧酸循环等生成草酰乙酸,并进入天冬氨酸支路途径;然后天冬氨酸在天冬氨酸激酶(lysC基因编码)、天冬氨酸半醛脱氢酶(asd基因编码)、高丝氨酸脱氢酶(hom基因编码)顺次作用下催化为L-高丝氨酸,最后在外排转运蛋白的作用下将L-高丝氨酸分泌到胞外环境。作为天冬氨酸家族的重要中间产物,L-高丝氨酸在野生型菌株较难高效积累,易被直接用作苏氨酸、蛋氨酸生物合成的前体物质。
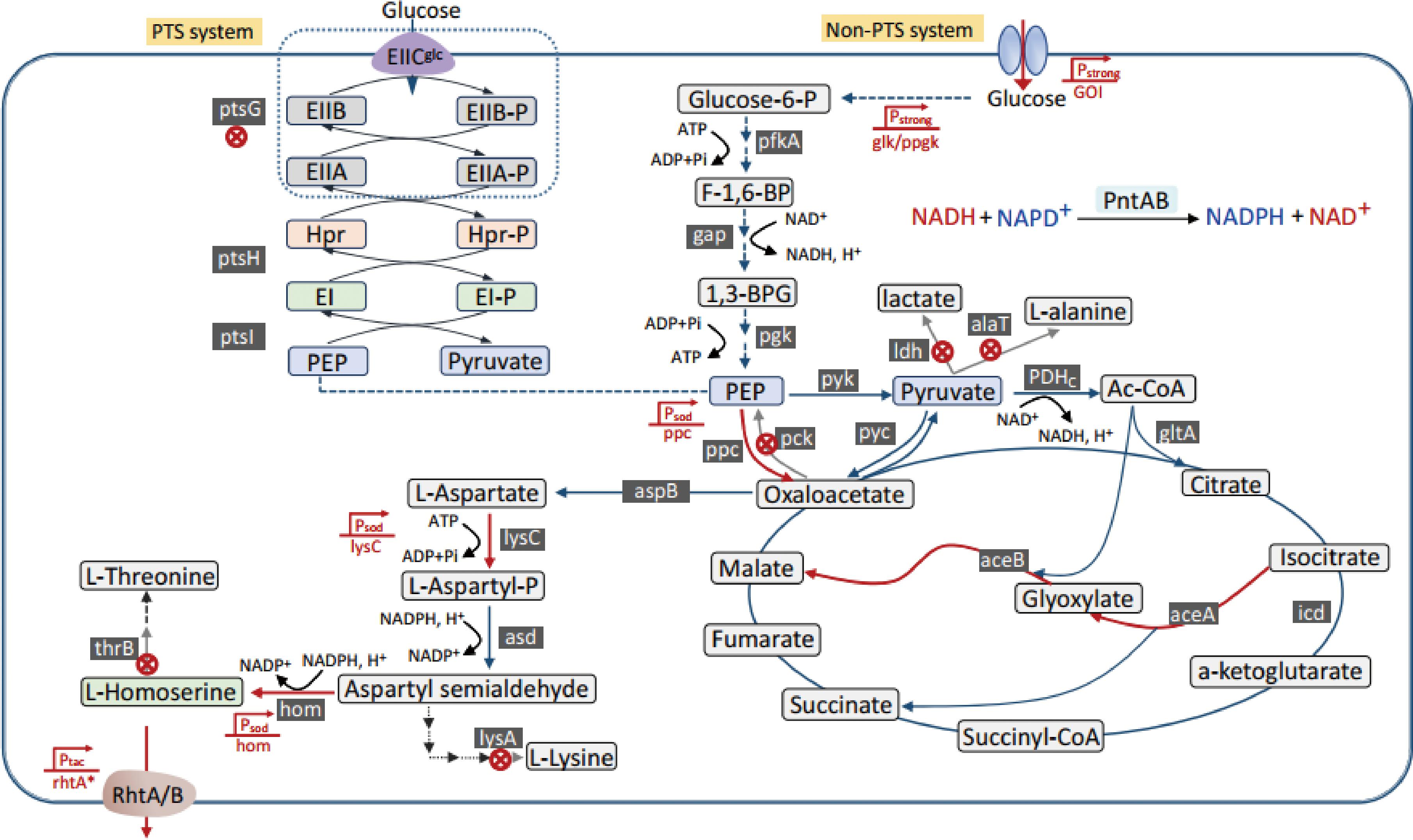
图1 谷氨酸棒杆菌L-高丝氨酸代谢合成途径改造示意图
Fig.1 Schematic overview of the strategies for engineering L-homoserine biosynthesis pathway in C.glutamcium
注:Glucose:葡萄糖;Glucose-6-P:葡萄糖-6-磷酸;F-1,6-BP:果糖-1,6-二磷酸;1,3-BPG:1,3-二磷酸甘油酸;Pyruvate:丙酮酸;Lactate:乳酸;L-alanine:L-丙氨酸;Ac-CoA:乙酰辅酶 A(Acetyl-CoA);Citrate:柠檬酸;Isocitrate:异柠檬酸;α-ketoglutarate:α-酮戊二酸;Succinyl-CoA:琥珀酰辅酶A;Succinate:琥珀酸;Fumarate:延胡索酸;Malate:苹果酸; Oxaloacetate:草酰乙酸;Glyoxylate:乙醛酸;L-Aspartate:L-天冬氨酸;L-Aspartyl-P,L-天冬氨酰磷酸;Aspartyl semialdehyde:天冬氨酰半醛;L-Homserine:L-高丝氨酸;L-Threonine:L-苏氨酸;L-Lysine:L-赖氨酸; NADH:还原型烟酰胺腺嘌呤二核苷酸;NAD+:烟酰胺腺嘌呤二核苷酸;NADPH:还原型烟酰胺腺嘌呤二核苷酸磷酸;NADP+:烟酰胺腺嘌呤二核苷酸磷酸;ATP:腺嘌呤核苷三磷酸;ADP:二磷酸腺苷;Pi:磷酸基团。
本研究以野生型谷氨酸棒杆菌ATCC 13032为出发菌株,通过一系列代谢途径设计和改造,提高菌株的L-高丝氨酸积累水平。首先,敲除L-高丝氨酸激酶编码基因thrB弱化L-高丝氨酸的降解途径,利用常用的组成型强启动子Psod过表达天冬氨酸激酶编码基因lysC和高丝氨酸脱氢酶编码基因hom以强化L-高丝氨酸生物合成代谢流通量,上述方案获得的谷氨酸棒杆菌工程菌命名为HOM1。如图2-A所示,和野生型出发菌株相比,上述代谢途径改造靶点的遗传操作并未明显降低HOM1重组菌的生长能力。在摇瓶发酵培养60 h后,野生型WT出发菌中未检测到明显的L-高丝氨酸含量,但HOM1重组菌能够积累4.35 g/L L-高丝氨酸(图2-B)。随后,在HOM1重组菌基础上,利用常用的组成型强启动子Psod过表达磷酸烯醇式丙酮酸羧化酶编码基因ppc以增强草酰乙酸前体供应(HOM2),以及人工合成强启动子PH7过表达TCA循环中乙醛酸回补途径的2个关键酶基因aceBA以加快中心碳代谢到目标产物的转化(HOM3),继续探究了上述改造策略对菌株生长和L-高丝氨酸积累水平的影响。如图2-C和2-D所示,相较于HOM1菌株,HOM2和HOM3重组菌在发酵条件下具有相似的生长能力,但是HOM2重组菌能够积累4.82 g/L L-高丝氨酸,比出发菌株提高了10.7%。然而,HOM3重组菌和HOM1菌株具有相似的L-高丝氨酸积累水平。最后,在HOM2优势重组菌基础上,通过阻断或弱化丙氨酸竞争途径(敲除alaT基因)和赖氨酸竞争途径(敲除ddh基因),以进一步增强高丝氨酸合成碳代谢流,将最终获得的工程菌株命名为HOM4。发酵结果表明,尽管菌株的生长能力有该菌株能够积累5.03 g/L L-高丝氨酸,比HOM1出发菌株提高15.6%(图2-D)。综上结果表明,本研究通过强化高丝氨酸合成碳代谢流,提高前体物供应,以及阻断或弱化高丝氨酸竞争途径,获得了具有优良L-高丝氨酸积累能力的谷氨酸棒杆菌底盘菌。
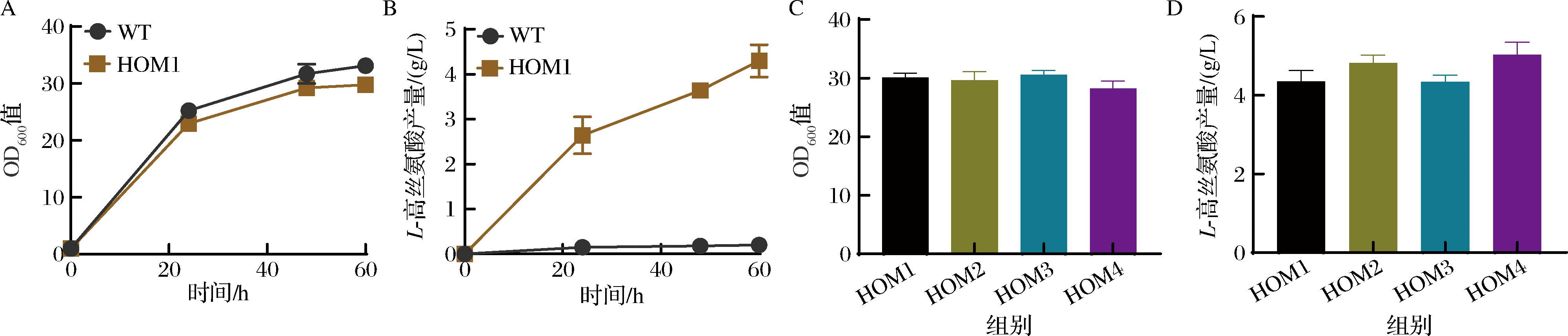
A-谷氨酸棒杆菌野生型与重组菌株HOM1生物量;B-谷氨酸棒杆菌野生型与重组菌株HOM1 L-高丝氨酸产量;C-重组菌株HOM1、HOM2、HOM3、HOM4生物量;D-重组菌株HOM1、HOM2、HOM3、HOM4 L-高丝氨酸产量
图2 谷氨酸棒杆菌野生型与重组菌株的生物量和L-高丝氨酸产量
Fig.2 Biomass and L-homoserine production of wild-type and recombinant strains of C.glutamicum
2.2 基于辅因子平衡工程提升L-高丝氨酸生产
NAD+/NADP+和NADH/NADPH是细胞内重要的核苷酸类辅酶,可为细胞生物合成与分解反应提供氧化还原载体,是细胞生长和能量代谢必不可少的辅因子[19]。维持胞内辅因子供应与平衡在目标代谢产物的合成效率中扮演着重要的角色,这种供应平衡也是氨基酸代谢工程改造中的一个重要策略[20]。在L-高丝氨酸生物合成过程中,天冬氨酸半醛脱氢酶Asd和高丝氨酸脱氢酶Hom催化的反应都需要辅因子NADPH参与。因此,代谢工程改造获得L-高丝氨酸高产菌的关键步骤之一在于改善胞内NADPH的供应。
大肠杆菌中pntAB基因编码的吡啶核苷酸转氢酶,能够催化NADH和NADPH间的转换,增强胞内NADPH的供应,解决胞内辅因子不平衡问题。但是,在谷氨酸棒杆菌基因组中尚未发现pntAB基因同源物。因此,为了解决高丝氨酸生物合成过程中NADPH供应不足问题,本研究在HOM4重组菌基础上,在染色体pck基因位点处引入大肠杆菌来源的pntAB基因,并分别用自身来源启动子Pself和常用强启动子Pgap控制pntAB基因表达,构建获得的谷氨酸棒杆菌工程菌分别命名为HOM5和HOM6。如图3-A所示,外源pntAB基因的引入对菌株的正常生长没有显著影响。在L-高丝氨酸发酵生产方面,利用自身启动子Pself表达大肠杆菌来源的pntAB基因能够轻微提高菌株L-高丝氨酸积累能力,但是利用强启动子Pgap控制pntAB基因表达则能够显著提升菌株L-高丝氨酸生产(图3-B)。相较于HOM4出发菌株,HOM6重组菌在摇瓶条件下可生产5.57 g/L L-高丝氨酸,比出发菌株提高了10.9%。通过对工程菌胞内NADPH含量进行测定,发现HOM6重组菌胞内具有更多的NADPH,相较于HOM4出发菌株,其胞内NADPH含量水平提高了34.7%(图3-C)。上述研究结果表明,通过过表达pntAB基因改善胞内NADPH供应和平衡,是构建高效L-高丝氨酸微生物细胞工厂的重要策略。
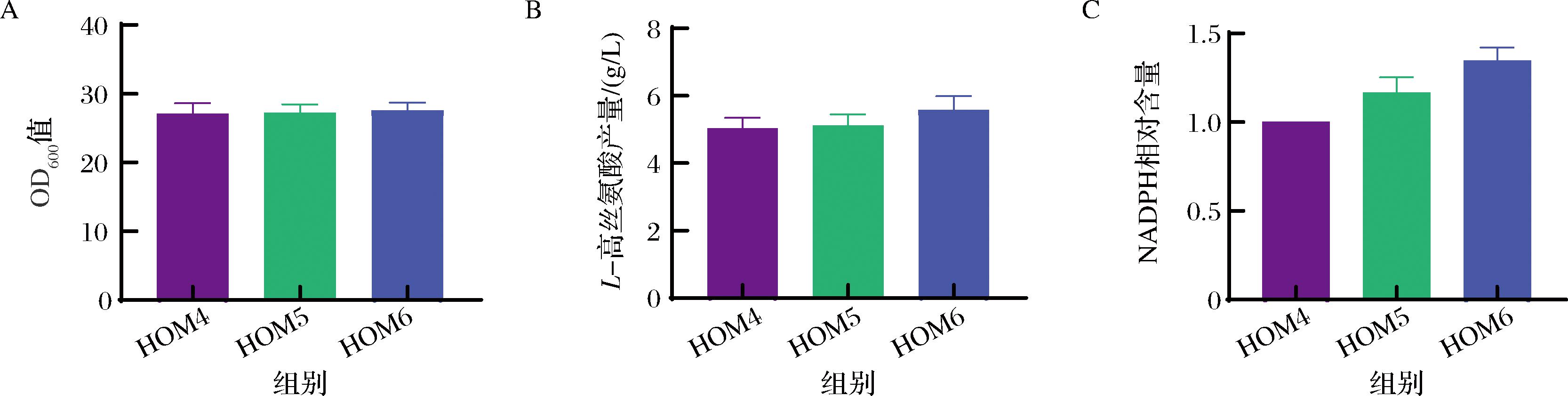
A-重组菌株生物量;B-重组菌株L-高丝氨酸产量;C-重组菌株NADPH相对含量
图3 辅因子平衡对谷氨酸棒杆菌L-高丝氨酸产量的影响
Fig.3 Effect of cofactor balance on L-homoserine production in C.glutamicum
2.3 基于高丝氨酸外排蛋白提升L-高丝氨酸生产
在设计和构建高效微生物底盘细胞工厂中,转运蛋白的鉴定和工程化改造是提高宿主细胞生长和目标产物效价的有效途径[21]。挖掘、筛选并增强外排转运蛋白,以降低目标产物在细胞内的浓度,有利于最大限度地减少代谢抑制效应,并提高底盘菌的生物制造效率。大肠杆菌中的氨基酸转运蛋白RhtA和RhtB,最初被发现可向胞外转运L-苏氨酸或支链氨基酸,但也被证实能够参与L-高丝氨酸的胞外排出过程(图4-A)。最近的一项研究也揭示,对氨基酸转运蛋白RhtA进行定向进化后获得的RhtAmut(A22V/P119L/T235I)突变体,具有增强的L-高丝氨酸外排活性[22]。
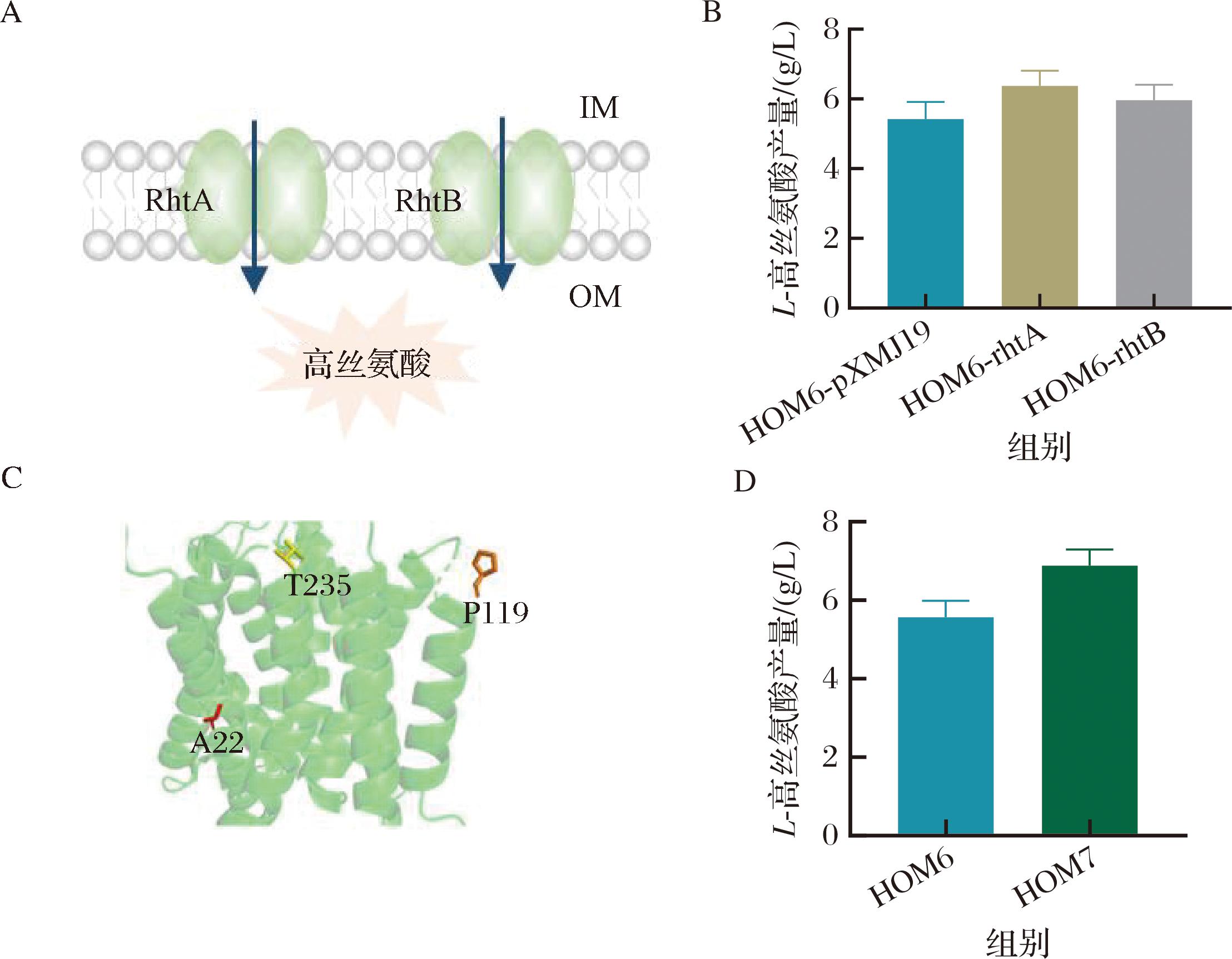
A-L-高丝氨酸外排示意图;B-HOM6-rhtA、HOM6-rhtB菌株L-高丝氨酸产量;C-氨基酸转运蛋白RhtAmut结构示意图;D-重组菌株HOM6、HOM7 L-高丝氨酸产量
图4 L-高丝氨酸外排蛋白系统对L-高丝氨酸产量的影响
Fig.4 Influence of engineering L-homoserine efflux system on L-homoserine production
本研究在HOM6重组菌基础上,探究了过表达大肠杆菌来源的氨基酸转运蛋白编码基因rhtA或rhtB,对菌株发酵生产L-高丝氨酸的影响。如图4-B所示,研究发现引入大肠杆菌中的氨基酸转运蛋白RhtA和RhtB能够不同程度地提高HOM6重组菌的L-高丝氨酸生产水平,其中rhtA基因过表达菌株的L-高丝氨酸积累量相比于对照菌株提升了17.5%。鉴于理性改造后的氨基酸转运蛋白RhtAmut(A22V/P119L/T235I)突变体具有更优异的L-高丝氨酸外排活性(图4-C),在HOM6重组菌染色体ldh基因位点处引入大肠杆菌来源的rhtAmut基因突变体,并使用强启动子Ptac控制该基因表达,将构建获得的谷氨酸棒杆菌工程菌命名为HOM7。发酵结果表明,相较于HOM6出发菌株,HOM7重组菌在摇瓶条件下可生产6.88 g/L L-高丝氨酸,比出发菌株提高了23.5%(图4-D)。上述研究结果表明,基于转运蛋白工程策略加快胞内氨基酸向外转运效率,也是构建高效L-高丝氨酸微生物细胞工厂的重要策略。
2.4 改造葡萄糖摄取利用系统提升L-高丝氨酸生产
谷氨酸棒杆菌利用葡萄糖作为底物生产L-高丝氨酸时,主要通过磷酸烯醇式丙酮酸-糖磷酸转移酶系统(phosphoenolpyruvate:sugar phosphotransferase system,PTS)完成葡萄糖的摄取及磷酸化[23]。由于PTS系统依赖磷酸烯醇式丙酮酸(phosphoenolpyruvate,PEP)作为磷酸供体基团,导致胞内可用的PEP含量减少,从而影响L-高丝氨酸的合成。此外,在谷氨酸棒杆菌中还存在一种由肌醇渗透酶和葡萄糖激酶组成的non-PTS葡萄糖摄取系统,但该系统对葡萄糖的亲和力较低。为了调节葡萄糖的转运和利用进一步改善L-高丝氨酸的产生,本研究在HOM7重组菌株基础上,利用常用的组成型强启动子Ptuf过表达来源于B.subtilis的葡萄糖激酶编码基因glk、强启动子Pgap过表达自身的葡萄糖激酶编码基因ppgk,取代PTS系统中葡萄糖转运及磷酸化编码基因ptsG,获得重组菌株HOM8。将获得的重组菌株HOM8在以葡萄糖为唯一碳源的CGX Ⅱ培养基中进行摇瓶发酵,结果如图5-A所示,由于PTS系统的破坏,重组菌株HOM8几乎失去了以葡萄糖为唯一碳源进行生长的能力。为了恢复重组菌株HOM8的葡萄糖摄取和细胞生长,本研究评估了31种与糖转运有关的转运体,其中包括内源性转运体26个,异源性转运体5个。其中内源性肌醇转运体的编码基因iolT1过表达可以最大程度地恢复HOM8菌株的生长能力(图5-B),生物量与ptsG回补菌株相比提升了21.2%,而其余基因未起到有效的作用。在异源转运体的筛选中,不同来源的转运体编码基因的过表达在恢复HOM8菌株的生长上展现出不同的效果,其中来源于大肠杆菌的半乳糖渗透酶编码基因galP 的过表达使得HOM8菌株生长较好(图5-C)。将筛选出来的菌株进行摇瓶发酵验证,最终HOM8-iolT1和HOM8-EcgalP重组菌株L-高丝氨酸产量分别达到9.08 g/L和9.29 g/L,与HOM8-ptsG重组菌株相比分别提高了26.28%和29.2%(图5-D)。将产量较高的HOM8-EcgalP菌株进行发酵罐培养,菌株生物量与L-高丝氨酸产量随着时间的延长逐步增加,最终在发酵培养60 h时,菌株生物量达到62.76,L-高丝氨酸产量达到77.53 g/L,生产强度达到1.29 g/(L·h)(图5-E),发酵过程中的主要副产物是赖氨酸和丙氨酸等,副产物产量低于5.0 g/L,相关技术指标为目前已报道谷氨酸棒杆菌发酵生产L-高丝氨酸的先进水平。
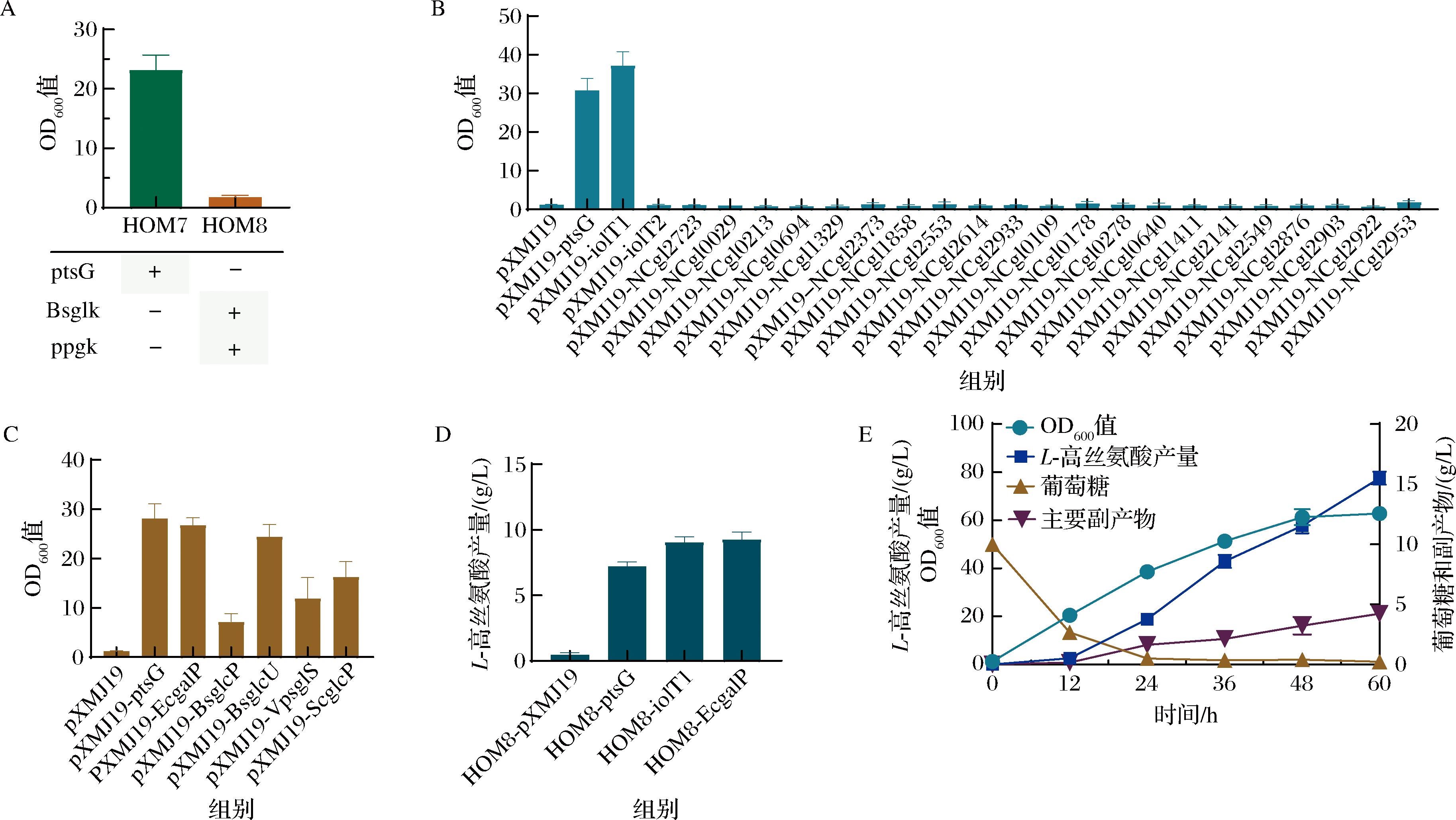
A-重组菌株HOM6、HOM7生物量;B-内源性糖转运基因筛选;C-异源性糖转运基因筛选;D-重组菌株L-高丝氨酸产量;E-HOM8-EcgalP菌株发酵罐培养分析
图5 改造葡萄糖摄取利用系统对L-高丝氨酸产量的影响
Fig.5 Effect of engineering glucose uptake and utilization system on L-homoserine production
3 结论与讨论
L-高丝氨酸是一种天然存在的非蛋白质氨基酸,是生物合成天冬氨酸衍生氨基酸的重要前体,同时也是合成许多重要C4平台化合物和新型农药L-草铵膦的关键中间体。尽管L-高丝氨酸具有潜在的生物和化学应用潜力,但由于生产强度、经济可行性、环境安全性等原因,其大规模生产尚未实现商业化。近年来,随着对L-高丝氨酸的市场需求不断增长,以及代谢工程和合成生物学的快速发展,越来越多的研究者开始关注开发适用于大规模工业生产的L-高丝氨酸微生物细胞工厂[24]。目前,主要研究集中在大肠杆菌底盘细胞上,该菌株被证实是生产L-高丝氨酸的最有效宿主之一,但是改造食品安全级微生物谷氨酸棒杆菌以便高效发酵生产L-高丝氨酸的实例仍然较少。本研究通过系统代谢工程策略,对谷氨酸棒杆菌中L-高丝氨酸生物合成途径进行改造,实现了工程菌中L-高丝氨酸的高效积累,L-高丝氨酸发酵水平达到77.53 g/L,生产强度达到1.29 g/(L·h),展现出一定的经济可行性。相关研究进一步证实了谷氨酸棒杆菌在氨基酸发酵生产方面的巨大潜力,将为后续改善和提升谷氨酸棒杆菌L-高丝氨酸生产性能提供一定理论参考。相信随着研究者不断开发构建新型L-高丝氨酸高产底盘工程菌,未来将很快实现利用微生物发酵法达成L-高丝氨酸高效生物合成和工业化生产的目标。
[1] SUN Y J, WU J P, XU J Q, et al.Metabolic engineering of Escherichia coli for the production of L-homoserine[J].Chem &Bio Engineering, 2024:1(3):223-230.
[2] MU Q X, ZHANG S S, MAO X J, et al.Highly efficient production of L-homoserine in Escherichia coli by engineering a redox balance route[J].Metabolic Engineering, 2021, 67:321-329.
[3] CAI M M, ZHAO Z Q, LI X F, et al.Development of a nonauxotrophic L-homoserine hyperproducer in Escherichia coli by systems metabolic engineering[J].Metabolic Engineering, 2022, 73:270-279.
[4] BABENKO L M, KOSAKIVSKA I V, ROMANENKO K O.Molecular mechanisms of N-acyl homoserine lactone signals perception by plants[J].Cell Biology International, 2022, 46(4):523-534.
[5] ZHOU C Z, LUO X X, CHEN N Y, et al.C-P natural products as next-generation herbicides:Chemistry and biology of glufosinate[J].Journal of Agricultural and Food Chemistry, 2020, 68(11):3344-3353.
[6] 牛坤, 高利平, 葛丽蓉, 等.大肠杆菌代谢工程改造合成 L-高丝氨酸及其衍生物研究进展[J].生物工程学报, 2022,38(12):4385-4402.NIU K, GAO L P, GE L R, et al.Advances in the biosynthesis of L-homoserine and its derivatives by metabolic engineering of Escherichia coli[J].Chinese Journal of Biotechnology, 2022, 38(12):4385-4402.
[7] ZHANG Y, WEI M H, ZHAO G H, et al.High-level production of L-homoserine using a non-induced, non-auxotrophic Escherichia coli chassis through metabolic engineering[J].Bioresource Technology, 2021, 327:124814.
[8] LIU Z F, CAI M M, ZHOU S Q, et al.High-efficient production of L-homoserine in Escherichia coli through engineering synthetic pathway combined with regulating cell division[J].Bioresource Technology, 2023, 389:129828.
[9] LIU P, ZHANG B, YAO Z-H, et al.Multiplex design of the metabolic network for production of L-homoserine in Escherichia coli[J].Applied and Environmental Microbiology, 2020, 86(20):e01477-20.
[10] SUN B Y, WANG F Q, ZHAO J, et al.Engineering Escherichia coli for L-homoserine production[J].Journal of Basic Microbiology, 2023, 63:168-178.
[11] VO T M, PARK S.Metabolic engineering of Escherichia coli W3110 for efficient production of homoserine from glucose[J].Metabolic Engineering, 2022, 73:104-113.
[12] LI N, XU S, DU G C, et al.Efficient production of L-homoserine in Corynebacterium glutamicum ATCC 13032 by redistribution of metabolic flux[J].Biochemical Engineering Journal, 2020, 161:107665.
[13] LI N, WANG M, YU S Q, et al.Optimization of CRISPR-Cas9 through promoter replacement and efficient production of L-homoserine in Corynebacterium glutamicum[J].Biotechnology Journal, 2021, 16(8):2100093.
[14] LI N, LI L H, YU S Q, et al.Dual-channel glycolysis balances cofactor supply for L-homoserine biosynthesis in Corynebacterium glutamicum[J].Bioresource Technology, 2023, 369:128473.
[15] JU Y, ZHANG H Y, DU X C, et al.DRAGON:Harnessing the power of DNA repair for accelerating genome evolution in Corynebacterium glutamicum[J].Metabolic Engineering, 2023, 79:182-191.
[16] P TEK M, N
TEK M, N EVERA J, GUYONVARCH A, et al.Promoters of Corynebacterium glutamicum[J].Journal of Biotechnology, 2003, 104(1-3):311-323.
EVERA J, GUYONVARCH A, et al.Promoters of Corynebacterium glutamicum[J].Journal of Biotechnology, 2003, 104(1-3):311-323.
[17] RYTTER J V, HELMARK S, CHEN J, et al.Synthetic promoter libraries for Corynebacterium glutamicum[J].Applied microbiology and biotechnology, 2014, 98:2617-2623.
[18] TSUGE Y, MATSUZAWA H.Recent progress in production of amino acid-derived chemicals using Corynebacterium glutamicum[J].World Journal of Microbiology and Biotechnology, 2021, 37(3):49.
[19] 贾男, 臧国伟, 李春, 等.辅因子在微生物细胞工厂中的代谢调控与应用[J].中国生物工程杂志, 2022, 42(7):79-89.JIA N, ZANG G W, LI C,et al.Metabolic regulations and applications of cofactors in microbial cell factories[J].China Biotechnology, 2022, 42(7):79-89.
[20] WANG M, CHEN B Q, FANG Y M, et al.Cofactor engineering for more efficient production of chemicals and biofuels[J].Biotechnology advances, 2017, 35(8):1032-1039.
[21] ZHU Y, ZHOU C, WANG Y, et al.Transporter engineering for microbial manufacturing[J].Biotechnology Journal, 2020, 15(9):1900494.
[22] DING C, ZHANG J W, QIAO J F, et al.Identification and engineering efflux transporters for improved L-homoserine production in Escherichia coli[J].Journal of Applied Microbiology, 2023, 134(4):lxad075.
[23] RUAN H Z, YU H B, XU J Z.The glucose uptake systems in Corynebacterium glutamicum:A review[J].World Journal of Microbiology and Biotechnology, 2020, 36(9):126.
[24] KO Y-S, KIM J W, LEE J A, et al.Tools and strategies of systems metabolic engineering for the development of microbial cell factories for chemical production[J].Chemical Society Reviews, 2020, 49(14):4615-4636.